抗黄萎病相关蛋白GbVIP1及其编码基因与应用的制作方法
本发明涉及生物技术领域,特别是涉及一种抗黄萎病相关蛋白gbvip1及其编码基因与应用。
背景技术:
黄萎病是我国棉花生产上危害最为严重的病害,我国西北内陆、黄河流域和长江流域三大棉区每年都不同程度的受到黄萎病的危害,年发生面积约300万hm2,年经济损失高达12亿元(朱荷琴,李志芳,冯自力等.我国棉花黄萎病研究十年回顾及展望,棉花学报,2017,29(增刊):37-50)。通过长期对棉花黄萎病的防治实践表明,选育和种植抗病品种才是防治棉花黄萎病最有效、最经济的措施。但我国陆地棉对黄萎病菌免疫及高抗的种质资源缺乏,而且抗黄萎病海岛棉无法大面积推广,加之传统育种周期长、效率低,导致传统育种方法选育抗黄萎病品种进展缓慢,所以很多研究人员运用现代基因工程技术,以期找到棉花抗黄萎病的关键蛋白,并利用抗病蛋白结合分子育种手段选育抗黄萎病棉花品种。
vire2互作蛋白1(vip1,vire2interactingproteins1)是植物碱性亮氨酸拉链(bzip,basicleucinezipper)转录因子,具有保守的bzip结构域,属于bzip转录因子家族的i亚家族(jakobym,weisshaarb,droge-laserw,vicente-carbajosaj,tiedemannj,krojt,parcyf.bziptranscriptionfactorsinarabidopsis.trendsplantssci,2002,7(3):106-111)。vip1蛋白通过与基因组中的vip1应答原件(vre,vip1responseelement)结合,可激活许多抗逆相关基因的转录,引起植物的抗性反应(lacroixb,citovskyv.characterizationofvip1activityasatranscriptionalregulatorinvitroandinplanta.scirep,2013,3(8):2440)。当植物受到病原菌侵害时,vip1蛋白可参与mapk(mitogen-activatedproteinkinases)途径,作为mapk蛋白的底物被磷酸化,磷酸化的vip1蛋白可定位于细胞核,激活病程相关基因pr1(pathogenesis-regulatedprotein)的表达,提高植物对病原菌的抗性(zaltsmana,krichevskya,kozlovskysv,yasminf,citovskyv,plantdefensepathwayssubvertedbyagrobacteriumforgenetictransformation,plantsignalingandbehavior,5:10,1245-1248.)。
技术实现要素:
本发明的目的是提供一种抗黄萎病相关蛋白gbvip1及其编码基因与应用。
本发明的技术方案具体如下:
一种抗黄萎病相关蛋白gbvip1,其氨基酸序列如seqidno:1所示。
本发明所述的蛋白gbvip1在棉花抗黄萎病中的应用。
本发明所述的蛋白gbvip1来源于抗黄萎病海岛棉品种海7124。
本发明所述的蛋白gbvip1通过基因工程手段培育抗黄萎病陆地棉品种的应用。
一种gbvip1基因,其核苷酸序列为下列所述之一:
(1)编码如seqidno:1所示蛋白质的多核苷酸序列;
(2)与上述多核苷酸序列根据碱基互补配对原则互补的多核苷酸序列。
本发明所述的gbvip1基因,其多核苷酸序列如seqidno:2所示。
一种重组载体,含有上述的gbvip1基因。
本发明所述的重组载体为在植物表达载体pbi121的酶切位点bamhi和saci之间插入gbvip1基因的重组载体。
一种遗传工程的宿主细胞,含有上述的重组载体。
本发明所述遗传工程的宿主细胞为上述重组载体的大肠杆菌或农杆菌。
本发明的gbvip1蛋白来自抗黄萎病海岛棉品种海7124,研究发现黄萎病菌侵染条件下gbvip1基因上调表达,沉默gbvip1基因降低了海岛棉对黄萎病菌的抵抗能力,所以gbvip1蛋白对棉花抗黄萎病育种具有重要的意义。
gbvip1蛋白由337个氨基酸残基组成,其氨基酸序列如seqidno:1所示,gbvip1蛋白理论分子量为37.5kda,理论等电点为7.89,包含保守的bzip结构域,bzip结构域中亮氨酸残基位置保守性高,每隔6个氨基酸残基就存在一个亮氨酸。本发明对不同物种的vip1蛋白进行进化树分析发现来自不同棉花品种的vip1基因聚在一起。同时,对来自不同物种的vip1蛋白进行结构域进行分析,发现来自不同物种的vip1蛋白都包含bzip结构域。
本发明对vip1基因在棉花中的组织表达特异性进行分析,发现gbvip1在棉花根、茎、叶组织中都有表达,其中,在根中的表达量最高。对vip1基因在棉花接种黄萎病菌后的表达模式进行分析发现在棉花接种黄萎病菌后gbvip1基因上调表达,在接菌后6h表达量最高,为对照的3.73倍。
本发明利用vigs技术沉默棉花中的gbvip1基因,结果表明,gbvip1基因沉默植株的叶片变黄萎蔫,叶脉呈现黄色网状,株形微显矮化。沉默植株比对照植株的抗黄萎病能力显著下降。接菌后25天调查病指,沉默植株的黄萎病病指为22.5,对照植株的病指为20.83,接菌后35天沉默植株的黄萎病病指为30.54,对照植株的病指为23.45。接菌后35天对棉花植株进行剖杆观察,发现沉默植株的茎秆的木质化程度减弱,维管束导管部发黄褐化。对黄萎病侵染后的棉花叶片进行台盼蓝染色发现沉默植株的叶片死亡细胞的比例更高。所以,gbvip1基因与棉花抗黄萎病能力正相关,可应用于棉花抗黄萎病育种研究中。
本发明获得了包含上述基因的重组表达载体,将获得的gbvip1基因构建到表达载体pbi121中,以35s启动子启动基因的表达,nos终止子终止,以卡那霉素基因作为筛选标记。将上述重组表达载体转入大肠杆菌和农杆菌中,获得了包含上述基因的重组细胞。将包含上述重组表达载体的农杆菌转入烟草中,获得了包含重组表达载体的转基因烟草,包含上述基因的重组表达载体、重组细胞和转基因烟草可为进一步利用该基因奠定基础。
下面结合附图说明和具体实施例对本发明所述的棉花抗黄萎病相关蛋白gbvip1及其编码基因与应用作进一步说明。
附图说明
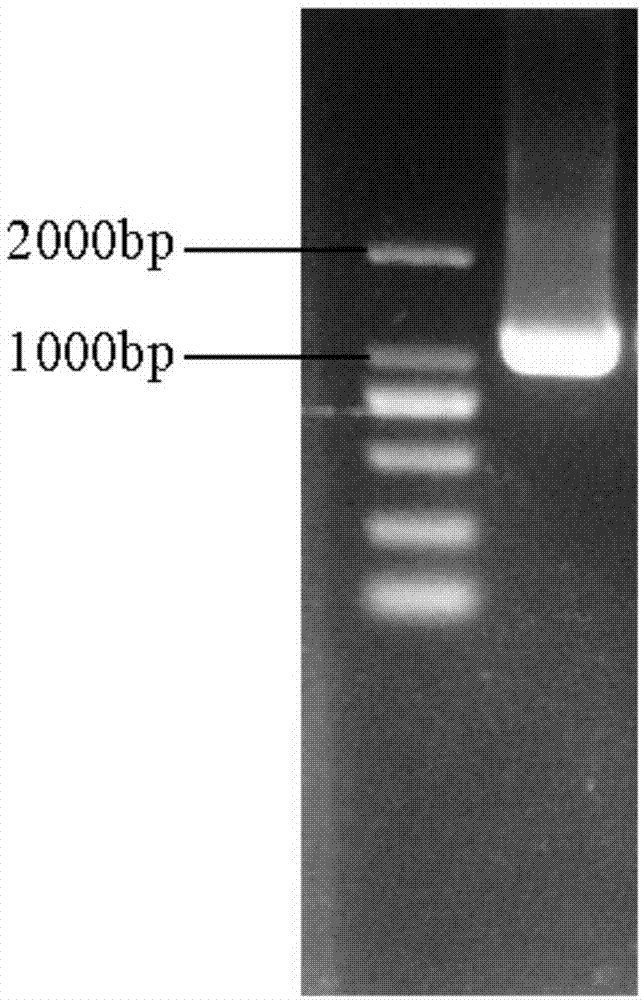
图1为克隆海岛棉gbvip1基因的pcr产物电泳图谱,左侧泳道为2000bpdna分子量标准,右侧泳道为扩增产物;
图2为黄萎病抗性和敏感品种中vip1蛋白的序列差异图,海岛棉品种海7124和pima为抗性品种,陆地棉品种tm-1为敏感性品种;
图3为gbvip1基因在棉花不同组织中的表达情况,误差线表示标准误差;
图4为gbvip1蛋白bzip保守结构域分析,箭头所指为亮氨酸;
图5来自不同物种的vip1蛋白的进化树分析;
图6来自不同物种的vip1蛋白的bzip结构域分析;
图7为包含gbvip1基因的重组表达载体图;
图8为转基因烟草检测的图片,泳道1为2000bpdna分子量标准,泳道2为阴性对照,泳道3为阳性对照,泳道4-6为转基因阳性烟草植株;
图9为gbvip1基因在棉花接种黄萎病菌后的表达情况,误差线表示标准误差,显著性分析***表示p<0.001;
图10为沉默载体pyl156-gbvip1图;
图11为利用vigs在海岛棉中沉默gbvip1基因的照片,沉默gbvip1基因后显著降低了海岛棉对黄萎病的抗性,pyl156:pds为阳性对照,pyl156:00为阴性对照;
图12为剖杆观察照片;
图13为台盼蓝染色结果照片。
具体实施方式
实施例1gbvip1基因的克隆、序列和表达分析
种植海岛棉品种海7124,在幼苗期取棉花叶片,用液氮把棉花叶片研磨成粉末,利用easyspinplus植物rna快速提取试剂盒(艾德莱)提取棉花叶片总rna,利用反转录试剂盒(takara)合成cdna。
以合成的cdna为模板,扩增gbvip1,所用引物序列为f:5’tacgccgccgcttcttccgcctaca3’;r:5’cttaccaagcatttccttccaacat3’(如seqidno:3-4所示)。
利用高保真taq酶kod-plus-neo(toyobo)扩增gbvip1,20ul反应体系如下,10×pcrbufferforkod-plus-neo,0.2mmdntps,1.5mmmgso4,0.3umforwardprimerand0.3umreverseprimer,0.4ukod-plus-neo,200mmcdna,扩增程序为94℃4min,98℃10s,60℃1min,68℃1min;34个循环,68℃5min,取扩增产物用于琼脂糖凝胶电泳,如图1所示,扩增出1000bp左右的片段。将条带切出,利用琼脂糖凝胶回收试剂盒(天根)回收。将回收产物连接到plbvector测序载体(天根)中,将重组载体gbvip1-plb利用热激法转入大肠杆菌感受态中,获得含有重组载体的大肠杆菌,并测序,最终获得gbvip1基因的序列信息。
利用同样的方法和引物,从抗性品种海岛棉pima和敏感品种陆地棉tm-1中扩增了vip1基因,发现抗感品种vip1蛋白序列存在一些碱基差异。(图2)选取幼苗期根、茎、叶组织,利用real-timepcr对gbvip1基因进行组织表达特异性分析。取材后利用上述提取rna和反转录的方法获得不同组织的cdna,利用荧光定量试剂sybrprimixextaqii(tlirnasehplus)(takara)进行real-timepcr。所用的引物序列如下:f:5’ccaatcccagtctgccacctr:5’tcaacccgagtccatcaaaga(如seqidno:5-6所示)。反应体系如下:10ul2×sybrpremixextaqii,2ulcdnatemplate,0.8ulpcrforwardprimer(10um),0.8ulpcrreverseprimer(10um),0.4ulroxand6ul灭菌水。反应程序:95℃30s,40个循环、95℃5s,60℃34s。利用2-δδct方法对得到的结果进行处理。结果发现gbvip1在根、茎、叶组织中都有表达,在根中的表达量最高(图3)。
对gbvip1蛋白的保守结构域进行分析发现gbvip1蛋白的bzip结构域中亮氨酸残基位置保守性高,每隔6个氨基酸残基就存在一个亮氨酸,如图4所示。从ncbi中下载了拟南芥、水稻、烟草等其他物种的vip1蛋白的氨基酸序列,利用mega软件对来自不同物种的vip1蛋白进行进化树分析发现来自不同棉花品种的vip1基因具有较高的亲缘关系,聚在一起,如图5所示。同时,对来自不同物种的vip1蛋白进行结构域进行分析,发现来自不同物种的vip1蛋白都包含bzip结构域,如图6所示。
实施例2gbvip1基因的表达载体、重组表达细胞和转基因烟草的获得
将gbvip1基因构建到表达载体pbi121中获得重组表达载体,以35s启动子启动基因的表达,以nos终止子终止基因的表达,以卡纳霉素基因作为筛选标记,具体方法如下,利用实施例1中所得重组载体gbvip1-plb为模板,以引物f:acgggggactctagaggatcctacgccgccgcttcttcc,r:cgatcggggaaattcgagctccttaccaagcatttccttccaac(如seqidno:7-8所示)进行扩增,获得gbvip1基因序列的pcr产物,利用限制性内切酶saci和bamhi对pbi121载体进行酶切,得到带酶切位点的线性化质粒,利用同源重组试剂盒(clonexpressiionestepcloningkit,诺唯赞)将pcr产物构建到沉过表达载体pbi121中,获得了表达载体pbi121-gbvip1(图7)。
将重组载体pbi121-gbvip1利用热激法转入大肠杆菌感受态中,获得含有重组载体的大肠杆菌,并测序,最终获得重组表达细胞。
将重组载体pbi121-gbvip1转入农杆菌细胞gv3101中,转化方法如下:在无菌条件下取50ul农杆菌感受态细胞,加入质粒dna(pbi121-gbvip1)1ug,轻轻混匀后,放置冰上30min,之后放于液氮中5min,28度热激5min,加入无抗生素的yep培养液体培养基,于28度,200r/min震荡培养2h,涂布卡那霉素抗性平板,28度静置培养36h后挑菌测序鉴定。利用农杆菌介导的遗传转化方法将重组载体pbi121-gbvip1转入烟草中获得转棉花gbvip1基因的烟草。具体操作如下:
(1)将本氏烟草种子在0.1%的次氯酸钠溶液中灭菌,再用灭菌水冲洗3次,点播于ms培养基中。待种子发芽后在无菌条件下移栽至培养瓶继续培养。
(2)选择叶面平展的本氏烟草叶片,剪成5.0mm×5.0mm大小方块,平铺于ms基本培养基(含ms维生素)+1.0mgl-16-ba+0.2mgl-1iaa的培养基上25℃、光照条件下预培养3d。
(3)侵染前,将含有重组载体pbi121-tavip1的农杆菌活化,待农杆菌菌液od600=0.5时,离心收集菌液,倒掉上清,用ms重悬液(ph6.0)重悬。
(4)将预培养的本氏烟草叶盘转至农杆菌菌液中侵染20min,取出后用无菌滤纸吸干叶片表面菌液,转移至ms基本培养基+1.0mgl-16-ba+0.2mgl-1iaa的培养皿中,25℃黑暗条件下共培养2d。
(5)将共培养后的叶盘转至筛选培养基(ms基本培养基+1.0mgl-16-ba+0.2mgl-1iaa+cb(羧苄青霉素)400mgl-1+cef(头孢青霉素)100mgl-1+kan100mgl-1),25℃、光照条件下培养,每隔3周更换一次培养基
(6)待烟草叶盘周边不定芽长至0.5cm时将不定芽转入生根培养基(1/2ms基本培养基+cb400mgl-1+cef100mgl-1+kan100mgl-1)进一步筛选、培养。
将高度为2-3cm的再生芽从愈伤组织上分割出来,转移到三角瓶中培养,获得抗性再生植株,利用卡那霉素筛选标记基因对t0代转基因烟草进行pcr检测,所用引物f:5’tacgccgccgcttcttccgcctaca3’r:5’cttaccaagcatttccttccaacat3’(如seqidno:9-10所示),能够扩增出1014bppcr产物的植株即为转海岛棉gbvip1基因的烟草植株(图8)。该转基因植株对后续gbvip1蛋白在抗黄萎病育种中的应用奠定了基础
实施例3gbvip1的功能分析
(1)gbvip1基因在棉花接种黄萎病菌后的表达模式分析
利用水培法种植棉花,在二叶一心期利用孢子浓度为1*107的棉花黄萎病菌vd853使用蘸根法侵染棉花幼苗的根部,然后分别取了接菌后0h,6h,12h,24h棉花幼苗的根组织,利用实施例1中所述方法提取了rna并反转录成cdna,利用real-timepcr研究了gbvip1基因在棉花接种黄萎病菌后的表达情况,所用引物和反应体系见实施例1。结果发现在接菌后gbvip1基因的表达量上调,在接菌后6h,gbvip1基因的表达量最高,为对照的3.73倍(图9)。由此可推断gbvip1基因与棉花抗黄萎病能力相关。
(2)利用病毒诱导的基因沉默(vigs:virusinducedgenesilencing)技术在海岛棉中沉默gbvip1基因
从gbvip1基因的cdna序列中选取了272bp保守序列反向构建到沉默载体pyl156中,获得沉默载体pyl156-gbvip1(图10),具体操作如下,首先,根据实施例1中所得的海岛棉叶片的cdna作为模板,以引物fggcctcgagacgcgtgagctcgccgttgatggtgttaagaaaac,r:agaaggcctccatggggatccatagcctgtagccgtagtttcagtt(如seqidno:11-12所示)进行扩增,获得了272bp保守序列的pcr产物。利用限制性内切酶saci和bamhi对pyl156载体进行酶切,得到带酶切位点的线性化质粒,利用同源重组试剂盒(clonexpressiionestepcloningkit,诺唯赞)将pcr产物构建到沉默载体pyl156中,获得了沉默载体pyl156-gbvip1。并将沉默载体转入农杆菌细胞gv3101中,转化方法如实施实例2中所示。将棉花种子用无菌水浸泡24h,露白后种到混好的营养土中(基质:蛭石=1:1),待幼苗子叶完全平展时进行vigs实验,接种前将携带pyl156-gbvip1载体的农杆菌、携带对照载体pyl156-ghpds的农杆菌及携带pyl156空载体和pyl192载体的农杆菌活化,待农杆菌菌液od600=1.5时,离心收集菌液,利用重悬液(10mmmgcl2,10mmmes,150um乙酰丁香酮)将农杆菌重悬,然后携带pyl192载体的农杆菌分别与携带pyl156-gbvip1,pyl156,pyl156-ghpds载体的农杆菌1:1混合均匀,静置4h后使用。用1ml的注射器针头在棉花幼苗子叶背面扎1个小孔,然后将菌液注射入棉花子叶中,将注射后的棉花幼苗用褐色塑料布遮盖,避光培养24h,然后光照培养,待对照植株出现白化表型时接种黄萎病菌,接菌后25天和35天调查病情并计算病指,病情调查采用五级划分标准划分田间黄萎病病株级别:0级:无病植株;1级:25%以下叶片发病的植株;2级:25%-50%叶片发病的植株;3级:50%-75%叶片发病的植株;4级:75%以上叶片发病的植株。病情指数计算公式为:
结果发现gbvip1基因沉默植株的叶片变黄萎蔫,叶脉呈现黄色网状,株形微显矮化。沉默植株比对照植株的抗黄萎病能力显著下降(图11)。接菌后25天调查病指,沉默植株的黄萎病病指为22.5,对照植株的病指为20.83,接菌后35天沉默植株的黄萎病病指为30.54,对照植株的病指为23.45。
用刀片把沉默植株和对照植株的茎秆剖开,在显微镜下观察维管束部位的表型(图12),对黄萎病侵染后的棉花叶片进行台盼蓝染色观察沉默和对照植株的叶片的死亡细胞比例,取沉默和对照植株相同部位的叶片,浸泡在台盼蓝染液(2.5g台盼蓝,25%乳酸,23%水溶苯酚,25%甘油)中,沸水浴染色2min,自然冷却后室温染色过夜,然后置于1.25g/ml水合氯醛溶液脱色3d,每天更换一次脱色液。在体式显微镜下观察细胞死亡情况(图13)。剖杆观察发现沉默植株的茎秆的木质化程度减弱,维管束导管部发黄褐化。台盼蓝染色试验发现沉默植株的叶片死亡细胞的比例更高。所以,gbvip1基因与棉花抗黄萎病能力正相关,可应用于棉花抗黄萎病育种研究中。
以上所述的实施例仅仅是对本发明的优选实施方式进行描述,并非对本发明的范围进行限定,在不脱离本发明设计精神的前提下,本领域普通技术人员对本发明的技术方案作出的各种变形和改进,均应落入本发明权利要求书确定的保护范围内。
序列表
<120>抗黄萎病相关蛋白gbvip1及其编码基因与应用
<141>2018-10-30
<160>12
<170>siposequencelisting1.0
<210>1
<211>337
<212>prt
<213>gbvip1氨基酸序列(gbvip1)
<400>1
metgluglnlysleuargmetgluileaspglnmetproproarggly
151015
alahishisargargalahisseraspthrthrpheargpheaspasp
202530
leuleuleupheaspproseraspleuaspleusercysleuaspleu
354045
proaspserserserasnproserleuproprovalalaprovalpro
505560
alagluserseraspaspsersercysasnglyproproargserthr
65707580
leuasnasnproarghisileargserleuservalaspseraspphe
859095
pheaspglyleuglyleuthrglyproalaileserglyglyalagly
100105110
aspglulyspheglyglylysargglyalaglyglulysargvalhis
115120125
hisarghisserasnsermetaspglyserthrthralaserpheasp
130135140
valgluserleumetalavalaspglyvallyslysthrmetalapro
145150155160
aspargleualagluleualaleuileaspprolysargalalysarg
165170175
ileleualaasnargglnseralaalaargserlysgluarglysile
180185190
argtyrthrasngluleuglulyslysvalglnthrleuglnthrglu
195200205
alathrasnleuseralaglnvalthrmetleuglnargaspthrthr
210215220
glyleuthrthrgluasnlysgluleulysleuargleuglnalamet
225230235240
gluglnglnalaglnleuargaspalaleuasnglulysleulysglu
245250255
gluvalglnargleuargileglnalaglyglnvalseralametasn
260265270
glyasnpropheasnargglyleuserproglntyrleuthrhisgln
275280285
proalaprohishispheglyvalleuglnthrprohisglnglngln
290295300
glnglnglnglnglnleuglnmetprohisserserthrasnasnpro
305310315320
thrleuasnglyglnproglnprohisphemetasppheasnglnarg
325330335
ala
<210>2
<211>1014
<212>dna
<213>gbvip1核苷酸序列(gbvip1)
<400>2
atggagcaaaagctgcgaatggagatagaccagatgccaccacgtggagctcaccatagg60
cgggctcattccgacacaactttccgtttcgatgatcttctcctcttcgacccctcggac120
ctcgatctttcctgccttgaccttccggattcctcttccaatcccagtctgccacctgtc180
gcacccgtacctgctgaatcctccgatgactcctcatgcaacggccctcctcgctccacc240
ctcaataaccctaggcatatccgtagcctttctgttgactctgatttctttgatggactc300
gggttgactggacccgcgatctccggaggagctggggatgagaaatttggcgggaaaaga360
ggtgcaggagagaagagggttcaccatagacatagtaactcgatggatggttcgactacg420
gcatcgtttgacgttgagtccttgatggccgttgatggtgttaagaaaactatggctcct480
gatagacttgctgagcttgcccttatcgatcccaagagagctaaaaggattctggctaac540
aggcaatcagctgcgcgttcaaaggagaggaagattcgatacacgaatgaactagagaag600
aaggttcagactcttcagacagaagctaccaatctctccgcacaagtcacaatgttacag660
agagacactactgggttgactactgagaacaaggaactgaaactacggctacaggctatg720
gagcaacaagcgcagcttagggatgctttgaatgaaaaattgaaggaagaagtgcaacgg780
cttaggatacaagctggtcaagtgtctgctatgaatggaaatcctttcaatagaggacta840
tctcctcaatatttaacccaccaacctgctccacatcactttggcgtcctgcagacaccg900
catcaacaacaacagcagcagcagcagctgcagatgcctcattcatccaccaataacccg960
actttgaacggacagcctcaacctcactttatggattttaaccagagggcatag1014
<210>3
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>3
tacgccgccgcttcttccgcctaca25
<210>4
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>4
cttaccaagcatttccttccaacat25
<210>5
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>5
ccaatcccagtctgccacct20
<210>6
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>6
tcaacccgagtccatcaaaga21
<210>7
<211>39
<212>dna
<213>人工序列(artificialsequence)
<400>7
acgggggactctagaggatcctacgccgccgcttcttcc39
<210>8
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>8
cgatcggggaaattcgagctccttaccaagcatttccttccaac44
<210>9
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>9
tacgccgccgcttcttccgcctaca25
<210>10
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>10
cttaccaagcatttccttccaacat25
<210>11
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>11
ggcctcgagacgcgtgagctcgccgttgatggtgttaagaaaac44
<210>12
<211>46
<212>dna
<213>人工序列(artificialsequence)
<400>12
agaaggcctccatggggatccatagcctgtagccgtagtttcagtt46
- 还没有人留言评论。精彩留言会获得点赞!