一套用于水稻的人工基因编辑系统的制作方法
本申请涉及一套用于水稻的人工基因编辑系统。
背景技术:
水稻(oryzasatival.)是世界主要粮食作物之一,养活了世界上近一半人口,包括几乎整个东亚和东南亚的人口。中国是世界上水稻总产量最高的国家,水稻产量占全球总量的30%左右。在生产过程中,以稻瘟病、稻曲病和纹枯病为主的水稻三大病害严重制约着水稻的生长发育,导致水稻产量和品质降低,威胁着全球的粮食安全。因此,提高产量,改善稻米品质,增加水稻植株抗病、抗逆性等研究以保证粮食的稳定供给是人类社会可持续性发展的重大课题。水稻作为单子叶植物的模式植物,其研究技术、方法、理论和成果对其它禾本科植物,如小麦、玉米、高粱等具有重要指导作用。
近年来发展起来的crispr/cas9系统因为可以对基因组进行定点修饰而极具应用性。不过crispr/cas9系统在进行核酸切割时,需要识别引导rna(grna)3’端保守的pam序列。现在最常用的spcas9所识别pam序列主要是ngg,尽管spcas9也可识别nag,以及spcas9(vqr)可识别nga等,但其编辑效率均较低;同时基于crispr/spcas9系统发展而来的碱基编辑技术也会因所编辑靶位点的特殊性及可能没有合适的pam序列导致碱基编辑效率受到限制,这些都在很大程度上限制了crispr/cas9系统在水稻基因组编辑中应用。
因此,如果能开发出一种可更高效、适用范围更广、通用性更强、dna特异性更强的对植物基因组,特别是水稻基因组进行定点编辑的crispr/cas9系统,不仅会大大提高对植物基因组编辑的效率,而且将被更广泛的应用于植物基因功能研究、作物育种等方面,将会极大地促进植物基因组编辑领域的进程。
技术实现要素:
本申请提供了一套人工基因编辑系统,所述人工基因编辑系统包括:
第i调节元件,其包括能够编码如氨基酸序列i的核苷酸序列;其中所述氨基酸序列i包括如i-1氨基酸序列、i-2氨基酸序列和i-3氨基酸序列中的一种,其中所述i-1氨基酸序列为如seqidno.1所示的氨基酸序列;所述i-2氨基酸序列包括依次从n端到c端串联的seqidno.2、seqidno.1和seqidno.3所示的氨基酸序列;所述i-3氨基酸序列包括依次从n端到c端串联的seqidno.4和seqidno.1所示的氨基酸序列;
第ii调节元件,其包括依次从5’端到3’端串联的第ii-1核苷酸序列和第ii-2核苷酸序列;所述第ii-1核苷酸序列包括靶核苷酸序列;所述靶核苷酸序列来源于目标生物的基因组中,并且所述靶核苷酸序列中含有目标生物基因组中待突变的靶位点;所述第ii-2核苷酸序列包括来源于化脓链球菌(streptococcuspyogenes)的sgrna核酸序列;所述第ii-1核苷酸序列和所述第ii-2核苷酸序列转录融合,其产物能引导第i调控元件编码的蛋白至目标生物基因组中待突变的靶位点处,并将靶位点处产生碱基进行突变;
当所述第ii调节元件为多个时,包含在每一个所述第ii调节元件中的第ii-1核苷酸序列两两不相同。另外,当所述第ii调节元件为多个时,这些第ii调节元件可以串联的形成连接在一起。
本申请中,人工基因编辑系统中的靶核苷酸序列由该人工基因编辑系统本身与目标生物基因组中的待突变的靶位点共同确定,并且,如上所述,靶核苷酸序列来源于目标生物的基因组中,因此,靶核苷酸序列上的所述靶位点与目标生物基因组中的待突变的靶位点序列一致,因此,为了表述简便起见,两者均称之为靶位点,但突变发生在目标生物的基因组的序列上,而非发生在人工基因编辑系统的序列上。
在一个具体实施方式中,当使用所述i-1氨基酸序列时,所述靶核苷酸序列中的靶位点处于所述靶核苷酸序列的从3′端到5′端方向的3至5位置中的任意一处;当使用所述i-2氨基酸序列时,所述靶核苷酸序列中的靶位点为处于所述靶核苷酸序列的从5′端到3′端方向的2至10位置中的碱基c;当使用所述i-3氨基酸序列时,所述靶核苷酸序列中的靶位点为处于所述靶核苷酸序列的从5′端到3′端方向的2至8位置中的碱基a。
当所述氨基酸序列i为如i-1氨基酸序列时,通过利用本申请的人工基因编辑系统,可以将水稻基因组中内源的特定位点处缺失或在其中插入一个或数个碱基,筛选得到水稻基因相应的缺失或插入突变体。对于这些缺失或插入突变体,有可能会使原基因的功能丢失,也有可能使原基因的功能发生减弱或增强,这取决于实际发生的情况,根据实际需要,选择保留或舍弃那些已完成基因序列检测的突变体。
或者,当所述氨基酸序列i为如i-2氨基酸序列时,当第i调节元件通过利用本申请的人工基因编辑系统,可以将水稻基因组中内源的特定碱基c定点突变为t、a或g中的一种,筛选得到水稻基因功能“矫正”突变体。或者对于其反向互补序列来讲,将g定点突变为a、t或c中的一种,筛选得到水稻基因功能“矫正”突变体,此时,使用的靶核苷酸序列为靶位点处为c那条链上的核苷酸序列。
或者,当所述氨基酸序列i为如i-3氨基酸序列时,通过利用本申请的人工基因编辑系统,可以将水稻基因组中内源的特定碱基a定点突变为g,筛选得到水稻基因功能“矫正”突变体。或对于其反向互补序列来讲,将t定点突变为c,筛选得到水稻基因功能“矫正”突变体,此时,使用的靶核苷酸序列为靶位点处为a那条链上的核苷酸序列。
在一个具体实施方式中,所述目标生物为水稻,所述第i调节元件的核苷酸序列为能够适于在水稻中表达的核苷酸序列,所述第ii调节元件的核苷酸序列为能够适于在水稻中发生转录的核苷酸序列。
在一个具体实施方式中,能够编码如seqidno.1所示氨基酸序列的核苷酸编码序列如seqidno.5所示。如seqidno.5所示核苷酸编码序列能够较优的在水稻中使用。
在一个具体实施方式中,能够编码如seqidno.2所示氨基酸序列的核苷酸编码序列如seqidno.6所示。如seqidno.6所示核苷酸编码序列能够较优的在水稻中使用。
在一个具体实施方式中,能够编码如seqidno.3所示氨基酸序列的核苷酸编码序列如seqidno.7所示。如seqidno.7所示核苷酸编码序列能够较优的在水稻中使用。
在一个具体实施方式中,能够编码如seqidno.4所示氨基酸序列的核苷酸编码序列如seqidno.8所示。如seqidno.8所示核苷酸编码序列能够较优的在水稻中使用。
在一个具体实施方式中,所述第ii-2核苷酸序列如seqidno.9所示。
在一个具体实施方式中,所述第ii-1核苷酸序列还包括含有iis型限制性内切酶的酶切位点的克隆位点,所述靶核苷酸序列通过所述第ii-1核苷酸序列上的所述克隆位点而被克隆到其中(例如通过酶切-连接的方式将所述靶核苷酸序列连接到所述克隆位点上),以使所述第ii-1核苷酸序列与第ii-2序列转录融合;当所述第ii调节元件为多个时,用于克隆不同靶核苷酸序列的所述iis型限制性内切酶的酶切位点两两不相同。
其中,由于所述靶核苷酸序列是根据碱基编辑位点而变化的,因此可以将包括事先克隆到相关位置的限制性内切酶的酶切位点在内的其他元件构建好。在使用之前,再根据碱基编辑目的将所述靶核苷酸序列通过限制性内切酶的酶切位点的切割而被克隆。当所述第ii调节元件为多个时,包含在其中的多个第ii-1核苷酸序列中的限制性内切酶的酶切位点两两不相同,如此,可以有效的保障不同的靶核苷酸顺利的被克隆到目标位置。多个靶核苷酸序列可用于目标生物基因组上的多个待突变的靶位点的碱基替换。
在一个具体实施方式中,优选所述克隆位点的核苷酸序列包括seqidno.10和/或seqidno.11。
在一个具体实施方式中,通过如下方式确定所述靶核苷酸序列:
1)确定水稻基因组上需要被改造的核苷酸序列;
2)判断步骤1)中所确定的需要被改造的核苷酸序列为基因组中的特异性序列(被改造的核苷酸序列的特异性越高,则在进行基因编辑时越准确,否则可能会产生错误识别),
并根据所述第i调节元件来判断待突变的核苷酸位点的碱基发生突变后引起的改变是否符合预期;或者根据所述第i调节元件来判断待突变的核苷酸位点的反向互补碱基发生突变后引起的改变是否符合预期;
对于符合预期者,所述待突变的核苷酸位点即为潜在的靶位点;
3)在需要被改造的核苷酸序列或其反向互补序列中筛选靶标序列:向潜在的靶位点的3′端方向搜索以确认存在能够被所述第i调节元件编码的氨基酸序列i识别的识别模序,并且
当所述氨基酸序列i为如i-1氨基酸序列时,所述靶位点处于所述识别模序5′端上游的-3至-5的位置,由此确定的所述识别模序5′端上游17至21个核苷酸序列为所述靶核苷酸序列;
当所述氨基酸序列i为如i-2氨基酸序列时,所述靶位点处于所述识别模序5′端上游的-19至-11的位置,由此确定的所述识别模序5′端上游17至21个核苷酸序列为所述靶核苷酸序列;
当所述氨基酸序列i为如i-3氨基酸序列时,所述靶位点处于所述识别模序5′端上游的-19至-13的位置,由此确定的所述识别模序5′端上游17至21个核苷酸序列为所述靶核苷酸序列。
在一个具体实施方式中,所识别模序为5′-n1gn2-3′,所述靶核苷酸序列上游的17至21个核苷酸序列,淘汰含有连续五个t的核苷酸序列;其中,所述n1和n2独立地为a、g、c和t中的一种。
在一个具体实施方式中,所述靶核苷酸序列为如seqidno.16、seqidno.17和seqidno.18中所示的至少一种。
在一个具体实施方式中,所述人工基因编辑系统还包括在所述第i调节元件的5’端的能够用于水稻中的,且能够启动所述第i调节元件转录的第一启动子;和/或所述人工基因编辑系统还包括在所述第ii调节元件的5’端的能够用于水稻中的,且能够启动所述第ii调节元件转录的第二启动子。
在一个具体实施方式中,所述第一启动子为rna聚合酶ii型启动子;和/或第二启动子为rna聚合酶iii型启动子。
在一个具体实施方式中,第一启动子为seqidno.12;和/或第二启动子为seqidno.13。
在一个具体实施方式中,所述人工基因编辑系统还包括在所述第i调节元件的3’端的能够终止所述第i调节元件转录的第一终止子;和/或所述人工基因编辑系统还包括在所述第ii调节元件的3’端的能够终止所述第ii调节元件转录的第二终止子。
在一个具体实施方式中,第一终止子为seqidno.14;和/或第二终止子为seqidno.15。
在一个具体实施方式中,所述第i调节元件和所述第ii元件能够被克隆到至少一个载体上。例如,所述第i调节元件表达盒和所述第ii调节元件转录盒能够被克隆或整合到同一个载体上。或第i调节元件表达盒和第ii调节元件转录盒分别位于不同的载体上时,可以采用基因枪法、农杆菌侵染法或peg介导转化法将两个盒或含有两个盒的载体导入到水稻愈伤组织或原生质体细胞中。
在一个具体实施方式中,所述第i调节元件能够被克隆到pcambia1300上;所述第ii调节元件被克隆到入门载体pentr4上。pcambia1300为基于gateway反应并用于水稻遗传转化的双元载体,也可以使用其他类似的双元载体。
在一个具体实施方式中,所述第一启动子、第i调节元件和第一终止子能够被克隆到pcambia1300载体上。
在一个具体实施方式中,第二启动子、第ii调节元件和第二终止子被克隆到pentr4载体上。当所述第ii调节元件为多个时,其5’端的第二启动子和其3’端的终止子也为多个。即第二启动子、第ii调节元件和第二终止子形成一组,成套出现。含有不同第ii调节元件的多个组可以串联的形成连接在一起。其中,第ii调节元件的不同,主要指的是第ii-1核苷酸序列的不同。
在一个具体实施方式中,所述第i调节元件和所述第ii调节元件能够被整合到同一个载体上,或被分布在两个载体上一起使用。
本申请之二提供了一种如本申请之一中任意一人工基因编辑系统在用于水稻基因组突变中的应用。
本申请之三提供了一种实现水稻基因组定点编辑方法,其包括如下步骤:
1)将本申请之一中任意一人工基因编辑系统通过农杆菌介导、基因枪轰击或peg介导转化的方法中的一种导入到水稻愈伤组织或水稻原生质体中,然后培养获得水稻植株;
2)筛选获得含有所需突变的水稻植株;进一步地,所述水稻植株能够产生含有所述突变的水稻种子。
在进行所述的人工基因编辑系统导入时,可以采用peg介导转化的方法,也可以采用基因枪法或农杆菌侵染法中的一种将所述的人工基因编辑系统导入到水稻原生质体或愈伤组织中,这是本领域技术人员容易理解的。本领域的技术人员公知,水稻基因组dna由两条链组成,因此,所述靶核苷酸序列可以在其中互补的任意一条链上。例如,当所述靶核苷酸序列位于某一功能基因的一正义链中时,如果在该功能基因的特定位点上发生一至数个碱基的缺失或插入,并且如果其中的一种突变能够获得预期的使该基因移码突变而产生基因失活,则可以采用此系统来实现,即可以通过直接进行正义链上的碱基缺失或插入,得到水稻基因敲除突变体;当所述靶核苷酸序列位于某一功能基因的正义条链中时,如果该功能基因的特定位点上的c被定点突变为t后,并且如果其中的一种突变能够获得预期的其对应的功能蛋白中的氨基酸,则可以采用此系统来实现,即可以通过直接进行正义链上的碱基替换来实现三联体密码子中的c替换为t,得到水稻基因功能“矫正”突变体;或当所述靶核苷酸序列位于某一功能基因的反义链中时,如果该功能基因的特定位点上的g被定点突变为a后,并且如果其中的一种突变能够获得预期的其对应的功能蛋白中的氨基酸,也可以采用此系统来实现,即可以通过将反义链中的c被定点突变为t,进而使正义链中的相应互补的g替换为a来改变正义链中的所述三联体密码子编码氨基酸,得到水稻基因功能“矫正”突变体;当所述靶核苷酸序列位于某一功能基因的反义链中时,如果该功能基因的特定位点上的t被定点突变为c后,并且如果其中的一种突变能够获得预期的其对应的功能蛋白中的氨基酸,则可以采用此系统来实现,即可以通过将该反义链中的a被定点突变为g,进而使正义链中的相应互补的t替换为c来改变正义链中的所述三联体密码子编码氨基酸,得到水稻基因功能“矫正”突变体;或当所述靶核苷酸序列位于某一功能基因的正义链中时,如果该功能基因的特定位点上的a被定点突变为g后,并且如果其中的一种突变能够获得预期的其对应的功能蛋白中的氨基酸,也可以采用此系统来实现,即可以通过直接进行正义链上的碱基替换来实现三联体密码子中的a替换为g,从而得到水稻基因功能“矫正”突变体。
本申请的有益效果在于:
a)第ii调节元件可以为多个,这样可以同时编辑水稻细胞内多个基因靶位点。
b)通过选用本申请的人工基因编辑系统中不同的第i调节元件可实现了对水稻基因组中的基因敲除(包括缺失或插入),或从碱基对at到碱基对gc的替换,或从碱基对gc到碱基对at的替换。
c)该全新基因编辑工具盒扩展了已有基因编辑工具盒的pam序列,具有更宽更广的pam序列,其能广泛地应用于水稻基因组中靶标基因的敲除或单碱基的定向突变,以此创制基因功能失活或获得性突变体材料。尤其是碱基编辑系统在植物中的应用比通过hr的基因替换或通过nhej的基因插入更加有效和经济;而广泛的pam序列使得实现任意位点碱基替换的可能性加大,为植物研究领域科研人员提供一个重要的基因功能研究工具,并在水稻基因功能研究和分子育种方向上为培育水稻新品种提供了新的策略。
附图说明
图1显示了使用pubi:cas9ng在oscerk1基因靶位点处的编辑效果图。
图2显示了使用pubi:rbe22在osrlck185基因靶位点处的编辑效果图。
图3显示了使用pubi:rbe23在os03g02040基因靶位点处的编辑效果图。
具体实施方式
以下通过优选的实施例的形式对本申请的上述内容作进一步的详细说明,但不构成对本发明的限制。
如无特别说明,本申请的实施例中的试剂均可通过商业途径购买。
pcambia1300来源于biovectorntcc典型培养物保藏中心。在pcambia1300中插入了attr1-ccdb-attr2模块,用于gateway反应接受来自于入门载体的attl1-靶向序列转录模块-attl2模块。
pentr4载体的来源:购自美国invitrogen公司。
pbluescriptsk载体的来源:购自clontech公司。
实施例1
重组质粒的构建
构建该载体的技术路线如下:
1.1pubi:cas9ng重组质粒构建
确定cas9ng的氨基酸序列如seqidno.1所示,并根据cas9ng的氨基酸序列确定用于在水稻中表达的基因序列seqidno.5,并人工合成如seqidno.5所示的4299bp的核苷酸序列,将其克隆至puc57上,命名为puc57:cas9ng(由北京擎科新业生物技术有限公司完成)。然后将seqidno.12(玉米泛素启动子ubip)、seqidno.5、seqidno.14(nos终止子)按照从5’到3’的方向克隆到pcambia1300载体,命名为pubi:cas9ng。
质粒pubi:cas9ng的主要构成如下:camv35s启动子(genebank登陆号为fj362600.1,第10382至第11162核苷酸序列),潮霉素基因(genebank登陆号为ky420085.1),nos终止子(seqidno.14),pvs1repa(genebank登陆号为ky420084.1,第5755至第6435核苷酸序列),pvs1复制起点(genebank登陆号为ky420084.1,第4066至第5066核苷酸序列),attr1(genbank登陆号为kr233518.1,第2055至第2174核苷酸序列),ccdb表达框genbank登陆号为kr233518.1,第3289至第3594核苷酸序列),attr2(genbank登陆号为kr233518.1,第3635至第3759核苷酸序列),ubip启动子(seqidno.12),cas9ng基因(seqidno.5),nos终止子(seqidno.14)。
1.2pubi:rbe22重组质粒构建
用ecori和spei对本实验室自有载体pubi:rbe9(improvedbaseeditorforefficientlyinducinggeneticvariationsinricewithcrispr/cas9.renbin,yanfang,kuangyongjie,lina,zhangdawei,zhouxueping,linhonghuiandzhouhuanbin.molecularplant,2018,11:623-626)进行双酶切,回收5.05kb的片段;用ecori和spei对克隆载体pbluescriptsk进行双酶切,回收3kb的线性化载体骨架;然后将两者连接,经转化、菌落pcr和酶切验证后备用,获得的重组质粒命名为pbs:rbe9。
以rapo-r1(seqidno.19:agcaagtccgattgaatact)和ugi-f1(seqidno.20:tccggcggaagtacaaac)为引物,以重组质粒pbs:rbe9为模板,利用i-5tm2×highfidelitymastermix(购自克劳宁(北京)生物科技有限公司)进行pcr扩增,获得约4.0kb的载体骨架;同时,以oscas9-fg1-f1(seqidno.21:attgggacaaactctgtgg和oscas9-fg2-r1(seqidno.22:gtcaccgcccaactgcga)为引物,以puc57:cas9ng为模板,利用i-5tm2×highfidelitymastermix进行pcr扩增,获得约4.3kb的cas9ng基因的pcr片段,该片段经纯化后进行磷酸化处理,与上述4.0kb载体骨架进行连接,经转化、菌落pcr和酶切验证、测序验证后备用,获得的重组质粒命名为pbs:rbe22。
利用bamhi和spei对pbs:rbe22进行双酶切并回收5.03kb的rbe22片段;利用bamhi和spei对载体pubi:cas9ng进行双酶切并回收约12kb的载体骨架;将两者连接,经转化、菌落pcr和酶切验证后备用,获得的重组质粒命名为pubi:rbe22。
质粒pubi:rbe22的构成如下:camv35s启动子(genebank登陆号为fj362600.1,第10382至第11162核苷酸序列),潮霉素基因(genebank登陆号为ky420085.1),nos终止子(seqidno.14),pvs1repa(genebank登陆号为ky420084.1,第5755至第6435核苷酸序列),pvs1复制起点(genebank登陆号为ky420084.1,第4066至第5066核苷酸序列),attr1(genbank登陆号为kr233518.1,第2055至第2174核苷酸序列),ccdb表达框genbank登陆号为kr233518.1,第3289至第3594核苷酸序列),attr2(genbank登陆号为kr233518.1,第3635至第3759核苷酸序列),ubip启动子(seqidno.12),aid基因(seqidno.6),cas9ng基因(seqidno.5),ugi基因(seqidno.7),nos终止子(seqidno.14)。
1.3pubi:rbe23重组质粒构建
根据氨基酸序列seqidno.4确定用于在水稻中表达的基因序列seqidno.8,并人工合成如seqidno.8所示的1191bp的核苷酸序列,将其克隆至puc57上,命名为puc57:tada(由北京擎科新业生物技术有限公司完成)。
以puc57-f1(seqidno.23:gcgcgcttggcgtaatca)和tada-r1(seqidno.24:agccagaccaattgagtattttttgtc)为引物,以载体puc57:tada为模板,利用i-5tm2×highfidelitymastermix进行pcr扩增,纯化后获得4.13kb的载体骨架;再以oscas9-fg1-f1(seqidno.21)和nls-r2(seqidno.25:cactagttcacccgccaac)为引物,以puc57:cas9ng为模板利用i-5tm2×highfidelitymastermix进行pcr扩增,获得约4.3kb的cas9ng基因的pcr片段,纯化后进行磷酸化处理,与上述4.13kb载体骨架进行连接,经转化、菌落pcr和酶切验证后测序备用,获得的重组质粒命名为puc57:rbe23。
利用bamhi和spei对puc57:rbe23进行双酶切并回收5.33kb的rbe23片段;利用bamhi和spei对载体pubi:cas9ng并回收约12kb的载体骨架;然后将两者连接,经转化、菌落pcr和酶切验证后测序备用,获得的重组质粒命名为pubi:rbe23。
质粒pubi:rbe23的构成如下:camv35s启动子(genebank登陆号为fj362600.1,第10382至第11162核苷酸序列),潮霉素基因(genebank登陆号为ky420085.1),nos终止子(seqidno.14),pvs1repa(genebank登陆号为ky420084.1,第5755至第6435核苷酸序列),pvs1复制起点(genebank登陆号为ky420084.1,第4066至第5066核苷酸序列),attr1(genbank登陆号为kr233518.1,第2055至第2174核苷酸序列),ccdb表达框genbank登陆号为kr233518.1,第3289至第3594核苷酸序列),attr2(genbank登陆号为kr233518.1,第3635至第3759核苷酸序列),ubip启动子(seqidno.12),tada基因(seqidno.8),cas9ng基因(seqidno.5),nos终止子(seqidno.14)。
1.4pentr4:sgrna的构建
按照从5’端到3’端的方向,将依次连接的u6启动子序列(seqidno.13)、含有两个btgzi酶切位点的核苷酸序列(seqidno.10)、grnascaffold序列(seqidno.9)、(t)8终止序列(seqidno.15)、u6启动子序列(seqidno.13)、含有两个bsai酶切位点的核苷酸序列(seqidno.11)、sgrna序列(seqidno.9)、(t)8终止序列(seqidno.15)进行人工合成,并克隆到pentr4载体中,命名为pentr4:sgrna。其中的两个btgzi或两个bsai酶切位点用于克隆中特定基因中的靶核苷酸序列。
实施例2:利用pubi:cas9ng进行水稻内源基因oscerk1的敲除
2.1针对oscerk1基因的识别序列设计和克隆
oscerk1(loc_os08g42580)基因的转录序列和基因组序列从msu/tigr水稻基因组数据库中获得(http://rice.plantbiology.msu.edu/)。
对于oscerk1基因,设计含有与btgzi酶切位点末端连接匹配的靶核苷酸序列(seqidno.16:
2.2peg介导的pubi:cas9ng系统转化粳稻品种kitaake原生质体及基因编辑检测
1)水稻原生质体的制备:
将去壳的成熟的水稻种子用50%的商业化消毒液处理25min;无菌水清洗3-5次,然后将种子转移至无菌的培养皿中,吸出多余的水份;将种子放置于1/2ms培养基上(2.2g/lms粉;30g/l蔗糖;6g/l植物凝胶;ph5.7),于光照培养室培养10天。用剪刀剪取水稻幼苗茎叶,用单面刀片横切茎部,越细越好,并切好的水稻材料转移至无菌三角瓶中,加入10ml酶解液(1.5%纤维素酶;0.3%离析酶r-10;0.4m甘露醇;2mm2-(n-吗啉)乙磺酸(mes);0.1×w5溶液;ph5.7),轻轻混匀后用锡箔纸包住瓶身,抽真空30min,之后置于水平摇床(转速约60rpm),酶解6h。酶解后,尼龙网(孔径为35μm)过滤收集原生质体溶液。原生质体溶液经室温离心(离心力1000g,时间5min)后弃上清,下层原生质体沉淀加入w5溶液(154mmnacl;125mmcacl2;25mmkcl;2mmmes;ph5.7)重悬并1000g离心5min后弃上清,加入适量mmg溶液(0.4m甘露醇;20mmcacl2;25mmmes;ph5.7)重悬原生质体。
2)peg介导的水稻原生质体转化及原生质体基因组dna提取
取新圆底离心管,依次加入20μl质粒pubi:cas9ng(浓度为1000ng/μl)、20μl质粒pentr4:sgrna-goscerk1(浓度为1000ng/μl)、400μl原生质体、440μl(等体积)40%peg4000溶液(40%(w/v)peg4000;0.4m甘露醇;100mmca(no3)2;ph5.7),轻轻混匀,放置15min。加入1mlw5溶液稀释中止转化反应,1000g离心2min。弃上清,加入1mlw5溶液,重悬原生质体,并将其转入12孔细胞培养板中,锡箔纸包裹避光,室温培养2天后收集原生质体,采用ctab法提取原生质体基因组dna。
3)靶位点突变类型的检测
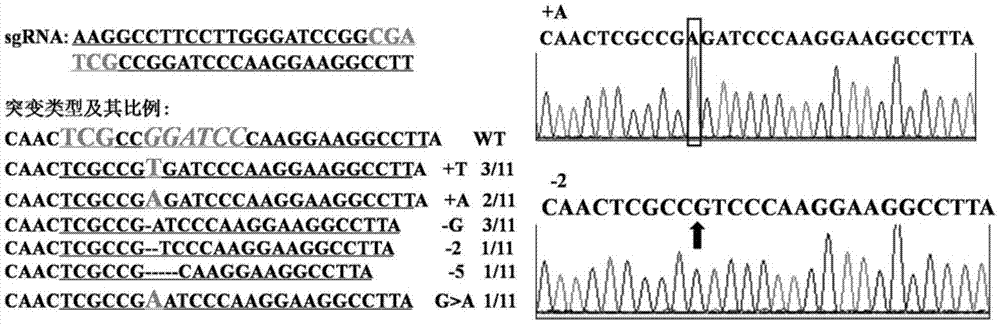
根据oscerk1基因的靶位点dna序列设计用于鉴定的特异性的pcr引物:oscerk1-f1(seqidno.28:gacgtctacgcctttggtgt),oscerk1-r1(seqidno.29:gtcagctgcaaaatgcaatg),pcr产物片段为393bp。首先利用bamhi对原生质体基因组dna进行酶切消解2h,再以酶切产物为模板,以oscerk1-f1(seqidno.28)和oscerk1-r1(seqidno.29)为引物,利用i-5tm2×highfidelitymastermix进行pcr扩增,获得393bp的pcr片段。该pcr产物再bamhⅰ酶解3h并利用琼脂糖凝胶电泳除去靶位点未被成功编辑的pcr产物,利用axyprep凝胶回收试剂盒回收靶位点发生碱缺失或插入的片段,连接ta克隆载体和sanger测序分析突变类型。如图1所示,随机测序获得了11个单克隆的序列,共检测到6种突变类型,分别为碱基缺失(-1、-2和-4bp)、碱基插入(+t和+a)以及碱基替换(g替换为a),这表明cas9ng可识别ngapam基序完成基因编辑。
实施例3:利用pubi:rbe22进行水稻内源基因osrlck185的碱基c向t替换
osrlck185(loc_os05g30870)基因的转录序列和基因组序列从msu/tigr水稻基因组数据库中获得(http://rice.plantbiology.msu.edu/)。
对于osrlck185基因,设计含有与bsai酶切位点末端连接匹配的靶核苷酸序列(seqidno.17:
其他操作同实施例2。
根据osrlck185基因的靶位点dna序列设计用于鉴定的特异性的pcr引物:osrlck185-f1(seqidno.32:tccatggccttgttcctctt),osrlck185-r1(seqidno.33:tgctgctagacacatccaca),pcr产物片段为484bp。首先利用alw44i对原生质体基因组dna进行酶切消解2h,再以酶切产物为模板,以osrlck185-f1(seqidno.32)和osrlck185-r1(seqidno.33)为引物,利用i-5tm2×highfidelitymastermix进行pcr扩增,获得484bp的pcr片段。该pcr产物再alw44i酶解3h并利用琼脂糖凝胶电泳除去靶位点未被成功编辑的pcr产物,利用axyprep凝胶回收试剂盒回收成功替换靶位点碱基的片段,连接ta克隆载体和sanger测序分析突变类型。如图2所示,随机测序获得了10个单克隆的序列,均检测到靶碱基g突变为a,其中有3种突变类型,分别为g4,6>a、g4,6,9>a以及g4,6,9,14>a,这表明rbe22可识别ngcpam基序完成碱基编辑。
实施例4:利用pubi:rbe23进行水稻内源基因os03g02040的碱基a向g替换
os03g02040基因的转录序列和基因组序列从msu/tigr水稻基因组数据库中获得(http://rice.plantbiology.msu.edu/)。
对于os03g02040基因,设计含有与bsai酶切位点末端连接匹配的靶核苷酸序列(seqidno.18:
其他操作同实施例2。
根据os03g02040基因的靶位点dna序列设计用于鉴定的特异性的pcr引物:os03g02040-f1(seqidno.36:cactagcacgacgcactttc),os03g02040-r1(seqidno.37:agaacacgcgcatcatatc),pcr产物片段为493bp。首先利用alw44i对原生质体基因组dna进行酶切消解2h,再以酶切产物为模板,以os03g02040-f1(seqidno.36)和os03g02040-r1(seqidno.37)为引物,利用i-5tm2×highfidelitymastermix进行pcr扩增,获得493bp的pcr片段。该pcr产物再xbai酶解3h后利用琼脂糖凝胶电泳除去靶位点未被成功编辑的pcr产物,利用axyprep凝胶回收试剂盒回收成功替换靶位点碱基的片段,连接ta克隆载体和sanger测序分析突变类型。如图3所示,测序结果显示检测到靶碱基t突变为c,其中,这表明rbe23可识别ngtpam基序完成碱基编辑。
序列表
<110>中国农业科学院植物保护研究所
<120>一套用于水稻的人工基因编辑系统
<130>lha1860907
<160>37
<170>siposequencelisting1.0
<210>1
<211>1417
<212>prt
<213>人工序列(non)
<400>1
metasptyrlysasphisaspglyasptyrlysasphisaspileasp
151015
tyrlysaspaspaspasplysmetalaprolyslyslysarglysval
202530
glyilehisglyvalproalaalaasplyslystyrserileglyleu
354045
aspileglythrasnservalglytrpalavalilethraspglutyr
505560
lysvalproserlyslysphelysvalleuglyasnthrasparghis
65707580
serilelyslysasnleuileglyalaleuleupheaspserglyglu
859095
thralaglualathrargleulysargthralaargargargtyrthr
100105110
argarglysasnargilecystyrleuglngluilepheserasnglu
115120125
metalalysvalaspaspserphephehisargleuglugluserphe
130135140
leuvalglugluasplyslyshisgluarghisproilepheglyasn
145150155160
ilevalaspgluvalalatyrhisglulystyrprothriletyrhis
165170175
leuarglyslysleuvalaspserthrasplysalaaspleuargleu
180185190
iletyrleualaleualahismetilelyspheargglyhispheleu
195200205
ilegluglyaspleuasnproaspasnseraspvalasplysleuphe
210215220
ileglnleuvalglnthrtyrasnglnleupheglugluasnproile
225230235240
asnalaserglyvalaspalalysalaileleuseralaargleuser
245250255
lysserargargleugluasnleuilealaglnleuproglyglulys
260265270
lysasnglyleupheglyasnleuilealaleuserleuglyleuthr
275280285
proasnphelysserasnpheaspleualagluaspalalysleugln
290295300
leuserlysaspthrtyraspaspaspleuaspasnleuleualagln
305310315320
ileglyaspglntyralaaspleupheleualaalalysasnleuser
325330335
aspalaileleuleuseraspileleuargvalasnthrgluilethr
340345350
lysalaproleuseralasermetilelysargtyraspgluhishis
355360365
glnaspleuthrleuleulysalaleuvalargglnglnleuproglu
370375380
lystyrlysgluilephepheaspglnserlysasnglytyralagly
385390395400
tyrileaspglyglyalaserglnglugluphetyrlyspheilelys
405410415
proileleuglulysmetaspglythrglugluleuleuvallysleu
420425430
asnarggluaspleuleuarglysglnargthrpheaspasnglyser
435440445
ileprohisglnilehisleuglygluleuhisalaileleuargarg
450455460
glngluaspphetyrpropheleulysaspasnargglulysileglu
465470475480
lysileleuthrpheargileprotyrtyrvalglyproleualaarg
485490495
glyasnserargphealatrpmetthrarglysserglugluthrile
500505510
thrprotrpasnpheglugluvalvalasplysglyalaseralagln
515520525
serpheilegluargmetthrasnpheasplysasnleuproasnglu
530535540
lysvalleuprolyshisserleuleutyrglutyrphethrvaltyr
545550555560
asngluleuthrlysvallystyrvalthrgluglymetarglyspro
565570575
alapheleuserglygluglnlyslysalailevalaspleuleuphe
580585590
lysthrasnarglysvalthrvallysglnleulysgluasptyrphe
595600605
lyslysileglucyspheaspservalgluileserglyvalgluasp
610615620
argpheasnalaserleuglythrtyrhisaspleuleulysileile
625630635640
lysasplysasppheleuaspasnglugluasngluaspileleuglu
645650655
aspilevalleuthrleuthrleuphegluaspargglumetileglu
660665670
gluargleulysthrtyralahisleupheaspasplysvalmetlys
675680685
glnleulysargargargtyrthrglytrpglyargleuserarglys
690695700
leuileasnglyileargasplysglnserglylysthrileleuasp
705710715720
pheleulysseraspglyphealaasnargasnphemetglnleuile
725730735
hisaspaspserleuthrphelysgluaspileglnlysalaglnval
740745750
serglyglnglyaspserleuhisgluhisilealaasnleualagly
755760765
serproalailelyslysglyileleuglnthrvallysvalvalasp
770775780
gluleuvallysvalmetglyarghislysprogluasnilevalile
785790795800
glumetalaarggluasnglnthrthrglnlysglyglnlysasnser
805810815
arggluargmetlysargileglugluglyilelysgluleuglyser
820825830
glnileleulysgluhisprovalgluasnthrglnleuglnasnglu
835840845
lysleutyrleutyrtyrleuglnasnglyargaspmettyrvalasp
850855860
glngluleuaspileasnargleuserasptyraspvalasphisile
865870875880
valproglnserpheleulysaspaspserileaspasnlysvalleu
885890895
thrargserasplysasnargglylysseraspasnvalproserglu
900905910
gluvalvallyslysmetlysasntyrtrpargglnleuleuasnala
915920925
lysleuilethrglnarglyspheaspasnleuthrlysalagluarg
930935940
glyglyleusergluleuasplysalaglypheilelysargglnleu
945950955960
valgluthrargglnilethrlyshisvalalaglnileleuaspser
965970975
argmetasnthrlystyraspgluasnasplysleuilearggluval
980985990
lysvalilethrleulysserlysleuvalseraspphearglysasp
99510001005
pheglnphetyrlysvalarggluileasnasntyrhishisalahis
101010151020
aspalatyrleuasnalavalvalglythralaleuilelyslystyr
1025103010351040
prolysleuglusergluphevaltyrglyasptyrlysvaltyrasp
104510501055
valarglysmetilealalyssergluglngluileglylysalathr
106010651070
alalystyrphephetyrserasnilemetasnphephelysthrglu
107510801085
ilethrleualaasnglygluilearglysargproleuilegluthr
109010951100
asnglygluthrglygluilevaltrpasplysglyargasppheala
1105111011151120
thrvalarglysvalleusermetproglnvalasnilevallyslys
112511301135
thrgluvalglnthrglyglypheserlysgluserileargprolys
114011451150
argasnserasplysleuilealaarglyslysasptrpaspprolys
115511601165
lystyrglyglyphevalserprothrvalalatyrservalleuval
117011751180
valalalysvalglulysglylysserlyslysleulysservallys
1185119011951200
gluleuleuglyilethrilemetgluargserserpheglulysasn
120512101215
proileasppheleuglualalysglytyrlysgluvallyslysasp
122012251230
leuileilelysleuprolystyrserleuphegluleugluasngly
123512401245
arglysargmetleualaseralaargpheleuglnlysglyasnglu
125012551260
leualaleuproserlystyrvalasnpheleutyrleualaserhis
1265127012751280
tyrglulysleulysglyserprogluaspasngluglnlysglnleu
128512901295
phevalgluglnhislyshistyrleuaspgluileilegluglnile
130013051310
serglupheserlysargvalileleualaaspalaasnleuasplys
131513201325
valleuseralatyrasnlyshisargasplysproileargglugln
133013351340
alagluasnileilehisleuphethrleuthrasnleuglyalapro
1345135013551360
argalaphelystyrpheaspthrthrileasparglysvaltyrarg
136513701375
serthrlysgluvalleuaspalathrleuilehisglnserilethr
138013851390
glyleutyrgluthrargileaspleuserglnleuglyglyasparg
139514001405
prolyslyslysarglysvalglygly
14101415
<210>2
<211>211
<212>prt
<213>人工序列(non)
<400>2
metaspserleuleumetasnargargglupheleutyrglnphelys
151015
asnvalargtrpalalysglyargarggluthrtyrleucystyrval
202530
vallysargargaspseralathrserpheserleuasppheglytyr
354045
leuargasnlysasnglycyshisvalgluleuleupheleuargtyr
505560
ileserasptrpaspleuaspproglyargcystyrargvalthrtrp
65707580
pheilesertrpserprocystyraspcysalaarghisvalalaasp
859095
pheleuargglyasnproasnleuserleuargilephethralaarg
100105110
leutyrphecysgluasparglysalagluprogluglyleuargarg
115120125
leuhisargalaglyvalglnilealailemetthrphelysasptyr
130135140
phetyrcystrpasnthrphevalgluasnhisglyargthrphelys
145150155160
alatrpgluglyleuhisgluasnservalargleuserargglnleu
165170175
argargileleuleuproleutyrgluvalaspaspleuargaspala
180185190
pheargthrserglysergluthrproglythrsergluseralathr
195200205
progluser
210
<210>3
<211>91
<212>prt
<213>人工序列(non)
<400>3
serglyglyserthrasnleuseraspileileglulysgluthrgly
151015
lysglnleuvalileglngluserileleumetleuproglugluval
202530
glugluvalileglyasnlysprogluseraspileleuvalhisthr
354045
alatyraspgluserthraspgluasnvalmetleuleuthrserasp
505560
alaproglutyrlysprotrpalaleuvalileglnaspserasngly
65707580
gluasnlysilelysmetleuserglyglyser
8590
<210>4
<211>397
<212>prt
<213>人工序列(non)
<400>4
metsergluvalglupheserhisglutyrtrpmetarghisalaleu
151015
thrleualalysargalatrpaspgluarggluvalprovalglyala
202530
valleuvalhisasnasnargvalileglygluglytrpasnargpro
354045
ileglyarghisaspprothralahisalagluilemetalaleuarg
505560
glnglyglyleuvalmetglnasntyrargleuileaspalathrleu
65707580
tyrvalthrleugluprocysvalmetcysalaglyalametilehis
859095
serargileglyargvalvalpheglyalaargaspalalysthrgly
100105110
alaalaglyserleumetaspvalleuhishisproglymetasnhis
115120125
argvalgluilethrgluglyileleualaaspglucysalaalaleu
130135140
leuseraspphepheargmetargargglngluilelysalaglnlys
145150155160
lysalaglnserserthraspserglyglyserserglyglyserser
165170175
glysergluthrproglythrsergluseralathrprogluserser
180185190
glyglyserserglyglysersergluvalglupheserhisglutyr
195200205
trpmetarghisalaleuthrleualalysargalaargaspgluarg
210215220
gluvalprovalglyalavalleuvalleuasnasnargvalilegly
225230235240
gluglytrpasnargalaileglyleuhisaspprothralahisala
245250255
gluilemetalaleuargglnglyglyleuvalmetglnasntyrarg
260265270
leuileaspalathrleutyrvalthrphegluprocysvalmetcys
275280285
alaglyalametilehisserargileglyargvalvalpheglyval
290295300
argasnalalysthrglyalaalaglyserleumetaspvalleuhis
305310315320
tyrproglymetasnhisargvalgluilethrgluglyileleuala
325330335
aspglucysalaalaleuleucystyrphepheargmetproarggln
340345350
valpheasnalaglnlyslysalaglnserserthraspserglygly
355360365
serserglyglyserserglysergluthrproglythrsergluser
370375380
alathrprogluserserglyglyserserglyglyser
385390395
<210>5
<211>4254
<212>dna
<213>人工序列(non)
<400>5
atggactataaggatcacgatggcgactacaaggatcatgacattgactataaggatgac60
gacgataagatggcacctaagaagaaaaggaaagtcggcattcatggcgttccggcagcc120
gacaaaaagtatagcatcggcctcgatattgggacaaactctgtgggctgggcggtaatt180
accgacgagtacaaggtgcctagtaagaaatttaaagtgctcggaaacactgacaggcac240
tctataaagaagaacctgatcggggcactgcttttcgactccggagagacggcggaggcg300
acgcgtctcaagcgtaccgcgcgccgcaggtacacaagaaggaagaataggatctgctac360
ttgcaggaaatcttcagtaacgagatggcgaaggtcgacgatagtttctttcatcggttg420
gaagaatcgttcctcgtagaggaggacaaaaagcacgagcgtcacccaatattcgggaat480
attgttgacgaggttgcctaccatgagaaatatcctacaatatatcacctccgtaagaag540
cttgtcgattcaactgataaggctgatctcagactcatctatcttgccctcgcacatatg600
attaagtttcgtggccacttcttgattgaaggcgacctcaacccggacaactcagatgtt660
gacaagctttttatacagctcgtccagacatataaccagctgtttgaagagaatcccatc720
aatgcgagtggggttgatgctaaagccattttgtccgccaggttgtccaaatctcgcaga780
ctggaaaacctgatcgcacagcttcccggtgaaaagaaaaacgggctcttcggcaatctc840
atcgcactgtccctcggcctcaccccaaacttcaagtctaacttcgacctggccgaggat900
gcgaagctccagctgtcaaaagatacatacgacgacgatttggacaatctgcttgcgcaa960
ataggcgaccagtatgcggacctgttcctggctgccaaaaatctgtcagatgcaatcctc1020
ctgtccgatatattgcgtgtgaacaccgaaatcacgaaggcaccgcttagcgcatccatg1080
atcaagagatacgacgagcaccatcaggacctcacactcctcaaggcgcttgttcgtcag1140
cagcttcccgagaaatataaggaaatttttttcgatcaaagcaagaatggatatgctggc1200
tatattgacggtggcgcttcgcaggaggagttctataaattcattaagccgattctggag1260
aagatggacggaacggaggagctcctcgtcaagcttaaccgggaagacctgttgcggaag1320
cagaggacttttgataacggctctattccgcaccaaatccatctgggtgagttgcacgca1380
atcttgagaagacaagaggatttctacccgttccttaaggataacagagagaagatagaa1440
aaaatactgaccttcaggataccatactatgtgggcccactggcgcgcggaaatagtcgt1500
ttcgcatggatgactagaaagtccgaagaaacgatcacgccatggaattttgaggaagtg1560
gtcgacaagggcgcctctgcccagagcttcatcgaaaggatgaccaattttgacaaaaat1620
ctgcctaacgaaaaggtgcttccgaagcacagcctgttgtatgaatacttcacagtttat1680
aacgagctcactaaggtcaagtacgtcacggagggcatgcgtaagcctgctttcctgtct1740
ggtgaacaaaaaaaggcgattgtggacctccttttcaagacgaaccgtaaagttactgtg1800
aagcaactgaaagaggattactttaagaaaattgagtgcttcgacagtgtggagatttcc1860
ggtgtcgaggaccggtttaacgccagcctgggtacgtatcatgacctgcttaaaattatc1920
aaggataaagatttcctggataatgaagagaacgaagatatactggaggacattgtgttg1980
actttgaccctcttcgaggacagagagatgattgaggaaagactgaagacctacgcacac2040
ctttttgatgacaaggtcatgaaacaactcaagcgccggcgctatactggctggggccgg2100
ctttctcgcaagctcatcaatgggattcgggataagcaatcaggcaagacaattttggac2160
ttcctcaaatccgacggattcgcaaataggaattttatgcagctgatacatgacgactct2220
ttgacattcaaagaagacatacagaaggctcaggtcagcggccaaggagattctttgcac2280
gagcatatcgctaacttggcaggtagccccgccataaaaaagggcattcttcaaacggta2340
aaagttgttgacgaactcgtgaaggttatgggccgtcataagccggaaaacattgttatt2400
gaaatggctagggaaaatcagacgacccagaagggacagaaaaatagcagggagcggatg2460
aagagaattgaagagggaattaaggagcttggatctcagattcttaaggagcaccctgtg2520
gagaacacccaacttcagaatgaaaagctctacctttactaccttcaaaacggccgggat2580
atgtacgtcgatcaggaacttgacattaaccggttgagcgattatgacgttgaccatatt2640
gtgccccaatctttccttaaagacgactctatcgacaataaagtgctgacgcgcagcgat2700
aaaaatcgcggtaagtcggataatgtcccgtcggaagaggtggttaaaaaaatgaagaac2760
tattggaggcaactcctgaatgccaagctgatcactcagaggaaattcgacaatctcacc2820
aaggcagaaaggggtggacttagcgagctcgacaaggccggttttatcaaaagacagctg2880
gtggagacacgccaaatcaccaaacacgttgcccagatcctggattcgaggatgaacacg2940
aagtatgacgagaacgacaagttgattagggaagtcaaggtcatcactttgaagtccaag3000
ctggtgagcgactttcgcaaagacttccagttttacaaagtcagggaaattaataactac3060
caccacgcccacgacgcctaccttaacgccgtggttggcacagcactcatcaagaaatac3120
cctaagctcgaatctgagttcgtctatggcgactataaggtctacgacgttagaaaaatg3180
atcgcgaaatctgagcaggaaataggcaaggcaactgccaagtacttcttctattccaat3240
atcatgaacttttttaagacggagattaccctggcgaatggtgagatccgcaagcgccct3300
ttgattgagacaaacggagaaacaggagagatcgtatgggacaaagggcgggactttgct3360
actgttaggaaggtgctctctatgccacaagttaacattgtcaaaaaaactgaagtgcag3420
acaggtgggtttagcaaggaatctatccgcccgaagaggaactctgacaagctgatcgcc3480
cgcaagaaagattgggacccgaaaaagtacggaggattcgtttcccccacagttgcgtac3540
tccgtgcttgtcgtggccaaagtggagaagggcaagtctaagaagctcaagagcgtcaaa3600
gagttgttggggatcacgattatggagcggtcgtctttcgaaaagaatccgatagatttt3660
ctcgaggccaagggttataaagaagtcaagaaggatcttatcatcaagctccctaagtac3720
tccctctttgagcttgaaaacggacggaaaagaatgctggcttcagcgcgctttcttcag3780
aagggtaatgaactcgctctgccctcaaaatatgtgaatttcctttacctggcatcacac3840
tatgagaagcttaagggttctccagaggacaacgagcagaagcaactgttcgttgaacaa3900
cacaagcactaccttgacgagattatcgagcaaatcagcgagtttagcaagcgcgttata3960
ctggcagacgcaaatcttgataaggtccttagcgcctacaacaagcatagagacaaaccc4020
atccgggagcaggccgagaacattattcatctcttcaccttgacgaatcttggggccccg4080
cgcgcgttcaagtacttcgatactaccatagacagaaaggtctatcgctcgacaaaggaa4140
gttcttgacgccacgctgatccaccaaagtataacaggcctctatgagacacgcatcgac4200
ctttcgcagttgggcggtgaccgccccaaaaagaagaggaaagttggcgggtga4254
<210>6
<211>633
<212>dna
<213>人工序列(non)
<400>6
atggatagccttctcatgaacagaagagagtttctctatcagtttaaaaatgttcggtgg60
gcgaaggggaggagagagacatatctctgctatgttgttaagcggagagattctgcgacc120
tcattctcactcgattttggttatttgaggaacaagaatggatgtcatgtcgaattgttg180
tttctccggtatatttccgactgggatttggacccagggcggtgttaccgggtcacatgg240
tttatttcctggagtccatgttacgactgtgcgcgccatgtcgccgacttcctcaggggt300
aatcctaacttgtccttgcggatttttacagccagactctatttctgtgaggatcggaag360
gcggaacccgaggggctgagaagactgcaccgcgctggcgtccaaatcgccatcatgact420
tttaaggattatttctactgttggaacacgttcgtcgagaaccacggtcggaccttcaaa480
gcctgggaagggctgcatgaaaattccgtgaggttgtcccggcaactccgcagaatactc540
ctgcccctttatgaggtcgacgatctcagagacgcctttagaactagcggaagcgagacg600
ccagggacttctgaatcggccacccccgagagc633
<210>7
<211>273
<212>dna
<213>人工序列(non)
<400>7
tccggcggaagtacaaacctttcagacattatagaaaaggaaaccggcaagcaactcgtc60
atccaggaatccatacttatgctccctgaagaggtggaagaagtgatcggtaataaacca120
gagagcgacatacttgtccacaccgcttatgacgaaagtacagacgaaaacgtcatgctt180
ctgacgagtgatgcccccgaatacaaaccttgggcgctcgtcatccaggattccaatggg240
gagaataaaataaagatgctctctggaggcagc273
<210>8
<211>1191
<212>dna
<213>人工序列(non)
<400>8
atgtccgaagtggaatttagccatgaatattggatgcggcacgccctcacgcttgccaag60
agagcctgggatgagagggaggttcccgtcggtgccgtgttggtccataacaacagggtg120
attggggaaggatggaacagacccattgggcgccatgatccaactgcccatgcagagatt180
atggcgctcaggcaaggggggttggttatgcaaaactaccggcttattgacgcaaccctg240
tatgtcacccttgaaccctgtgttatgtgcgcgggggccatgatacactctcggataggg300
cgggtggtgttcggggctcgggatgctaagaccggagctgctggttccctcatggatgtc360
ttgcatcatcctggtatgaaccatagagtcgagattactgaaggcattctcgcagacgaa420
tgcgctgcccttctctcagatttctttagaatgcgcagacaggaaataaaggctcaaaaa480
aaagcacagagttccacggattccggcgggtcgagcggtggcagctccggctccgagaca540
cccggtacgagtgaatccgctacgcccgaatcctcggggggaagctctggaggctcatca600
gaagtcgagttctcccatgagtattggatgaggcacgccctcactcttgcgaagagggcc660
agggacgagagggaggtgccggtcggtgctgtcctggtcttgaataacagggtgataggc720
gaaggttggaacagggctattggccttcatgaccctactgctcatgcggaaatcatggca780
cttagacaggggggcctcgttatgcaaaattaccgcctgatcgacgccactctttatgtc840
acatttgaaccatgtgttatgtgtgcgggcgctatgatccattcacgcataggtcgcgtg900
gtttttggagttcgcaacgcgaaaacaggggctgcaggctctctgatggacgttttgcac960
tatccgggaatgaaccatagagtcgaaatcacagaagggattttggcagacgaatgcgcg1020
gctcttctttgttattttttcagaatgccccgccaagtgtttaatgctcaaaagaaagcg1080
cagagtagcacagactcggggggatcttctgggggctcgtctggttccgagactcccgga1140
acttccgagtcggcaacacctgaatcctccggcggctcttcgggcggatct1191
<210>9
<211>76
<212>dna
<213>人工序列(non)
<400>9
gttttagagctagaaatagcaagttaaaataaggctagtccgttatcaacttgaaaaagt60
ggcaccgagtcggtgc76
<210>10
<211>25
<212>dna
<213>人工序列(non)
<400>10
tgtgtagagaccaaaggaggtctca25
<210>11
<211>41
<212>dna
<213>人工序列(non)
<400>11
tgttggctaggatccatcgcagtcagcgatgagtacagcaa41
<210>12
<211>1765
<212>dna
<213>人工序列(non)
<400>12
gcagcgtgacccggtcgtgcccctctctagagataatgagcattgcatgtctaagttata60
aaaaattaccacatattttttttgtcacacttgtttgaagtgcagtttatctatctttat120
acatatatttaaactttactctacgaataatataatctatagtactacaataatatcagt180
gttttagagaatcatataaatgaacagttagacatggtctaaaggacaattgagtatttt240
gacaacaggactctacagttttatctttttagtgtgcatgtgttctcctttttttttgca300
aatagcttcacctatataatacttcatccattttattagtacatccatttagggtttagg360
gttaatggtttttatagactaatttttttagtacatctattttattctattttagcctct420
aaattaagaaaactaaaactctattttagtttttttatttaataatttagatataaaata480
gaataaaataaagtgactaaaaattaaacaaataccctttaagaaattaaaaaaactaag540
gaaacatttttcttgtttcgagtagataatgccagcctgttaaacgccgtcgacgagtct600
aacggacaccaaccagcgaaccagcagcgtcgcgtcgggccaagcgaagcagacggcacg660
gcatctctgtcgctgcctctggacccctctcgagagttccgctccaccgttggacttgct720
ccgctgtcggcatccagaaattgcgtggcggagcggcagacgtgagccggcacggcaggc780
ggcctcctcctcctctcacggcacggcagctacgggggattcctttcccaccgctccttc840
gctttcccttcctcgcccgccgtaataaatagacaccccctccacaccctctttccccaa900
cctcgtgttgttcggagcgcacacacacacaaccagatctcccccaaatccacccgtcgg960
cacctccgcttcaaggtacgccgctcgtcctccccccccccccctctctaccttctctag1020
atcggcgttccggtccatggttagggcccggtagttctacttctgttcatgtttgtgtta1080
gatccgtgtttgtgttagatccgtgctgctagcgttcgtacacggatgcgacctgtacgt1140
cagacacgttctgattgctaacttgccagtgtttctctttggggaatcctgggatggctc1200
tagccgttccgcagacgggatcgatttcatgattttttttgtttcgttgcatagggtttg1260
gtttgcccttttcctttatttcaatatatgccgtgcacttgtttgtcgggtcatcttttc1320
atgctttttttttgtcttggttgtgatgatgtggtgtggttgggcggtcgttcattcgtt1380
ctagatcggagtagaatactgtttcaaactacctggtgtatttattaattttggaactgt1440
atgtgtgtgtcatacatcttcatagttacgagtttaagatggatggaaatatcgatctag1500
gataggtatacatgttgatgtgggttttactgatgcatatacatgatggcatatgcagca1560
tctattcatatgctctaaccttgagtacctatctattataataaacaagtatgttttata1620
attattttgatcttgatatacttggatgatggcatatgcagcagctatatgtggattttt1680
ttagccctgccttcatacgctatttatttgcttggtactgtttcttttgtcgatgctcac1740
cctgttgtttggtgttacttctgca1765
<210>13
<211>322
<212>dna
<213>人工序列(non)
<400>13
aagaacgaactaagccggacaaaaaaaggagcacatatacaaaccggttttattcatgaa60
tggtcacgatggatgatggggctcagacttgagctacgaggccgcaggcgagagaagcct120
agtgtgctctctgcttgtttgggccgtaacggaggatacggccgacgagcgtgtactacc180
gcgcgggatgccgctgggcgctgcgggggccgttggatggggatcggtgggtcgcgggag240
cgttgaggggagacaggtttagtaccacctcgcctaccgaacaatgaagaacccacctta300
taaccccgcgcgctgccgcttg322
<210>14
<211>253
<212>dna
<213>人工序列(non)
<400>14
gatcgttcaaacatttggcaataaagtttcttaagattgaatcctgttgccggtcttgcg60
atgattatcatataatttctgttgaattacgttaagcatgtaataattaacatgtaatgc120
atgacgttatttatgagatgggtttttatgattagagtcccgcaattatacatttaatac180
gcgatagaaaacaaaatatagcgcgcaaactaggataaattatcgcgcgcggtgtcatct240
atgttactagatc253
<210>15
<211>8
<212>dna
<213>人工序列(non)
<400>15
tttttttt8
<210>16
<211>22
<212>dna
<213>人工序列(non)
<400>16
ggccttccttgggatccggcga22
<210>17
<211>23
<212>dna
<213>人工序列(non)
<400>17
gtgcactgccaagctcacactgc23
<210>18
<211>22
<212>dna
<213>人工序列(non)
<400>18
agatctagaggttggtctacgt22
<210>19
<211>20
<212>dna
<213>人工序列(non)
<400>19
agcaagtccgattgaatact20
<210>20
<211>18
<212>dna
<213>人工序列(non)
<400>20
tccggcggaagtacaaac18
<210>21
<211>19
<212>dna
<213>人工序列(non)
<400>21
attgggacaaactctgtgg19
<210>22
<211>18
<212>dna
<213>人工序列(non)
<400>22
gtcaccgcccaactgcga18
<210>23
<211>18
<212>dna
<213>人工序列(non)
<400>23
gcgcgcttggcgtaatca18
<210>24
<211>27
<212>dna
<213>人工序列(non)
<400>24
agccagaccaattgagtattttttgtc27
<210>25
<211>18
<212>dna
<213>人工序列(non)
<400>25
actagttcacccgccaac18
<210>26
<211>23
<212>dna
<213>人工序列(non)
<400>26
tgttggccttccttgggatccgg23
<210>27
<211>23
<212>dna
<213>人工序列(non)
<400>27
aaacccggatcccaaggaaggcc23
<210>28
<211>20
<212>dna
<213>人工序列(non)
<400>28
gacgtctacgcctttggtgt20
<210>29
<211>19
<212>dna
<213>人工序列(non)
<400>29
tcagctgcaaaatgcaatg19
<210>30
<211>24
<212>dna
<213>人工序列(non)
<400>30
gtgtgtgcactgccaagctcacac24
<210>31
<211>22
<212>dna
<213>人工序列(non)
<400>31
aaacgtgtgattggcagtgcac22
<210>32
<211>20
<212>dna
<213>人工序列(non)
<400>32
tccatggccttgttcctctt20
<210>33
<211>20
<212>dna
<213>人工序列(non)
<400>33
tgctgctagacacatccaca20
<210>34
<211>24
<212>dna
<213>人工序列(non)
<400>34
tgttgagatctagaggttggtcta24
<210>35
<211>24
<212>dna
<213>人工序列(non)
<400>35
aaactagaccaacctctagatctc24
<210>36
<211>20
<212>dna
<213>人工序列(non)
<400>36
cactagcacgacgcactttc20
<210>37
<211>20
<212>dna
<213>人工序列(non)
<400>37
cagaacacgcgcatcatatc20
- 还没有人留言评论。精彩留言会获得点赞!