检测沙门菌的引物组和试剂盒的制作方法
本发明属于生物检测领域,具体涉及一种检测沙门菌的引物组和试剂盒。
背景技术:
沙门菌是寄居在人类动物肠道内,生化反应和抗原构造相似的革兰阴性杆菌。沙门菌是一种肠道致病菌,常常因为误食不洁食物引起感染,感染者会出现严重腹泻,它是人类食物中毒的主要病原之一。
目前,针对沙门菌鉴定的标准方法主要是传统血清学法,传统方法操作繁琐,受主观因素影响比较大,而且一次试验流程一般只能筛查1-2种菌,若要快速检测一系列的可疑致病菌,则需多次实验,工作量偏大,检测时间长。而采用常规pcr检测该些沙门菌时,一次鉴定的种类少,这样会造成效率低、成本高。
因此,现有技术有待改进。
技术实现要素:
本发明的目的在于克服现有技术的上述不足,提供一种检测沙门菌的引物组和试剂盒,旨在解决现有沙门菌的检测步骤繁琐、成本高和效率低问题。
为实现上述发明目的,本发明采用的技术方案如下:
本发明一方面提供一种检测沙门菌的引物组,所述引物组基于荧光探针熔解曲线法同时检测30种沙门菌,所述引物组包括:
针对a群抗原基因的杂交连接引物seqidno:1和seqidno:2;
针对b群抗原基因的杂交连接引物seqidno:3和seqidno:4;
针对c1群抗原基因的杂交连接引物seqidno:5和seqidno:6;
针对c2-c3群抗原基因的杂交连接引物seqidno:7和seqidno:8;
针对d群抗原基因的杂交连接引物seqidno:9和seqidno:10;
针对e群抗原基因的杂交连接引物seqidno:11和seqidno:12;
针对a抗原基因的杂交连接引物seqidno:13和seqidno:14;
针对b抗原基因的杂交连接引物seqidno:15和seqidno:16;
针对c抗原基因的杂交连接引物seqidno:17和seqidno:18;
针对d抗原基因的杂交连接引物seqidno:19和seqidno:20;
针对e,n,x/e,n,z15抗原基因的杂交连接引物seqidno:21和seqidno:22;
针对i抗原基因的杂交连接引物seqidno:23和seqidno:24;
针对v抗原基因的杂交连接引物seqidno:25和seqidno:26;
针对g,m抗原基因的杂交连接引物seqidno:27和seqidno:28;
针对z4,z23抗原基因的杂交连接引物seqidno:29和seqidno:30;
针对e,h抗原基因的杂交连接引物seqidno:31和seqidno:32;
针对f,g抗原基因的杂交连接引物seqidno:33和seqidno:34;
针对f,g,s/g,s,t抗原基因的杂交连接引物seqidno:35和seqidno:36;
针对r抗原基因的杂交连接引物seqidno:37和seqidno:38;
针对1,2抗原基因的杂交连接引物seqidno:39和seqidno:40;
针对1,5抗原基因的杂交连接引物seqidno:41和seqidno:42;
针对1,6抗原基因的杂交连接引物seqidno:43和seqidno:44;
针对1,7抗原基因的杂交连接引物seqidno:45和seqidno:46;
针对z6抗原基因的杂交连接引物seqidno:47和seqidno:48。
本发明另一方面提供一种检测沙门菌的试剂盒,所述试剂盒包括本发明的上述引物组。
本发明提供的检测沙门菌的引物组,以多重连接探针扩增技术的原理为基础,根据目前常见30种沙门菌血的清型o抗原和h抗原进行设计得到,这样,使得该引物组能够基于荧光探针熔解曲线法同时检测30种沙门菌,从而有效缩短了沙门菌的检测周期,提高了沙门菌的检测效率。
本发明试剂盒因含有本发明特有的引物组,所以利用该试剂盒可以在多重连接探针扩增技术的基础上采用实时荧光pcr熔解曲线法实现快速、简便和高通量的对30种沙门菌同时检测,从而有效缩短了沙门菌的检测周期,提高了沙门菌的检测效率,灵敏度高,成本低,可以用来代替操作繁琐、受主观影响较大的传统血清学法。
附图说明
图1为本发明实施例2的荧光探针熔解曲线法同时检测30种沙门菌的第一管rox荧光通道图;
图2为本发明实施例2的荧光探针熔解曲线法同时检测30种沙门菌的第一管fam荧光通道图;
图3为本发明实施例2的荧光探针熔解曲线法同时检测30种沙门菌的第一管cy5荧光通道图;
图4为本发明实施例2的荧光探针熔解曲线法同时检测30种沙门菌的第二管rox荧光通道图;
图5为本发明实施例2的荧光探针熔解曲线法同时检测30种沙门菌的第二管fam荧光通道图;
图6为本发明实施例2的荧光探针熔解曲线法同时检测30种沙门菌的第二管cy5荧光通道图;
图7为本发明的24对杂交连接引物中上游杂交连接引物和下游杂交连接引物的序列结构示意图。
具体实施方式
为了使本发明要解决的技术问题、技术方案及有益效果更加清楚明白,以下结合实施例,对本发明进行进一步详细说明。应当理解,此处所描述的具体实施例仅仅用以解释本发明,并不用于限定本发明。
一方面,本发明实施例提供了一种检测沙门菌的引物组,所述引物组基于荧光探针熔解曲线法同时检测30种沙门菌,所述引物组包括:
针对a群抗原基因的杂交连接引物seqidno:1和seqidno:2;
针对b群抗原基因的杂交连接引物seqidno:3和seqidno:4;
针对c1群抗原基因的杂交连接引物seqidno:5和seqidno:6;
针对c2-c3群抗原基因的杂交连接引物seqidno:7和seqidno:8;
针对d群抗原基因的杂交连接引物seqidno:9和seqidno:10;
针对e群抗原基因的杂交连接引物seqidno:11和seqidno:12;
针对a抗原基因的杂交连接引物seqidno:13和seqidno:14;
针对b抗原基因的杂交连接引物seqidno:15和seqidno:16;
针对c抗原基因的杂交连接引物seqidno:17和seqidno:18;
针对d抗原基因的杂交连接引物seqidno:19和seqidno:20;
针对e,n,x/e,n,z15抗原基因的杂交连接引物seqidno:21和seqidno:22;
针对i抗原基因的杂交连接引物seqidno:23和seqidno:24;
针对v抗原基因的杂交连接引物seqidno:25和seqidno:26;
针对g,m抗原基因的杂交连接引物seqidno:27和seqidno:28;
针对z4,z23抗原基因的杂交连接引物seqidno:29和seqidno:30;
针对e,h抗原基因的杂交连接引物seqidno:31和seqidno:32;
针对f,g抗原基因的杂交连接引物seqidno:33和seqidno:34;
针对f,g,s/g,s,t抗原基因的杂交连接引物seqidno:35和seqidno:36;
针对r抗原基因的杂交连接引物seqidno:37和seqidno:38;
针对1,2抗原基因的杂交连接引物seqidno:39和seqidno:40;
针对1,5抗原基因的杂交连接引物seqidno:41和seqidno:42;
针对1,6抗原基因的杂交连接引物seqidno:43和seqidno:44;
针对1,7抗原基因的杂交连接引物seqidno:45和seqidno:46;
针对z6抗原基因的杂交连接引物seqidno:47和seqidno:48。
本发明实施例提供的检测沙门菌的引物组,以多重连接探针扩增技术的原理为基础,根据目前常见30种沙门菌血的清型o抗原和h抗原进行设计得到,这样,使得该引物组能够基于荧光探针熔解曲线法同时检测30种沙门菌,从而有效缩短了沙门菌的检测周期,提高了沙门菌的检测效率。
通过调研,发现沙门菌血清型抗原基因wzx或wzy是鉴定不同的o抗原的特异基因;fljc和flib基因分别是h1和h2抗原特异基因。通过国内外文献调研发现沙门菌血清型抗原分子生物学机制:o抗原是由菌体最外层类脂-多糖-多肽复合物决定,导致o抗原特异性的核心生物学基础是多糖的寡糖单位,而wzx和wzy两个基因分别特异合成并识别转移寡糖单位,因此,wzx和wzy基因成为检测o抗原的基础。h抗原特异性是由鞭毛蛋白内氨基酸序列和空间结构决定,而fljc和flib基因分别合成h1和h2抗原。
表1为常见30种沙门菌血清型列单。本发明实施例为了鉴定分型该30种沙门菌学清型,在表1中的沙门菌血清型分型所需的24个抗原基因基础上,设计了包括seqidno:1-seqidno:48所示的24对杂交连接引物,表2为对应的血清型分子鉴定检测体系的杂交连接引物序列。
该24对杂交连接引物序列选择上,首先需要在ncbi上查找沙门菌血清型抗原基因并下载相关序列,使用软件mega7.0建立沙门菌血清型抗原基因数据库。通过比对得到每个抗原特异基因序列,并将所有特异序列导入ncbi检验特异性,确保不会和其他细菌基因组产生交叉。针对24种抗原的每种抗原基因,分别设计多对杂交连接引物,并筛选出峰值较高,不会产生非特异峰的引物。而该设计过程的难点在于,本发明实施例需要将24对杂交连接引物合理有效地分配到两管并混合,既要保证这些杂交连接引物之间不会或者非常低的干扰,还要能够识别出对应的基因,并且该过程需要在单对杂交连接引物设计有效的基础上不断添加新设计有效的杂交连接引物对,直到所有的杂交连接引物都能够特异性识别对应的抗原基因。本发明实施例中,通过试验不断探索得到这24对杂交连接引物,其可以使用两管就能够识别24个基因的荧光pcr技术,检测效率和灵敏度高,成本低。
该24对杂交连接引物(包括上游杂交连接引物和下游杂交连接引物)序列结构和反应原理为:上游杂交连接引物结构分为三部分,从5’端开始部分是通用引物(通用上游引物)结合序列,用于扩增整个引物,中间部分为人工熔点标签序列,用于对应的荧光探针杂交并产生对应的tm值,第三部分就是与待检测基因杂交的上游杂交序列;下游杂交连接引物结构分为两部分:从5’端开始的部分为与待检测基因杂交的下游杂交序列,此序列与上游杂交连接引物序列最终通过连接酶连接在一起,形成与待检测基因完全互补的序列片段,接下来是通用引物(通用下游引物)结合序列,用于扩增整个引物。反应原理:对杂交连接产物的富集和熔解曲线分析,通过第一步的连接反应得到杂交连接产物,通用引物会对此产物进行扩增,而荧光探针最后会与连接产物的扩增产物杂交,发出荧光并得到对应的tm值。具体引物结构如图7所示。
表1
表2
另一方面,本发明实施例还提供了一种检测沙门菌的试剂盒,所述试剂盒包括本发明实施例的上述引物组。
本发明实施例的试剂盒因含有本发明实施例特有的引物组,所以利用该试剂盒可以在多重连接探针扩增技术的基础上采用实时荧光pcr熔解曲线法实现快速、简便和高通量的对30种沙门菌同时检测,从而有效缩短了沙门菌的检测周期,提高了沙门菌的检测效率,灵敏度高,成本低,可以用来代替操作繁琐、受主观影响较大的传统血清学法。
进一步地,本发明一实施例中,所述试剂盒还包括连接酶和缓冲液,所述连接酶、连接酶缓冲液与所述引物组用于杂交连接反应体系。优选地,所述杂交连接反应系统的条件:95℃预变性5min,60℃连接80min,95℃变性5min。
进一步地,本发明一实施例中,所述试剂盒还包括荧光探针、pcr缓冲液、mgcl2、dntp和rtaq酶,所述荧光探针、pcr缓冲液、mgcl2、dntp和rtaq酶用于荧光探针溶解曲线检测体系。优选地,所述荧光探针包括rox探针、fam探针和cy5探针。具体地,所述rox探针的序列如seqidno:49所示;所述fam探针的序列如seqidno:50所示;所述cy5探针的序列如seqidno:51所示。
优选地,所述荧光探针溶解曲线检测体系中的pcr扩增条件为:95℃预变性3min;然后经过如下40个循环:95℃变性10s,57℃退火20s,同时采集rox、fam和cy5的荧光信号,72℃延伸20s。更优选地,所述荧光探针溶解曲线检测体系中的溶解曲线分析条件为:95℃变性1min,40℃杂交2min,从40℃-85℃逐步升温,采集rox、fam和cy5的荧光信号。为了更好地得到荧光信号,从40℃-85℃升温的过程中,每上升0.5℃采集一次荧光信号。
本发明先后进行过多次试验,现举一部分试验结果作为参考对发明进行进一步详细描述,下面结合具体实施例进行详细说明。
实施例1引物组设计
根据表1中的30种沙门菌血清型分子设计表2中的检测体系引物组。
实施例230种沙门菌的检测
表3为对应的30种沙门菌血清型分子鉴定检测体系,以常见30种沙门菌血清型dna提取液作为阳性样本,进行如下步骤:
表3
(1)普通pcr连接反应。
连接反应体系总体积为10ul,包括:连接酶(1u/μl)1μl、连接酶缓冲液1μl、表2中的杂交连接引物f/rmix(10nm)1.5μl、模板dna5μl,ddh2o1.5μl。
反应程序如下:
95℃预变性5min;75℃pause取出开盖,加杂交连接反应液。
60℃连接80min;95℃变性5min;4℃pause。
(2)荧光pcr扩增和熔解曲线分析
将上一步连接反应产物5ul加到荧光pcr反应体系中,上机运行(bio-radcfx96)。反应体系为50μl,包括:10×buffer5μl、dntp(2.5mm)4μl、mg2+(1.5mm)3ml、通用上游引物(2.5μm)0.3μl、通用下游引物(50μm)0.3μl、roxprobe(50μm)0.2ul、famprobe(50μm)0.2μl、cy5probe(50μm)0.2μl、rtaq(5u/μl)0.2μl、ddw31.4μl,杂交连接产物5ul。荧光探针序列如下表4:
表4
荧光pcr反应程序如下:
信号(即从40℃-85℃逐步升温,每上升0.5℃采集一次荧光信号),进行荧光熔解曲线分析。
(4)试验结果
表5为30种沙门菌血清型检测结果判读表,对结果判读说明:例如1-rox-70.5-o:4(b)群:1代表第一管,rox代表rox荧光通道,70.5代表熔解峰温度,o:4(b)群代表血清学上的o:4(b)菌群。如实验结果:1-rox-70.5-o:4(b)群、2-rox-59-h1-i、1-cy5-55.5-h2-1,2即代表检测到沙门菌抗原o:4(b)群、i、1,2,在沙门菌血清学上是鼠伤寒沙门菌。其中,沙门菌抗原e,n,x和e,n,z15,抗原f,g,s和f,g,t分别合并在一起检测,并不影响30种沙门菌血清型的鉴定。通过ncbi数据库调取,没有发现彻那利沙门菌h2抗原z35基因序列信息,使用其他含有z35抗原的沙门菌血清型序列信息设计引物也未能有效检出彻那利沙门菌z35抗原。在常见30种沙门菌血清型列单中,彻那利沙门菌与斯坦利沙门菌的o抗原和h1抗原相同,h2抗原不同,可通过是否能检出h2抗原来判断二者血清型。
表5
本实施例的检测体系最低检出限为1.04-1.56ng/ul,如表6所示,即本体系检测能力级别为1ng/ul级别。
表6
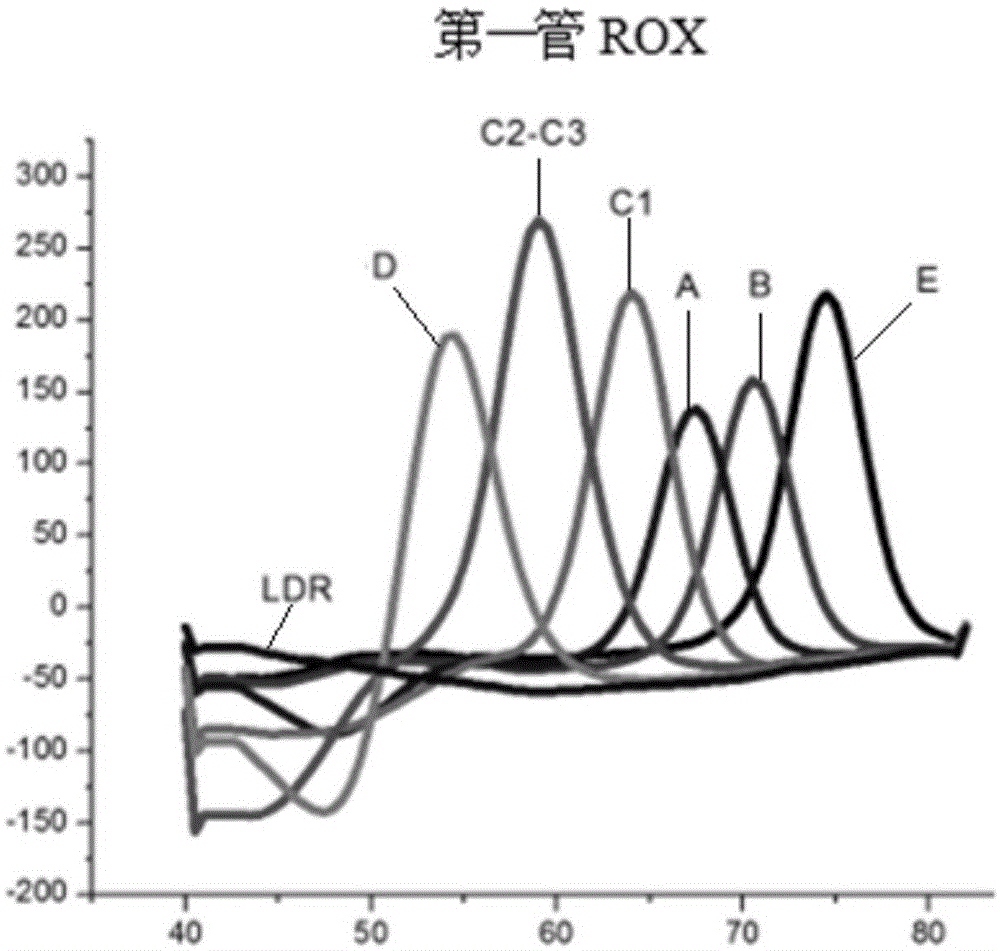
本实施例中,使用rox、fam、cy5三个荧光通道,建立两管24重鉴定体系。实验结果如图1-图6所示。
图1、图2、图3分别为第一管rox、fam、cy5三个荧光通道分别检测的抗原基因:按tm值从高到低,rox通道检测沙门菌血清型o抗菌群e群、b群、a群、c1群、c2-c3群、d群六个抗原基因;fam通道检测沙门菌h1抗原d、b、a、c四个抗原基因;cy5通道检测沙门菌血清型h2抗原1,7、1,2、z6三个抗原基因。图4、图5、图6分别为第二管rox、fam、cy5三个荧光通道分别检测的抗原基因:按tm值从高到低,rox通道检测沙门菌血清型h抗原e,n,x/e,n,z15、r、i、v、g,m五个抗原基因;fam通道检测沙门菌血清型h1抗原z4,z23、e,h、f,g、f,g,s/f,g,t四个抗原基因;cy5通道检测沙门菌血清型h2抗原1,5、1,6两个抗原基因。
实施例3方法学评价
使用本实施例2的检测体系对2017年深圳市疾病控制中心菌种库431株临床沙门菌菌株进行分子鉴定,与传统金标准方法“血清学”方法做比较。所有菌株经过水煮法提取核酸,上机进行检测,将得到的结果与菌种库中血清型信息做对比。
结果显示:各个沙门菌血清型一致性良好。以血清学方法为参考,本实施例的多重连接探针熔解曲线法的灵敏度为99.5%,特异度100%,kappa值0.89,结果见表7。其中,2株鼠伤寒沙门菌和9株未分型沙门菌未能检出。目前,部分鼠伤寒沙门菌存在h2相不凝集的现象,可能存在基因变异的情况,因此本实施例未能检出。而9株未分型沙门菌抗原,其对应的抗原可能超出本实施例抗原检测范围。
表7
注:多重连接探针熔解曲线灵敏度:阳性符合率=a/(a+b)×100%=99.5%
多重连接探针熔解曲线特异度:阴性符合率=d/(c+d)×100%=100%
多重连接探针熔解曲线kappa=(po-pe)/(1-pe)=0.8
以上所述仅为本发明的较佳实施例而已,并不用以限制本发明,凡在本发明的精神和原则之内所作的任何修改、等同替换和改进等,均应包含在本发明的保护范围之内。
序列表
<110>深圳市疾病预防控制中心
<120>检测沙门菌的引物组和试剂盒
<160>51
<170>siposequencelisting1.0
<210>1
<211>80
<212>dna
<213>人工序列(artificialsequence)
<400>1
gtggcagggcgctacgaacaatcctaacgactctgtcttctcgttcgtgacgttatctgg60
cattgttggttgtgctgcaa80
<210>2
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>2
ccgaacctggcaacataccattagatgtgagattggatcttgctgggc48
<210>3
<211>89
<212>dna
<213>人工序列(artificialsequence)
<400>3
gtggcagggcgctacgaacaatcctaacgactctagctgctcgttcgtgacggcaaatgt60
tataaaaccattaaagcttcttgatttgg89
<210>4
<211>57
<212>dna
<213>人工序列(artificialsequence)
<400>4
caataaaatatcgggcggatatctttttaaatacagtgagattggatcttgctgggc57
<210>5
<211>84
<212>dna
<213>人工序列(artificialsequence)
<400>5
gtggcagggcgctacgaacaatcctaacgactctagctgcttgttcgtgacgtattactt60
gtgcttggtgccattctatcattg84
<210>6
<211>58
<212>dna
<213>人工序列(artificialsequence)
<400>6
cctttgtcacattatttatcagatataatttcttccgtgagattggatcttgctgggc58
<210>7
<211>85
<212>dna
<213>人工序列(artificialsequence)
<400>7
gtggcagggcgctacgaacaatcctaactactctcgctgctcgttcgggacgggaacatc60
cagacgtttgttttataaatttacg85
<210>8
<211>55
<212>dna
<213>人工序列(artificialsequence)
<400>8
tttagaacatgtttacggtgagagggataaagcagtgagattggatcttgctggg55
<210>9
<211>91
<212>dna
<213>人工序列(artificialsequence)
<400>9
gtggcagggcgctacgaacaatcctaactactcttgctgctagttcgtgtcgcgagttta60
tatgcatatactaaacaaaaagcaaatgaac91
<210>10
<211>49
<212>dna
<213>人工序列(artificialsequence)
<400>10
tcgccgccgccattatagataaagtttgtgagattggatcttgctgggc49
<210>11
<211>86
<212>dna
<213>人工序列(artificialsequence)
<400>11
gtggcagggcgctacgaacaatcctaacgactctggctgctcgttcgtgacgggtgctga60
tttaaccgggtattatttattagtag86
<210>12
<211>57
<212>dna
<213>人工序列(artificialsequence)
<400>12
tacagttgatgggagtattaaatcttgctgagctagtgagattggatcttgctgggc57
<210>13
<211>83
<212>dna
<213>人工序列(artificialsequence)
<400>13
gtggcagggcgctacgaacaatcctatccgttctttatcgctcagccttcatcggtcacg60
ccttcggctacattaagcactac83
<210>14
<211>46
<212>dna
<213>人工序列(artificialsequence)
<400>14
tgcacttgatggtgctggcctcaaatgagattggatcttgctgggc46
<210>15
<211>85
<212>dna
<213>人工序列(artificialsequence)
<400>15
gtggcagggcgctacgaacaatcctatcggtccttcatggctcagtcttcaccggaagtg60
tacaggatgcctatacgccaaaagg85
<210>16
<211>53
<212>dna
<213>人工序列(artificialsequence)
<400>16
taccgctgttaccagagatgttaccacctatatgagattggatcttgctgggc53
<210>17
<211>85
<212>dna
<213>人工序列(artificialsequence)
<400>17
gtggcagggcgctacgaacaatcctatcggtccttcatggctcagtcttcaccggatgcc60
tatacgccaaaaggtaccgctgtta85
<210>18
<211>52
<212>dna
<213>人工序列(artificialsequence)
<400>18
ccagagatgttaccacctataaaaatggtggtgagattggatcttgctgggc52
<210>19
<211>88
<212>dna
<213>人工序列(artificialsequence)
<400>19
gtggcagggcgctacgaacaatcctatcggtccttcatcgctcagccttcaccgggccac60
ttatgatgaaactacaaagaaagttaat88
<210>20
<211>50
<212>dna
<213>人工序列(artificialsequence)
<400>20
attgatacgactgataaaactccgttggctgagattggatcttgctgggc50
<210>21
<211>85
<212>dna
<213>人工序列(artificialsequence)
<400>21
gtggcagggcgctacgaacaatcctaacgactctggctgctcgttcgtgacgtaaggatg60
gtaaatattatgccactgtaggtgg85
<210>22
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>22
ttataccgatgcgggggatactgccaatgagattggatcttgctgggc48
<210>23
<211>85
<212>dna
<213>人工序列(artificialsequence)
<400>23
gtggcagggcgctacgaacaatcctaactactctcgctgctcgttcgggacgctggtaaa60
gatggctattatgaagtttccgttg85
<210>24
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>24
ataagacgaacggtgaggtgactcttgtgagattggatcttgctgggc48
<210>25
<211>83
<212>dna
<213>人工序列(artificialsequence)
<400>25
gtggcagggcgctacgaacaatcctaacgactctagcttctcgttagtgacgaagttagt60
gttgcagatgatggcactgttac83
<210>26
<211>47
<212>dna
<213>人工序列(artificialsequence)
<400>26
aatgccgacaaccacgaaagtgacagtgagattggatcttgctgggc47
<210>27
<211>80
<212>dna
<213>人工序列(artificialsequence)
<400>27
gtggcagggcgctacgaacaatcctaactactcttgctgctagttcgtgtcgttaactgt60
cgctgatattgccactggcg80
<210>28
<211>49
<212>dna
<213>人工序列(artificialsequence)
<400>28
cgacggatgttaatgctgctaccttacatgagattggatcttgctgggc49
<210>29
<211>86
<212>dna
<213>人工序列(artificialsequence)
<400>29
gtggcagggcgctacgaacaatcctatcggtccttcatcgctcagccttcaccggggtag60
ttggtgatgtaaaaattgcggcagct86
<210>30
<211>49
<212>dna
<213>人工序列(artificialsequence)
<400>30
gatttcgataacgcaaaaacaactggtgtgagattggatcttgctgggc49
<210>31
<211>81
<212>dna
<213>人工序列(artificialsequence)
<400>31
gtggcagggcgctacgaacaatcctatcggtgcttcatagctcagccttcaccggaatcc60
cacggcaacaggcgattcctt81
<210>32
<211>55
<212>dna
<213>人工序列(artificialsequence)
<400>32
gtctgctacgctttcgtttaaagatggtaagtattgagattggatcttgctgggc55
<210>33
<211>83
<212>dna
<213>人工序列(artificialsequence)
<400>33
gtggcagggcgctacgaacaatcctatccgttctttatcgctcagccttcatcggtgacg60
atgcggaaaataacactgcggtt83
<210>34
<211>53
<212>dna
<213>人工序列(artificialsequence)
<400>34
gacctctttaagaccactaaatctactgctggtgagattggatcttgctgggc53
<210>35
<211>89
<212>dna
<213>人工序列(artificialsequence)
<400>35
gtggcagggcgctacgaacaatcctatcgctccttcatagctcagacttcatcgggatat60
taattcaggtgctgtagtaactgatgatg89
<210>36
<211>54
<212>dna
<213>人工序列(artificialsequence)
<400>36
cagcaccggataaagtatatgtaaatgcagcaatgagattggatcttgctgggc54
<210>37
<211>81
<212>dna
<213>人工序列(artificialsequence)
<400>37
gtggcagggcgctacgaacaatcctaacgactctagctgcttgttcgtgacggatggaaa60
agtcactttaactggcacacc81
<210>38
<211>47
<212>dna
<213>人工序列(artificialsequence)
<400>38
aacaggaccaattactgctggcttcctgagattggatcttgctgggc47
<210>39
<211>83
<212>dna
<213>人工序列(artificialsequence)
<400>39
gtggcagggcgctacgaacaatcctacggtgacgcccttggaaggttgtataccatgatg60
cagctattaaagcggctacgggt83
<210>40
<211>47
<212>dna
<213>人工序列(artificialsequence)
<400>40
ggtacgaatggtacggcttctgtaactgagattggatcttgctgggc47
<210>41
<211>78
<212>dna
<213>人工序列(artificialsequence)
<400>41
gtggcagggcgctacgaacaatcctacggtgacgcccttggaaggttgtatacccggcya60
ydggtggtacgactggta78
<210>42
<211>52
<212>dna
<213>人工序列(artificialsequence)
<400>42
cggctkctgtaacsggtrcrgttaaatttgatgagattggatcttgctgggc52
<210>43
<211>82
<212>dna
<213>人工序列(artificialsequence)
<400>43
gtggcagggcgctacgaacaatcctacggtaaggccattggcacgttgtataccaaagcg60
gctataggtggtacgcttggca82
<210>44
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>44
cggcttctgtaaccggtggtacagtgagattggatcttgctgggc45
<210>45
<211>77
<212>dna
<213>人工序列(artificialsequence)
<400>45
gtggcagggcgctacgaacaatcctacggtgaggccattggcaggttgtatacccggcta60
cgggtggtacgaatggt77
<210>46
<211>54
<212>dna
<213>人工序列(artificialsequence)
<400>46
gcacctagtgtaacaggtagtgcggttaaattttgagattggatcttgctgggc54
<210>47
<211>81
<212>dna
<213>人工序列(artificialsequence)
<400>47
gtggcagggcgctacgaacaatcctacggtaaggccattggcacgttgtataccttaaat60
ctggcgggattacagacccag81
<210>48
<211>49
<212>dna
<213>人工序列(artificialsequence)
<400>48
aaattgctgctgcccaggttgtcaaaattgagattggatcttgctgggc49
<210>49
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>49
acgactctggctgctcgttcgtgacg26
<210>50
<211>29
<212>dna
<213>人工序列(artificialsequence)
<400>50
tcggtccttcatcgctcagccttcaccgg29
<210>51
<211>31
<212>dna
<213>人工序列(artificialsequence)
<400>51
cggtgaggcccttggcaggttggtatcaccc31
- 还没有人留言评论。精彩留言会获得点赞!