赖氨酸环化脱氨酶及其应用的制作方法
本发明属于基因工程技术领域,具体涉及一种赖氨酸环化脱氨酶及其应用。
背景技术:
l-哌啶酸:中文名称为l-2-哌啶酸,英文名称为l-pipecolicacid,分子式是c6h11no2,分子量为129.16,熔点272℃,比旋光度-27.5°(c=5,h2o24℃)。其是亚氨基酸的一种,在大多数植物中以游离态存在,特别是在豆科植物中发现有较高的浓度。在从赖氨酸向α-氨基己二酸(adipicacid)的分解过程中,l-哌啶酸是重要的中间代谢产物。作为一种重要的刚性环状非蛋白质氨基酸,哌啶甲酸既可以限定多肽的构象,还可以作为不同化合物合成库中的多功能骨架,所以广泛应用于手性药物和生物活性物质的制备。如新一代局麻药罗哌卡因、抗精神病药物硫利达嗪、免疫抑制剂雷帕霉素以及抗肿瘤抗生素等均是以哌啶酸或其衍生物为主要材料制得的。由于哌啶酸及其衍生物具有的生物活性和广泛的应用前景,其合成已引起众多国外研究者的兴趣,去开发合成的新方法和新技术。
目前,l-哌啶酸合成方法主要分为生物化学催化、化学不对称合成和光催化合成。由于l-哌啶甲酸是手性分子,因此合成较困难。近年来发现的赖氨酸环化脱氨酶,可以一步将l-赖氨酸直接脱氨环化转化为l-哌啶甲酸,这一条路径相较现有的哌啶甲酸生产路径,具有更好的催化效率,更少的副产物产出,更小的成本投入,更绿色的工艺过程。目前现有的利用含赖氨酸环化脱氨酶催化效率较低,受到严重的底物与产物抑制,无法为工业化提供有效的基础。
技术实现要素:
本发明的目的在于提供一种新型的赖氨酸环化脱氨酶,以解决现有技术中赖氨酸环化脱氨酶受到底物和产物抑制的问题,提高赖氨酸环化脱氨酶的催化活性。
为解决上述技术问题,本发明采用如下技术方案:
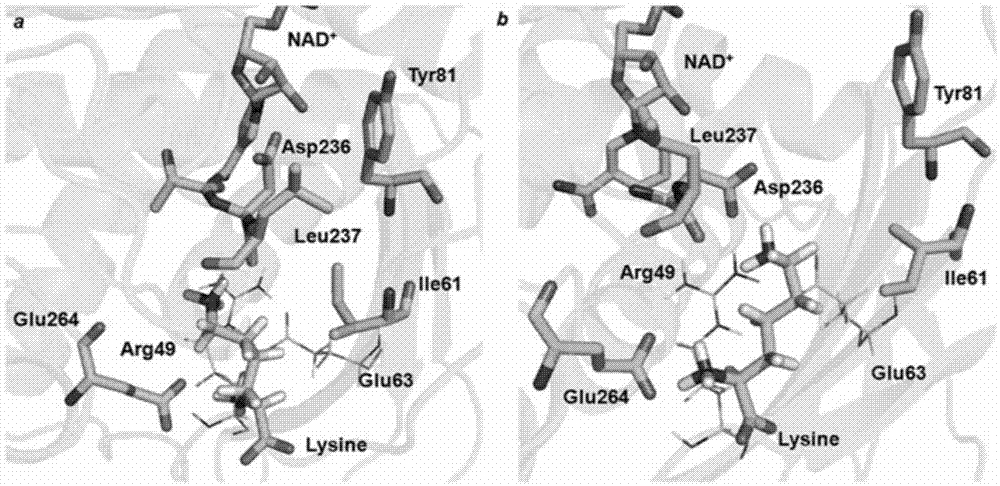
赖氨酸环化脱氨酶,其氨基酸序列如seqidno.1~3任一所示。通过对来源于s.pristinaespiralis(atcc25486)赖氨酸环化脱氨酶基因(splcd)的序列(seqidno.7)进行分析,找到了底物结合与产物释放过程中的瓶颈氨基酸位点ile61及ile94,从图1b中可以看出,ile61与asp236之间的距离为整个通道中最窄的,成为了限制底物小分子赖氨酸进入活性中心的瓶颈,从图2a中可以看出,ile61与nad+之间的距离造成了产物释放位点1较大的空间位阻,成为限制产物小分子l-哌啶甲酸离开活性中心的瓶颈。从图2b中可以看出,ile94与nad+之间的距离造成了产物释放位点2较大的空间位阻,成为限制产物小分子l-哌啶甲酸离开活性中心的瓶颈。
对ile61及ile94位点进行突变,将第61位ile突变为val,得到氨基酸序列如seqidno1所示,核苷酸序列如seqidno.4所示。第94位ile突变为val,得到氨基酸序列如seqidno.2所示,核苷酸序列如seqi,将ile61及ile94位点联合突变,得到的氨基酸seqidno.5所示序列如seqidno.3所示,核苷酸序列如seqidno.6所示。
包含seqidno.4~6任一所示的核苷酸序列的重组质粒在本发明的保护范围之内。
包含seqidno.4~6任一所示的核苷酸序列的重组细胞在本发明的保护范围之内。
作为优选,将了seqidno.4~6任一所示的核苷酸序列克隆到表达质粒上,得到重组质粒,将重组质粒转化宿主菌。
其中,所述的表达质粒为pet28a,所述宿主菌为大肠杆菌bl21。
上述赖氨酸环化脱氨酶在制备l-哌啶甲酸中的应用在本发明的保护范围之内。
上述重组细胞在制备l-哌啶甲酸中的应用在本发明的保护范围之内。
有益效果:
(1)通过对赖氨酸环化脱氨酶结构与功能的分析,找到了底物结合与产物释放过程中的瓶颈,通过突变筛选,使得改造后的赖氨酸环化脱氨酶的催化转化效率提高且底物与产物抑制效应减弱,提供了后续产业化过程的基础。
(2)利用基因工程技术获得的含赖氨酸环化脱氨酶突变株的高产重组菌株,得到了适合产业化的廉价易得的高活性生物催化剂,以休止细胞法高效催化赖氨酸的脱氨环化,哌啶甲酸的合成过程效率高。
(3)与目前广泛应用的化学法合成哌啶甲酸的方法相比,本方法不需要添加贵金属催化剂,不需要污染较大的有机溶剂,反应过程温和绿色,生产工艺简单可控。
(4)与目前应用的生物化学联合的合成哌啶甲酸的方法相比,本方法的底物成本低廉,且基本不产生任何副产物,主产物哌啶甲酸的ee值较高,可达到99%以上。
(5)与其他酶法合成哌啶甲酸的方法相比,利用基因工程技术获得的高活性赖氨酸环化脱氨酶休止细胞作为催化剂,不需要通过复杂的蛋白纯化步骤分离出纯酶,可直接使用细胞进行催化生产,进一步缩短了工艺步骤,降低了生产成本。
附图说明
图1为splcd中的底物结合转运过程ile61处于瓶颈位置,其中,a为底物转运通道1,b位底物转运通道2。
图2为splcd中的产物释放转运过程ile61与ile94处于瓶颈位置,其中,a位产物释放位点1,b为产物释放位点2。
具体实施方式:
下面结合实施例对本发明做进一步描述:
发明实施例中的赖氨酸环化脱氨酶基因的基因序列是由申请人通过ncbi数据库经查找得到,然后采用常规的重叠pcr技术对其进行合成获得,其氨基酸序列如seqidno:1所示。
实施例1:表达赖氨酸环化脱氨酶的重组质粒的构建。
克隆来自s.pristinaespiralis(atcc25486)赖氨酸环化脱氨酶基因(splcd)(基因由思普金公司合成,其氨基酸序列如seqidno.3所示,其核苷酸序列如seqidno.4所示。)两端加上xhoi和ncoi酶切位点,通过双酶切、连接,基因片段插入表达载体pet28a相应的酶切位点中,置于t7启动子的控制之下,获得了重组质粒pet28a-splcd。
实施例2:赖氨酸环化脱氨酶ile61的改造。
通过对splcd结构的分析,找到了底物结合与产物释放过程中的瓶颈氨基酸位点ile61。从图1b中可以看出,ile61与asp236之间的距离为整个通道中最窄的,成为了限制底物小分子赖氨酸进入活性中心的瓶颈。从图2a可以看出,ile61与nad+之间的距离造成了产物释放位点1较大的空间位阻,成为限制产物小分子l-哌啶甲酸离开活性中心的瓶颈。过全质粒pcr、饱和突变筛选,获得了最优突变株val61-splcd与重组质粒pet28a-val61-splcd。
实施例3:赖氨酸环化脱氨酶ile94的改造。
通过对splcd结构的分析,找到了底物结合与产物释放过程中的瓶颈氨基酸位点ile94。从图2b中可以看出,ile94与nad+之间的距离造成了产物释放位点2较大的空间位阻,成为限制产物小分子l-哌啶甲酸离开活性中心的瓶颈。通过全质粒pcr、饱和突变筛选,获得了最优突变株val94-splcd与重组质粒pet28a-val94-splcd。
实施例4:赖氨酸环化脱氨酶ile61与ile94的改造。
通过对splcd结构的分析,找到了底物结合与产物释放过程中的瓶颈氨基酸位点ile94与ile61,通过全质粒pcr、饱和突变筛选,获得了最优组合突变株val61-val94-splcd与重组质粒pet28a-val61-val94-splcd(在pet28a质粒中克隆seqidno.2所示的核苷酸序列,其对应的氨基酸序列如seqidno.1所示)。
实施例5:表达含赖氨酸环化脱氨酶突变株的重组基因工程菌株的构建。
将pet28a-val61-splcd,pet28a-val94-splcd与pet28a-val61-val94-splcd通过cacl2化学转化法转化到e.colibl21(de3)中,即得到表达了改造后赖氨酸环化脱氨酶的基因工程重组大肠杆菌e.colibl21(de3)-pet28a-val61-splcd,e.colibl21(de3)-pet28a-val94-splcd与e.colibl21(de3)-pet28a-val61-val94-splcd。
实施例6:重组大肠杆菌高密度发酵获取作为催化剂的休止细胞。
分别将重组大肠杆菌e.colibl21(de3)-pet28a-val61-splcd,e.colibl21(de3)-pet28a-val94-splcd与e.colibl21(de3)-pet28a-val61-val94-splcd接种至含100μg/ml卡那霉素的lb液体种子培养基中。
培养基成分具体为:蛋白胨10g/l;j、酵母粉5g/l、nacl10g/l在37℃、200rpm条件下培养过夜后,按1%(ml/ml)接种量接种至高密度发酵液体培养基中(含同等浓度卡那霉素)培养基成分具体为:葡萄糖30g/l、酵母粉20g/l、k2hpo48.7g/l;nah2po44.2g/l、(nh4)2so45.5g/l;mgso42.5g/l、微量营养液1.6g/l(cacl2·2h2o10g/l、znso4·7h2o0.50g/l、cucl2·2h2o0.25g/l、mnso4·h2o2.5g/l、cocl2·6h2o1.75g/l、h3bo30.125g/l、alcl3·6h2o2.5g/l、na2moo4·2h2o0.5g/l、feso4·7h2o10g/l,37℃、200rpm培养至od600为10时(约5h),加入诱导剂0.1mmiptg,25℃诱导表达16h后,菌液于8000rpm,4℃离心5min,弃上清,沉淀用中性缓冲溶液洗涤2-3次即为休止细胞,-20℃冰柜冻存备用。
实施例7:赖氨酸环化脱氨酶及其突变株的酶学性质对比。
将含赖氨酸环化脱氨酶突变株的基因工程重组大肠杆菌e.colibl21(de3)-pet28a-val61-splcd,e.colibl21(de3)-pet28a-val94-splcd与e.colibl21(de3)-pet28a-val61-val94-splcd休止细胞催化剂在37℃,1g/l赖氨酸及100mmhepes缓冲溶液(ph=7.2)的条件下进行了酶活力与表观动力学检测,结果如表1,相比野生型(wt-lcd,其他条件相同,但是赖氨酸环化脱氨酶未突变的大肠杆菌重组菌),所有的突变株的酶活力与催化转化数均有明显的提高,其中e.colibl21(de3)-pet28a-val61-val94-splcd的酶学表现最好,酶活力相比野生型提高了约1.84倍,催化转化数相比野生型提高了约9倍。
表1.赖氨酸环化脱氨酶野生型及其突变株的酶学性质对比
实施例8:25℃水相体系中1g/l赖氨酸为底物催化合成哌啶甲酸。
在反应体系中加入适量含赖氨酸环化脱氨酶突变株的重组大肠杆菌e.colibl21(de3)-pet28a-val61-val94-splcd休止细胞作为催化剂(细胞浓度5gdcw/l),随后在体系中依次加入1g/l赖氨酸及100mmhepes缓冲溶液(ph=7.2)至终体积为10ml,混合均匀后置于25℃加热搅拌器中,200rpm,搅拌反应4h。反应结束后,取1ml反应液,12000rpm离心2min,取上清液过滤膜后用高效液相色谱检测底物消耗与产物生成,得到哌啶甲酸浓度0.33g/l,摩尔转化率为37.5%。
hplc法色谱分析条件如下,以下实施例中的检测方法相同:
检测器:elsd检测器;
气体流速:3ml/min
色谱柱:c18(agilent)色谱柱(250mm×4.6mm);
流动相为三氟乙酸:七氟丁酸:水(7:0.6:993.4;v:v);
流速:1ml/min;
柱温:30℃。
实施例9:25℃水相体系中1g/l赖氨酸为底物催化4-24h合成哌啶甲酸。
在反应体系中加入适量含赖氨酸环化脱氨酶突变株的重组大肠杆菌e.colibl21(de3)-pet28a-val61-val94-splcd休止细胞作为催化剂(细胞浓度5gdcw/l)随后在体系中依次加入1g/l赖氨酸及100mmhepes缓冲溶液(ph=7.2)至终体积为10ml,混合均匀后置于25℃加热搅拌器中,200rpm,搅拌反应4-24h。反应结束后,取1ml反应液,12000rpm离心2min,取上清液过滤膜后用高效液相色谱检测底物消耗与产物生成,得到哌啶甲酸浓度0.33-0.61g/l,摩尔转化率为37.5%-69.3%。相比较仅转化4h的结果,可以看出转化时间的延长能够显著增加产物的积累量,而当继续延长转化时间时,底物浓度与转化率并没有显著提高,可能是较低温度对催化活力造成负面的影响。
实施例10:37℃水相体系中1g/l赖氨酸为底物催化合成哌啶甲酸。
在反应体系中加入适量含赖氨酸环化脱氨酶突变株的重组大肠杆菌e.colibl21(de3)-pet28a-val61-val94-splcd休止细胞催化剂(细胞浓度5gdcw/l),随后在体系中依次加入1g/l赖氨酸及100mmhepes缓冲溶液(ph=7.2)至终体积为10ml,混合均匀后置于37℃加热搅拌器中,200rpm,搅拌反应24h。反应结束后,取1ml反应液,12000rpm离心2min,取上清液过滤膜后用高效液相色谱检测底物消耗与产物生成,得到哌啶甲酸浓度0.82g/l,摩尔转化率为93.5%。相比较25℃的结果,可以看出转化温度的适宜能够显著增加产物的积累量。
实施例11:水相体系中20g/l赖氨酸为底物催化合成哌啶甲酸。
在反应体系中加入适量含赖氨酸环化脱氨酶突变株的重组大肠杆菌e.colibl21(de3)-pet28a-val61-val94-splcd休止细胞催化剂(细胞浓度25gdcw/l)随后在体系中依次加入20g/l赖氨酸及100mmhepes缓冲溶液(ph=7.2)至终体积为10ml,混合均匀后置于37℃加热搅拌器中,200rpm,搅拌反应24h。反应结束后,取1ml反应液,12000rpm离心2min,取上清液过滤膜后用高效液相色谱检测底物消耗与产物生成,得到哌啶甲酸浓度17.3g/l,摩尔转化率为98.3%。
实施例12:水相体系中50g/l赖氨酸为底物催化合成哌啶甲酸。
在反应体系中加入适量含赖氨酸环化脱氨酶突变株的重组大肠杆菌e.colibl21(de3)-pet28a-val61-val94-splcd休止细胞催化剂(细胞浓度25gdcw/l)随后在体系中依次加入50g/l赖氨酸及100mmhepes缓冲溶液(ph=7.2)至终体积为10ml,混合均匀后置于37℃加热搅拌器中,200rpm,搅拌反应48h。反应结束后,取1ml反应液,12000rpm离心2min,取上清液过滤膜后用高效液相色谱检测底物消耗与产物生成,得到哌啶甲酸浓度43.1g/l,摩尔转化率为97.7%。
实施例13:水相体系中70g/l赖氨酸为底物催化合成哌啶甲酸。
在反应体系中加入适量含赖氨酸环化脱氨酶突变株的重组大肠杆菌e.colibl21(de3)-pet28a-val61-val94-splcd休止细胞催化剂(细胞浓度25gdcw/l)随后在体系中依次加入70g/l赖氨酸及100mmhepes缓冲溶液(ph=7.2)至终体积为10ml,混合均匀后置于37℃加热搅拌器中,200rpm,搅拌反应48h。反应结束后,取1ml反应液,12000rpm离心2min,取上清液过滤膜后用高效液相色谱检测底物消耗与产物生成,得到哌啶甲酸浓度56.4g/l,摩尔转化率为91.5%,相较底物赖氨酸浓度为50g/l下降明显。
实施例14含0.1-0.5g/lfeso4水相体系中40g/l赖氨酸为底物催化合成哌啶甲酸。
在反应体系中加入适量含赖氨酸环化脱氨酶突变株的重组大肠杆菌e.colibl21(de3)-pet28a-val61-val94-splcd休止细胞(细胞浓度25gdcw/l)随后在体系中分别加入50g/l赖氨酸,0.1g/l,0.25g/l,0.5g/lfeso4及100mmhepes缓冲溶液(ph=7.2)至终体积为10ml,混合均匀后置于37℃加热搅拌器中,200rpm,搅拌反应12-24h。反应过程中,每2h取样取0.5ml反应液,12000rpm离心2min,取上清液过滤膜后用高效液相色谱检测底物消耗与产物生成,得到哌啶甲酸浓度达到平衡时间分别为22h,14h与24h,摩尔转化率为97.7%±0.5%,97.5%±0.1%,91.1%±0.2%。从结果可以看出,feso4添加量为0.25g/l时能够提高哌啶甲酸的合成效率,而过高的feso4添加量反而会降低哌啶甲酸的合成效率。
序列表
<110>南京工业大学
<120>赖氨酸环化脱氨酶及其应用
<160>8
<170>siposequencelisting1.0
<210>1
<211>355
<212>prt
<213>始旋链霉菌atcc25486(s.pristinaespiralisatcc25486)
<400>1
metgluthrtrpvalleuglyargargaspvalalagluvalvalala
151015
alavalglyargaspgluleumetargargileileaspargleuthr
202530
glyglyleualagluileglyargglygluarghisleuserproleu
354045
argglyglyleugluargsergluprovalproglyvaltrpglutrp
505560
metprohisarggluproglyasphisilethrleulysthrvalgly
65707580
tyrserproalaasnproglyargpheglyleuprothrileleugly
859095
thrvalalaargtyraspaspthrthrglyalaleuthralaleumet
100105110
aspglyvalleuleuthralaleuargthrglyalaalaseralaval
115120125
alaserargleuleualaargproaspserhisthrleuglyleuile
130135140
glythrglyalaglnalavalthrglnleuhisalaleuserleuval
145150155160
leuproleuglnargalaleuvaltrpaspthraspproalahisarg
165170175
gluserphealaargargalaalaphethrglyvalservalgluile
180185190
alagluproalaargilealaalaglualaaspvalileserthrala
195200205
thrservalalavalglyglnglyprovalleuproaspthrglyval
210215220
arggluhisleuhisileasnalavalglyalaaspleuvalglylys
225230235240
thrgluleuproleuglyleuleugluargalaphevalthralaasp
245250255
hisprogluglnalaleuarggluglyglucysglnglnleuserala
260265270
aspargleuglyproglnleualahisleucysalaaspproalaala
275280285
alaalaglyargglnaspthrleuservalpheaspserthrglyphe
290295300
alaphegluaspalaleualametgluvalpheleuglualaalaala
305310315320
gluargaspleuglyileargvalglyilegluhishisproglyasp
325330335
alaleuaspprotyralaleuglnproleuproleuproleualaala
340345350
proalahis
355
<210>2
<211>355
<212>prt
<213>始旋链霉菌atcc25486(s.pristinaespiralisatcc25486)
<400>2
metgluthrtrpvalleuglyargargaspvalalagluvalvalala
151015
alavalglyargaspgluleumetargargileileaspargleuthr
202530
glyglyleualagluileglyargglygluarghisleuserproleu
354045
argglyglyleugluargsergluprovalproglyiletrpglutrp
505560
metprohisarggluproglyasphisilethrleulysthrvalgly
65707580
tyrserproalaasnproglyargpheglyleuprothrvalleugly
859095
thrvalalaargtyraspaspthrthrglyalaleuthralaleumet
100105110
aspglyvalleuleuthralaleuargthrglyalaalaseralaval
115120125
alaserargleuleualaargproaspserhisthrleuglyleuile
130135140
glythrglyalaglnalavalthrglnleuhisalaleuserleuval
145150155160
leuproleuglnargalaleuvaltrpaspthraspproalahisarg
165170175
gluserphealaargargalaalaphethrglyvalservalgluile
180185190
alagluproalaargilealaalaglualaaspvalileserthrala
195200205
thrservalalavalglyglnglyprovalleuproaspthrglyval
210215220
arggluhisleuhisileasnalavalglyalaaspleuvalglylys
225230235240
thrgluleuproleuglyleuleugluargalaphevalthralaasp
245250255
hisprogluglnalaleuarggluglyglucysglnglnleuserala
260265270
aspargleuglyproglnleualahisleucysalaaspproalaala
275280285
alaalaglyargglnaspthrleuservalpheaspserthrglyphe
290295300
alaphegluaspalaleualametgluvalpheleuglualaalaala
305310315320
gluargaspleuglyileargvalglyilegluhishisproglyasp
325330335
alaleuaspprotyralaleuglnproleuproleuproleualaala
340345350
proalahis
355
<210>3
<211>355
<212>prt
<213>始旋链霉菌atcc25486(s.pristinaespiralisatcc25486)
<400>3
metgluthrtrpvalleuglyargargaspvalalagluvalvalala
151015
alavalglyargaspgluleumetargargileileaspargleuthr
202530
glyglyleualagluileglyargglygluarghisleuserproleu
354045
argglyglyleugluargsergluprovalproglyvaltrpglutrp
505560
metprohisarggluproglyasphisilethrleulysthrvalgly
65707580
tyrserproalaasnproglyargpheglyleuprothrvalleugly
859095
thrvalalaargtyraspaspthrthrglyalaleuthralaleumet
100105110
aspglyvalleuleuthralaleuargthrglyalaalaseralaval
115120125
alaserargleuleualaargproaspserhisthrleuglyleuile
130135140
glythrglyalaglnalavalthrglnleuhisalaleuserleuval
145150155160
leuproleuglnargalaleuvaltrpaspthraspproalahisarg
165170175
gluserphealaargargalaalaphethrglyvalservalgluile
180185190
alagluproalaargilealaalaglualaaspvalileserthrala
195200205
thrservalalavalglyglnglyprovalleuproaspthrglyval
210215220
arggluhisleuhisileasnalavalglyalaaspleuvalglylys
225230235240
thrgluleuproleuglyleuleugluargalaphevalthralaasp
245250255
hisprogluglnalaleuarggluglyglucysglnglnleuserala
260265270
aspargleuglyproglnleualahisleucysalaaspproalaala
275280285
alaalaglyargglnaspthrleuservalpheaspserthrglyphe
290295300
alaphegluaspalaleualametgluvalpheleuglualaalaala
305310315320
gluargaspleuglyileargvalglyilegluhishisproglyasp
325330335
alaleuaspprotyralaleuglnproleuproleuproleualaala
340345350
proalahis
355
<210>4
<211>1068
<212>dna
<213>始旋链霉菌atcc25486(s.pristinaespiralisatcc25486)
<400>4
atggaaacttgggttttaggtcgtcgtgatgttgcagaagttgttgcagcagttggtcgt60
gatgaattaatgcgtcgtattatcgatcgtttaactggtggtttagcagaaattggtcgt120
ggtgaacgtcatttatctccattacgtggtggtttagaacgttctgaaccagttccaggt180
gtgtgggaatggatgccacatcgtgaaccaggtgatcatattactttaaaaactgttggt240
tattctccagcaaatccaggtcgttttggtttaccaactattttaggtaccgttgcacgt300
tatgatgatactactggtgcattaactgcattaatggatggtgttttattaactgcatta360
cgtactggtgcagcatctgctgttgcatctcgtttattagcacgtccagattctcatact420
ttaggtttaattggtactggtgcacaagcagttactcaattgcatgcattatctttagtt480
ttaccattacaacgtgcattagtttgggatactgatccagcacatcgtgaatcttttgca540
cgtcgtgcagcatttactggtgtttctgttgaaattgcagaaccagcacgtattgcagca600
gaagcagatgttatttctactgcaacttctgttgcagttggtcaaggtccagttttacca660
gatactggtgttcgtgaacatttacatattaatgcagttggtgcagatttagttggtaaa720
actgaattaccattaggtttattagaacgtgcatttgttactgcagatcatccagaacaa780
gcattacgtgaaggtgaatgtcaacaattatctgctgatcgtttaggtccacaattagca840
catttatgtgcagatccagcagcagcagcaggtcgtcaagatactttatctgtttttgat900
tctactggttttgcatttgaagatgcattagcaatggaagtttttttagaagcagcagca960
gaacgtgatttaggtattcgtgttggtattgaacatcatccaggtgatgcattagatcca1020
tatgcattacaaccattaccattaccattagcagcaccagcacattaa1068
<210>5
<211>1068
<212>dna
<213>始旋链霉菌atcc25486(s.pristinaespiralisatcc25486)
<400>5
atggaaacttgggttttaggtcgtcgtgatgttgcagaagttgttgcagcagttggtcgt60
gatgaattaatgcgtcgtattatcgatcgtttaactggtggtttagcagaaattggtcgt120
ggtgaacgtcatttatctccattacgtggtggtttagaacgttctgaaccagttccaggt180
atttgggaatggatgccacatcgtgaaccaggtgatcatattactttaaaaactgttggt240
tattctccagcaaatccaggtcgttttggtttaccaactgtgttaggtaccgttgcacgt300
tatgatgatactactggtgcattaactgcattaatggatggtgttttattaactgcatta360
cgtactggtgcagcatctgctgttgcatctcgtttattagcacgtccagattctcatact420
ttaggtttaattggtactggtgcacaagcagttactcaattgcatgcattatctttagtt480
ttaccattacaacgtgcattagtttgggatactgatccagcacatcgtgaatcttttgca540
cgtcgtgcagcatttactggtgtttctgttgaaattgcagaaccagcacgtattgcagca600
gaagcagatgttatttctactgcaacttctgttgcagttggtcaaggtccagttttacca660
gatactggtgttcgtgaacatttacatattaatgcagttggtgcagatttagttggtaaa720
actgaattaccattaggtttattagaacgtgcatttgttactgcagatcatccagaacaa780
gcattacgtgaaggtgaatgtcaacaattatctgctgatcgtttaggtccacaattagca840
catttatgtgcagatccagcagcagcagcaggtcgtcaagatactttatctgtttttgat900
tctactggttttgcatttgaagatgcattagcaatggaagtttttttagaagcagcagca960
gaacgtgatttaggtattcgtgttggtattgaacatcatccaggtgatgcattagatcca1020
tatgcattacaaccattaccattaccattagcagcaccagcacattaa1068
<210>6
<211>1068
<212>dna
<213>始旋链霉菌atcc25486(s.pristinaespiralisatcc25486)
<400>6
atggaaacttgggttttaggtcgtcgtgatgttgcagaagttgttgcagcagttggtcgt60
gatgaattaatgcgtcgtattatcgatcgtttaactggtggtttagcagaaattggtcgt120
ggtgaacgtcatttatctccattacgtggtggtttagaacgttctgaaccagttccaggt180
gtgtgggaatggatgccacatcgtgaaccaggtgatcatattactttaaaaactgttggt240
tattctccagcaaatccaggtcgttttggtttaccaactgtgttaggtaccgttgcacgt300
tatgatgatactactggtgcattaactgcattaatggatggtgttttattaactgcatta360
cgtactggtgcagcatctgctgttgcatctcgtttattagcacgtccagattctcatact420
ttaggtttaattggtactggtgcacaagcagttactcaattgcatgcattatctttagtt480
ttaccattacaacgtgcattagtttgggatactgatccagcacatcgtgaatcttttgca540
cgtcgtgcagcatttactggtgtttctgttgaaattgcagaaccagcacgtattgcagca600
gaagcagatgttatttctactgcaacttctgttgcagttggtcaaggtccagttttacca660
gatactggtgttcgtgaacatttacatattaatgcagttggtgcagatttagttggtaaa720
actgaattaccattaggtttattagaacgtgcatttgttactgcagatcatccagaacaa780
gcattacgtgaaggtgaatgtcaacaattatctgctgatcgtttaggtccacaattagca840
catttatgtgcagatccagcagcagcagcaggtcgtcaagatactttatctgtttttgat900
tctactggttttgcatttgaagatgcattagcaatggaagtttttttagaagcagcagca960
gaacgtgatttaggtattcgtgttggtattgaacatcatccaggtgatgcattagatcca1020
tatgcattacaaccattaccattaccattagcagcaccagcacattaa1068
<210>7
<211>355
<212>prt
<213>始旋链霉菌atcc25486(s.pristinaespiralisatcc25486)
<400>7
metgluthrtrpvalleuglyargargaspvalalagluvalvalala
151015
alavalglyargaspgluleumetargargileileaspargleuthr
202530
glyglyleualagluileglyargglygluarghisleuserproleu
354045
argglyglyleugluargsergluprovalproglyiletrpglutrp
505560
metprohisarggluproglyasphisilethrleulysthrvalgly
65707580
tyrserproalaasnproglyargpheglyleuprothrileleugly
859095
thrvalalaargtyraspaspthrthrglyalaleuthralaleumet
100105110
aspglyvalleuleuthralaleuargthrglyalaalaseralaval
115120125
alaserargleuleualaargproaspserhisthrleuglyleuile
130135140
glythrglyalaglnalavalthrglnleuhisalaleuserleuval
145150155160
leuproleuglnargalaleuvaltrpaspthraspproalahisarg
165170175
gluserphealaargargalaalaphethrglyvalservalgluile
180185190
alagluproalaargilealaalaglualaaspvalileserthrala
195200205
thrservalalavalglyglnglyprovalleuproaspthrglyval
210215220
arggluhisleuhisileasnalavalglyalaaspleuvalglylys
225230235240
thrgluleuproleuglyleuleugluargalaphevalthralaasp
245250255
hisprogluglnalaleuarggluglyglucysglnglnleuserala
260265270
aspargleuglyproglnleualahisleucysalaaspproalaala
275280285
alaalaglyargglnaspthrleuservalpheaspserthrglyphe
290295300
alaphegluaspalaleualametgluvalpheleuglualaalaala
305310315320
gluargaspleuglyileargvalglyilegluhishisproglyasp
325330335
alaleuaspprotyralaleuglnproleuproleuproleualaala
340345350
proalahis
355
<210>9
<211>1068
<212>dna
<213>始旋链霉菌atcc25486(s.pristinaespiralisatcc25486)
<400>9
atggaaacttgggttttaggtcgtcgtgatgttgcagaagttgttgcagcagttggtcgt60
gatgaattaatgcgtcgtattatcgatcgtttaactggtggtttagcagaaattggtcgt120
ggtgaacgtcatttatctccattacgtggtggtttagaacgttctgaaccagttccaggt180
atttgggaatggatgccacatcgtgaaccaggtgatcatattactttaaaaactgttggt240
tattctccagcaaatccaggtcgttttggtttaccaactattttaggtaccgttgcacgt300
tatgatgatactactggtgcattaactgcattaatggatggtgttttattaactgcatta360
cgtactggtgcagcatctgctgttgcatctcgtttattagcacgtccagattctcatact420
ttaggtttaattggtactggtgcacaagcagttactcaattgcatgcattatctttagtt480
ttaccattacaacgtgcattagtttgggatactgatccagcacatcgtgaatcttttgca540
cgtcgtgcagcatttactggtgtttctgttgaaattgcagaaccagcacgtattgcagca600
gaagcagatgttatttctactgcaacttctgttgcagttggtcaaggtccagttttacca660
gatactggtgttcgtgaacatttacatattaatgcagttggtgcagatttagttggtaaa720
actgaattaccattaggtttattagaacgtgcatttgttactgcagatcatccagaacaa780
gcattacgtgaaggtgaatgtcaacaattatctgctgatcgtttaggtccacaattagca840
catttatgtgcagatccagcagcagcagcaggtcgtcaagatactttatctgtttttgat900
tctactggttttgcatttgaagatgcattagcaatggaagtttttttagaagcagcagca960
gaacgtgatttaggtattcgtgttggtattgaacatcatccaggtgatgcattagatcca1020
tatgcattacaaccattaccattaccattagcagcaccagcacattaa1068
- 还没有人留言评论。精彩留言会获得点赞!