一种环状RNAhsa-circ-0044508及其特异性扩增引物和应用的制作方法
本发明属于肿瘤分子生物学领域,尤其涉及一种用于乳腺癌诊断的环状rna,用于所述环状rna的特异性扩增引物及应用。
背景技术:
乳腺癌是一种常见的女性高危恶性肿瘤,严重影响人们的生活质量。近年来,全世界的女性癌症患者中大约有22.9%为乳腺癌患者,新发现的乳腺癌患者中,中国患者占12.2%。尽管在药物敏感性和外科技术上已有巨大的突破,乳腺癌的复发和转移仍频繁地发生,成为晚期乳腺癌患者死亡的主要原因。早期诊断、早期治疗能够大大减少乳腺癌恶化的进一步发生,随着分子水平的发展以及对乳腺癌发生、发展、治疗相关机制的进一步研究,对靶向诊断和靶向治疗分子标志物的筛选和研究成为乳腺癌研究最受关注的领域之一。
环状rna(circularrna,circrna)是一类不具有5'末端帽子和3'末端poly(a)尾巴、并以共价键形成环形结构的非编码rna分子,主要由前体rna(pre-mrna)通过可变剪切加工产生。circrna大多数定位于细胞质中,少数定位于细胞核内,大多数由外显子形成。circrna不易被核酸外切酶降解,与线性rna相比能更稳定地存在于生物体内,其在不同的物种中具有保守性,同时在不同组织及发育阶段具有表达特异性,大多数circrna在不同的物种间含量丰富且都具有组织特异性,在人体组织、唾液、血液和外泌体中都有稳定表达,这使得circrna成为肿瘤患者诊断、治疗和预后追踪的理想生物标志物。
近年来,关于circrna与肿瘤之间的关系研究已有很多报道,已经明确circrna具有调节肿瘤细胞增殖、凋亡、血管形成、代谢以及耐药等功能。zhang等通过对不同泌乳时期的两组老鼠的乳腺组织进行circrna芯片检测,发现circrna在不同泌乳期老鼠乳腺组织中的表达有很大差异,两个时期检测到circrna种类大多数不同。因此,circrna可能是乳腺癌精准诊疗的新靶点,发现和筛选乳腺癌相关的circrna标志物,对早期防治乳腺癌具有重要的意义和价值。
技术实现要素:
本申请的目的在于提供以下几个方面:
第一方面,本申请提供一种环状rna,所述环状rna的核苷酸序列如seqidno:1所示,其中,起始两个核苷酸与最末两个核苷酸为成环结合位点。
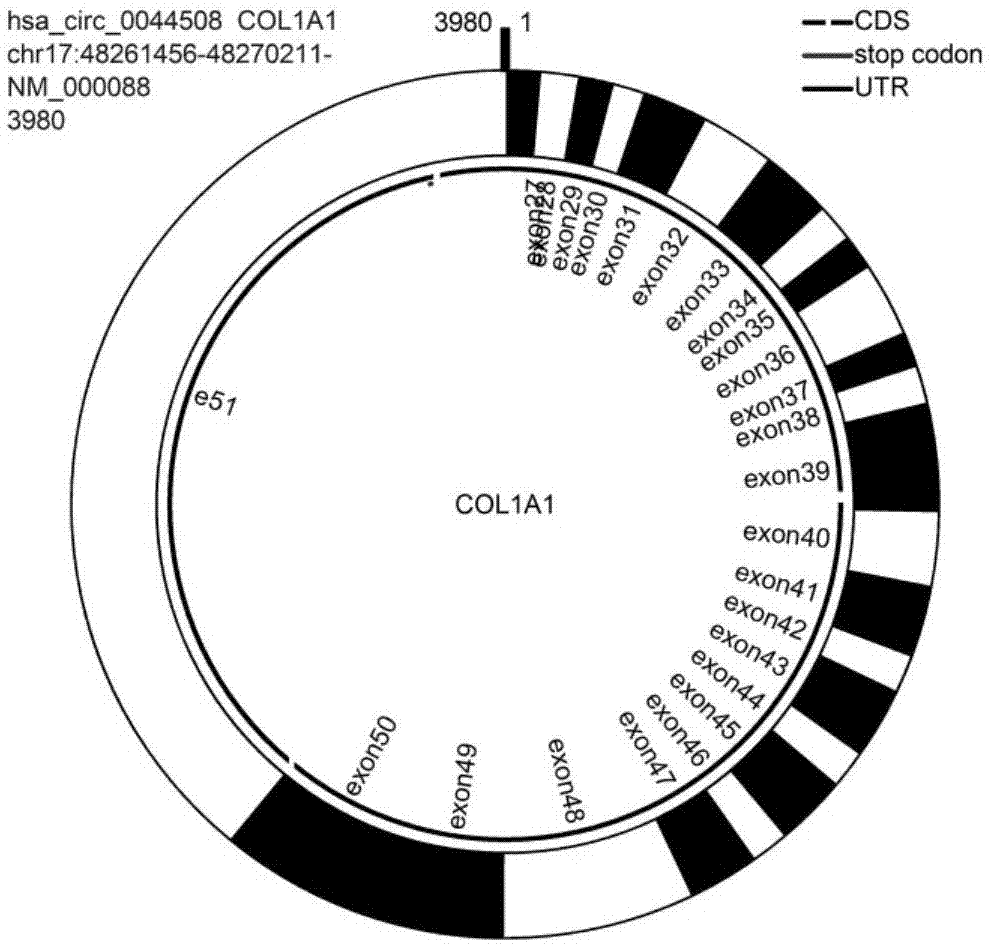
所述环状rna在circ-rna数据库中的全称为hsa-circ-0044508,简称circ-0044508,定位于人类17号染色体反义链48261456-48270211区域,由col1a1基因外显子(exon)27-50区剪切环化而成。
图1为所述环状rna的基因结构图,如图1所示,所述环状rnacirc-0044508的序列全长3980bp,其中,cds为蛋白质编码区。
根据本申请的第二方面,提供本发明第一方面所述环状rnacirc-0044508作为筛查乳腺癌的分子标志物的用途。
通过生物信息学预测,发明人发现环状rnacirc-0044508的应答原件mir-21,该基因能够通过pdcd4/mapk信号通路影响肿瘤细胞凋亡。
并且,本发明研究发现相对于乳腺癌患者癌旁正常组织,环状rnacirc-0044508在乳腺癌组织中显著上调;在血液样本中,相对健康人群,环状rnacirc-0044508在乳腺癌患者血液中也显著上调。
因此,所述环状rnacirc-0044508可用于乳腺癌筛查。
第二方面,本申请还提供所述环状rna用作筛查乳腺癌的分子标志物的用途。
第三方面,本申请还提供一种用于扩增第一方面所述环状rna的特异性扩增引物,所述特异性扩增引物包括上游引物和下游引物,其中,
所述上游引物的核苷酸序列如seqidno:2所示;
所述下游引物的核苷酸序列如seqidno:3所示。
具体地,所述上游引物的核苷酸序列为:5’-aaacataaggtcctgctggcaa-3’;
所述下游引物的核苷酸序列为:5’-gccttgttcacctctctcgc-3’。
在一种可实现的方式中,所述上游引物的gc含量为45.5%,所述下游引物的gc含量为60.0%,其中,所述gc含量是指在dna4种碱基中,鸟嘌呤和胞嘧啶所占的比率。
进一步地,所述上游引物的tm值为56.8,所述下游引物的tm值59.1,验证后上下游引物qpcr使用时的最适合tm值为60,其中,所述tm值是指上游引物或者下游引物的熔解温度。
本发明人发现,使用本申请提供的特异性扩增引物对第一方面所述rna进行扩增,如实施例1所示,将扩增结果进行分析,得到roc曲线,结果如图3所示,roc曲线下面积为0.972,这表明使用本申请提供的特异性扩增引物得到的扩增产物为单一条带,无非特异性扩增,所述特异性扩增引物能够作为该种检测的特异性标志物。
第四方面,本申请还提供一种扩增第一方面所述环状rna的方法,所述方法包括:
步骤1,合成第一链cdna:混合原料,所述原料包括第一方面所述环状rna、随机引物、ddh2o、dntp混合液、反转录缓冲液、rna酶抑制剂、反转录酶,按照第一程序控温反应;
步骤2,对步骤1制得的第一链cdna进行实时荧光定量聚合酶链式反应(即,qpcr)扩增:配制扩增体系,所述扩增体系包括步骤1制备的cdna、第三方面所示的上游引物、第三方面所示的下游引物、ddh2o、qpcr扩增mix(含sybgreen染料),按照第二程序控温反应。
在一种可实现的方式中,步骤1中,
所述原料的按照如下配比混合:1μl第一方面所述环状rna,1μl随机引物,10μlddh2o,2μldntp混合液,4μl反转录缓冲液,1μlrna酶抑制剂,1μl反转录酶,其中,所述dntp混合液包括datp,dgtp,dctp和dttp;
所述第一程序为:25℃条件下反应5min,42℃条件下反应60min,70℃条件下反应5min。使用abipcr扩增仪进行反转录,所述cdna置于-80℃储存备用。
进一步地,在步骤2中,
所述扩增体系按照如下配比配制:2μl步骤1制备的cdna,0.5μl如第三方面所述的上游引物,0.5μl第三方面所述的下游引物,7μlddh2o,10μlqpcr扩增mix(sybgreen染料);
所述第二程序为:95℃预变性10s,然后以95℃反应15s、60℃反应30s、72℃反应40s进行40个循环,以95℃反应5s、65℃反应1min、37℃反应1min,最后终止反应。
第五方面,本申请还提供前述特异性扩增引物在制备用于乳腺癌筛查的试剂盒中的应用。
第六方面,本申请还提供一种用于乳腺癌筛查的试剂盒,所述试剂盒包括第三方面所述的特异性扩增引物。
在一种可实现的方式中,所述试剂盒还包括反转录酶、缓冲液、ddh2o、dna聚合酶、荧光染料和dntp混合液中的至少一种。
所述试剂盒可使用qpcr的方法检测乳腺癌组织及外周血中的circ-0044508,用于筛查乳腺癌。
本申请人发现,本申请所述的环状rna在乳腺癌组织细胞中呈现高表达,即,乳腺癌组织中所述环状rna的含量远高于癌旁组织中该rna含量,因此,所述环状rnacirc-0044508可以用作乳腺癌筛查的分子标志物,而且,本申请提供的用于扩增所述环状rna的特异性扩增引物对所述环状rna具有高度稳定性、特异性和敏感性,其中灵敏度可达100%,特异性可达83.3%以上。
附图说明
图1示出circ-0044508基因结构图;
图2示出qpcr检测circ-0044508在乳腺癌组织及癌旁组织中差异表达图;
图3示出乳腺癌组织中circ-0044508检测结果的roc曲线。
具体实施方式
下面结合实施例对本发明进行详细说明,本发明的特点和优点将随着这些说明而变得更为清楚、明确。不过这些实例仅仅是范例性的,并不对本发明的保护范围构成任何限制。
实施例
在实施例中,
所述无rna酶无dna酶灭菌水(ddh2o)购自天根生物公司,所述pcr扩增混合体系(mix)及qpcr(含荧光染料)扩增混合体系(mix)购自takara公司,所述反转录酶购自thermo公司,所述dntp混合液购自invitrogen公司。
实施例1qpcr反应检测circ-0044508在乳腺癌组织中的表达
1、设计特异性扩增引物:
本申请人所设计的特异性扩增引物序列及相关信息如表1所示:
表1特异性扩增引物序列及相关信息
2、总rna的提取:
(1)trizol法提取乳腺癌组织/血液中的总rna:取0.2g乳腺癌组织/血液,在液氮条件下进行研磨,直至所述组织/血液呈粉末状态,在液氮中加入1mltrizol继续研磨,待将trizol研磨为干粉状态后,室温静置,待干粉化为液体后收集所有液体;
(2)步骤1制得的液体样品在室温下裂解5分钟后,按照每1ml的trizol研磨匀浆加入0.2ml氯仿的比例加入氯仿,盖紧管盖,涡旋震荡15s后,静置待出现分层后,4℃条件下12000rpm离心15分钟,离心后混合液体分层为下层氯仿相,中层蛋白层,上层无色水相,rna全部被分配于水相中;
(3)将水相转移到新的离心管中,按照每1ml的trizol研磨匀浆加入0.5ml异丙醇的比例向体系中加入异丙醇,混匀,于-20℃静置30min后,4℃条件下12000rpm离心15分钟,得沉淀,rna全部存在于沉淀中;
(4)移去上清液,向体系中加入1ml75%乙醇清洗rna沉淀,于4℃条件下7500rpm离心5分钟;
(5)去除乙醇溶液,室温下干燥5-10分钟,至乙醇挥发,但使rna未完全干燥,将无rna酶的ddh2o水加入离心管,60℃孵育5分钟,获得总rna;
(6)提取的总rna使用nanodropnd-2000测定rna的浓度和纯度,确定总rna的浓度和纯度能够满足后续实验的要求,总rna溶液分装并置于-80℃保存。
3、第一链cdna序列的合成:
取pcr管配置反转录体系,反转录体系为:1μl总rna(约500ng),1μl随机引物(thermo公司),10μlddh2o(天根生物公司),2μldntp混合液(datp,dgtp,dctp和dttp,invitrogen公司),4μl反转录缓冲液(thermo公司),1μlrna酶抑制剂(thermo公司),1μl反转录酶(thermo公司),总体积20μl;
反应条件为:25℃下反应5min;42℃下反应60min,70℃下反应5min。使用abipcr扩增仪器进行反转录。反转录得到的cdna置于-80℃储存备用。
4、circ-0044508基因cdna扩增验证:
取2μl步骤3反转录得到的cdna,配制体系进行pcr扩增;
pcr扩增体系为:2μlcdna,0.5μl表1中所示上游引物,0.5μ表1中所示下游引物,7μlddh2o、10μlpcr扩增mix,总体积20μl;
反应条件为94℃预变性5min;然后以94℃反应30s、60℃下反应20s、72℃反应20s进行35个循环;最后以72℃反应5min。取2μl反应产物在1.5g琼脂糖/100ml1×tae缓冲液,120v电压,25min条件下验证pcr产物是否为单一特异性扩增条带。
5、qpcr扩增反应:
使用反转录得到的cdna样品进行检测。
反应体系为:2μlcdna,0.8μl上游引物,0.8μl下游引物,6.4μlddh2o(天根生物公司),10μlqpcr扩增mix(sybgreen染料),总体积20μl;
qpcr反应条件:95℃预变性10s,然后以95℃反应15s、60℃反应30s、72℃反应40s进行40个循环,以95℃反应5s、65℃反应1min、37℃反应1min,最后终止反应。
扩增反应在实时荧光定量pcr仪lightcyler480上进行,在扩增目的基因的同时扩增β-actin作为内参对照,并通过2(-△△ct)法计算基因的相对表达量。
对于实施例1,分别取不同样本,重复6次。
其中,在内参β-actin基因及circ-0044508基因qpcr溶解曲线图中可以看出,扩增峰单一,无杂峰,证明引物特异性优秀,扩增实验正常。
实施例2qpcr反应检测circ-0044508在乳腺癌癌旁组织中的表达
重复实施例1的过程,区别在于:在步骤2中,提取乳腺癌癌旁组织中的总rna。
对于实施例2,分别取不同样本,重复6次。
实施例1~2的qpcr结果分析
1、基因表达分析
其中,circ-0044508的表达结果如图2~3所示:
在图2的6对组织中,相对于乳腺癌癌旁组织,环状rnacirc-0044508在乳腺癌组织中表达显著上调,上调约6倍。
以上结果表明,环状rnacirc-0044508在乳腺癌组织及癌旁组织中具有差异表达,具体地,所述环状rnacirc-0044508在乳腺癌组织中为高表达,因此,所述环状rnacirc-0044508可以用于乳腺癌的筛查。
2、roc曲线分析
对实施例1得到的试验数据进行分析,得到roc曲线,如图3所示,其中,曲线下面积auc为0.972,表示检测靶点能够作为检测该环状rna的特异性标志物,并且,检测灵敏度可达100%,特异性可达88.3%以上。
本领域技术人员知道,roc曲线下面积在1.0和0.5之间,在auc>0.5的情况下,auc越接近于1,说明诊断效果越好。auc在0.5-0.7时有较低准确性,auc在0.7-0.9时有一定准确性,auc在0.9以上时有较高准确性,该值大于0.7即表示检测靶点能够作为该种检测的特异性标示物。
以上结合具体实施方式和范例性实例对本申请进行了详细说明,不过这些说明并不能理解为对本申请的限制。本领域技术人员理解,在不偏离本申请精神和范围的情况下,可以对本申请技术方案及其实施方式进行多种等价替换、修饰或改进,这些均落入本申请的范围内。本申请的保护范围以所附权利要求为准。
序列表
<110>宁夏医科大学总医院
<120>一种环状rnahsa-circ-0044508及其特异性扩增引物和应用
<160>3
<170>siposequencelisting1.0
<210>1
<211>3980
<212>dna
<213>人类(homosapiens)
<400>1
ggtcctgctggcaaagatggagaggctggagctcagggaccccctggccctgctggtccc60
gctggcgagagaggtgaacaaggccctgctggctcccccggattccagggtctccctggt120
cctgctggtcctccaggtgaagcaggcaaacctggtgaacagggtgttcctggagacctt180
ggcgcccctggcccctctggagcaagaggcgagagaggtttccctggcgagcgtggtgtg240
caaggtccccctggtcctgctggtccccgaggggccaacggtgctcccggcaacgatggt300
gctaagggtgatgctggtgcccctggagctcccggtagccagggcgcccctggccttcag360
ggaatgcctggtgaacgtggtgcagctggtcttccagggcctaagggtgacagaggtgat420
gctggtcccaaaggtgctgatggctctcctggcaaagatggcgtccgtggtctgactggc480
cccattggtcctcctggccctgctggtgcccctggtgacaagggtgaaagtggtcccagc540
ggccctgctggtcccactggagctcgtggtgcccccggagaccgtggtgagcctggtccc600
cccggccctgctggctttgctggcccccctggtgctgacggccaacctggtgctaaaggc660
gaacctggtgatgctggtgctaaaggcgatgctggtccccctggccctgccggacccgct720
ggaccccctggccccattggtaatgttggtgctcctggagccaaaggtgctcgcggcagc780
gctggtccccctggtgctactggtttccctggtgctgctggccgagtcggtcctcctggc840
ccctctggaaatgctggaccccctggccctcctggtcctgctggcaaagaaggcggcaaa900
ggtccccgtggtgagactggccctgctggacgtcctggtgaagttggtccccctggtccc960
cctggccctgctggcgagaaaggatcccctggtgctgatggtcctgctggtgctcctggt1020
actcccgggcctcaaggtattgctggacagcgtggtgtggtcggcctgcctggtcagaga1080
ggagagagaggcttccctggtcttcctggcccctctggtgaacctggcaaacaaggtccc1140
tctggagcaagtggtgaacgtggtccccctggtcccatgggcccccctggattggctgga1200
ccccctggtgaatctggacgtgagggggctcctggtgccgaaggttcccctggacgagac1260
ggttctcctggcgccaagggtgaccgtggtgagaccggccccgctggaccccctggtgct1320
cctggtgctcctggtgcccctggccccgttggccctgctggcaagagtggtgatcgtggt1380
gagactggtcctgctggtcccaccggtcctgtcggccctgttggcgcccgtggccccgcc1440
ggaccccaaggcccccgtggtgacaagggtgagacaggcgaacagggcgacagaggcata1500
aagggtcaccgtggcttctctggcctccagggtccccctggccctcctggctctcctggt1560
gaacaaggtccctctggagcctctggtcctgctggtccccgaggtccccctggctctgct1620
ggtgctcctggcaaagatggactcaacggtctccctggccccattgggccccctggtcct1680
cgcggtcgcactggtgatgctggtcctgttggtccccccggccctcctggacctcctggt1740
ccccctggtcctcccagcgctggtttcgacttcagcttcctgccccagccacctcaagag1800
aaggctcacgatggtggccgctactaccgggctgatgatgccaatgtggttcgtgaccgt1860
gacctcgaggtggacaccaccctcaagagcctgagccagcagatcgagaacatccggagc1920
ccagagggcagccgcaagaaccccgcccgcacctgccgtgacctcaagatgtgccactct1980
gactggaagagtggagagtactggattgaccccaaccaaggctgcaacctggatgccatc2040
aaagtcttctgcaacatggagactggtgagacctgcgtgtaccccactcagcccagtgtg2100
gcccagaagaactggtacatcagcaagaaccccaaggacaagaggcatgtctggttcggc2160
gagagcatgaccgatggattccagttcgagtatggcggccagggctccgaccctgccgat2220
gtggccatccagctgaccttcctgcgcctgatgtccaccgaggcctcccagaacatcacc2280
taccactgcaagaacagcgtggcctacatggaccagcagactggcaacctcaagaaggcc2340
ctgctcctccagggctccaacgagatcgagatccgcgccgagggcaacagccgcttcacc2400
tacagcgtcactgtcgatggctgcacgagtcacaccggagcctggggcaagacagtgatt2460
gaatacaaaaccaccaagacctcccgcctgcccatcatcgatgtggcccccttggacgtt2520
ggtgccccagaccaggaattcggcttcgacgttggccctgtctgcttcctgtaaactccc2580
tccatcccaacctggctccctcccacccaaccaactttccccccaacccggaaacagaca2640
agcaacccaaactgaaccccctcaaaagccaaaaaatgggagacaatttcacatggactt2700
tggaaaatatttttttcctttgcattcatctctcaaacttagtttttatctttgaccaac2760
cgaacatgaccaaaaaccaaaagtgcattcaaccttaccaaaaaaaaaaaaaaaaaaaga2820
ataaataaataactttttaaaaaaggaagcttggtccacttgcttgaagacccatgcggg2880
ggtaagtccctttctgcccgttgggcttatgaaaccccaatgctgccctttctgctcctt2940
tctccacaccccccttggggcctcccctccactccttcccaaatctgtctccccagaaga3000
cacaggaaacaatgtattgtctgcccagcaatcaaaggcaatgctcaaacacccaagtgg3060
cccccaccctcagcccgctcctgcccgcccagcacccccaggccctgggggacctggggt3120
tctcagactgccaaagaagccttgccatctggcgctcccatggctcttgcaacatctccc3180
cttcgtttttgagggggtcatgccgggggagccaccagcccctcactgggttcggaggag3240
agtcaggaagggccacgacaaagcagaaacatcggatttggggaacgcgtgtcaatccct3300
tgtgccgcagggctgggcgggagagactgttctgttccttgtgtaactgtgttgctgaaa3360
gactacctcgttcttgtcttgatgtgtcaccggggcaactgcctgggggcggggatgggg3420
gcagggtggaagcggctccccattttataccaaaggtgctacatctatgtgatgggtggg3480
gtggggagggaatcactggtgctatagaaattgagatgcccccccaggccagcaaatgtt3540
cctttttgttcaaagtctatttttattccttgatatttttcttttttttttttttttttt3600
gtggatggggacttgtgaatttttctaaaggtgctatttaacatgggaggagagcgtgtg3660
cggctccagcccagcccgctgctcactttccaccctctctccacctgcctctggcttctc3720
aggcctctgctctccgacctctctcctctgaaaccctcctccacagctgcagcccatcct3780
cccggctccctcctagtctgtcctgcgtcctctgtccccgggtttcagagacaacttccc3840
aaagcacaaagcagtttttccccctaggggtgggaggaagcaaaagactctgtacctatt3900
ttgtatgtgtataataatttgagatgtttttaattattttgattgctggaataaagcatg3960
tggaaatgacccaaacataa3980
<210>2
<211>22
<212>dna
<213>人工序列
<400>2
aaacataaggtcctgctggcaa22
<210>3
<211>20
<212>dna
<213>人工序列
<400>3
gccttgttcacctctctcgc20
- 还没有人留言评论。精彩留言会获得点赞!