一种重组慢病毒表达载体、重组细胞及其在自然杀伤细胞培养中的应用的制作方法
本发明属于细胞工程技术领域,具体涉及一种重组慢病毒表达载体、重组细胞及其在自然杀伤细胞培养中的应用。
背景技术:
自然杀伤细胞(naturalkillercell,nk)是机体重要的免疫细胞,不仅与抗肿瘤、抗病毒感染和免疫调节有关,而且在某些情况下参与超敏反应和自身免疫性疾病的发生,能够识别靶细胞、杀伤介质。自然杀伤细胞确切的来源还不十分清楚,一般认为直接从骨髓中衍生。鉴于自然杀伤细胞的广泛用途,提供一种高效稳定的自然杀伤细胞诱导方法具有广阔的应用前景。
申请号为cn201710096536.1的中国发明专利公开了一种使用高表达膜蛋白cd19、cd137l、cd86、cd64及跨膜蛋白il-21的k562工程细胞联合人il-2突变体高效扩增nk细胞的方法,该方法虽然解决了nk细胞特异识别cd19+细胞的问题,但是不能有效刺激提高nk细胞肿瘤杀伤至关重要的nkg2d受体表达。申请号为cn201310173235.6的中国发明专利公开了一种利用cd8α-白介素21片段-cd137转染的k562细胞扩展、激活淋巴细胞和nk细胞的方法,该方法细胞激活水平低,限制了细胞的增殖走势和杀伤能力。
技术实现要素:
有鉴于背景技术中存在的问题,本发明的目的在于提供一种在自然杀伤细胞诱导过程中起到提高nk细胞激活水平、加速nk细胞增殖,延缓nk细胞凋亡作用的一种重组工程细胞,以及用于构建该重组工程细胞的重组慢病毒表达载体和应用。
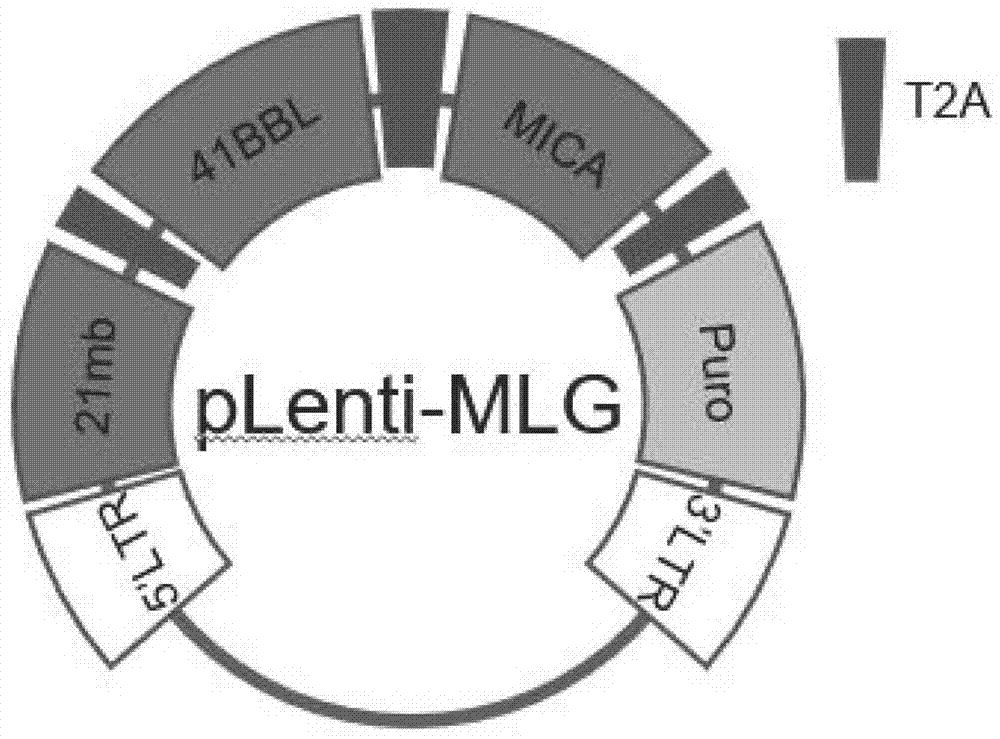
本发明提供了一种重组慢病毒表达载体,所述重组慢病毒表达载体以慢病毒载体为基础,包括依次串联的21mb基因、41bbl基因和mica基因;所述21mb基因由signalpeptide基因、il21基因、cd8hinge基因和cd8transmembrane基因依次串联得到。
优选的,所述21mb基因的核苷酸序列如seqidno.1所示。
优选的,所述41bbl基因的核苷酸序列如seqidno.2所示。
优选的,所述mica基因的核苷酸序列如seqidno.3所示。
优选的,所述21mb基因和所述41bbl基因通过t2a基因进行串联,所述41bbl基因和所述mica基因通过t2a基因进行串联,所述t2a基因的核苷酸序列如seqidno.4所示。
本发明提供了一种重组细胞,所述重组细胞由上述重组慢病毒表达载体感染宿主细胞获得。
优选的,所述宿主细胞为人白血病细胞系k562。
优选的,所述重组细胞通过含有puromycin的抗性培养基筛选获得。
本发明还提供了上述重组慢病毒表达载体或者重组细胞在自然杀伤细胞培养中的应用。
优选的,应用包括如下步骤:
(1)在rpmi-1640培养基中加入8~12%的fbs和800~1200iu/ml的il-2,得到用于外周血单个核细胞的诱导培养基;
(2)将上重组细胞与外周血单个核细胞按照1.5~2.5:1的比例加入到步骤(1)所述诱导培养基中,培养得到自然杀伤细胞。
有益效果:本发明提供了一种重组慢病毒表达载体,所述重组慢病毒表达载体以慢病毒载体为基础,包括依次串联的21mb基因、41bbl基因和mica基因;所述21mb基因由signalpeptide基因、il21基因、cd8hinge基因和cd8transmembrane基因依次串联得到。本发明提供的重组慢病毒表达载体转染宿主细胞后,能使宿主细胞表达mica,从而使宿主细胞在自然杀伤细胞的诱导培养过程中起到特异性激活nkg2d基因、进一步提高nkg2d表达量的作用;同时,由于21mb基因具有膜锚定功能,本发明提供的重组慢病毒表达载体转染宿主细胞后,能在mica阳性的细胞膜上融合表达(而非分泌在培养环境中)il21,从而能在自然杀伤细胞的诱导培养过程中为其提供特异性生长支持,避免mica结合nkg2d后导致的nk细胞过度激活;另外,本发明提供的重组慢病毒表达载体还具有表达41bbl基因的功能,进而能使宿主细胞起到延长nk细胞增殖时间,避免nk细胞凋亡的作用。
本发明还提供了上述重组慢病毒表达载体或者重组细胞在自然杀伤细胞培养中的应用。利用本发明提供的应用方法,既可以避免外周血单个核细胞对白介素的过分依赖,又能实现外周血单个核细胞向自然杀伤细胞的高效诱导。
附图说明
图1为本发明所述重组慢病毒表达载体的结构图;
图2为本发明所述重组慢病毒表达载体中21mb基因的结构图;
图3为本发明实施例1所述流式细胞术鉴定mlg滋养细胞中的蛋白表达结果;
图4为本发明实施例1所述不同mlg滋养细胞加入比例对培养获得的nk细胞纯度影响结果;
图5为本发明实施例1所述mlg滋养细胞处理后的nk细胞在体外细胞杀伤实验中的表现结果;
图6为本发明实施例1所述mlg滋养细胞培养nk细胞的结果数据图。
具体实施方式
本发明提供了一种重组慢病毒表达载体,所述重组慢病毒表达载体以慢病毒载体为基础,包括依次串联的21mb基因、41bbl基因和mica基因;所述21mb基因由signalpeptide基因、il21基因、cd8hinge基因和cd8transmembrane基因依次串联得到。
本发明所述重组慢病毒表达载体以慢病毒载体为基础,包括依次串联的21mb基因、41bbl基因和mica基因。本发明对所述慢病毒载体的类型不做特别限定,本领域常规市售产品均可。在本发明的实施例中,所述慢病毒载体为abm公司生产的lenti-mscv-puro。
本发明所述21mb基因由signalpeptide基因、il21基因、cd8hinge基因和cd8transmembrane基因依次串联得到。该结构可以使il21基因的表达具有膜锚定效应,使il21在宿主细胞的细胞膜上融合表达。在本发明中,所述signalpeptide基因的核苷酸序列优选如seqidno.5所示,其表达后的蛋白的氨基酸序列如seqidno.9所示;所述il21基因的核苷酸序列优选如seqidno.6所示,其表达后的蛋白的氨基酸序列如seqidno.10所示;所述cd8hinge基因的核苷酸序列优选如seqidno.7所示,其表达后的蛋白的氨基酸序列如seqidno.11所示;所述cd8transmembrane基因的核苷酸序列优选如seqidno.8所示,其表达后的蛋白的氨基酸序列如seqidno.12所示。所述21mb基因的核苷酸序列优选如seqidno.1所示,其表达后的蛋白的氨基酸序列如seqidno.13所示。
本发明所述重组慢病毒表达载体包括依次串联的21mb基因、41bbl基因和mica基因。在本发明中,所述41bbl基因的核苷酸序列优选如seqidno.2所示,其表达后的蛋白的氨基酸序列如seqidno.14所示。所述mica基因的核苷酸序列优选如seqidno.3所示,其表达后的蛋白的氨基酸序列如seqidno.15所示。在本发明中,所述21mb基因和所述41bbl基因优选通过t2a基因进行串联,所述t2a基因的核苷酸序列优选如seqidno.4所示,其表达后的蛋白的氨基酸序列如seqidno.16所示。在本发明中,所述41bbl基因和所述mica基因也优选通过t2a基因进行串联。
本发明提供了一种重组细胞,所述重组细胞由上述重组慢病毒表达载体感染宿主细胞获得。在本发明中,所述宿主细胞优选包括人白血病细胞系k562。本发明对所述感染的方法不作特别限定,本领域常规用于慢病毒载体感染宿主细胞的方法均可。利用所述重组慢病毒表达载体感染宿主细胞后,本发明优选将感染后的宿主细胞置于含有puromycin的抗性培养基中,利用重组慢病毒表达载体对puromycin的抗性,筛选得到具有高表达能力的重组细胞。
本发明还提供了上述重组慢病毒表达载体或者重组细胞在自然杀伤细胞培养中的应用。
在本发明中,所述应用优选包括如下步骤:
(1)在rpmi-1640培养基中加入8~12%的fbs和800~1200iu/ml的il-2,得到用于外周血单个核细胞的诱导培养基;
(2)将上述重组细胞与外周血单个核细胞按照1.5~2.5:1的比例加入到步骤(1)所述诱导培养基中,培养得到自然杀伤细胞。
本发明先在rpmi-1640培养基中加入fbs和il-2,配制诱导培养基。在本发明中,所述rpmi-1640培养基优选购买自gibco公司,货号为11875。在本发明中,所述fbs的添加量优选为8~12%,更优选为10%;所述il-2的添加量优选为800~1200iu/ml,更优选为1000iu/ml。本发明对所述fbs和所述il-2的来源不作特别限定,本领域常规市售产品即可。在本发明的实施例中,所述fbs购买自gibco公司,货号为10437。所述il-2购买自双鹭药业,商品名:欣吉尔;规格:100万单位每支。
得到诱导培养基后,本发明将上述重组细胞与外周血单个核细胞同时加入到步骤(1)所述诱导培养基中,培养得到自然杀伤细胞。在本发明中,所述重组细胞与所述外周血单个核细胞的数量比优选为1.5~2.5:1,更优选为2:1。
下面结合实施例对本发明提供的一种重组慢病毒表达载体、重组细胞及其在自然杀伤细胞培养中的应用进行详细的说明,但是不能把它们理解为对本发明保护范围的限定。
实施例1
1,mlg(membraneligandgrowthbooster跨膜配基生长支持体)滋养细胞构建:
人白血病细胞系k562(americantypeculturecollection;rockville,md)通过含10%fbs的rpmi-1640培养。通过将“膜锚定”il21(il-21与cd8融合)、41bbl和mica共同构建于慢病毒载体,进行病毒包装。病毒包装过程包括:通过悬浮293t表达体系,以脂质体或聚合物转染试剂(如pei25k,polyscience;transdux,sbi等)瞬时转染plenti-mlg慢病毒骨架质粒与慢病毒包装辅助质粒,然后待细胞表达慢病毒并分泌于细胞培养上清中。浓缩收获病毒上清,获得高滴度病毒,然后按供应商指导浓度添加transdux(sbi),感染k562细胞,从而构建得到k562-mbl21-41bbl-mica滋养细胞。载体具有抗性,经过含有puromycin(gibco)的抗性培养基筛选高表达细胞。
本发明以流式细胞术进行分选以筛选高表达细胞,结果如图3所示。图3结果表明:经流式细胞术鉴定,三种蛋白均在mlg细胞表面高表达。
本发明利用流式细胞仪对高表达的稳定转染细胞进行单细胞分离,建立稳定表达上述膜蛋白元件的滋养细胞单克隆细胞系。
2,mlg滋养细胞构建用于体外激活与扩增外周血单个核细胞
将滋养细胞离心收集并以pbs洗涤一次。取2×107个滋养细胞悬浮于2mlrpmi-1640培养基中(不含血清)。加入丝裂霉素c(购自sigma,使用浓度为50ug/ml),于37℃孵育30min以停止细胞的分裂。用完全培养基离心洗涤两次细胞沉淀,即可供激活培养外周血单个核细胞使用。
将新鲜分离或冻存的外周血单个核细胞与滋养细胞按照2:1的比例加入含有1000iu/mlil-2与10%fbs(gibco)的rpmi-1640培养基进行培养。每两天更换一次培养基(离心后弃去旧培养基,更换为含有il-2的新鲜培养基)。含有il-2的新鲜培养基,并调节细胞密度至0.5~1×106/ml。培养21天后,收获诱导成功的nk细胞。
3.本发明通过加入不同比例的mlg滋养细胞进行外周血pbmc的5天短时间培养,结果如图4所示。
由图4可知,不同滋养细胞的加入比例对培养获得的nk细胞纯度有影响。当mlg细胞加入比例为0.5倍pbmc时,诱导nk细胞效果最佳,为37.3,远大于其他比例的诱导方式。
4,体外细胞杀伤实验(验证产生nk的功能):
通过cfse染料标记k562细胞,在6孔板每孔中接种k562靶细胞2×105个,并且按照效靶比(effector:target,e:t)10:1加入培养得到的nk细胞。杀伤实验采用与扩增相同的培养基进行(含1000u/mlil-2)。在加入培养的nk细胞后4小时收集全部细胞,洗涤后加入pi染色30分钟,对cfse阳性群进行死亡细胞检测。结果如图5所示。
图5结果显示,通过mlg培养出的nk细胞比使用41bbl滋养细胞有更强的杀伤活力。导致靶细胞细胞死亡比例(即pi染色阳性比例)在mlg-nk杀伤后更高。
5.自然杀伤细胞的诱导相对于现有技术的优良效果:
常用的人外周血nk细胞培养常使用过表达41bbl基因的k562滋养细胞。本发明在此基础上进一步通过结合使用跨膜表达的il21和nkg2d配体mic-a构建了mlg滋养细胞并进行了外周血来源的nk细胞培养实验。结果如图6所示。
图6结果表明:使用mlg滋养细胞进行21天细胞培养后,cd56阳性,cd3阴性的nk细胞比例大幅增加,且比例(98.1%)远高于传统的41bbl-k562细胞。
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
序列表
<110>北京鼎成肽源生物技术有限公司
<120>一种重组慢病毒表达载体、重组细胞及其在自然杀伤细胞培养中的应用
<160>16
<170>siposequencelisting1.0
<210>1
<211>726
<212>dna
<213>人工序列(artificialsequence)
<400>1
atggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacgccgccagg60
ccgatggagaggattgtcatctgtctgatggtcatcttcttggggacactggtccacaaa120
tcaagctcccaaggtcaagatcgccacatgattagaatgcgtcaacttatagatattgtt180
gatcagctgaaaaattatgtgaatgacttggtccctgaatttctgccagctccagaagat240
gtagagacaaactgtgagtggtcagctttttcctgctttcagaaggcccaactaaagtca300
gcaaatacaggaaacaatgaaaggataatcaatgtatcaattaaaaagctgaagaggaaa360
ccaccttccacaaatgcagggagaagacagaaacacagactaacatgcccttcatgtgat420
tcttatgagaaaaaaccacccaaagaattcctagaaagattcaaatcacttctccaaaag480
atgattcatcagcatctgtcctctagaacacacggaagtgaagattccaccacgacgcca540
gcgccgcgaccaccaacaccggcgcccaccatcgcgtcgcagcccctgtccctgcgccca600
gaggcgtgccggccagcggcggggggcgcagtgcacacgagggggctggacttcgcctgt660
gatatctacatctgggcgcccttggccgggacttgtggggtccttctcctgtcactggtt720
atcacc726
<210>2
<211>762
<212>dna
<213>人工序列(artificialsequence)
<400>2
atggaatacgcctctgacgcttcactggaccccgaagccccgtggcctcccgcgccccgc60
gctcgcgcctgccgcgtactgccttgggccctggtcgcggggctgctgctgctgctgctg120
ctcgctgccgcctgcgccgtcttcctcgcctgcccctgggccgtgtccggggctcgcgcc180
tcgcccggctccgcggccagcccgagactccgcgagggtcccgagctttcgcccgacgat240
cccgccggcctcttggacctgcggcagggcatgtttgcgcagctggtggcccaaaatgtt300
ctgctgatcgatgggcccctgagctggtacagtgacccaggcctggcaggcgtgtccctg360
acggggggcctgagctacaaagaggacacgaaggagctggtggtggccaaggctggagtc420
tactatgtcttctttcaactagagctgcggcgcgtggtggccggcgagggctcaggctcc480
gtttcacttgcgctgcacctgcagccactgcgctctgctgctggggccgccgccctggct540
ttgaccgtggacctgccacccgcctcctccgaggctcggaactcggccttcggtttccag600
ggccgcttgctgcacctgagtgccggccagcgcctgggcgtccatcttcacactgaggcc660
agggcacgccatgcctggcagcttacccagggcgccacagtcttgggactcttccgggtg720
acccccgaaatcccagccggactcccttcaccgaggtcggaa762
<210>3
<211>1149
<212>dna
<213>人工序列(artificialsequence)
<400>3
atggggctgggcccggtcttcctgcttctggctggcatcttcccttttgcacctccggga60
gctgctgctgagccccacagtcttcgttataacctcacggtgctgtcctgggatggatct120
gtgcagtcagggtttctcactgaggtacatctggatggtcagcccttcctgcgctgtgac180
aggcagaaatgcagggcaaagccccagggacagtgggcagaagatgtcctgggaaataag240
acatgggacagagagaccagagacttgacagggaacggaaaggacctcaggatgaccctg300
gctcatatcaaggaccagaaagaaggcttgcattccctccaggagattagggtctgtgag360
atccatgaagacaacagcaccaggagctcccagcatttctactacgatggggagctcttc420
ctctcccaaaacctggagactaaggaatggacaatgccccagtcctccagagctcagacc480
ttggccatgaacgtcaggaatttcttgaaggaagatgccatgaagaccaagacacactat540
cacgctatgcatgcagactgcctgcaggaactacggcgatatctaaaatccggcgtagtc600
ctgaggagaacagtgccccccatggtgaatgtcacccgcagcgaggcctcagagggcaac660
attaccgtgacatgcagggcttctggcttctatccctggaatatcacactgagctggcgt720
caggatggggtatctttgagccacgacacccagcagtggggggatgtcctgcctgatggg780
aatggaacctaccagacctgggtggccaccaggatttgccaaggagaggagcagaggttc840
acctgctacatggaacacagcgggaatcacagcactcaccctgtgccctctgggaaagtg900
ctggtgcttcagagtcattggcagacattccatgtttctgctgttgctgctgctgctatt960
tttgttattattattttctatgtccgttgttgtaagaagaaaacatcagctgcagagggt1020
ccagagctcgtgagcctgcaggtcctggatcaacacccagttgggacgagtgaccacagg1080
gatgccacacagctcggatttcagcctctgatgtcagatcttgggtccactggctccact1140
gagggcgcc1149
<210>4
<211>54
<212>dna
<213>人工序列(artificialsequence)
<400>4
gagggcagaggaagtcttctaacatgcggtgacgtggaggagaatcccggccct54
<210>5
<211>63
<212>dna
<213>人工序列(artificialsequence)
<400>5
atggccttaccagtgaccgccttgctcctgccgctggccttgctgctccacgccgccagg60
ccg63
<210>6
<211>465
<212>dna
<213>人工序列(artificialsequence)
<400>6
atggagaggattgtcatctgtctgatggtcatcttcttggggacactggtccacaaatca60
agctcccaaggtcaagatcgccacatgattagaatgcgtcaacttatagatattgttgat120
cagctgaaaaattatgtgaatgacttggtccctgaatttctgccagctccagaagatgta180
gagacaaactgtgagtggtcagctttttcctgctttcagaaggcccaactaaagtcagca240
aatacaggaaacaatgaaaggataatcaatgtatcaattaaaaagctgaagaggaaacca300
ccttccacaaatgcagggagaagacagaaacacagactaacatgcccttcatgtgattct360
tatgagaaaaaaccacccaaagaattcctagaaagattcaaatcacttctccaaaagatg420
attcatcagcatctgtcctctagaacacacggaagtgaagattcc465
<210>7
<211>135
<212>dna
<213>人工序列(artificialsequence)
<400>7
accacgacgccagcgccgcgaccaccaacaccggcgcccaccatcgcgtcgcagcccctg60
tccctgcgcccagaggcgtgccggccagcggcggggggcgcagtgcacacgagggggctg120
gacttcgcctgtgat135
<210>8
<211>63
<212>dna
<213>人工序列(artificialsequence)
<400>8
atctacatctgggcgcccttggccgggacttgtggggtccttctcctgtcactggttatc60
acc63
<210>9
<211>21
<212>prt
<213>人工序列(artificialsequence)
<400>9
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargpro
20
<210>10
<211>155
<212>prt
<213>人工序列(artificialsequence)
<400>10
metgluargilevalilecysleumetvalilepheleuglythrleu
151015
valhislysserserserglnglyglnasparghismetileargmet
202530
argglnleuileaspilevalaspglnleulysasntyrvalasnasp
354045
leuvalproglupheleuproalaprogluaspvalgluthrasncys
505560
glutrpseralaphesercyspheglnlysalaglnleulysserala
65707580
asnthrglyasnasngluargileileasnvalserilelyslysleu
859095
lysarglysproproserthrasnalaglyargargglnlyshisarg
100105110
leuthrcysprosercysaspsertyrglulyslysproprolysglu
115120125
pheleugluargphelysserleuleuglnlysmetilehisglnhis
130135140
leuserserargthrhisglysergluaspser
145150155
<210>11
<211>45
<212>prt
<213>人工序列(artificialsequence)
<400>11
thrthrthrproalaproargproprothrproalaprothrileala
151015
serglnproleuserleuargproglualacysargproalaalagly
202530
glyalavalhisthrargglyleuaspphealacysasp
354045
<210>12
<211>21
<212>prt
<213>人工序列(artificialsequence)
<400>12
iletyriletrpalaproleualaglythrcysglyvalleuleuleu
151015
serleuvalilethr
20
<210>13
<211>242
<212>prt
<213>人工序列(artificialsequence)
<400>13
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargprometgluargilevalilecysleumetvalile
202530
pheleuglythrleuvalhislysserserserglnglyglnasparg
354045
hismetileargmetargglnleuileaspilevalaspglnleulys
505560
asntyrvalasnaspleuvalproglupheleuproalaprogluasp
65707580
valgluthrasncysglutrpseralaphesercyspheglnlysala
859095
glnleulysseralaasnthrglyasnasngluargileileasnval
100105110
serilelyslysleulysarglysproproserthrasnalaglyarg
115120125
argglnlyshisargleuthrcysprosercysaspsertyrglulys
130135140
lysproprolysglupheleugluargphelysserleuleuglnlys
145150155160
metilehisglnhisleuserserargthrhisglysergluaspser
165170175
thrthrthrproalaproargproprothrproalaprothrileala
180185190
serglnproleuserleuargproglualacysargproalaalagly
195200205
glyalavalhisthrargglyleuaspphealacysaspiletyrile
210215220
trpalaproleualaglythrcysglyvalleuleuleuserleuval
225230235240
ilethr
<210>14
<211>254
<212>prt
<213>人工序列(artificialsequence)
<400>14
metglutyralaseraspalaserleuaspproglualaprotrppro
151015
proalaproargalaargalacysargvalleuprotrpalaleuval
202530
alaglyleuleuleuleuleuleuleualaalaalacysalavalphe
354045
leualacysprotrpalavalserglyalaargalaserproglyser
505560
alaalaserproargleuarggluglyprogluleuserproaspasp
65707580
proalaglyleuleuaspleuargglnglymetphealaglnleuval
859095
alaglnasnvalleuleuileaspglyproleusertrptyrserasp
100105110
proglyleualaglyvalserleuthrglyglyleusertyrlysglu
115120125
aspthrlysgluleuvalvalalalysalaglyvaltyrtyrvalphe
130135140
pheglnleugluleuargargvalvalalaglygluglyserglyser
145150155160
valserleualaleuhisleuglnproleuargseralaalaglyala
165170175
alaalaleualaleuthrvalaspleuproproalasersergluala
180185190
argasnseralapheglypheglnglyargleuleuhisleuserala
195200205
glyglnargleuglyvalhisleuhisthrglualaargalaarghis
210215220
alatrpglnleuthrglnglyalathrvalleuglyleupheargval
225230235240
thrprogluileproalaglyleuproserproargserglu
245250
<210>15
<211>383
<212>prt
<213>人工序列(artificialsequence)
<400>15
metglyleuglyprovalpheleuleuleualaglyilepheprophe
151015
alaproproglyalaalaalagluprohisserleuargtyrasnleu
202530
thrvalleusertrpaspglyservalglnserglypheleuthrglu
354045
valhisleuaspglyglnpropheleuargcysaspargglnlyscys
505560
argalalysproglnglyglntrpalagluaspvalleuglyasnlys
65707580
thrtrpasparggluthrargaspleuthrglyasnglylysaspleu
859095
argmetthrleualahisilelysaspglnlysgluglyleuhisser
100105110
leuglngluileargvalcysgluilehisgluaspasnserthrarg
115120125
serserglnhisphetyrtyraspglygluleupheleuserglnasn
130135140
leugluthrlysglutrpthrmetproglnserserargalaglnthr
145150155160
leualametasnvalargasnpheleulysgluaspalametlysthr
165170175
lysthrhistyrhisalamethisalaaspcysleuglngluleuarg
180185190
argtyrleulysserglyvalvalleuargargthrvalpropromet
195200205
valasnvalthrargserglualasergluglyasnilethrvalthr
210215220
cysargalaserglyphetyrprotrpasnilethrleusertrparg
225230235240
glnaspglyvalserleuserhisaspthrglnglntrpglyaspval
245250255
leuproaspglyasnglythrtyrglnthrtrpvalalathrargile
260265270
cysglnglyglugluglnargphethrcystyrmetgluhissergly
275280285
asnhisserthrhisprovalproserglylysvalleuvalleugln
290295300
serhistrpglnthrphehisvalseralavalalaalaalaalaile
305310315320
phevalileileilephetyrvalargcyscyslyslyslysthrser
325330335
alaalagluglyprogluleuvalserleuglnvalleuaspglnhis
340345350
provalglythrserasphisargaspalathrglnleuglyphegln
355360365
proleumetseraspleuglyserthrglyserthrgluglyala
370375380
<210>16
<211>18
<212>prt
<213>人工序列(artificialsequence)
<400>16
gluglyargglyserleuleuthrcysglyaspvalglugluasnpro
151015
glypro
- 还没有人留言评论。精彩留言会获得点赞!