人体组织/细胞标本环状RNA的内参基因has_circ_0000284及其应用的制作方法
本发明属于分子生物学技术领域,具体涉及一种人体组织/细胞标本环状rna的rt-qpcr内参基因has_circ_0000284及其引物和应用。
背景技术:
环状rna(circularrnas,circrnas)是一类以共价键形成的单链闭合环形结构的分子,没有5’末端帽子和3’末端poly(a)尾巴,结构相对稳定,不受核糖核酸酶r(rnaser)的影响。1976年人类就已经发现环状rna分子的存在,但一直被视为是错误的剪切产物,或是逃脱内含子套索脱枝的中介物。近年来,随着高通量测序技术的发展和分析方法的更新,大量的环状rna分子被鉴定出来。越来越多的研究表明环状rna分子并不是基因剪切副产物,而是具有一定功能的分子,主要功能包括竞争性吸附mirna、结合蛋白而影响蛋白的功能、翻译成蛋白、竞争mrna的线性剪切、调控rna聚合酶ⅱ活性、以及作为疾病的生物标志物。
rt-qpcr作为一种检测rna的敏感方法已被广泛应用于基因的表达分析。在利用rt-qpcr技术检测目的基因的表达水平时,为了减少检测样本在rna产量、质量和反转录效率上可能存在的差异,需要内参基因进行标准化和校正。内参基因在很大程度上决定了rt-qpcr结果的准确性,所以找到可以在所有样本和实验条件下稳定表达的基因作为内参基因是rt-qpcr技术正确实施的关键。
目前,在检测环状rna表达水平时,研究人员仍使用线性的β-actin或gapdh作为内参基因。由于环状rna和其亲本线性基因有相同的序列,为了防止亲本线性基因的干扰,需要为环状rna设计背靠背引物(divergentprimer)。虽然背靠背引物在一定程度上排除了亲本线性基因的干扰,但是无法排除与环状rna有相同接头(back-splicejunction)序列的线性剪切异构体的干扰。最佳的做法是用rnaser来去除这些线性剪切异构体的干扰,然而这也会去除作为内参基因的β-actin或gapdh,所以无法再使用这些线性内参基因来校正目的基因的表达。而将rna等分为二,一份用rnaser处理用于环状rna的扩增,另外一份用于内参基因的扩增,再进行校正的做法会受rnaser缓冲液中mg2+的干扰。因此,在环状rna的研究领域迫切需要开发能够作为环状rna标准化的内参基因。
技术实现要素:
本发明的目的是针对上述现有技术的不足,提供一种人体组织/细胞标本环状rna的rt-qpcr内参基因hsa_circ_0000284及其引物和应用,该内参基因hsa_circ_0000284本身也是环状rna,可耐受rnaser的处理,克服了现有技术环状rna检测中rnaser处理后无内参的难题。
本发明的第一个目的是提供人体组织/细胞标本环状rna的rt-qpcr内参基因,其序列如seqidno.1所示。
本发明的第二个目的是提供上述人体组织/细胞标本环状rna的rt-qpcr内参基因hsa_circ_0000284的特异性引物,其正向引物和反向引物分别如seqidno.2和seqidno.3所示。
本发明的第三个目的是提供上述内参基因或特异性引物在制备rt-qpcr试剂盒中的应用。
进一步地,所述试剂盒还包括常规pcr试剂。
上述内参基因hsa_circ_0000284作为人体组织/细胞标本环状rna的rt-qpcr内参基因的应用。
上述特异性引物作为人体组织/细胞标本环状rna的rt-qpcr特异性引物的应用。
本发明提供了hsa_circ_0000284作为人体组织/细胞标本环状rna基因扩增的内参基因,因为hsa_circ_0000284基因本身也是环状rna,所以耐受rnaser的处理,攻克了环状rna检测中rnaser处理后无内参的难题。本发明首先在高通量测序结果中筛选,然后利用rt-qpcr在人体各种细胞中验证,证实hsa_circ_0000284及其引物用于人体相对表达分析的准确性和稳定性,并且其表达水平不受化疗药物、缺氧等实验条件的影响。hsa_circ_0000284可以作为单独的内参基因,无需与其他内参基因组合即可使用。
附图说明
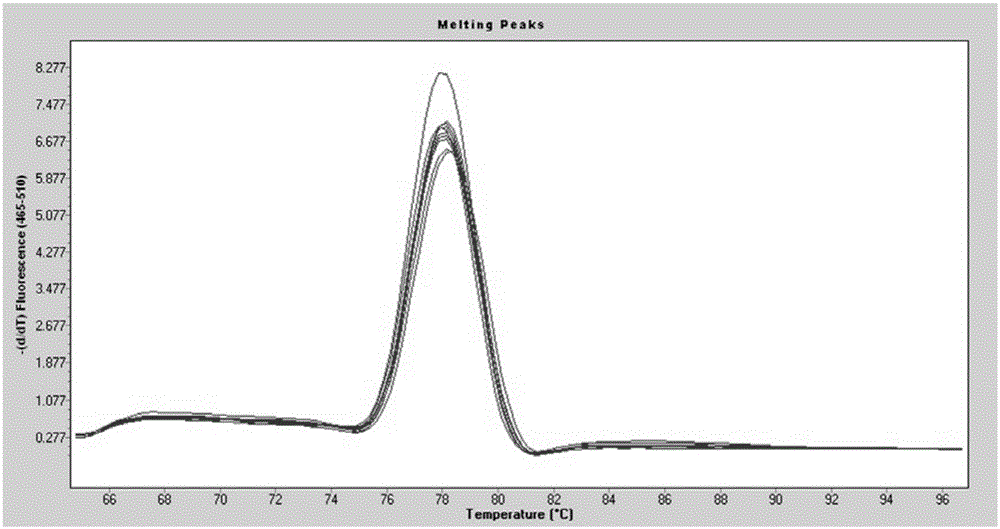
图1a为实施例2中内参基因hsa_circ_0000284的溶解曲线。
图1b为实施例2中所有候选内参基因的电泳图,其中:m为marker、1为hsa_circ_0000471、2为hsa_circ_0000284、3为hsa_circ_0002484、4为hsa_circ_0001445、5为hsa_circ_0000944、6为hsa_circ_0000567、7为gapdh、8为18srrna、9为β-actin。
具体实施方式
下面结合具体实施例对本发明的技术方案作进一步说明。
实施例1
1、筛选环状rna的候选内参基因
登录geneexpressionomnibus(geo)网站(https://www.ncbi.nlm.nih.gov/geo/)下载编号为gse77661的数据集(gse77661_circ_srpbm_union_samples_loci.txt.gz),该数据集中包括6例健康人体标本(脑、结肠、心、肝、肺和胃)和7对癌和癌旁人体组织标本(膀胱癌、乳腺癌、结直肠癌、胃癌、肝癌、透明细胞癌和前列腺癌)的环状rna测序结果。
数据处理方法如下:
(1)、只保留在所有样本中都表达的环状rna,共得到50个环状rna。
(2)、分别用genorm、normfinder和bestkeeper分析得到的50个环状rna在组织标本中的稳定性,取每一种方法稳定性最好的10个环状rna,然后将得到的3个数据集取交集,得到6个环状rna。
2、在细胞标本中进一步筛选稳定的候选内参基因
利用circprimer软件为6个候选环状rna设计rt-qpcr扩增引物,如表1所示。
表1rt-qpcr扩增所用的引物
收集表2中所示的人类肿瘤或正常细胞标本,利用rnasimpletotalrnakit按照说明书提取总rna,取500ng总rna利用thermoscientificrevertaidfirststrandcdnasynthesiskit试剂盒以随机引物为逆转录引物合成第一链cdna,稀释5倍后取1μlcdna作为模版利用light
表2筛选候选基因所用的细胞株
除了检测上述未经处理的人类细胞外,还检测了不同试剂处理后的细胞株中各个候选内参基因的表达情况。具体是分别用不同浓度的阿霉素(10、50和250nm)、氯化铵(25、50和100μm)和紫草素(5、10和15μm)处理mcf-7细胞24小时后,按照上述步骤提取rna、去线性rna、逆转录和检测。
根据所有细胞株去线性rna前后的rt-pcr扩增结果,先分析了22株未加药细胞株的rt-qpcr结果,得到了内参基因稳定性评价结果,如表3所示;然后,又分析了所有细胞株(含加药处理过的mcf-7细胞)的rt-qpcr结果,进一步得到了内参基因稳定性评价结果,如表4所示。因为genorm算法最终输出的是两个最稳定的基因,所以在本例中hsa_circ_0000284和hsa_circ_0000471最稳定。根据normfinder算法,hsa_circ_0000284和hsa_circ_0000471也是稳定性最佳的两个基因。根据bestkeeper的评价结果,表3显示未去线性的rna样本中hsa_circ_0001445稳定性最佳,然而其标准偏差和变异系数远远大于其它基因,此外在表3另外两组样本评价结果中hsa_circ_0000471最为稳定;表4结果显示,hsa_circ_0000284和hsa_circ_0000471是最为稳定的两个基因。
表3根据22株细胞的rt-qpcr结果得到的候选基因稳定性评价结果
基因按照稳定性由高到低排序
表4根据22株细胞及加药后mcf-7细胞的rt-qpcr结果得到的候选基因稳定性评价结果
基因按照稳定性由高到低排序
实施例2
hsa_circ_0000284的特异性和扩增效率评价
rt-qpcr的结果显示,hsa_circ_0000284的溶解曲线为单峰(图1a所示),说明其不存在非特异性扩增。为了进一步证实没有非特异性条带的扩增,利用2%的琼脂糖凝胶电泳扩增产物,其结果如图1b所示,未见杂带。
为了得到hsa_circ_0000284的扩增效率,首先将模板倍比稀释,然后上机扩增。得到扩增结果后,对结果进行直线拟合求得斜率(slope),然后用下列公式计算扩增效率:
e(%)=(2-1/slope-1)×100
最后求得hsa_circ_0000284的扩增效率是94.89%。
序列表
<110>江苏省肿瘤医院
<120>人体组织/细胞标本环状rna的内参基因has_circ_0000284及其应用
<130>20181221
<160>3
<170>siposequencelisting1.0
<210>1
<211>1099
<212>dna
<213>人工序列(artificialsequence)
<400>1
gtatggcctcacaagtcttggtctacccaccatatgtttatcaaactcagtcaagtgcct60
tttgtagtgtgaagaaactcaaagtagagccaagcagttgtgtattccaggaaagaaact120
atccacggacctatgtgaatggtagaaactttggaaattctcatcctcccactaagggta180
gtgcttttcagacaaagataccatttaatagacctcgaggacacaacttttcattgcaga240
caagtgctgttgttttgaaaaacactgcaggtgctacaaaggtcatagcagctcaggcac300
agcaagctcacgtgcaggcacctcagattggggcgtggcgaaacagattgcatttcctag360
aaggcccccagcgatgtggattgaagcgcaagagtgaggagttggataatcatagcagcg420
caatgcagattgtcgatgaattgtccatacttcctgcaatgttgcaaaccaacatgggaa480
atccagtgacagttgtgacagctaccacaggatcaaaacagaattgtaccactggagaag540
gtgactatcagttagtacagcatgaagtcttatgctccatgaaaaatacttacgaagtcc600
ttgattttcttggtcgaggcacgtttggccaggtagttaaatgctggaaaagagggacaa660
atgaaattgtagcaatcaaaattttgaagaatcatccttcttatgcccgtcaaggtcaaa720
tagaagtgagcatattagcaaggctcagtactgaaaatgctgatgaatataactttgtac780
gagcttatgaatgctttcagcaccgtaaccatacttgtttagtctttgagatgctggaac840
aaaacttgtatgactttctgaaacaaaataaatttagtcccctgccactaaaagtgattc900
ggcccattcttcaacaagtggccactgcactgaaaaaattgaaaagtcttggtttaattc960
atgctgatctcaagccagagaatattatgttggtggatcctgttcggcagccttacaggg1020
ttaaagtaatagactttgggtcggccagtcatgtatcaaagactgtttgttcaacatatc1080
tacaatctcggtactacag1099
<210>2
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>2
gtcggccagtcatgtatcaa20
<210>3
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>3
accaagacttgtgaggccat20
- 还没有人留言评论。精彩留言会获得点赞!