基于PAX3和ZIC4基因的肺癌诊断试剂及试剂盒的制作方法
本发明属于基因诊断领域,具体地,本发明涉及一种pax3和zic4基因在肺癌检测中的应用,以及含pax3和zic4基因的甲基化检测/诊断试剂,以及含有该试剂的试剂盒。
背景技术:
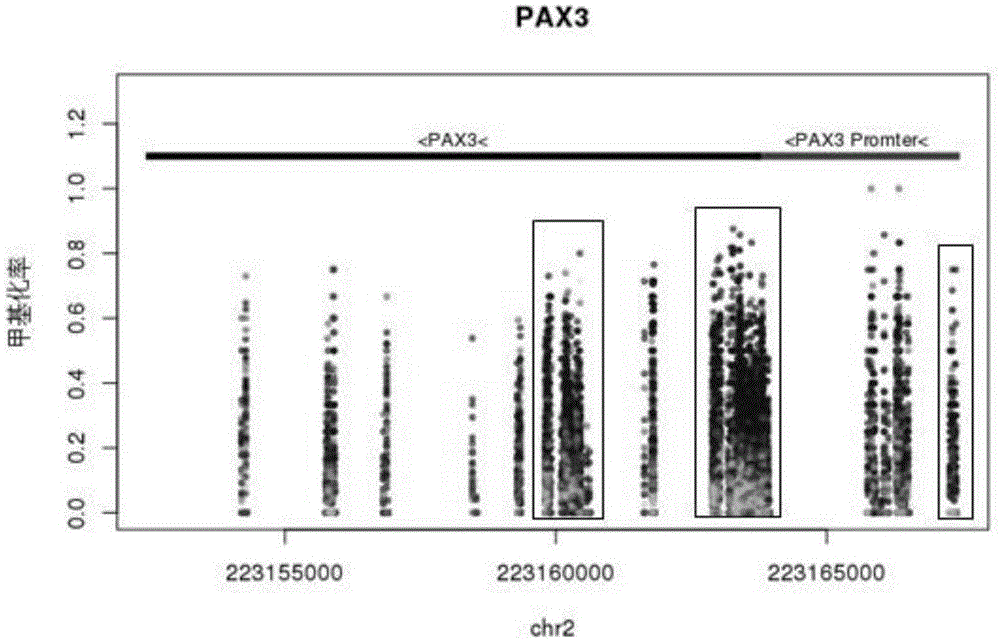
:肺癌是起源于支气管粘膜、腺体或肺泡上皮的肺部恶性肿瘤。按照病理类型可以分为:1)小细胞肺癌(smallcelllungcancer,sclc):一种特殊病理学类型的肺癌,有明显的远处转移倾向,预后较差,但多数病人对放化疗敏感;2)非小细胞肺癌(non-smallcelllungcancer,nsclc):除小细胞肺癌以外其他病理学类型的肺癌,包括鳞状细胞癌、腺癌、大细胞癌等。在生物学行为和临床病程方面具有一定差异。按照发生位置又可以分为:1)中心型肺癌(centrallungcancer):生长在肺段支气管开口及以上的肺癌;2)周围型肺癌(peripherallungcancer):生长在肺段支气管开口以远的肺癌。近年来,人口老龄化、大气污染及吸烟等因素的影响,致使中国肺癌的发病率和死亡率逐年递增,根据国家癌症中心发布的《2017中国肿瘤登记年报》显示,全国每分钟约7人确诊患癌,其中肺癌发病率与死亡率均为第一位。中国已成为世界上肺癌人数最多的国家,专家预测到2025年中国肺癌的人数将达到100万。而根据流行病学研究显示:吸烟是引起肺癌的重要因素。世界上约80%-90%的肺癌可归因于吸烟。与非吸烟者相比,45-64岁每日吸香烟1-19支和20支以上者患肺癌的相对危险度分别为4.27和8.61,与从不吸烟者比较,长期每日吸烟1-19支和20支以上者死于肺癌的相对危险度分别为6.14和10.73。虽然肺癌的治疗技术日新月异,但5年生存率从4%仅上升至12%左右,现有的抗肿瘤药物仍只能起到缓解病情作用,患者无进展生存期平均仅延长3个月~5个月,但对于ⅰ期肺癌患者,手术后5年生存率高达约60%~70%。因此肺癌的早期诊断与早期手术是提高肺癌5年生存率、降低死亡率最有效的方法之一。肺癌目前的临床辅助诊断主要有以下几种,但是他们都不能完全做到早发现,早诊断:(1)血液生化检查:对于原发性肺癌,目前无特异性血液生化检查。肺癌病人血液碱性磷酸酶或血钙升高考虑骨转移的可能,血液碱性磷酸酶、谷草转氨酶、乳酸脱氢酶或胆红素升高考虑肝转移的可能。肿瘤标志物检查:1)cea:30%~70%肺癌患者血清中有异常高水平的cea,但主要见于较晚期肺癌患者。目前血清中cea的检查主要用于估计肺癌预后以及对治疗过程的监控。2)nse:是小细胞肺癌首选标志物,用于小细胞肺癌的诊断和监测治疗反应,根据检测方法和使用试剂的不同,参考值不同。3)cyfra21-1:是非小细胞肺癌的首选标记物,对肺鳞癌诊断的敏感性可达60%,根据检测方法和使用试剂的不同,参考值不同。目前用于肺癌检测的甲基化标志物敏感性和特异性还有待提高。(2)影像学检查:1)胸部x线检查:应包括胸部正位和侧位片。在基层医院,胸部正侧位片仍是肺癌初诊时最基本和首选的影像诊断方法。一旦诊断或疑诊肺癌,即行胸部ct检查。2)ct检查:胸部ct是肺癌的最常用和最重要的检查方法,用于肺癌的诊断与鉴别诊断、分期及治疗后随诊。ct引导下肺穿刺活检是肺癌的重要诊断技术,有条件的医院可将其用于难以定性的肺内病变的诊断,以及临床诊断肺癌需经细胞学、组织学证实而其它方法又难以取材的病例。近年来,多层螺旋ct和低剂量ct(ldct)是发现早期肺癌和降低死亡率的有效筛查工具,全美国家肺癌筛查研究(nlst)已经表明ldct相比胸部x线筛查可降低20%肺癌的死亡率。低剂量螺旋ct被推荐为早期肺癌筛查的重要手段,但是人为影响因素较多,假阳性率非常高。3)超声检查:主要用于发现腹部重要器官及腹腔、腹膜后淋巴结有无转移,也用于颈部淋巴结的检查。对于贴邻胸壁的肺内病变或胸壁病变,可鉴别其囊实性及进行超声引导下穿刺活检;超声还常用于胸水抽取定位。4)骨扫描:对肺癌骨转移检出的敏感性较高,但有一定的假阳性率。可用于以下情况:肺癌的术前检查;伴有局部症状的病人。(3)其它检查:1)痰细胞学检查:目前肺癌简单方便的无创诊断方法,连续涂片检查可提高阳性率约达60%,是可疑肺癌病例的常规诊断方法。2)纤维支气管镜检查:肺癌诊断中最重要的手段之一,对于肺癌的定性定位诊断和手术方案的选择有重要的作用。对拟行手术治疗的患者为必需的常规检查项目。而经支气管镜穿刺活检检查(tbna),虽利于治疗前分期,但因技术难度和风险较大,有需要者应转上级医院进一步检查。3)其他:如经皮肺穿刺活检、胸腔镜活检、纵隔镜活检、胸水细胞学检查等,在有适应证的情况下,可根据现有条件分别采用以协助诊断。早期肺癌患者往往无明显的症状和体征,很容易被患者忽视,很少因症就诊。临床常规的胸片x光和痰脱落细胞学检查远远达不到筛查早期肺癌的要求,均未被证实能降低死亡率。胸部x线的筛查漏检率可高达54%-90%,痰脱落细胞学检查虽然成本低,不需要昂贵设备,但是漏检率高,结果判断的人为因素干扰较多,而且需要多次送检。目前,有很多研究检测血液,痰液,肺泡灌洗液中的细胞或者dna甲基化状态,以期找到用于肺癌早期诊断的标志物。尽管现有技术中已经发现了一些基因的dna甲基化于肺癌相关,但是目前本领域中仍然需要进一步研究能够切实地应用的于肺癌诊断的相关基因,以及开发出具有较高检测准确性的检测试剂。低剂量螺旋ct被推荐为早期肺癌筛查的重要手段,但是人为影响因素较多,假阳性率非常高。在临床实践工作中证明,任何肺癌筛查项目的成败取决于高危人群的识别,融合多重高危因素的风险预测模型已被世界公认是识别肺癌高危人群的方法之一。随着技术的飞速发展,肿瘤标志物检测成为继影像学诊断,病理诊断之后的肿瘤诊断治疗新领域,能够对肿瘤的诊断,检测,治疗产生重大的影响。肿瘤标志物可以在体液或者是组织中检测到,能够反映肿瘤的存在,分化程度,预后估计和个性化用药以及治疗效果等。早期肺癌患者没有明显的症状,难以被医生和患者察觉,再加之其在血液或生化项目上没有明显的特异性的标识物,因此难以通过常规的诊断方法进行早期发现和早期诊断,因此对肺癌早期诊断,尤其是大规模应用人群筛查上较为困难。越来越多的研究表明,在肿瘤形成过程中包含两大类机制。一个是通过dna核苷酸序列改变而形成突变,即遗传学机制。肿瘤作为一种遗传学疾病在分子生物学领域已经得到证实。另外一个就是表观遗传学(epigenetics)机制,即不依赖dna序列改变导致基因表达水平的变化,它在肿瘤形成过程中的作用越来越受到重视。遗传学与表观遗传学两种机制相互交叉存在,共同促进了肿瘤的形成。基因的异常甲基化在肿瘤发生的早期就可出现,并且在肿瘤逐步发展的过程中,基因异常甲基化的程度增加。对常见的98种人类原发肿瘤的基因组进行分析,发现每种肿瘤至少有600个异常甲基化的cpg岛。许多研究显示启动子异常甲基化在许多肿瘤的发生过程中是一个频发的早期事件,因此肿瘤相关基因的甲基化状态是肿瘤发生的一个早期敏感指标,被认为是一种有前景的肿瘤分子生物标志物(biomarker)。更为重要的是,癌变细胞可以释放dna到外周血中。正常人外周血中都存在纳克级的游离dna。研究发现外周血血浆/血清、肿瘤累及器官相关的体液(如唾液、痰等)中同样可以检测到肿瘤组织中存在的肿瘤相关基因的启动子异常甲基化。这些生物样品比较容易获得,并且通过pcr技术将其中的dna进行大量的扩增后能够很灵敏的检测到,因此检测其中某些肿瘤相关基因的启动子区甲基化状态,可以为肿瘤的早期诊断提供非常有价值的信息。与其它类型的肿瘤分子标记物相比,检测启动子异常甲基化有更多的优点。某一基因在不同类型肿瘤中,其启动子异常甲基化的区域是相同的,检测比较方便;另外与等位基因缺失这样的标志物相比,异常甲基化是一个阳性信号,很容易与正常组织中的阴性背景区分。esteller等检测了22例非小细胞肺癌(nsclc)的肿瘤组织和血清中p16、dapk、gstp1及mgmt等基因的启动子区异常甲基化状态,发现68%(15/22)的肿瘤组织中存在至少一种基因的启动子甲基化;而在15例组织阳性。病例中,有11例同时在血清中也检测到了启动子异常甲基化的存在。另有许多研究者也分别从肝癌、头颈部癌、食管癌及结肠癌患者的肿瘤组织和血清中同时检测出了某些肿瘤相关基因的启动子甲基化。palmisano等检测了21例肺鳞癌患者的肿瘤组织和痰液中p16、mgmt启动子异常甲基化情况,发现所有痰样品中都存在一种或两种基因的启动子区异常甲基化。其中10例痰样是在肿瘤确诊后采集的;另11例痰样来自有吸烟史或其它暴露的高危人群,这11例监测对象在随后5~35月都被确诊为肺癌。而这21例痰样经痰细胞形态学检验为阳性的仅有4例。因此检测基因启动子区异常甲基化是一个非常敏感的指标。这些研究结果表明:dna甲基化的检测可以作为癌症的早期症断和风险评估的手段。技术实现要素:本发明的目的在于提供一种pax3、zic4基因或它们的核酸片段在制备肿瘤检测/诊断试剂或试剂盒中的应用。本发明的另一个目的在于提供一种引物组合物在制备肿瘤检测/诊断试剂或试剂盒中的应用。本发明的另一个目的在于提供一种探针组合物在制备肿瘤检测/诊断试剂或试剂盒中的应用。本发明的进一步目的在于提供一种诊断人pax3、zic4基因甲基化的试剂、试剂盒和方法。本发明的进一步目的在于提供一种特异性强、敏感度高的肺癌检测/诊断试剂和试剂盒。本发明的进一步目的在于提供一种对肺癌应用范围广的肺癌检测/诊断试剂和试剂盒。本发明的进一步目的在于提供一种使用方便的肺癌检测/诊断试剂和试剂盒。本发明的上述目的通过以下技术手段实现:一方面,本发明提供了一种pax3基因或其核酸片段,以及zic4基因或其核酸片段在制备肿瘤检测/诊断试剂或试剂盒中的应用。pax3基因(pairedboxgene),pax3基因,配对盒子基因成员3,属于配对盒子基因家族成员,位于人类2号染色体。pax3基因是一类重要的转录调控因子,在胚胎发育过程中对组织和器官的分化起着重要的调控作用。已有研究表明pax3基因突变或表达失调会引起某些神经系统相关的出生缺陷和遗传综合征。zic4基因,锌指家族基因成员4(zicfamilymember4),位于人类3号染色体。锌指家族基因在心脏和神经的发育过程中起着重要作用,已有研究报道先天脑部异常dwm(dandy-walkermalformation)与缺乏zic1和zic4基因的单一拷贝有关。目前,未见将pax3基因和zic4基因联合作为肺癌的肿瘤标志物。本发明首次基于pax3以及zic4基因,或他们核酸片段,检测肺癌。作为优选的实施方式,进一步的,pax3基因的核酸片段选自seqidno:22、seqidno:24或seqidno:26;更为优选seqidno:22。zic4基因的核酸片段选自seqidno:28、seqidno:30、seqidno:32、seqidno:34或seqidno:36,更为优选,seqidno:28。另一方面,本发明还提供了一种引物组合,所述的引物含有引物对a和引物对b;所述引物对a选自以下的任意一对:seqidno:1和seqidno:2,seqidno:40和seqidno:41,seqidno:43和seqidno:44,seqidno:46和seqidno:47,seqidno:49和seqidno:50,seqidno:52和seqidno:53,seqidno:55和seqidno:56,seqidno:58和seqidno:59,seqidno:61和seqidno:62,seqidno:64和seqidno:65,seqidno:67和seqidno:68,seqidno:70和seqidno:71;所述引物对b选自以下的任意一对:seqidno:4和seqidno:5,seqidno:73和seqidno:74,seqidno:76和seqidno:77,seqidno:79和seqidno:80,seqidno:82和seqidno:83,seqidno:85和seqidno:86,seqidno:88和seqidno:89,seqidno:91和seqidno:92,seqidno:94和seqidno:95,seqidno:97和seqidno:98,seqidno:100和seqidno:101,seqidno:103和seqidno:104,seqidno:106和seqidno:107,seqidno:109和seqidno:110,seqidno:112和seqidno:113;优选地,所述的引物对a选自以下的任意一对:seqidno:1和seqidno:2,seqidno:40和seqidno:41,seqidno:43和seqidno:44,seqidno:46和seqidno:47,seqidno:49和seqidno:50,seqidno:67和seqidno:68;所述的引物对b选自以下的任意一对:seqidno:4和seqidno:5,seqidno:73和seqidno:74,seqidno:76和seqidno:77,seqidno:79和seqidno:80,seqidno:100和seqidno:101,seqidno:112和seqidno:113;更优选地,所述的引物对a选自:seqidno:1和seqidno:2,所述的引物对b选自:seqidno:4和seqidno:5。其中,引物a可用用以扩增pax3基因的核酸片段,引物b可以高效扩增zic4基因的核酸片段。另一方面,本发明还提供了一种核酸探针组合,所述的探针组合含有探针c和探针d;所述探针c选自以下任一序列:seqidno:3、seqidno:42,seqidno:45,seqidno:48,seqidno:51,seqidno:54,seqidno:57,seqidno:60,seqidno:63,seqidno:66,seqidno:69,seqidno:72;所述探针d选自以下任一序列:seqidno:6,seqidno:75,seqidno:78,seqidno:81,seqidno:84,seqidno:87,seqidno:90,seqidno:93,seqidno:96,seqidno:99,seqidno:102,seqidno:105,seqidno:108,seqidno:111,seqidno:114;优选地,所述探针c选自以下任一序列:seqidno:3、seqidno:42,seqidno:45,seqidno:48,seqidno:51,seqidno:69;所述探针d选自以下任一序列:seqidno:6,seqidno:75,seqidno:78,seqidno:81,seqidno:102,seqidno:114;更优选地,所述探针c选自seqidno:3,所述探针d选自seqidno:6。其中,探针c可以特异性结合pax3基因的核酸片段,探针d可以特异性结合zic4基因的核酸片段。另一方面,本发明还提供了上述的引物或核酸探针在制备肿瘤检测/诊断试剂或试剂盒中的应用。通过上述的引物和探针,基于msp,可以检测pax3和zic4基因或它们的核酸片段的甲基化情况。另一方面,本发明还提供了一种肿瘤检测/诊断试剂,所述的试剂含有所述的试剂含有pax3基因和zic4基因甲基化的检测试剂。进一步地,该试剂检测的是经亚硫酸氢盐或重亚硫酸氢盐或肼盐修饰后的序列。甲基化的发生是胞嘧啶上多了一个甲基,经过经亚硫酸氢盐或重亚硫酸氢盐或肼盐处理后,胞嘧啶会变成脲嘧啶,因为在进行pcr扩增时尿嘧啶与胸腺嘧啶相似而会被识别为胸腺嘧啶,体现在pcr扩增序列上就是没有发生甲基化的胞嘧啶变成了胸腺嘧啶(c变成t),甲基化的胞嘧啶(c)则不会发生变化。真核生物中dna甲基化主要发生于cpg二核苷酸中的c,并主要以5-甲基胞嘧啶(m5c)形式存在。cpg双核苷酸在人类基因组中的分布很不均一,在基因组的部分区域,cpg保持或高于正常概率。cpg岛主要位于基因的启动子和第一外显子,是富含cpg二核苷酸的区域,长度为300-3000bp。pcr检测甲基化基因的技术通常为甲基化特异性pcr(methylmionspecificpcr,msp),针对处理后的甲基化片段(即片段中未改变的c)设计引物,进行pcr扩增,如果有扩增则说明发生了甲基化,无扩增则没有甲基化。作为优选的实施方式,所述试剂针对pax3基因的检测区域如seqidno:22、seqidno:24或seqidno:26所示;更优选地,如seqidno:22所示;所述试剂针对zic4基因的检测区域如seqidno:28、seqidno:30、seqidno:32、seqidno:34或seqidno:36;更优选地,如seqidno:28所示。检测区域的选择会影响发明人经实验发现,pax3和zic4基因检测区域的选择,会对肿瘤的检测效能产生影响。针对本发明涉及的pax3基因,发明人进行了rrbs甲基化测序,得到了pax3基因自身以及该基因上游5000bp内的碱基甲基化情况。经过初步的分析能够明显的发现该基因的不同区域的甲基化情况在肺癌和非肺癌对照组中明显的不同。针对zic4基因,发明人进行了rrbs甲基化测序,得到了zic4基因自身以及该基因上游5000bp内的碱基甲基化情况。经过初步的分析能够明显的发现该基因的不同区域的甲基化情况在肺癌和非肺癌对照组中明显的不同,因此选择不同的区域设计引物和探针对肺癌和非肺癌的诊断具有重要的影响。如图1和图2所示。本发明的试剂中,含有扩增pax3和zic4基因引物。作为优选的实施方式,检测pax3基因的引物选自seqidno:1和seqidno:2,seqidno:40和seqidno:41,seqidno:43和seqidno:44,seqidno:46和seqidno:47,seqidno:49和seqidno:50,seqidno:52和seqidno:53,seqidno:55和seqidno:56,seqidno:58和seqidno:59,seqidno:61和seqidno:62,seqidno:64和seqidno:65,seqidno:67和seqidno:68,seqidno:70和seqidno:71中的任意一对;更优选地,选自seqidno:1和seqidno:2,seqidno:40和seqidno:41,seqidno:43和seqidno:44,seqidno:46和seqidno:47,seqidno:49和seqidno:50,seqidno:67和seqidno:68所示的引物对;最优选地,选自:seqidno:1和seqidno:2所示的引物对。检测zic4基因的引物选自seqidno:4和seqidno:5,seqidno:73和seqidno:74,seqidno:76和seqidno:77,seqidno:79和seqidno:80,seqidno:82和seqidno:83,seqidno:85和seqidno:86,seqidno:88和seqidno:89,seqidno:91和seqidno:92,seqidno:94和seqidno:95,seqidno:97和seqidno:98,seqidno:100和seqidno:101,seqidno:103和seqidno:104,seqidno:106和seqidno:107,seqidno:109和seqidno:110,seqidno:112和seqidno:113中的任意一对;更优选地,选自seqidno:4和seqidno:5,seqidno:73和seqidno:74,seqidno:76和seqidno:77,seqidno:79和seqidno:80,seqidno:100和seqidno:101,seqidno:112和seqidno:113所示的引物对;最优选地,选自:seqidno:4和seqidno:5所示的引物对。所述的引物用于扩增pax3和zic4基因的特定区域。本领域公知,引物的成功设计对于pcr是至关重要。相对于一般的pcr,在基因的甲基化检测中,引物的设计影响更为关键,这是由于甲硫化反应促使dna链中的“c”转化为“u”,导致gc含量降低,使pcr反应后在序列中出现长的连续“t”,容易引起dna链的断裂,导致很难选择具有合适的tm值及稳定的引物;另一方面,为了区别硫化处理和没有硫化处理以及未完全处理的dna,需要引物有足够数量的“c”,这些都增加了选择稳定引物的困难。因此,dna甲基化检测中,引物所针对的扩增片段的选择,如扩增片段长短和位置,以及引物的选择等等都对检测的灵敏度和特异性产生影响。发明人经实验也发现,不同的扩增目的片段和引物对检测效果有所区别。很多时候,发现了某些基因或核酸片段在肿瘤和非肿瘤中具有表达差异,然而其距离转化为肿瘤的标志物,应用到临床中,仍存在很长的距离。其中最主要的原因是因为检测试剂的限制,导致该潜在肿瘤标志物的检测灵敏度和特异性难以满足检测需求,或者检测方法操作复杂、成本高,难以在临床中大规模应用。作为一种优选的实施方式,本发明的试剂中,还含有特异性结合pax3和zic4基因的探针。进一步地,检测pax3基因的探针选自seqidno:3、seqidno:42,seqidno:45,seqidno:48,seqidno:51,seqidno:54,seqidno:57,seqidno:60,seqidno:63,seqidno:66,seqidno:69,seqidno:72中的任一序列;更优选地,选自seqidno:3、seqidno:42,seqidno:45,seqidno:48,seqidno:51,seqidno:69中的任一序列;最优选地,选自seqidno:3。检测zic4基因的探针选自seqidno:6,seqidno:75,seqidno:78,seqidno:81,seqidno:84,seqidno:87,seqidno:90,seqidno:93,seqidno:96,seqidno:99,seqidno:102,seqidno:105,seqidno:108,seqidno:111,seqidno:114中的任一序列;更优选地,选自seqidno:6,seqidno:75,seqidno:78,seqidno:81,seqidno:102,seqidno:114中的任一序列;最优选地,选自seqidno:6。作为优选的实施方式,所述的试剂还包括内参基因的检测试剂;,所述的内参基因为β-actin、col2a1。进一步地,所述的内参基因的检测试剂为针对内参基因的引物和探针。作为优选的实施方式,所述的内参基因β-actin的检测试剂为seqidno:19、seqidno:20所示的引物对和seqidno:21的探针。所述的内参基因col2a1的检测试剂为seqidno:115和seqidno:116所示的引物对,seqidno:117的探针。作为优选的实施方式,所述的试剂还包括亚硫酸氢盐、重亚硫酸氢盐或肼盐,以对pax3基因进行修饰,当然也可以不包括。作为优选的实施方式,所述的试剂含包括dna聚合酶、dntps、mg2+离子、缓冲液中的一种或几种,优选包括dna聚合酶、dntps、mg2+离子和缓冲液的pcr反应体系,用于对经修饰后的pax3基因和zic4基因扩增。另一方面,本发明还提供了一种检测pax3基因和zic4基因的dna甲基化的方法,其特征在于,包括以下步骤:(1)将待测样品进行亚硫酸氢盐或重亚硫酸氢盐或肼盐处理,获得经修饰的待测样品;(2)利用上述的试剂或试剂盒对步骤(1)经修饰的待测样品进行pax3基因和zic4基因甲基化情况检测;步骤(2)中,采用实时荧光定量甲基化特异性聚合酶链反应进行检测。另一方面,本发明还提供了一种肺癌的检测/诊断系统,其特征在于,所述的系统含有:a.pax3基因和zic4基因的dna甲基化检测构件,以及,b.结果判断系统;所述的pax3基因和zic4基因的dna甲基化检测构件含有上述任一所述试剂或试剂盒;所述的结果判断构件用于根据检测系统检测的pax3基因和zic4基因的dna甲基化结果,输出肺癌的患病风险和/或肺癌类型;更优选地,所述的患病风险是根据通过结果比较待测样本与正常样本的甲基化结果,当待测样本与正常样本的甲基化具有显著差异或极显著差异时,结果判断待测样本患病风险高。本发明中,所述的检测样品或待测样品可以选自肺泡灌洗液、组织、胸水、痰液、血液、血清、血浆、尿液、前列腺液或粪便。作为优选的实施方式,所述的样品选自组织、肺泡灌洗液、痰液;更优选地,所述样品选自肺泡灌洗液或痰液。本发明中所述的肿瘤选自肺癌;进一步地,所述的肺癌选自小细胞肺癌和非小细胞肺癌;所述的非小细胞肺癌选自鳞状细胞癌、腺癌或大细胞癌。本发明的有益效果:虽然,现有技术中,pax3基因和zic4基因的甲基化分别被报道了在肺癌中有甲基化。然而,有关肺癌的肿瘤标志物的报道,不计其数,真正能够用于临床中,作为肺癌检测的标志物的却少之又少。本发明在众多被报道的,跟肺癌可能相关甲基化基因中,首次将pax3基因和zic4基因联合,作为肺癌的肿瘤标志物,很大程度的提高了肺癌检测的可能性,且针对pax3基因和zic4基因,本发明还提供了优化的检测试剂,其具有很高的灵敏度和特异性,十分有希望应用到肺癌临床诊断中。本发明检测试剂,当检测样品为组织时,其中单独对pax3和zic4基因的对全部肺癌的特异性达到了95%,然而,即便是优化后的检测试剂,pax3基因对肺癌的敏感性为78.7%,而zic4为76.2%。通过联合pax3和zic4基因检测,对全部肺癌的敏感性提升到91%,如此高的灵敏度在现有的肺癌标志物是非常少见的。目前市面上已有基于shox2基因的肺癌检测试剂盒。在本发明的一个实施例中,对痰液标本进行检测,无论将肺癌作为一个整体进行比较分析,还是按照肺癌的亚型进行比较分析,pax3和zic4组合检测效果都要优于shox2基因的检测效果。特别是对腺癌的检测效果,pax3和zic4组合检测检出率为66.7%,而shox2基因检出率为0%。在本发明的另一个实施例中,对肺泡灌洗液进行检测,将肺癌作为一个整体进行比较分析,pax3和zic4组合检测可以明显提高肺癌的检出率。按照肺癌的亚型进行比较分析,特别是对腺癌的检测效果,其灵敏性达到90.9%,远远高于shox2的36.4%。此外,本发明的检测标志物针对不同类型的肺癌,包括小细胞肺癌和非小细胞肺癌中的鳞癌、大细胞癌、腺癌均具有很高的特异性和灵敏度,其适用范围广,基本上能作为所有肺癌的肿瘤标志物。而现有的用于临床的肺癌标志物,其一般仅能适用于一类肺癌的检测,如nse用于小细胞肺癌的诊断和监测治疗反应,而cyfra21-1是非小细胞肺癌的首选标记物。本发明提供的含有pax3基因和zic4的检测试剂和方法能非常方便、准确地判断出肺癌和肺部良性疾病患者,该基因的检测方法有望转化为基因检测试剂盒,并服务于肺癌的筛查、临床检测和预后监测。附图说明图1针对pax3不同的区域设计引物和探针的检测效果比较图2针对zic4不同的区域设计引物和探针的检测效果比较图3pax3&zic4、six3、pcdhga12、hoxd8、gata3、pax3、zic4基因检测肺癌的roc曲线图4pax3、zic4、pax3和zic4基因联合检测临床组织样品肺癌的roc曲线图5pax3和zic4基因联合、pax3、zic4和shox2基因在痰液样本中检测的roc曲线图6pax3和zic4基因联合、pax3、zic4和shox2基因在灌洗液样本中检测的roc曲线具体实施例实施例1:检测靶基因的选择甲基化dna作为检测靶标具有明显的优点,相比蛋白质类标志物,dna是可以扩增的,并且很容易检测到;与突变类标志物相比,dna发生甲基化的部位都位于基因的特定部位,一般在启动子区,使检测变得更容易和方便。为了完成本发明,发明人筛选了数百个基因,并从中选择出较好的pax3、zic4、six3、pcdhga12、hoxd8、gata3作为候选的检测基因,β-actin基因作为内参基因,研究各个基因甲基化位点分布情况,设计检测的引物探针分别用于检测。各基因检测引物探针如下:pax3的检测引物和探针为:seqidno:1pax3引物pax3-f1:tgggtatagcgtcggttagcseqidno:2pax3引物pax3-r1:ttcccgaaaatcatccgcgccgseqidno:3pax3探针pax3-p1:fam-aggtgaaggcgaaacggaaaggc-bq1zic4的检测引物和探针为:seqidno:4zic4引物zic4-f1:gatgcgcggttatgtttacseqidno:5zic4引物zic4-r1:gcgaccgaaacaatacgacseqidno:6zic4探针zic4-p1:fam-cgccgcaactacacgactacga-bq1six3的检测引物和探针为:seqidno:7six3引物f:cgttttatatttttggcgagtagcseqidno:8six3引物r:actccgccaacaccgseqidno:9six3探针:fam-cggcggcggcgcgggaggcgg-bq1pcdhga12的检测引物和探针为:seqidno:10pcdhga12引物f:ttggtttttacggttttcgacseqidno:11pcdhga12引物r:aaattctccgaaacgctcgseqidno:12pcdhga12探针:fam-attcggtgcgtataggtatcgcgc-bq1hoxd8的检测引物和探针为:seqidno:13hoxd8引物f:ttagtttcggcgcgtagcseqidno:14hoxd8引物r:cctaaaaccgacgcgatctaseqidno:15hoxd8探针:fam-aaaacttacgatcgtctaccctccg-bq1gata3的检测引物和探针为:seqidno:16gata3引物f:tttcggtagcgggtattgcseqidno:17gata3引物r:aaaataacgacgaaccaaccgseqidno:18gata3探针:fam-cgcgtttatgtaggagtggttgaggttc-bq1β-actin的检测引物和探针为:seqidno:19β-actin引物f:ttttggattgtgaatttgtgseqidno:20β-actin引物r:aaaacctactcctcccttaaaseqidno:21β-actin探针:fam-ttgtgtgttgggtggtggtt-bq1样本信息:肺组织样本共计36例,其中作为对照的肺组织标本共11例,包括4例癌旁正常组织和7例良性肺部疾病组织;癌组织样本25例,包括鳞癌4例,腺癌21例。试验过程:a.收集确诊为肺癌或者良性疾病肺部的手术切除标本,用石蜡进行包埋,进行病理组织切片染色,鉴定其组织类型和纯度。组织切片使用magen的dna提取试剂盒(hipureffpednakit,d3126-03)来提取dna。b.使用zymoresearch生物公司的dna转化试剂盒(ezdnamethylationkit,d5002)进行dna的亚硫酸氢盐修饰。c.扩增检测体系和检测体系如表:表1配液体系d.利用标准曲线分别计算基因在标本中的甲基化拷贝数,采用比值=目标基因拷贝数/actb拷贝数*100来进行判断组织的甲基化程度,最后设定一个pax3的阈值为6.38,zic4的阈值为8.49作为判断癌症组和对照组的标准,换算后比值超过或等于阈值可判断为阳性,小于阈值可判断为阴性。根据此标准,36例组织标本的检测结果如表3-4:表3组织中的检测结果注:“+”表示检测结果为阳性样本;“-”表示检测结果为阴性样本;在协同检测标本时,两者为阴性则判为阴性,一阴一阳或两者均为阳性则判为阳性。表4统计结果在11例肺组织对照标本中,包括4例癌旁正常组织和7例良性肺部疾病组织,pax3或zic4甲基化的阴性率(特异性)达到100%。在25例肺癌标本中,pax3和zic4甲基化的阳性率分别为88%(22/25)和72%(18/25),双阳性率为60%(15/25),任意阳性率(即pax3阳性或者zic4阳性)可高达100%(25/25),两个基因联合分析明显提高肺癌检出率。在鳞癌中,两种基因的甲基化阳性率都为100%。而在腺癌中,pax3基因甲基化阳性率为85.7%,zic4的阳性率为66.7%,联合检测可有互补作用,将阳性率提高到100%。故选择pax3和zic4基因甲基化进行协同检测,发明人还对其检测条件进行了优化,具体见实施例2。pax3、zic4、six3、pcdhga12、hoxd8、gata3、pax3和zic4组合基因检测肺癌的roc曲线见图3,pax3和zic4组合基因的roc曲线下面积是1。实施例2:pax3基因和zic4基因在临床组织标本中的检测a.检测引物探针如下:pax3的检测引物和探针为:seqidno:1pax3引物pax3-f1:tgggtatagcgtcggttagcseqidno:2pax3引物pax3-r1:ttcccgaaaatcatccgcgccgseqidno:3pax3探针pax3-p1:fam-aggtgaaggcgaaacggaaaggc-bq1zic4的检测引物和探针为:seqidno:4zic4引物zic4-f1:gatgcgcggttatgtttacseqidno:5zic4引物zic4-r1:gcgaccgaaacaatacgacseqidno:6zic4探针zic4-p1:fam-cgccgcaactacacgactacga-bq1β-actin的检测引物和探针为:seqidno:19β-actin引物f:ttttggattgtgaatttgtgseqidno:20β-actin引物r:aaaacctactcctcccttaaaseqidno:21β-actin探针:fam-ttgtgtgttgggtggtggtt-bq1b.样本信息:122对肺癌组织样本及其对应癌旁组织作为非肺癌对照,合共244例肺石蜡组织标本。其中包括23例鳞癌,93例腺癌,3例大细胞癌,1例混合性癌,2例未明确诊断肺癌类型。c.收集确诊为肺癌的手术切除标本,分离患癌组织和癌旁组织,分别用石蜡进行包埋,进行病理组织切片染色,鉴定其组织类型和纯度。组织切片使用magen的dna提取试剂盒(hipureffpednakit,d3126-03)来提取dna。d.使用zymoresearch生物公司的dna转化试剂盒(ezdnamethylationkit,d5002)进行dna的亚硫酸氢盐修饰。e.扩增检测体系和检测体系如表5和表6:表5配液体系f.检测结果利用标准曲线计算pax3基因在标本中的甲基化拷贝数,采用比值=拷贝数/actb拷贝数*100来进行判断两组组织的甲基化程度,最后选取pax3数值“19.5”或zic4数值“7.6”作为判断癌症组和对照组的标准,换算后的pax3比值超过“19.5”或zic4比值超过“7.6”可判断为阳性,pax3比值等于或小于“19.5”或zic4比值等于或小于“7.6”可判断为阴性。根据此标准,244例组织标本的检测结果如表7:表7检测结果注:“+”为甲基化dna检测阳性,“-”为甲基化dna检测阴性;在协同检测标本时,两者为阴性则判为阴性,一阴一阳或两者均为阳性则判为阳性。表8结果统计pax3、zic4以及pax3联合zic4在组织标本中检测肺癌的roc曲线见图4,pax3、zic4以及pax3联合zic4检测的roc曲线下面积分别是0.932、0.919、0.975。结果显示,在122例肺癌旁对照中,阴性检测率为95.0%的情况下,pax3和zic4甲基化的阳性率分别为78.7%和76.2%,两个基因联合检测特异性为90.2%,灵敏度高达91.0%,只有11例腺癌漏检,其他均可被检出,表明pax3和zic4联合可明显提高了肺癌的检出率。综合该实施例,能够充分的说明pax3和zic4联合在对肺癌检测诊断有更好的检测效果。实施例3:pax3基因和zic4基因在痰液标本中的检测大量文献显示shox2可用作检测肺癌的标志物,shox2在肺泡灌洗液、病变部位组织、胸水、痰液等样本中具有较高的检出率。为了验证pax3基因和zic4基因协同检测的效果,本发明人同时检测pax3基因、zic4基因与shox2基因在痰液中的检出效率。各基因检测引物探针如下:pax3的检测引物和探针为:seqidno:1pax3引物pax3-f1:tgggtatagcgtcggttagcseqidno:2pax3引物pax3-r1:ttcccgaaaatcatccgcgccgseqidno:3pax3探针pax3-p1:fam-aggtgaaggcgaaacggaaaggc-bq1zic4的检测引物和探针为:seqidno:4zic4引物zic4-f1:gatgcgcggttatgtttacseqidno:5zic4引物zic4-r1:gcgaccgaaacaatacgacseqidno:6zic4探针zic4-p1:fam-cgccgcaactacacgactacga-bq1shox2的检测引物和探针为:shox2_t_mf3引物f:tttaaagggttcgtcgtttaagtcshox2_t_mr3引物r:aaacgattactttcgcccgshox2_taq_p3_探针:fam-ttagaaggtaggaggcggaaaattag-bq1样本信息:测试痰样本共计60例,其中正常对照组样本31例,癌症组对照样本29例,29例癌症组样本中有鳞癌9例,小细胞癌6例,腺癌9例,大细胞癌1例,未明确分类的肺癌4例。试验过程:a.收集确诊为肺癌患者和非肺癌患者的痰液标本,使用dtt解稠后,离心取沉淀分离细胞,使用pbs洗涤2遍,然后使用magen的dna提取试剂盒(hipureffpednakit,d3126-03)提取dna。b.使用zymoresearch生物公司的dna转化试剂盒(ezdnamethylationkit,d5002)进行dna的重亚硫酸盐修饰。c.配液体系如表9:表9配液体系d.扩增体系如表10:表10pcr反应过程e.检测结果如下:利用标准曲线计算各基因在标本中的甲基化拷贝数,采用比值=拷贝数/actb拷贝数*100来进行判断两组组织的甲基化程度,最后选取pax3的阈值为4.5,zic4的阈值为2.2,shox2的阈值为5.1,作为判断癌症组和对照组的标准,换算后的比值超过设定阈值可判断为阳性,等于或小于设定阈值可判断为阴性,pax3与zic4联合检测,同为阴性则判断为阴性,只要有一个或两个为阳性则判断为阳性。根据此标准,60例痰液标本的检测结果如表11:表11痰液标本检测结果注:“+”表示检测结果为阳性样本;“-”表示检测结果为阴性样本。f.结果分析表12统计结果pax3、zic4、shox2以及pax3和zic4基因组合在痰液样本中检测肺癌的roc曲线见图5。pax3、zic4、shox2以及pax3和zic4基因组合联合检测的roc曲线下面积分别是0.804、0.905、0.847、0.903。从以上结果可以看出,无论将肺癌作为一个整体或按不同亚型进行比较分析,pax3和zic4的检测效果要优于shox2基因的检测效果。同时,当pax3与zic4联合检测时,均较大的提高鳞癌和腺癌的检出率,特别是腺癌的检出率达到66.7%,而shox2基因检出率为0%,腺癌一般为周围型,由于支气管的树状生理结构,肺深部的脱落细胞更加难以通过痰液咳出,因此对这部分的检测更加困难和有意义。实施例4:pax3基因和zic4基因在灌洗液标本中的检测样本信息:测试肺泡灌洗液样本共计79例,其中正常对照组样本58例,癌症组对照样本21例,21例癌症组样本中有鳞癌6例,小细胞癌4例,腺癌11例。试验过程:a.收集确诊为肺癌患者和非肺癌患者的肺泡灌洗液标本,离心分离细胞,然后使用magen的dna提取试剂盒(hipureffpednakit,d3126-03)提取dna。b.使用zymoresearch生物公司的dna转化试剂盒(ezdnamethylationkit,d5002)进行dna的亚硫酸氢盐修饰。c.配液体系如表9。d.扩增检测体系如表10.e.检测结果如下:利用标准曲线计算各基因在标本中的甲基化拷贝数,采用比值=拷贝数/actb拷贝数*100来进行判断两组组织的甲基化程度,最后选取pax3的阈值为2.4,zic4的阈值为1.8,shox2的阈值为0.6,作为判断癌症组和对照组的标准,换算后的比值超过设定阈值可判断为阳性,等于或小于设定阈值可判断为阴性,pax3与zic4联合检测,同为阴性则判断为阴性,只要有一个或两个为阳性则判断为阳性。根据此标准,79例灌洗液标本的检测结果如表13表13灌洗液标本检测结果注:“+”表示检测结果为阳性样本;“-”表示检测结果为阴性样本。在组合检测标本时,两者为阴性则判为阴性,一阴一阳或两者均为阳性则判为阳性。表14统计结果pax3、zic4、shox2以及pax3和zic4基因组合在肺泡灌洗液样本中检测肺癌的roc曲线见图6。pax3、zic4、shox2以及pax3和zic4基因组合联合检测的roc曲线下面积分别是0.841、0.838、0.784、0.880。从以上结果可以看出,同时检测pax3、zic4和shox2,无论将肺癌作为一个整体或者按亚型进行比较分析,pax3、zic4的检出率均远远高于shox2的47.6%,同时,对pax3和zic4进行联合分析发现,pax3和zic4的联合检测可以明显提高肺癌的检出率,特别是对腺癌的检测效果,其灵敏性达到90.9%,远远高于shox2的36.4%。因为腺癌一般为周围型,由于支气管的树状生理结构,肺泡灌洗液不容易接触到肺深部的肺泡或者癌组织,因此对这部分的检测更加困难和有意义。综合实施例1-4,能够充分的说明pax3和zic4基因的联合在对肺癌检测诊断,尤其是应用痰液,肺泡灌洗液等生物样本上具有更好的检测效果。能够更加容易的应用于大规模的人群筛查。具有更加优越的社会经济学价值。实施例5:pax3基因和zic4基因区域的选择各种研究资料表明,同一个基因的甲基化状态和分布并不均匀,因此对于同一个基因来说,选择不同的区域设计的甲基化引物、探针检测体系对同一样本,同一肿瘤的诊断检测效能并不一样,甚至有时候选择的区域不合适造成对肿瘤完全没有诊断效果。本发明人经过反复的研究和比较后,选择pax3和zic4基因的启动子区域序列如下:发明人选择了pax3和zic4基因的启动子的不同区域进行检测,具体见下表15。表15pax3和zic4基因检测区域的选择根据pax3序列的区域1、区域2、区域3,以及zic4序列区域1、区域2、区域3、区域4和区域5设计不同的甲基化引物和探针,各引物探针信息见表16。针对pax3,其中组1p、组2p、组3p、组4p、组5p、组6p是根据区域1设计的甲基化引物和探针;组7p、组8p、组9p是根据区域2设计的甲基化引物和探针;组10p、组11p、组12p是根据区域3设计的甲基化引物和探针。针对zic4,其中组1z、组2z、组3z、组4z、组5z是根据区域1设计的甲基化引物和探针;组6z、组7z是根据区域2设计的甲基化引物和探针;组8z、组9z、组10z是根据区域3设计的甲基化引物和探针;组11z、组12z是根据区域4设计的甲基化引物和探针;组13z、组14z、组15z是根据区域5设计的甲基化引物和探针。表16引物和探针在36例肺组织样本检测以上27组引物探针组合,其中正常组织样本11例,癌组织样本25例,25例癌症组样本中有鳞癌4例,腺癌21例。检测结果如下表17-18。样品处理、检测结果判断、统计方式同实施例1;pcr配液体系、反应过程为本领域常规操作。表17pax3在组织中的检测结果所处区域组别引物探针组合特异性灵敏性区域1组1ppax3-f1,pax3-r1,pax3-p1100%88%区域1组2ppax3-f2,pax3-r2,pax3-p2100%80%区域1组3ppax3-f3,pax3-r3,pax3-p3100%84%区域1组4ppax3-f4,pax3-r4,pax3-p4100%76%区域1组5ppax3-f5,pax3-r5,pax3-p5100%72%区域1组6ppax3-f6,pax3-r6,pax3-p6100%64%区域2组7ppax3-f7,pax3-r7,pax3-p7100%52%区域2组8ppax3-f8,pax3-r8,pax3-p8100%44%区域2组9ppax3-f9,pax3-r9,pax3-p9100%40%区域3组10ppax3-f10,pax3-r10,pax3-p10100%52%区域3组11ppax3-f11,pax3-r11,pax3-p11100%72%区域3组12ppax3-f12,pax3-r12,pax3-p12100%64%表18zic4在组织中的检测结果所处区域组别引物探针组合特异性灵敏性区域1组1zzic4-f1,zic4-r1,zic4-p1100%72%区域1组2zzic4-f2,zic4-r2,zic4-p2100%64%区域1组3zzic4-f3,zic4-r3,zic4-p3100%68%区域1组4zzic4-f4,zic4-r4,zic4-p4100%72%区域1组5zzic4-f5,zic4-r5,zic4-p5100%56%区域2组6zzic4-f6,zic4-r6,zic4-p6100%48%区域2组7zzic4-f7,zic4-r7,zic4-p7100%40%区域3组8zzic4-f8,zic4-r8,zic4-p8100%48%区域3组9zzic4-f9,zic4-r9,zic4-p9100%32%区域3组10zzic4-f10,zic4-r10,zic4-p10100%36%区域4组11zzic4-f11,zic4-r11,zic4-p11100%64%区域4组12zzic4-f12,zic4-r12,zic4-p12100%52%区域5组13zzic4-f13,zic4-r13,zic4-p13100%44%区域5组14zzic4-f14,zic4-r14,zic4-p14100%52%区域5组15zzic4-f15,zic4-r15,zic4-p15100%68%结果显示,针对pax3,组1p、组2p、组3p、组4p、组5p、组11p均有较高的检出率。无论采用本发明设计的何种引物和探针,区域1的检测灵敏度最低也可达到64%,最高达到88%,检出率要远远高于针对区域2设计的几对引物,且区域1的大部分引物比区域3的引物的检测灵敏性要高,因此,pax3区域1的检出率明显较其他区域高(见表17)。针对zic4,组1z、组2z、组3z、组4z、组11z、组15z均有较高的检出率。无论采用本发明设计的何种引物和探针,区域1的检测灵敏度最低也可达到56%,最高达到72%,检出率要远远高于针对区域2和区域3设计的几对引物,且区域1的大部分引物比区域4和区域5的引物的检测灵敏性要高,因此,zic4区域1的检出率明显较其他区域高(见表18)。实施例6引物和探针组合的选择为了进一步验证其在痰液中的检出率,我们选取了22例痰液标本,采用表19中的引物和探针进行验证,其中包括7例正常对照,15例肺癌对照,15例肺癌中有鳞癌7例,腺癌7例,大细胞癌1例,检测结果如下表19-20。表19pax3在痰液中的检测结果组别引物探针组合特异性灵敏性组1ppax3-f1,pax3-r1,pax3-p1100%73.3%组2ppax3-f2,pax3-r2,pax3-p2100%60%组3ppax3-f3,pax3-r3,pax3-p3100%46.7%组4ppax3-f4,pax3-r4,pax3-p4100%53.3%组5ppax3-f5,pax3-r5,pax3-p5100%40%组11ppax3-f11,pax3-r11,pax3-p11100%40%表20zic4在痰液中的检测结果从22例痰液标本的检测结果显示,pax3的组1p:pax3-f1,pax3-r1,pax3-p1的检出率最高,达到73.3%。虽然在组织样本中,组1p的灵敏度达到88%,组3p的灵敏度达到84%,但针对痰液检测样本,组3的灵敏度却大幅下降至46.7%。zic4的组1z:zic4-f1,zic4-r1,zic4-p1的检出率最高,达到66.7%。虽然在组织样本中,组1z和组4z的灵敏度达到72%,但针对痰液检测样本,组4z的灵敏度却大幅下降至46.7%。最终,根据各组引物探针的检测结果,最优选的引物探针序列如下表21。表21优化后的引物序列表<110>广州市康立明生物科技有限责任公司<120>基于pax3和zic4基因的肺癌诊断试剂及试剂盒<160>120<170>siposequencelisting1.0<210>1<211>20<212>dna<213>人工序列(artificialsequence)<400>1tgggtatagcgtcggttagc20<210>2<211>22<212>dna<213>人工序列(artificialsequence)<400>2ttcccgaaaatcatccgcgccg22<210>3<211>23<212>dna<213>人工序列(artificialsequence)<400>3aggtgaaggcgaaacggaaaggc23<210>4<211>19<212>dna<213>人工序列(artificialsequence)<400>4gatgcgcggttatgtttac19<210>5<211>19<212>dna<213>人工序列(artificialsequence)<400>5gcgaccgaaacaatacgac19<210>6<211>22<212>dna<213>人工序列(artificialsequence)<400>6cgccgcaactacacgactacga22<210>7<211>24<212>dna<213>人工序列(artificialsequence)<400>7cgttttatatttttggcgagtagc24<210>8<211>15<212>dna<213>人工序列(artificialsequence)<400>8actccgccaacaccg15<210>9<211>21<212>dna<213>人工序列(artificialsequence)<400>9cggcggcggcgcgggaggcgg21<210>10<211>21<212>dna<213>人工序列(artificialsequence)<400>10ttggtttttacggttttcgac21<210>11<211>19<212>dna<213>人工序列(artificialsequence)<400>11aaattctccgaaacgctcg19<210>12<211>24<212>dna<213>人工序列(artificialsequence)<400>12attcggtgcgtataggtatcgcgc24<210>13<211>18<212>dna<213>人工序列(artificialsequence)<400>13ttagtttcggcgcgtagc18<210>14<211>20<212>dna<213>人工序列(artificialsequence)<400>14cctaaaaccgacgcgatcta20<210>15<211>25<212>dna<213>人工序列(artificialsequence)<400>15aaaacttacgatcgtctaccctccg25<210>16<211>19<212>dna<213>人工序列(artificialsequence)<400>16tttcggtagcgggtattgc19<210>17<211>21<212>dna<213>人工序列(artificialsequence)<400>17aaaataacgacgaaccaaccg21<210>18<211>28<212>dna<213>人工序列(artificialsequence)<400>18cgcgtttatgtaggagtggttgaggttc28<210>19<211>20<212>dna<213>人工序列(artificialsequence)<400>19ttttggattgtgaatttgtg20<210>20<211>21<212>dna<213>人工序列(artificialsequence)<400>20aaaacctactcctcccttaaa21<210>21<211>20<212>dna<213>人工序列(artificialsequence)<400>21ttgtgtgttgggtggtggtt20<210>22<211>500<212>dna<213>homosapiens<400>22ccttaccttccagcgggaacccgctacgcgggtagttctgccccgggcccggccgcatca60tcctgggcacagcgccggccagcgtggtcatcctgggggcagcttcgctcggaaattata120tccaggtgaaggcgaaacggaaaggcgagtgcggcgcggatgaccctcgggaactatccg180gagcgtggagagcccctccccaaaacggctggagagagagggagggacgcggggaggggg240gctgtcggttcctagtccagaggccggagctggaacccgggaaaggggaggacggggagg300ccccggagtccaggatcccgagcccagggcggaaaagtttggtacgagtctgggcaaatg360ttccagcgactggggtccctgaaaagggggctcagagagccacggcgagccggggagcct420ggtgaggctggagcgcggcctgcctgagtctcctcctgtggtgacaccgagtgcggggat480ccgggctcgggagcatttat500<210>23<211>500<212>dna<213>人工序列(artificialsequence)<400>23ttttattttttagcgggaattcgttacgcgggtagttttgtttcgggttcggtcgtatta60ttttgggtatagcgtcggttagcgtggttattttgggggtagtttcgttcggaaattata120tttaggtgaaggcgaaacggaaaggcgagtgcggcgcggatgattttcgggaattattcg180gagcgtggagagtttttttttaaaacggttggagagagagggagggacgcggggaggggg240gttgtcggtttttagtttagaggtcggagttggaattcgggaaaggggaggacggggagg300tttcggagtttaggatttcgagtttagggcggaaaagtttggtacgagtttgggtaaatg360ttttagcgattggggtttttgaaaagggggtttagagagttacggcgagtcggggagttt420ggtgaggttggagcgcggtttgtttgagtttttttttgtggtgatatcgagtgcggggat480tcgggttcgggagtatttat500<210>24<211>769<212>dna<213>homosapiens<400>24ctcccgtgcgccctctcgctggccccgtgcttgcccctcttctccctccgcctcccccag60gctgccgtggcggggggctccggaccgtccctgagactctcggaggaaatcggggccgtt120gtggaagcctccacggctttgcgcacacggcaaagtccctcccggcgcgggccccatctc180ccttcggttggggttaccaaaacatttgtttctctttaaaagggaacatcaatattaata240aacgctctgcctccgcctcacgtttcctgccctgcctcctcgacagaaatcttctttggg300gcgtcctcagcggtggtctcgccaccctccgtccccaggacaagcagctcacccctccct360ccataaagtgccaagaacaccgggttggcaaatattgcagggcctcgggagaggccacct420cccaatagctgagatcgataattggggtgattacgtctgggtcgacgtgccggggtaata480gcgactgactgtcgcgcctcggggagaggttaatgggcctagtacctgacggcacggtgt540ttcgatcacagaccgcgtccttgagtaatttgtctcggatttcccagctgaacatgcccg600ggttctctcttttgtattcctcaattttcttctccacgtcaggcgttgtcacctgcttta660agagaacaggcgggcaggcgttggtacccggtaccctgggccaggtggcggcggccccac720cgcctctgggcctgtcgggatgctcctcgctgaatcctctgggacggtc769<210>25<211>769<212>dna<213>人工序列(artificialsequence)<400>25ttttcgtgcgttttttcgttggtttcgtgtttgtttttttttttttttcgttttttttag60gttgtcgtggcggggggtttcggatcgtttttgagattttcggaggaaatcggggtcgtt120gtggaagtttttacggttttgcgtatacggtaaagtttttttcggcgcgggttttatttt180ttttcggttggggttattaaaatatttgtttttttttaaaagggaatattaatattaata240aacgttttgttttcgttttacgttttttgttttgttttttcgatagaaattttttttggg300gcgtttttagcggtggtttcgttatttttcgtttttaggataagtagtttattttttttt360ttataaagtgttaagaatatcgggttggtaaatattgtagggtttcgggagaggttattt420tttaatagttgagatcgataattggggtgattacgtttgggtcgacgtgtcggggtaata480gcgattgattgtcgcgtttcggggagaggttaatgggtttagtatttgacggtacggtgt540ttcgattatagatcgcgtttttgagtaatttgtttcggattttttagttgaatatgttcg600ggtttttttttttgtattttttaatttttttttttacgttaggcgttgttatttgtttta660agagaataggcgggtaggcgttggtattcggtattttgggttaggtggcggcggttttat720cgtttttgggtttgtcgggatgtttttcgttgaattttttgggacggtt769<210>26<211>1001<212>dna<213>homosapiens<400>26cagcccaggccggtcaccaaagtgcgactgggctgtagacgcacgcatccaaacgggctg60cgagggagaactttcaagctaacaagggcacgctccactcaggcctggacagccagcccg120ctgttattttcacctgtagtgtctctgaaactgggggcagaggcgcagaggggacaatag180aagctatctcccccgcccaccttttttttttttttgtcaaattatccaaatatcgtcttc240aaggctcccgtttcatttcgaacgtgcacagagcgtgagtggaccctacactcgttcaca300acaccgttgtggagagtcccactgatgctttctgggtaatttgttagcccaggtaaaagt360cactgcctcccaaaccagggttcaaggatgcagggcgcgggccctcgccttccaagcccc420accgctgggtcatccctttaccaattagcgcctgtaccgcggccaaatttggcaccagga480ctccttggcccaccggctgcggatagggaagtggcctggactcccatccagctgcggggt540ttgggtgtggggtggcttgggctccggagagacttcagaaagaaataaaggggaggccca600agactcggagacagagaaagagtggctggaatcacccggcattcctgagacctgttgcgc660cctgttctgcggagtcacggctctgagccgggttaggtttggcggcgtgcgggcgcccta720ggccaggcagcctcagccaactttgctgtcctggccatcccaggtcggacttcggggaca780caggccacaagaaatttagattcattaagtctttggatgtggcccagctgtccagagcct840aagggtgccctcccacccctctccgggtgtccagggacctctggccactggccggtgaga900agggccgggctgatcatctgggtacatatgaatctcagagcgctcggcagaaaagaaaga960ggccggaaaggagagaaaatgagtcgcctcctgattccgga1001<210>27<211>1001<212>dna<213>人工序列(artificialsequence)<400>27tagtttaggtcggttattaaagtgcgattgggttgtagacgtacgtatttaaacgggttg60cgagggagaatttttaagttaataagggtacgttttatttaggtttggatagttagttcg120ttgttatttttatttgtagtgtttttgaaattgggggtagaggcgtagaggggataatag180aagttatttttttcgtttattttttttttttttttgttaaattatttaaatatcgttttt240aaggttttcgttttatttcgaacgtgtatagagcgtgagtggattttatattcgtttata300atatcgttgtggagagttttattgatgttttttgggtaatttgttagtttaggtaaaagt360tattgttttttaaattagggtttaaggatgtagggcgcgggttttcgttttttaagtttt420atcgttgggttatttttttattaattagcgtttgtatcgcggttaaatttggtattagga480ttttttggtttatcggttgcggatagggaagtggtttggatttttatttagttgcggggt540ttgggtgtggggtggtttgggtttcggagagattttagaaagaaataaaggggaggttta600agattcggagatagagaaagagtggttggaattattcggtatttttgagatttgttgcgt660tttgttttgcggagttacggttttgagtcgggttaggtttggcggcgtgcgggcgtttta720ggttaggtagttttagttaattttgttgttttggttattttaggtcggatttcggggata780taggttataagaaatttagatttattaagtttttggatgtggtttagttgtttagagttt840aagggtgtttttttattttttttcgggtgtttagggatttttggttattggtcggtgaga900agggtcgggttgattatttgggtatatatgaattttagagcgttcggtagaaaagaaaga960ggtcggaaaggagagaaaatgagtcgttttttgatttcgga1001<210>28<211>507<212>dna<213>homosapiens<400>28gttggcgcatgtagcggaagaaggcgccggcgccgtgatgcgcggccatgttcacgttca60tgggcccgtagccgtgcagctgcggcgcagcatagtgctccgaacgcgggctggtcacct120ggccgtactgctccggtcgcgggtacatgtcccccgagaagccgagcctcatctgcccgt180tgaccacgttaggcgacgcgtggccggcagcctgctcgtgaagcccggggaagaggaggt240ggcccgcggcgtccgtgtggccgtgtgggcccccgaagcccccggccgatgcagcaaaga300ggctgtgctgtgcgctggctgccgccgccgcgtcgccaaaaccccggttgcggaacagaa360agtcccgcgtggagttgaaggctgcgctggaataggagccgacgtggcccgggtgatggt420gatggcccagggccgcagcagccgcgtagcctggcgcctgcgacgtgaaggccgtctggc480cggccgaagccagctcgtgcgaactgg507<210>29<211>507<212>dna<213>人工序列(artificialsequence)<400>29gttggcgtatgtagcggaagaaggcgtcggcgtcgtgatgcgcggttatgtttacgttta60tgggttcgtagtcgtgtagttgcggcgtagtatagtgtttcgaacgcgggttggttattt120ggtcgtattgtttcggtcgcgggtatatgtttttcgagaagtcgagttttatttgttcgt180tgattacgttaggcgacgcgtggtcggtagtttgttcgtgaagttcggggaagaggaggt240ggttcgcggcgttcgtgtggtcgtgtgggttttcgaagttttcggtcgatgtagtaaaga300ggttgtgttgtgcgttggttgtcgtcgtcgcgtcgttaaaatttcggttgcggaatagaa360agtttcgcgtggagttgaaggttgcgttggaataggagtcgacgtggttcgggtgatggt420gatggtttagggtcgtagtagtcgcgtagtttggcgtttgcgacgtgaaggtcgtttggt480cggtcgaagttagttcgtgcgaattgg507<210>30<211>794<212>dna<213>homosapiens<400>30tttctacgcccttaacccggcctcctcaatttcctcagctctttcctttggcaaagtcct60cagtgaagtgcacggagagagatgttggcagcaagcaagggtttagatttgtctgcagcc120tgagcagctggcgcctccgcggctgccgggagccagccactttctccggcttcgaaagca180ccaaggtctccgccaaaccaggtttggcttcctgttctccgaccttttttgtttctcatt240taaaaaatctatatacccagctcctcctcgtccgccctgtgtgtgtgtgtgtgttttcca300taatttcaagccagaaactaaaaatcagcgcacactttccctgcctgcctgccggaacct360gacgcccgggatcccaccgaggctgcgtttgtgcgacctggccgctgactttcggtttgg420gtcccaggcccaagtgggaatgggtaggggtcctacagctgtttccgtacctgtgtgcgt480ccttttgtggatctttaaattctcggagcgcgcgaagaccttgccacagccagggaaggg540gcagggaaagggcttctcgcccgtgtgcacgcggatgtggttaaccagtttgtatttggc600tttgaagggcttgccctcgcgcggacactcctcccagaagcagatgtgattactctgctc660cgggccacctacgtgctccacggtgacgtgcgtaactagctcgtgcatggtgctgaaagt720tttgttgcacgactttttggggttggccagctgctcgggctcgatccacttgcagatgag780ctcttgcttgatgg794<210>31<211>794<212>dna<213>人工序列(artificialsequence)<400>31tttttacgtttttaattcggtttttttaatttttttagtttttttttttggtaaagtttt60tagtgaagtgtacggagagagatgttggtagtaagtaagggtttagatttgtttgtagtt120tgagtagttggcgttttcgcggttgtcgggagttagttatttttttcggtttcgaaagta180ttaaggttttcgttaaattaggtttggttttttgtttttcgattttttttgttttttatt240taaaaaatttatatatttagttttttttcgttcgttttgtgtgtgtgtgtgtgtttttta300taattttaagttagaaattaaaaattagcgtatattttttttgtttgtttgtcggaattt360gacgttcgggattttatcgaggttgcgtttgtgcgatttggtcgttgattttcggtttgg420gttttaggtttaagtgggaatgggtaggggttttatagttgttttcgtatttgtgtgcgt480ttttttgtggatttttaaattttcggagcgcgcgaagattttgttatagttagggaaggg540gtagggaaagggtttttcgttcgtgtgtacgcggatgtggttaattagtttgtatttggt600tttgaagggtttgttttcgcgcggatattttttttagaagtagatgtgattattttgttt660cgggttatttacgtgttttacggtgacgtgcgtaattagttcgtgtatggtgttgaaagt720tttgttgtacgattttttggggttggttagttgttcgggttcgatttatttgtagatgag780tttttgtttgatgg794<210>32<211>1062<212>dna<213>homosapiens<400>32ggttgagcttgaaggcgcccatgccgtcggcgaacgggttgatgcccaggcccacgtctc60gttcggccacgtcgcccgcggagtggtggcgggacgcgccaaaggtggtcacgccgatcg120ctgggtactgggggccggcgtccaggagcatcgtggctgctcggggcgagccccggcctc180ccccgccccccccaccctcgccagccgaggagggaaaatcgggaggaggaggaggaacaa240gaggaggaggaagaggaggaagaagaggaggagggagggggagtcgacccacctcgcgaa300gtcctagcctgcaggcaatggcgcggcgcgcccgggcgctggccgggccaaacgcaaagt360agccggacggctgcgggccgcgagtaacgcaggctccagcgcccgccaggcagattctgc420cggcccggctcgcactttctcgccgctcagtctctgccgagagggccgcgcgtcaaggct480gcccggggaaaggcatggagcatctcagccccctaaaaaaaaacttcctcctggatttgt540agcatagaggaatgtgagcgccagtgccgcgactgcttcgccctctctccgcctcgcgca600gcgcgcccctccttacttctccactagcccctcgccctttcttttctctctctctccttt660ctctcacgctcgcctcctcgctccttcactctccgtttttccctctctgctttttgaaaa720aaaaaaaaaaaaaagtgggtgggggggtgaagaaagaaagaaaaagccaaagaaaaggcg780caaatcatgcacaatattgtcttttgcggtttatcttcctggggagaaactttcacctcc840tcagccgggcggtgagcgcgagactgatagcagcaatcattcctgcagataaatgaattg900aaaggacgacaccgtcccccccttgatgaatgggagcaggggggaggtgtcacgtgctgc960cgacgctggcgcccattggtgcgcccgcattccccccgccctcagccgccgcgccaacgg1020agggcgcgccgaggcgccgcgcgcagctcattggcccgcccg1062<210>33<211>1062<212>dna<213>人工序列(artificialsequence)<400>33ggttgagtttgaaggcgtttatgtcgtcggcgaacgggttgatgtttaggtttacgtttc60gttcggttacgtcgttcgcggagtggtggcgggacgcgttaaaggtggttacgtcgatcg120ttgggtattgggggtcggcgtttaggagtatcgtggttgttcggggcgagtttcggtttt180tttcgttttttttattttcgttagtcgaggagggaaaatcgggaggaggaggaggaataa240gaggaggaggaagaggaggaagaagaggaggagggagggggagtcgatttatttcgcgaa300gttttagtttgtaggtaatggcgcggcgcgttcgggcgttggtcgggttaaacgtaaagt360agtcggacggttgcgggtcgcgagtaacgtaggttttagcgttcgttaggtagattttgt420cggttcggttcgtattttttcgtcgtttagtttttgtcgagagggtcgcgcgttaaggtt480gttcggggaaaggtatggagtattttagttttttaaaaaaaaatttttttttggatttgt540agtatagaggaatgtgagcgttagtgtcgcgattgtttcgtttttttttcgtttcgcgta600gcgcgtttttttttatttttttattagtttttcgtttttttttttttttttttttttttt660tttttacgttcgttttttcgtttttttatttttcgtttttttttttttgttttttgaaaa720aaaaaaaaaaaaaagtgggtgggggggtgaagaaagaaagaaaaagttaaagaaaaggcg780taaattatgtataatattgttttttgcggtttatttttttggggagaaatttttattttt840ttagtcgggcggtgagcgcgagattgatagtagtaattatttttgtagataaatgaattg900aaaggacgatatcgttttttttttgatgaatgggagtaggggggaggtgttacgtgttgt960cgacgttggcgtttattggtgcgttcgtatttttttcgtttttagtcgtcgcgttaacgg1020agggcgcgtcgaggcgtcgcgcgtagtttattggttcgttcg1062<210>34<211>801<212>dna<213>homosapiens<400>34gcgtcttcctcctcagctagacgctaggtagccggtcccttaactcagagcgctgggagg60cagccatttgtttaaagcgcccagaaaccttcttcccccatttccgcagcacttgcagct120ccggcaggaatctaggcttccgggttctcggggtcctttcctctcctacacgtcagcgaa180ttcagctggccgtgagctccccgattggggctcggtccggccaatattgtcactctaacg240tgcactcgcgccttccctcggctctacagggcatcgcagccaccgatcccagcccctccc300cggcctatgccagccgcctggtggccctccgggctgcggagccaggcctggcctgacagc360agcagctgttttcttcccgaagccgcagcgctcaagggcgtggttgcggtagacacctcg420gatcccatttggcctgtgcggaccctaccgcgccaacccctcgtcttaggaccgagaggc480ccgacagccaggtgggggtggtctgggcagaccggtcgacatcgcaagtcagtgcggact540gggagggccagctgcccacagcacggtctccaggaagcctctggagcagcgtaatgaaat600attattactggcgagcgtatctggagggatcccggaaggtttaggcaagatttgaagaat660gctgctaaatgtttgccctcagggggtagaaggaaagagggagggggctccattcctgta720aaaatctcttctgaagtgaatcattgctcagaatacctctccctgcagcacgaatccact780ttatgcacacacaatgtgcat801<210>35<211>801<212>dna<213>人工序列(artificialsequence)<400>35gcgtttttttttttagttagacgttaggtagtcggttttttaatttagagcgttgggagg60tagttatttgtttaaagcgtttagaaattttttttttttattttcgtagtatttgtagtt120tcggtaggaatttaggttttcgggttttcggggttttttttttttttatacgttagcgaa180tttagttggtcgtgagtttttcgattggggttcggttcggttaatattgttattttaacg240tgtattcgcgttttttttcggttttatagggtatcgtagttatcgattttagtttttttt300cggtttatgttagtcgtttggtggtttttcgggttgcggagttaggtttggtttgatagt360agtagttgtttttttttcgaagtcgtagcgtttaagggcgtggttgcggtagatatttcg420gattttatttggtttgtgcggattttatcgcgttaatttttcgttttaggatcgagaggt480tcgatagttaggtgggggtggtttgggtagatcggtcgatatcgtaagttagtgcggatt540gggagggttagttgtttatagtacggtttttaggaagtttttggagtagcgtaatgaaat600attattattggcgagcgtatttggagggatttcggaaggtttaggtaagatttgaagaat660gttgttaaatgtttgtttttagggggtagaaggaaagagggagggggttttatttttgta720aaaattttttttgaagtgaattattgtttagaatatttttttttgtagtacgaatttatt780ttatgtatatataatgtgtat801<210>36<211>1001<212>dna<213>homosapiens<400>36gcggcccggtacagggagctccggggagcgtgggtcggcaagcagccggccgccctctcc60tagccaagcggggcactgaaaatatatatggctggggcgcttcagaaccaccaccacctc120caacatcatcatttctgtcacctccactccccgcagggcatcgtgccccggagagccccg180gggtgttagccgccaaagtgcgcttggggctcagctcctacccctctggagcagaacccc240cagcaacccacagactcagccctgcgccttccattaccccttacatctactctgccgcta300ccatccctctctagctcatggcgcggcgggggacttggcccaacagcaattcggcccaat360ggcggcggaggtgtttggccttgctctgtccaccgggttcactgctggcgctgtctggct420ctgagttccacggctgactgcagaggccgtagtgcggcagggctgggcctgccccccggg480gcccagggaagagcgcgcagcccacgccacccgttctcatgcgcgctatttgtttcgaca540acaggtagcagctctggacaccatggcccccagctcaccgccgcctccagcccctcggtg600ttcccgggcctccacgaggagcctccccaggcctcccccagccgtcctttgaatggactc660ctgcgtctggggctccctggagacatgtacgcgcggccggagcccttcccgccagggcct720gcggcccgcagcgacgccctggcagctgccgcagccctgcatggctacgggggcatgaac780ctgacggtgaacctcgctgcgccccacggtcctggcgctttcttccgctacatgcgccag840cccatcaaacaggagctcatctgcaagtggctggcggccgacggcaccgcgaccccgagc900ctctgctccaaaactttcagcaccatgcacgagctggtcacgcacgtcaccgtggagcac960gtcggcggcccggaacaggccaaccacatttgcttctggga1001<210>37<211>1001<212>dna<213>人工序列(artificialsequence)<400>37gcggttcggtatagggagtttcggggagcgtgggtcggtaagtagtcggtcgtttttttt60tagttaagcggggtattgaaaatatatatggttggggcgttttagaattattattatttt120taatattattatttttgttatttttatttttcgtagggtatcgtgtttcggagagtttcg180gggtgttagtcgttaaagtgcgtttggggtttagtttttatttttttggagtagaatttt240tagtaatttatagatttagttttgcgttttttattattttttatatttattttgtcgtta300ttatttttttttagtttatggcgcggcgggggatttggtttaatagtaattcggtttaat360ggcggcggaggtgtttggttttgttttgtttatcgggtttattgttggcgttgtttggtt420ttgagttttacggttgattgtagaggtcgtagtgcggtagggttgggtttgtttttcggg480gtttagggaagagcgcgtagtttacgttattcgtttttatgcgcgttatttgtttcgata540ataggtagtagttttggatattatggtttttagtttatcgtcgtttttagtttttcggtg600ttttcgggtttttacgaggagtttttttaggttttttttagtcgttttttgaatggattt660ttgcgtttggggttttttggagatatgtacgcgcggtcggagtttttttcgttagggttt720gcggttcgtagcgacgttttggtagttgtcgtagttttgtatggttacgggggtatgaat780ttgacggtgaatttcgttgcgttttacggttttggcgttttttttcgttatatgcgttag840tttattaaataggagtttatttgtaagtggttggcggtcgacggtatcgcgatttcgagt900ttttgttttaaaatttttagtattatgtacgagttggttacgtacgttatcgtggagtac960gtcggcggttcggaataggttaattatatttgtttttggga1001<210>38<211>972<212>dna<213>homosapiens<400>38agcccggggcggggtggggctggagctcctgtctcttggccagctgaatggaggcccagt60ggcaacacaggtcctgcctggggatcaggtctgctctgcaccccaccttgctgcctggag120ccgcccacctgacaacctctcatccctgctctgcagatccggtcccatccccactgccca180ccccacccccccagcactccacccagttcaacgttccacgaacccccagaaccagccctc240atcaacaggcagcaagaagggccccccgcccatcgccccacaacgccagccgggtgaacg300ttggcaggtcctgaggcagctggcaagacgcctgcagctgaaagatacaaggccagggac360aggacagtcccatccccaggaggcagggagtatacaggctggggaagtttgcccttgcgt420ggggtggtgatggaggaggctcagcaagtcttctggactgtgaacctgtgtctgccactg480tgtgctgggtggtggtcatctttcccaccaggctgtggcctctgcaaccttcaagggagg540agcaggtcccattggctgagcacagccttgtaccgtgaactggaacaagcagcctccttc600ctggccacaggttccatgtccttatatggactcatctttgcctattgcgacacacactca660gtgaacacctactacgcgctgcaaagagccccgcaggcctgaggtgcccccacctcacca720ctcttcctatttttgtgtaaaaatccagcttcttgtcaccacctccaaggagggggagga780ggaggaaggcaggttcctctaggctgagccgaatgcccctctgtggtcccacgccactga840tcgctgcatgcccaccacctgggtacacacagtctgtgattcccggagcagaacggaccc900tgcccacccggtcttgtgtgctactcagtggacagacccaaggcaagaaagggtgacaag960gacagggtcttc972<210>39<211>972<212>dna<213>人工序列(artificialsequence)<400>39agttcggggcggggtggggttggagtttttgttttttggttagttgaatggaggtttagt60ggtaatataggttttgtttggggattaggtttgttttgtattttattttgttgtttggag120tcgtttatttgataattttttatttttgttttgtagattcggttttatttttattgttta180ttttatttttttagtattttatttagtttaacgttttacgaatttttagaattagttttt240attaataggtagtaagaagggtttttcgtttatcgttttataacgttagtcgggtgaacg300ttggtaggttttgaggtagttggtaagacgtttgtagttgaaagatataaggttagggat360aggatagttttatttttaggaggtagggagtatataggttggggaagtttgtttttgcgt420ggggtggtgatggaggaggtttagtaagttttttggattgtgaatttgtgtttgttattg480tgtgttgggtggtggttattttttttattaggttgtggtttttgtaatttttaagggagg540agtaggttttattggttgagtatagttttgtatcgtgaattggaataagtagtttttttt600ttggttataggttttatgtttttatatggatttatttttgtttattgcgatatatattta660gtgaatatttattacgcgttgtaaagagtttcgtaggtttgaggtgtttttattttatta720ttttttttatttttgtgtaaaaatttagttttttgttattatttttaaggagggggagga780ggaggaaggtaggtttttttaggttgagtcgaatgtttttttgtggttttacgttattga840tcgttgtatgtttattatttgggtatatatagtttgtgattttcggagtagaacggattt900tgtttattcggttttgtgtgttatttagtggatagatttaaggtaagaaagggtgataag960gatagggttttt972<210>40<211>22<212>dna<213>人工序列(artificialsequence)<400>40tagaggtcggagttggaattcg22<210>41<211>20<212>dna<213>人工序列(artificialsequence)<400>41acttttccgccctaaactcg20<210>42<211>24<212>dna<213>人工序列(artificialsequence)<400>42cggggaggtttcggagtttaggat24<210>43<211>22<212>dna<213>人工序列(artificialsequence)<400>43ggatgattttcgggaattattc22<210>44<211>24<212>dna<213>人工序列(artificialsequence)<400>44ctaaactcgaaatcctaaactccg24<210>45<211>22<212>dna<213>人工序列(artificialsequence)<400>45tcgggaaaggggaggacgggga22<210>46<211>20<212>dna<213>人工序列(artificialsequence)<400>46tttagcgggaattcgttacg20<210>47<211>21<212>dna<213>人工序列(artificialsequence)<400>47ccaaaataaccacgctaaccg21<210>48<211>24<212>dna<213>人工序列(artificialsequence)<400>48cgggtagttttgtttcgggttcgg24<210>49<211>21<212>dna<213>人工序列(artificialsequence)<400>49ggtagttttgtttcgggttcg21<210>50<211>18<212>dna<213>人工序列(artificialsequence)<400>50actcgcctttccgtttcg18<210>51<211>24<212>dna<213>人工序列(artificialsequence)<400>51cggttagcgtggttattttggggg24<210>52<211>22<212>dna<213>人工序列(artificialsequence)<400>52tagggcggaaaagtttggtacg22<210>53<211>18<212>dna<213>人工序列(artificialsequence)<400>53accaaactccccgactcg18<210>54<211>25<212>dna<213>人工序列(artificialsequence)<400>54cgattggggtttttgaaaagggggt25<210>55<211>22<212>dna<213>人工序列(artificialsequence)<400>55gggtaatagcgattgattgtcg22<210>56<211>22<212>dna<213>人工序列(artificialsequence)<400>56ctataatcgaaacaccgtaccg22<210>57<211>24<212>dna<213>人工序列(artificialsequence)<400>57cgtttcggggagaggttaatgggt24<210>58<211>24<212>dna<213>人工序列(artificialsequence)<400>58gttggtaaatattgtagggtttcg24<210>59<211>20<212>dna<213>人工序列(artificialsequence)<400>59cgaaacgcgacaatcaatcg20<210>60<211>27<212>dna<213>人工序列(artificialsequence)<400>60cgataattggggtgattacgtttgggt27<210>61<211>22<212>dna<213>人工序列(artificialsequence)<400>61ttgagattttcggaggaaatcg22<210>62<211>24<212>dna<213>人工序列(artificialsequence)<400>62caaatattttaataaccccaaccg24<210>63<211>27<212>dna<213>人工序列(artificialsequence)<400>63cgttgtggaagtttttacggttttgcg27<210>64<211>25<212>dna<213>人工序列(artificialsequence)<400>64gggttgtagacgtacgtatttaaac25<210>65<211>25<212>dna<213>人工序列(artificialsequence)<400>65taaacgaatataaaatccactcacg25<210>66<211>17<212>dna<213>人工序列(artificialsequence)<400>66ggtagaggcgtagaggg17<210>67<211>25<212>dna<213>人工序列(artificialsequence)<400>67ttttattaattagcgtttgtatcgc25<210>68<211>20<212>dna<213>人工序列(artificialsequence)<400>68gactcaaaaccgtaactccg20<210>69<211>21<212>dna<213>人工序列(artificialsequence)<400>69cggttgcggatagggaagtgg21<210>70<211>20<212>dna<213>人工序列(artificialsequence)<400>70ggagttacggttttgagtcg20<210>71<211>20<212>dna<213>人工序列(artificialsequence)<400>71acctatatccccgaaatccg20<210>72<211>24<212>dna<213>人工序列(artificialsequence)<400>72cgtgcgggcgttttaggttaggta24<210>73<211>20<212>dna<213>人工序列(artificialsequence)<400>73ttgcgttggaataggagtcg20<210>74<211>20<212>dna<213>人工序列(artificialsequence)<400>74caaactacgcgactactacg20<210>75<211>24<212>dna<213>人工序列(artificialsequence)<400>75cgggtgatggtgatggtttagggt24<210>76<211>20<212>dna<213>人工序列(artificialsequence)<400>76gatgcgcggttatgtttacg20<210>77<211>20<212>dna<213>人工序列(artificialsequence)<400>77aaataaccaacccgcgttcg20<210>78<211>24<212>dna<213>人工序列(artificialsequence)<400>78cgtagtcgtgtagttgcggcgtag24<210>79<211>22<212>dna<213>人工序列(artificialsequence)<400>79ggtagtttgttcgtgaagttcg22<210>80<211>22<212>dna<213>人工序列(artificialsequence)<400>80acacaacctctttactacatcg22<210>81<211>24<212>dna<213>人工序列(artificialsequence)<400>81cgtgtggtcgtgtgggttttcgaa24<210>82<211>19<212>dna<213>人工序列(artificialsequence)<400>82gtggtcggtagtttgttcg19<210>83<211>19<212>dna<213>人工序列(artificialsequence)<400>83aacttcgaaaacccacacg19<210>84<211>21<212>dna<213>人工序列(artificialsequence)<400>84cggggaagaggaggtggttcg21<210>85<211>22<212>dna<213>人工序列(artificialsequence)<400>85gtttgtttgtcggaatttgacg22<210>86<211>22<212>dna<213>人工序列(artificialsequence)<400>86cttaaacctaaaacccaaaccg22<210>87<211>24<212>dna<213>人工序列(artificialsequence)<400>87cgaggttgcgtttgtgcgatttgg24<210>88<211>19<212>dna<213>人工序列(artificialsequence)<400>88tgtcggaatttgacgttcg19<210>89<211>20<212>dna<213>人工序列(artificialsequence)<400>89acccaaaccgaaaatcaacg20<210>90<211>24<212>dna<213>人工序列(artificialsequence)<400>90cgaggttgcgtttgtgcgatttgg24<210>91<211>22<212>dna<213>人工序列(artificialsequence)<400>91tcgggttaaacgtaaagtagtc22<210>92<211>22<212>dna<213>人工序列(artificialsequence)<400>92aactaaacgacgaaaaaatacg22<210>93<211>24<212>dna<213>人工序列(artificialsequence)<400>93agtaacgtaggttttagcgttcgt24<210>94<211>20<212>dna<213>人工序列(artificialsequence)<400>94aaaggtggttacgtcgatcg20<210>95<211>22<212>dna<213>人工序列(artificialsequence)<400>95gattttccctcctcgactaacg22<210>96<211>24<212>dna<213>人工序列(artificialsequence)<400>96cggcgtttaggagtatcgtggttg24<210>97<211>21<212>dna<213>人工序列(artificialsequence)<400>97cgagtaacgtaggttttagcg21<210>98<211>22<212>dna<213>人工序列(artificialsequence)<400>98ctttccccgaacaaccttaacg22<210>99<211>26<212>dna<213>人工序列(artificialsequence)<400>99cgtcgtttagtttttgtcgagagggt26<210>100<211>20<212>dna<213>人工序列(artificialsequence)<400>100aagtcgtagcgtttaagggc20<210>101<211>24<212>dna<213>人工序列(artificialsequence)<400>101cactaacttacgatatcgaccgat24<210>102<211>18<212>dna<213>人工序列(artificialsequence)<400>102taggatcgagaggttcga18<210>103<211>25<212>dna<213>人工序列(artificialsequence)<400>103tattgttattttaacgtgtattcgc25<210>104<211>25<212>dna<213>人工序列(artificialsequence)<400>104caaataaaatccgaaatatctaccg25<210>105<211>21<212>dna<213>人工序列(artificialsequence)<400>105cggtttatgttagtcgtttgg21<210>106<211>21<212>dna<213>人工序列(artificialsequence)<400>106gtgaatttcgttgcgttttac21<210>107<211>24<212>dna<213>人工序列(artificialsequence)<400>107gataacgtacgtaaccaactcgta24<210>108<211>22<212>dna<213>人工序列(artificialsequence)<400>108gtaagtggttggcggtcgacgg22<210>109<211>20<212>dna<213>人工序列(artificialsequence)<400>109cgacgttttggtagttgtcg20<210>110<211>18<212>dna<213>人工序列(artificialsequence)<400>110cgccaaaaccgtaaaacg18<210>111<211>24<212>dna<213>人工序列(artificialsequence)<400>111cgggggtatgaatttgacggtgaa24<210>112<211>24<212>dna<213>人工序列(artificialsequence)<400>112gggatttggtttaatagtaattcg24<210>113<211>19<212>dna<213>人工序列(artificialsequence)<400>113cgacctctacaatcaaccg19<210>114<211>24<212>dna<213>人工序列(artificialsequence)<400>114cggcggaggtgtttggttttgttt24<210>115<211>24<212>dna<213>人工序列(artificialsequence)<400>115ttttggatttaaggggaagataaa24<210>116<211>27<212>dna<213>人工序列(artificialsequence)<400>116tttttccttctctacatctttctacct27<210>117<211>28<212>dna<213>人工序列(artificialsequence)<400>117aagggaaattgagaaatgagagaaggga28<210>118<211>24<212>dna<213>人工序列(artificialsequence)<400>118tttaaagggttcgtcgtttaagtc24<210>119<211>19<212>dna<213>人工序列(artificialsequence)<400>119aaacgattactttcgcccg19<210>120<211>26<212>dna<213>人工序列(artificialsequence)<400>120ttagaaggtaggaggcggaaaattag26当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1