一种成骨发育不全疾病的致病突变及其检测试剂的制作方法
本发明属于生物医药领域,涉及一种成骨发育不全疾病的致病突变及其检测试剂。
背景技术:
成骨不全症(osteogenesisimperfecta,oi),是一种少见的先天性骨骼发育障碍性疾病,又称脆骨病,瓷娃娃,或脆骨-蓝巩膜-耳聋综合征。特征为骨质脆弱、蓝巩膜、耳聋、关节松弛,是一种由于间充质组织发育不全,胶原形成障碍而造成的先天性遗传性疼痛。患儿易发骨折,轻微的碰撞,也会造成严重的骨折。目前oi无特殊治疗方法,主要是预防骨折,药物治疗疗效不肯定,干细胞治疗及基因治疗方法有待进一步研究、鉴定,短时间内还不能应用于临床。oi具有遗传性和家族性,但也有少数为单发病例。oi在我国乃至世界范围内均有较高的发病率,约10万分之3,发病男女的比例大约相同。我国是oi遗传资源大国,但目前oi相关的遗传学信息多来自西方国家,因此对我国oi患者进行深入的遗传学研究,探寻潜在的oi相关的新致病基因及致病突变显得尤为重要。
oi为单基因遗传病,其常见的遗传模式有常染色体显性遗传及常染色体隐性遗传。oi发生主要是由于组成i型胶原的α1或α2前胶原(pro-α1或pro-α2)链的基因(即col1a1和col1a2)的突变,导致i型胶原合成障碍,结缔组织中胶原量尤其是i型胶原含量下降,胶原是骨骼、皮肤、巩膜及牙本质等组织的主要胶原成分,因而这些部位的病变更明显。根据基因突变可分成4-11种类型,但有争议,且不同类型的成骨不全症严重程度和预后不同。。目前仍有很多oi患者的致病基因尚未找到,尤其在胎儿期,很多临床表现无法在子宫内通过超声等技术方法显现出来,因此有可能跟骨骼发育异常的多种遗传病混淆,提示存在大量oi等骨骼发育异常的新致病基因及新致病突变有待挖掘。
针对oi的分子遗传学研究必须建立在一定的分子生物学技术的基础上。研究oi致病基因的一个重要目的是进行oi的分子诊断,如何检测众多致病基因突变是目前的难题之一。基因连锁分析的定位克隆策略是鉴定单基因遗传病致病基因的经典方法,但是同时也面临一些困难:(1)通常需要多代家系,难以分析小家系和散发病例。(2)有时多代家系也不能定位致病位点。(3)难以在连锁区域内筛选出正确的致病基因。产前诊断依靠超声学检查胎儿的骨骼系统也仅可以发现少数骨发育障碍性疾病,且难以区分不同类型的骨骼发育异常。因此,鉴于胎儿期oi疾病难以区分的性质及传统分析技术的局限性,寻求一种全新的oi致病基因的研究方法显得尤为迫切。
col1a1位于17号染色体长臂17q21-22位置,该基因长18kb,有51个外显子。每个ⅰ型胶原的三胶螺旋区都含有338个连续重复的三氨基酸gxy,其中g为甘氨酸,x和y通常为脯氨酸和羟脯氨酸。甘氨酸的存在对三胶螺旋的形成是必须的,在这一区域中点突变引起的甘氨酸残基被替代,以及剪切位点突变引起的外显子丢失,是ⅰ型胶原基因突变的主要形式。其他突变如插入、缺失、重复等相对较少。col1a1基因外显子很多,迄今为止,已有百余个突变被报道,然而c.1822g>a基因突变引起oi从未被报道或得到证实。
技术实现要素:
本发明的目的是针对上述缺陷,提供一种成骨发育不全疾病的新致病突变。
本发明的另一目的是提供该致病突变的应用。
本发明的目的可通过如下技术方案实现:
一种用于检测成骨发育不全疾病的突变的col1a1基因,突变的col1a1为杂合突变或纯合突变c.1822g>a,野生型col1a1基因在ncbi数据库中的基因编号为:nm_000088.3,该基因cds第1822bp处的碱基由g突变为a,其他部分与野生型相同。野生型的col1a1基因cds序列如seqidno.1所示。
一种突变的col1a1蛋白,野生型col1a1蛋白在ncbi数据库中的基因转录本编号为:np_000079,突变的col1a1蛋白在该野生型蛋白的第608位氨基酸由甘氨酸突变为丝氨酸,其他部分与野生型相同。野生型的col1a1蛋白氨基酸序列如seqidno.2所示,
检测本发明所述的突变的col1a1基因或者所述的突变的col1a1蛋白的试剂在制备成骨发育不全疾病检测试剂或检测设备中的应用。
其所述的检测试剂优选自引物或引物对、探针、抗体、或核酸芯片、高通量测序、sanger测序中的一种或多种。
所述的检测设备优选包括含有检测突变的col1a1基因的基因芯片、高通量测序、sanger测序的检测平台。
一种检测成骨发育不全疾病的试剂盒,所述的试剂盒包括:
(1)检测col1a1基因cds第1822bp处核苷酸的试剂;或检测col1a1蛋白第608位氨基酸位点的试剂;
(2)产品使用说明书,其中明确记载col1a1基因cds第1822bp处核苷酸由g突变为a,或者col1a1蛋白第608位氨基酸位点由g变为s为成骨发育不全的致病突变。
其中,所述的试剂优选自引物或引物对、探针、抗体、或核酸芯片。
作为本发明的一种优选,所述的试剂为基于深度测序为平台的基因芯片杂交探针。
所述的试剂进一步优选检测col1a1基因cds第1822bp处核苷酸的的引物对;更进一步优选由5'-tggcgctgtcgtaagtat-3'(seqidno.3)和5'-cctgtaggtgggaaatgg-3'(seqidno.4)组成的引物对。
所述的试剂盒中检测col1a1基因cds第1822bp处核苷酸的基因芯片杂交探针序列优选如seqidno.5所示。
一种以深度测序为平台筛查oi患者中col1a1基因新突变,斑马鱼突变模型结合sift和polyphen蛋白功能预测来验证该基因突变为致病基因突变的方法:包含以下步骤:
(1)对于胎儿超声显示骨骼发育异常,或有oi遗传病史的家系,收集临床资料及血液、组织等含有dna的标本,提取基因组dna;
(2)检测与骨发育异常的一系列相关基因,包括基因adamtsl2,agps,ankh,arse,ccdc8,chst3,col10a1,col2a1,col9a1,col9a2,col9a3,comp,ctsk,cul7,dll3,ebp,evc,evc2,fbn1,fgfr1,fgfr2,fgfr3,flnb,gnas,gnpat,hes7,lfng,lmna,matn3,mesp2,obsl1,pex7,pth1r,ror2,runx2,slc26a2,slc35d1,smarcal1,sost,sox9,tgfb1,tnfrs,f11a,trappc2,trem2,tyrobp,wnt5a,wnt7a,zmpste24,col1a1,col1a2,crtap,p3h1,serpinf1,ifitm5,fkbp10,ppib,sp7,bmp1,serpinh1,tmem38b,wnt1b,wnt1b,wnt1。
(3)将dna打断并制备文库,然后通过芯片对目标基因编码区及临近剪切的dna进行捕获和富集,最后使用高通量测序平台进行突变检测。
(4)对测序结果进行优化的生物信息学分析,筛选到一个新的oi致病突变为col1a1.gly608ser。突变位于17号染色体,物理位置为48270211(ncbi数据库)的碱基由g突变为a;蛋白质水平:col1a1基因编码蛋白第608位氨基酸由甘氨酸突变为丝氨酸。
(5)步骤(3)所述的高通量测序,测序目标区长度为131271bp,目标区覆盖度达到至少99.95%,目标区平均深度至少171.42x,目标区平均深度>30x位点所占比例至少为96.99%。
(6)对于新突变位点col1a1.gly608ser,用sift和polyphen对其进行蛋白功能预测。
(7)利用斑马鱼模型验证col1a1基因上c.1822g>a点突变后影响骨骼发育,在ensembl数据库中找到与人col1a1基因高度相似的基因col1a1a(ensdarg00000012405),通过在野生型斑马鱼中表达col1a1a同样点突变基因来模拟在人体中的显性表达,观察斑马鱼胚胎的骨骼发育情况验证新突变位点col1a1.c.1822g>a导致骨骼发育异常oi。
有益效果
1.oi是严重的先天性骨骼发育障碍性疾病,在我国遗传病中的发病率较高,危害了国民健康。目前oi没有有效的治疗手段,预防主要是进行产前诊断,预防患儿的出生。oi具有明显的遗传异质性,分为常染色体显性遗传和常染色体隐性遗传,目前已知突变上百种,但仍存在大量未知的致病基因。挖掘oi新致病突变基因有利于进一步探索oi的分子遗传学病因,从而帮助了解发病机制、辅助临床诊断、产前诊断和转基因治疗。
2.首次报道了oi致病基因col1a1中新的突变位点c.1822g>a,为常染色体显性遗传病,无论杂合突变还是纯合突变,均致病。
3.提供了一种发现新突变的方案,即通过设计针对某种人体系统的系列基因检测panel,对生物样本提取dna进行高通量测序检测,生物信息学分析新发突变的有害性,在斑马鱼等动物模型上进行基因敲出验证。本发明提供了oi新的致病位点,为该疾病的诊断提供了新的分子生物学基础。
附图说明
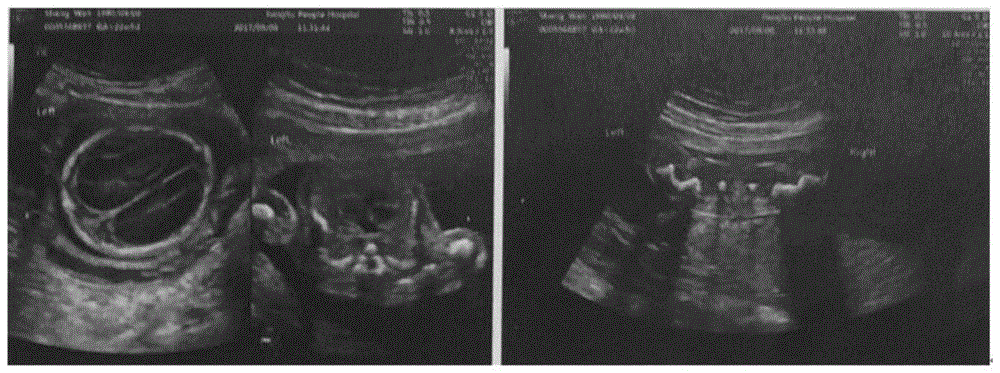
图1胎儿超声结果图
图2家系sanger测序结果
图3人col1a1基因与斑马鱼中的同源基因突变位置的保守性,本发明所述突变位点(g1822a)及其在斑马鱼中的对应突变位点用下划线表示
图4斑马鱼突变处序列的核苷酸和氨基酸对应图
图5构建斑马鱼转录及显微注射质粒
图6新位点在各数据库中的突变频率
图7新位点在各数据库中sift预测结果。
图8新位点在各数据库中polyphen预测结果。
图9野生型斑马鱼及col1a1基因c.1822g>a突变型斑马鱼
具体实施方式
本发明人经过广泛而深入的研究,发现了一种oi相关基因col1a1的新突变位点,可用于诊断上述疾病,以及用于开发对于上述疾病有效的基因治疗药物。
在检测相关位点的变异时,检测可以针对基因组dna,也可以针对cdna或mrna,或针对蛋白质。可用已有的技术如western印迹法、southern印迹法、dna序列分析、pcr和原位杂交检测突变等。
可采用各种技术来检测野生型col1a1基因(seqidno.1)第1822位是否存在g到a的突变,这些技术包含在本发明中。例如,基于相关位点制备基因芯片和高通量测序捕获探针。此外,可用相关位点特异的引物进行pcr来进行鉴定;或可根据相关位点设计可特异性结合的探针来进行鉴定;或可利用特异性的限制性内切酶来进行鉴定。
作为一种可选的方式,还可采用基于pcr技术的单碱基延伸技术来检测变异位点,其原理是设计一条引物,位于待测变异位点的上游,且该引物的3'端距离变异位点一个碱基。加入不同荧光标记的ddntp进行反应,或者通过焦磷酸测序加入dntp及相关反应用酶,只有当加入的ddntp或dntp与变异位点碱基互补时,引物才得以延伸。可通过检测延伸碱基所发出的荧光或者焦磷酸测序中系列酶反应发出的可见光来判断变异的类型。
本发明还包括用于在分析物中检测是否含有所述变异位点(col1a1基因cds第1822位是否存在g到a的突变)的试剂。所述的试剂例如是:对相关突变位点特异的引物,扩增出的扩增产物含有对应于col1a1基因第1822位的碱基;对相关突变位点特异的探针,可与发生突变区域发生特异性结合而不可与未发生突变区域特异性结合,且所述探针带有可检测信号;或对相关突变位点特异的限制性内切酶。
所述的试剂盒中还可包括用于提取dna、rna、杂交、显色等所需的各种试剂,包括但不限于:抽提液、扩增液、杂交液、酶、对照液、显色液、洗液等。
此外,所述的试剂盒中还可包含使用说明书和核酸序列分析软件等。
下面结合具体实施例,进一步阐述本发明。
实施例1
对一个超声提示骨骼发育异常的胎儿进行遗传检测。
实验方法:
1.孕妇系列超声结果的收集,及家族遗传史等病例资料的采集:收集该家系中各成员的临床资料和血液样本,胎儿的父母采集外周血,对孕妇行脐带血穿刺取脐带血对胎儿进行遗传诊断。用血液基因组dna提取试剂盒(天根生化科技有限公司)对家系中各成员的血液基因组dna进行提取。
2.采用高通量测序技术挖掘该家系的致病突变:检测与骨发育异常相关的61个基因,首先将基因组dna片段化,并进行末端标记,与基因组dna进行液相杂交,将目标基因组区域的dna片段进行富集后再利用第二代测序技术进行测序。具体方案是基于120mer的rna寡核苷酸探针或者叫“baits”。baits上连接的生物素,可以被链霉亲和素标记的磁珠吸附。打断后的基因组片段,与baits进行杂交,捕获目标片段。利用磁珠吸附出带有baits的dna片段后,进行磁珠洗脱、rna探针降解,最终获得目标区域dna片段。
3.标准信息分析的流程包括:去除接头污染和低质量数据,数据通过bwa与ucschg19数据库进行比对,数据产量统计分析、测序深度分析、覆盖度均一性分析,snp变异信息检测(samtools、soapsnp、gatk),snp的refgene注释,snp数据库分析(与dbsnp、千人基因组数据、esp外显子组数据库以及炎黄基因组(仅亚太地区)数据进行数据库注释分析),snp保守性预测、致病性分析(仅针对人类样本,软件:sift、polyphen-2、phylop、gerpscores、mutationassessor、condel、fathmm),snp在各基因功能元件上的分布统计,indel变异信息检测(samtools、gatk),indel的refgene注释,indel数据库分析(与dbsnp、千人基因组数据、esp外显子组数据库、炎黄基因组(仅亚太地区)进行数据库注释分析),indel在各基因功能元件上的分布统计。
4.经sanger测序验证,鉴定致病基因:pcr法分别针对筛选出的突变位点及邻近dna序列在相应家系中进行扩增,所用引物序列采用primer5引物设计软件设计,检测本发明所述致病突变的引物对序列如seqidno.3和seqidno.4所示。所用pcr的反应体系(50μl体系)为:10*buffer5μl,25mmmgcl23μl,taqdna聚合酶5u,dntp混合物2mm,正反向引物各1.2μm,灭菌蒸馏水加至50μl。放入pcr仪中,94℃,3min;(94℃,25s;55℃,25s;72℃15s)35个循环;72℃7min;4℃保存。2%琼脂糖电泳后凝胶成像仪检测,加入marker判定片段大小,单一条带且片段大小符合的样本进行sanger测序,判定位点是否发生突变。
实验结果:
1.超声科主任对孕妇腹中胎儿进行超声检测,发现胎儿颅骨形态异常,呈“草莓”型,胎儿双侧股骨弯曲成角,双侧胫骨弯曲,四肢发育不良,胎儿心胸比增大,主、肺动脉管径比例异常,三尖瓣轻度反流(图1),初步判断可能为成骨发育不全,而家族中未出现过类似症状的成员。
2.通过对胎儿脐血样本dna进行目标区域捕获测序及生物信息学分析后,发现胎儿在col1a1基因上存在c.1822g>a突变,为vous突变,即临床意义不明的突变,未发现其他可疑的致病基因突变位点。经sanger测序验证证实该基因位点的突变从未在oi患者中发现,也未在胎儿父母的外周血dna中发现(图2)。
3.根据本发明的设计方案,成功证实所检测到的该col1a1基因c.1822g>a突变为oi新致病位点。
实施例2:
针对实施例1中所检测出的致病基因进行功能学研究及基因敲除动物模型研究,此处以检测到的col1a1基因新突变c.1822g>a为例。
实验方法:
1.保守型分析:对该位点在各数据库中发生的频率进行评估。
2.根据sift和polyphen值预测突变的致病能力。
3.基因敲除的动物模型证实突变位点为致病突变位点。
(1)分析col1a1在斑马鱼中的同源基因及点突变位置,选择正确的斑马鱼中同源基因用于制备点突变;在ensembl网站中找到与humancol1a1基因高度相似基因,为ensdarg00000012405,分析突变位置的保守性,如图3。上述比对结果表明,该位点在斑马鱼的基因中保守,暗示其位点功能的重要性。为验证斑马鱼该位点突变导致类似的表型,选择斑马鱼中与humancol1a1相似度较高的基因col1a1a(ensdarg00000012405)进行实验。
(2)验证col1a1(g1822a)点突变功能的方法:在人类中,col1a1(g1822a)在胚胎期呈现显性的骨骼发育异常表型,因此可以通过在野生型斑马鱼中表达col1a1a同样点突变基因来模拟在人体中的显性表达,进而通过观察斑马鱼胚胎的骨骼发育情况来验证。以col1a1a基因的转录本ensdart00000009393.8的序列为参照,设计引物克隆该基因全长以及构建col1a1a(g1774a)点突变,突变处序列的核苷酸和氨基酸对应图如图4所示。
(3)构建斑马鱼中表达col1a1a(g1774a)的质粒:突变点:g1774a,突变引物:已原始质粒为模板,扩增载体骨架,选用引物col1a1a-vf(5'-gcccagtctgcttcttgtaaggatccaccggatctagata-3')和col1a1a-vr(5'-gcttaaacaagaatctctag-3');已原始质粒为模板,扩增启动子区,选用引物cmv-t7-f(5'-ctagagattcttgtttaagcgacattgattattgactag-3')和cmv-t7-r(5'-aatatccacaaagctgaacatggtggcaagcttaactagccagcttg-3');已斑马鱼基因组dna为模板,扩增col1a1am片段1,选用引物col1a1a-f1(5'-atgttcagctttgtggatat-3')和col1a1a-r1(5'-tagtggcaccaatagcaccc-3');为了克服非特异性扩增,在col1a1am片段2的两端延长一段序列设计引物,引物序列如下5'-gaggaaggcaagagaggacc-3'及5'-ccagggggattttacacgct-3',以斑马鱼基因组dna为模板,进行pcr扩增,以pcr回收产物为模板,用扩增col1a1am片段2引物col1a1a-f2(5'-gggtgctattggtgccactagcgctcctggtaaggatggt-3')和col1a1a-r2(5'-ttacaagaagcagactgggc-3')进行扩增获得col1a1am片段2。运用同源重组的方法合成质粒,质粒构建完成后,送至测序公司进行测序,经测序结果分析,质粒构建正确,构建成功的质粒序列如seqidno.5所示;菌种保存,质粒提取及纯化。质粒纯化后的浓度为400ng/μl。表达col1a1a(g1774a)的方法选用dna显微注射(利用tol2转座酶介导高效转基因),利用显微注射构建的过表达质粒(图5)和转座酶mrna来表达,通过表型来确认是否有影响。
(4)表达col1a1a(g1774a)后的表型观察:显微注射后,持续观察其整体形态发育,尤其关注躯干骨骼发育情况(有无弯曲)。
实验结果:
1.col1a1;nm_000088.3;c.1822g>a;p.gly608ser|p.g608s;ex27;cds27:错义突变,暂无该位点致病性的相关文献报道。用sift和polyphen对其进行蛋白功能预测,结果均为有害,该位点在正常人中发生的概率极低(图6-8)。col1a1基因相关的成骨不全1/2/3/4型均为常染色体显性遗传。
2.通过脊柱弯曲表型分析发现,注射col1a1a质粒导致斑马鱼脊柱发生弯曲(图9)。
序列表
<110>黄欢
<120>一种成骨发育不全疾病的致病突变及其检测试剂
<160>5
<170>siposequencelisting1.0
<210>1
<211>4395
<212>dna
<213>人类(homosapiens)
<400>1
atgttcagctttgtggacctccggctcctgctcctcttagcggccaccgccctcctgacg60
cacggccaagaggaaggccaagtcgagggccaagacgaagacatcccaccaatcacctgc120
gtacagaacggcctcaggtaccatgaccgagacgtgtggaaacccgagccctgccggatc180
tgcgtctgcgacaacggcaaggtgttgtgcgatgacgtgatctgtgacgagaccaagaac240
tgccccggcgccgaagtccccgagggcgagtgctgtcccgtctgccccgacggctcagag300
tcacccaccgaccaagaaaccaccggcgtcgagggacccaagggagacactggcccccga360
ggcccaaggggacccgcaggcccccctggccgagatggcatccctggacagcctggactt420
cccggaccccccggaccccccggacctcccggaccccctggcctcggaggaaactttgct480
ccccagctgtcttatggctatgatgagaaatcaaccggaggaatttccgtgcctggcccc540
atgggtccctctggtcctcgtggtctccctggcccccctggtgcacctggtccccaaggc600
ttccaaggtccccctggtgagcctggcgagcctggagcttcaggtcccatgggtccccga660
ggtcccccaggtccccctggaaagaatggagatgatggggaagctggaaaacctggtcgt720
cctggtgagcgtgggcctcctgggcctcagggtgctcgaggattgcccggaacagctggc780
ctccctggaatgaagggacacagaggtttcagtggtttggatggtgccaagggagatgct840
ggtcctgctggtcctaagggtgagcctggcagccctggtgaaaatggagctcctggtcag900
atgggcccccgtggcctgcctggtgagagaggtcgccctggagcccctggccctgctggt960
gctcgtggaaatgatggtgctactggtgctgccgggccccctggtcccaccggccccgct1020
ggtcctcctggcttccctggtgctgttggtgctaagggtgaagctggtccccaagggccc1080
cgaggctctgaaggtccccagggtgtgcgtggtgagcctggcccccctggccctgctggt1140
gctgctggccctgctggaaaccctggtgctgatggacagcctggtgctaaaggtgccaat1200
ggtgctcctggtattgctggtgctcctggcttccctggtgcccgaggcccctctggaccc1260
cagggccccggcggccctcctggtcccaagggtaacagcggtgaacctggtgctcctggc1320
agcaaaggagacactggtgctaagggagagcctggccctgttggtgttcaaggaccccct1380
ggccctgctggagaggaaggaaagcgaggagctcgaggtgaacccggacccactggcctg1440
cccggaccccctggcgagcgtggtggacctggtagccgtggtttccctggcgcagatggt1500
gttgctggtcccaagggtcccgctggtgaacgtggttctcctggccctgctggccccaaa1560
ggatctcctggtgaagctggtcgtcccggtgaagctggtctgcctggtgccaagggtctg1620
actggaagccctggcagccctggtcctgatggcaaaactggcccccctggtcccgccggt1680
caagatggtcgccccggacccccaggcccacctggtgcccgtggtcaggctggtgtgatg1740
ggattccctggacctaaaggtgctgctggagagcccggcaaggctggagagcgaggtgtt1800
cccggaccccctggcgctgtcggtcctgctggcaaagatggagaggctggagctcaggga1860
ccccctggccctgctggtcccgctggcgagagaggtgaacaaggccctgctggctccccc1920
ggattccagggtctccctggtcctgctggtcctccaggtgaagcaggcaaacctggtgaa1980
cagggtgttcctggagaccttggcgcccctggcccctctggagcaagaggcgagagaggt2040
ttccctggcgagcgtggtgtgcaaggtccccctggtcctgctggtccccgaggggccaac2100
ggtgctcccggcaacgatggtgctaagggtgatgctggtgcccctggagctcccggtagc2160
cagggcgcccctggccttcagggaatgcctggtgaacgtggtgcagctggtcttccaggg2220
cctaagggtgacagaggtgatgctggtcccaaaggtgctgatggctctcctggcaaagat2280
ggcgtccgtggtctgactggccccattggtcctcctggccctgctggtgcccctggtgac2340
aagggtgaaagtggtcccagcggccctgctggtcccactggagctcgtggtgcccccgga2400
gaccgtggtgagcctggtccccccggccctgctggctttgctggcccccctggtgctgac2460
ggccaacctggtgctaaaggcgaacctggtgatgctggtgctaaaggcgatgctggtccc2520
cctggccctgccggacccgctggaccccctggccccattggtaatgttggtgctcctgga2580
gccaaaggtgctcgcggcagcgctggtccccctggtgctactggtttccctggtgctgct2640
ggccgagtcggtcctcctggcccctctggaaatgctggaccccctggccctcctggtcct2700
gctggcaaagaaggcggcaaaggtccccgtggtgagactggccctgctggacgtcctggt2760
gaagttggtccccctggtccccctggccctgctggcgagaaaggatcccctggtgctgat2820
ggtcctgctggtgctcctggtactcccgggcctcaaggtattgctggacagcgtggtgtg2880
gtcggcctgcctggtcagagaggagagagaggcttccctggtcttcctggcccctctggt2940
gaacctggcaaacaaggtccctctggagcaagtggtgaacgtggtccccctggtcccatg3000
ggcccccctggattggctggaccccctggtgaatctggacgtgagggggctcctggtgcc3060
gaaggttcccctggacgagacggttctcctggcgccaagggtgaccgtggtgagaccggc3120
cccgctggaccccctggtgctcctggtgctcctggtgcccctggccccgttggccctgct3180
ggcaagagtggtgatcgtggtgagactggtcctgctggtcccgccggtcctgtcggccct3240
gttggcgcccgtggccccgccggaccccaaggcccccgtggtgacaagggtgagacaggc3300
gaacagggcgacagaggcataaagggtcaccgtggcttctctggcctccagggtccccct3360
ggccctcctggctctcctggtgaacaaggtccctctggagcctctggtcctgctggtccc3420
cgaggtccccctggctctgctggtgctcctggcaaagatggactcaacggtctccctggc3480
cccattgggccccctggtcctcgcggtcgcactggtgatgctggtcctgttggtcccccc3540
ggccctcctggacctcctggtccccctggtcctcccagcgctggtttcgacttcagcttc3600
ctgccccagccacctcaagagaaggctcacgatggtggccgctactaccgggctgatgat3660
gccaatgtggttcgtgaccgtgacctcgaggtggacaccaccctcaagagcctgagccag3720
cagatcgagaacatccggagcccagagggcagccgcaagaaccccgcccgcacctgccgt3780
gacctcaagatgtgccactctgactggaagagtggagagtactggattgaccccaaccaa3840
ggctgcaacctggatgccatcaaagtcttctgcaacatggagactggtgagacctgcgtg3900
taccccactcagcccagtgtggcccagaagaactggtacatcagcaagaaccccaaggac3960
aagaggcatgtctggttcggcgagagcatgaccgatggattccagttcgagtatggcggc4020
cagggctccgaccctgccgatgtggccatccagctgaccttcctgcgcctgatgtccacc4080
gaggcctcccagaacatcacctaccactgcaagaacagcgtggcctacatggaccagcag4140
actggcaacctcaagaaggccctgctcctccagggctccaacgagatcgagatccgcgcc4200
gagggcaacagccgcttcacctacagcgtcactgtcgatggctgcacgagtcacaccgga4260
gcctggggcaagacagtgattgaatacaaaaccaccaagacctcccgcctgcccatcatc4320
gatgtggcccccttggacgttggtgccccagaccaggaattcggcttcgacgttggccct4380
gtctgcttcctgtaa4395
<210>2
<211>1464
<212>prt
<213>人类(homosapiens)
<400>2
metpheserphevalaspleuargleuleuleuleuleualaalathr
151015
alaleuleuthrhisglyglnglugluglyglnvalgluglyglnasp
202530
gluaspileproproilethrcysvalglnasnglyleuargtyrhis
354045
aspargaspvaltrplysprogluprocysargilecysvalcysasp
505560
asnglylysvalleucysaspaspvalilecysaspgluthrlysasn
65707580
cysproglyalagluvalprogluglyglucyscysprovalcyspro
859095
aspglysergluserprothraspglngluthrthrglyvalglugly
100105110
prolysglyaspthrglyproargglyproargglyproalaglypro
115120125
proglyargaspglyileproglyglnproglyleuproglypropro
130135140
glyproproglyproproglyproproglyleuglyglyasnpheala
145150155160
proglnleusertyrglytyraspglulysserthrglyglyileser
165170175
valproglyprometglyproserglyproargglyleuproglypro
180185190
proglyalaproglyproglnglypheglnglyproproglyglupro
195200205
glygluproglyalaserglyprometglyproargglyproprogly
210215220
proproglylysasnglyaspaspglyglualaglylysproglyarg
225230235240
proglygluargglyproproglyproglnglyalaargglyleupro
245250255
glythralaglyleuproglymetlysglyhisargglyphesergly
260265270
leuaspglyalalysglyaspalaglyproalaglyprolysglyglu
275280285
proglyserproglygluasnglyalaproglyglnmetglyproarg
290295300
glyleuproglygluargglyargproglyalaproglyproalagly
305310315320
alaargglyasnaspglyalathrglyalaalaglyproproglypro
325330335
thrglyproalaglyproproglypheproglyalavalglyalalys
340345350
glyglualaglyproglnglyproargglysergluglyproglngly
355360365
valargglygluproglyproproglyproalaglyalaalaglypro
370375380
alaglyasnproglyalaaspglyglnproglyalalysglyalaasn
385390395400
glyalaproglyilealaglyalaproglypheproglyalaarggly
405410415
proserglyproglnglyproglyglyproproglyprolysglyasn
420425430
serglygluproglyalaproglyserlysglyaspthrglyalalys
435440445
glygluproglyprovalglyvalglnglyproproglyproalagly
450455460
glugluglylysargglyalaargglygluproglyprothrglyleu
465470475480
proglyproproglygluargglyglyproglyserargglyphepro
485490495
glyalaaspglyvalalaglyprolysglyproalaglygluarggly
500505510
serproglyproalaglyprolysglyserproglyglualaglyarg
515520525
proglyglualaglyleuproglyalalysglyleuthrglyserpro
530535540
glyserproglyproaspglylysthrglyproproglyproalagly
545550555560
glnaspglyargproglyproproglyproproglyalaargglygln
565570575
alaglyvalmetglypheproglyprolysglyalaalaglyglupro
580585590
glylysalaglygluargglyvalproglyproproglyalavalgly
595600605
proalaglylysaspglyglualaglyalaglnglyproproglypro
610615620
alaglyproalaglygluargglygluglnglyproalaglyserpro
625630635640
glypheglnglyleuproglyproalaglyproproglyglualagly
645650655
lysproglygluglnglyvalproglyaspleuglyalaproglypro
660665670
serglyalaargglygluargglypheproglygluargglyvalgln
675680685
glyproproglyproalaglyproargglyalaasnglyalaprogly
690695700
asnaspglyalalysglyaspalaglyalaproglyalaproglyser
705710715720
glnglyalaproglyleuglnglymetproglygluargglyalaala
725730735
glyleuproglyprolysglyaspargglyaspalaglyprolysgly
740745750
alaaspglyserproglylysaspglyvalargglyleuthrglypro
755760765
ileglyproproglyproalaglyalaproglyasplysglygluser
770775780
glyproserglyproalaglyprothrglyalaargglyalaprogly
785790795800
aspargglygluproglyproproglyproalaglyphealaglypro
805810815
proglyalaaspglyglnproglyalalysglygluproglyaspala
820825830
glyalalysglyaspalaglyproproglyproalaglyproalagly
835840845
proproglyproileglyasnvalglyalaproglyalalysglyala
850855860
argglyseralaglyproproglyalathrglypheproglyalaala
865870875880
glyargvalglyproproglyproserglyasnalaglyproprogly
885890895
proproglyproalaglylysgluglyglylysglyproargglyglu
900905910
thrglyproalaglyargproglygluvalglyproproglypropro
915920925
glyproalaglyglulysglyserproglyalaaspglyproalagly
930935940
alaproglythrproglyproglnglyilealaglyglnargglyval
945950955960
valglyleuproglyglnargglygluargglypheproglyleupro
965970975
glyproserglygluproglylysglnglyproserglyalasergly
980985990
gluargglyproproglyprometglyproproglyleualaglypro
99510001005
proglygluserglyarggluglyalaproglyalagluglyserpro
101010151020
glyargaspglyserproglyalalysglyaspargglygluthrgly
1025103010351040
proalaglyproproglyalaproglyalaproglyalaproglypro
104510501055
valglyproalaglylysserglyaspargglygluthrglyproala
106010651070
glyproalaglyprovalglyprovalglyalaargglyproalagly
107510801085
proglnglyproargglyasplysglygluthrglygluglnglyasp
109010951100
argglyilelysglyhisargglypheserglyleuglnglypropro
1105111011151120
glyproproglyserproglygluglnglyproserglyalasergly
112511301135
proalaglyproargglyproproglyseralaglyalaproglylys
114011451150
aspglyleuasnglyleuproglyproileglyproproglyproarg
115511601165
glyargthrglyaspalaglyprovalglyproproglyproprogly
117011751180
proproglyproproglyproproseralaglypheasppheserphe
1185119011951200
leuproglnproproglnglulysalahisaspglyglyargtyrtyr
120512101215
argalaaspaspalaasnvalvalargaspargaspleugluvalasp
122012251230
thrthrleulysserleuserglnglnilegluasnileargserpro
123512401245
gluglyserarglysasnproalaargthrcysargaspleulysmet
125012551260
cyshisserasptrplysserglyglutyrtrpileaspproasngln
1265127012751280
glycysasnleuaspalailelysvalphecysasnmetgluthrgly
128512901295
gluthrcysvaltyrprothrglnproservalalaglnlysasntrp
130013051310
tyrileserlysasnprolysasplysarghisvaltrppheglyglu
131513201325
sermetthraspglypheglnpheglutyrglyglyglnglyserasp
133013351340
proalaaspvalalaileglnleuthrpheleuargleumetserthr
1345135013551360
glualaserglnasnilethrtyrhiscyslysasnservalalatyr
136513701375
metaspglnglnthrglyasnleulyslysalaleuleuleuglngly
138013851390
serasngluilegluileargalagluglyasnserargphethrtyr
139514001405
servalthrvalaspglycysthrserhisthrglyalatrpglylys
141014151420
thrvalileglutyrlysthrthrlysthrserargleuproileile
1425143014351440
aspvalalaproleuaspvalglyalaproaspglnglupheglyphe
144514501455
aspvalglyprovalcyspheleu
1460
<210>3
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>3
tggcgctgtcgtaagtat18
<210>4
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>4
cctgtaggtgggaaatgg18
<210>5
<211>8733
<212>dna
<213>人工序列(artificialsequence)
<400>5
tatagtgagtcgtattacaattcactggccgtcgttttacaacgtcgtgactgggaaaac60
cctggcgttacccaacttaatcgccttgcagcacatccccctttcgccagctggcgtaat120
agcgaagaggcccgcaccgatcgcccttcccaacagttgcgcagcctgaatggcgaatgg180
acgcgccctgtagcggcgcattaagcgcggcgggtgtggtggttacgcgcagcgtgaccg240
ctacacttgccagcgccctagcgcccgctcctttcgctttcttcccttcctttctcgcca300
cgttcgccggctttccccgtcaagctctaaatcgggggctccctttagggttccgattta360
gtgctttacggcacctcgaccccaaaaaacttgattagggtgatggttcacgtagtgggc420
catcgccctgatagacggtttttcgccctttgacgttggagtccacgttctttaatagtg480
gactcttgttccaaactggaacaacactcaaccctatctcggtctattcttttgatttat540
aagggattttgccgatttcggcctattggttaaaaaatgagctgatttaacaaaaattta600
acgcgaattttaacaaaatattaacgcttacaatttcctgatgcggtattttctccttac660
gcatctgtgcggtatttcacaccgcatcaggtggcacttttcggggaaatgtgcgcggaa720
cccctatttgtttatttttctaaatacattcaaatatgtatccgctcatgagacaataac780
cctgataaatgcttcaataatattgaaaaaggaagagtatgagtattcaacatttccgtg840
tcgcccttattcccttttttgcggcattttgccttcctgtttttgctcacccagaaacgc900
tggtgaaagtaaaagatgctgaagatcagttgggtgcacgagtgggttacatcgaactgg960
atctcaacagcggtaagatccttgagagttttcgccccgaagaacgttttccaatgatga1020
gcacttttaaagttctgctatgtggcgcggtattatcccgtattgacgccgggcaagagc1080
aactcggtcgccgcatacactattctcagaatgacttggttgagtactcaccagtcacag1140
aaaagcatcttacggatggcatgacagtaagagaattatgcagtgctgccataaccatga1200
gtgataacactgcggccaacttacttctgacaacgatcggaggaccgaaggagctaaccg1260
cttttttgcacaacatgggggatcatgtaactcgccttgatcgttgggaaccggagctga1320
atgaagccataccaaacgacgagcgtgacaccacgatgcctgtagcaatggcaacaacgt1380
tgcgcaaactattaactggcgaactacttactctagcttcccggcaacaattaatagact1440
ggatggaggcggataaagttgcaggaccacttctgcgctcggcccttccggctggctggt1500
ttattgctgataaatctggagccggtgagcgtgggtctcgcggtatcattgcagcactgg1560
ggccagatggtaagccctcccgtatcgtagttatctacacgacggggagtcaggcaacta1620
tggatgaacgaaatagacagatcgctgagataggtgcctcactgattaagcattggtaac1680
tgtcagaccaagtttactcatatatactttagattgatttaaaacttcatttttaattta1740
aaaggatctaggtgaagatcctttttgataatctcatgaccaaaatcccttaacgtgagt1800
tttcgttccactgagcgtcagaccccgtagaaaagatcaaaggatcttcttgagatcctt1860
tttttctgcgcgtaatctgctgcttgcaaacaaaaaaaccaccgctaccagcggtggttt1920
gtttgccggatcaagagctaccaactctttttccgaaggtaactggcttcagcagagcgc1980
agataccaaatactgttcttctagtgtagccgtagttaggccaccacttcaagaactctg2040
tagcaccgcctacatacctcgctctgctaatcctgttaccagtggctgctgccagtggcg2100
ataagtcgtgtcttaccgggttggactcaagacgatagttaccggataaggcgcagcggt2160
cgggctgaacggggggttcgtgcacacagcccagcttggagcgaacgacctacaccgaac2220
tgagatacctacagcgtgagctatgagaaagcgccacgcttcccgaagggagaaaggcgg2280
acaggtatccggtaagcggcagggtcggaacaggagagcgcacgagggagcttccagggg2340
gaaacgcctggtatctttatagtcctgtcgggtttcgccacctctgacttgagcgtcgat2400
ttttgtgatgctcgtcaggggggcggagcctatggaaaaacgccagcaacgcggcctttt2460
tacggttcctggccttttgctggccttttgctcacatgttctttcctgcgttatcccctg2520
attctgtggataaccgtattaccgcctttgagtgagctgataccgctcgccgcagccgaa2580
cgaccgagcgcagcgagtcagtgagcgaggaagcggaagagcgcccaatacgcaaaccgc2640
ctctccccgcgcgttggccgattcattaatgcagctggcacgacaggtttcccgactgga2700
aagcgggcagtgagcgcaacgcaattaatgtgagttagctcactcattaggcaccccagg2760
ctttacactttatgcttccggctcgtatgttgtgtggaattgtgagcggataacaatttc2820
acacaggaaacagctatgaccatgattacgccaagctatttaggtgacactatagaatac2880
tcaagctatgcatccaacgcgttgggagctctcccatatggtcgagcagaggtgtaaaaa2940
gtactcaaaaattttactcaagtgaaagtacaagtacttagggaaaattttactcaatta3000
aaagtaaaagtatctggctagaatcttacttgagtaaaagtaaaaaagtactccattaaa3060
attgtacttgagtattaaggaagtaaaagtaaaagcaagaaagaaaactagagattcttg3120
tttaagcgacattgattattgactagttattaatagtaatcaattacggggtcattagtt3180
catagcccatatatggagttccgcgttacataacttacggtaaatggcccgcctggctga3240
ccgcccaacgacccccgcccattgacgtcaataatgacgtatgttcccatagtaacgcca3300
atagggactttccattgacgtcaatgggtggagtatttacggtaaactgcccacttggca3360
gtacatcaagtgtatcatatgccaagtacgccccctattgacgtcaatgacggtaaatgg3420
cccgcctggcattatgcccagtacatgaccttatgggactttcctacttggcagtacatc3480
tacgtattagtcatcgctattaccatggtgatgcggttttggcagtacatcaatgggcgt3540
ggatagcggtttgactcacggggatttccaagtctccaccccattgacgtcaatgggagt3600
ttgttttggcaccaaaatcaacgggactttccaaaatgtcgtaacaactccgccccattg3660
acgcaaatgggcggtaggcgtgtacggtgggaggtctatataagcagagctctctggcta3720
actagagaacccactgcttactggcttatcgaaattaatacgactcactatagggagacc3780
caagctggctagttaagcttgccaccatgttcagctttgtggatattcggctggcgctgt3840
tgctcagcgcaacggtgcttttggcaagaggacaaggcgaggacgatcgcactggcggca3900
gctgcacattggacggccaggtctacaatgacagggacgtctggaaaccagagccatgcc3960
aaatctgcgtgtgcgacagcggcaccgtaatgtgcgacgaagtgatctgcgaggacacaa4020
gcgactgccccaacccagtgattccccacgacgagtgctgccccgtctgcccagacgacg4080
atttccaggagcccagtgttgagggacctagaggttctcccggtgacaagggtgagaggg4140
gtcctgctggccctcccggcaatgatggaatccctggacagcctggcctccctgggcctc4200
caggccctcctggacctcctggccttggtggaaacttttctcctcagatgtctggtggct4260
ttgatgagaaatcctctccaatggctgtcccaggccccatgggacctatgggcccccgtg4320
gagcccctggacctcctggaccttccggaccccaaggatttactggtccccctggcgagc4380
ctggtgaggctggtgctcctggtccaatgggtccccgtggcgctgctggtccccctggaa4440
agaatggagaggatggtgagtctggcaaacctggtcgccctggtgagcgcggaccccctg4500
gaccacagggtgctcgtggattccccggaacccctggacttccaggcatcaagggacaca4560
gaggattcagcggtctagatggagctaagggggatgctggccctgctggacctaagggtg4620
agcctggtgcacctggtgagaatggaactcctggtgccatgggtccccgtggtctgcctg4680
gcgagagaggccgtgctggtcctcctggtgctgctggtgctcgtggtaatgatggtgccg4740
ctggagctgctggtcctcctggcccaactggccctgctggtcccccaggattccctggcg4800
gccctggatctaagggagaggttggtcctcagggatcccgtggtgcagagggacctcagg4860
gagcccgtggtgaagctggtaaccctggacctgctggtcctgctggtcccgctggtaaca4920
acggagctgatggtgcccctggtgccaagggtgctcctggcgctcctggtattgctggtg4980
ctcctggtttccctggaccccgtggtcctcctggagctgctggagcggctggtgcccctg5040
gccctaagggtaacaccggtgaggctggtgccccaggtgccaaaggagaggctggtgcta5100
agggagaggctggtgcccaaggagttcagggtccccctggtccccctggtgaggaaggca5160
agagaggaccccgtggtgagcccggtgctggtggtgcccgcggacctactggtgaacgtg5220
gtgctcctggtgctcgtggtttccctggtgctgatggagctgcaggtccaagaggtgccc5280
ctggtgagcgtggaggccctggagttgttggacctaagggtgccactggtgagcctggcc5340
gcaatggtgaacctggtatgccaggatccaagggtatgactggtagccctggcagccctg5400
gacctgatggaaagaccggacctggtggtgctcctggacaagatggccgccctggaccac5460
ctggccctgttggagccagaggacagcctggtgtcatgggattccctggacctaagggtg5520
ctgctggtgaggctggcaaacctggtgagagaggagtgatgggtgctattggtgccacta5580
gcgctcctggtaaggatggtgatgttggtgcccctggtgctcctggacctgctggacctg5640
ctggtgagagaggtgaacaaggagctgctggtcctcctggattccagggtctgccaggac5700
ctcagggtgctactggtgagccaggaaagtctggtgagcagggtgctcctggagaagctg5760
gagctcctggaccttctggttctagaggtgacagaggattccctggtgagcgtggtgccc5820
ctggccctgctggccctgttggtgcccgtggttctcctggttcagctggtaacgatggtg5880
ccaagggtgaaagtggtgccgcaggtgctcctggtgcccagggtcctcctggacttcaag5940
gaatgcctggagagcgtggggctgctggcctccctggtcttaagggtgacagaggtgacc6000
aaggtgccaagggtgctgatggtgctgctggtaaagatggcattcgtggtatgaccggac6060
caattggaccccccggacctgctggagctcctggtgacaagggagaatctggtgctcaag6120
gacttgttggacctactggtgcccgtggaccacctggtgagcgtggagagactggtgctc6180
ctggacctgccggatttgctggacctcctggtgctgatggtctgcctggtgccaagggag6240
aacctggagataatggtgctaagggtgatgctggtgctccaggacctgcaggagcaactg6300
gtgcccctggacctcagggtcccgttggtgctactggacccaagggtgcccgtggtgctg6360
ctggtcctcctggtgctactggcttccctggtgctgctggcagagttggacctcctggcc6420
cttctggaaattctggacccccaggacctcctggacccgctggtaaggagggccagaaag6480
gtaaccgtggtgagactggacctgctggtcgtactggtgaagttggtgctgctggaccac6540
ctggtgcccctggtgagaaaggaaatcccggagctgagggtgccactggccctgctggta6600
tccctggacctcaaggtattggtggtcagcgtggtattgtaggtctccctggacaaagag6660
gcgagcgtggtttccccggtctccctggcccatctggagagattggcaaacagggacctt6720
ctggtccatctggtgaacgtggacctcctggccccatgggaccccctggactggctggac6780
ctcctggtgagcctggtcgtgagggtactccaggaaatgagggctctgctggacgtgacg6840
gtgctgctggccccaagggtgaccgtggtgagactggcccatctggcactcctggagccc6900
ctggacctcctggtgctgctggacctattggccctgctggaaagactggtgatcgtggag6960
agaccggccctgctggtgtccccggccctgctggcccctctggtccccgcggaccctctg7020
gacccgctggagctcgtggagacaagggtgagactggtgaggctggtgagagaggcatga7080
agggacacagaggattcactggaatgccaggaccccctggtcctcctggaccctctggag7140
agtctggacccgctggtgcttctggacccgctggaccaagaggcccagctggatctgctg7200
gatcagctggtaaggatggtatgagtggcctccctggacccattggacctcctggacccc7260
gtggtcgcaatggagaaattggaccagctggacctcctggacctcccggcccccctggag7320
cacctggaccctctggtggtggattcgacattggcttcattgcccagccacaggagaagg7380
cccctgatcccttccgtcacttccgtgccgacgatgccaatgtgatgcgcgaccgtgatc7440
ttgaggttgacaccacccttaagtccctgagccagcagattgagagcatcatcagcccag7500
acggcaccaagaagaaccctgcccgcacttgccgtgacctgaagatgtgccacccagact7560
ggaagagcggcgagtactggattgaccctgaccagggctgcaaccaggatgccatcaagg7620
tctactgcaacatggaaactggcgagacttgcgtcaacccaactgagtctgctattccca7680
agaagaactggtacacaagcaagaacattaaggagaagaaacacgtctggttcggagagg7740
ccatgaccgatggcttccagttcgagtatggcagcgagggctccaagcctgaggatgtca7800
acattcagctcaccttcctgcgcctcatgtccactgaggcctcccagaacattacatacc7860
actgcaagaacagcattgcatacatggaccaggcttctggcaacctgaagaaggctcttc7920
tcctgcagggctccaacgaaattgagatcagagcagagggcaacagccgcttcacataca7980
gtgtcactgaggatggttgcacgtcgcacaccggtgcatggggcaagacagtcattgact8040
acaaaacaacgaaaacatcccgtctgcctattattgacatcgcccctatggacgttggtg8100
cacctaatcaggaatttggcattgaagttggcccagtctgcttcttgtaaggatccaccg8160
gatctagataactgatcataatcagccataccacatttgtagaggttttacttgctttaa8220
aaaacctcccacacctccccctgaacctgaaacataaaatgaatgcaattgttgttgtta8280
acttgtttattgcagcttataatggttacaaataaagcaatagcatcacaaatttcacaa8340
ataaagcatttttttcactgcattctagttgtggtttgtccaaactcatcaatgtatctt8400
aacgcgatccatggaattcactagtgcgcgcggccgctctagatggccagatctatttaa8460
attaaactgggcatcagcgcaattcaattggtttggtaatagcaagggaaaatagaatga8520
agtgatctccaaaaaataagtactttttgactgtaaataaaattgtaaggagtaaaaagt8580
acttttttttctaaaaaaatgtaattaagtaaaagtaaaagtattgatttttaattgtac8640
tcaagtaaagtaaaaatccccaaaaataatacttaagtacagtaatcaagtaaaattact8700
caagtactttacacctctgggcccaattcgccc8733
- 还没有人留言评论。精彩留言会获得点赞!