一种SNP-SNP标记的复合体系及其检测不平衡混合样本的方法和应用与流程
本发明属于分子生物
技术领域:
,具体涉及一种同时扩增人常染色体相邻连锁的snp-snp标记的复合检测体系及其对不平衡混合样本进行基因分型检测的方法和应用。
背景技术:
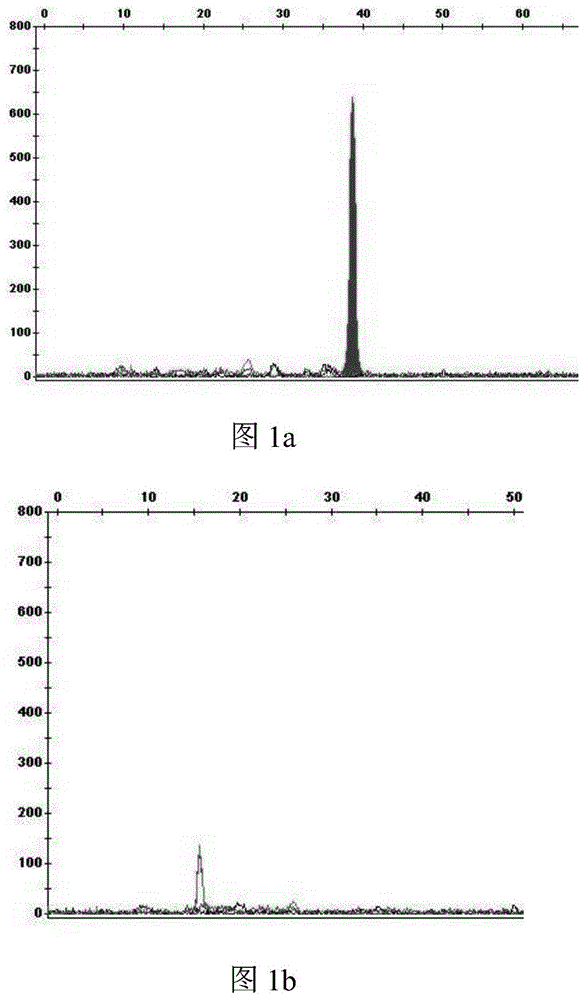
:单核苷酸多态性(singlenucleotidepolymorphism,snp)是继限制性片段长度多态性(rflp)和短串联重复序列(str)之后的第三代dna遗传标记。法医学领域一直在尝试将全基因组和群体数据库中的snps应用于法医dna分析、个体识别分析等。snps能提供一些比str更关键的优势,包括低突变率,在人类基因组中更高的丰度(在千人基因组计划中,有8470万snps、360万indels,在marshfieldgeneticmap中有7442个多态性好的strs,由此可见snps在基因组中分布最为广泛),短pcr扩增子长度适合高复合能力和降解dna的分析(可控制在200bp以下),二等位基因的特性也使其分型结果的分析简便易行,更加容易完成向自动化的转变。此外,snps可以提供身份、家系、祖先和表型信息。snps的遗传稳定、数量多、检测片段短、可以高通量、自动化检测等特点使其在医学遗传学领域同样有广泛的应用。arms(amplificationrefractorymutationsystem):针对复合遗传标记snp-snp的第一个snp(以下称为snp1),用arms方法设计等位基因特异引物,其原理是设计两种上游特异引物,即引物的3’端与snp序列是互补的,为了提高引物的特异扩增,在引物3’端倒数第二或倒数第三的碱基上引入刻意的不匹配。共用的反向引物则位于第二个snp(以下称为snp2)的下游区域。arms扩增产物长度限制在150bp以内,原因是法医学案件中大多检材都是降解样本,并且无创产前亲子鉴定(nipat)中cff的片段大多都不超过150bp-200bp。荧光标记单碱基延伸技术(snapshot)结合毛细管电泳(ce)技术:针对snp2引入的snapshot技术,是最常见的基于单碱基延伸(sbe)原则的微测序方法;再基于arms-pcr方法结合snapshot技术进行snp-snp分型,最后通过sanger测序来验证分型是否正确。与传统的设计引物相比,arms能够使具有特定碱基的snp成功地扩增,从而避免了在对不同碱基的两人混合斑的分析中常见的pcr偏差。在单个pcr反应中根据arms和sbe分别扩增两个snp,且snp-snp标记的等位基因可以由扩增产物的峰的移动位置和颜色来确定。另外,snapshot技术是具有多种用途的法医snp基因分型工具,可以很容易地在法医实验室中进行操作,而无需对额外设备进行任何投资,操作简单,费时短,易于普及。目前已有的snp-str和dip-str复合体系虽然能够将str与侧翼的snp或indel多态性联系在一起,并且在单一反应中分别对多个snp-str或dip-str进行基因分型,但其中snp-str对不均衡两人混合斑分辨率最多只能达到1:100,而dip-str中的indel在基因组中分布没有snp广泛,另外,最为关键的一点是由于str的存在,使这两种复合遗传标记在降解检材或者短片段检材的应用中存在较大的局限性,而目前还没有关于snp-snp体系的相关报道。技术实现要素:针对现有技术中的上述不足,本发明提供一种snp-snp标记的复合体系及其检测不平衡混合样本的方法和应用,为了补充现有遗传标记在降解检材应用方面的不足,本发明筛选出17个新型复合遗传标记snp-snp,这些snp-snp标记是基因组中间距50bp以内的遗传标记snp的组合,包含了snps双等位基因特异分型及扩增片段短等优势。为实现上述目的,本发明解决其技术问题所采用的技术方案是:一种snp-snp标记的复合体系,该复合体系包括扩增以下17个复合遗传标记snp-snp的复合扩增引物、单碱基延伸引物以及测序引物;复合扩增引物中每个位点均包括两条正向引物和一条反向引物,其序列如seqidno.1~51所示(见表1),单碱基延伸引物具体序列如seqidno.52~68所示(见表2),测序引物具体序列如seqidno.69~102所示(见表3);snp-snp位点包括rs66663660-rs9426355、rs3109851-rs6848611、rs7724803-rs6554864、rs68012481-rs2498233、rs35443929-rs6462431、rs2527748-rs2527749、rs10119697-rs10961215、rs1757217-rs2482113、rs3843625-rs12422436、rs59588112-rs8017285、rs12101725-rs55649144、rs9938522-rs9940690、rs12950438-rs12950190、rs10445426-rs57907290、rs380371-rs380410、rs2012094-rs220171以及rs468851-rs468852。表1复合扩增引物表2单碱基延伸引物表3sanger测序引物利用上述复合体系检测不平衡混合样本的方法,包括以下步骤:(1)提取待检样本dna;(2)采用权利要求1所述的复合扩增引物对待检样本dna进行pcr扩增,并纯化扩增产物;(3)再用权利要求1所述的单碱基延伸引物对纯化后的扩增产物进行单碱基延伸,再纯化延伸产物,然后使用abi3130xl遗传分析仪电泳分离上述延伸产物,获得snp-snp标记的单倍型基因。进一步地,步骤(2)中扩增体系包括2×qiagenmultiplexpcrmastermix5μl、primer1μl、dna1μl,最后用rnase-freewater补足至10μl。进一步地,步骤(2)中扩增条件为95℃,预变性15min;94℃,变性30s;55.2~61.4℃,退火90s;72℃,延伸60s;最后60℃,延伸30min;共30个循环。进一步地,步骤(2)中纯化体系包括1msap1μl、扩增产物2.5μl,以及10mexoι0.6μl;纯化条件为37℃,孵育1h后,80℃,灭活15min。进一步地,步骤(3)中单碱基延伸体系包括sbe-mix1.5μl、sbe-primer0.3μl、纯化后的扩增产物1.5μl,最后用rnase-freewater补足至5μl。进一步地,步骤(3)中单碱基延伸条件为96℃,变性10s;53℃,退火5s;60℃,延伸30s;共25个循环。进一步地,步骤(3)中纯化体系包括sbe产物5μl,以及1msap1μl;纯化条件为37℃,孵育1h后,80℃,灭活15min。上述方法在个体识别、产前亲子鉴定中的应用。一种用于进行不平衡混合样本检测分析的试剂盒,包括上述复合体系。更具体的,该试剂盒还可以包括以下组分:a)复合扩增反应混合液:含有pcr缓冲溶液、mgcl2、dntps、dna聚合酶等常用成分;b)复合引物:具体序列如seqidno.1~51所示;c)扩增产物纯化试剂:含有核酸外切酶1(exoi)及其缓冲溶液(exoibuffer)、虾碱性磷酸酶(sap)及其缓冲溶液(sapbuffer)等常用成分;用于将复合扩增后的产物进行纯化,以便于进行下一步操作;d)单碱基延伸引物:具体序列如seqidno.52~68所示的;e)单碱基延伸反应混合液:包括dna聚合酶、缓冲液、mgcl2、荧光标记双脱氧核糖核酸等常用成分;复合扩增反应混合液、扩增产物纯化试剂可按本领域常用的配方或按分子生物学手册进行配制,也可直接使用商业化的产品;至于提取待检测样本中的dna的模板,可使用本领域目前的各种常规试剂,提取dna模板可以参照现有的常规方法即可进行。本发明的有益效果为:1、本发明为了补充现有遗传标记在降解检材应用方面的不足,筛选出了全新的17个遗传复合标记snp-snp;本发明的17个新型复合遗传标记snp-snp是基于千人基因组数据库的全基因组常染色体snp以及dbsnp按照本发明制定的筛选原则逐步筛选出来的,而不是基于已经存在的snp位点将其随意组合。2、通过本发明设计的复合体系,将arms-pcr、snapshot和ce技术结合应用,以获得不平衡混合斑中的准确分型,极大的提高了检测速度和效率,满足实际公安案件的需要,极大了推动了案件排查力度;并且所使用的仪器设备均为可以很容易地在法医实验室中操作,而无需对额外设备进行任何投资,操作简单,费时短,易于普及。3、本发明中17个新型复合遗传标记snp-snp扩增产物长度均在50-150bp之间,且延伸产物长度均≤70bp,对于解决法医学领域降解检材的个体识别、不平衡两人混合检材的鉴定,以及临床领域的无创产前亲子鉴定等具有广泛的应用前景。附图说明图1为样本4的单倍分型检测结果;其中,图1a为单倍型rs9938522-rs9940690的snp1为t等位基因时的snapshot产物(即snp2);图1b为s9938522-rs9940690的snp1为a等位基因时的snapshot产物(即snp2);图2为样本4的正向sanger测序结果;图3为样本4的反向sanger测序结果;图4为样本721的单倍分型检测结果;其中,图4a为单倍型rs9938522-rs9940690的snp1为t等位基因时的snapshot产物(即snp2);图4b为单倍型rs9938522-rs9940690的snp1为a等位基因时的snapshot产物(即snp2);图5为样本723的单倍分型检测结果;其中,图5a为单倍型rs9938522-rs9940690的snp1为t等位基因时的snapshot产物(即snp2);图5b为单倍型rs9938522-rs9940690的snp1为a等位基因时的snapshot产物(即snp2);图6为样本721的正向sanger测序结果;图7为样本721的反向sanger测序结果;图8为样本723的正向sanger测序结果;图9为样本723的反向sanger测序结果。具体实施方式下面对本发明的具体实施方式进行描述,以便于本
技术领域:
的技术人员理解本发明,但应该清楚,本发明不限于具体实施方式的范围,对本
技术领域:
的普通技术人员来讲,只要各种变化在所附的权利要求限定和确定的本发明的精神和范围内,这些变化是显而易见的,一切利用本发明构思的发明创造均在保护之列。实施例1snp-snp复合遗传标记筛选1、筛选原则基于千人基因组数据库的全基因组常染色体snp以及dbsnp(http://www.1000genomes.org/home、http://www.ncbi.nlm.nih.gov/snp/)按照以下筛选原则:1)两个snp的间距≤50bp;2)两个snp的最小等位基因频率(maf)≥0.1且组成的单倍型个数≥3;3)通过hapmap数据库筛选出属于北京汉族人群(chb)和中国南方汉族(chs)的单倍型;4)通过haploview软件筛选出单倍型的最小频率≥0.2且处于连锁不平衡状态;5)通过在线primer3能够设计出扩增子在50-150bp内的arms引物;6)最后通过oligo7筛选arms引物在全基因的同源序列少并且发夹结构少的标记。2、根据上述筛选原则,共筛选得到17个新型复合遗传标记snp-snp,具体为rs66663660-rs9426355、rs3109851-rs6848611、rs7724803-rs6554864、rs68012481-rs2498233、rs35443929-rs6462431、rs2527748-rs2527749、rs10119697-rs10961215、rs1757217-rs2482113、rs3843625-rs12422436、rs59588112-rs8017285、rs12101725-rs55649144、rs9938522-rs9940690、rs12950438-rs12950190、rs10445426-rs57907290、rs380371-rs380410、rs2012094-rs220171、rs468851-rs468852。上述17个snp-snp标记均匀地分布于22条常染色体上,具体信息见表4,且snp-snp均紧密连锁,二者能够被同时检测。表4snp-snp标记信息snp-snp染色体物理位置rs66663660-rs9426355chr129774262-29774287rs3109851-rs6848611chr426473621-26473631rs7724803-rs6554864chr514950999-14951005rs68012481-rs2498233chr623007491-23007496rs35443929-rs6462431chr732935434-32935439rs2527748-rs2527749chr85396199-5396225rs10119697-rs10961215chr913741227-13741252rs1757217-rs2482113chr1018506467-18506478rs3843625-rs12422436chr1258858690-58858715rs59588112-rs8018285chr1425768631-25768641rs12101725-rs55649144chr1592575365-92575386rs9938522-rs9940690chr1513552242-13552251rs12950438-rs12950190chr1759846353-59846363rs10445426-rs57907290chr1812195773-12195803rs380371-rs380410chr2046968735-46968764rs2012094-rs220181chr2143561440-43561468rs468851-rs468852chr2229956137-29956156实施例2引物设计本发明所示的复合体系包括复合扩增引物、单碱基延伸引物,以及测序引物,具体设计过程如下。1、arms引物设计:复合扩增引物如seqidno.1~51所示,具体序列见表1。2、sbe引物设计:使用sbeprimer软件针对17个snp-snp标记的snp2设计单碱基延伸引物,通过引物5’端加尾(聚a、聚c或者聚gact)使不同标记的sbe引物的长度区分开,为复合体系延伸产物的分离做准备,单碱基延伸引物如seqidno.52~68所示,具体序列见表2。3、sanger测序引物设计:使用primer3引物在线设计工具(http://bioinfo.ut.ee/primer3/)对17个snp-snp标记设计sanger测序引物,测序引物如seqidno.69~102所示,具体序列见表3。实施例3复合体系个体识别检测1、获取22名中国汉族无关个体的外周血样本,样本来自四川大学基础医学与法医学院法医物证学实验室。使用百泰克全血基因组dna快速提取试剂盒提取所需样本的外周血基因组dna作为模板,所有样本对象的采集受适用条款监督,采集对象签署知情同意书。2、采用geneamp9700热循环仪,使用设计得到snp-snp标记的复合扩增引物组(见表1)对作为模板的22名中国汉族无关个体dna进行pcr扩增反应,将所述合成好的引物用1×te缓冲液稀释到100μm,再将17个snp-snp的引物分别与1×te缓冲液以1:9的比例混合,得到浓度为10μm的工作液浓度引物。从51管pcr引物中分别取不同体积加入到一个新的离心管中,作为17重pcr复合引物;pcr反应体系包括:2×qiagenmultiplexpcrmastermix5μl、primermix(10μm)1μl、dna(1ng/μl)1μl,最后用rnase-freewater补足至10μl;反应条件为:95℃,预变性15min;94℃,变性30s;57.8℃,退火90s;72℃,延伸60s;最后60℃,延伸30min;共30个循环;4℃保存。3、对扩增产物进行纯化,纯化体系包括1msap1μl、扩增产物2.5μl,以及10mexoι0.6μl;纯化条件为37℃,孵育1h后,80℃,灭活15min;然后用所述每个snp-snp标记的单碱基延伸引物组(见表2)对上述的扩增产物进行单碱基延伸,snapshot体系包括sbe-mix1.5μl、纯化产物1.5μl、sbe-primermix(10μm)0.3μl,以及rnase-freewater1.7μl;延伸条件为:96℃,变性10s;53℃,退火5s;60℃,延伸30s;共25个循环;4℃保存。4、使用abi3130xl遗传分析仪电泳分离上述延伸产物,其中毛细管长度为36cm,电泳胶为pop-7,上样条件包括:genescanliz-120内标与hiditmformamide混合液(3:200)9μl,以及延伸产物1μl;电泳程序为:进样时间为12s,进样时电压为1.5kv;电泳电压为15kv,电泳时间为18min。5、通过genemappertmid-xv1.2软件对电泳数据结果进行分析,得到所述22名中国汉族无关个体的17个snp-snp标记的分型结果,具体结果见表5(显示每个标记的几个代表样本),由表5结果可知,本发明设计的复合体系能完全分辨这22个不相关的个体;表5snp-snp代表样本单倍型分型由于检测了22个样本,以样本4、721和723为例,其snp1基因型均为两种等位基因的纯合子和杂合子,其检测结果见图1、图4和图5;如图1所示,图1a和图1b分别表示单倍型rs9938522-rs9940690的snp1为t和a等位基因时的snapshot产物,且该单倍型的sbe引物为正向引物,故只有t等位基因的snapshot产物显示为红色峰时,说明snp1的分型为tt,snp2的分型为tt,即样本4的单倍型分型为tt-tt;如图4所示,图4a和图4b分别表示单倍型rs9938522-rs9940690的snp1为t和a等位基因时的snapshot产物,且该单倍型的sbe引物为正向引物,故当a、t等位基因的snapshot产物都出峰,且分别显示为红色和绿色时,说明snp1的分型为at,snp2的分型为ta,即样本721的单倍型分型为at-ta;如图5所示,图5a和图5b表示单倍型rs9938522-rs9940690的snp1为t和a等位基因时的snapshot产物,且该单倍型的sbe引物为正向引物,故只有a等位基因的snapshot产物显示为红色峰时,说明snp1的分型为aa,snp2的分型为tt,即样本723的单倍型分型为aa-tt。6、测序验证:各snp-snp标记均选取snp1基因型为两种等位基因的纯合子和杂合子的三个样本,使用所述对应的sanger测序引物对其进行扩增,扩增产物用聚丙烯酰胺凝胶(page)电泳分离,切胶回收送tsingke公司进行sanger测序;sanger测序结果显示各snp-snp标记分型结果与snapshot的分型结果一致。由于检测了22个样本,以样本4、721和723为例,其snp1基因型均为两种等位基因的纯合子和杂合子,其结果见图2、图3、图6~9;如图2、图3和图6~9箭头所示,其检测结果与图1、图4和图5一致,表明snp-snp标记分型结果与snapshot的分型结果一致。实施例4不平衡混合样本检测1、模拟两人或多人混合样本dna若干;2、采用实施例2建立的复合体系,根据实施例3的检测过程,对模拟的两人或多人混合样本dna分别进行17个snp-snp分型检测;3、通过遗传分析仪,如3130型遗传分析仪(购自美国abi公司),对检测结果进行分析,通过检测结果表明,模拟的两人或多人混合样本均得到17个snp-snp的基因分型,说明该复合体系对于两人混合样本的鉴定具有很大的应用价值,能够增加法医检测过程中的有效物证量。实施例5产前亲子鉴定1、获取若干个怀孕妇女的血浆(在第1、2、3个妊娠期收集),无论性别类型,胎儿dna检测呈阳性,该样本由某医院提供;2、提取所述样本dna,基因组dna的提取参考《ga/t383-2014法庭科学dna实验室检验规范》进行;3、用所述实例2的复合检测体系及检测方法对提取的妇女血浆dna分别进行17个snp-snp的分型;4、所述妇女血浆中的胎儿dna均得到17个snp-snp的分型,和父亲的dna进行对比发现,该单倍型有两个同源等位基因来自于父亲,结果说明该复合检测体系对于无创产前亲子鉴定具有很大的应用价值。序列表<110>四川大学<120>一种snp-snp标记的复合体系及其检测不平衡混合样本的方法和应用<160>102<170>siposequencelisting1.0<210>1<211>22<212>dna<213>人工序列(artificialsequence)<400>1agaacacttagcctttcccgac22<210>2<211>22<212>dna<213>人工序列(artificialsequence)<400>2agaacacttagcctttcccgat22<210>3<211>20<212>dna<213>人工序列(artificialsequence)<400>3tcttgaacagccaccagaca20<210>4<211>21<212>dna<213>人工序列(artificialsequence)<400>4agccatccctctttgcctttc21<210>5<211>21<212>dna<213>人工序列(artificialsequence)<400>5agccatccctctttgcctgtt21<210>6<211>22<212>dna<213>人工序列(artificialsequence)<400>6tatgaatttgggaggggacaca22<210>7<211>19<212>dna<213>人工序列(artificialsequence)<400>7tggaagaatgcatcgagac19<210>8<211>19<212>dna<213>人工序列(artificialsequence)<400>8tggaagaatgcatcgaagt19<210>9<211>22<212>dna<213>人工序列(artificialsequence)<400>9gacatcctctcatcctggctac22<210>10<211>24<212>dna<213>人工序列(artificialsequence)<400>10cctcatctaagtccctaaagtaac24<210>11<211>24<212>dna<213>人工序列(artificialsequence)<400>11cctcatctaagtccctaaagtaat24<210>12<211>21<212>dna<213>人工序列(artificialsequence)<400>12gttgggtcttctgttacacca21<210>13<211>20<212>dna<213>人工序列(artificialsequence)<400>13ccaccaaccatgtttcctaa20<210>14<211>20<212>dna<213>人工序列(artificialsequence)<400>14ccaccaaccatgtttcctac20<210>15<211>20<212>dna<213>人工序列(artificialsequence)<400>15ccaccaaccatgtttcctac20<210>16<211>22<212>dna<213>人工序列(artificialsequence)<400>16atattttaatgcatttccctcg22<210>17<211>23<212>dna<213>人工序列(artificialsequence)<400>17catattttaatgcatttccctct23<210>18<211>20<212>dna<213>人工序列(artificialsequence)<400>18gccatcagcgaacaaatact20<210>19<211>23<212>dna<213>人工序列(artificialsequence)<400>19cttgttgtgggttgaaatgtatc23<210>20<211>23<212>dna<213>人工序列(artificialsequence)<400>20cttgttgtgggttgaaatgtatc23<210>21<211>23<212>dna<213>人工序列(artificialsequence)<400>21tgcaaatagggttttaacagagg23<210>22<211>25<212>dna<213>人工序列(artificialsequence)<400>22gtcaattgtttagtatggcaaactc25<210>23<211>25<212>dna<213>人工序列(artificialsequence)<400>23gtcaattgtttagtatggcaaactt25<210>24<211>20<212>dna<213>人工序列(artificialsequence)<400>24ccatcttgacctccctgctt20<210>25<211>20<212>dna<213>人工序列(artificialsequence)<400>25ccatcttgacctccctgctt20<210>26<211>21<212>dna<213>人工序列(artificialsequence)<400>26ggacaatgtttcctaagcgat21<210>27<211>22<212>dna<213>人工序列(artificialsequence)<400>27ctgcccttggtcaatttcctag22<210>28<211>21<212>dna<213>人工序列(artificialsequence)<400>28gtcctgcagtccttcaaaatc21<210>29<211>21<212>dna<213>人工序列(artificialsequence)<400>29gtcctgcagtccttcaaagtt21<210>30<211>20<212>dna<213>人工序列(artificialsequence)<400>30attttggggcagggaggaat20<210>31<211>21<212>dna<213>人工序列(artificialsequence)<400>31cttagtgtcgggtctctgtta21<210>32<211>21<212>dna<213>人工序列(artificialsequence)<400>32cttagtgtcgggtctctgatg21<210>33<211>20<212>dna<213>人工序列(artificialsequence)<400>33gggcttgagaaaatactgga20<210>34<211>22<212>dna<213>人工序列(artificialsequence)<400>34tgtatcagcaaatggaagcgaa22<210>35<211>22<212>dna<213>人工序列(artificialsequence)<400>35tgtatcagcaaatggaagctat22<210>36<211>20<212>dna<213>人工序列(artificialsequence)<400>36cccttccctccgctttatct20<210>37<211>18<212>dna<213>人工序列(artificialsequence)<400>37ccacgttgttgagagtgc18<210>38<211>18<212>dna<213>人工序列(artificialsequence)<400>38ccacgttgttgagagtgt18<210>39<211>20<212>dna<213>人工序列(artificialsequence)<400>39gggtccctcagtttgtttgc20<210>40<211>21<212>dna<213>人工序列(artificialsequence)<400>40acatggcttccttcttttgcg21<210>41<211>21<212>dna<213>人工序列(artificialsequence)<400>41acatggcttccttcttttgct21<210>42<211>22<212>dna<213>人工序列(artificialsequence)<400>42aaatggacctagacacagacat22<210>43<211>20<212>dna<213>人工序列(artificialsequence)<400>43aactggcccctgagaaccgc20<210>44<211>20<212>dna<213>人工序列(artificialsequence)<400>44aactggcccctgagaaccgt20<210>45<211>20<212>dna<213>人工序列(artificialsequence)<400>45tcccctcctgtccttaacct20<210>46<211>22<212>dna<213>人工序列(artificialsequence)<400>46tagctttgtctttcccgtcacc22<210>47<211>22<212>dna<213>人工序列(artificialsequence)<400>47tagctttgtctttcccgtccct22<210>48<211>22<212>dna<213>人工序列(artificialsequence)<400>48gtctatgctgtaagggtcctga22<210>49<211>22<212>dna<213>人工序列(artificialsequence)<400>49ggctccttctcatcctttaacc22<210>50<211>22<212>dna<213>人工序列(artificialsequence)<400>50ggctccttctcatcctttatct22<210>51<211>22<212>dna<213>人工序列(artificialsequence)<400>51ggggatctacattggtctaggt22<210>52<211>38<212>dna<213>人工序列(artificialsequence)<400>52gactgactgactgactcagcagacctccacacttttac38<210>53<211>51<212>dna<213>人工序列(artificialsequence)<400>53gactgactgactgactgactgactgactgactgaggggacacaagcattca51<210>54<211>57<212>dna<213>人工序列(artificialsequence)<400>54gactgactgactgactgactgactgactgactgactattgcctctttactgatcaca57<210>55<211>62<212>dna<213>人工序列(artificialsequence)<400>55gactgactgactgactgactgactgactgactgactgactgaggaggagatggaaacata60gg62<210>56<211>70<212>dna<213>人工序列(artificialsequence)<400>56gactgactgactgactgactgactgactgactgactgactgactgactgactggtctcat60ggtttgattg70<210>57<211>64<212>dna<213>人工序列(artificialsequence)<400>57gactgactgactgactgactgactgactgactgactgactactaagcatctttaagtaac60acca64<210>58<211>30<212>dna<213>人工序列(artificialsequence)<400>58gagactcccctcaaaaaatatattttgaat30<210>59<211>48<212>dna<213>人工序列(artificialsequence)<400>59gactgactgactgactgactgactcatctccattttctttttttctat48<210>60<211>35<212>dna<213>人工序列(artificialsequence)<400>60ctgactgactgaagaagaagataaagaattcatgg35<210>61<211>34<212>dna<213>人工序列(artificialsequence)<400>61actgactgactgactttggaaataattgctgcct34<210>62<211>31<212>dna<213>人工序列(artificialsequence)<400>62cccccccgctatagcaatgtttggccaagac31<210>63<211>31<212>dna<213>人工序列(artificialsequence)<400>63cccccccgctatagcaatgtttggccaagac31<210>64<211>26<212>dna<213>人工序列(artificialsequence)<400>64aaaaattaaaggttgcattcaggtcc26<210>65<211>25<212>dna<213>人工序列(artificialsequence)<400>65cggaaatgtatactgagctcatgac25<210>66<211>20<212>dna<213>人工序列(artificialsequence)<400>66cttttgctgaaccccaactg20<210>67<211>42<212>dna<213>人工序列(artificialsequence)<400>67ctgactgactgactgactgacttgagaccccagaatactgcg42<210>68<211>60<212>dna<213>人工序列(artificialsequence)<400>68ctgactgactgactgactgactgactgactgactgactagctctcagcttacattttact60<210>69<211>21<212>dna<213>人工序列(artificialsequence)<400>69tgtcattgcacaaatcatagg21<210>70<211>20<212>dna<213>人工序列(artificialsequence)<400>70tcttgaacagccaccagaca20<210>71<211>20<212>dna<213>人工序列(artificialsequence)<400>71atgttctggtgtgaagacgc20<210>72<211>19<212>dna<213>人工序列(artificialsequence)<400>72gactccaccctcatgactc19<210>73<211>20<212>dna<213>人工序列(artificialsequence)<400>73tttggtgttttcagggaggg20<210>74<211>20<212>dna<213>人工序列(artificialsequence)<400>74aaaatgcccggacatcctct20<210>75<211>23<212>dna<213>人工序列(artificialsequence)<400>75tgacacttaaataccagccatga23<210>76<211>21<212>dna<213>人工序列(artificialsequence)<400>76gttgggtcttctgttacacca21<210>77<211>22<212>dna<213>人工序列(artificialsequence)<400>77tcaatctggacaacatcttcgt22<210>78<211>22<212>dna<213>人工序列(artificialsequence)<400>78cctatgtgggtctcatggtttg22<210>79<211>22<212>dna<213>人工序列(artificialsequence)<400>79tctgtgatttgtgtgtgagaca22<210>80<211>21<212>dna<213>人工序列(artificialsequence)<400>80tctgtttccatctttctgcca21<210>81<211>20<212>dna<213>人工序列(artificialsequence)<400>81tgtctgcttgttgtgggttg20<210>82<211>20<212>dna<213>人工序列(artificialsequence)<400>82cctctccatctccctgttca20<210>83<211>18<212>dna<213>人工序列(artificialsequence)<400>83atcgtgccgctgtactcc18<210>84<211>21<212>dna<213>人工序列(artificialsequence)<400>84tcagtgaggaccatcttgacc21<210>85<211>20<212>dna<213>人工序列(artificialsequence)<400>85acaattacaatgccgtgcca20<210>86<211>20<212>dna<213>人工序列(artificialsequence)<400>86cagcacatggggaaaagaca20<210>87<211>21<212>dna<213>人工序列(artificialsequence)<400>87tcaggtgcattagtgactcct21<210>88<211>20<212>dna<213>人工序列(artificialsequence)<400>88accatcaccatgctagtcct20<210>89<211>20<212>dna<213>人工序列(artificialsequence)<400>89actctcaggattggcatggg20<210>90<211>20<212>dna<213>人工序列(artificialsequence)<400>90tcagttctcctcacacctgg20<210>91<211>20<212>dna<213>人工序列(artificialsequence)<400>91aatgtggctctcaggggatc20<210>92<211>20<212>dna<213>人工序列(artificialsequence)<400>92tcagccattgttgatcgagt20<210>93<211>21<212>dna<213>人工序列(artificialsequence)<400>93acaatgcacccctacagaaga21<210>94<211>20<212>dna<213>人工序列(artificialsequence)<400>94gggtccctcagtttgtttgc20<210>95<211>20<212>dna<213>人工序列(artificialsequence)<400>95gaggaaatgtgcagggaagc20<210>96<211>22<212>dna<213>人工序列(artificialsequence)<400>96tgcttaggaatcatgttgtggt22<210>97<211>18<212>dna<213>人工序列(artificialsequence)<400>97tacccgatgtgcctgcag18<210>98<211>20<212>dna<213>人工序列(artificialsequence)<400>98tcccctcctgtccttaacct20<210>99<211>20<212>dna<213>人工序列(artificialsequence)<400>99cgcctgtggatgtgattctg20<210>100<211>20<212>dna<213>人工序列(artificialsequence)<400>100gatagtgttggcctcatcgc20<210>101<211>20<212>dna<213>人工序列(artificialsequence)<400>101tccttgccacacctcaaaac20<210>102<211>20<212>dna<213>人工序列(artificialsequence)<400>102aaggcagggagagatgatgg20当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1