T7RNA聚合酶变体的制作方法
本申请要求于2017年6月30日提交的美国临时专利申请序号62/527,740和于2017年7月5日提交的美国临时专利申请序号62/528,840的优先权,这两项美国临时专利申请为了所有目的通过引用以其整体并入。
发明领域
本发明提供了工程化rna聚合酶变体和包含这些变体的组合物。本发明还提供了工程化t7rna聚合酶变体和包含这些变体的组合物。这些变体已经被演化用于在体外转录起始时相对于gtp选择性掺入m7g(5’)ppp(5’)m7g帽类似物。本发明还提供了用于对rna转录物选择性加帽的方法。
对序列表、表格或计算机程序的引用
序列表的正式副本作为ascii格式化文本文件经由efs-web与说明书同时提交,文件名为“cx8-168usp1_st25.txt”,创建日期为2017年7月5日,并且大小为184,320字节。经由efs-web提交的序列表为说明书的一部分并且通过引用以其整体并入本文。
发明背景
rna聚合酶,也称为“dna依赖性rna聚合酶”,将dna转录成rna转录物。这些酶代表驱动转录的主要机器(primarymachinery)。rna聚合酶已经被充分地分离和纯化,使得它们可用于体外产生rna。体外转录是这样的程序:允许任何序列的rna分子的dna引导的合成,所述任何序列的rna分子尺寸在从短寡核苷酸至几千碱基的范围内。通常,体外转录包括模板的工程化,随后使用相应的rna聚合酶进行转录,所述模板包括感兴趣序列上游的噬菌体启动子序列(例如,来自t7大肠杆菌噬菌体)。通常,这些rna转录物随后被修饰(例如,通过加帽、剪接、添加聚a尾等)。这些转录物用于分析技术(例如,杂交分析)、用于结构研究(例如,nmr和x射线晶体学)、用于生化和遗传学研究(例如,作为反义试剂)、用作功能性分子(例如,核酶和适体)以及用作治疗剂。
此外,作为酶替代疗法的治疗剂的mrna的开发,突显了在高成本效益、商业化产生mrna(包括有效产生修饰的mrna)的方面存在的重大商业障碍。
发明概述
本发明提供了工程化t7rna聚合酶变体及其组合物。这些变体已经被演化用于在体外转录起始时相对于gtp选择性掺入m7g(5’)ppp(5’)m7g帽类似物。本发明还提供了用于对rna转录物选择性加帽的方法。此外,本发明提供了包含本文提供的rna聚合酶变体的方法和组合物。
本发明提供了工程化rna聚合酶,该工程化rna聚合酶包含与参考序列seqidno:4和/或seqidno:15或其功能片段具有至少85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更高序列同一性的多肽序列,其中该工程化rna聚合酶在多肽序列中包含至少一个取代或取代集,并且其中多肽序列的氨基酸位置参考seqidno:4或seqidno:15编号。
在一些实施方案中,工程化rna聚合酶包含至少一个选自以下的取代或取代集:32/357、49/642、97/357、136、137、160/643、167/514、250、302/513、314/401、357、392、393、394、397、401、404、444、446、478、513、514、582、635、636、637、639、642、643、645、653、656、660、660/806、661和664,和/或其任何组合,其中氨基酸位置参考seqidno:4编号。在一些另外的实施方案中,工程化rna聚合酶包含至少一个选自以下的取代或取代集:32v/357i、49g/642l、97d/357g、136e/i、137w、160l/643s、167n/514l、250d、302v/513g、314c/401v、357g/k/l/m/n/q/r/s/t/v/w、392d、393l/y、394a/l/r、397f/m/q/w、401a/i/l/s/v、404e/y、444f/h/i/v、446w/y、478f/m/w、513c/f/k/l/r/t/w、514f/i/l/y、582n、635w、636l、637g/p/s、639h、642l、643a、645v、653c、656f/w、660a/c/m/s/t/w、660n/806y、661e/y和664w,和/或其任何组合,其中氨基酸位置参考seqidno:4编号。在一些另外的实施方案中,工程化rna聚合酶包含至少一个选自以下的取代或取代集:a32v/e357i、e49g/m642l、e97d/e357g、a136e/i、d137w、r160l/t643s、k167n/s514l、t250d、a302v/d513g、r314c/r401v、e357g/k/l/m/n/q/r/s/t/v/w、y392d、r393l/y、k394a/l/r、a397f/m/q/w、r401a/i/l/s/v、s404e/y、n444f/h/i/v、m446w/y、d478f/m/w、d513c/f/k/l/r/t/w、s514f/i/l/y、a582n、s635w、v636l、t637g/p/s、r639h、m642l、t643a、a645v、f653c、q656f/w、d660a/c/m/s/t/w、d660n/h806y、t661e/y和p664w,和/或其任何组合,其中氨基酸位置参考seqidno:4编号。
在一些实施方案中,工程化rna聚合酶包含至少一个选自以下的取代或取代集:397/513/635、397/513/635/660、513/660/664、513/635/660、513/635/664、513/660/664和660/664,和/或其任何组合,其中氨基酸位置参考seqidno:4编号。在一些另外的实施方案中,工程化rna聚合酶包含至少一个选自以下的取代或取代集:397f/513w/635w、397f/513y/635w、397w/513y/635w、397w/513w/635w、397w/513y/635w/660f、397w/513w/635w/660w、397y/513w/635w/660f、475v/513w/635w/660y、513f/660w/664y、513w/635w/660f、513w/635w/664w、513y/635w/660f、513y/635w/660y、513y/660w/664w和660y/664w,和/或其任何组合,其中氨基酸位置参考seqidno:4编号。在一些另外的实施方案中,工程化rna聚合酶包含至少一个选自以下的取代或取代集:a397f/d513w/s635w、a397f/d513y/s635w、a397w/d513y/s635w、a397w/d513w/s635w、a397w/d513y/s635w/d660f、a397w/d513w/s635w/d660w、a397y/d513w/s635w/d660f、a475v/d513w/s635w/d660y、d513f/d660w/p664y、d513w/s635w/d660f、d513w/s635w/p664w、d513y/s635w/d660f、d513y/s635w/d660y、d513y/d660w/p664w和d660y/p664w,和/或其任何组合,其中氨基酸位置参考seqidno:4编号。
在一些实施方案中,工程化rna聚合酶包含至少一个选自以下的取代或取代集:397、397/513、397/513/635、397/513/635、397/513/635/656、397/513/635/656/660、397/513/635/656/660/664、397/513/635/656/664、397/513/635/660、397/513/635/660/664、397/513/635/664、397/513/656/660、397/513/660、397/513/660/664、397/513/664、397/513、397/635、397/635/656/660/664、397/635/656/664、397/635/660、397/635/664、397/635/664/850、397/660、397/664、397/660/664、397/837、399/635/660、475/513/635/660、513、513/635、513/635/656、513/635/656/660、513/635/656/664、513/635/660、513/635/660/664、513/635/664、513/656/660、513/656/664、513/660、513/660/664、513/664、635、635/656、635/656/664、635/660、635/660/664、635/664、656/660/664、658、660、660/664和664,和/或其任何组合,其中氨基酸位置参考seqidno:4编号。在一些另外的实施方案中,工程化rna聚合酶包含至少一个选自以下的取代或取代集:397f、397f/513f、397f/513f/635f、397f/513f/635w、397f/513f/635w/660w、397f/513w/635w、397f/513w/635w/656f/664f、397f/513w/664w、397f/513y、397f/513y/635w、397f/513y/635w/656w、397f/513y/635w/664f、37f/513y/656h/660w、397f/635f/660w、397f/635w、397f/660w、397f/664w、397w、397w/513f、397w/513f/635f/656f/660f/664w、397w/513f/635f/660w、397w/513f/635w、397w/513f/660f、397w/513f/660w、397w/513f/660y/664f、397w/513w、397w/513w/635f、397w/513w/635f/656w/660f、397w/513w/635w、397w/513w/635w/656y、397w/513w/635w/660w、397w/513w/635w/660w/664y、397w/513w/635w/660y/664y、397w/513w/660w、397w/513w/660w/664y、397w/513w/664f、397w/513w/664w、397w/513y/635f、397w/513y/635w/656y/660w/664w、397w/513y/635w、397w/513y/635w/656y/664y、397w/513y/635w/660f、397w/635f、397w/635f/664f、397w/635w、397w/635w/656f/664f、397w/635w/656f/664w、397w/635w/656f/664y、397w/635w/660w、397w/635w/660y、397w/635w/664f、397w/635w/664w/850t、397w/660f/664f、397w/660f/664y、397w/660w、397w/837k、397y、397y/513f/635w/656f/660w/664f、397y/513f/635w/664w、397y/513w/635f/664y、397y/513w/635w/660f、397y/513w/656w/660w、397y/513y、397y/513y/635w、397y/635f、397y/635f/660w、397y/635w、397y/635w/656f/660y/664w、397y/635w/660f、397y/660f/664w、397y/664f、399e/635f/660w、475v/513w/635w/660y、513f、513f/635f、513f/635w、513f/635w/656w、513f/635w/664w、513f/660w、513f/660w/664f、513f/660w/664y、513w、513w/635f、513w/635w、513y/635f/660f/664y、513w/635w/656w/660f、513w/635w/660f、513w/635w/664w、513w/656w/664w、513w/656y/660w、513w/660f、513w/660w、513y/635f、513y/635f/664w、513y/635r/656f/664y、513y/635w、513y/635w/660f、513y/635w/660y、513y/635w/660y/664f、513y/660w、513y/660w/664w、513y/660y/664f、513y/664y、635f/656f/664y、635f/656y/664w、635f/660w/664f、635w、635w/656w、635w/660f、635w/660w、635w/664w、656w/660f/664y、656w/660w/664y、658p、660f、660f/664f、660f/664y、660w/664f、660y/664w、664f和664w,和/或其任何组合,其中氨基酸位置参考seqidno:4编号。在一些另外的实施方案中,工程化rna聚合酶包含至少一个选自以下的取代或取代集:a397f、a397f/d513f、a397f/d513f/s635f、a397f/d513f/s635w、a397f/d513f/s635w/d660w、a397f/d513w/s635w、a397f/d513w/s635w/q656f/p664f、a397f/d513w/p664w、a397f/d513y、a397f/d513y/s635w、a397f/d513y/s635w/q656w、a397f/d513y/s635w/p664f、a397f/d513y/q656h/d660w、a397f/s635f/d660w、a397f/s635w、a397f/d660w、a397f/p664w、a397w、a397w/d513f、a397w/d513f/s635f/q656f/d660f/p664w、a397w/d513f/s635f/d660w、a397w/d513f/s635w、a397w/d513f/d660f、a397w/d513f/d660w、a397w/d513f/d660y/p664f、a397w/d513w、a397w/d513w/s635f、a397w/d513w/s635f/q656w/d660f、a397w/d513w/s635w、a397w/d513w/s635w/q656y、a397w/d513w/s635w/d660w、a397w/d513w/s635w/d660w/p664y、a397w/d513w/s635w/d660y/p664y、a397w/d513w/d660w、a397w/d513w/p664f、a397w/d513w/d660w/p664y、a397w/d513w/p664w、a397w/d513y/s635f、a397w/d513y/s635w/q656y/d660w/p664w、a397w/d513y/s635w、a397w/d513y/s635w/q656y/p664y、a397w/d513y/s635w/d660f、a397w/s635f、a397w/s635f/p664f、a397w/s635w、a397w/s635w/q656f/p664f、a397w/s635w/q656f/p664w、a397w/s635w/q656f/p664y、a397w/s635w/d660w、a397w/s635w/d660y、a397w/s635w/p664f、a397w/s635w/p664w/a850t、a397w/d660f/p664f、a397w/d660f/p664y、a397w/d660w、a397w/e837k、a397y、a397y/d513f/s635w/q656f/d660w/p664f、a397y/d513f/s635w/p664w、a397y/d513w/s635f/p664y、a397y/d513w/s635w/d660f、a397y/d513w/q656w/d660w、a397y/d513y、a397y/d513y/s635w、a397y/s635f、a397y/s635f/d660w、a397y/s635w、a397y/s635w/q656f/d660y/p664w、a397y/s635w/d660f、a397y/d660f/p664w、a397y/p664f、k399e/s635f/d660w、a475v/d513w/s635w/d660y、d513f、d513f/s635f、d513f/s635w、d513f/s635w/q656w、d513f/s635w/p664w、d513f/d660w、d513f/d660w/p664f、d513f/d660w/p664y、d513w、d513w/s635f、d513w/s635w、d513y/s635f/d660f/p664y、d513w/s635w/q656w/d660f、d513w/s635w/d660f、d513w/s635w/p664w、d513w/q656w/p664w、d513w/q656y/d660w、d513w/d660f、d513w/d660w、d513y/s635f、d513y/s635f/p664w、d513y/s635r/q656f/p664y、d513y/s635w、d513y/s635w/d660f、d513y/s635w/d660y、d513y/s635w/d660y/p664f、d513y/d660w、d513y/d660w/p664w、d513y/d660y/p664f、d513y/p664y、s635f/q656f/p664y、s635f/q656y/p664w、s635f/d660w/p664f、s635w、s635w/q656w、s635w/d660f、s635w/d660w、s635w/p664w、q656w/d660f/p664y、q656w/d660w/p664y、l658p、d660f、d660f/p664f、d660f/p664y、d660w/p664f、d660y/p664w、p664f/w,和/或其任何组合,其中氨基酸位置参考seqidno:4编号。
在一些实施方案中,工程化rna聚合酶包含至少一个选自以下的取代或取代集:113/137/513、136/357/404/514、136/357/514、136/394/404/446、136/401、136/401/404、136/404/446、136/404/514、136/446、136/514、137、137/401、137/401/513、137/401/513、137/513、137/513/621、137/635、137/656、357/394/401/404/514、357/394/446/514、357/514、394/446/514、401/404、401/404/514、401/513/635、401/635、513/635、513/635/656、513/660、635/656、635/660和660,和/或其任何组合,其中氨基酸位置参考seqidno:15编号。在一些另外的实施方案中,工程化rna聚合酶包含至少一个选自以下的取代或取代集:113m/137w/513r、136e/357i/404y/514i、136i/357i/514f、136i/357k/514f、136i/394r/404y/446w、136i/401v、136i/401v/404y、136e/404y/446w、136e/404y/514f、136i/446w、136e/514f、136i/514i、137w、137w/401i、137w/401s、137w/401s/513r、137w/401s/513w、137w/401v、137w/513w、137w/513r/621r、137w/635w、137w/656f、357n/394r/446w/514i、357r/394r/401v/404y/514l、357r/514f、394r/446w/514i、401s/513r/635w、401s/635w、401v/404y、401v/404y/514l、513l/635w、513l/660w、513r/635w/656f、635w/656f、635w/660t和660s/t,和/或其任何组合,其中氨基酸位置参考seqidno:15编号。在一些另外的实施方案中,工程化rna聚合酶包含至少一个选自以下的取代或取代集:l113m/d137w/d513r、a136e/e357i/s404y/s514i、a136e/s404y/m446w、a136e/s404y/s514f、a136e/s514f、a136i/e357i/s514f、a136i/e357k/s514f、a136i/k394r/s404y/m446w、a136i/r401v、a136i/r401v/s404y、a136i/m446w、a136i/s514i、d137w、d137w/r401i、d137w/r401s、d137w/r401s/d513r、d137w/r401s/d513w、d137w/r401v、d137w/d513w、d137w/d513r/k621r、d137w/s635w、d137w/q656f、e357n/k394r/m446w/s514i、e357r/k394r/r401v/s404y/s514l、e357r/s514f、k394r/m446w/s514i、r401s/d513r/s635w、r401s/s635w、r401v/s404y、r401v/s404y/s514l、d513l/s635w、d513l/d660w、d513r/s635w/q656f、s635w/q656f、s635w/d660t和d660s/t,和/或其任何组合,其中氨基酸位置参考seqidno:15编号。
在一些另外的实施方案中,工程化rna聚合酶包含与至少一种表5.3、表5.4、表5.5和/或表5.6中列出的工程化rna聚合酶变体的序列至少85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更多相同的多肽序列。在一些另外的实施方案中,工程化rna聚合酶包含表5.3、表5.4、表5.5和/或表5.6中提供的变体工程化聚合酶。在一些另外的实施方案中,工程化rna聚合酶是表5.3、表5.4、表5.5和/或表5.6中提供的变体工程化聚合酶。
在还一些另外的实施方案中,工程化rna聚合酶包含与seqidno:4、15、17、19、21、23、25、27、29、31、33、35、37和/或39中列出的至少一种工程化rna聚合酶变体的序列至少85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更多相同的多肽序列。在一些另外的实施方案中,工程化rna聚合酶包括seqidno:4、15、17、19、21、23、25、27、29、31、33、35、37或39中列出的变体工程化聚合酶。在一些另外的实施方案中,工程化rna聚合酶是seqidno:4、15、17、19、21、23、25、27、29、31、33、35、37或39中列出的变体工程化聚合酶。
在一些另外的实施方案中,与野生型t7rna聚合酶相比,工程化聚合酶包含至少一种改进的性质。在一些另外的实施方案中,工程化rna聚合酶表现出至少一种改进的性质,该至少一种改进的性质选自转录起始期间相对于gtp对帽类似物改进的选择性、改进的蛋白质表达、储存缓冲液中改进的稳定性和反应条件下改进的稳定性。在一些另外的实施方案中,工程化rna聚合酶保持rna收率、转录保真性、热稳定性、蛋白质表达、在-20℃的稳定性或在反应条件中与野生型t7rna聚合酶相当的稳定性。在还一些另外的实施方案中,工程化rna聚合酶被纯化。在仍然一些另外的实施方案中,本发明提供了包含至少一种本文提供的工程化rna聚合酶的组合物。
本发明还提供了编码至少一种本文提供的工程化rna聚合酶的多核苷酸序列。在一些实施方案中,多核苷酸序列编码至少一种工程化rna聚合酶,所述工程化rna聚合酶包含与参考序列seqidno:4和/或seqidno:15或其功能片段至少85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更高的序列同一性,其中工程化rna聚合酶包含在一个或更多个氨基酸位置的至少一个取代。在一些另外的实施方案中,多核苷酸序列编码本文提供的工程化rna聚合酶,所述工程化rna聚合酶包含与seqidno:4、15、17、19、21、23、25、27、29、31、33、35、37和/或39或其功能片段至少85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更高的序列同一性。在一些另外的实施方案中,编码至少一种工程化rna聚合酶的多核苷酸序列包含与seqidno:3、14、16、18、20、22、24、26、28、30、32、34、36和/或38至少85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更高的序列同一性。在还一些另外的实施方案中,多核苷酸序列被可操作地连接至控制序列。在仍然一些另外的实施方案中,多核苷酸序列是密码子优化的。
本发明还提供了表达载体,该表达载体包含至少一种本文提供的多核苷酸序列。本发明还提供了包含至少一种本文提供的表达载体的宿主细胞。本发明还提供了用于在宿主细胞中产生工程化rna聚合酶的方法,该方法包括在合适的培养条件下培养本文提供的宿主细胞,从而产生至少一种工程化rna聚合酶。在一些实施方案中,所述方法还包括从培养物和/或宿主细胞回收至少一种工程化rna聚合酶。在还一些另外的实施方案中,所述方法还包括纯化至少一种工程化rna聚合酶的步骤。
本发明还提供了用于产生加帽的rna转录物的方法,该方法包括提供组合物,该组合物包含:i)至少一种本文提供的工程化rna聚合酶,二核苷酸帽类似物,和ii)dna模板;在使得工程化rna聚合酶产生加帽的rna转录物的条件下,将dna模板暴露于组合物。在所述方法的一些实施方案中,二核苷酸帽类似物是α,γ-双(n7-甲基鸟苷)三磷酸(m7g(5’)ppp(5’)m7g)或抗逆转(anti-reverse)帽类似物3’-o-me-m7g(5')ppp(5')g。在所述方法的一些另外的实施方案中,二核苷酸帽类似物是α,γ-双(n7-甲基鸟苷)三磷酸。在所述方法的还一些另外的实施方案中,将无机焦磷酸酶添加至反应中。
附图简述
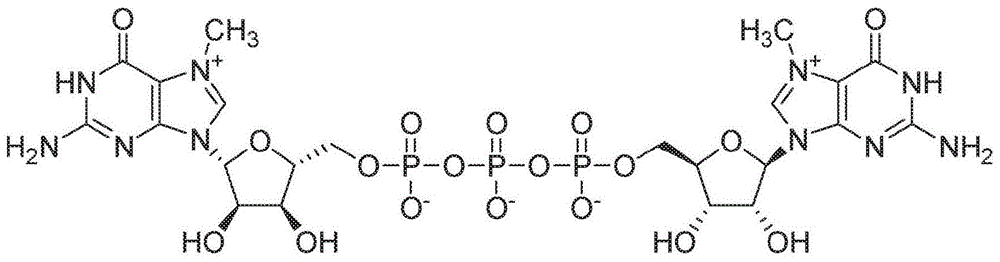
图1提供了“m7g(5’)ppp(5’)m7g”,也被称为“α,γ-双(n7-甲基鸟苷)三磷酸”、“加帽gtp”、“对称帽类似物”或“帽”的化学结构。
发明描述
本发明提供了工程化t7rna聚合酶变体及其组合物。这些变体已经被演化用于在体外转录起始时相对于gtp选择性掺入m7g(5’)ppp(5’)m7g帽类似物。本发明还提供了用于对rna转录物选择性加帽的方法。此外,本发明提供了包含本文提供的rna聚合酶变体的方法和组合物。
rna聚合酶、转录和加帽:
真核细胞具有多种类型的核rna聚合酶,每一种负责合成不同子集的rna。所有这些核rna聚合酶都在结构和机制上彼此相关,并且与细菌rna聚合酶(rnap)相关。rnapi合成前rrna45s(在酵母中是35s);rnapii合成mrna和大多数snrna和微rna的前体;rnapiii合成在细胞核和胞质溶胶中发现的trna、rrna5s和其他小rna;rnapiv合成植物中的sirna;并且rnapv合成参与植物中sirna指导的异染色质形成的rna。真核叶绿体含有在结构和机制上高度类似于细菌rnap的rnap(“质体编码的聚合酶”)。真核叶绿体还含有第二种结构和机制上不相关的rnap(“细胞核编码的聚合酶”;“单亚基rnap”蛋白质家族的成员)。真核线粒体含有结构和机制上不相关的rnap(“单亚基rnap”蛋白家族的成员)。
病毒(包括噬菌体)、叶绿体和线粒体典型的单亚基rnap已经被表征,并且尤其可用于体外转录。许多病毒使用单亚基dna依赖性rnap,该单亚基dna依赖性rnap在结构和机制上与真核叶绿体和线粒体的单亚基rnap相关,并且更远地,与dna聚合酶和逆转录酶相关。
转录以至少三个不同的阶段发生(即,起始、延伸和终止)。起始阶段开始于开放复合体的形成,并且在rnap造成向稳定延伸复合体的转变时结束(参见,hsu,biochim.biophys.acta,1577:191-207[2002];kinsella等人,biochem.,21:2719-23[1982];straney和crothers,j.mol.biol.,193:267-78[1987];和young等人,cell,109:417-20[2002])。在转录起始期间,rnap产生长度为2个至约8个核苷酸的短流产产物(abortiveproduct),同时与启动子保持稳定的相互作用(参见,carpousis和gralla,j.mol.biol.,183:165-77[1985];hieb等人,emboj.,25:3100-09[2006];ikeda和richardson,proc.natl.acad.sci.usa,83:3614-8[1986];ikeda和richardson,j.biol.chem.,262:3800-8[1987];以及krummel和chamberlin,biochem.,21:2719-23[1989])。当rna转录物的长度达到长度超过8个核苷酸时,rnap开始将它本身从启动子释放出来,因为其转变成延伸复合体(参见,bandwar等人,j.mol.biol.,360:466-83[2006];esposito和martin,j.biol.chem.,279:44270-6[2004];guo和sousa,j.biol.chem.,280:3474-82[2005];和temiakov等人,proc.natl.acad.sci.usa,97:14109-14[2000])。在一些rnap中(例如,单亚基t7rnap),从起始复合体(ic)向延伸复合体(ec)的转变涉及大规模的蛋白质结构再组织(参见,cheetham等人,coldspringharb.symp.quant.biol.,63:263-7[1998];cheetham等人,nature,399:80-83[1999];cheetham和steitz,science,286:2305-9[1999];guo等人,j.mol.biol.,353:256-70[2005];ma等人,j.biol.chem.,277:43206-15[2002];mukherjee等人,cell,110:81-91[2002];tahirov等人,nature,420:43-50[2002];以及yin和steitz,science,298:1387-95[2002])。
转录和随后的加工涉及多种过程和酶。如上文描述的,转录通过rna聚合酶的作用来实现。尽管加帽通常发生在转录仍在进行时,由于加帽酶和rna聚合酶之间的相互作用,该过程涉及单独的酶机制。
本发明提供了这样的rna聚合酶:该rna聚合酶已经被修饰以优先接受帽(也被称为rna帽、rna7-甲基鸟苷帽或rnam7g帽)或帽类似物(例如,“抗逆转帽类似物”(3'-o-me-m7g(5')ppp(5')g;“arca”),或具有转录起始位点的一个或更多个核苷酸的甲基化帽类似物(例如,m7g(5’)ppp(5’)n,其中n是任何核苷酸)以在转录起始期间开始转录。5’帽是在一些真核初级转录物诸如前体信使rna的5’端上的改变的核苷酸。典型的帽结构由经由5’-5’三磷酸桥连接至转录物的第一个核苷酸(“帽-1”位置)的7-甲基鸟苷组成。帽类似物可以在特定位置包含例如一个、两个或更多个甲基(或其他取代)基团。
因此,本发明提供了用于有效产生加帽的rna的组合物和方法。大多数真核细胞mrna转录物和大多数真核病毒mrna转录物,以及其他形式的真核rna(例如,小核rna[snrna]和前微rna[前mirna])在其5’末端被封端或“加帽”(参见例如,美国专利第8,846,348号,该美国专利通过引用以其整体并入本文)。帽是经由其5’碳连接至三磷酸基团的鸟嘌呤核苷,该三磷酸基团连接至初级mrna转录物的最5’端的核苷酸的5’碳。在大多数真核细胞中,帽核苷酸中的鸟嘌呤的7位的氮被甲基化。当mrna在长度上仅为约30个核苷酸时,加帽通常发生在通过酶过程起始之后。起点是rna分子的未改变的5’端,该未改变的5’端以羟基基团终止。随着新生的前mrna被合成,加帽过程在转录完成之前起始。加帽过程包括通过rna三磷酸酶除去末端磷酸基团中的一个,留下二磷酸基团。gtp通过mrna鸟苷转移酶添加至末端二磷酸,在该过程中从gtp底物失去焦磷酸。这导致5′–5′三磷酸键,产生g(5’)ppp(5’)n[pn]。鸟嘌呤的7-氮被mrna(鸟嘌呤-n7)-甲基转移酶甲基化,s-腺苷-l-甲硫氨酸被去甲基化,以产生s-腺苷基-l-高半胱氨酸和m7g(5’)ppp(5’)n[pn]。邻近帽的修饰可以发生,通常发生在新生rna转录物的第一和第二核苷酸位置(即,帽-1和帽-2位置)。如果最邻近帽的核苷酸是2’-o-核糖甲基-腺苷,则它可以在n6甲基位置进一步甲基化以形成n6-甲基腺苷。
这些rna分子的5’帽在rna稳定性和加工中具有重要作用。帽在mrna代谢中具有非常重要的作用,并且是细胞核中rna转录物的加工和成熟、mrna从细胞核至细胞质的转运、mrna稳定性以及mrna至蛋白质的有效翻译所需的(参见例如,lewis等人,eur.j.biochem.,247:461-9[1997].)。如上文指出的,5’帽结构参与真核mrna和病毒mrna的蛋白质合成的起始。5’帽还提供了对5’-核酸外切酶活性的耐受性,并且其缺失导致了mrna的快速降解(参见例如,美国专利第8,836,348号;和furiichi等人,nature,266:235-9[1997])。由于加帽提供的优点,加帽的rna转录物的有效合成为多种功能提供了相当大的价值,包括但不限于体外和体内蛋白质合成。
加帽的rna的体外合成实际上可以通过两种酶途径中的一种来完成。牛痘病毒加帽酶具有两个亚基和三种酶活性,将7-甲基鸟苷帽结构添加至体外转录的rna的5’磷酸(参见,mao和shuman,j.biol.chem.,269:24472-9[1994])。
t7rna聚合酶:
本发明提供了单亚基rnap(例如,修饰的t7rnap),该单亚基rnap已经被修饰,使得它们优先产生包含5’帽或5’帽类似物的加帽的rna转录物,而无需另外的酶加帽步骤。本发明提供了工程化t7rna聚合酶变体及其组合物。这些变体已经被演化用于在体外转录起始时相对于gtp选择性掺入m7g(5’)ppp(5’)m7g类似物。本发明还提供了使用本文提供的变体的方法。本发明还提供了本文提供的组合物的用途。
t7rna聚合酶(e.c.2.7.7.6),是一种单体噬菌体编码的dna指导的rna聚合酶,催化以5’至3’方向形成rna。在转录起始的过程中,t7rna聚合酶识别特定的启动子序列(即,t7启动子)。天然存在的t7rna聚合酶包含883个氨基酸。它与t3rna聚合酶高度同源,并且与sp6rna聚合酶在一定程度上同源。t7包含多个结构域,包括n末端结构域、“拇指”、“手掌”和“手指”(参见例如,sousa和mukherjee,prog.nucl.acidres.mol.biol.,73:1-41[2003],和美国专利第9,193,959号)。在功能酶的起始阶段和延伸阶段之间,n末端结构域的构象发生变化。t7rnap的ic结构和ec结构是转录中涉及的转变过程中的开始构象和最终构象。呈ec构象的t7rnap是高度续进性(processive)的dna依赖性rnap。处于其ic构象时,它识别特定的启动子序列,并且首先进入“流产阶段(abortivephase)”,在该阶段合成并释放了非常短的转录物,然后进行长rna链的续进性转录。ic的结构允许掺入帽或帽类似物,例如,arca,从而允许在mrna转录物的5’端处加载帽或帽类似物。已经报道了体外使用单亚基t7rnap,利用相对于gtp过量的m7g(5’)ppp(5’)g(或简单地m7gpppg)最高80%的加帽率(参见,nielsen和shapiro,nucl.acidsres.,14:5936,[1986])。已经对t7rnap的结合的鸟苷和m7gpppg的结构进行了确定(参见,kennedy等人,j.mol.biol.,370:256-68[2007]),指示相比鸟苷,m7gpppg仅有单一的另外的接触。因此,在一些实施方案中,在起始构象阶段中结构上接近结合位点的rnap中的氨基酸的改变被用于赋予使用帽或帽类似物来开始转录的进一步的特异性和效率(即,当rnap处于其ic构象时,帽或帽类似物在帽位置处优先掺入)。核苷酸帽或帽类似物的
尽管体外转录不完全依赖于使用加帽酶来产生加帽的转录物,但通常由目前使用的方法产生的80%收率往往太低而没有商业效用。
已经描述了编码t7rna聚合酶的基因的克隆和表达(参见例如,美国专利第4,952,496号)。由于其启动子特异性和高rna聚合酶活性,t7已经用于多种应用。它也可用于大肠杆菌(e.coli)中重组基因的高水平表达(参见,studier和moffat,j.mol.biol.,18:113-130[1986])。t7也用于多种核酸扩增方法,包括用于诊断方法的那些核酸扩增方法。由于稳定性和热稳定性通常是诊断方法组分的开发中的重要考虑因素,因此已经报道了改进t7的热稳定性和稳定性的工作(参见例如,美国专利第9,193,959号、第8,551,752号和第7,507,567号等)。然而,对于与天然存在的酶相比表现出改进的性质的变体t7酶,本领域仍然存在需求。
用于体外合成上文讨论的加帽的rna的可选择的方法是在体外转录期间使用帽类似物掺入帽结构。这些含7-甲基鸟苷的二核苷酸含有5’-5’三磷酸酯键,并且在转录起始位点处被掺入,产生7-甲基鸟苷加帽的rna产物。t7rna聚合酶随着相对于模板上的胞嘧啶残基掺入鸟苷而天然地起始转录。为了实现加帽的高效率,鸟苷帽类似物必须以与gtp相比高的浓度存在于体外转录反应中,鸟苷帽类似物与gtp竞争在mrna的第一个位置处掺入。体外共转录加帽的一个有用特征是,因为在起始期间实现了帽掺入,全长mrna的最终序列或二级结构不影响加帽的程度。此外,该过程仅需要在单步反应中使用一种酶(rna聚合酶)。化学合成的帽类似物的使用也增加了使用不能很好地被牛痘加帽酶识别甲基化的可选择的核苷酸帽结构的灵活性。然而,相对于核苷酸和其他反应组分,帽类似物是昂贵的,并且必须以高浓度存在于反应中。本发明提供了这样的t7rna聚合酶变体,该t7rna聚合酶变体在转录起始时相对于gtp选择性地用于掺入帽类似物,从而能够使用降低浓度的帽类似物进行有效的加帽,为加帽的rna提供了更高成本效益且可放大规模的产生方法。
在本发明的一些实施方案中,无机焦磷酸酶存在于体外转录反应期间,以将焦磷酸(pyrophosphate)降解成正磷酸,所述焦磷酸是rna聚合酶转录的产物抑制剂。
因此,本发明提供了修饰的rnap,该修饰的rnap优先掺入帽或帽类似物,以允许相对于未加帽的mrna大于约80%的加帽的mrna的收率。例如,修饰的rnap可以产生相对于未加帽的mrna大于85%或约85%、大于90%或约90%、大于95%或约95%、大于99%或约99%的加帽的mrna的mrna。
在一些实施方案中,修饰的rnap衍生自单亚基rnap(即,t7rnap)的序列和结构。本发明提供了修饰的t7rnap,该修饰的t7rnap具有提高的掺入“加帽g”(m7gpppg)、m7gpppn和/或arca的效率。改进的t7rnap变体有助于更有效且更高成本效益地制造用于药物用途和医疗用途的加帽的rna。相对于未修饰的rnap,这些修饰的rnap还提供了改进的加帽的rna收率,而不损害或仅最小程度地改变转录效率。
缩写和定义:
除非另外定义,否则本文使用的所有技术和科学术语通常具有与本发明所属领域的普通技术人员通常理解的相同的含义。通常,本文使用的命名法和下文描述的细胞培养、分子遗传学、微生物学、生物化学、有机化学、分析化学和核酸化学的实验室程序是本领域众所周知和常用的那些。这样的技术是众所周知的,并且在本领域技术人员熟知的许多教科书和参考文献中进行了描述。对于化学合成和化学分析,使用了标准技术或其修改形式。本文(上文和下文两者)提及的所有专利、专利申请、文章和出版物,在此通过引用明确地并入本文。
尽管本发明的实践中可使用类似于或等同于本文描述那些方法和材料的任何合适的方法和材料,本文描述了一些方法和材料。应理解本发明不限于所描述的特定方法论、方案和试剂,因为这些可以取决于本领域技术人员使用其的环境而变化。因此,下文紧接着定义的术语通过参考本申请作为整体而被更充分地描述。本文(上文和下文两者)提及的所有专利、专利申请、文章和出版物,在此通过引用明确地并入本文。
此外,除非上下文另外清楚地指示,否则如本文使用的单数“一(a)”、“一(an)”和“该(the)”包括复数指示物。
数值范围包括限定该范围的数字。因此,本文公开的每个数值范围意图涵盖落在此类较宽数值范围内的每一较窄数值范围,如同此类较窄数值范围在本文被全部明确地写出。还意图本文公开的每个最大(或最小)的数值限制包含每个较低(或较高)的数值限制,如同此类较低(或较高)的数值限制在本文中被明确地写出。
术语“约”意指特定值的可接受误差。在一些情况下,“约”意指给定值范围的0.05%、0.5%、1.0%或2.0%以内。在一些情况下,“约”意指给定值的1个、2个、3个或4个标准差以内。
此外,本文提供的标题不是本发明的各个方面或实施方案的限制,本发明的各个方面或实施方案可以通过参考作为整体的本申请而获得。
因此,下文紧接着定义的术语通过参考本申请作为整体而被更充分地定义。然而,为了帮助理解本发明,许多术语在下文中被定义。
除非另外指示,否则分别地,核酸以5’至3’方向从左至右书写;氨基酸序列以氨基至羧基方向从左至右书写。
如本文使用的,术语“包括/包含(comprising)”及其同根词以其包含性含义被使用(即,等同于术语“包括/包含(including)”及其相应的同根词)。
“ec”编号指的是生物化学和分子生物学国际联合命名委员会(nomenclaturecommitteeoftheinternationalunionofbiochemistryandmolecularbiology)(nc-iubmb)的酶命名法。该iubmb生物化学分类是基于酶催化的化学反应的酶的数字分类系统。
“atcc”指的是美国典型培养物保藏中心(americantypeculturecollection),其生物保藏收集物包括基因和菌株。
“ncbi”指的是美国国家生物技术信息中心(nationalcenterforbiologicalinformation)和在其中提供的序列数据库。
如本文使用的,“t7rna聚合酶”指的是单体t7噬菌体编码的dna指导的rna聚合酶,其催化以5’至3’方向形成rna。
如本文使用的,术语“帽”指的是鸟嘌呤核苷,该鸟嘌呤核苷经由其5’碳连接至三磷酸基团,该三磷酸基团继而连接至mrna转录物的最5’端的核苷酸的5’碳。在一些实施方案中,帽中鸟嘌呤的7位处的氮被甲基化。
如本文使用的,术语“加帽的rna”、“5’加帽的rna”和“加帽的mrna”分别指包含帽的rna和mrna。
如本文使用的,“多核苷酸”和“核酸”指的是共价连接在一起的两个或更多个核苷。多核苷酸可以完全包含核糖核苷酸(即,rna)、完全包含2’脱氧核糖核苷酸(即,dna)或包含核糖核苷酸和2’脱氧核糖核苷酸的混合物。虽然核苷典型地将经由标准磷酸二酯键连接在一起,但多核苷酸可以包含一个或更多个非标准键。多核苷酸可以是单链或双链的,或者可以包含单链区域和双链区域二者。此外,虽然多核苷酸典型地将包含天然存在的编码核苷碱基(即,腺嘌呤、鸟嘌呤、尿嘧啶、胸腺嘧啶和胞嘧啶),它可以包含一种或更多种修饰的和/或合成的核苷碱基,诸如例如肌苷、黄嘌呤、次黄嘌呤等。在一些实施方案中,这样的修饰的或合成的核苷碱基是编码氨基酸序列的核苷碱基。
“蛋白质”、“多肽”和“肽”在本文可互换使用,以表示通过酰胺键共价连接的至少两个氨基酸的聚合物,而不论长度或翻译后修饰(例如,糖基化或磷酸化)如何。
“氨基酸”通过其通常已知的三字母符号或通过iupac-iub生物化学命名委员会推荐的单字母符号在本文被提及。同样地,核苷酸可以通过其通常可接受的单字母代码被提及。用于遗传编码的氨基酸的缩写是常规的并且如下:丙氨酸(ala或a)、精氨酸(are或r)、天冬酰胺(asn或n)、天冬氨酸(asp或d)、半胱氨酸(cys或c)、谷氨酸(glu或e)、谷氨酰胺(gln或q)、组氨酸(his或h)、异亮氨酸(ile或i)、亮氨酸(leu或l)、赖氨酸(lys或k)、甲硫氨酸(met或m)、苯丙氨酸(phe或f)、脯氨酸(pro或p)、丝氨酸(ser或s)、苏氨酸(thr或t)、色氨酸(trp或w)、酪氨酸(tyr或y)和缬氨酸(val或v)。
当使用三字母缩写时,除非前面明确有“l”或“d”,或者从使用缩写的上下文清楚看出,否则氨基酸可以关于α-碳(cα)是l-构型或d-构型。例如,“ala”表示丙氨酸而不指定关于α-碳的构型,而“d-ala”和“l-ala”分别表示d-丙氨酸和l-丙氨酸。当使用单字母缩写时,大写字母表示关于α-碳的l-构型的氨基酸,并且小写字母表示关于α-碳的d-构型的氨基酸。例如,“a”表示l-丙氨酸并且“a”表示d-丙氨酸。当多肽序列以单字母缩写或三字母缩写(或其混合物)的串呈现时,根据常规惯例将序列以氨基(n)至羧基(c)方向呈现。
用于遗传编码核苷的缩写是常规的并且如下:腺苷(a);鸟苷(g);胞苷(c);胸苷(t);和尿苷(u)。除非具体阐明,否则缩写的核苷可以是核糖核苷或2'-脱氧核糖核苷。核苷可以基于个体或基于集合体(aggregate)被指定为核糖核苷或2'-脱氧核糖核苷。当核酸序列以单字母缩写串呈现时,根据常规惯例将序列以5'至3'方向呈现,并且不示出磷酸。
当提及细胞、多核苷酸或多肽使用时,术语“工程化”、“重组”、“非天然存在的”和“变体”指如下材料或与该材料的天然或自然形式对应的材料:已经以自然界中本来不存在的方式被修饰或与其相同但由合成材料和/或通过使用重组技术操作来产生或获得。
如本文使用的,“野生型”和“天然存在的”指的是在自然界中发现的形式。例如野生型多肽或多核苷酸序列是生物体中存在的序列,其可以从天然来源分离并且未通过人为操作被有意识地修饰。
“编码序列”指的是编码蛋白质的氨基酸序列的核酸的那部分(例如,基因)。
本文使用术语“序列同一性百分比(%)”以指多核苷酸和多肽之间的比较,并且通过跨比较窗比较两条最佳比对的序列来确定,其中为了两个序列的最佳比对,多核苷酸或多肽序列在比较窗中的部分与参考序列相比可以包含添加或缺失(即,空位)。百分比可以如下计算:确定两个序列中出现相同的核酸碱基或氨基酸残基的位置的数目,以得到匹配位置的数目,将匹配位置的数目除以比较窗中位置的总数目,并且将结果乘以100以得到序列同一性的百分比。可选择地,百分比可以如下计算:确定两个序列中出现相同的核酸碱基或氨基酸残基的位置的数目或者核酸碱基或氨基酸残基与空位对齐的位置的数目,以得到匹配位置的数目,将匹配位置的数目除以比较窗中位置的总数目,并且将结果乘以100以得到序列同一性的百分比。本领域技术人员理解,存在许多可用于比对两个序列的已建立的算法。用于比较的序列的最佳比对可以通过以下来进行,例如,smith和waterman的局部同源性算法(smith和waterman,adv.appl.math.,2:482[1981]),needleman和wunsch的同源性比对算法(needleman和wunsch,j.mol.biol.,48:443[1970),pearson和lipman的相似性方法的检索(pearson和lipman,proc.natl.acad.sci.usa85:2444[1988]),这些算法的计算机化实现(例如,gcgwisconsin软件包中的gap、bestfit、fasta和tfasta),或者通过视觉检查,如本领域已知的。适合用于确定序列同一性百分比和序列相似性的算法的实例包括但不限于blast和blast2.0算法,其由altschul等人描述(分别参见,altschul等人,j.mol.biol.,215:403-410[1990];和altschul等人,nucleicacidsres.,25:3389-3402[1977])。用于进行blast分析的软件是通过美国国家生物技术信息中心网站公共可得的。该算法包括首先通过鉴定查询序列中长度w的短字来鉴定高评分序列对(hsp),所述短字当与数据库序列中相同长度的字比对时匹配或满足某一正值的阈值评分t。t被称为邻近字评分阈值(参见,altschul等人,同上)。这些最初的邻近字击中(wordhit)充当用于启动检索的种子以找到更长的包含它们的hsp。然后字击中沿着每个序列的两个方向延伸直到累积比对评分不能增加的程度。对于核苷酸序列,累积评分使用参数m(对于匹配残基对的奖励评分;始终>0)和n(对于错配残基的惩罚评分;始终<0)计算。对于氨基酸序列,评分矩阵用于计算累积评分。当:累积比对评分从其最大达到值下降了量x;由于累积一个或更多个负评分残基比对,累积评分达到0或低于0;或到达任一序列末端时,字击中在每一个方向的延伸被终止。blast算法参数w、t和x决定比对的灵敏性和速度。blastn程序(对于核苷酸序列)使用以下作为缺省值:11的字长(w)、10的期望值(e)、m=5、n=-4、以及两条链的比较。对于氨基酸序列,blastp程序使用以下作为缺省值:3的字长(w),10的期望值(e)和blosum62评分矩阵(参见,henikoff和henikoff,proc.natl.acad.sci.usa89:10915[1989])。序列比对和序列同一性%的示例性确定可以使用gcgwisconsin软件包(accelrys,madisonwi)中的bestfit或gap程序,使用提供的缺省参数。
“参考序列”指的是用作序列比较的基础的指定序列。参考序列可以是较大序列的子集,例如,全长基因或多肽序列的区段。通常,参考序列为至少20个核苷酸或氨基酸残基的长度、至少25个残基的长度、至少50个残基的长度、至少100个残基的长度或核酸或多肽的全长。由于两个多核苷酸或多肽可以各自(1)包含两个序列之间相似的序列(即,完整序列的一部分),并且(2)还可以包含两个序列之间不同的序列,因此两个(或更多个)多核苷酸或多肽之间的序列比较典型地通过比较两个多核苷酸或多肽在“比较窗”上的序列来鉴定和比较序列相似性的局部区域来进行。在一些实施方案中,“参考序列”可以基于一级氨基酸序列,其中参考序列是可以在一级序列中具有一个或更多个变化的序列。
“比较窗”指的是至少约20个连续核苷酸位置或氨基酸残基的概念区段,其中序列可以与至少20个连续核苷酸或氨基酸的参考序列比较,并且其中与参考序列(其不包含添加或缺失)相比,为了两个序列的最佳比对,序列在比较窗中的部分可以包含20%或更少的添加或缺失(即,空位)。比较窗可以比20个连续残基更长,并且任选地包括30、40、50、100或更长的窗。
在用于给定的氨基酸或多核苷酸序列的编号的情况下,“对应于”、“参考于”或“相对于”指当给定的氨基酸或多核苷酸序列与参考序列相比较时指定的参考序列残基的编号。换言之,给定的聚合物的残基数目或残基位置关于参考序列被指定,而不是通过给定的氨基酸或多核苷酸序列内残基的实际数字位置被指定。例如,给定的氨基酸序列,诸如工程化t7rna聚合酶的氨基酸序列可以通过引入空位以与参考序列对齐而优化两个序列之间的残基匹配。在这些情况下,尽管存在空位,在给定的氨基酸或多核苷酸序列中的残基的编号根据与其比对的参考序列来做出。
“氨基酸差异”或“残基差异”指的是在多肽序列的一个位置处的氨基酸残基相对于参考序列中对应位置处的氨基酸残基的差异。氨基酸差异的位置通常在本文中被称为“xn”,其中n指的是残基差异所基于的参考序列中的对应位置。例如,“与seqidno:2相比在位置x93处的残基差异”指的是在对应于seqidno:2的位置93的多肽位置处的氨基酸残基的差异。因此,如果参考多肽seqidno:2在位置93处具有丝氨酸,则“与seqidno:2相比在位置x93处的残基差异”指的是在对应于seqidno:2的位置93的多肽位置处除了丝氨酸以外的任何残基的氨基酸取代。在本文的大多数情况下,在一个位置处的特定氨基酸残基差异被指示为“xny”,其中“xn”指定如上文描述的对应位置,并且“y”是在工程化多肽中发现的氨基酸(即,与参考多肽中不同的残基)的单字母标识符。在一些情况下(例如,在本文实施例中提供的表格中),本公开内容还提供由常规符号“anb”表示的特定氨基酸差异,其中a是参考序列中的残基的单字母标识符,“n”是在参考序列中的残基位置的编号,并且b是工程化多肽的序列中残基取代的单字母标识符。在一些情况下,本公开内容的多肽相对于参考序列可以包含一个或更多个氨基酸残基差异,其由相对于参考序列存在残基差异的一列指定位置指示。在一些实施方案中,在多于一个氨基酸可以在多肽的特定残基位置中使用的情况下,可以使用的多种氨基酸残基由“/”分开(例如,x10h/x10p或x10h/p)。在一些实施方案中,酶变体包含多于一个取代。为了便于阅读,这些取代由斜线分开(例如,c14a/k122a)。本申请包括含一个或更多个氨基酸差异的工程化多肽序列,该一个或更多个氨基酸差异包括保守和非保守的氨基酸取代的任一者/或两者。
“保守氨基酸取代”指的是用具有类似侧链的不同残基来取代残基,并且因此通常包括用相同或类似的氨基酸定义类别中的氨基酸取代多肽中的氨基酸。例如但不限于,具有脂肪族侧链的氨基酸可以被另一个脂肪族氨基酸(例如,丙氨酸、缬氨酸、亮氨酸和异亮氨酸)取代;具有羟基侧链的氨基酸被另一个具有羟基侧链的氨基酸(例如,丝氨酸和苏氨酸)取代;具有芳香族侧链的氨基酸被另一个具有芳香族侧链的氨基酸(例如,苯丙氨酸、酪氨酸、色氨酸和组氨酸)取代;具有碱性侧链的氨基酸被另一个具有碱性侧链的氨基酸(例如,赖氨酸和精氨酸)取代;具有酸性侧链的氨基酸被另一个具有酸性侧链的氨基酸(例如,天冬氨酸或谷氨酸)取代;和/或疏水性氨基酸或亲水性氨基酸分别被另一个疏水性氨基酸或亲水性氨基酸取代。
“非保守取代”指的是用具有显著不同的侧链性质的氨基酸取代多肽中的氨基酸。非保守取代可以使用定义的组之间而不是定义的组之内的氨基酸,并且影响:(a)取代区域中的肽骨架的结构(例如,用脯氨酸取代甘氨酸),(b)电荷或疏水性,或(c)侧链体积。例如但不限于,示例性非保守取代可以是用碱性氨基酸或脂肪族氨基酸取代酸性氨基酸;用小氨基酸取代芳香族氨基酸;和用疏水性氨基酸取代亲水性氨基酸。
“缺失”指的是通过从参考多肽除去一个或更多个氨基酸的多肽修饰。缺失可以包括除去1个或更多个氨基酸、2个或更多个氨基酸、5个或更多个氨基酸、10个或更多个氨基酸、15个或更多个氨基酸或者20个或更多个氨基酸、多达组成参考酶的氨基酸总数的10%、或多达组成参考酶的氨基酸总数的20%,同时保留酶活性和/或保留工程化酶的改进的性质。缺失可以涉及多肽的内部和/或端部。在多种实施方案中,缺失可以包括连续的区段或可以是不连续的。
“插入”指的是通过向参考多肽添加一个或更多个氨基酸的多肽修饰。插入可以在多肽的内部或插入至羧基或氨基末端。如本文使用的插入包括本领域已知的融合蛋白。插入可以是氨基酸的连续区段或由天然存在的多肽中的一个或更多个氨基酸分开。
“分离的多肽”指的是与其天然伴随的其他污染物(例如,蛋白质、脂质和多核苷酸)大体上分开的多肽。该术语包括已经从它们的天然存在的环境或表达系统(例如,宿主细胞或体外合成)中取出或纯化的多肽。重组t7rna聚合酶多肽可以存在于细胞内、存在于细胞培养基中,或以多种形式(诸如裂解物或分离的制备物)制备。因此,在一些实施方案中,重组t7rna聚合酶多肽可以是分离的多肽。
“大体上纯的多肽”指的是其中多肽物质是存在的优势物质的组合物(即,在摩尔或重量基础上,多肽物质比在该组合物中的任何其他单独的大分子物质更丰富),并且当目标物质构成存在的大分子物质的按摩尔或%重量计的至少约50%时,通常为大体上纯的组合物。通常,大体上纯的t7rna聚合酶组合物构成组合物中存在的所有大分子物质的按摩尔或%重量计的约60%或更多、约70%或更多、约80%或更多、约90%或更多、约95%或更多和约98%或更多。在一些实施方案中,将目标物质纯化至基本同质(即,通过常规检测方法不能在组合物中检测出污染物物质),其中该组合物基本上由单一大分子物质组成。溶剂物质、小分子(<500道尔顿)和元素离子物质不被认为是大分子物质。在一些实施方案中,分离的重组t7rna聚合酶多肽是大体上纯的多肽组合物。
t7rna聚合酶的“改进的酶性质”指的是与参考t7rna聚合酶多肽和/或野生型t7rna聚合酶多肽和/或另一种工程化t7rna聚合酶多肽相比表现出任何酶性质的改进的工程化t7rna聚合酶多肽。改进的性质包括但不限于诸如以下的性质:相比gtp对帽类似物增加的选择性、增加的复制保真性、增加的rna收率、增加的蛋白质表达、增加的热活性(thermoactivity)、增加的热稳定性、增加的ph活性、增加的稳定性、增加的酶活性、增加的底物特异性或亲和力、增加的比活性、增加的对底物或终产物抑制(包括焦磷酸盐)的耐受性、增加的化学稳定性、改进的溶剂稳定性、增加的对酸性ph或碱性ph的耐受性、增加的对蛋白水解活性的耐受性(即,降低的对蛋白水解的敏感性)、减少的聚集、增加的溶解度、和改变的温度曲线(temperatureprofile)。
“增加的酶活性”或“增强的催化活性”指的是工程化t7rna聚合酶的改进的性质,其可以被表示为与参考t7rna聚合酶相比,比活性(例如,产生的产物/时间/重量蛋白质)的增加或底物向产物的转化百分比(例如,在指定的时间段使用指定量的变体t7rna聚合酶,起始量的底物向产物的转化百分比)的增加。确定酶活性的示例性方法被提供在实施例中。与酶活性相关的任何性质可能受到影响。
“杂交严格性”涉及在核酸杂交中的杂交条件,诸如洗涤条件。通常,杂交反应在较低严格性的条件下进行,随后是不同的但较高严格性的洗涤。术语“中度严格杂交”指的是允许靶dna结合以下互补核酸的条件:所述互补核酸与靶dna具有约60%同一性,优选地约75%同一性,约85%同一性,与靶多核苷酸具有大于约90%同一性。示例性中度严格条件是等同于在42℃于50%甲酰胺、5×denhart溶液、5×sspe、0.2%sds中杂交,随后在42℃于0.2×sspe、0.2%sds中洗涤的条件。“高严格性杂交”通常指的是与定义的多核苷酸序列在溶液条件下确定的热解链温度tm相差约10℃或更小的条件。在一些实施方案中,高严格性条件指的是仅允许在65℃于0.018mnacl中形成稳定的杂合体的那些核酸序列的杂交的条件(即,如果杂合体在65℃于0.018mnacl中是不稳定的,它在如本文考虑的高严格性条件下将是不稳定的)。例如,可以通过在等同于在42℃于50%甲酰胺、5×denhart溶液、5×sspe、0.2%sds的条件杂交,随后在65℃于0.1×sspe和0.1%sds中洗涤来提供高严格性条件。另一个高严格性条件是在等同于在65℃于含有0.1%(w:v)sds的5×ssc中杂交并且在65℃于含有0.1%sds的0.1×ssc中洗涤的条件杂交。其他高严格性杂交条件以及中度严格条件在上文引用的参考文献中被描述。
“密码子优化的”指的是将编码蛋白质的多核苷酸的密码子改变为特定生物体中优先使用的那些密码子,使得所编码的蛋白质在感兴趣的生物体中被更有效地表达。尽管遗传密码是简并的,即大多数氨基酸由被称为“同义(synonyms)”或“同义(synonymous)”密码子的若干个密码子表示,但熟知的是,特定生物体的密码子使用是非随机的并且对于特定的密码子三联体是有偏好的。就给定的基因、具有共同功能或祖先起源的基因、高度表达的蛋白质相对于低拷贝数蛋白质以及生物体基因组的聚集蛋白质编码区域而言,这种密码子使用偏好可能更高。在一些实施方案中,编码t7rna聚合酶的多核苷酸可以被密码子优化用于从选择用于表达的宿主生物体的最佳产生。
“控制序列”在本文中指包括对本申请的多核苷酸和/或多肽的表达必要或有利的所有组分。每一个控制序列对于编码多肽的核酸序列可以是原生的(native)或外来的。这样的控制序列包括,但不限于,前导序列、多腺苷酸化序列、前肽序列、启动子序列、信号肽序列、起始序列和转录终止子。在最小程度上,控制序列包括启动子和转录及翻译终止信号。控制序列可以与接头一起被提供,以用于引入促进控制序列与编码多肽的核酸序列的编码区域的连接的特定限制性位点的目的。
“可操作地连接”在本文被定义为其中控制序列被适当地放置(即,以功能关系)在相对于感兴趣的多核苷酸的位置处,使得控制序列指导或调节感兴趣的多核苷酸和/或多肽的表达的配置。
“启动子序列”指的是被宿主细胞识别用于感兴趣的多核苷酸诸如编码序列的表达的核酸序列。启动子序列包含介导感兴趣的多核苷酸的表达的转录控制序列。启动子可以是在选择的宿主细胞中显示出转录活性的任何核酸序列,包括突变、截短的和杂合启动子,并且可以从编码与宿主细胞同源或异源的细胞外或细胞内多肽的基因来获得。
“合适的反应条件”指的是在酶促转化反应溶液中的那些条件(例如,酶载量、底物载量、温度、ph、缓冲液、助溶剂等的范围),在上述条件下本申请的t7rna聚合酶多肽能够将底物转化为期望的产物化合物。
在酶促转化反应过程的情况下,“底物”指的是由t7rna聚合酶多肽作用的化合物或分子。
在酶促转化过程的情况下,“产物”指的是从t7rna聚合酶多肽对底物的作用产生的化合物或分子。
如本文使用的术语“培养”指的是微生物细胞群体在任何合适的条件(例如,使用液体、凝胶或固体培养基)下的生长。
重组多肽可以使用本领域已知的任何合适的方法来产生。编码感兴趣的野生型多肽的基因可以被克隆在载体诸如质粒中,并且在期望的宿主诸如大肠杆菌、酿酒酵母(s.cerevisiae)等中表达。重组多肽的变体可以通过本领域已知的多种方法来产生。事实上,存在本领域技术人员熟知的多种不同的诱变技术。此外,诱变试剂盒也从许多商业分子生物学供应商可获得。在定义的氨基酸处(定点)产生特定取代、在基因的局部区域中(区域特异性)产生特定或随机突变、或在整个基因内产生随机诱变(例如,饱和诱变)的方法是可获得的。本领域技术人员已知许多合适的方法来产生酶变体,包括但不限于使用pcr对单链dna或双链dna的定点诱变、盒式诱变、基因合成、易错pcr、重排和化学饱和诱变,或本领域已知的任何其他合适的方法。用于dna和蛋白质工程化的方法的非限制性实例在以下专利中提供:美国专利第6,117,679号;美国专利第6,420,175号;美国专利第6,376,246号;美国专利第6,586,182号;美国专利第7,747,391号;美国专利第7,747,393号;美国专利第7,783,428号;和美国专利第8,383,346号。在变体产生之后,可以针对任何期望的性质(例如,高或增加的活性或低或降低的活性、增加的热活性、增加的热稳定性和/或酸性ph稳定性等)对它们进行筛选。
在一些实施方案中,可使用“重组t7rna聚合酶多肽”(本文中也称为“工程化t7rna聚合酶多肽”、“变体t7rna聚合酶”和“t7rna聚合酶变体”)。
如本文使用的,“载体”是用于将dna序列引入至细胞中的dna构建体。在一些实施方案中,载体是被可操作地连接至能够实现dna序列中编码的多肽在合适宿主中的表达的合适的控制序列的表达载体。在一些实施方案中,“表达载体”具有可操作地连接至dna序列(例如,转基因)以驱动在宿主细胞中的表达的启动子序列,并且在一些实施方案中,还包含转录终止子序列。
如本文使用的,术语“表达”包括参与多肽产生的任何步骤,包括但不限于,转录、转录后修饰、翻译和翻译后修饰。在一些实施方案中,该术语还涵盖多肽从细胞中的分泌。
如本文使用的,术语“产生”指的是蛋白质和/或其他化合物从细胞的产生。意图是,该术语涵盖参与多肽产生的任何步骤,包括但不限于,转录、转录后修饰、翻译和翻译后修饰。在一些实施方案中,该术语还涵盖多肽从细胞中的分泌。
如本文使用的,如果氨基酸或核苷酸序列(例如,启动子序列、信号肽、终止子序列等)与可操作地与其连接的另一个序列在自然界中未缔合,则这两个序列是异源的。
如本文使用的,术语“宿主细胞”和“宿主菌株”指的是用于包含本文提供的dna(例如,编码t7rna聚合酶变体的多核苷酸)的表达载体的合适的宿主。在一些实施方案中,宿主细胞是已经用使用如本领域已知的重组dna技术构建的载体转化或转染的原核细胞或真核细胞。
术语“类似物(analogue)”当用于提及多肽时,意指与参考多肽具有高于70%序列同一性但低于100%序列同一性(例如,高于75%、78%、80%、83%、85%、88%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%序列同一性)的多肽。在一些实施方案中,类似物意指包含一个或更多个非天然存在的氨基酸残基以及天然存在的氨基酸的多肽,所述一个或更多个非天然存在的氨基酸残基包括但不限于高精氨酸、鸟氨酸和正缬氨酸。在一些实施方案中,类似物还包含一个或更多个d-氨基酸残基和两个或更多个氨基酸残基之间的非肽键。
术语“有效量”意指足以产生期望的结果的量。本领域一般技术人员可以通过使用常规实验确定有效量是多少。
术语“分离的”和“纯化的”用于指从与其天然缔合的至少一种其他组分取出的分子(例如,分离的核酸、多肽等)或其他组分。术语“纯化的”不要求绝对纯度,而是意图作为相对定义。
如本文使用的,“组合物”和“制剂”涵盖包含至少一种本发明的工程化t7rna聚合酶的产物,该产物意图用于任何合适的用途(例如,研究、诊断等)。
术语“转录”用于指通过rna聚合酶的作用将dna模板的一部分拷贝成rna的过程。
术语“dna模板”用于指双链或单链dna分子,其包含启动子序列和编码转录的rna产物的序列。
术语“启动子”用于指被rna聚合酶识别为转录起始位点的dna序列。启动子募集rna聚合酶,并且在t7rna聚合酶的情况下,决定转录的起始位点。
术语“rna聚合酶”用于指dna指导的rna聚合酶,该dna指导的rna聚合酶通过将核苷酸三磷酸逐步掺入正在生长的rna聚合物中来将dna模板拷贝成rna多核苷酸。
术语“信使rna”和“mrna”用于指编码蛋白质的rna分子。该蛋白质通过翻译作用被解码。
术语“7-甲基鸟苷帽”、“7meg”、“5’端帽(five-primecap)”和“5’帽”用于指存在于真核mrna的5’端的特定修饰的核苷酸结构。7-甲基鸟苷帽结构通过5’至5’三磷酸酯键附接至mrna中的第一个核苷酸。在体内,这种帽结构通过多种酶的连续活动被添加至新生mrna的5’端。在体外,帽可以通过使用帽类似物在通过rna聚合酶的转录起始时直接被掺入。
术语“帽类似物”指的是含有5’-5’二、三或四磷酸酯键的二核苷酸。二核苷酸的一端终止于鸟苷或被取代的鸟苷残基;从这一端开始,rna聚合酶将通过从3’羟基延伸来起始转录。二核苷酸的另一端是鸟苷,该鸟苷模拟真核帽结构,并且通常将具有7-甲基取代、7-苄基取代或7-乙基取代和/或7-氨基甲基取代或7-氨基乙基取代。在一些情况下,该核苷酸还在3’羟基基团处被甲氧基取代,以防止转录从分子的帽端起始。
术语“arca”和“抗逆转帽类似物”指的是帽类似物的化学修饰形式,该帽类似物的化学修饰形式被设计成通过确保帽类似物以正确的方向正确地掺入转录物中,从而最大限度地提高体外翻译的效率。这些类似物可用于增强翻译。在一些实施方案中,可使用本领域中已知的arca(例如,peng等人,org.lett.,4:161-164[2002])。
术语“核糖开关(riboswitch)”用于指在配体存在下裂解自身或裂解另一种rna的自催化rna酶。
术语“保真性”用于指rna聚合酶在将dna模板转录或拷贝成rna多核苷酸的准确性。不准确的转录可以导致单核苷酸多态性(snp)或插入缺失(indel)。
术语“单核苷酸多态性”或“snp”指的是多核苷酸中的单个位置处存在的核苷酸的变化。在转录的情况下,snp可以是由rna聚合酶在dna模板上的位置处错误掺入了非互补核糖核苷酸(a、c、g或u)导致的。
术语“插入缺失”用于指一个或更多个多核苷酸的插入或缺失。在通过rna聚合酶转录的情况下,插入缺失误差可以是由在dna模板上的位置处添加一个或更多个额外的核糖核苷酸或未能掺入一个或更多个核苷酸导致的。
术语“选择性”用于指在催化反应期间,与另一种底物相比,酶对一种底物具有更高活性的特性。在共转录加帽的情况下,相比gtp,rna聚合酶对帽类似物可以具有高或低的选择性。
术语“无机焦磷酸酶”用于指将无机焦磷酸盐降解成正磷酸盐的酶。
工程化t7rna聚合酶活性:
在一些实施方案中,本文提供的表现出改进的性质的工程化t7rna聚合酶与seqidno:4和/或seqidno:15具有至少约85%、至少约88%、至少约90%、至少约91%、至少约92%、至少约93%、至少约94%、至少约95%、至少约96%、至少约97%、至少约98%、至少约99%或至少约100%的氨基酸序列同一性,并且与seqidno:4和/或seqidno:15相比在一个或更多个氨基酸位置处具有氨基酸残基差异(诸如与seqidno:4和/或seqidno:15或者与seqidno:4和/或seqidno:15具有至少85%、至少88%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或更高氨基酸序列同一性的序列相比,在1个、2个、3个、4个、5个、6个、7个、8个、9个、10个、11个、12个、14个、15个、20个或更多个氨基酸位置处具有氨基酸残基差异)。在一些实施方案中,与seqidno:4相比在一个或更多个位置处的残基差异将包括至少1个、2个、3个、4个、5个、6个、7个、8个、9个、10个或更多个保守氨基酸取代。在一些实施方案中,工程化t7rna聚合酶多肽是表5.3、表5.5和/或表5.6中列出的多肽。在一些实施方案中,工程化t7rna聚合酶多肽选自seqidno:4、15、17、19、21、23、25、27、29、31、33、35、37和/或39。在一些实施方案中,与seqidno:15相比在一个或更多个位置处的残基差异将包括至少1个、2个、3个、4个、5个、6个、7个、8个、9个、10个或更多个保守氨基酸取代。在一些实施方案中,工程化t7rna聚合酶多肽是表5.4中列出的多肽。在一些实施方案中,工程化t7rna聚合酶多肽选自seqidno:4、15、17、19、21、23、25、27、29、31、33、35、37和/或39。
在一些实施方案中,工程化t7rna聚合酶多肽包含由本发明涵盖的工程化t7rna聚合酶多肽的功能片段。功能片段具有其所源自的工程化t7rna聚合酶多肽(即,亲本工程化t7rna聚合酶)的活性的至少80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%。功能片段包含工程化t7rna聚合酶的亲本序列的至少80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%且甚至99%。在一些实施方案中,功能片段被截短了少于5个、少于10个、少于15个、少于20个、少于25个、少于30个、少于35个、少于40个、少于45个和少于50个氨基酸。
编码工程化多肽的多核苷酸、表达载体和宿主细胞:
本发明提供了编码本文描述的工程化t7rna聚合酶多肽的多核苷酸。在一些实施方案中,多核苷酸被可操作地连接至控制基因表达的一个或更多个异源调节序列,以创建能够表达多肽的重组多核苷酸。包含编码工程化t7rna聚合酶多肽的异源多核苷酸的表达构建体可以被引入适当的宿主细胞中以表达对应的t7rna聚合酶多肽。
如对技术人员将是明显的,蛋白质序列的可得性和对应于多种氨基酸的密码子的知识提供了能够编码目标多肽的所有多核苷酸的描述。遗传密码的简并性,其中相同氨基酸由可选择的或同义的密码子编码,允许制备极大数目的核酸,所有这些核酸编码工程化t7rna聚合酶多肽。因此,知道了特定的氨基酸序列后,本领域技术人员可以以不改变蛋白质的氨基酸序列的方式通过简单改变一个或更多个密码子的序列来制备任何数目的不同核酸。在这方面,本发明具体地考虑了可以通过基于可能的密码子选择来选择组合来制备以编码本文描述的多肽的多核苷酸的每一种可能的改变,并且对于本文描述的任何多肽,包括表5.3、表5.4、表5.5和/或表5.6中提供的变体,所有此类改变被认为具体地公开。
在多种实施方案中,密码子被优选地选择为符合在其中产生蛋白质的宿主细胞。例如,细菌中使用的优选的密码子被用于在细菌中的表达。因此,编码工程化t7rna聚合酶多肽的密码子优化的多核苷酸在全长编码区域的约40%、50%、60%、70%、80%或大于90%的密码子位置处包含优选的密码子。
在一些实施方案中,如上文描述的,多核苷酸编码具有t7rna聚合酶活性的工程化多肽,其具有本文公开的性质,其中所述多肽包含这样的氨基酸序列:与选自seqidno:4和seqidno:15的参考序列或者表5.3、表5.4、表5.5和/或表5.6中公开的任何变体的氨基酸序列具有至少80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更高同一性,以及与参考多肽seqidno:17、19、21、23、25、27、29、31、33、35、37和/或39或者表5.3、表5.4、表5.5和/或表5.6中公开的任何变体的氨基酸序列相比具有一个或更多个残基差异(例如1个、2个、3个、4个、5个、6个、7个、8个、9个、10个或更多个氨基酸残基位置)。在一些实施方案中,参考序列选自seqidno:4和/或seqidno:15。在一些实施方案中,多核苷酸编码具有t7rna聚合酶活性的工程化多肽,其具有本文公开的性质,其中所述多肽包含这样的氨基酸序列:当与seqidno:4和/或seqidno:15的多肽最佳比对时,与参考序列seqidno:4和/或seqidno:15具有至少80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更高序列同一性,以及与seqidno:4和/或seqidno:15相比,在选自表5.3、表5.4、表5.5和/或表5.6中提供的那些的残基位置处具有一个或更多个残基差异。
在一些实施方案中,编码工程化t7rna聚合酶多肽的多核苷酸包含选自编码seqidno:4、15、17、19、21、23、25、27、29、31、33、35、37和39的多核苷酸序列的多核苷酸序列。在一些实施方案中,编码工程化t7rna聚合酶多肽的多核苷酸与seqidno:3、14、16、18、20、22、24、26、28、30、32、34、36和38具有至少80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、93%、95%、96%、97%、98%、99%的核苷酸残基同一性。在一些实施方案中,编码工程化t7rna聚合酶多肽的多核苷酸包含选自seqidno:3、14、16、18、20、22、24、26、28、30、32、34、36和38的多核苷酸序列。
在一些实施方案中,多核苷酸能够在高度严格条件下与选自seqidno:3和/或seqidno:14的参考多核苷酸序列、或其互补序列、或编码本文提供的任何变体t7rna聚合酶多肽的多核苷酸序列杂交。在一些实施方案中,能够在高度严格条件下杂交的多核苷酸编码t7rna聚合酶多肽,该t7rna聚合酶多肽包含与seqidno:4和/或seqidno:15相比在选自表5.3、表5.4、表5.5和/或表5.6中列出的任何位置的残基位置处具有一个或更多个残基差异的氨基酸序列。
在一些实施方案中,编码本文提供的任何工程化t7rna聚合酶多肽的分离的多核苷酸以多种方式被操作,以提供多肽的表达。在一些实施方案中,编码多肽的多核苷酸作为表达载体来提供,其中存在一个或更多个控制序列来调节多核苷酸和/或多肽的表达。取决于表达载体,在分离的多核苷酸插入载体之前对分离的多核苷酸的操作可以是期望的或必要的。利用重组dna方法修饰多核苷酸和核酸序列的技术是本领域熟知的。
在一些实施方案中,控制序列包括,除了其他序列以外,启动子、前导序列、多腺苷酸化序列、前肽序列、信号肽序列和转录终止子。如本领域已知的,合适的启动子可以基于使用的宿主细胞来选择。用于丝状真菌宿主细胞的示例性启动子包括从以下的基因获得的启动子:米曲霉(aspergillusoryzae)taka淀粉酶、米黑根毛霉(rhizomucormiehei)天冬氨酸蛋白酶、黑曲霉(aspergillusniger)中性α-淀粉酶、黑曲霉酸稳定型α-淀粉酶、黑曲霉或泡盛曲霉(aspergillusawamori)葡糖淀粉酶(glaa)、米黑根毛霉脂肪酶、米曲霉碱性蛋白酶、米曲霉磷酸丙糖异构酶、构巢曲霉(aspergillusnidulans)乙酰胺酶和尖孢镰刀菌(fusariumoxysporum)胰蛋白酶样蛋白酶(参见,例如wo96/00787),以及na2-tpi启动子(来自黑曲霉中性α-淀粉酶基因和米曲霉磷酸丙糖异构酶基因的启动子的杂合体),和其突变启动子、截短启动子和杂合启动子。示例性酵母细胞启动子可以来自以下的基因:酿酒酵母(saccharomycescerevisiae)烯醇酶(eno-1)、酿酒酵母半乳糖激酶(gal1)、酿酒酵母乙醇脱氢酶/甘油醛-3-磷酸脱氢酶(adh2/gap)、和酿酒酵母3-磷酸甘油酸激酶。用于酵母宿主细胞的其他有用的启动子是本领域已知的(参见例如,romanos等人,yeast8:423-488[1992])。用于哺乳动物细胞的示例性启动子包括但不限于来自以下的那些启动子:巨细胞病毒(cmv)、猿猴空泡病毒40(sv40)、智人(homosapiens)磷酸甘油酸激酶、β肌动蛋白、延伸因子-1a或甘油醛-3-磷酸脱氢酶或原鸡(gallusgallus)β-肌动蛋白。
在一些实施方案中,控制序列是合适的转录终止子序列,转录终止子序列是由宿主细胞识别以终止转录的序列。终止子序列被可操作地连接至编码多肽的核酸序列的3’末端。在选择的宿主细胞中有功能的任何终止子可用于本发明中。例如,用于丝状真菌宿主细胞的示例性转录终止子可以从以下的基因获得:米曲霉taka淀粉酶、黑曲霉葡糖淀粉酶、构巢曲霉邻氨基苯甲酸合成酶、黑曲霉α-葡糖苷酶、和尖孢镰刀菌胰蛋白酶样蛋白酶。用于酵母宿主细胞的示例性终止子可以从以下的基因获得:酿酒酵母烯醇酶、酿酒酵母细胞色素c(cyc1)、和酿酒酵母甘油醛-3-磷酸脱氢酶。用于酵母宿主细胞的其他有用的终止子是本领域已知的(参见例如,romanos等人,同上)。用于哺乳动物细胞的示例性终止子包括但不限于来自巨细胞病毒(cmv)、猿猴空泡病毒40(sv40)或智人生长激素的那些终止子。
在一些实施方案中,控制序列是合适的前导序列,前导序列是一种对被宿主细胞翻译很重要的mrna非翻译区域。前导序列被可操作地连接至编码多肽的核酸序列的5’末端。可以使用在选择的宿主细胞中有功能的任何前导序列。用于丝状真菌宿主细胞的示例性前导序列从以下的基因获得:米曲霉taka淀粉酶和构巢曲霉磷酸丙糖异构酶。用于酵母宿主细胞的合适的前导序列包括,但不限于,从以下的基因获得的那些前导序列:酿酒酵母烯醇酶(eno-1)、酿酒酵母3-磷酸甘油酸激酶、酿酒酵母α-因子、和酿酒酵母乙醇脱氢酶/甘油醛-3-磷酸脱氢酶(adh2/gap)。
控制序列也可以是多腺苷酸化序列,多腺苷酸化序列是一种可操作地连接至核酸序列的3’末端的序列,并且其在转录时,被宿主细胞识别为将多腺苷残基添加至转录的mrna的信号。在选择的宿主细胞中有功能的任何多腺苷酸化序列可以用于本发明中。用于丝状真菌宿主细胞的示例性多腺苷酸化序列包括,但不限于来自以下的基因的那些多腺苷酸化序列:米曲霉taka淀粉酶、黑曲霉葡糖淀粉酶、构巢曲霉邻氨基苯甲酸合成酶、尖孢镰刀菌胰蛋白酶样蛋白酶和黑曲霉α-葡糖苷酶。用于酵母宿主细胞的有用的多腺苷酸化序列也是本领域已知的(参见例如,guo和sherman,mol.cell.bio.,15:5983-5990[1995])。
在一些实施方案中,控制序列是信号肽编码区域,其编码连接至多肽的氨基末端并且将编码的多肽引导至细胞的分泌途径中的氨基酸序列。核酸序列的编码序列的5’端可以固有地包含信号肽编码区域,其符合翻译阅读框地(intranslationreadingframe)与编码分泌的多肽的编码区域的区段天然地连接。可选择地,编码序列的5’端可以包含对编码序列而言外来的信号肽编码区域。将表达的多肽引导至选择的宿主细胞的分泌途径中的任何信号肽编码区域可用于本文提供的工程化t7rna聚合酶多肽的表达。对于丝状真菌宿主细胞有效的信号肽编码区域包括但不限于从以下的基因获得的信号肽编码区域:米曲霉taka淀粉酶、黑曲霉中性淀粉酶、黑曲霉葡糖淀粉酶、米黑根毛霉天冬氨酸蛋白酶、特异腐质霉(humicolainsolens)纤维素酶和柔毛腐质霉(humicolalanuginosa)脂肪酶。用于酵母宿主细胞的有用的信号肽包括但不限于来自以下的基因的那些信号肽:酿酒酵母α因子和酿酒酵母转化酶。用于哺乳动物宿主细胞的有用的信号肽包括但不限于来自免疫球蛋白γ(igg)的基因的那些信号肽。
在一些实施方案中,控制序列是前肽编码区域,其编码定位在多肽的氨基末端处的氨基酸序列。产生的多肽被称为“前酶(proenzyme)”、“前多肽(propolypeptide)”或在一些情况下被称为“酶原(zymogen)”。前多肽可以通过前肽从前多肽的催化或自催化裂解被转化为成熟活性多肽。
在另一个方面中,本发明还提供了重组表达载体,该重组表达载体包含编码工程化t7rna聚合酶多肽的多核苷酸和取决于多核苷酸将被引入的宿主的类型的一个或更多个表达调节区域,诸如启动子和终止子、复制起点等。在一些实施方案中,将上文描述的多种核酸和控制序列连接在一起以产生重组表达载体,其包含一个或更多个方便的限制位点以允许编码变体t7rna聚合酶多肽的核酸序列在此类位点处插入或取代。可选择地,本发明的多核苷酸序列通过将多核苷酸序列或包含多核苷酸序列的核酸构建体插入到用于表达的适当的载体来表达。在创建表达载体时,编码序列位于载体中使得编码序列与用于表达的适当的控制序列可操作地连接。
重组表达载体可以是任何载体(例如,质粒或病毒),其可以方便地经受重组dna程序并且可以导致变体t7rna聚合酶多核苷酸序列的表达。载体的选择通常将取决于载体与待引入载体的宿主细胞的相容性。载体可以是线性质粒或闭合的环状质粒。
在一些实施方案中,表达载体是自主复制载体(即,作为染色体外的实体存在的载体,其复制独立于染色体复制,诸如质粒、染色体外元件、微型染色体或人工染色体)。载体可以包含用于确保自我复制的任何工具(means)。在一些可选择的实施方案中,载体可以是当被引入到宿主细胞中时,被整合到基因组中并且与其被整合进的染色体一起复制的载体。此外,可以使用单一载体或质粒或者两种或更多种载体或质粒或者转座子,所述两种或更多种载体或质粒一起包含待引入到宿主细胞的基因组中的总dna。
在一些实施方案中,表达载体优选地包含一个或更多个可选择的标志物,其允许容易选择经转化的细胞。“可选择的标志物”是其产物提供杀生物剂或病毒耐受性、对重金属的耐受性、对营养缺陷型的原养型等的基因。用于酵母宿主细胞的合适的标志物包括但不限于ade2、his3、leu2、lys2、met3、trp1和ura3。用于在丝状真菌宿主细胞中使用的可选择的标志物包括但不限于amds(乙酰胺酶)、argb(鸟氨酸氨甲酰转移酶)、bar(膦丝菌素乙酰转移酶)、hph(潮霉素磷酸转移酶)、niad(硝酸还原酶)、pyrg(乳清酸核苷-5’-磷酸脱羧酶)、sc(硫酸腺苷转移酶(sulfateadenyltransferase))和trpc(邻氨基苯甲酸合成酶),以及其等同物。在另一个方面中,本发明提供了一种宿主细胞,所述宿主细胞包含编码本申请的至少一种工程化t7rna聚合酶多肽的多核苷酸,所述多核苷酸被可操作地连接至一个或更多个控制序列用于在宿主细胞中表达工程化t7rna聚合酶。用于表达由本发明的表达载体编码的多肽的宿主细胞在本领域中是众所周知的,并且包括但不限于真菌细胞,诸如酵母细胞(例如,酿酒酵母和巴斯德毕赤酵母(pichiapastoris)[例如,atcc保藏号201178]);昆虫细胞(例如,果蝇属s2细胞和斜纹夜蛾属sf9细胞)、植物细胞、动物细胞(例如,cho、cos和bhk)和人类细胞(例如,hek293t、人类成纤维细胞、thp-1、jurkat和bowes黑素瘤细胞系)。
因此,在另一个方面中,本发明提供了用于产生工程化t7rna聚合酶多肽的方法,其中所述方法包括将能够表达编码工程化t7rna聚合酶多肽的多核苷酸的宿主细胞在适合该多肽表达的条件下培养。在一些实施方案中,所述方法还包括分离和/或纯化如本文描述的t7rna聚合酶多肽的步骤。
用于上文描述的宿主细胞的适当的培养基和生长条件是本领域熟知的。用于表达t7rna聚合酶多肽的多核苷酸可以通过本领域已知的多种方法被引入到细胞中。技术包括,尤其是,电穿孔、生物颗粒轰击、脂质体介导的转染、氯化钙转染和原生质体融合。
具有本文公开的性质的工程化t7rna聚合酶可以通过使编码天然存在的或工程化的t7rna聚合酶多肽的多核苷酸经受本领域中已知的和如本文描述的诱变和/或定向进化方法来获得。示例性定向进化技术是诱变和/或dna重排(参见例如,stemmer,proc.natl.acad.sci.usa91:10747-10751[1994];wo95/22625;wo97/0078;wo97/35966;wo98/27230;wo00/42651;wo01/75767和美国专利6,537,746)。可以使用的其他定向进化程序包括,尤其是,交错延伸过程(step)、体外重组(参见例如,zhao等人,nat.biotechnol.,16:258–261[1998])、诱变pcr(参见例如,caldwell等人,pcrmethodsappl.,3:s136-s140[1994])和盒式诱变(参见如,black等人,proc.natl.acad.sci.usa93:3525-3529[1996])。
例如,诱变和定向进化方法可以容易地应用于多核苷酸,以产生可以被表达、筛选和测定的变体文库。诱变和定向进化方法是本领域熟知的(参见例如,美国专利第5,605,793号、第5,811,238号、第5,830,721号、第5,834,252号、第5,837,458号、第5,928,905号、第6,096,548号、第6,117,679号、第6,132,970号、第6,165,793号、第6,180,406号、第6,251,674号、第6,277,638号、第6,287,861号、第6,287,862号、第6,291,242号、第6,297,053号、第6,303,344号、第6,309,883号、第6,319,713号、第6,319,714号、第6,323,030号、第6,326,204号、第6,335,160号、第6,335,198号、第6,344,356号、第6,352,859号、第6,355,484号、第6,358,740号、第6,358,742号、第6,365,377号、第6,365,408号、第6,368,861号、第6,372,497号、第6,376,246号、第6,379,964号、第6,387,702号、第6,391,552号、第6,391,640号、第6,395,547号、第6,406,855号、第6,406,910号、第6,413,745号、第6,413,774号、第6,420,175号、第6,423,542号、第6,426,224号、第6,436,675号、第6,444,468号、第6,455,253号、第6,479,652号、第6,482,647号、第6,489,146号、第6,506,602号、第6,506,603号、第6,519,065号、第6,521,453号、第6,528,311号、第6,537,746号、第6,573,098号、第6,576,467号、第6,579,678号、第6,586,182号、第6,602,986号、第6,613,514号、第6,653,072号、第6,716,631号、第6,777,218号、第6,917,882号、第6,946,296号、第6,961,664号、第6,995,017号、第7,024,312号、第7,058,515号、第7,105,297号、第7,148,054号、第7,288,375号、第7,421,347号、第7,430,477号、第7,534,564号、第7,620,500号、第7,620,502号、第7,629,170号、第7,702,464号、第7,747,391号、第7,747,393号、第7,751,986号、第7,776,598号、第7,783,428号、第7,795,030号、第7,853,410号、第7,868,138号、第7,873,477号、第7,873,499号、第7,904,249号、第7,957,912号、第8,014,961号、第8,029,988号、第8,058,001号、第8,076,138号、第8,018,150号、第8,170,806号、第8,377,681号、第8,383,346号、第8,457,903号、第8,504,498号、第8,589,085号、第8,762,066号、第8,849,575号、第8,876,066号、第8,768,871号、第9,593,326号、第9,665,694号,以及所有相关的美国和非美国对应专利(counterpart);ling等人,anal.biochem.,254(2):157-78[1997];dale等人,meth.mol.biol.,57:369-74[1996];smith,ann.rev.genet.,19:423-462[1985];botstein等人,science,229:1193-1201[1985];carter,biochem.j.,237:1-7[1986];kramer等人,cell,38:879-887[1984];wells等人,gene,34:315-323[1985];minshull等人,curr.op.chem.biol.,3:284-290[1999];christians等人,nat.biotechnol.,17:259-264[1999];crameri等人,nature,391:288-291[1998];crameri,等人,nat.biotechnol.,15:436-438[1997];zhang等人,proc.nat.acad.sci.u.s.a.,94:4504-4509[1997];crameri等人,nat.biotechnol.,14:315-319[1996];stemmer,nature,370:389-391[1994];stemmer,proc.nat.acad.sci.usa,91:10747-10751[1994];美国专利申请公布第2008/0220990号、us2009/0312196、us2014/0005057、us2014/0214391、us2014/0221216、us2015/0050658、us2015/0133307、us2015/0134315和所有相关的美国和非美国对应申请;wo95/22625、wo97/0078、wo97/35966、wo98/27230、wo00/42651、wo01/75767和wo2009/152336;其全部通过引用并入本文)。
在一些实施方案中,诱变处理后获得的酶变体通过使酶变体经受指定的温度(或其他测定条件),并且测量热处理或其他测定条件之后剩余的酶活性的量来筛选。然后包含编码t7rna聚合酶多肽的多核苷酸的dna从宿主细胞中分离,被测序以鉴定核苷酸序列变化(如果有的话),并且用于在不同或相同的宿主细胞中表达酶。测量来自表达文库的酶活性可以使用本领域已知的任何合适的方法(例如,标准生物化学技术,诸如hplc分析)来进行。
对于已知序列的工程化多肽,编码酶的多核苷酸可以根据已知的合成方法通过标准的固相方法来制备。在一些实施方案中,多达约100个碱基的片段可以被单独地合成、然后被连接(例如,通过酶连接方法或化学连接方法或聚合酶介导的方法)以形成任何期望的连续序列。例如,本文公开的多核苷酸和寡核苷酸可以使用经典的亚磷酰胺方法(参见例如,beaucage等人,tetra.lett.,22:1859-69[1981]和matthes等人,emboj.,3:801-05[1984])通过化学合成来制备,如通常在自动合成方法中所实践的。根据亚磷酰胺方法,寡核苷酸被合成(例如,在自动的dna合成仪中)、纯化、退火、连接并且克隆入适当的载体。
因此,在一些实施方案中,用于制备工程化t7rna聚合酶多肽的方法可以包括:(a)合成编码多肽的多核苷酸,所述多肽包含选自以下的氨基酸序列:表5.3、表5.4、表5.5和/或表5.6中提供的任何变体的氨基酸序列,以及seqidno:15、17、19、21、23、25、27、29、31、33、35、37和39,以及(b)表达由该多核苷酸编码的t7rna聚合酶多肽。在所述方法的一些实施方案中,由该多核苷酸编码的氨基酸序列可以任选地具有一个或若干个(例如,多达3个、4个、5个或多达10个)氨基酸残基缺失、插入和/或取代。在一些实施方案中,氨基酸序列任选地具有1个-2个、1个-3个、1个-4个、1个-5个、1个-6个、1个-7个、1个-8个、1个-9个、1个-10个、1个-15个、1个-20个、1个-21个、1个-22个、1个-23个、1个-24个、1个-25个、1个-30个、1个-35个、1个-40个、1个-45个或1个-50个氨基酸残基缺失、插入和/或取代。在一些实施方案中,氨基酸序列任选地具有1个、2个、3个、4个、5个、6个、7个、8个、9个、10个、11个、12个、13个、14个、15个、16个、17个、18个、19个、20个、21个、22个、23个、24个、25个、30个、30个、35个、40个、45个或50个氨基酸残基缺失、插入和/或取代。在一些实施方案中,氨基酸序列任选地具有1个、2个、3个、4个、5个、6个、7个、8个、9个、10个、11个、12个、13个、14个、15个、16个、18个、20个、21个、22个、23个、24个或25个氨基酸残基缺失、插入和/或取代。在一些实施方案中,取代可以是保守取代或非保守取代。
可以使用本领域已知的任何合适的测定,包括但不限于本文描述的测定和条件,来评估所表达的工程化t7rna聚合酶多肽的任何期望的改进的性质(例如,活性、选择性、稳定性、酸耐受性、蛋白酶敏感性等)。
在一些实施方案中,使用用于蛋白质纯化的熟知技术中的任一种或更多种,将在宿主细胞中表达的任何工程化t7rna聚合酶多肽从细胞和/或培养基回收,所述用于蛋白质纯化的熟知技术包括尤其是,溶菌酶处理、超声处理、过滤、盐析、超离心和色谱法。
用于分离t7rna聚合酶多肽的色谱技术包括,尤其是,反相色谱法、高效液相色谱法、离子交换色谱法、疏水相互作用色谱法、凝胶电泳和亲和色谱法。用于纯化特定酶的条件部分地取决于因素诸如净电荷、疏水性、亲水性、分子量、分子形状等,并且对本领域技术人员将是明显的。在一些实施方案中,亲和技术可以用于分离改进的变体t7rna聚合酶。在利用亲和色谱法纯化的一些实施方案中,可使用特异性地结合变体t7rna聚合酶多肽的任何抗体。在利用亲和色谱法纯化的一些实施方案中,可使用与共价附接至t7rna聚合酶的聚糖结合的蛋白质。在利用亲和色谱法纯化的仍然其他实施方案中,可使用与t7rna聚合酶活性位点结合的任何小分子。为了产生抗体,多种宿主动物,包括但不限于兔、小鼠、大鼠等,通过注射t7rna聚合酶多肽(例如,t7rna聚合酶变体)或其片段进行免疫。在一些实施方案中,t7rna聚合酶多肽或片段借助于侧链官能团或附接至侧链官能团的接头附接至合适的载体,诸如bsa。
在一些实施方案中,工程化t7rna聚合酶多肽在宿主细胞中通过这样的方法来产生:该方法包括在有利于工程化t7rna聚合酶多肽产生的条件下培养宿主细胞(例如,酿酒酵母、胡萝卜(daucuscarota)、烟草(nicotianatabacum)、智人(h.sapiens)(例如,hek293t)或灰仓鼠(cricetulusgriseus)(例如,cho)),该宿主细胞包含编码本文描述的工程化t7rna聚合酶多肽的多核苷酸序列,并且从细胞和/或培养基回收工程化t7rna聚合酶多肽。
在一些实施方案中,本发明涵盖产生工程化t7rna聚合酶多肽的方法,该方法包括在合适的培养条件下培养重组真核细胞,以允许产生工程化t7rna聚合酶多肽,并且任选地从培养物和/或培养的细菌细胞回收工程化t7rna聚合酶多肽,所述重组真核细胞包含编码工程化t7rna聚合酶多肽的多核苷酸序列,所述工程化t7rna聚合酶多肽当与seqidno:4和/或seqidno:15的氨基酸序列最佳比对时,与参考序列seqidno:4和/或seqidno:15具有至少80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、95%、96%、97%、98%、99%或100%的序列同一性,并且与seqidno:4和/或seqidno:15和/或其组合相比,具有选自表5.3、表5.4、表5.5和/或表5.6中提供的那些的残基差异的一个或更多个氨基酸残基差异。
在一些实施方案中,一旦工程化t7rna聚合酶多肽从重组宿主细胞或细胞培养基回收,它们通过本领域已知的任何合适的方法进一步纯化。在一些另外的实施方案中,纯化的t7rna聚合酶多肽与其他成分和化合物组合以提供适当包含工程化t7rna聚合酶多肽用于不同应用和用途的组合物和制剂(例如,药物组合物)。在一些另外的实施方案中,纯化的t7rna聚合酶多肽或配制的t7rna聚合酶多肽被冻干。
组合物:
本发明提供了多种组合物和格式,包括但不限于下文描述的那些。在一些实施方案中,本发明提供了适合在用于诊断目的的组合物中使用的工程化t7rna聚合酶多肽。
实验
提供以下实施例,包括实验和获得的结果,仅用于说明的目的,而不应被解释为限制本发明。
在下文的实验公开内容中,应用了以下缩写:ppm(百万分率);m(摩尔/升);mm(毫摩尔/升);um和μm(微摩尔/升);nm(纳摩尔/升);mol(摩尔);gm和g(克);mg(毫克);ug和μg(微克);l和l(升);ml和ml(毫升);cm(厘米);mm(毫米);um和μm(微米);sec.(秒);min(s)(分钟);h(s)和hr(s)(小时);u(单位);mw(分子量);rpm(每分钟转数);rcf(相对离心力);℃(摄氏度);cds(编码序列);dna(脱氧核糖核酸);rna(核糖核酸);大肠杆菌w3110(e.coliw3110)(常用实验室大肠杆菌菌株,可得自coligeneticstockcenter[cgsc],newhaven,ct);hplc(高压液相色谱法);mwco(分子量截止值);sds-page(十二烷基硫酸钠聚丙烯酰胺凝胶电泳);t7rnap(t7rna聚合酶);pes(聚醚砜);cfse(羧基萤光素琥珀酰亚胺酯);iptg(异丙基β-d-1-硫代半乳糖苷);pmbs(硫酸多粘菌素b);nadph(烟酰胺腺嘌呤二核苷酸磷酸);gidh(谷氨酸脱氢酶);fiopc(相对于阳性对照的改进倍数);lb(luria肉汤);meoh(甲醇);athensresearch(athensresearchtechnology,athens,ga);neb(newenglandbiolabs,ipswich,ma);iontorrent(iontorrent,gilford,nh);prospec(prospectanytechnogene,eastbrunswick,nj);sigma-aldrich(sigma-aldrich,st.louis,mo);ramscientific(ramscientific,inc.,yonkers,ny);pallcorp.(pall,corp.,pt.washington,ny);millipore(millipore,corp.,billericama);difco(difcolaboratories,bddiagnosticsystems,detroit,mi);moleculardevices(moleculardevices,llc,sunnyvale,ca);kuhner(adolfkuhner,ag,basel,switzerland);axygen(axygen,inc.,unioncity,ca);torontoresearchchemicals(torontoresearchchemicalsinc.,toronto,ontario,canada);cambridgeisotopelaboratories,(cambridgeisotopelaboratories,inc.,tewksbury,ma);appliedbiosystems(appliedbiosystems,lifetechnologies,corp.的一部分,grandisland,ny);agilent(agilenttechnologies,inc.,santaclara,ca);thermoscientific(thermofisherscientific的一部分,waltham,ma);iontorrentngs(iontorrentnextgenerationsequencing);li-cor(li-cor,lincoln,ne);uvp(uvp,upland,ca);biotium(biotium,inc.,fremont,ca);corning(corning,inc.,paloalto,ca);megazyme(megazymeinternational,wicklow,ireland);enzo(enzolifesciences,inc.,farmingdale,ny);gehealthcare(gehealthcarebio-sciences,piscataway,nj);pierce(piercebiotechnology(现在是thermofisherscientific的一部分),rockford,il);li-cor(li-corbiotechnology,lincoln,ne);amicus(amicustherapeutics,cranbury,nj);phenomenex(phenomenex,inc.,torrance,ca);optimal(optimalbiotechgroup,belmont,ca);以及bio-rad(bio-radlaboratories,hercules,ca)。
实施例1
t7rna聚合酶基因获取和表达载体的构建
野生型(wt)t7rna聚合酶(seqidno:2)由噬菌体t7的基因组(seqidno:1)编码。编码t7rna聚合酶的加6-组氨酸标签版本(seqidno:4)的合成基因(seqidno:3)被构建,并且被亚克隆至大肠杆菌(escherichiacoli)表达载体pck100900i中(参见例如,美国专利第7,629,157号和美国专利申请公布2016/0244787,这两篇文献都通过引用特此并入)。将这些质粒构建体转化至源自w3110的大肠杆菌菌株中。使用本领域技术人员通常已知的定向进化技术从这些质粒产生基因变体的文库(参见例如,美国专利第8,383,346号和wo2010/144103,这两篇文献都通过引用特此并入)。本文描述的酶变体中的取代是参考加6his标签的wtt7rna聚合酶(即,seqidno:4)或其变体来指示的,如所指示的。
实施例2
t7rna聚合酶的高通量(htp)表达和纯化
在该实施例中,描述了针对t7rna聚合酶表达和t7rna聚合酶变体的纯化进行的实验。
t7rna聚合酶和变体的高通量(htp)生长
经转化的大肠杆菌细胞通过在含有1%葡萄糖和30μg/ml氯霉素的lb琼脂板上铺板来选择。在37℃孵育过夜之后,将菌落放入96孔浅平底nunctm(thermo-scientific)板的孔中,该板填充有180μl/孔的补充有1%葡萄糖和30μg/ml氯霉素的lb培养基。培养物被允许在振动器(200rpm,30℃,和85%相对湿度;kuhner)中生长过夜持续18-20小时。将过夜生长样品(20μl)转移至costar96孔深板中,该板填充有380μl的补充有30μg/ml氯霉素的terrific肉汤。将板在振动器(250rpm,30℃,和85%相对湿度;kuhner)中孵育120分钟,直到od600达到0.4-0.8之间。然后将细胞用40μl无菌水中的10mmiptg诱导,并且在振动器(250rpm,30℃,和85%相对湿度;kuhner)中孵育过夜持续18-20小时。将细胞沉淀(4000rpm×20min),丢弃上清液,并且将细胞在分析之前在-80℃冷冻。
htp沉淀物的裂解
将细胞沉淀物重悬在400μl的裂解缓冲液(50mm磷酸钠,ph7.5、10mm咪唑、0.1%tween
t7rnap从粗制裂解物的htp纯化
根据制造商的使用说明,使用hispurtmni-nta旋转板(thermofisher),通过金属亲和色谱法从澄清的大肠杆菌裂解物纯化t7rnap。用每孔总计800μl的洗涤缓冲液(50mm磷酸钠,ph7.5、300mmnacl、10mm咪唑、0.1%v/v
洗脱物使用zebatm旋转脱盐板(thermofisher)进行缓冲液交换。简言之,板用每孔375μl的2×t7rnap储存缓冲液(100mmtris.hcl,ph8.0、200mmnacl、2mmdtt、2mmedta、0.2%w/vtritonx-100)平衡两次,并且在4℃以1100rcf离心2min。将脱盐板加载90μl的hispurtmni-nta旋转板洗脱物,并且在4℃以1100rcf离心2min。将来自脱盐板的洗脱物保留并且与等体积的甘油混合,以得到50mmtrishcl,ph7.9、100mmnacl、1mmdtt、1mmedta、0.1%v/vtritonx-100和50%甘油(v/v)的最终储存缓冲液浓度。
使用rna酶预警测定(idt,lifetechnologies)确认纯化的预制物中不存在rna酶污染。t7rnap样品的sds-page分析示出大多数样品没有可检测到的污染条带。
对于使用htp纯化的t7rnap的体外转录反应,将纯化的聚合酶添加至总反应体积的10%的最终浓度(对于来自摇瓶培养物的酶是5%)。
实施例3
t7rna聚合酶的摇瓶表达和纯化
在该实施例中,描述了涉及t7rnap的摇瓶表达和纯化的实验。
摇瓶表达
将如实施例2中描述生长的选择的htp培养物铺板在具有1%葡萄糖和30μg/ml氯霉素的lb琼脂板上,并且在37℃生长过夜。将来自每个培养物的单个菌落转移至6ml的含1%葡萄糖和30μg/ml氯霉素的lb肉汤。培养物在30℃、以250rpm生长18h,并且以约1:10稀释至250ml的含有30μg/ml氯霉素的terrific肉汤中来传代培养至0.2的最终od600。培养物在30℃、以250rpm孵育约3小时,至0.6–0.8的od600,并且然后通过添加1mm最终浓度的iptg进行诱导。诱导的培养物在30℃、以250rpm孵育20h。该孵育期后,将培养物以4000rpm×10min离心。丢弃培养物上清液,并且将沉淀物重悬在35ml裂解缓冲液(50mmnah2po4,ph7.5、500mmnacl、0.1%tween-20、10mm咪唑)中。该细胞悬浮液在冰浴中冷却,并且使用microfluidizer细胞破碎仪(microfluidicsm-110l)裂解。粗制裂解物通过离心(在4℃以16,000rpm持续60min)沉淀,并且然后上清液通过0.2μmpes膜过滤以进一步澄清裂解物。
t7rna聚合酶从摇瓶裂解物的纯化
t7rnap裂解物使用aktastart纯化系统和5mlhistrapff柱(gehealthcare)使用acstephif设置(运行参数在下文中提供)进行纯化。sf洗涤缓冲液包含50mm磷酸钠,ph7.5、500mmnacl、0.1%v/v
通过uv吸收(a280)鉴定单一的五种最浓的3ml级分,并且在10kslide-a-lyzertm透析盒(thermofisher)中在2×t7rnap储存缓冲液(100mmtrishclph7.9、200mmnacl、2mmdtt、2mmedta、0.2%v/vtritonx-100)中透析过夜,进行缓冲液交换持续16小时,随后进行第二次缓冲液交换持续24小时。向经透析的材料添加等体积的甘油。制备物中的酶浓度通过凝胶密度测定和在280nm处的吸光度来测量。
实施例4
转录反应
转录反应用帽类似物α,γ-双(n7-甲基鸟苷)三磷酸(本文中也被称为“m7g(5’)ppp(5’)m7g”、“加帽gtp”或“帽”)进行组装(参见,grudzien等人,rna,10:1479-87[2004])。工程化转录dna模板glms-16a(seqidno:5)包含t7rnap启动子序列,其与炭疽杆菌(bacillusanthracis)glms核糖开关的编码序列(seqidno:6)偶联。在其配体葡糖胺-6-磷酸诱导下,glms核糖开关自我裂解并且从转录物的5’端释放16-merrna核苷酸(seqidno:7),所述转录物的5’端包括帽结构或未加帽的5’磷酸。这种小的16-merrna寡核苷酸裂解产物可通过lc-ms进行离子化和分析,用于区分加帽和未加帽的物质。
反应以每孔总计30ul组装,最终浓度为50mmtrishclph7.9、30mmmgcl2、10mmdtt、6mmatp、6mmctp、6mmutp、4.8mmgtp、1.2mmm7g(5’)ppp(5’)m7g、50ng/μlglms-16a转录模板、1u/μlrnasin抑制剂和6.4mm葡糖胺-6-磷酸,并且包含10%(v/v)(3ul)的来自实施例3的纯化的和脱盐的t7rnap。对于htp筛选,将反应在37℃孵育4h,并且用相等(30μl)体积的40mmedta猝灭。m7g(5’)ppp(5’)m7g和gtp的总和保持在6mm。反应开始时加入葡糖胺-6-磷酸,并且在4小时体外转录期间允许进行核糖开关的裂解。
实施例5
lc-ms活性测定
该实施例描述了针对使用thermoltqms系统分离较小的rna片段开发的lc-ms方法,用于分析较短的加帽的5’三磷酸裂解产物和未加帽的5’三磷酸裂解产物。
7meg加帽的和5’三磷酸未加帽的16-mer裂解产物(seqidno:7)用反相离子配对hplc方法从反应物和mw较高的rna色谱分离(参见,图1),所述反相离子配对hplc方法使用通常用于质谱寡核苷酸解析的c18柱和hfip流动相(参见,表5.1)。
对每种物质使用七种离子,包括多种带负电荷的离子和金属加合物,对加帽和未加帽的16-mer裂解产物进行基峰(base-peak)提取。这七种离子的峰强度用于导出每种加帽和未加帽的16-mer裂解产物的信号。这些离子包括多种带电荷的状态以及钠和钾加合物(参见,表5.2)。在期望值的1质量单位内观察到所有离子。
相对于wtt7rnap的加帽性能(即,“相对于亲本的改进倍数”或“fiop”)通过将每个样品的加帽/未加帽峰强度比率除以针对每个板上存在的wt亲本对照计算的比率来计算(n=6至10)。fiop值用于相对于wtt7rnap或文库亲本对变体性能进行排序,wtt7rnap或文库亲本作为对照被包含在每个板上。通过使用这种相对定量方法,可以消除加帽和未加帽的物质之间固有的离子化差异,以及由hfip流动相的批次和edta浓度引起的信号改变。体外转录反应如实施例4中描述进行,反应时间和m7g(5’)ppp(5’)m7g浓度如下表所示。表5.3、表5.4和表5.5报告了3至6次重复的平均活性改进。表5.6报告了可获得重复信息的变体的平均活性改进。
注意到变体85在lc-ms测定中未加帽的mrna的水平是不可检测的。
实施例6
rna收率的确定
体外转录反应如实施例4中描述进行,但使用萤光素酶模板(seqidno:10)。根据制造商针对quant-itrna测定试剂盒(宽范围,q-33140,thermofisher)的方案,进行从体外转录反应的mrna收率获取。使用为该测定试剂盒提供的mrna标准导出的标准曲线来计算mrna丰度。
实施例7
t7rnap变体转录保真性的确定
聚合酶保真性是基于直接测序大量源自从变体聚合酶转录的mrna的rt-pcr克隆来测量的。体外转录反应如实施例4中描述进行,使用0.5mmm7g(5’)ppp(5’)m7g、5.5mmgtp,并且省略葡糖胺-6-磷酸。m7g(5’)ppp(5’)m7g的包含允许测量在过程相关条件下该帽类似物(如果存在)对wt聚合酶和变体聚合酶的误差率的贡献。1.7kb萤光素酶模板dna(seqidno:8)用作使用野生型t7rnap和变体t7rnap转录的dna模板,以产生全长mrna转录物。rna使用zymornacleanandconcentrator-25试剂盒(zymoresearch)来分离,并且通过用dna-freednaasei试剂盒(ambion/thermofisher)连续两次处理从rna样品除去残余的dna。样品用accuprime逆转录酶(agilent)使用退火至萤光素酶模板上的聚(a)尾的oligo-(dt)25引物(seqidno:40)进行逆转录。rt反应然后经由pcr,使用退火至萤光素酶编码序列的基因特异性引物(seqidno:12、seqidno:13),使用hf缓冲液(newenglandbiolabs),使用
在iontorrentpgm平台(thermofisher)上,使用多重加条形码策略对单个克隆进行挑选和测序。条形码化的读段被解卷积,并且然后针对seqidno:12和seqidno:13之间的1632-bp区域的预期模板序列组装单个克隆的序列。记录包括小插入、缺失(即,插入缺失)和单核苷酸多态性的突变。观察到的大多数突变是取代,尽管也观察到插入和缺失。计算了测序的mrna来源的克隆的每个碱基的突变总数,并且在表7.1和表7.2中报告。基于文献的每个碱基的预期总突变率是由于t7rnap(1×10-4)(huang等人,biochem.,39:11571-11580[2000])、accuscript逆转录酶(6×10-5)(agilent;参见产品文献)和phusiondna聚合酶(1.2×10-5)(20个循环;参见neb产品文献)或整体1.72×10-4导致的误差的总和。大多数变体聚合酶显示出接近针对t7rnap报道的文献值的总误差率,即使在0.5mmm7g(5’)ppp(5’)m7g的存在下。
如果实验中的实际误差率等于表7.1和表7.2中呈现的单独实验中对t7rnap-wt观察到的总误差率,则使用单尾(右侧)二项检验来计算给定数目的测序碱基的观察到的误差数(或更多)的抽样概率。对于大于0.05的p值,给定变体的保真性被认为在该测定中与wtt7rnap无法区分。在表7.1中,观察误差率结果栏中的“+”是小于1.7*10-4,并且“-”是大于1.7*10-4,而二项检验结果栏中的“+”是p>0.05,并且“-”是p<0.05。在表7.2中,观察误差率结果栏中的“+”是小于1.5×10-4,并且“-”是大于1.5×10-4,而二项检验结果栏中的“+”是p>0.05,并且“-”是p<0.05。
尽管已经参考具体的实施方案描述了本发明,可以做出多种改变并且可以替换等同物,以适应特定的情况、材料、物质的组成、方法、方法步骤(processsteporsteps),从而实现本发明的益处,而不偏离所要求保护的范围。
本申请中引用的每一个和每个出版物和专利文件通过引用被并入本文用于在美国的所有目的,如同每个这样的出版物或文件被明确和单独指出通过引用并入本文。出版物和专利文件的引用不被认为是任何此类文件是相关的现有技术的指示,也不构成对其内容或日期的承认。
序列表
<110>科德克希思公司
阿雷克森制药公司
<120>t7rna聚合酶变体
<130>cx8-168usp1
<140>待指定
<141>随同提交
<160>40
<210>1
<211>2649
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>t7rnap野生型
<400>1
atgaacacgattaacatcgctaagaacgacttctctgacatcgaactggctgctatcccg60
ttcaacactctggctgaccattacggtgagcgtttagctcgcgaacagttggcccttgag120
catgagtcttacgagatgggtgaagcacgcttccgcaagatgtttgagcgtcaacttaaa180
gctggtgaggttgcggataacgctgccgccaagcctctcatcactaccctactccctaag240
atgattgcacgcatcaacgactggtttgaggaagtgaaagctaagcgcggcaagcgcccg300
acagccttccagttcctgcaagaaatcaagccggaagccgtagcgtacatcaccattaag360
accactctggcttgcctaaccagtgctgacaatacaaccgttcaggctgtagcaagcgca420
atcggtcgggccattgaggacgaggctcgcttcggtcgtatccgtgaccttgaagctaag480
cacttcaagaaaaacgttgaggaacaactcaacaagcgcgtagggcacgtctacaagaaa540
gcatttatgcaagttgtcgaggctgacatgctctctaagggtctactcggtggcgaggcg600
tggtcttcgtggcataaggaagactctattcatgtaggagtacgctgcatcgagatgctc660
attgagtcaaccggaatggttagcttacaccgccaaaatgctggcgtagtaggtcaagac720
tctgagactatcgaactcgcacctgaatacgctgaggctatcgcaacccgtgcaggtgcg780
ctggctggcatctctccgatgttccaaccttgcgtagttcctcctaagccgtggactggc840
attactggtggtggctattgggctaacggtcgtcgtcctctggcgctggtgcgtactcac900
agtaagaaagcactgatgcgctacgaagacgtttacatgcctgaggtgtacaaagcgatt960
aacattgcgcaaaacaccgcatggaaaatcaacaagaaagtcctagcggtcgccaacgta1020
atcaccaagtggaagcattgtccggtcgaggacatccctgcgattgagcgtgaagaactc1080
ccgatgaaaccggaagacatcgacatgaatcctgaggctctcaccgcgtggaaacgtgct1140
gccgctgctgtgtaccgcaaggacaaggctcgcaagtctcgccgtatcagccttgagttc1200
atgcttgagcaagccaataagtttgctaaccataaggccatctggttcccttacaacatg1260
gactggcgcggtcgtgtttacgctgtgtcaatgttcaacccgcaaggtaacgatatgacc1320
aaaggactgcttacgctggcgaaaggtaaaccaatcggtaaggaaggttactactggctg1380
aaaatccacggtgcaaactgtgcgggtgtcgataaggttccgttccctgagcgcatcaag1440
ttcattgaggaaaaccacgagaacatcatggcttgcgctaagtctccactggagaacact1500
tggtgggctgagcaagattctccgttctgcttccttgcgttctgctttgagtacgctggg1560
gtacagcaccacggcctgagctataactgctcccttccgctggcgtttgacgggtcttgc1620
tctggcatccagcacttctccgcgatgctccgagatgaggtaggtggtcgcgcggttaac1680
ttgcttcctagtgaaaccgttcaggacatctacgggattgttgctaagaaagtcaacgag1740
attctacaagcagacgcaatcaatgggaccgataacgaagtagttaccgtgaccgatgag1800
aacactggtgaaatctctgagaaagtcaagctgggcactaaggcactggctggtcaatgg1860
ctggcttacggtgttactcgcagtgtgactaagcgttcagtcatgacgctggcttacggg1920
tccaaagagttcggcttccgtcaacaagtgctggaagataccattcagccagctattgat1980
tccggcaagggtctgatgttcactcagccgaatcaggctgctggatacatggctaagctg2040
atttgggaatctgtgagcgtgacggtggtagctgcggttgaagcaatgaactggcttaag2100
tctgctgctaagctgctggctgctgaggtcaaagataagaagactggagagattcttcgc2160
aagcgttgcgctgtgcattgggtaactcctgatggtttccctgtgtggcaggaatacaag2220
aagcctattcagacgcgcttgaacctgatgttcctcggtcagttccgcttacagcctacc2280
attaacaccaacaaagatagcgagattgatgcacacaaacaggagtctggtatcgctcct2340
aactttgtacacagccaagacggtagccaccttcgtaagactgtagtgtgggcacacgag2400
aagtacggaatcgaatcttttgcactgattcacgactccttcggtaccattccggctgac2460
gctgcgaacctgttcaaagcagtgcgcgaaactatggttgacacatatgagtcttgtgat2520
gtactggctgatttctacgaccagttcgctgaccagttgcacgagtctcaattggacaaa2580
atgccagcacttccggctaaaggtaacttgaacctccgtgacatcttagagtcggacttc2640
gcgttcgcg2649
<210>2
<211>883
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>t7rnap野生型
<400>2
metasnthrileasnilealalysasnasppheseraspilegluleu
151015
alaalailepropheasnthrleualaasphistyrglygluargleu
202530
alaarggluglnleualaleugluhisglusertyrglumetglyglu
354045
alaargphearglysmetphegluargglnleulysalaglygluval
505560
alaaspasnalaalaalalysproleuilethrthrleuleuprolys
65707580
metilealaargileasnasptrppheglugluvallysalalysarg
859095
glylysargprothralapheglnpheleuglngluilelysproglu
100105110
alavalalatyrilethrilelysthrthrleualacysleuthrser
115120125
alaaspasnthrthrvalglnalavalalaseralaileglyargala
130135140
ilegluaspglualaargpheglyargileargaspleuglualalys
145150155160
hisphelyslysasnvalglugluglnleuasnlysargvalglyhis
165170175
valtyrlyslysalaphemetglnvalvalglualaaspmetleuser
180185190
lysglyleuleuglyglyglualatrpsersertrphislysgluasp
195200205
serilehisvalglyvalargcysileglumetleuilegluserthr
210215220
glymetvalserleuhisargglnasnalaglyvalvalglyglnasp
225230235240
sergluthrilegluleualaproglutyralaglualailealathr
245250255
argalaglyalaleualaglyileserprometpheglnprocysval
260265270
valproprolysprotrpthrglyilethrglyglyglytyrtrpala
275280285
asnglyargargproleualaleuvalargthrhisserlyslysala
290295300
leumetargtyrgluaspvaltyrmetprogluvaltyrlysalaile
305310315320
asnilealaglnasnthralatrplysileasnlyslysvalleuala
325330335
valalaasnvalilethrlystrplyshiscysprovalgluaspile
340345350
proalailegluargglugluleuprometlysprogluaspileasp
355360365
metasnproglualaleuthralatrplysargalaalaalaalaval
370375380
tyrarglysasplysalaarglysserargargileserleugluphe
385390395400
metleugluglnalaasnlysphealaasnhislysalailetrpphe
405410415
protyrasnmetasptrpargglyargvaltyralavalsermetphe
420425430
asnproglnglyasnaspmetthrlysglyleuleuthrleualalys
435440445
glylysproileglylysgluglytyrtyrtrpleulysilehisgly
450455460
alaasncysalaglyvalasplysvalpropheprogluargilelys
465470475480
pheileglugluasnhisgluasnilemetalacysalalysserpro
485490495
leugluasnthrtrptrpalagluglnaspserprophecyspheleu
500505510
alaphecyspheglutyralaglyvalglnhishisglyleusertyr
515520525
asncysserleuproleualapheaspglysercysserglyilegln
530535540
hispheseralametleuargaspgluvalglyglyargalavalasn
545550555560
leuleuprosergluthrvalglnaspiletyrglyilevalalalys
565570575
lysvalasngluileleuglnalaaspalaileasnglythraspasn
580585590
gluvalvalthrvalthraspgluasnthrglygluileserglulys
595600605
vallysleuglythrlysalaleualaglyglntrpleualatyrgly
610615620
valthrargservalthrlysargservalmetthrleualatyrgly
625630635640
serlysglupheglypheargglnglnvalleugluaspthrilegln
645650655
proalaileaspserglylysglyleumetphethrglnproasngln
660665670
alaalaglytyrmetalalysleuiletrpgluservalservalthr
675680685
valvalalaalavalglualametasntrpleulysseralaalalys
690695700
leuleualaalagluvallysasplyslysthrglygluileleuarg
705710715720
lysargcysalavalhistrpvalthrproaspglypheprovaltrp
725730735
glnglutyrlyslysproileglnthrargleuasnleumetpheleu
740745750
glyglnpheargleuglnprothrileasnthrasnlysaspserglu
755760765
ileaspalahislysglngluserglyilealaproasnphevalhis
770775780
serglnaspglyserhisleuarglysthrvalvaltrpalahisglu
785790795800
lystyrglyilegluserphealaleuilehisaspserpheglythr
805810815
ileproalaaspalaalaasnleuphelysalavalarggluthrmet
820825830
valaspthrtyrglusercysaspvalleualaaspphetyraspgln
835840845
phealaaspglnleuhisgluserglnleuasplysmetproalaleu
850855860
proalalysglyasnleuasnleuargaspileleugluseraspphe
865870875880
alapheala
<210>3
<211>2673
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>6his-t7rnap
<400>3
atgcaccatcaccatcaccatatgaacacgattaacatcgctaagaacgacttctctgac60
atcgaactggctgctatcccgttcaacactctggctgaccattacggtgagcgtttagct120
cgcgaacagttggcccttgagcatgagtcttacgagatgggtgaagcacgcttccgcaag180
atgtttgagcgtcaacttaaagctggtgaggttgcggataacgctgccgccaagcctctc240
atcactaccctactccctaagatgattgcacgcatcaacgactggtttgaggaagtgaaa300
gctaagcgcggcaagcgcccgacagccttccagttcctgcaagaaatcaagccggaagcc360
gtagcgtacatcaccattaagaccactctggcttgcctaaccagtgctgacaatacaacc420
gttcaggctgtagcaagcgcaatcggtcgggccattgaggacgaggctcgcttcggtcgt480
atccgtgaccttgaagctaagcacttcaagaaaaacgttgaggaacaactcaacaagcgc540
gtagggcacgtctacaagaaagcatttatgcaagttgtcgaggctgacatgctctctaag600
ggtctactcggtggcgaggcgtggtcttcgtggcataaggaagactctattcatgtagga660
gtacgctgcatcgagatgctcattgagtcaaccggaatggttagcttacaccgccaaaat720
gctggcgtagtaggtcaagactctgagactatcgaactcgcacctgaatacgctgaggct780
atcgcaacccgtgcaggtgcgctggctggcatctctccgatgttccaaccttgcgtagtt840
cctcctaagccgtggactggcattactggtggtggctattgggctaacggtcgtcgtcct900
ctggcgctggtgcgtactcacagtaagaaagcactgatgcgctacgaagacgtttacatg960
cctgaggtgtacaaagcgattaacattgcgcaaaacaccgcatggaaaatcaacaagaaa1020
gtcctagcggtcgccaacgtaatcaccaagtggaagcattgtccggtcgaggacatccct1080
gcgattgagcgtgaagaactcccgatgaaaccggaagacatcgacatgaatcctgaggct1140
ctcaccgcgtggaaacgtgctgccgctgctgtgtaccgcaaggacaaggctcgcaagtct1200
cgccgtatcagccttgagttcatgcttgagcaagccaataagtttgctaaccataaggcc1260
atctggttcccttacaacatggactggcgcggtcgtgtttacgctgtgtcaatgttcaac1320
ccgcaaggtaacgatatgaccaaaggactgcttacgctggcgaaaggtaaaccaatcggt1380
aaggaaggttactactggctgaaaatccacggtgcaaactgtgcgggtgtcgataaggtt1440
ccgttccctgagcgcatcaagttcattgaggaaaaccacgagaacatcatggcttgcgct1500
aagtctccactggagaacacttggtgggctgagcaagattctccgttctgcttccttgcg1560
ttctgctttgagtacgctggggtacagcaccacggcctgagctataactgctcccttccg1620
ctggcgtttgacgggtcttgctctggcatccagcacttctccgcgatgctccgagatgag1680
gtaggtggtcgcgcggttaacttgcttcctagtgaaaccgttcaggacatctacgggatt1740
gttgctaagaaagtcaacgagattctacaagcagacgcaatcaatgggaccgataacgaa1800
gtagttaccgtgaccgatgagaacactggtgaaatctctgagaaagtcaagctgggcact1860
aaggcactggctggtcaatggctggcttacggtgttactcgcagtgtgactaagcgttca1920
gtcatgacgctggcttacgggtccaaagagttcggcttccgtcaacaagtgctggaagat1980
accattcagccagctattgattccggcaagggtctgatgttcactcagccgaatcaggct2040
gctggatacatggctaagctgatttgggaatctgtgagcgtgacggtggtagctgcggtt2100
gaagcaatgaactggcttaagtctgctgctaagctgctggctgctgaggtcaaagataag2160
aagactggagagattcttcgcaagcgttgcgctgtgcattgggtaactcctgatggtttc2220
cctgtgtggcaggaatacaagaagcctattcagacgcgcttgaacctgatgttcctcggt2280
cagttccgcttacagcctaccattaacaccaacaaagatagcgagattgatgcacacaaa2340
caggagtctggtatcgctcctaactttgtacacagccaagacggtagccaccttcgtaag2400
actgtagtgtgggcacacgagaagtacggaatcgaatcttttgcactgattcacgactcc2460
ttcggtaccattccggctgacgctgcgaacctgttcaaagcagtgcgcgaaactatggtt2520
gacacatatgagtcttgtgatgtactggctgatttctacgaccagttcgctgaccagttg2580
cacgagtctcaattggacaaaatgccagcacttccggctaaaggtaacttgaacctccgt2640
gacatcttagagtcggacttcgcgttcgcgtaa2673
<210>4
<211>890
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>6his-t7rnap
<400>4
methishishishishishismetasnthrileasnilealalysasn
151015
asppheseraspilegluleualaalailepropheasnthrleuala
202530
asphistyrglygluargleualaarggluglnleualaleugluhis
354045
glusertyrglumetglyglualaargphearglysmetphegluarg
505560
glnleulysalaglygluvalalaaspasnalaalaalalysproleu
65707580
ilethrthrleuleuprolysmetilealaargileasnasptrpphe
859095
glugluvallysalalysargglylysargprothralapheglnphe
100105110
leuglngluilelysproglualavalalatyrilethrilelysthr
115120125
thrleualacysleuthrseralaaspasnthrthrvalglnalaval
130135140
alaseralaileglyargalailegluaspglualaargpheglyarg
145150155160
ileargaspleuglualalyshisphelyslysasnvalgluglugln
165170175
leuasnlysargvalglyhisvaltyrlyslysalaphemetglnval
180185190
valglualaaspmetleuserlysglyleuleuglyglyglualatrp
195200205
sersertrphislysgluaspserilehisvalglyvalargcysile
210215220
glumetleuilegluserthrglymetvalserleuhisargglnasn
225230235240
alaglyvalvalglyglnaspsergluthrilegluleualaproglu
245250255
tyralaglualailealathrargalaglyalaleualaglyileser
260265270
prometpheglnprocysvalvalproprolysprotrpthrglyile
275280285
thrglyglyglytyrtrpalaasnglyargargproleualaleuval
290295300
argthrhisserlyslysalaleumetargtyrgluaspvaltyrmet
305310315320
progluvaltyrlysalaileasnilealaglnasnthralatrplys
325330335
ileasnlyslysvalleualavalalaasnvalilethrlystrplys
340345350
hiscysprovalgluaspileproalailegluargglugluleupro
355360365
metlysprogluaspileaspmetasnproglualaleuthralatrp
370375380
lysargalaalaalaalavaltyrarglysasplysalaarglysser
385390395400
argargileserleugluphemetleugluglnalaasnlyspheala
405410415
asnhislysalailetrppheprotyrasnmetasptrpargglyarg
420425430
valtyralavalsermetpheasnproglnglyasnaspmetthrlys
435440445
glyleuleuthrleualalysglylysproileglylysgluglytyr
450455460
tyrtrpleulysilehisglyalaasncysalaglyvalasplysval
465470475480
propheprogluargilelyspheileglugluasnhisgluasnile
485490495
metalacysalalysserproleugluasnthrtrptrpalaglugln
500505510
aspserprophecyspheleualaphecyspheglutyralaglyval
515520525
glnhishisglyleusertyrasncysserleuproleualapheasp
530535540
glysercysserglyileglnhispheseralametleuargaspglu
545550555560
valglyglyargalavalasnleuleuprosergluthrvalglnasp
565570575
iletyrglyilevalalalyslysvalasngluileleuglnalaasp
580585590
alaileasnglythraspasngluvalvalthrvalthraspgluasn
595600605
thrglygluileserglulysvallysleuglythrlysalaleuala
610615620
glyglntrpleualatyrglyvalthrargservalthrlysargser
625630635640
valmetthrleualatyrglyserlysglupheglypheargglngln
645650655
valleugluaspthrileglnproalaileaspserglylysglyleu
660665670
metphethrglnproasnglnalaalaglytyrmetalalysleuile
675680685
trpgluservalservalthrvalvalalaalavalglualametasn
690695700
trpleulysseralaalalysleuleualaalagluvallysasplys
705710715720
lysthrglygluileleuarglysargcysalavalhistrpvalthr
725730735
proaspglypheprovaltrpglnglutyrlyslysproileglnthr
740745750
argleuasnleumetpheleuglyglnpheargleuglnprothrile
755760765
asnthrasnlysaspsergluileaspalahislysglnglusergly
770775780
ilealaproasnphevalhisserglnaspglyserhisleuarglys
785790795800
thrvalvaltrpalahisglulystyrglyilegluserphealaleu
805810815
ilehisaspserpheglythrileproalaaspalaalaasnleuphe
820825830
lysalavalarggluthrmetvalaspthrtyrglusercysaspval
835840845
leualaaspphetyraspglnphealaaspglnleuhisglusergln
850855860
leuasplysmetproalaleuproalalysglyasnleuasnleuarg
865870875880
aspileleugluseraspphealapheala
885890
<210>5
<211>3060
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>dna模板
<400>5
gatcttcttgagatcctttttttctgcgcgtaatctgctgcttgcaaacaaaaaaaccac60
cgctaccagcggtggtttgtttgccggatcaagagctaccaactctttttccgaaggtaa120
ctggcttcagcagagcgcagataccaaatactgttcttctagtgtagccgtagttagccc180
accacttcaagaactctgtagcaccgcctacatacctcgctctgctaatcctgttaccag240
tggctgctgccagtggcgataagtcgtgtcttaccgggttggactcaagacgatagttac300
cggataaggcgcagcggtcgggctgaacggggggttcgtgcacacagcccagcttggagc360
gaacgacctacaccgaactgagatacctacagcgtgagctatgagaaagcgccacgcttc420
ccgaagggagaaaggcggacaggtatccggtaagcggcagggtcggaacaggagagcgca480
cgagggagcttccagggggaaacgcctggtatctttatagtcctgtcgggtttcgccacc540
tctgacttgagcgtcgatttttgtgatgctcgtcaggggggcggagcctatggaaaaacg600
ccagcaacgcggcctttttacggttcctggccttttgctggccttttgctcacatgttct660
ttcctgcgttatcccctgattctgtggataaccgtattaccgcctttgagtgagctgata720
ccgctcgccgcagccgaacgaccgagcgcagcgagtcagtgagcgaggaagcggaaggcg780
agagtagggaactgccaggcatcaaactaagcagaaggcccctgacggatggcctttttg840
cgtttctacaaactctttctgtgttgtaaaacgacggccagtcttaagctcgggccccct900
gggcggttctgataacgagtaatcgttaatccgcaaataacgtaaaaacccgcttcggcg960
ggtttttttatggggggagtttagggaaagagcatttgtcagaatatttaagggcgcctg1020
tcactttgcttgatatatgagaattatttaaccttataaatgagaaaaaagcaacgcact1080
ttaaataagatacgttgctttttcgattgatgaacacctataattaaactattcatctat1140
tatttatgattttttgtatatacaatatttctagtttgttaaagagaattaagaaaataa1200
atctcgaaaataataaagggaaaatcagtttttgatatcaaaattatacatgtcaacgat1260
aatacaaaatataatacaaactataagatgttatcagtatttattatgcatttagaataa1320
attttgtgtcgcccttgtacttagtcgctgaataatacgactcactatagcggaacccaa1380
gctaagcgccagaactggcaccttcgggtgccagttgacgaggtggggtttatcgagatt1440
tcggcggatgactcccggttgttcatcacaaccgcaagcttttacttaaatcattaaggt1500
gacttagtggacaaaggtgaaagtgtgatgaaacccgacctggacggaggcgcgcccgag1560
atgagtaggctgtcccatcaggggaggaatcggggacggctgaaaggcgagggcgccgaa1620
gcgagcagagttcctcccgctctgcttggctgggggtgaggggaatacccttaccactgt1680
cgcgaaagcggagagccgtccaggatcccgtcaaaagggcgacaccccataattagcccg1740
ggcgaaaggcccagtctttcgactgagcctttcgttttatttgatgcctggcagttccct1800
actctcgcatggggagtccccacactaccatcggcgctacggcgtttcacttctgagttc1860
ggcatggggtcaggtgggaccaccgcgctactgccgccaggcaaacaaggggtgttatga1920
gccatattcaggtataaatgggctcgcgataatgttcagaattggttaattggttgtaac1980
actgacccctatttgtttatttttctaaatacattcaaatatgtatccgctcatgagaca2040
ataaccctgataaatgcttcaataatattgaaaaaggaagaatatgagtattcaacattt2100
ccgtgtcgcccttattcccttttttgcggcattttgccttcctgtttttgctcacccaga2160
aacgctggtgaaagtaaaagatgctgaagatcagttgggtgcacgagtgggttacatcga2220
actggatctcaacagcggtaagatccttgagagttttcgccccgaagaacgttttccaat2280
gatgagcacttttaaagttctgctatgtggcgcggtattatcccgtattgacgccgggca2340
agagcaactcggtcgccgcatacactattctcagaatgacttggttgagtactcaccagt2400
cacagaaaagcatcttacggatggcatgacagtaagagaattatgcagtgctgccataac2460
catgagtgataacactgcggccaacttacttctgacaacgatcggaggaccgaaggagct2520
aaccgcttttttgcacaacatgggggatcatgtaactcgccttgatcgttgggaaccgga2580
gctgaatgaagccataccaaacgacgagcgtgacaccacgatgcctgtagcgatggcaac2640
aacgttgcgcaaactattaactggcgaactacttactctagcttcccggcaacaattaat2700
agactggatggaggcggataaagttgcaggaccacttctgcgctcggcccttccggctgg2760
ctggtttattgctgataaatccggagccggtgagcgtggttctcgcggtatcatcgcagc2820
gctggggccagatggtaagccctcccgtatcgtagttatctacacgacggggagtcaggc2880
aactatggatgaacgaaatagacagatcgctgagataggtgcctcactgattaagcattg2940
gtaaaagcagagcattacgctgacttgacgggacggcgcaagctcatgaccaaaatccct3000
taacgtgagttacgcgcgcgtcgttccactgagcgtcagaccccgtagaaaagatcaaag3060
<210>6
<211>334
<212>rna
<213>人工序列(artificialsequence)
<220>
<223>rna转录物
<400>6
gcggaacccaagcuaagcgccagaacuggcaccuucgggugccaguugacgagguggggu60
uuaucgagauuucggcggaugacucccgguuguucaucacaaccgcaagcuuuuacuuaa120
aucauuaaggugacuuaguggacaaaggugaaagugugaugaaacccgaccuggacggag180
gcgcgcccgagaugaguaggcugucccaucaggggaggaaucggggacggcugaaaggcg240
agggcgccgaagcgagcagaguuccucccgcucugcuuggcugggggugaggggaauacc300
cuuaccacugucgcgaaagcggagagccguccag334
<210>7
<211>16
<212>rna
<213>人工序列(artificialsequence)
<220>
<223>rna裂解产物
<400>7
gcggaacccaagcuaa16
<210>8
<211>4671
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>底物等
<400>8
gagttacgcgcgcgtcgttccactgagcgtcagaccccgtagaaaagatcaaaggatctt60
cttgagatcctttttttctgcgcgtaatctgctgcttgcaaacaaaaaaaccaccgctac120
cagcggtggtttgtttgccggatcaagagctaccaactctttttccgaaggtaactggct180
tcagcagagcgcagataccaaatactgttcttctagtgtagccgtagttagcccaccact240
tcaagaactctgtagcaccgcctacatacctcgctctgctaatcctgttaccagtggctg300
ctgccagtggcgataagtcgtgtcttaccgggttggactcaagacgatagttaccggata360
aggcgcagcggtcgggctgaacggggggttcgtgcacacagcccagcttggagcgaacga420
cctacaccgaactgagatacctacagcgtgagctatgagaaagcgccacgcttcccgaag480
ggagaaaggcggacaggtatccggtaagcggcagggtcggaacaggagagcgcacgaggg540
agcttccagggggaaacgcctggtatctttatagtcctgtcgggtttcgccacctctgac600
ttgagcgtcgatttttgtgatgctcgtcaggggggcggagcctatggaaaaacgccagca660
acgcggcctttttacggttcctggccttttgctggccttttgctcacatgttctttcctg720
cgttatcccctgattctgtggataaccgtattaccgcctttgagtgagctgataccgctc780
gccgcagccgaacgaccgagcgcagcgagtcagtgagcgaggaagcggaaggcgagagta840
gggaactgccaggcatcaaactaagcagaaggcccctgacggatggcctttttgcgtttc900
tacaaactctttctgtgttgtaaaacgacggccagtcttaagctcgggccccctgggcgg960
ttctgataacgagtaatcgttaatccgcaaataacgtaaaaacccgcttcggcgggtttt1020
tttatggggggagtttagggaaagagcatttgtcagaatatttaagggcgcctgtcactt1080
tgcttgatatatgagaattatttaaccttataaatgagaaaaaagcaacgcactttaaat1140
aagatacgttgctttttcgattgatgaacacctataattaaactattcatctattattta1200
tgattttttgtatatacaatatttctagtttgttaaagagaattaagaaaataaatctcg1260
aaaataataaagggaaaatcagtttttgatatcaaaattatacatgtcaacgataataca1320
aaatataatacaaactataagatgttatcagtatttattatgcatttagaatacgtactc1380
agcgtctgggttccccatcggtgatgtcgtataagagacgtataggagacctatagtgtc1440
ttcggggtaatacgactcactatagcggaacccaagcttggcattccggtactgttggta1500
aagccaccatggaagatgcgaagaacataaagaaaggtcccgccccattttacccactcg1560
aggatggaacagctggggagcaactgcacaaggccatgaagcgctatgcgttggtgccgg1620
gaaccatcgcgttcaccgacgcacacatcgaagtgaacatcacttacgccgagtactttg1680
agatgagcgtcaggctggccgaggctatgaagcgatacggtctgaacaccaaccaccgga1740
tcgtggtctgctctgaaaacagcctgcagttcttcatgccggtcctgggggccctgttca1800
tcggcgtggccgtggcacccgccaacgatatctacaacgagagagaattgctgaactcga1860
tgaacatctcccagcctaccgtggtgttcgtgtcgaagaaggggttgcagaagatcctga1920
acgtgcagaagaagctgcccatcattcaaaagattatcattatggattccaaaaccgact1980
accagggtttccagtcaatgtataccttcgtgacctcccatctgccccctggcttcaacg2040
aatacgacttcgtgcctgaaagcttcgaccgcgacaagacgatcgccctcatcatgaact2100
cgtccggctcgaccgggctgcccaaaggagtggccctgccacaccggaccgcttgcgtgc2160
ggttctcccacgcccgggaccctattttcggcaatcagatcattccggacactgccatcc2220
tgagcgtggtccccttccatcacgggtttgggatgtttaccactctgggctacctcatct2280
gcggattcagggtggtgctgatgtaccggttcgaggaagaacttttcctgcggagcctgc2340
aggattacaagatccagtccgccctcctcgtgccaaccctcttctcattcttcgctaagt2400
ccactctcatcgataagtacgacctgtcgaatctccacgaaattgcgtccggtggtgcac2460
cgctgtccaaggaggtcggcgaagccgtggccaagcgcttccacctcccgggaatacgcc2520
agggatacggcctgactgaaacgaccagcgcgattctgatcaccccggagggcgacgaca2580
agccgggtgccgtggggaaagtggtgccgttcttcgaagcaaaggtcgtggatctggata2640
ccggaaagactctgggcgtgaaccagagaggggaactttgtgtgcgcggaccgatgatta2700
tgtccggatatgtcaacaaccccgaggccactaatgccctgatcgacaaggacggatggt2760
tgcatagcggcgacatcgcatactgggacgaggacgagcactttttcattgtggatcggc2820
tcaagtccctgatcaagtacaagggataccaggtcgcccctgccgaacttgagtccatcc2880
tgctgcaacatccgaacattttcgacgcgggcgtcgctggccttcctgatgatgacgccg2940
gagagctgcccgcggccgtggtggtgctcgaacacggaaaaactatgaccgagaaggaaa3000
tcgtggactacgtggcgtcacaagtcaccactgccaagaaactgcgcggcggagtcgtgt3060
tcgtggacgaggtgcccaagggcctgaccggaaagctggacgctagaaagatccgggaga3120
tcctgattaaggccaagaagggaggaaagtccaagctctgagatctagagggccctattc3180
tatagtgtcacctaaatgctaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaa3240
aaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaa3300
gaattcaaaagaagaccatatacgtctcctataggtctcgtatactgcttcctatacgac3360
atcaccgatggggaacacgaagcgatccagccccccttattagcccgggcgaaaggccca3420
gtctttcgactgagcctttcgttttatttgatgcctggcagttccctactctcgcatggg3480
gagtccccacactaccatcggcgctacggcgtttcacttctgagttcggcatggggtcag3540
gtgggaccaccgcgctactgccgccaggcaaacaaggggtgttatgagccatattcaggt3600
ataaatgggctcgcgataatgttcagaattggttaattggttgtaacactgacccctatt3660
tgtttatttttctaaatacattcaaatatgtatccgctcatgagacaataaccctgataa3720
atgcttcaataatattgaaaaaggaagaatatgagtattcaacatttccgtgtcgccctt3780
attcccttttttgcggcattttgccttcctgtttttgctcacccagaaacgctggtgaaa3840
gtaaaagatgctgaagatcagttgggtgcacgagtgggttacatcgaactggatctcaac3900
agcggtaagatccttgagagttttcgccccgaagaacgttttccaatgatgagcactttt3960
aaagttctgctatgtggcgcggtattatcccgtattgacgccgggcaagagcaactcggt4020
cgccgcatacactattctcagaatgacttggttgagtactcaccagtcacagaaaagcat4080
cttacggatggcatgacagtaagagaattatgcagtgctgccataaccatgagtgataac4140
actgcggccaacttacttctgacaacgatcggaggaccgaaggagctaaccgcttttttg4200
cacaacatgggggatcatgtaactcgccttgatcgttgggaaccggagctgaatgaagcc4260
ataccaaacgacgagcgtgacaccacgatgcctgtagcgatggcaacaacgttgcgcaaa4320
ctattaactggcgaactacttactctagcttcccggcaacaattaatagactggatggag4380
gcggataaagttgcaggaccacttctgcgctcggcccttccggctggctggtttattgct4440
gataaatccggagccggtgagcgtggttctcgcggtatcatcgcagcgctggggccagat4500
ggtaagccctcccgtatcgtagttatctacacgacggggagtcaggcaactatggatgaa4560
cgaaatagacagatcgctgagataggtgcctcactgattaagcattggtaagcagagcat4620
tacgctgacttgacgggacggcgcaagctcatgaccaaaatcccttaacgt4671
<210>9
<211>1697
<212>rna
<213>人工序列(artificialsequence)
<220>
<223>底物等
<400>9
gcggaacccaagcuuggcauuccgguacuguugguaaagccaccauggaagaugcgaaga60
acauaaagaaaggucccgccccauuuuacccacucgaggauggaacagcuggggagcaac120
ugcacaaggccaugaagcgcuaugcguuggugccgggaaccaucgcguucaccgacgcac180
acaucgaagugaacaucacuuacgccgaguacuuugagaugagcgucaggcuggccgagg240
cuaugaagcgauacggucugaacaccaaccaccggaucguggucugcucugaaaacagcc300
ugcaguucuucaugccgguccugggggcccuguucaucggcguggccguggcacccgcca360
acgauaucuacaacgagagagaauugcugaacucgaugaacaucucccagccuaccgugg420
uguucgugucgaagaagggguugcagaagauccugaacgugcagaagaagcugcccauca480
uucaaaagauuaucauuauggauuccaaaaccgacuaccaggguuuccagucaauguaua540
ccuucgugaccucccaucugcccccuggcuucaacgaauacgacuucgugccugaaagcu600
ucgaccgcgacaagacgaucgcccucaucaugaacucguccggcucgaccgggcugccca660
aaggaguggcccugccacaccggaccgcuugcgugcgguucucccacgcccgggacccua720
uuuucggcaaucagaucauuccggacacugccauccugagcgugguccccuuccaucacg780
gguuugggauguuuaccacucugggcuaccucaucugcggauucaggguggugcugaugu840
accgguucgaggaagaacuuuuccugcggagccugcaggauuacaagauccaguccgccc900
uccucgugccaacccucuucucauucuucgcuaaguccacucucaucgauaaguacgacc960
ugucgaaucuccacgaaauugcguccgguggugcaccgcuguccaaggaggucggcgaag1020
ccguggccaagcgcuuccaccucccgggaauacgccagggauacggccugacugaaacga1080
ccagcgcgauucugaucaccccggagggcgacgacaagccgggugccguggggaaagugg1140
ugccguucuucgaagcaaaggucguggaucuggauaccggaaagacucugggcgugaacc1200
agagaggggaacuuugugugcgcggaccgaugauuauguccggauaugucaacaaccccg1260
aggccacuaaugcccugaucgacaaggacggaugguugcauagcggcgacaucgcauacu1320
gggacgaggacgagcacuuuuucauuguggaucggcucaagucccugaucaaguacaagg1380
gauaccaggucgccccugccgaacuugaguccauccugcugcaacauccgaacauuuucg1440
acgcgggcgucgcuggccuuccugaugaugacgccggagagcugcccgcggccguggugg1500
ugcucgaacacggaaaaacuaugaccgagaaggaaaucguggacuacguggcgucacaag1560
ucaccacugccaagaaacugcgcggcggagucguguucguggacgaggugcccaagggcc1620
ugaccggaaagcuggacgcuagaaagauccgggagauccugauuaaggccaagaagggag1680
gaaaguccaagcucuga1697
<210>10
<211>4945
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>底物等
<400>10
ttaccaatgcttaatcagtgaggcacctatctcagcgatctgtctatttcgttcatccat60
agttgcctgactccccgtcgtgtagataactacgatacgggagggcttaccatctggccc120
cagcgctgcgatgataccgcgagaaccacgctcaccggctccggatttatcagcaataaa180
ccagccagccggaagggccgagcgcagaagtggtcctgcaactttatccgcctccatcca240
gtctattaattgttgccgggaagctagagtaagtagttcgccagttaatagtttgcgcaa300
cgttgttgccatcgctacaggcatcgtggtgtcacgctcgtcgtttggtatggcttcatt360
cagctccggttcccaacgatcaaggcgagttacatgatcccccatgttgtgcaaaaaagc420
ggttagctccttcggtcctccgatcgttgtcagaagtaagttggccgcagtgttatcact480
catggttatggcagcactgcataattctcttactgtcatgccatccgtaagatgcttttc540
tgtgactggtgagtactcaaccaagtcattctgagaatagtgtatgcggcgaccgagttg600
ctcttgcccggcgtcaatacgggataataccgcgccacatagcagaactttaaaagtgct660
catcattggaaaacgttcttcggggcgaaaactctcaaggatcttaccgctgttgagatc720
cagttcgatgtaacccactcgtgcacccaactgatcttcagcatcttttactttcaccag780
cgtttctgggtgagcaaaaacaggaaggcaaaatgccgcaaaaaagggaataagggcgac840
acggaaatgttgaatactcatattcttcctttttcaatattattgaagcatttatcaggg900
ttattgtctcatgagcggatacatatttgaatgtatttagaaaaataaacaaataggggt960
cagtgttacaaccaattaaccaattctgaacattatcgcgagcccatttatacctgaata1020
tggctcataacaccccttgtttgcctggcggcagtagcgcggtggtcccacctgacccca1080
tgccgaactcagaagtgaaacgccgtagcgccgatggtagtgtggggactccccatgcga1140
gagtagggaactgccaggcatcaaataaaacgaaaggctcagtcgaaagactgggccttt1200
cgcccgggctaattatggggtgtcgcccttcgctgaaggggtaatacgactcactatagg1260
gaaataagagagaaaagaagagtaagaagaaatataagagccaccatggaagatgcgaag1320
aacataaagaaaggtcccgccccattttacccactcgaggatggaacagctggggagcaa1380
ctgcacaaggccatgaagcgctatgcgttggtgccgggaaccatcgcgttcaccgacgca1440
cacatcgaagtgaacatcacttacgccgagtactttgagatgagcgtcaggctggccgag1500
gctatgaagcgatacggtctgaacaccaaccaccggatcgtggtctgctctgaaaacagc1560
ctgcagttcttcatgccggtcctgggggccctgttcatcggcgtggccgtggcacccgcc1620
aacgatatctacaacgagagagaattgctgaactcgatgaacatctcccagcctaccgtg1680
gtgttcgtgtcgaagaaggggttgcagaagatcctgaacgtgcagaagaagctgcccatc1740
attcaaaagattatcattatggattccaaaaccgactaccagggtttccagtcaatgtat1800
accttcgtgacctcccatctgccccctggcttcaacgaatacgacttcgtgcctgaaagc1860
ttcgaccgcgacaagacgatcgccctcatcatgaactcgtccggctcgaccgggctgccc1920
aaaggagtggccctgccacaccggaccgcttgcgtgcggttctcccacgcccgggaccct1980
attttcggcaatcagatcattccggacactgccatcctgagcgtggtccccttccatcac2040
gggtttgggatgtttaccactctgggctacctcatctgcggattcagggtggtgctgatg2100
taccggttcgaggaagaacttttcctgcggagcctgcaggattacaagatccagtccgcc2160
ctcctcgtgccaaccctcttctcattcttcgctaagtccactctcatcgataagtacgac2220
ctgtcgaatctccacgaaattgcgtccggtggtgcaccgctgtccaaggaggtcggcgaa2280
gccgtggccaagcgcttccacctcccgggaattcgccagggatacggcctgactgaaacg2340
accagcgcgattctgatcaccccggagggcgacgacaagccgggtgccgtggggaaagtg2400
gtgccgttcttcgaagcaaaggtcgtggatctggataccggaaagactctgggcgtgaac2460
cagagaggggaactttgtgtgcgcggaccgatgattatgtccggatatgtcaacaacccc2520
gaggccactaatgccctgatcgacaaggacggatggttgcatagcggcgacatcgcatac2580
tgggacgaggacgagcactttttcattgtggatcggctcaagtccctgatcaagtacaag2640
ggataccaggtcgcccctgccgaacttgagtccatcctgctgcaacatccgaacattttc2700
gacgcgggcgtcgctggccttcctgatgatgacgccggagagctgcccgcggccgtggtg2760
gtgctcgaacacggaaaaactatgaccgagaaggaaatcgtggactacgtggcgtcacaa2820
gtcaccactgccaagaaactgcgcggcggagtcgtgttcgtggacgaggtgcccaagggc2880
ctgaccggaaagctggacgctagaaagatccgggagatcctgattaaggccaagaaggga2940
ggaaagtccaagctctgaatctgctgccttctgcggggcttgccttctggccatgccctt3000
cttctctcccttgcacctgtacctcttggtctttgaataaagcctgagtaggaagtgagg3060
gaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaa3120
aaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaggaagagcgctgc3180
ctccttcagagtctgctcacgagctcggtctcggatcccctatacgacatcaccgatggg3240
gaacacgaagcgatccagccccccttattagcccgggcgaaaggcccagtctttcgactg3300
agcctttcgttttatttgatgcctggcagttccctactctcgcatggggagtccccacac3360
taccatcggcgctacggcgtttcacttctgagttcggcatggggtcaggtgggaccaccg3420
cgctactgccgccaggcaaacaaggggtgttatgagccatattcaggtataaatgggctc3480
gcgaaaacgtcaaaagggcgacacaaaatttattctaaatgcataataaatactgataac3540
atcttatagtttgtattatattttgtattatcgttgacatgtataattttgatatcaaaa3600
actgattttccctttattattttcgagatttattttcttaattctctttaacaaactaga3660
aatattgtatatacaaaaaatcataaataatagatgaatagtttaattataggtgttcat3720
caatcgaaaaagcaacgtatcttatttaaagtgcgttgcttttttctcatttataaggtt3780
aaataattctcatatatcaagcaaagtgacaggcgcccttaaatattctgacaaatgctc3840
tttccctaaactccccccataaaaaaacccgccgaagcgggtttttacgttatttgcgga3900
ttaacgattactcgttatcagaaccgcccagggggcccgagcttaagactggccgtcgtt3960
ttacaacacagaaagagtttgtagaaacgcaaaaaggccatccgtcaggggccttctgct4020
tagtttgatgcctggcagttccctactctcgccttccgcttcctcgctcactgactcgct4080
gcgctcggtcgttcggctgcggcgagcggtatcagctcactcaaaggcggtaatacggtt4140
atccacagaatcaggggataacgcaggaaagaacatgtgagcaaaaggccagcaaaaggc4200
caggaaccgtaaaaaggccgcgttgctggcgtttttccataggctccgcccccctgacga4260
gcatcacaaaaatcgacgctcaagtcagaggtggcgaaacccgacaggactataaagata4320
ccaggcgtttccccctggaagctccctcgtgcgctctcctgttccgaccctgccgcttac4380
cggatacctgtccgcctttctcccttcgggaagcgtggcgctttctcatagctcacgctg4440
taggtatctcagttcggtgtaggtcgttcgctccaagctgggctgtgtgcacgaaccccc4500
cgttcagcccgaccgctgcgccttatccggtaactatcgtcttgagtccaacccggtaag4560
acacgacttatcgccactggcagcagccactggtaacaggattagcagagcgaggtatgt4620
aggcggtgctacagagttcttgaagtggtgggctaactacggctacactagaagaacagt4680
atttggtatctgcgctctgctgaagccagttaccttcggaaaaagagttggtagctcttg4740
atccggcaaacaaaccaccgctggtagcggtggtttttttgtttgcaagcagcagattac4800
gcgcagaaaaaaaggatctcaagaagatcctttgatcttttctacggggtctgacgctca4860
gtggaacgacgcgcgcgtaactcacgttaagggattttggtcatgagcttgcgccgtccc4920
gtcaagtcagcgtaatgctctgctt4945
<210>11
<211>1955
<212>rna
<213>人工序列(artificialsequence)
<220>
<223>底物等
<400>11
gggaaauaagagagaaaagaagaguaagaagaaauauaagagccaccauggaagaugcga60
agaacauaaagaaaggucccgccccauuuuacccacucgaggauggaacagcuggggagc120
aacugcacaaggccaugaagcgcuaugcguuggugccgggaaccaucgcguucaccgacg180
cacacaucgaagugaacaucacuuacgccgaguacuuugagaugagcgucaggcuggccg240
aggcuaugaagcgauacggucugaacaccaaccaccggaucguggucugcucugaaaaca300
gccugcaguucuucaugccgguccugggggcccuguucaucggcguggccguggcacccg360
ccaacgauaucuacaacgagagagaauugcugaacucgaugaacaucucccagccuaccg420
ugguguucgugucgaagaagggguugcagaagauccugaacgugcagaagaagcugccca480
ucauucaaaagauuaucauuauggauuccaaaaccgacuaccaggguuuccagucaaugu540
auaccuucgugaccucccaucugcccccuggcuucaacgaauacgacuucgugccugaaa600
gcuucgaccgcgacaagacgaucgcccucaucaugaacucguccggcucgaccgggcugc660
ccaaaggaguggcccugccacaccggaccgcuugcgugcgguucucccacgcccgggacc720
cuauuuucggcaaucagaucauuccggacacugccauccugagcgugguccccuuccauc780
acggguuugggauguuuaccacucugggcuaccucaucugcggauucaggguggugcuga840
uguaccgguucgaggaagaacuuuuccugcggagccugcaggauuacaagauccaguccg900
cccuccucgugccaacccucuucucauucuucgcuaaguccacucucaucgauaaguacg960
accugucgaaucuccacgaaauugcguccgguggugcaccgcuguccaaggaggucggcg1020
aagccguggccaagcgcuuccaccucccgggaauucgccagggauacggccugacugaaa1080
cgaccagcgcgauucugaucaccccggagggcgacgacaagccgggugccguggggaaag1140
uggugccguucuucgaagcaaaggucguggaucuggauaccggaaagacucugggcguga1200
accagagaggggaacuuugugugcgcggaccgaugauuauguccggauaugucaacaacc1260
ccgaggccacuaaugcccugaucgacaaggacggaugguugcauagcggcgacaucgcau1320
acugggacgaggacgagcacuuuuucauuguggaucggcucaagucccugaucaaguaca1380
agggauaccaggucgccccugccgaacuugaguccauccugcugcaacauccgaacauuu1440
ucgacgcgggcgucgcuggccuuccugaugaugacgccggagagcugcccgcggccgugg1500
uggugcucgaacacggaaaaacuaugaccgagaaggaaaucguggacuacguggcgucac1560
aagucaccacugccaagaaacugcgcggcggagucguguucguggacgaggugcccaagg1620
gccugaccggaaagcuggacgcuagaaagauccgggagauccugauuaaggccaagaagg1680
gaggaaaguccaagcucugaaucugcugccuucugcggggcuugccuucuggccaugccc1740
uucuucucucccuugcaccuguaccucuuggucuuugaauaaagccugaguaggaaguga1800
gggaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaa1860
aaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaggaagagcgcu1920
gccuccuucagagucugcucacgagcucggucucg1955
<210>12
<211>57
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>底物等
<400>12
tctagaggccagcctggccataaggagatatacatcggtactgttggtaaagccacc57
<210>13
<211>50
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>底物等
<400>13
taatgaggccaaactggccaccatcaccatcagagcttggactttcctcc50
<210>14
<211>2673
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>14
atgcaccatcaccatcaccatatgaacacgattaacatcgctaagaacgacttctctgac60
atcgaactggctgctatcccgttcaacactctggctgaccattacggtgagcgtttagct120
cgcgaacagttggcccttgagcatgagtcttacgagatgggtgaagcacgcttccgcaag180
atgtttgagcgtcaacttaaagctggtgaggttgcggataacgctgccgccaagcctctc240
atcactaccctactccctaagatgattgcacgcatcaacgactggtttgaggaagtgaaa300
gctaagcgcggcaagcgcccgacagccttccagttcctgcaagaaatcaagccggaagcc360
gtagcgtacatcaccattaagaccactctggcttgcctaaccagtgctgacaatacaacc420
gttcaggctgtagcaagcgcaatcggtcgggccattgaggacgaggctcgcttcggtcgt480
atccgtgaccttgaagctaagcacttcaagaaaaacgttgaggaacaactcaacaagcgc540
gtagggcacgtctacaagaaagcatttatgcaagttgtcgaggctgacatgctctctaag600
ggtctactcggtggcgaggcgtggtcttcgtggcataaggaagactctattcatgtagga660
gtacgctgcatcgagatgctcattgagtcaaccggaatggttagcttacaccgccaaaat720
gctggcgtagtaggtcaagactctgagactatcgaactcgcacctgaatacgctgaggct780
atcgcaacccgtgcaggtgcgctggctggcatctctccgatgttccaaccttgcgtagtt840
cctcctaagccgtggactggcattactggtggtggctattgggctaacggtcgtcgtcct900
ctggcgctggtgcgtactcacagtaagaaagcactgatgcgctacgaagacgtttacatg960
cctgaggtgtacaaagcgattaacattgcgcaaaacaccgcatggaaaatcaacaagaaa1020
gtcctagcggtcgccaacgtaatcaccaagtggaagcattgtccggtcgaggacatccct1080
gcgattgagcgtgaagaactcccgatgaaaccggaagacatcgacatgaatcctgaggct1140
ctcaccgcgtggaaacgtgctgccgctgctgtgtaccgcaaggacaaggctcgcaagtct1200
cgccgtatcagccttgagttcatgcttgagcaagccaataagtttgctaaccataaggcc1260
atctggttcccttacaacatggactggcgcggtcgtgtttacgctgtgtcaatgttcaac1320
ccgcaaggtaacgatatgaccaaaggactgcttacgctggcgaaaggtaaaccaatcggt1380
aaggaaggttactactggctgaaaatccacggtgcaaactgtgcgggtgtcgataaggtt1440
ccgttccctgagcgcatcaagttcattgaggaaaaccacgagaacatcatggcttgcgct1500
aagtctccactggagaacacttggtgggctgagcaagattctccgttctgcttccttgcg1560
ttctgctttgagtacgctggggtacagcaccacggcctgagctataactgctcccttccg1620
ctggcgtttgacgggtcttgctctggcatccagcacttctccgcgatgctccgagatgag1680
gtaggtggtcgcgcggttaacttgcttcctagtgaaaccgttcaggacatctacgggatt1740
gttgctaagaaagtcaacgagattctacaagcagacgcaatcaatgggaccgataacgaa1800
gtagttaccgtgaccgatgagaacactggtgaaatctctgagaaagtcaagctgggcact1860
aaggcactggctggtcaatggctggcttacggtgttactcgcagtgtgactaagcgttca1920
gtcatgacgctggcttacgggtccaaagagttcggcttccgtcaacaagtgctggaagat1980
accattcagtgggctattgattccggcaagggtctgatgttcactcagccgaatcaggct2040
gctggatacatggctaagctgatttgggaatctgtgagcgtgacggtggtagctgcggtt2100
gaagcaatgaactggcttaagtctgctgctaagctgctggctgctgaggtcaaagataag2160
aagactggagagattcttcgcaagcgttgcgctgtgcattgggtaactcctgatggtttc2220
cctgtgtggcaggaatacaagaagcctattcagacgcgcttgaacctgatgttcctcggt2280
cagttccgcttacagcctaccattaacaccaacaaagatagcgagattgatgcacacaaa2340
caggagtctggtatcgctcctaactttgtacacagccaagacggtagccaccttcgtaag2400
actgtagtgtgggcacacgagaagtacggaatcgaatcttttgcactgattcacgactcc2460
ttcggtaccattccggctgacgctgcgaacctgttcaaagcagtgcgcgaaactatggtt2520
gacacatatgagtcttgtgatgtactggctgatttctacgaccagttcgctgaccagttg2580
cacgagtctcaattggacaaaatgccagcacttccggctaaaggtaacttgaacctccgt2640
gacatcttagagtcggacttcgcgttcgcgtaa2673
<210>15
<211>890
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>15
methishishishishishismetasnthrileasnilealalysasn
151015
asppheseraspilegluleualaalailepropheasnthrleuala
202530
asphistyrglygluargleualaarggluglnleualaleugluhis
354045
glusertyrglumetglyglualaargphearglysmetphegluarg
505560
glnleulysalaglygluvalalaaspasnalaalaalalysproleu
65707580
ilethrthrleuleuprolysmetilealaargileasnasptrpphe
859095
glugluvallysalalysargglylysargprothralapheglnphe
100105110
leuglngluilelysproglualavalalatyrilethrilelysthr
115120125
thrleualacysleuthrseralaaspasnthrthrvalglnalaval
130135140
alaseralaileglyargalailegluaspglualaargpheglyarg
145150155160
ileargaspleuglualalyshisphelyslysasnvalgluglugln
165170175
leuasnlysargvalglyhisvaltyrlyslysalaphemetglnval
180185190
valglualaaspmetleuserlysglyleuleuglyglyglualatrp
195200205
sersertrphislysgluaspserilehisvalglyvalargcysile
210215220
glumetleuilegluserthrglymetvalserleuhisargglnasn
225230235240
alaglyvalvalglyglnaspsergluthrilegluleualaproglu
245250255
tyralaglualailealathrargalaglyalaleualaglyileser
260265270
prometpheglnprocysvalvalproprolysprotrpthrglyile
275280285
thrglyglyglytyrtrpalaasnglyargargproleualaleuval
290295300
argthrhisserlyslysalaleumetargtyrgluaspvaltyrmet
305310315320
progluvaltyrlysalaileasnilealaglnasnthralatrplys
325330335
ileasnlyslysvalleualavalalaasnvalilethrlystrplys
340345350
hiscysprovalgluaspileproalailegluargglugluleupro
355360365
metlysprogluaspileaspmetasnproglualaleuthralatrp
370375380
lysargalaalaalaalavaltyrarglysasplysalaarglysser
385390395400
argargileserleugluphemetleugluglnalaasnlyspheala
405410415
asnhislysalailetrppheprotyrasnmetasptrpargglyarg
420425430
valtyralavalsermetpheasnproglnglyasnaspmetthrlys
435440445
glyleuleuthrleualalysglylysproileglylysgluglytyr
450455460
tyrtrpleulysilehisglyalaasncysalaglyvalasplysval
465470475480
propheprogluargilelyspheileglugluasnhisgluasnile
485490495
metalacysalalysserproleugluasnthrtrptrpalaglugln
500505510
aspserprophecyspheleualaphecyspheglutyralaglyval
515520525
glnhishisglyleusertyrasncysserleuproleualapheasp
530535540
glysercysserglyileglnhispheseralametleuargaspglu
545550555560
valglyglyargalavalasnleuleuprosergluthrvalglnasp
565570575
iletyrglyilevalalalyslysvalasngluileleuglnalaasp
580585590
alaileasnglythraspasngluvalvalthrvalthraspgluasn
595600605
thrglygluileserglulysvallysleuglythrlysalaleuala
610615620
glyglntrpleualatyrglyvalthrargservalthrlysargser
625630635640
valmetthrleualatyrglyserlysglupheglypheargglngln
645650655
valleugluaspthrileglntrpalaileaspserglylysglyleu
660665670
metphethrglnproasnglnalaalaglytyrmetalalysleuile
675680685
trpgluservalservalthrvalvalalaalavalglualametasn
690695700
trpleulysseralaalalysleuleualaalagluvallysasplys
705710715720
lysthrglygluileleuarglysargcysalavalhistrpvalthr
725730735
proaspglypheprovaltrpglnglutyrlyslysproileglnthr
740745750
argleuasnleumetpheleuglyglnpheargleuglnprothrile
755760765
asnthrasnlysaspsergluileaspalahislysglnglusergly
770775780
ilealaproasnphevalhisserglnaspglyserhisleuarglys
785790795800
thrvalvaltrpalahisglulystyrglyilegluserphealaleu
805810815
ilehisaspserpheglythrileproalaaspalaalaasnleuphe
820825830
lysalavalarggluthrmetvalaspthrtyrglusercysaspval
835840845
leualaaspphetyraspglnphealaaspglnleuhisglusergln
850855860
leuasplysmetproalaleuproalalysglyasnleuasnleuarg
865870875880
aspileleugluseraspphealapheala
885890
<210>16
<211>2673
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>16
atgcaccatcaccatcaccatatgaacacgattaacatcgctaagaacgacttctctgac60
atcgaactggctgctatcccgttcaacactctggctgaccattacggtgagcgtttagct120
cgcgaacagttggcccttgagcatgagtcttacgagatgggtgaagcacgcttccgcaag180
atgtttgagcgtcaacttaaagctggtgaggttgcggataacgctgccgccaagcctctc240
atcactaccctactccctaagatgattgcacgcatcaacgactggtttgaggaagtgaaa300
gctaagcgcggcaagcgcccgacagccttccagttcctgcaagaaatcaagccggaagcc360
gtagcgtacatcaccattaagaccactctggcttgcctaaccagtgctgataatacaacc420
gttcaggctgtagcaagcgcaatcggtcgggccattgaggacgaggctcgcttcggtcgt480
atccgtgaccttgaagctaagcacttcaagaaaaacgttgaggaacaactcaacaagcgc540
gtagggcacgtctacaagaaagcatttatgcaagttgtcgaggctgacatgctctctaag600
ggtctactcggtggcgaggcgtggtcttcgtggcataaggaagactctattcatgtagga660
gtacgctgcatcgagatgctcattgagtcaaccggaatggttagcttacaccgccaaaat720
gctggcgtagtaggtcaagactctgagactatcgaactcgcacctgaatacgctgaggct780
atcgcaacccgtgcaggtgcgctggctggcatctctccgatgttccaaccttgcgtagtt840
cctcctaagccgtggactggcattactggtggtggctattgggctaacggtcgtcgtcct900
ctggcgctggtgcgtactcacagtaagaaagcactgatgcgctacgaagacgtttacatg960
cctgaggtgtacaaagcgattaacattgcgcaaaacaccgcatggaaaatcaacaagaaa1020
gtcctagcggtcgccaacgtaatcaccaagtggaagcattgtccggtcgaggacatccct1080
gcgattgagcgtgaagaactcccgatgaaaccggaagacatcgacatgaatcctgaggct1140
ctcaccgcgtggaaacgtgctgccgctgctgtgtaccgcaaggacaaggctcgcaagtct1200
agacgtatcagccttgagttcatgcttgagcaagccaataagtttgctaaccataaggcc1260
atctggttcccttacaacatggactggcgcggtcgtgtttacgctgtgtcaatgttcaac1320
ccgcaaggtaacgatatgaccaaaggactgcttacgctggcgaaaggtaaaccaatcggt1380
aaggaaggttactactggctgaaaatccacggtgcaaactgtgcgggtgtcgataaggtt1440
ccgttccctgagcgcatcaagttcattgaggaaaaccacgagaacatcatggcttgcgct1500
aagtctccactggagaacacttggtgggctgagcaagattctccgttctgcttccttgcg1560
ttctgctttgagtacgctggggtacagcaccacggcctgagctataactgctcccttccg1620
ctggcgtttgacgggtcttgctctggcatccagcacttctccgcgatgctccgagatgag1680
gtaggtggtcgcgcggttaacttgcttcctagtgaaaccgttcaggacatctacgggatt1740
gttgctaagaaagtcaacgagattctacaagcagacgcaatcaatgggaccgataacgaa1800
gtagttaccgtgaccgatgagaacactggtgaaatctctgagaaagtcaagctgggcact1860
aaggcactggctggtcaatggctggcttacggtgttactcgctgggtgacaaagcgttca1920
gtcatgacgctggcttacgggtccaaagagttcggcttccgtcaacaagtgctggaaacc1980
accattcagtgggctattgattccggcaagggtctgatgttcactcagccgaatcaggct2040
gctggatacatggctaagctgatttgggaatctgtgagcgtgacggtggtagctgcggtt2100
gaagcaatgaactggcttaagtctgctgctaagctgctggctgctgaggtcaaagataag2160
aagactggagagattcttcgcaagcgttgcgctgtgcattgggtaactcctgatggtttc2220
cctgtgtggcaggaatacaagaagcctattcagacgcgcttgaacctgatgttcctcggt2280
cagttccgcttacagcctaccattaacaccaacaaagatagcgagattgatgcacacaaa2340
caggagtctggtatcgctcctaactttgtacacagccaagacggtagccaccttcgtaag2400
actgtagtgtgggcacacgagaagtacggaatcgaatcttttgcactgattcacgactcc2460
ttcggtaccattccggctgacgctgcgaacctgttcaaagcagtgcgcgaaactatggtt2520
gacacatatgagtcttgtgatgtactggctgatttctacgaccagttcgctgaccagttg2580
cacgagtctcaattggacaaaatgccagcacttccggctaaaggtaacttgaacctccgt2640
gacatcttagagtcggacttcgcgttcgcgtaa2673
<210>17
<211>890
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>17
methishishishishishismetasnthrileasnilealalysasn
151015
asppheseraspilegluleualaalailepropheasnthrleuala
202530
asphistyrglygluargleualaarggluglnleualaleugluhis
354045
glusertyrglumetglyglualaargphearglysmetphegluarg
505560
glnleulysalaglygluvalalaaspasnalaalaalalysproleu
65707580
ilethrthrleuleuprolysmetilealaargileasnasptrpphe
859095
glugluvallysalalysargglylysargprothralapheglnphe
100105110
leuglngluilelysproglualavalalatyrilethrilelysthr
115120125
thrleualacysleuthrseralaaspasnthrthrvalglnalaval
130135140
alaseralaileglyargalailegluaspglualaargpheglyarg
145150155160
ileargaspleuglualalyshisphelyslysasnvalgluglugln
165170175
leuasnlysargvalglyhisvaltyrlyslysalaphemetglnval
180185190
valglualaaspmetleuserlysglyleuleuglyglyglualatrp
195200205
sersertrphislysgluaspserilehisvalglyvalargcysile
210215220
glumetleuilegluserthrglymetvalserleuhisargglnasn
225230235240
alaglyvalvalglyglnaspsergluthrilegluleualaproglu
245250255
tyralaglualailealathrargalaglyalaleualaglyileser
260265270
prometpheglnprocysvalvalproprolysprotrpthrglyile
275280285
thrglyglyglytyrtrpalaasnglyargargproleualaleuval
290295300
argthrhisserlyslysalaleumetargtyrgluaspvaltyrmet
305310315320
progluvaltyrlysalaileasnilealaglnasnthralatrplys
325330335
ileasnlyslysvalleualavalalaasnvalilethrlystrplys
340345350
hiscysprovalgluaspileproalailegluargglugluleupro
355360365
metlysprogluaspileaspmetasnproglualaleuthralatrp
370375380
lysargalaalaalaalavaltyrarglysasplysalaarglysser
385390395400
argargileserleugluphemetleugluglnalaasnlyspheala
405410415
asnhislysalailetrppheprotyrasnmetasptrpargglyarg
420425430
valtyralavalsermetpheasnproglnglyasnaspmetthrlys
435440445
glyleuleuthrleualalysglylysproileglylysgluglytyr
450455460
tyrtrpleulysilehisglyalaasncysalaglyvalasplysval
465470475480
propheprogluargilelyspheileglugluasnhisgluasnile
485490495
metalacysalalysserproleugluasnthrtrptrpalaglugln
500505510
aspserprophecyspheleualaphecyspheglutyralaglyval
515520525
glnhishisglyleusertyrasncysserleuproleualapheasp
530535540
glysercysserglyileglnhispheseralametleuargaspglu
545550555560
valglyglyargalavalasnleuleuprosergluthrvalglnasp
565570575
iletyrglyilevalalalyslysvalasngluileleuglnalaasp
580585590
alaileasnglythraspasngluvalvalthrvalthraspgluasn
595600605
thrglygluileserglulysvallysleuglythrlysalaleuala
610615620
glyglntrpleualatyrglyvalthrargtrpvalthrlysargser
625630635640
valmetthrleualatyrglyserlysglupheglypheargglngln
645650655
valleugluthrthrileglntrpalaileaspserglylysglyleu
660665670
metphethrglnproasnglnalaalaglytyrmetalalysleuile
675680685
trpgluservalservalthrvalvalalaalavalglualametasn
690695700
trpleulysseralaalalysleuleualaalagluvallysasplys
705710715720
lysthrglygluileleuarglysargcysalavalhistrpvalthr
725730735
proaspglypheprovaltrpglnglutyrlyslysproileglnthr
740745750
argleuasnleumetpheleuglyglnpheargleuglnprothrile
755760765
asnthrasnlysaspsergluileaspalahislysglnglusergly
770775780
ilealaproasnphevalhisserglnaspglyserhisleuarglys
785790795800
thrvalvaltrpalahisglulystyrglyilegluserphealaleu
805810815
ilehisaspserpheglythrileproalaaspalaalaasnleuphe
820825830
lysalavalarggluthrmetvalaspthrtyrglusercysaspval
835840845
leualaaspphetyraspglnphealaaspglnleuhisglusergln
850855860
leuasplysmetproalaleuproalalysglyasnleuasnleuarg
865870875880
aspileleugluseraspphealapheala
885890
<210>18
<211>2673
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>18
atgcaccatcaccatcaccatatgaacacgattaacatcgctaagaacgacttctctgac60
atcgaactggctgctatcccgttcaacactctggctgaccattacggtgagcgtttagct120
cgcgaacagttggcccttgagcatgagtcttacgagatgggtgaagcacgcttccgcaag180
atgtttgagcgtcaacttaaagctggtgaggttgcggataacgctgccgccaagcctctc240
atcactaccctactccctaagatgattgcacgcatcaacgactggtttgaggaagtgaaa300
gctaagcgcggcaagcgcccgacagccttccagttcctgcaagaaatcaagccggaagcc360
gtagcgtacatcaccattaagaccactctggcttgcctaaccagtgctgataatacaacc420
gttcaggctgtagcaagcgcaatcggtcgggccattgaggacgaggctcgcttcggtcgt480
atccgtgaccttgaagctaagcacttcaagaaaaacgttgaggaacaactcaacaagcgc540
gtagggcacgtctacaagaaagcatttatgcaagttgtcgaggctgacatgctctctaag600
ggtctactcggtggcgaggcgtggtcttcgtggcataaggaagactctattcatgtagga660
gtacgctgcatcgagatgctcattgagtcaaccggaatggttagcttacaccgccaaaat720
gctggcgtagtaggtcaagactctgagactatcgaactcgcacctgaatacgctgaggct780
atcgcaacccgtgcaggtgcgctggctggcatctctccgatgttccaaccttgcgtagtt840
cctcctaagccgtggactggcattactggtggtggctattgggctaacggtcgtcgtcct900
ctggcgctggtgcgtactcacagtaagaaagcactgatgcgctacgaagacgtttacatg960
cctgaggtgtacaaagcgattaacattgcgcaaaacaccgcatggaaaatcaacaagaaa1020
gtcctagcggtcgccaacgtaatcaccaagtggaagcattgtccggtcgaggacatccct1080
gcgattgagcgtgaagaactcccgatgaaaccggaagacatcgacatgaatcctgaggct1140
ctcaccgcgtggaaacgtgctgccgctgctgtgtaccgcaaggacaaggctcgcaagtct1200
cgccgtatcagccttgagttcatgcttgagcaagccaataagtttgctaaccataaggcc1260
atctggttcccttacaacatggactggcgcggtcgtgtttacgctgtgtcaatgttcaac1320
ccgcaaggtaacgatatgaccaaaggactgcttacgctggcgaaaggtaaaccaatcggt1380
aaggaaggttactactggctgaaaatccacggtgcaaactgtgcgggtgtcgataaggtt1440
ccgttccctgagcgcatcaagttcattgaggaaaaccacgagaacatcatggcttgcgct1500
aagtctccactggagaacacttggtgggctgagcaattgtctccgttctgcttccttgcg1560
ttctgctttgagtacgctggggtacagcaccacggcctgagctataactgctcccttccg1620
ctggcgtttgacgggtcttgctctggcatccagcacttctccgcgatgctccgagatgag1680
gtaggtggtcgcgcggttaacttgcttcctagtgaaaccgttcaggacatctacgggatt1740
gttgctaagaaagtcaacgagattctacaagcagacgcaatcaatgggaccgataacgaa1800
gtagttaccgtgaccgatgagaacactggtgaaatctctgagaaagtcaagctgggcact1860
aaggcactggctggtcaatggctggcttacggtgttactcgcagtgtgactaagcgttca1920
gtcatgacgctggcttacgggtccaaagagttcggcttccgtcaacaagtgctggaatgg1980
accattcagtgggctattgattccggcaagggtctgatgttcactcagccgaatcaggct2040
gctggatacatggctaagctgatttgggaatctgtgagcgtgacggtggtagctgcggtt2100
gaagcaatgaactggcttaagtctgctgctaagctgctggctgctgaggtcaaagataag2160
aagactggagagattcttcgcaagcgttgcgctgtgcattgggtaactcctgatggtttc2220
cctgtgtggcaggaatacaagaagcctattcagacgcgcttgaacctgatgttcctcggt2280
cagttccgcttacagcctaccattaacaccaacaaagatagcgagattgatgcacacaaa2340
caggagtctggtatcgctcctaactttgtacacagccaagacggtagccaccttcgtaag2400
actgtagtgtgggcacacgagaagtacggaatcgaatcttttgcactgattcacgactcc2460
ttcggtaccattccggctgacgctgcgaacctgttcaaagcagtgcgcgaaactatggtt2520
gacacatatgagtcttgtgatgtactggctgatttctacgaccagttcgctgaccagttg2580
cacgagtctcaattggacaaaatgccagcacttccggctaaaggtaacttgaacctccgt2640
gacatcttagagtcggacttcgcgttcgcgtaa2673
<210>19
<211>890
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>19
methishishishishishismetasnthrileasnilealalysasn
151015
asppheseraspilegluleualaalailepropheasnthrleuala
202530
asphistyrglygluargleualaarggluglnleualaleugluhis
354045
glusertyrglumetglyglualaargphearglysmetphegluarg
505560
glnleulysalaglygluvalalaaspasnalaalaalalysproleu
65707580
ilethrthrleuleuprolysmetilealaargileasnasptrpphe
859095
glugluvallysalalysargglylysargprothralapheglnphe
100105110
leuglngluilelysproglualavalalatyrilethrilelysthr
115120125
thrleualacysleuthrseralaaspasnthrthrvalglnalaval
130135140
alaseralaileglyargalailegluaspglualaargpheglyarg
145150155160
ileargaspleuglualalyshisphelyslysasnvalgluglugln
165170175
leuasnlysargvalglyhisvaltyrlyslysalaphemetglnval
180185190
valglualaaspmetleuserlysglyleuleuglyglyglualatrp
195200205
sersertrphislysgluaspserilehisvalglyvalargcysile
210215220
glumetleuilegluserthrglymetvalserleuhisargglnasn
225230235240
alaglyvalvalglyglnaspsergluthrilegluleualaproglu
245250255
tyralaglualailealathrargalaglyalaleualaglyileser
260265270
prometpheglnprocysvalvalproprolysprotrpthrglyile
275280285
thrglyglyglytyrtrpalaasnglyargargproleualaleuval
290295300
argthrhisserlyslysalaleumetargtyrgluaspvaltyrmet
305310315320
progluvaltyrlysalaileasnilealaglnasnthralatrplys
325330335
ileasnlyslysvalleualavalalaasnvalilethrlystrplys
340345350
hiscysprovalgluaspileproalailegluargglugluleupro
355360365
metlysprogluaspileaspmetasnproglualaleuthralatrp
370375380
lysargalaalaalaalavaltyrarglysasplysalaarglysser
385390395400
argargileserleugluphemetleugluglnalaasnlyspheala
405410415
asnhislysalailetrppheprotyrasnmetasptrpargglyarg
420425430
valtyralavalsermetpheasnproglnglyasnaspmetthrlys
435440445
glyleuleuthrleualalysglylysproileglylysgluglytyr
450455460
tyrtrpleulysilehisglyalaasncysalaglyvalasplysval
465470475480
propheprogluargilelyspheileglugluasnhisgluasnile
485490495
metalacysalalysserproleugluasnthrtrptrpalaglugln
500505510
leuserprophecyspheleualaphecyspheglutyralaglyval
515520525
glnhishisglyleusertyrasncysserleuproleualapheasp
530535540
glysercysserglyileglnhispheseralametleuargaspglu
545550555560
valglyglyargalavalasnleuleuprosergluthrvalglnasp
565570575
iletyrglyilevalalalyslysvalasngluileleuglnalaasp
580585590
alaileasnglythraspasngluvalvalthrvalthraspgluasn
595600605
thrglygluileserglulysvallysleuglythrlysalaleuala
610615620
glyglntrpleualatyrglyvalthrargservalthrlysargser
625630635640
valmetthrleualatyrglyserlysglupheglypheargglngln
645650655
valleuglutrpthrileglntrpalaileaspserglylysglyleu
660665670
metphethrglnproasnglnalaalaglytyrmetalalysleuile
675680685
trpgluservalservalthrvalvalalaalavalglualametasn
690695700
trpleulysseralaalalysleuleualaalagluvallysasplys
705710715720
lysthrglygluileleuarglysargcysalavalhistrpvalthr
725730735
proaspglypheprovaltrpglnglutyrlyslysproileglnthr
740745750
argleuasnleumetpheleuglyglnpheargleuglnprothrile
755760765
asnthrasnlysaspsergluileaspalahislysglnglusergly
770775780
ilealaproasnphevalhisserglnaspglyserhisleuarglys
785790795800
thrvalvaltrpalahisglulystyrglyilegluserphealaleu
805810815
ilehisaspserpheglythrileproalaaspalaalaasnleuphe
820825830
lysalavalarggluthrmetvalaspthrtyrglusercysaspval
835840845
leualaaspphetyraspglnphealaaspglnleuhisglusergln
850855860
leuasplysmetproalaleuproalalysglyasnleuasnleuarg
865870875880
aspileleugluseraspphealapheala
885890
<210>20
<211>2673
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>20
atgcaccatcaccatcaccatatgaacacgattaacatcgctaagaacgacttctctgac60
atcgaactggctgctatcccgttcaacactctggctgaccattacggtgagcgtttagct120
cgcgaacagttggcccttgagcatgagtcttacgagatgggtgaagcacgcttccgcaag180
atgtttgagcgtcaacttaaagctggtgaggttgcggataacgctgccgccaagcctctc240
atcactaccctactccctaagatgattgcacgcatcaacgactggtttgaggaagtgaaa300
gctaagcgcggcaagcgcccgacagccttccagttcctgcaagaaatcaagccggaagcc360
gtagcgtacatcaccattaagaccactctggcttgcctaaccagtgcttggaatacaacc420
gttcaggctgtagcaagcgcaatcggtcgggccattgaggacgaggctcgcttcggtcgt480
atccgtgaccttgaagctaagcacttcaagaaaaacgttgaggaacaactcaacaagcgc540
gtagggcacgtctacaagaaagcatttatgcaagttgtcgaggctgacatgctctctaag600
ggtctactcggtggcgaggcgtggtcttcgtggcataaggaagactctattcatgtagga660
gtacgctgcatcgagatgctcattgagtcaaccggaatggttagcttacaccgccaaaat720
gctggcgtagtaggtcaagactctgagactatcgaactcgcacctgaatacgctgaggct780
atcgcaacccgtgcaggtgcgctggctggcatctctccgatgttccaaccttgcgtagtt840
cctcctaagccgtggactggcattactggtggtggctattgggctaacggtcgtcgtcct900
ctggcgctggtgcgtactcacagtaagaaagcactgatgcgctacgaagacgtttacatg960
cctgaggtgtacaaagcgattaacattgcgcaaaacaccgcatggaaaatcaacaagaaa1020
gtcctagcggtcgccaacgtaatcaccaagtggaagcattgtccggtcgaggacatccct1080
gcgattgagcgtgaagaactcccgatgaaaccggaagacatcgacatgaatcctgaggct1140
ctcaccgcgtggaaacgtgctgccgctgctgtgtaccgcaaggacaaggctcgcaagtct1200
agtcgtatcagccttgagttcatgcttgagcaagccaataagtttgctaaccataaggcc1260
atctggttcccttacaacatggactggcgcggtcgtgtttacgctgtgtcaatgttcaac1320
ccgcaaggtaacgatatgaccaaaggactgcttacgctggcgaaaggtaaaccaatcggt1380
aaggaaggttactactggctgaaaatccacggtgcaaactgtgcgggtgtcgataaggtt1440
ccgttccctgagcgcatcaagttcattgaggaaaaccacgagaacatcatggcttgcgct1500
aagtctccactggagaacacttggtgggctgagcaacggtctccgttctgcttccttgcg1560
ttctgctttgagtacgctggggtacagcaccacggcctgagctataactgctcccttccg1620
ctggcgtttgacgggtcttgctctggcatccagcacttctccgcgatgctccgagatgag1680
gtaggtggtcgcgcggttaacttgcttcctagtgaaaccgttcaggacatctacgggatt1740
gttgctaagaaagtcaacgagattctacaagcagacgcaatcaatgggaccgataacgaa1800
gtagttaccgtgaccgatgagaacactggtgaaatctctgagaaagtcaagctgggcact1860
aaggcactggctggtcaatggctggcttacggtgttactcgcagtgtgactaagcgttca1920
gtcatgacgctggcttacgggtccaaagagttcggcttccgtcaacaagtgctggaagat1980
accattcagtgggctattgattccggcaagggtctgatgttcactcagccgaatcaggct2040
gctggatacatggctaagctgatttgggaatctgtgagcgtgacggtggtagctgcggtt2100
gaagcaatgaactggcttaagtctgctgctaagctgctggctgctgaggtcaaagataag2160
aagactggagagattcttcgcaagcgttgcgctgtgcattgggtaactcctgatggtttc2220
cctgtgtggcaggaatacaagaagcctattcagacgcgcttgaacctgatgttcctcggt2280
cagttccgcttacagcctaccattaacaccaacaaagatagcgagattgatgcacacaaa2340
caggagtctggtatcgctcctaactttgtacacagccaagacggtagccaccttcgtaag2400
actgtagtgtgggcacacgagaagtacggaatcgaatcttttgcactgattcacgactcc2460
ttcggtaccattccggctgacgctgcgaacctgttcaaagcagtgcgcgaaactatggtt2520
gacacatatgagtcttgtgatgtactggctgatttctacgaccagttcgctgaccagttg2580
cacgagtctcaattggacaaaatgccagcacttccggctaaaggtaacttgaacctccgt2640
gacatcttagagtcggacttcgcgttcgcgtaa2673
<210>21
<211>890
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>21
methishishishishishismetasnthrileasnilealalysasn
151015
asppheseraspilegluleualaalailepropheasnthrleuala
202530
asphistyrglygluargleualaarggluglnleualaleugluhis
354045
glusertyrglumetglyglualaargphearglysmetphegluarg
505560
glnleulysalaglygluvalalaaspasnalaalaalalysproleu
65707580
ilethrthrleuleuprolysmetilealaargileasnasptrpphe
859095
glugluvallysalalysargglylysargprothralapheglnphe
100105110
leuglngluilelysproglualavalalatyrilethrilelysthr
115120125
thrleualacysleuthrseralatrpasnthrthrvalglnalaval
130135140
alaseralaileglyargalailegluaspglualaargpheglyarg
145150155160
ileargaspleuglualalyshisphelyslysasnvalgluglugln
165170175
leuasnlysargvalglyhisvaltyrlyslysalaphemetglnval
180185190
valglualaaspmetleuserlysglyleuleuglyglyglualatrp
195200205
sersertrphislysgluaspserilehisvalglyvalargcysile
210215220
glumetleuilegluserthrglymetvalserleuhisargglnasn
225230235240
alaglyvalvalglyglnaspsergluthrilegluleualaproglu
245250255
tyralaglualailealathrargalaglyalaleualaglyileser
260265270
prometpheglnprocysvalvalproprolysprotrpthrglyile
275280285
thrglyglyglytyrtrpalaasnglyargargproleualaleuval
290295300
argthrhisserlyslysalaleumetargtyrgluaspvaltyrmet
305310315320
progluvaltyrlysalaileasnilealaglnasnthralatrplys
325330335
ileasnlyslysvalleualavalalaasnvalilethrlystrplys
340345350
hiscysprovalgluaspileproalailegluargglugluleupro
355360365
metlysprogluaspileaspmetasnproglualaleuthralatrp
370375380
lysargalaalaalaalavaltyrarglysasplysalaarglysser
385390395400
serargileserleugluphemetleugluglnalaasnlyspheala
405410415
asnhislysalailetrppheprotyrasnmetasptrpargglyarg
420425430
valtyralavalsermetpheasnproglnglyasnaspmetthrlys
435440445
glyleuleuthrleualalysglylysproileglylysgluglytyr
450455460
tyrtrpleulysilehisglyalaasncysalaglyvalasplysval
465470475480
propheprogluargilelyspheileglugluasnhisgluasnile
485490495
metalacysalalysserproleugluasnthrtrptrpalaglugln
500505510
argserprophecyspheleualaphecyspheglutyralaglyval
515520525
glnhishisglyleusertyrasncysserleuproleualapheasp
530535540
glysercysserglyileglnhispheseralametleuargaspglu
545550555560
valglyglyargalavalasnleuleuprosergluthrvalglnasp
565570575
iletyrglyilevalalalyslysvalasngluileleuglnalaasp
580585590
alaileasnglythraspasngluvalvalthrvalthraspgluasn
595600605
thrglygluileserglulysvallysleuglythrlysalaleuala
610615620
glyglntrpleualatyrglyvalthrargservalthrlysargser
625630635640
valmetthrleualatyrglyserlysglupheglypheargglngln
645650655
valleugluaspthrileglntrpalaileaspserglylysglyleu
660665670
metphethrglnproasnglnalaalaglytyrmetalalysleuile
675680685
trpgluservalservalthrvalvalalaalavalglualametasn
690695700
trpleulysseralaalalysleuleualaalagluvallysasplys
705710715720
lysthrglygluileleuarglysargcysalavalhistrpvalthr
725730735
proaspglypheprovaltrpglnglutyrlyslysproileglnthr
740745750
argleuasnleumetpheleuglyglnpheargleuglnprothrile
755760765
asnthrasnlysaspsergluileaspalahislysglnglusergly
770775780
ilealaproasnphevalhisserglnaspglyserhisleuarglys
785790795800
thrvalvaltrpalahisglulystyrglyilegluserphealaleu
805810815
ilehisaspserpheglythrileproalaaspalaalaasnleuphe
820825830
lysalavalarggluthrmetvalaspthrtyrglusercysaspval
835840845
leualaaspphetyraspglnphealaaspglnleuhisglusergln
850855860
leuasplysmetproalaleuproalalysglyasnleuasnleuarg
865870875880
aspileleugluseraspphealapheala
885890
<210>22
<211>2673
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>22
atgcaccatcaccatcaccatatgaacacgattaacatcgctaagaacgacttctctgac60
atcgaactggctgctatcccgttcaacactctggctgaccattacggtgagcgtttagct120
cgcgaacagttggcccttgagcatgagtcttacgagatgggtgaagcacgcttccgcaag180
atgtttgagcgtcaacttaaagctggtgaggttgcggataacgctgccgccaagcctctc240
atcactaccctactccctaagatgattgcacgcatcaacgactggtttgaggaagtgaaa300
gctaagcgcggcaagcgcccgacagccttccagttcctgcaagaaatcaagccggaagcc360
gtagcgtacatcaccattaagaccactctggcttgcctaaccagtgcttggaatacaacc420
gttcaggctgtagcaagcgcaatcggtcgggccattgaggacgaggctcgcttcggtcgt480
atccgtgaccttgaagctaagcacttcaagaaaaacgttgaggaacaactcaacaagcgc540
gtagggcacgtctacaagaaagcatttatgcaagttgtcgaggctgacatgctctctaag600
ggtctactcggtggcgaggcgtggtcttcgtggcataaggaagactctattcatgtagga660
gtacgctgcatcgagatgctcattgagtcaaccggaatggttagcttacaccgccaaaat720
gctggcgtagtaggtcaagactctgagactatcgaactcgcacctgaatacgctgaggct780
atcgcaacccgtgcaggtgcgctggctggcatctctccgatgttccaaccttgcgtagtt840
cctcctaagccgtggactggcattactggtggtggctattgggctaacggtcgtcgtcct900
ctggcgctggtgcgtactcacagtaagaaagcactgatgcgctacgaagacgtttacatg960
cctgaggtgtacaaagcgattaacattgcgcaaaacaccgcatggaaaatcaacaagaaa1020
gtcctagcggtcgccaacgtaatcaccaagtggaagcattgtccggtcgaggacatccct1080
gcgattgagcgtgaagaactcccgatgaaaccggaagacatcgacatgaatcctgaggct1140
ctcaccgcgtggaaacgtgctgccgctgctgtgtaccgcaaggacaaggctcgcaagtct1200
agacgtatcagccttgagttcatgcttgagcaagccaataagtttgctaaccataaggcc1260
atctggttcccttacaacatggactggcgcggtcgtgtttacgctgtgtcaatgttcaac1320
ccgcaaggtaacgatatgaccaaaggactgcttacgctggcgaaaggtaaaccaatcggt1380
aaggaaggttactactggctgaaaatccacggtgcaaactgtgcgggtgtcgataaggtt1440
ccgttccctgagcgcatcaagttcattgaggaaaaccacgagaacatcatggcttgcgct1500
aagtctccactggagaacacttggtgggctgagcaatggtctccgttctgcttccttgcg1560
ttctgctttgagtacgctggggtacagcaccacggcctgagctataactgctcccttccg1620
ctggcgtttgacgggtcttgctctggcatccagcacttctccgcgatgctccgagatgag1680
gtaggtggtcgcgcggttaacttgcttcctagtgaaaccgttcaggacatctacgggatt1740
gttgctaagaaagtcaacgagattctacaagcagacgcaatcaatgggaccgataacgaa1800
gtagttaccgtgaccgatgagaacactggtgaaatctctgagaaagtcaagctgggcact1860
aaggcactggctggtcaatggctggcttacggtgttactcgcagtgtgactaagcgttca1920
gtcatgacgctggcttacgggtccaaagagttcggcttccgtcaacaagtgctggaagat1980
accattcagtgggctattgattccggcaagggtctgatgttcactcagccgaatcaggct2040
gctggatacatggctaagctgatttgggaatctgtgagcgtgacggtggtagctgcggtt2100
gaagcaatgaactggcttaagtctgctgctaagctgctggctgctgaggtcaaagataag2160
aagactggagagattcttcgcaagcgttgcgctgtgcattgggtaactcctgatggtttc2220
cctgtgtggcaggaatacaagaagcctattcagacgcgcttgaacctgatgttcctcggt2280
cagttccgcttacagcctaccattaacaccaacaaagatagcgagattgatgcacacaaa2340
caggagtctggtatcgctcctaactttgtacacagccaagacggtagccaccttcgtaag2400
actgtagtgtgggcacacgagaagtacggaatcgaatcttttgcactgattcacgactcc2460
ttcggtaccattccggctgacgctgcgaacctgttcaaagcagtgcgcgaaactatggtt2520
gacacatatgagtcttgtgatgtactggctgatttctacgaccagttcgctgaccagttg2580
cacgagtctcaattggacaaaatgccagcacttccggctaaaggtaacttgaacctccgt2640
gacatcttagagtcggacttcgcgttcgcgtaa2673
<210>23
<211>890
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>23
methishishishishishismetasnthrileasnilealalysasn
151015
asppheseraspilegluleualaalailepropheasnthrleuala
202530
asphistyrglygluargleualaarggluglnleualaleugluhis
354045
glusertyrglumetglyglualaargphearglysmetphegluarg
505560
glnleulysalaglygluvalalaaspasnalaalaalalysproleu
65707580
ilethrthrleuleuprolysmetilealaargileasnasptrpphe
859095
glugluvallysalalysargglylysargprothralapheglnphe
100105110
leuglngluilelysproglualavalalatyrilethrilelysthr
115120125
thrleualacysleuthrseralatrpasnthrthrvalglnalaval
130135140
alaseralaileglyargalailegluaspglualaargpheglyarg
145150155160
ileargaspleuglualalyshisphelyslysasnvalgluglugln
165170175
leuasnlysargvalglyhisvaltyrlyslysalaphemetglnval
180185190
valglualaaspmetleuserlysglyleuleuglyglyglualatrp
195200205
sersertrphislysgluaspserilehisvalglyvalargcysile
210215220
glumetleuilegluserthrglymetvalserleuhisargglnasn
225230235240
alaglyvalvalglyglnaspsergluthrilegluleualaproglu
245250255
tyralaglualailealathrargalaglyalaleualaglyileser
260265270
prometpheglnprocysvalvalproprolysprotrpthrglyile
275280285
thrglyglyglytyrtrpalaasnglyargargproleualaleuval
290295300
argthrhisserlyslysalaleumetargtyrgluaspvaltyrmet
305310315320
progluvaltyrlysalaileasnilealaglnasnthralatrplys
325330335
ileasnlyslysvalleualavalalaasnvalilethrlystrplys
340345350
hiscysprovalgluaspileproalailegluargglugluleupro
355360365
metlysprogluaspileaspmetasnproglualaleuthralatrp
370375380
lysargalaalaalaalavaltyrarglysasplysalaarglysser
385390395400
argargileserleugluphemetleugluglnalaasnlyspheala
405410415
asnhislysalailetrppheprotyrasnmetasptrpargglyarg
420425430
valtyralavalsermetpheasnproglnglyasnaspmetthrlys
435440445
glyleuleuthrleualalysglylysproileglylysgluglytyr
450455460
tyrtrpleulysilehisglyalaasncysalaglyvalasplysval
465470475480
propheprogluargilelyspheileglugluasnhisgluasnile
485490495
metalacysalalysserproleugluasnthrtrptrpalaglugln
500505510
trpserprophecyspheleualaphecyspheglutyralaglyval
515520525
glnhishisglyleusertyrasncysserleuproleualapheasp
530535540
glysercysserglyileglnhispheseralametleuargaspglu
545550555560
valglyglyargalavalasnleuleuprosergluthrvalglnasp
565570575
iletyrglyilevalalalyslysvalasngluileleuglnalaasp
580585590
alaileasnglythraspasngluvalvalthrvalthraspgluasn
595600605
thrglygluileserglulysvallysleuglythrlysalaleuala
610615620
glyglntrpleualatyrglyvalthrargservalthrlysargser
625630635640
valmetthrleualatyrglyserlysglupheglypheargglngln
645650655
valleugluaspthrileglntrpalaileaspserglylysglyleu
660665670
metphethrglnproasnglnalaalaglytyrmetalalysleuile
675680685
trpgluservalservalthrvalvalalaalavalglualametasn
690695700
trpleulysseralaalalysleuleualaalagluvallysasplys
705710715720
lysthrglygluileleuarglysargcysalavalhistrpvalthr
725730735
proaspglypheprovaltrpglnglutyrlyslysproileglnthr
740745750
argleuasnleumetpheleuglyglnpheargleuglnprothrile
755760765
asnthrasnlysaspsergluileaspalahislysglnglusergly
770775780
ilealaproasnphevalhisserglnaspglyserhisleuarglys
785790795800
thrvalvaltrpalahisglulystyrglyilegluserphealaleu
805810815
ilehisaspserpheglythrileproalaaspalaalaasnleuphe
820825830
lysalavalarggluthrmetvalaspthrtyrglusercysaspval
835840845
leualaaspphetyraspglnphealaaspglnleuhisglusergln
850855860
leuasplysmetproalaleuproalalysglyasnleuasnleuarg
865870875880
aspileleugluseraspphealapheala
885890
<210>24
<211>2673
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>24
atgcaccatcaccatcaccatatgaacacgattaacatcgctaagaacgacttctctgac60
atcgaactggctgctatcccgttcaacactctggctgaccattacggtgagcgtttagct120
cgcgaacagttggcccttgagcatgagtcttacgagatgggtgaagcacgcttccgcaag180
atgtttgagcgtcaacttaaagctggtgaggttgcggataacgctgccgccaagcctctc240
atcactaccctactccctaagatgattgcacgcatcaacgactggtttgaggaagtgaaa300
gctaagcgcggcaagcgcccgacagccttccagttcctgcaagaaatcaagccggaagcc360
gtagcgtacatcaccattaagaccactctggcttgcctaaccagtgcttggaatacaacc420
gttcaggctgtagcaagcgcaatcggtcgggccattgaggacgaggctcgcttcggtcgt480
atccgtgaccttgaagctaagcacttcaagaaaaacgttgaggaacaactcaacaagcgc540
gtagggcacgtctacaagaaagcatttatgcaagttgtcgaggctgacatgctctctaag600
ggtctactcggtggcgaggcgtggtcttcgtggcataaggaagactctattcatgtagga660
gtacgctgcatcgagatgctcattgagtcaaccggaatggttagcttacaccgccaaaat720
gctggcgtagtaggtcaagactctgagactatcgaactcgcacctgaatacgctgaggct780
atcgcaacccgtgcaggtgcgctggctggcatctctccgatgttccaaccttgcgtagtt840
cctcctaagccgtggactggcattactggtggtggctattgggctaacggtcgtcgtcct900
ctggcgctggtgcgtactcacagtaagaaagcactgatgcgctacgaagacgtttacatg960
cctgaggtgtacaaagcgattaacattgcgcaaaacaccgcatggaaaatcaacaagaaa1020
gtcctagcggtcgccaacgtaatcaccaagtggaagcattgtccggtcgaggacatccct1080
gcgattgagcgtgaagaactcccgatgaaaccggaagacatcgacatgaatcctgaggct1140
ctcaccgcgtggaaacgtgctgccgctgctgtgtaccgcaaggacaaggctcgcaagtct1200
agtcgtatcagccttgagttcatgcttgagcaagccaataagtttgctaaccataaggcc1260
atctggttcccttacaacatggactggcgcggtcgtgtttacgctgtgtcaatgttcaac1320
ccgcaaggtaacgatatgaccaaaggactgcttacgctggcgaaaggtaaaccaatcggt1380
aaggaaggttactactggctgaaaatccacggtgcaaactgtgcgggtgtcgataaggtt1440
ccgttccctgagcgcatcaagttcattgaggaaaaccacgagaacatcatggcttgcgct1500
aagtctccactggagaacacttggtgggctgagcaatggtctccgttctgcttccttgcg1560
ttctgctttgagtacgctggggtacagcaccacggcctgagctataactgctcccttccg1620
ctggcgtttgacgggtcttgctctggcatccagcacttctccgcgatgctccgagatgag1680
gtaggtggtcgcgcggttaacttgcttcctagtgaaaccgttcaggacatctacgggatt1740
gttgctaagaaagtcaacgagattctacaagcagacgcaatcaatgggaccgataacgaa1800
gtagttaccgtgaccgatgagaacactggtgaaatctctgagaaagtcaagctgggcact1860
aaggcactggctggtcaatggctggcttacggtgttactcgcagtgtgactaagcgttca1920
gtcatgacgctggcttacgggtccaaagagttcggcttccgtcaacaagtgctggaagat1980
accattcagtgggctattgattccggcaagggtctgatgttcactcagccgaatcaggct2040
gctggatacatggctaagctgatttgggaatctgtgagcgtgacggtggtagctgcggtt2100
gaagcaatgaactggcttaagtctgctgctaagctgctggctgctgaggtcaaagataag2160
aagactggagagattcttcgcaagcgttgcgctgtgcattgggtaactcctgatggtttc2220
cctgtgtggcaggaatacaagaagcctattcagacgcgcttgaacctgatgttcctcggt2280
cagttccgcttacagcctaccattaacaccaacaaagatagcgagattgatgcacacaaa2340
caggagtctggtatcgctcctaactttgtacacagccaagacggtagccaccttcgtaag2400
actgtagtgtgggcacacgagaagtacggaatcgaatcttttgcactgattcacgactcc2460
ttcggtaccattccggctgacgctgcgaacctgttcaaagcagtgcgcgaaactatggtt2520
gacacatatgagtcttgtgatgtactggctgatttctacgaccagttcgctgaccagttg2580
cacgagtctcaattggacaaaatgccagcacttccggctaaaggtaacttgaacctccgt2640
gacatcttagagtcggacttcgcgttcgcgtaa2673
<210>25
<211>890
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>25
methishishishishishismetasnthrileasnilealalysasn
151015
asppheseraspilegluleualaalailepropheasnthrleuala
202530
asphistyrglygluargleualaarggluglnleualaleugluhis
354045
glusertyrglumetglyglualaargphearglysmetphegluarg
505560
glnleulysalaglygluvalalaaspasnalaalaalalysproleu
65707580
ilethrthrleuleuprolysmetilealaargileasnasptrpphe
859095
glugluvallysalalysargglylysargprothralapheglnphe
100105110
leuglngluilelysproglualavalalatyrilethrilelysthr
115120125
thrleualacysleuthrseralatrpasnthrthrvalglnalaval
130135140
alaseralaileglyargalailegluaspglualaargpheglyarg
145150155160
ileargaspleuglualalyshisphelyslysasnvalgluglugln
165170175
leuasnlysargvalglyhisvaltyrlyslysalaphemetglnval
180185190
valglualaaspmetleuserlysglyleuleuglyglyglualatrp
195200205
sersertrphislysgluaspserilehisvalglyvalargcysile
210215220
glumetleuilegluserthrglymetvalserleuhisargglnasn
225230235240
alaglyvalvalglyglnaspsergluthrilegluleualaproglu
245250255
tyralaglualailealathrargalaglyalaleualaglyileser
260265270
prometpheglnprocysvalvalproprolysprotrpthrglyile
275280285
thrglyglyglytyrtrpalaasnglyargargproleualaleuval
290295300
argthrhisserlyslysalaleumetargtyrgluaspvaltyrmet
305310315320
progluvaltyrlysalaileasnilealaglnasnthralatrplys
325330335
ileasnlyslysvalleualavalalaasnvalilethrlystrplys
340345350
hiscysprovalgluaspileproalailegluargglugluleupro
355360365
metlysprogluaspileaspmetasnproglualaleuthralatrp
370375380
lysargalaalaalaalavaltyrarglysasplysalaarglysser
385390395400
serargileserleugluphemetleugluglnalaasnlyspheala
405410415
asnhislysalailetrppheprotyrasnmetasptrpargglyarg
420425430
valtyralavalsermetpheasnproglnglyasnaspmetthrlys
435440445
glyleuleuthrleualalysglylysproileglylysgluglytyr
450455460
tyrtrpleulysilehisglyalaasncysalaglyvalasplysval
465470475480
propheprogluargilelyspheileglugluasnhisgluasnile
485490495
metalacysalalysserproleugluasnthrtrptrpalaglugln
500505510
trpserprophecyspheleualaphecyspheglutyralaglyval
515520525
glnhishisglyleusertyrasncysserleuproleualapheasp
530535540
glysercysserglyileglnhispheseralametleuargaspglu
545550555560
valglyglyargalavalasnleuleuprosergluthrvalglnasp
565570575
iletyrglyilevalalalyslysvalasngluileleuglnalaasp
580585590
alaileasnglythraspasngluvalvalthrvalthraspgluasn
595600605
thrglygluileserglulysvallysleuglythrlysalaleuala
610615620
glyglntrpleualatyrglyvalthrargservalthrlysargser
625630635640
valmetthrleualatyrglyserlysglupheglypheargglngln
645650655
valleugluaspthrileglntrpalaileaspserglylysglyleu
660665670
metphethrglnproasnglnalaalaglytyrmetalalysleuile
675680685
trpgluservalservalthrvalvalalaalavalglualametasn
690695700
trpleulysseralaalalysleuleualaalagluvallysasplys
705710715720
lysthrglygluileleuarglysargcysalavalhistrpvalthr
725730735
proaspglypheprovaltrpglnglutyrlyslysproileglnthr
740745750
argleuasnleumetpheleuglyglnpheargleuglnprothrile
755760765
asnthrasnlysaspsergluileaspalahislysglnglusergly
770775780
ilealaproasnphevalhisserglnaspglyserhisleuarglys
785790795800
thrvalvaltrpalahisglulystyrglyilegluserphealaleu
805810815
ilehisaspserpheglythrileproalaaspalaalaasnleuphe
820825830
lysalavalarggluthrmetvalaspthrtyrglusercysaspval
835840845
leualaaspphetyraspglnphealaaspglnleuhisglusergln
850855860
leuasplysmetproalaleuproalalysglyasnleuasnleuarg
865870875880
aspileleugluseraspphealapheala
885890
<210>26
<211>2673
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>26
atgcaccatcaccatcaccatatgaacacgattaacatcgctaagaacgacttctctgac60
atcgaactggctgctatcccgttcaacactctggctgaccattacggtgagcgtttagct120
cgcgaacagttggcccttgagcatgagtcttacgagatgggtgaagcacgcttccgcaag180
atgtttgagcgtcaacttaaagctggtgaggttgcggataacgctgccgccaagcctctc240
atcactaccctactccctaagatgattgcacgcatcaacgactggtttgaggaagtgaaa300
gctaagcgcggcaagcgcccgacagccttccagttcctgcaagaaatcaagccggaagcc360
gtagcgtacatcaccattaagaccactctggcttgcctaaccagtgcttggaatacaacc420
gttcaggctgtagcaagcgcaatcggtcgggccattgaggacgaggctcgcttcggtcgt480
atccgtgaccttgaagctaagcacttcaagaaaaacgttgaggaacaactcaacaagcgc540
gtagggcacgtctacaagaaagcatttatgcaagttgtcgaggctgacatgctctctaag600
ggtctactcggtggcgaggcgtggtcttcgtggcataaggaagactctattcatgtagga660
gtacgctgcatcgagatgctcattgagtcaaccggaatggttagcttacaccgccaaaat720
gctggcgtagtaggtcaagactctgagactatcgaactcgcacctgaatacgctgaggct780
atcgcaacccgtgcaggtgcgctggctggcatctctccgatgttccaaccttgcgtagtt840
cctcctaagccgtggactggcattactggtggtggctattgggctaacggtcgtcgtcct900
ctggcgctggtgcgtactcacagtaagaaagcactgatgcgctacgaagacgtttacatg960
cctgaggtgtacaaagcgattaacattgcgcaaaacaccgcatggaaaatcaacaagaaa1020
gtcctagcggtcgccaacgtaatcaccaagtggaagcattgtccggtcgaggacatccct1080
gcgattgagcgtgaagaactcccgatgaaaccggaagacatcgacatgaatcctgaggct1140
ctcaccgcgtggaaacgtgctgccgctgctgtgtaccgcaaggacaaggctcgcaagtct1200
atacgtatcagccttgagttcatgcttgagcaagccaataagtttgctaaccataaggcc1260
atctggttcccttacaacatggactggcgcggtcgtgtttacgctgtgtcaatgttcaac1320
ccgcaaggtaacgatatgaccaaaggactgcttacgctggcgaaaggtaaaccaatcggt1380
aaggaaggttactactggctgaaaatccacggtgcaaactgtgcgggtgtcgataaggtt1440
ccgttccctgagcgcatcaagttcattgaggaaaaccacgagaacatcatggcttgcgct1500
aagtctccactggagaacacttggtgggctgagcaagattctccgttctgcttccttgcg1560
ttctgctttgagtacgctggggtacagcaccacggcctgagctataactgctcccttccg1620
ctggcgtttgacgggtcttgctctggcatccagcacttctccgcgatgctccgagatgag1680
gtaggtggtcgcgcggttaacttgcttcctagtgaaaccgttcaggacatctacgggatt1740
gttgctaagaaagtcaacgagattctacaagcagacgcaatcaatgggaccgataacgaa1800
gtagttaccgtgaccgatgagaacactggtgaaatctctgagaaagtcaagctgggcact1860
aaggcactggctggtcaatggctggcttacggtgttactcgcagtgtgactaagcgttca1920
gtcatgacgctggcttacgggtccaaagagttcggcttccgtcaacaagtgctggaagat1980
accattcagtgggctattgattccggcaagggtctgatgttcactcagccgaatcaggct2040
gctggatacatggctaagctgatttgggaatctgtgagcgtgacggtggtagctgcggtt2100
gaagcaatgaactggcttaagtctgctgctaagctgctggctgctgaggtcaaagataag2160
aagactggagagattcttcgcaagcgttgcgctgtgcattgggtaactcctgatggtttc2220
cctgtgtggcaggaatacaagaagcctattcagacgcgcttgaacctgatgttcctcggt2280
cagttccgcttacagcctaccattaacaccaacaaagatagcgagattgatgcacacaaa2340
caggagtctggtatcgctcctaactttgtacacagccaagacggtagccaccttcgtaag2400
actgtagtgtgggcacacgagaagtacggaatcgaatcttttgcactgattcacgactcc2460
ttcggtaccattccggctgacgctgcgaacctgttcaaagcagtgcgcgaaactatggtt2520
gacacatatgagtcttgtgatgtactggctgatttctacgaccagttcgctgaccagttg2580
cacgagtctcaattggacaaaatgccagcacttccggctaaaggtaacttgaacctccgt2640
gacatcttagagtcggacttcgcgttcgcgtaa2673
<210>27
<211>890
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>27
methishishishishishismetasnthrileasnilealalysasn
151015
asppheseraspilegluleualaalailepropheasnthrleuala
202530
asphistyrglygluargleualaarggluglnleualaleugluhis
354045
glusertyrglumetglyglualaargphearglysmetphegluarg
505560
glnleulysalaglygluvalalaaspasnalaalaalalysproleu
65707580
ilethrthrleuleuprolysmetilealaargileasnasptrpphe
859095
glugluvallysalalysargglylysargprothralapheglnphe
100105110
leuglngluilelysproglualavalalatyrilethrilelysthr
115120125
thrleualacysleuthrseralatrpasnthrthrvalglnalaval
130135140
alaseralaileglyargalailegluaspglualaargpheglyarg
145150155160
ileargaspleuglualalyshisphelyslysasnvalgluglugln
165170175
leuasnlysargvalglyhisvaltyrlyslysalaphemetglnval
180185190
valglualaaspmetleuserlysglyleuleuglyglyglualatrp
195200205
sersertrphislysgluaspserilehisvalglyvalargcysile
210215220
glumetleuilegluserthrglymetvalserleuhisargglnasn
225230235240
alaglyvalvalglyglnaspsergluthrilegluleualaproglu
245250255
tyralaglualailealathrargalaglyalaleualaglyileser
260265270
prometpheglnprocysvalvalproprolysprotrpthrglyile
275280285
thrglyglyglytyrtrpalaasnglyargargproleualaleuval
290295300
argthrhisserlyslysalaleumetargtyrgluaspvaltyrmet
305310315320
progluvaltyrlysalaileasnilealaglnasnthralatrplys
325330335
ileasnlyslysvalleualavalalaasnvalilethrlystrplys
340345350
hiscysprovalgluaspileproalailegluargglugluleupro
355360365
metlysprogluaspileaspmetasnproglualaleuthralatrp
370375380
lysargalaalaalaalavaltyrarglysasplysalaarglysser
385390395400
ileargileserleugluphemetleugluglnalaasnlyspheala
405410415
asnhislysalailetrppheprotyrasnmetasptrpargglyarg
420425430
valtyralavalsermetpheasnproglnglyasnaspmetthrlys
435440445
glyleuleuthrleualalysglylysproileglylysgluglytyr
450455460
tyrtrpleulysilehisglyalaasncysalaglyvalasplysval
465470475480
propheprogluargilelyspheileglugluasnhisgluasnile
485490495
metalacysalalysserproleugluasnthrtrptrpalaglugln
500505510
aspserprophecyspheleualaphecyspheglutyralaglyval
515520525
glnhishisglyleusertyrasncysserleuproleualapheasp
530535540
glysercysserglyileglnhispheseralametleuargaspglu
545550555560
valglyglyargalavalasnleuleuprosergluthrvalglnasp
565570575
iletyrglyilevalalalyslysvalasngluileleuglnalaasp
580585590
alaileasnglythraspasngluvalvalthrvalthraspgluasn
595600605
thrglygluileserglulysvallysleuglythrlysalaleuala
610615620
glyglntrpleualatyrglyvalthrargservalthrlysargser
625630635640
valmetthrleualatyrglyserlysglupheglypheargglngln
645650655
valleugluaspthrileglntrpalaileaspserglylysglyleu
660665670
metphethrglnproasnglnalaalaglytyrmetalalysleuile
675680685
trpgluservalservalthrvalvalalaalavalglualametasn
690695700
trpleulysseralaalalysleuleualaalagluvallysasplys
705710715720
lysthrglygluileleuarglysargcysalavalhistrpvalthr
725730735
proaspglypheprovaltrpglnglutyrlyslysproileglnthr
740745750
argleuasnleumetpheleuglyglnpheargleuglnprothrile
755760765
asnthrasnlysaspsergluileaspalahislysglnglusergly
770775780
ilealaproasnphevalhisserglnaspglyserhisleuarglys
785790795800
thrvalvaltrpalahisglulystyrglyilegluserphealaleu
805810815
ilehisaspserpheglythrileproalaaspalaalaasnleuphe
820825830
lysalavalarggluthrmetvalaspthrtyrglusercysaspval
835840845
leualaaspphetyraspglnphealaaspglnleuhisglusergln
850855860
leuasplysmetproalaleuproalalysglyasnleuasnleuarg
865870875880
aspileleugluseraspphealapheala
885890
<210>28
<211>2673
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>28
atgcaccatcaccatcaccatatgaacacgattaacatcgctaagaacgacttctctgac60
atcgaactggctgctatcccgttcaacactctggctgaccattacggtgagcgtttagct120
cgcgaacagttggcccttgagcatgagtcttacgagatgggtgaagcacgcttccgcaag180
atgtttgagcgtcaacttaaagctggtgaggttgcggataacgctgccgccaagcctctc240
atcactaccctactccctaagatgattgcacgcatcaacgactggtttgaggaagtgaaa300
gctaagcgcggcaagcgcccgacagccttccagttcctgcaagaaatcaagccggaagcc360
gtagcgtacatcaccattaagaccactctggcttgcctaaccagtatagacaatacaacc420
gttcaggctgtagcaagcgcaatcggtcgggccattgaggacgaggctcgcttcggtcgt480
atccgtgaccttgaagctaagcacttcaagaaaaacgttgaggaacaactcaacaagcgc540
gtagggcacgtctacaagaaagcatttatgcaagttgtcgaggctgacatgctctctaag600
ggtctactcggtggcgaggcgtggtcttcgtggcataaggaagactctattcatgtagga660
gtacgctgcatcgagatgctcattgagtcaaccggaatggttagcttacaccgccaaaat720
gctggcgtagtaggtcaagactctgagactatcgaactcgcacctgaatacgctgaggct780
atcgcaacccgtgcaggtgcgctggctggcatctctccgatgttccaaccttgcgtagtt840
cctcctaagccgtggactggcattactggtggtggctattgggctaacggtcgtcgtcct900
ctggcgctggtgcgtactcacagtaagaaagcactgatgcgctacgaagacgtttacatg960
cctgaggtgtacaaagcgattaacattgcgcaaaacaccgcatggaaaatcaacaagaaa1020
gtcctagcggtcgccaacgtaatcaccaagtggaagcattgtccggtcgaagacatccct1080
gcgattgagcgtgaagaactcccgatgaaaccggaagacatcgacatgaatcctgaggct1140
ctcaccgcgtggaaacgtgctgccgctgctgtgtaccgcagagacaaggctcgcaagtct1200
cgccgtatctatcttgagttcatgcttgagcaagccaataagtttgctaaccataaggcc1260
atctggttcccttacaacatggactggcgcggtcgtgtttacgctgtgtcaatgttcaac1320
ccgcaaggtaacgattggaccaaaggactgcttacgctggcgaaaggtaaaccaatcggt1380
aaggaaggttactactggctgaaaatccacggtgcaaactgtgcgggtgtcgataaggtt1440
ccgttccctgagcgcatcaagttcattgaggaaaaccacgagaacatcatggcttgcgct1500
aagtctccactggagaacacttggtgggctgagcaagattccccgttctgcttccttgcg1560
ttctgctttgagtacgctggggtacagcaccacggcctgagctataactgctcccttccg1620
ctggcgtttgacgggtcttgctctggcatccagcacttctccgcgatgctccgagatgag1680
gtaggtggtcgcgcggttaacttgcttcctagtgaaaccgttcaggacatctacgggatt1740
gttgctaagaaagtcaacgagattctacaagcagacgcaatcaatgggaccgataacgaa1800
gtagttaccgtgaccgatgagaacactggtgaaatctctgagaaagtcaagctgggcact1860
aaggcactggctggtcaatggctggcttacggtgttactcgcagtgtgactaagcgttca1920
gtcatgacgctggcttacgggtccaaagagttcggcttccgtcaacaagtgctggaagat1980
accattcagtgggctattgattccggcaagggtctgatgttcactcagccgaatcaggct2040
gctggatacatggctaagctgatttgggaatctgtgagcgtgacggtggtagctgcggtt2100
gaagcaatgaactggcttaagtctgctgctaagctgctggctgctgaggtcaaagataag2160
aagactggagagattcttcgcaagcgttgcgctgtgcattgggtaactcctgatggtttc2220
cctgtgtggcaggaatacaagaagcctattcagacgcgcttgaacctgatgttcctcggt2280
cagttccgcttacagcctaccattaacaccaacaaagatagcgagattgatgcacacaaa2340
caggagtctggtatcgctcctaactttgtacacagccaagacggtagccaccttcgtaag2400
actgtagtgtgggcacacgagaagtacggaatcgaatcttttgcactgattcacgactcc2460
ttcggtaccattccggctgacgctgcgaacctgttcaaagcagtgcgcgaaactatggtt2520
gacacatatgagtcttgtgatgtactggctgatttctacgaccagttcgctgaccagttg2580
cacgagtctcaattggacaaaatgccagcacttccggctaaaggtaacttgaacctccgt2640
gacatcttagagtcggacttcgcgttcgcgtaa2673
<210>29
<211>890
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>29
methishishishishishismetasnthrileasnilealalysasn
151015
asppheseraspilegluleualaalailepropheasnthrleuala
202530
asphistyrglygluargleualaarggluglnleualaleugluhis
354045
glusertyrglumetglyglualaargphearglysmetphegluarg
505560
glnleulysalaglygluvalalaaspasnalaalaalalysproleu
65707580
ilethrthrleuleuprolysmetilealaargileasnasptrpphe
859095
glugluvallysalalysargglylysargprothralapheglnphe
100105110
leuglngluilelysproglualavalalatyrilethrilelysthr
115120125
thrleualacysleuthrserileaspasnthrthrvalglnalaval
130135140
alaseralaileglyargalailegluaspglualaargpheglyarg
145150155160
ileargaspleuglualalyshisphelyslysasnvalgluglugln
165170175
leuasnlysargvalglyhisvaltyrlyslysalaphemetglnval
180185190
valglualaaspmetleuserlysglyleuleuglyglyglualatrp
195200205
sersertrphislysgluaspserilehisvalglyvalargcysile
210215220
glumetleuilegluserthrglymetvalserleuhisargglnasn
225230235240
alaglyvalvalglyglnaspsergluthrilegluleualaproglu
245250255
tyralaglualailealathrargalaglyalaleualaglyileser
260265270
prometpheglnprocysvalvalproprolysprotrpthrglyile
275280285
thrglyglyglytyrtrpalaasnglyargargproleualaleuval
290295300
argthrhisserlyslysalaleumetargtyrgluaspvaltyrmet
305310315320
progluvaltyrlysalaileasnilealaglnasnthralatrplys
325330335
ileasnlyslysvalleualavalalaasnvalilethrlystrplys
340345350
hiscysprovalgluaspileproalailegluargglugluleupro
355360365
metlysprogluaspileaspmetasnproglualaleuthralatrp
370375380
lysargalaalaalaalavaltyrargargasplysalaarglysser
385390395400
argargiletyrleugluphemetleugluglnalaasnlyspheala
405410415
asnhislysalailetrppheprotyrasnmetasptrpargglyarg
420425430
valtyralavalsermetpheasnproglnglyasnasptrpthrlys
435440445
glyleuleuthrleualalysglylysproileglylysgluglytyr
450455460
tyrtrpleulysilehisglyalaasncysalaglyvalasplysval
465470475480
propheprogluargilelyspheileglugluasnhisgluasnile
485490495
metalacysalalysserproleugluasnthrtrptrpalaglugln
500505510
aspserprophecyspheleualaphecyspheglutyralaglyval
515520525
glnhishisglyleusertyrasncysserleuproleualapheasp
530535540
glysercysserglyileglnhispheseralametleuargaspglu
545550555560
valglyglyargalavalasnleuleuprosergluthrvalglnasp
565570575
iletyrglyilevalalalyslysvalasngluileleuglnalaasp
580585590
alaileasnglythraspasngluvalvalthrvalthraspgluasn
595600605
thrglygluileserglulysvallysleuglythrlysalaleuala
610615620
glyglntrpleualatyrglyvalthrargservalthrlysargser
625630635640
valmetthrleualatyrglyserlysglupheglypheargglngln
645650655
valleugluaspthrileglntrpalaileaspserglylysglyleu
660665670
metphethrglnproasnglnalaalaglytyrmetalalysleuile
675680685
trpgluservalservalthrvalvalalaalavalglualametasn
690695700
trpleulysseralaalalysleuleualaalagluvallysasplys
705710715720
lysthrglygluileleuarglysargcysalavalhistrpvalthr
725730735
proaspglypheprovaltrpglnglutyrlyslysproileglnthr
740745750
argleuasnleumetpheleuglyglnpheargleuglnprothrile
755760765
asnthrasnlysaspsergluileaspalahislysglnglusergly
770775780
ilealaproasnphevalhisserglnaspglyserhisleuarglys
785790795800
thrvalvaltrpalahisglulystyrglyilegluserphealaleu
805810815
ilehisaspserpheglythrileproalaaspalaalaasnleuphe
820825830
lysalavalarggluthrmetvalaspthrtyrglusercysaspval
835840845
leualaaspphetyraspglnphealaaspglnleuhisglusergln
850855860
leuasplysmetproalaleuproalalysglyasnleuasnleuarg
865870875880
aspileleugluseraspphealapheala
885890
<210>30
<211>2673
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>30
atgcaccatcaccatcaccatatgaacacgattaacatcgctaagaacgacttctctgac60
atcgaactggctgctatcccgttcaacactctggctgaccattacggtgagcgtttagct120
cgcgaacagttggcccttgagcatgagtcttacgagatgggtgaagcacgcttccgcaag180
atgtttgagcgtcaacttaaagctggtgaggttgcggataacgctgccgccaagcctctc240
atcactaccctactccctaagatgattgcacgcatcaacgactggtttgaggaagtgaaa300
gctaagcgcggcaagcgcccgacagccttccagttcctgcaagaaatcaagccggaagcc360
gtagcgtacatcaccattaagaccactctggcttgcctaaccagtgctgacaatacaacc420
gttcaggctgtagcaagcgcaatcggtcgggccattgaggacgaggctcgcttcggtcgt480
atccgtgaccttgaagctaagcacttcaagaaaaacgttgaggaacaactcaacaagcgc540
gtagggcacgtctacaagaaagcatttatgcaagttgtcgaggctgacatgctctctaag600
ggtctactcggtggcgaggcgtggtcttcgtggcataaggaagactctattcatgtagga660
gtacgctgcatcgagatgctcattgagtcaaccggaatggttagcttacaccgccaaaat720
gctggcgtagtaggtcaagactctgagactatcgaactcgcacctgaatacgctgaggct780
atcgcaacccgtgcaggtgcgctggctggcatctctccgatgttccaaccttgcgtagtt840
cctcctaagccgtggactggcattactggtggtggctattgggctaacggtcgtcgtcct900
ctggcgctggtgcgtactcacagtaagaaagcactgatgcgctacgaagacgtttacatg960
cctgaggtgtacaaagcgattaacattgcgcaaaacaccgcatggaaaatcaacaagaaa1020
gtcctagcggtcgccaacgtaatcaccaagtggaagcattgtccggtcgaggacatccct1080
gcgattgagcgtgaagaactcccgatgaaaccggaagacatcgacatgaatcctgaggct1140
ctcaccgcgtggaaacgtgctgccgctgctgtgtaccgcaaggacaaggcgcgcaagtct1200
cgccgtatcagccttgagttcatgcttgagcaagccaataagtttgctaaccataaggcc1260
atctggttcccttacaacatggactggcgcggtcgtgtttacgctgtgtcaatgttcaac1320
ccgcaaggtaacgatatgaccaaaggactgcttacgctggcgaaaggtaaaccaatcggt1380
aaggaaggttactactggctgaaaatccacggtgcaaactgtgcgggtgtcgataaggtt1440
ccgttccctgagcgcatcaagttcattgaggaaaaccacgagaacatcatggcttgcgct1500
aagtctccactggagaacacttggtgggctgagcaatggtctccgttctgcttccttgcg1560
ttctgctttgagtacgctggggtacagcaccacggcctgagctataactgctcccttccg1620
ctggcgtttgacgggtcttgctctggcatccagcacttctccgcgatgctccgagatgag1680
gtaggtggtcgcgcggttaacttgcttcctagtgaaaccgttcaggacatctacgggatt1740
gttgctaagaaagtcaacgagattctacaagcagacgcaatcaatgggaccgataacgaa1800
gtagttaccgtgaccgatgagaacactggtgaaatctctgagaaagtcaagctgggcact1860
aaggcactggctggtcaatggctggcttacggtgttactcgctgggtgactaagcgttca1920
gtcatgacgctggcttacgggtccaaagagttcggcttccgtcaacaagtgctggaattc1980
accattcagcctgctattgattccggcaagggtctgatgttcactcagccgaatcaggct2040
gctggatacatggctaagctgatttgggaatctgtgagcgtgacggtggtagctgcggtt2100
gaagcaatgaactggcttaagtctgctgctaagctgctggctgctgaggtcaaagataag2160
aagactggagagattcttcgcaagcgttgcgctgtgcattgggtaactcctgatggtttc2220
cctgtgtggcaggaatacaagaagcctattcagacgcgcttgaacctgatgttcctcggt2280
cagttccgcttacagcctaccattaacaccaacaaagatagcgagattgatgcacacaaa2340
caggagtctggtatcgctcctaactttgtacacagccaagacggtagccaccttcgtaag2400
actgtagtgtgggcacacgagaagtacggaatcgaatcttttgcactgattcacgactcc2460
ttcggtaccattccggctgacgctgcgaacctgttcaaagcagtgcgcgaaactatggtt2520
gacacatatgagtcttgtgatgtactggctgatttctacgaccagttcgctgaccagttg2580
cacgagtctcaattggacaaaatgccagcacttccggctaaaggtaacttgaacctccgt2640
gacatcttagagtcggacttcgcgttcgcgtaa2673
<210>31
<211>890
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>31
methishishishishishismetasnthrileasnilealalysasn
151015
asppheseraspilegluleualaalailepropheasnthrleuala
202530
asphistyrglygluargleualaarggluglnleualaleugluhis
354045
glusertyrglumetglyglualaargphearglysmetphegluarg
505560
glnleulysalaglygluvalalaaspasnalaalaalalysproleu
65707580
ilethrthrleuleuprolysmetilealaargileasnasptrpphe
859095
glugluvallysalalysargglylysargprothralapheglnphe
100105110
leuglngluilelysproglualavalalatyrilethrilelysthr
115120125
thrleualacysleuthrseralaaspasnthrthrvalglnalaval
130135140
alaseralaileglyargalailegluaspglualaargpheglyarg
145150155160
ileargaspleuglualalyshisphelyslysasnvalgluglugln
165170175
leuasnlysargvalglyhisvaltyrlyslysalaphemetglnval
180185190
valglualaaspmetleuserlysglyleuleuglyglyglualatrp
195200205
sersertrphislysgluaspserilehisvalglyvalargcysile
210215220
glumetleuilegluserthrglymetvalserleuhisargglnasn
225230235240
alaglyvalvalglyglnaspsergluthrilegluleualaproglu
245250255
tyralaglualailealathrargalaglyalaleualaglyileser
260265270
prometpheglnprocysvalvalproprolysprotrpthrglyile
275280285
thrglyglyglytyrtrpalaasnglyargargproleualaleuval
290295300
argthrhisserlyslysalaleumetargtyrgluaspvaltyrmet
305310315320
progluvaltyrlysalaileasnilealaglnasnthralatrplys
325330335
ileasnlyslysvalleualavalalaasnvalilethrlystrplys
340345350
hiscysprovalgluaspileproalailegluargglugluleupro
355360365
metlysprogluaspileaspmetasnproglualaleuthralatrp
370375380
lysargalaalaalaalavaltyrarglysasplysalaarglysser
385390395400
argargileserleugluphemetleugluglnalaasnlyspheala
405410415
asnhislysalailetrppheprotyrasnmetasptrpargglyarg
420425430
valtyralavalsermetpheasnproglnglyasnaspmetthrlys
435440445
glyleuleuthrleualalysglylysproileglylysgluglytyr
450455460
tyrtrpleulysilehisglyalaasncysalaglyvalasplysval
465470475480
propheprogluargilelyspheileglugluasnhisgluasnile
485490495
metalacysalalysserproleugluasnthrtrptrpalaglugln
500505510
trpserprophecyspheleualaphecyspheglutyralaglyval
515520525
glnhishisglyleusertyrasncysserleuproleualapheasp
530535540
glysercysserglyileglnhispheseralametleuargaspglu
545550555560
valglyglyargalavalasnleuleuprosergluthrvalglnasp
565570575
iletyrglyilevalalalyslysvalasngluileleuglnalaasp
580585590
alaileasnglythraspasngluvalvalthrvalthraspgluasn
595600605
thrglygluileserglulysvallysleuglythrlysalaleuala
610615620
glyglntrpleualatyrglyvalthrargtrpvalthrlysargser
625630635640
valmetthrleualatyrglyserlysglupheglypheargglngln
645650655
valleugluphethrileglnproalaileaspserglylysglyleu
660665670
metphethrglnproasnglnalaalaglytyrmetalalysleuile
675680685
trpgluservalservalthrvalvalalaalavalglualametasn
690695700
trpleulysseralaalalysleuleualaalagluvallysasplys
705710715720
lysthrglygluileleuarglysargcysalavalhistrpvalthr
725730735
proaspglypheprovaltrpglnglutyrlyslysproileglnthr
740745750
argleuasnleumetpheleuglyglnpheargleuglnprothrile
755760765
asnthrasnlysaspsergluileaspalahislysglnglusergly
770775780
ilealaproasnphevalhisserglnaspglyserhisleuarglys
785790795800
thrvalvaltrpalahisglulystyrglyilegluserphealaleu
805810815
ilehisaspserpheglythrileproalaaspalaalaasnleuphe
820825830
lysalavalarggluthrmetvalaspthrtyrglusercysaspval
835840845
leualaaspphetyraspglnphealaaspglnleuhisglusergln
850855860
leuasplysmetproalaleuproalalysglyasnleuasnleuarg
865870875880
aspileleugluseraspphealapheala
885890
<210>32
<211>2673
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>32
atgcaccatcaccatcaccatatgaacacgattaacatcgctaagaacgacttctctgac60
atcgaactggctgctatcccgttcaacactctggctgaccattacggtgagcgtttagct120
cgcgaacagttggcccttgagcatgagtcttacgagatgggtgaagcacgcttccgcaag180
atgtttgagcgtcaacttaaagctggtgaggttgcggataacgctgccgccaagcctctc240
atcactaccctactccctaagatgattgcacgcatcaacgactggtttgaggaagtgaaa300
gctaagcgcggcaagcgcccgacagccttccagttcctgcaagaaatcaagccggaagcc360
gtagcgtacatcaccattaagaccactctggcttgcctaaccagtgctgacaatacaacc420
gttcaggctgtagcaagcgcaatcggtcgggccattgaggacgaggctcgcttcggtcgt480
atccgtgaccttgaagctaagcacttcaagaaaaacgttgaggaacaactcaacaagcgc540
gtagggcacgtctacaagaaagcatttatgcaagttgtcgaggctgacatgctctctaag600
ggtctactcggtggcgaggcgtggtcttcgtggcataaggaagactctattcatgtagga660
gtacgctgcatcgagatgctcattgagtcaaccggaatggttagcttacaccgccaaaat720
gctggcgtagtaggtcaagactctgagactatcgaactcgcacctgaatacgctgaggct780
atcgcaacccgtgcaggtgcgctggctggcatctctccgatgttccaaccttgcgtagtt840
cctcctaagccgtggactggcattactggtggtggctattgggctaacggtcgtcgtcct900
ctggcgctggtgcgtactcacagtaagaaagcactgatgcgctacgaagacgtttacatg960
cctgaggtgtacaaagcgattaacattgcgcaaaacaccgcatggaaaatcaacaagaaa1020
gtcctagcggtcgccaacgtaatcaccaagtggaagcattgtccggtcgaggacatccct1080
gcgattgagcgtgaagaactcccgatgaaaccggaagacatcgacatgaatcctgaggct1140
ctcaccgcgtggaaacgtgctgccgctgctgtgtaccgcaaggacaaggcgcgcaagtct1200
cgccgtatcagccttgagttcatgcttgagcaagccaataagtttgctaaccataaggcc1260
atctggttcccttacaacatggactggcgcggtcgtgtttacgctgtgtcaatgttcaac1320
ccgcaaggtaacgatatgaccaaaggactgcttacgctggcgaaaggtaaaccaatcggt1380
aaggaaggttactactggctgaaaatccacggtgcaaactgtgcgggtgtcgataaggtt1440
ccgttccctgagcgcatcaagttcattgaggaaaaccacgagaacatcatggcttgcgct1500
aagtctccactggagaacacttggtgggctgagcaatattctccgttctgcttccttgcg1560
ttctgctttgagtacgctggggtacagcaccacggcctgagctataactgctcccttccg1620
ctggcgtttgacgggtcttgctctggcatccagcacttctccgcgatgctccgagatgag1680
gtaggtggtcgcgcggttaacttgcttcctagtgaaaccgttcaggacatctacgggatt1740
gttgctaagaaagtcaacgagattctacaagcagacgcaatcaatgggaccgataacgaa1800
gtagttaccgtgaccgatgagaacactggtgaaatctctgagaaagtcaagctgggcact1860
aaggcactggctggtcaatggctggcttacggtgttactcgctgggtgactaagcgttca1920
gtcatgacgctggcttacgggtccaaagagttcggcttccgtcaacaagtgctggaatac1980
accattcagcctgctattgattccggcaagggtctgatgttcactcagccgaatcaggct2040
gctggatacatggctaagctgatttgggaatctgtgagcgtgacggtggtagctgcggtt2100
gaagcaatgaactggcttaagtctgctgctaagctgctggctgctgaggtcaaagataag2160
aagactggagagattcttcgcaagcgttgcgctgtgcattgggtaactcctgatggtttc2220
cctgtgtggcaggaatacaagaagcctattcagacgcgcttgaacctgatgttcctcggt2280
cagttccgcttacagcctaccattaacaccaacaaagatagcgagattgatgcacacaaa2340
caggagtctggtatcgctcctaactttgtacacagccaagacggtagccaccttcgtaag2400
actgtagtgtgggcacacgagaagtacggaatcgaatcttttgcactgattcacgactcc2460
ttcggtaccattccggctgacgctgcgaacctgttcaaagcagtgcgcgaaactatggtt2520
gacacatatgagtcttgtgatgtactggctgatttctacgaccagttcgctgaccagttg2580
cacgagtctcaattggacaaaatgccagcacttccggctaaaggtaacttgaacctccgt2640
gacatcttagagtcggacttcgcgttcgcgtaa2673
<210>33
<211>890
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>33
methishishishishishismetasnthrileasnilealalysasn
151015
asppheseraspilegluleualaalailepropheasnthrleuala
202530
asphistyrglygluargleualaarggluglnleualaleugluhis
354045
glusertyrglumetglyglualaargphearglysmetphegluarg
505560
glnleulysalaglygluvalalaaspasnalaalaalalysproleu
65707580
ilethrthrleuleuprolysmetilealaargileasnasptrpphe
859095
glugluvallysalalysargglylysargprothralapheglnphe
100105110
leuglngluilelysproglualavalalatyrilethrilelysthr
115120125
thrleualacysleuthrseralaaspasnthrthrvalglnalaval
130135140
alaseralaileglyargalailegluaspglualaargpheglyarg
145150155160
ileargaspleuglualalyshisphelyslysasnvalgluglugln
165170175
leuasnlysargvalglyhisvaltyrlyslysalaphemetglnval
180185190
valglualaaspmetleuserlysglyleuleuglyglyglualatrp
195200205
sersertrphislysgluaspserilehisvalglyvalargcysile
210215220
glumetleuilegluserthrglymetvalserleuhisargglnasn
225230235240
alaglyvalvalglyglnaspsergluthrilegluleualaproglu
245250255
tyralaglualailealathrargalaglyalaleualaglyileser
260265270
prometpheglnprocysvalvalproprolysprotrpthrglyile
275280285
thrglyglyglytyrtrpalaasnglyargargproleualaleuval
290295300
argthrhisserlyslysalaleumetargtyrgluaspvaltyrmet
305310315320
progluvaltyrlysalaileasnilealaglnasnthralatrplys
325330335
ileasnlyslysvalleualavalalaasnvalilethrlystrplys
340345350
hiscysprovalgluaspileproalailegluargglugluleupro
355360365
metlysprogluaspileaspmetasnproglualaleuthralatrp
370375380
lysargalaalaalaalavaltyrarglysasplysalaarglysser
385390395400
argargileserleugluphemetleugluglnalaasnlyspheala
405410415
asnhislysalailetrppheprotyrasnmetasptrpargglyarg
420425430
valtyralavalsermetpheasnproglnglyasnaspmetthrlys
435440445
glyleuleuthrleualalysglylysproileglylysgluglytyr
450455460
tyrtrpleulysilehisglyalaasncysalaglyvalasplysval
465470475480
propheprogluargilelyspheileglugluasnhisgluasnile
485490495
metalacysalalysserproleugluasnthrtrptrpalaglugln
500505510
tyrserprophecyspheleualaphecyspheglutyralaglyval
515520525
glnhishisglyleusertyrasncysserleuproleualapheasp
530535540
glysercysserglyileglnhispheseralametleuargaspglu
545550555560
valglyglyargalavalasnleuleuprosergluthrvalglnasp
565570575
iletyrglyilevalalalyslysvalasngluileleuglnalaasp
580585590
alaileasnglythraspasngluvalvalthrvalthraspgluasn
595600605
thrglygluileserglulysvallysleuglythrlysalaleuala
610615620
glyglntrpleualatyrglyvalthrargtrpvalthrlysargser
625630635640
valmetthrleualatyrglyserlysglupheglypheargglngln
645650655
valleuglutyrthrileglnproalaileaspserglylysglyleu
660665670
metphethrglnproasnglnalaalaglytyrmetalalysleuile
675680685
trpgluservalservalthrvalvalalaalavalglualametasn
690695700
trpleulysseralaalalysleuleualaalagluvallysasplys
705710715720
lysthrglygluileleuarglysargcysalavalhistrpvalthr
725730735
proaspglypheprovaltrpglnglutyrlyslysproileglnthr
740745750
argleuasnleumetpheleuglyglnpheargleuglnprothrile
755760765
asnthrasnlysaspsergluileaspalahislysglnglusergly
770775780
ilealaproasnphevalhisserglnaspglyserhisleuarglys
785790795800
thrvalvaltrpalahisglulystyrglyilegluserphealaleu
805810815
ilehisaspserpheglythrileproalaaspalaalaasnleuphe
820825830
lysalavalarggluthrmetvalaspthrtyrglusercysaspval
835840845
leualaaspphetyraspglnphealaaspglnleuhisglusergln
850855860
leuasplysmetproalaleuproalalysglyasnleuasnleuarg
865870875880
aspileleugluseraspphealapheala
885890
<210>34
<211>2673
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>34
atgcaccatcaccatcaccatatgaacacgattaacatcgctaagaacgacttctctgac60
atcgaactggctgctatcccgttcaacactctggctgaccattacggtgagcgtttagct120
cgcgaacagttggcccttgagcatgagtcttacgagatgggtgaagcacgcttccgcaag180
atgtttgagcgtcaacttaaagctggtgaggttgcggataacgctgccgccaagcctctc240
atcactaccctactccctaagatgattgcacgcatcaacgactggtttgaggaagtgaaa300
gctaagcgcggcaagcgcccgacagccttccagttcctgcaagaaatcaagccggaagcc360
gtagcgtacatcaccattaagaccactctggcttgcctaaccagtgcggacaatacaacc420
gttcaggctgtagcaagcgcaatcggtcgggccattgaggacgaggctcgcttcggtcgt480
atccgtgaccttgaagctaagcacttcaagaaaaacgttgaggaacaactcaacaagcgc540
gtagggcacgtctacaagaaagcatttatgcaagttgtcgaggctgacatgctctctaag600
ggtctactcggtggcgaggcgtggtcttcgtggcataaggaagactctattcatgtagga660
gtacgctgcatcgagatgctcattgagtcaaccggaatggttagcttacaccgccaaaat720
gctggcgtagtaggtcaagactctgagactatcgaactcgcacctgaatacgctgaggct780
atcgcaacccgtgcaggtgcgctggctggcatctctccgatgttccaaccttgcgtagtt840
cctcctaagccgtggactggcattactggtggtggctattgggctaacggtcgtcgtcct900
ctggcgctggtgcgtactcacagtaagaaagcactgatgcgctacgaagacgtttacatg960
cctgaggtgtacaaagcgattaacattgcgcaaaacaccgcatggaaaatcaacaagaaa1020
gtcctagcggtcgccaacgtaatcaccaagtggaagcattgtccggtcgaggacatccct1080
gcgattgagcgtgaagaactcccgatgaaaccggaagacatcgacatgaatcctgaggct1140
ctcaccgcgtggaaacgtgctgccgctgctgtgtaccgcaaggacaaggctcgcaagtct1200
gttcgtatctatcttgagttcatgcttgagcaagccaataagtttgctaaccataaggcc1260
atctggttcccttacaacatggactggcgcggtcgtgtttacgctgtgtcaatgttcaac1320
ccgcaaggtaacgatatgaccaaaggactgcttacgctggcgaaaggtaaaccaatcggt1380
aaggaaggttactactggctgaaaatccacggtgcaaactgtgcgggtgtcgataaggtt1440
ccgttccctgagcgcatcaagttcattgaggaaaaccacgagaacatcatggcttgcgct1500
aagtctccactggagaacacttggtgggctgagcaagatctcccgttctgcttccttgcg1560
ttctgctttgagtacgctggggtacagcaccacggcctgagctataactgctcccttccg1620
ctggcgtttgacgggtcttgctctggcatccagcacttctccgcgatgctccgagatgag1680
gtaggtggtcgcgcggttaacttgcttcctagtgaaaccgttcaggacatctacgggatt1740
gttgctaagaaagtcaacgagattctacaagcagacgcaatcaatgggaccgataacgaa1800
gtagttaccgtgaccgatgagaacactggtgaaatctctgagaaagtcaagctgggcact1860
aaggcactggctggtcaatggctggcttacggtgttactcgcagtgtgactaagcgttca1920
gtcatgacgctggcttacgggtccaaagagttcggcttccgtcaacaagtgctggaagac1980
accattcagtgggctattgattccggcaagggtctgatgttcactcagccgaatcaggct2040
gctggatacatggctaagctgatttgggaatctgtgagcgtgacggtggtagctgcggtt2100
gaagcaatgaactggcttaagtctgctgctaagctgctggctgctgaggtcaaagataag2160
aagactggagagattcttcgcaagcgttgcgctgtgcattgggtaactcctgatggtttc2220
cctgtgtggcaggaatacaagaagcctattcagacgcgcttgaacctgatgttcctcggt2280
cagttccgcttacagcctaccattaacaccaacaaagatagcgagattgatgcacacaaa2340
caggagtctggtatcgctcctaactttgtacacagccaagacggtagccaccttcgtaag2400
actgtagtgtgggcacacgagaagtacggaatcgaatcttttgcactgattcacgactcc2460
ttcggtaccattccggctgacgctgcgaacctgttcaaagcagtgcgcgaaactatggtt2520
gacacatatgagtcttgtgatgtactggctgatttctacgaccagttcgctgaccagttg2580
cacgagtctcaattggacaaaatgccagcacttccggctaaaggtaacttgaacctccgt2640
gacatcttagagtcggacttcgcgttcgcgtaa2673
<210>35
<211>890
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>35
methishishishishishismetasnthrileasnilealalysasn
151015
asppheseraspilegluleualaalailepropheasnthrleuala
202530
asphistyrglygluargleualaarggluglnleualaleugluhis
354045
glusertyrglumetglyglualaargphearglysmetphegluarg
505560
glnleulysalaglygluvalalaaspasnalaalaalalysproleu
65707580
ilethrthrleuleuprolysmetilealaargileasnasptrpphe
859095
glugluvallysalalysargglylysargprothralapheglnphe
100105110
leuglngluilelysproglualavalalatyrilethrilelysthr
115120125
thrleualacysleuthrseralaaspasnthrthrvalglnalaval
130135140
alaseralaileglyargalailegluaspglualaargpheglyarg
145150155160
ileargaspleuglualalyshisphelyslysasnvalgluglugln
165170175
leuasnlysargvalglyhisvaltyrlyslysalaphemetglnval
180185190
valglualaaspmetleuserlysglyleuleuglyglyglualatrp
195200205
sersertrphislysgluaspserilehisvalglyvalargcysile
210215220
glumetleuilegluserthrglymetvalserleuhisargglnasn
225230235240
alaglyvalvalglyglnaspsergluthrilegluleualaproglu
245250255
tyralaglualailealathrargalaglyalaleualaglyileser
260265270
prometpheglnprocysvalvalproprolysprotrpthrglyile
275280285
thrglyglyglytyrtrpalaasnglyargargproleualaleuval
290295300
argthrhisserlyslysalaleumetargtyrgluaspvaltyrmet
305310315320
progluvaltyrlysalaileasnilealaglnasnthralatrplys
325330335
ileasnlyslysvalleualavalalaasnvalilethrlystrplys
340345350
hiscysprovalgluaspileproalailegluargglugluleupro
355360365
metlysprogluaspileaspmetasnproglualaleuthralatrp
370375380
lysargalaalaalaalavaltyrarglysasplysalaarglysser
385390395400
valargiletyrleugluphemetleugluglnalaasnlyspheala
405410415
asnhislysalailetrppheprotyrasnmetasptrpargglyarg
420425430
valtyralavalsermetpheasnproglnglyasnaspmetthrlys
435440445
glyleuleuthrleualalysglylysproileglylysgluglytyr
450455460
tyrtrpleulysilehisglyalaasncysalaglyvalasplysval
465470475480
propheprogluargilelyspheileglugluasnhisgluasnile
485490495
metalacysalalysserproleugluasnthrtrptrpalaglugln
500505510
aspleuprophecyspheleualaphecyspheglutyralaglyval
515520525
glnhishisglyleusertyrasncysserleuproleualapheasp
530535540
glysercysserglyileglnhispheseralametleuargaspglu
545550555560
valglyglyargalavalasnleuleuprosergluthrvalglnasp
565570575
iletyrglyilevalalalyslysvalasngluileleuglnalaasp
580585590
alaileasnglythraspasngluvalvalthrvalthraspgluasn
595600605
thrglygluileserglulysvallysleuglythrlysalaleuala
610615620
glyglntrpleualatyrglyvalthrargservalthrlysargser
625630635640
valmetthrleualatyrglyserlysglupheglypheargglngln
645650655
valleugluaspthrileglntrpalaileaspserglylysglyleu
660665670
metphethrglnproasnglnalaalaglytyrmetalalysleuile
675680685
trpgluservalservalthrvalvalalaalavalglualametasn
690695700
trpleulysseralaalalysleuleualaalagluvallysasplys
705710715720
lysthrglygluileleuarglysargcysalavalhistrpvalthr
725730735
proaspglypheprovaltrpglnglutyrlyslysproileglnthr
740745750
argleuasnleumetpheleuglyglnpheargleuglnprothrile
755760765
asnthrasnlysaspsergluileaspalahislysglnglusergly
770775780
ilealaproasnphevalhisserglnaspglyserhisleuarglys
785790795800
thrvalvaltrpalahisglulystyrglyilegluserphealaleu
805810815
ilehisaspserpheglythrileproalaaspalaalaasnleuphe
820825830
lysalavalarggluthrmetvalaspthrtyrglusercysaspval
835840845
leualaaspphetyraspglnphealaaspglnleuhisglusergln
850855860
leuasplysmetproalaleuproalalysglyasnleuasnleuarg
865870875880
aspileleugluseraspphealapheala
885890
<210>36
<211>2673
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>36
atgcaccatcaccatcaccatatgaacacgattaacatcgctaagaacgacttctctgac60
atcgaactggctgctatcccgttcaacactctggctgaccattacggtgagcgtttagct120
cgcgaacagttggcccttgagcatgagtcttacgagatgggtgaagcacgcttccgcaag180
atgtttgagcgtcaacttaaagctggtgaggttgcggataacgctgccgccaagcctctc240
atcactaccctactccctaagatgattgcacgcatcaacgactggtttgaggaagtgaaa300
gctaagcgcggcaagcgcccgacagccttccagttcctgcaagaaatcaagccggaagcc360
gtagcgtacatcaccattaagaccactctggcttgcctaaccagtgcggacaatacaacc420
gttcaggctgtagcaagcgcaatcggtcgggccattgaggacgaggctcgcttcggtcgt480
atccgtgaccttgaagctaagcacttcaagaaaaacgttgaggaacaactcaacaagcgc540
gtagggcacgtctacaagaaagcatttatgcaagttgtcgaggctgacatgctctctaag600
ggtctactcggtggcgaggcgtggtcttcgtggcataaggaagactctattcatgtagga660
gtacgctgcatcgagatgctcattgagtcaaccggaatggttagcttacaccgccaaaat720
gctggcgtagtaggtcaagactctgagactatcgaactcgcacctgaatacgctgaggct780
atcgcaacccgtgcaggtgcgctggctggcatctctccgatgttccaaccttgcgtagtt840
cctcctaagccgtggactggcattactggtggtggctattgggctaacggtcgtcgtcct900
ctggcgctggtgcgtactcacagtaagaaagcactgatgcgctacgaagacgtttacatg960
cctgaggtgtacaaagcgattaacattgcgcaaaacaccgcatggaaaatcaacaagaaa1020
gtcctagcggtcgccaacgtaatcaccaagtggaagcattgtccggtccgggacatccct1080
gcgattgagcgtgaagaactcccgatgaaaccggaagacatcgacatgaatcctgaggct1140
ctcaccgcgtggaaacgtgctgccgctgctgtgtaccgcagagacaaggctcgcaagtct1200
gttcgtatctatcttgagttcatgcttgagcaagccaataagtttgctaaccataaggcc1260
atctggttcccttacaacatggactggcgcggtcgtgtttacgctgtgtcaatgttcaac1320
ccgcaaggtaacgatatgaccaaaggactgcttacgctggcgaaaggtaaaccaatcggt1380
aaggaaggttactactggctgaaaatccacggtgcaaactgtgcgggtgtcgataaggtt1440
ccgttccctgagcgcatcaagttcattgaggaaaaccacgagaacatcatggcttgcgct1500
aagtctccactggagaacacttggtgggctgagcaagatctcccgttctgcttccttgcg1560
ttctgctttgagtacgctggggtacagcaccacggcctgagctataactgctcccttccg1620
ctggcgtttgacgggtcttgctctggcatccagcacttctccgcgatgctccgagatgag1680
gtaggtggtcgcgcggttaacttgcttcctagtgaaaccgttcaggacatctacgggatt1740
gttgctaagaaagtcaacgagattctacaagcagacgcaatcaatgggaccgataacgaa1800
gtagttaccgtgaccgatgagaacactggtgaaatctctgagaaagtcaagctgggcact1860
aaggcactggctggtcaatggctggcttacggtgttactcgcagtgtgactaagcgttca1920
gtcatgacgctggcttacgggtccaaagagttcggcttccgtcaacaagtgctggaagat1980
actattcagtgggctattgattccggcaagggtctgatgttcactcagccgaatcaggct2040
gctggatacatggctaagctgatttgggaatctgtgagcgtgacggtggtagctgcggtt2100
gaagcaatgaactggcttaagtctgctgctaagctgctggctgctgaggtcaaagataag2160
aagactggagagattcttcgcaagcgttgcgctgtgcattgggtaactcctgatggtttc2220
cctgtgtggcaggaatacaagaagcctattcagacgcgcttgaacctgatgttcctcggt2280
cagttccgcttacagcctaccattaacaccaacaaagatagcgagattgatgcacacaaa2340
caggagtctggtatcgctcctaactttgtacacagccaagacggtagccaccttcgtaag2400
actgtagtgtgggcacacgagaagtacggaatcgaatcttttgcactgattcacgactcc2460
ttcggtaccattccggctgacgctgcgaacctgttcaaagcagtgcgcgaaactatggtt2520
gacacatatgagtcttgtgatgtactggctgatttctacgaccagttcgctgaccagttg2580
cacgagtctcaattggacaaaatgccagcacttccggctaaaggtaacttgaacctccgt2640
gacatcttagagtcggacttcgcgttcgcgtaa2673
<210>37
<211>890
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>37
methishishishishishismetasnthrileasnilealalysasn
151015
asppheseraspilegluleualaalailepropheasnthrleuala
202530
asphistyrglygluargleualaarggluglnleualaleugluhis
354045
glusertyrglumetglyglualaargphearglysmetphegluarg
505560
glnleulysalaglygluvalalaaspasnalaalaalalysproleu
65707580
ilethrthrleuleuprolysmetilealaargileasnasptrpphe
859095
glugluvallysalalysargglylysargprothralapheglnphe
100105110
leuglngluilelysproglualavalalatyrilethrilelysthr
115120125
thrleualacysleuthrseralaaspasnthrthrvalglnalaval
130135140
alaseralaileglyargalailegluaspglualaargpheglyarg
145150155160
ileargaspleuglualalyshisphelyslysasnvalgluglugln
165170175
leuasnlysargvalglyhisvaltyrlyslysalaphemetglnval
180185190
valglualaaspmetleuserlysglyleuleuglyglyglualatrp
195200205
sersertrphislysgluaspserilehisvalglyvalargcysile
210215220
glumetleuilegluserthrglymetvalserleuhisargglnasn
225230235240
alaglyvalvalglyglnaspsergluthrilegluleualaproglu
245250255
tyralaglualailealathrargalaglyalaleualaglyileser
260265270
prometpheglnprocysvalvalproprolysprotrpthrglyile
275280285
thrglyglyglytyrtrpalaasnglyargargproleualaleuval
290295300
argthrhisserlyslysalaleumetargtyrgluaspvaltyrmet
305310315320
progluvaltyrlysalaileasnilealaglnasnthralatrplys
325330335
ileasnlyslysvalleualavalalaasnvalilethrlystrplys
340345350
hiscysprovalargaspileproalailegluargglugluleupro
355360365
metlysprogluaspileaspmetasnproglualaleuthralatrp
370375380
lysargalaalaalaalavaltyrargargasplysalaarglysser
385390395400
valargiletyrleugluphemetleugluglnalaasnlyspheala
405410415
asnhislysalailetrppheprotyrasnmetasptrpargglyarg
420425430
valtyralavalsermetpheasnproglnglyasnaspmetthrlys
435440445
glyleuleuthrleualalysglylysproileglylysgluglytyr
450455460
tyrtrpleulysilehisglyalaasncysalaglyvalasplysval
465470475480
propheprogluargilelyspheileglugluasnhisgluasnile
485490495
metalacysalalysserproleugluasnthrtrptrpalaglugln
500505510
aspleuprophecyspheleualaphecyspheglutyralaglyval
515520525
glnhishisglyleusertyrasncysserleuproleualapheasp
530535540
glysercysserglyileglnhispheseralametleuargaspglu
545550555560
valglyglyargalavalasnleuleuprosergluthrvalglnasp
565570575
iletyrglyilevalalalyslysvalasngluileleuglnalaasp
580585590
alaileasnglythraspasngluvalvalthrvalthraspgluasn
595600605
thrglygluileserglulysvallysleuglythrlysalaleuala
610615620
glyglntrpleualatyrglyvalthrargservalthrlysargser
625630635640
valmetthrleualatyrglyserlysglupheglypheargglngln
645650655
valleugluaspthrileglntrpalaileaspserglylysglyleu
660665670
metphethrglnproasnglnalaalaglytyrmetalalysleuile
675680685
trpgluservalservalthrvalvalalaalavalglualametasn
690695700
trpleulysseralaalalysleuleualaalagluvallysasplys
705710715720
lysthrglygluileleuarglysargcysalavalhistrpvalthr
725730735
proaspglypheprovaltrpglnglutyrlyslysproileglnthr
740745750
argleuasnleumetpheleuglyglnpheargleuglnprothrile
755760765
asnthrasnlysaspsergluileaspalahislysglnglusergly
770775780
ilealaproasnphevalhisserglnaspglyserhisleuarglys
785790795800
thrvalvaltrpalahisglulystyrglyilegluserphealaleu
805810815
ilehisaspserpheglythrileproalaaspalaalaasnleuphe
820825830
lysalavalarggluthrmetvalaspthrtyrglusercysaspval
835840845
leualaaspphetyraspglnphealaaspglnleuhisglusergln
850855860
leuasplysmetproalaleuproalalysglyasnleuasnleuarg
865870875880
aspileleugluseraspphealapheala
885890
<210>38
<211>2673
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>38
atgcaccatcaccatcaccatatgaacacgattaacatcgctaagaacgacttctctgac60
atcgaactggctgctatcccgttcaacactctggctgaccattacggtgagcgtttagct120
cgcgaacagttggcccttgagcatgagtcttacgagatgggtgaagcacgcttccgcaag180
atgtttgagcgtcaacttaaagctggtgaggttgcggataacgctgccgccaagcctctc240
atcactaccctactccctaagatgattgcacgcatcaacgactggtttgaggaagtgaaa300
gctaagcgcggcaagcgcccgacagccttccagttcctgcaagaaatcaagccggaagcc360
gtagcgtacatcaccattaagaccactctggcttgcctaaccagtgctgacaatacaacc420
gttcaggctgtagcaagcgcaatcggtcgggccattgaggacgaggctcgcttcggtcgt480
atccgtgaccttgaagctaagcacttcaagaaaaacgttgaggaacaactcaacaagcgc540
gtagggcacgtctacaagaaagcatttatgcaagttgtcgaggctgacatgctctctaag600
ggtctactcggtggcgaggcgtggtcttcgtggcataaggaagactctattcatgtagga660
gtacgctgcatcgagatgctcattgagtcaaccggaatggttagcttacaccgccaaaat720
gctggcgtagtaggtcaagactctgagactatcgaactcgcacctgaatacgctgaggct780
atcgcaacccgtgcaggtgcgctggctggcatctctccgatgttccaaccttgcgtagtt840
cctcctaagccgtggactggcattactggtggtggctattgggctaacggtcgtcgtcct900
ctggcgctggtgcgtactcacagtaagaaagcactgatgcgctacgaagacgtttacatg960
cctgaggtgtacaaagcgattaacattgcgcaaaacaccgcatggaaaatcaacaagaaa1020
gtcctagcggtcgccaacgtaatcaccaagtggaagcattgtccggtcgaggacatccct1080
gcgattgagcgtgaagaactcccgatgaaaccggaagacatcgacatgaatcctgaggct1140
ctcaccgcgtggaaacgtgctgccgctgctgtgtaccgcaaggacaaggcgcgcaagtct1200
cgccgtatcagccttgagttcatgcttgagcaagccaataagtttgctaaccataaggcc1260
atctggttcccttacaacatggactggcgcggtcgtgtttacgctgtgtcaatgttcaac1320
ccgcaaggtaacgatatgaccaaaggactgcttacgctggcgaaaggtaaaccaatcggt1380
aaggaaggttactactggctgaaaatccacggtgcaaactgtgcgggtgtcgataaggtt1440
ccgttccctgagcgcatcaagttcattgaggaaaaccacgagaacatcatggcttgcgct1500
aagtctccactggagaacacttggtgggctgagcaatattctccgttctgcttccttgcg1560
ttctgctttgagtacgctggggtacagcaccacggcctgagctataactgctcccttccg1620
ctggcgtttgacgggtcttgctctggcatccagcacttctccgcgatgctccgagatgag1680
gtaggtggtcgcgcggttaacttgcttcctagtgaaaccgttcaggacatctacgggatt1740
gttgctaagaaagtcaacgagattctacaagcagacgcaatcaatgggaccgataacgaa1800
gtagttaccgtgaccgatgagaacactggtgaaatctctgagaaagtcaagctgggcact1860
aaggcactggctggtcaatggctggcttacggtgttactcgctgggtgactaagcgttca1920
gtcatgacgctggcttacgggtccaaagagttcggcttccgtcaacaagtgctggaattc1980
accattcagcctgctattgattccggcaagggtctgatgttcactcagccgaatcaggct2040
gctggatacatggctaagctgatttgggaatctgtgagcgtgacggtggtagctgcggtt2100
gaagcaatgaactggcttaagtctgctgctaagctgctggctgctgaggtcaaagataag2160
aagactggagagattcttcgcaagcgttgcgctgtgcattgggtaactcctgatggtttc2220
cctgtgtggcaggaatacaagaagcctattcagacgcgcttgaacctgatgttcctcggt2280
cagttccgcttacagcctaccattaacaccaacaaagatagcgagattgatgcacacaaa2340
caggagtctggtatcgctcctaactttgtacacagccaagacggtagccaccttcgtaag2400
actgtagtgtgggcacacgagaagtacggaatcgaatcttttgcactgattcacgactcc2460
ttcggtaccattccggctgacgctgcgaacctgttcaaagcagtgcgcgaaactatggtt2520
gacacatatgagtcttgtgatgtactggctgatttctacgaccagttcgctgaccagttg2580
cacgagtctcaattggacaaaatgccagcacttccggctaaaggtaacttgaacctccgt2640
gacatcttagagtcggacttcgcgttcgcgtaa2673
<210>39
<211>890
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>t7rnap变体
<400>39
methishishishishishismetasnthrileasnilealalysasn
151015
asppheseraspilegluleualaalailepropheasnthrleuala
202530
asphistyrglygluargleualaarggluglnleualaleugluhis
354045
glusertyrglumetglyglualaargphearglysmetphegluarg
505560
glnleulysalaglygluvalalaaspasnalaalaalalysproleu
65707580
ilethrthrleuleuprolysmetilealaargileasnasptrpphe
859095
glugluvallysalalysargglylysargprothralapheglnphe
100105110
leuglngluilelysproglualavalalatyrilethrilelysthr
115120125
thrleualacysleuthrseralaaspasnthrthrvalglnalaval
130135140
alaseralaileglyargalailegluaspglualaargpheglyarg
145150155160
ileargaspleuglualalyshisphelyslysasnvalgluglugln
165170175
leuasnlysargvalglyhisvaltyrlyslysalaphemetglnval
180185190
valglualaaspmetleuserlysglyleuleuglyglyglualatrp
195200205
sersertrphislysgluaspserilehisvalglyvalargcysile
210215220
glumetleuilegluserthrglymetvalserleuhisargglnasn
225230235240
alaglyvalvalglyglnaspsergluthrilegluleualaproglu
245250255
tyralaglualailealathrargalaglyalaleualaglyileser
260265270
prometpheglnprocysvalvalproprolysprotrpthrglyile
275280285
thrglyglyglytyrtrpalaasnglyargargproleualaleuval
290295300
argthrhisserlyslysalaleumetargtyrgluaspvaltyrmet
305310315320
progluvaltyrlysalaileasnilealaglnasnthralatrplys
325330335
ileasnlyslysvalleualavalalaasnvalilethrlystrplys
340345350
hiscysprovalgluaspileproalailegluargglugluleupro
355360365
metlysprogluaspileaspmetasnproglualaleuthralatrp
370375380
lysargalaalaalaalavaltyrarglysasplysalaarglysser
385390395400
argargileserleugluphemetleugluglnalaasnlyspheala
405410415
asnhislysalailetrppheprotyrasnmetasptrpargglyarg
420425430
valtyralavalsermetpheasnproglnglyasnaspmetthrlys
435440445
glyleuleuthrleualalysglylysproileglylysgluglytyr
450455460
tyrtrpleulysilehisglyalaasncysalaglyvalasplysval
465470475480
propheprogluargilelyspheileglugluasnhisgluasnile
485490495
metalacysalalysserproleugluasnthrtrptrpalaglugln
500505510
tyrserprophecyspheleualaphecyspheglutyralaglyval
515520525
glnhishisglyleusertyrasncysserleuproleualapheasp
530535540
glysercysserglyileglnhispheseralametleuargaspglu
545550555560
valglyglyargalavalasnleuleuprosergluthrvalglnasp
565570575
iletyrglyilevalalalyslysvalasngluileleuglnalaasp
580585590
alaileasnglythraspasngluvalvalthrvalthraspgluasn
595600605
thrglygluileserglulysvallysleuglythrlysalaleuala
610615620
glyglntrpleualatyrglyvalthrargtrpvalthrlysargser
625630635640
valmetthrleualatyrglyserlysglupheglypheargglngln
645650655
valleugluphethrileglnproalaileaspserglylysglyleu
660665670
metphethrglnproasnglnalaalaglytyrmetalalysleuile
675680685
trpgluservalservalthrvalvalalaalavalglualametasn
690695700
trpleulysseralaalalysleuleualaalagluvallysasplys
705710715720
lysthrglygluileleuarglysargcysalavalhistrpvalthr
725730735
proaspglypheprovaltrpglnglutyrlyslysproileglnthr
740745750
argleuasnleumetpheleuglyglnpheargleuglnprothrile
755760765
asnthrasnlysaspsergluileaspalahislysglnglusergly
770775780
ilealaproasnphevalhisserglnaspglyserhisleuarglys
785790795800
thrvalvaltrpalahisglulystyrglyilegluserphealaleu
805810815
ilehisaspserpheglythrileproalaaspalaalaasnleuphe
820825830
lysalavalarggluthrmetvalaspthrtyrglusercysaspval
835840845
leualaaspphetyraspglnphealaaspglnleuhisglusergln
850855860
leuasplysmetproalaleuproalalysglyasnleuasnleuarg
865870875880
aspileleugluseraspphealapheala
885890
<210>40
<211>25
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>底物等
<400>40
ttttttttttttttttttttttttt25
- 还没有人留言评论。精彩留言会获得点赞!