用于提高碱基编辑精度的融合蛋白的制作方法
背景技术:
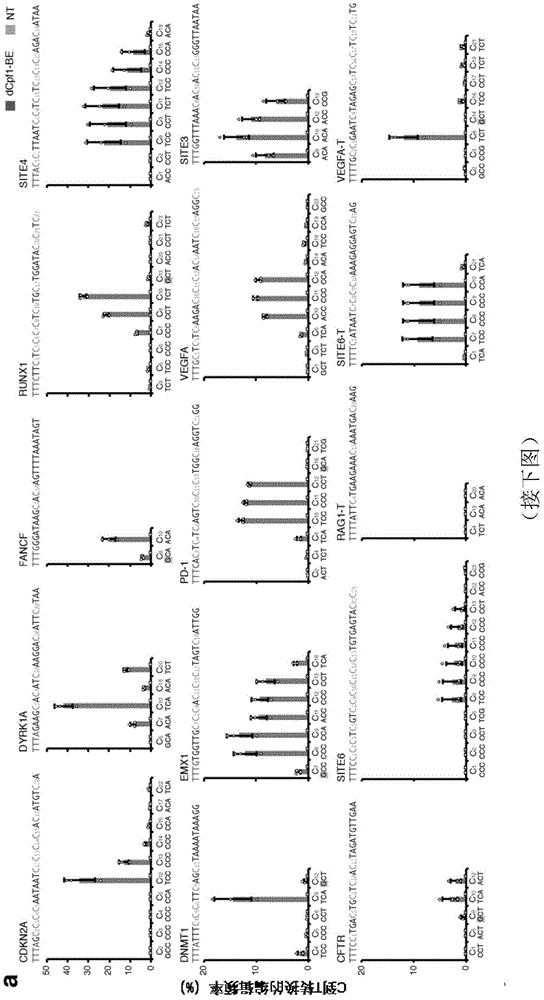
:可用于从遗传学角度操作细胞和活生物体基因组的基因组编辑在生命科学研究、生物技术、农业技术发展以及药物和临床开发中具有广泛的应用价值。例如,基因组编辑可用于纠正遗传疾病中的驱动突变,从而完全治愈活生物体中的这些疾病。crispr/cas(成簇的规律间隔的短回文重复序列/crispr相关蛋白)系统已成为最强大的基因组编辑工具,得益于其无与伦比的编辑效率、便利性以及在生物体中的潜在应用前景。由向导rna(grna)引导cas核酸酶,使其可以在各种细胞(细胞系和源自活生物体的细胞)的靶基因组位点产生dna双链断裂(dsb)。然后通过内源性dna修复系统修复这些dsb,可将其用于执行所需的基因组编辑。通常,dsb可以激活两种主要的dna修复途径,即非同源末端连接(nhej)和同源定向修复(hdr)。nhej可以在dsb周围的基因组dna区域中引入随机插入/缺失(indels),从而导致开放阅读框(orf)移位并最终导致基因失活。相反地,当触发hdr时,靶位点的基因组dna序列可以通过同源重组机制被外源供体dna模板序列所取代,从而可以纠正基因突变。近期发明了将crispr/cas系统与apobec(载脂蛋白bmrna编辑酶催化多肽样)胞苷脱氨酶家族整合的碱基编辑器(be),该编辑器极大地提高了crispr/cas9介导的基因纠正的效率。通过与cas9切口酶(ncas9)融合,大鼠apobec1(ra1)的胞嘧啶(c)脱氨基活性可以被定向到基因组中的靶碱基,并在这些碱基处催化c转化为胸腺嘧啶(t)的过程。但是,在当前最活跃的碱基编辑器中,依赖于cas9切口酶作为脱氨酶融合伴侣,会导致不必要的插入/缺失和非c到t的碱基取代的频率增加,并将编辑限制在富含g/c的前间区序列邻近基序(pam)序列。发明概述在一些实施方案中,本公开提供了可用于基因组编辑的碱基编辑器,其将无催化活性的毛螺旋菌(lachnospiraceaebacterium)cpf1(dlbcpf1)与胞苷脱氨酶进行组合。这样的碱基编辑器识别人类细胞中富含t的pam序列,并且以高的效率将c转换为t,同时插入/缺失、非c到t取代和脱靶编辑的水平低。这些都是与基于cas9的碱基编辑器相比的重大改进。此外,除了apobec1(a1)之外,当lbcpf1与apobec3(a3或apobec3a)融合时,还可以取得更高的编辑效率。除了大大提高编辑效率和精度外,基于lbcpf1的碱基编辑器还具有与基于cas9的碱基编辑器不同的编辑窗口。本发明中另一个有趣的发现是,游离的尿嘧啶dna糖基化酶抑制剂(ugi)结构域的存在可以进一步提高碱基编辑的效率和保真度。因此,本公开的一个实施方案提供了包含第一片段和第二片段的融合蛋白,所述第一片段包含胞苷脱氨酶,所述第二片段包含无催化活性的毛螺旋菌(lachnospiraceaebacterium)cpf1(dlbcpf1)。在一些实施方案中,胞苷脱氨酶是载脂蛋白bmrna编辑酶催化多肽样(apobec)蛋白。在一些实施方案中,apobec蛋白选自由apobec1、apobec2、apobec3a、apobec3b、apobec3c、apobec3d、apobec3f、apobec3g、apobec3h、apobec4和活化诱导的(胞苷)脱氨酶组成的群组。在一个实施方案中,apobec蛋白是apobec1。在一个实施方案中,apobec1蛋白包含w90y或r126e突变,或其组合。在一些实施方案中,apobec蛋白是apobec3a。在一些实施方案中,apobec3a蛋白具有一种或多种选自w104a、y130f、d131y、d31e和/或y132d突变的突变;组合突变的例子包括y130f-d131e-y132d、y130f-d131y-y132d。在一些实施方案中,融合蛋白还包含一个或多个尿嘧啶dna糖基化酶抑制剂(ugi)。在一些实施方案中,融合蛋白包含至少两个ugi。在一些实施方案中,至少一个ugi通过蛋白酶割切位点与第一片段和第二片段分开。在一些实施方案中,蛋白酶切割位点是自剪切肽。在一些实施方案中,融合蛋白进一步包含一个或多个核定位序列(nls)。在一些实施方案中,至少一个inls位于第二片段和第一ugi之间。在一些实施方案中,至少两个inls位于第二片段和第一ugi之间。在一些实施方案中,至少一个nls位于第一片段和第二片段的n末端。在一些实施方案中,至少一个nls位于第一片段和第二片段的c末端。在一些实施方案中,融合蛋白从n末端到c末端包含:第一nls、第一片段、第二片段、第二nls、第一ugi、第三nls、自剪切肽和第二ugi。在一些实施方案中,融合蛋白还包含第四nls,所述第四nls在第二片段和第一ugi之间。在一些实施方案中,融合蛋白还包含在第二自剪切肽和第三ugi,所述第二自剪切肽位于所述第二ugi的n末端。在一个实施方案中,还提供了一种编辑样品中核酸序列上的胞嘧啶的方法,其包括使样品与合适的指导rna(grna)和本发明的融合蛋白或编码该融合蛋白的多核苷酸接触。在一些实施方案中,胞嘧啶在核酸序列上的前间区序列邻近基序(pam)序列从3’端计的第6位和第22位核苷酸之间。在一些实施方案中,胞苷脱氨酶是apobec3a。在一些实施方案中,apobec3a蛋白具有一种或多种选自w104a、y130f、d131y、d31e和/或y132d突变的突变;组合突变的例子包括y130f-d131e-y132d、y130f-d131y-y132d。在一些实施方案中,胞嘧啶在核酸序列上的前间区序列邻近基序(pam)序列从3’端计的第8位和第13位核苷酸之间。在一些实施方案中,胞苷脱氨酶是apobec1蛋白。在一些实施方案中,胞嘧啶在pam序列从3’端计的第10位和第12位核苷酸之间。在一些实施方案中,胞苷脱氨酶是包含w90y或r126e突变或其组合的apobec1蛋白。在一些实施方案中,pam序列是富含t的pam序列。在一些实施方案中,该方法进一步包括使样品与未融合至cas蛋白的ugi或编码该ugi的多核苷酸接触。在另一个实施方案中,提供了一种融合蛋白,其包括:含有胞苷脱氨酶的第一片段,含有cas蛋白的第二片段,和通过蛋白酶切割位点与所述第一片段和所述第二片段分开的尿嘧啶dna糖基化酶抑制剂(ugi)。在一些实施方案中,蛋白酶切割位点是自剪切肽。在一些实施方案中,融合蛋白还包含通过第二蛋白酶切割位点与所述第一片段和所述第二片段分开的第二ugi。在一些实施方案中,融合蛋白还包含通过第三蛋白酶切割位点与第二ugi分开的第三ugi。在一些实施方案中,cas蛋白是cas9或cpf1。非限制性地,本发明还提供了编码本发明融合蛋白的多核苷酸、包含多核苷酸的构建体,包含多核苷酸或该构建体的细胞以及包含以上任何一种的组合物。附图说明图1,a-b。dcpf1-be介导的碱基编辑。(a)在所示间隔区中的每个胞嘧啶上测定dcpf1-be诱导的碱基编辑频率。dcpf1-be在g(阴影)后的胞嘧啶上显示出低效率的c到t转换的碱基编辑。胞嘧啶的计数以pam位置附近的碱基为位置1。(b)比较基于dcpf1和基于cas9的be介导的碱基编辑。在不同条件下,在指定的基因组靶位点分别确定了所示胞嘧啶的c到t转换的编辑频率、胞嘧啶取代率和插入/缺失频率。图中显示了dcpf1-be和cas9-be的靶位点序列和编辑窗口。nt为未转染。星号表示在未转染的293ft细胞中被检测的runx1位点的异常高的碱基插入/缺失频率(或扩增、测序、比对人为假象(alignmentartifact))。平均值±s.d.来自三次独立的实验。图2,a-h。dcpf1-be的改进。(a)使dcpf1-be中的apobec1突变以缩小编辑窗口。在不同条件下,在指定的基因组靶位点分别确定每个胞嘧啶的c到t转换的编辑频率。图中显示了dcpf1-be的靶位点序列和缩小的编辑窗口。主要编辑位点(c10-c12)为橙色,次要编辑位点(c1-c9和c13-c23)为绿色。(b)在指定的基因组靶位点确定主要编辑位点与次要编辑位点的比率。(c)对主要编辑位点与次要编辑位点的标准化比率进行统计分析,将dcpf1-be诱导的比率设为100%。dcpf1-be-ye诱导的主要编辑位点与次要编辑位点的比率明显更高。图中显示了中位数、四分位数间距(iqr)和1.5×iqr。n=来自3次独立实验的15个独立样本。(d-g)添加游离的ugi可提高dcpf1-bes诱导的编辑结果的纯度。不同条件下指定编辑位置胞嘧啶取代率(d,f)和统计分析(e,g)。(e,g)为:与dcpf1-be和dcpf1-be-ye相比,dcpf1-ebe和dcpf1-ebe-ye诱导的c到t转换的编辑结果明显更纯。图中显示了中位数和iqr。n=来自3次独立实验的9个独立样本。(h)基于cas9和基于dcpf1的be的概括。左侧示意图显示了cas9-be/sgrna/靶标dna和dcpf1-be/crrna/靶标dna的复合物。右侧列出了基于cas9和基于dcpf1的be系统中的相关功能。基于dyrk1a、fancf和runx1靶位点的碱基编辑进行比较。(a,b)平均值±s.d.来自三次独立的实验。(c,e,g)p值,单尾student’st检验。图3,a-c。在游离穿梭载体系统中,dlbcpf1-be0诱导了c到t转换的碱基编辑,但dascpf1-be0却没有。(a)为说明确定游离穿梭载体中dlbcpf1-be0或dascpf1-be0诱导的碱基编辑的程序的示意图。(b)包含由dascpf1-be0或dlbcpf1-be0诱导的突变的穿梭载体的大肠杆菌菌落数。(c)确定指定的胞嘧啶上c到t转换的编辑频率。胞嘧啶的计数以pam位置附近的碱基为位置1。根据(b)中的数据计算频率。平均值±s.d.来自3次独立的实验。图4,a-b。crrna间隔区长度对编辑效率的影响。(a)包含由dcpf1-be0和不同长度的crrna诱导的突变穿梭载体的菌落数。(b)在游离穿梭载体中所示的胞嘧啶上确定了由dcpf1-be0和不同长度的crrna诱导的c到t转换的编辑频率。间隔区长度在19到27个nt之间的crrna在大多数编辑位置显示出相似的碱基编辑效率。根据(a)中的数据计算频率。平均值±s.d.来自3次独立的实验。图5,a-e。dcpf1和ugi之间的内部nls(inls)对于dcpf1-be0诱导的碱基编辑很重要。(a)为dcpf1-be0和dcpf1-be0δinls的表达载体的设计示意图。(b)在用dcpf1-be0(蓝色)或dcpf1-be0δinls(绿色)处理后,在不同的基因组靶位点分别确定了指定的胞嘧啶的c到t转换的编辑频率。(c)归一化在基因组dna中dcpf1-be0和dcpf1-be0δinls诱导的c到t转换的编辑频率,将dcpf1-be0诱导的频率设置为100%。(d)对归一化的c到t转换的编辑频率的统计分析。与dcpf1-be0δinls相比,dcpf1-be0诱导的c到t转换的编辑频率更高。p值,单尾student’st检验。图中显示了中位数、四分位数间距(iqr)和1.5×iqr。n=来自3次独立实验的54个独立样本。(e)在基因组dna中的指定基因座处确定插入/缺失频率。293ft细胞经过dcpf1-be0处理(蓝色)、dcpf1-be0δinls处理(绿色)或未转染处理(灰色),然后进行深度测序。星号表示在未转染的293ft细胞中被检测的runx1位点的异常高的碱基插入/缺失频率(或扩增、测序、比对人为假象)。平均值±s.d.来自3次独立的实验。图6,a-e。额外的n末端nls增强了dcpf1-be在基因组dna中的碱基编辑效率。(a)说明dcpf1-be0和dcpf1-be表达载体的设计示意图。(b)在不同的基因组靶点分别确定了所示胞嘧啶的c到t转换的编辑频率。将293ft细胞用dcpf1-be0处理(蓝色)、dcpf1-be处理(紫色)或未转染(灰色)处理,之后进行深度测序。(c)归一化dcpf1-be0和dcpf1-be在基因组dna中诱导的c到t转换的编辑频率,将dcpf1-be0诱导的频率设置为100%。(d)对归一化的c到t转换的编辑频率的统计分析。与dcpf1-be0相比,dcpf1-be诱导的c到t转换的编辑频率更高。p值,单尾student’st检验。图中显示了中位数iqr和1.5×iqr。n=来自3次独立实验的54个独立样本。(e)在不同条件下,在基因组dna中指定的基因座处确定插入/缺失频率。星号表示未转染的293ft细胞中被检测的runx1位点异常高的碱基插入/缺失频率(或扩增、测序、比对人为假象)。平均值±s.d.来自3次独立的实验。图7,a-d。dcpf1-be诱导的碱基编辑的特征。(a)为指定的14个crrna在间隔区的每个胞嘧啶的碱基编辑频率汇总。这些数据表明,主要编辑窗口的范围是间隔区域中的位置8到13。(b)在不同条件下,在基因组dna中指定的基因座处确定插入/缺失频率。将293ft细胞用dcpf1-be处理(紫色)或未转染处理(灰色),之后进行深度测序。星号表示在未转染的293ft细胞中被检测的runx1位点的异常高的碱基插入/缺失频率(或扩增、测序、比对人为假象)。平均值±s.d.来自3次独立的实验。(c)在指定的胞嘧啶上单独确定由dcpf1-be诱导的胞嘧啶取代率。(d)统计分析表明,dcpf1-be诱导的碱基编辑结果的c到t的转换率显著高于ncas9-be3诱导的结果。图中显示了中位数和iqr。p值,单尾student’st检验。n=来自3次独立实验的42个独立样本。图8,a-c。在u2os细胞中dcpf1-be诱导了碱基编辑。(a)在指定的基因组靶位点分别确定指定胞嘧啶的c到t转换的编辑频率。用dcpf1-be处理(紫色)处理u2os细胞或不转染(灰色)u2os细胞,之后进行深度测序。(b)在不同条件下,在基因组dna中指定的基因座处确定插入/缺失频率。星号表示未转染的u2os细胞中被检测的runx1位点异常高的碱基插入/缺失频率(或扩增、测序、比对人为假象)。(c)在指定的胞嘧啶上单独确定由dcpf1-be诱导的胞嘧啶取代率。(a,b)平均值±s.d.来自3次独立的实验。图9,a-b。在预测的脱靶位点测定dcpf1-be诱导的碱基编辑。(a)指定crrna的靶位点和脱靶位点的序列。胞嘧啶的计数以pam位置附近的碱基为位置1。(b)在指定的靶位点、脱靶位点分别确定指定的胞嘧啶的c到t转换的编辑频率。将293ft细胞用dcpf1-be处理(紫色)处理或不进行转染(灰色),之后进行深度测序。平均值±s.d.来自3次独立的实验。图10,a-b。在间隔区以外的区域中未检测到大量的c到t的转换编辑。(a)为示意图,显示了pam区域、pam上游的20个nt区域和间隔区下游的20个nt区域。(b)在指定位点分别确定在间隔区以外的指定胞嘧啶的c到t转换的编辑频率。将293ft细胞用dcpf1-be处理(紫色)或作未转染处理(灰色),之后进行深度测序。平均值±s.d.来自3次独立的实验。图11,当间隔区中有多个胞嘧啶时,dcpf1-be引起多个c到t转换的编辑。确定不同的基因组靶位点上指定的胞嘧啶处dcpf1-be诱导的单个和多个c到t转换的编辑频率。深度测序的数据与图1a中的相同。平均值±s.d.来自3次独立的实验。图12,a-e。大鼠apobec1(ra1)中的w90y和r126e突变体缩小碱基编辑窗口至3个nt。(a)说明dcpf1-be、dcpf1-be-ye和dcpf1-be-yee表达载体设计的示意图。(b)dcpf1-be(紫色)和dcpf1-be-ye(洋红色)诱导的主要编辑位点与次要编辑位点的归一化比率,将dcpf1-be诱导的比率设为100%。(c)dcpf1-be和dcpf1-be-ye诱导的单个和多个c到t的转换率。(d)统计分析表明,dcpf1-be-ye诱导的单个c到t的转换率明显高于dcpf1-be诱导的单个c到t的转换率。p值,单尾student’st检验。图中显示了中位数和iqr。n=来自3次独立实验的15个独立样本。(e)在转染dcpf1-be(紫色)、转染dcpf1-be-ye(品红色)、转染dcpf1-be-yee(黄色)或未转染(灰色)的293ft细胞的指定基因组位点确定插入/缺失频率。星号表示在未转染的293ft细胞中被检测的runx1位点的异常高的碱基插入/缺失频率(或扩增、测序、比对人为假象)。(b,e)平均值±s.d.来自3次独立的实验。图13,a-f。三拷贝2a-ugi序列的融合基本上不影响编辑效率,也没有诱导可检测到的插入/缺失形成。(a)说明dcpf1-be和dcpf1-ebe表达载体设计的示意图。(b)在基因组dna的指定位置确定由dcpf1-be(紫色)和dcpf1-ebe(绿色)诱导的碱基编辑频率。(c)在指定的基因组位点确定插入/缺失频率。将293ft细胞用dcpf1-be处理(紫色)、dcpf1-ebe处理(绿色)或不进行转染(灰色),之后进行深度测序。(d)说明dcpf1-be-ye和dcpf1-ebe-ye的表达载体设计的示意图。(e)在基因组dna的指定位置确定由dcpf1-be-ye(品红色)和dcpf1-ebe-ye(棕色)诱导的碱基编辑频率。(f)在指定的基因组上确定插入/缺失频率。将293ft细胞用dcpf1-be-ye处理(品红色)、dcpf1-ebe-ye处理(棕色)或不转染(灰色),之后进行深度测序。星号表示在未转染的293ft细胞中被检测的runx1位点的异常高的碱基插入/缺失频率(或扩增、测序、比对人为假象)。平均值±s.d.来自3次独立的实验。图14,a-b。(a)说明cpf1-be和cpf1-a3-be表达载体设计的示意图。(b)在基因组dna的指定位点确定了cpf1-a3-be和cpf1-be诱导的碱基编辑效率。cpf1-a3-be的碱基编辑效率高于cpf1-be(在dyrk1a位点上的位置7和10,在runx1位点上位置7、8和10)。图15为cpf1-a3-be的编辑窗口。cpf1-a3-be的编辑窗口从被测试的前间隔区域中的位置6到22。图16,a-r。增强的碱基编辑系统。(a-i)通过在293ft细胞中共表达be3和来自不同载体的ugi来增强碱基编辑。说明sgrna、be3和ugi表达载体(a)设计的示意图。在指定的基因组位点针对指定的条件分别确定插入/缺失频率(b)、在sgrna靶标区域指定位置的c到t转换的编辑频率(d)、所需c到t转换的编辑与不需要的插入/缺失的比率(f)以及c到t、c到a和c到g的取代率(h),并作图如下:橙色代表be3,淡蓝色代表be+低水平ugi,蓝色代表be3+中水平ugi,深蓝色代表be3+高水平ugi,黑色代表dcas9。图中指示了sgfancf、sgsite2和sgrnf2靶区域中已编辑的cs的位置,将距pam远端的碱基作为位置1。统计分析突出显示了be3(橙色)和be3+高ugi(深蓝色)之间在插入/缺失频率(c)、在sgrna靶区域内指定位置的c到t转换的编辑频率(e)、所需c到t转换的编辑与不想要的插入/缺失的比率(g)以及c到t转换的取代率(i)的显著差异。(j-r)通过在293ft细胞中的ebe-s1和ebe-s3增强碱基编辑。说明sgrna、be3、ebe-s1和ebe-s3表达载体设计的示意图(j)。在指定的基因组位点分别确定be3(橙色)、ebe-s1(淡青色)和ebe-s3(青色)的插入/缺失频率(k)、c到t转换的编辑频率(m)、所需c到t转换的编辑与不想要的插入/缺失的比率(o)以及c到t、c到a和c到g的取代率(q)。图中标出了在sgemx1、sgfancf、sgsite2、sgsite4和sgrnf2靶区域中编辑的cs的位置,将距pam旁边的碱基作为位置1。统计分析突出显示了be3(橙色)和ebe-s3(青色)之间在插入/缺失频率(l)、c到t转换的编辑频率(n)、所需c到t转换的编辑与不需要的插入/缺失的比率(p),以及c到t转换的取代率(r)的显著差异。(b、d、f、k、m和o)误差线(±),3次重复的标准偏差。(c、e、g、i、l、n、p和r)p值,单尾student’st检验。发明详述定义应当注意的是,术语“一种”实体是指一种或多种该实体,例如“一种抗体”应当被理解为一种或多种抗体,因此,术语“一种”(或“一个”)、“一种或多种”和“至少一种”可以在本文中互换使用。在本发明中,术语“多肽”旨在涵盖单数的“多肽”以及复数的“多肽”,并且是指由通过酰胺键(也称为肽键)线性连接的单体(氨基酸)组成的分子。术语“多肽”是指两个或更多个氨基酸的任何单条链或多条链,并且不涉及产物的特定长度。因此,“多肽”的定义中包括肽、二肽、三肽、寡肽、“蛋白质”、“氨基酸链”或用于指两个或多个氨基酸链的任何其他术语,并且术语“多肽”可以用来代替上述任何一个术语,或者与上述任何一个术语交替使用。术语“多肽”也旨在指多肽表达后修饰的产物,包括但不限于糖基化、乙酰化、磷酸化、酰胺化、通过已知的保护/封闭基团衍生化、蛋白水解切割或非天然发生的氨基酸修饰。多肽可以源自天然生物来源或通过重组技术产生,但其不必从指定的核酸序列翻译所得。它可能以包括化学合成的任何方式产生。在本发明中,术语“重组”涉及多肽或多核苷酸,意指非天然存在的多肽或多核苷酸的形式,其非限制性实施例可以通过组合通常并不同时存在的多核苷酸或多肽来产生。“同源性”或“同一性”或“相似性”是指两个肽之间或两个核酸分子之间的序列相似性。可以通过比较每个序列中可以比对的位置来确定同源性。当被比较的序列中的位置被相同的碱基或氨基酸占据时,则分子在该位置是同源的。序列之间的同源程度是由序列共有的匹配或同源位置的数目组成的一个函数。术语“不相关的”或“非同源的”序列表示与本发明公开的序列之一有小于40%的同一性,但优选小于25%的同一性。多核苷酸或多核苷酸区域(或多肽或多肽区域)与另一序列具有一定百分比(例如,60%、65%、70%、75%、80%、85%、90%、95%、98%或者99%)的“序列同一性”是指当序列比对时,所比较的两个序列中该百分比的碱基(或氨基酸)相同。该比对和同源性百分比或序列同一性可以使用本领域已知的软件程序,比如ausubeletal.eds.(2007)currentprotocolsinmolecularbiology中所述的软件程序来确定。优选使用默认参数进行比对。其中一种比对程序是使用默认参数的blast。术语“等价的核酸或多核苷酸”是指具有与核酸或其互补序列的核苷酸序列具有一定程度的同源性或序列同一性的核苷酸序列的核酸。双链核酸的同源物意指包括具有与其或其互补序列具有一定同源性的核苷酸序列的核酸。一方面,核酸的同源物能够与核酸或其互补序列杂交。同样地,“等价的多肽”是指与参考多肽的氨基酸序列具有一定同源性或序列同一性的多肽。在某些方面,序列同一性为至少约70%、75%、80%、85%、90%、95%、98%或99%。在某些方面,与参考的多肽或多核苷酸相比,等价的多肽或多核苷酸具有1、2、3、4或5个添加、缺失、取代及其组合。在某些方面,等价的序列保留参考序列的活性(例如表位结合)或结构(例如盐桥)。杂交反应可以在不同的“严谨性”条件下进行。通常在约40℃条件下,在约10×ssc或相同等离子强度/温度的溶液中进行低严谨的杂交反应。通常在约50℃条件下,在约6×ssc中进行中度严谨的杂交反应,通常在约60℃条件下,在约1×ssc中进行高度严谨的杂交反应。杂交反应也可以在本领域技术人员熟知的“生理条件”下进行。生理条件的非限制性实施例指在细胞中通常存在的温度、离子强度、ph和mg2+浓度。多核苷酸由四个核苷酸碱基的特定序列组成:腺嘌呤(a)、胞嘧啶(c)、鸟嘌呤(g)、胸腺嘧啶(t)、和当多核苷酸是rna时用于置换胸腺嘧啶的尿嘧啶(u)。因此,术语“多核苷酸序列”是多核苷酸分子的字母表示。该字母表示可以被输入到具有中央处理单元的计算机中的数据库中,并用于生物信息学应用,例如用于功能基因组学和同源性搜索。术语“多态性”是指多种形式的基因或其部分的共存,具有至少两种不同形式(即两种不同的核苷酸序列)的基因的一部分被称为“基因的多态性区域”。多态性区域可以是单核苷酸,在不同的等位基因中其具有不同的同一性。术语“多核苷酸”和“寡核苷酸”可互换使用,是指无论是脱氧核糖核苷酸还是核糖核苷酸或其类似物的任何长度的核苷酸的聚合形式。多核苷酸可以具有任何三维结构并且可以执行已知或未知的任何功能。以下是非限制性的多核苷酸的实施例:基因或基因片段(例如探针、引物、est或sage标签)、外显子、内含子、信使rna(mrna)、转运rna、核糖体rna、核糖酶、cdna、dsrna、sirna、mirna、重组多核苷酸、分支的多核苷酸、质粒、载体、任何序列的分离的dna、任何序列的分离的rna、核酸探针和引物。多核苷酸可以包含修饰的核苷酸,例如甲基化的核苷酸和核苷酸类似物。如果存在该修饰,则对核苷酸的结构修饰可以在组装多核苷酸之前或之后进行。核苷酸的序列可以被非核苷酸组分中断。聚合后可以进一步修饰多核苷酸,例如通过与标记组分缀合。这个术语也指双链和单链分子。除另有说明或要求外,本公开的任何多核苷酸的实施例包括双链形式和已知或预测构成双链形式的两种可互补单链形式中的每一种。术语“编码”应用于多核苷酸时,是指被称为“编码”多肽的多核苷酸,如果在其天然状态或当通过本领域技术人员公知的方法操作时,其可以被转录和/或翻译以产生多肽和/或其片段的mrna。反义链是这种核酸的互补序列,其编码序列可以从中推导出来。融合蛋白如实施例1所示,通过将大鼠胞苷脱氨酶apobec1融合到无催化活性形式的毛螺旋菌(lachnospiraceaebacterium)cpf1(lbcpf1)中,开发了一种基于crispr-cpf1的碱基编辑器。该碱基编辑器识别人类细胞中富含t的pam序列,并以高的效率将c转换为t,同时插入/缺失、非c到t取代和脱靶编辑的效率低。这些都是与基于cas9的碱基编辑器相比的重大改进。此外,除了apobec1(a1)外,当lbcpf1与apobec3(a3或apobec3a)融合时,可以实现更高的编辑效率。除了大大提高了编辑效率和精度外,基于lbcpf1的碱基编辑器在编辑窗口方面还与基于cas9的碱基编辑器有所不同。通常,基于cas9的碱基编辑器的编辑窗口是从位置4到位置8,观察到的基于cpf1的碱基编辑器的编辑窗口是从位置8到位置13(cpf1-a1碱基编辑器)以及从位置6到位置22(cpf1-a3碱基编辑器)。当cpf1与apobec突变体(例如,具有w90y和r126e突变的a1)融合时,可以将编辑窗口缩小到位置10到位置12,从而提供了用于更精确地针对位置进行编辑的工具。本公开中的另一个有趣的发现是,游离尿嘧啶dna糖基化酶抑制剂(ugi)结构域的存在可以进一步提高碱基编辑的效率和保真度。ugi已被用作碱基编辑器中的融合部分,通常将其置于碱基编辑器的c端。但是,添加游离的ugi带来的额外好处是令人惊讶和预料不到的。为了方便和良好的控制,在本公开的一个实施方案中,通过包含蛋白酶切割位点的接头将ugi与碱基编辑器融合,从而使得在表达时产生游离的ugi。本公开的另一个有趣的发现是,在碱基编辑器中添加更多的内部sv40核定位序列(inls)可以进一步提高编辑效率。可以将一个、两个或多个inls插入cpf1或cas9与ugi之间。在一些实施方案中,可以将inls添加至胞苷脱氨酶和cpf1或cas9的n末端或c末端侧。因此,本公开的一个实施方案提供了一种融合蛋白,其包括:含有胞苷脱氨酶的第一片段,和含有无催化活性的毛螺旋菌(lachnospiraceaebacterium)cpf1(dlbcpf1)的第二片段。“胞苷脱氨酶”是指分别催化胞苷和脱氧胞苷不可逆水解为尿苷和脱氧尿苷的酶。胞苷脱氨酶维持细胞内的嘧啶存储。胞苷脱氨基酶家族是apobec(“载脂蛋白bmrna编辑酶,催化多肽样”)。该家族的成员是c到u转换的编辑酶。apobec样蛋白的n末端结构域是催化结构域,而c末端结构域是伪催化结构域。更具体地,该催化结构域是锌依赖性胞苷脱氨酶结构域,并且对于胞苷脱氨是重要的。通过apobec-1进行rna编辑需要同二聚体作用,该复合物与rna结合蛋白相互作用形成编辑体。apobec蛋白的非限制性实例包括apobec1、apobec2、apobec3a、apobec3b、apobec3c、apobec3d、apobec3f、apobec3g、apobec3h、apobec4和激活诱导的(胞苷)脱氨酶。已知apobec蛋白质的各种突变体为碱基编辑器带来了不同的编辑特征。例如,对于人类apobec3a而言,其某些突变体(例如y130f、y132d、w104a和d131y)的编辑效率甚至超过野生型的人类apobec3a。因此,术语apobec及其每个家族成员也涵盖与相应的野生型apobec蛋白序列具有一定程度同一性(例如70%、75%、80%、85%、90%、95%、98%、99%)的变体和突变体,并且保留了胞苷脱氨活性。变体和突变体可以通过氨基酸的添加、缺失和/或取代衍生。在一些实施方案中,此类取代是保守取代。毛螺旋菌(lachnospiraceaebacterium)cpf1(lbcpf1)是一大组cpf1蛋白中的其中一种。cpf1是一种cas蛋白。术语“cas蛋白”或“成簇的规律间隔的短回文重复序列(cas)蛋白”是指化脓性链球菌(streptococcuspyogenes)以及其他细菌中与crispr(成簇的规律间隔的短回文重复序列)自适应免疫系统相关的rna引导的dna核酸内切酶。cas蛋白包括cas9蛋白、cas12a(cpf1)蛋白、cas13蛋白和各种工程改造的对应物。下表提供了示例性的cas蛋白。表a.示例性cas蛋白在一些实施方案中,融合蛋白包括:含有apobec1蛋白的第一片段和含有无催化活性的lbcpf1的第二片段。在一些实施方案中,融合蛋白包括:含有apobec1蛋白的第一片段和含有无催化活性lbcpf1的第二片段。在一些实施方案中,融合蛋白包括:含有apobec3a蛋白的第一片段和含有无催化活性的lbcpf1的第二片段。在一些实施方案中,融合蛋白包括:含有apobec3a蛋白的第一片段和含有无催化活性的lbcpf1的第二片段。在一些实施方案中,胞苷脱氨酶是人蛋白。在一些实施方案中,胞苷脱氨酶是大鼠蛋白。在一些实施方案中,胞苷脱氨酶是小鼠蛋白。在一些实施方案中,胞苷脱氨酶包括一个、两个或三个氨基酸取代,同时保留胞苷脱氨酶活性(例如具有w90y和/或r126e突变的apobec1)。融合蛋白可以包括其他片段,例如尿嘧啶dna糖基化酶抑制剂(ugi)和核定位序列(nls)。可以从枯草芽孢杆菌噬菌体pbs1中制备得到的“尿嘧啶糖基化酶抑制剂”(ugi)是一种小蛋白(9.5kda),其可以抑制大肠杆菌尿嘧啶-dna糖基化酶(udg)以及其他物种的udg。udg的抑制是通过可逆的蛋白质结合(以1:1ugd:ugi化学计量比进行)发生的。ugi能够解离udg-dna复合物。在芽孢杆菌噬菌体ar9(yp_009283008.1)中发现了ugi的非限制性实例。在一些实施方案中,ugi包含如seqidno:8所示的氨基酸序列或与seqidno:8具有至少70%、75%、80%、85%、90%或95%同一性的序列并保留尿嘧啶糖基化酶抑制活性。在一些实施方案中,将ugi置于胞苷脱氨酶-cpf1部分的c末端侧。在一些实施方案中,融合蛋白包含至少两个ugi。在一些实施方案中,至少一个ugi通过蛋白酶切割位点与脱氨酶-cpf1部分分开。因此在表达后,ugi可能会从融合蛋白上切割下来成为独立的蛋白,与脱氨酶-cpf1部分分开。如实施例2所示,这种游离的ugi(即未与脱氨酶-cpf1融合蛋白融合的ugi蛋白)可以进一步提高碱基编辑器的效率和特异性。在一些实施方案中,融合蛋白包括至少两个这样的切割位点分离的ugi单元。在一些实施方案中,蛋白酶切割位点是自剪切肽,例如2a肽。“2a肽”是18-22个氨基酸长度的病毒寡肽,其在真核细胞的翻译过程中介导多肽的“切割”。名称“2a”是指病毒基因组的特定区域,通常以其源自的病毒来命名不同的病毒2a。最早发现的2a是f2a(口蹄疫病毒),其后还鉴定了e2a(马甲鼻炎病毒)、p2a(猪破伤风病毒12a)和t2a(胸海蛇病毒2a)。seqidno:9-11中提供了2a肽的一些非限制性实例。在一些实施方案中,融合蛋白可包括一个或多个核定位序列(nls)。“核定位信号或序列”(nls)是标记通过核转运输入到细胞核中的蛋白质的一种氨基酸序列。通常,该信号由暴露于蛋白质表面的一个或多个带正电荷的赖氨酸或精氨酸的短序列组成。不同的核定位蛋白可能共享相同的nls。nls具有与核输出信号(nes)相反的功能,后者将蛋白质靶向于核外。nls的非限制性实例是内部sv40核定位序列(inls)。在一些实施方案中,nls包含如seqidno:7所示的氨基酸序列或与seqidno:7具有至少70%、75%、80%、85%、90%或95%同一性的序列,并保留了核定位活性。在一些实施方案中,至少一个nls位于第一片段和第二片段(胞苷脱氨酶-cpf1部分)的c末端,例如,在第二片段(包括cpf1)和ugi之间。在一些实施方案中,至少两个nls位于第二片段和ugi之间。在一些实施方案中,至少三个nls位于第二片段和ugi之间。在一些实施方案中,至少一个nls位于第一片段和第二片段(胞苷脱氨酶-cpf1部分)的n末端。融合蛋白中组分排列的非限制性实例,从n端至c端包括,(a)nls,胞苷脱氨酶,cpf1,nls,ugi,nls,2a和ugi;(b)nls,胞苷脱氨酶,cpf1,nls,nls,ugi,nls,2a和ugi;(c)nls,胞苷脱氨酶,cpf1,nls,ugi,nls,2a,ugi,2a和ugi;(d)nls,胞苷脱氨酶,cpf1,nls,ugi,nls,2a,ugi,2a,ugi,2a和ugi。在一些实施方案中,任选地在融合蛋白中的每个片段之间提供肽接头。在一些实施方案中,肽接头具有1-100个(或3-20个、4-15个,无限制)氨基酸残基。在一些实施方案中,肽接头的氨基酸残基的至少10%、20%、30%、40%、50%、60%、70%、80%或90%是选自由丙氨酸、甘氨酸、半胱氨酸和丝氨酸组成的群组的氨基酸残基。表1.示例性序列对于本发明的任何融合蛋白,还提供了其生物等效物。在一些实施方案中,生物等效物与参考的融合蛋白具有至少约70%、75%、80%、85%、90%、95%、98%或99%的序列同一性。优选地,生物等效物保留了参考融合蛋白的期望活性。在一些实施方案中,生物等效物通过包括1个、2个、3个、4个、5个或更多个氨基酸的添加、缺失、取代或其组合而衍生。在一些实施方案中,该取代是保守氨基酸取代。“保守性氨基酸取代”是指其中氨基酸残基被具有相似侧链的氨基酸残基置换。本领域已经定义了具有相似侧链的氨基酸家族,包括碱性侧链(例如赖氨酸、精氨酸、组氨酸)、酸性侧链(例如天冬氨酸、谷氨酸)、不带电荷的极性侧链(例如甘氨酸、天冬酰胺、谷氨酰胺、丝氨酸、苏氨酸、酪氨酸、半胱氨酸)、非极性侧链(例如丙氨酸、缬氨酸、亮氨酸、异亮氨酸、脯氨酸、苯丙氨酸、甲硫氨酸、色氨酸)、β-分支侧链(例如苏氨酸、缬氨酸、异亮氨酸)和芳族侧链(例如酪氨酸、苯丙氨酸、色氨酸、组氨酸)。因此,优选将免疫球蛋白多肽中的非必需氨基酸残基置换为来自同一侧链家族的另一种氨基酸残基。在另一个实施方案中,一串氨基酸可以用结构相似的串代替,该串在侧链家族成员的顺序和/或组成上不同。下表中提供了保守的氨基酸取代的非限制性实施例,其中0或更高的类似得分表明了两个氨基酸之间的保守的可取代性。表b氨基酸相似性矩阵cgpsatdenqhkrvmilfyww-8-7-6-2-6-5-7-7-4-5-3-32-6-4-5-20017y0-5-5-3-3-3-4-4-2-40-4-5-2-2-1-1710f-4-5-5-3-4-3-6-5-4-5-2-5-4-10129l-6-4-3-3-2-2-4-3-3-2-2-3-32426i-2-3-2-1-10-2-2-2-2-2-2-2425m-5-3-2-2-1-1-3-20-1-20026v-2-1-1-100-2-2-2-2-2-2-24r-4-300-2-1-1-101236k-5-2-10-10001105h-3-20-1-1-111236q-5-10-10-12214n-40-1100212e-50-100034d-51-10004t-200113a-21112s0111p-3-16g-35c12表c保守的氨基酸取代融合蛋白的用途如本发明所提供的,胞苷脱氨酶-lbcpf1融合蛋白是一种高效、高保真的碱基编辑器。因此,这种碱基编辑器可用于生物样品中有效的基因组编辑。此外,鉴于胞苷脱氨酶-lbcpf1碱基编辑器具有不同的编辑窗口,并且采用了与基于cas9的系统中不同的pam序列,因此这些新的碱基编辑器是对cas9系统的扩充。本公开提供了组合物和方法。这种组合物包含有效量的融合蛋白和可接受的载体。在一些实施方案中,所述组合物还包含与靶dna具有所需互补性的指导rna。这样的组合物可以用于样本中的碱基编辑。融合蛋白及其组合物可用于碱基编辑。在一个实施方案中提供了一种用于编辑靶多核苷酸的方法,该方法包括将本公开的靶多核苷酸融合蛋白和与靶多核苷酸具有至少部分序列互补性的指导rna与该靶多核苷酸接触,其中所述编辑包括将靶多核苷酸中胞嘧啶(c)脱氨基。在一个实施方案中,提供了一种编辑样品中核酸序列上的胞嘧啶的方法。在一些实施方案中,该方法需要使样品接触本公开的融合蛋白或编码该融合蛋白的多核苷酸。在一些实施方案中,进一步添加合适的指导rna。指导rna的设计是技术人员容易获得的。在一些实施方案中,胞嘧啶在核酸序列上的前间区序列邻近基序(pam)序列3’端的第8位和第13位核苷酸之间。用于该编辑窗口的胞苷脱氨酶可以是apobec1。在一些实施方案中,胞嘧啶在pam序列3’端的第10位和第12位核苷酸位置之间。为了缩小编辑范围,可能需要突变的apobec1蛋白(例如,具有w90y和r126e突变的apobec1蛋白)。在一些实施方案中,胞嘧啶在核酸序列上前间区序列邻近基序(pam)序列3’端的第6位和第22位核苷酸位置之间。用于该编辑窗口的胞苷脱氨酶可以是apobec3。在一些实施方案中,编辑窗口更窄。为了缩小编辑范围,可能需要突变的apobec3a蛋白(例如,带有w104a、y130f、d131y、d31e和/或y132d突变的apobec3a蛋白;组合突变的示例包括y130f-d131e-y132d、y130f-d131y-y132d;参见seqidno:12-17)。在一些实施方案中,pam序列是富含t的pam序列。在一些实施方案中,还添加了不与cas蛋白融合的游离ugi、或编码该游离ugi的多核苷酸。融合蛋白(和指导rna)与靶多核苷酸之间的接触可以在体外,特别是在细胞培养物中。当接触是离体或在体内时,融合蛋白可表现出临床的/治疗的意义。体内接触可以不受限制地施用于活的受试者,例如人、动物、酵母、植物、细菌、病毒。游离的ugi和使用本公开的发现是,游离尿嘧啶dna糖基化酶抑制剂(ugi)结构域的存在可以进一步提高碱基编辑的效率和保真度。在一个实施方案中,提供了一种编辑靶多核苷酸的方法,其包括使碱基编辑器和未与cas蛋白融合的ugi与靶多核苷酸接触。在一些实施方案中,进一步添加合适的指导rna。指导rna的设计是本领域技术人员容易获得的。本发明还提供了一种融合蛋白,所述融合蛋白包括:含有胞苷脱氨酶的第一片段,含有cas蛋白的第二片段,以及通过蛋白酶切割位点与所述第一片段和所述第二片段分开的尿嘧啶dna糖基化酶抑制剂(ugi)。在一些实施方案中,蛋白酶切割位点是自剪切肽,例如a2肽。在一些实施方案中,融合蛋白还包括通过第二蛋白酶切割位点连接的第二ugi。在一些实施方案中,融合蛋白还包括通过第三蛋白酶切割位点连接的第三ugi。以上描述了各种胞苷脱氨酶和cas蛋白。在一些实施方案中,胞苷脱氨酶选自由apobec1、apobec2、apobec3a、apobec3b、apobec3c、apobec3d、apobec3f、apobec3g、apobec3h、apobec4和激活诱导的(胞苷)组成的群组。在一些实施方案中,cas蛋白是cas9或cpf1。在一个实施方案中,提供了一种编辑样品中核酸序列的方法。在一些实施方案中,该方法需要使样品接触本公开的融合蛋白或编码该融合蛋白的多核苷酸。在一些实施例中,进一步添加合适的指导rna。本发明还提供了组合物和方法。此类组合物包含有效量的融合蛋白和可接受的载体。在一些实施方案中,所述组合物还包含与靶dna具有所需互补性的指导rna。这样的组合物可以用于样本中的碱基编辑。融合蛋白(和指导rna)与靶多核苷酸之间的接触可以在体外,特别是在细胞培养物中。当该接触是离体的或在体内时,融合蛋白可表现出临床/治疗意义。体内接触可以不限制于施用在活的受试者,例如人、动物、酵母、植物、细菌、病毒。具体实施方式实施例1:融合蛋白能够精确编辑人类基因组中富含a/t的区域中的单个碱基crispr-cas9碱基编辑器(be)的靶向范围局限在富含g/c的pam序列。为了克服这一局限性,该实施例通过将大鼠胞苷脱氨酶apobec1与毛螺旋菌(lachnospiraceaebacterium)cpf1的无催化活性形式融合,开发了一种基于crispr-cpf1的碱基编辑器。该碱基编辑器识别人细胞中富含t的pam序列,并将其中的c转换为t,且插入/缺失、非c到t的取代和脱靶编辑的水平很低。方法与材料质粒构建pst1374-lb-cpf1-nls为商业合成。使用两个引物对(lb_d971a_f/lb_r4635)(lb_d971a_r/lb_f2096)扩增含d832a的片段lbcpf1-d832a。接着使用两个引物对(lb_e1006a_f/lb_e1006a_r)(lb_d1225a_f/lb_d1225a_r)引入突变e925a和d1148a。使用质粒重组试剂盒(vazyme,c112-02)将含有d832a、e925a和d1148a的dlbcpf1克隆到psti和apai线性化的pst1374-lbcpf1-nls中,以产生dlbcpf1表达质粒pst1374-dlbcpf1-nls。使用两个引物对(lb_be3_f1/lb_be3_r1)(lb_be3_f2/cpf_be3_fu_r2)扩增dlbcpf1-sv40nls-ugi片段,将其克隆到smai和pmei线性化的pcmv-be3中以产生dlbcpf1-be0(dcpf1-be0)表达载体pcmv-apobec1-xten-dlbcpf1(d832a/e925a/d1148a)-sv40nls-sggs-ugi-sv40nls。pst1374-as-cpf1-nls为商业合成。使用两个引物对(as_d917a_f/as_r4871)(as_d917a_r/as_f2155)扩增含d908a的片段ascpf1-d908a。然后使用两个引物对(as_e1006a_f/as_e1006a_r)(as_d1225a_f/as_d1225a_r)引入突变e993a和d1235a。将含有d908a、e993a和d1235a的片段dascpf1克隆到psti和apai线性化的pst1374-ascpf1-nls,以产生dascpf1表达质粒pst1374-dascpf1-nls。使用两个引物对(as_be3_f1/as_be3_r1)(as_be3_f2/cpf_be3_fu_r2)扩增dascpf1-sv40nls-ugi片段,将其克隆到smai和pmei线性化的pcmv-be3中以产生dascpf1-a0表达载体pcmv-apobec1-xten-dascpf1(d908a/e993a/d1235a)-sv40nls-sggs-ugi-sv40nls。将寡核苷酸(l079_lbcpf1scaffold_for/l080_lbcpf1scaffold_rev,l081_ascpf1scaffold_for/l082_ascpf1scaffold_rev)退火并连接到bsai和ecori线性化的pgl3-u6-sgrna-pgk-puromycin(addgene,51133),以产生lb-crrna和as-crrna表达载体plb-cpf1-pgl3-u6-sgrna和pas-cpf1-pgl3-u6-sgrna。将寡核苷酸supf_cpf1_sg1_for/supf_cpf1_sg1_rev,supf_cpf1_sg2_for/supf_cpf1_sg2_rev,supf_cpf1_sg3_for/supf_cpf1_sg3_rev或其他具有不同长度的寡核苷酸对退火并连接到bsai线性化的plb-cpf1-pgl3-u6-sgrna或pas-cpf1-pgl3-u6-sgrna,以产生穿梭载体psp189中靶向supf基因的lb-crrna或as-crrna的表达载体。使用两个引物对(lb_be3_f1/lb_r)(ugi_f/cpf_be3_fu_r2)扩增dlbcpf1-sggs-ugi片段,将其克隆到smai和pmei线性化的dlbcpf1-be0(dcpf1-be0)表达载体中以产生dlbcpf1-be0δinls表达载体pcmv-apobec1-xten-dlbcpf1(d832a/e925a/d1148a)-sggs-ugi-sv40nls。使用引物对(1xnls_pcrf/1xnls_pcrr)从pcmv-be3中扩增出片段nls-apobec1,并将跑胶纯化后的nls-apobec1片段连接至smai和noti线性化的dcpf1-be0表达载体,以产生dcpf1-be表达载体pcmv-sv40nls-apobec1-xten-dlbcpf1(d832a/e925a/d1148a)-sv40nls-sggs-ugi-sv40nls。使用两个引物对(apobec_w90y_f1/1xnls_pcrr)(1xnls_pcrf/apobec_w90y_r1)扩增含w90y的片段apobec-y。使用两个引物对(apobec_r126e_f/apobec_r126e_r)(apobec_r132e_f/apobec_r132e_r)引入突变r126e和r132e。将apobec-ye和apobec-yee片段分别连接到noti和smai线性化的dcpf1-be表达载体中,以产生dcpf1-be-ye和dcpf1-be-yee表达载体pcmv-sv40nls-apobec1(w90y/r126e)-xten-dlbcpf1(d832a/e925a/d1148a)-sv40nls-sggs-ugi-sv40nls和pcmv-sv40nls-apobec1(w90y/r126e/r132e)-xten-dlbcpf1(d832a/e925a/d1148a)-sv40nls-sggs-ugi-sv40nls。使用引物对(lb_f2096/be8.1_pmei_apai_r)将apai位点引入dcpf1-be表达载体中以产生pcmv-dcpf1-be-apai。使用引物对(apai_1t2augi_f/pmei_3t2augi_r)从商业合成的dna片段3×2a-ugi中扩增3×2a-ugi片段,并将3×2a-ugi片段连接到pmei和apai线性化的pcmv-dcpf1-be-apai,以产生dcpf1-ebe表达载体pcmv-sv40nls-apobec1-xten-dlbcpf1(d832a/e925a/d1148a)-sv40nls-sggs-ugi-sv40nls-t2a-ugi-sv40nls-p2a-ugi-sv40nls-t2a-ugi-sv40nls。将apobec1-ye片段连接到noti和smai线性化的dcpf1-ebe表达载体中,以产生dcpf1-ebe-ye表达载体pcmv-sv40nls-apobec1(w90y/r126e)-xten-dlbcpf1(d832a/e925a/d1148a)-sv40nls-sggs-ugi-sv40nls-t2a-ugi-sv40nls-p2a-ugi-sv40nls-t2a-ugi-sv40nls。将寡核苷酸hcdkn2a_cpf1_sg1_for/hcdkn2a_cpf1_sg1_rev退火并连接到bsai线性化的plb-cpf1-pgl3-u6-sgrna中,以产生crcdkn2a表达载体pcrcdkn2a。将寡核苷酸hcdkn2a_cpfsp_sg1_for/hcdkn2a_cpfsp_sg1_rev退火并连接到bsai线性化的pgl3-u6-sgrna-pgk-puromycin(嘌呤霉素)中,以产生sgcdkn2a表达载体psgcdkn2a。通过相同的方式构建其他crrna和sgrna表达载体。细胞培养和转染购自atcc的293ft和u2os培养在dmem(10566,gibco/thermofisherscientific)+10%fbs(16000-044,gibco/thermofisherscientific)中,并经过检测以排除有支原体污染。为了在游离的穿梭载体中进行碱基编辑,将293ft细胞以每孔5×105的密度铺在6孔板中,并用500μl不含血清的opti-mem转染,所述opti-mem中含有4μllipofectamineltx(life,invitrogen)、2μllipofectamineplus(life,invitrogen)、1μgdlbcpf1-be0表达载体(或dascpf1-be0表达载体)、0.5μgcrrna表达质粒和0.5μg穿梭载体psp189。48小时后,用tianprepmini质粒试剂盒(dp103-a,tiangen)从细胞中提取质粒。为了在基因组dna中进行碱基编辑,将293ft和u2os细胞以每孔2×105的密度铺到24孔板中,并用500μl不含血清的opti-mem转染,所述opti-mem中含有5.04μllipofectamineltx(life,invitrogen)、1.68μllipofectamineplus(life,invitrogen)、1μgdcpf1-be0表达载体(dcpf1-be0δinls,dcpf1-be,dcpf1-be-ye,dcpf1-be-yee,dcpf1-ebe,dcpf1-ebe-ye表达载体或pcmv-be2,pcmv-be3)和0.68μgcrrna或sgrna表达质粒。72小时后,用quickextracttmdna提取溶液(qe09050,epicentre)从细胞中提取基因组dna。蓝/白菌落筛选从转染的细胞中提取的质粒用dpni消化(去除未复制的输入质粒),并转化到大肠杆菌mbm7070(laczuag_amber)中,使其在含有50μg/ml卡那霉素、1mmiptg和0.03%bluo-gal(invitrogen/lifetechnologies,纽约州格兰德岛)的lb平板上生长,37℃过夜,然后在室温下放置一天(以最大程度地改善颜色)。为了确定突变谱,随机选择白色菌落用于sanger测序。dna文库的制备和测序靶基因组位点通过高保真dna聚合酶primestarhs(clonetech)进行pcr扩增,其引物侧翼于每个检测的sgrna靶位点。通过使用truseqchip样品制备试剂盒(illumina)进行了一些小修改,制备索引的dna文库。简而言之,从基因组dna区域扩增的pcr产物通过covariss220进行片段化处理。然后使用truseqchip样品制备试剂盒(illumina)pcr扩增片段化的dna。用qubit高灵敏度dna试剂盒(invitrogen)定量后,将具有不同标签的pcr产物汇集在一起,通过中国上海cas-mpg计算生物学合作伙伴研究所量化生物学平台(omicscore)的illuminahiseq2500(2x150)或hiseqx-10(2x150)进行深度测序。原始读数的质量通过fastqc(www.bioinformatics.babraham.ac.uk/projects/fastqc/,v0.11.4)进行评估。对于配对的末端测序,仅使用r1读数。剪切两端具有phred质量值低于28的接头序列和读段序列。然后使用bwa-mem算法(bwav0.7.9a)将剪切的读段映射到靶序列。将其与samtools(v0.1.18)合并在一起后,进一步计算出插入/缺失和碱基取代。插入/缺失频率计算对于cpf1,根据pam位点(55bp),在从上游3个核苷酸到下游48个核苷酸的比对区域估计插入/缺失。对于cas9,根据pam位点(50bp),在从上游8个核苷酸到靶位点到下游19个核苷酸的比对区域中估计插入/缺失。随后通过将包含至少一个插入和/或缺失的核苷酸的读段除以同一区域的所有映射读段来计算插入/缺失频率。碱基取代计算在所检测的sgrna(或crrna)靶位点的每个位置挑选出碱基取代,这些位点至少映射了1,000个独立的读数,并且仅在靶碱基编辑位点观察到了明显的碱基取代。通过将碱基取代读数除以总读数来计算碱基取代频率。统计分析该项研究中通过单尾student'st-检验计算得出p值。结果cpf1(cas12a)是某些方面与cas9不同的另一种cas蛋白。本实施例检测了两个不同的cpf1进行碱基编辑的能力。大鼠apobec1与无催化活性的氨基酸球菌(acidaminococcussp)cpf1(dascpf1)或无催化活性的毛螺旋菌(lachnospiraceaebacterium)cpf1(dlbcpf1)以及尿嘧啶dna糖基化酶抑制剂(ugi)融合,以生成两种基于dcpf1的碱基编辑器(be):dascpf1-be0和dlbcpf1-be0(图3a)。该实施例首先在大肠杆菌质粒衍生的游离型穿梭载体报告系统中测试了它们的编辑潜力(图3a),该系统已被证明是探测人类细胞中碱基取代的灵敏工具。dlbcpf1-be0在靶区域诱导了高水平的c到t转换的碱基编辑(单胞嘧啶的编辑频率范围为44%至74%),而dascpf1-be0在相似条件下未显示可检测的碱基编辑(图3b,3c)。因此,本实施例中在本研究的其余部分中使用dlbcpf1-be,并且为简单起见,将它们称为dcpf1-be。本发明发现间隔区范围为19个nt至27个nt的crrna显示出相似的编辑频率(图4)。接着,该实施例分析了dcpf1-be0在哺乳动物细胞的内源性基因组位点的性能。dcpf1-be0还可以在靶基因组位点诱导碱基编辑,从而产生6%-37%的c到t转换的编辑频率(平均为20%,计算每个靶标中单胞嘧啶的最高编辑频率,参见图5a和5b)。dcpf1和ugi之间的内部sv40核定位序列(inls)的删除显著降低了碱基编辑效率(图5b-5d,将dcpf1-be0δinls与dcpf1-be0比较)。具有n末端sv40nls额外拷贝的dcpf1-be在所有测试的基因组基因座上均提高了碱基编辑效率(图6a-6d,p=3×10-11)。为了总体上评估其功效,该实施例在具有tttvpam序列的12个靶位点和具有ttttpam序列的3个靶位点上测试了dcpf1-be(图1a)。在具有tttvpam序列的12个靶位点中,dcpf1-be在10个位点上诱导了碱基编辑(最高的单c到t转换频率范围为11%-46%,平均为22%)并且在2个位点上诱导了无效的碱基编辑(频率低于5%,平均为3%)。在具有ttttpam序列的位点上,dcpf1-be诱导的效率相对较低(两个位点的编辑频率为~10%,而另一个位点的编辑检测不到)。同时,dcpf1-be的主要编辑窗口从位置8到13,将pam旁边的碱基计数为位置1(图7a),并且dcpf1-be在鸟苷之后的胞嘧啶上几乎不引起c到t转换的碱基编辑(图1a)。值得注意的是,由于在dcpf1-be中使用了dcpf1,通常不会诱导不想要的插入/缺失,并且在检测位点实现了高比例的c到t的转换(图5e、6e、7b和7c)。同样地,dcpf1-be在另一个人类细胞系u2os中所有受检测的部位均诱导碱基编辑(单个c到t转换的最高频率范围为10%-33%,平均为20%),且没有不需要的插入/缺失和非c到t的取代(图8)。此外,该实施例检测了由dcpf1-be诱导的在八个crrna上的40个预测ot位点(每个crrna五个ot位点)可能的ot碱基编辑,并且发现在一个crrna上的三个位点有ot碱基编辑(图9)。最后,通过分析间隔序列外的44个nt区域,该实施例很少能够检测到c到t碱基的转换(图10)。接下来,该实施例比较了在dcpf1-be(位置8-13)和cas9-be(位置4-8)的编辑窗口重叠的8个靶位置上,dcpf1-be的编辑效率与不同cas9-be的编辑效率。如图1b所示,dcpf1-be通常在14个可编辑的胞嘧啶上诱导的编辑频率比dcas9-be2高,并且在14个可编辑的胞嘧啶中的5个达到或超过ncas9-be3诱导的编辑水平(图1b,c到t转换的编辑频率)。同时,不能排除的是,与ncas9-be3相比,nls数量的增加以及dcpf1和ugi之间更长的连接子均有助于dcpf1-be的性能。在其他9个常见可编辑的胞嘧啶上,dcpf1-be诱导的碱基编辑水平低于基于ncas9的be3(图1b)。值得注意的是,在所有情况下,dcpf1-be诱导的插入/缺失和非c到t的取代都少于ncas9-be3(图1b和图7d,p=5×10-10)。为了进一步缩小dcpf1-be的6个nt编辑窗口(图7a的位置8至13)并减少多个c到t的碱基转换(图11),此实施例在apobec结构域处引入了突变(w90y和r126e)进入(图12a)。在五分之四的测试基因组位点中,dcpf1-be-ye在其高度偏好的编辑位置(主要编辑位点,位置10至12,图2a)保留了约30%至90%的原始编辑效率,但显示出在间隔区中其他位置(次要编辑位点,位置1至9和13-23,图2a)上大大降低的编辑效率,这导致了主要编辑位点与次要编辑位点的比率增加(图2b)。在被归一化为dcpf1-be之后(图12b),由dcpf1-be-ye诱导的主要编辑位点与次要编辑位点的比率增加了约2至3倍(图2c,p=0.0005)。当在编辑窗口中有两个或更多个胞嘧啶时,dcpf1-be-ye还产生了高于dcpf1-be的单个c到t的转换率(图12c,12d)。带有第三个突变(r132e)的dcpf1-be-yee将所有编辑位置的碱基编辑频率降低到了接近背景水平(图2a)。与dcpf1-be相似,dcpf1-be-ye和dcpf1-be-yee几乎都不引起不想要的插入/缺失(图12e)。总体而言,dcpf1-be-ye可以将碱基编辑特定地缩小为3个nt窗口(位置10至12)。dcpf1-be诱导的非c到t的取代少于由ncas9-be3诱导的取代(图1b),但在某些编辑位点上仍值得关注(dyrk1a-c10,fancf-c10和runx1-c10,图1b,胞嘧啶取代率)。如实施例2中所示,共表达额外的ugi蛋白可以显著减少这些非预期的非c到t的取代。因此,该实施例将三个拷贝的自剪切肽2a(2a)-ugi序列添加到dcpf1-be编码区的3’末端以构建dcpf1-ebe(图13a)。在dcpf1-ebe介导的编辑中抑制了非c到t取代的形成(图2d)。从而导致c到t取代率进一步增加(图2e,p=0.0002),而编辑效率基本上保持不变(将dcpf1-ebe与dcpf1-be进行比较,图13b)。同样地,在dcpf1-ebe-ye介导的碱基编辑中,c到t的转换率也有所增加(图2f,2g,p=0.007和图13d),而对编辑效率的影响很小(图13e)。一致地是,dcpf1-ebe和dcpf1-ebe-ye在所有检测的基因组基因座上都几乎未诱导能够检测到的插入/缺失(图13c,13f)。在另一个实验中,将dcpf1与apobec3(a3)融合以产生dcpf1-a3-be编辑器(图14a)。与基于cpf1-a1的编辑器一样,此cpf1-a3融合编辑器与基于cas9的编辑器相比,大大提高了编辑频率(图14b)。cpf1-a3-be的编辑窗口是从位置6到22(图15)。本发明人最近发现,突变w104a、y130f、d131y、d131e和y132d可以缩小a3的编辑窗口,这与在a1中检测的突变一致。总之,此实施例开发了一系列基于crispr-cpf1的be,可以以极低水平的插入/缺失形成和非c到t的取代(图2h)执行靶碱基编辑,并有助于在富含a/t的区域进行碱基编辑。实施例2:通过共表达游离尿嘧啶dna糖基化酶抑制剂来增强碱基编辑与前期的be(be1和be2)相比,最新的be3通过用cas9切口酶(ncas9)代替无催化作用的cas9(dcas9)实现了更高的碱基编辑频率。由于be在不引入dna双链断裂(dsb)的情况下实现基因校正,因此在碱基编辑中排除了通过非同源末端连接(nhej)从dsb转换而来的不需要的插入/缺失。然而,在be3介导的碱基编辑中仍然观察到不可忽略的插入/缺失水平(~4%-12%)。另外还观察到不需要的非c到t取代(即c到a取代或c到g取代),并且c到a/c到g取代的频率可能与在某些检测过的案例中从c到t取代的频率一样高。不需要的插入/缺失和c到a/c到g取代的存在损害了碱基编辑结果的保真度。尽管在be3中ugi与ncas9融合,但在报道的研究中仍然可以观察到插入/缺失。预期额外的ugi活性对于进一步提高be3介导的碱基编辑的效率和保真度可能是有用的。然后,该实施例中将ugi与be3反式共表达。在293ft细胞中将ugi与sgrna/be3反式共转染后(图16a),该实施例应用深度测序以确定在三个sgrna靶位点的插入/缺失和碱基取代频率。与单独的be3相比,反式共表达be3和ugi明显降低了插入/缺失频率(图16b和16c,p<10-6)并提高了靶碱基处的c到t转换的编辑频率(图16d和16e,p<10-5)。具体地,ugi的表达水平与c到t转换的编辑与插入/缺失的比率呈正相关(图16f)。当存在高水平的游离ugi时,所需碱基编辑与不需要的插入/缺失的比率增加了约6倍(图16g,p<10-4)。同时,在大多数检测的案例中,游离ugi的表达也抑制了不必要的c到a/c到g的取代,从而导致c到t较于c到a/c到g的取代显著增加(图16h和16i,p<10-6)。应注意生物学复孔之间的变化不是微不足道的(图16b、16d和16f,标准差以误差线表示),这可以通过复孔之间的转染效率不同来解释。为了排除不同生物复孔之间转染效率的影响,本实施例将be3/ugi共表达中诱导的插入/缺失频率、c到t转换的编辑频率和插入与缺失的比率根据平行对照的be3测试中诱导的那些数值进行归一化。在be3/ugi共表达中观察到的碱基编辑效果始终优于be3。此外,统计分析表明,高水平的游离ugi所带来的改善效果非常显著(图16c,16e和16g),p值均在10-6-10-4的范围内。这些结果表明,额外的游离ugi可以减少单链nts上ap位点的形成,从而抑制不需要的插入/缺失和c到a/c到g取代的产生,并同时增加所需的c到t转换的编辑。接着,本实施例试图通过使用单个载体与2a-ugi序列的一个(ebe-s1)或三个(ebe-s3)拷贝共同表达be3,来更方便地建立增强型be(ebe)(图16j)。在与五个靶向不同基因组基因座的sgrna一起转染到293ft细胞后,这两个ebe都显示出比原始be3更低的插入/缺失频率和更高的c到t转换的编辑频率(图16k和16m)。具有三个拷贝的2a-ugi和最高水平的ugi表达的ebe-s3表现出最强大和最显著的作用(图16k-16n,p<10-8-10-4)。一致地是,当使用任何一种ebe时,c到t转换的编辑与插入/缺失的比率均得到提高(图16o和16p,ebe-s3的p<10-4)。此外,ebe还可抑制c到a/c到g的取代,ebe-s3诱导的c到t转换率比c到a/c到g的转换率显著增加(图16q和16r,p<10-9)。值得注意的是,即使存在大量的游离ugi,ncas9融合的ugi结构域对于实现高保真的碱基编辑仍然非常重要。接着,本实施例测试了在另一种细胞系hela中共同表达be3和游离ugi的作用。与be3相比,从单独的或相同的载体中共表达游离ugi均可诱导显著的更低的插入/缺失频率、更高的c到t转换的编辑频率、更高的c到t转换的编辑与插入/缺失的比率、以及更高的超过c到a/c到g转换率的c到t转换率。综上所述,这些结果表明我们增强的碱基编辑系统可以提高碱基编辑的效率和结果保真度,从而在单碱基水平上实现更准确的基因编辑。综上所述,此实施例通过将be3与游离ugi一起共表达,开发了一种增强的碱基编辑系统。增强的碱基编辑系统不仅抑制了不需要的插入/缺失的形成,而且还提高了c到t转换的编辑频率,从而提高了碱基编辑的保真度和效率。在诸如be的治疗相关的应用等情况下,编辑的“纯度”是一种被追求的。因此,该发现提供了一种进一步改善be以便得到纯度更高的编辑结果的方法。鉴于最近开发了利用具有改变的pam的ncas9的新be,因此这种增强的碱基编辑策略也可以用于提高这些新出现的be的保真度和效率。本发明的范围并不受所述旨在作为各个方面的单个说明的具体实施例的限制,并且在功能上等同的任何组合物或方法均在本发明的保护范围内。对本领域的技术人员来说显而易见的是,在不脱离本发明的精神或范围的情况下,可以对本发明的方法和组合物进行各种修改和变化。因此,落入本发明所附的权利要求及其等同物范围内的修改和变化均属于本发明保护范围。本发明中提及的所有出版物和专利申请通过引用结合于此,其程度相当于每个单独的出版物或专利申请被具体地和单独地指示通过引用结合于此。sequencelisting<110>上海科技大学<120>用于提高碱基编辑精度的融合蛋白<130>p20110519cp<160>17<170>patentinversion3.5<210>1<211>229<212>prt<213>artificialsequence<220><223>合成的<400>1metsersergluthrglyprovalalavalaspprothrleuargarg151015argilegluprohisgluphegluvalphepheaspproarggluleu202530arglysgluthrcysleuleutyrgluileasntrpglyglyarghis354045seriletrparghisthrserglnasnthrasnlyshisvalgluval505560asnpheileglulysphethrthrgluargtyrphecysproasnthr65707580argcysserilethrtrppheleusertrpserprocysglyglucys859095serargalailethrglupheleuserargtyrprohisvalthrleu100105110pheiletyrilealaargleutyrhishisalaaspproargasnarg115120125glnglyleuargaspleuileserserglyvalthrileglnilemet130135140thrgluglngluserglytyrcystrpargasnphevalasntyrser145150155160proserasnglualahistrpproargtyrprohisleutrpvalarg165170175leutyrvalleugluleutyrcysileileleuglyleuproprocys180185190leuasnileleuargarglysglnproglnleuthrphephethrile195200205alaleuglnsercyshistyrglnargleuproprohisileleutrp210215220alathrglyleulys225<210>2<211>229<212>prt<213>artificialsequence<220><223>合成的<400>2metsersergluthrglyprovalalavalaspprothrleuargarg151015argilegluprohisgluphegluvalphepheaspproarggluleu202530arglysgluthrcysleuleutyrgluileasntrpglyglyarghis354045seriletrparghisthrserglnasnthrasnlyshisvalgluval505560asnpheileglulysphethrthrgluargtyrphecysproasnthr65707580argcysserilethrtrppheleusertyrserprocysglyglucys859095serargalailethrglupheleuserargtyrprohisvalthrleu100105110pheiletyrilealaargleutyrhishisalaaspprogluasnarg115120125glnglyleuargaspleuileserserglyvalthrileglnilemet130135140thrgluglngluserglytyrcystrpargasnphevalasntyrser145150155160proserasnglualahistrpproargtyrprohisleutrpvalarg165170175leutyrvalleugluleutyrcysileileleuglyleuproprocys180185190leuasnileleuargarglysglnproglnleuthrphephethrile195200205alaleuglnsercyshistyrglnargleuproprohisileleutrp210215220alathrglyleulys225<210>3<211>229<212>prt<213>artificialsequence<220><223>合成的<400>3metsersergluthrglyprovalalavalaspprothrleuargarg151015argilegluprohisgluphegluvalphepheaspproarggluleu202530arglysgluthrcysleuleutyrgluileasntrpglyglyarghis354045seriletrparghisthrserglnasnthrasnlyshisvalgluval505560asnpheileglulysphethrthrgluargtyrphecysproasnthr65707580argcysserilethrtrppheleusertyrserprocysglyglucys859095serargalailethrglupheleuserargtyrprohisvalthrleu100105110pheiletyrilealaargleutyrhishisalaaspprogluasnarg115120125glnglyleugluaspleuileserserglyvalthrileglnilemet130135140thrgluglngluserglytyrcystrpargasnphevalasntyrser145150155160proserasnglualahistrpproargtyrprohisleutrpvalarg165170175leutyrvalleugluleutyrcysileileleuglyleuproprocys180185190leuasnileleuargarglysglnproglnleuthrphephethrile195200205alaleuglnsercyshistyrglnargleuproprohisileleutrp210215220alathrglyleulys225<210>4<211>199<212>prt<213>artificialsequence<220><223>合成的<400>4metglualaserproalaserglyproarghisleumetaspprohis151015ilephethrserasnpheasnasnglyileglyarghislysthrtyr202530leucystyrgluvalgluargleuaspasnglythrservallysmet354045aspglnhisargglypheleuhisasnglnalalysasnleuleucys505560glyphetyrglyarghisalagluleuargpheleuaspleuvalpro65707580serleuglnleuaspproalaglniletyrargvalthrtrppheile859095sertrpserprocysphesertrpglycysalaglygluvalargala100105110pheleuglngluasnthrhisvalargleuargilephealaalaarg115120125iletyrasptyraspproleutyrlysglualaleuglnmetleuarg130135140aspalaglyalaglnvalserilemetthrtyraspgluphelyshis145150155160cystrpaspthrphevalasphisglnglycyspropheglnprotrp165170175aspglyleuaspgluhisserglnalaleuserglyargleuargala180185190ileleuglnasnglnglyasn195<210>5<211>1237<212>prt<213>artificialsequence<220><223>合成的<400>5metserlysleuglulysphethrasncystyrserleuserlysthr151015leuargphelysalaileprovalglylysthrglngluasnileasp202530asnlysargleuleuvalgluaspglulysargalagluasptyrlys354045glyvallyslysleuleuaspargtyrtyrleuserpheileasnasp505560valleuhisserilelysleulysasnleuasnasntyrileserleu65707580phearglyslysthrargthrglulysgluasnlysgluleugluasn859095leugluileasnleuarglysgluilealalysalaphelysglyasn100105110gluglytyrlysserleuphelyslysaspileilegluthrileleu115120125proglupheleuaspasplysaspgluilealaleuvalasnserphe130135140asnglyphethrthralaphethrglyphepheaspasnarggluasn145150155160metphesergluglualalysserthrserilealapheargcysile165170175asngluasnleuthrargtyrileserasnmetaspilepheglulys180185190valaspalailepheasplyshisgluvalglngluilelysglulys195200205ileleuasnserasptyraspvalgluaspphephegluglygluphe210215220pheasnphevalleuthrglngluglyileaspvaltyrasnalaile225230235240ileglyglyphevalthrgluserglyglulysilelysglyleuasn245250255glutyrileasnleutyrasnglnlysthrlysglnlysleuprolys260265270phelysproleutyrlysglnvalleuserasparggluserleuser275280285phetyrglygluglytyrthrseraspglugluvalleugluvalphe290295300argasnthrleuasnlysasnsergluilepheserserilelyslys305310315320leuglulysleuphelysasnpheaspglutyrserseralaglyile325330335phevallysasnglyproalaileserthrileserlysaspilephe340345350glyglutrpasnvalileargasplystrpasnalaglutyraspasp355360365ilehisleulyslyslysalavalvalthrglulystyrgluaspasp370375380argarglysserphelyslysileglyserpheserleugluglnleu385390395400glnglutyralaaspalaaspleuservalvalglulysleulysglu405410415ileileileglnlysvalaspgluiletyrlysvaltyrglyserser420425430glulysleupheaspalaaspphevalleuglulysserleulyslys435440445asnaspalavalvalalailemetlysaspleuleuaspservallys450455460serphegluasntyrilelysalaphepheglygluglylysgluthr465470475480asnargaspgluserphetyrglyaspphevalleualatyraspile485490495leuleulysvalasphisiletyraspalaileargasntyrvalthr500505510glnlysprotyrserlysasplysphelysleutyrpheglnasnpro515520525glnphemetglyglytrpasplysasplysgluthrasptyrargala530535540thrileleuargtyrglyserlystyrtyrleualailemetasplys545550555560lystyralalyscysleuglnlysileasplysaspaspvalasngly565570575asntyrglulysileasntyrlysleuleuproglyproasnlysmet580585590leuprolysvalphepheserlyslystrpmetalatyrtyrasnpro595600605sergluaspileglnlysiletyrlysasnglythrphelyslysgly610615620aspmetpheasnleuasnaspcyshislysleuileaspphephelys625630635640aspserileserargtyrprolystrpserasnalatyrasppheasn645650655phesergluthrglulystyrlysaspilealaglyphetyrargglu660665670valglugluglnglytyrlysvalserphegluseralaserlyslys675680685gluvalasplysleuvalglugluglylysleutyrmetpheglnile690695700tyrasnlysasppheserasplysserhisglythrproasnleuhis705710715720thrmettyrphelysleuleupheaspgluasnasnhisglyglnile725730735argleuserglyglyalagluleuphemetargargalaserleulys740745750lysglugluleuvalvalhisproalaasnserproilealaasnlys755760765asnproaspasnprolyslysthrthrthrleusertyraspvaltyr770775780lysasplysargphesergluaspglntyrgluleuhisileproile785790795800alaileasnlyscysprolysasnilephelysileasnthrgluval805810815argvalleuleulyshisaspaspasnprotyrvalileglyileala820825830argglygluargasnleuleutyrilevalvalvalaspglylysgly835840845asnilevalgluglntyrserleuasngluileileasnasnpheasn850855860glyileargilelysthrasptyrhisserleuleuasplyslysglu865870875880lysgluargpheglualaargglnasntrpthrserilegluasnile885890895lysgluleulysalaglytyrileserglnvalvalhislysilecys900905910gluleuvalglulystyraspalavalilealaleualaaspleuasn915920925serglyphelysasnserargvallysvalglulysglnvaltyrgln930935940lyspheglulysmetleuileasplysleuasntyrmetvalasplys945950955960lysserasnprocysalathrglyglyalaleulysglytyrglnile965970975thrasnlysphegluserphelyssermetserthrglnasnglyphe980985990ilephetyrileproalatrpleuthrserlysileaspproserthr99510001005glyphevalasnleuleulysthrlystyrthrserilealaasp101010151020serlyslyspheileserserpheaspargilemettyrvalpro102510301035glugluaspleuphegluphealaleuasptyrlysasnpheser104010451050argthraspalaasptyrilelyslystrplysleutyrsertyr105510601065glyasnargileargilepheargasnprolyslysasnasnval107010751080pheasptrpglugluvalcysleuthrseralatyrlysgluleu108510901095pheasnlystyrglyileasntyrglnglnglyaspileargala110011051110leuleucysgluglnserasplysalaphetyrserserphemet111511201125alaleumetserleumetleuglnmetargasnserilethrgly113011351140argthraspvalalapheleuileserprovallysasnserasp114511501155glyilephetyraspserargasntyrglualaglngluasnala116011651170ileleuprolysasnalaaspalaasnglyalatyrasnileala117511801185arglysvalleutrpalaileglyglnphelyslysalagluasp119011951200glulysleuasplysvallysilealaileserasnlysglutrp120512101215leuglutyralaglnthrservallyshisglyserprolyslys122012251230lysarglysval1235<210>6<211>1316<212>prt<213>artificialsequence<220><223>合成的<400>6metthrglnphegluglyphethrasnleutyrglnvalserlysthr151015leuargphegluleuileproglnglylysthrleulyshisilegln202530gluglnglypheileglugluasplysalaargasnasphistyrlys354045gluleulysproileileaspargiletyrlysthrtyralaaspgln505560cysleuglnleuvalglnleuasptrpgluasnleuseralaalaile65707580aspsertyrarglysglulysthrglugluthrargasnalaleuile859095glugluglnalathrtyrargasnalailehisasptyrpheilegly100105110argthraspasnleuthraspalaileasnlysarghisalagluile115120125tyrlysglyleuphelysalagluleupheasnglylysvalleulys130135140glnleuglythrvalthrthrthrgluhisgluasnalaleuleuarg145150155160serpheasplysphethrthrtyrpheserglyphetyrgluasnarg165170175lysasnvalpheseralagluaspileserthralaileprohisarg180185190ilevalglnaspasnpheprolysphelysgluasncyshisilephe195200205thrargleuilethralavalproserleuarggluhisphegluasn210215220vallyslysalaileglyilephevalserthrserileglugluval225230235240pheserpheprophetyrasnglnleuleuthrglnthrglnileasp245250255leutyrasnglnleuleuglyglyileserargglualaglythrglu260265270lysilelysglyleuasngluvalleuasnleualaileglnlysasn275280285aspgluthralahisileilealaserleuprohisargpheilepro290295300leuphelysglnileleuseraspargasnthrleuserpheileleu305310315320glugluphelysseraspglugluvalileglnserphecyslystyr325330335lysthrleuleuargasngluasnvalleugluthralaglualaleu340345350pheasngluleuasnserileaspleuthrhisilepheileserhis355360365lyslysleugluthrileserseralaleucysasphistrpaspthr370375380leuargasnalaleutyrgluargargilesergluleuthrglylys385390395400ilethrlysseralalysglulysvalglnargserleulyshisglu405410415aspileasnleuglngluileileseralaalaglylysgluleuser420425430glualaphelysglnlysthrsergluileleuserhisalahisala435440445alaleuaspglnproleuprothrthrleulyslysglngluglulys450455460gluileleulysserglnleuaspserleuleuglyleutyrhisleu465470475480leuasptrpphealavalaspgluserasngluvalaspprogluphe485490495seralaargleuthrglyilelysleuglumetgluproserleuser500505510phetyrasnlysalaargasntyralathrlyslysprotyrserval515520525glulysphelysleuasnpheglnmetprothrleualaserglytrp530535540aspvalasnlysglulysasnasnglyalaileleuphevallysasn545550555560glyleutyrtyrleuglyilemetprolysglnlysglyargtyrlys565570575alaleuserphegluprothrglulysthrsergluglypheasplys580585590mettyrtyrasptyrpheproaspalaalalysmetileprolyscys595600605serthrglnleulysalavalthralahispheglnthrhisthrthr610615620proileleuleuserasnasnpheilegluproleugluilethrlys625630635640gluiletyraspleuasnasnproglulysgluprolyslysphegln645650655thralatyralalyslysthrglyaspglnlysglytyrarggluala660665670leucyslystrpileaspphethrargasppheleuserlystyrthr675680685lysthrthrserileaspleuserserleuargproserserglntyr690695700lysaspleuglyglutyrtyralagluleuasnproleuleutyrhis705710715720ileserpheglnargilealaglulysgluilemetaspalavalglu725730735thrglylysleutyrleupheglniletyrasnlysaspphealalys740745750glyhishisglylysproasnleuhisthrleutyrtrpthrglyleu755760765pheserprogluasnleualalysthrserilelysleuasnglygln770775780alagluleuphetyrargprolysserargmetlysargmetalahis785790795800argleuglyglulysmetleuasnlyslysleulysaspglnlysthr805810815proileproaspthrleutyrglngluleutyrasptyrvalasnhis820825830argleuserhisaspleuseraspglualaargalaleuleuproasn835840845valilethrlysgluvalserhisgluileilelysaspargargphe850855860thrserasplysphephephehisvalproilethrleuasntyrgln865870875880alaalaasnserproserlyspheasnglnargvalasnalatyrleu885890895lysgluhisprogluthrproileileglyilealaargglygluarg900905910asnleuiletyrilethrvalileaspserthrglylysileleuglu915920925glnargserleuasnthrileglnglnpheasptyrglnlyslysleu930935940aspasnargglulysgluargvalalaalaargglnalatrpserval945950955960valglythrilelysaspleulysglnglytyrleuserglnvalile965970975hisgluilevalaspleumetilehistyrglnalavalvalvalleu980985990alaasnleuasnpheglyphelysserlysargthrglyilealaglu99510001005lysalavaltyrglnglnpheglulysmetleuileasplysleu101010151020asncysleuvalleulysasptyrproalaglulysvalglygly102510301035valleuasnprotyrglnleuthraspglnphethrserpheala104010451050lysmetglythrglnserglypheleuphetyrvalproalapro105510601065tyrthrserlysileaspproleuthrglyphevalaspprophe107010751080valtrplysthrilelysasnhisgluserarglyshispheleu108510901095gluglypheasppheleuhistyraspvallysthrglyaspphe110011051110ileleuhisphelysmetasnargasnleuserpheglnarggly111511201125leuproglyphemetproalatrpaspilevalpheglulysasn113011351140gluthrglnpheaspalalysglythrpropheilealaglylys114511501155argilevalprovalilegluasnhisargphethrglyargtyr116011651170argaspleutyrproalaasngluleuilealaleuleugluglu117511801185lysglyilevalpheargaspglyserasnileleuprolysleu119011951200leugluasnaspaspserhisalaileaspthrmetvalalaleu120512101215ileargservalleuglnmetargasnserasnalaalathrgly122012251230glualatyrileasnserprovalargaspleuasnglyvalcys123512401245pheaspserargpheglnasnproglutrpprometaspalaasp125012551260alaasnglyalatyrhisilealaleulysglyglnleuleuleu126512701275asnhisleulysgluserlysaspleulysleuglnasnglyile128012851290serasnglnasptrpleualatyrileglngluleuargasngly129513001305serprolyslyslysarglysval13101315<210>7<211>7<212>prt<213>artificialsequence<220><223>合成的<400>7prolyslyslysarglysval15<210>8<211>83<212>prt<213>artificialsequence<220><223>合成的<400>8thrasnleuseraspileileglulysgluthrglylysglnleuval151015ileglngluserileleumetleuproglugluvalglugluvalile202530glyasnlysprogluseraspileleuvalhisthralatyraspglu354045serthraspgluasnvalmetleuleuthrseraspalaproglutyr505560lysprotrpalaleuvalileglnaspserasnglygluasnlysile65707580lysmetleu<210>9<211>22<212>prt<213>artificialsequence<220><223>合成的<400>9glyserglyalathrasnpheserleuleulysglnalaglyaspval151015glugluasnproglypro20<210>10<211>23<212>prt<213>artificialsequence<220><223>合成的<400>10glyserglyglncysthrasntyralaleuleulysleualaglyasp151015valgluserasnproglypro20<210>11<211>23<212>prt<213>artificialsequence<220><223>合成的<400>11glyserglyglncysthrasntyralaleuleulysleualaglyasp151015valgluserasnproglypro20<210>12<211>199<212>prt<213>artificialsequence<220><223>合成的<400>12metglualaserproalaserglyproarghisleumetaspprohis151015ilephethrserasnpheasnasnglyileglyarghislysthrtyr202530leucystyrgluvalgluargleuaspasnglythrservallysmet354045aspglnhisargglypheleuhisasnglnalalysasnleuleucys505560glyphetyrglyarghisalagluleuargpheleuaspleuvalpro65707580serleuglnleuaspproalaglniletyrargvalthrtrppheile859095sertrpserprocysphesertrpglycysalaglygluvalargala100105110pheleuglngluasnthrhisvalargleuargilephealaalaarg115120125ilepheasptyraspproleutyrlysglualaleuglnmetleuarg130135140aspalaglyalaglnvalserilemetthrtyraspgluphelyshis145150155160cystrpaspthrphevalasphisglnglycyspropheglnprotrp165170175aspglyleuaspgluhisserglnalaleuserglyargleuargala180185190ileleuglnasnglnglyasn195<210>13<211>199<212>prt<213>artificialsequence<220><223>合成的<400>13metglualaserproalaserglyproarghisleumetaspprohis151015ilephethrserasnpheasnasnglyileglyarghislysthrtyr202530leucystyrgluvalgluargleuaspasnglythrservallysmet354045aspglnhisargglypheleuhisasnglnalalysasnleuleucys505560glyphetyrglyarghisalagluleuargpheleuaspleuvalpro65707580serleuglnleuaspproalaglniletyrargvalthrtrppheile859095sertrpserprocysphesertrpglycysalaglygluvalargala100105110pheleuglngluasnthrhisvalargleuargilephealaalaarg115120125iletyraspaspaspproleutyrlysglualaleuglnmetleuarg130135140aspalaglyalaglnvalserilemetthrtyraspgluphelyshis145150155160cystrpaspthrphevalasphisglnglycyspropheglnprotrp165170175aspglyleuaspgluhisserglnalaleuserglyargleuargala180185190ileleuglnasnglnglyasn195<210>14<211>199<212>prt<213>artificialsequence<220><223>合成的<400>14metglualaserproalaserglyproarghisleumetaspprohis151015ilephethrserasnpheasnasnglyileglyarghislysthrtyr202530leucystyrgluvalgluargleuaspasnglythrservallysmet354045aspglnhisargglypheleuhisasnglnalalysasnleuleucys505560glyphetyrglyarghisalagluleuargpheleuaspleuvalpro65707580serleuglnleuaspproalaglniletyrargvalthrtrppheile859095sertrpserprocyspheseralaglycysalaglygluvalargala100105110pheleuglngluasnthrhisvalargleuargilephealaalaarg115120125iletyrasptyraspproleutyrlysglualaleuglnmetleuarg130135140aspalaglyalaglnvalserilemetthrtyraspgluphelyshis145150155160cystrpaspthrphevalasphisglnglycyspropheglnprotrp165170175aspglyleuaspgluhisserglnalaleuserglyargleuargala180185190ileleuglnasnglnglyasn195<210>15<211>199<212>prt<213>artificialsequence<220><223>合成的<400>15metglualaserproalaserglyproarghisleumetaspprohis151015ilephethrserasnpheasnasnglyileglyarghislysthrtyr202530leucystyrgluvalgluargleuaspasnglythrservallysmet354045aspglnhisargglypheleuhisasnglnalalysasnleuleucys505560glyphetyrglyarghisalagluleuargpheleuaspleuvalpro65707580serleuglnleuaspproalaglniletyrargvalthrtrppheile859095sertrpserprocysphesertrpglycysalaglygluvalargala100105110pheleuglngluasnthrhisvalargleuargilephealaalaarg115120125iletyrtyrtyraspproleutyrlysglualaleuglnmetleuarg130135140aspalaglyalaglnvalserilemetthrtyraspgluphelyshis145150155160cystrpaspthrphevalasphisglnglycyspropheglnprotrp165170175aspglyleuaspgluhisserglnalaleuserglyargleuargala180185190ileleuglnasnglnglyasn195<210>16<211>199<212>prt<213>artificialsequence<220><223>合成的<400>16metglualaserproalaserglyproarghisleumetaspprohis151015ilephethrserasnpheasnasnglyileglyarghislysthrtyr202530leucystyrgluvalgluargleuaspasnglythrservallysmet354045aspglnhisargglypheleuhisasnglnalalysasnleuleucys505560glyphetyrglyarghisalagluleuargpheleuaspleuvalpro65707580serleuglnleuaspproalaglniletyrargvalthrtrppheile859095sertrpserprocysphesertrpglycysalaglygluvalargala100105110pheleuglngluasnthrhisvalargleuargilephealaalaarg115120125ilephegluaspaspproleutyrlysglualaleuglnmetleuarg130135140aspalaglyalaglnvalserilemetthrtyraspgluphelyshis145150155160cystrpaspthrphevalasphisglnglycyspropheglnprotrp165170175aspglyleuaspgluhisserglnalaleuserglyargleuargala180185190ileleuglnasnglnglyasn195<210>17<211>199<212>prt<213>artificialsequence<220><223>合成的<400>17metglualaserproalaserglyproarghisleumetaspprohis151015ilephethrserasnpheasnasnglyileglyarghislysthrtyr202530leucystyrgluvalgluargleuaspasnglythrservallysmet354045aspglnhisargglypheleuhisasnglnalalysasnleuleucys505560glyphetyrglyarghisalagluleuargpheleuaspleuvalpro65707580serleuglnleuaspproalaglniletyrargvalthrtrppheile859095sertrpserprocysphesertrpglycysalaglygluvalargala100105110pheleuglngluasnthrhisvalargleuargilephealaalaarg115120125ilephetyraspaspproleutyrlysglualaleuglnmetleuarg130135140aspalaglyalaglnvalserilemetthrtyraspgluphelyshis145150155160cystrpaspthrphevalasphisglnglycyspropheglnprotrp165170175aspglyleuaspgluhisserglnalaleuserglyargleuargala180185190ileleuglnasnglnglyasn195当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1