用于基因组编辑和调节转录的CRISPR/CAS系统和方法与流程
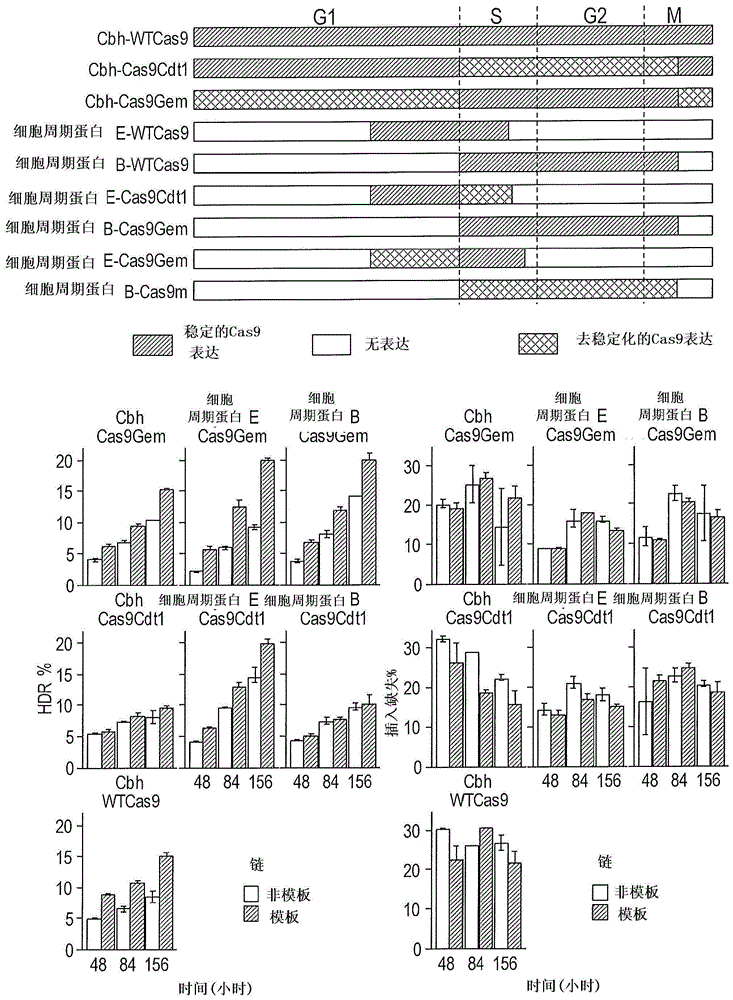
引言本申请要求2017年9月26日提交的美国临时申请号62/563,128;2017年9月26日提交的美国临时申请号62/563,131;和2017年9月26日提交的美国临时申请号62/563,133的优先权权益,这些美国临时申请的内容以引用的方式整体并入本文。本发明是在政府支持下根据由国家卫生研究院(nationalinstitutesofhealth)授予的基金号r01hd081534作出的。政府拥有本发明的某些权利。
背景技术:
:在使用哺乳动物细胞的早期研究中,发现使用归巢核酸内切酶i-scei在基因组中的独特位置处引入dna双链断裂(dsb)会通过同源重组而刺激基因靶向。随后,已经使用人工序列特异性核酸酶(如锌指核酸酶和tale核酸酶)和最近的rna引导的成簇的规律间隔的短回文重复序列(crispr)/crispr相关(cas)(crispr/cas)核酸酶来靶向预定的基因组位点。在crispr/cas9的情况下,具有与靶dna互补的间隔区序列的单向导rna(sgrna或grna)引导cas9核酸内切酶对dna的切割。在dsb修复期间发生基因组序列的修饰,并且起作用的分子途径决定了序列变化的类型。规范的非同源末端连接(nhej)和诸如微同源介导的末端连接(mmej)的替代的末端连接途径在dna末端已经被加工之后通过连接所述dna末端而进行,并且引起靶向但不精确的插入缺失(一般是小的插入或缺失)。两个或更多个核苷酸的微同源性可以在dna切割之后通过切除而暴露并且可以在通过mmej进行修复期间使用。与末端连接途径相反,使用外源性dna修复模板的同源性依赖性修复(hdr)支持精确的基因组编辑。通常,可以使用具有与侧接dsb的序列同源的臂的转基因并且所述转基因因此将被精确地整合。限制基于crispr/cas9的基因组编辑的效率的障碍有非编码区或弱转录基因的存在,它们对于crispr/cas9诱变来说似乎是难处理的;以及相对于nhej修复,hdr的频率较低,这使得难以对基因组序列产生精确的变化。为了增强通过hdr进行的基因组编辑,已经开发了不同的策略。例如,当将细胞在s/g2期同步化时,通过同源重组进行dna修复的细胞周期被限制,并且hdr可以增加到高达五倍。例如在连接酶4失活后的nhej抑制也可以增加hdr。另一种方法是将规范的cdh1或cdc20降解决定子(如联会蛋白降解决定子)与cas9融合以诱导它在g1中降解并且将靶dna切割限制到s/g2期(gutschner等,(2016)cellrep.14:1555-1566;maji等,(2017)nat.chem.biol.13:9-11;howden等,(2016)stemcellrep.7:508-517)。还参见wo2017/024047a1、us2016/0376610和cn105647885b。类似地,已经将pest降解决定子与cas9融合以缩短cas9的半衰期(cn201410656081)。当前的crispr方法的另一个限制是当cas9和它的sgrna共表达时的组成型核酸内切酶活性。这在靶向在发育上对于活力来说重要的或必要的基因时可特别成问题。此外,已经证实,cas9的组成型表达可增加脱靶突变的数量并且可触发dna损伤响应。解决该问题的一种方法是将fkbp12衍生的去稳定化结构域与cas9融合,这会有条件地调节蛋白质稳定性(senturk等,(2017)nat.commun.8:14370;us2016/0298096)。当表达多个sgrna时,可以引导cas9以同时操纵多个基因组基因座,这可以通过在单独的构建体中共转染多个sgrna而实现。尽管这种方法是高效的,但是对于其中载体容量和/或载体数量对于同时产生多个grna是有限的某些应用来说,它将是一个挑战。已经开发了几种策略来从单个转录物表达多个grna。一种策略使用csy4核糖核酸内切酶,其可以加工含有与csy4可切割的rna融合的grna的转录物(nissim等,(2014)mol.cell.54:698-710;tsai等,(2014)nat.biotechnol.32:569-576)。此外,已经使用自切割核酶在u6启动子的控制下从单个表达盒表达多个sgrna(xu等,(2017)nucl.acidsres.45(5):e28)。技术实现要素:本发明提供了一种crispr/cas系统,其包括cas核酸内切酶和同源单向导rna(sgrna),其中:(a)所述cas核酸内切酶与一个或多个降解决定子序列融合,其中所述降解决定子具有(i)非规范的cdc20或chd1识别基序,或(ii)由连接酶靶向的序列,所述连接酶选自emi1、trp1、cbl-ptk、cbl-met、cop1、crl4-cdt2、kelchkeap1、kelchklhl3、mdm2-swib、odph-vhl、scf-skp2、scf-skp2-cks1、scf-cullin、scf-fbw7、scf-fbx05、scf-trcp1、scf-cul4、e6-ap、siah、hect结构域家族、环指家族(ringfingerfamily)、u框家族(uboxfamily)和其组合;(b)所述同源sgrna是可激活的sgrna,其在所述可激活的sgrna的非必需区域中带有失活序列,其中所述失活序列包含一个或多个核酸内切酶识别位点;或(c)(a)和(b)的组合。在一些实施方式中,所述系统包括可激活的sgrna的阵列,其中所述同源sgrna靶向所述可激活的sgrna的阵列中的至少一个可激活的sgrna。在所述cas融合蛋白的某些方面,所述cas核酸内切酶是cas9核酸内切酶。在所述cas融合蛋白的一些实施方式中,所述一个或多个降解决定子序列与所述cas核酸内切酶融合,在它们之间设置有一个或多个接头。在所述cas融合蛋白的其它实施方式中,所述一个或多个降解决定子序列在所述cas核酸内切酶的n末端、c末端或n末端和c末端处与所述cas核酸内切酶融合。在所述可激活的sgrna的某些方面,所述可激活的sgrna靶向核酸分子的转录链。在所述可激活的sgrna的其它方面,所述失活序列是顺式作用核酶。在一些实施方式中,所述顺式作用核酶由seqidno:2、seqidno:3或seqidno:4的核酸分子编码。在其它实施方式中,所述可激活的sgrna由seqidno:14-22的核酸分子编码。还包括一种核酸,所述核酸具有编码序列、5'非翻译区和3'非翻译区,其中所述核酸在5'非翻译区或3'非翻译区中插入有可激活的sgrna。还提供了编码所述cas融合蛋白和/或可激活的sgrna的多核苷酸和载体,以及一种使用所述crispr/cas系统以通过将所述cas融合蛋白和/或可激活的sgrna引入到细胞中来编辑或调节细胞的基因组的转录的方法。当用于本发明的方法中时,可以通过一种或多种可调节的启动子来控制cas核酸内切酶、可激活的sgrna或cas核酸内切酶和可激活的sgrna的表达。此外,在一些实施方式中,将靶向两个或更多个序列的至少两个同源sgrna引入到细胞中,其中所述至少两个同源sgrna任选地是可以被顺序地激活以在基因组中引入两个或更多个编辑或调节所关注的基因的转录的可激活的sgrna。此外,在其它实施方式中,所述至少一个同源sgrna插入在编码基因的5'非翻译区或3'非翻译区的核酸中。附图说明图1描绘了经验证的cas9变体构建体和它们的表达/降解特征(顶部)。在靶向ctnnb1以插入egfp之后,在7天的过程中,对hdr频率(左下)和插入缺失频率(右下)进行测量。用对应的hdr试剂瞬时转染小鼠胚胎干细胞(mesc)并且将cas9靶向模板或非模板链以评估链偏好性对精确基因组编辑的影响。通过流式细胞术分析hdr频率并且通过对非hdr等位基因进行深度测序来测量插入缺失频率。当与组成型表达的wt-cas9相比时,使用细胞周期蛋白e启动子表达cas9变体基本上增加了hdr频率,同时减少了插入缺失。使用cdt1变体将cas9去稳定化增强了非模板sgrna的插入缺失活性,同时降低了hdr频率,这表明了能够克服非模板sgrna通常遇到的链偏好性阻碍的动力学的能力。一般,模板sgrna介导与非模板相似的插入缺失诱变,但是对于hdr是优越的。图2示出了在hek293细胞中使用两个混杂的sgrna由cas9变体引起的中靶和组合脱靶突变频率。图3是描绘了未修饰的化脓性链球菌(streptococcuspyogenes)sgrna(seqidno:1)的结构的图示。图4是描绘了可激活的sgrna的示意图。将顺式作用核酶序列插入到发夹1中引起在转录时对sgrna的切割。然后通过cas9靶向侧接或编码核酶的dna序列以引入突变而破坏它的顺式切割活性并且恢复sgrna功能。图5示出了使用包括核酶作为失活序列的可激活的sgrna变体的egfp破坏测定的结果。变体1:seqidno:5;变体2:seqidno:7;和变体3:seqidno:6。所有这三种含有核酶的变体都不能在哺乳动物细胞中的egfp转基因内介导显著的突变,从而在可激活的sgrna条件下产生接近100%的egfp阳性细胞。对于所有条件,n=3个生物学平行测定,通过流式细胞术进行测量。图6是描绘了通过核酶的诱变将可激活的sgrna转化成功能性sgrna的两种策略的示意图。产生带有靶向egfp转基因的变体1的基因组整合的细胞系,然后仅转染cas9或转染靶向切除整个核酶(策略1)或使核酶活性位点突变(策略2)的cas9和sgrna。图表示出了由直接靶向egfp的野生型sgrna(未修饰)或由靶向可激活的sgrna的dna的sgrna通过策略1或策略2介导的egfp破坏频率。图7是描绘了在可激活的sgrna阵列内sgrna的顺序激活的示意图。每一个sgrna靶向直接在它下游的sgrna,从而使得阵列中的每一个sgrna依赖于它上游的可激活的sgrna而通过基因组编辑转化成功能状态。该事件顺序最终以顺序方式控制cas9活性。可以通过深度测序直接分析每一个可激活的sgrna的dna,或每一个的rna可以由寡聚dt结合而转化成cdna以通过一般rna测序或单细胞rna测序来分析。阵列内sgrna的激活过程是细胞条形编码过程,并且对阵列中诱变事件的分析允许进行谱系树构建。这些sgrna还可以具有靶向图8中所描绘的“条形码”基因座、靶向如例如图9中的另一个可激活的sgrna或内源性基因的次要功能。图8提供了条形码基因座示意图。可以将所述条形码基因座通过pcr扩增并且通过深度测序进行分析,或将它的rna转化成cdna以通过单细胞rna测序进行分析。图9描绘了可激活的sgrna的阵列,其中每一个sgrna靶向下一个sgrna以进行激活。1号sgrna具有一个功能:即激活阵列中的下一个sgrna。图10描绘了图9中的可激活的sgrna的阵列,其中所述sgrna被编程以执行多个功能。其中1号sgrna1激活2号sgrna,2号sgrna具有三个功能并且因此具有三个靶标。功能1是靶向阵列中下一个可激活的sgrna(3号sgrna)以继续顺序细胞条形编码,并且功能2和功能3是靶向位于整个基因组中的其它可激活的sgrna的dna。在对2号sgrna的第二靶标和第三靶标进行激活基因组编辑之后,产生功能性的可激活的sgrna,所述功能性的可激活的sgrna与dcas9转录激活因子或阻遏因子结合,它们由于pam序列特异性而具有生物化学特异性。pam识别中的这种生物化学差异仅使dcas9分子靶向所关注的位点,而阻止核酸酶活性cas9靶向相同的位点。这些sgrna结合dcas9并且靶向内源性基因的启动子,其中dcas9募集rna聚合酶以引发基因的转录。值得注意的是,2号sgrna同时激活3号sgrna,所述3号sgrna可以通过激活4号sgrna的唯一功能而继续细胞条形编码。图11描绘了使用细胞周期控制的cas9核酸酶以调节可激活的sgrna阵列的激活。如顶部部分中所示,g1-cas9(pam:ngg)存在并且与功能性sgrna结合。在结合后,cas9:sgrna靶向阵列中的下一个sgrna。这去除了它的失活序列并且引起功能性的可激活的sgrna的表达以及sgrnadna阵列中的独特突变。由于pam特异性要求,因此新功能性可激活的sgrna不能被g1-cas9使用,而仅可以被g2-cas9使用。随着细胞进入g2,g2-cas9(pam:nag)与功能性sgrna结合。g2-cas9:sgrna靶向阵列中的下一个sgrna,去除它的失活序列,并且引入独特的突变。新功能性sgrna由于pam特异性差异而对g1-cas9具有生物化学特异性并且靶向阵列中新生的sgrna。这些pam特异性差异使得阵列由于cas9变体的细胞周期调节而依赖于细胞周期进程并且使得每个细胞周期激活两个sgrna。此外,这些激活过程使得每个细胞周期发生两个条形编码事件。图12是描绘了利用链选择来影响每个细胞周期被激活的可激活的sgrna的数量的示意图。与非模板链结合的sgrna将通过dna复制活动被逐出,从而使得过程动力学取决于细胞周期并且导致每个细胞周期的一个激活基因组编辑。因此,如果在阵列中存在10个可激活的sgrna,这些sgrna全部都靶向非模板链,那么为了耗尽所述阵列需要完成10个细胞周期。与模板链结合的sgrna将具有不同的动力学,这取决于基因的转录水平,通常允许阵列的更快激活和条形编码。图13提供了示出了将可激活的sgrna的阵列进行编程以通过将图11或图12中所示的过程与图10中所示的过程组合而在特定的细胞分裂次数之后进行受控的转录调节的示意图。根据图10中所示的策略,采用可激活的sgrna对转录的控制。为了引入细胞周期调节,所述过程还包括如图11中所描绘的细胞周期控制的可激活的sgrna阵列。图14示出了当cas9靶向模板(转录)链时在存在或不存在t7rnap的情况下cas9的动力学。从两次重复实验计算平均动力学值。图15示出了t7rnap介导的cas9从dsb中的逐出具有模板链和距离依赖性。示出了在模板链取向或非模板取向上带有t7rnap启动子的靶dna的cas9消化反应的结果。值表示由于添加t7rnap而引起的切割dna倍数变化的平均值±s.d.,n=3。图16示出了体外模板链偏好性在多个sgrna(即靶向mcherry和gfp的sgrna)(n=3)中是一致的。所述示意图描绘了转化成转录模板的mcherry靶dna,所述转录模板介导sgrna的模板或非模板碰撞。图17示出了通过sgrna与模板链退火而进行转录,从而增强了基因组编辑。所述图表示出了由靶向不同基因的17个模板sgrna和23个非模板sgrna引起的插入缺失频率,其中每一个点代表独立转染的突变频率,每一个sgrna的n=2。图18示出了通过靶向模板链的sgrna的主动转录增强了诱变频率。通过t7e1测定测量了靶向多西环素诱导型mcherry的17个sgrna的诱变频率。值表示平均值±s.d.,n=3。**p<0.01,***p<0.001。图19示出了当靶向模板链时,间隔区rc1-4介导多周转cas9活性,如通过分别针对在任一末端上带有t7rnap启动子的φnm1靶dna的体外cas9消化反应所测量。值表示由于添加t7rnap而引起的切割dna倍数变化的平均值±s.d.,n=3。图20示出了在基因组编辑期间cas9核酸内切酶的链偏好性引起不同的基因组编辑动力学和差异突变结果。所述图示描绘了在基因组编辑期间模板链与非模板链的偏好性。特别是,上图示出了当cas9靶向模板链时,易位rna聚合酶通过与聚合酶的碰撞而被逐出,从而引起非细胞周期依赖性cas9介导的dsb修复。相比之下,与非模板结合的sgrna不会被rna聚合酶逐出(中图)。然而,与非模板结合的sgrna会在dna复制期间通过在细胞周期的hdr活跃期的期间内发生的dna复制复合体的各种活动而从双链断裂(dsb)中被逐出,这潜在地使突变结果具有偏好性。图21描绘了通过生物处理器技术使细胞行为自动化的方法,所述技术使用可激活的sgrna的阵列以在一段延长的时间内跟踪细胞历史,即世代条形编码系统。将与单克隆抗体(mab)的产生相关的细胞活动与世代条形编码系统相关联。一旦已经跟踪了各种细胞历史并且检测到影响mab产生的特定事件(例如1号事件、4号事件、9号事件等),就将数据用于修改生物处理器以使得对细胞进行预编程以规避所检测到的问题。这通过将指令程序嵌入到细胞的基因组中而产生高性能的产生mab的细胞。此外,核心生物处理器可以通过可激活的sgrna进行补充,所述可激活的sgrna可以通过细胞表面受体-配体相互作用而被激活以执行一系列程序,这些程序各自在细胞培养物的生命周期期间的特定阶段执行,从而优化mab的产生。图22示出了将cas9-vpr与14ntsgrna和20ntsgrna组合在诱变和转录激活方面的结果。cas9-vpr是与转录激活因子vpr融合的核酸酶活性cas9。通过将sgrna截短到14nt,消除了cas9核酸酶活性,但是维持了dna结合,从而允许募集rna聚合酶ii而不引入dsb。示出了描绘了cas9-vpr靶向沉默内源性基因ttn以进行转录激活和诱变的示意图。在ttn处通过使用cas9-vpr构建体合成激活人类ttn基因的转录来测试链偏好性。使用14ntsgrna,将核酸酶活性cas9-vpr靶向激活转录,但是不引入dsb。同时,提供靶向模板或非模板链的20ntsgrna以驱动由14nt-cas9-vpr介导的通过cas9切割位点的转录。在转染后48小时收获基因组dna,并且显示突变频率,如通过t7e1测定所确定。通过rna的定量实时pcr分析来评估由靶向ttn的14ntsgrna产生的转录水平并且观测到转录物水平的约5倍变化。图23示出了嵌入基因的3'-utr中的一系列sgrna可以用于调节cas9活性。sgrna嵌入于所关注的基因的开放阅读框的下游以允许在表达所述基因时转录所述基因和sgrna。sgrna被放置在多聚腺苷酸尾的下游并且由两个顺式作用核酶侧接(被称作rgr,即核酶-向导-核酶)以使得sgrna被切除并且产生用于cas9结合的功能性分子。使得编码mrna的转录物具有功能性,并且由于丧失rgr但维持适当的多聚腺苷酸尾而可以被翻译。使用这种方法,将1个、2个、3个或4个rgr嵌入mcherry的3'-utr中。rgr靶向hek293基因组内的egfp序列。使用流式细胞术测量egfp荧光的损失作为cas9活性的读数,并且测量mcherry的存在,所述mcherry是rgr上游的编码序列。egfp阴性细胞中高水平的mcherry确认了rgr被正确切除的mrna的翻译。具体实施方式cas9核酸内切酶由于它的通用性和易编程性而已经被用于在许多物种中进行基因组编辑。然而,cas9在本质上是单周转核酸酶,这是因为它保持与它产生的双链断裂结合,并且基因组的许多区域对于基因组编辑来说是难处理的。现在已经证实,cas9的难处理性是由于cas9阻断了在它的核酸酶活性之后的步骤(dna修复),因此抑制了基因组编辑的完成。在这方面,如果使sgrna与rna聚合酶的模板链退火,那么易位的rna聚合酶将与cas9-dsb复合物发生碰撞并且将cas9从dna末端逐出,最终促进了dna修复并且为基因组编辑产生了链偏好性。为了减轻链偏好性和对rna聚合酶的需要,通过去稳定化对cas9进行修饰还通过增加从dna中去除cas9的速率来减轻这种“阻断”现象。此外,通过控制cas9的表达和降解以对应于细胞周期,可以影响基因组编辑的类型(易错的“nhej”或精确的“hdr”)。除了修饰cas9之外,现在还已经证实,通过专门设计与基因的转录链退火的sgrna,可以对突变频率产生积极的影响。与模板链退火的sgrna的设计会显著增加突变频率/动力学,而靶向非模板链由于基因组编辑动力学较慢能导致基本上较低的突变频率/动力学。此外,通过控制特定sgrna的功能,基因组编辑活动不依赖于cas蛋白的转录或翻译控制。实际上,现在已经证实,实现一系列可激活的sgrna,可以从靶位点到靶位点更多地以时间方式调节基因组编辑,可以执行顺序基因组编辑以发挥类似于多米诺效应的功能,并且可以对细胞进行条形编码。因此,本发明提供了一种crispr/cas系统和其用于编辑细胞,特别是真核细胞的基因组的用途。本发明的crispr/cas系统包括cas核酸内切酶和可激活的同源sgrna,其中所述cas核酸内切酶与一个或多个降解决定子序列融合和/或所述可激活的同源sgrna在所述可激活的同源sgrna的非必需区域中带有失活序列。鉴于它的改善的基因编辑能力,本发明的crispr/cas系统可用于基因治疗、药物筛选、遗传记录、遗传回路以及疾病诊断和预后。术语“crispr/cas”、“crispr/cas系统”和“核酸靶向系统”可以可互换地使用。crispr-cas系统是本领域已知的。参见例如美国专利号8,697,359;8,771,945;8,795,965;8,865,406;8,871,445;8,889,356;8,895,308;8,906,616;8,932,814;8,945,839;8,999,641;美国申请号14/704,551;15/192,095;以及pct公布号wo2016106244和wo2014093622。还参见komor等,(2017)cell168(1-2):20-36。本发明的crispr/cas系统包括rna引导的核酸酶(在本文中也被称为cas核酸内切酶或cas蛋白)和同源向导rna。如本领域中常规的那样,向导rna在本文中也被称为单向导rna、sgrna或grna,将同源cas蛋白引导到基因组中的特定位点以进行靶向切割。如本文所用的“同源”指的是能够形成核蛋白复合物的cas蛋白和sgrna,所述核蛋白复合物直接结合与sgrna中存在的核酸序列互补的靶核酸分子。如本领域中常规的那样,“互补性”指的是核酸分子与另一核酸分子形成氢键的能力(例如通过传统的沃森-克里克碱基配对(watson-crickbase-pairing))。互补百分比表示在核酸分子中可以与第二核酸序列形成氢键的残基的百分比。当两个多核苷酸序列具有100%互补性时,这两个序列是完全互补的,即第一多核苷酸的所有连续残基与第二多核苷酸中相同数量的连续残基形成氢键。cas融合蛋白。如所示,本发明提供了一种crispr/cas系统,所述crispr/cas系统包括与一个或多个降解决定子序列融合的cas核酸内切酶,其在本文中一般被称为“cas融合蛋白”或“融合蛋白”。cas核酸内切酶是本领域已知的并且包括但不限于cas蛋白,如cas1、cas1b、cas2、cas3、cas4、cas5、cas6、cas7、cas8、cas9(也被称为csn1和csx12)、cas10、cas12a、csy1、csy2、csy3、cse1、cse2、csc1、csc2、csa5、csn2、csm2、csm3、csm4、csm5、csm6、cmr1、cmr3、cmr4、cmr5、cmr6、csb1、csb2、csb3、csx17、csx14、csx10、csx16、csax、csx3、csx1、csx15、csf1、csf2、csf3、csf4和其同源物或其修饰型式。在一些实施方式中,所述cas是dcas9蛋白(无核酸内切酶活性的cas9蛋白,例如具有点突变d10a和h840a)或其融合蛋白。尽管dcas9缺乏核酸内切酶活性,但是它仍然能够与它的向导rna和靶核酸分子结合。这种结合dna的能力可以用于通过将dcas9与转录阻遏肽,如krab融合来阻断靶向基因的转录或通过将dcas9与激活结构域,如vp64-p65-rta(vpr)融合来激活转录。这些cas蛋白的核酸序列和氨基酸序列可容易地从包括基因库(genbank)和swissprot的公共来源获得。作为说明,化脓性链球菌cas9蛋白质序列可在swissprot登录号q99zw2下获得。同样,猪放线杆菌(actinobacillussuis)cas9可在基因库登录号wp_014991277下获得。优选的是,所述crispr酶是cas9蛋白。代表性cas9蛋白包括但不限于来自包括化脓性链球菌、肺炎链球菌(s.pneumoniae)等的链球菌属(streptococci)的cas9以及来自金黄色葡萄球菌(staphylococcusaureus)、脑膜炎奈瑟氏球菌(neisseriameningitides)和齿垢密螺旋体(treponemadenticola)的cas9。另外的rna引导的核酸酶包括cas12a核酸酶(“cas12a”,以前的“cpf1”),包括来自普罗威登斯菌属(providicella)、弗朗西斯氏菌属(francisella)、氨基酸球菌属(acidaminococcus)和毛螺菌科(lachnospiracae)的cas12a。为了调节cas蛋白的稳定性并且因此调节从dna中去除cas9的速率,本发明包括由与一个或多个降解决定子序列融合的cas核酸内切酶构成的融合蛋白。如本文所用的“降解决定子”指的是蛋白质中赋予降解的代谢不稳定性的特定氨基酸序列。降解决定子序列可以存在于n末端或c末端区域处,并且这些分别被称作n-降解决定子或c-降解决定子。降解决定子可以是向与它融合的蛋白质赋予降解作用的任何氨基酸序列或序列的组合。通过细胞周期的有序进展取决于通过泛素介导的蛋白水解来调节几种蛋白质的丰度。降解是精确定时的和特异性的。这样的降解系统的一个实例是后期促进复合物(apc),其是一种泛素蛋白连接酶。apc在有丝分裂期间和有丝分裂/g1后期两者分别由wd重复蛋白cdc20和cdh1激活。这些激活因子靶向不同组的底物。apc/c-cdc20和apc/c-cdh1共激活因子复合物通过识别以下规范基序来识别它们大部分的底物:d(破坏)框降解决定子(arg-xaa-xaa-leu-asp或arg-xaa-xaa-leu)和ken框降解决定子(lys-glu-asn的三氨基酸基序)。在这方面,“规范的cdc20或chd1识别基序”指的是经典表征的cdc20和cdh1识别基序,即d框和ken框基序。在一些实施方式中,所述降解决定子含有规范的cdc20或chd1识别基序。包括d框降解决定子的天然存在的蛋白质的实例是细胞周期依赖性细胞周期蛋白(cdk),如细胞周期蛋白a和细胞周期蛋白b,以及其它细胞周期调节蛋白,如hsl1、cdc6、fin1、p21和联会蛋白。代表性的d框降解决定子氨基酸序列包括rpalsd、rpals和rlald。包括ken框降解决定子的天然存在的蛋白质的实例是cdc20、sgo1、nek2和b99。含有d框或ken框的其它蛋白质是本领域公知的(glotzer等,(1991)991,nature349:132-138;pfleger和kirschner(2000)genesdev.14(6):655-665)。在其它实施方式中,所述降解决定子不是规范的cdc20或chd1识别基序。因此,在一些方面,所述降解决定子是非规范的cdc20或chd1识别基序。“非规范的cdc20或chd1识别基序”指的是作为除d框或ken框基序以外的基序的cdc20或chd1基序。非规范的cdc20和chd1识别基序包括lxxk基序、a基序、cry框和rl尾。“lxxk”降解决定子是一种非规范的apc/cdh1靶标,其使得它的蛋白质可靶向蛋白酶体。lxxk降解决定子包括氨基酸序列lxxk,其中x可以是任何氨基酸残基。代表性的lxxk降解决定子序列是lask。a基序是增强apc-cdh1结合以有助于靶向蛋白酶体的序列。a基序降解决定子包括氨基酸序列eet。代表性的a基序降解决定子序列是eetae。cry框基序是由apc/c识别以靶向蛋白酶体的降解决定子。该降解决定子包括氨基酸序列cryxps,其中x可以是任何氨基酸残基。代表性的cry框降解决定子序列是cryips。emi1/emi2的rl尾基序用作apc/c的停泊位点,从而促进apc/c-chd1的相互作用和抑制。该降解决定子包括氨基酸序列(t/s)kkskknl(r/q)rl(seqidno:56),其中x可以是任何氨基酸残基。代表性的rl尾降解决定子序列是tkkskknlrrl(seqidno:57)。在本发明的其它方面,所述降解决定子既不是规范的,也不是非规范的cdc20或chd1识别基序。根据本发明的这个方面,所述降解决定子可以介导泛素依赖性降解或非泛素依赖性降解(例如通过本文公开的pest降解决定子)。在一些实施方式中,所述降解决定子具有由连接酶靶向的序列,所述连接酶选自emi1、trp1、cbl-ptk、cbl-met、cop1、crl4-cdt2、kelchkeap1、kelchklhl3、mdm2-swib、odph-vhl、scf-skp2、scf-skp2-cks1、scf-cullin、scf-fbw7、scf-fbx05、scf-trcp1、scf-cul4、e6-ap、siah、hect结构域家族、环指家族、u框家族和其组合。不用作cdc20或chd1的识别基序的降解决定子的实例包括例如phd结构域、pip基序、cy基序、abba基序等(表1)。表1***共有序列。x表示任何氨基酸残基。还参见guharoy等,(2016)nat.commun.7:10239。在一些实施方式中,所述降解决定子是从cdt1的氨基酸序列中获得的,并且优选地具有如seqidno:76所示的氨基酸序列。在另一个实施方式中,所述降解决定子是从联会蛋白的氨基酸序列中获得的,并且优选地具有如seqidno:77所示的氨基酸序列。在其它实施方式中,所述降解决定子是从鸟氨酸脱羧酶的氨基酸序列中获得的,并且优选地具有如seqidno:78或seqidno:79所示的氨基酸序列。尽管任何生物体的蛋白质的降解决定子可以与任何细胞类型中的cas蛋白融合以向核酸酶赋予降解作用,但是使用宿主细胞内源性蛋白质的降解决定子或与宿主细胞最密切相关的物种的蛋白质的降解决定子是优选的。可以通过常规的重组蛋白产生方法将降解决定子添加到cas蛋白的c末端、n末端或两个末端上。在这方面,所述融合蛋白是与至少一个降解决定子序列缔合的非天然存在或工程化的crispr酶。所述降解决定子可以作为单个拷贝或多个拷贝插入。此外,cas融合蛋白可以包括不止一种类型的降解决定子。在某些实施方式中,编码cas融合蛋白的核酸可以经过密码子优化以在真核细胞中表达。在一些实施方式中,本发明的cas9融合蛋白切割dna的两条链以产生双链断裂(dsb)。在某些实施方式中,所述cas9融合蛋白是切口酶。在其它实施方式中,所述cas9融合蛋白是双切口酶。在另外的实施方式中,所述cas9融合蛋白是无活性cas9(dcas9),例如基本上没有核酸酶活性,例如与野生型cas9或没有突变的cas9相比,不超过5%核酸酶活性的cas9。此外,所述cas9融合蛋白可以与一个或多个功能结构域(例如转录阻遏因子或激活因子)缔合。更具体地,所述cas9融合蛋白是dcas9和/或与一个或多个功能结构域(例如转录调节因子,如vpr或krab)缔合。在一些方面,所述cas9融合蛋白具有rec2或hd2截短。在一些实施方式中,所述降解决定子可以例如使用诸如标记系统,如链霉亲和素-生物素系统的系统,通过连接头蛋白与cas核酸内切酶缔合。因而,提供了cas核酸内切酶与连接头蛋白的融合体,所述连接头蛋白对该连接头的高亲和力配体具有特异性,而所述降解决定子与所述高亲和力配体结合。例如,链霉亲和素可以是与cas核酸内切酶融合的连接头,而生物素可以与降解决定子结合。在共定位时,链霉亲和素将与生物素结合,从而将cas核酸内切酶与降解决定子连接。优选的是,所述cas核酸内切酶与降解决定子融合或共价连接。在一些实施方式中,所述融合可以是与cas核酸内切酶的n末端融合。在一些实施方式中,至少一个降解决定子与cas核酸内切酶的n末端融合。在其它实施方式中,所述融合可以是与cas核酸内切酶的c末端融合。在另外的实施方式中,至少一个降解决定子与cas核酸内切酶的c末端融合。在一些实施方式中,一个降解决定子可以与cas核酸内切酶的n末端融合,而另一个降解决定子可以与cas核酸内切酶的c末端融合。在一些实施方式中,所述cas核酸内切酶与至少两个降解决定子缔合,并且其中第一降解决定子与cas核酸内切酶的n末端融合并且第二降解决定子与cas核酸内切酶的c末端融合,所述第一降解决定子和第二降解决定子是相同的或不同的。在一些实施方式中,所述融合可以是与降解决定子的n末端融合。在其它实施方式中,所述融合可以是与降解决定子的c末端融合。在一些实施方式中,所述融合可以在cas核酸内切酶的c末端与降解决定子的n末端之间。在其它实施方式中,所述融合可以在降解决定子的c末端与cas核酸内切酶的n末端之间。在一些实施方式中,一个或两个降解决定子可以与cas核酸内切酶的n末端融合,而一个或两个降解决定子可以与cas核酸内切酶的c末端融合。在一些实施方式中,至少两个降解决定子与cas核酸内切酶缔合并且所述降解决定子是相同的降解决定子,即所述降解决定子是同源的。在一些实施方式中,至少两个降解决定子与cas核酸内切酶缔合并且所述降解决定子是不同的降解决定子,即所述降解决定子是异源的。具有两个或更多个异源的降解决定子可以是有利的,这是因为它将提供更大水平的降解控制。在n末端或c末端处多于一个降解决定子的串联融合可以增强降解。设想的是,高水平的降解将在不存在任一种稳定化配体的情况下发生,中间水平的降解将在不存在一种稳定化配体而存在另一种(或另外的)稳定化配体的情况下发生,而低水平的降解将在这两种(或两种以上)稳定化配体的两者都存在的情况下发生。所述融合蛋白可以包括与降解决定子直接融合的cas蛋白,或可选地,所述cas蛋白通过接头与降解决定子融合。在一些实施方式中,所述接头是gly-ser接头。在其它实施方式中,所述融合蛋白还包括至少一个核输出信号(nes)。在一些实施方式中,所述融合蛋白包括两个或更多个nes。在一些实施方式中,所述融合蛋白包括至少一个核定位信号(nls)。这可以是除nes之外另加的。在一些实施方式中,所述融合蛋白包含以下各项,由以下各项组成,或基本上由以下各项组成:cas蛋白、降解决定子和定位(核输入或输出)信号,所述信号作为cas蛋白与降解决定子之间的接头或作为所述接头的一部分。ha或标签也可以用作接头。在某些方面,所述融合蛋白包括nls和/或nes和/或gly-ser接头,所述gly-ser接头具有氨基酸序列gsggsgs或(ggggs)3(seqidno:66)。融合蛋白的表达可以处在组成型启动子或调节型启动子的控制之下,例如组织特异性启动子、化学诱导型启动子或细胞周期调节的启动子,如受rb或e2f转录因子控制的细胞周期蛋白b、e、a或d基因的启动子。使融合蛋白的表达处在细胞周期启动子的控制之下将调节融合蛋白的表达时间和随后的活性。“在调节型启动子的控制之下”或“受调节型启动子的控制”意指编码所关注的产物的dna序列将在控制它的rna产物转录的启动子元件的下游。“调节型启动子”是活性受顺式作用因子或反式作用因子影响的任何启动子。本发明的调节型启动子可以通过细胞周期来调节,即通过在细胞周期中的特定时间点,诸如e2f或rb的内源性转录因子被募集到启动子dna元件以阻遏或开启rna聚合酶ii对该基因的转录;通过组织特异性诱导;化学诱导,即通过与毒素、生长因子、类固醇、重金属等接触;或环境刺激,例如通过光或温度来调节。本发明还提供了一种编码所述cas-降解决定子融合蛋白的多核苷酸。在一些实施方式中,编码的融合蛋白与第一调节元件可操作地连接。在一些实施方式中,降解决定子也被编码并且与第二调节元件可操作地连接。有利的是,这里的降解决定子将“扫除”稳定化配体并且有利地是与和融合蛋白缔合的降解决定子相同的降解决定子(即相同类型的结构域)。如本文所用的术语“扫除”意指在执行以促进或结束活动的意义上。在一些实施方式中,第一调节元件是启动子并且可以任选地包括增强子。在一些实施方式中,第二调节元件是启动子并且可以任选地包括增强子。在一些实施方式中,第一调节元件是早期启动子。在一些实施方式中,第二调节元件是晚期启动子。在一些实施方式中,第二调节元件是以下各项,包含以下各项,或基本上由以下各项组成:诱导型控制元件,任选地是tet系统;或阻遏型控制元件,任选地是tetr系统。诱导型启动子可以是有利的,例如rtta,以在多西环素存在下诱导tet。本发明还提供了一种用于递送本发明的cas-降解决定子融合蛋白或编码所述融合蛋白的多核苷酸的手段。这样的递送手段可以包括例如融合蛋白的粒子、带有编码融合蛋白的多核苷酸的载体;和/或crispr/cas复合物的rna。载体可以包括dna质粒、细菌人工染色体(bac)、酵母人工染色体(yac)、病毒载体或与递送运载体,如脂质体或泊洛沙姆复合的核酸。在一些实施方式中,载体可以是质粒或病毒载体,如aav或慢病毒。使用质粒瞬时转染到例如hek细胞中可以是有利的,特别是考虑到aav的尺寸限制。虽然spcas9适合aav,但是在编码一个或多个降解决定子的核酸的情况下可能会达到上限。可激活的sgrna。如本领域已知的那样,单向导rna(“sgrna”或“grna”)指的是嵌合rna分子,其由crisprrna(crrna)和反式编码的crisprrna(tracrrna)构成。常规的sgrna(图3)具有(i)5'起点(通常是g,鸟苷核苷酸);(ii)dna引导元件或间隔区序列,其与靶核酸分子互补;(iii)下和上茎、凸起和连结,其与cas相互作用(即cas结合元件);和(iv)一个或多个发夹结构(briner等,(2014)mol.cell56(2):333-339)。根据本发明,从5'起点到dna引导元件或间隔区序列的距离的长度可以是0nt(即所述起点是引导元件的一部分)至40nt(核苷酸),优选地是0nt至20nt,或0nt至10nt,或甚至1nt至5nt。dna引导元件或序列可以具有8nt至50nt的长度,优选地9nt、10nt、11nt、12nt、13nt、14nt、15nt、16nt、17nt、18nt、19nt、20nt、21nt、22nt、23nt、24nt、25nt、26nt、27nt、28nt、29nt、30nt、31nt、32nt、33nt、34nt、35nt、36nt、37nt、38nt、40nt、42nt、44nt、46nt、48nt的长度或之间的任何范围;优选的长度是15nt-30nt,甚至更优选的长度是17nt-19nt,这使脱靶事件减到最低限度(wiles等,(2015)mamm.genome26:501-510)。dna引导元件或序列与cas结合元件或序列之间的距离的长度可以是0nt(直接相邻)至30nt,优选地是0nt至20nt或2nt至10nt。可以自由选择dna引导元件或间隔区序列以与靶核酸分子杂交。sgrna/crispr靶向和选择合适的dna引导序列的方法以及向导rna的修饰公开于例如wo2014/093709、wo2014/144761、wo2015/089486、wo2015/048577、wo2015/123339、wo2015/089486、wo2015/089427和wo2015/113063中。引导元件或间隔区序列通常与靶序列共有100%的序列同一性,尽管也考虑与靶序列具有至少80%、85%、90%和95%的序列同一性,只要sgrna为crispr/cas系统提供必要的特异性即可。如本文所用的“靶核酸”、“靶核酸分子”、“靶序列”或“靶dna分子”指的是待由crispr/cas系统编辑的基因组或线粒体dna位点。sgrna的序列允许许多变异,只要它能够结合cas并且具有与靶核酸结合的互补区即可。在特定的实施方式中,sgrna具有与靶核酸分子的模板或转录链互补或特异性杂交的dna引导元件。本文提供的数据表明可以通过将sgrna特异性靶向靶核酸分子的模板链来增加诱变频率。更特别是,在基因组编辑期间,通过sgrna将cas核酸酶靶向基因组dna序列。在sgrna与靶核酸分子进行沃森-克里克碱基配对后,cas引入双链断裂。cas:sgrna-dsb(cas:dsb)状态是异常稳定的并且保持在复合物中,除非有很大的力,如易位rna聚合酶(rnap)与它碰撞。只有当sgrna与rnap的模板链退火时,rnap与cas-dsb的碰撞才会破坏复合物。破坏cas-dsb复合物的这种机制基本上增加了诱变频率并且为cas-dsb复合物的解离是基因组编辑的限速步骤的观测结果提供了解释。因此,靶向转录链的sgrna的选择可以用于增加基因组编辑的动力学,而非模板的选择可以用于降低基因组编辑的动力学。通过模板或非模板链选择来调节动力学在调节本文所述的条形编码和/或生物处理技术方面是有用的。为了选择单个基因的模板链,首先确定在基因组内基因的取向。具体地,评估由rna聚合酶用作模板的链是在基因组的正链还是负链上取向。通常,使用者可以通过从公共数据库中访问所关注的基因的dna序列来鉴定模板sgrna。值得注意的是,公共数据库一般会将基因进行取向以使得编码链处于5'->3'取向。可以通过使用在线sgrna选择工具(例如crispor)或在模板链上找到合适的前间区序列邻近基序(pam)位点(例如5'-ngg-3'、5'-nnnngatt-3'、5'-nnagaaw-3'或5'-naaaac-3')来选择用于引入基因敲除的预期sgrna以与位于起始密码子下游(但是在外显子,优选地外显子1、2或3内)的序列杂交。对于每一个实验都应当对许多潜在的sgrna进行选择,并且应当检查每一个sgrna的脱靶活性。此外,使用者应当确认已知所关注的基因被表达并且所述基因是否以封闭的染色质的状态存在(例如通过查看dna酶超敏感位点数据)。封闭的染色质可对突变频率产生负面影响,并且缺乏通过cas靶位点的转录将会降低链选择的效果。sgrna的cas结合元件或序列优选地包括茎环或至少双链区。优选的是,茎环具有6nt至50nt的长度,例如7nt、8nt、9nt、10nt、11nt、12nt、13nt、14nt、15nt、16nt、17nt、18nt、19nt、20nt、21nt、22nt、23nt、24nt、25nt、26nt、27nt、28nt、30nt、32nt、34nt、36nt、38nt、40nt、45nt、50nt或更长的长度或介于这些值之间的任何范围。茎环可以具有60%至100%的互补核苷酸,包括a-u(由dsdna的有义链上的t编码)、g-c和g-u(由t编码)碱基对。优选的是,互补核苷酸是70%至95%或80%至90%互补的。环的序列不是必要的并且甚至可以突出cas结合区,并且仅在cas结合口袋中留下茎区。在通过sgrna与特定靶核酸结合后,cas在靶核酸中在pam位点处产生断裂(例如双链断裂或单链断裂)。sgrna的3'部分可以具有20nt至600nt的长度。通常,可以相对自由地设计该区域。如果太长,那么它甚至可能被天然存在的酶消化,这将自动产生crispr/cas系统的合适尺寸。可能的长度是20nt至400nt、25nt至300nt或30nt至200nt。总体上,sgrna优选地具有60nt至2000nt长度,优选地70nt至1500nt,或80nt至1000nt、90nt至800nt、100nt至500nt、120nt至300nt长度的尺寸。对cas9的结构分析表明尽管同源sgrna的凸起和连结区是对于cas9靶向来说最关键的特征,但是发夹1、发夹2以及上和下茎环区相对能耐受序列变异,包括核苷酸取代、插入和缺失以及甚至预测的结构破坏(briner等,(2014)mol.cell56(2):333-339)。因此,本发明利用sgrna的这些非必需区域来插入失活序列,所述失活序列允许产生这样的sgrna,所述sgrna是非功能性的直到被激活为止。在这方面,除了cas-降解决定子融合蛋白之外或作为cas-降解决定子融合蛋白的替代,本发明还提供了一种可激活的sgrna。如本文所用的“可激活的sgrna”指的是修饰的sgrna分子,其以其中cas不能与所述修饰的sgrna结合并且引入dna断裂的状态存在。在某些实施方式中,将可激活的sgrna整合或插入到所关注的基因组中。在无活性状态期间,可激活的sgrna被转录,但是由于位于sgrna的非必需区域中的失活序列的存在,因此所述rna分子对于cas结合/活性是非功能性的。失活序列位于sgrna内部或外部的可以处理修饰,但是对于cas功能来说并非可有可无的区域中。在某些实施方式中,非必需区域指的是sgrna的发夹(例如cas9sgrna的发夹1或发夹2)、上茎环和/或下茎。在特定的实施方式中,sgrna的非必需区域是发夹。“失活序列”指的是通过以下方式使sgrna无活性的核酸序列:(i)通过例如破坏sgrna折叠、固有rna结构或募集的蛋白质结合的庞大结构对cas结合造成空间位阻;(ii)顺式或反式核酶插入;或(iii)细胞(内源性或外源性)rna酶活性(例如rna酶p/z)。可以通过将cas或特定核酸酶靶向每一个失活序列的dna来激活无活性的可激活的sgrna。该靶向引入了破坏失活序列的关键区域的突变,并且恢复了cas结合新修饰的和功能性的可激活的sgrna的能力。在某些实施方式中,可激活的sgrna在sgrna的非必需区域中带有失活序列,其中所述失活序列包括一个或多个核酸内切酶识别位点。术语“核酸内切酶识别位点”指的是由核酸内切酶识别的核酸序列或结构基序,所述核酸内切酶切割所述结构基序处或附近的核酸。在某些实施方式中,使用一对核酸内切酶识别位点,其被相同或不同的核酸内切酶靶向。理想地,这对核酸内切酶识别位点处于失活序列的末端处(即侧接失活序列)以允许核酸内切酶介导的失活序列的去除。在某些实施方式中,核酸内切酶识别位点被cas9识别并且切割。值得注意的是,将可激活的sgrna插入到内源性基因的5'-utr或3'-utr中的功能在于通过将sgrna表达(以及因此对于与cas9结合的可用性)与其中插入有sgrna的基因的表达相联系来调节cas9活性。在这方面,可激活的sgrna以“关闭状态”表达,rna引导的核酸酶(例如cas9)从可激活的sgrna切除失活序列,从而将可激活的sgrna转化成“开启状态”,其中它可以与它的同源cas核酸内切酶结合。在本发明的某些实施方式中,所述失活序列是顺式作用核酶。“顺式作用核酶”是催化性rna分子,其可以作用于与它的位置相邻或邻近的靶rna。核酶切割具有位点特异性并且由靶区域处互补碱基之间的氢键键合来介导。顺式作用核酶是本领域已知的并且包括例如锤头型核酶、丁型肝炎病毒(hdv)核酶、发夹型核酶、varkud卫星(vs)核酶、i型内含子核酶和ii型内含子核酶(doudna和cech(2002)nature418:222-228)。在某些实施方式中,所述失活序列是由seqidno:2、seqidno:3或seqidno:4的核酸分子编码的顺式作用核酶。在本发明的特定实施方式中,可激活的sgrna由如表2中所示的核酸分子编码,其中“n^n”表示核酸内切酶识别位点的核苷酸序列,它们中的每一个可以由相同的或不同的核酸内切酶识别;并且“x”表示失活序列。在某些实施方式中,“x”表示顺式作用核酶并且“n^n”表示cas9核酸内切酶的核酸内切酶识别位点。表2sp:化脓性链球菌;sth:嗜热链球菌(streptococcusthermophilus);sa:金黄色葡萄球菌。为了用于本发明的系统和方法中,sgrna可以作为编码sgrna的分离的多核苷酸(例如dna分子)被提供,所述多核苷酸可以任选地被包括在载体(例如质粒)中。在某些实施方式中,优选的是,将可激活的sgrna整合到基因组中。本发明的可激活的sgrna可用于多种应用中,包括例如谱系追踪,其中可激活的sgrna的阵列以时间控制的方式相互靶向;顺序基因敲除,其中随时间推移以受控方式引入突变;使用cas9-vpr和14ntsgrna、具有改变的pam特异性的cas9核酸酶,或正交rna引导的核酸酶,使用可激活的sgrna的阵列进行转录编程,其中一些或所有sgrna具有结合或激活另一个sgrna的次要功能,所述另一个sgrna的功能在于引导dcas9(无核酸酶活性的cas9)或cas9-14ntsgrna复合物对靶基因的转录活性;和/或生物回路,即“如果基因a开启,则关闭基因b”。因此,在本发明的某些实施方式中,所述系统还提供了可激活的sgrna的阵列,其中所述可激活的同源sgrna靶向所述可激活的sgrna的阵列中的至少一个可激活的sgrna。参见例如图7、图9和图10。在一些实施方式中,所述阵列的可激活的sgrna彼此相邻。在其它实施方式中,所述阵列的可激活的sgrna彼此不相邻。本发明的crispr/cas系统可以包括使用常规的cas蛋白与可激活的同源sgrna的组合;cas-降解决定子融合蛋白与常规的sgrna的组合;或cas-降解决定子融合蛋白和可激活的sgrna这两者。crispr/cas系统的一个或这两个组分可以在用于基因组编辑的试剂盒中被提供,所述试剂盒包括编码cas-降解决定子融合蛋白和可激活的sgrna中的一者或这两者的核酸,所述核酸例如呈质粒的形式;以及关于使用crispr/cas系统进行基因组编辑的说明书。所述试剂盒可以任选地包括例如连接组分、储存缓冲液、反应缓冲液和转化感受态细胞。可以通过插入合适的dna引导元件将所述系统和试剂盒的sgrna定制成特异性地靶向所关注的基因。在某些实施方式中,所述sgrna靶向模板或非模板链,其中模板链增加激活动力学,而非模板降低动力学。除了crispr/cas系统、cas-降解决定子融合蛋白、可激活的sgrna和试剂盒之外,本发明还提供了一种编辑或调节细胞基因组(例如核dna或线粒体dna)转录的方法,所述方法是通过向真核细胞中引入或递送至少一种cas核酸内切酶和靶向基因组的核酸序列的至少一个同源sgrna而实现的,其中所述cas核酸内切酶与一个或多个降解决定子序列融合和/或所述同源sgrna在所述同源sgrna的非必需区域中带有失活序列。在某些实施方式中,至少一种cas核酸内切酶和至少一个同源sgrna(例如可激活的sgrna)的引入包括将cas核酸内切酶和/或同源sgrna整合到正在被编辑或转录调节的细胞基因组中。因此,本发明的系统、试剂盒和方法在操纵内源性基因以及异源性基因方面是有用的。“细胞”意图包括原核细胞和真核细胞,如真菌细胞(如酵母)、植物细胞、动物细胞、哺乳动物细胞和人类细胞。在某些实施方式中,所述细胞是真核细胞。根据所选择的cas蛋白,基因组编辑和转录调节可以包括靶核酸的插入、缺失、甲基化或转录中的改变(例如通过dcas9或dcas9与转录阻遏因子或激活因子的融合体)。转录中的改变包括转录的抑制、转录的激活或增加,这可以通过选择合适的cas9蛋白,例如与阻遏因子或激活因子的融合蛋白,或使用顺序激活的可激活的sgrna来促进(参见例如实施例8、实施例10和实施例12)。在某些实施方式中,本发明的方法的cas蛋白是cas9核酸内切酶。在其它实施方式中,通过本文所述的一种或多种调节型启动子,例如细胞周期、组织特异性、化学或环境调节型启动子来控制cas核酸内切酶、同源sgrna或cas核酸内切酶和同源sgrna的表达。在涉及使用细胞周期调节型启动子来调节sgrna表达的实施方式中,优选的是,所述sgrna由两个顺式作用核酶侧接以允许sgrna的适当合成。在所述方法的一些方面,同源sgrna靶向基因组的核酸序列的转录链。在其它实施方式中,同源sgrna插入或嵌入编码所关注的异源性基因或内源性基因的5'-非翻译区或3'-非翻译区(utr)的核酸中。当引入5'-utr或3'-utr中时,所插入的sgrna优选地不破坏由所述基因编码的蛋白质的基因表达(即转录)或功能。将可激活的sgrna插入到异源性基因或内源性基因的utr中提供了所述基因的表达以及功能性或非功能性的可激活的sgrna的存在。在这方面,本发明将cas9活性与其中嵌入有sgrna的基因的表达相联系。在特定的实施方式中,同源sgrna的失活序列是顺式作用核酶,例如由seqidno:2、seqidno:3或seqidno:4的核酸分子编码。在特定的实施方式中,所述同源sgrna由seqidno:14、seqidno:15、seqidno:16、seqidno:17、seqidno:18、seqidno:19、seqidno:20、seqidno:21或seqidno:22的核酸分子编码。在所述方法的其它方面,将靶向基因组中的两个或更多个序列的至少两个同源sgrna引入或递送到细胞中。在一个实施方式中,所述至少两个可激活的sgrna被顺序激活以在基因组中引入两个或更多个编辑(参见例如实施例7)。在另一个实施方式中,所述至少两个可激活的sgrna中的第一可激活的sgrna靶向编码第二可激活的sgrna的dna,因此在激活后,第二sgrna例如通过与启动子结合来调节所关注的基因的转录(参见例如实施例8、实施例10和实施例12)。出于多种原因,可以使用本发明的方法来编辑或调节细胞基因组内的转录,所述原因包括例如增强真核细胞中生物治疗剂的产生、增强原核细胞或真核细胞中商品化学品的产生、生物体或细胞培养物中的基因驱动、体内或离体基因治疗、用于诊断或研究的离体细胞转染、离体细胞分化以及转基因生物体的产生。在一个优选的实施方式中,从受试者生物体中分离细胞,将所述细胞用sgrna和cas蛋白或编码sgrna和cas蛋白的核酸(基因或cdna)转染,并且重新输注回受试者生物体(例如患者)体内。适用于离体转染的各种细胞类型是本领域技术人员公知的。在另一个实施方式中,对分离的细胞进行所述发明的基因组整合(例如cas9介导的hdr或慢病毒转导),其中经整合的dna编码程序来控制在生物体外部或在进入生物体内部后的细胞行为。当用于产生转基因动物时,本发明的方法可以包括产生作为疾病模型的转基因动物以及具有所期望的性状的动物。可以使用本发明的方法和组合物处理胚胎以产生转基因动物。在一些实施方式中,合适的胚胎可以包括来自小型哺乳动物(例如啮齿类动物、兔等)、伴侣动物、牲畜和灵长类动物的胚胎。啮齿类动物的非限制性实例可以包括小鼠、大鼠、仓鼠、沙鼠和豚鼠。伴侣动物的非限制性实例可以包括猫、狗、兔、刺猬和雪貂。牲畜的非限制性实例可以包括马、山羊、绵羊、猪、美洲驼、羊驼和牛。灵长类动物的非限制性实例可以包括卷尾猴、黑猩猩、狐猴、猕猴、狨猴、绢毛猴、蜘蛛猴、松鼠猴和长尾黑颚猴。在其它实施方式中,合适的胚胎可以包括来自鱼类、爬行动物、两栖动物或鸟类的胚胎。可选地,合适的胚胎可以是昆虫胚胎,例如果蝇胚胎或蚊子胚胎。由本发明的方法和组合物考虑的转基因生物体还包括转基因植物和种子。用于引入的合适的转基因的实例包括编码一种或多种功能性多肽或rna的外源性核酸分子,其带有或不带有一个或多个启动子,赋予生物体所期望的性状。植物中的这些性状包括但不限于除草剂抗性或耐受性;昆虫抗性或耐受性;疾病抗性或耐受性(病毒、细菌、真菌、线虫);胁迫耐受性和/或抗性,如通过对干旱、高温、寒冷、冰冻、水分过多、盐胁迫的抗性或耐受性来举例说明;氧化胁迫;增加产量;食物含量和组成;物理外观;雄性不育;抗倒伏性;淀粉、油或蛋白质的量和/或质量;氨基酸组成;等。当然,可以根据需要使用任何描述的任何两种或更多种外源性核酸,如赋予除草剂、昆虫、疾病或干旱抗性、雄性不育、多产性、淀粉特性、油的量和质量的那些,或增加产量或营养质量的那些。提供以下非限制性实施例以进一步说明本发明。实施例1:cas9融合蛋白材料和方法重组cas9纯化。根据常规方法,使cas9(pmj806,addgene公司)表达并且通过亲和、离子交换和尺寸排阻色谱步骤的组合对其进行纯化(anders和jinek(2014)methodsenzymol.546:1-20)。sgrna质粒和rna产生。将所有sgrna克隆到pspgrna(addgene公司)中。通过pcr介导的t7rna聚合酶(rnap)启动子与sgrna序列的5'末端的融合产生rna。将柱纯化的pcr产物在含有5.0μg/ml纯化的重组t7rnap和以下1×转录缓冲液的反应中用作转录模板:40mmtris(ph8.0)、2mm亚精胺、10mmmgcl2、5mmdtt和2.5mmrntp。将反应物用dna酶i处理并且使用rnaclean&concentratortm试剂盒(zymoresearch公司)纯化。cas9变体构建体和ctnnb1::egfp供体dna的产生。编码cdt1和联会蛋白的核苷酸序列是以gblock基因片段(idt公司)的形式订购的,使用gibson(neb公司)将其与编码cas9的核苷酸序列(addgene公司)的3'末端融合。编码所得的cas9-cdt1和cas9-gem融合蛋白的核酸在本文中分别如seqidno:67和seqidno:68所示。将编码cas9-cdt1和cas9-gem融合蛋白的核酸分别插入到小鼠细胞周期蛋白b启动子和小鼠细胞周期蛋白e启动子(分别参见seqidno:69和seqidno:70)的下游。cas9-cdt1和cas9-gem融合蛋白的氨基酸序列分别如seqidno:71和seqidno:72所示。通过对ctnnb1::egfp基因座进行pcr扩增(shy等,(2016)nucl.acidsres.44(16):7997-8010)以包括500bp同源臂而产生ctnnb1::egfp供体dna。对pcr产物进行ta克隆并且验证序列。细胞培养。小鼠胚胎干(es)细胞:为了使野生型、rex1:egfpd2或rosa26::teton-otx2-mcherryes细胞正常传代,将1-2×106个c57bl/6小鼠es细胞的单细胞悬浮液平板接种到先前包被有0.1%明胶的10cm培养皿(密理博公司(millipore))上。使细胞在补充有以下的knockouttm杜氏改良伊格尔氏培养基(dulbecco'smodifiedeaglemedium,dmem;gibco公司)中生长:15%的knockouttm血清替代品(gibco公司)、2mm的l-谷氨酰胺(gibco公司)、1000u/ml的penstrep(gibco公司)、1mmhepes(thermoscientific公司)、1×mem非必需氨基酸(gibco公司)、55μm的2-巯基乙醇(gibco公司)、100u/ml的lif(密理博公司)和3μm的chir99021(糖原合酶激酶3抑制剂,西格玛公司(sigma))。每2天-3天使用0.25%的胰蛋白酶-edta(gibco公司)将细胞1:10分瓶。hek293和neuro2a:hek293细胞是本领域已知的。将所有细胞维持在补充有10%胎牛血清(fbs;西格玛公司)和100μg/ml青霉素-链霉素的dmem中。通过深度测序来验证hek293的细胞系身份。转染和选择条件。对于es细胞,在平板接种到12孔培养皿中约18小时之后不久转染1×105个细胞。对于每一个孔,将5μl的lipofectaminetm2000和相关的dna在250μl的opti-memtm(gibco公司)中孵育。在适当的情况下,转染包括250ng的ppgkpuro(addgene公司)以允许消除未转染的细胞。此外,用250ng的px330cas9(野生型或变体)表达质粒和250ng相应的pspgrna质粒转染细胞。对于ctnnb1::egfp测定,添加另外400ng的ctnnb1::egfp质粒并且在48小时之后,将细胞分瓶到2μg/ml嘌呤霉素中并且施加选择48小时。对于neuro2a细胞,在转染前一晚将0.5×105个细胞平板接种到6孔培养皿中。对于每一个孔,将6μl的lipofectaminetm2000和相关的dna在500μl的opti-memtm(gibco公司)中孵育,之后添加到孔中。ctnnb1::egfp测定含有400ng的cas9表达质粒、400ng相应的pspgrna质粒、400ng的ppgkpuro(以允许消除未转染的细胞)和800ng的ctnnb1::egfp质粒。在48小时之后,将细胞分瓶到4μg/ml嘌呤霉素中并且施加选择96小时。对于hek293细胞,在转染前一晚将0.5×105个细胞平板接种到6孔培养皿中。对于每一个孔,将6μl的lipofectaminetm2000和相关的dna在500μl的opti-memtm(gibco公司)中孵育,之后添加到孔中。所有转染都含有1μg的cas9表达质粒和1μg相应的pspgrna质粒。对于深度测序噪音控制,用空的pspgrna质粒转染细胞。t7核酸内切酶i测定。通过t7核酸内切酶i(t7e1)或限制性内切酶消化来监测编辑。简单地说,在转染后36小时收获经编辑的小鼠胚胎干细胞(mesc)并且使用含有蛋白酶k的bradley裂解缓冲液提取基因组dna,继而进行etoh/nacl介导的沉淀和随后的etoh洗涤。在使用高gc缓冲液(neb公司)和标准pcr条件(98℃持续30秒;98℃持续5秒,64℃持续10秒和72℃持续15秒的30个循环;和72℃持续5分钟的1个循环)的pcr反应中,使用基因组dna(50ng)作为模板。使纯化的pcrdna的等分试样(200ng)经受t7ei并且在1.5%琼脂糖凝胶上拆分。使用imagej对凝胶图像进行定量。流式细胞术。通过进行胰蛋白酶消化并且重悬在2%fbs/pbs/2mmedta中来制备单细胞悬浮液。在lsrfortessatm流式细胞仪上分析细胞。使用flowjov9.3.2进行数据分析。通过前向角散射和侧向角散射面积对活细胞进行门控,通过侧向角散射面积和侧向角散射宽度对单细胞进行门控。对于每一个样品计数至少5×105个单活细胞(计数越高,事件的频率越低)。通过使用荧光阴性细胞作为对照,对适当的通道进行门控来定量绿色荧光事件。在转染后一周,对用于ctnnb1::egfp测定的细胞进行流式细胞术。在转染后40小时,对用于rex1::egfpd2破坏测定的细胞进行流式细胞术。蛋白质印迹法和免疫荧光法。对于蛋白质印迹分析,分别用等摩尔浓度的cas9质粒转染neuro2a、hek293和mesc,并且在转染后48小时产生裂解物。使用抗体(西格玛公司)检测标记的cas9。对于免疫荧光法,在18mm玻璃盘上将neuro2a细胞用对应的cas9和mcherry表达质粒共转染。在转染后48小时,在室温下将细胞用4%多聚甲醛固定并且透化10分钟。然后在室温下用0.3%tritontmx-100、2%fbs和1%牛血清白蛋白(bsa)将细胞封闭45分钟。用抗体的1:3000稀释液探测cas9,用αcdt1抗体(艾博抗公司(abcam))的1:1000稀释液探测内源性cdt1,并且用α联会蛋白抗体(proteintech公司)的1:500稀释液探测内源性联会蛋白。将所有抗体在封闭缓冲液中稀释并且在4℃下孵育过夜。然后在室温下,用α小鼠-fitc缀合的二抗探测细胞以进行cas9检测,并且用α兔-cy5缀合物探测cdt1/gem,持续45分钟。然后将细胞洗涤3次,第二次洗涤含有300nmdapi。然后将细胞封片并且在zeisslsm710共聚焦显微镜上成像。cas9-降解决定子融合蛋白的表达和分析已经提出了降解决定子标记的去稳定化的cas9变体(gutschner等,(2016)cellrep.14:1555-1566;maji等,(2017)nat.chem.biol.13:9-11;howden等,(2016)stemcellrep.7:508-517)。然而,已经测试了这些标记的cas9变体用于灭活cas9,而不是刺激基因组编辑的用途。为了评估将cas9蛋白去稳定化是否可以减少基因组编辑的限速解离步骤,使用来自cdt1(scfskp2的靶标)和联会蛋白(apccdh1的靶标)的降解决定子作为细胞周期传感器(tada(2007)front.biosci.12:1629-41)。除了作为传感器之外,cdt1和联会蛋白在核中也具有活性,从而使得活性cas9能够在降解之前进入核中。实际上,cas9cdt1和cas9gem融合蛋白的表达证实了,当通过免疫荧光法检查时,cas9融合蛋白的水平在整个细胞周期的不同阶段有波动。此外,蛋白质印迹分析揭示了,由于去稳定化和/或细胞周期调节的启动子,因此在变体之间蛋白质水平是不同的。为了评估基因组编辑,最初使用小鼠胚胎干(es)细胞,这是因为它们在整个细胞周期中的振荡快速,而基于细胞周期阶段特异性的影响最小(ballabeni等,(2011)proc.natl.acad.sci.usa108:19252-7)。去稳定化的cas9融合蛋白的表达水平低于未标记的cas9。然而,当靶向es细胞的rex1:egfpd2中的egfp时,去稳定化的cas9融合蛋白更有效地产生插入缺失突变。在使用靶向mcherry的非模板sgrna(sgm3)的情况下,将cas9去稳定化也增加了诱变作用,从而支持了以下结论,即cas9去稳定化克服了基因组编辑的限速解离步骤。为了测试在细胞周期的s/g2期中cas9的细胞周期调节和去稳定化是否可以增加hdr介导的编辑,在产生egfp在ctnnb1基因中的中靶插入(shy等,(2016)nucl.acidsres.44(16):7997-8010)以及中靶突变(插入缺失)方面,将组成型表达(启动子:cbh)的cas9gem、cas9cdt1以及细胞周期启动子(细胞周期蛋白e或b)控制的cas9gem、cas9cdt1和wt-cas9与组成型表达的wt-cas9进行比较。用对应的cas9构建体(图1)和模板或非模板靶向sgrna转染mesc。在转染后48小时、84小时和156小时,分别通过流式细胞术或靶向深度测序来分析hdr和插入缺失频率(图1)。cbh-wtcas9表现出模板链偏好性,hdr在所有时间点上都是一致的。这种条件下的互补插入缺失数据显示,模板和非模板的表现相似,这表明了在hdr中观测到的模板链偏好性不是非模板sgrna表现不佳的结果。cbh-cas9cdt1减轻了hdr模板链偏好性,并且插入缺失数据显示该变体增强了非模板sgrna的活性。对于细胞周期蛋白b-cas9cdt1观测到这种减少的hdr,所述细胞周期蛋白b-cas9cdt1被表达并且仅使cas9去稳定化。这两个发现表明,由于插入缺失频率增加,因此由cdt1变体而引起的去稳定化可降低hdr频率。有趣的是,细胞周期蛋白e-cascdt1在它在g1期间被稳定化时表达cas9,但是随后在s期突然去稳定化,然后停止表达。该特征使得对于非模板sgrna观测到最高的hdr频率,这表明了表达时间是至关重要的。此外,由细胞周期蛋白e引起的cas9gem表达使得模板sgrna的hdr频率最高,并且大大降低了非模板的hdr频率。该变体的插入缺失数据是变体中最低的,这表明了当cas9gem在g1期间被去稳定化,然后在s期短暂稳定化时,它的表达有利于增加hdr:插入缺失的比率。最后,在s/g2期由细胞周期蛋白b启动子引起的casgem表达增加了非模板sgrna的hdr频率而胜过wt-cas9。这些发现证实了,细胞周期调节的转录和cas9稳定化降低了蛋白质水平并且可以增加hdr水平,同时减少中靶插入缺失的积累。此外,如用cdt1变体所证实,cas9的去稳定化增加了非模板sgrna的活性而减轻模板链偏好性。增加cas9dsb的频率可潜在地导致脱靶突变增加。为了测试这种可能性,使用具有脱靶活性的sgrna。该分析使用靶向内源性emx1和zscan2基因中的位点的sgrna,所述位点先前已经通过guide-seq进行了表征(kleinstiver等,(2016)nature529(7487):490-5)。在hek293细胞中通过对来自转染细胞的基因组dna的pcr扩增子进行深度测序来测量中靶和脱靶突变。两种去稳定化的cas9融合蛋白都显示出emx1sgrna的脱靶突变减少(图2)。对于zscan2sgrna,cbh-cas9cdt1显示出中靶和脱靶突变增加,并且cbh-cas9gem显示出脱靶突变减少(图2)。因此,去稳定化显然没有增加脱靶诱变,而实际上,对于所检查的大多数良好表征的位点来说,减少了脱靶诱变。在基因组中的较低亲和力位点处的脱靶核酸酶活性已经与高水平的cas9表达相关。本文所提供的数据表明,去稳定化的cas9变体不会增加脱靶活性,这是因为降低细胞中的cas9水平会降低在低亲和力脱靶位点处的结合频率。总之,本文的发现证实了基因组在基因组编辑过程中的重要性,并且显示了靶位点处的活性如何能够影响基因组编辑程序中的限速步骤。在这些实验中,cas9没有从dsb移去的位点发生突变的频率更低,并且可以通过rnapolii通过位点的易位来预测。当靶位点存在于用于基因组编辑的细胞中表达的基因内或需要靶向非模板链的sgrna时,cas9的去稳定化可以增加基因组编辑的效率。对于需要快速的基因组编辑结果的实验,使用去稳定化的cas9变体可以缩短重复进行断裂和修复循环所需的时间,从而增加靶位点处的诱变。将细胞周期特异性去稳定化与细胞周期特异性表达相组合提供了将基因组编辑靶向细胞周期中的特定时间点的潜能。示例性泛素依赖性去稳定化的cas9融合蛋白包括但不限于cas9-cdt1(seqidno:71)和cas9-gem(seqidno:72)融合蛋白。还考虑的是,也可以使用非泛素依赖性降解决定子,包括例如来自小鼠鸟氨酸脱羧酶(modc)的pest序列,已知其通过引入这些另外的蛋白水解信号来缩短gfp的半衰期。cas9-pest融合蛋白在本文中如seqidno:73、seqidno:74和seqidno:75所示。实施例2:核酶介导的crisprsgrna失活化脓性链球菌cas9的sgrna(图3)已经被广泛表征以了解所述rna的哪些区域可以突变。该分析已经表明,尽管sgrna的凸起和连结区是对于cas9靶向来说最关键的特征,但是发夹1、发夹2以及上和下茎环区相对能耐受序列变异,包括核苷酸取代、插入和缺失以及甚至预测的结构破坏(briner等,(2014)mol.cell56(2):333-339)。为了证实sgrna可以被顺式作用核酶激活,选择了用于cas9的sgrna的发夹1模块。sgrna的发夹1出于以下两个原因对于产生可激活的sgrna是特别令人关注的:(1)发夹1对于cas9功能来说不是可有可无的并且因此可以被修饰以破坏cas9活性;和(2)发夹1可以处理广泛的突变,只要保留发夹二级结构的碱基即可。鉴于发夹1的这些特征,将核酶插入发夹1的中间(图4)。核酶基本上在转录后将sgrna切割成两半。为了在维持功能性发夹1的同时破坏核酶的切割活性,将突变引入到核酶的dna序列中。使用这种方法,产生了三种不同的顺式作用核酶-sgrna变体。具体地,将编码锤头型核酶(hammann等,(2012)rna18(5):871-85;seqidno:2)、扭转核酶(liu等,(2014)nat.chem.biol.10:739-744;seqidno:3)或丁型肝炎病毒融合核酶序列(seqidno:4)的核酸插入到化脓性链球菌sgrna的发夹1中(分别参见seqidno:5、seqidno:6和seqidno:7)。在体外转录后,证实了这些分子中的每一个都有效地自我切割。随后通过将所述变体靶向小鼠67n细胞中的gfp来测试这些核酶-sgrna变体促进活细胞中cas9介导的突变的能力。所有三种变体均靶向gfp内六个不同的序列,并且均未介导基因的显著破坏,如通过gfp荧光的流式细胞术检测所测量(图5)。随后确定了侧接核酶序列或在核酶序列内部的cas9识别位点是否可以通过对核酶进行诱变而将可激活的sgrna转化成功能性sgrna(图6)。进行了靶向切除整个核酶(策略1)或使核酶活性位点突变(策略2)的cas9和sgrna的转染。该分析比较了由野生型sgrna或策略1或策略2介导的egfp破坏频率(图6)。鉴于这些结果,可以将编码顺式作用核酶的序列插入到sgrna内的任何位置,只要核酶起到在转录期间正确折叠成它的二级结构后立即(在mg2+存在下几秒内)切割sgrna的功能即可。更具体地,可以将编码顺式作用核酶的核苷酸序列克隆到sgrna分子的非必需区域(例如茎环和发夹)中,从而用作在其转录后激活sgrna分子的手段。可激活的sgrna可以是结合来自例如化脓性链球菌、嗜热链球菌、金黄色葡萄球菌、新凶手弗朗西斯氏菌(francisellanovicida)、脑膜炎奈瑟氏球菌或侧孢短芽孢杆菌(brevibacilluslaterosporus)、毛螺菌科细菌或氨基酸球菌属菌种的任何rna引导的核酸酶的rna。像顺式作用核酶一样,假定反式作用核酶可以异源表达并且被设计成靶向sgrna的任何区域以介导sgrna的切割。编码包括插入非必需区域中的核酶的sgrna变体的核酸分子的实例示于表3中。表3sp:化脓性链球菌;sth:嗜热链球菌;sa:金黄色葡萄球菌。与核酶序列相似,可以将其它类型的失活靶位点引入sgrna序列中以使sgrna失活。例如,可以将空间位阻策略放置在sgrna内部或外部的任何位置,如添加核苷酸序列,所述核苷酸序列引起sgrnarna分子内抑制cas9结合的二级rna结构。此外,也可以将募集阻断cas9结合的rna结合蛋白、募集rna酶或结合内源性/外源性配体/化学物质的序列放置在sgrna内部或外部的任何位置。此外,将rna酶活性靶向sgrna的外在rna酶募集序列可以放置在sgrna内部或外部的任何位置。具体地,可以将trna分子放置在sgrna内,以使得rna酶p/z作用于sgrna以将它降解并且使它对于cas9是非功能性的。含有一个或多个可激活的sgrna的细胞系可以通过慢病毒转导,或对小鼠(rosa26)或人类细胞(aavs1)中的安全港基因座进行靶向同源定向修复而产生。通过对应试剂的合子注射或将小鼠胚胎干细胞注射到胚泡中而建立小鼠模型。实施例3:可激活的sgrna阵列产生了一系列可激活的sgrna以含有一个或多个sgrna,所述sgrna的功能在于彼此靶向并且彼此激活(图7、图9);靶向“条形码”基因座以用于遗传谱系记录;和/或靶向不同的sgrna进行激活,所述不同的sgrna用来结合具有转录目的或靶向核酸酶活性的rna引导的核酸酶(图8、图10)。可以将一系列可激活的sgrna构建成多顺反子阵列,其由trna侧接并且通过rna酶p/z活性分离或由在转录后切割的核酶侧接。一系列可激活的sgrna也可以是受单个启动子,如u6、cmv、四环素响应元件或细胞周期调节的启动子控制的不同基因。此外,sgrna可以位于内源性基因内并且由两个顺式作用核酶侧接,以使得它们的表达受到细胞活动的控制(图23)。可激活的sgrna的表达可以在异源性基因或内源性基因内的任何rnapolii或rnapoliii启动子的控制之下。使用可激活的sgrna对细胞进行的一般条形编码。条形编码以给予每一个可激活的sgrna至少两个靶位点的方式使用可激活的sgrna阵列。靶位点1是可激活的sgrna阵列中的后续和失活的sgrna,靶位点2(和之后的靶位点)是相同的确切靶序列,但是在基因组中的不同位点处(图10)。所述不同位点可以带有许多可激活的sgrna的靶位点,并且用作条形码区。条形码显著小于可激活的sgrna的阵列,从而允许进行下一代测序。所述条形码也可以被设计成使得可以通过本文所述的链选择来影响突变结果。此外,条形码可以位于内源性基因或异源性转录基因的3'非翻译区内,并且该条形码可以并入单细胞rna测序中以同时进行谱系追踪和转录谱分析(图8)。细胞周期控制的细胞条形编码。来自一个或多个物种的可激活的sgrna构建体可以在由cas9(或任何rna引导的核酸酶)驱动的多调节盒中发挥功能,其中所述核酸酶活性受细胞周期的调节(例如受细胞周期启动子元件控制的去稳定化的cas9)。例如,可以在细胞基因组中编码来自两个不同物种(例如化脓性链球菌和嗜热链球菌)的两个可激活sgrna阵列,所述细胞基因组还带有两种不同的细胞周期调节的核酸酶。这些核酸酶可以在细胞周期的相反阶段存在,以使得它们的核酸酶活性不重叠。每一种核酸酶只能使用已经被激活的来自它的物种的sgrna,并且每一个激活的sgrna将靶向另一物种的sgrna。使用相似的策略,受细胞周期调节的两种cas9核酸酶可以激活单个阵列中可激活的sgrna(图11)。再次,这些核酸酶可以在细胞周期的相反阶段发挥功能,以使得它们的核酸酶活性不重叠。可选地,阵列内的每一个可激活的sgrna可以靶向阵列中另一个无活性sgrna的非模板链(图12)。该靶向策略引起cas9与dsb结合直到发生dna复制为止,然后允许修复断裂。由于修复仅在dna复制期间才开始发生,因此每个细胞周期被激活的sgrna的数量显著降低并且最有可能将是一个。相反,靶向模板链将允许sgrna阵列的连续的不受细胞周期影响的激活。使用可激活的sgrna的转录编程。与为了在同时条形编码的同时作用于阵列上而具有双重功能相似,可激活的sgrna可以用于结合和/或激活另一个sgrna,所述另一个sgrna的功能在于通过dcas9激活因子/阻遏因子调节转录活性(图13)。该双重功能用于在时间上调节转录程序,并且掺入来自其它物种的rna引导的核酸酶(例如dcas12a)以严格区分转录调节与基因组编辑调节。实施例4:cas9核酸内切酶的链偏好性材料和方法重组cas9纯化。通过常规方法,使cas9(pmj806)表达并且通过亲和、离子交换和尺寸排阻色谱步骤的组合对其进行纯化(anders等,(2015)meth.enzymol.558:515-537)。用于体外cas9-rnp的sgrna合成。遵循针对px330基础质粒优化的方案,将所有sgrna克隆到pspgrna(addgene公司)中(cong等,(2013)science339:819-823)。使用适当的pspgrna作为反应模板dna,通过pcr介导的t7rnap启动子与sgrna序列的5'末端的融合而产生用于体外转录的模板。使用高gc缓冲液(neb公司)和标准pcr条件(98℃持续30秒;98℃持续5秒,64℃持续10秒和72℃持续15秒的30个循环;和72℃持续5分钟的1个循环)进行pcr反应。然后将pcr产物进行柱纯化(快而精公司(qiagen))并且在te(10mmtris-hcl(ph8.0)、1mmedta)中洗脱。使用nanodroptm2000(赛默飞世尔科技公司(thermofisherscientific))确定dna浓度,然后当用作体外转录反应的模板时将其稀释到200nm。转录反应含有5.0μg/ml纯化的重组t7rnap和1×转录缓冲液(40mmtris-hcl(ph8.0)、2mm亚精胺、10mmmgcl2、5mmdtt、2.5mmrntp)。在37℃下孵育1小时后,将反应物用不含rna酶的dna酶i(赛默飞世尔科技公司)处理并且使用rnaclean&concentratortm试剂盒(zymoresearch公司)遵循制造商的方案进行柱纯化。将纯化的rna产物在15μl水中从柱中洗脱。用于体外cas9核酸酶反应的dna模板。用于杂合体消化和转录测定的线性靶dna包括小鼠lef1、mcherry和gfp靶dna,它们是在使用高gc缓冲液(neb公司)和标准pcr条件(98℃持续30秒;98℃持续5秒,64℃持续10秒和72℃持续15秒的30个循环;和72℃持续5分钟的1个循环)的反应中,使用来自mescrosa26::teton-otx2-mcherry细胞的50ng基因组dna,通过pcr扩增而产生的。除了使用hf缓冲液之外,使用相同的参数扩增φnm1基因组dna。将所有pcr产物进行柱纯化(快而精公司),在te中洗脱,并且使用nanodroptm2000(赛默飞世尔科技公司)确定浓度。将小鼠lef1基因的不同片段用于测试t7rnap对cas9的滴定、模板dna的滴定、dsb产生的动力学、tss与cas9切割位点之间的距离、模板链碰撞和非模板链碰撞的影响的实验中。对于需要环状dsdna模板的反应(测试核酸内切酶或连接酶的可及性的实验),使用ta克隆制备质粒靶dna。将先前描述的lef1::pgk-neo和ctnnb1::egfpdna序列(shy等,(2016)nucl.acids.res.44:7997-8010)的pcr产物克隆到载体(赛默飞世尔科技公司)中。体外cas9dsb形成测定。在含有最终浓度100nm的cas9的1×cas9消化缓冲液(40mmtris(ph8.0)、10mmmgcl2、5mmdtt)中准备基本cas9dsb形成测定。在添加dna模板之前,以摩尔过量添加sgrna,并且在室温下孵育10分钟以确保形成cas9-rnp。添加靶dna达到200nm的最终浓度和50μl的最终反应体积。将反应物在37℃下孵育25分钟,然后在75℃下热失活10分钟或在37℃下用蛋白酶k处理15分钟。将来自每一个反应物的一部分(约15μl)的dna片段通过在1.5%琼脂糖凝胶上电泳而分离,并且通过溴化乙锭染色而可视化。通过在imagej中测量单个dna条带的光密度测定,然后将总切割dna除以总dna来确定切割百分比值。除非另有说明,否则对于涉及t7rnap转录的反应,除了使用1×转录缓冲液之外,应用基本cas9消化条件。在添加靶dna后,添加t7rnap达到5.0μg/ml的最终浓度。除非另有说明,否则将反应物在37℃下放置25分钟,然后在75℃下热失活10分钟。将不含dna酶的rna酶a(neb公司)添加到所有反应中并且在37℃下孵育30分钟,之后在1.5%琼脂糖凝胶上分离dna片段。除非另有说明,否则在1×cas9消化缓冲液中进行t7和t5核酸外切酶测定。使用lef1::pgk-neo质粒进行t7核酸外切酶测定并且使用sglef1消化。使用ctnnb1::egfp质粒进行t5核酸外切酶测定并且使用sgg2消化。在1×转录缓冲液中进行含有t7rnap的t5核酸外切酶测定。所有反应均含有100nm的cas9:rnp、200nm的靶dna和10u的适当核酸外切酶。对反应物进行蛋白酶k处理,之后上样到1%琼脂糖凝胶上。在含有atp的t4dna连接酶缓冲液中进行t4dna连接酶和cas9消化测定。使用200nm的cas9:rnp(sglef1)和100nm的lef1:pgk-neo质粒进行含有cas9的反应。将含有cas9的反应物在37℃下孵育30分钟,然后将温度降低到16℃并且添加40u的t4dna连接酶。将反应进行30分钟并且随后在3次连续稀释中转化成感受态dh5a。在37℃下过夜孵育后,确定氨苄西林(ampicillin)抗性菌落形成单位。使用sgm2并且通过滴定mcherry靶dna来进行cas9杂合体消化和转录反应。使用imagej测量切割频率。计算速率(v)并且绘制与底物浓度的关系图以产生米-曼氏常数(michaelis-mentenconstant)。ku70/80竞争测定。通过常规方法纯化重组人类ku70/80(hanakahi(2007)proteinexpr.purif.52:139-145)。使用生物素化的引物(5'-gcctcacacggaatct-3';seqidno:23)和fitc缀合的引物(5'-gagagccctctcccaatcttc-3';seqidno:24)(集成dna技术公司(integrateddnatechnologies))来扩增650bp的lef1靶dna,将pcr产物进行柱纯化(快而精公司)并且在te中洗脱。如制造商所述来准备myonetm(赛默飞世尔公司)以将750ng的靶dna固定到约4μl的珠粒上。将cas9和sgrna在1×cas9消化缓冲液(40mmtris(ph8.0)、10mmmgcl2、5mmdtt)中在室温下预孵育30分钟,以5:1摩尔比添加到固定的dna中,并且在37℃下孵育25分钟。同时准备没有cas9,但是含有dna酶、pmei和/或ku70/80的对照反应,并且在37℃下孵育25分钟。然后对含有cas9的反应物进行蛋白酶k处理或添加过量的ku70/80,并且在37℃下孵育15分钟。然后通过将反应管放置在磁体上来收集与珠粒结合的dna片段,并且将10μl的可溶性级分在技术上按一式三份转移到384孔板中。使用tecanpro200测量fitc荧光水平。在减去缺乏fitc标记的dna的反应的背景荧光水平之后进行计算。对于每一个反应条件进行三次独立设置的反应。细胞培养。将带有rex1:egfpd2插入(kalkan和smith(2014)philos.trans.r.soc.lond.bbiol.sci.369(1657))或rosa26::teton-otx2-mcherry插入(yang等,(2014)cellrep.7:1968-1981)的小鼠胚胎干(es)细胞在先前包被有0.1%明胶的10cm培养皿(密理博公司)中维持在补充有15%knockouttm血清替代品(gibco公司)、2mml-谷氨酰胺(gibco公司)、1000u/mlpenstrep(gibco公司)、1mmhepes(赛默科技公司(thermoscientific))、1×mem非必需氨基酸(gibco公司)、55μm2-巯基乙醇(gibco公司)、100u/mllif(密理博公司)和3μmchir99021(西格玛公司)的knockouttmdmem(gibco公司)培养基中。通常每2天-3天将细胞培养物用0.25%胰蛋白酶-edta(gibco公司)1:10分瓶。转染和选择条件。在转染2小时内,将0.25×105个es细胞新鲜平板接种到24孔培养皿的每一个孔中。对于每一个孔,将2.5μl的lipofectaminetm2000和相关的dna在125μl的opti-memtm(gibco公司)中孵育,之后添加到孔中。对于es细胞中40个不同基因的cas9诱变,转染包括150ng的ppgkpuro(addgene公司)、150ng的px330(缺乏sgrna插入序列)和150ng相关的pspgrna质粒。为了评估由于可能的深度测序或扩增错误而导致的背景突变率,将含有具有空sgrna位点的pspgrna的转染与其它含有sgrna的转染一起评估。在转染后两天,将细胞分瓶到2μg/ml嘌呤霉素中并且施加选择48小时,之后通过用含有1mg/ml蛋白酶k的bradley裂解缓冲液(10mmtris-hcl、10mmedta、0.5%sds、10mmnacl)过夜裂解来分离基因组dna,继而进行etoh/nacl沉淀,2次etoh洗涤,并且在50μl的te中洗脱。对于mcherry靶向,转染含有相同的dna,不同的是pspgrna靶向mcherry基因组插入,在转染后48小时在50μl的quickextracttm溶液(epicentre公司)中分离基因组dna以用于t7核酸内切酶1测定。表4提供了sgrna序列、距四环素响应元件(tre)的预测转录起始位点(tss)的近似距离和计算预测的sgrna中靶分数的列表(doench等,(2015)nat.biotech.34(2):184-191)。表4t7核酸内切酶1(t7e1)测定。在使用聚合酶(neb公司)和标准pcr条件(98℃持续30秒;98℃持续5秒,55℃持续10秒和72℃持续25秒的30个循环;和72℃持续5分钟的1个循环)的pcr反应中,使用基因组dna作为模板。将柱纯化(快而精公司)的pcr产物制备成于20μl的1×nebuffer2(neb公司)中200ng的dna,在95℃下变性10分钟,然后通过每秒降低1℃的温度使其降到室温。将半微升的t7e1(neb公司)添加到每一个反应中,并且将其在37℃下孵育25分钟。通过1.5%琼脂糖凝胶进行电泳来分离dna片段。分析凝胶图像并且使用imagej定量插入缺失频率。通过使用两样本t检验产生每一个sgrna的p值以比较加上多西环素和减去多西环素来进行统计分析,然后在rstudio1.0.136版中通过邦费罗尼校正(bonferronicorrection)来调整所有p值。rnaseq与插入缺失频率的生物信息学分析。大规模插入缺失诱变和rna-seq数据的来源是来自先前公开的报道(chari等,(2015)nat.methods12:823-826;chavez等,(2016)nat.methods13:563-567)。将blat和bedtools命令行工具(quinlan和hall(2010)bioinformatics26:841-842)用于将由chari等((2015)nat.methods12:823-826)使用的sgrna中的每一个分类为靶向模板或非模板基因链。使用rstudio1.0.136版(软件包:ggplot2)将所有数据合并并且可视化,从而允许确定fpkm和链取向对插入缺失频率的影响。使用平均值的多重比较和图凯对比(tukeycontrast)(软件包:multcomp)确定统计分析和显著性。流式细胞术。通过进行胰蛋白酶消化并且重悬在2%fbs/pbs/2mmedta中来制备单细胞悬浮液。在lsrfortessatm流式细胞仪上分析细胞。使用flowjov9.3.2进行数据分析。通过前向角散射和侧向角散射面积对活细胞进行门控。通过侧向角散射面积和侧向角散射宽度对单细胞进行门控。对于每一个样品计数至少5×105个单活细胞。通过使用荧光阴性细胞作为对照,对适当的通道进行门控来定量mcherry荧光事件。靶向深度测序准备和分析。在转染后四天收获基因组dna并且在pcr中使用约100ng的dna来扩增对应的靶位点,同时连接衔接子序列以进行后续的条形编码步骤。通过琼脂糖凝胶分析pcr产物,然后基于imagej定量,分别以等量为每一个平行测定汇集不同的扩增子。使用ampure珠粒(安捷伦公司(agilent))纯化汇集的pcr产物,并且使用accuprimetmii(赛默飞世尔科技公司)pcr混合物(95℃持续5分钟;95℃持续30秒,60℃持续30秒和72℃持续30秒的8个循环;和72℃持续7分钟的1个循环),用fluidigmaccessarraytm条形码对5ng的纯化池进行条形编码。在2轮0.6×固相可逆固定珠粒纯化以排除引物二聚体之前和之后,在2200tapestation(安捷伦公司)上分析条形编码的pcr产物。产生最终的扩增子池并且将其加到illuminaminiseq上,从而产生150bp的配对末端读段。插入缺失频率的确定利用在适当情况下通过扩增子解复用的crispresso命令行工具,然后通过与参考扩增子文件进行比对来确定插入缺失频率(pinello等,(2016)nat.biotechnol.34:695-697)。使用自定义命令行、python和r脚本对输出进行组装和分析。靶向φnm1的间隔区的产生。如先前所述(heler等,(2015)nature519:199-202),通过bsai克隆到pdb114上来构建带有cas9、tracrrna和靶向φnm1的单间隔区阵列的质粒。具体地,构建了间隔区rc1(质粒prh320)、rc2(prh322)、rc3(prh324)和rc4(prh326)。φnm1感染测定。将噬菌体φnm1h1分离为用间隔区4b对φnm1进行iii型crispr靶向的逃避者(goldberg等,(2014)nature514:633-637)。受噬菌体感染的细菌的板读数器生长曲线如先前所述进行(goldberg等,(2014)nature514:633-637),但稍有改动。将过夜培养物1:100稀释到2ml补充有适当抗生素和5mmcacl2的新鲜bhi肉汤中并且生长到约0.2的od600。将携带靶向间隔区的免疫细胞用缺乏crispr-cas的细胞以1:10,000的比例稀释并且用φnm1h1或φnm1g6以moi1感染。为了产生板读数器生长曲线,将针对od600归一化的200μl受感染的培养物按一式三份转移到96孔板中。每10分钟收集一次od600测量值,持续24小时。基因组链选择影响突变频率对cas9的生物化学和生物物理学表征已经鉴定出这种核酸酶的独特特性。cas9与dna结合的酶产物复合物是非常稳定的,据报道其在体外的寿命是5.5小时(richardson等,(2016)nat.biotechnol.34:339-344)。考虑到二倍体哺乳动物基因组通常仅具有cas9的两个(在dna复制之前)至四个(在dna复制之后)中靶底物,有理由认为,cas9解离速率可以是基因组编辑中的限速步骤并且cas9结合会阻止dsb修复。使用纯化的蛋白质,除非通过热变性去除cas9,否则cas9产生的dsb不能用作噬菌体修复酶、t4dna连接酶和t7核酸外切酶的底物。通过测量3'-荧光标签从通过5'-生物素与珠粒连接的dsdna分子中的释放,cas9产生的dsb没有从所述珠粒释放dna末端。当将纯化的哺乳动物dna末端结合蛋白(复合的人类ku70/80)添加到固定的dna中时,ku70/80结合可用的dna末端并且用珠粒沉淀。相反,添加ku70/80没有增加由cas9切割的dna的可溶性荧光,这表明ku70/80不能从dsb置换cas9。因此,cas9与dna的持久结合使得dsb无法由其它dna末端结合蛋白触及。鉴于该分析,假定在切割后cas9的解离速率的增加可以增加基因组编辑效率。为了证实这一点,需要一种方法以实验方式在体内操纵cas9的解离速率。因此,测试了几种体外条件以找到从dsb去除cas9的方式。通过4nm至250mmnacl的盐浓度或通过4℃至59℃的温度没有消除对dsb的保护。相比之下,使t7rnap与cas9碰撞使得dsb可由核酸外切酶活性触及。对dna末端的可及性表明了,随着聚合酶通过sgrna靶位点易位,与cas9的碰撞会将所述核酸酶从dsb移去。有趣的是,通过t7rnap置换cas9没有消除被移去的cas9rnp分子的核酸酶活性。从它的dsb置换cas9似乎也没有刺激sgrna从cas9rnp中的交换或丧失。实际上,被移去的cas9rnp的保留的核酸酶活性使得能够在体外切割另外的dna分子(图14)。由t7rnap引起的多周转效应对rntp浓度(0.125μm至2.5μm)相对不敏感,但是它的有效性在高底物浓度下以及在t7启动子与dsb之间的距离小于150bp时降低(图15)。cas9识别被称为pam或前间区序列邻近基序的短基序以便将自身与非自身区分开。先前的生物化学分析证实了与cas9缔合的dna链的自由度的不对称性(richardson等,(2016)nat.biotechnol.34:339-344);来自cas9的pam近侧的dsdna保持与cas9稳定结合,而非靶向dna链从pam远侧释放。为了确定t7rnap刺激多周转核酸酶活性的能力是否取决于它撞击cas9的哪个表面,将t7启动子移动到线性dna底物的相对端。使用这些底物的反应表明了cas9对于t7rnap从pam近端方向的易位来说是难处理的(图15)。t7rnap的不对称效应在不同的sgrna之间是一致的(图16)并且与sgrna靶位点的gc含量或与pam相邻的gc含量无关。实际上,所测试的sgrna序列的gc含量百分比与多周转cas9效率水平没有强相关性(表5;皮尔森相关系数(pearsoncorrelation):0.36)。因此,使得能够实现多周转活性的取向在本文中被称作“模板”取向,这是因为只有当sgrna与由rnap用作模板的dna链退火时cas9才被置换。表5*当可处理时。在细胞中,单个sgrna对基因组编辑显示出可变的效率。一些可变性已经归因于cas9rnp本身的形成和功能(wang等,(2014)science343:80-84)。一些染色质特征,如核小体的存在或低水平的靶基因转录,与低sgrna活性相关(chari等,(2014)natmethods12:823-826;hinz等,(2015)biochemistry54:7063-7066;horlbeck等,(2016)elife5,pii:e12677)。假定模板链偏好性也影响了cas9介导的基因组编辑的效率,这是因为cas9的解离速率可以是基因组编辑中的限速步骤。如果是这样,则与转录基因的模板链退火的sgrna将比与非模板链退火的sgrna更有效。这通过三种不同的方式得到证实。首先,通过为sgrna分配模板/非模板链状态并且基于靶基因的相对mrna水平对sgrna进行分箱,分析了先前报道的在hek293细胞中由975个sgrna引起的插入缺失突变率(chari等,(2014)natmethods12:823-826)。当由模板链sgrna靶向时,mrna水平的前两个四分位数中的基因突变的频率更高。其次,单独地靶向小鼠es细胞中表达的40个基因(17个模板sgrna、23个非模板sgrna)并且通过靶向深度测序来测量每一个靶标的插入缺失频率。与非模板链sgrna相比,模板链sgrna的插入缺失频率百分比显著更高(图17)。再次,通过使用10个模板sgrna和7个非模板sgrna靶向单个多西环素诱导型转基因mcherry,消除了基因之间的可变性(表6)。表6sgrna名称链插入缺失%-dox插入缺失%+doxsgm1模板14.539.1sgm2模板16.752.3sgm3非模板19.726.7sgm4模板11.023.1sgm5模板7.219.9sgm6模板6.713.2sgm7非模板4.35.5sgm8模板6.517.5sgm9非模板3.62.5sgm10模板11.519.6sgm11非模板21.025.2sgm12模板19.239.5sgm13非模板27.017.6sgm14非模板12.615.5sgm15模板20.358.6sgm16模板11.020.1sgm17非模板6.74.9sgm18非模板8.36.9sgm19模板15.514.7sgm20模板18.046.8通过t7核酸内切酶1测定来测量插入缺失形成显示,刺激转录仅显著增加模板链sgrna的插入缺失形成(图18)。对于用于测试t7rnap的体外效应的三个mcherrysgrna观测到相同的链偏好性(图16)。总之,这些数据证实,当rnapolii与cas9的pam远端表面碰撞时,rnapolii通过靶位点的转录可以刺激基因组编辑效率。用作多周转核酸酶的cas9可以显著增强细菌中的噬菌体免疫力,在存在许多噬菌体基因组时,所述细菌可需要在感染后存活下来。有趣的是,噬菌体基因组的核苷酸组成在正链和负链中有所不同(jin等,(2014)bmcgenomics15:793;kwan等,(2005)proc.natl.acad.sci.usa102:5174-5179;lobry(1996)mol.biol.evol.13:660-665;uchiyama等,(2008)appl.environ.microbiol.74:4149-4163),这通过在感染化脓性链球菌和嗜热链球菌的链球菌噬菌体的正链中朝向腺嘌呤和鸟嘌呤偏移(34%腺嘌呤:27%苏氨酸,和22%鸟嘌呤:17%胞嘧啶)来说明(表7)。因此,spcas9(ngg)和stcas9(nnagaaw)的pam位点以约2:1的比例优先靶向模板链,这类似于在噬菌体不敏感性突变株中靶向噬菌体的模板链的crrna的高频率(achigar等,(2017)sci.rep.7:43438;levin等,(2013)plosgenet.9:e1003312)。表7数据表明,stcas9和spcas9的链偏好的pam序列进化为靶向噬菌体基因组的正链,这是因为它为细菌提供了针对噬菌体的选择性优势。与改变噬菌体中全基因组gc偏移的进化挑战性障碍相比,改变由cas9使用的pam序列相对更简单并且仅需要少量突变(kleinstiver等,(2015)nature523:481-485)。靶向噬菌体正链提供了优势,这是因为它将在裂解基因转录后更频繁地产生多周转核酸酶。为了直接测试链偏好性对细菌免疫力的影响,使用了φnm1噬菌体的两种毒性型式。一种含有使溶源性盒的转录所需的启动子失活的突变(φnm1γ6;goldberg等,(2014)nature514:633-637),而另一种表达溶源性盒,但是在ci阻遏基因内带有失活性缺失(φnm1h1)。因此,这两种噬菌体都不能建立溶源性,但是它们在溶源性盒的转录上不同。产生了不同的细菌菌株,这些菌株带有靶向在φnm1γ6和φnm1h1这两者中均存在的阻遏基因内的模板或非模板链序列的间隔区。用每一种噬菌体感染每一种菌株并且通过随时间推移而测量od600来确定它们的存活率。从受感染的细菌培养物的板读数器生长曲线来解释每一个间隔区对这两种噬菌体的干扰效率。靶向非模板链的两个间隔区(rc2和rc4)对任一种噬菌体均显示出相似的干扰。相反,靶向模板链的间隔区(rc1或rc3)在切割φnm1h1的转录靶标时显著更为有效。针对t7rnap易位在体外将cas9转变成多周转核酸酶的能力,测试了φnm1内相同的四个靶位点(图19),证实了模板链偏好性对噬菌体基因组的影响。这些结果表明,跨越cas9的主动转录通过将cas9转变为多周转酶来改善crispr免疫力。综上所述,从它的dsb置换cas9刺激了基因组编辑效率并且增加了crispr免疫力。数据表明,rnap对cas9的取向依赖性置换提供了一种测量链偏好性对基因组的影响的方法。除了rnap通过cas9的易位之外,包括复制和修复活动的其它dna代谢过程也可能会产生足够的力以移去cas9(图20)。与rnap沿它的模板的模式化易位相反,在基因组内那些其它活动的位置和时间不太可预测。开发修饰的cas9酶或刺激cas9从dsb中的去除的处理可以增加基因组编辑过程的速率并且降低sgrna有效性的可变性。这在非转录位点处和在具有低dna代谢活性的细胞中应当特别有用。实施例5:另外的方法合成引导通过cas9靶位点的转录。转录因子cas9s:将无核酸酶活性(例如dcas9)的rna引导的核酸酶(例如cas9或cas12a)与转录激活结构域融合或它的sgrna含有转录激活结构域(例如dcas9-vp64),并且功能在于将rna聚合酶募集到基因组中的特定区域。将rna聚合酶引导到cas9靶位点:转录因子cas9s靶向实际的基因组编辑靶位点(核酸酶活性cas9所针对的序列)的上游。转录因子cas9和核酸酶活性cas9这两者都同时存在于细胞中,并且转录因子cas9通过与模板结合的核酸酶活性cas9引导rna聚合酶活性。转录因子cas9s与核酸酶活性cas9是不同的物种以避免sgrna的共用。例如,来自嗜热链球菌的转录因子cas9具有化脓性链球菌不能利用的sgrna。因此,化脓性链球菌cas9可以是核酸酶活性cas9并且这两者可以在该系统内协作。小分子影响cas9从dsb的解离。可以通过高通量筛选来鉴定破坏cas9-dsb复合物的小分子,所述高通量筛选包括其中对该复合物的破坏介导了从cas9释放dsb而解离的cas9是非功能性的,或介导了从dsb释放cas9以使得cas9是功能性的并且是多周转酶的测定。合适的测定包括来自所期望的物种的重组cas9、纯化的sgrna和发荧光的靶dna。当荧光靶dna处于cas9-dsb状态或在未切割状态下游离时,它以非荧光形式存在。与cas9解离的经切割的dna将发荧光作为破坏cas9-dsb复合物的读数。该测定是可以用于检测合适的小分子的许多荧光测定的实例。可选地,可以设计以引起cas9从dsb解离而不会完全抑制cas9与靶dna缔合的能量破坏cas9-dsb复合物的小分子。更特别是,小分子被设计成与一般的cas9-dsb复合物特异性相互作用,以使得它们破坏性地结合:i)在靶dnar环内(插入dna链之间);ii)在sgrna:dna杂合体内(插入rna链与dna链之间);iii)在ruvc结构域内和非靶链相互作用(插入氨基酸残基与dna之间);iv)在hnh结构域内和靶链相互作用(插入氨基酸残基与dna之间);v)pam序列和pam相互作用结构域(插入氨基酸残基与dna之间);vi)在sgrna的支架区(不可编程区域)内以引起sgrna或cas9去折叠;vii)在ruvc或hnh核酸酶结构域内或外表面上以破坏sgrna或cas9折叠;或viii)在cas9或sgrna的任何区域内部或外部而破坏其折叠和/或结合dsb的能力。sgrna:dna杂合体的酶促破坏。与具有rna:dna降解活性的rna酶h酶、结构域或任何rna:dna结合酶融合的cas9核酸酶用于通过破坏sgrna:dna杂合体来解离cas9-dsb复合物。一旦cas9与dsb形成复合物,所述rna酶就会将其识别为底物并且将它降解,从而引起cas9从dsb释放。rna酶通过以长形式或短形式存在的接头(甘氨酸-丝氨酸型)与cas9的n末端或c末端连接。根据所使用的cas9的物种,所述长度可由于酶接触到它的底物所需的“到达”量而变化。周围dna序列的酶促破坏。将cas9核酸酶与具有特异性或非特异性活性的一个或多个核酸酶融合。所述另外的核酸酶的功能在于切割紧邻的基因组序列以使得cas9-dsb复合物被完全去除并且真正地漂走。这些另外的核酸酶可以使用短或长接头(甘氨酸-丝氨酸)与cas9的n末端或c末端融合并且是具有小的共有序列(少于5个或6个核苷酸)的限制性内切酶。cas9核酸酶的突变修饰。通过改变/缺失/插入一个或多个氨基酸残基来使cas9核酸酶突变以使得所述酶的功能在于释放dsb而不是保持结合,并且所述核酸酶也可具有新发现的多周转酶活性。这样的突变可以存在于pam相互作用结构域、ruvc核酸酶结构域和/或hnh核酸酶结构域内或周围。这些突变可以通过pcr或gibson组装产生,或可以通过易错pcr产生cas9突变体的文库,然后针对多周转核酸酶活性和/或与dsb的快速解离来筛选突变体。实施例6:通过生物处理器介导的遗传记录和细胞编程来实现时间调节的、顺序作用的、预定的细胞行为细胞过程的遗传记录。可以使用本文所述的模块,即cas融合蛋白、可激活的sgrna和链偏好性的组合将在细胞的生命周期中发生的动态事件记录到它的基因组中。引起转录的所关注的细胞事件(天然或合成)可以与模块关联以用作所述事件的直接或间接遗传记录的输入。例如,在它的3'-utr内含有嵌入的正常sgrna的基因一旦被转录,所述基因就将在不同的基因组位点处通过所述sgrna引起诱变活动,从而最终形成所述事件的遗传记录。在同时记录所关注的事件的同时,与细胞周期进程相关的遗传记录器将记录产生特定基因组条形码的每一次细胞分裂,这在下文中被称作“世代条形编码”(图11和图12)。该条形码允许重建基于世代的谱系树,并且与其它细胞事件相关联的记录数据可以在所述谱系树的范围内被映射以确定事件发生的时间和持续时间。遗传记录事件的过程发挥了对细胞群体进行被动条形编码的功能并且引起了预编程的生物处理器的活动。生物处理器和生物处理。生物处理器由与本文所述的一个或多个其它模块相关联的一个或多个可激活的sgrna构成,目的在于执行一系列顺序的遗传事件。可激活的sgrna用作“程序”,并且由这些sgrna引导的对cas9活动的调节用作中心代码。cas9活动直接受以“开启”状态存在的可激活的sgrna或存在的正常sgrna以及sgrna靶位点的控制。生物处理器程序的性质提供了cas9活动的顺序调节,从而使得特定的cas9活动依赖于另一cas9活动的完成,如生物处理器内可激活的sgrna的转化(图7)。所述依赖性允许通过世代条形编码系统来调节生物处理器的编程(图13),这使得编程的cas9活动视细胞完成特定数量的细胞周期而定。生物处理器活动的启动取决于生物处理器被编程为对其作出响应的细胞事件。启动生物处理器活动的一般直接输入需要正常或开启状态的可激活的sgrna的转录。这些sgrna可以嵌入内源性或合成基因本身,或可以由它们自身的启动子控制。引起活性sgrna转录的细胞事件的存在将引起直接遗传记录事件,其中所述细胞事件引起sgrna的活性;或间接遗传记录事件,其中所述细胞事件引起sgrna活性的下游控制,如信号传导级联。该遗传记录事件将可激活的sgrna从“关闭”状态转变成“开启”状态,从而允许在生物处理器内编程的下一个事件开始。生物处理的作用是通过所需的模块顺序完成预编程的事件,并且生物处理器的中心程序是可激活的sgrna的阵列(图7和图9)。每一个生物处理器的输出通常需要其它模块来影响可激活的sgrna从“关闭”状态转变成“开启”状态的速率。所述速率受链偏好性cas9活性(图12)和细胞周期cas9(图11)的影响。输出。生物处理器的直接输出是cas9介导的诱变(遗传变化和条形编码)或转录激活或阻遏(表观遗传调节)。这些活动引起细胞行为的改变,并且可以通过生物处理器的设计而多重化。重要的是,每一个输出可以通过细胞周期完成的数量来调节,如在细胞生命周期中特定或多个时间点使内源性基因表达或突变。输出可以通过生物处理器对内源性转录的依赖性来进一步调节。例如,当所关注的基因表达时,嵌入3'-utr的sgrna介导了对细胞进行条形编码或突变/转录调节一系列靶基因的过程。对特定输出的依赖性可以涵盖很少或许多细胞活动并且唯一的主要要求是在所关注的物种中验证的cas9活性。实施例7:在特定数量的细胞周期完成后进行基因敲除以诱导细胞凋亡世代条形编码。跟踪的细胞过程是细胞周期进程。世代条形编码生物处理器使用一系列可激活的sgrna与通过靶向非模板链而获得的链偏好性(图12、图20)或细胞周期cas9(图11)的组合。链偏好性。将10个可激活的sgrna放置在阵列中以计数10次细胞分裂,其中每一个都含有它们自身的用于哺乳动物细胞的人类u6启动子或用于酵母的snr52启动子。每一个可激活的sgrna都靶向它自身的dna直接相邻和下游的可激活的sgrna的dna。每一个sgrna都与非模板链结合,从而允许每个细胞周期将一个可激活的sgrna从“关闭”状态转变成“开启”状态。阵列中第一个可激活的sgrna将通过激活性sgrna的转染而被激活,或它将是正常的sgrna并且将通过诱导型启动子来控制转录。由第一个sgrna引起的活动将开始启动记录10次细胞分裂的过程。细胞周期cas9。将20个sgrna放置在阵列中,其中奇数编号的sgrna对应于g1期特异性cas9(pam=ngg),而偶数编号对应于g2期特异性cas9(pam=nag)。如图11中所示,阵列中的每一个sgrna都将靶向新生sgrna的dna,并且遵循图11中所述的机制,每个细胞周期两个sgrna将从“关闭”状态转变成“开启”状态。生物处理。在从“关闭”状态转变成“开启”状态后,阵列中的最后一个sgrna(sgrnaks1)靶向在另一个位置处另一个sgrna的基因组整合dna。第二位置处的sgrna(sgrnaks2)被转变成“开启”状态并且将cas9活性靶向位于基因组中的其它三个位置的三个不同的sgrna(sgrnaks3、sgrnaks4和sgrnaks5)。与sgrnaks2结合的cas9将sgrnaks3、sgrnaks4和sgrnaks5转变成“开启”状态。输出。sgrnaks3靶向bcl2开放阅读框(orf)的模板链,sgrnaks4靶向mdm2orf的模板链,并且sgrnaks5靶向aktorf的模板链。模板链靶向确保了快速的基因组编辑动力学以使对这些蛋白质的遗传破坏速率达到最大。bcl2、mdm2和akt都通过不同的机制促进细胞存活。破坏所有这些基因会引起内在凋亡途径的有效激活。在酵母中,这些蛋白的同源物是bcl2(bcl2)和sch9p(akt),并且mdm2被靶向tps1的sgrna替代。此外,可以对世代条形编码基因座进行测序以从存活的细胞重建谱系树。实施例8:在特定数量的细胞周期完成后进行基因敲除和转录激活以诱导细胞静止细胞过程的遗传记录。跟踪的过程是细胞周期进程,并且所使用的世代条形编码系统基本上与实施例7中所述的系统相似,不同的是在本实施例中仅使用链偏好性。在该系统中,所利用的cas9核酸酶是与转录激活结构域“vpr”融合的核酸酶活性cas9(kiani等,(2015)naturemeth.12(11):1051-4)。通过改变sgrna的长度来调节核酸酶活性和rna聚合酶ii(rnap)的募集。20ntsgrna允许cas9切割dna并且介导诱变,而14ntsgrna消除了cas9活性,但是核酸酶仍然与dna结合并且可以募集rnap以转录靶基因座(参见例如图22)。生物处理。世代条形编码系统中的最后一个sgrna(sgrnaqt2)靶向另一个sgrna的dna(sgrnaqt2),并且激活的qt2随后靶向三个不同sgrna的dna(sgrnaqt3、sgrnaqt4和sgrnaqt5)。在本实施例中,sgrnaqt3是靶向e2f的20ntsgrna,sgrnaqt4是靶向p16ink4a的启动子的14ntsgrna,并且sgrnaqt5是靶向p21的启动子的14ntsgrna。14ntsgrna靶向距转录起始位点(tss)200bp之内的启动子以有效激活转录。输出。sgrnaqt3靶向e2f的模板链以诱导遗传破坏性突变。e2f的丧失会引起永久性细胞周期停滞在g1期或进入g0期。sgrnaqt4介导的p16ink4a表达和sgrnaqt5介导的p21表达诱导进入g0期,从而造成细胞生长抑制和有效的静止。对于酵母同源物,e2f(e2f)、p16ink4a和p21的转录激活被成视网膜细胞瘤蛋白rb替代。此外,可以对世代条形编码基因座进行测序以重建谱系树。实施例9:检测细胞应激并且诱导细胞凋亡的生物处理器细胞过程的遗传记录。在该系统中记录的细胞过程是代谢应激后果,即活性氧簇(ros)。为了监测该过程,将正常的sgrna嵌入到3个对应的ros诱导的基因nf-κb、atf和ap-1中的3'非翻译区(3'-utr)中。嵌入的sgrna由两个顺式切割核酶(rgr)侧接,从而使得在基因完全转录时产生功能性sgrna(参见例如图23)。然而,这些基因是沉默的直到发生ros的积累为止。整合了该处理器的细胞表达两种cas9变体:野生型cas9(wt-cas9,pam=ngg)和dcas9-krab(pam=nag)。dcas9-krab是募集内源性转录阻遏机制的无核酸酶活性的cas9。生物处理。在这三个基因中的任一个转录时,产生功能性sgrna。该sgrna(sgrnanf1)靶向三个不同的可激活的sgrna(nf2、nf3和nf4)的dna。sgrnanf1、sgrnanf2和sgrnanf3均与含有nggpam序列的dna互补,从而使得它们能够介导wt-cas9特异性诱变。sgrnanf4结合含有nagpam的dna,从而使它的活性对dcas9-krab具有特异性。sgrnanf2成功转变成“开启”状态可将cas9诱变活性靶向bcl2,sgrnanf3的转变将活性靶向mdm2,并且sgrnanf4的转变通过在tss后50bp之内结合而阻遏tnfα的转录。这些活动也可以在世代条形编码存在下进行以确定ros积累相对于细胞老化的时间,只要对sgrnanf2、sgrnanf3和sgrnanf4的dna进行测序即可。输出。bcl2和mdm2的诱变将引起内在凋亡途径的诱导。tnfα基因座的阻遏将排除tnfα的分泌,从而最终减少附近细胞的炎症反应和坏死。在酵母中,这些蛋白的同源物是bcl2(bcl2),mdm2被靶向tps1的sgrna替代,并且tnfα没有替代。实施例10:赋予细胞存活基因的转录和炎症基因的阻遏的所关注的异源性基因的时间编码诱导细胞过程的遗传记录。被记录并且用作生物处理器的主要输入的主要过程是细胞周期进程。在该系统中,世代条形编码过程通过使用链偏好性记录了10次细胞分裂。条形编码和转录激活事件由cas9-vpr(pam=ngg)介导,并且细胞还表达dcas9-krab(pam=nag)。生物处理1。在完成10个细胞周期后,世代条形编码阵列中的最后一个sgrna(sgrnah1)是靶向14ntsgrna(sgrnah2)的dna的20ntsgrna。sgrnah2靶向转录沉默的异源性的所关注基因(goi)的启动子。14ntsgrnah2结合具有nggpam的序列,从而使得cas9-vpr靶向该位点并且对异源性基因产物进行转录激活。输出1。发生异源性基因的表达,所述基因在3'-utr中编码两个正常的sgrna,即rgr(rgrh3和rgrh4)。生物处理2。所述rgr在转录后变成功能性sgrna,从而转变成sgrnah3和sgrnah4。sgrnah3将cas9-vpr靶向bcl2启动子,并且sgrnah5将dcas9-krab靶向干扰素α的tss附近。输出2。sgrnah4的活性引起bcl2的转录,从而通过抑制细胞凋亡而促进细胞存活。sgrnah5阻遏干扰素α,从而减少潜在的细胞应激反应和炎性细胞因子向周围细胞的分泌。实施例11:生物处理器介导的两个独立的内源性途径的关联以产生新的细胞行为细胞过程的遗传记录。所记录的过程是一个途径的受体介导的信号传导,以及第二途径的转录活动,其中第二途径依赖于第一途径。所述记录是由cas9-vpr活性介导的,所述活性是由受到受体信号传导级联调节的基因(基因1)中的3'-utr正常sgrna以及基因2中嵌入3'-utr的可激活的sgrna转变成“开启”状态而引导的。生物处理。基因2被不断转录并且在它的3'-utr中含有无活性的可激活的rgr(sgrna2)。sgrna2向“开启”状态的转变取决于sgrna1的表达,所述sgrna1是嵌入基因1的3'-utr中的正常sgrna。在受体-配体结合后,所关注的信号传导级联诱导基因1的转录,从而引起sgrna1的表达。sgrna1将sgrna2转变成“开启”状态。sgrna2靶向可激活的sgrna3的dna,从而将它转变成“开启”状态。sgrna3是14ntsgrna并且靶向所关注的靶基因的启动子。输出。sgrna3引起某个靶基因表达的活性完全取决于这两个途径同时表达,从而允许将两个通常无关的途径相关联。如果在世代条形编码的背景下进行,那么可以对可激活的sgrna的dna进行测序以确定过程的动力学和时间。实施例12:在完成特定数量的细胞周期时诱导基因以操纵细胞适应性细胞过程的遗传记录。利用链偏好性,通过cas9-vpr介导的世代条形编码在10个细胞周期内记录细胞周期进程。在完成5次(sgrnac1)、7次(sgrnac2)和10次(sgrnac3)细胞分裂之后被转变成“开启”状态的可激活的sgrna具有在不同的内源性基因处介导转录或突变的次要靶标。生物处理。在完成5次细胞分裂后,sgrnac1被激活并且靶向sgrnaf1的dna。sgrnaf1是14ntsgrna并且靶向所关注的异源性基因的启动子以促进转录。在完成7个细胞周期后,sgrnac2被转变成“开启”状态并且靶向sgrnaf2的dna。sgrnaf2是20ntsgrna,其靶向三个不同的14nt可激活的sgrna(sgrnae1、e2和e3)的dna。该靶向将这些sgrna转变成“开启”状态,并且它们结合苹果酸酶(sgrnae1)、过氧化氢酶(sgrnae2)和超氧化物歧化酶(sgrnae3)的启动子来激活转录。在第10次且最后一次记录的细胞分裂后,sgrnac3被激活并且靶向sgrnaf3(20nt)的dna。sgrnaf3通过在相关dna处进行基因组编辑而将sgrnae4、e5和e6转变成“开启”状态。sgrnae4靶向bcl2的orf,sgrnae5靶向mdm2的orf,并且sgrnae6靶向akt的orf。输出。在第5次细胞分裂时,所关注的异源性基因在细胞存活期的整个其余部分中连续表达。在第7个细胞周期之后,靶向产生napdh的苹果酸酶以与过氧化氢酶和超氧化物歧化酶同时增加表达水平。这些靶向努力试图减少在产生异源性基因产物时由ros积累而引起的细胞应激。在10个细胞周期之后,诱导细胞凋亡。实施例13:通过生物处理器技术跟踪产生生物治疗剂的细胞的细胞历史以使细胞行为自动化已知的是,在7天内,产生单克隆抗体(mab)的cho细胞培养物的活力和生产力会在约第5天-第6天达到最高的mab滴度时降低。本发明的系统在细胞分裂之前对细胞进行条形编码并且将细胞分裂与其它细胞行为,如mab表达和毒性水平相结合。通过说明的方式,尽管第1次-第9次细胞分裂没有表现出任何毒性迹象并且维持高mab表达,但是在第13次细胞分裂时,检测到中等量的毒性、mab表达减少和抗体聚集,并且在第17次细胞分裂时,检测到高水平的毒性、没有产物分泌和开始细胞死亡(图21)。根据该分析,可以推断的是,由于代谢应激,因此连续的产物表达随时间推移而具有毒性;在整个细胞群体中生长速率由于生物反应器而不同;使产物基因沉默的细胞获得适应性优势并且生长更快;中等细胞培养密度提供最佳的收率,但是以细胞健康为代价;并且在第9次细胞分裂时诱导产物表达降低了毒性并且使收率达到最大。因此,使用从遗传记录产生的数据,可以通过使用可激活的sgrna将指令程序嵌入到细胞的基因组中而产生高性能的mab产生细胞。例如,在第1次细胞分裂时,可以执行程序1,其促进细胞繁殖。在第9次细胞分裂时,可以执行程序2,其促进基因产物的诱导。在第10次细胞分裂时,可以执行程序3,其中诱导细胞周期停滞,伴随有细胞存活基因的表达和基因产物的表达。在第11次细胞分裂时,执行程序4以使得分泌所有产物并且诱导细胞死亡。参见图21。序列表<110>伊利诺伊大学理事会(theboardoftrusteesoftheuniversityofillinois)<120>用于基因组编辑和调节转录的crispr/cas系统和方法<130>uic0076wo<150>us62/563,128<151>2017-09-26<150>us62/563,131<151>2017-09-26<150>us62/563,133<151>2017-09-26<160>79<170>patentinversion3.5<210>1<211>100<212>rna<213>化脓性链球菌(streptococcuspyogenes)<400>1ggggccacuagggacaggauguuuuagagcuagaaauagcaaguuaaaauaaggcuaguc60cguuaucaacuugaaaaaguggcaccgagucggugcuuuu100<210>2<211>65<212>dna<213>人工序列<220><223>合成寡核苷酸<400>2ggggtgcttcggatgctgatgagtccgtgaggacgaaacagggcaacctgtccatccggt60atccc65<210>3<211>54<212>dna<213>人工序列<220><223>合成寡核苷酸<400>3ggtgcctaacactgccaatgccggtcccaagcccggataaaagtggagggggca54<210>4<211>52<212>dna<213>人工序列<220><223>合成寡核苷酸<400>4cctgtcaccggatgtgctttccggtctgatgagtccgtgaggacgaaacagg52<210>5<211>186<212>dna<213>人工序列<220><223>合成多核苷酸<400>5gttttagagctagaaatagcaagttaaaataaggctagtccgttatcaacttcgggcgcc60tcagatacggtcgcctgtcaccggatgtgctttccggtctgatgagtccgtgaggacgaa120acaggcgattttttaacttcgggcgcctcagatacgggcccgaagtggcaccgagtcggt180gctttt186<210>6<211>182<212>dna<213>人工序列<220><223>合成多核苷酸<400>6gttttagagctagaaatagcaagttaaaataaggctagtccgttatcaacttcgggcgcc60tcagatacggggtgcctaacactgccaatgccggtcccaagcccggataaaagtggaggg120ggcattttttaacttcgggcgcctcagatacgggcccgaagtggcaccgagtcggtgctt180tt182<210>7<211>192<212>dna<213>人工序列<220><223>合成多核苷酸<400>7gttttagagctagaaatagcaagttaaaataaggctagtccgttatcaacttcgggcgcc60tcagatacggggggtgcttcggatgctgatgagtccgtgaggacgaaacagggcaacctg120tccatccggtatcccttttttaacttcgggcgcctcagatacgggcccgaagtggcaccg180agtcggtgcttt192<210>8<211>194<212>dna<213>人工序列<220><223>合成多核苷酸<400>8gttttagagctgggtcgggcgcctcagatacggggggtgcttcggatgctgatgagtccg60tgaggacgaaacagggcaacctgtccatccggtatcccaacttcgggcgcctcagatacg120ggcccgacccagcgagttaaaataaggcttagtccgtactcaacttgaaaaggtggcacc180gattcggtgttttt194<210>9<211>194<212>dna<213>人工序列<220><223>合成多核苷酸<400>9gttttagagctgggtacccagcgagttaaaataaggcttagtccgtactcaacttgaacg60ggcgcctcagatacggggggtgcttcggatgctgatgagtccgtgaggacgaaacagggc120aacctgtccatccggtatcccaacttcgggcgcctcagatacgggcccgaaggtggcacc180gattcggtgttttt194<210>10<211>194<212>dna<213>人工序列<220><223>合成多核苷酸<400>10gtttttgtactctggtcgggcgcctcagatacggggggtgcttcggatgctgatgagtcc60gtgaggacgaaacagggcaacctgtccatccggtatcccaacttcgggcgcctcagatac120gggcccgaccagaagctacaaagataaggcttcatgccgaaatcaacaccctgtcatttt180atggcaggtgtttt194<210>11<211>194<212>dna<213>人工序列<220><223>合成多核苷酸<400>11gtttttgtactctggtaccagaagctacaaagataaggcttcatgccgaaatcaacaccc60tgtcattcgggcgcctcagatacggggggtgcttcggatgctgatgagtccgtgaggacg120aaacagggcaacctgtccatccggtatcccaacttcgggcgcctcagatacgggcccgtt180atggcaggtgtttt194<210>12<211>215<212>dna<213>人工序列<220><223>合成多核苷酸<400>12gttttagtactctggaaacagaatctactaaaacaaggcaacgggcgcctcagatacggt60cgcctgtcaccggatgtgctttccggtctgatgagtccgtgaggacgaaacaggcgaaac120ttcgggcgcctcagatacgggcccgaagtggcaccgagtcggtgcttttgacgaatgccg180tgtttatctcgtcaacttgttggcgagattttttt215<210>13<211>215<212>dna<213>人工序列<220><223>合成多核苷酸<400>13gttttagtactctggaaacagaatctactaaaacaaggcaagacgaatgccgtgtttatc60tcgtcaactcgggcgcctcagatacggtcgcctgtcaccggatgtgctttccggtctgat120gagtccgtgaggacgaaacaggcgaaacttcgggcgcctcagatacgggcccgaagtggc180accgagtcggtgctttttgttggcgagattttttt215<210>14<211>81<212>dna<213>人工序列<220><223>合成寡核苷酸<220><221>misc_feature<222>(53)..(57)<223>n是a、c、g或t<400>14gttttagagctagaaatagcaagttaaaataaggctagtccgttatcaacttnnnnnaag60tggcaccgagtcggtgctttt81<210>15<211>88<212>dna<213>人工序列<220><223>合成寡核苷酸<220><221>misc_feature<222>(16)..(20)<223>n是a、c、g或t<400>15gttttagagctgggtnnnnnacccagcgagttaaaataaggcttagtccgtactcaactt60gaaaaggtggcaccgattcggtgttttt88<210>16<211>88<212>dna<213>人工序列<220><223>合成寡核苷酸<220><221>misc_feature<222>(59)..(63)<223>n是a、c、g或t<400>16gttttagagctgggtacccagcgagttaaaataaggcttagtccgtactcaacttgaann60nnnaaggtggcaccgattcggtgttttt88<210>17<211>88<212>dna<213>人工序列<220><223>合成寡核苷酸<220><221>misc_feature<222>(17)..(21)<223>n是a、c、g或t<400>17gtttttgtactctggtnnnnnaccagaagctacaaagataaggcttcatgccgaaatcaa60caccctgtcattttatggcaggtgtttt88<210>18<211>88<212>dna<213>人工序列<220><223>合成寡核苷酸<220><221>misc_feature<222>(68)..(72)<223>n是a、c、g或t<400>18gtttttgtactctggtaccagaagctacaaagataaggcttcatgccgaaatcaacaccc60tgtcattnnnnnttatggcaggtgtttt88<210>19<211>24<212>dna<213>人工序列<220><223>合成寡核苷酸<220><221>misc_feature<222>(20)..(24)<223>n是a、c、g或t<400>19tagatgttgtcatctttaannnnn24<210>20<211>24<212>dna<213>人工序列<220><223>合成寡核苷酸<220><221>misc_feature<222>(15)..(19)<223>n是a、c、g或t<400>20tagatgttgtcatcnnnnntttaa24<210>21<211>92<212>dna<213>人工序列<220><223>合成寡核苷酸<220><221>misc_feature<222>(42)..(46)<223>n是a、c、g或t<400>21gttttagtactctggaaacagaatctactaaaacaaggcaannnnngacgaatgccgtgt60ttatctcgtcaacttgttggcgagattttttt92<210>22<211>92<212>dna<213>人工序列<220><223>合成寡核苷酸<220><221>misc_feature<222>(70)..(74)<223>n是a、c、g或t<400>22gttttagtactctggaaacagaatctactaaaacaaggcaagacgaatgccgtgtttatc60tcgtcaactnnnnntgttggcgagattttttt92<210>23<211>16<212>dna<213>人工序列<220><223>合成寡核苷酸<400>23gcctcacacggaatct16<210>24<211>21<212>dna<213>人工序列<220><223>合成寡核苷酸<400>24gagagccctctcccaatcttc21<210>25<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>25ggagccgtacatgaactgag20<210>26<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>26ggcaccaacttcccctccga20<210>27<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>27gtaatgcagaagaagaccat20<210>28<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>28gccgagggccgccactccac20<210>29<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>29ccatgccgccggtggagtgg20<210>30<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>30ctacaacgtcaacatcaagt20<210>31<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>31caacttgatgttgacgttgt20<210>32<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>32tgaagggcgagatcaagcag20<210>33<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>33tctgcttgatctcgcccttc20<210>34<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>34gacccaggactcctccctgc20<210>35<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>35gaactcgccgtcctgcaggg20<210>36<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>36cttgaagctgtccttccccg20<210>37<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>37ccacttgaagccctcgggga20<210>38<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>38gaagggcagggggccaccct20<210>39<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>39aagctgaaggtgaccaaggg20<210>40<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>40gggcgagggccgcccctacg20<210>41<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>41tctgggtgccctcgtagggg20<210>42<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>42ctcgaactcgtggccgttca20<210>43<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>43catgcgcttcaaggtgcaca20<210>44<211>20<212>dna<213>人工序列<220><223>合成寡核苷酸<400>44ggataacatggccatcatca20<210>45<211>23<212>dna<213>人工序列<220><223>合成寡核苷酸<400>45gttcaatctgatttcttttatgg23<210>46<211>23<212>dna<213>人工序列<220><223>合成寡核苷酸<400>46tagattgtctaaagttgagatgg23<210>47<211>23<212>dna<213>人工序列<220><223>合成寡核苷酸<400>47gtttctaaaggttatctcttagg23<210>48<211>23<212>dna<213>人工序列<220><223>合成寡核苷酸<400>48tcgatttcgctatcaaattctgg23<210>49<211>23<212>dna<213>人工序列<220><223>合成寡核苷酸<400>49gtaatgcagaagaagaccatggg23<210>50<211>22<212>dna<213>人工序列<220><223>合成寡核苷酸<400>50tgtcccctcagttcatgtacgg22<210>51<211>23<212>dna<213>人工序列<220><223>合成寡核苷酸<400>51gcctcaacacgaacagagaaagg23<210>52<211>22<212>dna<213>人工序列<220><223>合成寡核苷酸<400>52ggagccgtacatgaactgaggg22<210>53<211>22<212>dna<213>人工序列<220><223>合成寡核苷酸<400>53tcgtgaccaccctgacctacgg22<210>54<211>23<212>dna<213>人工序列<220><223>合成寡核苷酸<400>54gtgaaccgcatcgagctgaaggg23<210>55<211>22<212>dna<213>人工序列<220><223>合成寡核苷酸<400>55ctgaagcactgcacgccgtagg22<210>56<211>11<212>prt<213>人工序列<220><223>合成肽<220><221>misc_feature<222>(1)..(1)<223>xaa表示thr或ser。<220><221>misc_feature<222>(9)..(9)<223>xaa表示arg或gln。<400>56xaalyslysserlyslysasnleuxaaargleu1510<210>57<211>11<212>prt<213>人工序列<220><223>合成肽<400>57thrlyslysserlyslysasnleuargargleu1510<210>58<211>13<212>prt<213>人工序列<220><223>合成肽<400>58thrsermetthraspphetyrhisserlysargargleu1510<210>59<211>13<212>prt<213>人工序列<220><223>合成肽<400>59proleuileseraspphephealalysarglysargser1510<210>60<211>12<212>prt<213>人工序列<220><223>合成肽<400>60argargvalthraspphephealaargargargpro1510<210>61<211>12<212>prt<213>人工序列<220><223>合成肽<400>61arglysleuthraspphetyrprovalargargser1510<210>62<211>10<212>prt<213>人工序列<220><223>合成肽<400>62gluproglugluproglualaaspglnhis1510<210>63<211>10<212>prt<213>人工序列<220><223>合成肽<400>63glucysglugluthrgluvalaspglnhis1510<210>64<211>14<212>prt<213>人工序列<220><223>合成肽<400>64leualaprotyrileprometaspglygluasppheglnleu1510<210>65<211>14<212>prt<213>人工序列<220><223>合成肽<400>65leualaproalaalaglyaspthrileileserleuaspphe1510<210>66<211>15<212>prt<213>人工序列<220><223>合成肽<400>66glyglyglyglyserglyglyglyglyserglyglyglyglyser151015<210>67<211>4494<212>dna<213>人工序列<220><223>合成多核苷酸<400>67atggactataaggaccacgacggagactacaaggatcatgatattgattacaaagacgat60gacgataagatggccccaaagaagaagcggaaggtcggtatccacggagtcccagcagcc120gacaagatacagcatcggcctggacatcggcaccaactctgtgggctgggccgtgatcac180cgacgagtacaaggtgcccagcaagaaattcaaggtgctgggcaacaccgaccggcacag240catcaagaagaactatcggagccctgctgttcgacagcggcgaaacagccgaggccaccc300ggctgaagagaaccgccagaagaagatacaccagacggaagaaccggatctgctatctgc360aagagatcttcagcaacgaatgccaaggtggacgacagcttcttccacagactggaagag420tccttcctggtggaagaggataagaagcacgagcggcaccccatcttcggcaacatcgtg480gacgaggtggcctaccacgagaagtcccaccatctaccacctgagaaagaaactggtgga540cagcaccgacaaggccgacctgcggctgatctatctggccctggcccacatgatcaagtt600ccggggccacttcctgatcgagggcgacctgacccgacaacagcgacgtggacaagctgt660tcatccagctggtgcagacctacaaccagctgttcgaggaaaaccccatcaacgccagcg720gcgtggacgccaaggccatcctgtctgccagactgagaagagagacggctggaaaatctg780atcgcccagctgcccggcgagaagaagaatggcctgttcggaaacctgattgccctgagc840ctgggcctgacccccaacttcaagagcaacttcgacctggccgggatgcaaactgcagct900gagcaaggacacctacgacgacgacctggacaacctgctggcccagatcggcgaccagta960cgccgacctgtttctggccgccaagaacctgtccgacgccatcctgctggcgacatctga1020gagtgaacaccgagatcaccaaggcccccctgagcgcctctatgatcaagagatacgacg1080agcaccaccaggacctgaccctgctgaaagctctcgtgcggcagcagctgcctgaaagta1140caagagattttcttcgaccagagcaagaacggctacgccggctacattgacggcggagcc1200agccaggaagagttctacaagttcatcaagcccatcctggaaaagatggacggcaccgag1260gactgctcgtaagctgaacagagaggacctgctgcggaagcagcggaccttcgacaacgg1320cagcatcccccaccagatccacctgggagagctgcacgccattctgcggcggcaggaaga1380tttttaccattcctgaagacaaccgggaaaagatcgagaagatcctgaccttccgcatcc1440cctactacgtgggccctctggccaggggaaacagcagattcgcctggatgaccagaaaga1500gcgaggaaaccataccccctggaattcgaggaagtggtggacaagggcgcttccgcccag1560agcttcatcgagcggatgaccaacttcgataagaacctgcccaacgagaaggtgctgccc1620aagcacagcctgctgtacggtacttcaccgttataacgagctgaccaaagtgaaatacgt1680gaccgagggaatgagaaagcccgccttcctgagcggcgagcagaaaaaggccatcgtgga1740cctgctgttcaagaccaaccggaaatgaccgtgaagcactgaaagaggactacttcaaga1800aaatcgagtgcttcgactccgtggaaatctccggcgtggaagatcggttcaacgcctccc1860tgggcacataccacgatctgctgaaaattatcaaggacaaggacttcctggacaatgagg1920aaaacgaggacattctggaagatatcgtgctgaccctgacactgtttggacagagagatg1980atcgaggaacggctgaaaacctatgcccacctgttcgacgacaaagtgatgaagcagctg2040aagcggcggagatacaccggctggggcaggctgagccggaagctgatcaacggaccggga2100caagcagtccggcaagacaatcctggatttcctgaagtccgacggcttcgccaacagaaa2160cttcatgcagctgatccacgacgacagcctgacctttaaagaggacatccagaaagcccg2220ggtccggccagggcgatagcctgcacgagcacattgccaatctggccggcagccccgcca2280ttaagaagggcatcctgcagacagtgaaggtggtggacgagctcgtgaaagtgatgggcc2340ggcacagccgagaacatcgtgatcgaaatggccagagagaaccagaccacccagaaggga2400cagaagaacagccgcgagagaatgaagcggatcgaagagggcatcaaagagctgggcagc2460cagatcctgaagaacccccgtggaaaacacccagctgcagaacgagaagctgtacctgta2520ctacctgcagaatgggcgggatatgtacgtggaccaggaactggacatcaaccggctgtc2580cgactacgatgtggacctatcggcctcagagctttctgaaggacgactccatcgacaaca2640aggtgctgaccagaagcgacaagaaccggggcaagagcgacaacgtgccctccgaagagg2700tcgtgaagaagatgaagaactacggcggcgctgctgaacgccaagctgattacccagaga2760aagttcgacaatctgaccaaggccgagagaggcggcctgagcgaactggataaggccggc2820ttcatcaagagacagctggtggaaacccgcagatcaaaagcacgtggcacagatcctgga2880ctcccggatgaacactaagtacgacgagaatgacaagctgatccgggaagtgaaagtgat2940caccctgaagtccaagctggtgtccgatttccggaggatttccgttttacaaagtgcgcg3000agatcaacaactaccaccacgcccacgacgcctacctgaacgccgtcgtgggaaccgccc3060tgatcaaaaagtaccctaagctggaaagcgagttcgtgtacgcgactacaggtgtacgac3120gtgcggaagatgatcgccaagagcgagcaggaaatcggcaaggctaccgccaagtacttc3180ttctacagcaacatcatgaactttttcaagaccgagattaccctggcaacggcgagaccg3240gaagcggcctctgatcgagacaaacggcgaaaccggggagatcgtgtgggataagggccg3300ggattttgccaccgtgcggaaagtgctgagcatgccccaagtgaatatcgtgaaaagacc3360gagggcagacaggcggcttcagcaaagagtctatcctgcccaagaggaacagcgataagc3420tgatcgccagaaagaaggactgggaccctaagaagtacggcggcttcgacagccccacct3480ggcctattctggctggtggtggccaaagtggaaaagggcaagtccaagaaactgaagagt3540gtgaaagagctgctggggatcaccatcatggaaagaagcagcttcgagaagaatcccatc3600gacttctggaagccaaggctacaaagaagtgaaaaaggacctgatcatcaagctgcctaa3660gtactccctgttcgagctggaaaacggccggaagagaatgctggcctctgccggcgaact3720gcagaagggaacgaactggccctgcctccaaatatgtgaacttcctgtacctggccagcc3780actatgagaagctgaagggctcccccgaggataatgagcagaaacagctgtttgtggaac3840agcacaagcactacctgacgagatcatcgagcgatcagcgagttctccaagagagtgatc3900ctggccgacgctaatctggacaaagtgctgtccgcctacaacaagcaccgggataagccc3960atcagagagcaggccgagaatatatccacctgtttacccgaccaatctgggagcccctgc4020cgccttcaagtactttgacaccaccatcgaccggaagaggtacaccagcaccaaagaggt4080gctggacgccaccctgatccaccagagcacaccggcctgtacgagaacggatcgacctgt4140ctcagctgggaggcgacaaaaggccggcggccacgaaaaaggccggccaggcaaaaaaga4200aaaaggggagtggcggttctggaccttcccctgctggcctgctctgcgcgctcagcttct4260gccacatctgggagtcgaaaacgcgctcgacctcccgccgctccgggacgcgaccaggcc4320aggcctcccgccagacggcggctgcgactgtccgtggacgagtgagctcaccctccacac4380tgaagcccctgatatccccgcttgtccatctcctgggcagaaaatcaaaaagagcacccc4440ggctgcagggcagcctccacaccttacttctgcccaagatcaggatacatctaa4494<210>68<211>5357<212>dna<213>人工序列<220><223>合成多核苷酸<400>68acccacgcgccggactcggcccggaactctgcgtctcaggggcggggaggacgagtcagg60agggacagtgcgcgcgcgcgggagaccggcggatgacgggttcttaactccgggccctcc120cgtgcctctccagtcaagcccggccgccgatcattgcgatctgccctgctgacattccac180ttgggcggcccatgccgctcctcccggccccgcacgctcagaatccttcaggcgctcgcg240ccaagattctcccctcagcgagcccgccccgcagccgcgcgcgccagacgactctcccgc300gtcccgccccgcgcaggccccgcctaccacgtccgcaccccgcccccggcgcgcaggccc360tgacatctagccccaccccttgtgcatgccacgcccgtaaaagaacacgccccccgggag420gccacgcccccaccagagctcctcgctggtccgcgcggcgacggggcgggccccgggcct480gcggcggccgctgaggggctcgcagccctcggggcggggcgcgagggcgggacggggccg540gtgccgcgcgctgggatttttaaatgtcccgctcgaagccgggcgcaggagcagccggct600cgacagccagcggtgtagggggcaggcgcggatcccgccagcgccgcgcgctcggccgcc660gcctcccgcctcctgcttcccgctcgccgtcggcctcctgcccgctcgagcgcgcctgcc720tgcggcggacacagctgggtacgggctgcctggactcccgggactcccgggactcccacg780actcccaggcgtccggatggactataaggaccacgacggagactacaaggatcatgatat840tgattacaaagacgatgacgaaaatggccccaaagaagaagcggaaggtcggtatccacg900gagtcccagcagccgacaagaagtacagcatcggcctggacatcggcaccaactctgtgg960gctgggccgtgatcaccgacgagtacaggtcccagcaagaaattcaaggtgctgggcaac1020accgaccggcacagcatcaagaagaacctgatcggagccctgctgttcgacagcggcgaa1080acagccgaggccacccggctgaagagaaccgccgaagagatacaccagacggaagaaccg1140gatctgctatctgcaagagatcttcagcaacgagatggccaaggtggacgacagcttctt1200ccacagactggaagagtccttcctggtggaagaggataaaagcagagcggcaccccatct1260tcggcaacatcgtggacgaggtggcctaccacgagaagtaccccaccatctaccacctga1320gaaagaaactggtggacagcaccgacaaggccgacctgcggctgactatctgccctggcc1380cacatgatcaagttccggggccacttcctgatcgagggcgacctgaaccccgacaacagc1440gacgtggacaagctgttcatccagctggtgcagacctacaaccagctgttcaggaaaacc1500catcaacgccagcggcgtggacgccaaggccatcctgtctgccagactgagcaagagcag1560acggctggaaaatctgatcgcccagctgcccggcgagaagaagaatggcctgttcggaac1620ctgatgccctgagcctgggcctgacccccaacttcaagagcaacttcgacctggccgagg1680atgccaaactgcagctgagcaaggacacctacgacgacgacctggacaacctgctggccc1740agacggcgaccatacgccgacctgtttctggccgccaagaacctgtccgacgccatcctg1800ctgagcgacatcctgagagtgaacaccgagatcaccaaggcccccctgagcgcctctatg1860atcaagagaacgacgagcacaccaggacctgaccctgctgaaagctctcgtgcggcagca1920gctgcctgagaagtacaaagagattttcttcgaccagagcaagaacggctacgccggcta1980cattgacggcggagcagccaggaagattctacaagttcatcaagcccatcctggaaaaga2040tggacggcaccgaggaactgctcgtgaagctgaacagagaggacctgctgcggaagcagc2100ggaccttcgacaacggcagcacccccaccagatcacctgggagagctgcacgccattctg2160cggcggcaggaagatttttacccattcctgaaggacaaccgggaaaagatcgagaagatc2220ctgaccttccgcatcccctactacgtggccctctggccagggaaacagcagattcgcctg2280gatgaccagaaagagcgaggaaaccatcaccccctggaacttcgaggaagtggtggacaa2340gggcgcttccgcccagagcttcatcgagcggataccaacttcgataaaacctgcccaacg2400agaaggtgctgcccaagcacagcctgctgtacgagtacttcaccgtgtataacgagctga2460ccaaagtgaaatacgtgaccgagggaatgagaaagcccgcttcctgagcggcgacagaaa2520aaggccatcgtggacctgctgttcaagaccaaccggaaagtgaccgtgaagcagctgaaa2580gaggactacttcaagaaaatcgagtgcttcgactccgtggaaatcccggcgtggaagatc2640gttcaacgcctccctgggcacataccacgatctgctgaaaattatcaaggacaaggactt2700cctggacaatgaggaaaacgaggacattctggaagatatcgtgctgaccctacactgttt2760gaggacaggagatgatcgaggaacggctgaaaacctatgcccacctgttcgacgacaaag2820tgatgaagcagctgaagcggcggagatacaccggctggggcaggctgagccggaagcgat2880caacggcatccgggaaagcagtccggcaagacaatcctggatttcctgaagtccgacggc2940ttcgccaacagaaacttcatgcagctgatccacgacgacagcctgacctttaaagaggac3000atcagaaagcccaggtgtccggcagggcgatagcctgcacgagcacattgccaatctggc3060cggcagccccgccattaagaagggcatcctgcagacagtgaaggtggtggacgagctcgt3120gaaagtgatggccggcacaagcccgagaaatcgtgatcgaaatggccagagagaaccaga3180ccacccagaagggacagaagaacagccgcgagagaatgaagcggatcgaagagggcatca3240aagagctgggcagccgatcctgaaagaacaccccgtgaaaacacccagctgcagaacgag3300aagctgtacctgtactacctgcagaatgggcgggatatgtacgtggaccaggaactggac3360atcaaccggctgtccgactacatgtggaccatatcgtgcctcaagctttctgaaggacga3420ctccatcgacaacaaggtgctgaccagaagcgacaagaaccggggcaagagcgacaacgt3480gccctccgaagaggtcgtgaagaagataagaactactggcggcagctgctaacgccaagc3540tgattacccagagaaagttcgacaatctgaccaaggccgagagaggcggcctgagcgaac3600tggataaggccggcttcatcaagagacagctggggaaacccggcagatcacaaagcagtg3660gcacagatcctggactcccggatgaacactaagtacgacgagaatgacaagctgatccgg3720gaagtgaaagtgatcaccctgaagtccaagctggtgtccatttccggaaggatttccagt3780tttacaaagtgcgcgagatcaacaactaccaccacgcccacgacgcctacctgaacgccg3840tcgtgggaaccgccctgatcaaaaagtaccctaagctggaaagcgagttcgtgtacggcg3900actacaaggtgtcgacgtgcggaagatgatcgccaagagcgagcaggaaatcggcaaggc3960taccgccaagtacttcttctacagcaacatcatgaactttttcaagaccgagattaccct4020ggccaacggcgagatccggagcggcctctgatcgagacaaacggcgaaaccggggagatc4080gtgtgggataagggccgggattttgccaccgtgcggaaagtgctgagcatgccccaagtg4140aatatcgtgaaaaagaccgaggtgcaacaggcggcttcagcaaagagtctatcctgccca4200agaggaacagcgataagctgatcgccagaaagaaggactgggaccctaagaagtacggcg4260gcttcgacagccccaccgtggcctattctgtgcggtggtggccaaagtggaaaagggcaa4320gtccaagaaactgaagagtgtgaaagagctgctggggatcaccatcatggaaagaagcag4380cttcgagaagaatcccatcgactttctggaagccaagggcacaaagaagtgaaaaaggac4440ctgatcatcaagctgcctaagtactccctgttcgagctggaaaacggccggaagagaatg4500ctggcctctgccggcgaactgcagaagggaaacgaactggccctgcctccaaatatgtga4560acttcctgtacctggccagccactatgagaagctgaagggctcccccgaggataatgagc4620agaaacagctgtttgtggaacagcacaagcactacctggacgagatcatcgagcgatcag4680cgagttctccaagagagtgatcctggccgacgctaatctggacaaagtgctgtccgccta4740caacaagcaccgggataagcccatcagagagcaggccgagaatatcatccacctgtttac4800ctgaccaatctgggagcccctgccgccttcaagtactttgacaccaccatcgaccggaag4860aggtacaccagcaccaaagaggtgctggacgccaccctgatccaccagagcatcaccggc4920ctgtacgaacacggatcgacctgtctcagctgggaggcgacaaaaggccggcggccacga4980aaaaggccggccaggcaaaaaagaaaaaggggagtggcggttctggaatgaatcccagta5040tgaagcagaaacaagagaaatcaaagagaatataaagaatagttctgtcccaagaagaac5100tctgaagatgattcagccttctgcatctggatctcttgttggaagagaaaatgagctgtc5160cgcaggcttgtccaaaaggaaaatcggaatgaccacttaacatctacaacttccagccct5220ggggttattgtcccagaatctagtgaaaataaaaatcttggaggagtcacccaggagtca5280tttgatcttatgattaaagaaaatccatctctcagtattggaaggaagtggcagaaaaac5340ggagaaaggcgctgtaa5357<210>69<211>5479<212>dna<213>人工序列<220><223>合成多核苷酸<400>69taaggagtttgacatagaaagatagagagttgcaatagagatctttgttaaataagattt60tacaaaactatatttttggagtaatcttaaacttagagcaaaattaagcataaggcacat120tttcttttcactggggcagtcgctattggggagcttggtatgttaatctatttgcttata180ataaccatttacagctgggcggtggtggcacacgcctttaatcccagcacttgggaggca240gaggcaggtggatttgagttcgaggccagcctggtatacagagtgagtttcaggacagcc300aggcctatacagagaaaccctgtctcgaaaaaacaaaaagcaaaaacaaaacaaaaaaca360tttactatgtgtatgtgtgtaaacttcatgctatcaacctcaaaatccatcccagatagc420tgttggaacagtcatattaaagaccgtctctgcaacaaagctttcgggaactcttaagat480agtattggcacttctatagaagaggtgaaaacaataaataggacaacagcaaaactgata540tgcgtactccccacagtcattttcctaaagtccaacggagtcgcctgggctagcttggac600agcacacaagtgagcactgcacgccatgcctcctaggcaggtggcagcaatgcgacttgt660gcgcaggcatagagcctgacctcgcgaggggtgggcggtgctgccagcggcagcctcgct720ctggccccgcccccgccccacccccgccgaacttgggtcgcggatcgcccaggaaacgca780ttctacgggaacccgcggcgccaatgggaagagagcgagtgccacgaactggccaatgag840gagcgagcggcccggggtttaaacctaagcccggcagactcctccggcggctgcggagga900acggctgttagtgtttagctgtggatagccagaggttagggtgtcttctcgaatcgggga960acctctgattttggaggagccatggactataaggaccacgacggagactcaaggatatga1020tattgattacaaagacgatgacgataagatggccccaaagaagaagcggaaggtcggtat1080ccacggagtcccagcagccgacaagaagtacagcatcggcctggacatcggcaccactct1140gtggctgggccgtgatcaccgacgagtacaaggtgcccagcaagaaattcaaggtgctgg1200gcaacaccgaccggcacagcatcaagaagaacctgatcggagccctgctgttcgacagcg1260ggaaacagccaggccacccggctgaagagaaccgccagaagaagatacaccagacggaag1320aaccggatctgctatctgcaagagatcttcagcaacgagatggccaaggtggacgacagc1380ttcttcccagactggaaagtccttcctggtggaagaggataagaagcacgagcggcaccc1440catcttcggcaacatcgtggacgaggtggcctaccacgagaagtaccccaccatctacca1500cctgagaaagaaatggtggacagcccgacaaggccgacctgcggctgatctatctggccc1560tggcccacatgatcaagttccggggccacttcctgatcgagggcgacctgaaccccgaca1620acagcgacgtggacaagctgttcatccagctggtgcagacctacaaccagctgttcgagg1680aaaaccatcaacgccagcggcgtggacgccaaggccatcctgtctgccagactgagcaag1740agcagacggctggaaaatctgatcgcccagctgcccggcgagaagaagaatggcctgttc1800ggaaacctgatccctgagcctgggcctgacccccaacttcaagagcaacttcgacctggc1860cgaggatgccaaactgcagctgagcaaggacacctacgacgacgacctggacaacctgct1920ggcccagatcggcgacagacgccgacctgtttctggccgccaagaacctgtccgacgcca1980tcctgctgagcgacatcctgagagtgaacaccgagatcaccaaggcccccctgagcgcct2040ctatgatcaagagatacgacgacacaccaggacctgaccctgctgaaagctctcgtgcgg2100cagcagctgcctgagaagtacaaagagattttcttcgaccagagcaagaacggctacgcc2160ggctacattgacggcggagccagccaggagagtctacaagttcatcaagcccatcctgga2220aaagatggacggcaccgaggaactgctcgtgaagctgaacagagaggacctgctgcggaa2280gcagcggaccttcgacaacggcagcatcccccacagatcacctgggagagctgcacgcca2340ttctgcggcggcaggaagatttttacccattcctgaaggacaaccgggaaaagatcgaga2400agatcctgaccttccgcatcccctactacgtgggccctctgccagggaaacagcagattc2460gcctggatgaccagaaagagcgaggaaaccatcaccccctggaacttcgaggaagtggtg2520gacaagggcgcttccgcccagagcttcatcgagcggatgaccaactcgataagacctgcc2580caacgagaaggtgctgcccaagcacagcctgctgtacgagtacttcaccgtgtataacga2640gctgaccaaagtgaaatacgtgaccgagggaatgagaaagcccgccttcctggcggcgag2700agaaaaaggccatcgtggacctgctgttcaagaccaaccggaaagtgaccgtgaagcagc2760tgaaagaggactacttcaagaaaatcgagtgcttcgactccgtggaaatctccggcgtga2820agatcggtcaacgcctccctgggcacataccacgatctgctgaaaattatcaaggacaag2880gacttcctggacaatgaggaaaacgaggacattctggaagatatcgtgctgaccctgaca2940ctgttgaggacagaagatgatcgaggaacggctgaaaacctatgcccacctgttcgacga3000caaagtgatgaagcagctgaagcggcggagatacaccggctggggcaggctgagccggaa3060gctgatcaacgcatccgggacagcagtccggcaagacaatcctggatttcctgaagtccg3120acggcttcgccaacagaaacttcatgcagctgatccacgacgacagcctgacctttaaag3180aggacatccagaaagccaggtgtccggcagggcgatagcctgcacgagcacattgccaat3240ctggccggcagccccgccattaagaagggcatcctgcagacagtgaaggtggtggacgag3300ctcgtgaaagtgatgggccggccaagcccgagaactcgtgatcgaaatggccagagagaa3360ccagaccacccagaagggacagaagaacagccgcgagagaatgaagcggatcgaagaggg3420catcaaagagctgggcagccagatcctgaagaacaccccgtgaaaacacccagctgcaga3480acgagaagctgtacctgtactacctgcagaatgggcgggatatgtacgtggaccaggaac3540tggacatcaaccggctgtccgactacgatgtggacatatcgtgcctcaggctttctgaag3600gacgactccatcgacaacaaggtgctgaccagaagcgacaagaaccggggcaagagcgac3660aacgtgccctccgaagaggtcgtgaagaagatgaagaactctggcggcagctgctgacgc3720caagctgattacccagagaaagttcgacaatctgaccaaggccgagagaggcggcctgag3780cgaactggataaggccggcttcatcaagagacagctggtggaaaccggcagatcacaaag3840cactggcacagatcctggactcccggatgaacactaagtacgacgagaatgacaagctga3900tccgggaagtgaaagtgatcaccctgaagtccaagctggtgtccgatttccgaaggattt3960ccagttttacaagtgcgcgagatcaacaactaccaccacgcccacgacgcctacctgaac4020gccgtcgtgggaaccgccctgatcaaaaagtaccctaagctggaaagcgagttcgtgtcg4080gcgactacaaggtgtacacgtgcggaagatgatcgccaagagcgagcaggaaatcggcaa4140ggctaccgccaagtacttcttctacagcaacatcatgaactttttcaagaccgagattac4200cctgccaacggcgagatccggaagggcctctgatcgagacaaacggcgaaaccggggaga4260tcgtgtgggataagggccgggattttgccaccgtgcggaaagtgctgagcatgccccaag4320tgaatatcgtaaaaagaccgaggtgcagacagcggcttcagcaaagagtctatcctgccc4380aagaggaacagcgataagctgatcgccagaaagaaggactgggaccctaagaagtacggc4440ggcttcgacagccccacgtggcctattctgtgctggtgtggccaaagtggaaaagggcaa4500gtccaagaaactgaagagtgtgaaagagctgctggggatcaccatcatggaaagaagcag4560cttcgagaagaatcccatcgacttctggaagccaagggctacaaaaagtgaaaaaggacc4620tgatcatcaagctgcctaagtactccctgttcgagctggaaaacggccggaagagaatgc4680tggcctctgccggcgaactgcagaagggaacgaactggccctgccctccaaaatgtgaac4740ttcctgtacctggccagccactatgagaagctgaagggctcccccgaggataatgagcag4800aaacagctgtttgtggaacagcacaagcactacctggacgagatcatcgagcagatcagc4860gagttctccaagagagtgatcctggccgacgctaatctggacaaagtgctgtccgccaca4920acaagcaccgggataagcccatcagagagcaggccgagaatatcatccacctgtttaccc4980tgaccaatctgggagcccctgccgccttcaagtactttgacaccaccatcgaccggaaga5040ggtaaccagcaccaaagaggtgctggacgccaccctgatccaccagagcatcaccggcct5100gtacgagacacggatcgacctgtctcagctgggaggcgacaaaaggccggcggccacgaa5160aaaggccggccggcaaaaaagaaaaaggggagtggcggttctggaccttcccctgctagg5220cctgctctgcgcgctccagcttctgccacatctgggagtcgaaaacgcgctcgacctccc5280gccgctccgggacgcgacaggccaggcctcccgccagacggcggctgcgactgtccgtgg5340acgaggtgagctcaccctccacacctgaagcccctgatatccccgcttgtccatctcctg5400ggcagaaaatcaaaaagagcaccccgctgcagggcagcctccacaccttacttctgccca5460agatcaggataccatctaa5479<210>70<211>5343<212>dna<213>人工序列<220><223>合成多核苷酸<400>70acccacgcgccggactcggcccggaactctgcgtctcaggggcggggaggacgagtcagg60agggacagtgcgcgcgcgcgggagaccggcggatgacgggttcttaactccgggccctcc120cgtgcctctccagtcaagcccggccgccgatcattgcgatctgccctgctgacattccac180ttgggcggcccatgccgctcctcccggccccgcacgctcagaatccttcaggcgctcgcg240ccaagattctcccctcagcgagcccgccccgcagccgcgcgcgccagacgactctcccgc300gtcccgccccgcgcaggccccgcctaccacgtccgcaccccgcccccggcgcgcaggccc360tgacatctagccccaccccttgtgcatgccacgcccgtaaaagaacacgccccccgggag420gccacgcccccaccagagctcctcgctggtccgcgcggcgacggggcgggccccgggcct480gcggcggccgctgaggggctcgcagcctggggcggggcgcgagggcgggacggggccggt540gccgcgcgctgggatttttaaatgtcccgctcgaagccgggcgcaggagcagccggctcg600acagccagcgcggtgtagggggcaggcgcggtcccccagcgccgcgcgctcggccgccgc660ctcccgcctcctgcttcccgctcgccgtcggcctcctgcccgctcgagcgcgcctgcctg720cggcggacacagcttcgggtacgggctgcctggactccgggatcccgggactcccacgac780tcccaggcgtccggatggactataaggaccacgacggagactacaaggatcatgatattg840attacaaagacgatgacgataagatggccccaaagaagaagcgaaggtcgtatccacgga900gtcccagcagccgacaagaagtacagcatcggcctggacatcggcaccaactctgtgggc960tgggccgtgatcaccgacgagtacaaggtgcccagcaagaaattcaagggctgggcacac1020cgaccggcacagcatcaagaagaacctgatcggagccctgctgttcgacagcggcgaaac1080agccgaggccacccggctgaagagaaccgccagaagaagatacaccagacggaagaccgg1140atcgctatctgcaagagatcttcagcaacgagatggccaaggtggacgacagcttcttcc1200acagactggaagagtccttcctggtggaagaggataagaagcacgagcggcaccccatct1260tggcaacatctggacgaggtggcctaccacgagaagtaccccaccatctaccacctgaga1320aagaaactggtggacagcaccgacaaggccgacctgcggctgatctatctggccctggcc1380cacatgacaagttccgggccacttcctgatcgagggcgacctgaaccccgacaacagcga1440cgtggacaagctgttcatccagctggtgcagacctacaaccagctgttcgaggaaaaccc1500catcaacgccagcgcgtggacgccaggccatcctgtctgccagactgagcaagagcagac1560ggctggaaaatctgatcgcccagctgcccggcgagaagaagaatggcctgttcggaaacc1620tgattgccctgagcctgggctgacccccaactcaagagcaacttcgacctggccgaggat1680gccaaactgcagctgagcaaggacacctacgacgacgacctggacaacctgctggcccag1740atcggcgaccagtacgccgacctgttctggccgccaagacctgtccgacgccatcctgct1800gagcgacatcctgagagtgaacaccgagatcaccaaggcccccctgagcgcctctatgat1860caagagatacgacgagcaccaccaggacctgccctgctgaaagcttcgtgcggcagcagc1920tgcctgagaagtacaaagagattttcttcgaccagagcaagaacggctacgccggctaca1980ttgacggcggagccagccaggaagagttctacaagttatcaagcccatcctgaaaagatg2040gacggcaccgaggaactgctcgtgaagctgaacagagaggacctgctgcggaagcagcgg2100accttcgacaacggcagcatcccccaccagatccacctgggaggctgcacgccattctgg2160gcggcaggaagatttttacccattcctgaaggacaaccgggaaaagatcgagaagatcct2220gaccttccgcatcccctactacgtgggccctctggccaggggaaacagcgattcgcctgg2280atgaccgaaagagcgaggaaaccatcaccccctggaacttcgaggaagtggtggacaagg2340gcgcttccgcccagagcttcatcgagcggatgaccaacttcgataagaacctgccaacga2400gaaggtgctgcccagcacagcctgctgtacgagtacttcaccgtgtataacgagctgacc2460aaagtgaaatacgtgaccgagggaatgagaaagcccgccttcctgagcggcgagcagaaa2520aggccatcgtggacctgctgttcaagaccaaccggaaagtgaccgtgaagcagctgaaag2580aggactacttcaagaaaatcgagtgcttcgactccgtggaaatctccggcgtggaagatc2640ggttcaacgcctccctgggcacataacgatctgctgaaaattatcaaggacaaggacttc2700ctggacaatgaggaaaacgaggacattctggaagatatcgtgctgaccctgacactgttt2760gaggacagagagatgatcgaggaacggctgaacctatgcccacctgttcgacgacaaagt2820gatgaagcagctgaagcggcggagatacaccggctggggcaggctgagccggaagctgat2880caacggcatccgggacaagcagtccggcaagacaatctgatttcctgaagtccgacggct2940tcgccaacagaaacttcatgcagctgatccacgacgacagcctgacctttaaagaggaca3000tccagaaagcccaggtgtccggccagggcgatagcctgcacgacacttgccaatctggcc3060ggcagccccgccattaagaagggcatcctgcagacagtgaaggtggtggacgagctcgtg3120aaagtgatgggccggcacaagcccgagaacatcgtgatcgaaatggccaagagaccagac3180cacccagaagggacagaagaacagccgcgagagaatgaagcggatcgaagagggcatcaa3240agagctgggcagccagatcctgaaagaacaccccgtggaaaacacccagctgcagacgag3300agctgtacctgtactacctgcagaatgggcgggatatgtacgtggaccaggaactggaca3360tcaaccggctgtccgactacgatgtggaccatatcgtgcctcagagctttctgaaggacg3420atccatcacaacaaggtgctgaccagaagcgacaagaaccggggcaagagcgacaacgtg3480ccctccgaagaggtcgtgaagaagatgaagaactactggcggcagctgctgaacgccaag3540ctgattaccagagaagttcgacaatctgaccaaggccgagagaggcggcctgagcgaact3600ggataaggccggcttcatcaagagacagctggtggaaacccggcagatcacaaagcacgt3660ggcacagatcctgactcccggtgaacactaagtacgacgagaatgacaagctgatccggg3720aagtgaaagtgatcaccctgaagtccaagctggtgtccgatttccggaaggatttccagt3780tttacaaagtgcgcgagataacaactacaccacgcccacgacgcctacctgaacgccgtc3840gtgggaaccgccctgatcaaaaagtaccctaagctggaaagcgagttcgtgtacggcgac3900tacaaggtgtacgacgtgcggaagagatcgccaaggcgagcaggaaatcggcaaggctac3960cgccaagtacttcttctacagcaacatcatgaactttttcaagaccgagattaccctggc4020caacggcgagatccggaagcggcctctgatcagacaaacggcaaaccggggagatcgtgt4080gggataagggccgggattttgccaccgtgcggaaagtgctgagcatgccccaagtgaata4140tcgtgaaaaagaccgaggtgcagacaggcggcttcagaaagagtctatctgcccaagagg4200aacagcgataagctgatcgccagaaagaaggactgggaccctaagaagtacggcggcttc4260gacagccccaccgtggcctattctgtgctggtggtggccaaagggaaaagggcaagccaa4320gaaactgaagagtgtgaaagagctgctggggatcaccatcatggaaagaagcagcttcga4380gaagaatcccatcgactttctggaagccaagggctacaaagaagtgaaaaggacctgatc4440atcagctgcctaagtactccctgttcgagctggaaaacggccggaagagaatgctggcct4500ctgccggcgaactgcagaagggaaacgaactggccctgccctccaaatatgtgaattcct4560gtacctggccgccactatgagaagctgaagggctcccccgaggataatgagcagaaacag4620ctgtttgtggaacagcacaagcactacctggacgagatcatcgagcagatcagcgagttc4680tcaagagagtgatcctgccgacgctaatctggacaaagtgctgtccgcctacaacaagca4740ccgggataagcccatcagagagcaggccgagaatatcatccacctgtttaccctgaccaa4800tctgggacccctgccgccttcaagactttgacaccaccatcgaccggaagaggtacacca4860gcaccaaagaggtgctggacgccaccctgatccaccagagcatcaccggcctgtacgaga4920cacggatcgaccttctcagctgggaggcgacaaaggccggcggccacgaaaaaggccggc4980caggcaaaaaagaaaaaggggagtggcggttctggaatgaatcccagtatgaagcagaaa5040caagaagaaatcaaagagatataaagaatagttctgtccaagaagaactctgaagatgat5100tcagccttctgcatctggatctcttgttggaagagaaaatgagctgtccgcaggcttgtc5160caaaaggaaacatcggaatgaccactaacatctacaacttccagcctggggttattgtcc5220cagaatctagtgaaaataaaaatcttggaggagtcacccaggagtcatttgatcttatga5280ttaaagaaaatccatcctctcagtattggaagaagtggcagaaaaacggagaaggcgctg5340taa5343<210>71<211>1520<212>prt<213>人工序列<220><223>合成多肽<400>71metasptyrlysasphisaspglyasptyrlysasphisaspileasp151015tyrlysaspaspaspasplysmetalaprolyslyslysarglysval202530glyilehisglyvalproalaalaasplyslystyrserileglyleu354045aspileglythrasnservalglytrpalavalilethraspglutyr505560lysvalproserlyslysphelysvalleuglyasnthrasparghis65707580serilelyslysasnleuileglyalaleuleupheaspserglyglu859095thralaglualathrargleulysargthralaargargargtyrthr100105110argarglysasnargilecystyrleuglngluilepheserasnglu115120125metalalysvalaspaspserphephehisargleuglugluserphe130135140leuvalglugluasplyslyshisgluarghisproilepheglyasn145150155160ilevalaspgluvalalatyrhisglulystyrprothriletyrhis165170175leuarglyslysleuvalaspserthrasplysalaaspleuargleu180185190iletyrleualaleualahismetilelyspheargglyhispheleu195200205ilegluglyaspleuasnproaspasnseraspvalasplysleuphe210215220ileglnleuvalglnthrtyrasnglnleupheglugluasnproile225230235240asnalaserglyvalaspalalysalaileleuseralaargleuser245250255lysserargargleugluasnleuilealaglnleuproglyglulys260265270lysasnglyleupheglyasnleuilealaleuserleuglyleuthr275280285proasnphelysserasnpheaspleualagluaspalalysleugln290295300leuserlysaspthrtyraspaspaspleuaspasnleuleualagln305310315320ileglyaspglntyralaaspleupheleualaalalysasnleuser325330335aspalaileleuleuseraspileleuargvalasnthrgluilethr340345350lysalaproleuseralasermetilelysargtyraspgluhishis355360365glnaspleuthrleuleulysalaleuvalargglnglnleuproglu370375380lystyrlysgluilephepheaspglnserlysasnglytyralagly385390395400tyrileaspglyglyalaserglnglugluphetyrlyspheilelys405410415proileleuglulysmetaspglythrglugluleuleuvallysleu420425430asnarggluaspleuleuarglysglnargthrpheaspasnglyser435440445ileprohisglnilehisleuglygluleuhisalaileleuargarg450455460glngluaspphetyrpropheleulysaspasnargglulysileglu465470475480lysileleuthrpheargileprotyrtyrvalglyproleualaarg485490495glyasnserargphealatrpmetthrarglysserglugluthrile500505510thrprotrpasnpheglugluvalvalasplysglyalaseralagln515520525serpheilegluargmetthrasnpheasplysasnleuproasnglu530535540lysvalleuprolyshisserleuleutyrglutyrphethrvaltyr545550555560asngluleuthrlysvallystyrvalthrgluglymetarglyspro565570575alapheleuserglygluglnlyslysalailevalaspleuleuphe580585590lysthrasnarglysvalthrvallysglnleulysgluasptyrphe595600605lyslysileglucyspheaspservalgluileserglyvalgluasp610615620argpheasnalaserleuglythrtyrhisaspleuleulysileile625630635640lysasplysasppheleuaspasnglugluasngluaspileleuglu645650655aspilevalleuthrleuthrleuphegluaspargglumetileglu660665670gluargleulysthrtyralahisleupheaspasplysvalmetlys675680685glnleulysargargargtyrthrglytrpglyargleuserarglys690695700leuileasnglyileargasplysglnserglylysthrileleuasp705710715720pheleulysseraspglyphealaasnargasnphemetglnleuile725730735hisaspaspserleuthrphelysgluaspileglnlysalaglnval740745750serglyglnglyaspserleuhisgluhisilealaasnleualagly755760765serproalailelyslysglyileleuglnthrvallysvalvalasp770775780gluleuvallysvalmetglyarghislysprogluasnilevalile785790795800glumetalaarggluasnglnthrthrglnlysglyglnlysasnser805810815arggluargmetlysargileglugluglyilelysgluleuglyser820825830glnileleulysgluhisprovalgluasnthrglnleuglnasnglu835840845lysleutyrleutyrtyrleuglnasnglyargaspmettyrvalasp850855860glngluleuaspileasnargleuserasptyraspvalasphisile865870875880valproglnserpheleulysaspaspserileaspasnlysvalleu885890895thrargserasplysasnargglylysseraspasnvalproserglu900905910gluvalvallyslysmetlysasntyrtrpargglnleuleuasnala915920925lysleuilethrglnarglyspheaspasnleuthrlysalagluarg930935940glyglyleusergluleuasplysalaglypheilelysargglnleu945950955960valgluthrargglnilethrlyshisvalalaglnileleuaspser965970975argmetasnthrlystyraspgluasnasplysleuilearggluval980985990lysvalilethrleulysserlysleuvalseraspphearglysasp99510001005pheglnphetyrlysvalarggluileasnasntyrhishisala101010151020hisaspalatyrleuasnalavalvalglythralaleuilelys102510301035lystyrprolysleuglusergluphevaltyrglyasptyrlys104010451050valtyraspvalarglysmetilealalyssergluglngluile105510601065glylysalathralalystyrphephetyrserasnilemetasn107010751080phephelysthrgluilethrleualaasnglygluilearglys108510901095argproleuilegluthrasnglygluthrglygluilevaltrp110011051110asplysglyargaspphealathrvalarglysvalleusermet111511201125proglnvalasnilevallyslysthrgluvalglnthrglygly113011351140pheserlysgluserileleuprolysargasnserasplysleu114511501155ilealaarglyslysasptrpaspprolyslystyrglyglyphe116011651170aspserprothrvalalatyrservalleuvalvalalalysval117511801185glulysglylysserlyslysleulysservallysgluleuleu119011951200glyilethrilemetgluargserserpheglulysasnproile120512101215asppheleuglualalysglytyrlysgluvallyslysaspleu122012251230ileilelysleuprolystyrserleuphegluleugluasngly123512401245arglysargmetleualaseralaglygluleuglnlysglyasn125012551260gluleualaleuproserlystyrvalasnpheleutyrleuala126512701275serhistyrglulysleulysglyserprogluaspasnglugln128012851290lysglnleuphevalgluglnhislyshistyrleuaspgluile129513001305ilegluglnileserglupheserlysargvalileleualaasp131013151320alaasnleuasplysvalleuseralatyrasnlyshisargasp132513301335lysproilearggluglnalagluasnileilehisleuphethr134013451350leuthrasnleuglyalaproalaalaphelystyrpheaspthr135513601365thrileasparglysargtyrthrserthrlysgluvalleuasp137013751380alathrleuilehisglnserilethrglyleutyrgluthrarg138513901395ileaspleuserglnleuglyglyasplysargproalaalathr140014051410lyslysalaglyglnalalyslyslyslysglyserglyglyser141514201425glyproserproalaargproalaleuargalaproalaserala143014351440thrserglyserarglysargalaargproproalaalaprogly144514501455argaspglnalaargproproalaargargargleuargleuser146014651470valaspgluvalserserproserthrproglualaproaspile147514801485proalacysproserproglyglnlysilelyslysserthrpro149014951500alaalaglyglnproprohisleuthrseralaglnaspglnasp150515101515thrile1520<210>72<211>1380<212>prt<213>人工序列<220><223>合成多肽<400>72metasptyrlysasphisaspglyasptyrlysasphisaspileasp151015tyrlysaspaspaspasplysmetalaprolyslyslysarglysval202530glyilehisglyvalproalaalaasplyslystyrserileglyleu354045aspileglythrasnservalglytrpalavalilethraspglutyr505560lysvalproserlyslysphelysvalleuglyasnthrasparghis65707580serilelyslysasnleuileglyalaleuleupheaspserglyglu859095thralaglualathrargleulysargthralaargargargtyrthr100105110argarglysasnargilecystyrleuglngluilepheserasnglu115120125metalalysvalaspaspserphephehisargleuglugluserphe130135140leuvalglugluasplyslyshisgluarghisproilepheglyasn145150155160ilevalaspgluvalalatyrhisglulystyrprothriletyrhis165170175leuarglyslysleuvalaspserthrasplysalaaspleuargleu180185190iletyrleualaleualahismetilelyspheargglyhispheleu195200205ilegluglyaspleuasnproaspasnseraspvalasplysleuphe210215220ileglnleuvalglnthrtyrasnglnleupheglugluasnproile225230235240asnalaserglyvalaspalalysalaileleuseralaargleuser245250255lysserargargleugluasnleuilealaglnleuproglyglulys260265270lysasnglyleupheglyasnleuilealaleuserleuglyleuthr275280285proasnphelysserasnpheaspleualagluaspalalysleugln290295300leuserlysaspthrtyraspaspaspleuaspasnleuleualagln305310315320ileglyaspglntyralaaspleupheleualaalalysasnleuser325330335aspalaileleuleuseraspileleuargvalasnthrgluilethr340345350lysalaproleuseralasermetilelysargtyraspgluhishis355360365glnaspleuthrleuleulysalaleuvalargglnglnleuproglu370375380lystyrlysgluilephepheaspglnserlysasnglytyralagly385390395400tyrileaspglyglyalaserglnglugluphetyrlyspheilelys405410415proileleuglulysmetaspglythrglugluleuleuvallysleu420425430asnarggluaspleuleuarglysglnargthrpheaspasnglyser435440445ileprohisglnilehisleuglygluleuhisalaileleuargarg450455460glngluaspphetyrpropheleulysaspasnargglulysileglu465470475480lysileleuthrpheargileprotyrtyrvalglyproleualaarg485490495glyasnserargphealatrpmetthrarglysserglugluthrile500505510thrprotrpasnpheglugluvalvalasplysglyalaseralagln515520525serpheilegluargmetthrasnpheasplysasnleuproasnglu530535540lysvalleuprolyshisserleuleutyrglutyrphethrvaltyr545550555560asngluleuthrlysvallystyrvalthrgluglymetarglyspro565570575alapheleuserglygluglnlyslysalailevalaspleuleuphe580585590lysthrasnarglysvalthrvallysglnleulysgluasptyrphe595600605lyslysileglucyspheaspservalgluileserglyvalgluasp610615620argpheasnalaserleuglythrtyrhisaspleuleulysileile625630635640lysasplysasppheleuaspasnglugluasngluaspileleuglu645650655aspilevalleuthrleuthrleuphegluaspargglumetileglu660665670gluargleulysthrtyralahisleupheaspasplysvalmetlys675680685glnleulysargargargtyrthrglytrpglyargleuserarglys690695700leuileasnglyileargasplysglnserglylysthrileleuasp705710715720pheleulysseraspglyphealaasnargasnphemetglnleuile725730735hisaspaspserleuthrphelysgluaspileglnlysalaglnval740745750serglyglnglyaspserleuhisgluhisilealaasnleualagly755760765serproalailelyslysglyileleuglnthrvallysvalvalasp770775780gluleuvallysvalmetglyarghislysprogluasnilevalile785790795800glumetalaarggluasnglnthrthrglnlysglyglnlysasnser805810815arggluargmetlysargileglugluglyilelysgluleuglyser820825830glnileleulysgluhisprovalgluasnthrglnleuglnasnglu835840845lysleutyrleutyrtyrleuglnasnglyargaspmettyrvalasp850855860glngluleuaspileasnargleuserasptyraspvalasphisile865870875880valproglnserpheleulysaspaspserileaspasnlysvalleu885890895thrargserasplysasnargglylysseraspasnvalproserglu900905910gluvalvallyslysmetlysasntyrtrpargglnleuleuasnala915920925lysleuilethrglnarglyspheaspasnleuthrlysalagluarg930935940glyglyleusergluleuasplysalaglypheilelysargglnleu945950955960valgluthrargglnilethrlyshisvalalaglnileleuaspser965970975argmetasnthrlystyraspgluasnasplysleuilearggluval980985990lysvalilethrleulysserlysleuvalseraspphearglysasp99510001005pheglnphetyrlysvalarggluileasnasntyrhishisala101010151020hisaspalatyrleuasnalavalvalglythralaleuilelys102510301035lystyrprolysleuglusergluphevaltyrglyasptyrlys104010451050valtyraspvalarglysmetilealalyssergluglngluile105510601065glylysalathralalystyrphephetyrserasnilemetasn107010751080phephelysthrgluilethrleualaasnglygluilearglys108510901095argproleuilegluthrasnglygluthrglygluilevaltrp110011051110asplysglyargaspphealathrvalarglysvalleusermet111511201125proglnvalasnilevallyslysthrgluvalglnthrglygly113011351140pheserlysgluserileleuprolysargasnserasplysleu114511501155ilealaarglyslysasptrpaspprolyslystyrglyglyphe116011651170aspserprothrvalalatyrservalleuvalvalalalysval117511801185glulysglylysserlyslysleulysservallysgluleuleu119011951200glyilethrilemetgluargserserpheglulysasnproile120512101215asppheleuglualalysglytyrlysgluvallyslysaspleu122012251230ileilelysleuprolystyrserleuphegluleugluasngly123512401245arglysargmetleualaseralaglygluleuglnlysglyasn125012551260gluleualaleuproserlystyrvalasnpheleutyrleuala126512701275serhistyrglulysleulysglyserprogluaspasnglugln128012851290lysglnleuphevalgluglnhislyshistyrleuaspgluile129513001305ilegluglnileserglupheserlysargvalileleualaasp131013151320alaasnleuasplysvalleuseralatyrasnlyshisargasp132513301335lysproilearggluglnalagluasnileilehisleuphethr134013451350leuthrasnleuglyalaproalaalaphelystyrpheaspthr135513601365thrileasparglysargtyrthrserthrlysglu137013751380<210>73<211>1469<212>prt<213>人工序列<220><223>合成多肽<400>73metasptyrlysasphisaspglyasptyrlysasphisaspileasp151015tyrlysaspaspaspasplysmetalaprolyslyslysarglysval202530glyilehisglyvalproalaalaasplyslystyrserileglyleu354045aspileglythrasnservalglytrpalavalilethraspglutyr505560lysvalproserlyslysphelysvalleuglyasnthrasparghis65707580serilelyslysasnleuileglyalaleuleupheaspserglyglu859095thralaglualathrargleulysargthralaargargargtyrthr100105110argarglysasnargilecystyrleuglngluilepheserasnglu115120125metalalysvalaspaspserphephehisargleuglugluserphe130135140leuvalglugluasplyslyshisgluarghisproilepheglyasn145150155160ilevalaspgluvalalatyrhisglulystyrprothriletyrhis165170175leuarglyslysleuvalaspserthrasplysalaaspleuargleu180185190iletyrleualaleualahismetilelyspheargglyhispheleu195200205ilegluglyaspleuasnproaspasnseraspvalasplysleuphe210215220ileglnleuvalglnthrtyrasnglnleupheglugluasnproile225230235240asnalaserglyvalaspalalysalaileleuseralaargleuser245250255lysserargargleugluasnleuilealaglnleuproglyglulys260265270lysasnglyleupheglyasnleuilealaleuserleuglyleuthr275280285proasnphelysserasnpheaspleualagluaspalalysleugln290295300leuserlysaspthrtyraspaspaspleuaspasnleuleualagln305310315320ileglyaspglntyralaaspleupheleualaalalysasnleuser325330335aspalaileleuleuseraspileleuargvalasnthrgluilethr340345350lysalaproleuseralasermetilelysargtyraspgluhishis355360365glnaspleuthrleuleulysalaleuvalargglnglnleuproglu370375380lystyrlysgluilephepheaspglnserlysasnglytyralagly385390395400tyrileaspglyglyalaserglnglugluphetyrlyspheilelys405410415proileleuglulysmetaspglythrglugluleuleuvallysleu420425430asnarggluaspleuleuarglysglnargthrpheaspasnglyser435440445ileprohisglnilehisleuglygluleuhisalaileleuargarg450455460glngluaspphetyrpropheleulysaspasnargglulysileglu465470475480lysileleuthrpheargileprotyrtyrvalglyproleualaarg485490495glyasnserargphealatrpmetthrarglysserglugluthrile500505510thrprotrpasnpheglugluvalvalasplysglyalaseralagln515520525serpheilegluargmetthrasnpheasplysasnleuproasnglu530535540lysvalleuprolyshisserleuleutyrglutyrphethrvaltyr545550555560asngluleuthrlysvallystyrvalthrgluglymetarglyspro565570575alapheleuserglygluglnlyslysalailevalaspleuleuphe580585590lysthrasnarglysvalthrvallysglnleulysgluasptyrphe595600605lyslysileglucyspheaspservalgluileserglyvalgluasp610615620argpheasnalaserleuglythrtyrhisaspleuleulysileile625630635640lysasplysasppheleuaspasnglugluasngluaspileleuglu645650655aspilevalleuthrleuthrleuphegluaspargglumetileglu660665670gluargleulysthrtyralahisleupheaspasplysvalmetlys675680685glnleulysargargargtyrthrglytrpglyargleuserarglys690695700leuileasnglyileargasplysglnserglylysthrileleuasp705710715720pheleulysseraspglyphealaasnargasnphemetglnleuile725730735hisaspaspserleuthrphelysgluaspileglnlysalaglnval740745750serglyglnglyaspserleuhisgluhisilealaasnleualagly755760765serproalailelyslysglyileleuglnthrvallysvalvalasp770775780gluleuvallysvalmetglyarghislysprogluasnilevalile785790795800glumetalaarggluasnglnthrthrglnlysglyglnlysasnser805810815arggluargmetlysargileglugluglyilelysgluleuglyser820825830glnileleulysgluhisprovalgluasnthrglnleuglnasnglu835840845lysleutyrleutyrtyrleuglnasnglyargaspmettyrvalasp850855860glngluleuaspileasnargleuserasptyraspvalasphisile865870875880valproglnserpheleulysaspaspserileaspasnlysvalleu885890895thrargserasplysasnargglylysseraspasnvalproserglu900905910gluvalvallyslysmetlysasntyrtrpargglnleuleuasnala915920925lysleuilethrglnarglyspheaspasnleuthrlysalagluarg930935940glyglyleusergluleuasplysalaglypheilelysargglnleu945950955960valgluthrargglnilethrlyshisvalalaglnileleuaspser965970975argmetasnthrlystyraspgluasnasplysleuilearggluval980985990lysvalilethrleulysserlysleuvalseraspphearglysasp99510001005pheglnphetyrlysvalarggluileasnasntyrhishisala101010151020hisaspalatyrleuasnalavalvalglythralaleuilelys102510301035lystyrprolysleuglusergluphevaltyrglyasptyrlys104010451050valtyraspvalarglysmetilealalyssergluglngluile105510601065glylysalathralalystyrphephetyrserasnilemetasn107010751080phephelysthrgluilethrleualaasnglygluilearglys108510901095argproleuilegluthrasnglygluthrglygluilevaltrp110011051110asplysglyargaspphealathrvalarglysvalleusermet111511201125proglnvalasnilevallyslysthrgluvalglnthrglygly113011351140pheserlysgluserileleuprolysargasnserasplysleu114511501155ilealaarglyslysasptrpaspprolyslystyrglyglyphe116011651170aspserprothrvalalatyrservalleuvalvalalalysval117511801185glulysglylysserlyslysleulysservallysgluleuleu119011951200glyilethrilemetgluargserserpheglulysasnproile120512101215asppheleuglualalysglytyrlysgluvallyslysaspleu122012251230ileilelysleuprolystyrserleuphegluleugluasngly123512401245arglysargmetleualaseralaglygluleuglnlysglyasn125012551260gluleualaleuproserlystyrvalasnpheleutyrleuala126512701275serhistyrglulysleulysglyserprogluaspasnglugln128012851290lysglnleuphevalgluglnhislyshistyrleuaspgluile129513001305ilegluglnileserglupheserlysargvalileleualaasp131013151320alaasnleuasplysvalleuseralatyrasnlyshisargasp132513301335lysproilearggluglnalagluasnileilehisleuphethr134013451350leuthrasnleuglyalaproalaalaphelystyrpheaspthr135513601365thrileasparglysargtyrthrserthrlysgluvalleuasp137013751380alathrleuilehisglnserilethrglyleutyrgluthrarg138513901395ileaspleuserglnleuglyglyasplysargproalaalathr140014051410lyslysalaglyglnalalyslyslyslysglyserglyglyser141514201425glyserhisglypheproprogluvalglugluglnaspaspgly143014351440thrleuprometsercysalaglngluserglymetasparghis144514501455proalaalacysalaseralaargileasnval14601465<210>74<211>1471<212>prt<213>人工序列<220><223>`合成多肽<400>74metasptyrlysasphisaspglyasptyrlysasphisaspileasp151015tyrlysaspaspaspasplysmetalaprolyslyslysarglysval202530glyilehisglyvalproalaalaasplyslystyrserileglyleu354045aspileglythrasnservalglytrpalavalilethraspglutyr505560lysvalproserlyslysphelysvalleuglyasnthrasparghis65707580serilelyslysasnleuileglyalaleuleupheaspserglyglu859095thralaglualathrargleulysargthralaargargargtyrthr100105110argarglysasnargilecystyrleuglngluilepheserasnglu115120125metalalysvalaspaspserphephehisargleuglugluserphe130135140leuvalglugluasplyslyshisgluarghisproilepheglyasn145150155160ilevalaspgluvalalatyrhisglulystyrprothriletyrhis165170175leuarglyslysleuvalaspserthrasplysalaaspleuargleu180185190iletyrleualaleualahismetilelyspheargglyhispheleu195200205ilegluglyaspleuasnproaspasnseraspvalasplysleuphe210215220ileglnleuvalglnthrtyrasnglnleupheglugluasnproile225230235240asnalaserglyvalaspalalysalaileleuseralaargleuser245250255lysserargargleugluasnleuilealaglnleuproglyglulys260265270lysasnglyleupheglyasnleuilealaleuserleuglyleuthr275280285proasnphelysserasnpheaspleualagluaspalalysleugln290295300leuserlysaspthrtyraspaspaspleuaspasnleuleualagln305310315320ileglyaspglntyralaaspleupheleualaalalysasnleuser325330335aspalaileleuleuseraspileleuargvalasnthrgluilethr340345350lysalaproleuseralasermetilelysargtyraspgluhishis355360365glnaspleuthrleuleulysalaleuvalargglnglnleuproglu370375380lystyrlysgluilephepheaspglnserlysasnglytyralagly385390395400tyrileaspglyglyalaserglnglugluphetyrlyspheilelys405410415proileleuglulysmetaspglythrglugluleuleuvallysleu420425430asnarggluaspleuleuarglysglnargthrpheaspasnglyser435440445ileprohisglnilehisleuglygluleuhisalaileleuargarg450455460glngluaspphetyrpropheleulysaspasnargglulysileglu465470475480lysileleuthrpheargileprotyrtyrvalglyproleualaarg485490495glyasnserargphealatrpmetthrarglysserglugluthrile500505510thrprotrpasnpheglugluvalvalasplysglyalaseralagln515520525serpheilegluargmetthrasnpheasplysasnleuproasnglu530535540lysvalleuprolyshisserleuleutyrglutyrphethrvaltyr545550555560asngluleuthrlysvallystyrvalthrgluglymetarglyspro565570575alapheleuserglygluglnlyslysalailevalaspleuleuphe580585590lysthrasnarglysvalthrvallysglnleulysgluasptyrphe595600605lyslysileglucyspheaspservalgluileserglyvalgluasp610615620argpheasnalaserleuglythrtyrhisaspleuleulysileile625630635640lysasplysasppheleuaspasnglugluasngluaspileleuglu645650655aspilevalleuthrleuthrleuphegluaspargglumetileglu660665670gluargleulysthrtyralahisleupheaspasplysvalmetlys675680685glnleulysargargargtyrthrglytrpglyargleuserarglys690695700leuileasnglyileargasplysglnserglylysthrileleuasp705710715720pheleulysseraspglyphealaasnargasnphemetglnleuile725730735hisaspaspserleuthrphelysgluaspileglnlysalaglnval740745750serglyglnglyaspserleuhisgluhisilealaasnleualagly755760765serproalailelyslysglyileleuglnthrvallysvalvalasp770775780gluleuvallysvalmetglyarghislysprogluasnilevalile785790795800glumetalaarggluasnglnthrthrglnlysglyglnlysasnser805810815arggluargmetlysargileglugluglyilelysgluleuglyser820825830glnileleulysgluhisprovalgluasnthrglnleuglnasnglu835840845lysleutyrleutyrtyrleuglnasnglyargaspmettyrvalasp850855860glngluleuaspileasnargleuserasptyraspvalasphisile865870875880valproglnserpheleulysaspaspserileaspasnlysvalleu885890895thrargserasplysasnargglylysseraspasnvalproserglu900905910gluvalvallyslysmetlysasntyrtrpargglnleuleuasnala915920925lysleuilethrglnarglyspheaspasnleuthrlysalagluarg930935940glyglyleusergluleuasplysalaglypheilelysargglnleu945950955960valgluthrargglnilethrlyshisvalalaglnileleuaspser965970975argmetasnthrlystyraspgluasnasplysleuilearggluval980985990lysvalilethrleulysserlysleuvalseraspphearglysasp99510001005pheglnphetyrlysvalarggluileasnasntyrhishisala101010151020hisaspalatyrleuasnalavalvalglythralaleuilelys102510301035lystyrprolysleuglusergluphevaltyrglyasptyrlys104010451050valtyraspvalarglysmetilealalyssergluglngluile105510601065glylysalathralalystyrphephetyrserasnilemetasn107010751080phephelysthrgluilethrleualaasnglygluilearglys108510901095argproleuilegluthrasnglygluthrglygluilevaltrp110011051110asplysglyargaspphealathrvalarglysvalleusermet111511201125proglnvalasnilevallyslysthrgluvalglnthrglygly113011351140pheserlysgluserileleuprolysargasnserasplysleu114511501155ilealaarglyslysasptrpaspprolyslystyrglyglyphe116011651170aspserprothrvalalatyrservalleuvalvalalalysval117511801185glulysglylysserlyslysleulysservallysgluleuleu119011951200glyilethrilemetgluargserserpheglulysasnproile120512101215asppheleuglualalysglytyrlysgluvallyslysaspleu122012251230ileilelysleuprolystyrserleuphegluleugluasngly123512401245arglysargmetleualaseralaglygluleuglnlysglyasn125012551260gluleualaleuproserlystyrvalasnpheleutyrleuala126512701275serhistyrglulysleulysglyserprogluaspasnglugln128012851290lysglnleuphevalgluglnhislyshistyrleuaspgluile129513001305ilegluglnileserglupheserlysargvalileleualaasp131013151320alaasnleuasplysvalleuseralatyrasnlyshisargasp132513301335lysproilearggluglnalagluasnileilehisleuphethr134013451350leuthrasnleuglyalaproalaalaphelystyrpheaspthr135513601365thrileasparglysargtyrthrserthrlysgluvalleuasp137013751380alathrleuilehisglnserilethrglyleutyrgluthrarg138513901395ileaspleuserglnleuglyglyasplysargproalaalathr140014051410lyslysalaglyglnalalyslyslyslysglyserglyglyser141514201425glylysleuserhisglypheproprogluvalglugluglnasp143014351440aspglythrleuprometsercysalaglngluserglymetasp144514501455arghisproalaalacysalaseralaargileasnval146014651470<210>75<211>1624<212>prt<213>人工序列<220><223>合成多肽<400>75metasptyrlysasphisaspglyasptyrlysasphisaspileasp151015tyrlysaspaspaspasplysmetalaprolyslyslysarglysval202530glyilehisglyvalproalaalaasplyslystyrserileglyleu354045aspileglythrasnservalglytrpalavalilethraspglutyr505560lysvalproserlyslysphelysvalleuglyasnthrasparghis65707580serilelyslysasnleuileglyalaleuleupheaspserglyglu859095thralaglualathrargleulysargthralaargargargtyrthr100105110argarglysasnargilecystyrleuglngluilepheserasnglu115120125metalalysvalaspaspserphephehisargleuglugluserphe130135140leuvalglugluasplyslyshisgluarghisproilepheglyasn145150155160ilevalaspgluvalalatyrhisglulystyrprothriletyrhis165170175leuarglyslysleuvalaspserthrasplysalaaspleuargleu180185190iletyrleualaleualahismetilelyspheargglyhispheleu195200205ilegluglyaspleuasnproaspasnseraspvalasplysleuphe210215220ileglnleuvalglnthrtyrasnglnleupheglugluasnproile225230235240asnalaserglyvalaspalalysalaileleuseralaargleuser245250255lysserargargleugluasnleuilealaglnleuproglyglulys260265270lysasnglyleupheglyasnleuilealaleuserleuglyleuthr275280285proasnphelysserasnpheaspleualagluaspalalysleugln290295300leuserlysaspthrtyraspaspaspleuaspasnleuleualagln305310315320ileglyaspglntyralaaspleupheleualaalalysasnleuser325330335aspalaileleuleuseraspileleuargvalasnthrgluilethr340345350lysalaproleuseralasermetilelysargtyraspgluhishis355360365glnaspleuthrleuleulysalaleuvalargglnglnleuproglu370375380lystyrlysgluilephepheaspglnserlysasnglytyralagly385390395400tyrileaspglyglyalaserglnglugluphetyrlyspheilelys405410415proileleuglulysmetaspglythrglugluleuleuvallysleu420425430asnarggluaspleuleuarglysglnargthrpheaspasnglyser435440445ileprohisglnilehisleuglygluleuhisalaileleuargarg450455460glngluaspphetyrpropheleulysaspasnargglulysileglu465470475480lysileleuthrpheargileprotyrtyrvalglyproleualaarg485490495glyasnserargphealatrpmetthrarglysserglugluthrile500505510thrprotrpasnpheglugluvalvalasplysglyalaseralagln515520525serpheilegluargmetthrasnpheasplysasnleuproasnglu530535540lysvalleuprolyshisserleuleutyrglutyrphethrvaltyr545550555560asngluleuthrlysvallystyrvalthrgluglymetarglyspro565570575alapheleuserglygluglnlyslysalailevalaspleuleuphe580585590lysthrasnarglysvalthrvallysglnleulysgluasptyrphe595600605lyslysileglucyspheaspservalgluileserglyvalgluasp610615620argpheasnalaserleuglythrtyrhisaspleuleulysileile625630635640lysasplysasppheleuaspasnglugluasngluaspileleuglu645650655aspilevalleuthrleuthrleuphegluaspargglumetileglu660665670gluargleulysthrtyralahisleupheaspasplysvalmetlys675680685glnleulysargargargtyrthrglytrpglyargleuserarglys690695700leuileasnglyileargasplysglnserglylysthrileleuasp705710715720pheleulysseraspglyphealaasnargasnphemetglnleuile725730735hisaspaspserleuthrphelysgluaspileglnlysalaglnval740745750serglyglnglyaspserleuhisgluhisilealaasnleualagly755760765serproalailelyslysglyileleuglnthrvallysvalvalasp770775780gluleuvallysvalmetglyarghislysprogluasnilevalile785790795800glumetalaarggluasnglnthrthrglnlysglyglnlysasnser805810815arggluargmetlysargileglugluglyilelysgluleuglyser820825830glnileleulysgluhisprovalgluasnthrglnleuglnasnglu835840845lysleutyrleutyrtyrleuglnasnglyargaspmettyrvalasp850855860glngluleuaspileasnargleuserasptyraspvalasphisile865870875880valproglnserpheleulysaspaspserileaspasnlysvalleu885890895thrargserasplysasnargglylysseraspasnvalproserglu900905910gluvalvallyslysmetlysasntyrtrpargglnleuleuasnala915920925lysleuilethrglnarglyspheaspasnleuthrlysalagluarg930935940glyglyleusergluleuasplysalaglypheilelysargglnleu945950955960valgluthrargglnilethrlyshisvalalaglnileleuaspser965970975argmetasnthrlystyraspgluasnasplysleuilearggluval980985990lysvalilethrleulysserlysleuvalseraspphearglysasp99510001005pheglnphetyrlysvalarggluileasnasntyrhishisala101010151020hisaspalatyrleuasnalavalvalglythralaleuilelys102510301035lystyrprolysleuglusergluphevaltyrglyasptyrlys104010451050valtyraspvalarglysmetilealalyssergluglngluile105510601065glylysalathralalystyrphephetyrserasnilemetasn107010751080phephelysthrgluilethrleualaasnglygluilearglys108510901095argproleuilegluthrasnglygluthrglygluilevaltrp110011051110asplysglyargaspphealathrvalarglysvalleusermet111511201125proglnvalasnilevallyslysthrgluvalglnthrglygly113011351140pheserlysgluserileleuprolysargasnserasplysleu114511501155ilealaarglyslysasptrpaspprolyslystyrglyglyphe116011651170aspserprothrvalalatyrservalleuvalvalalalysval117511801185glulysglylysserlyslysleulysservallysgluleuleu119011951200glyilethrilemetgluargserserpheglulysasnproile120512101215asppheleuglualalysglytyrlysgluvallyslysaspleu122012251230ileilelysleuprolystyrserleuphegluleugluasngly123512401245arglysargmetleualaseralaglygluleuglnlysglyasn125012551260gluleualaleuproserlystyrvalasnpheleutyrleuala126512701275serhistyrglulysleulysglyserprogluaspasnglugln128012851290lysglnleuphevalgluglnhislyshistyrleuaspgluile129513001305ilegluglnileserglupheserlysargvalileleualaasp131013151320alaasnleuasplysvalleuseralatyrasnlyshisargasp132513301335lysproilearggluglnalagluasnileilehisleuphethr134013451350leuthrasnleuglyalaproalaalaphelystyrpheaspthr135513601365thrileasparglysargtyrthrserthrlysgluvalleuasp137013751380alathrleuilehisglnserilethrglyleutyrgluthrarg138513901395ileaspleuserglnleuglyglyasplysargproalaalathr140014051410lyslysalaglyglnalalyslyslyslyslysleuserhisgly141514201425pheproprogluvalglugluglnaspaspglythrleupromet143014351440sercysalaglngluserglymetasparghisproalaalacys144514501455alaseralaargileasnvalvalleuaspalathrleuilehis146014651470glnserilethrglyleutyrgluthrargileaspleusergln147514801485leuglyglyasplysargproalaalathrlyslysalaglygln149014951500alalyslyslyslysglyserglyglyserglymetasnproser150515101515metlysglnlysglnglugluilelysgluasnilelysasnser152015251530servalproargargthrleulysmetileglnproseralaser153515401545glyserleuvalglyarggluasngluleuseralaglyleuser155015551560lysarglyshisargasnasphisleuthrserthrthrserser156515701575proglyvalilevalproglusersergluasnlysasnleugly158015851590glyvalthrglngluserpheaspleumetilelysgluasnpro159516001605serserglntyrtrplysgluvalalaglulysargarglysala161016151620leu<210>76<211>91<212>prt<213>人工序列<220><223>合成多肽<400>76proserproalaargproalaleuargalaproalaseralathrser151015glyserarglysargalaargproproalaalaproglyargaspgln202530alaargproproalaargargargleuargleuservalaspgluval354045serserproserthrproglualaproaspileproalacysproser505560proglyglnlysilelyslysserthrproalaalaglyglnpropro65707580hisleuthrseralaglnaspglnaspthrile8590<210>77<211>110<212>prt<213>人工序列<220><223>合成多肽<400>77metasnprosermetlysglnlysglnglugluilelysgluasnile151015lysasnserservalproargargthrleulysmetileglnproser202530alaserglyserleuvalglyarggluasngluleuseralaglyleu354045serlysarglyshisargasnasphisleuthrserthrthrserser505560proglyvalilevalproglusersergluasnlysasnleuglygly65707580valthrglngluserpheaspleumetilelysgluasnproserser859095glntyrtrplysgluvalalaglulysargarglysalaleu100105110<210>78<211>40<212>prt<213>人工序列<220><223>合成肽<400>78serhisglypheproprogluvalglugluglnaspaspglythrleu151015prometsercysalaglngluserglymetasparghisproalaala202530cysalaseralaargileasnval3540<210>79<211>42<212>prt<213>人工序列<220><223>合成肽<400>79lysleuserhisglypheproprogluvalglugluglnaspaspgly151015thrleuprometsercysalaglngluserglymetasparghispro202530alaalacysalaseralaargileasnval3540当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1