一种耐低温几丁质酶的耐酸突变体及其应用的制作方法
本发明属于基因筛选改造技术领域,具体涉及一种几丁质酶酶(chitinase)及其应用,具体涉及一种通过基因突变的方法筛选获得几丁质酶的突变体以及几丁质酶耐酸突变体的在几丁质降解中的应用。
背景技术:
几丁质又称甲壳素、甲壳质,它广泛存在于甲壳纲动物、软体动物和节肢动物外壳中,如虾、螃蟹、蝗虫等,此外在藻类、贝类、真菌类的细胞壁及高等植物中也广泛存在,是目前自然界中仅次于纤维素的第二大生物资源,但比纤维素具有更多的特殊功能。几丁质是一种白色或灰白色、半透明片状晶体,无味,为天然粘多糖。化学名称为1,4-2-乙酰氨基-2-脱氧-β-d葡聚糖,基本单位是乙酰氨基葡萄糖(glcnac)为一种直链状聚合物化学结构如下,根据分子链的排列方式,它存在α,β,γ三种晶型物。由于该多聚糖大分子间有较强的氢键作用,所以其理化性质非常稳定。含量最大的α-几丁质,由两条反向平行链组成,多氢键相连,浸水不胀;β-几丁质,由两条同向平行链构成,分子链间的氢键较小几丁质少的多,遇水会膨胀;γ-几丁质,为三条链,两条同向,一条反向,类似于α-和β-几丁质的混和晶体。
来自海洋生物圈的几丁质占几丁质总量的绝大部分,然而几丁质在海底沉积却很少,这主要与海洋微生物对几丁质的有效分解有关。海洋微生物通过分泌几丁质酶分解几丁质作为自身生长繁殖的碳源和氮源,推动了几丁质的再循环。几丁质是海洋环境中一种丰富而且重要的营养和能量物质,海洋微生物通过几丁质酶对几丁质进行降解和利用是海洋环境中营养物质循环的关键,对维持海洋环境生态系统的平衡也很重要。
最近几丁质引起了研究者的极大兴趣,将它们转化成低聚糖,低聚糖不但溶解于水,而且拥有多种生物功能,如抗肿瘤活性和抗真菌活性。目前,几丁质低聚糖在工业上是通过化学法制备的,这存在很多问题,如有很多短链低聚糖产生,低聚糖产量低,分离成本高,而且易对环境产生污染。相对的,酶解几丁质有许多优点,环境兼容性,成本低,可重复。近年来,几丁质的酶法水解变得越来越受欢迎。
与陆地几丁质酶相比,海洋几丁质酶有更高的ph和盐度忍受力,有助于特别的应用。stefanidi等从生活在南太平洋1200m处的样品中分离到的嗜冷细菌能够分泌几种几丁质酶降解几丁质。几丁质酶还发现于alteromonassp.straino-7、vibrioanguillarum、v.parahaemolyticus。
酸酶主要有以下四个生理生化特点:(1)在酸下的具有高周转率(kcat)和高催化效率(kcat/km)是酸酶的主要适应特性。它们可以弥补酸导致化学反应速率降低带来的影响,为生物提够足够的代谢活力并维持足够的代谢通量。高催化效率(包括km值的下降)对酸酶尤为重要,因为她可以保证酸下底物与酶活性位点的充分结合。(2)最适作用温度低或在酸下有高催化活性是酶归类为酸酶的先决条件。酸酶的最适反应温度一般低于40℃,与普通的中温酶相比,低了10℃到20℃。目前已报道的酸几丁质酶的最适作用温度最低在30℃。(3)结构柔顺性是酸酶的另一个特点。酸酶柔顺、松散和开放的结构特征,有助于提高酶分子在催化过程中的构象变化能力。当温度下降,随着蛋白结构柔顺性的降低以及冷变性作用,酶的催化速率随之下降。而酸酶的高柔顺性,一方面为底物到达酶的活性位点提供更佳的通道和容纳位置,提高酶活性位点与底物之间的结合互补性,并有利于底物的释放:另一方面可降低活化能以补偿反应分子的低动能,减少容纳庞大底物所耗费的能量,在低能耗情况下保证酶的高催化效率(4)酸酶的热稳定性差。酸酶结构的高度柔顺性可能造成蛋白的离子配位松散,并且由于缺乏二硫键及刚性的二极结构,导致酸酶对热变性和化学变性敏感,稳定性降低。此外,酸酶所处的自然环境缺乏热选择性压力,可能也是造成蛋白结构热不稳定性的原因。
与中高温环境相比,酸环境下,水的粘度明显增大,热运动显著降低。在这样的环境下,由于热运动的降低,酶与底物的结合与分离,酶分子构象的改变都比中高温下困难,温度会显著影响酶与底物的结合。酶分子要在这样的环境下有较高的催化率,必须使分子结构变的更加柔顺(flexible),从而增加构象改变的速度。与中温酶相比,酸酶的共同特征是:盐桥的数量减少,疏水核内芳香环的相互作用减弱,脯氨酸与精氨酸残基数量减少,酶的疏水性减弱,使酶与溶剂的相互作用增强。这些结构的改变都有利于酶分子柔顺性的增加。
由于酸几丁质酶具有反应温度低,对热敏感等特点,所以在产业化利用几丁质酶降解几丁质生产几丁寡糖过程中有着中温几丁质酶无法比拟的优越性,应用酸几丁质酶可以省却加热与冷却两步,既节省能量又节省时间。同时,在利用几丁质酶处理自然界废物与污水处理时,由于酸几丁质酶有着接近自然环境温度的最适反应温度,因此应用的效果更好。这对环境保护有着重要意义。
目前,已报道的酸几丁质酶对酸相对敏感。为解决上述问题,本发明将从现有技术公开的已知几丁质酶基础上,通过随机突变并筛选的方法,在耐低温的几丁质酶基础上获得耐酸的几丁质酶。
技术实现要素:
本发明人发现了现有技术公开的耐酸的几丁质酶,其筛选自假交替单胞菌,最适作用温度为35℃,在10℃还能保持25%的活性,属于耐酸的酶,但是其最适ph值为中性的7,对酸性环境敏感,因此,发明人对该酶进行了突变体的筛选,以期获得具有耐酸性的环境的同时耐酸的几丁质酶。
本发明的目的是提供一种几丁质酶突变体其应用。本发明通过大量的筛选,最终获得一种耐酸更强几丁质酶突变体,并构建得到了重组表达几丁质酶的基因工程菌株,为实现其应用奠定了基础。
本发明第一方面涉及一种几丁质酶耐酸突变体,是氨基酸序列为与seqidno:1所示的氨基酸序列对比,其具有如下的任一个或多个的突变,所述的突变选自:第861位的ile突变为ser,第875位的ala突变为cys,第894位的val突变为pro,第903位的gla突变为arg,第914位的his突变为lys,第927位的ala突变为asn,第942位的met突变为trp。
本发明第二方面涉及一种几丁质酶耐酸突变体,是氨基酸序列为seqidno:3所示。
本发明的第三方面,提供编码上述几丁质酶耐酸突变体编码基因的核酸序列;在一个具体的实施例中,所述的核苷酸序列为seqidno:4所示。
本发明的第四方面,本发明提供了一种载体,其含有本发明第二个方面所述的多核苷酸。本文中的载体包括表达载体,例如,细菌质粒、粘粒、噬菌粒、酵母质粒、植物细胞病毒、动物病毒及其它各种病毒载体。本发明中适用的载体包括但不限于:在细菌中表达用的载体(原核表达载体)、在酵母中表达用的载体(如毕赤酵母载体、汉逊酵母载体等)、在昆虫细胞中表达的杆状病毒载体、在哺乳动物细胞中表达用的载体(痘苗病毒载体、逆转录病毒载体、腺病毒载体、腺伴病毒载体等)、在植物中表达用的植物病毒载体以及在哺乳动物乳腺中表达用的各种载体。总之,只要能在宿主细胞中稳定复制,任何质粒和载体都可使用。优选表达载体包含选择标记基因,如细菌的氨苄青霉素抗性基因、四环素抗性基因、卡那霉素抗性基因、链霉素抗性基因、氯霉素抗性基因;酵母菌的新霉素抗性基因、zeocin抗性基因,酵母菌的缺陷选择标志,如his,leu,trp等;真核细胞的新霉素抗性基因、zeocin抗性基因、二氢叶酸还原酶基因及荧光蛋白标记基因等。本发明的载体优选为原核载体,更优选是大肠杆菌表达载体;更优选为pet系列质粒;更优选为pet-28a(+)质粒。
本发明第五方面,本发明提供了细胞,其含有本发明第三个方面所述的多核苷酸。该细胞可以用本发明第四个方面所述的载体转化或转染而获得。细胞可以是原核细胞,也可以是真核细胞,如,细菌细胞、酵母细胞、植物细胞、昆虫细胞、哺乳动物细胞等。细胞在转化或转染含本发明所述编码融合蛋白的基因序列后,即构成工程化细胞或细胞株,可用于生产所需融合蛋白。合适的转化或转染方法包括但不限于:用于细菌细胞,如氯化钙法、电穿孔法;用于酵母细胞,如电穿孔法和原生质体融合法;用于哺乳动物细胞等,如质体包裹、磷酸钙共沉淀、电融合法以及显微注射法。优选本发明的细胞是大肠杆菌细胞。本发明还涉及一种工程菌株,其携带有上述重组质粒。所述工程菌株为细菌,优选为革兰氏阴性菌,优选为大肠杆菌,在一个具体的实施例中,所述的宿主为e.colibl21。
本发明还涉及一种含有几丁质酶高耐酸突变体的组合物,其中所述的几丁质酶耐酸突变体是由上述含有所述表达质粒的基因工程菌发酵制备得到的。
本发明还涉及上述的几丁质酶耐酸突变体、编码基因、表达载体、重组细胞在水解几丁质中的应用。
本发明的有益效果是通过基因工程手段,构建大样本的突变文库,然后从中筛选获得了大量筛选获得了一种几丁质酶耐酸突变体,该酶相对于野生型酶在各自最适宜的条件下绝对酶活有一定的提升,另外,所述突变体的最适作用ph为5.5,最适温度与野生型类似,仍然为35℃,且在ph值在4.5时仍然能保持50%以上的酶活,在ph3.5时仍然保持近30%的活性,具有耐酸特性;因此更加有利于其在几丁质降解过程中的应用。
附图说明
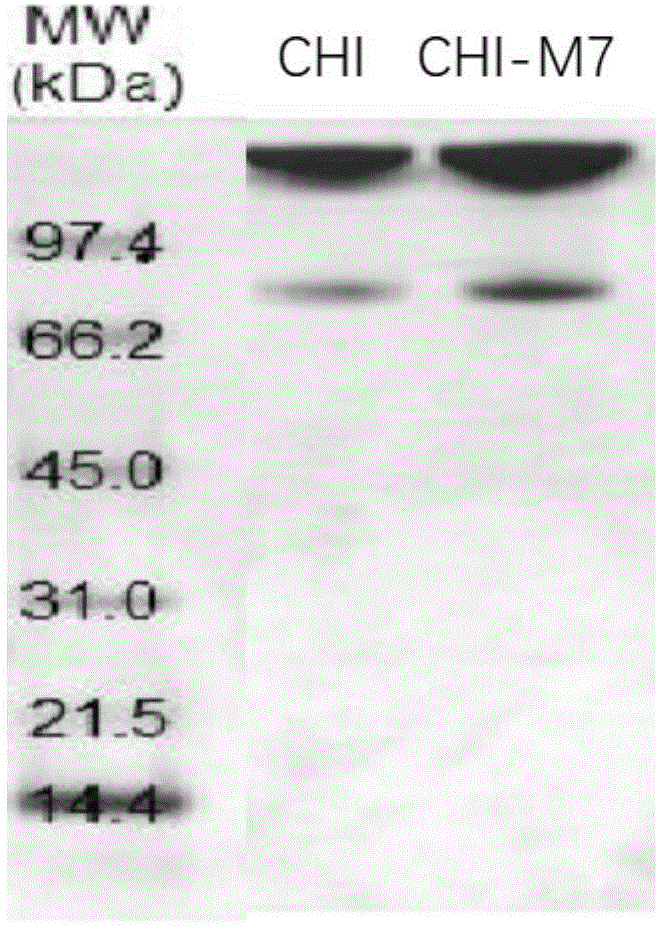
图1:sds-page电泳检测图,其中泳道chi为野生型蛋白,chi-m7为突变型蛋白,分子量大小为127kd。
图2:几丁质酶相对酶活-作用ph值曲线图,对照为野生型几丁质酶。
具体实施方式
下面结合实例对本发明的方法做进一步说明。在实施例中未注明具体条件的实验方法,通常可按分子生物学领域的常规实验,冷泉港实验室等编写的《分子克隆实验指南》中所述的条件,或按照质粒、菌株等商品化生产厂商的说明书进行操作。本领域相关的技术人员可以借助实施例更好地理解和掌握本发明。
突变pcr为本领域技术人员所熟悉的易错pcr。
实施例1野生型几丁质酶的基因克隆
本发明人发现了现有技术公开的耐酸的几丁质酶,其筛选自假交替单胞菌(pseudoalteromonassp.),最适作用温度为35℃,在10℃还能保持25%的活性,属于耐酸的酶,其氨基酸序列如seqidno:1所示的几丁质酶,其编码核苷酸如seqidno:2所示,在大肠杆菌中表达制备。简而言之,委托上海捷瑞生物技术有限公司人工合成该基因,然后连接入pet-28(+)载体(newenglandbiolabs公司)的ecori和hindiii的酶切位点之间,转化入大肠杆菌dh5α,于含氨苄青霉素50mg/l的lb平板上筛选,筛选阳性的菌中包含的表达载体被命名为pet-chi,抽提质粒,测序鉴定正确。
实施例2突变筛选
申请人为了进一步提高上述几丁质酶(氨基酸序列为seqidno:1)的耐酸性,以实施例1中构建得到的重组质粒pet-chi为模板,分别设计随机突变引物,进行易错pcr,对目的基因进行多点随机突变,结果发现有些突变对其耐酸性没有影响,有些突变甚至使酸稳定性更差了,另外还有些突变,虽然能提高耐酸性,但突变后其耐低温的性能有所降低,均不符合要求。最终,申请人获得了既能显著提高耐酸性,又能维持与野生型耐酸活性相当的酶,具体突变组合为:i861s、a875c、v894p、g903r、h914k、a927n、m942w;具体为第861位的ile突变为ser,第875位的ala突变为cys,第894位的val突变为pro,第903位的gla突变为arg,第914位的his突变为lys,第927位的ala突变为asn,第942位的met突变为trp。
将上述七点突变体命名为chi-mut7,其氨基酸序列为seqidno:3,参照该序列合成一个编码核苷酸序列seqidno:4。该序列是依照大肠杆菌的密码偏爱性而优化合成,并且在合成序列5’和3’两端分别加上bamhi和hindiii两个酶切位点,由上海生工生物工程股份有限公司完成。
采用实施例1中所述操作构建含上述七点突变体基因的重组质粒,命名为pet-chi-mut7。
实施例3表达纯化
本领域公知的方法制备大肠杆菌bl21(de3)的感受态细胞,并用热激法将实施例1和实施例2构建的野生型表达载体和突变体的表达载体分别转化入感受态细胞,筛选阳性克隆pcr及测序验证后备用。
接种针挑取取阳性菌接种于5mllb培养基中,以30℃、200r/min培养12h,然后以按2%(v/v)接种量接种于400mllb培养基中,以30℃、200r/min培养8h。当细菌密度od600达到0.6时,加入0.5mm的iptg进行诱导表达,为防止包涵体的形成,表达条件为16℃低温诱导,过夜。
4℃离心收集菌体,pbs重悬,冰域超声破菌,离心收集上清,采用akta亲和层析系统,利用ni2+层析柱对上述表达的两种蛋白(野生型和突变型)进行亲和层析纯化,用咪唑洗脱后经过脱盐柱脱盐,蛋白保存在pbs缓冲液中备用,野生型蛋白命名为chi,突变型蛋白命名为chi-m7,纯化获得蛋白进行sds-page检测,结果见图1。
实施例4几丁质酶及其耐酸突变体的酶活测定
1、酶活测定方法
1)胶体几丁质制备:称取25g几丁质,加250ml85%磷酸,30℃反应1.5d,加蒸馏水待胶体沉淀后,反复水洗直至中性。用20mm磷酸缓冲液(ph7.0)调整终浓度至2%。
2)酶活测定:采用3,5-二硝基水杨酸(dns)比色定还原糖法。酶活力单位定义为该酶在最适条件下(即35℃,ph7.0的20mm磷酸缓冲液中)每分钟产生1微摩尔还原糖所需酶量。将0.25ml0.5%胶体几丁质与0.25ml适量稀释的待测酶液混合,于35℃温育1hr,离心去沉淀,上清加1.5ml蒸馏水与1.5mldns溶液,溶液,混匀,沸水育5min后,冷却至室温,补加21.5ml蒸馏水,混匀,测od540。的比色值,用糖做标准曲线。
3)蛋白浓度测定:蛋白质浓度的测定依照lowry方法,以牛血清白蛋白作为标准。
2、最适作用ph值
分别在ph值设置3.0,3.5,4.0,4.5,5.0,5.5,6.0,6.5,7.0,7.5,8.0,8.5,9.0,在温度35℃条件下测定上述纯化获得的野生型几丁质酶和突变体的活性,以最高酶活为100%,计算相对酶活,做温度-相对酶活曲线。结果如图2所示,野生型几丁质酶的最适作用温度为35℃,具有耐酸特性,最适的ph值为7.0,而突变体的最适作用ph为5.5,最适温度仍然为35℃,且在ph值在4.5时仍然能保持50%以上的酶活,在ph3.5时仍然保持近30%的活性,具有耐酸特性。
3、酶活测定
利用20mm磷酸缓冲液将酶蛋白稀释到1mg/ml分别在最适宜条件下(野生型35℃,ph7,突变型为35℃,ph5.5)水解胶体,具体为:
将0.25ml0.5%胶体几丁质与0.25ml适量稀释的待测酶液混合,于35℃(突变型为30℃)温育1hr,离心去沉淀,上清加1.5ml蒸馏水与1.5mldns溶液,溶液,混匀,沸水育5min后,冷却至室温,补加21.5ml蒸馏水,混匀,测od540。的比色值,用糖做标准曲线。
结果野生型的酶活在11.48u/mg,而突变型的在12.45u/mg,即绝对酶活有一定的提升。
该突变的筛选获得了可以更加适应于工业几丁质水解的几丁质酶。
序列表
<110>大连大学
<120>一种耐低温几丁质酶的耐酸突变体及其应用
<130>1
<150>2018115317313
<151>2018-12-14
<160>4
<170>siposequencelisting1.0
<210>1
<211>1054
<212>prt
<213>pseudoalteromonassp.
<400>1
metserasnlysleuvalglylysargleuphelysmetservalleu
151015
serleualavalthrglytrpalaalahisalaalavalaspcysthr
202530
asnilealaglutrpglnserglyvalalatyrthrglyglyglngln
354045
valglnlysalaasnthralatyrglualalystrptrpthrglnala
505560
aspprovalthrhisserglyglntyrglnglutrplysasnleugly
65707580
alacysseraspglyglnvalasngluserproilealagluvalleu
859095
serprometserglyserserphethrgluglyaspservalvalile
100105110
glualaglnthrseraspseraspglythrvalvallysleugluval
115120125
leuvalaspglyvalalailealathrleuserseralaprotyrser
130135140
thrthrtrpleualavalthrglyglnhisglnileservalargala
145150155160
thraspasplysglyalagluthrthrserproalaasnservalthr
165170175
valalagluvalvalproaspasntyralaprothralaserleuthr
180185190
serasnalaserserasnthrleuasnleuasnaspasnvalvalleu
195200205
asnleuserglyseraspglnaspglythrvalthrserleugluleu
210215220
pheglnaspglyalaservalalaglnileserglyalathrglyser
225230235240
pheasnphethralathrseralaglyasnthrglnpheserleuval
245250255
alathraspasplysglyalathrseraspalaglnthrleuserphe
260265270
argilevalaspglythrthrglnproglyaspargaspalacyslys
275280285
progluglyleutyrglnthrproglyvalasnthrprotyrcysthr
290295300
iletyraspalaaspglyarggluaspmetglyalaasphisproarg
305310315320
argvalileglytyrphethrsertrpargasnglyalaasnglygln
325330335
prosertyrleuvalaspaspileprotrpasplysilethrhisile
340345350
asntyralaphealahisvalaspalaasnasnlysvalserilegly
355360365
aspproseralaalaglyasnproalathrasnmetglutrpprogly
370375380
valalaglyalaglumetaspprothrleuprotyrlysglyhisphe
385390395400
asnleuleuasnlystyrlyslysleuhisproaspvallysthrleu
405410415
ileservalglyglytrpalagluthrglyglytyrpheaspalaasn
420425430
glyasnargvalalaserglyglyphetyrthrmetthrthrasnala
435440445
aspglyservalasnthralaglyileasnalaphealalysserala
450455460
valglupheilegluthrtyrglypheaspglyvalaspileasptyr
465470475480
glutyrprosersermetasnaspserglyhisproaspaspphepro
485490495
ileserasnalaargargalaglyleuasnalasertyrglnvalleu
500505510
metlysthrleuargglugluleuasplysalaglygluleualagly
515520525
lyshistyrmetleuthrilealaserproserserglytyrleuleu
530535540
argglymetgluthrpheglnthrthrlystyrleuasptyrvalasn
545550555560
ilemetsertyraspleuhisglyalatrpasnasphisvalglyhis
565570575
asnalaalaleutyraspthrglyleuaspsergluleualaglntrp
580585590
asnvaltyrglythrlysgluphegluglyileglytyrleuasnthr
595600605
asptrpalavalthrtyrpheargglyglyleuseralaglyargile
610615620
asnileglyvalprotyrtyrthrargglyphelysaspvalsergly
625630635640
glygluasnglyleutrpglyargalaalaleuproasnglnalaasp
645650655
cysalalysglythrglyvalglyglulysasnlyscysglyasngly
660665670
alavalglyileaspasnmettrphisaspleuaspgluasnglyasn
675680685
glumetproalaglyserasnproleutrphisvallysasnleugln
690695700
asnglyileleuprosertyrleualaalatyrglyleuaspproala
705710715720
asnaspprothrasplysleuvalglythrtyrthrarghistyrasp
725730735
servalalavalalaprotrpleutrpasnalaasplysasnvalphe
740745750
leuserilegluaspgluglusermetglythrlysleuasptyrval
755760765
ileasnlysglyleuglyglyilemetphetrpgluleualaglyasp
770775780
pheasptyraspseralalyslysglutyrphemetglyserthrmet
785790795800
thrserleualatyrasnlysphealaglnalaglyserlystyrasp
805810815
ileasnlysglyglulysthrhisglnvalproalaglualavalasp
820825830
valserphegluvallysasppheproileglyaspaspasntyrpro
835840845
ilealaprothrpheserphethrasnasnserasnileaspleuser
850855860
glyalalysvalserpheaspvalprovalalathrseralailephe
865870875880
lysserasntrpasnalaglnglulysleuglymetalavalaspval
885890895
asnargserasnalaalaglyasnasnileglyglyphegluasnglu
900905910
phehisargpheserilethrphevalasnglutrpglyglyalaala
915920925
lysserphealaproglygluthrileasnalaglnvalmettyrtyr
930935940
metproilethrglyprovalasnleuvalpheglulysaspglylys
945950955960
glntyralaalalyspheglutyrprotyrleuproaspalathrala
965970975
glyasnglyglyglyasnaspglyglyglyasnthraspthrcysglu
980985990
glyvalalavalalaaspileprovaltyrproasnpheproglnthr
99510001005
asptrpalaglyasnproserhisalaalaglyglyaspleumetval
101010151020
aspglyalaserthralavaltyrlysalalystrptrpthrserthr
1025103010351040
gluproglythrseralaasptrpaspservalcysthrleu
10451050
<210>2
<211>3165
<212>dna
<213>pseudoalteromonassp.
<400>2
atgagtaacaaattagtaggcaagcgtttatttaaaatgagcgtgttgtcgttagctgta60
actgggtgggcagcccatgcagcagtggactgtacaaacattgccgaatggcaatcaggc120
gtggcctacactggcgggcagcaagtgcaaaaagccaatactgcttatgaggcgaagtgg180
tggacacaagccgatccagtaacccattcagggcaatatcaagagtggaaaaacttaggt240
gcgtgtagtgatggtcaagtaaacgaatcacctattgcagaggttttatcacctatgagc300
ggtagttcttttactgaaggtgacagcgttgtcattgaggcgcaaacatcagattctgac360
ggcaccgttgtgaaactagaagtactggttgatggtgtggcgattgcaacactaagctct420
gcgccatattcaacaacttggcttgcagtgacagggcagcatcaaatttcggtacgcgct480
accgatgataaaggtgctgaaacaacatcccctgcaaatagtgtgactgtggctgaagta540
gtgccagataactatgcaccaacggcaagccttactagcaatgcgagttcaaataccttg600
aacttaaatgacaacgttgtcttaaatttgagcggtagcgatcaagatggcacagtaaca660
agtcttgaattgtttcaagatggcgcgtctgtagcccaaatttctggcgcaacaggcagt720
tttaattttaccgcaacaagcgcaggtaatacgcaatttagtcttgttgcaaccgacgac780
aaaggcgcaacatctgatgcacaaacattaagtttccgtattgttgatggcactacacaa840
ccgggtgaccgcgatgcctgtaagccggaaggtttgtaccaaacaccgggtgtaaatacg900
ccgtattgtactatttacgatgctgatggccgtgaagatatgggtgccgatcacccacgt960
cgtgtcattggttatttcaccagttggcgcaacggtgccaatggccaaccgtcttaccta1020
gtagatgatattccttgggataaaatcacccatattaactacgcgtttgcccatgttgat1080
gccaataacaaggtttcaattggcgatccaagtgcggcgggtaaccctgctaccaatatg1140
gaatggccaggtgtagcaggtgctgaaatggacccaacactgccgtacaaaggtcatttc1200
aacttactgaataaatataaaaagctccatccagacgttaaaacactgatctctgtaggt1260
ggttgggcagaaacgggcggttactttgacgcaaacggcaaccgtgttgcaagcggtggt1320
ttctacactatgacaaccaatgctgatggctcagtaaatactgcgggcattaatgcgttt1380
gcaaaaagtgcggttgagtttattgaaacgtacggtttcgatggggttgatattgattat1440
gaatacccatcatcaatgaatgattcgggtcaccctgatgatttccctatttcaaatgca1500
cgccgagcagggctaaatgcctcttaccaagtgttaatgaaaaccctacgtgaagagctt1560
gataaagcgggtgaattagctggtaaacactacatgcttaccattgcatcgccgtcatca1620
ggttacttattacgtggtatggaaactttccaaacaactaagtacctagattacgtaaac1680
attatgtcttacgacttgcacggtgcttggaacgaccacgtgggtcataacgcagcgctt1740
tacgatacagggttagactctgagcttgcgcagtggaatgtgtatggcaccaaagagttt1800
gaaggcattggctatttaaataccgattgggcggttacgtatttccgcggtggtttaagc1860
gcaggtcgcatcaacattggtgtgccgtactatacccgtggttttaaagatgtatcgggt1920
ggcgaaaacggtttatgggggcgcgctgcgttacctaaccaagctgattgtgccaaaggt1980
actggcgtaggtgagaaaaacaaatgtggtaatggcgcagttggtatcgacaacatgtgg2040
catgaccttgacgaaaacggtaatgaaatgcctgcaggtagcaatccgttatggcatgtt2100
aaaaacctacaaaatggcattttaccaagttaccttgcggcttacggcttagatccagca2160
aatgatccaactgataagctagtgggcacttacactcgtcattacgattcagtggcagta2220
gcgccgtggctttggaatgctgataaaaacgtgttcttgtcgattgaagatgaagagtca2280
atgggcactaagcttgattacgtaatcaacaaaggccttggcggtattatgttctgggag2340
ctggcaggtgactttgactacgacagcgcgaagaaagagtactttatgggttctactatg2400
acctcgcttgcttacaataagtttgcacaagcgggttctaagtacgacatcaacaaaggt2460
gaaaaaactcaccaagtaccagcagaggcagttgacgtaagctttgaagtgaaagacttc2520
ccaattggtgacgacaactacccaattgcgccaacctttagttttactaacaactcaaac2580
attgatttgagcggtgcaaaagtatcgtttgatgtaccagtggctacatcggcaattttc2640
aaatcaaactggaatgcgcaagaaaaactcggcatggcagttgacgtaaatcgttcaaac2700
gcagctggcaacaacattggtggctttgaaaacgagttccaccgtttctcaattaccttt2760
gttaacgagtggggcggcaatgctaagtcgtttgcgccgggcgaaaccattaatgcgcaa2820
gtatggtactacatgccaattactggcccagtgaaccttgtatttgaaaaagacggtaag2880
caatacgccgctaaatttgaatacccatatctaccagatgcaacggccggtaacggtggt2940
ggcaatgatggcggcggtaacaccgacacatgtgaaggtgttgcagtagctgatattcca3000
gtttatccaaacttcccacaaacagattgggcaggcaacccgtcacacgctgcaggcggt3060
gacttgatggttgatggcgcatctactgcggtttacaaagccaagtggtggacatcaact3120
gagcctggcacatccgccgattgggatagtgtgtgtactttataa3165
<210>3
<211>1054
<212>prt
<213>pseudoalteromonassp.
<400>3
metserasnlysleuvalglylysargleuphelysmetservalleu
151015
serleualavalthrglytrpalaalahisalaalavalaspcysthr
202530
asnilealaglutrpglnserglyvalalatyrthrglyglyglngln
354045
valglnlysalaasnthralatyrglualalystrptrpthrglnala
505560
aspprovalthrhisserglyglntyrglnglutrplysasnleugly
65707580
alacysseraspglyglnvalasngluserproilealagluvalleu
859095
serprometserglyserserphethrgluglyaspservalvalile
100105110
glualaglnthrseraspseraspglythrvalvallysleugluval
115120125
leuvalaspglyvalalailealathrleuserseralaprotyrser
130135140
thrthrtrpleualavalthrglyglnhisglnileservalargala
145150155160
thraspasplysglyalagluthrthrserproalaasnservalthr
165170175
valalagluvalvalproaspasntyralaprothralaserleuthr
180185190
serasnalaserserasnthrleuasnleuasnaspasnvalvalleu
195200205
asnleuserglyseraspglnaspglythrvalthrserleugluleu
210215220
pheglnaspglyalaservalalaglnileserglyalathrglyser
225230235240
pheasnphethralathrseralaglyasnthrglnpheserleuval
245250255
alathraspasplysglyalathrseraspalaglnthrleuserphe
260265270
argilevalaspglythrthrglnproglyaspargaspalacyslys
275280285
progluglyleutyrglnthrproglyvalasnthrprotyrcysthr
290295300
iletyraspalaaspglyarggluaspmetglyalaasphisproarg
305310315320
argvalileglytyrphethrsertrpargasnglyalaasnglygln
325330335
prosertyrleuvalaspaspileprotrpasplysilethrhisile
340345350
asntyralaphealahisvalaspalaasnasnlysvalserilegly
355360365
aspproseralaalaglyasnproalathrasnmetglutrpprogly
370375380
valalaglyalaglumetaspprothrleuprotyrlysglyhisphe
385390395400
asnleuleuasnlystyrlyslysleuhisproaspvallysthrleu
405410415
ileservalglyglytrpalagluthrglyglytyrpheaspalaasn
420425430
glyasnargvalalaserglyglyphetyrthrmetthrthrasnala
435440445
aspglyservalasnthralaglyileasnalaphealalysserala
450455460
valglupheilegluthrtyrglypheaspglyvalaspileasptyr
465470475480
glutyrprosersermetasnaspserglyhisproaspaspphepro
485490495
ileserasnalaargargalaglyleuasnalasertyrglnvalleu
500505510
metlysthrleuargglugluleuasplysalaglygluleualagly
515520525
lyshistyrmetleuthrilealaserproserserglytyrleuleu
530535540
argglymetgluthrpheglnthrthrlystyrleuasptyrvalasn
545550555560
ilemetsertyraspleuhisglyalatrpasnasphisvalglyhis
565570575
asnalaalaleutyraspthrglyleuaspsergluleualaglntrp
580585590
asnvaltyrglythrlysgluphegluglyileglytyrleuasnthr
595600605
asptrpalavalthrtyrpheargglyglyleuseralaglyargile
610615620
asnileglyvalprotyrtyrthrargglyphelysaspvalsergly
625630635640
glygluasnglyleutrpglyargalaalaleuproasnglnalaasp
645650655
cysalalysglythrglyvalglyglulysasnlyscysglyasngly
660665670
alavalglyileaspasnmettrphisaspleuaspgluasnglyasn
675680685
glumetproalaglyserasnproleutrphisvallysasnleugln
690695700
asnglyileleuprosertyrleualaalatyrglyleuaspproala
705710715720
asnaspprothrasplysleuvalglythrtyrthrarghistyrasp
725730735
servalalavalalaprotrpleutrpasnalaasplysasnvalphe
740745750
leuserilegluaspgluglusermetglythrlysleuasptyrval
755760765
ileasnlysglyleuglyglyilemetphetrpgluleualaglyasp
770775780
pheasptyraspseralalyslysglutyrphemetglyserthrmet
785790795800
thrserleualatyrasnlysphealaglnalaglyserlystyrasp
805810815
ileasnlysglyglulysthrhisglnvalproalaglualavalasp
820825830
valserphegluvallysasppheproileglyaspaspasntyrpro
835840845
ilealaprothrpheserphethrasnasnserasnseraspleuser
850855860
glyalalysvalserpheaspvalprovalcysthrseralailephe
865870875880
lysserasntrpasnalaglnglulysleuglymetalaproaspval
885890895
asnargserasnalaalaargasnasnileglyglyphegluasnglu
900905910
phelysargpheserilethrphevalasnglutrpglyglyasnala
915920925
lysserphealaproglygluthrileasnalaglnvaltrptyrtyr
930935940
metproilethrglyprovalasnleuvalpheglulysaspglylys
945950955960
glntyralaalalyspheglutyrprotyrleuproaspalathrala
965970975
glyasnglyglyglyasnaspglyglyglyasnthraspthrcysglu
980985990
glyvalalavalalaaspileprovaltyrproasnpheproglnthr
99510001005
asptrpalaglyasnproserhisalaalaglyglyaspleumetval
101010151020
aspglyalaserthralavaltyrlysalalystrptrpthrserthr
1025103010351040
gluproglythrseralaasptrpaspservalcysthrleu
10451050
<210>4
<211>3165
<212>dna
<213>pseudoalteromonassp.
<400>4
atgagtaacaaattagtaggcaagcgtttatttaaaatgagcgtgttgtcgttagctgta60
actgggtgggcagcccatgcagcagtggactgtacaaacattgccgaatggcaatcaggc120
gtggcctacactggcgggcagcaagtgcaaaaagccaatactgcttatgaggcgaagtgg180
tggacacaagccgatccagtaacccattcagggcaatatcaagagtggaaaaacttaggt240
gcgtgtagtgatggtcaagtaaacgaatcacctattgcagaggttttatcacctatgagc300
ggtagttcttttactgaaggtgacagcgttgtcattgaggcgcaaacatcagattctgac360
ggcaccgttgtgaaactagaagtactggttgatggtgtggcgattgcaacactaagctct420
gcgccatattcaacaacttggcttgcagtgacagggcagcatcaaatttcggtacgcgct480
accgatgataaaggtgctgaaacaacatcccctgcaaatagtgtgactgtggctgaagta540
gtgccagataactatgcaccaacggcaagccttactagcaatgcgagttcaaataccttg600
aacttaaatgacaacgttgtcttaaatttgagcggtagcgatcaagatggcacagtaaca660
agtcttgaattgtttcaagatggcgcgtctgtagcccaaatttctggcgcaacaggcagt720
tttaattttaccgcaacaagcgcaggtaatacgcaatttagtcttgttgcaaccgacgac780
aaaggcgcaacatctgatgcacaaacattaagtttccgtattgttgatggcactacacaa840
ccgggtgaccgcgatgcctgtaagccggaaggtttgtaccaaacaccgggtgtaaatacg900
ccgtattgtactatttacgatgctgatggccgtgaagatatgggtgccgatcacccacgt960
cgtgtcattggttatttcaccagttggcgcaacggtgccaatggccaaccgtcttaccta1020
gtagatgatattccttgggataaaatcacccatattaactacgcgtttgcccatgttgat1080
gccaataacaaggtttcaattggcgatccaagtgcggcgggtaaccctgctaccaatatg1140
gaatggccaggtgtagcaggtgctgaaatggacccaacactgccgtacaaaggtcatttc1200
aacttactgaataaatataaaaagctccatccagacgttaaaacactgatctctgtaggt1260
ggttgggcagaaacgggcggttactttgacgcaaacggcaaccgtgttgcaagcggtggt1320
ttctacactatgacaaccaatgctgatggctcagtaaatactgcgggcattaatgcgttt1380
gcaaaaagtgcggttgagtttattgaaacgtacggtttcgatggggttgatattgattat1440
gaatacccatcatcaatgaatgattcgggtcaccctgatgatttccctatttcaaatgca1500
cgccgagcagggctaaatgcctcttaccaagtgttaatgaaaaccctacgtgaagagctt1560
gataaagcgggtgaattagctggtaaacactacatgcttaccattgcatcgccgtcatca1620
ggttacttattacgtggtatggaaactttccaaacaactaagtacctagattacgtaaac1680
attatgtcttacgacttgcacggtgcttggaacgaccacgtgggtcataacgcagcgctt1740
tacgatacagggttagactctgagcttgcgcagtggaatgtgtatggcaccaaagagttt1800
gaaggcattggctatttaaataccgattgggcggttacgtatttccgcggtggtttaagc1860
gcaggtcgcatcaacattggtgtgccgtactatacccgtggttttaaagatgtatcgggt1920
ggcgaaaacggtttatgggggcgcgctgcgttacctaaccaagctgattgtgccaaaggt1980
actggcgtaggtgagaaaaacaaatgtggtaatggcgcagttggtatcgacaacatgtgg2040
catgaccttgacgaaaacggtaatgaaatgcctgcaggtagcaatccgttatggcatgtt2100
aaaaacctacaaaatggcattttaccaagttaccttgcggcttacggcttagatccagca2160
aatgatccaactgataagctagtgggcacttacactcgtcattacgattcagtggcagta2220
gcgccgtggctttggaatgctgataaaaacgtgttcttgtcgattgaagatgaagagtca2280
atgggcactaagcttgattacgtaatcaacaaaggccttggcggtattatgttctgggag2340
ctggcaggtgactttgactacgacagcgcgaagaaagagtactttatgggttctactatg2400
acctcgcttgcttacaataagtttgcacaagcgggttctaagtacgacatcaacaaaggt2460
gaaaaaactcaccaagtaccagcagaggcagttgacgtaagctttgaagtgaaagacttc2520
ccaattggtgacgacaactacccaattgcgccaacctttagttttactaacaactcaaac2580
agtgatttgagcggtgcaaaagtatcgtttgatgtaccagtgtgtacatcggcaattttc2640
aaatcaaactggaatgcgcaagaaaaactcggcatggcacctgacgtaaatcgttcaaac2700
gcagctcgcaacaacattggtggctttgaaaacgagttcaagcgtttctcaattaccttt2760
gttaacgagtggggcggcgctgctaagtcgtttgcgccgggcgaaaccattaatgcgcaa2820
gtaatgtactacatgccaattactggcccagtgaaccttgtatttgaaaaagacggtaag2880
caatacgccgctaaatttgaatacccatatctaccagatgcaacggccggtaacggtggt2940
ggcaatgatggcggcggtaacaccgacacatgtgaaggtgttgcagtagctgatattcca3000
gtttatccaaacttcccacaaacagattgggcaggcaacccgtcacacgctgcaggcggt3060
gacttgatggttgatggcgcatctactgcggtttacaaagccaagtggtggacatcaact3120
gagcctggcacatccgccgattgggatagtgtgtgtactttataa3165
- 还没有人留言评论。精彩留言会获得点赞!