阳离子脂肽、药物组合物及其应用的制作方法
本发明属于生物技术领域,具体涉及阳离子脂肽、药物组合物及其应用。
背景技术:
细菌耐药危机日趋严峻,已经成为全球范围内的重大公共安全问题,严重危害人类的健康。在2016年的二十国集团峰会(g20)上,各国领导人明确提到:“抗生素耐药性严重威胁公共健康、经济增长和全球经济稳定”。传统的抗菌药物开发手段已很难应对当前危机,急需新思路来研究抗生素耐药问题和开发新型抗生素。近年来,基因测序技术的革新把天然药物开发推向了基因时代,为微生物来源的新型抗生素开发带来了新希望。在传统抗生素失效的后抗生素时代,挖掘微生物天然产物依然是解决抗生素危机的前沿热点。
由多重耐药革兰氏阴性菌引起的感染已成为公众健康的主要威胁(参见文献1-2)。在过去的40年中,尚没有真正新型的用于抗革兰氏阴性菌的抗生素面市(参见文献3)。这一因素、以及抗生素耐药性的出现,使得对新抗生素的迫切需求变得尤为突出(参见文献1-4)。阳离子肽代表了一个巨大的抗生素家族,并以其多样的化学结构和巨大的抗革兰氏阴性病原体的潜力而引起了广泛的兴趣(参见文献5-8)。阳离子非核糖体肽(包括多粘菌素(polymyxins)和短杆菌肽s(gramicidins))是结构最多样化的阳离子肽家族,具有很大的抗菌潜力,但是在很大程度上仍然是未知的(参见文献6、9、10)。
阳离子肽存在于所有形式的生命(哺乳动物、植物和细菌)中,并且是自然界中最广泛且结构最多样化的抗生素之一。由于阳离子肽具有独特的化学性质,不仅使其便于渗透穿过革兰氏阴性菌的高度抗渗性外膜,而且还使其能够与多个阴离子细胞内靶点相互作用,因此阳离子肽对耐药革兰氏阴性病原体高度有效(参见文献5-9)。在过去的40年中,已经发现了数千种具有广泛抗菌活性的阳离子肽,其中大多数是天然的或天然来源于多细胞生物的宿主防御肽。然而,由于其供应成本高、不稳定以致于蛋白质降解、以及毒理学特征不明,因此这些阳离子肽中只有少数进入临床应用(参见文献8、9、11-15)。供应充足并且具有蛋白质水解稳定性的细菌阳离子非核糖体肽(cnrp)是很有吸引力的治疗候选物。由杆菌类细菌产生的多粘菌素和短杆菌肽s实际上是少数几种有临床疗效的先例中的两种(参见文献9、14-17)。然而,由于cnrp的多样性和复杂性使得系统化研究变得困难,因此,细菌中绝大多数基因编码的cnrp被忽视、并且仍然是未知的。
技术实现要素:
为解决上述现有技术中所存在的问题,本发明提供了阳离子脂肽、药物组合物及其应用。
具体而言,本发明提供了:
(1)一种阳离子脂肽,其特征在于,所述阳离子脂肽包含:
具有6-12个碳的n端脂肪酰基;
含有3-12个氨基酸的线性肽段,其中该线性肽段通过其n端与所述n端脂肪酰基相连,并且含有≥2个带正电荷的氨基酸;和
含有4-9个氨基酸的c端亲脂性环肽,其中该c端亲脂性环肽与所述线性肽段的c端相连;并且
其中所述阳离子脂肽具有革兰氏阴性菌抗菌性。
(2)根据(1)所述的阳离子脂肽,其中所述线性肽段含有4-9个氨基酸。
(3)根据(1)所述的阳离子脂肽,其中所述线性肽段含有2-3个带正电荷的氨基酸。
(4)根据(1)-(3)中任一项所述的阳离子脂肽,其中所述带正电荷的氨基酸为orn或lys。
(5)根据(1)所述的阳离子脂肽,其中所述n端脂肪酰基包括直链和支链的烷酰基。
(6)根据(5)所述的阳离子脂肽,其中所述烷酰基的碳原子个数为7-9个。
(7)根据(1)所述的阳离子脂肽,其中所述c端亲脂性环肽为由四个氨基酸或五个氨基酸形成的环肽。
(8)根据(1)所述的阳离子脂肽,其中所述阳离子脂肽的结构式如下式i或ii所示:
(9)一种药物组合物,包含根据(1)-(8)中任一项所述的阳离子脂肽。
(10)根据(1)-(8)中任一项所述的阳离子脂肽在制备革兰氏阴性菌抗生素药物中的应用。
本发明与现有技术相比具有以下优点和积极效果:
本发明在传统的活性和化学导向的天然产物研究方法基础上,进一步采用全基因组挖掘策略,利用生物学信息学大数据在基因组导向下鉴别出潜在的可作为革兰氏阴性菌抗生素的cnrp,并结合经典的化学分析及活性评价,筛选并验证了有效的可作为抗革兰氏阴性菌抗生素的cnrp,其为阳离子脂肽。本发明通过上述系统化的研究和筛选策略,发现具有本发明所述的结构特征的阳离子脂肽对革兰氏阴性病原体具有广泛且强的杀菌活性,低毒高效,耐药性风险低,并且由于具有脂肪酸侧链和内酯环(亲脂性环肽),其在蛋白水解方面也是稳定的。
特别地,本发明还发现其中的两种化合物(本发明将其分别命名为brevicidine和laterocidine)具有最优的抗革兰氏阴性菌性质,特别适合开发为抗革兰氏阴性菌抗生素。
附图说明
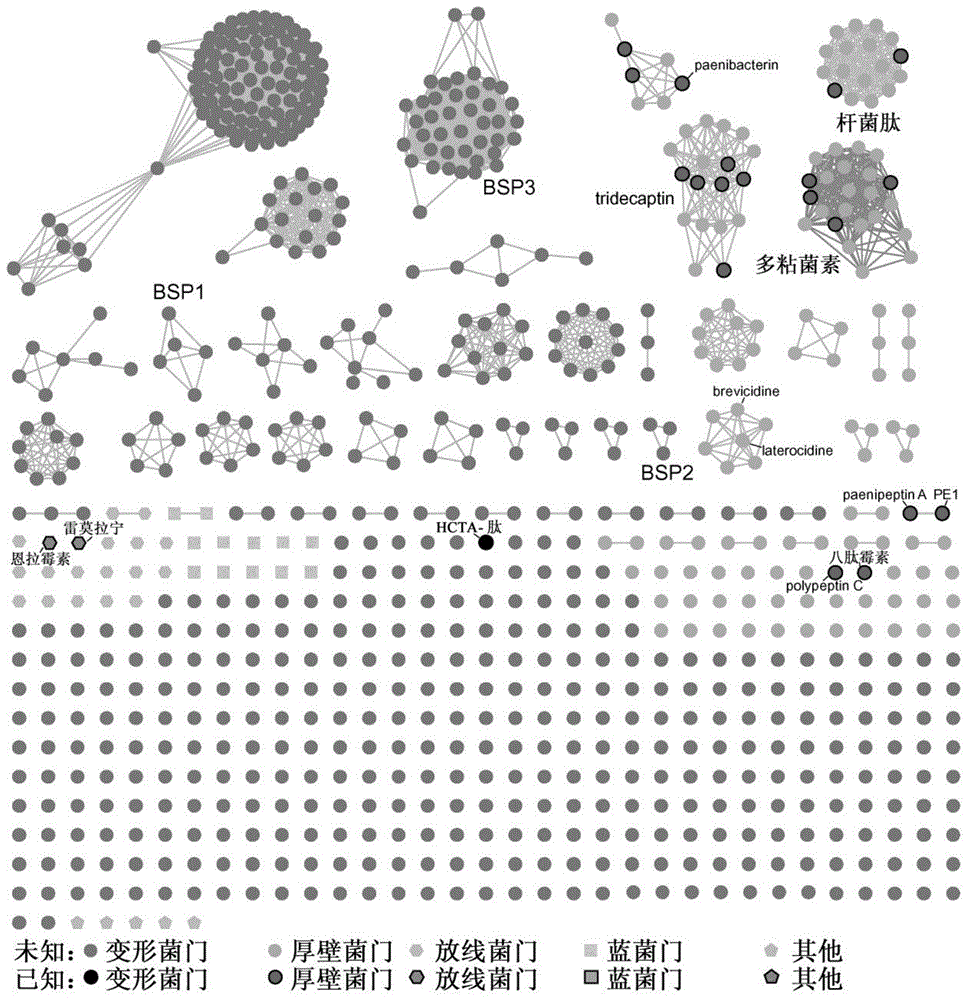
图1示出cnrp的肽相似性网络(peptidesimilaritynetwork),其显示了它们的多样性、分布和发现状态。显然,大部分cnrp还处于未被探索的状态,特别是厚壁菌门(firmicutes)和变形菌门(proteobacteria)中的那些cnrp更是如此。
图2示出通过全基因组挖掘发现的阳离子抗菌肽的基因簇和结构。其中:图2a从上到下分别示出brevicidine和laterocidine的cnrp的基因簇,基因簇上方标出了所预测的编码氨基酸,阳离子残基被标记成浅灰色;图2b示出brevicidine、laterocidine的结构,该结构是通过ms/nmr分析鉴定的。
图3示出brevicidine和laterocidine具有杀菌活性、耐药性风险低、以及在小鼠大腿感染模型中的效力,其中,图3a示出时间-杀菌试验结果,图中数据代表三次独立实验±s.d.;图3b示出细菌生长动力学,图中数据为暴露于2倍mic的相应抗生素的大肠杆菌细胞的光密度;图3c示出cnrp处理导致大肠杆菌的裂解,该图为三次独立实验的代表;图3d示出在亚mic水平的抗菌药物的存在下,连续传代过程中的耐药性获得情况;图3e示出在10倍mic的cnrp抗生素的存在下,在37℃下培养至中对数期(od约为0.5)的大肠杆菌的原子力显微镜图像,白色方框突出显示为了获得经brevicidine处理的细胞表面的高分辨率轮廓图像而扫描的区域,该区域在图3e右上方示出;图示扫描是两次独立实验的代表;图3f示出brevicidine和laterocidine在用大肠杆菌感染的小鼠大腿模型中的药效:用brevicidine(30mgkg-1)或laterocidine(15mgkg-1)处理(2次)大肠杆菌感染的大腿(5只小鼠)导致在26h后活菌数量减少。通过mann-whitney检验来分析组与组之间的差异显著性(*p<0.01)。在a-f中,con:阴性对照,bre:brevicidine,lat:laterocidine,ply:多粘菌素,lev:左氧氟沙星,cpf:环丙沙星。
图4示出cnrpbgc全基因组挖掘的计算机工作流程。其中,步骤a示出从genbank下载7,395份完整的或者作为草稿的细菌基因组,并利用单机版的antismash进行bgc分析和nrp序列预测,从而鉴别出29,461个nrpbgc,其中有817个nrpbgc编码长度大于或等于6的cnrp;步骤b示出将预测的肽序列提交给efi-酶相似性工具(efi-est),以构建肽相似性网络;步骤c示出通过传统的基于发酵的分离法获得具有理想化学特征的cnrp。
图5示出通过全基因组挖掘揭示的cnrpbgc在细菌中的系统发育分布。其中图5a示出对细菌完整基因组(5,585份)进行的全bgc分析揭示出cnrp大量分布于变形菌门、放线菌门和厚壁菌门中;图5b示出cnrpbgc在菌属水平上的广泛分布。
图6示出通过对7,395份细菌基因组进行全基因组挖掘揭示出的cnrp的多样性,其中:图6a示出cnrp在肽长度方面的多样性:cnrp(n=1,818)的长度为2至34不等,平均为6.6,中值为5,这显示了它们的高度多样性;虚线以下(长度≥6)的是位于图1的相似性网络中的cnrp(n=817);这些cnrp的平均长度为9.7,中值长度为9;图6b示出cnrp在带正电荷残基百分比方面的多样性:带正电荷残基的百分比为6.3%至78.6%,平均为33.0%(中值为30.0%)。
图7示出n-酰化cnrp(阳离子脂肽)的序列相似性网络,其显示了它们的多样性、分布和发现状态。显然,大部分n-酰化cnrp还处于未被探索的状态。
图8示出推定的n-酰化cnrp在肽长度方面的多样性。在其bgc中具有缩合启动结构域(cs)的cnrp被认为是n-酰化cnrp。n-酰化cnrp(n=260)的长度为2至34不等,平均为11.7,中值为11。虚线以下(长度≥6)的是位于图7的序列相似性网络中的n-酰化cnrp(n=239),这些n-酰化cnrp的平均长度为12.38,中值长度为12。
图9示出brevicidine的ms、nmr、以及marfey型分析。其中图9a示出化合物1的msn分析;图9b示出化合物1的关键1h-1h-cosy与1h-13c-hmbc关联;图9c示出brevicidine的marfey型分析(经d/l-fdla衍生化的化合物1组成氨基酸的保留时间(min)):在brukerav500波谱仪(500mhz)上,采用dmso-d6记录化合物1的1h、1h-1h-cosy、1h-13c-hsqc和1h-13c-hmbcnmr数据(1h-nmrmeoh-d4:δ=3.31ppm;dmso-d6:δ=2.50ppm;13c-nmr:meoh-d4:δ=49.00ppm;dmso-d6:δ=39.8ppm)。获得的化合物1为白色非晶固体。基于hresims数据(m/z:760.4046[m+2h]2+,calc760.4064m/z:507.2729[m+3h]3+,calc507.2734),将其分子式建立为c74h106n18o17。1hnmr谱中酰胺nh和α-氨基质子的特征信号以及13cnmr谱中羰基的特征信号表明了该化合物的肽属性。通过分析1h、13c、1h-1hcosy、hmqc、以及hmbcnmr波谱数据,进一步建立了化合物1的总体结构(图9b),揭示出其结构中存在orn(3)、trp(2)、gly(2)、ile(1)、ser(1)、thr(1)、asn(1)、tyr(1)以及经鉴定为4-甲基己酸的脂类残基。通过nmr分析建立的总体结构也得到了ms/ms分析的证实。采用高级marfey法确定化合物1的氨基酸构型。将化合物1及其水解产物4、6、7(0.1-0.2mg)在6mhcl中于110℃下水解过夜。将各溶液蒸发至干燥,将残留物溶于100μl水中并且分成两份。用20μlnahco3(1m)和50μl的1-氟-2,4-二硝基苯基-5-l-亮氨酸酰胺(l-fdla)或d-fdla(1m)将每份样品在40℃下处理2h。用5μl的hcl(1m)将反应淬灭,并且用200μl的meoh稀释。采用uplc-ms分析,通过比较l-/dfdla衍生化样品来确定立体化学性质。基于这四种化合物的高级marfey分析所产生的数据的综合,确定天然产物brevicidine为脂缩酚酸肽(lipodepsipeptide):4-甲基-己酰基-d-asn-d-tyr-d-trp-d-orn-orn-gly-d-orn-trp-thr-ile-gly-ser(通过thr与c末端之间内酯化而成环)。化合物1的结构与基于生物合成途径分析所预测的结构一致(见图12)。
图10示出lacterocidine的ms、nmr和marfey型分析。其中图10a示出laterocidine的msn分析;图10b示出化合物2的关键1h-1h-cosy与1h-13c-hmbc关联;图10c示出laterocidine的marfey型分析(经d/l-fdla衍生化的化合物2组成氨基酸的保留时间(min)):在brukerav500波谱仪(500mhz)上,采用dmso-d6记录化合物2的1h、1h-1h-cosy、1h-13c-hsqc和1h-13c-hmbcnmr数据(1h-nmr:dmso-d6:δ=2.50ppm;13c-nmr:dmso-d6:δ=39.8ppm)。获得的化合物2为白色非晶固体。基于hresims数据(m/z:802.9345[m+2h]2+,calc802.9329),将其分子式建立为c78h113n19o18。1hnmr谱中酰胺nh和α-氨基质子的特征信号以及13cnmr谱中羰基的特征信号表明了该化合物的肽属性。通过分析1h、13c、1h-1hcosy、hmqc和hmbcnmr波谱数据,进一步建立了laterocidine的总体结构(图10b),揭示出其结构中存在orn(3)、trp(2)、gly(3)、ile(1)、ser(1)、thr(1)、asn(1)、tyr(1)以及经鉴定为7-甲基辛酸的脂类残基。通过nmr分析建立的总体结构也得到了ms/ms分析的证实。通过对化合物2及其水解产物7、8、10进行如上文所述的marfey分析来确定化合物2的氨基酸构型。基于高级marfey分析和生物合成途径分析(见图13)所产生的数据的综合,确定天然产物laterocidine为脂缩酚酸肽:7-甲基-辛酰基-d-ser-d-tyr-d-trp-d-orn-orn-gly-d-orn-trp-thr-ile-asn-gly-gly(通过thr与c末端之间内酯化而成环)。
图11示出paenibacterinb的ms分析。其中图11a示出paenibacterinb(paea-g,来自nz_ambz01000008,57,959-110,917nt))与paenibacterina(pbta-c)的bgc对比;图11b示出通过ms/ms分析鉴定出的paenibacterinb的结构;图11c示出paenibacterinb的msn分析。
图12示出预测的brevicidine的生物合成途径(结构域缩写:c,缩合;a,腺苷酰化;t,硫醇化(载体);e,差向异构化;te,硫酯酶;基因簇位置:kb894285,144,281-196,331nt)。brevicidine的生物合成是从典型的nrp启动模块开始的,其具有使n末端与脂肪酸发生酰化的缩合启动结构域。根据共线性规律,12个氨基酸被引入该线性肽中,在产物卸载过程中,由硫酯酶结构域催化最后的ser12与thr9之间发生闭环,从而得到brevicidine。
图13示出预测的laterocidine的生物合成途径(结构域缩写:c,缩合;a,腺苷酰化;t,硫醇化(载体);e,差向异构化;te,硫酯酶;基因簇位置:nz_cp007806.1,2,835,471-2,890,522)。laterocidine的生物合成是从典型的nrps启动模块开始的,其具有使n末端与脂肪酸发生酰化的缩合启动结构域。根据共线性规律,13个氨基酸被引入该线性肽中,在产物卸载过程中,由硫酯酶结构域催化最后的gly13与thr9之间发生闭环,从而得到laterocidine。
图14示出化合物1、2、4-11的化学结构。
图15示出brevicidine和laterocidine对真核细胞的细胞毒性和溶血活性。其中,图15a示出在浓度最高达128μgml-1的brevicidine或laterocidine的存在下孵育兔红细胞,通过血红蛋白的释放来监测细胞溶解情况;阴性对照是未使用测试化合物的细胞;阳性对照是在10%tritonx-100中孵育细胞,以使其完全溶解;图中数据为两次独立实验的代表;图15b示出brevicidine和laterocidine的细胞毒性,图中数据为三次独立实验的代表,图中bogorol为阳性对照。
图16示出经brevicidine和laterocidine处理的大肠杆菌atcc25922的原子力显微镜图像,其显示出对外膜结构的显著影响。图中“对照”为不经测试化合物处理的细胞图像,白色方框突出显示为了获得经处理的细胞表面的高分辨率轮廓图像而扫描的区域,该区域在右侧示出,图中数据是三次独立实验的代表。
图17示出经brevicidine和laterocidine处理的铜绿假单胞菌pao1的原子力显微镜图像,其显示出对外膜结构的显著影响。图中“对照”为不经测试化合物处理的细胞图像,白色方框突出显示为了获得经处理的细胞表面的高分辨率轮廓图像而扫描的区域,该区域在右侧示出,图中数据是三次独立实验的代表。
图18示出通过等温滴定量热(itc)实验揭示了brevicidine和多粘菌素b对源自大肠杆菌的lps的结合作用。其中图18a为多粘菌素b+lps;图18b为brevicidine+lps。所有结果均显示出对lps的强结合作用。
图19示出brevicidine的1hnmr(dmso-d6,500mhz)。
图20示出brevicidine的13c-nmr(dmso-d6,100mhz)。
图21示出brevicidine的1h-1hcosy(dmso-d6,500mhz)。
图22示出brevicidine的1h-13chsqc(dmso-d6,500mhz)。
图23示出brevicidine的1h-13chmbc(dmso-d6,500mhz)。
图24示出brevicidine的1h-1htocsy(dmso-d6,500mhz)。
图25示出laterocidine的1hnmr(dmso-d6,500mhz)。
图26示出laterocidine的1h-1hcosy(dmso-d6,500mhz)。
图27示出laterocidine的1h-13chsqc(dmso-d6,500mhz)。
图28示出laterocidine的1h-13chmbc(dmso-d6,500mhz)。
图29示出laterocidine的1h-1htocsy(dmso-d6,500mhz)。
图30示出化合物4的1hnmr(cd3od,500mhz)。
图31示出化合物4的13nmr(cd3od,100mhz)。
图32示出化合物4的1h-1hcosy(cd3od,500mhz)。
图33示出化合物4的1h-13chsqc(cd3od,500mhz)。
图34示出化合物4的1h-13chmbc(cd3od,500mhz)。
具体实施方式
以下通过具体实施方式的描述并参照附图对本发明作进一步说明,但这并非是对本发明的限制,本领域技术人员根据本发明的基本思想,可以做出各种修改或改进,但是只要不脱离本发明的基本思想,均在本发明的范围之内。
为了系统化地研究阳离子非核糖体肽(cnrp),并筛选找到能够有效地作为革兰氏阴性菌抗生素的cnrp,本发明在传统的活性和化学导向的天然产物研究方法基础上,利用生物学信息学大数据在基因组导向下鉴别出了潜在的可作为革兰氏阴性菌抗生素的cnrp,并结合经典的化学分析及活性评价,筛选并验证了有效的可作为抗革兰氏阴性菌抗生素的cnrp,其为一类阳离子脂肽。本发明通过上述系统化的研究和筛选策略,发现具有本发明所述的结构特征的阳离子脂肽对革兰氏阴性病原体具有广泛且强的杀菌活性,低毒高效,耐药性风险低,并且由于具有脂肪酸侧链和内酯环(亲脂性环肽),其在蛋白水解上也是稳定的。
由此,本发明提供了一种阳离子脂肽,其特征在于,所述阳离子脂肽包含:
具有6-12个(例如6、7、8、9、10、11、12个)碳的n端脂肪酰基;
含有3-12个(例如3、4、5、6、7、8、9、10、11、12个)氨基酸的线性肽段,其中该线性肽段通过其n端与所述n端脂肪酰基相连,并且含有≥2个(例如≥3、≥4、≥5、≥6个)带正电荷的氨基酸;和
含有4-9个(例如4、5、6、7、8、9个)氨基酸的c端亲脂性环肽,其中该c端亲脂性环肽与所述线性肽段的c端相连;并且
其中所述阳离子脂肽具有革兰氏阴性菌抗菌性。
在本文中,术语“生物信息学(bioinformatics)”在本领域是已知的,其是指在生命科学的研究中,以计算机为工具对生物信息进行储存、检索和分析的科学。通常而言,生物信息学将分子生物学与信息技术(尤其是因特网技术)结合在一起。生物信息学的研究材料和结果包括各种各样的生物学数据,其研究工具包括计算机,研究方法包括对生物学数据的搜索(收集和筛选)、处理(编辑、整理、管理和显示)及利用(计算、模拟)。
在本文中,术语“非核糖体肽(nrp)”的含义包括“阳离子非核糖体肽(cnrp)”和“非阳离子非核糖体肽”,也就是说,阳离子非核糖体肽(cnrp)属于非核糖体肽(nrp)中的一类。
在本文中,术语“阳离子脂肽”是指具有n-脂肪酰基且包含阳离子氨基酸的多肽。
在本文中,在提及“线性肽段”或“线性阳离子片段”时,术语“线性”是指线性结构,可以具有或不具有侧链,但不具有氨基酸残基之间形成的环。术语“线性阳离子片段”是指cnrp或阳离子脂肽中所具有的呈线性结构的氨基酸链,由于具有带正电荷的氨基酸残基,因此该氨基酸链总体上带有阳离子。
在本文中,术语“环肽”是指通过两个氨基酸残基形成环状结构的肽段。
本发明所述的阳离子脂肽所含的氨基酸包括l-型氨基酸和d-型氨基酸。
优选地,所述线性肽段含有4-9个氨基酸。
所述线性肽段优选含有2-3个带正电荷的氨基酸。并且还优选地,所述带正电荷的氨基酸为orn或lys。
所述n端脂肪酰基可以包括直链和支链的烷酰基,该烷酰基可用下式表示:
还优选地,所述c端亲脂性环肽为由四个氨基酸或五个氨基酸形成的环肽。
在一个实施方案中,本发明所述的阳离子脂肽是通过下述方法筛选得到的。具体而言,所述方法包括:1)通过基因分析鉴定编码能够作为革兰氏阴性菌抗生素的cnrp的生物合成基因簇(bgc);2)通过细菌培养分离能够作为革兰氏阴性菌抗生素的cnrp。
所述步骤1可以包括以下步骤:
a)提供n条不同细菌的基因组序列,其中n≥2,并且通过生物信息学方法针对每条所述基因组序列分别独立地鉴定出编码非核糖体肽(nrp)的生物合成基因簇(bgc);
b)针对上一步所得的所有nrpbgc,通过生物信息学方法预测氨基酸构建块,从而判断每个nrpbgc的nrp生物合成能力,排除被预测为不具有生物合成能力的bgc;
c)从上一步所得的所有nrpbgc中排除编码赖氨酸/鸟氨酸n-单加氧酶的bgc;以及
d)针对上一步所得的所有nrpbgc,得到其各自所编码的nrp序列,根据以下规则对所有nrp序列进行筛选,排除不符合所述规则的nrpbgc,所述规则为:每条nrp序列中具有≥2个(优选≥3个)带正电荷的氨基酸,且序列长度≥6个(优选≥11个)氨基酸;
其中所述步骤b)至d)在步骤a)之后分别独立且不按序地进行。
所述步骤2)可以包括:对经过所述步骤1)筛选得到的每个nrpbgc各自所属的细菌进行培养,分离出符合步骤d)中所述规则的cnrp。然后,对所分离出的所述cnrp进行抗革兰氏阴性菌检测,从而验证所分离出的cnrp的抗生素活性,排除不具有抗革兰氏阴性菌性质的cnrp。所述抗革兰氏阴性菌检测包括本领域已知的各种抗菌性检验和/或耐药性检验。本发明发现,具有线性阳离子片段和c端疏水环肽对cnrp的抗菌活性至关重要。另外,n-酰化cnrp(又称为阳离子脂肽)对革兰氏阴性病原体引起的感染非常有效。
在上述方法中,所述不同细菌可以指不同的细菌物种,也可以指同一种细菌的不同菌株。
在步骤a)中,从基因组中鉴定nrp的生物合成基因簇(bgc)可以利用已知的计算机软件或算法进行,例如antismashv3.0.5。
在步骤b)中,可以利用已知的计算机软件或算法预测氨基酸构建块,从而判断bgc的nrp生物合成能力。
在本发明方法的步骤d)中,由nrpbgc得到其各自所编码的nrp序列的方法是已知的。在处理大数据时,可以采用已知的计算机软件或算法进行。
在步骤2)中,分离出所需的cnrp可以通过本领域已知的化学分析和分离技术进行。化学分析和分离的例子包括但不限于:超高效液相色谱法(uplc)、质谱法(ms)、串联质谱法(ms/ms)、核磁共振波谱法(nmr)和marfey型检测等。
本领域技术人员可以理解,本发明所述的阳离子脂肽可以通过已知的方法获得,例如通过人工合成或微生物发酵分离获得。
优选地,所述阳离子脂肽为上述brevicidine,其分子式为c74h106n18o17,化学结构为:4-甲基-己酰基-d-asn-d-tyr-d-trp-d-orn-orn-gly-d-orn-trp-thr-ile-gly-ser,其中ser12与thr9之间发生闭环,结构式如下式i所示:
还优选的是,所述阳离子脂肽为上述laterocidine,其分子式为c78h113n19o18,化学结构为:7-甲基-辛酰基-d-ser-d-tyr-d-trp-d-orn-orn-gly-d-orn-trp-thr-ile-asn-gly-gly,其中gly13与thr9之间发生闭环,结构式如下式ii所示:
本发明发现,这两种化合物具有最优的抗革兰氏阴性菌性质,特别适合开发为抗革兰氏阴性菌抗生素。
本发明还提供了一种药物组合物,包含根据本发明所述的阳离子脂肽。
所述药物组合物还可以包含药用辅料。所述药用辅料包括本领域已知常见的那些。
本领域技术人员可以理解,所述药物组合物可以根据实际情况包含所需量(例如治疗有效量)的所述阳离子脂肽。所述药物组合物还可以根据实际情况包含所需量的药用辅料。
本发明还提供了根据本发明所述的阳离子脂肽在制备革兰氏阴性菌抗生素药物中的应用。
以下通过例子的方式进一步解释或说明本发明的内容,但这些例子不应被理解为对本发明的保护范围的限制。
例子
方法
cnrp的全基因组挖掘
从ncbi基因组中检索出总共5,585份完整的细菌基因组,并利用单机版的antismashv3.0.5(https://antismash.secondarymetabolites.org/#!/start)进行bgc分析(参见文献33)。从所述完整的细菌基因组中跨越细菌的全部结构域,共鉴别出6,879份nrpbgc。为了深入了解细菌cnrp的生物合成能力,创建了一系列脚本,以便从antismash注释的文件中收集bgc信息(常规技术,故不赘述)。脚本costumenode.js是为了用于从nrp基因簇中的腺苷酰化(a)结构域提取预测的氨基酸构建块(aminoacidbuildingblock)。收集了antismash内含的minowa和stachelhaus编码算法预测的氨基酸,并统计了它们在每个nrp基因簇中的出现次数。在分析了所有完整的基因组之后,将菌属根据其含有两个以上带正电荷的氨基酸(即精氨酸、组氨酸、赖氨酸、鸟氨酸和2,4-二氨基丁酸)的cnrpbgc的丰度进行排序。选择来自位列前20名菌属的基因组草稿(draftgenomes)(1,810份)以及所有完整的基因组进行cnrp的全基因组挖掘。位列前20名的菌属如下:游动放线菌属(actinoplanes)、食烷菌属(alcanivorax)、节杆菌属(arthrobacter)、固氮杆菌属(azotobacter)、杆菌属(bacillus)、短芽孢杆菌属(brevibacillus)、柄球菌属(corallococcus)、蓝杆藻属(cyanothece)、拟胞囊菌属(kibdelosporangium)、粘球菌属(myxococcus)、亚硝化单胞菌属(nitrosomonas)、类芽孢杆菌属(paenibacillus)、潘多拉菌属(pandoraea)、光杆状菌属(photorhabdus)、多囊菌属(polyangium)、假单胞菌属(pseudomonas)、糖丝菌属(saccharothrix)、堆囊菌属(sorangium)、弧菌属(vibrio)和致病杆菌属(xenorhabdus)。利用酶功能主动-酶相似性工具(enzymefunctioninitiative-enzymesimilaritytool(efi-est);参见文献19)建立肽序列相似性网络。本研究将含有蛋白smcog1080(赖氨酸/鸟氨酸n-单加氧酶)的基因簇排除在外,因为它们的编码产物主要是具有n-甲酰基-n-oh-lys/orn残基的nrps-依赖性铁载体(参见文献18)。开发了脚本costumenode.js来询问预测的肽序列并将氨基酸转化为它们的缩写形式。基于结构相似性或生物合成来源,将非蛋白质源(non-proteinogenic)氨基酸归属到它们的蛋白质源对应物(表1)。针对每个基因簇,将两种算法(minowa和stachelhaus编码)预测的肽序列关联在一起,以提高预测的准确性。根据以下规则对所有基因簇的关联的肽序列进行筛选:带正电荷的氨基酸个数≥2,并且序列长度≥6。总共筛选出817个肽序列,用efi-est进行分析(e值=10e-3,分数=1)。通过cytoscape将最终得到的完整网络可视化(参见文献34)。特别指出的是,多粘菌素家族含有极高比例的dab和有限的氨基酸组成,这会导致efi-est中的e值不准确。因此,对多粘菌素家族(23bgc)分别采用blastp多重比对法进行人工筛选和分析,由此构建网络(e值<10e-3)。
表1.预测的氨基酸残基以及为它们分配的简写形式
通过antismash预测的蛋白质源和非蛋白质源的氨基酸都根据本表为其分配简写形式。基于结构相似性或生物合成来源,将非蛋白质源氨基酸归属到它们的蛋白质源对应物。
uplc-ms分析和化合物的分离
在代谢分析中,对所有溶解于100μlmeoh中的样品均采用与brukermicrotof-qii质谱仪(德国bremen市brukerdaltonicsgmbh公司)联用的超高效液相色谱(uplc)系统(watersacquity公司,配有watersbehc18反相uplc柱)进行分析。
在5个各装有1.0l改良胰蛋白胨大豆肉汤(缩写mtsb,30g/l胰蛋白胨大豆肉汤、20.0g/l淀粉、2.0g/lmgso4·7h2o、10.0g/lcaco3)的2.5升瓶中,将侧孢芽孢杆菌(b.laterosporus)dsm25在30℃下培养1天,以产生brevicidine。用
在10个各装有1.0lmtsb的2.5l瓶中,将侧孢芽孢杆菌(b.laterosporus)atcc9141在27℃下培养2天,以产生laterocidine(2)。用
化合物结构分析
在brukerav500波谱仪(500mhz,brukerdaltonicsgmbh公司)上,采用meoh-d4(1h-nmrmeoh-d4:δh=3.31ppm;meoh-d4:δc=49.00ppm)记录化合物1、2、4的1h、1h-1h-cosy、1h-13c-hsqc、以及1h-13c-hmbcnmr波谱。采用高级marfey法确定化合物1-2的氨基酸构型(参见文献35)。为了进一步确认化合物1-2的氨基酸构型,在25%的磷酸缓冲盐溶液(pbs,ph7.4,80μl)中,用100nmd-orn/lys特异性肽酶bogq36和100μm底物(1-2)在37℃下分别进行化合物1-2的d-orn/lys特异性水解,水解时间为2h。向所得样品中加入3体积的冷甲醇(240μl),然后在-80℃下孵育1h,以析出蛋白质。将所得样品在15,000rpm下离心10分钟,将上清液(2μl)注入上文所述的同一个uplc-ms系统中。采用半制备型rp-hplc柱,用含有0.1%三氟乙酸的25%mecn水溶液分别将化合物1和2的bogq水解产物纯化,得到化合物4-7和7-10。将纯化后的化合物1-2、4-10(0.1-0.2mg)在6mhcl中水解过夜,水解温度为120℃。将所得的各种溶液在干n2流下蒸发至干燥,将残留物溶解在100μl的水中并且分成两份。用5μl的nahco3(1m)和50μl的1-氟-2,4-二硝基苯基-5-l-亮氨酸酰胺(l-fdla)或d-fdla(1m)将每份样品在40℃下处理2h。用5μl的hcl(1m)将反应淬灭,并且用200μl的meoh稀释。采用uplc-ms分析,通过比较l-/dfdla衍生化样品来确定立体化学性质。
抗菌试验
根据clsi指南,通过肉汤微量稀释法来测定最低抑菌浓度(mic)。大部分菌种的试验培养基为mueller-hinton肉汤(mhb)。在96孔微量滴定板的孔中,将细菌培养过夜至前稳定期(earlystationaryphase),并在mhb中调节成每毫升5.0×105个菌落形成单位(c.f.u./ml),将其与不同浓度的测试化合物混合,并在37℃下孵育24h。通过检测595nm下的光密度(采用美国马萨诸塞州waltham市thermoscientific公司的multiskanfc多板光度计)来评价细胞生长情况,并将显示出没有细菌生长时的最低化合物浓度定义为mic。另外采用含有10%胎牛血清(gibco)、21mmmgcl、或1.0mgml-1lps(sigma-aldrich公司的源自大肠杆菌o55:b5的脂多糖)的mhb来测试它们的抑菌效果。
细菌生长动力学
将大肠杆菌(e.coli)top10的一个单独的新鲜菌落接种到mhb(5ml)中,在225rpm的摇动速度、28℃下培养过夜。在96孔板的孔中,用新鲜的mhb将细菌稀释至1x105c.f.u./ml,培养过夜至前对数期(od=0.15),并且用所关注的化合物加以处理。最终的抗生素浓度为1×mic至8×mic。然后将板子在30℃下孵育过夜,并且每30秒进行一次od600的测量。
时间-杀菌试验
将大肠杆菌top10或铜绿假单胞菌pao1细胞的过夜培养物(培养温度为37℃)在mhb中稀释至1:10,000,并在180rpm的搅拌条件下于37℃孵育2h(前指数期(earlyexponential))。然后,在室温并且不摇动的条件下,用10×mic的brevicidine(20μgml-1)、laterocidine(20μgml-1)或者多粘菌素b(10μgml-1)来攻击细菌。用未经处理的细胞样品作为阴性对照。每隔一段时间,取出10μl等分试样,在10,000g下离心1分钟,然后再悬浮于1.0ml的无菌pbs中。将10倍系列稀释的悬浮液涂于mhb琼脂平板上,并在37℃下孵育过夜。将菌落计数,并计算c.f.u./ml值。
细胞溶解试验
为了分析裂解情况,将300μl大肠杆菌培养物(od600=1.0)用10×mic的抗生素处理24h。实验一式三份。
耐药性研究
在37℃并且连续摇动的条件下,将大肠杆菌(escherichiacoli)atcc25922接种到mhb中过夜培养。将细胞在mhb中稀释10,000倍至约1x106c.f.u./ml。将10μl等分试样移至96孔板,该板容纳有90μl的brevicidine、laterocidine或环丙沙星的系列稀释液,终浓度分别为0.25x、0.5x、1x、1.5x、2x和4xmic。将带菌板在37℃并且不摇动的条件下孵育。24h后记录mic,从使用表现出可见性生长的第二高的抗生素浓度的培养物中取出1μl等分试样,将其在mhb中稀释1,000倍,以用于随后的试验。这个过程重复30天,通过上文所述的同样的抗菌试验确认最终的mic。实验一式四份,并且每天对产生最高耐药性的样品进行测量,以绘制曲线。
细胞毒性和溶血活性
采用人hela细胞进行试验。将90μl的1×105个/ml细胞置于96微孔板的孔内(37℃下培养)。12h后,加入10μl含有不同浓度(1、2、4、8、16、32、64、128μg/ml)的测试化合物的细胞培养基,并在37℃下孵育48h。然后,移出上清液,并向每个孔中加入20μl处于1xpbs中的mtt(2.5mgml-1)。在37℃下孵育3h后,向每个孔中加入80μl的二甲基亚砜(dmso),并且再孵育15分钟。然后用thermoscientificmultiskanfc多板光度计测量570nm下的吸光度。
采用从健康家兔的血液中新鲜分离出的兔红细胞进行溶血活性测定。向2%(v/v)的处于1×pbs内的红细胞中分别加入在0.5%dmso中的终浓度为128、64、32、16μgml-1的brevicidine和laterocidine。将细胞在37℃下孵育1h,并且在10,000g下离心5分钟。将上清液转移到96孔板中,并采用thermoscientificmultiskanfc多板光度计测量570nm波长下的吸光度。将该吸光度与采用10%tritonx-100处理的阳性对照的吸光度之比定义为溶血百分比。
通过itc确定lps结合作用
用美国马萨诸塞州northampton市microcal公司的mcs等温滴定量热仪(itc)在37℃下进行肽与lps结合的微量热法实验。通过悬浮、声波处理、以及在5℃和70℃之间进行温度循环,将lps制备成50μm的处于20mmhepes(ph7.0)缓冲液中的含水悬浮液。在使用前将该悬浮液在4℃下储存过夜。在同样的缓冲液中制备100μm肽溶液(brevicidine和多粘菌素b),所有溶液在37℃的真空条件下搅拌5分钟脱气后使用。在热平衡后,在37℃并且不断搅拌的条件下,每3分钟向含脂质的样品池中加一次肽溶液的5μl等分试样。实时记录滴定过程中热量的变化,并使用仪器配备的
细菌细胞的原子力显微镜成像
将100μl的在lb培养基中并处于对数期的大肠杆菌atcc25922细胞(od600=0.5)在37℃下、在没有或具有测试化合物(10×mic)的条件下孵育1.5h。然后在4℃下将所得样品在8000g下离心10分钟,将所得的小球用100μl无菌水洗涤两次。将细菌再悬浮于50μl的无菌水中,并将其施加到云母片上,在28℃下干燥过夜后成像。用带有在接触模式下工作的nanoscopeiii控制器的veecomulitmodeafm记录原子力显微图像。用美国veeco公司的nanoscopeanalysis软件v.1.40分析数据。在25℃下以1.0hz的速率和256个样本/线分辨率的条件进行扫描。采用bruker公司的nanoscope软件进行下游图像处理和分析。高度图像被平坦化以抵消细胞曲率的影响,而轮廓截面(topographicalsections)则用来生成表面纹理的二维重建。
小鼠大腿感染模型
用小鼠大腿感染模型(参见文献38)检测brevicidine和laterocidine的体内活性。动物研究是由台湾药学发现服务有限公司实施的,总体上根据动物福利标准指南(参见文献39)在经过aaalac认证的设施中进行,该协议经实验动物照护及使用委员会审查和批准。
每组采用5只无特定病原体的icr(cd-1)雌性小鼠,小鼠重22±2g(约5周龄)。通过两次腹腔内注射环磷酰胺对动物进行免疫抑制,第一次注射是在感染之前4天的时候(第-4天),剂量为150mg/kg,第二次注射是在感染之前1天的时候(第-1天),剂量为100mg/kg。在第0天时,在动物右大腿中肌内接种(0.1ml/大腿)大肠杆菌atcc25922悬浮液。然后,在2h和8h后皮下注射赋形剂(10%dmso、1%吐温80、0.9%nacl)和抗生素。由于化合物1和2的量有限,所以本研究中仅采用了一种浓度的化合物1(30mgkg-1)或化合物2(15mgkg-1)。在接种后26h的时候,对动物实施co2窒息安乐死,然后取下每只实验动物的右大腿肌肉。采用polytron匀化器,将取下的肌肉在5ml的pbs(ph7.4)中匀化。用0.1ml的匀化物进行连续十倍稀释,并涂在琼脂营养培养基上,以进行菌落计数(c.f.u./g)测定。
实施例1:筛选能够作为革兰氏阴性菌抗生素的阳离子非核糖体肽(cnrp)
对7,395份细菌基因组(其中5,585份为完整的基因组)进行了针对编码cnrp的生物合成基因簇(bgc)的生物信息学分析,共鉴定出8,567个编码带正电荷残基的非核糖体肽的bgc,其中有2,676个被推定为是含有两个以上带正电荷残基的cnrp。进一步对这些推定的cnrp进行bgc分析,从cnrp基因簇中的腺苷酰化(a)结构域提取预测的氨基酸构建块,以了解细菌cnrp的生物合成能力,淘汰被预测为不具有生物合成能力的bgc,进一步排除编码具有n-甲酰基-n-oh-lys/orn残基的肽(即铁载体(siderophores),参见文献18)或较短的肽的bgc,最终识别出817种cnrp(≥6个残基,具有≥2个带正电荷的残基)。它们极其多样化并且在细菌中广泛分布(图5)。所述推定的cnrp的长度为6至34不等,平均为9.7(中值为9),而其带正电荷的残基的百分比为6.3%至78.6%,平均为33.0%(中值为30.0%)(图6)。然后,利用序列相似性网络算法(参见文献19),基于所鉴定的cnrp肽序列对基因编码的cnrp进行分类,将它们的多样性、分布和发现状态可视化(图1)。由此可显而易见地看出,大部分cnrp还处于未被探索的状态,其中大部分主要存在于变形菌门(proteobacteria)和厚壁菌门(firmicutes)中。在所选的817种cnrp中,有495种来自假单胞菌属(pseudomonas),其中大多数尚没有任何被分离出的成员。
然后进行传统的基于发酵的分离过程,以发现具有杀死革兰氏阴性病原体的能力的cnrp(图4)。n-酰化cnrp又称为阳离子脂肽(包括多粘菌素和tridecaptin;参见文献20),其对革兰氏阴性病原体引起的感染非常有效,代表了一个越来越有吸引力的抗生素候选物家族(图7-8)。于是,后续筛选集中于具有三个以上带正电荷残基的那些推定的阳离子脂肽,这是因为带正电荷的残基使其能够与带负电荷的细胞膜发生静电相互作用,而疏水性酰基链则限定了膜裂解(membranolytic)和细胞穿透(cell-penetrating)性质(参见文献7)。虽然已熟知杆菌可产生cnrp抗生素(参见文献20),但选择这个类别中含有理想的bgc的侧孢芽孢杆菌(brevibacilluslaterosporus)dsm25、atcc9141和蜂房芽孢杆菌(paenibacillusalvei)dsm29来进行传统的基于发酵的探索(图2和图11)。基于基因组挖掘和代谢分析,从三种菌株培养物中分离出阳离子脂肽及其衍生物,并通过串联质谱法(ms/ms)、核磁共振波谱法(nmr)和marfey型检测对其进行结构表征。这些cnrp被命名为brevicidine(化合物1,分离自侧孢芽孢杆菌dsm25)、laterocidine(化合物2,分离自侧孢芽孢杆菌atcc9141)和paenibacterinb(化合物3,分离自蜂房芽孢杆菌dsm29),它们是新的环状缩酚酸肽(cyclicdepsipeptide),其具有n-酰化侧链、以及通过c端残基与苏氨酸(thr)残基缩合而形成的内酯环(图2和图9-11)。paenibacterinb是paenibacterin的新衍生物(图11),paenibacterin是一种与细菌外膜结合的杀菌性抗生素(参见文献21)。brevicidine和laterocidine是一类新的cnrp,其表现出三个关键的结构特征:疏水性的n端脂肪酰基链;具有三个带正电荷的鸟氨酸残基的线性阳离子片段;以及疏水性的四肽/五肽环(图2b)。据预测,brevicidine的生物合成是从n-酰化nrp的典型的启动模块开始的,其具有使第一个氨基酸与脂肪酸发生酰化的缩合启动结构域。根据共线性规则,12个氨基酸(包括带正电荷的残基d-orn4、l-orn5、d-orn7)被引入该线性肽中,在产物卸载过程中,由硫酯酶结构域催化最后的ser12与thr9之间发生闭环(图12)。laterocidine的生物合成途径遵循类似的生物合成组装线(图13)。
brevicidine、laterocidine及其一种衍生物(化合物4)的nmr数据分别如表2、3、4所示;brevicidine、laterocidine的结构及相应的nmr归属如下式v-vi所示;化合物4的关键1h-1hcosy与hmbc关联如式vii所示。
表2.brevicidine的nmr数据(d6-dmso)
表3.laterocidine的nmr数据(d6-dmso)
表4.化合物4的nmr数据(cd3od)
brevicidine、laterocidine和paenibacterinb及其衍生物(化合物1-11)的ms分析以进行化合物表征结果如下表5所示。
表5:通过ms分析进行化合物表征
brevicidine的1hnmr(dmso-d6,500mhz)、13c-nmr(dmso-d6,100mhz)、1h-1hcosy(dmso-d6,500mhz)、1h-13chsqc(dmso-d6,500mhz)、1h-13chmbc(dmso-d6,500mhz)、1h-1htocsy(dmso-d6,500mhz)分别如图19-24所示。
laterocidine(2)的1hnmr(dmso-d6,500mhz)、1h-1hcosy(dmso-d6,500mhz)、1h-13chsqc(dmso-d6,500mhz)、1h-13chmbc(dmso-d6,500mhz)、1h-1htocsy(dmso-d6,500mhz)分别如图25-29所示。
化合物4的1hnmr(cd3od,500mhz)、13nmr(cd3od,100mhz)、1h-1hcosy(cd3od,500mhz)、1h-13chsqc(cd3od,500mhz)、1h-13chmbc(cd3od,500mhz)分别如图30-34所示。
实施例2:抗菌试验
对所有分离出的化合物1-11(化合物4-11如图14所示)都进行抗eskape细菌(屎肠球菌(enterococcusfaecium)、金黄色葡萄球菌(staphylococcusaureus)、肺炎克雷伯氏菌(klebsiellapneumoniae)、鲍氏不动杆菌(acinetobacterbaumannii)、铜绿假单胞菌(pseudomonasaeruginosa)、以及肠杆菌物种(enterobacterspecies))的生物活性评估,大多数医院获得性感染都是由eskape细菌导致的(参见文献2)。brevicidine和laterocidine对革兰氏阴性菌(包括机会性致病菌,例如难以治疗的铜绿假单胞菌(pseudomonasaeruginosa)和耐粘菌素的大肠杆菌(colistin-resistante.coli))均表现出广泛的抗菌活性。最低抑菌浓度(mic)值在微摩尔范围内(1–16μgml-1;0.66–10.5μm)证明了其高效性。此外,在胎牛血清的存在下,它们的活性没有受到损害(表6)。由于化合物1和2含有非天然氨基酸如d-氨基酸、脂肪酸侧链和内酯环,这两种环状脂肽在蛋白水解上也是稳定的。初步的结构-活性关系研究表明,线性阳离子片段和疏水性内酯环这二者对其活性都是至关重要的。例如,缺失内酯环或阳离子侧链中任意一者都会使mic值增加8至32倍(化合物1与4-5相比,化合物2与9-11相比)(表7)。如大肠杆菌生长动力学、时间-杀菌试验(time-killassay)和细胞溶解试验(lysisassay)所显示,cnrp1和2对大肠杆菌都具有杀菌作用(图3a-c)。它们既不会引起红细胞的溶解,在最高达128μgml-1的浓度下也不会对人类系hela细胞显示出明显的毒性(图15),这些都表明了化合物1-2是革兰氏阴性菌抗生素的有力候选物。
表6.cnrp的抗微生物活性
a:大肠杆菌atcc25922携带有mcr-1。
表7.cnrp及其衍生物的抗菌活性
nd表示:>128μg/ml
“-”表示:实验未测
ec=大肠杆菌(escherichiacoli)atcc25922,pa=铜绿假单胞菌(pseudomonasaeruginosa)pao1,ab=鲍氏不动杆菌(acinetobacterbaumannii),kp=肺炎克雷伯氏菌(klebsiellapneumoniae)nrrl-b-408,en=阴沟肠杆菌(enterobactercloacae)nrrl-b-413,bs=枯草芽孢杆菌(b.subtilis)168,mrsa=耐甲氧西林的金黄色葡萄球菌(staphylococcusaureus)atcc43300,ca=白色念珠菌(candidaalbicans),sc=酿酒酵母(saccharomycescerevisiae)vl-48,nd>64μgml-1。
实施例3:进一步的生物活性评价
为了探索化合物1(brevicidine)和2(laterocidine)的作用方式,起初尝试了针对大肠杆菌atcc25922耐药突变体来进行鉴定。令人感兴趣的是,在亚抑菌浓度的化合物1或2中任意一者的存在下,在30天的连续系列传代过程中,在大肠杆菌中没有观察到耐药性的发展(图3d)。相反,在暴露于环丙沙星的情况中,大肠杆菌对环丙沙星的耐药性迅速发展(图3d)。当将细胞在外源脂多糖(lps)或高浓度mg2+的存在下培养时,如预期一样,化合物1和2都没有表现出抗菌作用(表6),这表明了这些阳离子肽是通过与lps结合来破坏外膜的完整性。这一推论得到了原子力显微镜(afm)和等温滴定量热法(itc)分析的进一步支持。afm分析显示,当将大肠杆菌atcc25922和铜绿假单胞菌pao1用化合物1或2处理之后,处理过的细胞失去了它们规则的棒状形态,变得粗糙而不平坦(图3e以及图16-17)。用所述化合物处理过的细胞,其形态受到了广泛的破坏,表现为幅度在大约30nm左右的起伏,而未经处理的大肠杆菌细胞则保持均匀一致。
实际上,化合物1在体外表现出对lps的高亲和力(解离常数kd为6.5μm),类似于多粘菌素b(图18)。值得注意的是,brevicidine和laterocidine对携带mcr-1(参见文献22)的耐粘菌素大肠杆菌atcc25922都依然具有抗菌活性(表6),这表明了这两种cnrp的作用机制不依赖于磷酸乙醇胺(phosphoethanolamine)对脂质a的修饰(参见文献22)。
大肠杆菌细胞的生长动力学实验显示,在暴露于化合物1或2的情况下,开始的50分钟细胞以不断降低的速率生长着,而在那之后细胞数量则开始以稳定的速率下降(图3b)。这些数据不同于多粘菌素的数据,这是因为多粘菌素会在细菌的细胞膜中造成大的非特异性的孔,所以当细菌暴露于多粘菌素时,多粘菌素通常会在几分钟内表现出杀菌的作用(参见文献23-24)。在时间-杀菌试验中,当大肠杆菌暴露于10倍mic浓度的化合物1或2中时,15分钟后细胞的活力显示有略微的下降,在30分钟后则表现出了显著的下降。而多粘菌素b作用更加快速,5分钟的暴露便能观测到活细胞种群的显著减少,30分钟内所有细胞均被杀死。这些结果表明了化合物1和2是细胞膜靶向的杀菌肽,但是它们并不是像多粘菌素b那样通过普通的细胞膜裂解机制起作用。这两种cnrp对革兰氏阴性菌的选择性(表6)进一步支持了这一观点,因为通过普通的细胞裂解机制发挥作用的杀菌剂对革兰氏阳性菌也会产生杀菌作用(参见文献23)。在用两倍mic浓度的化合物1和2处理的情况下,没有观测到明显的细胞膜破坏现象,这表明了这两种cnrp存在潜在的细胞内靶点。
考虑到化合物1和2的低耐药性风险以及具有吸引力的作用模式,接下来在小鼠大腿模型中进行了动物药效研究。用1.50×106c.f.u.大肠杆菌atcc25922对小鼠进行肌内感染。在感染后两小时,以两个剂量皮下注射brevicidine(30mgkg-1)或laterocidine(15mgkg-1)。作为阳性对照,将左氧氟沙星(mic=0.032μgml-1)以3.0mgkg-1的剂量给药。化合物1和2在中性粒细胞减少的小鼠大腿感染模型中对大肠杆菌表现出了有效的杀菌作用。特别是,化合物1对大肠杆菌感染的小鼠非常有效,在24小时的时候,相较于媒介物对照(vehiclecontrol),化合物1导致大腿内的菌落数降低5log10c.f.u.(图3f)。
结论:
多重耐药革兰氏阴性病原体(特别是耐粘菌素病原体)所引起的感染发病率不断上升,威胁着世界各地的医疗保健工作。在本发明中,显示了全基因组挖掘在系统研究细菌cnrp中的应用,并且为基于基因组测序的cnrp的定向发现提供了一个平台。本发明发现的两种cnrp对革兰氏阴性病原体(特别是耐粘菌素的病原体)的抗菌效果成功地得到证明,这无疑将使细菌cnrp作为革兰氏阴性菌抗生素的新来源而引人注目。本发明通过cnrp的全基因组挖掘鉴别出有效的抗革兰氏阴性菌的抗生素,证实了这是一种用于找到合适的药物候选物来对抗已产生抗生素耐药性的革兰氏阴性病原体的强有力的策略。
参考文献:
1.laxminarayan,r.etal.antibioticresistance-theneedforglobalsolutions.lancetinfect.dis.13,1057-1098(2013).
2.boucher,h.w.etal.badbugs,nodrugs:noeskape!anupdatefromtheinfectiousdiseasessocietyofamerica.clin.infect.dis.48,1-12(2009).
3.fischbach,m.a.&walsh,c.t.antibioticsforemergingpathogens.science325,1089-1093(2009).
4.lewis,k.platformsforantibioticdiscovery.nat.rev.drugdiscov.12,371-387(2013).
5.yeung,a.t.y.,gellatly,s.l.&hancock,r.e.w.multifunctionalcationichostdefencepeptidesandtheirclinicalapplications.cell.mol.lifesci.68,2161-2176(2011).
6.hancock,r.e.w.&lehrer,r.cationicpeptides:anewsourceofantibiotics.trendsbiotechnol.16,82-88(1998).
7.epand,r.m.&vogel,h.j.diversityofantimicrobialpeptidesandtheirmechanismsofaction.biochim.biophys.acta,biomembr.1462,11-28(1999).
8.marr,a.k.,gooderham,w.j.&hancock,r.e.w.antibacterialpeptidesfortherapeuticuse:obstaclesandrealisticoutlook.curr.opin.pharmacol.6,468-472(2006).
9.hancock,r.e.w.&sahl,h.g.antimicrobialandhost-defensepeptidesasnewanti-infectivetherapeuticstrategies.nat.biotechnol.24,1551-1557(2006).
10.blin,k.,medema,m.h.,kottmann,r.,lee,s.y.&weber,t.theantismashdatabase,acomprehensivedatabaseofmicrobialsecondarymetabolitebiosyntheticgeneclusters.nucleicacidsres.45,d555-d559(2017).
11.kosikowska,p.&lesner,a.antimicrobialpeptides(amps)asdrugcandidates:apatentreview(2003-2015).expertopin.ther.pat.26,689-702(2016).
12.fjell,c.d.,hiss,j.a.,hancock,r.e.w.&schneider,g.designingantimicrobialpeptides:formfollowsfunction.nat.rev.drugdiscov.11,37-51(2012).
13.mahlapuu,m.,hakansson,j.,ringstad,l.&bjorn,c.antimicrobialpeptides:anemergingcategoryoftherapeuticagents.front.cell.infect.microbiol.6,194(2016).
14.law,v.etal.drugbank4.0:sheddingnewlightondrugmetabolism.nucleicacidsres.42,d1091-d1097(2014).
15.gordon,y.j.,romanowski,e.g.&mcdermott,a.m.areviewofantimicrobialpeptidesandtheirtherapeuticpotentialasanti-infectivedrugs.curr.eyeres.30,505-515(2005).
16.clardy,j.,fischbach,m.a.&walsh,c.t.newantibioticsfrombacterialnaturalproducts.nat.biotechnol.24,1541-1550(2006).
17.schwarzer,d.,finking,r.&marahiel,m.a.nonribosomalpeptides:fromgenestoproducts.nat.prod.rep.20,275-287(2003).
18.crosa,j.h.&walsh,c.t.geneticsandassemblylineenzymologyofsiderophorebiosynthesisinbacteria.microbiol.mol.biol.rev.66,223-249(2002).
19.gerlt,j.a.etal.enzymefunctioninitiative-enzymesimilaritytool(efi-est):awebtoolforgeneratingproteinsequencesimilaritynetworks.biochim.biophys.acta,proteinsproteomics1854,1019-1037(2015).
20.cochrane,s.a.&vederas,j.c.lipopeptidesfrombacillusandpaenibacillusspp.:agoldmineofantibioticcandidates.med.res.rev.36,4-31(2016).
21.huang,e.&yousef,a.e.thelipopeptideantibioticpaenibacterinbindstothebacterialoutermembraneandexertsbactericidalactivitythroughcytoplasmicmembranedamage.appl.environ.microbiol.80,2700-2704(2014).
22.liu,y.y.etal.emergenceofplasmid-mediatedcolistinresistancemechanismmcr-1inanimalsandhumanbeingsinchina:amicrobiologicalandmolecularbiologicalstudy.lancetinfect.dis.16,161-168(2016).
23.cochrane,s.a.etal.antimicrobiallipopeptidetridecaptina1selectivelybindstogram-negativelipidii.proc.natl.acad.sci.u.s.a.113,11561-11566(2016).
24.trimble,m.j.,mlynarcik,p.,kolar,m.&hancock,r.e.w.polymyxin:alternativemechanismsofactionandresistance.coldspringharb.perspect.med.6(2016).
25.chu,j.etal.discoveryofmrsaactiveantibioticsusingprimarysequencefromthehumanmicrobiome.nat.chem.biol.12,1004-1006(2016).
26.gross,h.&loper,j.e.genomicsofsecondarymetaboliteproductionbypseudomonasspp.nat.prod.rep.26,1408-1446(2009).
27.bode,h.b.entomopathogenicbacteriaasasourceofsecondarymetabolites.curr.opin.chem.biol.13,224-230(2009).
28.moon,s.h.etal.novellinearlipopeptidepaenipeptinswithpotentialforeradicatingbiofilmsandsensitizinggram-negativebacteriatorifampicinandclarithromycin.j.med.chem.60,9630-9640(2017).
29.vaara,m.etal.anovelpolymyxinderivativethatlacksthefattyacidtailandcarriesonlythreepositivechargeshasstrongsynergismwithagentsexcludedbytheintactoutermembrane.antimicrob.agentschemother.54,3341-3346(2010).
30.forst,s.,dowds,b.,boemare,n.&stackebrandt,e.xenorhabdusandphotorhabdusspp.:bugsthatkillbugs.annu.rev.microbiol.51,47-72(1997).
31.fuchs,s.w.etal.neutrallossfragmentationpatternbasedscreeningforarginine-richnaturalproductsinxenorhabdusandphotorhabdus.anal.chem.84,6948-6955(2012).
32.stokes,j.m.etal.pentamidinesensitizesgram-negativepathogenstoantibioticsandovercomesacquiredcolistinresistance.nat.microbial.2,17028(2017).
33.weber,t.etal.antismash3.0-acomprehensiveresourceforthegenomeminingofbiosyntheticgeneclusters.nucleicacidsres.43,w237-w243(2015).
34.smoot,m.e.,ono,k.,ruscheinski,j.,wang,p.l.&ideker,t.cytoscape2.8:newfeaturesfordataintegrationandnetworkvisualization.bioinformatics27,431-432(2010).
35.fujii,k.,ikai,y.,oka,h.,suzuki,m.&harada,k.anonempiricalmethodusinglc/msfordeterminationoftheabsoluteconfigurationofconstituentaminoacidsinapeptide:combinationofmarfey'smethodwithmassspectrometryanditspracticalapplication.anal.chem.69,5146-5151(1997).
36.lietal.resistancetononribosomalpeptideantibioticsmediatedbyd-stereospecificpeptidase.nat.chem.biol.accepted.
37.odds,f.c.synergy,antagonism,andwhatthechequerboardputsbetweenthem.j.antimicrob.chemother.52,1(2003).
38.deryke,c.a.,banevicius,m.a.,fan,h.w.,&nicolau,d.p.(2007).bactericidalactivitiesofmeropenemandertapenemagainstextended-spectrum-β-lactamase-producingescherichiacoliandklebsiellapneumoniaeinaneutropenicmousethighmodel.antimicrob.agentschemother.51(4),1481-1486.
39.nationalresearchcouncil.guideforthecareanduseoflaboratoryanimals.nationalacademiespress(2010).
- 还没有人留言评论。精彩留言会获得点赞!