一种组蛋白脱乙酰化酶基因HDA19的CRISPR_Cas9载体构建及其应用的制作方法
本发明涉及红麻逆境胁迫调控及分子育种领域,特别是指一种组蛋白脱乙酰化酶基因hda19的crispr_cas9载体构建及其应用。
背景技术:
表观遗传(epigenetic)是指dna序列不发生变化,但基因表达改变引起的表型变异却发生了可遗传的改变。这种改变并不依赖于dna序列的改变,而是dna与染色体上的修饰发生了改变,且这种修饰改变可以通过减数分裂和有丝分裂稳定遗传给后代。目前,表观遗传学中研究较多的有:dna甲基化、组蛋白修饰、非编码rna调控及染色质重塑等,它们在植物生长发育、应对生物和非生物胁迫以及适应环境变化中发挥着极其重要的作用。蛋白质翻译后修饰(post-translationalmodification,ptm)是指蛋白质翻译后的化学修饰,几乎参与了细胞所有的正常生命活动过程,并发挥着十分重要的调控作用,蛋白质乙酰化是一种重要的蛋白质翻译后修饰,尤其是组蛋白乙酰化修饰在基因表达调控方面扮演重要角色,组蛋白通过组蛋白乙酰转移酶(hats)促进基因表达,而组蛋白脱乙酰化酶(hdacs)抑制基因表达。研究表明,组蛋白乙酰化和脱乙酰化主要参与植物的生长发育,其中包括根发育、花发育,配子体发育及器官生长过程中细胞的增殖等;且也参与植物应对外界环境变化,如光信号、盐胁迫、冷害、热胁迫、aba信号途径以及一些其他的激素信号途径。
红麻具有耐盐碱、耐旱、纤维产量高等特性,可用于盐渍土的修复,是极具潜力的耐盐、脱盐和高产的经济作物。组蛋白脱乙酰化酶基因hda19在植物的生长发育过程中起到非常重要的作用,拟南芥中hda19蛋白参与多种生物过程,包括调控盐胁迫、开花时间、干旱响应,种子发育及休眠,花发育,下胚轴的生长,乙烯、ja、sa通路,叶片形态建成等。组蛋白乙酰化是一种重要的表观遗传修饰,在应激反应中调节基因表达活性。到目前为止,还没有关于红麻非生物胁迫机制中组蛋白乙酰化修饰的报道。
为探索组蛋白乙酰化修饰与红麻生长发育及红麻细胞质雄性不育发生的关系,克隆红麻组蛋白脱乙酰化酶基因hda19,构建其pylcrispr-cas9pubi-h-hda19载体并转化红麻来对其基因的功能进行研究,可为组蛋白乙酰化修饰调控红麻雄性不育发生的分子机制研究提供理论依据,进一步丰富红麻细胞质雄性不育机理的研究,为将来利用工程技术创制雄性不育材料,更好的开发红麻价值奠定研究基础。
技术实现要素:
本发明提出一种组蛋白脱乙酰化酶基因crispr_cas9载体的构建及其应用,解决了红麻组蛋白脱乙酰化酶基因crispr_cas9载体的构建及组蛋白乙酰化修饰对逆境胁迫调控及雄性不育影响的研究中的问题。
本发明的技术方案是这样实现的:
一种组蛋白脱乙酰化酶基因crispr_cas9载体,所述载体为在载体pylcrispr-cas9pubi-h上插入红麻hda19基因三个靶点的t1-sgrna表达盒、t2-sgrna表达盒及t3-sgrna表达盒。
所述t1-sgrna表达盒位于tnos元件和t2-sgrna表达盒之间,t2-sgrna表达盒位于t1-sgrna表达盒和t3-sgrna表达盒之间,t3-sgrna表达盒位于t2-sgrna表达盒和sgrnascaffold元件之间。
所述tnos元件、t1-sgrna表达盒、t2-sgrna表达盒及t3-sgrna表达盒和sgrnascaffold元件均为载体pylcrispr-cas9pubi-h-hda19上的结构。
所述t1靶点序列序列如seqidno:15所示;t2靶点序列如seqidno:16所示;t3靶点序列如seqidno:17所示。
所述的组蛋白脱乙酰化酶基因crispr_cas9载体的构建方法,步骤如下:
(1)靶点选择及靶点接头设计:根据红麻hda19基因的序列选择靶点序列t1、t2和t3,并根据atu3bsgrna、atu6-29sgrna和atu6-1sgrna表达盒中间载体经bsai酶切后产生的粘性末端序列以及g为转录起始位点的原则,设计3个靶点对应的正反向引物对hda19-s1/hda19-a1、正反向引物对hda19-s2/hda19-a2和正反向引物对hda19-s3/hda19-a3;
(2)靶点接头制备:将合成的靶点正反向引物分别用ddh2o溶解成10μm母液,每个靶点取正向、反向引物各10μl,加入到80μl的ddh2o中,于90℃,30s后室温自然冷却,分别形成3个带有粘性末端的靶点接头;
(3)靶点接头与sgrna表达盒连接:将t1、t2和t3靶点接头分别于与bsai酶切后的atu3bsgrna表达盒中间载体、atu6-29sgrna表达盒中间载体和atu6-1sgrna表达盒中间载体连接,分别得到三个靶点接头与sgrna表达盒连接的产物;
(4)靶标sgrna表达盒扩增:
第一轮反应:以连接产物为模板进行扩增,每个靶点分两个独立的反应,t1靶点分别以正反引物对u-f/hda19a1和正反引物对hda19-s1/gr-r作为引物进行扩增,回收产物ⅰ、产物ⅱ,t2靶点分别以正反引物对u-f/hda19-a2和正反引物对hda19-s2/gr-r作为引物进行扩增,回收产物ⅲ、产物ⅳ,t3靶点分别以正反引物对u-f/hda19-a3和正反引物对hda19-s3/gr-r作为引物进行扩增,回收产物ⅴ、产物ⅵ;
第二轮反应:分别取各靶点的两个回收产物2μl混合均匀,稀释10倍后,得产物混合模板,t1靶点产物混合模板以引物对b1/b1'为引物、t2靶点产物混合模板以引物对b2/b2'为引物、t3靶点产物混合模板以引物对b3/bl为引物,进行扩增,分别回收产物得到t1-sgrna表达盒片段、t2-sgrna表达盒片段及t3-sgrna表达盒片段,并测量浓度;
(5)靶标sgrna表达盒与pylcrispr-cas9pubi-h载体的连接:将t1-sgrna表达盒片段、t2-sgrna表达盒片段及t3-sgrna表达盒片段按每表达盒15ng的量混合得sgrna表达盒混合物,然后把sgrna表达盒混合物与经bsai酶切的pylcrispr-cas9pubi-h载体进行连接后得到组蛋白脱乙酰化酶基因crispr_cas9载体pylcrispr-cas9pubi-h-hda19。
所述步骤(1)中,正反向引物对hda19-s1/hda19-a1序列如seqidno:1、seqidno:2所示,正反向引物对hda19-s2/hda19-a2序列如seqidno:3、seqidno:4所示,正反向引物对hda19-s3/hda19-a3序列如seqidno:5、seqidno:6所示。
所述步骤(4)中,引物u-f序列如seqidno:7所示,引物gr-r序列如seqidno:8所示,引物对b1/b1'序列如seqidno:9、seqidno:10所示,引物对b2/b2'序列如seqidno:11、seqidno:12所示,引物对b3/bl序列如seqidno:13、seqidno:14所示。
所述组蛋白脱乙酰化酶基因crispr_cas9载体,通过针刺—真空渗透辅助农杆菌介导红麻种子的遗传转化方法制备转基因红麻植株,研究组蛋白乙酰化修饰调控红麻逆境胁迫及雄性不育发生的分子机制。
本发明的有益效果在于:
(1)本申请发明人在研究中发现红麻hda19的亚细胞定位结果(图9)表明其不仅分布在细胞核中,还分布在细胞质中,说明红麻hda19不仅在细胞核中对组蛋白起作用,还可能对核外多种蛋白质起到重要的催化作用,预示着hda19功能的广泛性,本课题组致力于研究与红麻雄性不育相关的基因,而与雄性不育相关的基因,有不少是在细胞质中起作用的,同一个基因在细胞核内和在细胞质内可能发挥着截然不同的作用;细胞质雄性不育(cytoplasmicmalesterility,cms)表现为母体遗传、花粉败育和雌蕊正常,高纤维产量和较强抗性是红麻育种和杂种优势利用的主要目标,cms是红麻育种和杂种优势利用的主要手段之一,然而红麻cms不育机理尚未完全明确。为探索红麻hda19基因与cms发生的关系,发明人克隆红麻hda19基因并构建其crispr_cas9载体后转化红麻来对其基因的功能进行研究,可为组蛋白乙酰化修饰调控红麻雄性不育发生的分子机制研究提供理论依据,进一步丰富红麻细胞质雄性不育机理的研究,为利用工程技术创制雄性不育材料,更好的开发红麻价值提供可行性依据。
(2)本发明根据crispr_cas9技术设计靶位点的原则,把三个靶位点设计在红麻hda19基因的三个外显子上,成功构建了pylcrispr-cas9pubi-h-hda19载体,为研究hda19基因在红麻雄性不育发生中的作用以及对盐和干旱等逆境胁迫的调控机理奠定了基础。本发明还摸索了一整套红麻遗传转化方法,能够将pylcrispr-cas9pubi-h-hda19载体成功转化红麻并在后代中稳定遗传。目前,在红麻基因功能验证及分子育种中,由于其遗传转化体系尚未建立,造成很多基因的功能无法验证;虽然采用花粉管通道法来转化红麻可以弥补由于红麻再生体系造成的缺陷,但是花粉管通道法的转化机理不清楚很难得到国际同行的认可,本发明中的遗传转化方法的建立为研究红麻基因的功能作出实质性的贡献。
(3)本申请发明人前期对hda19基因的qrt-pcr研究结果表明,红麻hda19基因的表达量受到盐和干旱处理的显著诱导,表明其参与了盐和干旱抗性基因的表观遗传调控。因此,红麻组蛋白脱乙酰化酶基因hda19的crispr_cas9载体的构建及其应用为进一步研究hda19基因的功能奠定了基础,也为从基因表达方面研究红麻组蛋白脱乙酰化酶hda19对盐和干旱逆境胁迫的调控,从而进一步解析红麻对逆境胁迫适应的机理奠定了基础。
附图说明
为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
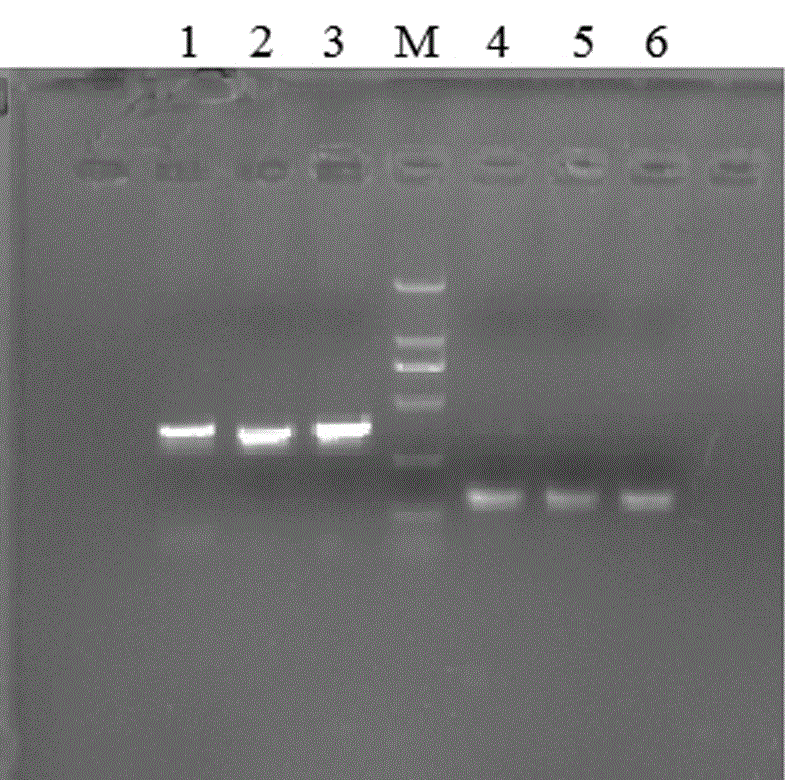
图1为hda19的t1、t2及t3-sgrna表达盒目标片段第一轮扩增结果,其中,泳道1:u-f/hda19-a1引物对扩增片段、泳道2:u-f/hda19-a2引物对扩增片段、泳道3:u-f/hda19-a3引物对扩增片段、泳道4:hda19-s1/gr-r引物对扩增片段、泳道5:hda19-s2/gr-r引物对扩增片段、泳道6:hda19-s3/gr-r引物对扩增片段、泳道m:markerdl2000。
图2为hda19的t1、t2及t3-sgrna表达盒目标片段第二轮扩增结果,其中,泳道1:t1-sgrna表达盒片段、泳道2:t2-sgrna表达盒片段、泳道3:t3-sgrna表达盒片段、泳道m:markerdl2000。
图3为载体pylcrispr-cas9pubi-h-hda19的结构示意图。
图4为红麻遗传转化过程示意图。
图5为载体pylcrispr-cas9pubi-h-hda19转化后幼苗提取dna进行pcr鉴定的电泳结果图,其中,泳道m:markerdl5000、泳道1:阳性对照,以载体为模板进行pcr扩增、泳道3和5:阴性对照,以未转化红麻植株dna进行pcr扩增、泳道2、4、6:阳性转化株。
图6为阳性植株t1代及t2代的形态图。
图7为阳性植株t1代植株提取dna后pcr鉴定的电泳结果图,其中,泳道m:markerdl5000、泳道1:阴性对照,以未转化红麻植株dna进行pcr扩增、泳道2:阳性对照,以载体为模板进行pcr扩增、泳道3至8:阳性转化株。
图8为阳性植株t2代植株提取dna后pcr鉴定的电泳结果,其中,泳道m:markerdl5000、泳道1:阴性对照,以未转化红麻植株dna进行pcr扩增、泳道2:阳性对照,以载体为模板进行pcr扩增、泳道3至11:阳性转化株。
图9为红麻hda19的亚细胞定位图。
具体实施方式
下面将结合本发明实施例,对本发明的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有付出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
实施例1
一、植物材料:红麻722b,由广西大学周瑞阳教授提供。
二、菌种:农杆菌eha105,pylcrispr-cas9pubi-h载体,atu3bsgrna、atu6-29sgrna和atu6-1sgrna表达盒中间载体(由华南农业大学刘耀光教授提供)。
三、药品:胰蛋白脉、琼脂粉、酵母提取物、ms培养基等试剂购自北京酷来博科技有限公司;taqplusdna聚合酶、乙酰丁香酮、卡那霉素、头孢霉素、利福平、硫酸链霉素、表面活性剂silwetl-77等购试剂购自生工生物工程(上海)股份有限公司;transstartfastpfudnapolymerase、transstarttaqdnapolymerase、peasy-bluntcloningkit购至北京全式金生物技术有限公司;fastpuregeldnaextractionminikit及高保真酶2×phantamaxmastermix购至南京诺唯赞生物技术有限公司;eha105农杆菌电转感受态细胞购至北京博迈德基因技术有限公司;bsai购至newenglandbiolabs公司。
构建pylcrispr-cas9pubi-h-hda19载体并利用针刺—真空渗透辅助农杆菌介导红麻种子遗传转化的方法,将其转化到红麻722b,步骤如下:
1、红麻hda19基因序列的扩增
根据本实验室对红麻转录组的高通量测序结果设计引物hda19-f序列如seqidno:19所示和hda19-r序列如seqidno:20所示。采用高保真酶2×phantamaxmastermix,以提取的红麻722b基因组dna为模板扩增红麻hda19基因序列。扩增程序为:95℃预变性3min;95℃15s,58℃15s,72℃90s,35个循环;72℃延伸5min。pcr产物纯化回收后连到peasy-blunt克隆载体上,转化到trans5αchemicallycompetentcell中,挑选阳性克隆测序。
、pylcrispr-cas9pubi-h-hda19载体的构建
载体系统包含了载体pylcrispr-cas9pubi-h和针对t1靶点、t2靶点和t3靶点的atu3bsgrna、atu6-29sgrna和atu6-1sgrna表达盒中间载体。
(1)靶点选择及靶点接头设计
根据红麻hda19基因见seqidno:21所示的序列、cg含量和靶点位置,选择合适的靶点序列t1序列如seqidno:15所示,t2序列如seqidno:16所示和t3序列如seqidno:17所示,其中靶点序列的后三个碱基为前间区序列邻近基序位点,根据atu3bsgrna、atu6-29sgrna和atu6-1sgrna表达盒中间载体经bsai酶切后产生的粘性末端序列以及g为转录起始位点的原则,设计3个靶点对应的正反向引物对hda19-s1/hda19-a1序列如seqidno:1、seqidno:2所示,正反向引物对hda19-s2/hda19-a2序列如seqidno:3、seqidno:4所示,正反向引物对hda19-s3/hda19-a3序列如seqidno:5、seqidno:6所示;
(2)靶点接头制备
将合成的靶点正反向引物用ddh2o溶解成10μm母液,t1靶点、t2靶点和t3靶点分别取正向s和反向a引物10μl,加入到80μl的ddh2o中,90℃,30s后室温自然冷却,分别形成3个带有粘性末端的靶点接头。
(3)靶点接头与sgrna表达盒连接
t1靶点、t2靶点和t3靶点的靶点接头分别与bsai酶切过的sgrna表达盒中间载体atu3bsgrna、atu6-29sgrna和atu6-1sgrna连接,分别得到三个靶点接头与sgrna表达盒连接的产物,反应体系及程序如下表:
(4)靶标sgrna表达盒扩增
第一轮反应:以连接产物为模板进行扩增,每一个靶点分2个独立的反应,靶点1:引物对u-f/hda19a1(反应1)和hda19-s1/gr-r(反应2);靶点2:u-f/hda19-a2(反应1)和hda19-s2/gr-r(反应2),靶点3:u-f/hda19-a3(反应1)和hda19-s3/gr-r(反应2),其中引物u-f序列如seqidno:7所示,引物gr-r序列如seqidno:8所示。
反应1体系及程序如下:
反应2体系及程序如下:
反应结束后,取5μl产物用2%琼脂凝胶电泳检测,结果见图1。
第二轮pcr用sgrna表达盒中间载体上的位置特异性引物扩增,每一个靶点各取第一轮反应1和2中pcr产物2μl,加入16μlddh2o(相当于稀释10倍),以此混合物为模板分别用位置特异性引物对b1/b1'和b2/b2'和b3/bl扩增获得相应的t1-sgrna表达盒片段、t2-sgrna表达盒片段及t3-sgrna表达盒片段。引物对b1/b1'序列如seqidno:9、seqidno:10所示,引物对b2/b2'序列如seqidno:11、seqidno:12所示,引物对b3/bl序列如seqidno:13、seqidno:14所示。第二轮反应体系及程序如下:
pcr产物电泳后经fastpuregeldnaextractionminikit胶纯化回收试剂盒纯化后测量浓度,电泳结果见图2。
(5)靶标sgrna表达盒与pylcrispr-cas9pubi-h载体的连接
将t1-sgrna表达盒片段、t2-sgrna表达盒片段及t3-sgrna表达盒片段按每表达盒15ng的量混合得sgrna表达盒混合物,然后把sgrna表达盒混合物与经bsai酶切的pylcrispr-cas9pubi-h载体进行连接后得到组蛋白脱乙酰化酶基因crispr_cas9载体pylcrispr-cas9pubi-h-hda19。反应体系及程序如下:
取6μl反应产物转化大肠杆菌,涂布在含kan(100mg/ml)的lb平板上,37℃培养12-18h后挑选阳性菌落,菌液经测序验证后提取质粒再进行酶切验证,将测序及酶切验证正确的表达载体质粒结构如图3所示,转入农杆菌eha105感受态细胞。
实施例2
1、转化用农杆菌菌液的制备
将-80℃保存的含有pylcrispr-cas9pubi-h-hda19载体的农杆菌菌液在含有kan(100mg/ml)、rif(50mg/ml)及str(25mg/ml)的lb平板上划线,28℃倒置暗培养。从平板上挑取单菌落加入1.5ml含有kan(100mg/ml)、rif(50mg/ml)及str(25mg/ml)的lb液体培养基中28℃,220rpm震荡培养过夜,第二天将其加入到150ml含相应抗生素的lb液体培养基中继续培养。将培养的od600达到对数生长期的农杆菌,5000rmp离心10min收集菌液,弃上清,用侵染培养基(以1/2ms为基本培养基,添加乙酰丁香酮100mg/l和表面活性剂silwetl-770.04%-0.06%,ph5.8-6.0)重悬,使最后的od600达到0.6-0.8。
、种子预处理
挑选籽粒饱满的红麻种子,先用2%的次氯酸钠消毒5-10min,再用无菌水冲洗4-5次后放在40℃无菌水浸泡30-40min,然后放到铺有2层滤纸的培养皿(φ9cm)里暗培养18-24h,见图4。
、针刺
在无菌的超净台中,用1ml(针头φ0.45mm)规格的一次性无菌注射器蘸取农杆菌悬浮菌液,小心的将针头刺向红麻种子的上胚轴,每粒种子刺2-3次,每次针刺前先蘸取菌液,针刺的深度约1mm,注意控制针刺的力度,不要损伤胚芽顶端的分生组织。
、农杆菌侵染
将针刺好的种子放在盛悬浮好的菌液的大培养皿(φ12cm)中,将培养皿放在真空压缩机中,抽真空负压30-50kpa处理10-15min,放气2-5min,再次负压30-50kpa处理2-5min。抽真空结束后,将培养皿置于摇床上28℃,60-80rpm摇30min。
、共培养
取出摇床里侵染的种子,用无菌滤纸吸干红麻种子表面的菌液,放到铺有2层滤纸盛有1/2ms培养基的培养皿(φ9cm)中,28℃黑暗条件下共培养3天。
、移栽
将暗培养后的种子取出,用无菌水冲洗3-4次后,用无菌滤纸吸干红麻种子表面的水分,浸泡在1000mg/l的头孢霉素中1h以去除种子表面的农杆菌,再用无菌水清洗3-4次,用无菌滤纸吸干红麻种子表面的水分,然后用80孔育苗盆(规格:上口47mm×下口22mm×高45mm)装进口丹麦品氏托普泥炭土,每孔移栽一粒红麻种子,10h光照/14h黑暗,28℃培养箱中培养15天后将幼苗移栽至试验地中,见图4。
、检测
从移栽至试验地中成活的植株中剪取新长出的叶片,用ctab法提取dna,在pylcrispr-cas9pubi-h-hda19载体上的cas9p表达元件区域,序列如seqidno:18所示,设计扩增引物,提取的dna经pcr检测及测序验证,含有该序列的阳性株有2株,转化率为1%,见图5。阳性株t0代收种后种子全部播种,t1代收种后种子全部播种,t1及t2代均提取植株见图6叶片dna进行pcr检测及测序验证,结果表明阳性株t1及t2代植株见图7、图8均含有载体上cas9p表达元件区域序列,说明pylcrispr-cas9pubi-h-hda19载体在红麻阳性株中稳定的遗传。
以上所述仅为本发明的较佳实施例而已,并不用以限制本发明,凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
<110>广西大学
<120>一种组蛋白脱乙酰化酶基因hda19的crispr_cas9载体构建及其应用
<160>21
<210>1
<211>23
<212>dna
<213>人工序列
<220>
<221>misc_difference
<222>(1)…(23)
<400>1
gtcatttctatgacccggaggtg23
<210>2
<211>23
<212>dna
<213>人工序列
<220>
<221>misc_difference
<222>(1)…(23)
<400>2
aaaccacctccgggtcatagaaa23
<210>3
<211>24
<212>dna
<213>人工序列
<220>
<221>misc_difference
<222>(1)…(24)
<400>3
attgagcccttccctgccagggat24
<210>4
<211>24
<212>dna
<213>人工序列
<220>
<221>misc_difference
<222>(1)…(24)
<400>4
aaacatccctggcagggaagggct24
<210>5
<211>24
<212>dna
<213>人工序列
<220>
<221>misc_difference
<222>(1)…(24)
<400>5
attgtatgcaggtggctccgttgg24
<210>6
<211>24
<212>dna
<213>人工序列
<220>
<221>misc_difference
<222>(1)…(24)
<400>6
aaacccaacggagccacctgcata24
<210>7
<211>22
<212>dna
<213>人工序列
<220>
<221>misc_difference
<222>(1)…(22)
<400>7
ctccgttttacctgtggaatcg22
<210>8
<211>20
<212>dna
<213>人工序列
<220>
<221>misc_difference
<222>(1)…(20)
<400>8
cggaggaaaattccatccac20
<210>9
<211>42
<212>dna
<213>人工序列
<220>
<221>misc_difference
<222>(1)…(42)
<400>9
ttcagaggtctctctcgactagtatggaatcggcagcaaagg42
<210>10
<211>37
<212>dna
<213>人工序列
<220>
<221>misc_difference
<222>(1)…(37)
<400>10
agcgtgggtctcgtcagggtccatccactccaagctc37
<210>11
<211>38
<212>dna
<213>人工序列
<220>
<221>misc_difference
<222>(1)…(38)
<400>11
ttcagaggtctctctgacactggaatcggcagcaaagg38
<210>12
<211>38
<212>dna
<213>人工序列
<220>
<221>misc_difference
<222>(1)…(38)
<400>12
agcgtgggtctcgtcttcactccatccactccaagctc38
<210>13
<211>38
<212>dna
<213>人工序列
<220>
<221>misc_difference
<222>(1)…(38)
<400>13
ttcagaggtctctaagactttggaatcggcagcaaagg38
<210>14
<211>42
<212>dna
<213>人工序列
<220>
<221>misc_difference
<222>(1)…(42)
<400>14
agcgtgggtctcgaccgacgcgtatccatccactccaagctc42
<210>15
<211>23
<212>dna
<213>hibiscuscannabinus
<220>
<221>misc_difference
<222>(1)…(23)
<400>15
atttctatgacccggaggtgggg23
<210>16
<211>20
<212>dna
<213>hibiscuscannabinus
<220>
<221>misc_difference
<222>(1)…(20)
<400>16
agcccttccctgccagggat20
<210>17
<211>23
<212>dna
<213>hibiscuscannabinus
<220>
<221>misc_difference
<222>(1)…(23)
<400>17
tatgcaggtggctccgttggcgg23
<210>18
<211>506
<212>dna
<213>hibiscuscannabinus
<220>
<221>cds
<222>(1)…(506)
<400>18
taccttctggaactgatggcgtgaagaggaaggtttgctatttctatgacccggaggtgg60
ggaactattattatggccaaggtcaccctatgaagcctcacagaattcgaatgactcatg120
cccttctagcacattatggattacttcagcacatgcaagttcacaagcccttccctgcca180
gggatcgggatctttgccgcttccatgctgatgattatgtgaactttctccgcaccgtaa240
cccctgaaacgcagcaagatcagctcagacagctcaagcgctttaatgtcggtgaggatt300
gtcccgtctttgatggcctttactccttctgtcaaacttatgcaggtggctccgttggcg360
gtgctgtcaaattaaaccatggtctatgtgacattgccattaactgggctggcggcctgc420
atcatgcaaagaagtgtgaggcatcggggttttgctatgtcaatgatattgtccttgcaa480
ttctagaacttcttaagcagcatcag506
<210>19
<211>20
<212>dna
<213>hibiscuscannabinus
<220>
<221>misc_difference
<222>(1)…(20)
<400>19
atggaaaccggagggaattc20
<210>20
<211>24
<212>dna
<213>hibiscuscannabinus
<220>
<221>misc_difference
<222>(1)…(24)
<400>20
agctttcggatcatcgatagacat24
<210>21
<211>1384
<212>dna
<213>hibiscuscannabinus
<220>
<221>gene
<222>(1)…(1384)
<400>21
taccttctggaactgatggcgtgaagaggaaggtttgctatttctatgacccggaggtgg60
ggaactattattatggccaaggtcaccctatgaagcctcacagaattcgaatgactcatg120
cccttctagcacattatggattacttcagcacatgcaagttcacaagcccttccctgcca180
gggatcgggatctttgccgcttccatgctgatgattatgtgaactttctccgcaccgtaa240
cccctgaaacgcagcaagatcagctcagacagctcaagcgctttaatgtcggtgaggatt300
gtcccgtctttgatggcctttactccttctgtcaaacttatgcaggtggctccgttggcg360
gtgctgtcaaattaaaccatggtctatgtgacattgccattaactgggctggcggcctgc420
atcatgcaaagaagtgtgaggcatcggggttttgctatgtcaatgatattgtccttgcaa480
ttctagaacttcttaagcagcatcagcgtgttctatatgttgatattgatatccaccatg540
gagatggtgtggaggaggcattttatacaaccgacagggtcatgactgtttcatttcata600
aatttggagattattttcccggtacaggtgatattcgtgatatcggatattcgaaaggga660
aatactattccctcaatgttcctttggatgatgggattgatgatgagagttatcattgtt720
tgtttaagccaattatcggtaaagttatggaagtttttaagcctggtgctgtagttcttc780
aatgtggtgctgactctctatctggagataggttggggtgtttcaatctttcaatcaagg840
gtcatgcagagtgtgttaaatttatgagatcattcaatgtcccgctgttgcttttaggtg900
gcggcgggtataccattcggaatgtcgctcgctgttggtgctatgagacaggagttgcac960
ttggaatggaagttaacgacaagatgcctcaacacgagtattatgagtattttggtccag1020
attatacccttcatgttgcccctagtaacatggaaaacaaaaactcgcgccagttacttg1080
aagaaattcgtaataggctactcgacaatctctcaaagcttcagcattcaccaagtgtcc1140
agtttcaagaaaggccacctgatacggagcttcctgaggctgatgaagatcaggataatg1200
gagatgaaagatgggactcggattcagacatggacgttgatgaggaccgtaagctcattc1260
caagtagagtaaagagagaagtggttgaaacagatatgaaagatccggaggtccaaaaag1320
gaacagctgagcatggtagaggatctgtgaccgatgagactggtgtagatgccagtccaa1380
tgtc1384
- 还没有人留言评论。精彩留言会获得点赞!
- 一种药物中间体2-芳酰基吲哚衍生物的合成方法
- 双重hdac/brd4抑制剂及其制备方法和应用
- 含有色氨酸基本骨架的邻苯二胺类选择性组蛋白去乙酰化酶抑制剂及其制备方法和应用
- 一种生产贝利司他中间体的工艺方法
- 组蛋白去乙酰化酶抑制剂治疗β亚科疱疹病毒的新用图
- 一种含有苯甘氨酸的肉桂酰胺类组蛋白去乙酰化酶抑制剂及其制备方法和应用
- 2-(1-(4-氯苯甲酰基)-5-甲氧基-2-甲基-1氢-吲哚-3-基)-n-羟基乙酰胺作为组蛋白去 ...的制作方法
- 一种组蛋白去乙酰酶抑制剂2-(1-(4-氯苯甲酰基)-5-甲氧基-2-甲基-1氢-吲哚-3-基-n ...的制作方法
- 一种组蛋白去乙酰酶抑制剂(e)-3-(2-(1-(4-氯苯甲酰基)-5-甲氧基-2-甲基-1氢-吲哚 ...的制作方法
- 小麦组蛋白去乙酰化酶基因TaHD2C及其应用