本发明涉及分子生物学检测技术领域,更具体的说是涉及一种利用pcr-elisa检测csfv的方法。
背景技术:
猪瘟(classicalswinefever,csf)是由猪瘟病毒(classicalswinefevervirus,csfv)引起的一种高度接触性传染病,具有急性、高传染性、高致死率等特性,是严重威胁猪健康的重要传染病之一。
目前常用的检测方法有酶联免疫吸附试验(enzyme-linkedimmunosorbentassay,elisa),聚合酶链式反应(polymerasechainreaction,pcr),包含常规rt-pcr、多重rt-pcr、real-timepcr等多种pcr技术。其中,elisa方法操作简单,不需要昂贵的仪器,适合大规模临床样品的检验检疫,但由于其特异性低、灵敏性差,容易出现假阳性或者假阴性结果;而普通pcr凝胶电泳敏感性较低;实时荧光定量pcr虽可以进行定量检测,但需要特殊的设备和探针,实验费用昂贵。
因此,提供一种特异性强、灵敏度高并且检测方法操作简单、快速的pcr-elisa检测csfv的方法,是本领域技术人员亟需解决的问题。
技术实现要素:
有鉴于此,本发明提供了一种利用pcr-elisa检测csfv的方法,具有特异性强、灵敏度高并且检测方法操作简单、快速等特点。
为了实现上述目的,本发明采用如下技术方案:
一种以非诊断治疗为目的利用pcr-elisa检测csfv的特异性引物,所述引物序列如下:
csfv-f:5’-ctgcaaggaagattacaggtacgc-3’;seqidno.1;
csfv-r:5’-ttccactgtggtggtcacacaatc-3’;seqidno.2;
所述上游引物csfv-f5’端用地高辛标记,所述下游引物csfv-r5’端用生物素标记。
进一步,一种以非诊断治疗为目的利用pcr-elisa检测csfv的方法,具体步骤如下:
(1)提取样品rna,反转录成cdna,得到扩增模板;
(2)利用所述的特异性引物,对步骤(1)获得的模板进行pcr扩增,获得pcr产物;
(3)确定pcr-elisa链霉亲和素最佳包被浓度;
(4)确定pcr-elisa二抗最佳浓度;
(5)确定pcr-elisa的临界值。
进一步,步骤(2)所述pcr扩增的反应体系为:94℃预变性4min;94℃30s,55℃30s,72℃40s,30个循环;72℃延伸5min。
进一步,步骤(3)所述确定pcr-elisa链霉亲和素最佳包被浓度的具体步骤为:将链霉亲和素用cbs倍比稀释从1:50-1:1600,pcr产物作为一抗,1:2000hrp标记的抗地高辛抗体为二抗,100μl/孔,进行pcr-elisa检测。
进一步,步骤(4)所述确定pcr-elisa二抗最佳浓度:根据筛选出来的最佳链霉亲和素包被浓度,将hrp标记的抗地高辛抗体从1:1000依次倍比稀释到1:16000,100μl/孔,进行pcr-elisa检测。
进一步,步骤(5)所述确定pcr-elisa的临界值:检测样品od450值大于或者等于0.244时判定为阳性样品,小于0.244时则为阴性样品。
本发明针对csfve2基因高度保守序列设计特异性引物,分别标记生物素和地高辛,利用链霉亲和素与生物素之间特异性结合的原理,使pcr产物牢牢的结合在酶标板上。pcr-elisa将pcr与elisa高灵敏性的特性有效的结合,消除了pcr凝胶电泳灵敏度低和elisa样品制备复杂等过程,降低了试验难度,有效提高了检测的特异性和灵敏性,为猪瘟的预防与控制提供了一定的技术支撑。
经由上述的技术方案可知,与现有技术相比,本发明公开提供了一种利用pcr-elisa检测csfv的方法,最低检测到4.51×104copies/μl,普通pcr最低检测到4.51×106copies/μl,pcr-elisa灵敏性比普通pcr高约100倍;通过对临床27份未知样品进行检测,普通pcr检测出阳性样品15份,阴性12份,阳性率为55.5%;而pcr-elisa检出阳性病料20份,阴性7份,阳性率为74%,阳性检出率增加18.5%。通过建立的real-timepcr进行验证,检测结果与pcr-elisa一致,两者的符合度为100%。然后将普通pcr未检测出的阳性病料接种pk15细胞,进行病毒分离培养,通过间接免疫荧光(ifa)及普通pcr检测,该5份样品均为阳性。
附图说明
为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据提供的附图获得其他的附图。
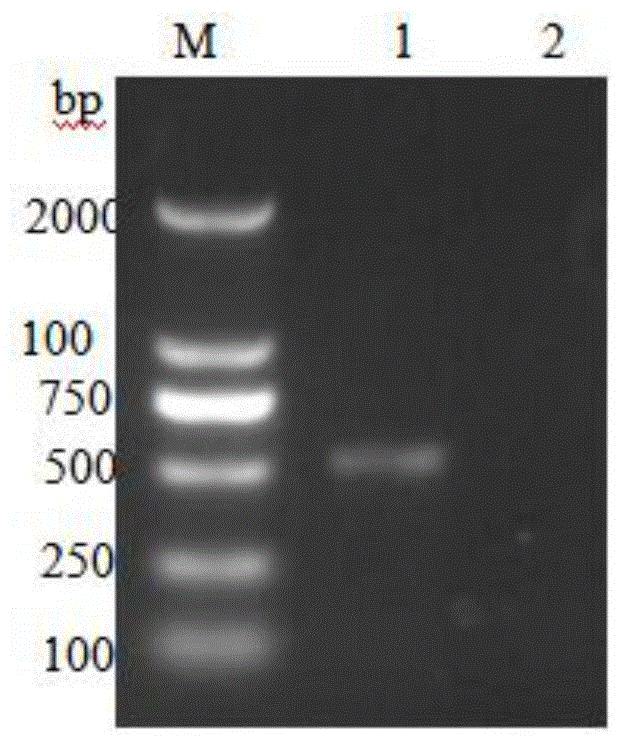
图1附图为本发明csfvpcr扩增结果;
其中,m:dl2000,1:目的基因,2:阴性对照(蒸馏水);
图2附图为本发明重组质粒双酶切结果;
其中,m:dl2000,1:重组质粒,2:质粒对照;
图3附图为本发明普通pcr灵敏性实验结果;
其中,m:dl2000,1-12:4.51×109copies/μl-4.51×10-2copies/μl;
图4附图为本发明荧光定量pcr标准曲线;
图5附图为本发明荧光定量pcr扩增曲线;
图6附图为本发明pcr检测病料结果;
其中,m:dl2000,1-27:临床病料;
图7附图为本发明临床样品pcr检测结果;
其中,m:dl2000;1-5:5份临床样品;6:阴性对照(蒸馏水);
图8附图为本发明间接免疫荧光试验结果;
其中,a:阳性对照(实验室保存的已知的csfv病毒);b、c、d、e、f:样品感染的pk15细胞;g:阴性对照(蒸馏水)。
具体实施方式
下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
猪瘟病毒、pk15细胞、猪圆环病毒、猪伪狂犬病毒、猪繁殖与呼吸综合征病毒、猪细小病毒由实验室保存并提供。
实施例1引物设计
根据genbank猪瘟石门株序列(登录号:ay775178.2),设计特异性引物csfv-f、csfv-r,用于普通pcr扩增,基因片段为511bp;然后分别在引物上下游的5’端分别标记地高辛(dig)和生物素(bio);同时设计1对特异性引物csfv-if、csfv-ir,用于荧光定量pcr扩增,基因长度为114bp;引物由上海生工生物工程股份有限公司合成并标记,如表1所示。
表1引物
实施例2csfv普通pcr扩增
从-80℃冰箱取出实验室保存的猪瘟病毒组织病料,取大约10mg淋巴结,根据takaraminibestviralrna/dnaextractionkitver.5.0说明书提取病毒rna,然后反转录cdna。以csfvcdna为模板,csfv-f、csfv-r为引物进行pcr扩增。pcr反应体系及条件为94℃预变性4min;94℃30s,55℃30s,72℃40s,30个循环;72℃延伸5min;结束后取5μl的pcr产物进行琼脂糖凝胶电泳,结果如图1所示,扩增出大小约为511bp的目的条带,与预期目的片段大小一致。
实施例3阳性质粒的构建
将上述pcr产物与克隆载体pmd19-tvector连接。连接体系为pmd19-tvector0.5μl,目的基因4.5μl,solutioni5.0μl,总体积10.0μl,混匀后放于16℃恒温槽中过夜连接。次日,从-80℃冰箱取出大肠杆菌dh5α感受态细胞,放置冰水混合物中溶解,加入连接产物5μl,冰浴30min,42℃热击90s立即取出,冰上放置1~2min,加入900μl不含抗性的lb液体培养基,放入37℃培养箱中振荡培养45min,4000rpm离心5min,保留约100μl上清,均匀涂布在含amp抗性(100μg/ml)的lb固体培养基平板上,将平板倒置于37℃恒温培养箱过夜培养,通过菌液pcr和双酶切(kpnⅰ和hindⅲ)验证质粒构建结果,双酶切鉴定结果如图2所示,与预期结果一致。
实施例4csfvpcr-elisa方法的建立
(1)pcr扩增:以构建的csfvcdna为模板,标记dig的上游引物csfv-f,标记bio的下游引物csfv-r为引物进行pcr扩增;pcr反应体系为:premixtaq12.5μl,上下游引物各0.5μl,模板1μl,双蒸水10.5μl;pcr反应条件为94℃预变性4min;94℃30s,55℃30s,72℃40s,30个循环;72℃延伸5min;
(2)确定pcr-elisa链霉亲和素最佳包被浓度:将链霉亲和素用cbs(包被液)倍比稀释从1:50-1:1600,pcr产物作为一抗,1:2000hrp标记的抗地高辛抗体为二抗,100μl/孔,进行pcr-elisa检测,结果如表2所示;链霉亲和素包被浓度1:100(1μg/孔),包被时间1h时od450接近于1.0并且p/n值最大,最符合预期要求;
表2pcr-elisa链霉亲和素包被浓度的筛选
注:p表示od450阳性值,n表示od450阴性值;
(3)确定pcr-elisa二抗最佳浓度:根据筛选出来的最佳链霉亲和素包被浓度,将hrp标记的抗地高辛抗体从1:1000依次倍比稀释到1:16000,100μl/孔,进行pcr-elisa检测;二抗稀释浓度1:2000时od450接近于1.0并且p/n值最大,最符合预期要求;
(4)确定pcr-elisa的临界值:将30份csfv阴性样品,进行pcr-elisa检测,确定pcr-elisa的阳性判断结果;
阳性对照od值为1.95,空白对照od值为0.054;根据30份阴性样品检测结果的od450值计算出平均值(x)等于0.112,标准方差(sd)等于0.044,根据公式x+3sd=0.244得到pcr-elisa判定标准为:检测样品od450值大于或者等于0.244时判定为阳性样品,小于0.244时则为阴性样品。
实施例5普通pcr与pcr-elisa灵敏性实验
将构建好的csfv阳性质粒od260nm值为158.5ng/μl,计算得出其拷贝数为4.51×1010copies/μl,按10倍梯度稀释,10-1-10-12依次是4.51×109-4.51×10-2copies/μl,进行pcr-elisa和普通pcr检测,比较其灵敏性。
灵敏性实验结果显示,pcr-elisa最低检测到4.51×104copies/μl(表3),而普通pcr最低检测到4.51×106copies/μl(图3),pcr-elisa灵敏性比普通pcr高100倍。
表3pcr-elisa的灵敏性
注:p表示od450阳性值,n表示od450阴性值。
实施例6csfv荧光定量pcr检测方法的建立
以csfvcdna为模板,csfv-if、csfv-ir为引物进行pcr扩增,质粒构建参照实施例3,获得荧光定量pcr标准质粒。将标准质粒浓度换算成拷贝数,然后按照10倍梯度稀释10-1-10-10,制作标准曲线;然后进行csfv荧光定量pcr灵敏性、特异性、重复性分析。
根据质粒od260nm值为55.8ng/μl,计算得出其拷贝数为1.81×1010copies/μl,10倍梯度稀释后,分别作为模板进行sybrgreenireal-timepcr扩增,结果显示线性关系良好,标准曲线如图4所示,扩增曲线如图5所示。将已知阳性样品csfv、ppv、prv、pcv2、prrsv作为模板(表4)进行sybrgreenireal-timepcr检测,结果显示,只有csfv出现特异性扩增,sybrgreenireal-timepcr最低检出量为1.81×103copies/μl,其灵敏性约为常规pcr的1000倍。
表4sybrgreenⅰreal-timepcr特异性检测结果
实施例7临床病料检测
从本地周边疑似感染猪瘟的猪场采集口腔粘液和肛门粘液27份,分别用普通pcr、pcr-elisa、荧光定量pcr3种检测方法对27份样品进行临床检测。
结果显示检测的27份样品,其中普通pcr有15份为阳性,12份为阴性,阳性率为55.5%,如图6所示。pcr-elisa和real-timepcr检出了20份阳性样品,两种检测方法符合度100%,阳性率为74%,阳性率增加18.5%,检测结果如表5所示。
表5pcr-elisa、rt-pcr和real-timepcr临床样品检测结果
实施例8病毒分离
为进一步验证pcr-elisa的灵敏性和准确性,选取普通pcr检测为阴性而pcr-elisa检测为阳性的临床样品进行病毒分离,将5份样品分别加入灭菌的pbs1ml,12000rpm离心5min,取上清液用0.22μm滤膜进行过滤。接种pk15细胞,72h后收获病毒,进行普通pcr检测,结果如图7所示,收获的病毒液里均含有csfv。
实施例9间接免疫荧光检测(ifa)
将生长状态良好的pk15细胞平铺于96孔细胞培养板中,待细胞生长至80%,接种上述实验收获的病毒液,设立阳性对照。置于37℃5%co2的培养箱中培养72h,进行ifa检测,具体步骤如下:
(1)固定:用预冷的3%h2o2甲醇固定,4℃20min,50μl/孔,弃去固定液,pbst洗6遍;
(2)封闭:用5%脱脂奶粉,300μl/孔,37℃恒温培养箱温育1h,pbst洗涤6遍;
(3)加一抗:加入1:100csfv阳性血清,50μl/孔,37℃恒温培养箱作用1h,pbst洗涤6遍;
(4)加二抗:加入1:100兔抗猪lgg-fitc,50μl/孔,37℃恒温培养箱作用1h,pbst洗涤6遍;
(5)每孔加入50μl的pbs,在荧光显微镜下观察结果。
结果如图8所示,5份普通pcr检测结果为阴性的csfv病毒,ifa检测均为阳性,与pcr-elisa和real-timepcr结果一致,从而说明pcr-elisa灵敏度较高,可用于临床样品的检测。
对所公开的实施例的上述说明,使本领域专业技术人员能够实现或使用本发明。对这些实施例的多种修改对本领域的专业技术人员来说将是显而易见的,本文中所定义的一般原理可以在不脱离本发明的精神或范围的情况下,在其它实施例中实现。因此,本发明将不会被限制于本文所示的这些实施例,而是要符合与本文所公开的原理和新颖特点相一致的最宽的范围。
序列表
<110>新乡学院
<120>一种利用pcr-elisa检测csfv的方法
<160>4
<170>siposequencelisting1.0
<210>1
<211>24
<212>dna
<213>artificialsequence
<400>1
ctgcaaggaagattacaggtacgc24
<210>2
<211>24
<212>dna
<213>artificialsequence
<400>2
ttccactgtggtggtcacacaatc24
<210>3
<211>19
<212>dna
<213>artificialsequence
<400>3
agcccacctcgagatgcta19
<210>4
<211>23
<212>dna
<213>artificialsequence
<400>4
ctatcaggtcgtactcccatcac23
