用于发酵生产1,5-戊二胺的重组DNA、菌株及其应用的制作方法
本发明属于微生物工程技术领域,具体地说,涉及用于发酵生产1,5-戊二胺的重组dna、菌株及其应用。
背景技术:
通过dna重组技术改造具有生产l-赖氨酸能力的棒杆菌属和埃希杆菌属的细菌,现已实现工业化生产。这些细菌的l-赖氨酸生产率的提高是通过过表达l-赖氨酸合成途径的相关基因和反馈抑制脱敏的相关基因,或者从葡萄糖代谢开始的能量供应途径的强化而实现的。赖氨酸脱羧酶可以将l-赖氨酸脱去一个羧基生成1,5-戊二胺(尸胺)和co2。例如,在大肠杆菌(e.coli)中,尸胺是通过两种赖氨酸脱羧酶多肽cada和ldcc直接从l-赖氨酸生物合成的。
目前,1,5-戊二胺的生物合成主要利用两种策略进行:发酵生产或体外酶催化。在l-赖氨酸的发酵生产路径中,将赖氨酸脱羧酶(通常为cada或ldcc),添加至产赖氨酸的微生物(例如谷氨酸棒杆菌和大肠杆菌)中,以便将赖氨酸生物合成途径延长至1,5-戊二胺生物合成途径。或者,对于体外酶催化,可以将细菌工程化或诱导以产生赖氨酸脱羧酶(通常为cada或ldcc),然后其可用于通过脱羧作用将赖氨酸转化为1,5-戊二胺。1,5-戊二胺的用途相当广泛,例如可以与二元酸进行聚合反应合成新型尼龙,在工业生产中具有很高的应用价值。
但是,目前l-赖氨酸延长发酵生产1,5-戊二胺的路径中,1,5-戊二胺产量有限且收率较低。因此,亟需开发一种具有较高收率的生产尸胺的方法。
技术实现要素:
本发明的目的是提供l-赖氨酸透性酶基因在微生物发酵生产1,5-戊二胺中的应用。
本发明的另一目的是提供用于发酵生产1,5-戊二胺的重组dna、菌株及其应用。
本发明构思如下:在大肠杆菌中,l-赖氨酸透性酶lysp和膜上整合的ph感应器cadc,共同诱导l-赖氨酸依赖的菌株在酸性压力条件下的适应性。由lysp基因编码的l-赖氨酸透性酶(l-lysinepermease,lysp)是将细胞外l-赖氨酸转运至细胞内的专一的通透酶。本发明通过增加l-赖氨酸转运蛋白lysp的含量,加速l-赖氨酸向细胞内的运输过程,改善菌株对高浓度l-赖氨酸的耐受能力,促进l-赖氨酸的生产,进而提高代谢产物1,5-戊二胺的产量。
为了实现本发明目的,第一方面,本发明提供l-赖氨酸透性酶基因在微生物发酵生产1,5-戊二胺中的应用。
本发明中,所述l-赖氨酸透性酶基因,通过质粒导入微生物中或通过基因工程手段整合到微生物染色体上。
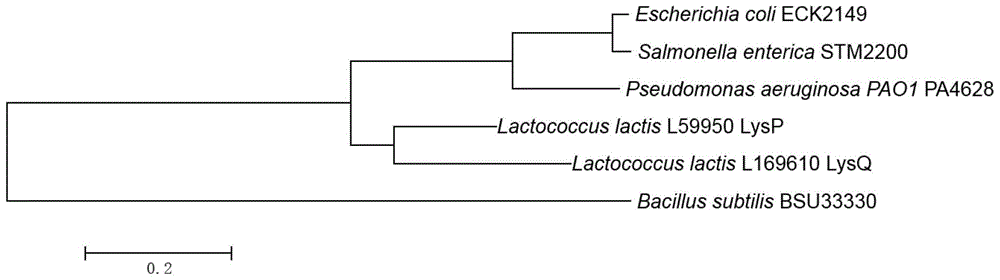
本发明所述l-赖氨酸透性酶(l-赖氨酸转运蛋白)来自于微生物;优选来自大肠杆菌(escherichiacoli)、沙门氏菌属(salmonella)、绿脓假单胞菌(pesudomonaspyocyaneum)、乳球菌属(lactococcus)、枯草芽孢杆菌(bacillussubtilis)。
不同微生物来源的l-赖氨酸透性酶的氨基酸序列相似性比对结果见图1。
第二方面,本发明提供用于发酵生产1,5-戊二胺的重组dna,所述重组dna至少包括l-赖氨酸透性酶基因,以及1,5-戊二胺生物合成途径的相关基因和/或反馈抑制脱敏的相关基因。所述基因可以由相同质粒或不同质粒携带。某些基因可以由相同质粒携带。当使用两种或两种以上质粒时,优选使用具有稳定的分配系统的质粒,该系统使得这些质粒能在细胞中稳定的共存。导入所述基因的顺序没有特别限制。
优选地,所述重组dna至少包括l-赖氨酸透性酶基因和l-赖氨酸脱羧酶基因。
更优选地,所述l-赖氨酸透性酶基因是来自大肠杆菌的lysp基因(seqidno:1)或lysp基因的片段,所述l-赖氨酸脱羧酶基因是来自大肠杆菌的cada基因或ldcc基因。所述l-赖氨酸透性酶还可以是上述来源的l-赖氨酸透性酶的突变体(包括天然突变体和人工重组突变体)或者活性片段。大肠杆菌l-赖氨酸透性酶lysp的氨基酸序列见seqidno:2。
第三方面,本发明提供含有所述重组dna的生物材料,所述生物材料包括但不限于表达盒、转座子、质粒载体、噬菌体载体、病毒载体或工程菌。
第四方面,本发明提供一种表达载体,包含l-赖氨酸透性酶基因和l-赖氨酸脱羧酶基因,以及能够在宿主细胞中自主复制的骨架质粒。
优选地,所述宿主细胞选自大肠杆菌(escherichiacoli)、嗜热菌(thermus.thermophilus)、蜂房哈夫尼菌(hafniaalvei)、枯草芽孢杆菌(bacillussubtilis)、谷氨酸棒状杆菌(corynebacteriumglutamicum),或经过诱变或随机突变之后的菌株或基因工程菌的细胞。
优选地,所述骨架质粒选自puc18、puc19、pbr322、pacyc、pet、psc101以及它们的衍生质粒。
优选地,所述表达载体含有启动子、cada、lysp的dna序列或含有启动子、ldcc、lysp的dna序列。
更优选地,所述表达质粒载体包含启动子-cada-启动子-lysp、启动子-ldcc-启动子-lysp、启动子-lysp-启动子-cada或启动子-lysp-启动子-ldcc的dna序列。
第五方面,本发明提供高产1,5-戊二胺的工程菌,其包含所述重组dna,且所述工程菌的出发菌株为具有生产l-赖氨酸能力的菌株。
优选地,所述出发菌株选自埃希氏菌属(escherichia)、棒杆菌属(corynebacterium)、短杆菌属(brevibacterium)、哈夫尼菌属(hafnia)中的菌种。
更优选地,所述出发菌株选自大肠杆菌(escherichiacoli)、嗜热菌(thermus.thermophilus)、蜂房哈夫尼菌(hafniaalvei)、枯草芽孢杆菌(bacillussubtilis)、谷氨酸棒状杆菌(corynebacteriumglutamicum),或经过诱变或随机突变之后的菌株或基因工程菌。
在本发明的一个具体实施方式中,所述出发菌株为大肠杆菌(escherichiacoli)m11-a3,保藏编号为cctccno:m2018456。其是以大肠杆菌mg1655(e.colimg1655k12)为出发菌株,通过artp物理诱变获得的l-赖氨酸生产水平提高的突变株m11-a3。根据布达佩斯条约,大肠杆菌(escherichiacoli)m11-a3现已保藏于中国典型培养物保藏中心(cctcc),地址:中国武汉,武汉大学,邮编430072,保藏编号cctccno:m2018456,保藏日期2018年7月6日。
所述工程菌中包含:启动子-cada-启动子-lysp的dna序列或启动子-ldcc-启动子-lysp的dna序列。
其中,所述启动子选自plac、trp、tac、trc或pl等。
优选地,所述工程菌中包含:plac-cada-plac-lysp的dna序列plac-ldcc-plac-lysp的dna序列。
第六方面,本发明提供所述工程菌在发酵生产1,5-戊二胺中的应用。
第七方面,本发明提供1,5-戊二胺的生产方法,包括在发酵培养基中对高产1,5-戊二胺的工程菌进行培养以生产1,5-戊二胺。
借由上述技术方案,本发明至少具有下列优点及有益效果:
本发明提供了一种高效生产1,5-戊二胺的方法,通过增加l-赖氨酸转运蛋白lysp的含量,加速l-赖氨酸向细胞内的运输过程,改善菌株对高浓度l-赖氨酸的耐受能力,同时促进l-赖氨酸的生产,进而提高代谢产物1,5-戊二胺的产量。
附图说明
图1为本发明几种微生物来源的l-赖氨酸透性酶的氨基酸序列相似性比对结果。
具体实施方式
以下实施例用于说明本发明,但不用来限制本发明的范围。若未特别指明,实施例均按照常规实验条件,如sambrook等分子克隆实验手册(sambrookj&russelldw,molecularcloning:alaboratorymanual,2001),或按照制造厂商说明书建议的条件。
本发明中涉及到的百分号“%”,若未特别说明,是指质量百分比;但溶液的百分比,除另有规定外,是指100ml溶液中含有溶质的克数。
实施例1诱变获得的l-赖氨酸生产水平提高的突变株
从大肠杆菌mg1655(e.colimg1655k12,购自北京天恩泽基因科技有限公司)出发,通过artp物理诱变的方法,以及l-赖氨酸类似物添加平板进行筛选。
接种mg1655菌株至50ml的lb培养基,待菌体浓度到od600=0.6时,收集菌体。将菌体转移至artp仪器(无锡源清天木生物科技有限公司),选取70秒进行诱变处理(致死率达到98%),收集诱变后的菌体,稀释102和103倍后,全部涂板m9培养基(其中培养基中添加4mg/l的l-赖氨酸类似物aec)。在未添加aec(5-(2-氨基乙基)-l-半胱氨酸)的平板上分别涂布未诱变菌(稀释105倍的菌液),以及诱变处理后的菌体(稀释103倍的菌液)。37℃培养两天后,共获得l-赖氨酸类似物耐受能力提高的菌株36株。挑取单菌落至30μl无菌生理盐水中,从筛选平板上蘸取菌体获得浑浊的菌液。取3μl划线至不同浓度的aec平板进行复筛。通过使用不断增加浓度的l-赖氨酸类似物的添加平板继续重复筛选。最后确认m11菌株耐受能力最强。
从新突变株m11出发,通过artp物理诱变和高浓度l-赖氨酸类似物添加平板进行筛选,共获得24株新的突变菌株。将获得的24个新突变株和出发株m11,分别接种于不含抗生素的5ml种子培养基[(含有4%葡萄糖,0.1%kh2po4,0.1%mgso4,1.6%(nh4)2so4,0.001%feso4,0.001%mnso4,0.2%酵母提取物(购自英格兰oxoidltd.),0.01%l-苏氨酸,0.005%l-异亮氨酸)]进行培养,37℃、225rpm过夜。第二天,每个菌株再分别转接至50ml新鲜的含有30g/l葡萄糖、0.7%ca(hco3)2和100μg/ml氨苄青霉素的发酵培养基中,于37℃、170rpm继续培养72h,利用核磁检测并计算各培养基中的赖氨酸的含量(表1)。
表1核磁检测24株新突变株与出发菌株m11相比的赖氨酸产量及od600
注:d25表示发酵液稀释25倍之后的测定结果。
如表1所示,发酵72h后,突变株m11-a3的赖氨酸的产量为3.57g/kg,相比于出发株m11的l-赖氨酸产量提升26%,且突变株与对照相比生长无显著差异。为提高筛选条件,将发酵时间缩短为48h。三次重复发酵出发株和该突变株,确认突变株m11-a3的产量为3.00g/kg,相对l-赖氨酸产量提升维持在24~26%。m11-a3菌株保藏于武汉大学中国典型培养物保藏中心(cctcc),保藏编号为cctccno:m2018456。
实施例2构建表达载体puc18-plac-cada、puc18-plac-ldcc、puc18-plac-cada-plac-lysp和puc18-plac-ldcc-plac-lysp
首先,构建重组表达质粒puc18-plac-cada和puc18-plac-ldcc。根据多片段一步法克隆试剂盒(购自南京诺唯赞生物科技有限公司)产品说明书设计引物,在plac序列的左侧引入与puc18载体hindiii单酶切后上游重合20~30bp的dna序列(seqidno:3)获得引物plac-f(seqidno:4)。在cada基因序列的两端引入与plac序列及载体hindiii单酶切后下游重合20~30bp的dna序列(seqidno:6)和(seqidno:7)获得引物对cada-f/r(seqidno:8和9)。在ldcc基因序列的两端引入与plac序列及载体hindiii单酶切后下游重合20~30bp的dna序列(seqidno:10)和(seqidno:11)获得引物对ldcc-f/r(seqidno:12和13)。以puc18质粒dna作为模板,使用引物对plac-f/rseqidno:4)和(seqidno:5),通过pcr扩增获得plac片段(seqidno:14)。以mg1655的基因组dna作为模板,分别使用引物对cada-f/r和ldcc-f/r,通过pcr扩增获得cada片段(seqidno:15)和ldcc片段(seqidno:16)。将plac、cada片段、ldcc片段和hindiii单酶切后的puc18载体进行切胶回收纯化,使用多片段一步法克隆试剂盒将plac、cada(或ldcc)片段和载体进行重组。重组后的混合物分别转化至e.colijm109(购自宝生物工程(大连)有限公司)感受态细胞中,在含有氨苄青霉素的lb平板上进行筛选,获得多个单菌落。通过菌落pcr和测序验证证实plac-cada和plac-ldcc表达序列已分别插入到质粒载体puc18的hindiii位点,得到正确的表达载体puc18-plac-cada和puc18-plac-ldcc。
然后,构建重组表达质粒puc18-plac-cada-plac-lysp和puc18-plac-ldcc-plac-lysp。根据多片段一步法克隆试剂盒(购自南京诺唯赞生物科技有限公司)产品说明书设计引物,在lysp基因序列的两端引入与puc18-plac-cada或puc18-plac-ldcc载体saci/xbai双酶切后重合20~30bp的dna序列(seqidno:17)和(seqidno:18),获得引物对lysp-f/r(seqidno:19和20)。以大肠杆菌mg1655基因组dna作为模板,使用引物对lysp-f/r(seqidno:19和20),通过pcr扩增获得lysp基因片段(seqidno:21)。将lysp基因片段分别与saci和xbai酶切后的puc18-plac-cada和puc18-plac-ldcc载体进行切胶回收纯化,使用多片段一步法克隆试剂盒将lysp基因片段分别与上述两个载体进行重组。重组后的混合物全部转化至e.colijm109(购自宝生物工程(大连)有限公司)感受态细胞中,在含有氨苄青霉素的lb平板上进行筛选,获得多个单菌落。通过菌落pcr和测序验证证实lysp基因已插入到质粒载体puc18-plac-cada和puc18-plac-ldcc的saci和xbai位点之间,得到正确的表达载体puc18-plac-cada-plac-lysp和puc18-plac-ldcc-plac-lysp。
实施例3构建1,5-戊二胺生产菌株和生产1,5-戊二胺
首先,构建l-赖氨酸生产菌株:使用实施例1筛选获得的m11-a3突变株,制备感受态。将实施例2构建得到的表达载体puc18-plac-cada、puc18-plac-ldcc、puc18-plac-cada-plac-lysp和puc18-plac-ldcc-plac-lysp分别转入受体菌m11-a3突变株中。涂布在含有100μg/ml氨苄青霉素的lb抗性平板中进行筛选。挑取12个单菌落至添加有氨苄青霉素的600μllb培养基中,37℃培养3h后,取1μl菌体作为模板进行pcr验证筛选正确的突变株,并甘油保种。
上述含有puc18-plac-cada、puc18-plac-ldcc、puc18-plac-cada-plac-lysp和puc18-plac-ldcc-plac-lysp质粒的突变株各选取3个转化子和出发株m11-a3,分别涂布于含有100μg/ml氨苄青霉素和不含抗生素的种子培养基(同实施例1)平板上进行筛选。进一步地,分别各挑取3个单克隆,利用5ml种子培养基进行培养,37℃、225rpm过夜。第二天,每个菌株再分别转接至50ml新鲜的含有30g/l葡萄糖、0.7%ca(hco3)2和100μg/ml氨苄青霉素的发酵培养基中于37℃、170rpm继续培养48h,利用核磁检测并计算各培养基中的戊二胺的含量(表2)。
表2核磁检测突变株与出发菌株相比的戊二胺产量及od600
如表2所示,含有质粒puc18-plac-cada和puc18-plac-cada-plac-lysp的转化株的戊二胺的产量分别为1.47g/kg和2.07g/kg。两个转化株的l-赖氨酸转化率分别为82.5%和93.1%。含有质粒puc18-plac-ldcc和puc18-plac-ldcc-plac-lysp的转化株的戊二胺的产量分别为1.31g/kg和1.87g/kg,两个转化株的l-赖氨酸转化率分别为78.0%和91.5%,且转化株与对照相比生长无显著差异。由此分析,lysp促进胞外lysine向细胞内的运送,lysp和cada基因或ldcc基因同时过表达均能够加速l-赖氨酸高效转化为戊二胺,菌株m11-a3/puc18-plac-cada-plac-lysp和m11-a3/puc18-plac-ldcc-plac-lysp可用于戊二胺的生产。
虽然,上文中已经用一般性说明及具体实施方案对本发明作了详尽的描述,但在本发明基础上,可以对之做一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。
序列表
<110>上海凯赛生物技术研发中心有限公司
cibt美国公司
<120>用于发酵生产1,5-戊二胺的重组dna、菌株及其应用
<130>khp181118795.1
<160>21
<170>siposequencelisting1.0
<210>1
<211>1470
<212>dna
<213>大肠杆菌(escherichiacoli)
<400>1
atggtttccgaaactaaaaccacagaagcgccgggcttacgccgtgaattaaaggcgcgt60
cacctgacgatgattgccattggcggttccatcggtacaggtctttttgttgcctctggc120
gcaacgatttctcaggcaggtccgggcggggcattgctctcgtatatgctgattggcctg180
atggtttacttcctgatgaccagtctcggtgaactggctgcatatatgccggtttccggt240
tcgtttgccacttacggtcagaactatgttgaagaaggctttggcttcgcgctgggctgg300
aactactggtacaactgggcggtgactatcgccgttgacctggttgcagctcagctggtc360
atgagctggtggttcccggatacaccgggctggatctggagtgcgttgttcctcggcgtt420
atcttcctgctgaactacatctcagttcgtggctttggtgaagcggaatactggttctca480
ctgatcaaagtcacgacagttattgtctttatcatcgttggcgtgctgatgattatcggt540
atcttcaaaggcgcgcagcctgcgggctggagcaactggacaatcggcgaagcgccgttt600
gctggtggttttgcggcgatgatcggcgtagctatgattgtcggcttctctttccaggga660
accgagctgatcggtattgctgcaggcgagtccgaagatccggcgaaaaacattccacgc720
gcggtacgtcaggtgttctggcgaatcctgttgttctatgtgttcgcgatcctgattatc780
agcctgattattccgtacaccgatccgagcctgctgcgtaacgatgttaaagacatcagc840
gttagtccgttcaccctggtgttccagcacgcgggtctgctctctgcggcggcggtgatg900
aacgcagttattctgacggcggtgctgtcagcgggtaactccggtatgtatgcgtctact960
cgtatgctgtacaccctggcgtgtgacggtaaagcgccgcgcattttcgctaaactgtcg1020
cgtggtggcgtgccgcgtaatgcgctgtatgcgacgacggtgattgccggtctgtgcttc1080
ctgacctccatgtttggcaaccagacggtatacctgtggctgctgaacacctccgggatg1140
acgggttttatcgcctggctggggattgccattagccactatcgcttccgtcgcggttac1200
gtattgcagggacacgacattaacgatctgccgtaccgttcaggtttcttcccactgggg1260
ccgatcttcgcattcattctgtgtctgattatcactttgggccagaactacgaagcgttc1320
ctgaaagatactattgactggggcggcgtagcggcaacgtatattggtatcccgctgttc1380
ctgattatttggttcggctacaagctgattaaaggaactcacttcgtacgctacagcgaa1440
atgaagttcccgcagaacgataagaaataa1470
<210>2
<211>489
<212>prt
<213>大肠杆菌(escherichiacoli)
<400>2
metvalsergluthrlysthrthrglualaproglyleuargargglu
151015
leulysalaarghisleuthrmetilealaileglyglyserilegly
202530
thrglyleuphevalalaserglyalathrileserglnalaglypro
354045
glyglyalaleuleusertyrmetleuileglyleumetvaltyrphe
505560
leumetthrserleuglygluleualaalatyrmetprovalsergly
65707580
serphealathrtyrglyglnasntyrvalglugluglypheglyphe
859095
alaleuglytrpasntyrtrptyrasntrpalavalthrilealaval
100105110
aspleuvalalaalaglnleuvalmetsertrptrppheproaspthr
115120125
proglytrpiletrpseralaleupheleuglyvalilepheleuleu
130135140
asntyrileservalargglypheglyglualaglutyrtrppheser
145150155160
leuilelysvalthrthrvalilevalpheileilevalglyvalleu
165170175
metileileglyilephelysglyalaglnproalaglytrpserasn
180185190
trpthrileglyglualaprophealaglyglyphealaalametile
195200205
glyvalalametilevalglypheserpheglnglythrgluleuile
210215220
glyilealaalaglyglusergluaspproalalysasnileproarg
225230235240
alavalargglnvalphetrpargileleuleuphetyrvalpheala
245250255
ileleuileileserleuileileprotyrthraspproserleuleu
260265270
argasnaspvallysaspileservalserprophethrleuvalphe
275280285
glnhisalaglyleuleuseralaalaalavalmetasnalavalile
290295300
leuthralavalleuseralaglyasnserglymettyralaserthr
305310315320
argmetleutyrthrleualacysaspglylysalaproargilephe
325330335
alalysleuserargglyglyvalproargasnalaleutyralathr
340345350
thrvalilealaglyleucyspheleuthrsermetpheglyasngln
355360365
thrvaltyrleutrpleuleuasnthrserglymetthrglypheile
370375380
alatrpleuglyilealaileserhistyrargpheargargglytyr
385390395400
valleuglnglyhisaspileasnaspleuprotyrargserglyphe
405410415
pheproleuglyproilephealapheileleucysleuileilethr
420425430
leuglyglnasntyrglualapheleulysaspthrileasptrpgly
435440445
glyvalalaalathrtyrileglyileproleupheleuileiletrp
450455460
pheglytyrlysleuilelysglythrhisphevalargtyrserglu
465470475480
metlyspheproglnasnasplyslys
485
<210>3
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>3
tctagagtcgacctgcaggcatgcaagctt30
<210>4
<211>52
<212>dna
<213>人工序列(artificialsequence)
<400>4
tctagagtcgacctgcaggcatgcaagcttggataaccgtattaccgccttt52
<210>5
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>5
agctgtttcctgtgtgaaattgtta25
<210>6
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>6
gcggataacaatttcacacaggaaacagct30
<210>7
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>7
aagcttggcactggccgtcgttttacaacg30
<210>8
<211>55
<212>dna
<213>人工序列(artificialsequence)
<400>8
gcggataacaatttcacacaggaaacagctatgaacgttattgcaatattgaatc55
<210>9
<211>54
<212>dna
<213>人工序列(artificialsequence)
<400>9
cgttgtaaaacgacggccagtgccaagcttccacttcccttgtacgagctaatt54
<210>10
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>10
gcggataacaatttcacacaggaaacagct30
<210>11
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>11
aagcttggcactggccgtcgttttacaacg30
<210>12
<211>55
<212>dna
<213>人工序列(artificialsequence)
<400>12
gcggataacaatttcacacaggaaacagctatgaacatcattgccattatgggac55
<210>13
<211>50
<212>dna
<213>人工序列(artificialsequence)
<400>13
cgttgtaaaacgacggccagtgccaagcttccggaagccgctctggcaag50
<210>14
<211>338
<212>dna
<213>大肠杆菌(escherichiacoli)
<400>14
tctagagtcgacctgcaggcatgcaagcttggataaccgtattaccgcctttgagtgagc60
tgataccgctcgccgcagccgaacgaccgagcgcagcgagtcagtgagcgaggaagcgga120
agagcgcccaatacgcaaaccgcctctccccgcgcgttggccgattcattaatgcagctg180
gcacgacaggtttcccgactggaaagcgggcagtgagcgcaacgcaattaatgtgagtta240
gctcactcattaggcaccccaggctttacactttatgcttccggctcgtatgttgtgtgg300
aattgtgagcggataacaatttcacacaggaaacagct338
<210>15
<211>2230
<212>dna
<213>大肠杆菌(escherichiacoli)
<400>15
gcggataacaatttcacacaggaaacagctatgaacgttattgcaatattgaatcacatg60
ggggtttattttaaagaagaacccatccgtgaacttcatcgcgcgcttgaacgtctgaac120
ttccagattgtttacccgaacgaccgtgacgacttattaaaactgatcgaaaacaatgcg180
cgtctgtgcggcgttatttttgactgggataaatataatctcgagctgtgcgaagaaatt240
agcaaaatgaacgagaacctgccgttgtacgcgttcgctaatacgtattccactctcgat300
gtaagcctgaatgacctgcgtttacagattagcttctttgaatatgcgctgggtgctgct360
gaagatattgctaataagatcaagcagaccactgacgaatatatcaacactattctgcct420
ccgctgactaaagcactgtttaaatatgttcgtgaaggtaaatatactttctgtactcct480
ggtcacatgggcggtactgcattccagaaaagcccggtaggtagcctgttctatgatttc540
tttggtccgaataccatgaaatctgatatttccatttcagtatctgaactgggttctctg600
ctggatcacagtggtccacacaaagaagcagaacagtatatcgctcgcgtctttaacgca660
gaccgcagctacatggtgaccaacggtacttccactgcgaacaaaattgttggtatgtac720
tctgctccagcaggcagcaccattctgattgaccgtaactgccacaaatcgctgacccac780
ctgatgatgatgagcgatgttacgccaatctatttccgcccgacccgtaacgcttacggt840
attcttggtggtatcccacagagtgaattccagcacgctaccattgctaagcgcgtgaaa900
gaaacaccaaacgcaacctggccggtacatgctgtaattaccaactctacctatgatggt960
ctgctgtacaacaccgacttcatcaagaaaacactggatgtgaaatccatccactttgac1020
tccgcgtgggtgccttacaccaacttctcaccgatttacgaaggtaaatgcggtatgagc1080
ggtggccgtgtagaagggaaagtgatttacgaaacccagtccactcacaaactgctggcg1140
gcgttctctcaggcttccatgatccacgttaaaggtgacgtaaacgaagaaacctttaac1200
gaagcctacatgatgcacaccaccacttctccgcactacggtatcgtggcgtccactgaa1260
accgctgcggcgatgatgaaaggcaatgcaggtaagcgtctgatcaacggttctattgaa1320
cgtgcgatcaaattccgtaaagagatcaaacgtctgagaacggaatctgatggctggttc1380
tttgatgtatggcagccggatcatatcgatacgactgaatgctggccgctgcgttctgac1440
agcacctggcacggcttcaaaaacatcgataacgagcacatgtatcttgacccgatcaaa1500
gtcaccctgctgactccggggatggaaaaagacggcaccatgagcgactttggtattccg1560
gccagcatcgtggcgaaatacctcgacgaacatggcatcgttgttgagaaaaccggtccg1620
tataacctgctgttcctgttcagcatcggtatcgataagaccaaagcactgagcctgctg1680
cgtgctctgactgactttaaacgtgcgttcgacctgaacctgcgtgtgaaaaacatgctg1740
ccgtctctgtatcgtgaagatcctgaattctatgaaaacatgcgtattcaggaactggct1800
cagaatatccacaaactgattgttcaccacaatctgccggatctgatgtatcgcgcattt1860
gaagtgctgccgacgatggtaatgactccgtatgctgcattccagaaagagctgcacggt1920
atgaccgaagaagtttacctcgacgaaatggtaggtcgtattaacgccaatatgatcctt1980
ccgtacccgccgggagttcctctggtaatgccgggtgaaatgatcaccgaagaaagccgt2040
ccggttctggagttcctgcagatgctgtgtgaaatcggcgctcactatccgggctttgaa2100
accgatattcacggtgcataccgtcaggctgatggccgctataccgttaaggtattgaaa2160
gaagaaagcaaaaaataattagctcgtacaagggaagtggaagcttggcactggccgtcg2220
ttttacaacg2230
<210>16
<211>2222
<212>dna
<213>大肠杆菌(escherichiacoli)
<400>16
gcggataacaatttcacacaggaaacagctatgaacatcattgccattatgggaccgcat60
ggcgtcttttataaagatgagcccatcaaagaactggagtcggcgctggtggcgcaaggc120
tttcagattatctggccacaaaacagcgttgatttgctgaaatttatcgagcataaccct180
cgaatttgcggcgtgatttttgactgggatgagtacagtctcgatttatgtagcgatatc240
aatcagcttaatgaatatctcccgctttatgccttcatcaacacccactcgacgatggat300
gtcagcgtgcaggatatgcggatggcgctctggttttttgaatatgcgctggggcaggcg360
gaagatatcgccattcgtatgcgtcagtacaccgacgaatatcttgataacattacaccg420
ccgttcacgaaagccttgtttacctacgtcaaagagcggaagtacaccttttgtacgccg480
gggcatatgggcggcaccgcatatcaaaaaagcccggttggctgtctgttttatgatttt540
ttcggcgggaatactcttaaggctgatgtctctatttcggtcaccgagcttggttcgttg600
ctcgaccacaccgggccacacctggaagcggaagagtacatcgcgcggacttttggcgcg660
gaacagagttatatcgttaccaacggaacatcgacgtcgaacaaaattgtgggtatgtac720
gccgcgccatccggcagtacgctgttgatcgaccgcaattgtcataaatcgctggcgcat780
ctgttgatgatgaacgatgtagtgccagtctggctgaaaccgacgcgtaatgcgttgggg840
attcttggtgggatcccgcgccgtgaatttactcgcgacagcatcgaagagaaagtcgct900
gctaccacgcaagcacaatggccggttcatgcggtgatcaccaactccacctatgatggc960
ttgctctacaacaccgactggatcaaacagacgctggatgtcccgtcgattcacttcgat1020
tctgcctgggtgccgtacacccattttcatccgatctaccagggtaaaagtggtatgagc1080
ggcgagcgtgttgcgggaaaagtgatcttcgaaacgcaatcgacccacaaaatgctggcg1140
gcgttatcgcaggcttcgctgatccacattaaaggcgagtatgacgaagaggcctttaac1200
gaagcctttatgatgcataccaccacctcgcccagttatcccattgttgcttcggttgag1260
acggcggcggcgatgctgcgtggtaatccgggcaaacggctgattaaccgttcagtagaa1320
cgagctctgcattttcgcaaagaggtccagcggctgcgggaagagtctgacggttggttt1380
ttcgatatctggcaaccgccgcaggtggatgaagccgaatgctggcccgttgcgcctggc1440
gaacagtggcacggctttaacgatgcggatgccgatcatatgtttctcgatccggttaaa1500
gtcactattttgacaccggggatggacgagcagggcaatatgagcgaggaggggatcccg1560
gcggcgctggtagcaaaattcctcgacgaacgtgggatcgtagtagagaaaaccggccct1620
tataacctgctgtttctctttagtattggcatcgataaaaccaaagcaatgggattattg1680
cgtgggttgacggaattcaaacgctcttacgatctcaacctgcggatcaaaaatatgcta1740
cccgatctctatgcagaagatcccgatttctaccgcaatatgcgtattcaggatctggca1800
caagggatccataagctgattcgtaaacacgatcttcccggtttgatgttgcgggcattc1860
gatactttgccggagatgatcatgacgccacatcaggcatggcaacgacaaattaaaggc1920
gaagtagaaaccattgcgctggaacaactggtcggtagagtatcggcaaatatgatcctg1980
ccttatccaccgggcgtaccgctgttgatgcctggagaaatgctgaccaaagagagccgc2040
acagtactcgattttctactgatgctttgttccgtcgggcaacattaccccggttttgaa2100
acggatattcacggcgcgaaacaggacgaagacggcgtttaccgcgtacgagtcctaaaa2160
atggcgggataacttgccagagcggcttccggaagcttggcactggccgtcgttttacaa2220
cg2222
<210>17
<211>36
<212>dna
<213>人工序列(artificialsequence)
<400>17
ttgtgagcggataacaatttcacacaggaggagctc36
<210>18
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>18
tctagagtcgacctgcaggcatgcaagctt30
<210>19
<211>60
<212>dna
<213>人工序列(artificialsequence)
<400>19
ttgtgagcggataacaatttcacacaggaggagctcatggtttccgaaactaaaaccaca60
<210>20
<211>56
<212>dna
<213>人工序列(artificialsequence)
<400>20
aagcttgcatgcctgcaggtcgactctagattatttcttatcgttctgcgggaact56
<210>21
<211>1539
<212>dna
<213>大肠杆菌(escherichiacoli)
<400>21
ttgtgagcggataacaatttcacacaggaggagctcatggtttccgaaactaaaaccaca60
gaagcgccgggcttacgccgtgaattaaaggcgcgtcacctgacgatgattgccattggc120
ggttccatcggtacaggtctttttgttgcctctggcgcaacgatttctcaggcaggtccg180
ggcggggcattgctctcgtatatgctgattggcctgatggtttacttcctgatgaccagt240
ctcggtgaactggctgcatatatgccggtttccggttcgtttgccacttacggtcagaac300
tatgttgaagaaggctttggcttcgcgctgggctggaactactggtacaactgggcggtg360
actatcgccgttgacctggttgcagctcagctggtcatgagctggtggttcccggataca420
ccgggctggatctggagtgcgttgttcctcggcgttatcttcctgctgaactacatctca480
gttcgtggctttggtgaagcggaatactggttctcactgatcaaagtcacgacagttatt540
gtctttatcatcgttggcgtgctgatgattatcggtatcttcaaaggcgcgcagcctgcg600
ggctggagcaactggacaatcggcgaagcgccgtttgctggtggttttgcggcgatgatc660
ggcgtagctatgattgtcggcttctctttccagggaaccgagctgatcggtattgctgca720
ggcgagtccgaagatccggcgaaaaacattccacgcgcggtacgtcaggtgttctggcga780
atcctgttgttctatgtgttcgcgatcctgattatcagcctgattattccgtacaccgat840
ccgagcctgctgcgtaacgatgttaaagacatcagcgttagtccgttcaccctggtgttc900
cagcacgcgggtctgctctctgcggcggcggtgatgaacgcagttattctgacggcggtg960
ctgtcagcgggtaactccggtatgtatgcgtctactcgtatgctgtacaccctggcgtgt1020
gacggtaaagcgccgcgcattttcgctaaactgtcgcgtggtggcgtgccgcgtaatgcg1080
ctgtatgcgacgacggtgattgccggtctgtgcttcctgacctccatgtttggcaaccag1140
acggtatacctgtggctgctgaacacctccgggatgacgggttttatcgcctggctgggg1200
attgccattagccactatcgcttccgtcgcggttacgtattgcagggacacgacattaac1260
gatctgccgtaccgttcaggtttcttcccactggggccgatcttcgcattcattctgtgt1320
ctgattatcactttgggccagaactacgaagcgttcctgaaagatactattgactggggc1380
ggcgtagcggcaacgtatattggtatcccgctgttcctgattatttggttcggctacaag1440
ctgattaaaggaactcacttcgtacgctacagcgaaatgaagttcccgcagaacgataag1500
aaataatctagagtcgacctgcaggcatgcaagcttctt1539
- 还没有人留言评论。精彩留言会获得点赞!