通过整合发光报告基因反映体外无细胞蛋白表达水平的方法与流程
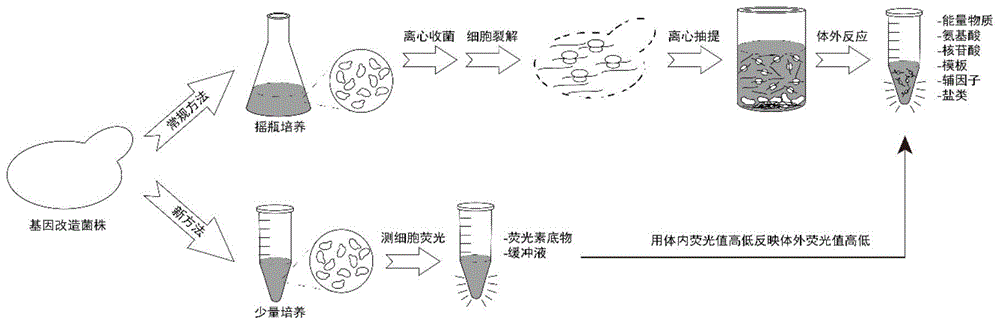
本发明属于生物技术领域,具体而言,是通过整合发光报告基因反映体外无细胞外源蛋白表达水平的方法及其相应的应用。
背景技术:
蛋白质是细胞中的重要分子,几乎参与了细胞所有功能的执行。蛋白的序列和结构不同,决定了其功能的不同。在细胞内,蛋白可以作为酶类催化各种生化反应,可以作为信号分子协调生物体的各种活动,可以支持生物形态,储存能量,运输分子,并使生物体运动。在生物医学领域,蛋白质抗体作为靶向药物,是治疗癌症等疾病的重要手段。
除了人们对于细胞内蛋白质合成的了解之外,蛋白质合成也可以在细胞外进行。蛋白质体外无细胞合成系统一般是指在细菌、真菌、植物细胞或动物细胞的裂解体系中,加入mrna或者dna模板、rna聚合酶及氨基酸和atp等组分,完成外源蛋白的快速高效翻译。体外翻译系统中表达蛋白质无需进行细胞转化、细胞培养、细胞收集和破碎步骤,是一种快速、省时、便捷的蛋白质表达方式。目前,经常实验的商业化体外无细胞蛋白表达系统包括大肠杆菌系统(e.coliextract,ece)、兔网织红细胞(rabbitreticulocytelysate,rrl)、麦胚(wheatgermextract,wge)、昆虫(insectcellextract,ice)和人源系统。与传统的体内重组表达系统相比,蛋白质的体外无细胞合成系统具有多种优点,如可表达对细胞有毒害作用或含有非天然氨基酸(如d-氨基酸)的特殊蛋白质,能够直接以pcr产物作为模板同时平行合成多种蛋白质,开展高通量药物筛选和蛋白质组学的研究。
体外无细胞蛋白合成体系(cell-freeproteinsynthesis,cfps)中,细胞裂解液或细胞提取物是很重要的一个组分,直接来自于菌株细胞,通过基因组编辑技术可以获得各式各样的改造菌株,但是对于任何改造效果的评估最后都需要实验数据来检测。例如将改造后的菌株应用于体外无细胞蛋白合成,常规的实验步骤包括摇瓶或发酵罐发酵培养、细胞收集、细胞破碎、固液分离、构建无细胞蛋白合成体系、蛋白合成、分析检测等,完成整个流程至少需要约一周的时间,耗时耗力成本高,对于菌株的筛选效率不高。因此,在无细胞蛋白合成领域中迫切需要一种可以快速检测基因改造效果,提高菌株筛选效率的方法。
技术实现要素:
本发明的目的是,提供一种可以快速检测基因改造效果,提高菌株筛选效率的方法。具体为,通过整合发光报告基因反映体外无细胞蛋白表达水平。
为达到上述目的,本发明的第一方面提供一种反映菌株体外无细胞外源蛋白合成能力的方法,包括以下步骤:
(i)将发光报告基因整合到第一目标菌株的基因组中管家基因的5’端和/或3’端,获得整合发光报告基因的第一对照菌株,所述第一目标菌株是原始菌株或由原始菌株经未整合所述发光报告基因的其他任意基因改造的非原始菌株;
(ii)在所述第一对照菌株和第一目标菌株的基础上,分别进行除整合所述发光报告基因外的其他任意相同的基因改造,由第一对照菌株获得第二对照菌株,由第一目标菌株获得第二目标菌株;如所述第一目标菌株为非原始菌株,则步骤(i)中的基因改造与本步骤中的基因改造不相同;
(iii)扩大培养步骤(i)中的第一对照菌株或步骤(ii)中的第二对照菌株,使得所述发光报告基因在第一对照菌株或第二对照菌株内表达;
(iv)不经细胞破碎,检测培养后菌株的发光信号强度,通过所述第一对照菌株的发光信号强度反映所述第一目标菌株或第一对照菌株的体外无细胞外源蛋白合成能力;通过所述第二对照菌株的发光信号强度反映所述第二目标菌株或第二对照菌株的体外无细胞外源蛋白合成能力。
优选的,所述第一对照菌株的发光信号强度与所述第一对照菌株或第一目标菌株的体外无细胞外源蛋白合成能力正相关;或所述第二对照菌株的发光信号强度与所述第二对照菌株或第二目标菌株的体外无细胞外源蛋白合成能力正相关。
进一步优选的,所述管家基因为肌动蛋白基因、微管蛋白基因、核糖体蛋白基因或甘油醛-3-磷酸脱氢酶基因。微管蛋白基因包括但不限于β-tubulin(β-微管蛋白)基因、α-tubulin(α-微管蛋白)基因,甘油醛-3-磷酸脱氢酶基因包括但不限于gapdh1、gapdh2基因。优选的为α-tubulin基因、β-tubulin基因。
优选的,所述发光报告基因的5’端非翻译区具有与体外无细胞蛋白合成体系中外源蛋白的dna模板的5’端非翻译区相同的多核苷酸序列。
进一步优选的,所述发光报告基因的3’端非翻译区具有与体外无细胞蛋白合成体系中外源蛋白的dna模板的3’端非翻译区相同的多核苷酸序列。
优选的,所述发光信号强度是将检测到的发光值与扩大培养后菌液的od值相比后获得的比值。
其中,不同的所述第一对照菌株进行扩大培养前的初始od值可以相同也可以不相同,优选具有相同的od值;或不同的所述第二对照菌株进行扩大培养前的初始od值可以相同也可以不相同,优选具有相同的od值。
优选的,所述发光报告基因为荧光素酶基因或荧光蛋白基因。进一步优选的,所述荧光素酶基因为荧光素酶nanoluc基因。
优选的,不同的整合有荧光素酶nanoluc基因的第一对照菌株或第二对照菌株的发光信号强度,是在扩大培养后菌液中加入荧光素酶nanoluc特异性的底物后立即进行检测,或者反应相同时间后进行检测。
其中,不同菌株的扩大培养过程(培养条件、时间等)可以相同也可以不相同。优选的,所述第一目标菌株的扩大培养条件及时间,与所述第一对照菌株的扩大培养条件及时间一致;或所述第二目标菌株的扩大培养条件及时间,与所述第二对照菌株的扩大培养条件及时间一致。
优选的,不同第一目标菌株或不同第一对照菌株或不同第二目标菌株或不同第二对照菌株的扩大培养条件及时间一致。
优选的,所述第一对照菌株或第一目标菌株或第二目标菌株或第二对照菌株来源于大肠杆菌、酵母细胞;所述酵母细胞来源于酿酒酵母、毕赤酵母、克鲁维酵母;所述克鲁维酵母为乳酸克鲁维酵母。
本发明第二方面提供一种用于反映体外无细胞外源蛋白合成能力的菌株,所述菌株的基因组中管家基因的5’端和/或3’端整合有发光报告基因,所述发光报告基因可以在所述菌株内表达报告蛋白,通过检测所述报告蛋白发光信号强度反映体外无细胞外源蛋白合成能力。
优选的,本发明第二方面所述的菌株可以用于反映其自身的体外无细胞外源蛋白合成能力。进一步优选的,在本发明第二方面所述菌株的基础上,进行经未整合所述发光报告基因的其他任意基因改造,由此获得的改造菌株可以通过检测该改造菌株细胞内发光反映前述的其他任意基因改造对于体外无细胞外源蛋白合成能力的影响。
优选的,所述管家基因为肌动蛋白基因、微管蛋白基因、核糖体蛋白基因或甘油醛-3-磷酸脱氢酶基因。
优选的,所述发光报告基因为荧光素酶基因或荧光蛋白基因。进一步优选的,所述荧光素酶基因为荧光素酶nanoluc基因。
优选的,所述菌株来源于大肠杆菌、酵母细胞;所述酵母细胞来源于酿酒酵母、毕赤酵母、克鲁维酵母;所述克鲁维酵母为乳酸克鲁维酵母。
本发明第三方面提供一种如本发明第二方面所述的菌株在反映体外无细胞外源蛋白合成能力中的应用。
本发明的有益效果主要如下:
(1)将体外无细胞体系的体外检测转变为细胞内检测,无需细胞破碎等繁琐工作,可以在一天时间内得到检测结果,更方便快捷地获得能够初步反应体外无细胞蛋白合成体系活性的荧光数据,减少时间和人力成本,提高了筛选效率,也可用于高通量实验筛选。
(2)选择新型化学发光蛋白荧光素酶nanoluc(简写为nluc)基因作为报告基因,它是一种荧光素酶,其对应底物在该酶的作用下产生辉光型信号,其信号强度是传统荧光素酶(firefly荧光素酶和renilla荧光素酶)的100倍,且信号更持久,使得检测过程更加灵敏。
(3)选择转录强度比较稳定的位点作为报告基因的整合位点,不受或几乎不受环境等因素的调控。具体的,管家基因是组成性表达并且相当稳定的,不容易受到环境影响。
附图说明
图1细胞内发光报告基因表达来反映体外无细胞蛋白合成体系活性的方法示意图。
图2pkmcas9-βtubulin-grna1质粒图谱示意图。
图3pcas9-53nluc质粒图谱示意图。
图4pkmd1-βtubulin-53nluc质粒图谱示意图。
图5tnl1,tnl2,tnl3三株菌细胞内发光信号强度(a)与体外无细胞蛋白合成体系活性(b)以及km3a,kmg5,kmp9三株菌体外无细胞蛋白合成体系活性(c)的对比。
图6pkmcas9-tpk1-grna质粒图谱示意图。
图7pkmd1-tpk1-s237d质粒图谱示意图。
图8pkmcas9-edc3-grna1质粒图谱示意图。
图9pkmcas9-edc3-grna2质粒图谱示意图。
图10pkmd1-edc3-ko质粒图谱示意图。
图11ynl1,l2t4,l2e1三株菌细胞内发光信号强度(a)与体外无细胞蛋白合成体系活性(b)以及y1140、tpk1s237d、δedc3三株菌体外无细胞蛋白合成体系活性(c)的比较图。
具体实施方式
本发明中的“体外无细胞蛋白合成体系”、“无细胞蛋白合成体系”、“体外蛋白合成体系”、“无细胞体外蛋白表达体系”、“体外蛋白合成系统”、“蛋白质体外合成系统”、“体外翻译系统”、“体外蛋白表达系统”、“cfps”等表述具有相同的含义。
本发明中,原始菌株可以作为出发菌株,原始菌株为未经过任何基因改造的菌株。
本发明所称的无细胞蛋白合成体系的活性,指的是该无细胞蛋白合成体系用于外源蛋白合成时,所能够合成外源蛋白的能力,可以用外源蛋白的浓度、产量等来表示,也可以用任何与外源蛋白产量相关的可检测的信号值来表示;例如外源蛋白是荧光蛋白时,可以用相对荧光单位值(rfu)来表示。
本发明中,所述发光报告基因是指整合到菌株基因组中的基因,该基因可以在细胞内转录、翻译成报告蛋白,该报告蛋白的发光可以通过实验仪器检测或肉眼直接观测到。比如荧光素酶基因、各种荧光蛋白基因等,经过转录翻译,获得荧光素酶或荧光蛋白,可经过反应后或直接或发出光信号,并可被检测发光信号强度(或发光值)。
对于荧光素酶来说,发光信号强度(或发光值)是指荧光素酶与其特异性底物发生反应,反应过程中检测到的发光信号强度(或发光值)。对于荧光蛋白来说,发光信号强度(或发光值)是指荧光蛋白分子受到激发(如光激发)后发出荧光,检测到的该荧光强度(或荧光值)。
虽然nluc荧光素酶相比于其他荧光素酶具有较好的优点,但并不排除其他荧光素酶作为报告蛋白的可能。细胞内发光报告基因表达的报告蛋白并不仅限于nluc荧光素酶,也可利用其他荧光素酶如firefly荧光素酶、renilla荧光素酶,或者各种荧光蛋白如青色荧光蛋白(cyanfluorescentproteins)、增强型绿色荧光蛋白egfp、绿色荧光蛋白(greenfluorescentproteins)、黄色荧光蛋白(yellowfluorescentproteins)、蓝色荧光蛋白(bluefluorescentproteins)、橙色荧光蛋白(orangefluorescentproteins)、红色荧光蛋白(redfluorescentproteins)等,具体的,荧光蛋白可以选自emerald、superfoldergfp、azamigreen、mwasabi、taggfp、turbogfp、acgfp、zsgreen、t-sapphire、ebfp、ebfp2、azurite、mtagbfp、ecfp、mecfp、cerulean、cypet、amcyan1、midori-ishicyan、tagcfp、eyfp、topaz、venus、mcitrine、ypet、tagyfp、phiyfp、zsyellow1、mbanana、kusabiraorange、kusabiraorange2、morange、morange2、tdtomato、tagrfp、dsred、mtangerine、mruby、mapple、mstrawberry、asred2、mrfp1、jred、mplum、aq143、moxcerulean3、amcyan1、micy、megfp、clover、mvenus、meos3.2、mko2、turborfp、tdtomato、mscarlet-i、eqfp611、mkate1.3、mneptune2、mirfp670、mtagbfp2、pamcherry、mametrine、azamigreen等。
本发明中,选择具有比较稳定转录强度的基因作为报告基因整合的位点基因,优选的,选择管家基因,由于管家基因是组成性表达并且相当稳定的,不容易受到环境影响。进一步优选的,所述管家基因选自actin(肌动蛋白)基因、tubulin(微管蛋白)基因、gapdh(甘油醛-3-磷酸脱氢酶)基因、核糖体蛋白基因,这些基因在western-blot和real-timepcr技术中经常作为内参而被使用。优选的,将转录水平较低的βtubulin(β微管蛋白)基因位点作为整合发光报告基因的位点。转录水平相对较高的位点基因同样适应于本发明技术方案。
本发明中,所述发光报告基因的上游5’端序列元件及下游3’端序列元件,可以对基因的表达起到调控作用,上游5’端序列元件可以包括启动子、5’utr(5’端非翻译区)及其他调控或连接元件,下游3’端序列元件可以包括3’utr(3’端非翻译区,3’utr包含终止子序列)。优选的,发光报告基因的5’端非翻译区具有与体外无细胞蛋白合成体系中外源蛋白的dna模板的5’端非翻译区相同的多核苷酸序列。优选的,发光报告基因的3’端非翻译区具有与体外无细胞蛋白合成体系中外源蛋白的dna模板的3’端非翻译区相同的多核苷酸序列。进一步优选的,发光报告基因的5’端序列元件的至少一部分,具有与体外无细胞蛋白合成体系中外源蛋白的dna模板的5’端相同的多核苷酸序列。进一步优选的,发光报告基因的3’端序列元件的至少一部分,具有与体外无细胞蛋白合成体系中外源蛋白的dna模板的3’端相同的多核苷酸序列。
本发明中,以未插入报告基因的经过其他基因改造后的菌株为基础菌株,进一步在其基因组中管家基因的5’端和/或3’端整合发光报告基因,经过扩大培养后检测发光信号,从而评估基础菌株体外无细胞蛋白合成体系的活性,甚至是基础菌株的体外外源蛋白合成能力。更合适的方法是,将基因组中管家基因的5’端和/或3’端整合了发光报告基因的菌株作为出发菌株,在此基础上做各种基因改造,经过细胞培养后检测发光信号强度,用于评估相应出发菌株的各种基因改造对无细胞蛋白合成体系活性乃至细胞蛋白合成能力的影响,即反映各种基因改造菌株的无细胞蛋白合成体系的活性,或者说各种基因改造菌株的无细胞蛋白合成能力。
本发明的技术方案可以应用于各种不同的菌株,包括但不限于大肠杆菌、酵母等,酵母可以是酿酒酵母、毕赤酵母、克鲁维酵母等,克鲁维酵母如乳酸克鲁维酵母、马克斯克鲁维酵母等。本发明的技术方案也可以应用于各种不同的细胞株,包括但不限于哺乳动物细胞(如hf9、hela、cho、hek293)、小麦胚芽细胞、兔网织红细胞、昆虫细胞等。
在本发明中,体外无细胞蛋白合成体系不受特别限制,往往以细胞提取物或细胞裂解液的来源进行区分,来源可以是大肠杆菌、小麦胚芽细胞、兔网织红细胞、昆虫细胞、哺乳动物细胞、酵母细胞等;一种优选的无细胞蛋白合成体系为酵母(如酿酒酵母、毕赤酵母、克鲁维酵母等)体外蛋白合成体系,较佳的为克鲁维酵母体外蛋白合成体系,更佳地,乳酸克鲁维酵母(kluyveromyceslactis,k.lactis)体外蛋白合成体系。
酵母(yeast)兼具培养简单、高效蛋白质折叠、和翻译后修饰的优势。其中酿酒酵母(saccharomycescerevisiae)和毕氏酵母(pichiapastoris)是表达复杂真核蛋白质和膜蛋白的模式生物,酵母也可作为制备体外翻译系统的原料。
克鲁维酵母(kluyveromyces)是一种子囊孢子酵母,其中的马克斯克鲁维酵母(kluyveromycesmarxianus)和乳酸克鲁维酵母(kluyveromyceslactis)是工业上广泛使用的酵母。与其他酵母相比,乳酸克鲁维酵母具有许多优点,如超强的分泌能力,更好的大规模发酵特性、食品安全的级别、以及同时具有蛋白翻译后修饰的能力等。
本发明的体外无细胞蛋白合成体系包括:细胞提取物或细胞裂解液、用于合成蛋白的底物、用于合成rna的底物和编码外源蛋白的dna模板。
优选的,所述蛋白合成体系还包括下组的一种或多种组分:能量再生系统、聚乙二醇、镁离子、钾离子、缓冲剂、rna聚合酶、抗氧化剂、任选的水性溶剂。
在本发明中,所述细胞提取物或细胞裂解液的含量和纯度没有特别限制。优选的,所述细胞提取物或细胞裂解液的浓度(v/v)为20%-70%,较佳地,30-60%,更佳地,40%-50%,以所述蛋白合成体系的总体积计。
在本发明中,所述的细胞提取物不含完整的细胞,典型的细胞提取物包括用于rna合成所需的各种类型的rna聚合酶,和用于蛋白翻译的核糖体、转运rna、氨酰trna合成酶、蛋白质合成需要的起始因子和延伸因子以及终止释放因子。此外,细胞提取物中还含有一些源自细胞的细胞质中的其他蛋白,尤其是可溶性蛋白。
在本发明中,所述的细胞提取物的制备方法不受限制,一种优选的制备方法包括以下步骤:
(i)提供细胞;
(ii)对细胞进行洗涤处理,获得经洗涤的细胞;
(iii)对经洗涤的细胞进行破细胞处理,从而获得细胞粗提物;
(iv)对所述细胞粗提物进行固液分离,获得液体部分,即为细胞提取物。
在本发明中,所述的固液分离方式不受特别限制,一种优选的方式为离心。
在一优选实施方式中,所述离心在液态下进行。
在本发明中,所述离心条件不受特别限制,一种优选的离心条件为5000-100000g,较佳地,8000-30000g。
在本发明中,所述离心时间不受特别限制,一种优选的离心时间为0.5min-2h,较佳地,20min-50min。
在本发明中,所述离心的温度不受特别限制,优选的,所述离心在1-10℃下进行,较佳地,在2-6℃下进行。
在本发明中,所述的洗涤处理方式不受特别限制,一种优选的洗涤处理方式为采用洗涤液在ph为7-8(较佳地,7.4)下进行处理,所述洗涤液没有特别限制,典型的所述洗涤液选自下组:4-羟乙基哌嗪乙磺酸钾、醋酸钾、醋酸镁之一或其组合。
在本发明中,所述破细胞处理的方式不受特别限制,一种优选的所述的破细胞处理包括高压破碎、冻融(如液氮低温)破碎。
在另一优选例中,所述能量再生系统是能够产生atp的体系,包括但不限于:磷酸肌酸和磷酸肌酸酶系统、糖酵解途径及其中间产物能量系统、多糖和磷酸盐能量体系或其组合。进一步的,所述多糖选自淀粉、糊精;所述磷酸盐选自正磷酸盐、磷酸二氢盐、磷酸氢二盐、偏磷酸盐、焦磷酸盐或其组合。
进一步的,所述细胞提取物为对细胞的水性提取物。
进一步的,所述细胞提取物不含内源性的长链核酸分子。
进一步的,所述的合成rna的底物包括:核苷单磷酸、核苷三磷酸之一或其组合。
进一步的,所述的合成蛋白质的底物包括:20种天然氨基酸以及非天然氨基酸。
进一步的,所述的抗氧化剂为二硫苏糖醇(dtt)。
进一步的,所述镁离子来源于镁离子源,所述镁离子源选自下组:醋酸镁、谷氨酸镁之一或其组合。
进一步的,所述钾离子来源于钾离子源,所述钾离子源选自下组:醋酸钾、谷氨酸钾之一或其组合。
进一步的,所述缓冲剂选自下组:4-羟乙基哌嗪乙磺酸、三羟甲基氨基甲烷之一或其组合。
下面结合具体实施例,进一步阐述本发明。应理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。其中,各个实施例中描述核苷三磷酸混合物及氨基酸混合物的浓度,指的是混合物中单一物质浓度,而非混合物总物质浓度。实施例中以乳酸克鲁维酵母为例进行试验,但并不代表该试验仅能用于乳酸克鲁维酵母,本发明的实验设计、技术思路及技术方案同样可应用于其它真核细胞(如植物细胞、其它酵母细胞、昆虫细胞、动物细胞等)与原核细胞(如大肠杆菌细胞)。通过对野生型乳酸克鲁维酵母kluyveromyceslactisnrrly-1140(简称y1140)进行了转录组分析,获得了绝大多数基因的转录水平数据,其中包括actin(肌动蛋白)基因、βtubulin(β微管蛋白)基因、α-tubulin(α-微管蛋白)基因、核糖体蛋白基因、gapdh1和gapdh2(均为甘油醛-3-磷酸脱氢酶)基因。实施例中选择转录水平相对较低的βtubulin基因的下游作为nluc基因的整合位点,可以在nluc蛋白的细胞内表达提升时,测得的发光信号强度的增强更加明显。但这并不代表该技术方案仅能用于转录水平较低的基因位点,本发明的技术方案同样适用于具有稳定转录水平的任何其它管家基因位点。
实施例1
1.1crisprgrna序列的确定
本实施例中grna选择的原则为:靠近βtubulin基因的3’端,gc含量适中(40%-60%),避免polyt结构的存在。在本实施例中,βtubulingrna1序列为cagtagcatcttgatattgt。
质粒构建及转化方法如下:使用引物pcas9-βtubulin-grna1-pf:cagtagcatcttgatattgtgttttagagctagaaatagc和pcas9-βtubulin-grna1-pr:acaatatcaagatgctactggattcgaactgccgagaaagtaac,以pcas质粒为模板,进行pcr扩增。取扩增产物17μl,加入1μldpni,2μl10×digestionbuffer,混匀后37℃温浴3h。取dpni处理后产物10μl加入50μldh5α感受态细胞中,冰上放置30min,42℃热激45s后,加入1mllb液体培养基37℃振荡培养1h,涂布于kan抗性lb固体培养,37℃倒置培养至单克隆长出。挑取2个单克隆在lb液体培养基中振荡培养,pcr检测阳性并测序确认后,提取质粒保存,命名为pkmcas9-βtubulin-grna1(图2)。
1.2构建供体质粒pkmd1-βtubulin-53nluc
以pmd18t质粒为模板,引物pmd18t-pf:atcgtcgacctgcaggcatg和pmd18t-pr:atctctagaggatccccggg进行pcr扩增,取扩增产物17μl,加入1μldpni,2μl10×digestionbuffer,混匀后37℃水浴3h,获得质粒骨架线性片段pmd18t-vector。
以k.lactis基因组dna为模板,用引物βtubulin-hr1-pf:ggtatgggtactttgttgatctc和βtubulin-grna1-m-pr:cttcaactgttgcgtcctggtactgctgatattcacttactaagtcattc进行pcr扩增,产物名为βtubulin-hr1-l;以k.lactis基因组dna为模板,用引物βtubulin-grna1-m-pf:gtgaatatcagcagtaccaggacgcaacagttgaagatgacgaagaattg和βtubulin-hr1-pr:ttactcgaagttttcagcca进行pcr扩增,产物名为βtubulin-hr1-r;以pcas9-53nluc质粒(如图3,pcas9为质粒骨架,该质粒骨架仅起到使53nluc成环的作用,53nluc为插入片段,其结构依次包含t7启动子、5’utr、nluc编码框、3’utr,nluc编码框序列可在promega公司网站查到)为模板,用引物βtubulin-53nluc-pf:gaacaaccaatggctgaaaacttcgagtaaggcgtagaggatcgagatct和引物βtubulin-53nluc-pr:ttttcctgctgaagttttcctttatattctatccggatatagttcctcct进行pcr扩增,产物名为53nluc(核苷酸序列如seqidno:1所示);以k.lactis基因组dna为模板,用引物βtubulin-hr2-pf:agaatataaaggaaaacttc和βtubulin-hr2-pr:gatcagttactggcgagttctcc进行pcr扩增,产物名为βtubulin-hr2。
将扩增产物βtubulin-hr1-l、βtubulin-hr1-r、βtubulin-hr2、53nluc、pmd18t-vector各1μl,加入5μlcloningmix,混匀后50℃水浴1h。水浴结束后置于冰上2min,将10μl反应液全部加入50μltrans-t1感受态细胞中,冰上放置30min,42℃热激30s后,加入1mllb液体培养基37℃振荡培养1h,涂布于amp抗性lb固体培养,37℃倒置培养至单克隆长出。挑取6个单克隆在lb液体培养基中振荡培养,pcr检测阳性并测序确认后,提取质粒保存,命名为pkmd1-βtubulin-53nluc(图4)。
1.3乳酸克鲁维酵母的电转化
将不同基因型的k.lactis菌株(分别命名为km3a,kmg5,kmp9)分别制作成电转化感受态,加入400ngpkmcas9-βtubulin-grna1质粒和1000ngβtubulin-53nluc片段(以质粒pkmd1-βtubulin-53nluc为模板,用引物βtubulin-pf-n1:cgttgtagaaccatacaatg与引物βtubulin-pr-n1:ccgtccgacatcctagattg进行pcr扩增获得βtubulin-53nluc片段,其核苷酸序列如seqidno:2所示)片段,混匀后全部转入电击杯中,冰浴2min;将电击杯放入电转仪中进行电击(参数为1.5kv,200ω,25μf);电击结束后立即加入700μlypd,30℃,200rpm摇床孵育3h;取100μl涂布于ypd(含g418抗性)平板,30℃培养1~2天至单菌落出现。
1.4阳性鉴定
在细胞转化后的平板上挑取12-24个单克隆,以细胞为模板,用鉴定引物βtubulin-cf:cattttcggtcaatcttcag和βtubulin-cr:ctaatgaagaacttggcatg对样品进行pcr检测。pcr结果阳性并经测序鉴定的细胞株,确定为阳性细胞株并分别命名为tnl1,tnl2,tnl3。
1.5检测tnl1,tnl2,tnl3的nluc发光信号强度
将tnl1,tnl2,tnl3这三株菌转接含有4mlypd培养基的试管,使初始od600一致,30℃培养18h左右。将菌液分别测量od600后,都用d-pbs缓冲液稀释20倍,各自混匀后加10μl至白色酶标板的孔中,每个菌株做三个平行样品。在每个孔中同时加入10μl工作浓度的底物furimazine溶液(furimazine购买自promega公司,casno.1374040-24-0,溶于d-pbs缓冲液)后,立即置于酶标仪中读数,荧光值与od值的比值结果如图5a。
1.6体外无细胞蛋白合成体系合成外源蛋白
与此同时,对上述六株不同基因型的k.lactis菌株(分别命名为km3a,kmg5,kmp9,以及tnl1,tnl2,tnl3)按照常规的cfps,利用体外蛋白质合成反应体系分别测定它们的无细胞蛋白合成活性。
所使用的体外蛋白质合成反应体系为:终浓度为9.78mmph为8.0的三羟甲基氨基甲烷(tris-hcl),80mm醋酸钾,5.0mm醋酸镁,1.5mm核苷三磷酸混合物(腺嘌呤核苷三磷酸、鸟嘌呤核苷三磷酸、胞嘧啶核苷三磷酸和尿嘧啶核苷三磷酸,每种核苷三磷酸的浓度均为1.5mm),0.7mm的氨基酸混合物(甘氨酸、丙氨酸、缬氨酸、亮氨酸、异亮氨酸、苯丙氨酸、脯氨酸、色氨酸、丝氨酸、酪氨酸、半胱氨酸、蛋氨酸、天冬酰胺、谷氨酰胺、苏氨酸、天冬氨酸、谷氨酸、赖氨酸、精氨酸和组氨酸,各自浓度均为0.7mm),1.7mm二硫苏糖醇(dtt),2%的聚乙二醇,15mm葡萄糖,24mm磷酸三钾,50%体积的酵母细胞提取物(六种细胞提取物分别来自于前述km3a,kmg5,kmp9,以及tnl1,tnl2,tnl3六株菌),15ng/μl增强型绿色荧光蛋白dna模板(dna模板中绿色荧光蛋白的编码序列上游5’端序列如seqidno:3所示,下游3’端序列如seqidno:4所示)。
体外蛋白质合成反应条件:将上述的反应体系中混匀后放置在室温(20-30℃)的环境中反应20小时。
荧光值测定:将待测样品放置于envision2120多功能酶标仪(perkinelmer),检测获得相对荧光单位值(relativefluorescenceunit,rfu),用rfu值表示无细胞蛋白合成体系的活性,结果如图5b、5c所示。
由图5可以看出,含有nanoluc基因的三株菌tnl1、tnl2、tnl3的发光报告值(图5a)与体外无细胞蛋白合成体系的活性(图5b、图5c)正相关。
实施例2
2.1构建菌株ynl1
参照实施例1.3的步骤,以野生型乳酸克鲁维酵母kluyveromyceslactisnrrly-1140(简称为y1140)作为感受态细胞进行转化,再参照1.4的步骤进行阳性鉴定,获得的阳性细胞株命名为ynl1。
2.2crisprgrna序列的确定
已申请专利(专利申请号:2018114520758)中的实验表明k.lactis中tpk1基因的s237d改造(获得的菌株为tpk1s237d)能够提高cfps活性,通过搜索该s237残基附近的pam序列(ngg),确定对应的grna以切割造成双链断裂,grna选择的原则为:gc含量适中(40%-60%),避免polyt结构的存在。最终,本实施例的tpk1的grna序列为gtctcaaaggttcccaaacc。
用引物为tpk1-grna-pf:gtctcaaaggttcccaaaccgttttagagctagaaatagc和引物tpk1-grna-pr:ggtttgggaacctttgagacgattcgaactgccgagaaagtaac。以pcas质粒为模板,进行pcr扩增。取扩增产物17μl,加入0.2μldpni,2μl10×digestionbuffer,混匀后37℃水浴3h。取dpni处理后产物10μl加入50μldh5α感受态细胞中,冰上放置30min,42℃热激45s后,加入1mllb液体培养基37℃振荡培养1h,涂布于kan抗性lb固体培养,37℃倒置培养至单克隆长出。挑取2个单克隆在lb液体培养基中振荡培养,pcr检测阳性并测序确认后,提取质粒保存,命名为pkmcas9-tpk1-grna(图6)。
2.3构建供体质粒pkmd1-tpk1-s237d
以pmd18t质粒为模板,引物pmd18t-pf:atcgtcgacctgcaggcatg和pmd18t-pr:atctctagaggatccccggg进行pcr扩增,取扩增产物17μl,加入1μldpni,2μl10×digestionbuffer,混匀后37℃水浴3h,获得质粒骨架线性片段pmd18t-vector。
以k.lactis基因组dna为模板,用引物kltpk1-hr1-pf:gagctcggtacccggggatcctctagagatgaaccgctatatcttgcatg和kltpk1-mutant-pr:gactggattaggaaaacgctggtctttccttaaaagagagaatag进行pcr扩增,产物名为tpk1-hr1;
以k.lactis基因组dna为模板,用引物kltpk1-mutant-pf:aaggaaagaccagcgttttcctaatccagtcgcgaagttttatgcagcag和kltpk1-hr2-pr:gcatgcctgcaggtcgacgattaacggcagcgtttctgaag进行pcr扩增,产物名为tpk1-hr2;
将扩增产物tpk1-hr1、tpk1-hr2和pmd18t-vector各1μl,加入3μlcloningmix(transgenepeasy-uniseamlesscloningandassemblykit,全式金公司,下同),混匀后50℃水浴1h。水浴结束后置于冰上2min,将6μl反应液全部加入50μltrans-t1感受态细胞中,冰上放置30min,42℃热激30s后,加入1mllb液体培养基37℃振荡培养1h,涂布于amp抗性lb固体培养,37℃倒置培养至单克隆长出。挑取6个单克隆在lb液体培养基中振荡培养,pcr检测阳性并测序确认后,提取质粒保存,命名为pkmd1-tpk1-s237d(图7)。
2.4乳酸克鲁维酵母的电转化
将实施例2.1中得到的ynl1菌株制作成电转化感受态,加入400nggrna&cas9质粒(或grna/cas9片段)和1000ng供体dna片段(以质粒pkmd1-tpk1-s237d为模板,用引物kltpk1-hr1-pf与引物kltpk1-hr2-pr进行pcr扩增获得供体dna片段,其核苷酸序列如seqidno:5所示),混匀后全部转入电击杯中,冰浴2min;将电击杯放入电转仪中进行电击(参数为1.5kv,200ω,25μf);电击结束后立即加入700μlypd,30℃,200rpm摇床孵育1~3h;取2~200μl接种于ypd(含g418抗性)平板,30℃培养2~3天至单菌落出现。
2.5阳性鉴定
在细胞转化后的平板上挑取12-24个单克隆,以细胞为模板,用鉴定引物f:agtctcaaaggttcccaaac和r:ataagattattgcatcgagc对样品进行pcr检测。阴性菌株无pcr条带。pcr结果阳性并用引物对f:gggtatttcgaataagggac、r:ataagattattgcatcgagc经测序鉴定的细胞株,确定为阳性细胞株并命名为l2t4。
实施例3
3.1crisprgrna序列的确定
已申请专利(专利申请号:2018116083534)的实验表明k.lactis中edc3基因的敲除(获得的菌株为δedc3)能够提高cfps活性,本实施例中grna选择的原则为:靠近edc3基因内部的两端,gc含量适中(40%-60%),避免polyt结构的存在。在本实施例中,edc3grna1序列为tcaaattgagatcgaattga,edc3grna2序列为ggacatatacccgggtttct。
质粒构建及转化方法如下:使用引物pcas9-edc3-grna1-pf:tcaaattgagatcgaattgagttttagagctagaaatagc和pcas9-edc3-grna1-pr:tcaattcgatctcaatttgagattcgaactgccgagaaagtaac,以pcas质粒为模板,进行pcr扩增。取扩增产物17μl,加入1μldpni,2μl10×digestionbuffer,混匀后37℃温浴3h。取dpni处理后产物10μl加入50μldh5α感受态细胞中,冰上放置30min,42℃热激45s后,加入1mllb液体培养基37℃振荡培养1h,涂布于kan抗性lb固体培养,37℃倒置培养至单克隆长出。挑取2个单克隆在lb液体培养基中振荡培养,pcr检测阳性并测序确认后,提取质粒保存,命名为pkmcas9-edc3-grna1(图8)。
同理,使用引物pcas9-edc3-grna2-pf:ggacatatacccgggtttctgttttagagctagaaatagc和pcas9-edc3-grna2-pr:agaaacccgggtatatgtccgattcgaactgccgagaaagtaac,构建质粒命名为pkmcas9-edc3-grna2(图9)。
3.2构建供体质粒pkmd1-edc3-ko
以k.lactis基因组dna为模板,用引物edc3-hr1-pf:ggtagctccaataatccaag和edc3-hr1-pr:acacatctatctttacagatataagtcaaaagtgacttgttctataaact进行pcr扩增,产物名为edc3-hr1;以k.lactis基因组dna为模板,用引物edc3-hr2-pf:agaagctataagtttatagaacaagtcacttttgacttatatctgtaaag和edc3-hr2-pr:aactccaacaacgacgtcac进行pcr扩增,产物名为edc3-hr2。
将扩增产物edc3-hr1、edc3-hr2、pmd18t-vector各1μl,加入3.5μlcloningmix和0.5μl水,混匀后50℃水浴1h。水浴结束后置于冰上2min,将10μl反应液全部加入50μltrans-t1感受态细胞中,冰上放置30min,42℃热激30s后,加入1mllb液体培养基37℃振荡培养1h,涂布于amp抗性lb固体培养,37℃倒置培养至单克隆长出。挑取6个单克隆在lb液体培养基中振荡培养,pcr检测阳性并测序确认后,提取质粒保存,命名为pkmd1-edc3-ko(图10)。
3.3乳酸克鲁维酵母的电转化
将实施例2.1中得到的ynl1菌株制作成电转化感受态,加入pkmcas9-edc3-grna1质粒和pkmcas9-edc3-grna2质粒各200ng,再加1000ngedc3-ko片段(以质粒pkmd1-edc3-ko为模板,用引物edc3-hr1-pf与引物edc3-hr2-pr进行pcr扩增获得,核苷酸序列如seqidno:6所示)片段,混匀后全部转入电击杯中,冰浴2min;将电击杯放入电转仪中进行电击(参数为1.5kv,200ω,25μf);电击结束后立即加入700μlypd,30℃,200rpm摇床孵育3h;取100μl涂布于ypd(含g418抗性)平板,30℃培养1~2天至单菌落出现。
3.4阳性鉴定
在细胞转化后的平板上挑取12-24个单克隆,以细胞为模板,用鉴定引物edc3-cf:cgaatcaaacgttaaacatc和edc3-cr:agaaattcccatttccattg对样品进行pcr检测。pcr结果阳性并经测序鉴定的细胞株,确定为阳性细胞株并分别命名为l2e1。
实施例4
4.1ynl1,l2t4,l2e1的nluc荧光值测定
将实施例2.1中获得的ynl1菌株,实施例2中获得的l2t4菌株,以及实施例3中获得的l2e1菌株分别转接含有4mlypd培养基的试管,使初始od600一致,30℃培养18h左右。将菌液分别测量od600后,都用d-pbs缓冲液稀释20倍,各自混匀后加10μl至白色酶标板的孔中,每个菌株做三个平行样品。在每个孔中同时加入10μl工作浓度的底物furimazine溶液(furimazine购买自promega公司,casno.1374040-24-0,溶于d-pbs缓冲液)后,立即置于酶标仪中读数,荧光值与od值的比值结果如图11a。
4.2体外无细胞蛋白合成体系合成外源蛋白
与此同时,对上述六株菌株(ynl1、l2t4、l2e1,以及y1140、tpk1s237d、δedc3)按照常规的cfps,利用体外蛋白质合成反应体系分别测定它们的无细胞蛋白合成活性。
所使用的体外蛋白质合成反应体系为:终浓度为9.78mmph为8.0的三羟甲基氨基甲烷(tris-hcl),80mm醋酸钾,5.0mm醋酸镁,1.5mm核苷三磷酸混合物(腺嘌呤核苷三磷酸、鸟嘌呤核苷三磷酸、胞嘧啶核苷三磷酸和尿嘧啶核苷三磷酸,每种核苷三磷酸的浓度均为1.5mm),0.7mm的氨基酸混合物(甘氨酸、丙氨酸、缬氨酸、亮氨酸、异亮氨酸、苯丙氨酸、脯氨酸、色氨酸、丝氨酸、酪氨酸、半胱氨酸、蛋氨酸、天冬酰胺、谷氨酰胺、苏氨酸、天冬氨酸、谷氨酸、赖氨酸、精氨酸和组氨酸,各自浓度均为0.7mm),1.7mm二硫苏糖醇(dtt),2%的聚乙二醇,15mm葡萄糖,24mm磷酸三钾,50%体积的酵母细胞提取物(六种细胞提取物分别来自于前述的ynl1、l2t4、l2e1,以及y1140、tpk1s237d、δedc3六株菌),15ng/μl增强型绿色荧光蛋白dna模板(dna模板中绿色荧光蛋白的编码序列上游序列如seqidno:3所示,下游序列如seqidno:4所示)。
体外蛋白质合成反应条件:将上述的反应体系中混匀后放置在室温(20-30℃)的环境中反应20小时。
荧光值测定:将待测样品放置于envision2120多功能酶标仪(perkinelmer),检测获得相对荧光单位值(relativefluorescenceunit,rfu),用rfu值表示无细胞蛋白合成体系的活性,结果如图11b、11c所示。
由图11可以看出,含有nanoluc基因的三株菌ynl1、l2t4、l2e1的发光报告值(图11a)与体外无细胞蛋白合成体系的活性(图11b、图11b)正相关。
从图5、图11来看,本发明的技术方案,可以通过在菌株的基因组中整合发光报告基因(如nluc基因),通过报告基因的细胞内表达,来反映经过某种基因改造后的菌株的无细胞蛋白合成体系的活性,也即无细胞蛋白合成体系中外源蛋白的表达水平,大大方便了基因改造后菌株的无细胞蛋白合成体系的活性检测。
在本发明提及的所有文献都在本申请中引用作为参考,就如同每一篇文献被单独引用作为参考那样。此外应理解,在阅读了本发明的上述内容之后,本领域技术人员可以对本发明作各种改动或修改,这些等价形式同样落于本申请所附权利要求书所限定的范围。
序列表
<110>康码(上海)生物科技有限公司
<120>通过整合发光报告基因反映体外无细胞蛋白表达水平的方法
<130>2019
<141>2019-04-03
<160>6
<170>siposequencelisting1.0
<210>1
<211>1074
<212>dna
<213>人工序列(artificialsequence)
<400>1
gaacaaccaatggctgaaaacttcgagtaaggcgtagaggatcgagatctcgcgaaatta60
atacgactcactatagggaaaaaagaaatctctcaagctgaaattaaaccaaaactctaa120
tataagaaaaaaaaatagaaaggtatttttacaacaattaccaacaacaacaaacaacaa180
acaacattacaattactatttacaattacaaaaaaaaaaaatggtcttcacactcgaaga240
tttcgttggggactggcgacagacagccggctacaacctggaccaagtccttgaacaggg300
aggtgtgtccagtttgtttcagaatctcggggtgtccgtaactccgatccaaaggattgt360
cctgagcggtgaaaatgggctgaagatcgacatccatgtcatcatcccgtatgaaggtct420
gagcggcgaccaaatgggccagatcgaaaaaatttttaaggtggtgtaccctgtggatga480
tcatcactttaaggtgatcctgcactatggcacactggtaatcgacggggttacgccgaa540
catgatcgactatttcggacggccgtatgaaggcatcgccgtgttcgacggcaaaaagat600
cactgtaacagggaccctgtggaacggcaacaaaattatcgacgagcgcctgatcaaccc660
cgacggctccctgctgttccgagtaaccatcaacggagtgaccggctggcggctgtgcga720
acgcattctggcgtaaataaggattaattacttggatgccaataaaaaaaaaaaagcgac780
atagccaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaa840
aaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaacgaactcgagcaccaccaccacc900
accactgagatccggctgctaacaaagcccgaaaggaagctgagttggctgctgccaccg960
ctgagcaataactagcataaccccttggggcctctaaacgggtcttgaggggttttttgc1020
tgaaaggaggaactatatccggatagaatataaaggaaaacttcagcaggaaaa1074
<210>2
<211>2571
<212>dna
<213>人工序列(artificialsequence)
<400>2
cgttgtagaaccatacaatgccactctatctgtacaccaattagtcgaacacgctgatga60
aactttctgtatcgataatgaagccctttacgatatatgtcaaactactctaaaattacc120
acaaccagattatgcgaacttgaaccatttagtttcaagtgtcatgtctggtgtcactac180
atccctacgttacccaggtcaattgaactcagatctaagaaaattggccgtgaatttggt240
gcccttcccaagattacacttcttcatggttggttatgctccattaacctcgattggtgc300
ccaatctttcagatccctatctgtatcagaattgacccaacaaatgttcgattcaagaaa360
tatgatggctgcatctgatccaagaaacggaaggtacttgactgttgctgcattcttccg420
tggtaaagtaagcgtgaaagaagttgaagatgaaatgcataaggttcaaactagaaactc480
gtcgtacttcgtagaatggattccaaacaacgtgcaaacagccgtctgttctgtacctcc540
aaaggacttagacatgtctgctactttcattggtaactctacatccatccaagaattatt600
cagaagggtcggagatcaatttagtgttatgttcaagaagagagctttcttgcattggta660
tactaatgaaggtatggaagaaattgaattttctgaagctgaatctaacatgaatgactt720
agtaagtgaatatcagcagtaccaggacgcaacagttgaagatgacgaagaattggaatt780
cgttgacgaacaaccaatggctgaaaacttcgagtaaggcgtagaggatcgagatctcgc840
gaaattaatacgactcactatagggaaaaaagaaatctctcaagctgaaattaaaccaaa900
actctaatataagaaaaaaaaatagaaaggtatttttacaacaattaccaacaacaacaa960
acaacaaacaacattacaattactatttacaattacaaaaaaaaaaaatggtcttcacac1020
tcgaagatttcgttggggactggcgacagacagccggctacaacctggaccaagtccttg1080
aacagggaggtgtgtccagtttgtttcagaatctcggggtgtccgtaactccgatccaaa1140
ggattgtcctgagcggtgaaaatgggctgaagatcgacatccatgtcatcatcccgtatg1200
aaggtctgagcggcgaccaaatgggccagatcgaaaaaatttttaaggtggtgtaccctg1260
tggatgatcatcactttaaggtgatcctgcactatggcacactggtaatcgacggggtta1320
cgccgaacatgatcgactatttcggacggccgtatgaaggcatcgccgtgttcgacggca1380
aaaagatcactgtaacagggaccctgtggaacggcaacaaaattatcgacgagcgcctga1440
tcaaccccgacggctccctgctgttccgagtaaccatcaacggagtgaccggctggcggc1500
tgtgcgaacgcattctggcgtaaataaggattaattacttggatgccaataaaaaaaaaa1560
aagcgacatagccaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaa1620
aaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaacgaactcgagcaccac1680
caccaccaccactgagatccggctgctaacaaagcccgaaaggaagctgagttggctgct1740
gccaccgctgagcaataactagcataaccccttggggcctctaaacgggtcttgaggggt1800
tttttgctgaaaggaggaactatatccggatagaatataaaggaaaacttcagcaggaaa1860
aaaaaaggaataaggaacaaagatcagatgtgtacagaagataagtttatcattttgaga1920
gcagtaccactgaaagcaaattatttggacacgagataaacaataaacaatgtacttcag1980
aaagatattttctttcttcttatagttttcctttcgttacttctactattctaatactat2040
tccccacaccactagtagaagcggaaaatttcgaaatatgataccacccttactataatt2100
tatatgctttttgtttctctgctcaatgatatgtactatcaatgtgacccaaaacatttc2160
tttagtatttgtttctatttttttttaataatatcatatcttactacataattttagtct2220
tcacattataatacgtgattcttcatgatatatttgactcagatgtcaacacagtagtat2280
gtgaatgtttcaatgacttatcctcttccccaggcctgatcaatcatttcacagatcaac2340
gaaacataccaagaacaaaaagaaataaatttttcacaatagtacccgaagaaaaaaatt2400
aataaggaactaaactgatgtgaaatattgaactgaagctcatctcccctctcatctctc2460
aaaaatagaccaacaaccaaggactaccagttgatcagtctacttggaataggtgatata2520
aaggtagcggcatgctcaggatatctaggaccaatctaggatgtcggacgg2571
<210>3
<211>170
<212>dna
<213>人工序列(artificialsequence)
<400>3
cgcgaaattaatacgactcactatagggaaaaaagaaatctctcaagctgaaattaaacc60
aaaactctaatataagaaaaaaaaatagaaaggtatttttacaacaattaccaacaacaa120
caaacaacaaacaacattacaattactatttacaattacaaaaaaaaaaa170
<210>4
<211>283
<212>dna
<213>人工序列(artificialsequence)
<400>4
ataaggattaattacttggatgccaataaaaaaaaaaaagcgacatagccaaaaaaaaaa60
aaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaaa120
aaaaaaaaaaaaaaaaaaaaacgaactcgagcaccaccaccaccaccactgagatccggc180
tgctaacaaagcccgaaaggaagctgagttggctgctgccaccgctgagcaataactagc240
ataaccccttggggcctctaaacgggtcttgaggggttttttg283
<210>5
<211>1571
<212>dna
<213>人工序列(artificialsequence)
<400>5
gaaccgctatatcttgcatgaggcaagacctgcaaagagaagaaagatgagaccttttga60
caacgaatccaagggatttctttctggccgaacagatcaaattgaattacctgtggtaca120
ggctgtttcaacaagttcaaattctcaagaaccacttctttctagcaaagacccactacg180
agatggaagcaacgtggggaataggttgagtggaaacagcgaaactctgataaccaactc240
tgatgctgatcatcctatggagacggataataataactctaatcttaataatactaattc300
ctctactaatactaatgataacaaagattcgtcatccaattccagcaataatgaaaatgg360
ggacagtagcaacaattcaaatagaaggcatagcaaccattgtcacaagcataaccatct420
aagcagcacttctaccctcaaggcaagagttacttctggaaaatatgcattatatgattt480
tcagattctgagaactttgggtacaggttcgtttggtagagttcatttggtgagatccaa540
tcacaacggcaggttctatgcaatgaaagttttgaaaaaaaacaccgttgtcaaattgaa600
acaagtggagcataccaacgatgaaagaaatatgctaagtatagtatcccatcctttcat660
aattagaatgtggggaactttccaggactcacagcaattgtttatgatcatggattacat720
cgaaggtggtgaactattctctcttttaaggaaagaccagcgttttcctaatccagtcgc780
gaagttttatgcagcagaagtttgtcttgcgttggaatacctacattctaaaggcatcat840
ttacagagatttgaaaccagaaaatatccttttagataagaacggacatatcaagttaac900
tgatttcgggttcgctaaatatgttcctgatgtcacttacactctctgtggaacaccgga960
ctatatcgcacctgaagtggtaagcacaaaaccttacaacaaatctgtggattggtggag1020
ttttggtgttttaatctacgaaatgttagcaggatatacacctttctacgattcaaacac1080
aattaagacctatgagaatattctaaatgctccagtaagattcccaccatttttccattc1140
tgatgctcaagatctaatatcgaaactcatcacaagagacttaagtcaacggctaggtaa1200
tttacaaaatggaagtgaggacgtaaagaatcatccgtggttcagcgaggttgtgtggga1260
aaaactactctgcaaaaacattgaaactccatatgaacctcctattcaggccggacaagg1320
cgatacctctcaatacgataggtatccagaggaagaggttaattatggcattcaaggcga1380
agatccgtaccatagtattttcaccaacttttagcaggcatacttgctcatgaaattcag1440
atatcaacatgcctcttatcttgcatctacacaatatgtaaactttaacacgcctttccc1500
cccgttagtcccttttctttttttttttgttttgatatatcggctcattgccttcagaaa1560
cgctgccgtta1571
<210>6
<211>1767
<212>dna
<213>人工序列(artificialsequence)
<400>6
ggtagctccaataatccaagcatatctaacattttgtctctgatcaactcaactatctca60
aaactatgtctattcgcaacgctgtacatcaagatacaccctgcaacaccaagcatagac120
ttggcattgatcatagagaattcatcttgtcctgacgtatccacgatatctaggttaaat180
tttaactcggttccgttgggtttctttaatacgaattcatgtatgaactgattctcgatc240
gttggataatacgattcgacaaaatgagactctgtaagttgtaccaataaagttgtcttg300
ccgacattccgcgaccccaagacaataatctttctatccctgactgacatatttcctaaa360
aaatccctactgtatcgattcaaaagtcgcttaccagatcggaaaacactcaaaaagcta420
aatggaaagttgtaacaaagaggaaaaaacgagccgaaaccagtaaatccaaacaagaaa480
gctttaacggttatagcactgatataccttgcccaaaacaaatgatccctgatatactta540
taggtctagaaatcaaaatatacatgtctatgtcggagcgttctgtccagaagtgatgga600
tgatgtatcacattttttataatttgaagtttttgtaaaaaaaaaatattgaaaagtatg660
aaggcatgaaaagtgtcgaatcaaacgttaaacatctaatagatatagctctaaattctg720
agagatccgactgttcttcgctgtcaataggcttatacaaagttgctataattttatttg780
tgaacgatatactagtgattgtccaaaaacctttggttgcttgaaagaaagagagagaga840
tagaaatttagaagctataagtttatagaacaagtcacttttgacttatatctgtaaaga900
tagatgtgtaatatatagtaattgtaatagagacgataccaagatctaatagtccaatga960
aatgggccaggaccatccggaggatggtccttgccatgagctggtcaagcatataccgat1020
gccagcgtccatcccaacgtccatcactctctacgagccgcgtttttcagtgcattccaa1080
tggaaatgggaatttctctttactcgcgcaaagggggcgaaaaggcagttttccaaagaa1140
aggaactaaagaaaaaaatctttaattacagaaacacagaaaacaaacagacagaaacaa1200
caacaacaataagacaggtgagagacagaggtctgtatacagtgactgctgtgttattgc1260
gagcggcttctgtactggttatttttgtgtttgcacagggttagtcaggttgaaaaagat1320
ttgagatatagagagagagagagagagagacgcgcgcgcttgtgagtgttctgtgtgtat1380
ccggttttgtgtgcaaaattgagagaacggaattataagaggaaacgaataaataaagca1440
gagacaagaagtgcagttgggttgttgaacgtgacatttaaagttagctgtgtttcgtga1500
gtgtagagtttagtctgttttgttttgtctcttaacgtttcgaggattaataaactgaat1560
taaacgaacatattaaagaaagtttgagacagggaagagaaaagaaaaaaagaaaattac1620
tataactgtaaaaggaagataaatattacaatacaaggctatggggttatgctgtagttg1680
tttgagaggtgaaagttctctcgaggattcgacagggttgcccattgcagaaaatgaaag1740
ggaagcagtgacgtcgttgttggagtt1767
- 该技术已申请专利。仅供学习研究,如用于商业用途,请联系技术所有人。
- 技术研发人员:郭敏;许乃庆;姜灵轩;于雪
- 技术所有人:康码(上海)生物科技有限公司
- 我是此专利的发明人
- 该领域下的技术专家
- 如您需求助技术专家,请点此查看客服电话进行咨询。
- 1、薛老师:1.CRISPR-Cas系统 2.基因编辑 3.基因修复 4.天然产物合成 5.单分子技术开发与应用
- 2、张老师:1.探索新型氧化还原酶结构-功能关系,电催化反应机制 2.酶电催化导向的酶分子改造 3.纳米材料、生物功能多肽对酶-电极体系的影响4. 生物电化学传感和生物电合成体系的设计与应用。
- 3、豆老师:1.环境纳米材料及挥发性有机化合物(VOCs) 2.CO污染物的催化氧化 3.低温等离子体 4.吸脱附等控制技术
- 4、赵老师:1.高分子材料改性及加工技术 2.微孔及过滤材料 3.环境友好高分子材料
- 5、邬老师:1.高分子材料的共混与复合 2.涉及材料功能化及结构与性能的研究; 高分子热稳定剂的研发
- 如您是高校老师,可以点此联系我们加入专家库。
- 还没有人留言评论。精彩留言会获得点赞!