一种基于微生物溯源解析水体污染源的方法与流程
本发明涉及一种基于微生物溯源解析水体污染源的方法,属于水体环境监测和水污染溯源技术领域。
背景技术:
随着人类社会的发展,城市化工业化的推进,人类生产活动产生的污染物,通过点源和面源的方式进入水体,造成水体污染事件的频发,对水体的生态安全和人类健康都构成重要的威胁。因此,对水体污染区域进行溯源,准确掌握污染来源信息,对于水体生态环境的保护和治理有重要作用。
随着生活水平提高和水体治理技术的发展,相对于物理、化学污染,水体中微生物造成的水体污染,如致病细菌,抗生素抗性基因逐渐成为水体污染溯源的研究热点和难点。水体中的微生物会随着水和泥沙在水体中与污染物一起迁移转化,通过找寻宿主特异性或相关性的微生物,通过对微生物的溯源也可以帮助我们追踪其他污染物的来源。传统水质指纹溯源方法对水体相关微生物污染的不适用,也促进了水体微生物溯源技术的发展。
微生物溯源起源于20世纪80年代,最初是为了诊断水源污染物的来源(如牲畜粪便或人类来源),是利用宿主特异性或相关性的微生物指标,建立区别、鉴定污染源的流程。由于不同宿主饮食结构和生存环境的差异,不同污染物的指示微生物的基因型会存在一定差异,基于这种差异可进行污染物的微生物溯源。因此,指示微生物及生物标记的选择对于微生物溯源尤为重要。
微生物溯源的传统方法多数是基于不同菌株基因分型,通过判断不同来源菌株基因相似度进而判断指定微生物的来源。传统的微生物溯源方法如脉冲场凝胶电泳等由于操作复杂、费时费力等因素,无法满足日益增长的对环境微生态的认识。
技术实现要素:
本发明所要解决的技术问题是,提供一种适用广泛、准确、高效的基于微生物溯源解析水体污染源的方法。
为解决上述技术问题,本发明采用的技术方案为:
一种基于微生物溯源解析水体污染源的方法,包括以下步骤:
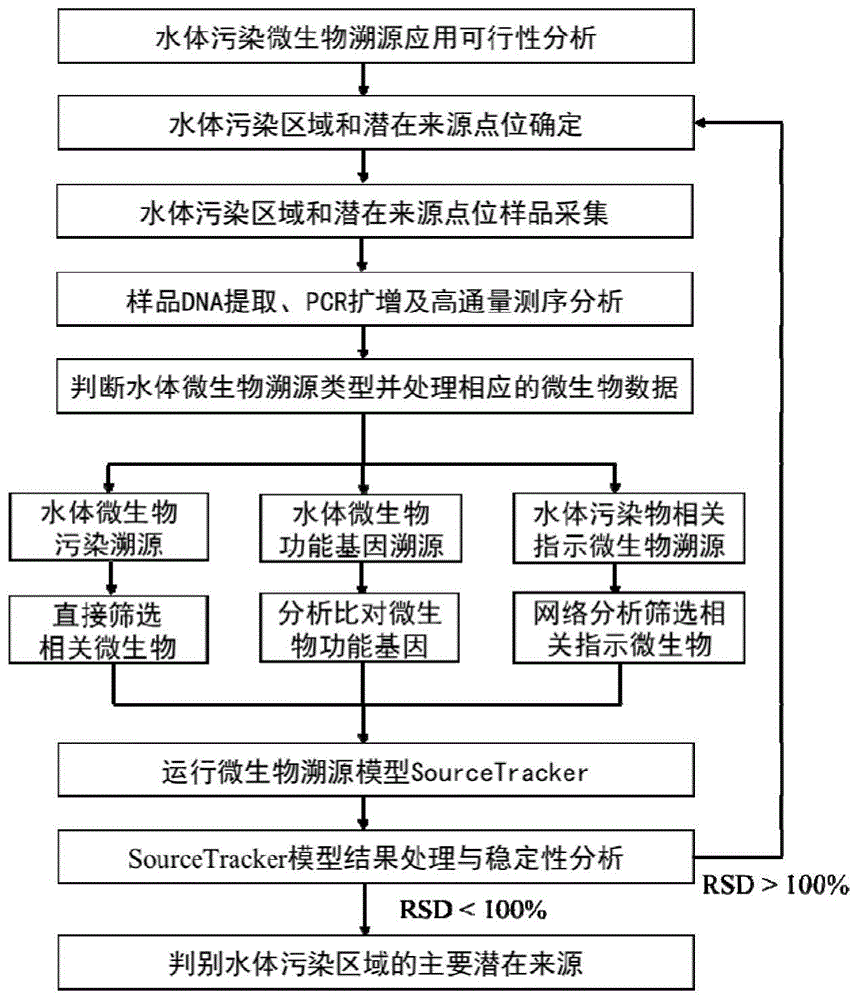
步骤1,根据水体污染物环境调查情况,对水体污染物类型与微生物溯源应用进行可行性分析;
步骤2,确定水体污染区域和潜在来源点位,具体的,可根据水体的点源,面源污染输入情况,水文水资源状况,自然地理区域特征,明确水体污染物的潜在来源点位;
步骤3,采集水体污染区域和潜在来源点位样品;
步骤4,对采集样品进行dna提取、pcr扩增以及测序分析,并基于微生物数据库进行微生物比对分类;
步骤5,基于步骤4所得微生物分类结果判断微生物溯源类型并处理相应的微生物数据;
步骤6,将步骤5处理后的数据,源和汇点位安排输入微生物溯源模型中进行检测;
步骤7,对微生物溯源模型处理结果进行稳定性分析,不符合要求的需重新采样分析;
步骤8,基于检测结果,根据判断方案判别水体污染区域的潜在来源。
进一步的,步骤3中所述样品包括水样,泥样,微生物膜,粪便或活性污泥。
进一步的,步骤4中基于illuminamiseq平台进行高通量测序。
进一步的,步骤5中微生物溯源类型分为以下三类:
a.水体微生物污染溯源,如致病细菌,粪便细菌等,直接通过选择合适引物进行扩增测序筛选出相关微生物数据,输入到微生物溯源模型中;
b.水体微生物功能基因溯源,如抗生素抗性基因,氮循环功能基因等,将高通量测序得到的微生物数据进行功能基因分析或与相关基因库比对得到微生物功能基因数据,输入到微生物溯源模型中;
c.水体污染物相关指示微生物溯源,如氮、磷等相关指示细菌溯源,通过水体污染物与微生物的相关网络分析,筛选出与污染物有极大相关性的微生物数据,输入到微生物溯源模型中。
进一步的,步骤5中采用spearman相关性分析进行数据处理。
进一步的,步骤6中微生物溯源模型采用sourcetracker。
进一步的,步骤7中稳定性分析采用重复多次检测结果的标准差sd和相对标准差rsd进行评估。
进一步的,步骤8中判断方案为结合实际情况和潜在来源的贡献比例,判别水体污染区域的潜在来源。
本发明所达到的有益效果:本发明通过溯源水体微生物的高通量测序得到的微生物数据,不仅可以直接识别水体中微生物造成的水体污染,如致病细菌,抗生素抗性基因,还可通过找寻宿主特异性或相关性的指示微生物,通过对微生物的溯源帮助我们追踪其他污染物的来源,采用sourcetracker作为基于高通量测序技术的微生物溯源模型,解决了传统水质指纹溯源方法对水体相关微生物污染不适用的问题,提高了该方法估计的可靠程度,本发明方法新颖独特,可以高效准确的追踪水体污染来源,为水体生态环境的保护和治理提供了重要理论依据。
附图说明
图1为本发明提供的一种基于微生物溯源解析水体污染来源方法的流程图;
图2为本发明实施例中相关网络分析筛选出与p相关的指示微生物示意图。
具体实施方式
下面结合附图对本发明作进一步描述。以下实施例仅用于更加清楚地说明本发明的技术方案,而不能以此来限制本发明的保护范围。
在本实施例中,研究区域为洞庭湖流域的西洞庭湖和南洞庭湖区域。图1为本发明提供的一种基于微生物溯源解析水体污染来源方法的流程图,该方法主要包括如下步骤:
(1)水体污染类型及微生物溯源应用可行性分析:根据水体污染物环境现状调查,研究区域洞庭湖流域富营养化水平偏高,n、p营养元素均超过国家标准,湖区存在蓝藻爆发的风险。营养元素磷是控制湖泊蓝藻爆发的关键因素,因此急需对洞庭湖流域水体污染p的来源,进行溯源。磷元素是微生物生长的必要元素,磷大部分以颗粒磷的形式与微生物一起吸附在悬浮泥沙中在河湖系统中传播,因此通过相关网络分析,可以筛选出磷的相关指示微生物,从而进行微生物溯源分析。
(2)水体污染物的潜在来源判断:洞庭湖流域大部分磷通过颗粒磷的形式吸附在入湖河流泥沙中输入湖区沉积在底泥中,在水力扰动和合适自然环境下释放造成湖区污染,因此,各条入湖河流是主要的p污染来源,具体的源、汇点位安排如表1所示。
表1洞庭湖流域底泥磷溯源的源和汇的点位安排
(3)水体污染区域和潜在来源点位样品采集:根据实地调研,采集洞庭湖流域湖心区和各条河流入湖口的底泥样品,利用采泥器对研究点位底泥进行取样,每个取样点设置3个重复,沉积物样品采自沉积物-水交界面以下0-25cm,样品采集后,记录样品的感官指标和原位监测常规水质指标。将表层沉积物样品充分混合,分别用灭菌的50ml聚丙烯管收集,一份保存于4℃冰箱中,用于底质样品的理化因子测定;另一份保存于-20℃冰箱中,用于底泥基因组dna提取。实验室进行泥沙样品有机质(om)、总氮(tn)、总磷(tp)、无机磷(ip)、有机磷(op)、非磷灰石无机磷(naip)、磷灰石磷(ap)的检测。
(4)样品dna提取、pcr扩增及高通量测序分析:采集的泥沙样品经过预处理后,用土壤基因组dna提取试剂盒从0.3g底泥样品中提取总dna并取3μl进行1%琼脂糖凝胶电泳检测。16srrna基因v3-v4区使用引物341f和806r进行克隆。pcr过程使用三份20-μl体系的反应物。提取的dna样品进行illuminamiseq测序。在质量检查后,16srrna序列按照97%相似性被分成各个操作分类单元(otu),序列使用silvav128参考数据库进行比对安排微生物分类,得到基于高通量测序的微生物数据。
(5)判断水体微生物溯源类型并处理相应的微生物数据:在本实施例中,水体微生物溯源类型为水体污染物相关指示微生物溯源。水体污染物各种形式p与微生物的相关网络分析(如图2所示),其中运用spearman相关性分析展现不同形式p与微生物的关系,r>0.6,p<0.05的微生物被筛选出来作为与污染物p有极大相关性的微生物数据,输入到微生物溯源模型sourcetracker中。
(6)运行微生物溯源模型sourcetracker:将经过质量过滤和网络分析筛选处理的高通量测序数据,源和汇的点位安排输入到模型sourcetrackerv1.0.1中,使用模型的初始参数运行。模型初始参数设置为:ararefactiondepthof1000,burn-in100,restart10,alpha(0.001)andbeta(0.01)。为了保证模型结果的稳定性,模型重复运行5次,得到各潜在来源微生物对污染区域微生物的贡献比例。
(7)模型结果处理与稳定性分析:各潜在来源微生物对污染区域微生物的贡献比例取5次平均值为a。计算来源比例的标准差sd和相对标准差rsd来评估模型稳定性,本实施例中,所有数据的相对标准差rsd<100%,证明该模型准确性较高。
(8)判别水体污染区域的潜在来源:湖区p潜在来源的贡献比例如表2所示,西洞庭湖流域l1,l2点位p的主要贡献河流为松滋河(sz);南洞庭湖流域l3点位p的主要贡献河流为藕池河(oc),l4点位p的主要贡献河流为汨罗河(ml)。
表2洞庭湖流域底泥磷溯源的源和汇的点位安排
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明技术原理的前提下,还可以做出若干改进和变形,这些改进和变形也应视为本发明的保护范围。
- 还没有人留言评论。精彩留言会获得点赞!