一种同时检测32个Y染色体基因座的复合扩增试剂盒的制作方法
本发明属于法医dna鉴定领域,尤其涉及一种同时检测32个y染色体基因座的复合扩增试剂盒。
背景技术:
短串联重复序列(shorttandemrepeats,str),也称微卫星dna(microsatellitedna),是由2~6个碱基作为核心基序,串联重复形成的一类dna序列,广泛分布于人类基因组中,具有多态性高、信息量大以及易于检测等优点。自上世纪90年代发现以来,现已广泛应用于法医dna鉴定、物种鉴别和遗传病诊断等领域。
y染色体str遗传标记是指存在于y染色体特异区的短串联重复序列。在减数分裂时,y染色体特异区不与x染色体进行重组交换,所以y染色体str遗传标记具有男性特有、父系单倍型遗传等特点。
目前,在法医领域,y染色体str遗传标记主要应用于家系排查和辅助个体识别。而满足上述应用的前提,则需要ystr试剂盒具有足够高的整体多态性。而增加ystr试剂盒位点数,是提高ystr试剂盒的整体多态性,增加累计非父排除率和个体识别能力的有效途径。但是在现有技术条件下,ystr试剂盒位点数的增加,具有一定的局限性。因此,当前市场上,主流的ystr建库试剂盒均是以公安部发布的20个核心ystr基因座为基础,然后再从公安部发布的优选和备选基因座中,选择10到20个基因座进行优化组合。此外,随着市场上差异化需求的不断增长,用户对于试剂盒包含的基因座数量、兼容性等提出了更高的要求。
技术实现要素:
鉴于上述现有技术存在的缺陷,本发明的目的是提出一种同时检测32个y染色体基因座的复合扩增试剂盒。本发明中的基因座是具有高多态性的基因座,其在实际应用过程中,不仅可用于家系排查,也可用于对主流建库试剂盒的功能进行补充,以满足客户的差异化需求。
本发明的目的将通过以下技术方案得以实现:
一种同时检测32个染色体基因座的复合扩增试剂盒,其中,包括56条扩增引物,分别依次为:seqidno:1~2、seqidno:3~4、seqidno:5~6、seqidno:7~8、seqidno:9~10、seqidno:11~12、seqidno:13~14、seqidno:15~16、seqidno:17~18、seqidno:19~20、seqidno:21~22、seqidno:23~24、seqidno:25~26、seqidno:27~28、seqidno:29~30、seqidno:31~32、seqidno:33~34、seqidno:35~36、seqidno:37~38、seqidno:39~40、seqidno:41~42、seqidno:43~44、seqidno:45~46、seqidno:47~48、seqidno:49~51、seqidno:52~53、seqidno:54~56。
在一些实施例中,所述56条扩增引物对应的y染色体基因座依次分别为:dys531、dys630、dys622、dys510、dys533、dyf404s1a/b、dys459a/b、dys446、dys443、dys388、dys587、dys522、dys626、yindel、dys460、y_gata_a10、dys520、dys557、dys527a/b、dys481、dys508、dys444、dys385a/b、dys552、dys526a/b、dys617和dys713,其中,所述dyf404s1a/b、所述dys527a/b、所述dys385a/b和所述dys526a/b为双等位基因基因座。
在一些实施例中,所述y染色体基因座所对应的引物对在扩增体系中的终浓度为0.027μm~0.82μm。
在一些实施例中,所述y染色体基因座所对应的引物对在扩增体系中的终浓度依次分别为dys531:0.52μm、dys630:0.08μm、dys622:0.82μm、dys522:0.02μm、dys460:0.78μm、y_gata_a10:0.06μm、dys520:0.8μm、dys481:0.41μm、dys444:0.76μm、dys552:0.24μm、dys526a/b:0.3μm、和dys510:0.124μm、dys533:0.021μm、dyf404s1a/b:0.144μm、dys459a/b:0.7μm、dys446:0.09μm、dys443:0.027μm、dys388:0.53μm、dys557:0.071μm、dys587:0.184μm、dys626:0.41μm、dys527a/b:0.051μm、dys508:0.14μm、dys385a/b:0.17μm、dys617:0.15μm、dys713:0.45μm和yindel:0.5μm。
在一些实施例中,所述每个基因座中的至少一条引物的5’端标记有荧光染料。
在一些实施例中,所述荧光染料包括6-fam、hex、sum、lyn、pur、和/或siz。
在一些实施例中,所述每个y染色体基因座对应的扩增引物的长度为90-480bp。
一种根据上述所述的试剂盒的应用,其中,所述试剂盒用于亲权鉴定、个体识别。
与现有技术相比,本发明提供的一种同时检测32个y染色体基因座的复合扩增试剂盒,达到的技术效果是:本发明通过提供市场上未被广泛使用的新y染色体基因座位点,如dyf404s1a/b、dys626、dys508、dys713、dys617、dys526a/b等,与市场上主流建库试剂盒,如
附图说明
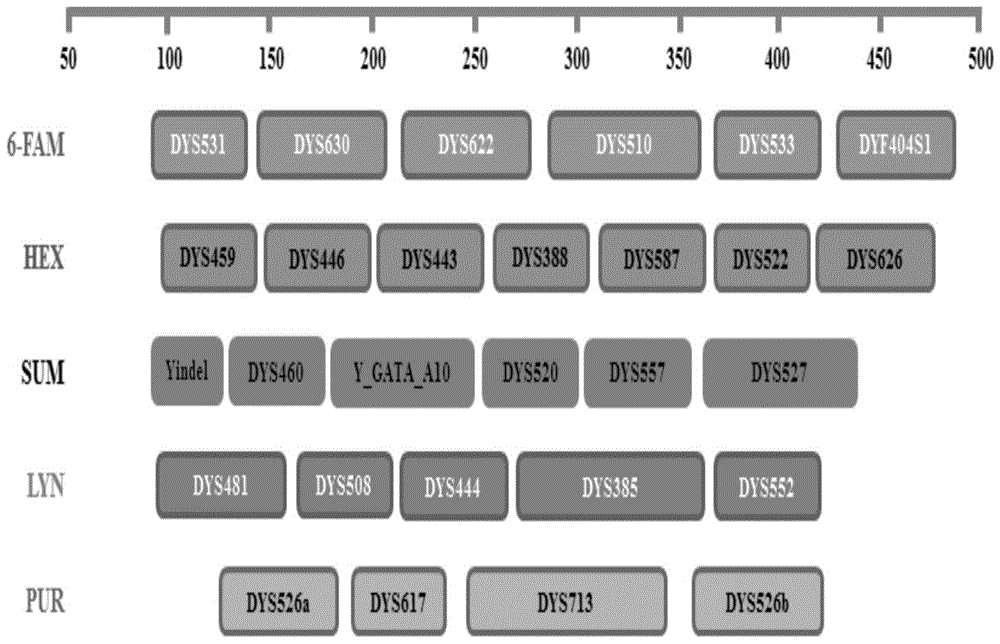
图1是根据本申请一些实施例所示的基因座的排布示意图;
图2是根据本申请一些实施例所示的等位基因ladder分型图;
图3是根据本申请一些实施例所示的标准dna9948的分型图;
图4是根据本申请一些实施例所示的试剂盒对棉签提取样本的分型图;
图5是根据本申请一些实施例所示的试剂盒对fta血卡样本的分型图;
图6是根据本申请一些实施例所示的试剂盒对唾液卡样本的分型图。
以下便结合实施例,对本发明的具体实施方式作进一步的详述,以使技术方案更易于理解、掌握。
具体实施方式
下面通过具体实施例对本发明进行说明,但本发明并不局限于此。下述实施例中所述实验方法,如无特殊说明,均为常规方法;所述试剂和材料,如无特殊说明,均可从商业途径获得,下面实施例并非用以限制本发明的专利范围,凡未脱离本发明所为的等效实施或变更,均应包含于本专利保护范围中。
实施例132个y染色体基因座的筛选
在一些实施例中,y染色体基因座可以基于现有文献等记载进行获取,并对其进行筛选。在一些实施例中,可以基于获取的y染色体基因座的多态性进行调查,筛选出高多态性基因座。其中,高多态性基因座可以包括低突变的高多态性基因座,或高突变的高多态性基因座等。在一些实施例中,低突变的高多态性基因座可以指突变率低于1%,多态性高于0.6的基因座为低突变的高多态性基因座(如表1);高突变的高多态性的基因座可以指突变率高于1%,多态性高于0.6的基因座为高突变的高多态性基因座(如表1)。
为了便于理解的目的,本实施例可以通过对dys271、dyf380s1、dys566、dys611、dyf396s1、dys441、dys67、dys11、dys66、dys551、dys282、dys564、dys507、dys148、dys507、dys712、dys559、dys512、dys385a/b、dys481、dys533、dys643、dys460、dys549、dys387s1a/b、dys449、dys518、dys627、dys570、dys527a/b、dys447、dys444、dys557、dys596、dys446、dys510、dys622、dys443、dys587、dys522、y_gata_a10、dys520、dys552、dys593、dys531、dys459a/b、dys508、dys388、dys617、dys645、dys713、dys630、dyf404s1a/b、dys626、dys526a/b等63个基因座,在中国人群中的多态性调查,优选出27个低突变、高多态性基因座:dys531、dys481、dys510、dys533、dys459a/b、dys446、dys443、dys388、dys557、dys587、dys522、dys520、dys527a/b、dys460、y_gata_a10、dys508、dys444、dys385a/b、dys552、dys526a、dys617、dys630、dys622、dys713和4个高突变、高多态性基因座:dyf404s1a/b、dys526b、dys626以及一个yindel。其中上述31个基因座(除yindel)的详细信息见表1。
表1:31个ystr基因座信息
为了进一步说明本发明优选的31个ystr基因座和一个yindel,与目前市场上主流的y建库试剂盒(如表2所示的4个试剂盒)形成较好的互补,本发明将所筛选的31个ystr基因座和一个yindel与下述4个市场上主流的y建库试剂盒中的基因座进行比较。详见表2,其中“+”指的是本发明可以识别的基因座,空格为本发明不能识别该str基因座。其中,4个市场上主流的y建库试剂盒可分别包括
表2:本发明所提供的试剂盒与国内外试剂盒比较
实施例2:基因座排布
根据实施例1中优选的32个y染色体基因座序列及其多态性等特征,结合荧光染料的特性,可以设计独特的基因座排布方式。
在一些实施例中,荧光染料用于将每个基因座的扩增引物对中的至少一条引物5′端进行标记。
在一些实施例中,本发明可以将32个y染色体基因座分为五组:第一组:dys510、dys533、dys531、dys630、dys622、dyf404s1a/b;第二组:dys443、dys388、dys459a/b、dys446、dys557、dys587、dys522、dys626;第三组:y_gata_a10、dys460、dys527a/b、dys520、yindel;第四组:dys385a/b、dys481、dys508、dys444、dys552;第五组:dys617、dys526a/b、dys713。
在一些实施例中,本发明可以将32个y染色体基因座分为五组:第一组:dys510、dys533、dys531、dys630、dys622、dyf404s1a/b,采用蓝色荧光染料;第二组:dys443、dys388、dys459a/b、dys446、dys557、dys587、dys522、dys626,采用绿色荧光染料;第三组:y_gata_a10、dys460、dys527a/b、dys520、yindel,采用黄色荧光染料;第四组:dys385a/b、dys481、dys508、dys444、dys552,采用红色荧光染料;第五组:dys617、dys526a/b、dys713,采用紫色荧光染料。
在一些实施例中,本发明可以将32个y染色体基因座分为五组:第一组:dys510、dys531、dys622、dys630、dys533、dyf404s1a/b,蓝色荧光染料为:6-fam;第二组:dys446、dys557、dys443、dys388、dys587、dys459a/b、dys522、dys626,绿色荧光染料为:hex;第三组:y_gata_a10、dys520、dys460、dys527a/b、yindel,黄色荧光染料为:sum;第四组:dys444、dys385a/b、dys481、dys508、dys552,红色荧光染料为:lyn;第五组:dys617、dys526a/b、dys713,紫色荧光染料为pur,如图1所示,即图1为本发明的基因座排布图。
实施例3:基因座引物设计和复合扩增体系建立
引物设计时需要注意一下几点:a、避免引物间二聚体和发卡结构,以免影响引物的扩增效率;b、确保引物序列中不包含snp位点,特别是引物3’端序列尤为重要,如果存在snp点可能会出现扩增丢失;c、引物序列需要利用ncbi进行blast比对,保证序列的特异性;d、确保所有引物具有相近的熔炼温度,确保同一退火温度下,具有相近的扩增效率。
随着复合扩增系统中引物数量的增加,不同基因座引物之间的相互干扰也越来越严重,反应体系的动力学变得越来越复杂,因此需要设计大量的引物序列进行复杂的测试,最终保证试剂盒的扩增特异性及效率。
便于说明的目的,本申请仅仅提供一个实施例来保证试剂盒的扩增特异性及效率。
具体的,根据ncbigenbank中公布的上述基因座的序列信息,采用primer5.0软件设计引物,然后使用oligo7软件对设计的引物进行评价,优选扩增效率高的引物,最后,再通过ncbi数据库中的primerblast在线引物比对工具,进一步筛选高特异性引物。通过上述步骤设计的引物,再经单扩测试,作进一步优选。
将上述筛选的32个基因座引物进行混合测试,排查可能引起非特异扩增的引物,然后重新设计,直至每个基因座引物的扩增子大小为90~480bp之间,且无二聚体峰和非特异扩增现象。最后,根据每个基因座的引物扩增效率,调整复合扩增体系中的引物浓度至表3所示。
表3:32个y染色体基因座引物信息
其中,表3中的每个基因座的扩增引物序列中,至少一条采用荧光染料标记其5′端。关于荧光染料标记引物的详细描述可以在本申请的其他地方找到,如实施例2中所述。
在确定32个基因座的复合扩增引物体系的基础上,通过大量重复实验进一步确定扩增中的其他参数,如:酶量、缓冲液离子强度、模板dna量、复合引物量、循环参数、退火温度以及延伸温度和时间等。以期达到试剂盒的分型精准度高、均衡性好、特异性强和灵敏度优等目标要求(如表4和表5)。
在一些实施例中,本申请的试剂盒可以包括反应混合液、热启动酶、32个基因座的复合引物、32个基因座的等位基因分型标准物、dna标准品、荧光分子内标、和/或sdh2o。
在一些实施例中,人基因组dna的提取方法可以包括利用chelex法、磁珠提取法或有机抽提法。在一些实施例中,人基因组dna可以采用免提取的滤纸、fta卡、棉签、唾液卡、或纱布中一种或其任意组合的载体收集的含有人基因组dna人类血液或口腔细胞。在一些实施例中,人基因组dna的来源可以包括含有人基因组dna的血液、血痕、精液、唾液、体液、毛发、肌肉或组织器官等中的一种或其任意组合。例如,人基因组dna可以是1.0或1.2mm孔径的血斑;又例如,人基因组dna可以是1.0或1.2mm孔径的唾液斑;再例如,人基因组dna可以是采用磁珠法或者chelex-100法或者有机纯化法提取的0.1ng-2ng人基因组。
表4:pcr扩增体系及其组分
表5:pcr循环参数
实施例4试剂盒的应用
基于基因座引物设计和复合扩增体系建立,本申请的试剂盒可以应用于法医鉴定、亲权鉴定或dna家谱构建。
本申请试剂盒具体使用步骤可以包括如下:
1、提取人基因组dna,具体的操作方式可以参考实施例3中表4及其所述;
2、目标等位基因扩增:参照表4所示的组分,配制扩增体系,置于pcr仪上,然后参照表5的扩增程序,扩增目标等位基因,得到扩增产物;
3、扩增产物的电泳检测:取荧光分子量内标agcumarkersiz-500与去离子甲酰胺,按体积比1/25的比例混合组成上样混合物;将12.5μl的上样混合物分别与1.0μl的扩增产物、32个基因座的等位基因分型标准物混合,于离心机中4000rmp离心3min,去除气泡;将其置于pcr仪上,95℃变性3min,然后转至低温冰箱中,静置3min,最后使用遗传分析仪进行电泳检测;
4、分型分析:采用片段分析软件genemapperid-x分析上述遗传分析仪检测收集的数据。
实施例5:等位基因分型标准物组配
等位基因分型标准物为分型的标准,用于对个人进行分型鉴定。根据最终确定的每个基因座的引物特点,将上述收集的每个基因座的不同分型集合到一起。本发明所述的试剂盒的等位基因分型标准物的分型图,包含目前已发现的所有标准等位基因,如图2所示。
实施例6:种属特异性
试剂盒检验马、狗、猪、牛、羊、猫、鸡、鸭、鼠、兔、鱼和大肠杆菌dna,在分型范围内,不出现特异的dna分型,说明本发明具有较好的种属特异性。
实施例7:检材适用性
本发明检材适用性广,对人源的棉签采集提取样本、fta血卡、唾液卡样本均能正确分型。如图4至图6所示。具体如下:
图3所示为标准dna9948的分型图,该图表明本发明可对阳性dna标准品9948进行正确分型;
图4所示为试剂盒对棉签提取样本的分型图;其表明本发明可对棉签采集类样本,在提取的基础上进行正确分型;
图5所示为试剂盒对fta血卡样本的分型图;其表明本发明可对血卡采集类样本,在不经提取的情况下进行直接检测,且能正确分型;
图6是所示的试剂盒对唾液卡样本的分型图;其表明本发明可对唾液卡采集类样本,在不经提取的情况下进行直接检测,且能正确分型。
上述说明示出并描述了本发明的若干优选实施例,但如前所述,应当理解本发明并非局限于本文所披露的形式,不应看作是对其他实施例的排除,而可用于各种其他组合、修改和环境,并能够在本文所述发明构想范围内,通过上述教导或相关领域的技术或知识进行改动。而本领域人员所进行的改动和变化不脱离本发明的精神和范围,则都应在本发明所附权利要求的保护范围内。
序列表
<110>无锡市公安局刑事科学技术研究所
无锡中德美联生物技术有限公司
<120>一种同时检测32个y染色体基因座的复合扩增试剂盒
<141>2019-04-04
<160>56
<170>siposequencelisting1.0
<210>1
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>1
ctgagacgcactggcattcaaatc24
<210>2
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>2
ctgagacgcactggcattcaaatc24
<210>3
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>3
agagcaagactccacctcaaaag23
<210>4
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>4
catggatagctgtgagttccataaact27
<210>5
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>5
gctgtgtatgtcccagaaatgtaggt26
<210>6
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>6
cctcggtgataagagtgaaac21
<210>7
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>7
tcccttaccacagataggtaagac24
<210>8
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>8
gcttatacagcagatgataataggtat27
<210>9
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>9
caccagttagaggagatggaaagc24
<210>10
<211>31
<212>dna
<213>人工序列(artificialsequence)
<400>10
gtctacctattcatctaacatctttgtcatc31
<210>11
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>11
ttgagtttcccagaaggcctaatgcaca28
<210>12
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>12
ccaggtattctggttgaggct21
<210>13
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>13
agcaagacttaatcacaccccact24
<210>14
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>14
cctgatatatttgcagttaatttgatcc28
<210>15
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>15
caattattttcagtcttgtcctgtcc26
<210>16
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>16
attattgctgagcttgtaccactg24
<210>17
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>17
agattgaattattcagtgcttttcat26
<210>18
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>18
ttatctttttattagctttttgcagcc27
<210>19
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>19
ggcggagcttttagtgagcca21
<210>20
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>20
tctgggcagcgagtttaaggg21
<210>21
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>21
tgatgagtaagacacagatttgttgta27
<210>22
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>22
ggtttcaggaaaatattcaaaaatgatc28
<210>23
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>23
aatcctttgaaatcattcataatgc25
<210>24
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>24
ggaagacagagtcataaacagag23
<210>25
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>25
caatgaaaccatacaagagcac22
<210>26
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>26
cagcctgggtgacagagtgc20
<210>27
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>27
ccaaatcaactcaactccagtga23
<210>28
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>28
ctgatacctttgtttctgttcatt24
<210>29
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>29
gttatatcatctatcctctgcctatc26
<210>30
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>30
aataagaataccagaggaatctgaca26
<210>31
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>31
aatggagatagtgggtggat20
<210>32
<211>29
<212>dna
<213>人工序列(artificialsequence)
<400>32
gcaacctgccatctctatttatcttgcat29
<210>33
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>33
gtcaagcaatttgctttcgtcaac24
<210>34
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>34
gtcgagttagagaacagcct20
<210>35
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>35
gcccagcatgtgttttgacta21
<210>36
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>36
actgtcacaatgggtcctgta21
<210>37
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>37
attctaggaagattagccacaa22
<210>38
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>38
cttattcgtgagccaagatcgc22
<210>39
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>39
tttaccagaaggttgcaagactc23
<210>40
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>40
gaatgtggctaacgctgttc20
<210>41
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>41
ttatacaatggcaatcccaaatt23
<210>42
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>42
acaaataaggtgggatggat20
<210>43
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>43
gagcccatgccattcaaac19
<210>44
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>44
gctcattttctctcttctccactttaa27
<210>45
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>45
ctattccaattacatagtcctcc23
<210>46
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>46
gttgaaatgatggcactgca20
<210>47
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>47
cttcaatgtccatagtgccg20
<210>48
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>48
atcgatgttgagcaggatcc20
<210>49
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>49
gaaagaaagtaaacgtttgggtta24
<210>50
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>50
tctttaccttctggtgaactgatcc25
<210>51
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>51
tctttaccttctgggaaactgatcc25
<210>52
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>52
tgttctttctgcttagtactgtg23
<210>53
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>53
tgcgggattggggagtgata20
<210>54
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>54
actgtactccagcctgggtgac22
<210>55
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>55
accagaaatagatttattcacgcttg26
<210>56
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>56
accagaactagatttattcacggttg26
- 还没有人留言评论。精彩留言会获得点赞!