检测单基因突变的CRISPR高通量生物芯片的制作方法
本发明属于生物技术领域,具体地说,本发明涉及一种检测单基因突变的crispr高通量生物芯片及构建方法。
背景技术:
单基因病(monogenicdisease)主要指由一对等位基因突变导致的疾病,分为由显性基因和隐性基因突变所致的两种情况。在临床诊断中,有许多的疾病由单基因的突变而引起。由于单基因疾病有其独一无二的特征基因突变位点,因此可以开发出针对特定单基因疾病的核酸分子检测。核酸诊断(nadsnucleicaciddiagnostics)是用分子生物学的理论和技术,通过直接探查不同物种特定核酸的存在状态,从核酸自身结构、复制、转录或翻译过程分析核酸的功能,从而有针对性的做出诊断的方法。与此相类似的还有单核苷酸多态性(snps,singlenucleotidepolymorphisms)检测,snps广泛存在于人类基因组中,其发生频率约为1%或更高,snps检测技术的发展对于基因样品检测尤为重要。
但上述的单基因突变疾病检测不仅存在非人为操作的技术问题,而且每次只能检测少数几种甚至一种的单基因疾病,加上检测技术成本过高,这些技术在单基因突变疾病检测上并没有被广泛应用。
由于dna分子稳定性较高,当前的nads方法大多是对dna分子进行检测,也有部分方法针对rna分子进行检测。进行核酸分子检测的步骤大致为两步:第一步是目标dna的扩增,第二步是目标dna的检测。snp中核苷酸的变化有两种形式:一种是转换,另一种是颠换。随着生物技术的不断发展,出现了一类高通量、自动化程度较高的snp检测方法,如直接测序、dna芯片等。目前,美国昂菲公司利用分子杂交和原位荧光检测,已经制成snps检测的高通量生物芯片,但是芯片设计成本高,而且有些snp不能被检测。
生物芯片是根据生物分子间特异相互作用的原理,将生化分析过程集成于芯片表面,从而实现对dna、rna、多肽、蛋白质以及其他生物成分的高通量快速检测。可作为各种检测方法的操作平台,即能够同时进行大量不同反应条件的实验分析并输出数据,实现对不同靶标的同时、高速的检测。
疏水蛋白是一种高等丝状真菌在特定时期产生的一类具有特殊理化性质的小分子量(12kd左右)蛋白质,其蛋白结构比较特殊,既含有亲水结构,也含有疏水结构,整体结构上表现为双亲性。疏水蛋白的双亲性使其像表面活性剂一样,主要功能为能够自主装在任何亲水/疏水表面形成一层两亲性的膜以改变表面性质。疏水蛋白是已知的表面活性最高的蛋白质之一,具有耐高温、耐酸碱等特性,可试用于调整实际操作中蛋白分子排列的结构,以实现更高的反应效率,在实际生产中具有很高的理论价值和应用价值。
crispr-cas12a蛋白是一种rna指导的酶,作为细菌适应性免疫系统的组分,其拥有t富集的前间区序列邻近基序(pam),能催化他们自己的引导rna(crrna)成熟并产生一个pam末端不规则的双链dna断点。cas12a蛋白在特异性切割后,还能不加区分的非特异性切割dna单链,应用这个独特性质,我们便可以通过荧光探针检测系统是否完成切割功能。
技术实现要素:
本发明的目的在于提供一种检测单基因突变的crispr高通量生物芯片,克服现有技术生物芯片的不足如低通量、高成本、技术缺陷等问题。
本发明的技术方案为:
检测单基因突变的crispr高通量生物芯片,构建方法包括如下步骤:
(1)化学合成含有cas12a蛋白序列的大肠杆菌表达载体、含有pam(pam指前间区序列邻近基序,是cas12a行使切割功能所必须的识别序列,fncas12a的pam序列为ttn)的特异性底物、含有pam序列的pcr上游引物、下游引物、荧光探针,以及对应于fncas12a和相应单基因突变疾病的crrna(crrna,即向导rna,是指引导fncas12a蛋白特异性结合靶标dna的rna,crrna长约43bp),确定cas12a蛋白在大肠杆菌重组质粒中的表达;
(2)cas12a和crrna的复合物对特异性底物(包括合成的质粒和通过设计含有pam的上游引物pcr扩增得到的长140bp左右的片段)——双链dna进行特异性切割的体系的构建,包括对双链dna切割实验比例的优化、对pcr扩增片段的切割;
(3)cas12a和crrna的复合物对单链dna(包括合成的单链dna和荧光探针)进行非特异性切割的体系的构建,包括对普通单链dna的切割、对荧光探针单链dna的切割;
(4)将芯片基底进行疏水蛋白修饰;
(5)构建高通量芯片基底,包括疏水蛋白对fncas12a-crrna复合物的固定、复合物对特异性底物的特异性切割和对荧光探针的非特异性切割。
所述步骤(4)疏水蛋白指一些双亲性蛋白,可在两相界面处通过自我装配形成纳米级厚度的两性蛋白膜。所述步骤(4)疏水蛋白指hgfⅰ、hfbⅰ。
所述cas12a蛋白优选为fncas12a蛋白。
检测单基因突变的crispr高通量生物芯片的构建方法,包括如下步骤:
(1)化学合成含有cas12a蛋白序列的大肠杆菌表达载体、特异性底物、含有pam序列的pcr上游引物、下游引物、荧光探针,以及对应于cas12a和相应单基因突变疾病的crrna,确定fncas12a蛋白在大肠杆菌重组质粒中的表达;
(2)cas12a和crrna的复合物对特异性底物——双链dna进行特异性切割的体系的构建,包括对双链dna切割实验比例的优化、对pcr扩增片段的切割;
(3)cas12a和crrna的复合物对单链dna进行非特异性切割的体系的构建,包括对普通单链dna的切割、对荧光探针单链dna的切割;
(4)将芯片基底进行疏水蛋白修饰;
(5)构建高通量芯片基底,包括疏水蛋白对cas12a-crrna复合物的固定、复合物对特异性底物的特异性切割和对荧光探针的非特异性切割。
所述步骤(4)疏水蛋白指一些双亲性蛋白,可在两相界面处通过自我装配形成纳米级厚度的两性蛋白膜。所述步骤(4)疏水蛋白指hgfⅰ、hfbⅰ。
所述cas12a蛋白优选为fncas12a蛋白。
检测单基因突变的crispr高通量生物芯片的应用。
本发明有益效果:本发明利用疏水蛋白对芯片基底进行修饰,经过疏水蛋白修饰的芯片基底与普通基底相比,不仅使亲水蛋白cas12a在芯片表面均匀铺展,还避免了蛋白和crrna的浪费,保证目的基因在低浓度下仍有较好的检测结果,并实现了目的基因的1个拷贝的检测,与普通单基因疾病检测相比具有效率高、成本低、周期短、特异性强的特点,且检测材料稳定、操作难度低,结果直观可靠,十分有望投入实际应用。
附图说明
图1显示了2种不同来源的cas12a——fncas12a、lbcas12a的westernblot图;
图2显示了特异性底物的琼脂糖凝胶电泳图;其中:
图2a显示了两种质粒pdsred-monomer-n1、pacgfp1-n1的琼脂糖凝胶电泳图;
图2b显示了转录激活因子gata4表达载体的琼脂糖凝胶电泳图;
图2c显示了质粒pacgfp1-n1的pcr片段的琼脂糖凝胶电泳图;
图3显示了fncas12a切割特异性双链dna的特性;其中:
图3a为fncas12a特异性切割质粒的蛋白质梯度电泳结果图;
图3b为fncas12a特异性切割质粒的质粒梯度电泳结果图;
图3c为fncas12a特异性切割质粒的优化样品比例梯度电泳结果图;
图3d为fncas12a特异性切割质粒的最优样品比例电泳结果图;
图3e为fncas12a特异性切割两种质粒的最优样品比例电泳结果图;
图3f为fncas12a特异性切割质粒的pcr片段的电泳结果图;
图4显示了fncas12a切割非特异性单链dna的特性;其中:
图4a显示了fncas12a切割非特异性单链dna——egfp的特性;
图4b显示了fncas12a切割非特异性单链dna的单链dna梯度电泳结果图;
图5显示了fncas12a非特异性切割荧光探针的荧光数值;其中:
图5a显示了fncas12a非特异性切割荧光探针的探针梯度荧光数值;
图5b显示了fncas12a切割荧光探针的探针最低检测线荧光数值;
图5c显示了fncas12a非特异性切割荧光探针的材料影响荧光数值;
图6显示了疏水蛋白修饰芯片的crispr切割反应的流程示意图;
图7显示了疏水蛋白修饰和未经疏水蛋白修饰的芯片上crispr切割反应的荧光检测结果图。
具体实施方式
下面将结合本发明具体实施例中的附图,对本发明中的技术方案进行进一步的说明。
术语:
术语“向导rna”是指引导cas蛋白特异性结合靶标dna序列的rna。
术语“crrna”是指crisprrna,是短的引导cas12a到结合到靶标dna序列的rna。
术语“crispr”是指成簇的、规律间隔的短回文重复序列(clusteredregularlyinterspacedshortpalindromicrepeats),该序列是许多原核生物的免疫系统。
术语“cas蛋白”是指crispr-associated蛋白,它是crispr系统中的相关蛋白。
术语“cas12a”(旧称“cpf1”)是指crrna依赖的内切酶,它是crispr系统分类中v型(typev)的酶。
术语“pam“是指前间区序列邻近基序(protospacer-adjacentmotif),是cas12a切割所必须,fncas12a的pam为ttn序列,lbcas12a的pam为tttn序列。
材料:
rna酶抑制剂购自solarbio公司;引物(寡核苷酸)、单链dna片段dnmt、向导rna包括pacgfp1-n1-1、pdsred-monomer-n1-1、dnmt-1、fncas12a蛋白表达载体、lbcas12a蛋白表达载体均购自genewiz公司;荧光探针(fam-ttatt-bhq1)购自北京赛百盛基因技术有限公司;
pacgfp1-n1、pdsred-monomer-n1,均为市售。
大肠杆菌(e.coli)dh5α、bl21,均为市售。
实施例1:fncas12a特异性切割pacgfp1-n1质粒的优化样品比的梯度实验
一、fncas12a蛋白表达载体的大肠杆菌(e.coli)bl21的转化与fncas12a蛋白的纯化,具体步骤如下:
fncas12a蛋白表达载体的大肠杆菌(e.coli)bl21的转化,按如下方法进行:
1、取一管感受态细胞e.colibl21于冰上缓慢溶解,加入要转化的fncas12a蛋白表达载体质粒(10μl),轻混后冰浴30min;
2、42℃热激90s后,迅速冰浴5min;
3、加入900μl,37℃温浴的lb培养基,37℃摇床培养1h;
4、3000rpm,3min离心,在无菌操作台内,用移液枪丢弃500ul的菌液后轻混,取200微升涂布于含卡那霉素(100μg/ml)的lb选择平板,37℃倒置培养12-16h;
5、挑取单克隆,进行后续质粒提取以及验证实验。
在上述步骤中,所述的质粒载体阳性克隆验证,按如下方法进行:
1、从平板上挑取fncas12a蛋白表达载体的转化子,接入5mllb液体培养基中(含100μg/ml卡那霉素),37℃摇床培养12-16h;
2、取5ml菌液,用质粒小量提取试剂盒提取质粒;
3、取1μl质粒提取液进行nanodrop检测,检测提取的质粒浓度和纯度;
4、将提取好的质粒放入-20℃保存。
在上述步骤中,fncas12a蛋白纯化,按如下方法进行:
1、挑取平板上明显的周围菌落适量的单克隆,接入5mllb液体培养基中(含100μg/ml卡那霉素),37℃摇床培养6h;
2、将od值约为0.5的菌液接入接入1llb液体培养瓶中(含100μg/ml卡那霉素),37℃摇床培养6h;
3、将摇床温度调至16℃,降温30min,每瓶培养基中加入500μliptg诱导,16℃摇床培养16h;
4、4000rpm,4℃下离心收菌15min,用洗杂缓冲液[50mmtris-hcl(ph8.0),1.5mnacl,5%glycerol]重悬菌体,破菌器破菌;
5、18000rpm,4℃下离心40min,收集上清,进行亲和层析,跑胶验证;用50mmtris-hcl(ph8.0),1mmdtt,5%glycerol换液,稀释至盐浓度在80mm以下;
6、进行蛋白质层析纯化,sds-page电泳跑胶,图1显示了fncas12a、lbcas12a的westernblot图。
二、pacgfp1-n1、pdsred-monomer-n1质粒的大肠杆菌(e.coli)dh5α的转化,具体步骤如下:
1、取2管感受态细胞e.colidh5α于冰上缓慢溶解,分别加入要转化的pacgfp1-n1、pdsred-monomer-n1质粒(各10μl),轻混后冰浴30min;
2、42℃热激90s后,迅速冰浴5min;
3、加入900μl,37℃温浴的lb培养基,37℃摇床培养1h;
4、3000rpm,3min离心,在无菌操作台内,用移液枪丢弃500ul的菌液后轻混,各取200微升涂布于含卡那霉素(100μg/ml)的lb选择平板,37℃倒置培养12-16h;
5、挑取单克隆,进行后续质粒提取以及验证实验。
在上述步骤中,所述的质粒载体阳性克隆验证,按如下方法进行:
1、分别从平板上挑取pacgfp1-n1、pdsred-monomer-n1的转化子,接入5mllb液体培养基中(含100μg/ml卡那霉素),37℃摇床培养12-16h;
2、分别取5ml菌液,用质粒小量提取试剂盒提取质粒;
3、分别取1μl质粒提取液进行nanodrop检测,检测提取的质粒浓度和纯度;
4、将提取好的质粒放入-20℃保存。图2a显示了两种质粒pdsred-monomer-n1、pacgfp1-n1的琼脂糖凝胶电泳图。
三、fncas12a特异性切割pacgfp1-n1质粒的蛋白质梯度优化实验,具体步骤如下:
1、四种20ul反应体系中,分别加入步骤一纯化的fncas12a(62.5nm、125nm、187.5nm、250nm),向导rna——pacgfp1-n1-1(0.5um),pacgfp1-n1质粒1ul,rna酶抑制剂0.5ul,缓冲液nebbuffer3补齐。
2、在37℃反应中反应15min,然后98℃2min终止反应。
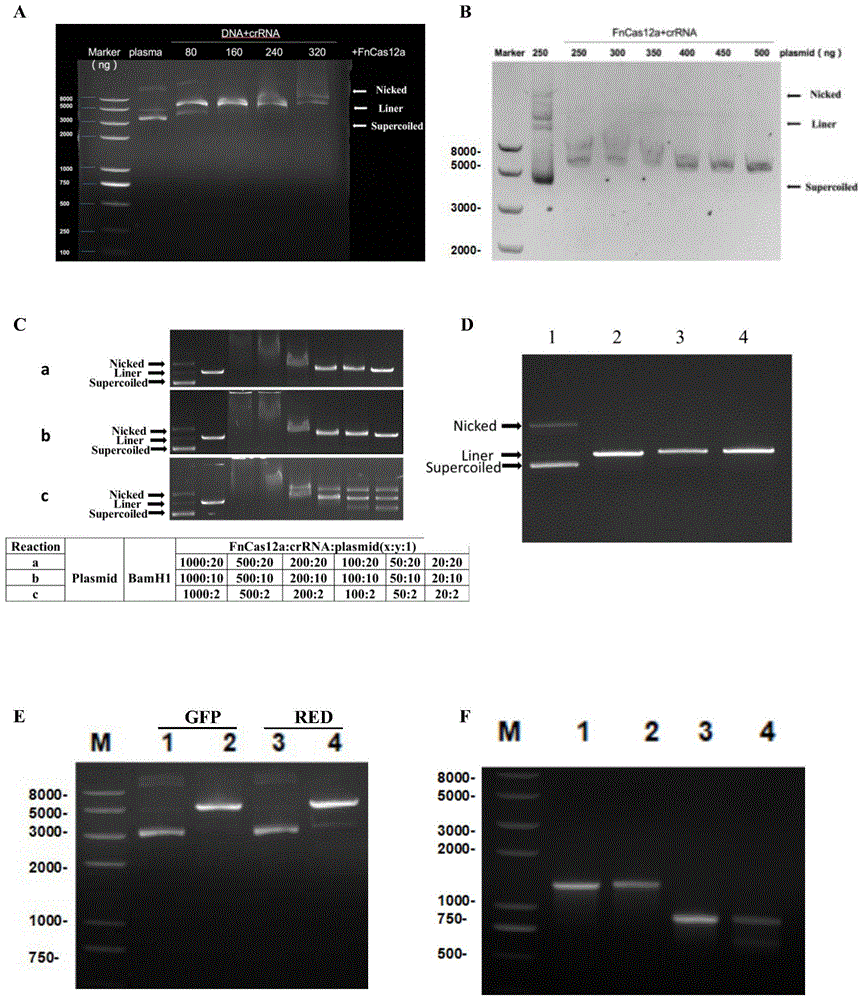
3、进行聚丙烯酰胺凝胶电泳,图3a显示了fncas12a特异性切割质粒的蛋白质梯度电泳结果图。结果说明,特异性底物能否完全切割与fncas12a蛋白的含量有关。
四、fncas12a特异性切割pacgfp1-n1的质粒梯度优化实验,具体步骤如下:
1、六种20ul反应体系中,分别加入步骤一纯化的fncas12a(125nm),向导
rna——pacgfp1-n1-1(0.5um),pacgfp1-n1质粒(250ng、300ng、350ng、400ng、450ng、500ng),rna酶抑制剂0.5ul,缓冲液nebbuffer3补齐。
2、在37℃反应中反应15min,然后98℃2min终止反应。
3、进行聚丙烯酰胺凝胶电泳,图3b显示了fncas12a特异性切割质粒的质粒梯度电泳结果图。结果说明,特异性底物的用量要与材料的用量存在一定的合适的比例。
五、fncas12a特异性切割pacgfp1-n1质粒的样品比例梯度优化实验,具体步骤如下:
1、三组20ul反应体系中,分别加入步骤一纯化的fncas12a(0.5um、1.25um、2.5um、5um、12.5um、25um),其中,第一组向导rna——pacgfp1-n1-1(500nm),第二组向导rna——pacgfp1-n1-1(250nm),第三组向导rna——pacgfp1-n1-1(50nm),pacgfp1-n1质粒(1ul),rna酶抑制剂0.5ul,缓冲液nebbuffer3补齐。
2、在37℃反应中反应15min,然后98℃2min终止反应。
3、进行聚丙烯酰胺凝胶电泳,图3c显示了fncas12a特异性切割质粒的样品比例梯度优化电泳结果图。结果说明,对特异性底物切割结果起决定性作用的是向导rna和底物的比例。六、fncas12a特异性切割pacgfp1-n1质粒的样品最优比实验,具体步骤如下:
1、20ul反应体系中,加入步骤一纯化的fncas12a(0.25um),向导
rna——pacgfp1-n1-1(0.25um),pacgfp1-n1质粒(1ul),rna酶抑制剂0.5ul,缓冲液nebbuffer3补齐。
2、在37℃反应中反应15min,然后98℃2min终止反应。
3、进行聚丙烯酰胺凝胶电泳,图3d、3e显示了fncas12a特异性切割pacgfp1-n1、pdsred-monomer-n1质粒的样品最优比电泳结果图(10:10:1)。
实施例2:fncas12a特异性切割pacgfp1-n1、pdsred-monomer-n1质粒的pcr片段的实验设计与构建,包括如下步骤:
一、同实施例1步骤一。
二、同实施例1步骤二。
三、利用pcr得到pacgfp1-n1、pdsred-monomer-n1质粒片段,具体步骤如下:
设计上游引物gfp-f/red-f和下游引物gfp-r/red-r,以纯化的pacgfp1-n1、pdsred-monomer-n1质粒为模板扩增pacgfp1-n1、pdsred-monomer-n1质粒片段,pcr完成后,直接用于fncas12a切割反应。图2c显示了质粒pacgfp1-n1的pcr片段的琼脂糖凝胶电泳图;
gfp-f:gtggcgataagtcgtgtcttaccgggt
gfp-r:gtcaaaccgctatccacgcccattgatg
red-f:gtacaaggccaagaagcccgtgcag
red-r:gtctcttgatcgatctttgcaaaagcctaggc
四、fncas12a特异性切割pacgfp1-n1、pdsred-monomer-n1质粒片段,具体步骤如下:
1、两种30ul反应体系中,分别加入纯化的fncas12a(250nm),向导rna——pacgfp1-n1-1(250nm)、pdsred-monomer-n1-1(250nm),pacgfp1-n1质粒片段(10ul)、pdsred-monomer-n1质粒片段(10ul),rna酶抑制剂0.5ul,缓冲液nebbuffer3补齐。
2、在37℃反应中反应15min,然后98℃2min终止反应。
3、进行聚丙烯酰胺凝胶电泳,图3f显示了fncas12a特异性切割pacgfp1-n1、pdsred-monomer-n1质粒片段电泳结果图。结果说明,fncas12a可以特异性切割dna片段。
实施例3:fncas12a非特异性切割单链dna——dnmt的短序列电泳的实验设计与构建,包括如下步骤:
一、同实施例1步骤一。
二、同实施例1步骤二。
三、fncas12a非特异性切割单链dna的性质验证,具体步骤如下:
1、30ul反应体系中,按图4a表格所示加入纯化的fncas12a(1um),向导rna——pacgfp1-n1-1(0.5um)、dnmt-1(0.5um),pacgfp1-n1质粒(1ul)、dnmt的dna片段(1ul),单链dna——egfp(10um),rna酶抑制剂0.5ul,缓冲液nebbuffer3补齐。
2、在37℃反应中反应15min,然后98℃2min终止反应。
3、配置浓度为12%的非变性聚丙烯酰胺凝胶,进行聚丙烯酰胺凝胶电泳,图4a显示了fncas12a非特异性切割单链dna——dnmt的短序列非变性电泳结果图。结果说明,fncas12a只能非特异性切割单链dna,不能非特异性切割双链dna。
四、fncas12a非特异性切割单链dna的实验优化,具体步骤如下:
1、四种30ul反应体系中,按图4b表格所示分别加入纯化的fncas12a(0.5um),向导rna——dnmt-1(0.5um),dnmt的dna片段(0.5um),单链dna——egfp(1um、2um、5um、6.25um),rna酶抑制剂0.5ul,缓冲液nebbuffer3补齐。
2、在37℃反应中反应15min,然后98℃2min终止反应。
3、配置浓度为20%的变性聚丙烯酰胺凝胶,进行聚丙烯酰胺凝胶电泳,图4b显示了fncas12a非特异性切割单链dna——dnmt的短序列变性电泳结果图。结果说明,fncas12a非特异性切割单链dna的量与fncas12a、向导rna和特异性底物的量有关。
实施例4:fncas12a非特异性切割荧光探针的荧光检测的实验设计与构建,包括如下步骤:
一、同实施例1步骤一。
二、同实施例1步骤二。
三、fncas12a非特异性切割荧光探针的探针梯度荧光数值检测实验构建,包括如下步骤:
1、8组100ul反应体系中,分别加入纯化的fncas12a(250nm),向导
rna——pacgfp1-n1-1(250nm),pacgfp1-n1质粒(4ul),荧光探针(其组成序列是ttatt,并在5,端标记荧光基团fam,在3,端标记猝灭基团bhq1,即fam-ttatt-bhq1)(0.5um、1um、1.5um、2um、2.5um、3um、3.5um、4um),rna酶抑制剂2ul,缓冲液nebbuffer3补齐,每组分别重复三次。
2、在37℃反应中反应60min,然后98℃2min终止反应。
3、用酶标仪检测(激发光492nm,发射光515nm),图5a显示了fncas12a非特异性切割荧光探针的探针梯度荧光数值,结果说明,荧光数值和荧光探针的量成正比。
四、fncas12a切割荧光探针的探针最低检测线荧光数值实验构建,包括如下步骤:
1、8组100ul反应体系中,分别加入纯化的fncas12a(250nm),向导rna——pacgfp1-n1-1(250nm),pacgfp1-n1质粒(4ul),荧光探针(fam-ttatt-bhq1)(10um、1um、0.1um、0.01um、0.001um、0.0001um、0.00001um、0.000001um),rna酶抑制剂2ul,缓冲液nebbuffer3补齐,每组分别重复三次。
2、在37℃反应中反应60min,然后98℃2min终止反应。
3、用酶标仪检测(激发光492nm,发射光515nm),图5b显示了fncas12a切割荧光探针的探针最低检测线荧光数值,结果说明,探针浓度过低会影响荧光检测灵敏度,探针的最低检测限可达60pm。
五、fncas12a非特异性切割荧光探针的材料影响探究的实验构建,包括如下步骤:
1、八组100ul反应体系中,按图5c表格所示加入纯化的fncas12a(250nm),向导rna——pacgfp1-n1-1(250nm),pacgfp1-n1质粒(4ul),荧光探针(fam-ttatt-bhq1)(4um),rna酶抑制剂2ul,缓冲液nebbuffer3补齐。
2、在37℃反应中反应60min,然后98℃2min终止反应。
3、用酶标仪检测(激发光492nm,发射光515nm),,图5c显示了fncas12a非特异性切割荧光探针的材料影响的荧光数值,结果说明,fncas12a蛋白对空白实验中荧光数值的检测误差比向导rna的影响大。
实施例5:疏水蛋白修饰的芯片的crispr切割反应的构建及应用,包括如下步骤:
一、同实施例1步骤一。
二、同实施例1步骤二。
三、芯片基底的实验设计与进行疏水蛋白修饰,包括如下步骤:
1、实验流程如图6所示,将芯片基底进行疏水蛋白修饰,在通风处中晾干。
2、将fncas12a蛋白和向导rna加到风干的疏水蛋白上进行孵育,然后加入特异性底物(pacgfp1-n1质粒、pdsred-monomer-n1质粒)和荧光探针(fam-ttatt-bhq1),37℃反应15min。
四、疏水蛋白修饰的芯片的crispr切割反应的构建及应用,包括如下步骤:
1、设置三组平行实验,同时设置三组无疏水蛋白修饰的平行实验,每组平行实验中,共设置五个5ul反应体系,pacgfp1-n1、pdsred-monomer-n1实验组分别加入纯化的fncas12a(250nm),向导rna——pacgfp1-n1-1(250nm)、pdsred-monomer-n1-1(250nm),pacgfp1-n1质粒(0.75ul)、pdsred-monomer-n1质粒(0.75ul),荧光探针(fam-ttatt-bhq1)(1um),rna酶抑制剂0.25ul,缓冲液nebbuffer3补齐;pacgfp1-n1、pdsred-monomer-n1对照组将两种crrna互换,其他实验条件不变;阴性对照组不加fncas12a蛋白,其他实验条件不变。
2、荧光共聚焦显微镜下观察结果,图7显示了疏水蛋白修饰和未经疏水蛋白修饰的芯片上crispr切割反应的荧光检测结果图,结果说明,疏水蛋白和fncas12a蛋白极有可能存在蛋白质互作作用,并会加强fncas12a蛋白的切割能力。
检测单基因突变疾病的crispr高通量生物芯片方法的建立:
利用fncas12a的切割特性和疏水蛋白的修饰能力,我们发明了特异性检测单基因突变疾病的crispr高通量生物芯片,该芯片具有效率高、成本低、周期短、特异性强的特点,且检测材料稳定、操作难度低,结果直观可靠。
整个反应体系可大致分为三个步骤,一是cas12a蛋白的切割反应,二是疏水蛋白hfbⅰ修饰的芯片基底荧光反应,三是hfbⅰ固定的cas12a的高通量检测。对于cas12a蛋白的切割反应阶段,其中3个材料是实验的关键,分别为cas12a,crrna和荧光探针。除了实施例中用到的fncas12a蛋白,其他cas12a蛋白同样适用于该方法。
对于疏水蛋白修饰的芯片基底荧光反应,本发明选用了hfbⅰ疏水蛋白对芯片基底进行修饰并对cas蛋白进行芯片固定,与fncas12a蛋白发生互作后,可增强cas12a蛋白行驶切割的能力,并选用fam和bhq1标记的短ssdna,其他可检测单链切割的标记方式理论上都是适用的,只要该荧光探针被切割后产生可检测的差异。
我们不仅创新性的实现了疏水蛋白hfbⅰ对crisprcas蛋白的固定,还实现了对单基因疾病的高通量检测。在保证cas蛋白生物活性的前提下,我们利用蛋白质互作和高通量检测提高了疾病检测的效率,该方法学的提出将在基因突变疾病检测方面有广阔的前景和应用。
表1本发明中涉及到的基因序列
表2本发明中涉及到的fncas12a蛋白和lbcas12a蛋白的名称和gi号
尽管上面结合附图对本发明的优选实施例进行了描述,但是本发明并不局限于上述的具体实施方式,上述的具体实施方式仅仅是示意性的,并不是限制性的,本领域的普通技术人员在本发明的启示下,在不脱离本发明宗旨和权利要求所保护的范围情况下,还可以做出很多形式,这些均属于本发明的保护范围之内。
<110>天津大学
<120>检测单基因突变的crispr高通量生物芯片
<130>
<160>12
<170>
<210>1
<211>4726
<212>dna
<213>人工序列
<400>1
tagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccg60
cgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccatt120
gacgtcaataatgacgtatgttcccatagtaacgccaatagggactttccattgacgtca180
atgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgcc240
aagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagta300
catgaccttatgggactttcctacttggcagtacatctacgtattagtcatcgctattac360
catggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacgggg420
atttccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaacg480
ggactttccaaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgt540
acggtgggaggtctatataagcagagctggtttagtgaaccgtcagatccgctagcgcta600
ccggactcagatctcgagctcaagcttcgaattctgcagtcgacggtaccgcgggcccgg660
gatccaccggtcatggtgagcaagggcgccgagctgttcaccggcatcgtgcccatcctg720
atcgagctgaatggcgatgtgaatggccacaagttcagcgtgagcggcgagggcgagggc780
gatgccacctacggcaagctgaccctgaagttcatctgcaccaccggcaagctgcctgtg840
ccctggcccaccctggtgaccaccctgagctacggcgtgcagtgcttctcacgctacccc900
gatcacatgaagcagcacgacttcttcaagagcgccatgcctgagggctacatccaggag960
cgcaccatcttcttcgaggatgacggcaactacaagtcgcgcgccgaggtgaagttcgag1020
ggcgataccctggtgaatcgcatcgagctgaccggcaccgatttcaaggaggatggcaac1080
atcctgggcaataagatggagtacaactacaacgcccacaatgtgtacatcatgaccgac1140
aaggccaagaatggcatcaaggtgaacttcaagatccgccacaacatcgaggatggcagc1200
gtgcagctggccgaccactaccagcagaatacccccatcggcgatggccctgtgctgctg1260
cccgataaccactacctgtccacccagagcgccctgtccaaggaccccaacgagaagcgc1320
gatcacatgatctacttcggcttcgtgaccgccgccgccatcacccacggcatggatgag1380
ctgtacaagtgagcggccgcgactctagatcataatcagccataccacatttgtagaggt1440
tttacttgctttaaaaaacctcccacacctccccctgaacctgaaacataaaatgaatgc1500
aattgttgttgttaacttgtttattgcagcttataatggttacaaataaagcaatagcat1560
cacaaatttcacaaataaagcatttttttcactgcattctagttgtggtttgtccaaact1620
catcaatgtatcttaaggcgtaaattgtaagcgttaatattttgttaaaattcgcgttaa1680
atttttgttaaatcagctcattttttaaccaataggccgaaatcggcaaaatcccttata1740
aatcaaaagaatagaccgagatagggttgagtgttgttccagtttggaacaagagtccac1800
tattaaagaacgtggactccaacgtcaaagggcgaaaaaccgtctatcagggcgatggcc1860
cactacgtgaaccatcaccctaatcaagttttttggggtcgaggtgccgtaaagcactaa1920
atcggaaccctaaagggagcccccgatttagagcttgacggggaaagccggcgaacgtgg1980
cgagaaaggaagggaagaaagcgaaaggagcgggcgctagggcgctggcaagtgtagcgg2040
tcacgctgcgcgtaaccaccacacccgccgcgcttaatgcgccgctacagggcgcgtcag2100
gtggcacttttcggggaaatgtgcgcggaacccctatttgtttatttttctaaatacatt2160
caaatatgtatccgctcatgagacaataaccctgataaatgcttcaataatattgaaaaa2220
ggaagagtcctgaggcggaaagaaccagctgtggaatgtgtgtcagttagggtgtggaaa2280
gtccccaggctccccagcaggcagaagtatgcaaagcatgcatctcaattagtcagcaac2340
caggtgtggaaagtccccaggctccccagcaggcagaagtatgcaaagcatgcatctcaa2400
ttagtcagcaaccatagtcccgcccctaactccgcccatcccgcccctaactccgcccag2460
ttccgcccattctccgccccatggctgactaattttttttatttatgcagaggccgaggc2520
cgcctcggcctctgagctattccagaagtagtgaggaggcttttttggaggcctaggctt2580
ttgcaaagatcgatcaagagacaggatgaggatcgtttcgcatgattgaacaagatggat2640
tgcacgcaggttctccggccgcttgggtggagaggctattcggctatgactgggcacaac2700
agacaatcggctgctctgatgccgccgtgttccggctgtcagcgcaggggcgcccggttc2760
tttttgtcaagaccgacctgtccggtgccctgaatgaactgcaagacgaggcagcgcggc2820
tatcgtggctggccacgacgggcgttccttgcgcagctgtgctcgacgttgtcactgaag2880
cgggaagggactggctgctattgggcgaagtgccggggcaggatctcctgtcatctcacc2940
ttgctcctgccgagaaagtatccatcatggctgatgcaatgcggcggctgcatacgcttg3000
atccggctacctgcccattcgaccaccaagcgaaacatcgcatcgagcgagcacgtactc3060
ggatggaagccggtcttgtcgatcaggatgatctggacgaagagcatcaggggctcgcgc3120
cagccgaactgttcgccaggctcaaggcgagcatgcccgacggcgaggatctcgtcgtga3180
cccatggcgatgcctgcttgccgaatatcatggtggaaaatggccgcttttctggattca3240
tcgactgtggccggctgggtgtggcggaccgctatcaggacatagcgttggctacccgtg3300
atattgctgaagagcttggcggcgaatgggctgaccgcttcctcgtgctttacggtatcg3360
ccgctcccgattcgcagcgcatcgccttctatcgccttcttgacgagttcttctgagcgg3420
gactctggggttcgaaatgaccgaccaagcgacgcccaacctgccatcacgagatttcga3480
ttccaccgccgccttctatgaaaggttgggcttcggaatcgttttccgggacgccggctg3540
gatgatcctccagcgcggggatctcatgctggagttcttcgcccaccctagggggaggct3600
aactgaaacacggaaggagacaataccggaaggaacccgcgctatgacggcaataaaaag3660
acagaataaaacgcacggtgttgggtcgtttgttcataaacgcggggttcggtcccaggg3720
ctggcactctgtcgataccccaccgagaccccattggggccaatacgcccgcgtttcttc3780
cttttccccaccccaccccccaagttcgggtgaaggcccagggctcgcagccaacgtcgg3840
ggcggcaggccctgccatagcctcaggttactcatatatactttagattgatttaaaact3900
tcatttttaatttaaaaggatctaggtgaagatcctttttgataatctcatgaccaaaat3960
cccttaacgtgagttttcgttccactgagcgtcagaccccgtagaaaagatcaaaggatc4020
ttcttgagatcctttttttctgcgcgtaatctgctgcttgcaaacaaaaaaaccaccgct4080
accagcggtggtttgtttgccggatcaagagctaccaactctttttccgaaggtaactgg4140
cttcagcagagcgcagataccaaatactgtccttctagtgtagccgtagttaggccacca4200
cttcaagaactctgtagcaccgcctacatacctcgctctgctaatcctgttaccagtggc4260
tgctgccagtggcgataagtcgtgtcttaccgggttggactcaagacgatagttaccgga4320
taaggcgcagcggtcgggctgaacggggggttcgtgcacacagcccagcttggagcgaac4380
gacctacaccgaactgagatacctacagcgtgagctatgagaaagcgccacgcttcccga4440
agggagaaaggcggacaggtatccggtaagcggcagggtcggaacaggagagcgcacgag4500
ggagcttccagggggaaacgcctggtatctttatagtcctgtcgggtttcgccacctctg4560
acttgagcgtcgatttttgtgatgctcgtcaggggggcggagcctatggaaaaacgccag4620
caacgcggcctttttacggttcctggccttttgctggccttttgctcacatgttctttcc4680
tgcgttatcccctgattctgtggataaccgtattaccgccatgcat4726
<210>2
<211>4691
<212>dna
<213>人工序列
<400>2
tagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccg60
cgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccatt120
gacgtcaataatgacgtatgttcccatagtaacgccaatagggactttccattgacgtca180
atgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgcc240
aagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagta300
catgaccttatgggactttcctacttggcagtacatctacgtattagtcatcgctattac360
catggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacgggg420
atttccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaacg480
ggactttccaaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgt540
acggtgggaggtctatataagcagagctggtttagtgaaccgtcagatccgctagcgcta600
ccggactcagatctcgagctcaagcttcgaattctgcagtcgacggtaccgcgggcccgg660
gatccaccggtcgccaccatggacaacaccgaggacgtcatcaaggagttcatgcagttc720
aaggtgcgcatggagggctccgtgaacggccactacttcgagatcgagggcgagggcgag780
ggcaagccctacgagggcacccagaccgccaagctgcaggtgaccaagggcggccccctg840
cccttcgcctgggacatcctgtccccccagttccagtacggctccaaggcctacgtgaag900
caccccgccgacatccccgactacatgaagctgtccttccccgagggcttcacctgggag960
cgctccatgaacttcgaggacggcggcgtggtggaggtgcagcaggactcctccctgcag1020
gacggcaccttcatctacaaggtgaagttcaagggcgtgaacttccccgccgacggcccc1080
gtaatgcagaagaagactgccggctgggagccctccaccgagaagctgtacccccaggac1140
ggcgtgctgaagggcgagatctcccacgccctgaagctgaaggacggcggccactacacc1200
tgcgacttcaagaccgtgtacaaggccaagaagcccgtgcagctgcccggcaaccactac1260
gtggactccaagctggacatcaccaaccacaacgaggactacaccgtggtggagcagtac1320
gagcacgccgaggcccgccactccggctcccagtagagcggccgcgactctagatcataa1380
tcagccataccacatttgtagaggttttacttgctttaaaaaacctcccacacctccccc1440
tgaacctgaaacataaaatgaatgcaattgttgttgttaacttgtttattgcagcttata1500
atggttacaaataaagcaatagcatcacaaatttcacaaataaagcatttttttcactgc1560
attctagttgtggtttgtccaaactcatcaatgtatcttaaggcgtaaattgtaagcgtt1620
aatattttgttaaaattcgcgttaaatttttgttaaatcagctcattttttaaccaatag1680
gccgaaatcggcaaaatcccttataaatcaaaagaatagaccgagatagggttgagtgtt1740
gttccagtttggaacaagagtccactattaaagaacgtggactccaacgtcaaagggcga1800
aaaaccgtctatcagggcgatggcccactacgtgaaccatcaccctaatcaagttttttg1860
gggtcgaggtgccgtaaagcactaaatcggaaccctaaagggagcccccgatttagagct1920
tgacggggaaagccggcgaacgtggcgagaaaggaagggaagaaagcgaaaggagcgggc1980
gctagggcgctggcaagtgtagcggtcacgctgcgcgtaaccaccacacccgccgcgctt2040
aatgcgccgctacagggcgcgtcaggtggcacttttcggggaaatgtgcgcggaacccct2100
atttgtttatttttctaaatacattcaaatatgtatccgctcatgagacaataaccctga2160
taaatgcttcaataatattgaaaaaggaagagtcctgaggcggaaagaaccagctgtgga2220
atgtgtgtcagttagggtgtggaaagtccccaggctccccagcaggcagaagtatgcaaa2280
gcatgcatctcaattagtcagcaaccaggtgtggaaagtccccaggctccccagcaggca2340
gaagtatgcaaagcatgcatctcaattagtcagcaaccatagtcccgcccctaactccgc2400
ccatcccgcccctaactccgcccagttccgcccattctccgccccatggctgactaattt2460
tttttatttatgcagaggccgaggccgcctcggcctctgagctattccagaagtagtgag2520
gaggcttttttggaggcctaggcttttgcaaagatcgatcaagagacaggatgaggatcg2580
tttcgcatgattgaacaagatggattgcacgcaggttctccggccgcttgggtggagagg2640
ctattcggctatgactgggcacaacagacaatcggctgctctgatgccgccgtgttccgg2700
ctgtcagcgcaggggcgcccggttctttttgtcaagaccgacctgtccggtgccctgaat2760
gaactgcaagacgaggcagcgcggctatcgtggctggccacgacgggcgttccttgcgca2820
gctgtgctcgacgttgtcactgaagcgggaagggactggctgctattgggcgaagtgccg2880
gggcaggatctcctgtcatctcaccttgctcctgccgagaaagtatccatcatggctgat2940
gcaatgcggcggctgcatacgcttgatccggctacctgcccattcgaccaccaagcgaaa3000
catcgcatcgagcgagcacgtactcggatggaagccggtcttgtcgatcaggatgatctg3060
gacgaagagcatcaggggctcgcgccagccgaactgttcgccaggctcaaggcgagcatg3120
cccgacggcgaggatctcgtcgtgacccatggcgatgcctgcttgccgaatatcatggtg3180
gaaaatggccgcttttctggattcatcgactgtggccggctgggtgtggcggaccgctat3240
caggacatagcgttggctacccgtgatattgctgaagagcttggcggcgaatgggctgac3300
cgcttcctcgtgctttacggtatcgccgctcccgattcgcagcgcatcgccttctatcgc3360
cttcttgacgagttcttctgagcgggactctggggttcgaaatgaccgaccaagcgacgc3420
ccaacctgccatcacgagatttcgattccaccgccgccttctatgaaaggttgggcttcg3480
gaatcgttttccgggacgccggctggatgatcctccagcgcggggatctcatgctggagt3540
tcttcgcccaccctagggggaggctaactgaaacacggaaggagacaataccggaaggaa3600
cccgcgctatgacggcaataaaaagacagaataaaacgcacggtgttgggtcgtttgttc3660
ataaacgcggggttcggtcccagggctggcactctgtcgataccccaccgagaccccatt3720
ggggccaatacgcccgcgtttcttccttttccccaccccaccccccaagttcgggtgaag3780
gcccagggctcgcagccaacgtcggggcggcaggccctgccatagcctcaggttactcat3840
atatactttagattgatttaaaacttcatttttaatttaaaaggatctaggtgaagatcc3900
tttttgataatctcatgaccaaaatcccttaacgtgagttttcgttccactgagcgtcag3960
accccgtagaaaagatcaaaggatcttcttgagatcctttttttctgcgcgtaatctgct4020
gcttgcaaacaaaaaaaccaccgctaccagcggtggtttgtttgccggatcaagagctac4080
caactctttttccgaaggtaactggcttcagcagagcgcagataccaaatactgtccttc4140
tagtgtagccgtagttaggccaccacttcaagaactctgtagcaccgcctacatacctcg4200
ctctgctaatcctgttaccagtggctgctgccagtggcgataagtcgtgtcttaccgggt4260
tggactcaagacgatagttaccggataaggcgcagcggtcgggctgaacggggggttcgt4320
gcacacagcccagcttggagcgaacgacctacaccgaactgagatacctacagcgtgagc4380
tatgagaaagcgccacgcttcccgaagggagaaaggcggacaggtatccggtaagcggca4440
gggtcggaacaggagagcgcacgagggagcttccagggggaaacgcctggtatctttata4500
gtcctgtcgggtttcgccacctctgacttgagcgtcgatttttgtgatgctcgtcagggg4560
ggcggagcctatggaaaaacgccagcaacgcggcctttttacggttcctggccttttgct4620
ggccttttgctcacatgttctttcctgcgttatcccctgattctgtggataaccgtatta4680
ccgccatgcat4691
<210>3
<211>43
<212>dna
<213>人工序列
<400>3
cgtcaatgggtggagtatttacggatctacaacagtagaaatt43
<210>4
<211>43
<212>dna
<213>人工序列
<400>4
agttccgcgttacataacttacggatctacaacagtagaaatt43
<210>5
<211>42
<212>dna
<213>人工序列
<400>5
gagtaacagacatggaccatcagatctacaacagtagaaatt42
<210>6
<211>324
<212>dna
<213>homosapiens(human)
<400>6
ggtcgcgagtgcgttaattgcggggcaatgtctaccccactgtggcgtcgtgacggtacc60
ggtcattatctgtgtaacgcctgcggtctgtaccataaaatgaacggcatcaaccgtccg120
ctgattaaaccgcaacgtcgtctgtctgcaagtcgtcgcgttggtctgagttgcgcaaat180
tgtcaaaccaccaccaccaccctgtggcgtcgtaacgcagaaggcgaaccagtttgtaac240
gcttgcggcctgtatatgaaactgcacggcgttccacgtccactggcaatgcgtaaagaa300
ggcatccagacccgcaaacgcaaa324
<210>7
<211>50
<212>dna
<213>homosapiens(human)
<400>7
gtcacgccacttgacaggcgagtaacagacatggaccatcaggaaacatt50
<210>8
<211>55
<212>dna
<213>人工序列
<400>8
gccggggtggtgcccatcctggtcgagctggacggcgacgtaaacggccacaagc55
<210>9
<211>27
<212>dna
<213>人工序列
<400>9
gtggcgataagtcgtgtcttaccgggt27
<210>10
<211>28
<212>dna
<213>人工序列
<400>10
gtcaaaccgctatccacgcccattgatg28
<210>11
<211>25
<212>dna
<213>人工序列
<400>11
gtacaaggccaagaagcccgtgcag25
<210>12
<211>32
<212>dna
<213>人工序列
<400>12
gtctcttgatcgatctttgcaaaagcctaggc32
- 还没有人留言评论。精彩留言会获得点赞!