一种暗纹东方鲀快速生长相关的SNP位点与应用的制作方法
本发明属于鱼类遗传选育技术领域,具体涉及一种暗纹东方鲀快速生长相关的snp位点与应用。
背景技术:
暗纹东方鲀(takifuguobscurus)隶属于鲀形目,鲀科,东方鲀属,主要分布于我国长江中下游以及渤海、黄海等地。暗纹东方鲀肉质鲜美,营养价值丰富,古代民间有“拼死吃河豚”的说法。2016年10月起,我国有条件地放开红鳍东方鲀和暗纹东方鲀的生产养殖。暗纹东方鲀生长速度很慢,明显小于红鳍东方鲀。2年龄养殖型红鳍东方鲀平均体长30.4cm左右,平均体重1062g左右,而同龄养殖型暗纹东方鲀平均体长只有24.9cm左右,平均体重只有611g左右。低生长速度直接导致暗纹东方鲀的产量低,影响着养殖效益,制约着暗纹东方鲀养殖业的发展。因此,改良暗纹东方鲀生长速度工作势在必行。
分子标记辅助育种是快速获得理想家系、品系或品种的现代生物育种的理想方法之一。单核苷酸多态性(singlenucleotidepolymorphism,snp)是指基因组dna上由单个核苷酸突变所引起的dna序列多态性,通过确定snp及基因型分型进行选种是成熟的分子生物技术,已在畜禽及水生动物中得以应用,与传统方法相比,提高了选育效率。但在暗纹东方鲀育种领域中还缺少可以应用的辅助分子标记技术。
技术实现要素:
本发明的目的是提供一种暗纹东方鲀快速生长相关的snp位点与应用,即筛查出基因中与体重、体长和体全长生长性状相关的snp位点,并用于选育快速生长暗纹东方鲀。
本发明首先提供一种与暗纹东方鲀生长性状相关的snp位点,所述的位点位于核苷酸序列为seqidno:1的mstn基因的第724位,其碱基为c或t;
本发明提供的snp位点用于选育具有快速生长潜力的暗纹东方鲀个体。
本发明另一个方面提供一种筛选快速生长潜力的暗纹东方鲀个体的方法,是通过检测上述的snp位点来实现的;
所述的方法,是通过pcr扩增方法检测暗纹东方鲀个体,pcr产物进行测序分析后确定待检测的个体是否具有快速生长潜力;
所述的pcr扩增方法,其中所使用的引物的序列信息如下:
snp-f:5’-atgcaactgtctccgagcat-3’(seqidno:2),
snp-r:5’-ttgagctcaccaggccttcct-3’(seqidno:3);
所述的测序分析,是确定snp位点的基因型分型,其中基因型为tt纯合型个体的体重、体长和体全长显著高于cc和ct基因型个体生长性状的表型值(p<0.05)。
本发明通过分析位点基因型频率与暗纹东方鲀生长性状的相关性,发现暗纹东方鲀的mstn基因的724碱基处存在与生长性状相关的snp位点,基因型为tt纯合型个体的体重、体长和体全长显著高于cc和ct基因型个体生长性状的表型值(p<0.05)。因此,生产中可优先选择该位点基因型为tt型个体作为亲本或者进行规模养殖。
附图说明
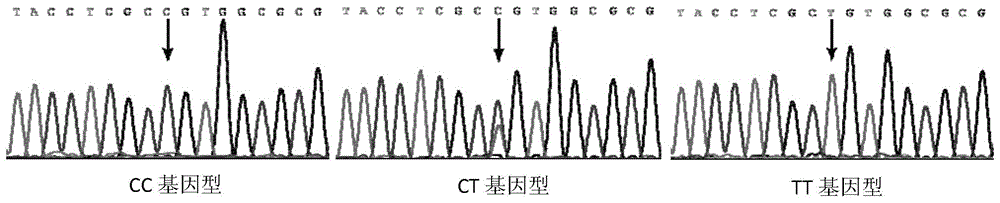
图1:本发明snp标记位点处cc、ct和tt三种基因型的测序峰图。
具体实施方式
本发明通过测序和clustal比对,筛查snp位点,并分析位点基因型频率与暗纹东方鲀生长性状的相关性,发现序列seqidno.1的肌肉生长抑制素基因(myostatin,mstn)第724碱基处,基因型为tt纯合型个体的体重、体长和体全长显著高于cc和ct基因型个体生长性状的表型值(p<0.05)。因此,生产中可优先选择该位点基因型为tt型个体作为亲本或者进行规模养殖。
下面通过实施例对本发明做进一步说明。
实施例1克隆暗纹东方鲀mstn基因核苷酸序列
肌肉生长抑制素基因(myostatin,mstn),又名生长分化因子8(growthanddifferentiationfactor8),属于转化生长因子β(transforminggrowthfactorbeta)超家族,是肌肉生长发育的负调控因子。
本发明克隆mstn基因的步骤如下:
a)暗纹东方鲀基因组的提取。采用常规酚仿法抽提暗纹东方鲀鳍条中的基因组dna。取暗纹东方鲀尾鳍组织120mg于1.5ml的离心管中,并用眼科剪将其剪碎;向已剪碎尾鳍的离心管中加入700μldna提取液;加蛋白酶k(20mg/ml)5μl,轻轻混匀,放入55℃水浴中消化2小时;将溶液冷却至室温,加等体积苯酚,抽提10分钟,12000rpm离心10分钟;取上清加入一新的1.5ml离心管中,加等体积酚,仿抽提10分钟,12000rpm离心10分钟;取上清加入一新的1.5ml离心管中,加等体积氯仿抽提10分钟,12000rpm离心10分钟;取上清加入一新的离心管中,加2倍体积无水乙醇,12000rpm离心5分钟;弃掉乙醇溶液,用70%乙醇洗沉淀2次,5000rpm离心5分钟;弃掉乙醇,室温放置10分钟,用100μlte溶解沉淀dna。用1%的琼脂糖凝胶电泳检测dna,dna溶液于-4℃或-20℃保存备用。
b)引物设计。根据genbank已报道的红鳍东方鲀、斑马鱼、鲤鱼、大西洋鲑、牙鲆、白斑狗鱼、小鼠和人类等物种的mstn基因核苷酸序列,选择保守性高的,且能够扩增目的基因序列全长的区域设计引物:
mstn-f1:5’-cgttgcgcaatcaccgtaag-3’,
mstn-r1:5’-ctacggcagctgataaggtg-3’,克隆暗纹东方鲀的mstn基因核苷酸序列全长。
c)目的基因pcr扩增。反应体系为25μl:10×buffer2.5μl,dntp2μl,mstn-f1引物1μl,mstn-r1引物1μl,基因组dna1μl,extaq酶0.25μl,双蒸水补足至25μl。pcr反应程序为:98℃,30s;98℃10s,65℃30s,72℃2min30s,30个循环;72℃3min。扩增产物使用1%琼脂糖电泳检验。
d)目的片段的ta克隆。pcr产物使用uniq-10柱式胶回收试剂盒回收目的片段,再与pmd18-t载体(大连宝生物工程有限公司)连接,转化至大肠杆菌dh5α中,挑选阳性克隆,接种于600μllb培养基中,200rpm振荡培养过夜,送生工生物工程(上海)有限公司进行sanger测序。获得的暗纹东方鲀mstn基因全长2373bp(seqidno:1),与红鳍东方鲀mstn基因序列比对,同源性为99.73%。利用dnaman8.0软件推导氨基酸序列、expasy(https://www.expasy.org/)网站分析氨基酸理化性质及蛋白质疏水性、signalp4.1(http://www.cbs.dtu.dk/services/signalp/)在线软件预测信号肽位点。分析表明该基因包括三个外显子和两个内含子,5’utr长度327bp,3’utr长度156bp;orf1131bp,编码376个氨基酸,在第23-24氨基酸处有信号肽剪切位点;mstn蛋白分子量42658.97da,等电点6.38,总平均亲水性-0.391,为亲水性不稳定蛋白。
实施例2:筛选获得snp位点
确定暗纹东方鲀mstn基因与生长性状关联的snp的位点,包括下列步骤:
a)暗纹东方鲀样品的获取:所采用的群体为大连天正实业有限公司养殖场孵化的一个家系的暗纹东方鲀,从该家系随机选出109个暗纹东方鲀个体。
b)数据的采集与基因组dna的提取:对109个个体的体重、体长和体全长的表型值进行测量和记录,同时剪取尾鳍部分鳍条用于基因组dna的提取。
c)pcr反应及测序。反应体系为25μl:10×buffer2.5μl,dntp2μl,snp-f引物(seqidno.2)1μl,snp-r引物(seqidno.3)1μl,基因组dna1μl,extaq酶0.25μl,双蒸水补足至25μl。pcr反应程序为:98℃,30s;98℃10s,65℃30s,72℃1min10s,30个循环;72℃3min。扩增产物使用1.5%琼脂糖电泳检验。
使用abi3730测序仪对纯化后的pcr产物进行sanger测序,筛选出
dna序列seqidno.1第724碱基处存在snp位点,等位基因为c
和t,有cc、ct和tt三种基因型;结果见图1。
snp基因型与生长性状表型值关联分析:
利用spss18.0软件对该snp位点的三种基因型与暗纹东方鲀体重、体长和体全长的性状表型值分别进行最小二乘的统计分析关联分析,计算该snp位点基因型与生长性状的关联性,结果见表1。
采用的模型如下:
yij=μ+gi+eij
其中yij表示i基因型的第j个个体的体长性状测定值;μ是测定值的均值;gi是基因型i的遗传效应;eij表示随机误差效应。
表1:暗纹东方鲀mstn基因snp与生长性状的相关信息(均值±标准差)
注:同列中字母相同为差异不显著,相邻字母为差异显著(p<0.05)
由表1可知,基因型为tt纯合型个体的体重、体长和体全长显著高于cc和ct基因型个体生长性状的表型值(p<0.05)。
利用引物snp-f(seqidno.2)和snp-r(seqidno.3)扩增亲本mstn基因序列,测序并检测snp位点基因型,挑选出符合基因型为tt快速生长的亲鱼。利用基因型tt快速生长的亲鱼构建全同胞和半同胞家系,用未筛选亲鱼构建家系作为对照组。在家系培育4个月后,对快速生长型亲鱼和未筛选亲鱼的f1代进行测序及snp分型,并对f1代个体生长指标检测。结果表明,基因型为tt的亲鱼子代的生长指标也显著高于未筛选亲鱼的子代。
综上结果表明,通过使用本发明的snp引物和方法筛选的暗纹东方鲀tt基因型个体,其生长速度显著高于同家系cc和ct型个体,对暗纹东方鲀良品种的选育工作提供了快速准确的检测方法。
序列表
<110>大连海洋大学
<120>一种暗纹东方鲀快速生长相关的snp位点与应用
<160>3
<170>siposequencelisting1.0
<210>1
<211>2373
<212>dna
<213>人工序列(artificialsequence)
<400>1
cgttgcgcaatcaccgtaagcgcggttaatttatccatctggggacgcgttcagcacatg60
ctcgcgctctccgtccctgtatgggttgacaaggaaaaaaacttccctgtcagtcggttt120
aaattcattgttgtctgtccagccaatcatggtttttgacgacacaaaagcgactaaagt180
tggagtataaaaaggtgcgcgctaataaagcatgatgcctctcagtgtgggacattaatc240
caaacccagcccaggctgcgcgtcagaaccagaacacgccaagggatatactttttatag300
aacgcatttccacgtcttcaggagacaatgcaactgtctccgagcatgctgcattttagc360
ctgatgatttctttgagtttggtggtgttgagtggccaagagacgcaccagcagccgccc420
gtcggcagcccagaggacacggagcagtgcgtcacctgcgacgtccgacagcacatcaaa480
accatgcgattaaacgccatcaagtctcagattctgagcaagctgcggatgaaagaggct540
ccgaacatcagccgggacacggtgaagcagctcctgcccaaagcgccgccgctgcagcag600
ctcctcgaccagtacgacgtgctgggcgatgacaacagggacgtggtcacggaggaggac660
gacgagcacgccatcacggagaccatcatgatgatggccactgaacgtgagtgtttacct720
cgccgtggcgcgcgccagcaacggtgcgctaaagtctccaaaagcgctttacgcgcgctg780
acggcaggaaatgttcgtttcagagcgctgccgtaaattaattgtaaattaaaaatgtaa840
gacagcaacggatagcgacgcgtaataatactgtgtcggttaattgtggagagaatgaaa900
gctaaaaggctcctcgccgactcatttgtgacactattaacgcgcgcgttcgttctcttc960
tctctccagccgcgtccgtcgtccaagtgaacggggagcccaaatgctgccatttctcct1020
tcactcaaaagtttcaagtcagccggctggtccgcgcgcagctctgggtgcatctgcgcc1080
cggcggccgaggccaccaccgtgttcctgcaaatctcccgcctgatgccggtcacagacg1140
ggaaccggcacatccgcatccgctccctgaagctggacgtgaaggccggggcgagctcct1200
ggcagagcatagacgtcaagcaagtgctgagcgtgtggctgcggcagccggagaccaact1260
ggggcatcgagattaacgcgttcgactcgagaggaaaagacttggccgtgacctccacac1320
agcccggagaggaaggcctggtgagctcaaccgcttcttcctttccttagcatgaaactt1380
taggtgagaccttgtagttgcgcacgcacgaaaacaagctccccgcgcgccattgttttc1440
accttcagggggccgaggagctttgaaaggtccgcaggtggttaaacattcctgcttttc1500
actgccgctgcagagaatctagacttttgcaaagcacttaatcaaccccgatcacatgtg1560
ggcaaacatctggatttggacagctgcccggtcagcaacaaccccgaatctgatggctgg1620
attatataaacgttcacctgctgcacattccttacagtattctgtatgactgtgcaaatc1680
agagtaattggctgcacacacacacacacacacacacacacacatgcgcgctggtgctta1740
atcagccataatctaacttctgcaagactttcataatattatcaggctgtgtttgctcgg1800
tcagcgtcctcgctcacatgcattctgtcgcttcagcaacctttcatggaggtgaagatc1860
tcggaggggcccaggcgtgtccgcagagacctgggactggactgcgacgagaactctcca1920
gagtcccgttgctgccgctacccgctcacggtggactttgaagactttggctgggactgg1980
attatagccccaaagcgctacaaggccaactattgctccggggagtgtgagtacatgcac2040
ttgcagaaatacccgcacacccacctggtgaacaaggccaaccccagagggaccgcgggc2100
ccttgctgtacccccaccaagatgtcgcccatcaacatgctctacttcaaccaagaacag2160
cagatcatctacggaaagatcccctccatggtggtggaccgttgtggatgtctttaagtc2220
gggggggccttctagtgtcagactttttgatgatctaccagttccagcgctttttttact2280
ccttcctgcagcgacacggtgcaatagaaccagagtagaggcctcaaaccagtccgacct2340
tcctgcagggcaacaccttatcagctgccgtag2373
<210>2
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>2
atgcaactgtctccgagcat20
<210>3
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>3
ttgagctcaccaggccttcct21
- 还没有人留言评论。精彩留言会获得点赞!