一种褐藻胶裂解酶突变体及其应用的制作方法
本发明涉及一种褐藻胶裂解酶突变体及其应用,属于酶工程和基因工程领域。
背景技术:
褐藻胶裂解酶是一种降解褐藻中海藻酸盐的裂解酶,主要通过4-o-糖苷键的β-消除反应催化藻酸盐的解聚,并在非还原端c-4和c-5之间产生具有不饱和双键的寡聚糖醛酸结构。根据褐藻胶裂解酶的底物偏好性可分为polym,polyg和polymg特异性裂解酶;根据作用方式的不同,又可分为内切酶和外切酶,内切酶主要切割海藻酸聚合物内部的糖苷键,产生不饱和低聚糖,而外切酶则是进一步将褐藻寡糖降解为单糖。
目前随着对褐藻寡糖研究的深入,越来越多的生物活性被发现,如抗肿瘤、抗过敏、抗菌、凝血、抗逆和抗氧化等,其广泛的应用价值促进了研究人员对褐藻寡糖制备方法的进一步探索。传统的褐藻寡糖降解法主要有:酸解、碱解、氧化降解,这些降解方法不仅容易造成环境污染,而且所得到的寡糖生物活性低且效果差。相反,酶法制备褐藻寡糖不仅条件温和,得率高、易受控制、不污染环境,还具有较好的寡糖生物活性,因此逐渐取代传统化学降解法成为主要的寡糖制备方式。然而当前筛选到的多是底物特异性差、酶活性低的褐藻胶裂解酶,应用于工业化的褐藻胶裂解酶产品少之又少,所以获得具有高活性和广泛底物特异性的褐藻胶裂解酶成了当前的研究重点,尤其是通过基因工程及蛋白质工程手段提高褐藻胶裂解酶的催化活性,更是受到了大多数研究者的青睐。
本发明采用结构序列分析和discoverystudio软件辅助筛选相结合的方法,确定突变位点,并通过pcr定点突变得到催化活性明显提高的突变体,为其进一步的工业化应用奠定基础。
技术实现要素:
本发明的目的是提供一种褐藻胶裂解酶突变体及其应用,采用结构序列分析和discoverystudio软件辅助筛选相结合的方法,确定突变位点,并通过pcr定点突变得到催化活性明显提高的突变体,为其进一步的工业化应用奠定基础。
为实现上述目的采用以下技术方案:
本发明中的褐藻胶裂解酶基因algl(核苷酸序列如seqidno.1所示)来源于分离自舟山群岛海水的pseudoalteromonassp.zb7-4,全长1203bp,编码400个氨基酸,第1-31位氨基酸为信号肽,32-131位氨基酸为碳水化合物结合结构域(即cbm结构域),197-385位氨基酸为催化结构域。在157-158位氨基酸之间进行截断,截除第1-157位氨基酸,得到褐藻胶裂解酶截短酶algl-t157n,其氨基酸序列如seqidno.3所示。
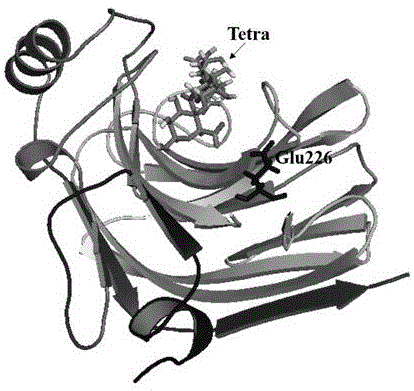
提交截短酶algl-t157n氨基酸序列到swissmodel在线服务器上进行同源建模,采用saves服务器中的verify-3d、errat及procheck程序进行蛋白模型结构评价,采用autodocktools将模型与甘露糖醛酸四糖小分子tetra进行分子对接,得到对接结果(见图1)。采用结构序列分析和discoverystudio软件辅助筛选相结合的方法,确定突变位点,在algl-t157n氨基酸序列seqidno.3基础上,将第226位谷氨酸定点突变为赖氨酸,得到催化活性明显提高的褐藻胶裂解酶突变体e226k,其氨基酸序列如seqidno.4所示,核苷酸序列如seqidno.5所示。
突变体e226k酶活较algl-t157n提高了1.11倍,比活提高了1.03倍;最适温度为55℃,较algl-t157n提高了5℃,在ph6.0-8.0范围内保温1h后残余酶活力达90%以上,具有较强的ph稳定性。动力学参数测定发现,e226k对褐藻酸钠、polym和polyg这3种底物的km值分别下降了89.69%、88.14%、61.11%,表明底物亲和力明显增强;kcat分别为algl-t157n的118.02%、114.62%、151.37%,kcat/km值分别较algl-t157n提高了10.22、8.59、2.97倍。
分别采用突变体e226k酶解和酸解制备褐藻寡糖,并对褐藻寡糖产物进行esi-ms质谱分析,结果发现,酶解法得到的褐藻寡糖为不饱和寡糖,由单糖、二糖和三糖组成;酸解法得到的褐藻寡糖为饱和寡糖,由单糖和二糖组成,以单糖为主。据研究发现,不饱和寡糖比饱和寡糖具有更高的生物活性,因此酶解法制备褐藻寡糖能更好地保留寡糖的生物活性。
本发明的优点在于:
本发明突变体e226k酶活较algl-t157n提高了1.11倍,比活提高了1.03倍;突变体e226k最适温度为55℃,较algl-t157n提高了5℃,在ph6.0-8.0范围内保温1h后残余酶活力达90%以上,具有较强的ph稳定性;而且突变体对褐藻酸钠、polym和polyg这3种底物的催化效率kcat/km分别比algl-t157n提高了10.22、8.59、2.97倍。采用酶解方法制备褐藻寡糖能更好地保留寡糖的生物活性。
附图说明
图1截短酶algl-t157n与甘露糖醛酸四糖分子tetra的对接结构图。注:glu226为定点突变位点。
图2截短酶algl-t157n和突变体e226k的重组质粒核酸电泳图。m:1kbdnaladder;泳道1:algl-t157n重组质粒;泳道2:e226k重组质粒。
图3截短酶algl-t157n和突变体e226k的诱导表达和纯化的sds-page分析。m:蛋白分子量标准品(180kda蛋白marker);泳道1:未诱导重组菌上清液;泳道2:iptg诱导后的algl-t157n上清液;泳道3:iptg诱导后的e226k上清液;泳道4:纯化后的algl-t157n目的蛋白;泳道5:纯化后的e226k目的蛋白。
图4截短酶algl-t157n和突变体e226k的最适反应ph。
图5截短酶algl-t157n和突变体e226k的最适反应温度。
图6截短酶algl-t157n和突变体e226k的ph稳定性。
图7截短酶algl-t157n和突变体e226k的温度稳定性。
图8酶解产物的电喷雾质谱分析图。
图9酸解产物的电喷雾质谱分析图。
图10褐藻酸钠、酶解褐藻寡糖产物、酸解褐藻寡糖产物对羟自由基清除作用比较。
图11褐藻酸钠、酶解褐藻寡糖产物、酸解褐藻寡糖产物对abts自由基清除作用比较。
图12褐藻酸钠、酶解褐藻寡糖产物、酸解褐藻寡糖产物还原力比较。
具体实施方式
实施例1褐藻胶裂解酶突变体e226k的构建
本发明中的褐藻胶裂解酶基因algl(核苷酸序列如seqidno.1所示)来源于分离自舟山群岛海水的pseudoalteromonassp.zb7-4,全长1203bp,编码400个氨基酸,第1-31位氨基酸为信号肽,32-131位氨基酸为碳水化合物结合结构域(即cbm结构域),197-385位氨基酸为催化结构域。在157-158位氨基酸之间截断,截除第1-157位氨基酸,得到褐藻胶裂解酶截短酶algl-t157n,序列如seqidno.3所示。
通过序列比对,发现algl-t157n与4q8k在催化结构域部位有相同的氨基酸序列,因此在4q8k蛋白结构和催化机理研究基础上结合对接结果(图1),利用discoverystudio2016软件对algl-t157n催化腔附近8å内的氨基酸进行虚拟丙氨酸扫描和饱和突变,确定最佳突变氨基酸,最终筛选得到了突变体e226k。
设计含有突变位点的引物,以重组质粒pet-22b(+)-algl-t157n为模板,采用pcr技术扩增突变重组质粒pet-22b(+)-e226k,转化至e.colibl21(de3)中,获得突变体重组工程菌。具体步骤如下:
以含有重组质粒pet-22b(+)-algl-t157n的实验室保存菌株e.colitop10为出发菌株,使用omega公司质粒试剂盒提取重组质粒。设计特异性突变引物,将褐藻胶裂解酶的第226位谷氨酸突变为赖氨酸,正反引物如下:
e226k-f:5’-aggttaaaaagagtttacgcgttgctatga-3’,
e226k-r:5’-cgcgtaaactctttttaaccttatactcatgac-3’。
划线字母代表突变氨基酸赖氨酸的密码子。以提取的pet-22b(+)-algl-t157n质粒为模板,采用pcr技术对algl-t157n基因进行定点突变,pcr扩增体系:5×transstartfastpfuflybuffer10μl,highpuredntps(2.5mmol/l)4μl,pcrstimulant(5×)5μl,mgso41μl,pet-22b(+)-e226k1μl,e226k-f(10µmol/l)2μl,e226k-r(10µmol/l)2μl,transstartfastpfuflydnapolymerase1μl,ddh2o补至50μl。
pcr反应参数:98℃预变性2min;98℃变性15s,55℃退火15s,72℃延伸2min,30个循环;72℃保温5min;4℃保存。取10ulpcr扩增产物用1%琼脂糖凝胶电泳检测(见图2)。往剩余的pcr产物中加入1µl的dmt酶,混合均匀,在37℃条件下保温1h,消化原始模板。取2μl消化产物加入到100μle.colitop10感受态细胞中,轻微吹打混匀后冰浴30min;42℃热激45-90s,于冰上放置2min;加入800µllb培养基,37℃、200r/min培养45-60min;6000×g离心2min,吸取80μl上清重悬菌体,涂布在lb(含100μg/mlamp+)平板上,37℃培养12h。挑取克隆子测序验证。
突变体e226k核苷酸序列如seqidno.5所示,相应氨基酸序列见seqidno.4所示。
实施例2截短酶algl-t157n和突变体e226k的诱导表达与纯化
提取重组质粒pet-22b(+)-algl-t157n和pet-22b(+)-e226k,将其分别转入e.colibl21(de3)感受态细胞中,挑取阳性重组子于5mllb(amp+)液体培养基中,37℃200r/min培养12h,再以1%接种量接种至25mllb(amp+)培养基中,37℃200r/min培养od600至0.6-0.8之间,加入iptg(终浓度0.2mmol/l)于20℃200r/min诱导24h,测定褐藻胶裂解酶活力。以同样接种后不加iptg的lb培养基作为空白对照。测得突变体e226k粗酶液酶活为7.14±0.09u/ml,较algl-t157n(3.38±0.11u/ml)提高了1.11倍。
褐藻胶裂解酶活力采用dns法测定。取0.1ml酶液加入到0.9ml0.3%海藻酸钠的底物中,50℃下保温15min,加入1.5mldns终止反应,煮沸5min显色,待冷却后测定od540。酶活力单位定义:在一定反应条件下,每min催化海藻酸钠产生1μmol还原糖所需的酶量作为一个酶活力单位u。
酶活力计算公式为:
式中:
u:酶活力,单位u/ml
△a:减去空白对照后的od540
n:稀释倍数
k:标准曲线斜率
v:酶液体积,ml
t:反应时间,min
194.14:葡萄糖醛酸的相对分子量
1000:质量换算倍数。
实施例3截短酶algl-t157n和突变体e226k的分离纯化
(1)deae阴离子柱纯化:使用akta纯化系统纯化蛋白,首先利用deaeff(hitraptm,5ml)预装柱对蛋白进行纯化,以5ml/min流速及浓度为50mmol/l、ph7.0的mcilvaine缓冲液平衡柱子,用0.22µm滤膜过滤粗酶液。将得到的滤液上载阴离子柱,再以2ml/min流速平衡柱子,待平衡4-5个柱体积后,用事先配制好的1mol/lnacl溶液(50mmol/l,ph7.0的mcilvaine缓冲液配制)梯度洗脱柱子,通过uv检测器检测蛋白,收集不同梯度洗脱下的蛋白峰,进行酶活检测,并进行sds-page检测蛋白纯度。
(2)镍柱纯化:将经过deae初步纯化的蛋白上载到镍柱(histraptm,5ml)进行纯化,使用nta0(20mmol/l磷酸盐缓冲液,0.5mol/lnacl,10%甘油)作为平衡缓冲液,并以nta0为溶解液配制500mmol/l咪唑,使用配制好的咪唑梯度洗脱镍柱,收集不同浓度的洗脱蛋白,测定酶活,并进行sds-page检测蛋白纯度。
(3)脱盐:采用脱盐柱(hitrapdesalting,5ml)对蛋白进行去咪唑,取1ml镍柱纯化后的酶液上载脱盐柱,以50mmol/l、ph7.0的mcilvaine缓冲液为平衡液,流速设置为2ml/min,收集蛋白峰,进行酶活检测,并进行sds-page电泳,得到单一条带蛋白(见图3)。纯化后测得截短酶algl-t157n和突变体e226k的比活力分别为77.81u/ml、157.93u/ml,经突变后比活提高了1.03倍。
实施例4截短酶algl-t157n和突变体e226k的酶学性质
最适反应ph测定:将截短酶algl-t157n和突变体e226k分别加入到由不同ph缓冲液(ph为4.0~7.0的mcilvaine缓冲液,ph为7.0~8.0的na2hpo4-nah2po4缓冲液,ph为8.0~11.0的gly-naoh缓冲液)配制的浓度为3%的海藻酸钠底物中进行反应,反应温度为50℃,缓冲液浓度为50mmol/l。采用dns法测定酶活,将吸光值最高的酶活力设定为100%,计算不同ph下的相对酶活力,测定其最适反应ph。结果如图4显示,两种酶的最适反应ph均为7.0。
最适反应温度测定:在最适反应ph条件下,分别测定在不同温度下algl-t157n和e226k的残余酶活力,将测定的最高酶活力设定为100%。结果如图5显示,algl-t157n的最适反应温度为50℃,e226k的最适反应温度为55℃。
ph稳定性测定:将algl-t157n和e226k以一定的比例与上述不同ph的缓冲液混合,于37℃下保温1h,测定其在最适反应温度与最适反应ph条件下的酶活力,将测定的最高酶活力定义为100%,计算不同ph下的残余酶活力。结果如图6所示,37℃下保温1h后,在ph6.0-8.0范围内algl-t157n和e226k的残余酶活力达90%以上,说明酶在该ph范围内具有较强的稳定性。
温度稳定性测定:在最适反应温度与最适反应ph条件下,分别测定algl-t157n和e226k在不同温度下保温30min的残余酶活力,分别以未进行处理的酶活力作为对照。结果如图7显示,两种酶在40℃以下稳定性较好,40℃以上酶活开始下降。
实施例5褐藻胶裂解酶的催化动力学参数测定
催化动力学参数测定的具体方法如下:
以50mmol/l的mcilvaine缓冲液分别配制不同终浓度(0.3、0.45、0.6、1、2、3mg/ml)的褐藻酸钠、聚甘露糖醛酸钠(polym)和聚古罗糖醛酸钠(polyg)底物。测定algl-t157n和e226k在45℃条件下的酶活力。采用lineweaver-burk法,以反应速率倒数为纵坐标,底物浓度倒数为横坐标作图,计算km、vmax、kcat以及kcat/km。
催化动力学参数测定结果见表1,与截短酶algl-t157n相比,突变体e226k对褐藻酸钠、polym和polyg这3种底物的km值分别下降了89.69%、88.14%、61.11%,说明突变导致酶的底物亲和力明显提高。通过催化常数kcat比较发现,突变后催化常数分别为algl-t157n的118.02%、114.62%、151.37%,3种底物的kcat/km值分别提高了10.22、8.59、2.97倍。
表1截短酶algl-t157n与突变体e226k的催化动力学参数
实施例6褐藻寡糖的制备及成分分析
褐藻寡糖的酶解制备。将褐藻酸钠用50mmol/l、ph7.0的mcilvaine缓冲液配制成浓度为1.2%的反应底物。40℃预热5min后,加入一定量(0.5u/ml)的酶液进行反应,酶解24h取出0.9ml酶解底物,加入0.1ml酶液于40℃条件下测定od235,待吸光值基本不变,底物完全酶解,沸水浴5min终止反应。用sevage法反复抽提除蛋白,离心得到上清液进行旋蒸浓缩,用无水乙醇沉淀寡糖,并将样品干燥备用,苯酚-硫酸法定量。
寡糖的酸解制备。配制浓度为1.5%的褐藻酸钠底物,用盐酸调ph至4.0,于120℃条件下酸解4h,酸解结束后冷却至室温,再调ph至7.0,离心去除沉淀,得到上清液旋蒸浓缩,用无水乙醇沉淀寡糖,并将样品干燥备用,采用苯酚-硫酸法定量。
esi-ms分析。对酶解产物和酸解产物分别进行质谱分析。质谱条件为离子化模式:esi-,高分辨全扫描模式:m/z100-800。酶解结果如图8所示,esi-ms检测出3种聚合度寡糖的存在,分别为单糖175.0327(m-1na+),二糖351.0565(m+h+-2na+)、373.0385(m-1na+),三糖527.0886(m+2h+-3na+)、549.0706(m+h+-2na+)、571.0525(m-1na+),为不饱和寡糖。酸解结果如图9所示,检测出2种聚合度的寡糖,分别为单糖193.0348(m-1na+),二糖369.0678(m+h+-2na+)、391.0500(m-1na+),为饱和寡糖。
实施例7褐藻寡糖生物活性比较
将褐藻酸钠、酶解得到的褐藻寡糖产物、酸解得到的褐藻寡糖产物进行抗氧化性(羟自由基清除作用、abts自由基清除作用、还原力)比较,结果(图10、11、12)显示,酶解得到的褐藻寡糖抗氧化性要强于酸解产物和未水解的褐藻酸钠多糖。可见,酶解法制备褐藻寡糖能更好地保留寡糖的生物活性,这可能是由于酶解得到的不饱和寡糖具有更高的生物活性。
以上所述仅为本发明的较佳实施例,凡依本发明申请专利范围所做的均等变化与修饰,皆应属本发明的涵盖范围。
sequencelisting
<110>福州大学
<120>一种褐藻胶裂解酶突变体及其应用
<130>7
<160>7
<170>patentinversion3.3
<210>1
<211>1203
<212>dna
<213>人工序列
<400>1
atgttcaggtttaaaggaataaggataatgattaaccataaaaaactgtttttttacagc60
gcaattgcgacaagttcagcgctatctcatgctgcaacaattaataatgcaggctttgaa120
agtggctttagtaactggaacgaaaccgacccagccgctatttcttcagatgcttacagt180
ggctcaaaatcgttaaaaattcagggcagtccagcacgggtttatcaagtggtagatata240
cagcctaacactgaatacaccctaagtgcttatgtgttgggtaaagggcaaattggtgta300
aacgatttaaatggtttatttaaaaaccaaacctttaatgtttcttcgtggactaaagta360
acaaaaacatttacctcagcaaacaccaattcacttcaggtttttgctaaacattacaac420
aacaccagcgatgtaaggtttgataattttgccttggttgagggcagcggcagtaatgat480
ggtggctcagatggcggcagcgataactcaaatggttcaacaattcctagcagcataacc540
agtggtagcatttttgatttagaaggtgataacccaaatcctctcgttgacgatagcacc600
ttagtgtttgtgccgttagaggcacaacatattacgcctaatggtaatggctggcgtcat660
gagtataaggttaaagaaagtttacgcgttgctatgactcaaacctatgaagtgttcgaa720
gctacggtaaaagttgagatgtctgatggcggaaaaacaattatatcgcagcaccatgct780
agcgataccggcactatatctaaagtgtatgtgtcggatactgatgaatcgggctttaat840
gatagcgtagcgaacaacggaatttttgatgtgtacgtacgtttacgtaataccagcggt900
aatgaagaaaaatttgctttgggtacaatgaccagcggtgagacatttaacttgcgggta960
gttaataactacggcgatgtagaggttacggcattcggtaactcgttcggtataccagta1020
gaggatgattcgcagtcatactttaagtttggtaactacctgcaatcgcaagacccatac1080
acattagataaatgtggtgaggccggaaactctaactcgtttaaaaactgttttgaggat1140
ttaggcattacagagtcaaaagtgacgatgaccaatgtgagttatacgcgtgaaactaat1200
taa1203
<210>2
<211>400
<212>prt
<213>人工序列
<400>2
metpheargphelysglyileargilemetileasnhislyslysleu
151015
phephetyrseralailealathrserseralaleuserhisalaala
202530
thrileasnasnalaglyphegluserglypheserasntrpasnglu
354045
thraspproalaalaileserseraspalatyrserglyserlysser
505560
leulysileglnglyserproalaargvaltyrglnvalvalaspile
65707580
glnproasnthrglutyrthrleuseralatyrvalleuglylysgly
859095
glnileglyvalasnaspleuasnglyleuphelysasnglnthrphe
100105110
asnvalsersertrpthrlysvalthrlysthrphethrseralaasn
115120125
thrasnserleuglnvalphealalyshistyrasnasnthrserasp
130135140
valargpheaspasnphealaleuvalgluglyserglyserasnasp
145150155160
glyglyseraspglyglyseraspasnserasnglyserthrilepro
165170175
serserilethrserglyserilepheaspleugluglyaspasnpro
180185190
asnproleuvalaspaspserthrleuvalphevalproleugluala
195200205
glnhisilethrproasnglyasnglytrparghisglutyrlysval
210215220
lysgluserleuargvalalametthrglnthrtyrgluvalpheglu
225230235240
alathrvallysvalglumetseraspglyglylysthrileileser
245250255
glnhishisalaseraspthrglythrileserlysvaltyrvalser
260265270
aspthraspgluserglypheasnaspservalalaasnasnglyile
275280285
pheaspvaltyrvalargleuargasnthrserglyasngluglulys
290295300
phealaleuglythrmetthrserglygluthrpheasnleuargval
305310315320
valasnasntyrglyaspvalgluvalthralapheglyasnserphe
325330335
glyileprovalgluaspaspserglnsertyrphelyspheglyasn
340345350
tyrleuglnserglnaspprotyrthrleuasplyscysglygluala
355360365
glyasnserasnserphelysasncysphegluaspleuglyilethr
370375380
gluserlysvalthrmetthrasnvalsertyrthrarggluthrasn
385390395400
<210>3
<211>243
<212>prt
<213>人工序列
<400>3
serasnaspglyglyseraspglyglyseraspasnserasnglyser
151015
thrileproserserilethrserglyserilepheaspleuglugly
202530
aspasnproasnproleuvalaspaspserthrleuvalphevalpro
354045
leuglualaglnhisilethrproasnglyasnglytrparghisglu
505560
tyrlysvallysgluserleuargvalalametthrglnthrtyrglu
65707580
valpheglualathrvallysvalglumetseraspglyglylysthr
859095
ileileserglnhishisalaseraspthrglythrileserlysval
100105110
tyrvalseraspthraspgluserglypheasnaspservalalaasn
115120125
asnglyilepheaspvaltyrvalargleuargasnthrserglyasn
130135140
gluglulysphealaleuglythrmetthrserglygluthrpheasn
145150155160
leuargvalvalasnasntyrglyaspvalgluvalthralaphegly
165170175
asnserpheglyileprovalgluaspaspserglnsertyrphelys
180185190
pheglyasntyrleuglnserglnaspprotyrthrleuasplyscys
195200205
glyglualaglyasnserasnserphelysasncysphegluaspleu
210215220
glyilethrgluserlysvalthrmetthrasnvalsertyrthrarg
225230235240
gluthrasn
<210>4
<211>243
<212>prt
<213>人工序列
<400>4
serasnaspglyglyseraspglyglyseraspasnserasnglyser
151015
thrileproserserilethrserglyserilepheaspleuglugly
202530
aspasnproasnproleuvalaspaspserthrleuvalphevalpro
354045
leuglualaglnhisilethrproasnglyasnglytrparghisglu
505560
tyrlysvallyslysserleuargvalalametthrglnthrtyrglu
65707580
valpheglualathrvallysvalglumetseraspglyglylysthr
859095
ileileserglnhishisalaseraspthrglythrileserlysval
100105110
tyrvalseraspthraspgluserglypheasnaspservalalaasn
115120125
asnglyilepheaspvaltyrvalargleuargasnthrserglyasn
130135140
gluglulysphealaleuglythrmetthrserglygluthrpheasn
145150155160
leuargvalvalasnasntyrglyaspvalgluvalthralaphegly
165170175
asnserpheglyileprovalgluaspaspserglnsertyrphelys
180185190
pheglyasntyrleuglnserglnaspprotyrthrleuasplyscys
195200205
glyglualaglyasnserasnserphelysasncysphegluaspleu
210215220
glyilethrgluserlysvalthrmetthrasnvalsertyrthrarg
225230235240
gluthrasn
<210>5
<211>732
<212>dna
<213>人工序列
<400>5
agtaatgatggtggctcagatggcggcagcgataactcaaatggttcaacaattcctagc60
agcataaccagtggtagcatttttgatttagaaggtgataacccaaatcctctcgttgac120
gatagcaccttagtgtttgtgccgttagaggcacaacatattacgcctaatggtaatggc180
tggcgtcatgagtataaggttaaaaagagtttacgcgttgctatgactcaaacctatgaa240
gtgttcgaagctacggtaaaagttgagatgtctgatggcggaaaaacaattatatcgcag300
caccatgctagcgataccggcactatatctaaagtgtatgtgtcggatactgatgaatcg360
ggctttaatgatagcgtagcgaacaacggaatttttgatgtgtacgtacgtttacgtaat420
accagcggtaatgaagaaaaatttgctttgggtacaatgaccagcggtgagacatttaac480
ttgcgggtagttaataactacggcgatgtagaggttacggcattcggtaactcgttcggt540
ataccagtagaggatgattcgcagtcatactttaagtttggtaactacctgcaatcgcaa600
gacccatacacattagataaatgtggtgaggccggaaactctaactcgtttaaaaactgt660
tttgaggatttaggcattacagagtcaaaagtgacgatgaccaatgtgagttatacgcgt720
gaaactaattaa732
<210>6
<211>30
<212>dna
<213>人工序列
<400>6
aggttaaaaagagtttacgcgttgctatga30
<210>7
<211>33
<212>dna
<213>人工序列
<400>7
cgcgtaaactctttttaaccttatactcatgac33
- 还没有人留言评论。精彩留言会获得点赞!