一种调控柽柳耐盐性的关键基因TcNAC1及其应用的制作方法
本发明属于植物基因工程技术领域,具体涉及一种调控柽柳耐盐性的关键基因tcnac1及其应用。
背景技术:
据联合国粮农组织统计,陆地总面积的6.5%为无法耕种的盐碱地,我国盐碱地面积约20多万平方公里,盐碱地产生高盐胁迫,直接影响作物产量,通过抗逆育种手段充分利用盐碱地,对增加作物产量、解决粮食危机意义重大。尽管在在拟南芥、水稻等模式物种中已经阐明了一些普遍的耐盐机制,但很多耐盐基因研究表明,目前应用这些基因的转基因植物耐盐性提高的效果并不理想,不能满足在实际生产中的需求,难以推广(zhu,2002)。相比草本植物和禾本科作物,耐盐树种具有独特的高度耐盐的优良特性,可以在高度盐碱化的盐渍化土壤、滩涂上正常生长并完成整个生活史,目前仅仅在生理层面解析了相关机制,如泌盐、离子区域化等特有机制,但是其分子层面了解较少,迫切需要阐明其高耐盐调控机理,为获取高耐盐性的转基因植株提供有效的理论指导。
中国柽柳(tamarixchinenseslour.)作为耐盐性最强的树种之一,是研究植物耐盐机制的良好材料。柽柳是我国乡土树种,在维持黄河三角洲等海滨湿地盐渍化地区的生态稳定中发挥了重要作用重,也是我国沿海防护林构建的重要资源(李艳华等,2000;赵明范等,1997)。柽柳具有独特的耐高盐胁迫特有机制,挖掘其耐盐性调控关键基因,对植物耐盐机理研究意义重大,可以为盐渍地脱盐、抗逆植物育种提供理论基础,目前在柽柳属的其他物种中已经克隆得到了几十个相关的抗逆基因,为植物耐盐育种提供了重要分子工具,但在我国分布面积最广的柽柳中尚未有耐盐调控基因的报道,急需开展其耐盐调控基因的分离鉴定。
nac是成员最多的转录因子家族之一。nac家族在拟南芥中有138个成员,粳稻中有170个成员,杨树中有289个成员,在高等植物中广泛分布(牟桂萍等,2013)。nac转录因子的命名源于nam(noapicalmeristem,‘无顶端分生组织’)、atafl/2(arabidopsisthalianaactivatingfactor;‘拟南芥激活因子’)以及cuc2(cup-shapedcotyledon;‘杯状子叶’)三类基因的缩写。nam最早发现于矮牵牛(petuniahybrida),ataf和cuc来自拟南芥(arabidopsisthaliana)(彭辉等,2010)。nac包含保守的n端和高度可变的c端,n端含有约160个氨基酸残基组成nac结构域,c端有转录激活作用,nac大多以同源或异源二聚体形式发挥功能,c端序列影响寡聚化作用。nac家族广泛参与植物的生物胁迫响应、非生物胁迫响应、器官发育、次生生长、果实成熟等生物过程相关调控,功能广泛,但相对于数量巨大的nac家族成员,对其功能研究仍然不足(olsenetal.,2005)。
拟南芥中5个nac参与植株的营养器官的形成、分生组织边界确立、侧根发生和衰老,cucl/2双突变体,相比野生型萼片变形和花瓣较少,超表达cuc1和cuc2,植株分别出现叶片发育畸形和器官边缘扩张的表型,表明nac控制着着组织发育中边界相关的功能。非生物胁迫方面,水稻中nac作为mir164的靶基因,负调控植株抗旱性;在胡杨中,三个nac表达模式有不一致甚至相反的情况,相对矛盾的胁迫相关性暗示着nac在调控胡杨非生物胁迫方面存在复杂的调控模式。拟南芥atnac4(anac080)最初报道为影响侧根发生和叶片衰老进程,近年研究发现和n、p等营养元素胁迫密切相关(leeetal.,2016)且含有保守的mir164响应元件,暗示着atnac4可能在受到转录后调控水平影响胁迫响应;拟南芥anac019、anac055受到aba和干旱诱导(buetal.,2009),提高了超表达植株的抗旱能力,刚毛柽柳thnac13异源表达提高了拟南芥耐盐性和渗透胁迫耐受能力(wang,li,luandwang,2017),刚毛柽柳thnac7的过表达类似thnac13提高了植株的耐盐性(张明意,2015)。
由上述研究结果可以看出,nac在调控植物非生物胁迫方面存在复杂的调控模式,一些nac转录因子被鉴定为耐盐调控的关键因子。目前,尚未有中国柽柳的nac转录因子相关报道,克隆和开发利用中国柽柳nac转录因子,不仅有助于阐明柽柳高度耐盐的分子机制,而且可以为筛选重要耐盐基因和抗逆遗传育种提供理论基础和分子工具,对盐碱地利用和农业综合生产能力提高具有重要应用价值。
技术实现要素:
发明目的:针对现有技术中存在的不足,本发明的目的是提供一种柽柳基因tcnac1,是调控柽柳耐盐性的关键基因。本发明的另一目的是提供一种上述调控柽柳耐盐性的关键基因tcnac1的应用。
技术方案:为了实现上述发明目的,本发明采用的技术方案如下:
一种调控柽柳耐盐性的关键基因tcnac1,其核苷酸序列如seqidno.1所示。
所述调控柽柳耐盐性的关键基因tcnac1的表达蛋白,其氨基酸序列如seqidno.2所示。
含有所述调控柽柳耐盐性的关键基因tcnac1的载体。
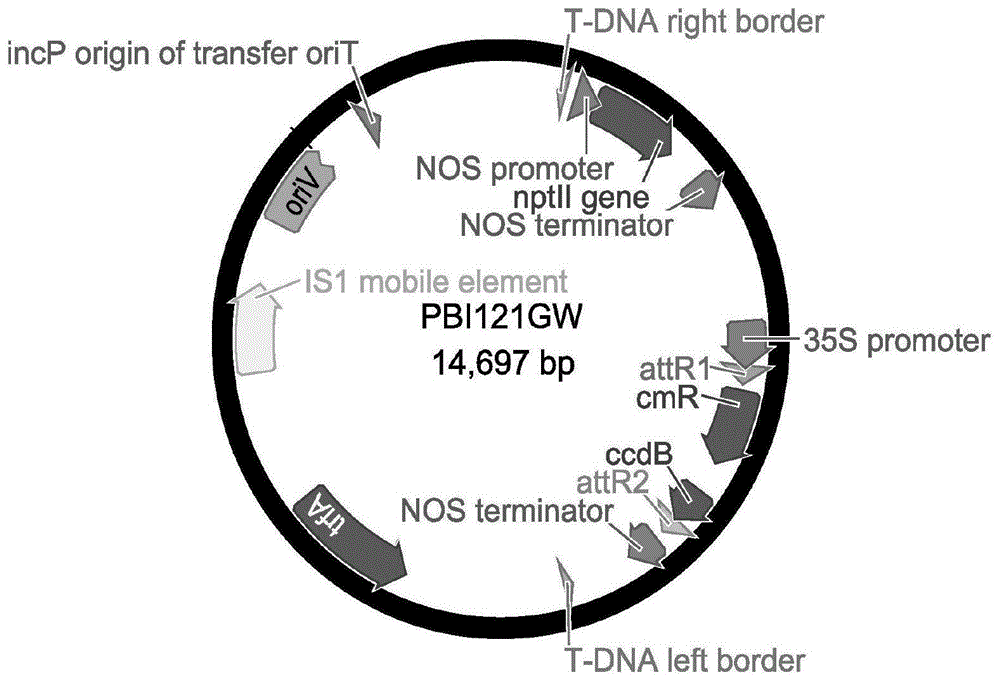
采用通路克隆技术构建植物超表达载体pbi121gw-tcnac1,组装通路克隆attr1和attr2元件,快速组装tcnac1基因表达框并确保准确翻译;组装lb和rb序列,促使组装于其间的tcnac1基因和筛选标记基因nptii整合至侵染的拟南芥染色体中;基因tcnac1位于启动子p35s之后,在启动子p35s的驱动下,tcnac1可在拟南芥体内高效表达,从而调控耐盐性;在tcnac1基因的5′端组装组成型强表达启动子p35s,使tcnac1基因在植物体内高效表达;在tcnac1基因的3′端组装了强终止子nos,可有效终止tcnac1基因的转录。
含有所述调控柽柳耐盐性的关键基因tcnac1的宿主细胞。
所述调控柽柳耐盐性的关键基因tcnac1在植物耐盐性育种中的应用。
有益效果:与现有技术相比,本申请的优点在于:本申请公开了一种调控柽柳盐耐性的关键基因tcnac1,将tcnac1基因转入拟南芥,超表达tcnac1基因的拟南芥植株耐盐性显著下降,其种子的耐盐萌发率显著下降,说明tcnac1基因是调控柽柳耐盐性的关键调节因子,可用于土壤盐度指示植物的育种,为柽柳耐盐性的基因表达调控分子机制提供理论研究依据和基因序列,在林木基因工程和耐盐性育种领域有重要应用价值。
附图说明
图1是植物超表达载体pbi121gw的结构示意图;
图2是10株成功转化的转基因拟南芥的tcnac1插入片段的pcr检测的1%琼脂糖凝胶电泳图;
图3是超表达tcnac1基因的拟南芥和野生型拟南芥形态比较图:1为转基因拟南芥0.3%nacl培养基生长28d,2为转基因拟南芥0.3%nacl培养基生长14天后转入无nacl的ms培养基脱盐处理14天,3为野生型0.3%nacl培养基生长28d,4为野生型0.3%nacl培养基生长14天后转入无nacl的ms培养基脱盐处理14天;
图4是超表达tcnac1基因的拟南芥和野生型拟南芥种子萌发率比较图,右为野生型,左为超表达tcnac1基因的拟南芥。
具体实施方式
下面结合具体实施例对本发明进一步进行描述。以下实施例中未作具体说明的分子生物学实验方法,均可参照《分子克隆实验指南》(第三版)j.萨姆布鲁克一书中所列的方法或本领域的常规方法进行,或者按照试剂盒和产品说明书进行。
下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
实施例1:通过race技术克隆tcnac1基因
基于柽柳rna-seq数据的tcnac1序列,设计5′和3′的race引物,通过巢式pcr扩增两段特异性产物,通过t-载体克隆测序,测序结果通过重叠区拼接,得到cnda全长。
具体如下:
i.引物设计
3′race正向引物为:
outerprimerf:5′-tcaacgccaacagcaatcactacat-3′;
innerprimerf:5′-cggcgttgaactcattctatgg-3′;
3′race反向引物为:
outerprimerr:
5′-ctaatacgactcactatagggcaagcagtggtatcaacgcagagt-3′;
innerprimerr:5′-ctaatacgactcactatagggc-3′;
5′race正向引物为
outerprimerf:
5′-ctaatacgactcactatagggcaagcagtggtatcaacgcagagt-3′;
innerprimerf:5′-ctaatacgactcactatagggc-3′;
5′race反向引物为:
outerprimerr:5′-agtaggtagacttggcttcgttgt-3′;
innerprimerr:5′-tccgctcctgactccacttact-3′;
ii.3′race反应过程:
(1)反转录,将以下成分加入到一个置于冰上的rnase-free的小离心管中:1μgtotalrna(植物材料为柽柳叶片),4μldntpmix,2μl3′raceadapter,2μl10xrtbuffer,1μlrnaseinhibitor,1μlm-mlvreversetranscriptase,nuclease-freewater补至20μl。
(2)轻轻混匀,短暂离心,42℃温育1小时,进入pcr步骤;
(3)3′race巢式pcr;
3′raceouterpcr反应体系(50μl)组成:5.0μl10×lapcrbuffer(mg2+free),5.0μlmgcl2(25mm),8.0μldntpmixture(each2.5mm),2.0μl3′raceouterprimerf(10μm),2.0μl3′outerprimerr(10μm),1μlrtreactionproduct,0.5μltakaralataq(5u/μl),26.5μlnuclease-freewater。
3′raceinnerpcr反应体系(50μl)组成:5.0μl10×lapcrbuffer(mg2+free),5.0μlmgcl2(25mm),8.0μldntpmixture(each2.5mm),2.0μl3′raceinnerprimerf(10μm),2.0μl3′raceinnerprimerr(10μm),1μlouter3′racepcrproduct,0.5μltakaralataq(5u/μl),26.5μlnuclease-freewater。
反应程序:94℃3分钟;94℃30秒,60℃30秒,72℃1分钟,35cycles;72℃7分钟。
(4)纯化片段的连接反应
采用takara公司的pmd19-tsimplevetor克隆目的dna分子,反应体系(5μl):2.2μl纯化回收的pcr产物,0.3μlpmd-19simplevector,2.5μlsolutioni。反应条件:16℃30分钟;4℃过夜。
(5)大肠杆菌转化:将新鲜制备或-70℃冻存的大肠杆菌top10感受态细胞在冰上融化;取5μl纯化片段与克隆载体的连接产物,加入到100μl感受态细胞中,并轻轻混匀,冰浴30分钟;42℃水浴中热击90秒,迅速置于冰上3-5分钟;加入800μllb液体培养基,37℃100转/分钟摇菌1小时;4000转/分钟离心3分钟,吸掉上层800μl培养基,混匀剩余菌液;将菌液涂抹于含有amp的lb筛选培养板上,37℃倒置培养过夜。
(6)阳性克隆筛选及测序分析
从筛选培养板上挑选单菌落接种于lb液体培养基中,37℃250转/分钟摇菌过夜;直接以培养过夜的菌液为模板进行重组转化子的pcr检测。
反应体系(20μl):2.0μl10×pcrbuffer(mg2+free),1.5μlmgcl2(25mm),1.3μldntpmixture(each2.5mm),1.0μl3′racegenespecificinnerprimer(10μm),1.0μl3′raceinnerprimer(10μm),0.1μl菌液,1.0μlrtaq,12.1μlmilli-qwater。反应程序:94℃3分钟;94℃30秒,60℃30秒,72℃1分钟,28cycles;72℃7分钟。
阳性克隆通过sanger测序法测序得到碱基序列。
iii.5′race反应过程
(1)rna处理:cip反应,将如下成分加入到rnase-free的小离心管中:10μgtotalrna,2μl10xcipbuffer,2μlcalfintestinealkalinephosphatase(cip),nuclease-freewater至20μl。
(2)轻轻混匀,短暂离心;37℃温育1小时;
(3)加入以下试剂到cip反应离心管:15μlammoniumacetatesolution,115μlnuclease-freewater,150μlacidphenol:chloroform。
(4)充分涡旋,室温高速离心(≥10000g)5分钟;
(5)转移上层水相到一个新的离心管中,加入150μl氯仿,充分涡旋,室温高速离心(≥10000g)5分钟;
(6)转移上层水相到一个新的离心管中,加入150μl异丙醇,充分涡旋,冰浴10分钟;
(7)最大转速离心20分钟,用0.5ml预冷的70%乙醇冲洗沉淀,最大转速离心5分钟,小心弃乙醇,气干沉淀;
(8)以11μlnuclease-freewater重悬沉淀,即得cip’rna,冰上放置进一步用于tap反应,或者-20℃保存;
(9)tap反应,把以下成分加入到一个rnase-free的小离心管中:5μlcip’drna,1μl10xtapbuffer,2μltobaccoacidpyrophosphatase(tap),2μlnuclease-freewater:
(10)轻轻混匀,短暂离心,37℃温育1小时,即得cip/tap-treatedrna;进入接头连接步骤,或-20℃保存反应物;
(11)5′race接头连接,把以下成分加入到一个rnase-free的小离心管中:2μlcip/tap-treatedrna,1μl5′raceadapter,1μl10×rnaligasebuffer,2μlt4rnaligase(2.5u/μl),4μlnuclease-freewater。
(12)轻轻混匀,短暂离心,37℃温育1小时,即得ligatedrna;进入反转录步骤,或-20℃保存反应物。
(13)将以下成分加入到一个置于冰上的rnase-free的小离心管中:2μlligatedrna,4μldntpmix,2μlrandomdecamers,2μl10xrtbuffer,1μlrnaseinhibitor,1μlm-mlvreversetranscriptase,nuclease-freewater补至20μl。
(14)轻轻混匀,短暂离心;42℃温育1小时,即得rtreaction;进入pcr步骤,或-20℃保存反应物。
(15)5′race巢式pcr:反应体系、反应条件与3′race的巢式pcr一致。
(16)pcr产物克隆测序,操作与3′race克隆一致。
iv.orf扩增
对3′race和5′race序列进行拼接,并利用ncbi-orffinder工具预测其阅读框。根据基因全长序列设计引物(扩增子包含起始密码子及终止密码子),进行tcnac1基因的全长克隆。其中,tcnac1orf正向引物:5′-atggaaaacattcctggattt-3′,tcnac1orf反向引物:5′-ttaatagtaaccccaaaggtc-3′,高保真pcr反应体系如下:10×lapcrbuffer5.0μl;2.5mmdntpmixture8.0μl;25mmmg2+5.0μl;lataqdnapolymerase(5u/μl)0.5μl;正向引物(10μm)2μl;反向引物(10μm)2μl;模板(柽柳cdna)1μl;加无菌ddh2o补足50μl。反应程序:预变性94℃3分钟-(94℃40秒-55℃30秒-72℃30秒)×35个循环-72℃10分钟。
tcnac1全长cdna序列为2033bp,其序列如seqidno.1所示,包含一个1164bp的完整阅读框,对应的tcnac1蛋白的氨基酸序列为387aa,其序列如seqidno.2所示。
实施例2:tcnac1基因植物表达载体构建
利用通路克隆技术构建tcnac1基因的过量表达载体。使用特异pcr引物(实施例1的tcnac1orf引物),以cdna为模板,进行pcr扩增,将tcnac1基因orf构建到入门载体。入门载体为pcr8/gw/topotmvector(invitrogen)。反应体系为:freshpcrproduct(purified)10-20ng;saltsolution1μl;pcr8/gw/topotmvector1μl;加无菌ddh2o补足6μl。反应程序为:室温静置30分钟。
从筛选培养板上挑取阳性克隆进行测序验证,阳性入门载体与植物表达载体pbi121gw进行lr反应。载体质粒如图1所示。反应体系为:入门载体100ng;pbi121gwvector(100ng/μl)1.5μl;lrclonaseiienzymemix2μl;加te(ph8.0)补足10μl;。反应条件:25℃1小时。经lr反应后,tcnac1基因导入植物表达载体pbi121gw中。pbi121gw组装有通路克隆attr1和attr2元件,快速组装tcnac1基因表达框并确保准确翻译;组装有lb和rb序列,促使组装于其间的tcnac1基因和筛选标记基因nptii整合至侵染的植物染色体中。另外,在tcnac1基因的5′端组装组成型强表达启动子p35s,它能使tcnac1高效表达。通过pcr检测及测序验证,确认过量表达载体构建成功,命名为pbi121gw-tcnac1,该基因位于启动子p35s之后,在启动子p35s的驱动下,tcnac1可在植物体内高效表达。
实施例3:tcnac1基因的遗传转化
通过液氮冻融法将所构建的pbi121gw-tcnac1过量表达载体转入农杆菌菌株eha105,通过农杆菌的花絮侵染法将tcnac1基因转入拟南芥。得到的阳性拟南芥种子,在含有0.3%nacl(51mm)的ms固体培养基上进行发芽率测定和植株表型观察测。图2为10株成功转化的转基因拟南芥的tcnac1插入片段的pcr检测;图3为超表达tcnac1的转基因拟南芥与野生型整体形态比较,生长基质为含有0.3%nacl(51mm)的ms固体培养基,1为转基因拟南芥0.3%nacl培养基生长28d,2为转基因拟南芥0.3%nacl培养基生长14天后转入无nacl的ms培养基脱盐处理14天,3为野生型0.3%nacl培养基生长28d,4为野生型0.3%nacl培养基生长14天后转入无nacl的ms培养基脱盐处理14天;图4是超表达tcnac1的转基因拟南芥和野生型拟南芥种子萌发率比较图,培养基质为含有0.3%nacl(51mm)的ms固体培养基,右为野生型种子,左为超表达tcnac1基因的拟南芥种子。从结果可以明显得出,过量表达tcnac1基因使得拟南芥出现盐敏感表型,转基因拟南芥耐盐性显著下降,说明tcnac1基因是调控植物耐盐性的关键因子,tcnac1及其过量表达技术可以作为高效的分子工具加速植物耐盐性育种的进程。
以上说明对本发明而言只是说明性的,而非限制性的,本领域普通技术人员理解,在不脱离所附权利要求所限定的精神和范围的情况下,可做出许多修改、变化或等效,但都将落入本发明的保护范围。
序列表
<110>南京林业大学
<120>一种调控柽柳耐盐性的关键基因tcnac1及其应用
<130>100
<160>12
<170>siposequencelisting1.0
<210>1
<211>2033
<212>dna
<213>tamarixchinenseslour.
<400>1
gattgcagttttttctttttcttgtttgaaagctctgtttttcgtttccctccttttgag60
ctgctcgtcttcttcaagatcacaactttattaatagcgcgtcggtttctttccttcttt120
ctcttagttcattaagccatactctttacagtttttcctcatccatatccaaatccaatt180
ccccatccctgcccctatctctctccatctctctctatatattgtatagaactcccctcc240
ctttcaactcatttctacaattccatagcacccacatccctagttagcagtattggcccg300
gaacacagaagactagattgagctggttagattggataggactagtaagtggagtcagga360
gcggataggaagtataagaaatggaaaacattcctggatttgttggtggttgtggaggtg420
atcaggaagaacgaatggaattaccacctggattccgattccatcccacagatgaagagc480
tcatcactcactacctttctcctaaagtagttgataacagcttctctgctagagctatcg540
gcgaggttgatttgaacaagtctgaaccatgggaattgcgtgaacgggcgaagatgggag600
aaaaggaatggtatttcttctgtgtcagagaccggaaatacccaactggtttaagaacga660
acagggcgacagaagctggttattggaaagctactgggaaggacaaagaaattcgtagag720
gaaaaccacttgttggtatgaagaaaactctggttttctacaagggtcgagctccaaagg780
gggagaaatcaaattgggtcatgcatgaatatagactggagggtaaattatccctgcaaa840
atcttcctaattcagctaagcagaacgagtgggtgatatgccgggtctttcagaagagtg900
caggatgtaagaagattcaatacccaggtttaatccgatccgaatcatcggaattcggtc960
cttcgtcaggtttaccaccattgacggactcttcaccttactttgacccccaacaaacga1020
agccaagtctacctactgattcccttcacgtgtcctgcttctccaaccccaccatcatgt1080
ctaatcctcaatcaaacatcaacgccaacagcaatcactacatgttcgattcctacgaaa1140
atagtaacacgattagtcccttcatgcccggttatcatgccaaattaggaggaggaggag1200
gaccgacgacgtcggcgttgaactcattctatggatctgaattcaatccgaatccgttgt1260
cgtacccgggagcaggtggaggaggatacgtgtcggagcagtcaatcttaaggtctatcc1320
tggagaggaacggaacggagggcgtgatgaaggcggagagccatatgatctctgtttctc1380
aagacaccgttttgtcaggtgatatgaatgctgaaatctcatctgttgtctcaaattatg1440
agatgggaggtaggagcgcatttgatgatcaacctcagcatccttctgttgctgctgctg1500
ctgctggacctgtggatgtgggagacctttggggttactattaattgaatctgaatctga1560
atctgaatctgaatctacgaggatgatgcagcagcagaagagattagctaggtgagctca1620
tatagactgataaaaatcattggaagaagaagaagaagaagaagctgtttagcgtcaata1680
cctatgttagtatattggtaagtgaagtcacttgtggtaattgatatagtttgtgtatag1740
tatttctctgtatcagaaggtgggggtgggggaacaggaaaaagaaagaaggaagaagag1800
ggtacacatacaattatgtaacatataatccattatagtatagtatcagcttcttctttt1860
atttttgtcatttttcgtttgcaaattatgggcgtagttttgttatcttggtagcactgg1920
taatatgtttagcggttgccatagtatatactatactgtcttttagttcttattaaaata1980
aaattgaatgtaaaatatttgatcaccaaaaaaaaaaaaaaaaaaaaaaaaaa2033
<210>2
<211>387
<212>prt
<213>tamarixchinenseslour.
<400>2
metgluasnileproglyphevalglyglycysglyglyaspglnglu
151015
gluargmetgluleuproproglypheargphehisprothraspglu
202530
gluleuilethrhistyrleuserprolysvalvalaspasnserphe
354045
seralaargalaileglygluvalaspleuasnlyssergluprotrp
505560
gluleuarggluargalalysmetglyglulysglutrptyrphephe
65707580
cysvalargasparglystyrprothrglyleuargthrasnargala
859095
thrglualaglytyrtrplysalathrglylysasplysgluilearg
100105110
argglylysproleuvalglymetlyslysthrleuvalphetyrlys
115120125
glyargalaprolysglyglulysserasntrpvalmethisglutyr
130135140
argleugluglylysleuserleuglnasnleuproasnseralalys
145150155160
glnasnglutrpvalilecysargvalpheglnlysseralaglycys
165170175
lyslysileglntyrproglyleuileargserglusersergluphe
180185190
glyproserserglyleuproproleuthraspserserprotyrphe
195200205
aspproglnglnthrlysproserleuprothraspserleuhisval
210215220
sercyspheserasnprothrilemetserasnproglnserasnile
225230235240
asnalaasnserasnhistyrmetpheaspsertyrgluasnserasn
245250255
thrileserprophemetproglytyrhisalalysleuglyglygly
260265270
glyglyprothrthrseralaleuasnserphetyrglysergluphe
275280285
asnproasnproleusertyrproglyalaglyglyglyglytyrval
290295300
sergluglnserileleuargserileleugluargasnglythrglu
305310315320
glyvalmetlysalagluserhismetileservalserglnaspthr
325330335
valleuserglyaspmetasnalagluileserservalvalserasn
340345350
tyrglumetglyglyargseralapheaspaspglnproglnhispro
355360365
servalalaalaalaalaalaglyprovalaspvalglyaspleutrp
370375380
glytyrtyr
385
<210>3
<211>25
<212>dna
<213>3'raceouterprimerf引物序列(artificial)
<400>3
tcaacgccaacagcaatcactacat25
<210>4
<211>22
<212>dna
<213>3'raceinnerprimerf引物序列(artificial)
<400>4
cggcgttgaactcattctatgg22
<210>5
<211>45
<212>dna
<213>3'raceouterprimerr引物序列(artificial)
<400>5
ctaatacgactcactatagggcaagcagtggtatcaacgcagagt45
<210>6
<211>22
<212>dna
<213>3'raceinnerprimerr引物序列(artificial)
<400>6
ctaatacgactcactatagggc22
<210>7
<211>45
<212>dna
<213>5'raceouterprimerf引物序列(artificial)
<400>7
ctaatacgactcactatagggcaagcagtggtatcaacgcagagt45
<210>8
<211>22
<212>dna
<213>5'raceinnerprimerf引物序列(artificial)
<400>8
ctaatacgactcactatagggc22
<210>9
<211>25
<212>dna
<213>5'raceouterprimerr引物序列(artificial)
<400>9
agtaggtagacttggcttcgtttgt25
<210>10
<211>22
<212>dna
<213>5'raceinnerprimerr引物序列(artificial)
<400>10
tccgctcctgactccacttact22
<210>11
<211>21
<212>dna
<213>tcnac1orf正向引物(artificial)
<400>11
atggaaaacattcctggattt21
<210>12
<211>21
<212>dna
<213>tcnac1orf反向引物(artificial)
<400>12
ttaatagtaaccccaaaggtc21
- 还没有人留言评论。精彩留言会获得点赞!