一种SIRT1基因敲除的IPEC-J2细胞系的构建方法与流程
本发明涉及基因敲除技术领域,尤其是一种sirt1基因敲除的ipec-j2细胞系的构建方法。
背景技术:
ipec-j2细胞是从新生仔猪中空肠中分离的猪肠柱状上皮细胞。当在0.4μm孔径滤器上培养时,该细胞系形成具有高跨上皮电阻的极化单层。与常见的人结肠衍生系ht-29,t84和caco-2相比,ipec-j2细胞的独特之处在于它来自猪小肠组织并且未被转化(与ipi-2i相比)。猪肠上皮细胞比啮齿类动物的肠上皮细胞系(例如iec-6或iec-18)更加接近的模拟人类肠上皮生理学,这在人畜共患病感染的研究中尤为重要。此外,ipec-j2细胞越来越多地用于肠道氧化应激研究,它们为研究断奶仔猪肠道健康提供了特异性模型,以检测各种外源抗氧化剂与肠上皮细胞之间的相互作用,被用作潜在饲用抗氧化剂的初始筛选工具。
sirt1(silentmatingtypeinformationregulation2homologue1)是哺乳动物中高度保守的烟酰胺腺嘌呤二核苷酸(nad+)依赖性蛋白脱乙酰基酶,在代谢调节、发育、肿瘤发生以及衰老等生理病理过程中至关重要。作为哺乳动物组织中主要的核sirtuin,sirt1是一种关键的代谢传感器,通过调节细胞应激反应、能量代谢、细胞凋亡、炎症等生物过程,以响应外界环境信号。在过去几年中,sirt1已被鉴定为多种组织/细胞中氧化应激和炎症的关键阻遏物。最近的一些研究表明sirt1在调节肠道炎症和组织氧化-还原稳态中起着关键作用。然而,sirt1是否刺激或抑制肠道应激仍存在争议,肠上皮sirt1如何调节复杂的环境-宿主相互作用以调节肠上皮完整性尚不清楚。
技术实现要素:
基于上述问题,本发明的目的在于克服上述现有技术的不足之处而提供一种构建sirt1基因敲除的ipec-j2细胞系的构建方法,采用该方法能筛选得到稳定的单克隆sirt1基因敲除ipec-j2纯合细胞系,可作为研究仔猪肠道氧化应激的细胞模型。
为实现上述目的,本发明采取的技术方案为:
一种sirt1基因敲除的ipec-j2细胞系的构建方法,包括如下步骤:
(1)针对sirt1基因的第一外显子设计并合成grna探针,grna的碱基序列如seqidno.1所示,其反向互补序列如seqidno.2所示;
(2)将如seqidno.1和2所示的dna链退火后形成反向互补的双链grna探针克隆到如图2所示的pspcas9-2a-puro(px459)v2.0载体中,构建同时表达如seqidno.1所示sgrna和cas9蛋白的质粒;
(3)将步骤(2)构建好的质粒转染到ipec-j2细胞;
(4)从步骤(3)转染后的ipec-j2细胞筛选单克隆突变细胞系,得到sirt1基因敲除的ipec-j2细胞系,其相比野生型细胞系,缺少了sirt1基因第一外显子中的5个碱基(5’-gcgg-3’),使sirt1蛋白在翻译过程中发生移码突变,提前终止翻译。需要说明的是,本发明的构建方法中难点在于,从众多grna位点中筛选起作用的grna,然后从突变的ipec-j2嵌合体细胞群中筛选出sirt1基因敲除的纯合细胞系,该过程经过反复试验,最后才构建成功本发明的相比野生型细胞系,缺少了sirt1基因第一外显子中的5个碱基(5’-gcggc-3’)的ipec-j2细胞系。
作为上述技术方案的进一步优化,所述步骤(3)中利用lipo3000(lipofectamine3000)进行转染。
作为上述技术方案的进一步优化,所述步骤(4)中,筛选单克隆突变细胞系的方法包括嘌呤毒素结合有限稀释法。
作为上述技术方案的进一步优化,所述嘌呤毒素的浓度为2μg/ml。
作为上述技术方案的进一步优化,所述嘌呤毒素的使用方法为:用含有嘌呤毒素的细胞培养基培养细胞,且每天更换新的含有嘌呤毒素的细胞培养基,一共培养7天。
作为上述技术方案的进一步优化,所述步骤(4)中,筛选单克隆突变细胞系的方法还包括高分辨率熔解曲线分析(hrma)。
作为上述技术方案的进一步优化,所述高分辨率熔解曲线分析的包括:
1)hrma扩增条件为:98℃2min;98℃15sec,60℃20sec(收集信号),进行40个循环;95℃10sec;
2)扩增结束后进行熔解曲线分析,从55℃至95℃,每10sec升高0.2℃,收集信号。
作为本发明的另一方面,本发明提供了采用上述方法构建的sirt1基因敲除的ipec-j2细胞系。
作为本发明的又一方面,本发明提供了一种sirt1基因敲除的ipec-j2细胞系,所述细胞系相比野生型ipec-j2细胞系,缺少了sirt1基因第一外显子中的5个碱基(gcggc)。
综上所述,本发明的有益效果为:
(1)首次利用crispr/cas9基因编辑技术构建sirt1基因敲除的ipec-j2细胞系,筛选得到稳定的单克隆sirt1基因敲除ipec-j2纯合细胞系,可作为研究仔猪肠道氧化应激的细胞模型;
(2)利用sirt1基因敲除的ipec-j2细胞系研究sirt1在肠上皮细胞中的作用,有望解析肠道应激反应的信号传导途径,为缓解仔猪断奶应激提供新的研究模型和思路。
附图说明
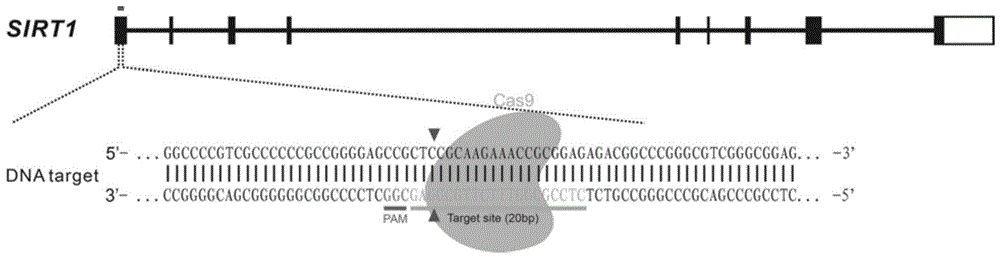
图1为猪sirt1基因组结构及crispr/cas9靶位点示意图;
图2为pspcas9-2a-puro(px459)v2.0载体图谱;
图3为px459(sirt1-sgrna)质粒测序图谱;
图4为sirt1野生型ipec-j2细胞系crispr靶位点测序峰图;
图5为sirt1突变型ipec-j2细胞系crispr靶位点测序峰图;
图6为ipec-j2细胞sirt1野生型与突变型单克隆细胞系序列比对图。
具体实施方式
本发明公开了一种利用crispr/cas9基因编辑技术结合嘌呤毒素筛选sirt1基因敲除单细胞ipec-j2纯合细胞系的构建方法,首先针对sirt1基因开放阅读框(orf)中第一外显子共设计若干个sirt1基因sgrna(singleguiderna)识别位点;根据识别位点设计合成grna探针序列,将grna探针克隆到pspcas9-2a-puro(px459)v2.0载体中,构建同时表达sgrna和cas9蛋白的质粒px459(sirt1-sgrna);利用lipo3000(lipofectamine3000)将构建好的质粒分别转染到ipec-j2细胞,嘌呤毒素(puromycin)结合有限稀释法筛选单克隆突变细胞系,hrma(high-resolutionmeltanalysis)检测突变结果。
本发明的构建方法能够在基因组sirt1靶位点造成移码突变的嵌合体细胞系,通过嘌呤毒素筛选和单细胞克隆培养达到在ipec-j2细胞中稳定敲除sirt1基因的目的;建立的sirt1基因敲除小肠上皮细胞系可为深入揭示仔猪断奶应激引起的肠上皮屏障功能受损及其保护机制的研究提供模型,还可用于新型抗氧化和抗生素替代产品的作用机制提供研究模型。
在一些实施例中,本发明的sirt1基因敲除的ipec-j2细胞系的构建方法包括以下步骤:
(1)根据sirt1的基因序列,在第一外显子上设计sirt1基因sgrna靶位点(如图1所示);
(2)将退火后形成反向互补序列的双链grna探针克隆到pspcas9-2a-puro(px459)v2.0载体(参见图2)中,构建同时表达sgrna和cas9蛋白的质粒px459(sirt1-sgrna);
(3)克隆筛选px459(sirt1-sgrna)阳性质粒,测序鉴定grna探针插入结果(参见图3);
(4)利用lipo3000(lipofectamine3000)将构建好的质粒转染到ipec-j2细胞;
(5)利用嘌呤毒素(puromycin)结合有限稀释法筛选单克隆突变细胞系;
(6)提取细胞基因组dna,利用hrma、测序鉴定突变结果。
为更好的说明本发明的目的、技术方案和优点,下面将结合附图和具体实施例对本发明作进一步说明。如无特别说明,本发明中的材料、试剂等均可从市场或其它公开渠道获得。如无特别说明,本发明中的实验方法均为常规方法。
实施例1
本发明的sirt1基因敲除的ipec-j2细胞系的构建方法的一种实施例,包括如下步骤:
1、sirt1靶位点设计及sgrna序列合成
根据ncbi数据库获取猪sirt1基因(geneid:nm_001145750.2)所有转录本的orf序列,并找出orf区所处的第一个外显子进行靶位点设计。
利用在线工具(http://tools.genomeengineering.org)设计筛选sgrna向导序列:5’-ctccgcggtttcttgcggag-3’,其中sgrna作用位点位于猪sirt1基因第一外显子中,在其5’端添加caccg,形成正向oligodna。
同时合成sgrna序列的反向互补序列:
5’-ctccgcaagaaaccgcggag-3’,并在5’端添加aaac,在3’端添加c,形成反向oligodna。sirt1-sgrna的碱基序列如下所示:
5’-caccgctccgcggtttcttgcggag-3’(seqidno.1)
3’-cgaggcgccaaagaacgcctccaaa-5’(seqidno.2)
2、构建px459(sirt1-sgrna)质粒
将以上正链oligodna(即seqidno.1)和负链oligodna(即seqidno.2)退火形成dsdna:
表1反应体系
将上述表1的反应体系指弹混匀,瞬时离心后置于pcr反应仪中按照如下反应程序反应:37℃30min;95℃5min,梯度降至25℃,5℃/min。
取1μl退火后形成的dsdna与bbsi酶切后线性化的pspcas9-2a-puro(px459)v2.0载体连接,连接体系如下表2所示:
表2连接体系
将上述反应体系室温放置10min反应。
3、转化及筛选鉴定px459(sirt1-sgrna)阳性克隆
取10μl上述连接产物加入stbl3感受态细胞中,冰上放置30min;42℃水浴90sec;冰上放置3~5min,加入900μl不含amp的lb液体培养基,200rpm振荡培养1h;取200μl菌液均匀涂布于amp+的lb固体培养平板上;37℃正置培养1h后,倒置培养过夜。
用灭菌牙签挑取若干单菌落,接种至1mlamp+的lb液体培养基中,37℃、200rpm培养2~3h;pcr鉴定阳性克隆,反应体系如下表3所示:
表3反应体系
pcr反应条件为:94℃预变性3min;94℃变性30s,55℃退火30s,72℃延伸1min,扩增29个循环;最后72℃延伸5min。pcr结束后,取3μl扩增的pcr产物进行琼脂糖凝胶分析,挑选阳性的克隆菌送生工测序。
测序结果如图3所示,插入片段序列与sgrna位点序列吻合,证明所得质粒为阳性质粒,可用于后续敲除实验。
将阳性质粒扩大培养后,利用无内毒素质粒提取试剂盒提取质粒,进行细胞转染。
4、细胞转染
将状态良好的的ipec-j2细胞按照106个细胞/孔的量加入6孔板中,待细胞密度为80%左右开始转染。转染前将培养基更换为无抗生素的完全培养基(2ml/孔)。
配置转染复合物:
a:用150μl的opti-mem稀释2μg//孔转染用质粒px459(sirt1-sgrna);
b:用150μl的opti-mem稀释8μllipofectamine3000/孔;
将a和b按照体积比为1:1的比例充分混匀,室温孵育5min;每孔加入300μl的转染复合物,37℃,5%co2培养箱中培养。
5、筛选、鉴定sirt1阳性突变的ipec-j2单细胞克隆
转然后6h,去除含转染复合物的细胞培养液,更换新鲜的细胞培养基。培养24h后,加入2μg/ml浓度的嘌呤毒素进行筛选。每天更换新的含2μg/ml嘌呤毒素的完全培养基,7天后将筛选的细胞消化下来,用有限稀释法接种到若干96孔细胞培养板中,37℃,5%co2培养箱中静置培养7~14天,期间观察单细胞克隆生长情况,做好标记。
14天后,将长起来的单细胞克隆转移至24孔板中继续扩大培养,待细胞达到80%左右时将细胞分别接种于两块12孔细胞培养板中,其中一块板用于继续扩大培养冻存使用,另一块板抽提细胞dna,利用hrma筛选潜在的单细胞克隆突变株,将pcr产物送生工进行测序分析。
根据sirt1基因序列,在敲除位点附近设计特异的hrma引物:
f引物:5’-aggcggcgctcgcccttcag-3’(seqidno.3)
r引物:5’-tccaaggccgccccaccaggctc-3‘(seqidno.4)
hrma反应体系如下表4所示:
表4hrma反应体系
hrma扩增条件为:98℃2min;98℃15sec,60℃20sec(收集信号),进行40个循环;95℃10sec;扩增结束后进行熔解曲线分析,从55℃至95℃,每10sec升高0.2℃,收集信号。反应完成后,分析结果,将潜在的突变克隆细胞株pcr产物纯化后送生工进行测序分析。
潜在的阳性克隆测序峰图如图5所示,与野生型(参见图4)进行比对,突变的细胞株sirt1基因缺失了5bp碱基(参见图6),在第一外显子区域造成移码突变,提前引入终止密码子。
将筛选鉴定过的突变细胞系进一步扩大培养,冻存一部分,剩余进行后续相关实验研究。
最后应当说明的是,以上实施例仅用以说明本发明的技术方案而非对本发明保护范围的限制,尽管参照较佳实施例对本发明作了详细说明,本领域的普通技术人员应当理解,可以对本发明的技术方案进行修改或者等同替换,而不脱离本发明技术方案的实质和范围。
sequencelisting
<110>广东省农业科学院动物科学研究所
<120>一种sirt1基因敲除的ipec-j2细胞系的构建方法
<130>2019
<160>4
<170>patentinversion3.5
<210>1
<211>25
<212>dna
<213>人工序列
<400>1
caccgctccgcggtttcttgcggag25
<210>2
<211>25
<212>dna
<213>人工序列
<400>2
cgaggcgccaaagaacgcctccaaa25
<210>3
<211>20
<212>dna
<213>人工序列
<400>3
aggcggcgctcgcccttcag20
<210>4
<211>23
<212>dna
<213>人工序列
<400>4
tccaaggccgccccaccaggctc23
- 还没有人留言评论。精彩留言会获得点赞!
- 一种沉默人c2orf68基因的shRNA及其医药用途
- 使用人工microrna以沉默基因家族的方法和组合物的制作方法
- 基于基因沉默技术的烟粉虱防御素基因片段及其dsRNA的制作方法
- 导入特异shRNA得到的CypA基因被沉默的重组哺乳动物细胞的制作方法
- 一种使用病毒诱导的基因沉默系统培育雄性不育植株的方法
- 修饰的基因沉默rna及其用途的制作方法
- 基于基因沉默技术的灰飞虱致死基因片段Alpha1-tubulin及其dsRNA的制作方法
- 基于基因沉默技术的灰飞虱致死基因片段Chitinase及其dsRNA的制作方法
- 基于基因沉默技术的灰飞虱致死基因片段ADP-ribosylation factor及其dsRNA的制作方法
- 基于基因沉默技术的灰飞虱致死基因片段Chitinase 7及其dsRNA的制作方法