芜菁花叶病毒P3蛋白插入标签的方法及其重组载体和应用与流程
本发明涉及基因工程领域,特别是涉及一种芜菁花叶病毒p3蛋白插入标签的方法及构建的重组载体。
背景技术:
芜菁花叶病毒(turnipmosaicvirus,tumv)是单链、正义的植物rna病毒,属于马铃薯y病毒科病毒。tumv能够侵染十子花科多种蔬菜作物与油料作物,是我国及世界农业生产上重要的病原物。一般研究植物病毒蛋白的功能,是利用基于35s启动子的瞬时表达载体,通过分析病毒蛋白的亚细胞定位、蛋白表达及其他功能。然而,基于35s启动子表达载体研究病毒蛋白功能存在以下问题:(1)35s启动子是花椰菜花叶病毒的启动子,具有很高的表达活性,造成目标蛋白在细胞内高表达、形成聚集体,并非呈现出病毒蛋白真实的亚细胞定位及表达情况;(2)病毒蛋白之间存在复杂的互作关系,通过35s启动子基于的单独外源表达病毒蛋白与在病毒侵染下,其表达水平、在细胞中的分布情况并不一定吻合,会产生假象。
病毒的侵染性克隆是依据分子生物学体外产生能够侵染植物的有活性病毒,一般用于农杆菌介导的方法将其接种到植物体内,用于研究植物病毒生物学特性重要的工具。
本发明通过体外构建带有标签myc及myc-gfp的tumv侵染性克隆,将这些标签插入到其病毒蛋白p3的n端,产生有活性并表达myc及myc-gfp的侵染性克隆,具有以下特征和优势:(1)能够表达出特异的标签融合蛋白,可用于后续分析tumvp3蛋白的功能;(2)由于融合蛋白的高表达性,证明p3是tumv重要的功能基因,可利用标签抗体进行免疫共沉淀,筛选出与p3互作的寄主蛋白;(3)插入具有gfp荧光标签的p3蛋白,能够观察在病毒侵染下的tumvp3蛋白真实的功能。这些为研究tumv及其编码的p3蛋白的生物学功能提供了有用的工具。
技术实现要素:
本发明的目的是提供一种芜菁花叶病毒p3蛋白插入标签的方法及其重组载体和应用。
为实现上述目的,本发明采用的技术方案具体如下:
一种芜菁花叶病毒p3蛋白插入标签的方法,将myc插入到pcambia2300-tumvp3蛋白的n端。
具体包括以下步骤:
(1)以tumv-kpni-f和hcpro-2a-myc-p3-r为引物,以含有tumv序列的质粒为模板,利用高保真dna聚合酶进行pcr扩增出第一段特异的pcr条带,并回收特异的pcr条带,称为pcr1;
tumv-kpni-f:tacgagacaagagcggtaccaaatgggtcacggaaac,
tumv-snabi-r:aagacatactactacgtaatctattacctatac,如seqid:no.3-4所示;
(2)以hcpro-2a-myc-p3-f和tumv-snabi-r为引物,以含有tumv序列的质粒为模板,利用高保真dna聚合酶进行pcr扩增出第二段特异的pcr条带,并回收特异的pcr条带,称为pcr2;
hcpro-2a-myc-p3-f:acactaccgcgttggaggaacagagcagaagctgatctcagaggaggacctgggaacagaatgggaggac,
hcpro-2a-myc-p3-r:gtcctcccattctgttcccaggtcctcctctgagatcagcttctgctctgttcctccaacgcggtagtgt,如seqid:no.5-6所示;
(3)利用引物tumv-kpni-f与tumv-snabi-r为引物,以pcr产物pcr1和pcr2为模板,利用高保真dna聚合酶进行overlapping-pcr扩增出特异的pcr条带,并回收特异的pcr条带,称为pcr12;
(4)构建重组的pcambia2300-tumv:myc-p3质粒:通过限制性内切酶kpni和snabi酶切pcambia2300-tumv质粒与pcr12,凝胶回收kpni-snabi切割的pcambia2300-tumv载体与pcr12;通过t4连接酶连接kpni-snabi切割的pcambia2300-tumv载体和pcr12;将连接产物转化大肠杆菌;利用引物tumv-kpni-f与tumv-snabi-r筛选阳性克隆;挑选阳性克隆,进行培养;提取培养目标克隆的质粒;通过dna测序进行验证;验证成功后,将其导入农杆菌中;
(5)活化农杆菌,接种含有pcambia2300-tumv:myc-p3质粒的农杆菌液到野生型本氏烟上,观察植物症状,确定改造的病毒具有侵染性;
(6)抽取本氏烟植株新叶片上的总蛋白,通过westernblot分析,确定重组蛋白myc-p3的存在。
通过上述方法构建的重组载体pcambia2300-tumv:myc-p3含有如seqid:no.1所示的序列。
一种芜菁花叶病毒p3蛋白插入标签的方法,将myc-gfp插入到pcambia2300-tumvp3蛋白的n端。
具体包括以下步骤:
(1)以tumv-kpni-f和gfp-myc-hc-r为引物,以含有tumv序列的质粒为模板,利用高保真dna聚合酶进行pcr扩增出第一段特异的pcr条带,并回收特异的pcr条带,称为pcr1;
tumv-kpni-f:tacgagacaagagcggtaccaaatgggtcacggaaac,
gfp-myc-hc-r:aagttcttctcctttactcaggtcctcctctgagatcagcttctgctctgttcctccaacgcggta,如seqid:no.3和no.8所示;
(2)以hc-myc-gfp-f和p3-2a-gfp-r为引物,以含有gfp序列的质粒为模板,利用高保真dna聚合酶进行pcr扩增出第二段特异的pcr条带,并回收特异的pcr条带,称为pcr2;
hc-myc-gfp-f:taccgcgttggaggaacagagcagaagctgatctcagaggaggacctgagtaaaggagaagaactt,
p3-2a-gfp-r:agtgtcctcccattctgttcctttgtatagttcatccatgcc,如seqid:no.7和no.10所示;
(3)以gfp-2a-p3-f和tumv-snabi-r为引物,以含有tumv序列的质粒为模板,利用高保真dna聚合酶进行pcr扩增出第三段特异的pcr条带,并回收特异的pcr条带,称为pcr3;
gfp-2a-p3-f:ggcatggatgaactatacaaaggaacagaatgggaggacact,如seqid:no.9所示;
(4)利用引物tumv-kpni-f与p3-2a-gfp-r为引物,以pcr产物pcr1和pcr2为模板,利用高保真dna聚合酶进行overlapping-pcr扩增出特异的pcr条带,并回收特异的pcr条带,称为pcr12;
(5)利用引物tumv-kpni-f与tumv-snabi-r为引物,以pcr产物pcr12和pcr3为模板,利用高保真dna聚合酶进行overlapping-pcr扩增出特异的pcr条带,并回收特异的pcr条带,称为pcr123;
tumv-snabi-r:aagacatactactacgtaatctattacctatac,如seqid:no.4所示;
(6)构建重组的pcambia2300-tumv:myc-gfp-p3质粒:通过限制性内切酶kpni和snabi酶切pcambia2300-tumv质粒与pcr123,凝胶回收kpni-snabi切割的pcambia2300-tumv载体与pcr123;通过t4连接酶连接kpni-snabi切割的pcambia2300-tumv载体和pcr123;将连接产物转化大肠杆菌;利用引物tumv-kpni-f与tumv-snabi-r筛选阳性克隆;挑选阳性克隆,进行培养;提取培养目标克隆的质粒;通过dna测序进行验证;验证成功后,将其导入农杆菌中;
(7)活化农杆菌,接种含有pcambia2300-tumv:myc-gfp-p3质粒的农杆菌液到野生型本氏烟上,观察植物症状,确定改造的病毒具有侵染性;
(8)抽取本氏烟植株新叶片上的总蛋白,通过westernblot分析,确定重组蛋白myc-gfp-p3的存在。
上述方法构建的重组载体pcambia2300-tumv:myc-gfp-p3含有如seqid:no.2所示的序列。
改造具有标签的侵染性克隆有利于进一步研究病毒蛋白的功能、实时观察病毒在细胞体内的变化情况、客观真实反应在病毒侵染的条件下,病毒蛋白的表达、定位等功能的变化、可通过免疫共沉淀利用标签抗体筛选出与病毒互作的寄主蛋白等。因此插入myc标签和myc-gfp标签到tumvp3蛋白n端是一种有效研究p3蛋白功能的手段。
tumv是严重危害农业生产的病原物,本发明产生和获得的p3蛋白n端具有myc或myc-gfp标签的重组性侵染性克隆tumv,有利于进一步研究tumv病毒蛋白与寄主蛋白的功能,挖掘出的抗病毒寄主蛋白为病毒病害的防治提供新的抗病策略,具有重要的生产意义。
本发明构建出的重组载体pcambia2300-tumv:myc-p3和pcambia2300-tumv:myc-gfp-p3具有以下特征和优势:
(1)具有侵染性,插入的标签不会影响tumv的侵染性;
(2)能够表达出特异的标签融合蛋白,可用于后续分析tumvp3蛋白的功能;
(3)由于融合蛋白的高表达性,证明p3是tumv重要的功能基因,可利用标签抗体进行免疫共沉淀,筛选出与p3互作的寄主蛋白;
(4)插入具有gfp荧光标签的p3蛋白,能够观察到在病毒侵染下的tumvp3蛋白真实的功能。
下面结合附图说明和具体实施例对本发明所述的芜菁花叶病毒p3蛋白插入标签的方法及其重组载体和应用作进一步说明。
附图说明
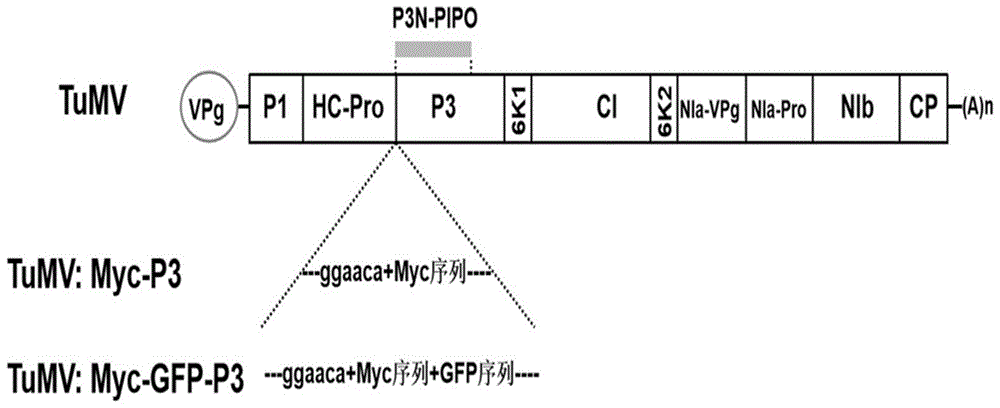
图1为野生型侵染性克隆pcambia2300-tumv(简称tumv),重组侵染性克隆pcambia2300-tumv:myc-p3(简称tumv:myc-p3)和pcambia2300-tumv:myc-gfp-p3(简称tumv:myc-gfp-p3)载体构建示意图。
图2为接种重组侵染性克隆tumv:myc-p3和tumv:myc-gfp-p3到本氏烟上7天后,植物感染病毒的症状,接种缓冲液的作为对照(mock)。
图3为westernblot检测tumv:myc-p3和tumv:myc-gfp-p3系统侵染的本氏烟中myc-p3蛋白及myc-gfp-p3蛋白的表达情况。使用myc抗体和gfp抗体进行westernblot分析。
(a)使用myc抗体(wb:myc)检测mock和tumv:myc-p3侵染本氏烟系统叶片中myc-p3蛋白的表达情况,myc标签大小为2kda,p3蛋白大小约为40kda,因此myc-p3大小约为42kda。
(b,c)使用myc抗体(b,wb:myc)和gfp抗体(c,wb:gfp)检测mock和tumv:myc-gfp-p3侵染本氏烟系统叶片中myc-gfp-p3蛋白的表达情况,myc标签大小为2kda,gfp标签大小为26kda,p3蛋白大小约为40kda,因此myc-gfp-p3大小约为68kda。考马斯亮蓝染色的rubisco大小亚基作为上样的对照(cbb)(a-c)。
图4为接种重组侵染性克隆tumv:myc-gfp-p3到本氏烟3天(接种叶片)和7天(系统新出现的叶片)上的myc-gfp-p3的荧光表达情况。激光共聚焦显微镜拍摄,所用激光为gfp,bright为白光,图中标尺大小为50μm。
具体实施方式
实施例1
如图1所示,一种芜菁花叶病毒p3蛋白插入标签的方法,将myc插入到pcambia2300-tumvp3蛋白的n端。具体包括以下步骤:
(1)以tumv-kpni-f和hcpro-2a-myc-p3-r为引物,以含有tumv序列的质粒为模板,利用高保真dna聚合酶进行pcr扩增出第一段特异的pcr条带,并回收特异的pcr条带,称为pcr1;
tumv-kpni-f:tacgagacaagagcggtaccaaatgggtcacggaaac,
tumv-snabi-r:aagacatactactacgtaatctattacctatac,如seqid:no.3-4所示;
(2)以hcpro-2a-myc-p3-f和tumv-snabi-r为引物,以含有tumv序列的质粒为模板,利用高保真dna聚合酶进行pcr扩增出第二段特异的pcr条带,并回收特异的pcr条带,称为pcr2;
hcpro-2a-myc-p3-f:acactaccgcgttggaggaacagagcagaagctgatctcagaggaggacctgggaacagaatgggaggac,
hcpro-2a-myc-p3-r:gtcctcccattctgttcccaggtcctcctctgagatcagcttctgctctgttcctccaacgcggtagtgt,如seqid:no.5-6所示;
(3)利用引物tumv-kpni-f与tumv-snabi-r为引物,以pcr产物pcr1和pcr2为模板,利用高保真dna聚合酶进行overlapping-pcr扩增出特异的pcr条带,并回收特异的pcr条带,称为pcr12;
(4)构建重组的pcambia2300-tumv:myc-p3质粒:通过限制性内切酶kpni和snabi酶切pcambia2300-tumv质粒与pcr12,凝胶回收kpni-snabi切割的pcambia2300-tumv载体(回收切割出的大片段)与pcr12;通过t4连接酶连接kpni-snabi切割的pcambia2300-tumv载体和pcr12;将连接产物转化大肠杆菌;利用引物tumv-kpni-f与tumv-snabi-r筛选阳性克隆;挑选阳性克隆,进行培养;提取培养目标克隆的质粒;通过dna测序进行验证;验证成功后,将其导入农杆菌中;
(5)活化农杆菌,接种含有pcambia2300-tumv:myc-p3质粒的农杆菌液到野生型本氏烟上,观察植物症状,如图2所示,确定改造后的病毒具有侵染性;
(6)抽取本氏烟植株新叶片上的总蛋白,通过westernblot分析,如图3所示,确定重组蛋白myc-p3的存在。
通过上述方法构建的重组载体pcambia2300-tumv:myc-p3含有如seqid:no.1所示的序列。
实施例2
如图1所示,一种芜菁花叶病毒p3蛋白插入标签的方法,将myc-gfp插入到pcambia2300-tumvp3蛋白的n端。具体包括以下步骤:
(1)以tumv-kpni-f和gfp-myc-hc-r为引物,以含有tumv序列的质粒为模板,利用高保真dna聚合酶进行pcr扩增出第一段特异的pcr条带,并回收特异的pcr条带,称为pcr1;
tumv-kpni-f:tacgagacaagagcggtaccaaatgggtcacggaaac,
gfp-myc-hc-r:aagttcttctcctttactcaggtcctcctctgagatcagcttctgctctgttcctccaacgcggta,如seqid:no.3和no.8所示;
(2)以hc-myc-gfp-f和p3-2a-gfp-r为引物,以含有gfp序列的质粒为模板,利用高保真dna聚合酶进行pcr扩增出第二段特异的pcr条带,并回收特异的pcr条带,称为pcr2;
hc-myc-gfp-f:taccgcgttggaggaacagagcagaagctgatctcagaggaggacctgagtaaaggagaagaactt,
p3-2a-gfp-r:agtgtcctcccattctgttcctttgtatagttcatccatgcc,如seqid:no.7和no.10所示;
(3)以gfp-2a-p3-f和tumv-snabi-r为引物,以含有tumv序列的质粒为模板,利用高保真dna聚合酶进行pcr扩增出第三段特异的pcr条带,并回收特异的pcr条带,称为pcr3;
gfp-2a-p3-f:ggcatggatgaactatacaaaggaacagaatgggaggacact,如seqid:no.9所示;
(4)利用引物tumv-kpni-f与p3-2a-gfp-r为引物,以pcr产物pcr1和pcr2为模板,利用高保真dna聚合酶进行overlapping-pcr扩增出特异的pcr条带,并回收特异的pcr条带,称为pcr12;
(5)利用引物tumv-kpni-f与tumv-snabi-r为引物,以pcr产物pcr12和pcr3为模板,利用高保真dna聚合酶进行overlapping-pcr扩增出特异的pcr条带,并回收特异的pcr条带,称为pcr123;
tumv-snabi-r:aagacatactactacgtaatctattacctatac,如seqid:no.4所示;
(6)构建重组的pcambia2300-tumv:myc-gfp-p3质粒:通过限制性内切酶kpni和snabi酶切pcambia2300-tumv质粒与pcr123,凝胶回收kpni-snabi切割的pcambia2300-tumv载体与pcr123;通过t4连接酶连接kpni-snabi切割的pcambia2300-tumv载体和pcr123;将连接产物转化大肠杆菌;利用引物tumv-kpni-f与tumv-snabi-r筛选阳性克隆;挑选阳性克隆,进行培养;提取培养目标克隆的质粒;通过dna测序进行验证;验证成功后,将其导入农杆菌中;
(7)活化农杆菌,接种含有pcambia2300-tumv:myc-gfp-p3质粒的农杆菌液到野生型本氏烟上,如图2所示,观察植物症状,确定改造的病毒具有侵染性;
(8)抽取本氏烟植株新叶片上的总蛋白,通过westernblot分析,如图3所示,确定重组蛋白myc-gfp-p3的存在。
(9)如图4所示,通过激光共聚焦显微镜观察新叶片gfp-p3荧光的表达,表明p3蛋白在病毒侵染的情况下,主要定位在细胞质中并可能形成病毒复制复合物的场所。
以上所述的实施例仅仅是对本发明的优选实施方式进行描述,并非对本发明的范围进行限定,在不脱离本发明设计精神的前提下,本领域普通技术人员对本发明的技术方案作出的各种变形和改进,均应落入本发明权利要求书确定的保护范围内。
序列表
<110>中国农业科学院植物保护研究所
<120>芜菁花叶病毒p3蛋白插入标签的方法及其重组载体和应用
<160>10
<170>siposequencelisting1.0
<210>1
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>1
taccgcgttggaggaacagagcagaagctgatctcagaggaggacctg48
<210>2
<211>759
<212>dna
<213>人工序列(artificialsequence)
<400>2
taccgcgttggaggaacagagcagaagctgatctcagaggaggacctgagtaaaggagaa60
gaacttttcactggagttgtcccaattcttgttgaattagatggtgatgttaatgggcac120
aaattttctgtcagtggagagggtgaaggtgatgcaacatacggaaaacttacccttaaa180
tttatttgcactactggaaaactacctgttccgtggccaacacttgtcactactttctct240
tatggtgttcaatgcttttcaagatacccagatcatatgaagcggcacgacttcttcaag300
agcgccatgcctgagggatacgtgcaggagaggaccatcttcttcaaggacgacgggaac360
tacaagacacgtgctgaagtcaagtttgagggagacaccctcgtcaacaggatcgagctt420
aagggaatcgatttcaaggaggacggaaacatcctcggccacaagttggaatacaactac480
aactcccacaacgtatacatcatggccgacaagcaaaagaacggcatcaaagccaacttc540
aagacccgccacaacatcgaagacggcggcgtgcaactcgctgatcattatcaacaaaat600
actccaattggcgatggccctgtccttttaccagacaaccattacctgtccacacaatct660
gccctttcgaaagatcccaacgaaaagagagaccacatggtccttcttgagtttgtaaca720
gctgctgggattacacatggcatggatgaactatacaaa759
<210>3
<211>37
<212>dna
<213>人工序列(artificialsequence)
<400>3
tacgagacaagagcggtaccaaatgggtcacggaaac37
<210>4
<211>33
<212>dna
<213>人工序列(artificialsequence)
<400>4
aagacatactactacgtaatctattacctatac33
<210>5
<211>70
<212>dna
<213>人工序列(artificialsequence)
<400>5
acactaccgcgttggaggaacagagcagaagctgatctcagaggaggacctgggaacaga60
atgggaggac70
<210>6
<211>70
<212>dna
<213>人工序列(artificialsequence)
<400>6
gtcctcccattctgttcccaggtcctcctctgagatcagcttctgctctgttcctccaac60
gcggtagtgt70
<210>9
<211>66
<212>dna
<213>人工序列(artificialsequence)
<400>9
taccgcgttggaggaacagagcagaagctgatctcagaggaggacctgagtaaaggagaa60
gaactt66
<210>10
<211>66
<212>dna
<213>人工序列(artificialsequence)
<400>10
aagttcttctcctttactcaggtcctcctctgagatcagcttctgctctgttcctccaac60
gcggta66
<210>11
<211>42
<212>dna
<213>人工序列(artificialsequence)
<400>11
ggcatggatgaactatacaaaggaacagaatgggaggacact42
<210>12
<211>42
<212>dna
<213>人工序列(artificialsequence)
<400>12
agtgtcctcccattctgttcctttgtatagttcatccatgcc42
- 还没有人留言评论。精彩留言会获得点赞!