LOC100507599作为喉鳞癌诊断的分子标记的制作方法
本发明属于生物医学领域,涉及loc100507599作为喉鳞癌诊断的分子标记。
背景技术:
喉癌是头颈部常见的恶性肿瘤,占头颈部肿瘤的第二位,其发病率约占全身恶性肿瘤的5%,鳞状细胞癌约占喉癌总数的93%-99%,喉鳞状细胞癌原发于声带部位者居多,约占60%。黄新辉等对306例喉恶性肿瘤进行回顾性分析,发现鳞状细胞癌占全部恶性肿瘤的99.7%,其中又以高、中分化鳞癌为主;喉癌的原发部位以声门型居多,其次为声门上型,声门下型;吸烟、饮酒与肿瘤的发生密切相关。虽然手术、放疗和化疗可以在不同程度上缓解患者的痛苦,但是治疗后常造成功能障碍、局部畸形,而且复发率较高,严重威胁患者的身心健康。1971年knudson提出的二次突变假说是一个为肿瘤遗传学研究工作者所普遍接受的有关肿瘤发生机制的科学预言,指导了30多年的肿瘤发生分子机制的研究。探讨不同类型肿瘤相关基因是认识肿瘤发生分子机制、开展基因诊断和基因治疗的基本前提,包括肿瘤在内的疾病致病基因的克隆和定位仍是人类基因组计划完成后的一项重要内容。srinivus等研究发现,肿瘤在很早期即可伴随肿瘤标志物的改变,检测其含量变化有助于肿瘤的早期发现。基因水平的研究工作将是根治包括喉癌在内的所有肿瘤的关键。因此,寻找并克隆喉癌特异性相关基因将为喉癌的分子机制、基因诊断和基因治疗的研究提供广阔前景。
虽然肿瘤诊断与治疗的手段日新月异,但全球的喉癌总体生存率并未得到明显改善,尤其是晚期病人。其原因主要表现在三个方面:首先,喉癌早期临床表现多样,给喉癌的早期诊断带来了较大的难度,多数患者早期未能接受及时有效的治疗,如何找到喉癌早期诊断的分子标记物,是提高喉癌早期诊断率的关键。二,喉癌的治疗以手术为主,放疗、化疗为辅,尽管喉部分切除术及声门型喉癌早期采用c02激光治疗比例的提高,使得喉功能完全保留或部分保留,术后并发症减少,从而使患者的生活质量得到提高,但术后复发率仍然很高,如果能找出喉癌促瘤基因或肿瘤抑制因子,从中筛选出潜在的药物作用靶点,将会为开发出喉癌相关特异性的疫苗或/和抗癌新药奠定基础。三,不同患者所患喉癌在病理类型、肿瘤分化程度、就诊时病变严重程度等方面有差异,不同喉癌患者有不同的临床表现及体质状况,对手术后放疗、化疗的敏感性各有差异,如何找到疗效监测,预后判断的分子标志物,将为实现对喉癌的个性化治疗奠定基础。
技术实现要素:
本发明的目的在于提供一种用于诊断喉鳞癌的长链非编码rna标志物。本发明利用测序和qpcr实验证明loc100507599在喉鳞癌患者癌组织中的表达水平明显高于癌旁组织,因此可以将loc100507599作为诊断喉鳞癌的分子标志物。
为了实验上述目的,本发明采用了如下技术方案:
本发明提供了检测长链非编码rna表达的试剂在制备喉鳞癌诊断产品中的应用;所述长链非编码rna的编码基因geneid:100507599(nc_000022.11(26512532..26514568))。
本发明的长链非编码rna在ncbi中的命名为loc100507599。本发明的属于基因id:100507599编码产物的loc100507599的转录本如下:genbank登录号nr_144529.1(具有1673bp的长度)。
进一步,所述试剂包括利用sybrgreen、taqman探针、分子信标、双杂交探针、或复合探针检测loc100507599表达量时使用的试剂。
更进一步,所述试剂包括检测loc100507599表达量时使用的pcr扩增引物。
在本发明的具体实施方案中,所述pcr扩增引物序列如seqidno.1和seqidno.2所示。
本发明提供了一种用于喉鳞癌诊断的产品,所述产品通过检测样本中前面所述的长链非编码rna的表达来诊断喉鳞癌。
进一步,所述产品包括但不限于芯片、试剂盒、试纸、或高通量测序平台;高通量测序平台是一种特殊的诊断工具,随着高通量测序技术的发展,对一个人的rna表达谱的构建将成为十分便捷的工作。通过对比疾病患者和正常人群的rna表达谱,容易分析出哪个rna的异常与疾病相关。因此,在高通量测序中获知loc100507599表达异常与喉鳞癌相关也属于loc100507599的用途,同样在本发明的保护范围之内。
所述试剂盒包括检测loc100507599表达量的试剂,所述试剂包括与loc100507599或其dna序列结合的核酸,所述核酸包括sybrgreen、taqman探针、分子信标、双杂交探针、或复合探针检测loc100507599表达量时使用的引物和/或探针。
所述芯片包括检测loc100507599表达量的试剂,所述试剂包括与loc100507599或其dna序列结合的核酸,所述核酸包括能够检测loc100507599表达量的引物和/或探针。
所述试纸包括检测loc100507599表达量的试剂,所述试剂包括与loc100507599或其dna序列结合的核酸,所述核酸包括能够检测loc100507599表达量的引物和/或探针。
进一步,所述引物序列如seqidno.1和seqidno.2所示。
进一步,本发明的所述样本来源于组织。
本发明还提供了一种诊断喉鳞癌的方法,所述方法包括如下步骤:
(1)获取受试者喉部疑似患病组织;
(2)检测步骤1组织中loc100507599的表达水平;
(3)将测得的loc100507599的表达水平与受试者的患病与否关联起来。
(4)与正常对照相比,受试者喉部疑似患病组织中loc100507599的表达水平显著升高,则该受试者被判断患有喉鳞癌、或判断该受试者患有喉鳞癌的风险高、或者喉鳞癌患者被判断为复发、或者喉鳞癌患者被判断为预后不良。
在本发明的上下文中,“正常对照”可以指不患有喉鳞癌的正常人,也可以指患有喉鳞癌的人的癌旁组织。
在本发明的上下文中,“诊断”包括判断受试者是否已经患病、判断受试者是否存在患病的风险、判断患者是否已经复发、判断患者对药物治疗的反应性、或者判断患者的预后情况。
附图说明
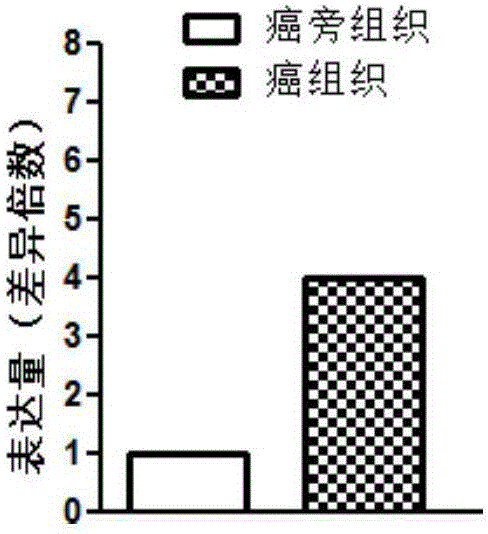
图1显示利用qpcr检测loc100507599的差异表达情况的统计图。
具体的实施方式
下面结合附图和实施例对本发明作进一步详细的说明。以下实施例仅用于说明本发明而不用于限制本发明的范围。实施例中未注明具体条件的实验方法,通常按照常规条件,例如sambrook等人,分子克隆:实验室手册(newyork:coldspringharborlaboratorypress,1989)中所述的条件,或按照制造厂商所建议的条件。
实施例1筛选差异表达分子
一、研究对象
收集5例喉鳞癌患者的癌组织及相应的癌旁组织。纳入排除标准:①患者除原发肿瘤外,无其他部位原发肿瘤,原发肿瘤未转移;②无糖尿病、高血压等心脑血管疾病史;③无肝炎、梅毒、结核、hiv等传染病病史;④无家族遗传性疾病史;⑤术前未行放疗、化疗及生物治疗等。
取样注意事项:切取肿瘤标本时注意切取肿瘤中央区域非坏死肿瘤组织,切取癌旁正常组织时尽可能远离肿瘤,距肿瘤边缘1.5cm以上,尽量切取粘膜上皮组织避免深层结缔组织。
二、实验步骤
利用norgenrna提取试剂盒提取组织总rna:
组织裂解
1)在较少rnase干扰的清洁区,使用含适量液氮的研钵称取离体组织样本约20mg,用杵棒研磨至粉末状;
2)将样本转移到一个不含rna酶的2ml的离心管中;
3)加入300ullysissolution,置于匀浆器内,充分研磨1-5min;
4)12000g,4℃,离心10min,转移上清至新的1.5ml的离心管中;
5)加入600ulrnase-freewater,用漩涡器混匀;
6)加入20ul蛋白酶k,在55℃温浴15min,不断涡旋混匀;
7)14000g,室温,离心1min,使细胞碎片沉淀于离心管底部,取上清转移到另外一个不含rna酶1.5ml的离心管中;
8)加入450ul的95%乙醇,涡旋混匀;
rna吸附:
9)取650ul含乙醇的裂解液加到离心柱中,14000g离心1min;
10)弃下层,重置收集管于柱上;
11)依照裂解液的容量,重复9)~10)步;
12)加入400ulwashsolution,14000g离心2min;
13)弃下层,将柱置于一新的收集管上;
dnase处理:
14)加入100ulenzymeincubationbuffer和15uldnasei,14000g离心1min;
15)将收集管中的溶液重新移入柱中;
16)室温放置15min;
rna洗涤:
17)加入400ulwashsolution,14000g离心1min,弃下层,重置收集管于柱上;
18)加入400ulwashsolution,14000g离心2min,弃收集管;
rna洗脱:
19)把柱子放入1.7mlelution管中;
20)加入30ulelutionbuffer;
21)200g离心2min,使溶液充分与柱结合,然后14000g离心1min。
2、rna质量检测
使用nanodropnd-1000型紫外分光光度计测定组织总rna的浓度和纯度。
3、组织总rna完整性测定
经1%甲醛变性琼脂糖凝胶电泳,紫外透射光下观察,检测rna的完整性。
4、agilent2100测定rin值。
lncrna测序要求:样品需求量:≥200ng;样品浓度:c≥20ng/μl;样品纯度:rin≥7.0,28s/18s≥1.0。
5、去除rrna
细胞内一部分(>24%)的长链非编码rna都是缺少传统polya尾的,因此采用去除rrna的方式建库可以获得更加全面的lncrna信息。
6、片段化rna
illumina平台是针对短序列片段进行测序,去除rrna得到的mrna和lncrna是完整的rna序列,平均长度可能达几kb,因此需要对其进行随机打断。利用金属离子,可以将rna随机断裂成200bp左右的小片段。
7、反转合成cdna
在逆转录酶的作用下,利用随机引物,以mrna为模板反转合成一链cdna,进行二链合成时,dntps试剂中用dutp代替dttp,使cdna第二链中碱基包含a/u/c/g。
8、连接adaptor
双链的cdna结构为粘性末端,加入endrepairmix将其补成平末端,随后在3’末端加上一个a碱基,用于连接y字形的接头。
9、ung酶消化cdna二链
在pcr扩增前,用ung酶将cdna第二链消化,从而使文库中仅包含cdna第一链。
10、illuminahiseq2500上机测序
illuminahiseq2500测序平台,进行2*150bp测序。
11、生物信息学分析
测序数据获得以后的rawdata分析过程如下所示:
(1)用cutadapt对reads的5’和3’段进行trim,trim掉质量<20的碱基,并且删掉n大于10%的reads;
(2)tophat比对到参考基因组上。所用的参考基因组版本为grch38.p7,fasta和gff文件下载自ncbi;
(3)cuffquant定量lncrna的表达量并标准化输出;
(4)cuffdiff比较癌旁组织跟癌组织lncrna的表达差异。
12、结果
显著差异lncrna筛选条件:fdr<0.01。用以上标准筛选得到以下结果:与癌旁组织相比,喉鳞癌患者癌组织中差异表达lncrna为247个,其中上调的为96个,下调的为151个。
实施例2大样本验证筛选出的差异表达分子
1、样本收集
按照实施例1的方法收集30例喉鳞癌患者的癌组织和癌旁组织。
2、在转录水平上进行验证
试剂:反转录试剂盒(ddr037a)购自宝生物工程(大连)有限公司。荧光实时(real-time)定量pcr(polymerasechainreaction)所用的sybrpremixextaqtm(tlirnasehplus)试剂盒为日本takara公司生产。
2.1提取组织总rna
步骤同实施例1。
2.2逆转录
以提取的总rna(1μg)为模板,加入以下反应体系,具体为:
2.3qpcr
按照日本takara公司的
反应体系:sybrpremixextaqtm(2×)25μl,roxreferencedye(50×)1μl,pcr上游引物(10μm)1μl,pcr下游引物(10μm)1μl,cdna4μl,灭菌ddh2o18μl。
反应程序:95℃20s预变性,按95℃10s变性、55℃20s退火、70℃10s延伸过程循环40次,获得ct值。
结果釆用相对定量法,运用公式2-△△ct计算。实验重复3次。
引物设计:根据loc1005075997序列,通过ncbi的引物设计工具(primerblast)设计针对cdna的引物,引物序列如下所示:上游引物:5’-ttccagcagattcaagag-3’(seqidno.1);下游引物:5’-acagtcatcattcctaagc-3’(seqidno.2)。
根据gapdh(内参基因)序列设计引物,上游引物:5’-ctctggtaaagtggatattgt-3’(seqidno.3);5’-ggtggaatcatattggaaca-3’(seqidno.4)。
3、结果
结果显示,30例喉鳞癌患者中有26例患者癌组织loc1005075997表达水平显著高于癌旁组织。统计结果如图1所示,与癌旁组织相比,喉鳞癌患者癌组织loc1005075997表达水平明显升高,差异具有统计学意义(p<0.05)。
上述实施例的说明只是用于理解本发明的方法及其核心思想。应当指出,对于本领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以对本发明进行若干改进和修饰,这些改进和修饰也将落入本发明权利要求的保护范围内。
序列表
<110>北京泱深生物信息技术有限公司
<120>loc100507599作为喉鳞癌诊断的分子标记
<160>4
<170>siposequencelisting1.0
<210>1
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>1
ttccagcagattcaagag18
<210>2
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>2
acagtcatcattcctaagc19
<210>3
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>3
ctctggtaaagtggatattgt21
<210>4
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>4
ggtggaatcatattggaaca20
- 还没有人留言评论。精彩留言会获得点赞!