抗草鱼呼肠孤Ⅰ型病毒的核酸适配体及其构建方法和应用与流程
本发明属于水产病原菌检测技术领域,具体地,涉及一种抗草鱼呼肠孤Ⅰ型病毒的核酸适配体及其构建方法和应用。
背景技术:
广西是我国的水产养殖大省,主要养殖品种包括草鱼、斑点叉尾鮰、石斑鱼、草鱼、对虾、牡蛎、马氏珠母贝和各种食用植物。据统计,截止2017年底,广西的渔业经济总产值已超过621亿元,水产养殖总量超过320.76万吨。然而,水产养殖规模的不断扩大和养殖密度的持续增加导致养殖环境恶化,各类水产疫病病原频繁暴发。2018年广西南宁、河池、柳州等地池塘和网箱养殖的草鱼暴发疑似草鱼出血症,通过对患病草鱼中病原微生物进行聚合酶链式反应检测技术(polymerase chain reation,PCR)分析鉴定,证明草鱼呼肠孤Ⅰ型病毒(Grass carp reovirus,GCRVⅠ)是主要致病病原。草鱼出血症是草鱼养殖中最常见的病毒病,其致病病原为草鱼呼肠孤病毒(Grass carp reovirus,GCRV),又称为草鱼出血病病毒(GCHV),隶属呼肠孤病毒科,水生动物呼肠孤病毒属。GCRV病毒作为一种高致病性鱼类传染病毒,通常表现为爆发式感染,即会在极短时间内导致鱼大量死亡。目前GCRV病毒引起的草鱼病毒性出血症在我国广泛存在,严重影响我国的水产养殖业的发展。因此,研发新型高效的抗病毒药物,同时,针对GCRV病毒发展操作便捷、成本低、耗时短、准确度高的检测技术,对于防控草鱼GCRV病毒的危害来说至关重要。
核酸适配体是利用指数富集的配基系统进化技术(Systematic Evolution of Ligands by Exponential Enrichment technology,SELEX),在体外经过多轮筛选获得的、能够特异性识别靶物质的单链寡核苷酸。核酸适配体具有特异性高、亲和性高、稳定性强、易化学合成和化学修饰等诸多优点。目前核酸适配体作为一类新型的、广受关注的新型检测和治疗工具,在人类医学研究、疾病诊断、病毒侵染机制研究等领域展现出广阔的应用前景。
技术实现要素:
本发明的目的是提供一种抗草鱼呼肠孤Ⅰ型病毒的核酸适配体及其构建方法和应用,以实现草鱼呼肠孤Ⅰ型病毒的高灵敏、特异性检测。
根据本发明的一个方面,提供一种抗草鱼呼肠孤Ⅰ型病毒的核酸适配体:包括SEQ ID NO.1的核苷酸序列。
优选地,包括SEQ ID NO.2的核苷酸序列。
优选地,其核苷酸序列上的至少一个核苷酸被磷酸化、巯基化、甲基化、氨基化或同位素化。
优选地,其核苷酸序列上结合有标志物,标志物选自荧光物质、发光材料、生物素或酶中的一种或一种以上。
优选地,荧光物质选自羟基荧光素、异硫氰酸荧光素或羧基四甲基罗丹明中的一种或一种以上。
根据本发明的另一个方面,提供一种构建上述抗草鱼呼肠孤Ⅰ型病毒的核酸适配体的方法,采用SELEX技术,包括以下步骤:(1)建立单链DNA随机文库Library50,单链DNA随机文库Library50的核苷酸序列为:
5’-GTCTGAAGTAGACGCAGGAG(50N)AGTCACACCTGAGTAAGCGT;(2)利用草鱼呼肠孤Ⅰ型病毒感染细胞筛选单链DNA随机文库Library50,得出草鱼呼肠孤Ⅰ型病毒的特异性DNA文库;(3)以特异性DNA文库作为模板,与核苷酸序列为SEQ ID NO.3的引物、核苷酸序列为SEQ ID NO.4的引物进行PCR扩增。
优选地,PCR扩增的扩增程序是:94℃5分钟,94℃1分钟,56℃30秒,72℃1分钟,25轮循环;72℃5分钟。
根据本发明的另一个方面,提供上述抗草鱼呼肠孤Ⅰ型病毒的核酸适配体在检测草鱼呼肠孤Ⅰ型病毒中的应用。
根据本发明的另一个方面,提供一种草鱼呼肠孤Ⅰ型病毒的荧光定量PCR快速检测试剂盒:包括荧光分子检测探针,以上述抗草鱼呼肠孤Ⅰ型病毒的核酸适配体作为荧光分子检测探针,标志物为羟基荧光素。
本发明提供的抗草鱼呼肠孤Ⅰ型病毒的核酸适配体的分子量较小,制备周期短,重现性好,便于体外化学合成,便于标记,序列稳定易于运输和保存。该核酸适配体对草鱼呼肠孤Ⅰ型病毒具有较高的特异性和亲和力,并且无免疫原性。另外,组成该核酸适配体的核苷酸序列容易被取代或修饰。经部分取代或者经过修饰后的上述核酸适配体能够保持与原核酸适配体基本相同或者类似的分子结构、理化性质和功能,同样能够有效实现草鱼呼肠孤Ⅰ型病毒的检测。利用羟基荧光素标记本发明提供的核酸适配体,由此,将该核酸适配体制作成荧光探针,将其与实时荧光定量PCR检测技术相结合,能够实现对草鱼呼肠孤Ⅰ型病毒的高特异性、高灵敏度的实时定性、定量检测。
附图说明
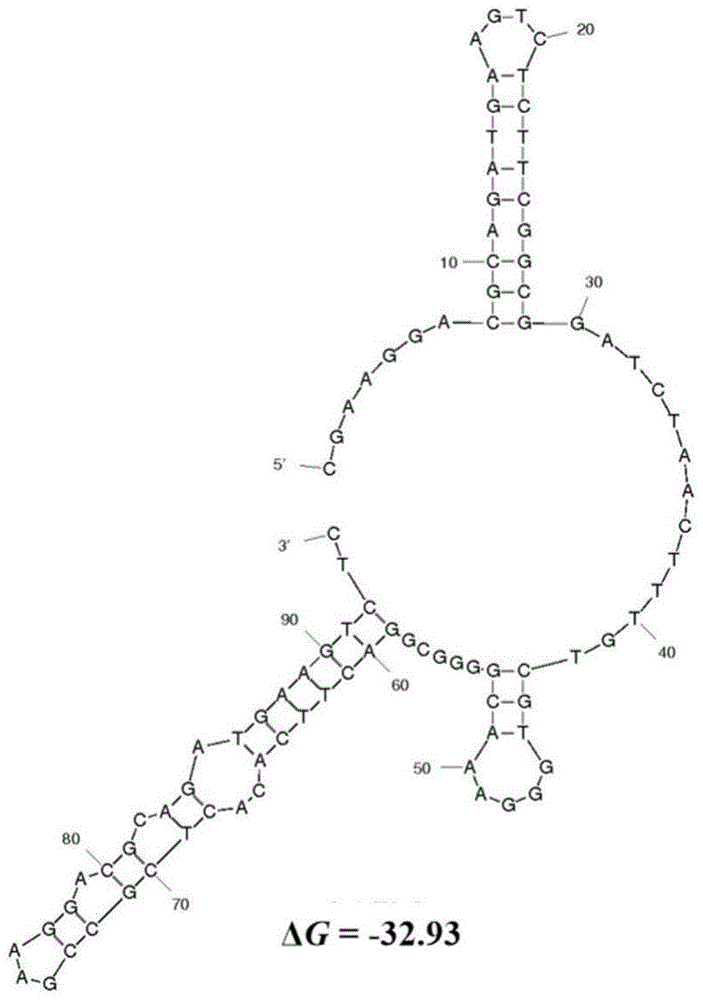
图1为核苷酸序列为SEQ ID NO.2的抗草鱼呼肠孤Ⅰ型病毒的核酸适配体的二级结构预测图;
图2为实施例2中流式细胞仪检测的样品FAM荧光值测试结果;
图3为实施例2中激光扫描共聚焦显微镜的观测结果:(a)对照组样品的光镜图,(b)对照组样品的荧光图,(c)试验组样品的光镜图,(d)试验组样品的荧光图。
具体实施方式
为了使本技术领域的人员更好地理解本发明方案,下面将对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分的实施例,而不是全部的实施例。
实施例1检测抗草鱼呼肠孤Ⅰ型病毒的核酸适配体的筛选和制备
S1.随机单链DNA(ssDNA)文库的构建和引物的合成
构建随机ssDNA文库Library50,两端为固定序列,中间50个核苷酸为随机序列,其核苷酸序列如下:
5’-GTCTGAAGTAGACGCAGGAG(50N)AGTCACACCTGAGTAAGCGT。
5’引物(SEQ ID NO.3):5’-FAM-GTCTGAAGTAGACGCAGGAG-3’。
3’引物(SEQ ID NO.4):5’-Biotin-ACGCTTACTCAGGTGTGACT-3’。
随机ssDNA文库和引物均由上海生工生物有限公司合成。
S2.SELEX筛选(相当于阳性筛选)
将10nmol上述随机ssDNA文库溶解在500μL PBS中,92℃恒温水浴5min,然后迅速插入冰中,冰浴10min,将处理后的随机ssDNA文库与草鱼GCRVⅠ型病毒感染细胞在冰上孵育1h。
孵育结合完成后,离心移除上清,用10mL的经过PBS洗涤草鱼GCRVⅠ型病毒感染细胞,92℃恒温水浴10min,12000g离心收集上清,即为特异性识别草鱼GCRVⅠ型病毒感染细胞的ssDNA核酸文库。
S3.PCR扩增
取100μL筛选得到的识别草鱼GCRVⅠ型病毒感染细胞的ssDNA文库和5’引物、3’引物进行PCR扩增。PCR反应体系如下(1000μL):10×Buffer 100μL,dNTP Mix(2.5mM)80μL,SEQ ID NO.3的引物40μL,SEQ ID NO.4的引物,ssDNA文库100μL,rTaq酶12.5μL,ddH2O 627.5μL。PCR的扩增程序为:94℃5min,94℃1min,56℃30sec,72℃1min,经过25轮循环;72℃5min。第一轮SELEX筛选后得到的上清,要全部用于进行PCR扩增,扩增后得到双链核酸(dsDNA)文库。
S4.ssDNA文库的制备
将100μL链酶亲和素标记的磁珠与S3制得的dsDNA文库在常温下孵育20min,利用dsDNA上的生物素与磁珠上链酶亲和素的亲和作用,将dsDNA结合到磁珠表面,利用磁性分离器上除去上清,用2mL PBS洗涤磁珠,然后在EP管中加入200μL NaOH溶液(200mM),常温反应10min,使dsDNA变性解链,带有生物素的一条链与链亲和素结合留在磁珠上,反应完毕后利用磁性分离架回收得到上清液;将上清液加入经过无菌水洗涤后的除盐柱,在重力的作用下自然滴完。向滤液中加入500μL PBS,收集到含有ssDNA文库的溶液,用于下一轮的筛选。
S5.ssDNA文库的反复筛选
将S4中得到的ssDNA文库代替S2中的随机ssDNA文库,重复S2–S4所示的SELEX筛选、PCR扩增及ssDNA文库的制取过程9次。
S6.阴性筛选
将S5的第二轮及第二轮以后筛选得到的ssDNA文库溶解、92℃恒温水浴、冰浴后,与草鱼正常细胞在冰上孵育1h,孵育完成后离心收集上清溶液;然后将此上清溶液与草鱼正常细胞进行冰浴结合;孵育结合完成后,收集上清,此时,收集到的上清即为经过阴性筛选的ssDNA文库。
本步骤采用草鱼正常细胞为对照,将S5后筛选得到的ssDNA文库进行阴性筛选以提高ssDNA文库的筛选效率。
S7.9轮筛选
将S6中收集到的上清液,经过S3的PCR扩增和S4的ssDNA文库制备后,依次重复进行S6、S2、S3和S4的过程,利用流式细胞仪检测所得ssDNA文库对草鱼GCRVⅠ型病毒感染细胞识别能力的变化情况,重复进行9轮筛选,所得到的ssDNA文库对草鱼GCRVⅠ型病毒感染细胞识别能力达到最强。将所得扩增产物经克隆测序分析后,最终得到可用于检测草鱼GCRVⅠ型病毒感染细胞的ssDNA核酸适配体,其核苷酸序列为:
TTCGGCGGATCTAACTTTGTCGTGGGAAACGGGGCGGACTTCACACTCGC(SEQ ID NO.1),
或,
GTCTGAAGTAGACGCAGGAGTTCGGCGGATCTAACTTTGTCGTGGGAAACGGGGCGGACTTCACACTCGCAGTCACACCTGAGTAAGCGT(SEQ ID NO.2)。
利用MFOLD软件(http://mfold.rna.albany.edu/?q=mfold/DNA-Folding-Form)在线预测核苷酸序列为SEQ ID NO.2的核酸适配体的二级结构,预测结果如图1所示,核酸适配体形成特殊的茎环结构和发卡结构。
实施例2
2.1主要仪器
Attune NxT流式细胞仪(赛默飞世尔科技)、FV3000激光扫描共聚焦显微镜(奥林巴斯)。
2.2实验操作
利用羟基荧光素(FAM)分别标记实施例1制备的SEQ ID NO.2核酸适配体和随机ssDNA文库Library50。
试验组:将0.1nmol FAM标记的SEQ ID NO.2核酸适配体溶解在500μL PBS中,92℃恒温水浴5min,然后迅速插入冰中,冰浴10min,将处理后的SEQ ID NO.2核酸适配体与草鱼GCRVⅠ型病毒感染细胞在冰上孵育1h。孵育结合完成后,离心移除上清。
对照组:将0.1nmol FAM标记的随机ssDNA文库Library50溶解在500μL PBS中,92℃恒温水浴5min,然后迅速插入冰中,冰浴10min,将处理后的随机ssDNA文库Library50与草鱼GCRVⅠ型病毒感染细胞在冰上孵育1h。孵育结合完成后,离心移除上清。
分别利用流式细胞仪和激光扫描共聚焦显微镜检测试验组和对照组经过孵育所得到的细胞沉淀物。
2.3实验结果
流式细胞仪的检测结果如图2所示,测得与试验组对应的荧光值明显高于对照组。激光扫描共聚焦显微镜的检测结果如图3所示,对照组几乎观察不到明显的荧光效果,相对而言,实验组所对应的荧光效果明显,细胞样品发出强烈的绿光。
流式细胞仪和激光扫描共聚焦显微镜的测试结果说明,与随机ssDNA文库Library50相比,FAM标记的SEQ ID NO.2核酸适配体对草鱼GCRVⅠ型病毒感染细胞具有更高的亲和力和特异性。
以上实施例仅用以说明本发明的技术方案而非对本发明保护范围的限制,尽管参照较佳实施例对本发明作了详细说明,本领域的普通技术人员应当理解,可以对本发明的技术方案进行修改或者等同替换,而不脱离本发明技术方案的实质和范围。
SEQUENCE LISTING
<110> 广西科学院
<120> 抗草鱼呼肠孤Ⅰ型病毒的核酸适配体及其构建方法和应用
<130>
<160> 4
<170> PatentIn version 3.5
<210> 1
<211> 50
<212> DNA
<213> 人工序列
<400> 1
ttcggcggat ctaactttgt cgtgggaaac ggggcggact tcacactcgc 50
<210> 2
<211> 90
<212> DNA
<213> 人工序列
<400> 2
gtctgaagta gacgcaggag ttcggcggat ctaactttgt cgtgggaaac ggggcggact 60
tcacactcgc agtcacacct gagtaagcgt 90
<210> 3
<211> 20
<212> DNA
<213> 人工序列
<400> 3
gtctgaagta gacgcaggag 20
<210> 4
<211> 20
<212> DNA
<213> 人工序列
<400> 4
acgcttactc aggtgtgact 20
- 还没有人留言评论。精彩留言会获得点赞!
- 一种基于CRISPR‑Cas9系统的基因编辑载体及其应用的制造方法与工艺
- 一种基于CRISPR/Cas9技术制备水稻OsPIL15突变体的方法及应用与流程
- 检测黄曲霉毒素M1的核酸适配体、传感器、试剂盒及应用的制造方法与工艺
- 一组肌酸激酶同工酶核酸适配体及其应用的制造方法与工艺
- 用于抑制KRAS靶基因mRNA表达的寡核酸分子及其成套组合物的制造方法与工艺
- 用于抑制EIF4E靶基因mRNA表达的寡核酸分子及其成套组合物的制造方法与工艺
- 用于抑制VEGFA靶基因mRNA表达的寡核酸分子及其成套组合物的制造方法与工艺
- 一种新型向导RNA表达盒及在CRISPR/Cas系统中的应用的制造方法与工艺
- 一种具备双单链末端PCR产物的获得方法及其检测方法与流程
- 一种通过生物技术扩增脱氧胸苷三磷酸的方法与流程