一种米曲霉及其在制备单宁酶中的应用的制作方法
本发明属于生物工程
技术领域:
,具体涉及一种米曲霉及其在制备单宁酶中的应用。
背景技术:
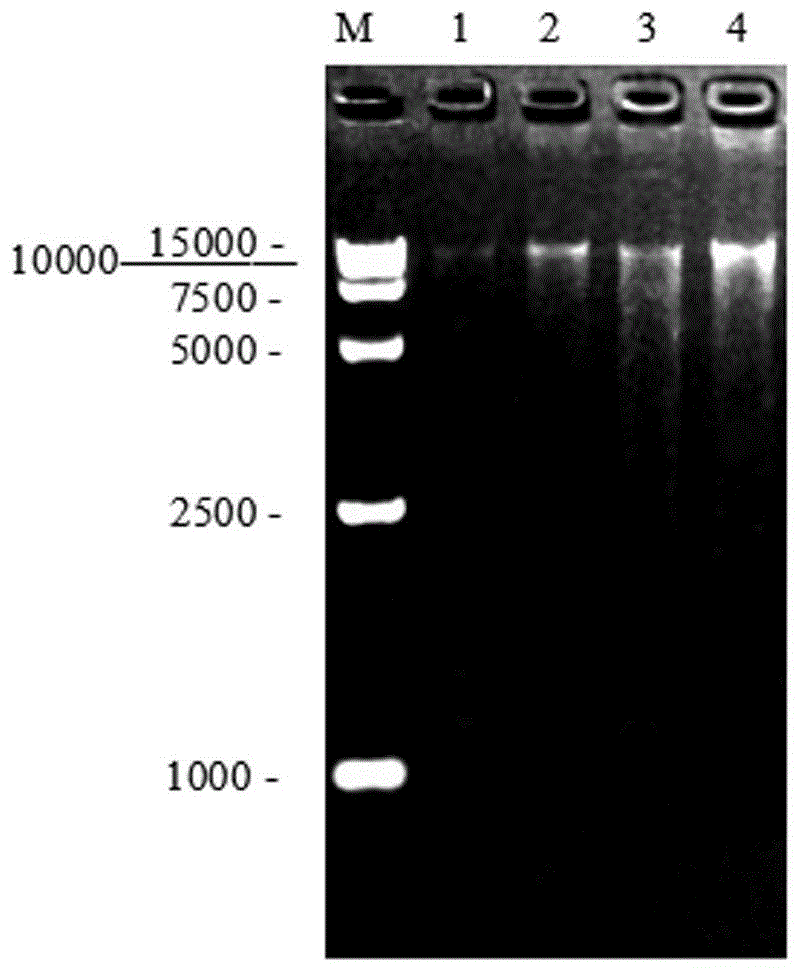
:没食子酸是一种重要的精细化学品,广泛应用在食品、化工、医药等领域。目前工业化制备没食子酸方法(酸法和碱法),虽然具有工艺成熟等优点,但也存在着不少缺点,如设备腐蚀较为严重,单宁水解不彻底,没食子酸收率交底,产品质量不稳定等。因此,近年来,国内外相继开展了单宁酶法的研究,单宁酶酶法制备没食子酸的反应时间短,单宁水解转化率高,生产成本低。但是,相关技术生产的单宁酶的酶活仍然较低、热稳定性也较差,且产量低。因而现有的单宁酶生产技术仍有待改进。技术实现要素:本发明旨在至少在一定程度上解决上述技术中的技术问题之一。在本发明的第一方面,本发明提出了一种米曲霉。根据本发明的实施例,所述米曲霉(aspergillusoryzaesp.fj0123),于2019年05月16日保藏于中国典型培养物保藏中心,保藏编号为cctccno:m2019357,分类命名为aspergillusoryzaesp.fj0123黑曲霉fj0123,保藏地址为:中国.武汉.武汉大学。根据本发明实施例的米曲霉可以利用其单宁酶基因制备出酶活高,酶活可达到560.0u/ml,热稳定好的单宁酶,在较高的温度范围内仍能保持酶活较长时间,并且该单宁酶的产量高,生物量可达到307mg/ml,使得利用该米曲霉制备出的单宁酶可较好的应用于工业化量产没食子酸。在本发明的第二方面,本发明提出了一种编码单宁酶的基因,从前述的米曲霉中进行pcr扩增以获得所述编码单宁酶的基因,所述基因的核酸序列如seqidno:1和seqidno:2所示。在本发明的第三方面,本发明提出了一种制备单宁酶的方法,包括以下步骤:1)、从前述米曲霉中提取基因;2)、将所述的基因进行pcr扩增,以获得前述的编码单宁酶的基因;3)将所述编码单宁酶的基因进行双酶切,以获得酶切片段;4)将所述酶切片段与经同样双酶切的毕赤酵母ppic9k连接,以获得表达载体ppic9k-tan1和ppic9k-tan2;5)将所述表达载体ppic9k-tan1和ppic9k-tan2分别转化毕赤酵母gs115,以获得两转化体;6)在适于单宁酶表达的条件下培养所述转化体,分离纯化,以获得单宁酶tan1和tan2。本发明基于pcr的方法分离克隆了米曲霉单宁酶tan1和tan2的基因,dna全序列分析结果表明,米曲霉单宁酶tan1和tan2的基因全长分别为1722bp和1767bp。根据本发明实施例,本发明通过设计了两对特异引物,将编码米曲霉单宁酶成熟蛋白的核苷酸序列(如seqidno:3所示)和的核苷酸序列(如seqidno:4所示)用pcr方法从米曲霉基因中扩增出来,克隆到毕赤酵母表达载体ppic9k,构建表达载体ppic9k-tan1和ppic9k-tan2,采用电击转化毕赤酵母gs115获得转化体,经甲醇发酵诱导表达出单宁酶tan1和tan2。利用本发明实施例的方法,能够制备出酶活高,热稳定好的单宁酶,并且酶产量高,使得制备出的单宁酶可较好的应用于工业化量产没食子酸。根据本发明的实施例,上述方法还可以包括如下附加技术特征至少之一:根据本发明的实施例,所述单宁酶tan1的氨基酸序列如seqidno:3所示,所述单宁酶tan2的氨基酸序列如seqidno:4所示。其中,该单宁酶tan1包括554个氨基酸,单宁酶tan2包括570个氨基酸。tan1酶活为560.0u/ml,tan1酶活为64u/ml,从而提供了一种酶活较高的单宁酶,且这两单宁酶具有较好的热稳定性,在较高的温度范围内仍保持酶活较长时间。根据本发明的实施例,所述步骤6)中,将所述转化体接种于罐上发酵培养基中发酵,待生物量达到180g/l后,加入甲醇进行诱导产酶,诱导时间为96h,以获得酶液。根据本发明的实施例,所述步骤6)的分离纯化为:采用1.6×20cm的阴离子交换柱纯化蛋白,用柠檬酸缓冲液平衡柱子,流速为1ml/min;加入酶液,流速为1ml/min,孵育20min;用含nacl的柠檬酸缓冲液洗脱,流速为1ml/min,收集并浓缩各步洗脱液,sds-page分析蛋白纯化情况。在本发明的第四方面中,本发明提出了前述方法制得的单宁酶。如前所述,根据本发明的实施例,可制备出酶活高,热稳定好的单宁酶,并且酶产量高,使得利用该米曲霉制备出的单宁酶可较好的应用于工业化量产没食子酸。在本发明的第五方面中,本发明提出了前述单宁酶在酶解塔拉生产没食子酸中的应用,将前述单宁酶用在塔拉粉中生产没食子酸,可在15min内生产大量的没食子酸,并且单宁酶tan1可多次酶解塔拉单宁生产没食子酸,说明该单宁酶可应用与工业化量产没食子酸。在本发明的第六方面中,本发明提出了前述单宁酶在茶饮料的巴氏消毒中的应用。试验说明,茶饮料经过巴氏消毒时,添加单宁酶tan1和tan2,均能改善茶饮料的感官品质,提高茶饮料贮藏性能。在本发明的第七方面中,本发明提出了前述单宁酶在葡萄酒、苹果酒和啤酒等饮料酒的巴氏消毒中的应用。试验说明,酒饮料经过巴氏消毒时,添加单宁酶tan1和tan2,均能改善酒饮料的感官品质,提高酒饮料贮藏性能。本发明的附加方面和优点将在下面的描述中部分给出,部分将从下面的描述中变得明显,或通过本发明的实践了解到。附图说明图1为提取米曲霉基因的结果;图2是从米曲霉基因中pcr扩增的单宁酶tan1基因结果;图3是从米曲霉基因中pcr扩增的单宁酶tan2基因结果;图4是表达载体ppic9k-tan1;图5是表达载体ppic9k-tan2;图6是表达载体ppic9k-tan1鉴定电泳结果;图7是表达载体ppic9k-tan2鉴定电泳结果;图8是线性化表达载体ppic9k-tan1鉴定电泳结果;图9是线性化表达载体ppic9k-tan2鉴定电泳结果;图10是整合了表达载体的转化体pcr鉴定电泳结果;图11是整合了表达载体的另一转化体pcr鉴定电泳结果;图12是转化体经甲醇诱导表达产单宁酶曲线图;图13是另一转化体经甲醇诱导表达产单宁酶曲线图;图14是单宁酶tan1分离纯化结果;图15是单宁酶tan2分离纯化结果;图16是单宁酶tan1热稳定性曲线图;图17是单宁酶tan2热稳定性曲线图;图18是单宁酶tan1的最适反应温度曲线图;图19是单宁酶tan1的最适ph曲线图;图20是单宁酶tan2的最适反应温度曲线图;图21是单宁酶tan2的最适ph曲线图;图22是单宁酶tan1酶法提取没食子酸结果图;图23是单宁酶tan2酶法提取没食子酸结果图;图24是单宁酶tan1多次酶解提取没食子酸结果图;图25是单宁酶tan2多次酶解提取没食子酸结果图。具体实施方式下面详细描述本发明的实施例,所述实施例的示例在附图中示出,其中自始至终相同或类似的标号表示相同或类似的元件或具有相同或类似功能的元件。下面通过参考附图描述的实施例是示例性的,旨在用于解释本发明,而不能理解为对本发明的限制。下文的公开提供了许多不同的实施例或例子用来实现本发明的不同实施方式。为了简化本发明的公开,下文中对特定实施例或示例进行描述。当然,他们仅仅为示例,并且目的不在于限制本发明。此外,本发明提供的各种特定工艺和材料的例子,本领域普通技术人员可以意识到其他工艺的可应用性和/或其他材料的使用。除非另有说明,本发明的实施将采用本领域技术人员的能力范围之内的化学、分子生物学等领域的传统技术。另外,除非另有说明,在本文中,核酸以5’至3’的方向从左向右书写,氨基酸序列则以氨基端到羧基端的方向从左向右书写。下面通过说明性的具体实施例对本发明进行描述,这些实施例并不以任何方式限制本发明的范围。特别说明的是:本发明所用到的试剂除特别说明外均有市售。实施例1米曲霉dna提取米曲霉dna提取pda培养基:去皮土豆200g,用蒸馏水煮至软而不烂后用八层纱布过滤取汁,加入20g葡萄糖(加入20g琼脂为固体培养基),定容至1l,分装至试管后121℃高压蒸汽灭菌20min,摆斜面冷却备用。用plantgenomicdnaextractionkit试剂盒(购自杭州博日科技有限公司)提取米曲霉基因组dna。1)从厦门红树林采集土壤样品,通过平板初筛,固体发酵产酶复筛,根据酶活筛选,获得稳定传代菌株,对其进行菌种鉴定后,保藏于中国典型培养物保藏中心,保藏编号为cctccno:m2019357,分类命名为aspergillusoryzaesp.fj0123米曲霉fj0123。2)将米曲霉(aspergillusoryzaesp.fj0123)接种于固体pda培养基斜面上,30℃恒温培养4-5天。3)将斜面上的孢子用无菌生理盐水洗下,用无菌玻璃珠打散,再用蒸馏水调整其od600为2.0,接种于带有玻璃珠的液体pda培养基中,30℃、180r/min培养24h。4)抽滤取菌丝,用去离子水清洗三次后,将菌丝体置于预冷的研钵中,边添加液氮边研磨,直至研磨成细小的白色粉末。5)用plantgenomicdnaextractionkit试剂盒(购自杭州博日科技有限公司)提取米曲霉基因组dna,提取步骤按照plantgenomicdnaextractionkit试剂盒提供的说明书进行。提取完后,用1%琼脂糖凝胶电泳检测总dna,并将米曲霉基因组dna保存于-20℃环境备用,结果如图1所示,1-4泳道是提取的4管米曲霉基因组dna,都达到15000bp,且条带单一,提取效果理想,可用于后续实验。实施例2单宁酶基因的pcr扩增根据米曲霉单宁酶基因编码的成熟蛋白酶两端序列和毕赤酵母表达载体ppic9k(购自invitrogen公司)的多酶切位点,合成两对引物,序列如下:tan1-f:5’-cgccctagggctagccttagcgatgtttgcac-3’(单下划线部分为avrii酶切序列)(seqidno:5)tan1-r:5’-atttgcggccgcttagtatagaggaatgcgataggcatcaaattc-3’(单下划线部分为noti酶切序列)(seqidno:6)tan2-f:5’-cgccctagggcttcttttaccgatgtgtgcacc-3’(单下划线部分为avrii酶切序列)(seqidno:7)tan2-r:5’-atttgcggccgcctagtatacagggaccttgaaggctg-3’(单下划线部分为noti酶切序列);(seqidno:8)以实施例1获取的米曲霉基因组dna作为模板,对单宁酶基因进行扩增。反应总体积为50μl,在0.2mlpcr管中加入下列成分:米曲霉基因组dna1μl上游引物tan1-f1μl下游引物tan1-r1μl无菌水32.5μlprimerstarenzyme0.5μl5×primestarbuffer(mg2+plus)10μl10mmol/l的4种dntps混合液4μl米曲霉基因组dna1μl上游引物tan2-f1μl下游引物tan2-r1μl无菌水32.5μlprimerstarenzyme0.5μl5×primestarbuffer(mg2+plus)10μl10mmol/l的4种dntps混合液4μl混匀,瞬时离心,反应条件为:95℃变性5min,然后94℃变性55sec,56℃退火45sec,72℃延伸2min,30个循环后72℃保温10min,4℃保存。扩增后进行琼脂糖凝胶电泳验证,电泳条件:电压:1520v,电流:110ma,时间25min,50μl上样量。如图2和图3所示,对tan1和tan2去前导肽同时添加酶切位点序列后,所得到的tan1和tan2序列大约有1500bp左右,图中除了marker泳道,两种单宁酶基因的其他各泳道都为单一条带,说明tan1和tan2均被扩增成功,可用于后续实验。实施例3两种单宁酶表达载体ppic9k-tan构建1)米曲霉单宁酶基因双酶切及回收用小量dna片段快速胶回收试剂盒(购自takara公司),操作按产品说明书提供的步骤进行回收实施例2pcr扩增的两种米曲霉单宁酶基因片段。根据设计的上下游引物所带的酶切位点,选用avrⅱ和notⅰ对所回收纯化的单宁酶基因pcr产物进行双酶切,20μl酶切体系,如下:avrⅱ1μl无菌水1μlnotⅰ1μl回收纯化pcr产物15μl10×hbuffer2μl混匀,离心,37℃酶切12h。酶切产物经1.0%琼脂糖凝胶电泳后,观察结果。在紫外灯下,用干净的刀片切下目的条带,放入1.5ml离心管中。双酶切产物的回收与纯化:用小量dna片段快速胶回收试剂盒(购自takara公司),操作按产品说明书提供的步骤进行。2)毕赤酵母表达载体ppic9k双酶切及其回收根据构建表达载体时选用的酶切位点avrⅱ和notⅰ,对ppic9k进行双酶切,20μl酶切体系,如下:无菌水1μlavrⅱ1μlnotⅰ1μlppic9k15μl10×hbuffer2μl混匀,离心,37℃酶切12h。酶切产物经1.0%琼脂糖凝胶电泳后,观察结果:在紫外灯下,用干净的刀片切下目的条带,放入1.5ml离心管中,按照小量dna片段快速胶回收试剂盒产品说明书提供的步骤进行双酶切产物的回收与纯化。3)酶切片段与表达载体的连接将经avrⅱ和notⅰ双酶切的米曲霉单宁酶基因,插入到经同样双酶切的表达载体ppic9k中,构建成表达载体ppic9k-tan1和ppic9k-tan2。连接体系如下:混匀,离心,16℃连接反应16h;得如图4和图5所示的表达载体ppic9k-tan1和ppic9k-tan2。4)大肠杆菌dh5α化学转化感受态细胞的制备(用takara公司的试剂盒)从lb平板(在胰蛋白胨10g、酵母提取物5g、氯化钠10g及琼脂粉15g中,加入蒸馏水900ml,充分溶解后,定容至1l,121℃高压灭菌20min)上挑取新活化的e.colidh5α单菌落,接种于5mllb液体培养基(在胰蛋白胨10g、酵母提取物5g、氯化钠10g中,加入蒸馏水900ml,充分溶解后,定容至1l,121℃高压灭菌20min)中,37℃振荡培养12h。将上述培养物以1:100的比例接种于100mllb液体培养基中,37℃振荡培养至od600=0.5左右。将菌液转入4℃预冷的离心管中,冰上放置10min,4000rpm4℃离心10min,弃去上清。用100μl预冷的solutiona溶液轻轻悬浮细胞,冰上放置5min,4000rpm4℃离心10min。弃去上清,用100μl预冷的solutionb溶液轻轻悬浮细胞,冰上放置30min。4000rpm4℃离心10min,即成感受态。冰上将感受态细胞分装成50μl/管,-80℃保存。5)连接产物转入大肠杆菌感受态细胞dh5α从-80℃冰箱中取上述的感受态细胞dh5α,迅速冰浴解冻。取连接产物(表达载体ppic9k-tan1和ppic9k-tan2)加入大肠杆菌感受态细胞dh5α中,冰浴30min,42℃水浴热击90s,取出后立即冰浴2min,加入1mllb液体培养基,37℃培养过夜。挑取阳性单菌落接种到氨苄青霉素抗性lb培养基中,37℃、180rpm过夜培养,通过菌液pcr验证阳性单菌落和测序分析表明克隆的目的基因为目的基因。结果如图6和图7所示,从ppic9k-tan1的大肠杆菌菌液pcr电泳图中可知,菌落1、2、4、8和9能被克隆出2000bp的目的条带,为阳性菌落,5个阳性菌落中tan1成功的插入ppic9k载体中,表达载体ppic9k-tan1构建成功。其他菌落为阴性菌落,阴性菌落中tan1未能与ppic9k连接在一起。从ppic9k-tan2的大肠杆菌菌液pcr电泳图中可知,菌落1-5和7-9能被克隆出2000bp的目的条带,为阳性菌落,8个阳性菌落中tan2成功的插入ppic9k载体中,表达载体ppic9k-tan2构建成功。其他菌落为阴性菌落,阴性菌落中tan2未能与ppic9k连接在一起。测序结果显示两种单宁酶基因均成功的插入ppic9k载体中,可用于后续实验。实施例4表达载体ppic9k-tan1和ppic9k-tan2转化毕赤酵母gs1151)单酶切线性化表达载体ppic9k-tan1和ppic9k-tan2用限制性核酸内切酶salⅰ对表达载体ppic9k-tan1单酶切线性化,体系如下:无菌水7ul10×hbuffer2ul表达载体ppic9k-tan1(300ng/μl)10ulsalⅰ1ul混匀,瞬时离心,反应条件:37℃反应1h。反应结束后,进行琼脂糖凝胶电泳进行分析鉴定,结果如图8所示,salⅰ对表达载体ppic9k-tan1单酶切线性化电泳图中,泳道1是未线性化的ppic9k-tan1,因提取的过程中对大肠杆菌基因组dna去除不完全,故有两条带,第二条带为环状ppic9k-tan1,泳道2和3是被sal1线性化后的ppic9k-tan1,第一条带和泳道1中的第一条带一样都是未被除净的大肠杆菌基因组dna,第二条带为线性化之后的ppic9k-tan1,ppic9k-tan1线性化成功。用限制性内切酶pmei对表达载体ppic9k-tan2单酶切线性化,体系如下:cutsmartbuffer2ul表达载体ppic9k-tan2(200ng/μl)15ulpmeⅰ1ul混匀,瞬时离心,反应条件:37℃反应1h。反应结束后,进行琼脂糖凝胶电泳进行分析鉴定,结果如图9所示,pmei对表达载体ppic9k-tan2单酶切线性化电泳图中,泳道1是未线性化的ppic9k-tan2,泳道2是被pmei线性化后的ppic9k-tan2,单一条带显示线性化比较完全,因为环状dna比线装dna电泳时,移动速度快,线性化后的ppic9k-tan2条带较未线性化的处于电泳图靠上的位置,ppic9k-tan2线性化成功。2)毕赤酵母gs115(购自北京华越洋生物科技有限公司)感受态细胞制备:将存放于-80℃保种的gs115菌液在ypd平板上划线,在30℃培养箱中倒置培养2-3天。从平板上挑取单菌落置于10mlypd液体培养基中,30℃,200rpm培养18-20h。0.1%接种量接种至100mlypd液体培养基中,30℃,200rpm培养过夜,至od600达到1.3-1.5,取出置于冰上10min,使之完全冷却至0℃。在4℃,3000rpm条件下离心5min,将上清倒掉。用40ml预冷的无菌水轻轻重悬菌体,在4℃,3000rpm条件下离心5min,将上清倒掉。40ml预冷的无菌水:先加10ml吸打混匀,再补足至40ml。用20ml预冷的无菌水轻轻重悬菌体,在4℃,3000rpm条件下离心5min,将上清倒掉。20ml预冷的无菌水:先加10ml吸打混匀,再加10ml。用1ml预冷的1mol/ld-山梨醇溶液轻轻重悬菌体,在4℃,3000rpm条件下离心5min,将上清倒掉。1ml预冷的无菌水:每个50ml离心管加1ml,分装到2个1.5mlep管中。用300ul预冷的1mol/ld-山梨醇溶液轻轻重悬菌体,置于冰上,当天使用。300ul预冷的1mol/ld-山梨醇溶液:先加300ul预冷的1mol/ld-山梨醇溶液轻轻重悬菌体,加入第二管中,吸打混匀,不要有气泡。3)线性化表达载体ppic9k-tan1和ppic9k-tan2电转化毕赤酵母gs115感受态细胞:根据异源表达载体电击转化毕赤酵母gs115感受态细胞按照说明书将线性化的含有目的基因的9k表达载体转入毕赤酵母感受态细胞中;然后在md平板上,30℃培养箱中倒置培养2-3天,观察单菌落生长情况。4)用g418抗性平板筛选阳性转化子:从md平板上挑选单菌落点于ypd(已加入g418抗性)平板上,平板底部用直尺划线,分成不同的区域以方便点板。放置在30℃恒温培养箱倒置培养2天。5)挑选阳性转化子于试管培养并进行破壁菌液pcr并送测序:将挑选的阳性转化子放入ypd液体试管(5-7ml)中,培养18h。做破壁pcr鉴定,送测序鉴定。破壁菌液pcr体系如下:无菌水4.5μl5’aox1μl3’aox1μl破壁菌液1μl预混estaqdnapolymerase7.5μlpcr反应条件:94℃℃变性5min;94℃变性30sec,62℃退火30sec,72℃延伸4min,28个循环;接下来72℃保温5min,4℃保存。反应结束后,进行琼脂糖凝胶电泳进行分析鉴定,结果如图10和图11所示,sal1-9k-tan1酵母破壁pcr电泳图中,泳道1-5为用通用引物进行酵母菌落pcr的结果,在2200bp处均只有一条带,这一条带中由两种序列构成,一种是2.2kb的aox1基因,另一种是序列tan1(1722bp)+492bp序列,因两种序列大小相同,用通用引物不能验证此酵母为阳性转化子。泳道6-10为用特异引物进行酵母菌落pcr的结果,酵母菌落6-10在1600bp处均有单一条带,证明此五个酵母菌落为阳性转化子。pme1-9k-tan2酵母破壁pcr电泳图中,泳道1-5为用通用引物进行酵母菌落pcr的结果,在2200bp处均只有一条带,这一条带中由两种序列构成,一种是2.2kb的aox1基因,另一种是序列tan2(1767bp)+492bp序列,因两种序列大小相同,用通用引物不能验证此酵母为阳性转化子。泳道6-10为用特异引物进行酵母菌落pcr的结果,酵母菌落7-10在1600bp处均有单一条带,证明此四个酵母菌落为阳性转化子。琼脂糖凝胶电泳检测显示图,pcr扩增结果与预期大小相符。实施例5两株转化体在发酵罐上诱导表达出重组米曲霉单宁酶tan1和tan2罐上发酵培养基:85%磷酸80.1ml,caso42.79g,k2so454.6g,koh12.39g,mgso4·7h2o44.7g,甘油120g,酵母膏15g,蛋白胨15g,消泡油3ml,溶解后定容至3l,注入发酵罐中,121℃高压灭菌20min,冷却后备用。(1)将两株转化体分别接种于ypd培养基中进行活化,30℃,200rpm培养16-18h。(2)将活化后的菌种按1%的接种量接种于ypd种子培养基内,30℃,200rpm培养16-18h。(3)按5%的接种量接种于3l罐上发酵培养基中进行发酵,待生物量达到180g/l后加入甲醇进行诱导产酶,诱导时间为96h。发酵完成后,离心获取酶液,得单宁酶tan1和tan2。(4)单宁酶活性测定1)取空白管、对照管、测试管,各加入0.25ml的底物没食子酸丙酯(0.01mol/l)。3)空白管中加入ph5.0的柠檬酸缓冲液0.25ml,测试管中加入经稀释一定倍数的重组单宁酶粗酶液0.25ml,对照管中加入经沸水浴灭活的重组单宁酶酶液0.25ml,所有试管30℃水浴5min。4)三支试管中均加入6.67g/l甲醇绕丹宁溶液0.3ml,置于30℃水浴5min。5)三支试管均加入0.7mol/lkoh0.2ml,置于30℃水浴5min。6)所有试管中加入蒸馏水4ml,振荡均匀后30℃水浴10min。7)所有试管在520nm处以蒸馏水为空白测吸光值,测三组平行,结果取算术平均值。8)将30℃条件下每分钟产生1μmol没食子酸所需要的酶量定义为一个酶活单位u。9)测定上述获得的两单宁酶tan1和tan2的酶活,结果如图12和图13所示,单宁酶tan1诱导发酵图,随着甲醇诱导发酵时间的延长,酵母的生物量逐渐稳步增加,酶活整体呈增长趋势,在诱导88h达到最高生物量307mg/ml,此时检测到的单宁酶活力也达到最高值560u/ml,直至发酵96h,单宁酶活力和生物量略有下降。从单宁酶tan2诱导发酵图中可知,随着甲醇诱导发酵时间的延长,酵母的生物量整体呈逐渐增长趋势,发酵前期,测得单宁酶活力缓慢增加;在发酵至40-64h时,单宁酶迅速地在发酵液中富集,最高生物量达到231mg/ml,最高单宁酶活力也达到值64u/ml;之后单宁酶活力和生物量开始减少。实施例6单宁酶分离纯化用在线软件为:https://web.expasy.org/compute_pi/分析单宁酶tan1和tan2等电点,s-tan1等电点为4.98,分子量为61.36kda,p-tan2等电点为4.65,分子量为62.29kda。配制10l,pka=7.0,ph=6.0的0.02mssc的缓冲液。采用1.6×20cm的阴离子交换柱纯化蛋白,用ssc缓冲液平衡柱子,流速为1ml/min,粗酶液上样量2.5ml,流速为1ml/min,孵育20min;配制0.05m,0.1m和2m的nacl的柠檬酸缓冲液洗脱进行洗脱,流速为1ml/min,收集各步洗脱液并浓缩洗脱液,sds-page分析纯化情况。结果如图14和图15所示,从tan1聚丙烯酰胺凝胶电泳图,相比于原酶液(泳道1)的两条蛋白质条带,纯化后酶液(泳道2)只有一条蛋白质条带,分子量在100kda左右,说明tan1纯化成功。从tan2聚丙烯酰胺凝胶电泳图,泳道1是tan2原酶液,泳道2是纯化后tan2酶液,tan2原酶液具有两条蛋白质条带,一条分子量在110kda左右,另一条在78kda左右。而纯化后的tan2酶液,只有一条分子量在110kda左右的蛋白质条带,杂带被去除,说明tan2纯化成功。实施例7两单宁酶热稳定性测定两单宁酶酶液在60℃、70℃、80℃条件下处理一段时间后,在30℃,ph=5.0条件下测定酶活力。以最高酶活力为100%,灭活10min的酶液为空白对照,温度稳定性结果见图16和图17。单宁酶tan1在80℃的半衰期为8min,在70℃处理26min后,依然保留50%以上的酶活力,60℃保温120min不会对单宁酶tan1活力造成显著性影响。单宁酶tan2在70℃的水浴环境中处理10min后,单宁酶活力剩余10%左右,而在60℃和50℃的半衰期分别为6min和20min。如此,因工业化量产没食子酸等物质反应的温度较高,且量产持续的时间较长,而本实施例中两单宁酶的热稳定性可满足工业化的量产没食子酸等物质。实施例8两单宁酶生物学性质最适反应温度:保持底物添加量为0.25ml,与0.25ml单宁酶酶液混匀,使反应体系为0.5ml,分别在30℃、40℃、50℃、60℃、70℃、80℃、90℃条件下反应5min,测定酶活力。以最高酶活力为100%,灭活10min的酶液为空白对照。结果如图18和图20所示,单宁酶tan1和tan2的最适反应温度分别为80℃和70℃。最适ph:将酶液用ph3.0、4.0、5.0、6.0、7.0、8.0、9.0、10.0的缓冲液稀释到合适的倍数后,取0.25ml与等体积底物混匀,在30℃下测定单宁酶活力。以最高酶活力为100%,不同ph条件下灭活10min的酶液为空白对照。结果如图19和图21所示,单宁酶tan1和tan2的最适反应ph分别为6和5。实施例9单宁酶tan1和tan2在酶解塔拉生产没食子酸中的应用用40ml蒸馏水在50℃的磁力搅拌器上溶解2g塔拉粉10min,然后向溶解液中加入1500u的单宁酶tan1或tan2,固定时间取样(5000rpm,1min离心,取上清液)检测没食子酸的生成量,此过程中维持反应温度为50℃。并用薄层检测浸提液中塔拉单宁和没食子酸的变化,展开剂:6%乙酸,显色剂:3%fecl3。结果如图22和图23所示,两种单宁酶水解塔拉单宁的薄层层析图。两张图中,1号:没食子酸和单宁酸的标品,上面和下面两条带分别为没食子酸和单宁酸的条带。2号为塔拉粉水溶液,可以看出塔拉粉水溶液中绝大部分是单宁酸,少量的没食子酸。3号为塔拉粉水溶液15min酶解液,可以看出在被单宁酶tan1或tan2酶解15min后,单宁酸的量明显减少,没食子酸大量生成。4号为塔拉粉水溶液120min酶解液,在塔拉粉水溶液被单宁酶tan1或tan2处理2h后,体系里面大部分都是没食子酸,只有少量未被水解的塔拉单宁。实施例10单宁酶tan1及tan2多次酶解塔拉生产没食子酸对比用40ml蒸馏水在50℃的磁力搅拌器上溶解2g塔拉粉10min,然后向溶解液中加入1500u的单宁酶,20min固定时间取样,并在取样后立即向反应液中加入2g塔拉粉,操作同上,整个过程一共添加5次塔拉粉,反应过程中维持反应温度为50℃。样品经5000rpm离心1min分离上清液和固体残渣,应用薄层色谱检测上清液和塔拉残渣中塔拉单宁消耗和没食子酸生成情况,展开剂:6%乙酸,显色剂:3%fecl3。结果如图24和图25所示,分别为单宁酶tan1和tan2水解塔拉单宁的薄层层析图。1号:没食子酸和单宁酸的标品,上面和下面两条带分别为没食子酸和单宁酸的条带。2号和3号分别为第一次酶解塔拉单宁后的上清液和残渣,可以看出tan1上清液中塔拉单宁全被水解成没食子酸;而tan2上清液中塔拉单并未完全水解,还有少量单宁存在。两个酶水解的固体残渣中已无塔拉单宁。4号和5号为第二次酶解塔拉单宁后的上清液和残渣,可以看出此时tan1上清液中塔拉单宁全被水解成没食子酸,因浓度较高条带比较靠下;而tan2上清液中塔拉单还未完全水解。两个酶水解的固体残渣中已无塔拉单宁。6号和7号为第三次酶解塔拉单宁后被稀释过的上清液和残渣,可以看出此时tan1上清液中塔拉单宁全被水解成没食子酸;而tan2由于过长时间的高温,酶活力下降不能完全水解单宁,上清中残余单宁含量上升。tan1水解残渣有明显的没食子酸条带,说明反应液中没食子酸浓度高,已经被析出,无单宁酸条带,说明塔拉单宁被全部水解;而tan2水解残渣有明显的没食子酸和单宁酸条带,说明反应液中没食子酸浓度高,已经被析出,但塔拉单宁未被完全水解。8号和9号为第四次酶解塔拉单宁后被稀释过的上清液和残渣,可以看出此时tan1上清液中塔拉单宁全被水解成没食子酸;而tan2上清中还有大量单宁残留。tan1水解残渣有明显的没食子酸条带,无单宁酸条带,说明塔拉单宁被全部水解;而tan2水解残渣有明显的没食子酸和单宁酸条带,说明反应液中没食子酸浓度高,已经被析出,但塔拉单宁未被完全水解。10号和11号为第五次酶解塔拉单宁后被稀释过的上清液和残渣,可以看出此时tan1上清液中塔拉单宁全被水解成没食子酸;而tan2上清中还有大量单宁残留。tan1水解残渣有明显的没食子酸条带且无单宁酸条带,说明塔拉单宁被全部水解;而tan2水解残渣有明显的没食子酸和单宁酸条带,塔拉单宁还未被完全水解。12号和13号为第六次酶解塔拉单宁后被稀释过的上清液和残渣,可以看出此时tan1上清液中塔拉单宁全被水解成没食子酸;而tan2上清中还有大量单宁残留。tan1水解残渣有明显的没食子酸条带且无单宁酸条带,说明塔拉单宁被全部水解;而tan2水解残渣有明显的没食子酸和单宁酸条带,且塔拉单宁残留量上升。以上结果表明热稳定较好的tan1在多次酶解塔拉生产没食子酸的应用上更具有优势。实施例11单宁酶tan1和tan1分别在普洱茶、绿茶、红茶、乌龙茶和铁观音茶饮料巴氏消毒中的应用单宁酶tan1和tan1分别用于普洱茶、绿茶、红茶、乌龙茶和铁观音茶饮料的巴氏消毒过程中,以处理饮料中出现絮状沉淀(茶乳酪),避免出现饮料“冷后浑”的现象。向未经灭菌的茶饮料1000ml分别加入500u的单宁酶tan1或tan1,65℃保持30min,后取样检测剩余酶活力,并将灭菌后的饮料在25℃的室温下连续放置30天,每天观察检测茶饮料感官品质,以未添加单宁酶巴氏消毒灭菌的茶饮料为对照。其结果发现经过持续30min,65℃处理之后:单宁酶tan1酶活力均剩余51-55%,经单宁酶tan1处理后的饮料,口感均更加柔和醇香,苦涩口感消失,其中红茶、普洱茶和铁观音效果最好,并且在室温放置30天,饮料一直处于澄清透明状态,口感保持醇香,未再出现苦涩感。对照组茶饮料均在冷却后出现浑浊,放置第一天后出现沉淀,喝起来生涩感明显,红茶苦味最明显。单宁酶tan1能改善茶饮料的感官品质,提高茶饮料贮藏性能。单宁酶tan2酶活力都还剩有9-12%,经单宁酶tan2处理后的饮料,口感均更加柔和醇香,苦涩厚重感消失,其中铁观音、普洱茶和红茶效果最好,并且在室温放置30天,饮料一直处于澄清透明状态,口感保持醇香,未再出现苦涩感。对照组茶饮料均在冷却后出现浑浊,放置第一天后出现沉淀,喝起来生涩感明显,红茶苦味最明显。实施例12单宁酶tan1和tan1分别在葡萄酒、苹果酒和啤酒的巴氏消毒中的应用单宁酶tan1和tan1分别用于葡萄酒、苹果酒和生啤酒的巴氏消毒过程中,以解决饮料酒中单宁和蛋白质形成的浑浊沉淀现象,从而改善其口感,使其保持澄清透明,延长其的保质期。向未经灭菌的葡萄酒、苹果酒和生啤酒1000ml分别加入500u的单宁酶tan1或tan1,65℃保持30min,后取样检测剩余酶活力,并对在25℃的室温下连续放置60天,每天观察检测经酶处理和灭菌后的的饮料酒感官品质,以未添加单宁酶巴氏消毒灭菌的饮料酒为对照。其结果发现经过持续30min,65℃处理之后:单宁酶tan1酶活力剩余36-41%,经单宁酶tan1处理后的葡萄酒、苹果酒和生啤酒,涩口感消失,口感协调性更佳,并且在室温放置60天,饮料酒一直处于澄清透明状态,口感保持醇香,未有苦涩感。单宁酶tan1能改善饮料酒的感官品质,提高饮料酒贮藏性能。单宁酶tan2酶活力剩余14-18%,经单宁酶tan2处理后的葡萄酒、苹果酒和生啤酒,涩口感消失,口感协调性更佳,并且在室温放置60天,饮料酒一直处于澄清透明状态,口感保持醇香,未出现苦涩感。单宁酶tan2能改善饮料酒的感官品质,提高饮料酒稳定性和透明度,增加其贮藏性能,延长保质期。在本说明书的描述中,参考术语“一个实施例”、“一些实施例”、“示例”、“具体示例”、或“一些示例”等的描述意指结合该实施例或示例描述的具体特征、结构、材料或者特点包含于本发明的至少一个实施例或示例中。在本说明书中,对上述术语的示意性表述不应理解为必须针对的是相同的实施例或示例。而且,描述的具体特征、结构、材料或者特点可以在任何的一个或多个实施例或示例中以合适的方式结合。此外,本领域的技术人员可以将本说明书中描述的不同实施例或示例进行接合和组合。尽管上面已经示出和描述了本发明的实施例,可以理解的是,上述实施例是示例性的,不能理解为对本发明的限制,本领域的普通技术人员在本发明的范围内可以对上述实施例进行变化、修改、替换和变型。序列表<110>集美大学<120>一种米曲霉及其在制备单宁酶中的应用<130>2019<160>8<170>siposequencelisting1.0<210>1<211>1722<212>dna<213>人工序列(artificialsequence)<400>1atgaggtcggcgctaaacatcctcgcgggagcagccactctcacagtggcacaggctgct60agccttagcgatgtttgcacgaacagccatgtcaagtcggctctgccctccattgacctc120atcaatggtcttgtaattgatccatcttctattaccaccaatgccgtctacaatgcttcc180acccccggaggtgactacttcccagcagcatccgcctatgatttctgcaacgtgactctc240acgtacgcccaccctggacgcaatgatcgagttcatcttaaattatggatgccagctcca300gaccagttccaaaaccggtggctatccactggtggaggtggttttgcgatcaatcacgac360gaacagcagcttccgggtggtgttcaatatggcgccgcggctggcatcactgacggaggt420ttcggcagcttttacacccagtttgaccaagtattcctgctggccaatggcacgatcaac480tatgaagcactttatatgtttggctaccaggcgcaccatgagctgagtgtcatcggaaag540gctttgacaaagaacttttatggcacgggagacgcaaaactgtatgcctattggcaggga600tgctccgagggtggtcgcgagggattcagccaggtgcagcgatttcaggagttcgacggc660gcagtgatcggcgcccctgcccttcggtatggccaacaacaggccaaccatctgtatgga720aacttggttgagcatacactcaagtactaccctccgccctgtgaattggagaagatcgtt780aatttgaccattacggcatgcgatcgcctggatggaaggtcggacggtgtggtgtcgcgg840accgacctgtgcaagctgcatttcaatatcaattcaacgattggagcaccctactcctgc900ccagcctcaacgacaacaactggtacaaccccggcgcagaacggaaccgtgtctgcgctc960ggtgcagcagcagccaggaagatgttagatggccttcgcacccttgacagccgtcgggcc1020tatatcttctaccagccttccgccaccttcgatgatgcgcaaacgaaatataaccccgaa1080accaagcaattcgaactcgaggtctctgcctatgccgccgaatggatcccccgcttcctg1140cagctacagaattacactctatcttccctggaaaacgtaacatacgacaccttaaaagac1200tggatggaacttggatggcagcgctacgaagacgtcctgcagaccacatggcccgattta1260actcctttccagtccgcgggtggaaaggtgcttcactaccatggtgaatcggatccaagt1320attccggctggatcctcggtacactatcatgagtctgttcgtaagactatgtaccccaac1380atgtctttcaacgaaagtaaccaggcgttgaatgagtggaaccgcttattcttggtcccc1440ggcgccgcgcactgcgcctccagtaccgatcaacccaatggccccttcccccaggcaact1500ctcaagacgctcatcgaatgggtcgaaaatagtattgttccagaaactttgaatggtacg1560gtgctggacggtgatcacaagggtgaacagcagcagatctgcgcttggcccttgcgtccc1620ttatggacagagaacggcaccgttatgaactgtgtgtacgaccaggcttccttggacacc1680tgggactatgaatttgatgcctatcgcattcctctatactaa1722<210>2<211>1767<212>dna<213>人工序列(artificialsequence)<400>2atgcgccaacactcgcgcatggccgttgctgctttggcagcaggagcgaacgcagcttct60tttaccgatgtgtgcaccgtgtctaacgtgaaggctgcattgcctgccaacggaactctg120ctcggaatcagcatgcttccgtccgccgtcacggccaaccctctctacaaccagtcggct180ggcatgggtagcaccactacctatgactactgcaatgtgactgtcgcctacacgcatacc240ggcaagggtgataaagtggtcatcaagtacgcattccccaagccctccgactacgagaac300cgtttctacgttgctggtggtggtggcttttccctctctagcgatgctaccggaggtctc360gcctatggcgctgtgggaggtgccaccgatgctggatacgacgcattcgataacagctac420gacgaggtagtcctctacggaaacggaaccattaactgggacgccacatacatgttcgca480taccaggcactgggagagatgacccggatcggaaagtacatcaccaagggcttttatggc540cagtccagcgacagcaaggtctacacctactacgagggttgctccgatggaggacgtgag600ggtatgagtcaagtccagcgctggggtgaggagtatgacggtgcgattactggtgccccg660gctttccgtttcgctcagcaacaggttcaccatgtgttctcgtccgaagtggagcaaact720ctggactactacccgcctccatgtgagttgaagaagatcgtgaacgccaccattgctgct780tgcgacccgcttgatggaagaaccgacggtgttgtgtcccggacggatctttgcaagctt840aacttcaatttgacctctatcatcggtgagccttactactgtgctgcgggaactagcact900tcgcttggtttcggcttcagcaatggcaagcgcagcaatgtcaagcgtcaggccgagggc960agcaccaccagctaccagcccgcccagaacggcacggtcaccgcacgtggtgtagctgtc1020gcccaggccatctacgatggtctccacaacagcaagggcgagcgcgcgtacctctcctgg1080cagattgcctctgagctgagcgatgctgagaccgagtacaactctgacactggcaagtgg1140gagctcaacatcccgtcgaccggtggtgagtacgtcaccaagttcattcagctcctgaac1200ctcgacaacctttcggatctgaacaacgtgacctacgacaccctggtcgactggatgaac1260actggtatggtgcgctacatggacagccttcagaccacccttcccgatctgactcccttc1320caatcgtccggcggaaagctgctgcactaccacggtgaatctgaccccagtatccccgct1380gcctcctcggtccactactggcaggcggttcgttccgtcatgtacggcgacaagacggaa1440gaggaggccctggaggctctcgaggactggtaccagttctacctaatccccggtgccgcc1500cactgcggaaccaactctctccagcccggaccttaccctgagaacaacatggagattatg1560atcgactgggtcgagaacggcaacaagccgtcccgtctcaatgccactgtttcttcgggt1620acctacgccggcgagacccagatgctttgccagtggcccaagcgtcctctctggcgcggc1680aactccagcttcgactgtgtcaacgacgagaagtcgattgacagctggacctacgagttc1740ccagccttcaaggtccctgtatactag1767<210>3<211>554<212>prt<213>人工序列(artificialsequence)<400>3alaserleuseraspvalcysthrasnserhisvallysseralaleu151015proserileaspleuileasnglyleuvalileaspproserserile202530thrthrasnalavaltyrasnalaserthrproglyglyasptyrphe354045proalaalaseralatyraspphecysasnvalthrleuthrtyrala505560hisproglyargasnaspargvalhisleulysleutrpmetproala65707580proaspglnpheglnasnargtrpleuserthrglyglyglyglyphe859095alaileasnhisaspgluglnglnleuproglyglyvalglntyrgly100105110alaalaalaglyilethraspglyglypheglyserphetyrthrgln115120125pheaspglnvalpheleuleualaasnglythrileasntyrgluala130135140leutyrmetpheglytyrglnalahishisgluleuservalilegly145150155160lysalaleuthrlysasnphetyrglythrglyaspalalysleutyr165170175alatyrtrpglnglycyssergluglyglyarggluglyphesergln180185190valglnargpheglnglupheaspglyalavalileglyalaproala195200205leuargtyrglyglnglnglnalaasnhisleutyrglyasnleuval210215220gluhisthrleulystyrtyrproproprocysgluleuglulysile225230235240valasnleuthrilethralacysaspargleuaspglyargserasp245250255glyvalvalserargthraspleucyslysleuhispheasnileasn260265270serthrileglyalaprotyrsercysproalaserthrthrthrthr275280285glythrthrproalaglnasnglythrvalseralaleuglyalaala290295300alaalaarglysmetleuaspglyleuargthrleuaspserargarg305310315320alatyrilephetyrglnproseralathrpheaspaspalaglnthr325330335lystyrasnprogluthrlysglnphegluleugluvalseralatyr340345350alaalaglutrpileproargpheleuglnleuglnasntyrthrleu355360365serserleugluasnvalthrtyraspthrleulysasptrpmetglu370375380leuglytrpglnargtyrgluaspvalleuglnthrthrtrpproasp385390395400leuthrpropheglnseralaglyglylysvalleuhistyrhisgly405410415gluseraspproserileproalaglyserservalhistyrhisglu420425430servalarglysthrmettyrproasnmetserpheasngluserasn435440445glnalaleuasnglutrpasnargleupheleuvalproglyalaala450455460hiscysalaserserthraspglnproasnglypropheproglnala465470475480thrleulysthrleuileglutrpvalgluasnserilevalproglu485490495thrleuasnglythrvalleuaspglyasphislysglygluglngln500505510glnilecysalatrpproleuargproleutrpthrgluasnglythr515520525valmetasncysvaltyraspglnalaserleuaspthrtrpasptyr530535540glupheaspalatyrargileproleutyr545550<210>4<211>570<212>prt<213>人工序列(artificialsequence)<400>4alaserphethraspvalcysthrvalserasnvallysalaalaleu151015proalaasnglythrleuleuglyilesermetleuproseralaval202530thralaasnproleutyrasnglnseralaglymetglyserthrthr354045thrtyrasptyrcysasnvalthrvalalatyrthrhisthrglylys505560glyasplysvalvalilelystyralapheprolysproserasptyr65707580gluasnargphetyrvalalaglyglyglyglypheserleuserser859095aspalathrglyglyleualatyrglyalavalglyglyalathrasp100105110alaglytyraspalapheaspasnsertyraspgluvalvalleutyr115120125glyasnglythrileasntrpaspalathrtyrmetphealatyrgln130135140alaleuglyglumetthrargileglylystyrilethrlysglyphe145150155160tyrglyglnserseraspserlysvaltyrthrtyrtyrgluglycys165170175seraspglyglyarggluglymetserglnvalglnargtrpglyglu180185190glutyraspglyalailethrglyalaproalapheargphealagln195200205glnglnvalhishisvalphesersergluvalgluglnthrleuasp210215220tyrtyrproproprocysgluleulyslysilevalasnalathrile225230235240alaalacysaspproleuaspglyargthraspglyvalvalserarg245250255thraspleucyslysleuasnpheasnleuthrserileileglyglu260265270protyrtyrcysalaalaglythrserthrserleuglypheglyphe275280285serasnglylysargserasnvallysargglnalagluglyserthr290295300thrsertyrglnproalaglnasnglythrvalthralaargglyval305310315320alavalalaglnalailetyraspglyleuhisasnserlysglyglu325330335argalatyrleusertrpglnilealasergluleuseraspalaglu340345350thrglutyrasnseraspthrglylystrpgluleuasnileproser355360365thrglyglyglutyrvalthrlyspheileglnleuleuasnleuasp370375380asnleuseraspleuasnasnvalthrtyraspthrleuvalasptrp385390395400metasnthrglymetvalargtyrmetaspserleuglnthrthrleu405410415proaspleuthrpropheglnserserglyglylysleuleuhistyr420425430hisglygluseraspproserileproalaalaserservalhistyr435440445trpglnalavalargservalmettyrglyasplysthrglugluglu450455460alaleuglualaleugluasptrptyrglnphetyrleuileprogly465470475480alaalahiscysglythrasnserleuglnproglyprotyrproglu485490495asnasnmetgluilemetileasptrpvalgluasnglyasnlyspro500505510serargleuasnalathrvalserserglythrtyralaglygluthr515520525glnmetleucysglntrpprolysargproleutrpargglyasnser530535540serpheaspcysvalasnaspglulysserileaspsertrpthrtyr545550555560glupheproalaphelysvalprovaltyr565570<210>5<211>32<212>dna<213>人工序列(artificialsequence)<400>5cgccctagggctagccttagcgatgtttgcac32<210>6<211>45<212>dna<213>人工序列(artificialsequence)<400>6atttgcggccgcttagtatagaggaatgcgataggcatcaaattc45<210>7<211>45<212>dna<213>人工序列(artificialsequence)<400>7atttgcggccgcttagtatagaggaatgcgataggcatcaaattc45<210>8<211>38<212>dna<213>人工序列(artificialsequence)<400>8atttgcggccgcctagtatacagggaccttgaaggctg38当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1