CRISPR/Sa-SauriCas9基因编辑系统及其应用的制作方法
本发明属于基因编辑技术领域,具体涉及一种能在细胞中进行基因编辑的crispr/sa-sauricas9系统以及其相关应用。
背景技术:
crispr/cas9是细菌和古细菌为抵御外源病毒或质粒入侵而进化的一种获得性免疫系统。在crispr/cas9系统中,crrna(crispr-derivedrna)、tracrrna(trans-activatingrna)以及cas9蛋白形成复合体后,识别靶位点的pam(protospaceradjacentmotif)序列,crrna会与靶dna序列形成互补结构,cas9蛋白行使切割dna的功能,使dna发生断裂损伤。其中,tracrrna和crrna通过连接序列可以融合成为单链指导rna(singleguiderna,sgrna)。当dna发生断裂损伤后,细胞内的两种主要dna损伤修复机制负责修复:非同源末端连接(non-homologousend-joining,nhej)和同源重组(homologousrecombination,hr)。nhej修复的结果会引起碱基的缺失或插入,可以进行基因敲除;在提供同源模板的情况下,利用hr修复可以进行基因的定点插入和碱基的精确替换。
除了基础科研外,crispr/cas9还具有广泛的临床应用前景。利用crispr/cas9系统做基因治疗时,需要把cas9和sgrna导入到体内。目前做基因治疗最有效的递送载体是aav病毒。但是aav病毒包装的dna一般不超过4.5kb。spcas9因为pam序列简单(识别ngg)和活性高而得到广泛应用。但是spcas9蛋白有1367个氨基酸,加上sgrna和启动子,无法有效的包装到aav病毒中,限制了其在临床中的应用。为了克服这个问题,几个小的cas9被发明出来了,包括sacas9(pam序列为nngrrt);st1cas9(pam序列为nnagaw);nmcas9(pam序列为nnnngatt);nme2cas9(pam序列为nnnncc);cjcas9(pam序列为nnnnryac),但是这些cas9或者pam序列复杂(在基因组中可靶向的dna序列少),或者编辑效率低,难以广泛应用。寻找cas9蛋白小,pam序列简单crispr/cas9系统是解决上述问题的希望所在。
技术实现要素:
针对上述问题,本发明目的在于提供一种编辑活性高、cas9蛋白小、pam序列简单的crispr/cas9新的基因编辑系统及其应用。
本发明提供的crispr/cas9基因编辑系统,为sa-sauricas9蛋白与sgrna形成的复合体,记为crispr/sa-sauricas9基因编辑系统(即sa-sauricas9蛋白,它和singleguiderna(sgrna)共同作用实现基因编辑);能精确靶向dna序列,并产生切割,使dna发生双链断裂损伤;所述基因编辑为在细胞中或体外进行基因编辑;sa-sauricas9蛋白小,为1053个氨基酸,识别的pam序列简单(nngg),所述sa-sauricas9蛋白具有seqidno:1所示的氨基酸序列,或与seqidno:1所示的氨基酸序列至少80%相同的氨基酸序列;所述sgrna具有seqidno:2所示的核苷酸序列,或者基于seqidno:2改造的sgrna序列。
本发明中,所述细胞包括真核细胞和原核细胞;所述真核生物细胞包括哺乳动物细胞和植物细胞。
本发明中,所述哺乳动物细胞包括中国仓鼠卵巢细胞、幼仓鼠肾细胞、小鼠sertoli细胞、小鼠乳腺瘤细胞、buffalo大鼠肝细胞、大鼠肝瘤细胞、由sv40转化的猴肾cvi系、猴肾细胞、犬肾细胞、人宫颈癌细胞、人肺细胞、人肝细胞、hih/3t3细胞、人u2-os骨肉瘤细胞、人a549细胞、人k562细胞、人hek293细胞、人hek293t细胞、人hct116细胞或人mcf-7细胞或tri细胞。
本发明中,所述crispr/cas9系统中sa-sauricas9是融合蛋白,是将sacas9的pi结构域替换为sauricas9的pi结构域,sauricas9为staphylococcusauriculariscas9。sa-sauricas9融合蛋白和singleguiderna(sgrna)共同作用实现基因编辑。
本发明中,所述sauricas9蛋白属于耳氏葡萄球菌属(staphylococcusauricularis),所述sauricas9蛋白的uniprot的检索号为a0a2t4m4r5。
本发明中,所述sa-sauricas9蛋白包括无切割活性或仅具有单链切割活性或具有双链切割活性的sa-sauricas9蛋白。
本发明中,所述精确定位dna序列包括sgrna中的5’端20bp或21bp序列与靶向dna序列能形成碱基互补配对结构。
本发明中,所述精确定位靶向dna序列包括sa-sauricas9蛋白和sgrna复合体识别靶向dna序列上的pam序列。
本发明中,所述pam序列为nngg,所述靶向dna序列为:
nnnnnnnnnnnnnnnnnnnnnnngg(seqidno:3)。
本发明中,所述sa-sauricas9蛋白和sgrna复合体能精确靶向dna序列指sa-sauricas9蛋白和sgrna复合体可以识别并结合特定dna序列,或指将与sa-sauricas9蛋白融合的其他蛋白或特异性识别sgrna的蛋白带至靶向dna的位置。
本发明中,所述sa-sauricas9蛋白和sgrna复合体或与sa-sauricas9蛋白融合的其他蛋白或特异性识别sgrna的蛋白可以对靶向dna区域进行修饰和调控,所述修饰和调控包括但不限定于基因转录水平的调控、dna甲基化调控、dna乙酰化修饰、组蛋白乙酰化修饰、单碱基转换器或染色质成像追踪。
本发明中,所述单碱基转换器包括但不限定于碱基腺嘌呤到鸟嘌呤、或胞嘧啶到胸腺嘧啶、或胞嘧啶到尿嘧啶、或其它碱基之间的转换。
本发明提供的基因编辑系统编辑活性高,与之前的cas9相比具有明显的优势。
本发明通过基因合成、分子克隆、细胞转染、pcr产物深度测序、生物信息学分析等技术检测crispr/sa-sauricas9系统的编辑效率。
本发明提供的crispr/sa-sauricas9基因编辑系统能够在细胞中进行基因编辑,包括通过sa-sauricas9蛋白和sgrna的复合物识别定位靶向dna,对dna进行编辑;最后检测编辑效率。
具体步骤为:
(1)合成人源化的sa-sauricas9基因序列;并克隆到表达载体上,得到paav2_sa-sauricas9_itr;
(2)合成sgrna对应的寡核苷酸单链dna,即oligo-f和oligo-r序列,退火后连接至质粒paav2_sa-sauricas9_u6_bsai的bsai酶切位点,得到paav2_sa-sauricas9-hu6-sgrna;
(3)将表达sa-sauricas9蛋白、sgrna的载体递送至含有靶位点的细胞中;
(4)将编辑后的靶位点进行pcr扩增,t7ei酶切或者二代测序检测编辑效率。
本发明中,可以根据具体需要,针对待编辑的dna序列设计任意靶向的sgrna,并一定程度上对sgrna进行本领域所熟知的修饰,所述修饰包括但不限定于磷酸化、缩短、加长、硫化、甲基化、羟基化。
本发明中,可以根据具体需要,递送至细胞的crispr/sa-sauricas9系统包括但不限定于表达sa-sauricas9蛋白或sgrna的质粒、逆转录病毒、腺病毒、腺相关病毒载体或rna或sa-sauricas9蛋白。
本领域技术人员可以理解的是,碱基n表示a、t、c或g四种碱基中的任何一种。
进一步地,步骤(3)中,所述递送的手段包括但不限定于脂质体、阳离子多聚物、纳米颗粒、多功能信封式纳米以及病毒载体。
进一步地,步骤(3)中,所述细胞包括但不限定于人细胞、动物细胞、植物细胞、细菌细胞、真菌细胞。
进一步地,步骤(2)中,所述sgrna具有seqidno:2所示的核苷酸序列,或与seqidno:2所示的核苷酸序列至少80%相同的核苷酸序列,或基于核苷酸序列进行的修饰,修饰包括但不限定于磷酸化、缩短、加长、硫化或甲基化。
更具体地,在一个具体实施例中,合成sgrna对应的寡核苷酸单链dna序列,即oligo-f和oligo-r,该寡核苷酸单链dna序列如下:
oligo-fcaccgctcggagatcatcattgcg,(seqidno:4)
oligo-raaaccgcaatgatgatctccgagc。(seqidno:5)。
更具体地,在一个具体实施例中,本领域技术人员可以理解的是,oligo-f和oligo-r需要进行退火变为双链dna,退火反应体系为1μl100μmoligo-f,1μl100μmoligo-r,28μl水,震荡混匀后,放置于pcr仪中运行退火程序;退火程序如下:95℃_5min,85℃_1min,75℃_1min,65℃_1min,55℃_1min,45℃_1min,35℃_1min,25℃_1min,4℃保存,降温速率0.3℃/s。
更具体地,在一个具体实施例中,所述质粒paav2_sa-sauricas9_itr需要经过bsai限制性内切酶(neb)进行线性化处理。
更具体地,在一个具体实施例中,所述oligo-f和oligo-r退火后的产物与线性化处理后的paav2_sa-sauricas9_itr骨架载体通过dna连接酶进行连接反应。
更具体地,在一个具体实施例中,将连接产物转化至感受态细胞后,经sanger测序验证正确的克隆,然后提取质粒备用。
更具体地,在一个具体实施例中,步骤(3)中的细胞为hek293t,其包含的靶位点具有seqidno:6所示的核苷酸序列。
更具体地,在一个具体实施例中,步骤(3)中的递送手段为脂质体,包括
更具体地,在本发明第一方面的一个具体实施例中,所述实验步骤(4)中pcr的模板为编辑后的hek293t基因组dna。
更具体地,在一个具体实施例中,步骤(4)中pcr扩增的引物序列为:
f1-acactctttccctacacgacgctcttccgatctnnnngcgagaaaagccttgttt(seqidno:7)
r1-actggagttcagacgtgtgctcttccgatctctgaacttgtggccgtttac(seqidno:8)
f2-aatgatacggcgaccaccgagatctacactctttccctacacgac(seqidno:9)
r2-caagcagaagacggcatacgagatcactgtgtgactggagttcagacgtgtg(seqidno:10)。
本发明还提供一种用于基因编辑的crispr/sa-sauricas9系统试剂盒,该试剂盒包括sa-sauricas9蛋白或靶向dna序列的sgrna或靶向的dna。
本发明还提供crispr/sa-sauricas9基因编辑系统的应用,包括基因敲除、定点碱基的改变、定点插入、基因转录水平的调控、dna甲基化调控、dna乙酰化修饰、组蛋白乙酰化修饰、单碱基转换器或染色质成像追踪。
附图说明
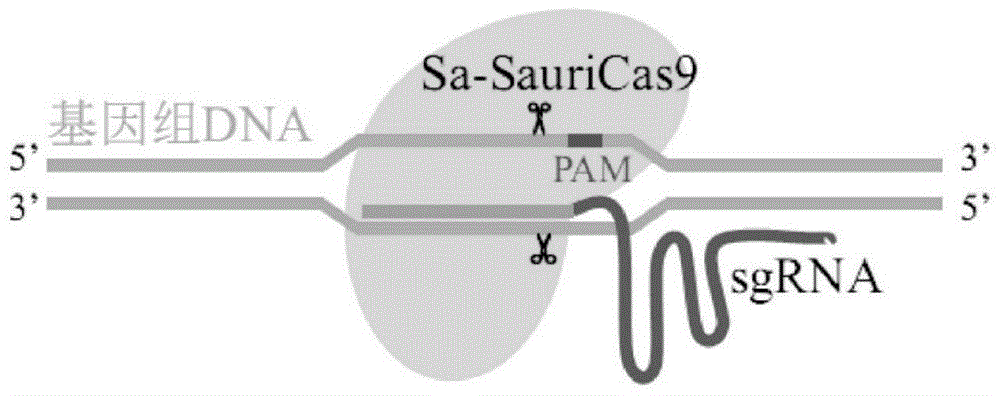
图1为crispr/sa-sauricas9基因编辑系统切割靶向dna示意图。其中,灰色椭圆形表示sa-sauricas9蛋白,黑色弯曲状表示sgrna序列,基因组上链中加深区域表示pam序列nngg。
图2为质粒paav2_sa-sauricas9_u6_bsai图谱示意图。其中,包括aav2itr、cmv增强子、cmv启动子、sv40nls、sa-sauricas9、nucleoplasminnls、3xha、bghpoly(a)、人u6启动子(hu6)、bsai内切酶位点、sgrnascaffold序列等元件。
图3为靶位点dna序列被编辑后的部分二代测序结果。其中编辑结果有缺失、插入或错配,最后4bp表示pam序列nngg。
图4为内源位点的t7endonucleasei酶切结果。其中,箭头表示切开的片段大小。
具体实施方式
以下将通过实施例进一步说明本发明,但实施例并不对本发明做任何形式的限定。
除非特别说明,本发明采用的试剂、方法和设备为本技术领域常规试剂、方法和设备。除非特别说明,以下实施例所用试剂和材料均为市购。未注明具体条件的实验方法,通常按照常规条件,或制造厂商所建议条件实施。
在一具体实施方式中,本发明提供的crispr/sa-sauricas9系统为一种新的基因编辑系统、方法、试剂盒及其应用。
在本发明一具体实施例中,crispr/sa-sauricas9系统能够在细胞中进行基因编辑,所述方法包括如下步骤:
1、构建质粒paav2_sa-sauricas9_itr
步骤(1),根据sa-sauricas9基因在uniprot上的检索号a0a2t4m4r5,下载其氨基酸序列,如seqidno:1所示。
步骤(2),将sa-sauricas9的氨基酸序列进行密码子优化,获得了sa-sauricas9在人细胞中高表达的编码序列,如seqidno:11所示。
步骤(3),将获得基因sa-sauricas9的编码序列在公司进行基因合成,并构建至paav2_itr骨架质粒上,得到质粒paav2_sa-sauricas9_itr,如图2所示。
2、制备线性化质粒paav2_sa-sauricas9_itr
步骤(1),用basi限制性内切酶将质粒paav2_sa-sauricas9_itr进行酶切线性化,酶切体系:1μg质粒paav2_sa-sauricas9_itr,5μl10xcutsmart缓冲液,1μlbasi内切酶,水补足至50μl,37℃反应1小时。
步骤(2),将酶切后的产物在1%的琼脂糖凝胶上电泳,120v电泳30分钟。
步骤(3),切除7433bp大小的dna片段,用胶回收试剂盒依据厂家提供的步骤进行回收,最终用超纯水进行洗脱。
步骤(4),将回收的线性化质粒paav2_sa-sauricas9_itr用nanodrop测定dna浓度,备用或置于-20℃进行长期保存。
3、构建质粒paav2_sa-sauricas9-hu6-sgrna
步骤(1),设计sgrna序列。
步骤(2),在设计的sgrna序列对用的正义链和反义链上分别加上线性化质粒paav2_
sa-sauricas9_itr两侧对应的粘性末端序列,并在公司合成两条寡核苷酸单链dna,具体序列为:
oligo-fcaccgctcggagatcatcattgcg,(seqidno:4)
oligo-raaaccgcaatgatgatctccgagc。(seqidno:5);
步骤(3),寡核苷酸单链dna进行退火变为双链dna,退火反应体系:1μl100μmoligo-f,1μl100μmoligo-r,28μl水,震荡混匀后,放置于pcr仪中运行退火程序:95℃_5min,85℃_1min,75℃_1min,65℃_1min,55℃_1min,45℃_1min,35℃_1min,25℃_1min,4℃保存,降温速率0.3℃/s。
步骤(4),将退火后的产物与线性化质粒paav2_sa-sauricas9_itr在dna连接酶的作用下依据产品提供的步骤进行连接。
步骤(5),取1μl连接产物进行化学感受态转化,将生长的细菌克隆进行sanger测序验证。
步骤(6),将测序验证连接正确的克隆摇菌,提取质粒paav2_sa-sauricas9-hu6-sgrna备用。
4、转染表达sa-sauricas9蛋白和sgrna的质粒paav2_sa-sauricas9-hu6-sgrna
步骤(1),第0天,根据转染所需,将含有sgrna靶向位点的hek293t细胞系在6孔板进行铺板,细胞密度约30%左右,靶位点序列如seqidno:6所示。
步骤(2),第1天,进行转染,转染体系如下,
i.取2μg待转染质粒paav2_sa-sauricas9-hu6-sgrna加入至100μlopti-mem培养基中,轻轻吹打混匀;
ii.将
iii.将稀释的
步骤(3),将细胞置于37℃,5%的co2培养箱中继续培养。
5、制备二代测序文库
步骤(1),收集编辑3天后的hek293t细胞,用dna试剂盒依据产品提供的步骤提取基因组dna。
步骤(2),进行pcr建库第一轮pcr,用2xq5mastermix进行pcr反应,pcr引物如seqidno:7-seqidno:8所示,反应体系如下:
pcr运行程序如下:
步骤(3),进行pcr建库第二轮pcr,用2xq5mastermix进行pcr反应,pcr引物如seqidno:9--seqidno:10所示,反应体系如下:
pcr运行程序如下:
步骤(4),将第二轮的pcr产物用胶回收试剂盒依据厂家提供的步骤,纯化366bp大小的dna片段,二代测序文库制备完毕。
6、二代测序结果分析
步骤(1),将制备好的二代测序文库交由公司在hiseqxten上进行双端测序。
步骤(2)生物信息学分析二代测序结果,部分编辑结果如图2和图3所示。
7、内源位点验证
步骤(1),将表达sa-sauricas9和sgrna的质粒paav2_sa-sauricas9-hu6-sgrna通过
sgrna序列为:atagggttaggggccccaggc,(seqidno:12)
靶位点的具体序列为:atagggttaggggccccaggccggg;(seqidno:13);
步骤(2),提取编辑5天后的细胞基因组dna,通过2xq5mastermix,用引物test-f和test-r将靶向dna序列进行扩增;其中:
test-f的具体序列为:acgcagtgggtcataggctc,(seqidno:14)
test-r的具体序列为:ggactcaggcccttcctcct;(seqidno:15);
步骤(3),将pcr产物通过琼脂糖凝胶回收,纯化509bp大小的dna片段;
步骤(4),依照t7endonucleasei的说明书对纯化的dna片段进行酶切,然后跑胶检测,结果如图4所示,左侧为阴性对照组,转染时无sgrna,t7endonucleasei切靶向序列后无切开的片段,说明未发生编辑;右侧为实验组,转染时有sgrna,t7endonucleasei切靶向序列后出现切开的片段,说明发生了编辑。
序列表
<110>复旦大学
<120>crispr/sa-sauricas9基因编辑系统及其应用
<130>1113
<160>15
<170>patentinversion3.5
<210>1
<211>1056
<212>prt
<213>人工序列
<400>1
metlysargasntyrileleuglyleuaspileglyilethrserval
151015
glytyrglyileileasptyrgluthrargaspvalileaspalagly
202530
valargleuphelysglualaasnvalgluasnasngluglyargarg
354045
serlysargglyalaargargleulysargargargarghisargile
505560
glnargvallyslysleuleupheasptyrasnleuleuthrasphis
65707580
sergluleuserglyileasnprotyrglualaargvallysglyleu
859095
serglnlysleuserglugluglupheseralaalaleuleuhisleu
100105110
alalysargargglyvalhisasnvalasngluvalglugluaspthr
115120125
glyasngluleuserthrlysgluglnileserargasnserlysala
130135140
leugluglulystyrvalalagluleuglnleugluargleulyslys
145150155160
aspglygluvalargglyserileasnargphelysthrserasptyr
165170175
vallysglualalysglnleuleulysvalglnlysalatyrhisgln
180185190
leuaspglnserpheileaspthrtyrileaspleuleugluthrarg
195200205
argthrtyrtyrgluglyproglygluglyserpropheglytrplys
210215220
aspilelysglutrptyrglumetleumetglyhiscysthrtyrphe
225230235240
proglugluleuargservallystyralatyrasnalaaspleutyr
245250255
asnalaleuasnaspleuasnasnleuvalilethrargaspgluasn
260265270
glulysleuglutyrtyrglulyspheglnileilegluasnvalphe
275280285
lysglnlyslyslysprothrleulysglnilealalysgluileleu
290295300
valasnglugluaspilelysglytyrargvalthrserthrglylys
305310315320
progluphethrasnleulysvaltyrhisaspilelysaspilethr
325330335
alaarglysgluileilegluasnalagluleuleuaspglnileala
340345350
lysileleuthriletyrglnsersergluaspileglnglugluleu
355360365
thrasnleuasnsergluleuthrglnglugluilegluglnileser
370375380
asnleulysglytyrthrglythrhisasnleuserleulysalaile
385390395400
asnleuileleuaspgluleutrphisthrasnaspasnglnileala
405410415
ilepheasnargleulysleuvalprolyslysvalaspleusergln
420425430
glnlysgluileprothrthrleuvalaspasppheileleuserpro
435440445
valvallysargserpheileglnserilelysvalileasnalaile
450455460
ilelyslystyrglyleuproasnaspileileilegluleualaarg
465470475480
glulysasnserlysaspalaglnlysmetileasnglumetglnlys
485490495
argasnalaalathrasngluargileglugluileileargthrthr
500505510
glylysgluasnalalystyrleuileglulysilelysleuhisasp
515520525
metglngluglylyscysleutyrserleuglualaileproleuglu
530535540
aspleuleuasnasnpropheasntyrgluvalasphisileilepro
545550555560
argservalserpheaspasnserpheasnasnlysvalleuvallys
565570575
glnglugluasnserlyslysglyasnargthrpropheglntyrleu
580585590
serserseraspserlysilesertyrgluthrphelyslyshisile
595600605
leuasnleualalysglylysglyargileserlysthrlyslysglu
610615620
tyrleuleuglugluargaspileasnargpheservalglnlysasp
625630635640
pheileasnargasnleuvalaspthrargtyralathralaalaleu
645650655
metasnleuleuargsertyrpheargvalasnasnleuaspvallys
660665670
vallysserileasnglyglyphethrserpheleuargarglystrp
675680685
lysphelyslysgluargasnlysglytyrlyshishisalagluasp
690695700
alaleuileilealaasnalaasppheilephelysglutrplyslys
705710715720
leuasplysalalyslysvalmetgluasnglnmetphegluglulys
725730735
glnalaglusermetprogluilegluthrgluglnglutyrlysglu
740745750
ilepheilethrprohisglnilelyshisilelysaspphelysasp
755760765
tyrlystyrserhisargvalasplyslysproasnarggluleuile
770775780
asnaspthrleutyrserthrarglysaspasplysglyasnthrleu
785790795800
ilevalasnasnleuasnglyleutyrasplysaspasnasplysleu
805810815
lyslysleuileasnlysserproglulysleuleumettyrhishis
820825830
aspproglnthrtyrglnlysleulysleuilemetgluglntyrgly
835840845
aspglulysasnproleutyrlystyrtyrglugluthrglyasntyr
850855860
leuthrlystyrserlyslysaspasnglyprovalilelyslysile
865870875880
lystyrtyrglyasnlysleuasnalahisleuaspilethraspasp
885890895
tyrproasnserargasnlysvalvallysleuserleulysserphe
900905910
argpheaspiletyrlyscysgluglnglytyrlysmetvalserile
915920925
glytyrleuaspvalleulyslysaspasntyrtyrtyrileprolys
930935940
asplystyrglualaglulysglnlyslyslysilelysgluserasp
945950955960
leuphevalglyserphetyrtyrasnaspleuilemettyrgluasp
965970975
gluleupheargvalileglyvalasnseraspileasnasnleuval
980985990
gluleuasnmetvalaspilethrtyrlysaspphecysgluvalasn
99510001005
asnvalthrglyglulysargilelyslysthrileglylysarg
101010151020
valvalleuileglulystyrthrthraspileleuglyasnleu
102510301035
tyrlysthrproleuprolyslysproglnleuilephelysarg
104010451050
glygluleu
1055
<210>2
<211>101
<212>rna
<213>人工序列
<220>
<221>misc_feature
<222>(1)..(20)
<223>nisa,c,g,oru
<400>2
nnnnnnnnnnnnnnnnnnnnguuuuaguacucuggaaacagaaucuacuaaaacaaggca60
aaaugccguguuuaucucgucaacuuguuggcgagauuuuu101
<210>3
<211>25
<212>dna
<213>人工序列
<220>
<221>misc_feature
<222>(1)..(23)
<223>nisa,c,g,ort
<400>3
nnnnnnnnnnnnnnnnnnnnnnngg25
<210>4
<211>24
<212>dna
<213>人工序列
<400>4
caccgctcggagatcatcattgcg24
<210>5
<211>24
<212>dna
<213>人工序列
<400>5
aaaccgcaatgatgatctccgagc24
<210>6
<211>25
<212>dna
<213>人工序列
<220>
<221>misc_feature
<222>(21)..(25)
<223>nisa,c,g,ort
<400>6
gctcggagatcatcattgcgnnnnn25
<210>7
<211>55
<212>dna
<213>人工序列
<220>
<221>misc_feature
<222>(34)..(37)
<223>nisa,c,g,ort
<400>7
acactctttccctacacgacgctcttccgatctnnnngcgagaaaagccttgttt55
<210>8
<211>51
<212>dna
<213>人工序列
<400>8
actggagttcagacgtgtgctcttccgatctctgaacttgtggccgtttac51
<210>9
<211>45
<212>dna
<213>人工序列
<400>9
aatgatacggcgaccaccgagatctacactctttccctacacgac45
<210>10
<211>52
<212>dna
<213>人工序列
<400>10
caagcagaagacggcatacgagatcactgtgtgactggagttcagacgtgtg52
<210>11
<211>3168
<212>dna
<213>人工序列
<400>11
atgaagcggaactacatcctgggcctggacatcggcatcaccagcgtgggctacggcatc60
atcgactacgagacacgggacgtgatcgatgccggcgtgcggctgttcaaagaggccaac120
gtggaaaacaacgagggcaggcggagcaagagaggcgccagaaggctgaagcggcggagg180
cggcatagaatccagagagtgaagaagctgctgttcgactacaacctgctgaccgaccac240
agcgagctgagcggcatcaacccctacgaggccagagtgaagggcctgagccagaagctg300
agcgaggaagagttctctgccgccctgctgcacctggccaagagaagaggcgtgcacaac360
gtgaacgaggtggaagaggacaccggcaacgagctgtccaccaaagagcagatcagccgg420
aacagcaaggccctggaagagaaatacgtggccgaactgcagctggaacggctgaagaaa480
gacggcgaagtgcggggcagcatcaacagattcaagaccagcgactacgtgaaagaagcc540
aaacagctgctgaaggtgcagaaggcctaccaccagctggaccagagcttcatcgacacc600
tacatcgacctgctggaaacccggcggacctactatgagggacctggcgagggcagcccc660
ttcggctggaaggacatcaaagaatggtacgagatgctgatgggccactgcacctacttc720
cccgaggaactgcggagcgtgaagtacgcctacaacgccgacctgtacaacgccctgaac780
gacctgaacaatctcgtgatcaccagggacgagaacgagaagctggaatattacgagaag840
ttccagatcatcgagaacgtgttcaagcagaagaagaagcccaccctgaagcagatcgcc900
aaagaaatcctcgtgaacgaagaggatattaagggctacagagtgaccagcaccggcaag960
cccgagttcaccaacctgaaggtgtaccacgacatcaaggacattaccgcccggaaagag1020
attattgagaacgccgagctgctggatcagattgccaagatcctgaccatctaccagagc1080
agcgaggacatccaggaagaactgaccaatctgaactccgagctgacccaggaagagatc1140
gagcagatctctaatctgaagggctataccggcacccacaacctgagcctgaaggccatc1200
aacctgatcctggacgagctgtggcacaccaacgacaaccagatcgctatcttcaaccgg1260
ctgaagctggtgcccaagaaggtggacctgtcccagcagaaagagatccccaccaccctg1320
gtggacgacttcatcctgagccccgtcgtgaagagaagcttcatccagagcatcaaagtg1380
atcaacgccatcatcaagaagtacggcctgcccaacgacatcattatcgagctggcccgc1440
gagaagaactccaaggacgcccagaaaatgatcaacgagatgcagaagcggaacgccgcc1500
accaacgagcggatcgaggaaatcatccggaccaccggcaaagagaacgccaagtacctg1560
atcgagaagatcaagctgcacgacatgcaggaaggcaagtgcctgtacagcctggaagcc1620
atccctctggaagatctgctgaacaaccccttcaactatgaggtggaccacatcatcccc1680
agaagcgtgtccttcgacaacagcttcaacaacaaggtgctcgtgaagcaggaagaaaac1740
agcaagaagggcaaccggaccccattccagtacctgagcagcagcgacagcaagatcagc1800
tacgaaaccttcaagaagcacatcctgaatctggccaagggcaagggcagaatcagcaag1860
accaagaaagagtatctgctggaagaacgggacatcaacaggttctccgtgcagaaagac1920
ttcatcaaccggaacctggtggataccagatacgccaccgccgccctgatgaacctgctg1980
cggagctacttcagagtgaacaacctggacgtgaaagtgaagtccatcaatggcggcttc2040
accagctttctgcggcggaagtggaagtttaagaaagagcggaacaaggggtacaagcac2100
cacgccgaggacgccctgatcattgccaacgccgatttcatcttcaaagagtggaagaaa2160
ctggacaaggccaaaaaagtgatggaaaaccagatgttcgaggaaaagcaggccgagagc2220
atgcccgagatcgaaaccgagcaggagtacaaagagatcttcatcaccccccaccagatc2280
aagcacattaaggacttcaaggactacaagtacagccaccgggtggacaagaagcctaat2340
agagagctgattaacgacaccctgtactccacccggaaggacgacaagggcaacaccctg2400
atcgtgaacaatctgaacggcctgtacgacaaggacaatgacaagctgaaaaagctgatc2460
aacaagagccccgaaaagctgctgatgtaccaccacgacccccagacctaccagaaactg2520
aagctgattatggaacagtacggcgacgagaagaatcccctgtacaagtactacgaggaa2580
accgggaactacctgaccaagtactccaaaaaggacaacggccccgtgatcaagaagatt2640
aagtattacggcaacaaactgaacgcccatctggacatcaccgacgactaccccaacagc2700
agaaacaaggtcgtgaagctgtccctgaagagcttccgcttcgacatctacaagtgcgag2760
cagggctacaagatggtgagcatcggctacctggacgtgctgaagaaggacaactactac2820
tacatccccaaggacaagtacgaggccgagaagcagaagaagaagataaaggagagcgac2880
ctgttcgtgggcagcttctactacaacgacctgataatgtacgaggacgagctgttccgc2940
gtgataggcgtgaacagcgacatcaacaacctggtggagctgaacatggtggacatcacc3000
tacaaggacttctgcgaggtgaacaacgtgaccggcgagaagcgcatcaagaagaccatc3060
ggcaagcgcgtggtgctgatagagaagtacaccaccgacatcctgggcaacctgtacaag3120
acccccctgcccaagaagccccagctgatattcaagcgcggcgagctg3168
<210>12
<211>21
<212>dna
<213>人工序列
<400>12
atagggttaggggccccaggc21
<210>13
<211>25
<212>dna
<213>人工序列
<400>13
atagggttaggggccccaggccggg25
<210>14
<211>20
<212>dna
<213>人工序列
<400>14
acgcagtgggtcataggctc20
<210>15
<211>20
<212>dna
<213>人工序列
<400>15
ggactcaggcccttcctcct20
- 还没有人留言评论。精彩留言会获得点赞!