一种牡丹皮的转录组SSR分子标记体系及其应用的制作方法
本发明属于生物领域,涉及一种牡丹皮的转录组ssr分子标记体系及其应用。
背景技术:
中药牡丹皮为毛茛科植物牡丹paeoniasuffruticosaandr.的干燥根皮,主要分布于我国的重庆、安徽、陕西、河南、山东等地。牡丹皮为常用的大宗药材,临床应用广泛,具有清热凉血、活血化瘀的功效,也是中药临床大品种制剂如六味地黄丸的原材料之一。重庆垫江为川丹皮的传统道地产区;安徽凤丹(p.ostiit.hongetj.x.zhang)虽未收入药典,但因药材品质好,也是作为药材市场上牡丹皮的主要来源之一。
现有的研究发现,牡丹有近1000个观赏培育品种,由于观赏品种与药用品种在品种选育与栽培模式上有较大差异,一般药用品种为单瓣单色花,牡丹皮质量为佳;观赏品种多为重瓣鲜艳的花,若根皮入药质量不如药用品种。但随着市场对牡丹皮的需求增加,经常有观赏牡丹来源的牡丹皮充斥于药材市场,会导致不同品种牡丹来源的牡丹皮存在明显的质量差异,进而影响到临床疗效。由于不同品种牡丹皮在外观形态上极为相似,难以通过外部形态特征进行区分。因此,亟需建立可靠的鉴定方法来区分不同来源的牡丹皮品种,对药用牡丹与观赏牡丹来源的牡丹皮进行鉴定,以保证牡丹皮药材质量的稳定性。
牡丹皮的品质主要体现在有效成分的含量,而有效成分的积累依赖于植物体内生物合成通路中的功能性基因,通过解析功能性基因的特征属性,可为药材品质形成及评价提供重要参考。而对功能基因的特征标记,可作为药材品质评价及优良品种培育的指标。
分子标记已成为揭示物种的遗传多样性和品种鉴定最重要的工具之一。目前常用的分子标记类型包括限制性片段长度多态性(rflp)、随机扩增多态性dna(rapd)、酶切扩增多态性序列(caps)、单核甘酸多态性(snp)、简单重复序列(ssr)等。其中,ssr(simplesequencerepeat)又称为微卫星序列,是由1~6个核苷酸为重复基元重复多次组成的dna序列,它们的长度大约为100~200个碱基对,广泛存在于真核生物基因组中。ssr序列具有分布广泛、共显性遗传、多态性位点多、信息含量丰富、物种间转移性好、易于检测和可重复性好的特点,已被广泛应用于品种鉴定,种质资源保存和利用、遗传多样性分析、分子标记辅助选育等研究领域。基于转录组中的ssr分子标记既可避免基因组ssr(genomic-ssr)周期长、成本高,以及est-ssr的数据量少的问题,又具有genic-ssr的优点;同时,这种技术也充分利用了转录组测序的结果。从已经开展的研究发现基于转录组ssr开发的分子标记多态性和扩增效果较好,说明转录组ssr适宜用来进行分子标记的开发。牡丹作为重要的观赏植物,具有“多地、多种、多元”起源的特点,有着极其丰富的遗传多样性。在作物育种中,深入了解分子多样性,是研究作物遗传多样性、鉴定品种和选择杂交亲本的基础,但牡丹在这方面的研究偏少,主要集中在对牡丹品种鉴定、起源等研究,研究则偏重于牡丹花的观赏性,而忽略了该植物的药用价值。部分地区的牡丹品种通过分子标记研究发现其具有丰富的遗传多样性。采用rapd技术对四川彭州地区的牡丹品种进行分析,发现多数来源地相同的品种表现为较为密切的亲缘关系;在构建中原牡丹核心种质资源库研究中,根据品种来源、分类体系等基本数据和形态、分子标记特征数据,建立了中原牡丹品种的表型数据库和品种的分子数据库。并通过构建中原牡丹品种的初级核心种质资源,进行issr和aflp标记证明构建的核心种质具有丰富的遗传多样性。cddp标记也被尝试用于牡丹的遗传多样性分析,品种特异性分析和一致性检验,以筛选区分牡丹种质资源的核心引物和探讨牡丹分子身份证的构建方法。于海萍等以凤丹为实验材料,利用磁珠富集法开发ssr标记,探讨了凤丹在芍药组植物中的交叉扩增能力,发现紫斑牡丹是西北牡丹品种群的主要原种之一,且二者与四川牡丹有较近的亲缘关系;凤丹参与了中原牡丹品种群的起源;系个体与中原牡丹品种群个体也有一定的亲缘关系。
技术实现要素:
有鉴于此,本发明的目的在于提供一种牡丹皮的转录组ssr分子标记体系及其应用,通过功能基因的遗传多态性对不同来源的牡丹皮药材及饮片品种及品质进行有效的鉴定和区分。
为达到上述目的,本发明提供如下技术方案:
1、一种牡丹皮的转录组ssr分子标记体系,包括以下10个引物对:
引物对p1,其核苷酸序列如seqidno.1和seqidno.2所示;
引物对p2,其核苷酸序列如seqidno.3和seqidno.4所示;
引物对p3,其核苷酸序列如seqidno.5和seqidno.6所示;
引物对p4,其核苷酸序列如seqidno.7和seqidno.8所示;
引物对p5,其核苷酸序列如seqidno.9和seqidno.10所示;
引物对p6,其核苷酸序列如seqidno.11和seqidno.12所示;
引物对p7,其核苷酸序列如seqidno.13和seqidno.14所示;
引物对p8,其核苷酸序列如seqidno.15和seqidno.16所示;
引物对p9,其核苷酸序列如seqidno.17和seqidno.18所示;
引物对p10,其核苷酸序列如seqidno.19和seqidno.20所示。
2、上述一种牡丹皮的转录组ssr分子标记体系在牡丹皮的品种鉴定和品质评价中的应用。
优选的,所述牡丹皮包括但不限于单瓣红、重瓣红、凤丹或洛阳观赏牡丹品种,所述洛阳观赏牡丹品种包括但不限于赵粉、葛巾紫、丁香紫、大棕紫、萍实艳、海黄、姚黄、洛阳锦、黑牡丹、白牡丹。
3、上述一种牡丹皮的转录组ssr分子标记体系的构建方法,具体步骤如下:
(1)提取新鲜牡丹皮样品rna,构建牡丹皮转录组文库并进行测序分析,获得牡丹皮转录组序列,并用trinity软件对转录本进行拼接得到unigene;
(2)unigene功能注释使用blast软件将unigene序列与nr、swiss-prot、go、cog、kog、eggnog4.5、kegg数据库比对,使用kobas2.0得到unigene在kegg中的keggorthology结果,预测完unigene的氨基酸序列之后使用hmmer软件与pfam数据库比对,获得unigene的注释信息,得到参与牡丹皮中有效成分如丹皮酚、芍药苷、没食子酸等生物合成通路中的功能基因或片段,独立建成功能基因数据库;
(3)使用misa软件对功能基因数据库中核苷酸长度大于1000bp的unigene进行ssr位点的筛选和分析;从得到的总ssr中再设置条件进行筛选,筛选标准:二核苷酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸最少重复次数分别为9、6、5、4、4;
(4)采用primer5软件设计ssr引物,引物设计参数为引物长度为18~25bp;pcr扩增产物长度为100~400bp;gc含量为40%~60%;
(5)对步骤(4)所设计的引物在不同牡丹皮样本中进行可扩增性和通用性鉴定,得到在不同品种牡丹皮中具有可扩增性和通用性的上述ssr分子标记体系。
本发明的有益效果在于:
本发明根据牡丹皮转录组分析首先筛选序列在1000bp以上的基因片段,并基于这些片段再寻找与有效成分生物合成功能基因相关的ssr分子标记引物,共得到10对引物体系,该引物体系多态性高、通用性好,且能够在多个牡丹样品中体现多态性,不但能对不同品种来源的牡丹皮品种来源进行有效鉴别,同时也可以作为牡丹皮质量优劣评价的一个指标。
附图说明
为了使本发明的目的、技术方案和有益效果更加清楚,本发明提供如下附图进行说明:
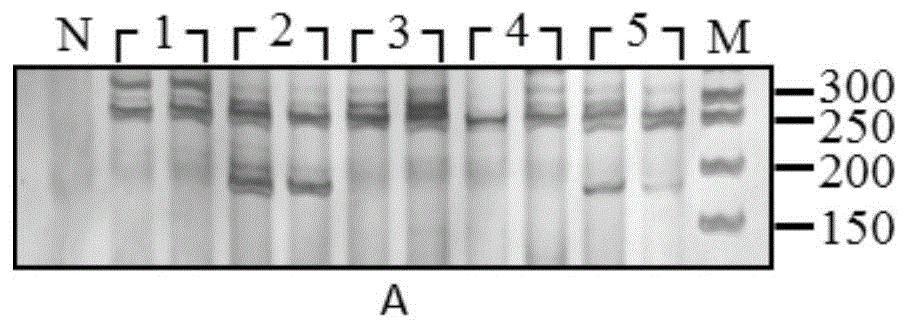
图1为引物对p1扩增部分牡丹皮样品后的凝胶电泳图谱,其中,n—阴性对照;1—重庆垫江太平红单瓣花牡丹皮样品;2—重庆垫江太平红重瓣花牡丹皮样品;3—重庆垫江凤丹白牡丹皮样品;4—安徽凤丹白牡丹皮样品;5—洛阳观赏用牡丹皮样品;m—dnamarker;
图2为引物对p2扩增部分牡丹皮样品后的凝胶电泳图谱,其中,n—阴性对照;1—重庆垫江太平红单瓣花牡丹皮样品;2—重庆垫江太平红重瓣花牡丹皮样品;3—重庆垫江凤丹白牡丹皮样品;4—安徽凤丹白牡丹皮样品;5—洛阳观赏用牡丹皮样品;m—dnamarker;
图3为引物对p3扩增部分牡丹皮样品后的凝胶电泳图谱,其中,n—阴性对照;1—重庆垫江太平红单瓣花牡丹皮样品;2—重庆垫江太平红重瓣花牡丹皮样品;3—重庆垫江凤丹白牡丹皮样品;4—安徽凤丹白牡丹皮样品;5—洛阳观赏用牡丹皮样品;m—dnamarker;
图4为引物对p4扩增部分牡丹皮样品后的凝胶电泳图谱,其中,n—阴性对照;1—重庆垫江太平红单瓣花牡丹皮样品;2—重庆垫江太平红重瓣花牡丹皮样品;3—重庆垫江凤丹白牡丹皮样品;4—安徽凤丹白牡丹皮样品;5—洛阳观赏用牡丹皮样品;m—dnamarker;
图5为引物对p5扩增部分牡丹皮样品后的凝胶电泳图谱,其中,1—重庆垫江太平红单瓣花牡丹皮样品;2—重庆垫江太平红重瓣花牡丹皮样品;3—重庆垫江凤丹白牡丹皮样品;4—安徽凤丹白牡丹皮样品;5—洛阳观赏用牡丹皮样品;m—dnamarker;
图6为引物对p6扩增部分牡丹皮样品后的凝胶电泳图谱,其中,n—阴性对照;1—重庆垫江太平红单瓣花牡丹皮样品;2—重庆垫江太平红重瓣花牡丹皮样品;3—重庆垫江凤丹白牡丹皮样品;4—安徽凤丹白牡丹皮样品;5—洛阳观赏用牡丹皮样品;m—dnamarker;
图7为引物对p7扩增部分牡丹皮样品后的凝胶电泳图谱,其中,n—阴性对照;1—重庆垫江太平红单瓣花牡丹皮样品;2—重庆垫江太平红重瓣花牡丹皮样品;3—重庆垫江凤丹白牡丹皮样品;4—安徽凤丹白牡丹皮样品;5—洛阳观赏用牡丹皮样品;m—dnamarker;
图8为引物对p8扩增部分牡丹皮样品后的凝胶电泳图谱,其中,n—阴性对照;1—重庆垫江太平红单瓣花牡丹皮样品;2—重庆垫江太平红重瓣花牡丹皮样品;3—重庆垫江凤丹白牡丹皮样品;4—安徽凤丹白牡丹皮样品;5—洛阳观赏用牡丹皮样品;m—dnamarker;
图9为引物对p9扩增部分牡丹皮样品后的凝胶电泳图谱,其中,n—阴性对照;1—重庆垫江太平红单瓣花牡丹皮样品;2—重庆垫江太平红重瓣花牡丹皮样品;3—重庆垫江凤丹白牡丹皮样品;4—安徽凤丹白牡丹皮样品;5—洛阳观赏用牡丹皮样品;m—dnamarker;
图10为引物对p10扩增部分牡丹皮样品后的凝胶电泳图谱,其中,n—阴性对照;1—重庆垫江太平红单瓣花牡丹皮样品;2—重庆垫江太平红重瓣花牡丹皮样品;3—重庆垫江凤丹白牡丹皮样品;4—安徽凤丹白牡丹皮样品;5—洛阳观赏用牡丹皮样品;m—dnamarker;
图11为基于ssr多态性结果的不同来源牡丹皮样品聚类分析。
具体实施方式
下面将结合附图,对本发明的优选实施例进行详细的描述。
本发明实施例中的方法若无特殊说明,均为该类技术中常规性方法,下列实施例中的实验材料、试剂和仪器设备均通过商业途径获得。
实施例1牡丹皮转录组ssr多态性引物对的开发
1总rna提取及纯化
挖取新鲜牡丹根,清洗泥土,迅速投入液氮罐中速冻,放入-80℃冰箱保存。
采用rna提取试剂盒(invitrogentm,ribominustmplantkitforrna-seq)提取牡丹皮样品总rna,分别采用nanodrop、qubit2.0、aglient2100方法检测rna样品的纯度、浓度和完整性等,以保证使用合格的样品进行转录组测序。
2文库构建
样品检测合格后,进行文库构建,根据
(1)用带有oligo(dt)的磁珠富集真核生物mrna;
(2)加入fragmentationbuffer将mrna进行随机打断;
(3)以mrna(5μl)为模板,用六碱基随机引物(randomhexamers,1μl)、nebnextfirststrandsynthesisreactionbuffer(4μl)、nebnextfirststrandsynthesisenzymemix(2μl)等混合通过梯度升温合成第一条cdna链,反应产物加入nebnextsecondstrandsynthesisreactionbuffer(10x)(8μl)、nebnextsecondstrandsynthesisenzymemix(4μl)、水等试剂通过16℃温育1小时合成第二条cdna链,利用ampurexp磁珠纯化cdna;
(4)纯化的双链cdna再进行末端修复、加a尾并连接测序接头,然后用ampurexp磁珠进行片段大小选择;
(5)最后通过pcr富集得到cdna文库。
3文库质控
文库构建完成后,分别使用qubit2.0和agilent2100对文库的浓度和插入片段大小(insertsize)进行检测,使用q-pcr方法对文库的有效浓度进行准确定量,以保证文库质量。
4上机测序
库检合格后,用illuminahiseq进行高通量测序。
对rawdata进行数据过滤,去除其中的接头序列及低质量reads获得高质量的cleandata。将cleandata进行序列组装,获得该物种的unigene库。
5unigene注释
使用blast软件将unigene序列与nr、swiss-prot、go、cog、kog、eggnog4.5、kegg数据库比对,使用kobas2.0得到unigene在kegg中的keggorthology结果。选择blast参数e-value不大于1e-5和hmmer参数e-value不大于1e-10,最终获得46,398个有注释信息的unigene。
基因注释的统计结果见表1。
表1.unigene注释统计表
注:annotateddatabases:表示各功能数据库;annotated_number:表示注释到该数据库的unigene数;300≤length<1000:表示注释到该数据库的unigene长度大于等于300且小于1000碱基的unigene数;length≥1000:表示注释到该数据库的长度大于1000个碱基的unigene数。
6ssr分子
利用misa软件对筛选得到的1kb以上的unigene做ssr分析,结果见表2。
表2.ssr重复基序类型及分布
从牡丹皮转录组数据鉴定出的6235个ssr位点中,首先进行引物筛选,筛选标准:二核苷酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸最少重复次数分别为9、6、5、4、4;引物长度为18~25bp;pcr扩增产物长度为150~300bp;gc含量为40%~60%。然后筛选后的引物随机选择132对进行可扩增性和多态性及通用性验证。
具体方法为:
dna提取
挖取垫江牡丹新鲜根,洗净泥土,去除木心,50℃干燥备用;
采用改良的ctab法提取牡丹皮dna,操作步骤如下:
(1)精密称取100mg牡丹皮药材置于研钵,加少量pvp共研磨;
(2)药材研磨成粉末后转移至2.0ml离心管中,加入1.5ml在4℃预冷的tne缓冲液(0.1mol·l-1tris,0.15mol·l-1氯化钠,0.02mol·l-1edta),4℃浸提2次,每次30分钟,12000rpm离心10分钟,弃掉上清液;
(3)管中加入900μl在65℃预热的2×ctab提取缓冲液(0.1mol·l-1tris-hcl,0.02mol·l-1edta,1.4mol·l-1氯化钠,2%(w/v)ctab,1%(v/v)pvp),然后加入14μlβ-巯基乙醇,置于65℃水浴保温90分钟,水浴期间翻动样品2~3次;
(4)水浴结束之后,待样品冷却至45℃以下,精密吸取900μl氯仿-异戊醇(体积比24:1)加入离心管中,温和摇动3~5分钟使成乳状液,12000rpm离心10分钟;
(5)静置分层后,吸取700μl上清液至另一干净的2.0ml离心管中,加入等体积的氯仿-异戊醇(体积比24:1),温和摇动2~3分钟使之充分混合,12000rpm离心10分钟;
(6)吸取500μl上清液至另一干净的1.5ml离心管中,加入占上清液2/3体积的异丙醇(﹣20℃提前预冷),﹣20℃条件下放置30分钟。12000rpm离心10分钟。弃掉上清液;
(7)离心管中加入500μl70%乙醇,12000rpm离心5分钟,弃掉上清液。重复操作一次;
(8)离心管中加入500μl无水乙醇,12000rpm离心5分钟,弃掉上清液。重复操作一次;
(9)用吸水纸吸干管中残留的乙醇,管口水平放置于37℃干燥箱15~30分钟,待乙醇完全挥发;
(10)离心管中加入50μlddh2o,充分溶解dna。置于﹣20℃冰箱保存备用。
将垫江太平红(含单瓣红和重瓣红)牡丹皮共24份;垫江产凤丹白8份;安徽产凤丹共8份;洛阳产各种观赏品种(赵粉、葛巾紫、丁香紫、大棕紫、萍实艳、海黄、姚黄、洛阳锦、黑牡丹、白牡丹)共10份;按上述dna提取方法进行样品基因组dna的提取。
dna质量检测
采用dycp-31dn型电泳仪对dna质量进行琼脂糖凝胶电泳检测。取5μldna样品,加入1μl6×loadingbuffer,轻轻混匀。混匀后用移液枪将混合液及3μldnamarker点入上样孔;接通电泳仪电源与电泳仪之间的电泳导线,设定电泳参数,电压为120v·cm-1,电泳时间40分钟;电泳结束后,使用zf-2型紫外分析仪检测琼脂糖凝胶,并对电泳图谱进行标记、保存。
pcr扩增
为达到良好的扩增效果,在pcr体系中使用了pcr增强剂(2%(w/v)bsa、1%(w/v)pvp)。pcr扩增体系配制如表3所示。
表3.pcr扩增体系
对上述配置好的pcr反应液,其扩增程序如下:94℃预变性,4min;94℃变性,30s;53℃退火,30s;72℃延伸,30s;重复变性至延伸步骤30-35个循环,72℃延伸5min,温度回落至25℃时取出备用。扩增产物用2%琼脂糖凝胶电泳进行检测,若出现100-400bp的条带,则该ssr引物具备可扩增性,共筛选出83对可扩增性引物。
接着对该83对可扩增ssr引物对扩大应用到4份不同品种牡丹皮样品中,pcr扩增体系及程序如上述所示。pcr扩增产物用质量浓度8%非变性聚丙烯酰胺凝胶电泳结合银染检测,筛选出多态性较好的ssr引物。
具体操作如下:
(1)清洗玻璃板、试样格、灌胶用量筒和烧杯,玻璃板气干;
(2)将平口玻璃板和凹口玻璃板放在桌面的支撑物上,玻璃板滴少量100%乙醇,用镜头纸均匀擦洗制胶面,气干;
(3)平口玻璃板和凹口玻璃板的三个边均匀的涂抹适量凡士林,两板重叠,用手将两块玻璃板在桌面上竖起,使两玻璃板底面平齐从而形成胶室;
(4)将胶室放入电泳仪主体,使凹口玻璃板的一面向内,插入楔形插板,将电泳仪主体放入原位制胶器内,根据实验要求的胶的厚度,旋转制胶器手柄至相应刻度,使楔形插板压紧胶室;
非变性聚丙烯酰胺凝胶配制:
(5)8%聚丙烯酰胺凝胶混合液配制(50ml):26.4ml去离子水,13.3ml30%丙烯酰胺,10ml5×tbe缓冲液,灌胶前迅速加入350μl10%过硫酸铵(催化剂),33μltemed(加速剂)。
(6)配胶、灌胶。针对所选用的胶的大小和厚度,量取适量8%聚丙烯酰胺凝胶混合液倒入干净烧杯,摇匀,灌胶,缓慢插入与胶厚度一致的试样格,待胶凝固30分钟以上。
采用dycz-24dn型电泳仪进行非变性聚丙烯酰胺凝胶电泳:
(1)凝胶聚合后,将电泳仪主体从原位制胶器上取出,放入电泳仪下槽中;
(2)将140ml0.5×tbe电泳缓冲液倒入由胶室和电泳仪主体形成的上槽中,使缓冲液没过凹口玻璃板;
(3)将200ml0.5×tbe电泳缓冲液倒入电泳仪下槽中;
(4)缓慢地将试样格垂直拔出;
(5)取3.5μlpcr扩增样品,加入0.7μl6×dna/rnaloadingbuffer,混匀;混匀后用移液枪将混合液及3μl50bpladderdnamarker点入上样孔;
(6)接通电泳仪电源与电泳仪之间的电泳导线,设定电泳参数。电压为200v·cm-1,电泳时间45分钟。
银染显色
(1)电泳结束后,缓慢取下凝胶,用去离子水漂洗2次,每次5分钟;
(2)将凝胶置于银染液(100ml水,1ml20%硝酸银)中,盖上遮光板使凝胶处于黑暗状态,在低速摇床上轻轻摇动13分钟染色。
(3)缓慢取下凝胶,用去离子水漂洗2次,每次5分钟;
(4)将凝胶转移到显影液(100ml3%氢氧化钠,1ml甲醛,4℃预冷)中,放置在低速摇床上轻轻摇动直至出现dna条带,
(5)将凝胶用去离子水迅速漂洗1次,室温干燥后进行凝胶成像及dna片段条带数目统计。
将所筛选出的多态性引物扩展到本发明所述所有牡丹皮dna中进行检测,基于以上实验结果,共筛选出10对多态性ssr引物对(图1~10):其序列信息如表4所示。
表4.序列信息
不同来源牡丹皮ssr引物对体系多态性结果:
根据dnamarker和引物信息中目标片段大小,人工判读10对多态性ssr引物在50份牡丹皮样本c小到大分别命名为a/b/c/d,纯合子命名为aa/bb/cc/dd,杂合子命名为ab/bd/cd/bd。
表5.牡丹皮样本情况
利用popgene32软件对10对多态性ssr引物在不同产地的50份牡丹皮样本dna中的扩增情况进行分析。等位基因数(na)的变化范围为2~4,平均2.8000,其中引物p10的最高,引物p2、p4和p9的最低。平均每个ssr位点有效等位基因数(ne)为2.0045。根据nei’s1973法计算观测杂合度(ho)范围为0.0816~0.6800,平均0.3947。期望杂合度(he)范围为0.3232~0.6455,平均0.4863。nei’s期望杂合度(h)范围为0.3200~0.6390,平均0.4814。多态信息含量(pic)从0.2688到0.5711,平均值为0.4069。引物p8和p10为高度多态性位点(pic>0.5),其余8个均为中度多态性位点(0.25<pic<0.5)。shannon信息指数(i)范围为0.5004~1.0944,平均0.7741。以上结果表明牡丹资源的遗传多样性比较丰富。(表6)
表6.ssr引物的遗传多样性分析
根据地理来源和品种将50份牡丹皮样本划分为5组进行群体间遗传多样性分析。以重庆垫江采集的“太平红”单瓣牡丹皮样本(编号s1~s12)作为第一组,重瓣样本(编号s13~s24)作为第二组,“凤丹”样本(编号s25~s32)作为第三组,安徽采集的“凤丹”样本(编号s33~s40)作为第四组,河南洛阳采集的牡丹皮样本(编号s41~s50)作为第五组。
popgene32分析结果显示,观测杂合度(ho)范围为0.2750~0.4917。期望杂合度(he)范围为0.2692~0.4263。nei’s期望杂合度(h)范围为0.2580~0.3993,h是反映群体遗传多样性的常用指标,根据遗传多样性对5个牡丹群体排序:第四组>第五组>第三组>第一组>第二组。等位基因数(na)的范围为1.700~2.400,第四组和第五组数量较多,第二组数量最少。shannon信息指数(i)范围为0.3639~0.6587,最高为第四组,最低为第二组。综合以上结果,可以看出安徽“凤丹”在5个牡丹群体中的遗传多样性最高(h=0.3993,i=0.6587),重庆垫江“太平红”重瓣牡丹的遗传多样性最低(h=0.2580,i=0.3639)。
第一组和第二组的观测杂合度(ho)均高于期望杂合度(he),近交系数(fis)均为负值(第一组:fis=﹣0.8656,第二组:fis=﹣0.9704),表明群体内的杂合子过剩,纯合子缺乏。第三组、第四组和第五组的观测杂合度(ho)均低于期望杂合度(he),近交系数均为正值(fis=0.0356~0.2129),表明群体内的杂合子缺失,纯合子过剩。5组牡丹的近交系数平均值为负值(fis=﹣0.2794),也表明群体杂合子过剩。(表7)
表7.不同来源牡丹皮的遗传多样性相关参数
根据50份牡丹皮样本间相应的nei’s遗传距离,在powermarkerv3.25软件上采用upgma法构建树状图。如图11所示,50份样本整体分为三个明显的分支cluster1、cluster2和cluster3。其中cluster1包括a和b两个分支,共有24份样本,均来自重庆垫江。a由所有的“太平红”重瓣牡丹(编号s13~s24)组成,b由所有的单瓣牡丹(编号s1~s12)组成。cluster1的聚类情况说明产地相同、花色相同的牡丹品种有较近的亲缘关系。cluster2中共有16份牡丹皮样本,包括8份采自重庆垫江“凤丹”样本(编号s25~s32)和8份采自安徽的“凤丹”样本(编号s33~s40))。从河南洛阳采集的10个观赏牡丹所产牡丹皮样本(编号s41~s50)都聚集在cluster3中。重庆垫江“太平红”。因而,基于ssr分子标记体系显示的牡丹皮样品多态性分析,通过聚类分析能有效的区分与鉴定不同来源的牡丹皮样品。
最后说明的是,以上优选实施例仅用以说明本发明的技术方案而非限制,尽管通过上述优选实施例已经对本发明进行了详细的描述,但本领域技术人员应当理解,可以在形式上和细节上对其作出各种各样的改变,而不偏离本发明权利要求书所限定的范围。
序列表
<110>西南大学
<120>一种牡丹皮的转录组ssr分子标记体系及其应用
<160>20
<170>siposequencelisting1.0
<210>1
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>1
ggtcctatctcttcacgaaaatc23
<210>2
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>2
catgaggttaacgcaaagga20
<210>3
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>3
cgtccgacgtacaactttga20
<210>4
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>4
gtgctcacctcttcctcgtc20
<210>5
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>5
atgtagccgaccccatgtag20
<210>6
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>6
tttgcacacaaaacatgcac20
<210>7
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>7
tcctctaaattatccgcccc20
<210>8
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>8
cggtgttgcagttgtagtcg20
<210>9
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>9
agggagggatggacaaaact20
<210>10
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>10
tcactagaaggagtgggagcat22
<210>11
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>11
ggggagatcagaaacacacaac22
<210>12
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>12
tcaaagaaaccccagttctcac22
<210>13
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>13
gagaaccatcattccattcaag22
<210>14
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>14
ctcaatctcttcttcctttgcc22
<210>15
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>15
ggcaaacaatttggcgtagt20
<210>16
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>16
cgcacccttccacaaataat20
<210>17
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>17
tgaagaaggtggtgaggagg20
<210>18
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>18
tggacaatgagagcaaaacg20
<210>19
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>19
tgtagctgaacctgtcgtgc20
<210>20
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>20
ctccccatcagtaactcgga20
- 还没有人留言评论。精彩留言会获得点赞!