一组绿豆InDel分子标记及其开发方法与流程
本发明涉及分子生物学、遗传学和基因组学技术领域的一种分子标记开发方法,尤其涉及一种一组绿豆indel分子标记的开发方法,还涉及该方法开发的一组绿豆indel分子标记。
背景技术:
绿豆是豆科菜豆族豇豆属中的一个栽培种,营养丰富,籽粒蛋白质含量高达19.5%~33.1%,高于水稻、小麦、玉米等禾谷类作物,属高蛋白、中淀粉、低脂肪类食物。不仅如此,绿豆还富含多种矿质元素、维生素及活性物质,具有解毒、抗菌抑菌、抗过敏、降血脂、降血压、抗肿瘤、预防癌症等功效,属于医食两用作物。绿豆根系中与其共生的根瘤菌有固氮能力,不仅满足自身生长的需要,也可供后茬作物使用。种植绿豆亦可改善土壤肥力和结构。此外,绿豆还具有喜温、生育期短、播种弹性大、耐贫瘠、耐荫,经济效益高等特点,使其深受广大农户的喜爱。因此,对于绿豆基础研究和育种研究十分重要。
目前,分子标记已广泛应用于绿豆遗传多样性分析、遗传图谱构建、目的基因定位和标记辅助育种等研究工作。但早期的分子标记如rflp、rapd等标记类型操作复杂且重复性较差。ssr标记虽然具有数量丰富,操作简单的优点,但需要对品种基因组中微卫星重复序列检索后,设计大批量的引物再进行多态性引物的筛选,多态性引物的比率较低,导致工作量较大。而snp标记虽然具有标记密度高,分布均匀等优点,但需要专用的基因分型设备,成本高,操作复杂,不适宜多数研究平台使用。因此,现有的绿豆分子标记存在操作复杂或多态性较差的问题。
技术实现要素:
为解决现有的绿豆分子标记存在操作复杂或多态性较差的技术问题,本发明提供一组绿豆indel分子标记及其开发方法。
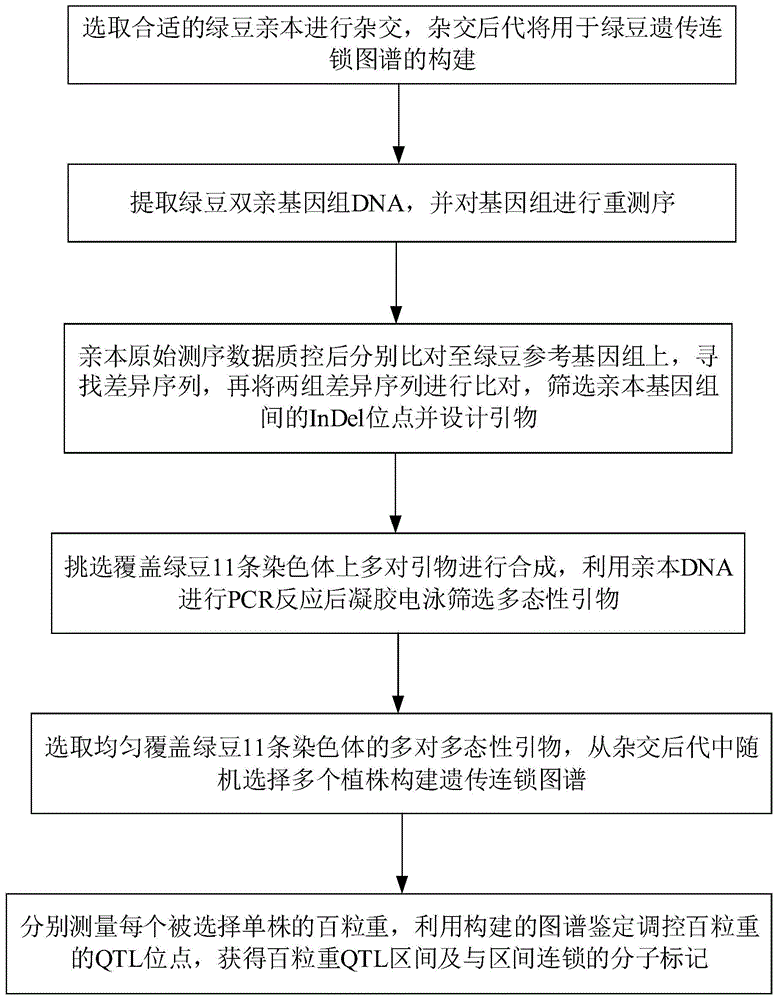
本发明采用以下技术方案实现:一种一组绿豆indel分子标记的开发方法,其包括以下步骤:
(1)选取绿豆双亲进行杂交,并利用杂交后代构建绿豆遗传作图群体;
(2)先提取所述绿豆双亲的基因组dna,再将提取的两个基因组dna片段化,以构建相应的两个基因组文库,最后对两个基因组文库进行双末端测序;
(3)先将步骤(2)中获得的测序数据质控后比对到绿豆参考基因组上,以获取所述绿豆双亲与所述参考基因组的差异位点,再将两组差异位点比对,筛选亲本基因组间的indel位点,最后在满足预设尺寸标准的indel位点两端设计引物,获得多对引物;
(4)挑选覆盖绿豆11条染色体的多对引物进行合成,再对合成引物进行pcr扩增效果评价,以选取在亲本基因组间表现出多态性的多对多态性引物;
(5)挑选均匀覆盖绿豆11条染色体的多对多态性引物,同时从所述杂交后代中随机选择多个植株,以构建遗传连锁图谱;
(6)在步骤(5)选择的植株收种后,先测量出每棵单株的百粒重,最后利用qtl分析软件鉴定调控百粒重的qtl位点,获得百粒重qtl区间及与区间连锁的分子标记。
本发明先选择合适的绿豆双亲进行杂交后构建作图群体,而后对作图亲本进行基因组重测序,随后将测序数据与参考基因组进行对比筛选差异序列,将获得的差异序列再进行对比以筛选亲本基因组间的indel位点,再针对大小合适的indel位点的两端设计引物,然后挑选出多对引物进行合成,同时进行pcr扩增,并根据电泳结果筛选多态性引物。再然后进一步选取多态性引物,从杂交后代中随机选择部分植株以构建遗传连锁图谱,最后测量出每棵单株的百粒重,并对调控绿豆百粒重的qtl位点进行检测,从而获得调控百粒重的qtl区间及区间连锁的分子标记。该发明解决了现有的绿豆分子标记存在开发困难、操作复杂或多态性较差的技术问题,得到了操作简单、多态性较好、遗传稳定、数量丰富和重复性好的技术效果。同时,本发明也为绿豆粒重的遗传改良提供分子标记,有利于绿豆分子标记辅助育种。
作为上述方案的进一步改进,对所述绿豆双亲进行杂交的方法包括以下步骤:
(1.1)根据所述绿豆双亲的花期,调整父本和母本的播种时间或分期播种,使所述父本和所述母本的花期相遇;
(1.2)在所述母本生长至初花期后,去除健康植株上的幼荚和已开花朵;
(1.3)去除母本植株上的待开放花朵中的雄蕊,并在隔天后使用所述父本的花粉对去雄的花朵进行授粉以获得杂交荚;
(1.4)待杂交荚成熟后,先单荚收获所述杂交荚,最后再单荚脱粒后播种;
(1.5)观察步骤(1.4)中播种幼苗的幼茎色,筛选出真杂种植株,并将f1代收获后脱粒播种以构建f2代绿豆遗传作图群体。
进一步地,所述父本的绿豆品种为潍绿11号,所述母本的绿豆品种为苏绿16-10;在步骤(1.1)中,所述父本在所述母本播种20天后播种或分2~3期播种;在步骤(1.5)中,在所述幼茎色为紫色时,判定相应的幼苗为真杂种植株,在所述幼茎色为绿色时,判定相应的幼苗为假杂种植株;在步骤(1.3)中,去雄的方法包括以下步骤:先去除待开放花朵的旗瓣和翼瓣,再从花朵背部打开龙骨瓣,最后去除花朵中的雄蕊并对花柄系线进行标记。
再进一步地,在步骤(1.3)中,对去雄的花朵进行授粉的方法包括以下步骤:先选取旗瓣微微张开的父本花朵,再去除所述父本花朵的旗瓣、翼瓣以及龙骨瓣,最后夹住所述父本的花柄并将花粉涂抹于所述母本外露柱头的绒毛上;在授粉后3~4d,可检查杂交是否成功,并在确定杂交成功后执行步骤(1.4),否则执行步骤(1.3)。
再进一步地,在步骤(3)中,还对测序数据进行质量过滤,且过滤方法包括以下步骤:先去除接头污染,再去除不满足预设质量标准和长度标准的原始测序序列。
作为上述方案的进一步改进,在步骤(4)中,先挑选所述indel长度在15~30bp,产物长度在100~300bp的引物进行合成;在评价pcr扩增效果时,pcr反应体系的体积为10μl,且包括1μl浓度为50ng/μl基因组dna、0.5μl浓度为10μmol/l的正向引物、0.5μl浓度为10μmol/l的反向引物、5μl2×pcrmastermix以及3μlddh2o;pcr反应扩增参数为:94℃预变性4min;94℃变性30s,退火(55~60℃)30s,72℃延伸30s,35个循环;最后72℃充分延伸10min,保存温度为8℃。
进一步地,调控绿豆百粒重的qtl位点包含3个染色体区间,且分别与所述三个区间连锁的引物对分别为id3-7和id3-8、id7-10和id7-7、id10-3和id10-4。
本发明还提供一组绿豆indel分子标记,该分子标记通过上述任意所述的一组绿豆indel分子标记的开发方法开发获得。
相较于现有的绿豆分子标记,本发明的一组绿豆indel分子标记及其开发方法,其具有以下有益效果:
1、该一组绿豆indel分子标记的开发方法,其首先选取绿豆双亲进行杂交,并利用杂交后代构建绿豆遗传作图群体,随后提取绿豆双亲的基因组dna并进行基因组重测序,然后将亲本质控后的测序数据分别与参考基因组进行对比,筛选出差异序列,再将两组差异序列进行比对筛选亲本基因组间的indel位点,针对indel位点两端序列设计引物。而后选取覆盖绿豆11条染色体的多对引物进行合成,同时对合成引物进行pcr扩增,并评价扩增的多态性效果获得多对多态性引物。再然后从表现多态性引物对中选取均匀覆盖绿豆11条染色体的多对多态性引物,同时从杂交后代中随机选取部分植株以构建遗传连锁图谱。最后测量出每棵单株的百粒重,并对调控百粒重的qtl位点进行检测,从而获得调控百粒重的染色体区间及与区间连锁的分子标记。该开发方法利用重测序技术对作图亲本的基因组重测序,仅针对双亲的基因组差异位点设计引物,极大了提高了引物设计的成功率和多态性引物的比率,从而使得分子标记的多态性更佳。同时,在分子标记的提供过程中,基于不同品种的dna序列,利用差异序列进行indel标记的开发,提高了多态性引物的成功率。
2、该绿豆分子标记为indel标记,具有数量丰富、遗传稳定、重复性好、操作简单的优点,而且为多态性较好的indel标记,可用于绿豆遗传多样性分析、遗传连锁图谱的构建以及目的基因定位和分子标记辅助育种,弥补目前绿豆indel标记较为缺乏的不足。
3、利用该方法开发indel标记构建的遗传连锁图谱可对数量性状的qtl位点进行检测,可同时鉴定出主效位点和微效位点,有益于绿豆重要农艺性状基因的克隆和绿豆基因组学的发展。
附图说明
图1为本发明实施例1的一组绿豆indel分子标记的开发方法的流程图;
图2为图1所示出的绿豆indel分子标记的开发方法所获得绿豆遗传连锁图谱的示意图;
图3为本发明实施例1中绿豆杂交后代百粒重频次分布图;
图4为本发明实施例1中调控百粒重的qtl在染色体上的分布及lod值曲线图。
具体实施方式
为了使本发明的目的、技术方案及优点更加清楚明白,以下结合附图及实施例,对本发明进行进一步详细说明。应当理解,此处所描述的具体实施例仅用以解释本发明,并不用于限定本发明。
实施例1
请参阅图1,本发明提供了一种一组绿豆indel分子标记的开发方法,该方法用于提供现有的绿豆分子标记所不具有的一种绿豆indel分子标记,该分子标记针对不同亲本基因组间插入/缺失差异而设计的标记。在本实施例中,该绿豆indel分子标记的开发方法及其应用包括以下步骤,即包括步骤(1)-(6)。
(1)选取绿豆双亲进行杂交,并利用杂交后代构建绿豆遗传作图群体,即选择合适的双亲杂交后构建作图群体。在本实施例中,父本是潍绿11号,为矮秆中粒紫色茎秆的绿豆品种。母本为苏绿16-10,是高杆大粒绿色茎秆的绿豆品种,双亲经多代自交纯化后单株收获并播种。具体而言,绿豆双亲进行杂交的方法包括以下步骤,即包括步骤(1.1)-(1.5)。
(1.1)根据绿豆双亲的花期,调整父本和母本的播种时间或分期播种,使父本和母本的花期相遇。由于双亲花期相差约20d左右,父本在母本播种后20天后播种或分2~3期播种,即潍绿11号可在苏绿16-10播种后20d左右播种或分2~3期播种。
(1.2)在母本生长至初花期后,去除健康植株的幼荚和已开花朵,即选择生长健壮无病虫害的植株,去除幼荚和已开过的花。目的是防止这些幼荚和已开花朵长成成熟荚后影响试验中正常杂交产生的果实,造成假杂交种的产生,因此进行本步骤后可以大大提高杂交荚的纯度。另一方面也保证了真杂交荚的养分供应。
(1.3)去除母本植株上的待开放花朵中的雄蕊,并在隔天后使用父本的花粉对去雄后的花朵进行授粉。本实施例中去雄的方法包括以下步骤:先去除待开放花朵的旗瓣和翼瓣,再从花朵背部打开龙骨瓣,最后去除花朵中的雄蕊并对花柄进行标记。去雄时间最好选择在下午4:00以后,并使用镊子尖小心去除旗瓣和翼瓣,从花朵背部打开龙骨瓣去除雄蕊,用较细的彩色线系于花柄处以示标记,并登记好每种颜色的线代表的杂交时间和父、母本。
其中,对去雄的花朵进行授粉的方法包括以下步骤:先选取旗瓣打开的父本花朵,再去除父本花朵的旗瓣、翼瓣以及龙骨瓣,最后夹住父本花朵的基部并将花粉涂抹于母本的花外露柱头的绒毛上。在授粉后3~4d,还可检查杂交是否成功,并在确定杂交成功后执行步骤(1.4),否则执行步骤(1.3)。授粉的时间在母本去雄后的第二天,授粉时选择旗瓣微微打开的父本花朵,去除旗瓣、翼瓣和龙骨瓣,用镊子夹住花朵基部将花粉涂抹于母本花外露柱头的绒毛上,若花粉量较少可用多个父本花朵多次涂抹,授粉在上午8:00之前完成。同一绿豆植株可多次做杂交。授粉后3~4d即可检查杂交是否成功。杂交全部完成后,去除杂交植株上后续长出的幼荚,保证杂交荚的养分供应。
(1.4)待杂交荚成熟后,先单荚收获杂交荚,最后再单荚脱粒后播种。在本实施例中,单荚收获苏绿16-10植株上的杂交荚,共收获10个杂交荚,每荚粒数在1~10个之间。
(1.5)观察步骤(1.4)中播种幼苗的幼茎色,并将f1代收获后脱粒播种以构建f2代绿豆遗传作图群体。其中,单荚脱粒后播种于大田里,苗期观察幼茎色。其中,在幼茎色为紫色时,判定相应的幼苗为真杂种植株,在幼茎色为绿色时,判定相应的幼苗为假杂种植株。经鉴定,此次杂交获得种子全部为真杂交种,f1代收获后脱粒播种约1200粒种子于大田构建f2代作图群体。
(2)先提取所述绿豆双亲的基因组dna,再将提取的两个基因组dna片段化,以构建相应的两个基因组文库,最后对两个基因组文库进行双末端测序。而且,本步骤中还对测序数据进行质量过滤,且过滤方法包括以下步骤:先去除接头污染,再去除不满足预设质量标准和长度标准的原始测序序列。
具体地,本实施例中可以利用基因组dna提取试剂盒提取潍绿11和苏绿16-10的基因组dna,将基因组总dna片段化,然后末端修复、3'端加a、连接测序接头,构建标准的基因组文库(文库的插入片段大小控制在400bp左右)。并且,本实施例采用第二代测序技术,并基于illuminahiseq测序平台,对构建的两个dna文库进行双末端测序。测序结果显示潍绿11的dna文库共获得原始测序序列106896086条,包含16141308986bp,q30为87.92%;而苏绿16-10基因组文库获得原始测序序列103902578条,包含15689289278bp,q30为86.96%。
由于原始测序数据包含一些带接头、低质量的序列,这些序列会对后续的信息分析造成很大的干扰,为了保证后续信息分析质量,本实施例需要对下机数据进行进一步过滤,即进行质量过滤。质量过滤主要包括以下几步:1)去除接头污染,采用adapterremoval去除3'端的接头污染;2)质量过滤,采用滑动窗口法进行质量过滤,窗口大小设置为5bp,步长设置为1bp,去除低质量的原始序列;3)长度过滤,若双末端测序的序列中任意一条序列的长度≤50bp,则去除该双末端序列。数据过滤后潍绿11文库包含高质量的序列92882068条,有13314018337个高质量碱基,占原始测序碱基总数的82.48%。苏绿16-10文库有88882540个高质量序列,包含12671857644个高质量碱基,占原始测序碱基总数的80.77%。
(3)先将步骤(2)中获得的测序数据质控后比对到绿豆参考基因组上,以获取所述绿豆双亲与所述参考基因组的差异位点,再将两组差异位点比对,筛选亲本基因组间的indel位点,最后针对大小合适(即满足预设尺寸标准)的indel位点(5-150bp)两端设计引物,获得多对引物并进行pcr验证。其中,产物长度控制在100~400bp,退火温度为55~60℃。
在本实施例中,采用bwamem程序将经过过滤后得到的高质量数据比对到参考基因组上,比对的参数均按照bwamem的默认参数。潍绿11文库测序数据可比对至基因组上的序列有92018265条,比对率为99.07%。这其中包含9745593607个碱基,测序深度为22.66×,基因组覆盖度为97.27%(参考基因组中至少被1条序列覆盖的位点占基因的百分比),与参考基因组相比有79,391个差异位点。
苏绿16-10文库测序数据可比对至基因组上的序列有88233697条,比对率为99.27%,包含10,558,443,206个碱基,测序深度为24.55×。基因组覆盖度为97.33%,与参考基因组相比有102,875个差异位点。将比对后获得的两组差异位点再次比对,筛选亲本基因组间的indel位点。并利用primer3针对indel大小在5~150bp的位点两端设计引物,产物长度控制在100~400bp,退火温度在55~60℃,成功设计出15213对引物。
(4)挑选覆盖绿豆11条染色体的多对引物进行合成,再对合成引物进行pcr扩增效果评价,以选取在亲本基因组间表现出多态性的多对多态性引物。其中,优先挑选indel位点大小在15~30bp,产物大小在100~300bp的引物进行合成,本实施例中优先选取了209对引物进行合成。
利用潍绿11号和苏绿16-10对合成的引物进行pcr扩增效果评价。pcr反应体系的体积为10μl,包括1μl浓度为50ng/μl基因组dna、0.5μl浓度为10μmol/l的正向引物、0.5μl浓度为10μmol/l的反向引物、5μl2×pcrmastermix以及3μlddh2o。在pcr反应程序为:94℃预变性4min;94℃变性30s,退火(根据引物设定,通常为55~60℃)30s,72℃延伸30s,35个循环;72℃充分延伸10min,8℃保存。用8%的聚丙烯酰胺凝胶电泳进行pcr产物的分离,电泳结束后,拍照并读带。统计结果表明有180对引物可扩增出多态性片段,占比86.12%,其余有4对引物表现出非特异性扩增或无扩增产物,25对引物可扩增出目的条带但无多态性。统计结果表明180对引物扩增效果良好,而且符合预期。
(5)挑选均匀覆盖绿豆11条染色体的多对多态性引物,同时从所述杂交后代中随机选择多个植株,以构建遗传连锁图谱。在本实施例中,挑选145对扩增效果较好,且在染色体上均匀分布的多态性引物用于构建遗传连锁图谱。从f2代群体中随机选择136个植株用于图谱的构建。本实施例分别对145对引物在子代中的分离情况进行χ2检验,分析其是否符合孟德尔分离比,去除严重偏分离的标记19对,偏分离比率为13.10%。剔除偏分离分子标记,利用qtlicimapping软件对126个标记进行连锁分析与图谱构建,其引物信息如表1所示。请参阅图2以及表2,最终得到一张覆盖基因组长度为692.56cm,平均图距为6.02cm的绿豆遗传连锁图谱。
表1引物信息表
表2绿豆遗传连锁图谱的信息表
(6)在步骤(5)选择的植株收种后,先测量出每棵单株的百粒重,最后利用qtl分析软件鉴定调控百粒重的qtl位点,获得百粒重qtl区间及与区间连锁的分子标记。在本实施例中,对选取的136个f2植株单株收种后,调查每棵单株的百粒重。正态分布检测表明单株百粒重近似正态分布,可用于qtl分析,请参阅图3。利用qtlicimapping软件进行百粒重qtl位点的检测,扫描步长设置为1cm,lod阈值设置为2.5。请参阅图4和表3,共检测到三个影响百粒重的qtl位点,包括1个主效qtl和两个微效qtl。实际上,调控绿豆百粒重的qtl位点包含3个染色体区间,且分别与所述三个区间连锁的引物对分别为id3-7和id3-8、id7-10和id7-7、id10-3和id10-4。
表3qtl位点检测数据统计表
综上所述,本实施例的绿豆indel分子标记的提供方法,其具有以下优点:
该绿豆indel分子标记的开发方法,其首先选取绿豆双亲进行杂交,并利用杂交后代构建绿豆遗传作图群体。随后提取绿豆双亲的基因组dna并进行片段化,构建两个亲本基因组文库并对其进行双末端测序。然后将前一步的测序数据与参考基因组进行对比分别获得亲本与参考基因组的差异序列,将两组差异序列进一步进行对比,从而筛选出亲本基因组间的indel位点,以便于针对indel位点两端设计引物。而后选取分布在绿豆11条染色体上的多对引物进行合成,同时对合成引物进行pcr扩增,并评价扩增效果,进而选取出多对多态性引物。再然后从多态性引物对中选取均匀覆盖绿豆11条染色体的多态性引物对,同时从杂交后代中选取植株以构建遗传连锁图谱,测量出每棵单株的百粒重,并进行正态性分布检测和qtl位点检测,从而获得调控百粒重染色体区间及与区间连锁的分子标记。该提供方法利用重测序技术对作图亲本的基因组重测序,仅针对双亲的基因组差异位点设计引物,极大了提高了引物设计的成功率和多态性引物的比率,从而使得分子标记的多态性更佳。同时,在分子标记的开发过程中,基于不同品种的dna序列,利用差异序列进行indel标记的开发,提高了多态性引物的成功率。
实施例2
本实施例提供了一组绿豆indel分子标记,该分子标记通过实施例1中的绿豆indel分子标记的开发方法获得。在本实施例中,该绿豆indel分子标记采用实施例1中的表1中所展示的引物对的任意一组或几组引物对进行pcr扩增得到。
该绿豆indel分子标记为indel标记,具有数量丰富、遗传稳定、重复性好、操作简单的优点,而且为多态性较好的indel标记,可为绿豆遗传多样性分析、遗传连锁图谱的构建以及目的基因定位和分子标记辅助育种提供indel标记,弥补目前绿豆indel标记较为缺乏的不足。
实施例3
本实施例提供了一种绿豆杂交方法,该绿豆杂交方法可应用于实施例1中的绿豆遗传作图群体的获得。其中,绿豆杂交方法包括以下步骤。
1、提供两种绿豆亲本,一种亲本作为父本,另一种亲本作为母本。这两种绿豆亲本可以采用实施例1中所使用的潍绿11号和苏绿16-10,也可以使用其他绿豆品种,具体种类可以根据实际需要进行选择,但是保证绿豆双亲的农艺性状差异比较大,以便于后续进行的表型鉴定和qtl位点鉴定工作。
2、根据绿豆双亲的花期,调整父本和母本的播种时间或分期播种,使父本和母本的花期相遇。这里,由于绿豆双亲的开花期差距较大,同时播种时会使双亲开花时间错开,无法进行后续的授粉工作。为此,在实际操作中,可以延后部分短花期的绿豆品种的播种时间,或者进行分期播种,使双亲开花时间能够位于同一个时段内,以便后续进行授粉。例如,当本实施例的双亲采用实施例1中的绿豆双亲品种时,由于双亲花期相差约20d左右,潍绿11号可在苏绿16-10播种后20d左右播种或分2~3期播种。
3、在母本生长至初花期后,去除健康植株的幼荚和已开花朵,即选择生长健壮无病虫害的植株,去除幼荚和已开过的花。目的是防止这些幼荚和已开花朵长成成熟荚影响试验中正常杂交产生的果实,造成假杂交种的产生,因此进行本步骤后可以大大提高杂交荚的纯度,同时也保证真杂交荚的营养供应。
4、去除植株的待开放花朵中的雄蕊,并在隔天后使用父本的花粉对去雄的花朵进行授粉。本实施例中去雄的方法包括以下步骤:先去除待开放花朵的旗瓣和翼瓣,再从花朵背部打开龙骨瓣,最后去除花朵中的雄蕊并对花柄进行标记。在去雄的时间选择上优先选择下午4:00以后,并使用镊子尖小心去除旗瓣和翼瓣,从花朵背部打开龙骨瓣去除雄蕊,用较细的彩色线系于花柄处以示标记,并登记好每种颜色的线代表的杂交时间和父本、母本。
其中,对去雄的花朵进行授粉的方法包括以下步骤:先选取旗瓣打开的父本花朵,再去除父本花朵的旗瓣、翼瓣以及龙骨瓣,最后夹住父本花朵的基部并将花粉涂抹于母本的花外露柱头的绒毛上。在授粉后3~4天,还可检查杂交是否成功,并在确定杂交成功后进行后续步骤,否则需重新进行杂交工作。授粉的时间在母本去雄后的第二天,授粉时选择旗瓣微微打开的父本花朵,去除旗瓣、翼瓣和龙骨瓣,用镊子夹住花朵基部将花粉涂抹于母本花外露柱头的绒毛上,若花粉量较少可用多个父本花粉多次涂抹,授粉在上午8:00之前完成。同一株绿豆植株可多次做杂交。授粉后3~4d即可检查杂交是否成功。杂交全部完成后,去除杂交苗上后续长出的幼荚。
5、单荚收获杂交荚,脱粒后播种。在本实施例中,单荚收获苏绿16-10植株上的杂交荚,共收获10个杂交荚,每荚粒数在1~10个之间。
6、观察上一步骤播种后幼苗的幼茎色,判定是否为真杂种植株,并将f1代收获后脱粒播种以构建f2代遗传作图群体。其中,单荚脱粒后播种于大田里,苗期观察幼茎色。其中,在幼茎色为紫色时,判定相应的幼苗为真杂种植株,在幼茎色为绿色时,判定相应的幼苗为假杂种植株。经鉴定,此次杂交获得种子全部为真杂交种,f1代收获后脱粒播种约1200粒种子于大田构建f2代作图群体。
本实施例的这种绿豆杂交方法,其能够获取到纯正的杂交后代,而且经过实施例1中对杂交种的鉴定,真杂交种含量高,杂交效果非常好。因此,该绿豆杂交方法还可以作为其他的绿豆育种方法中,可以提高绿豆的育种效果和效率,提升绿豆育种质量。
实施例4
本实施例提供了一张绿豆indel标记遗传连锁图谱,该绿豆遗传连锁图谱中的引物对采用实施例1中表1所示引物对。请继续参阅图2,染色体1(chromosome1)中包含id1-1、id1-2、id1-3、id1-11、id1-10和id1-9、id1-7、id1-5、id1-4。染色体2(chromosome2)中包含id2-1、id2-2、id2-7和id2-3、id2-4、id2-5、id2-6、id2-8、id2-9。染色体3(chromosome3)中包含id3-1、id3-2、id3-3、id3-4、id3-5和id3-6、id3-7、id3-8、id3-9。染色体4(chromosome4)中包含id4-5、id4-4、id4-1、id4-3、id4-2、id4-6和id4-7、id4-8、id4-9、id4-10、id4-11。染色体5(chromosome5)中包含id5-1、id5-2、id5-3、id5-4、id5-5、id5-6和id5-7、id5-8、id5-9、id5-10、id5-11、id5-12、id5-13。染色体6(chromosome6)中包含id6-1、id6-2、id6-3、id6-4、id6-7、id6-8、id6-6、id6-5、id6-9、id6-10和id6-11、id6-12、id6-13、id6-14、id6-17、id6-16。染色体7(chromosome7)中包含id7-20、id7-19、id7-18、id7-17和id7-16、id7-1、id7-2、id7-3、id7-4、id7-5、id7-6、id7-10、id7-7、id7-8、id7-9、id7-13、id7-12、id7-11、id7-15、id7-14。染色体8(chromosome8)中包含id8-1、id8-2、id8-3、id8-4、id8-5和id8-6、id8-7、id8-8、id8-9、id8-10、id8-11、id8-12。染色体9(chromosome9)中包含id9-1、id9-2、id9-3、id9-4、id9-5、id9-12和id9-11、id9-10、id9-9、id9-8、id9-7、id9-6。染色体10(chromosome10)中包含id10-1、id10-2和id10-3、id10-4、id10-5、id10-6。染色体11(chromosome11)中包含id11-1、id11-2和id11-3、id11-4、id11-5、id11-6、id11-7、id11-8、id11-9。这样,通过将表1中的引物对构建,组成成了本实施例中的绿豆遗传连锁图谱,该图谱包含绿豆11条染色体,覆盖基因组长度为692.56cm,平均图距为6.02cm。
实施例5
本发明提供了一种鉴定调控绿豆百粒重qtl位点的方法,利用该方法鉴定出调控百粒重的3个qtl区间,可用于绿豆百粒重的遗传改良。与百粒重区间连锁的分子标记可用于标记辅助育种。该方法也可用于其他数量性状如花期、株高等调控位点的鉴定,可同时鉴定出主效基因和微效基因。
以上所述仅为本发明的较佳实施例而已,并不用以限制本发明,凡在本发明的精神和原则之内所作的任何修改、等同替换和改进等,均应包含在本发明的保护范围之内。
- 还没有人留言评论。精彩留言会获得点赞!