一种用于法医学个体识别的59个微单倍型遗传标记分型体系及其应用的制作方法
本发明属于法医学领域,具体涉及一组用于法医学检测的59个微单倍型遗传标记及其在混合样本检测中的应用。
背景技术:
在过去三十年里,随着分子生物学的进步,法医dna分型技术已成为司法部门强有力的打击犯罪工具。目前,基于复合pcr扩增和毛细管电泳(capillaryelectrophoresis,ce)的短串联重复序列(shorttandemrepeat,str)分型方法是法医物证鉴定的金标准。常染色体str被广泛应用于个体识别、亲权鉴定和混合样本分析等法医实践中。其中,混合样本检验及其结果解释是目前法医物证实验室公认的技术难题之一,也是法医物证领域亟需解决的科学难题。
混合样本指由两名或两名以上不同个体的同种或不同种生物检材(如血液、唾液、精液、阴道分泌物及脱落上皮细胞等)组成的样本。混合样本在法医dna检验中非常常见,最典型的是强奸案或轮奸案中的精液与阴道分泌物组成的混合斑或混合精斑。另外还有凶杀、斗殴案现场的混合血斑、混合体液斑等。除此之外,随着近年来现场勘查技术的进步及dna检测灵敏度的提高,接触dna被广泛应用于案件侦破中。接触dna通常混合着大量背景dna,属于不平衡混合样本,其分析难度大,为案件的顺利侦破带来了很大挑战。由于混合样本包含两名或多名提供者的dna信息,样本提供者数量常是未知数,加之案发现场检材质量不稳定,混合样本在实际应用中的检测效率较低,尚无法满足法庭科学质证标准的要求,使得混合样本在刑事案件调查中所能发挥的证据价值有限。新遗传标记的开发、检测及应用不断推动着法医遗传学的发展进步,因而高效能的混合样本分析遗传标记的开发及对应分型体系的建立将是混合样本研究的关键突破口,对凶杀案和轮奸案等棘手刑事案件的定性和侦破至关重要。
目前,常染色体ce-str分型方法被广泛应用于个体识别、亲权鉴定和混合样本分析等法医实践中。然而,该技术应用于混合样本、尤其是不平衡混合样本分析时具有明显的局限性:首先,荧光标记多基因座str复合扩增检测技术中经常存在影子带(stutter)、等位基因共享(allelesharing)、等位基因丢失或插入(alleledrop-out/in)以及pcr扩增不均衡等多种问题。其中,str结构特点导致的影子带峰高可达主峰的15%,是混合样本分型解释中最大的干扰因素。基于下一代测序(next-generationsequencing,ngs)平台的str分型技术较ce分型虽优势明显,但仍然无法避免影子带等劣势,平均影子带比例甚至较ce检测高出约3%,影响混合物各组分分型的推断。其次荧光标记复合扩增技术的难度使得可混合的遗传标记数量和种类有限,由于等位基因共享现象的存在,使得个体识别能力不足。此外,复合扩增体系中基因座的pcr扩增效率不同导致的峰高或峰面积等定量信息的偏向性和等位基因丢失现象均增加了混合样本分型数据分析的难度,进一步增加了结果解释的不确定性。因此,就目前的检测技术,当次要组分占总dna至少5-10%时混合样本才能被有效地检测,如需获得全部分型数据,次要组分所占比例至少需要20%,这与法医实践中常见的混合样本组成不符。
单核苷酸多态性(single-nucleotidepolymorphism,snp)遗传标记也可应用于混合样本分析。snp属于基因序列多态性,扩增时不会出现stutter峰,是法医物证领域另一类常用遗传标记。与str相比,snp具有突变率低、扩增片段短等优点。但snp多为二等位基因,多态性不高,往往需要多达上千个snp才能有较好的混合样本分析效果,实验成本较高,数据分析难度大,限制了其在混合样本分型中的应用。近年来,也有研究人员将str与插入缺失多态性(deletionandinsertionpolymorphism,dip)或snp联合应用,形成了dip-str或snp-str等新型的复合遗传标记。相较于常染色体str,dip-str遗传标记虽不受混合性别的限制、检测灵敏度高、系统效能更佳,但其分析效果依赖于dna提供者之间的不匹配,对混合样本的基因型组合有所要求,如果次要组分只有一个有效等位基因,其个体识别力将大打折扣。
因此,以kidd为代表的学者们提出微单倍型(microhaplotype)是理想的法医学遗传标记,在法医学各个领域,尤其是始祖推断和混合样本分析中具有广泛的应用前景,受到了广泛关注。微单倍型指由2个或多个snp组成,至少存在3种单倍型的紧密连锁的snp群组(<250bp)。微单倍型是多个snp等位基因的组合,每一种组合方式即可视为一种等位基因,因此其多态性更高。目前,应用有效等位基因数ae值(effectivenumberofalleles)评估微单倍型应用于混合样本分析的实用价值。据kidd等人结果显示,由4个或4个以上snp组成的微单倍型具有很高的ae值(通常大于3),snp数越多其ae越高,越适合应用于混合样本分析。结合ngs技术的微单倍型分型技术并非特异性扩增次要组分dna,故可获得混合样本中的全部信息,其等位基因测序深度(doc,degreeofcoverage)可作为混合样本各组分来源区分和混合比例(mx)的有力提示。因此,基于ngs的微单倍型分型技术在混合样本分析应用中不仅在避免影子带等方面比str、snp-str和dip-str等标记更具优势,也利于后续数据分析。
但目前无论是kidd团队发表的微单倍型,还是后续其他团队筛选的微单倍型遗传标记,其微单倍型多态性仍不够高,ae值通常在3左右,包含的snp数量小于5个,对于复杂的混合样本分析效果有限,不能完全满足混合样本检测需求,且鉴于ngs测序技术存在的测序错误问题(错误率介于0.1-1%之间),微单倍型中所包含snp数量太少可能存在假阳性。
技术实现要素:
针对目前的微单倍型遗传标记所包含snp少、ae值不够高的问题,利用千人基因组数据信息,筛选包含snp数量≥6个、ae值更高的微单倍型,是本发明需要解决的第一个技术难题。另外,由于法医学样本多为少量或微量dna样本,固构建复合扩增体系,以节约dna用量,提高该体系的使用范围是本发明所需解决的第二个技术难题。通过上述突破,以期建立系统效能更好的混合样本分析体系,进一步攻破混合样本分析的难题。
为实现上述目的,本发明采取的技术方案为:
本发明用于法医学检测的59个微单倍型遗传标记通过以下方法获得:
1.微单倍型筛选方法
(1)编写perl程序,从千人基因组数据库(phase2)中筛选微单倍型遗传标记。筛选规则:长度小于200bp,至少包含6个snp位点,上下游各有60bp的保守侧翼序列。
(2)根据筛选规则获取微单倍型基因型、等位基因频率,并计算目标位点在不同人群中的ae值。ae的计算公式为1/σpi2,pi为第i个等位基因的频率。ae值越高,反映该基因座上等位基因数目越多且等位基因频率分布均匀,其混合样本检测效能越高。
(3)对于在中国汉族群体中ae值高的微单倍型标记,结合等位基因频率大于0.1的位点个数,进一步进行人工筛选和序列检查,选择ae值高且频率大于0.1的等位基因数目多的位点,同时排除包含高gc序列、单碱基重复序列或复杂重复序列等序列特征的位点。
2.所选59个微单倍型标记具体信息见表1。
表1:59个微单倍型标记的具体信息
3.基因组dna提取及定量
(1)基因组dna提取:利用qiaampdnabloodminikit,根据试剂盒说明书从全血样本中提取全基因组dna。此发明还适用于从其他人类组织或检材中提取的基因组dna。具体可为血斑、精液、精斑、口腔上皮细胞、骨骼、毛发、唾液、唾液斑或汗液等。
(2)基因组dna定量:利用qubittmdsdnahsassaykit进行样本dna定量。
4.复合扩增、测序文库构建及测序
(1)复合pcr扩增:
a、复合扩增体系引物对共59对,用于微单倍型分型,其法医学应用范围包括:单一个体微单倍型分型,用于个体识别;单一个体微单倍型分型,用于亲权鉴定;混合样本微单倍型分型,用于混合斑分析。每一对引物原始浓度100μm,每管取1μl混合后加水稀释至1μm,用于下一步复合扩增。引物合成时可根据测序平台加不同的测序接头序列。本发明使用了illumina平台测序接头序列。扩增59个微单倍型所用引物见表2。
表2:59个微单倍型所用引物序列
b、复合扩增使用kapa2gfastmultiplexpcrkit进行。
第一轮pcr体系为:模板dna50ng,2×kapa2gfastmultiplexmix扩增缓冲液(含mg2+)12.5μl,正向引物混合物(1μm)1μl,反向引物混合物(1μm)1μl,ddh2o补足至25μl。第一轮复合扩增pcr程序为:94℃,3min;98℃,10s,58℃,5min,68℃,30s,6个循环;68℃,5min;4℃,贮存。
第二轮pcr体系为:第一轮pcr反应纯化产物22μl,2×kapa2gfastmultiplexmix扩增缓冲液(含mg2+)25μl,i5(10pmol/μl)1μl,i7(10pmol/μl)1μl,ddh2o补足至50μl。第二轮复合扩增pcr程序为:94℃,3min;98℃,10s,60℃,30s,68℃,30s,15个循环;68℃,5min;4℃,贮存。
(2)二代测序文库构建及测序:利用vahtstmdnacleanbeads磁珠,根据产品说明书对两轮pcr产物进行纯化。用qubit3.0测定纯化后的文库浓度,并进行agilent2100检测,正常文库主峰片段为380bp,无接头及大片段污染。文库在illuminanextseq500平台上机测序,测序模式为pe150。
(3)二代测序数据分析:
对测序得到的fastq文件使用soapnuke软件过滤低质量的read,进行质量控制;使用bwa完成比对,并用samtools将比对结果进行格式转换和排序。用samtools和perl编写的小程序统计比对率和目标区域覆盖情况。用samtools和perl编写的小程序筛选比对到目标区域的read,并根据每条read的比对信息得到单体型结果。此处剔除比对质量低和目标位点碱基情况不明的read。每个片段的snp组合到一起,即为该个体在该染色体上的单倍型分型。
微单倍型频率计算:先用samtoolsview将比对结果进行读取,然后用perl程序根据需要检测的单体型区域将潜在有用的read与扩增子相对应,对于每一条有用的read根据比对结果中cigar项进行解析,得到该read所支持的基因型,统计每个扩增子中得到的基因型(该处为原始的分型结果),频率是某个基因型的read数除以该扩增子中所有有效的read数。
本发明的有益效果:
与目前常用的遗传标记str或snp相比较,由连锁snp组成的微单倍型具有突变率低、稳定性高、等位基因多和分型难度等同单个snp(可通过短片段扩增)等特点,既可弥补str的突变率高、影子带干扰、等位基因丢失等缺陷,也可弥补snp多态性不高的劣势,在解决法医混合样本分型的疑难检案中更具优势。
与初步报道的微单倍型遗传标记相比较,本发明所报道的微单倍型遗传标记具有以下优点:
(1)多态性高,即ae值高。目前报道的微单倍型ae值多为3左右,较少有ae值超过5的微单倍型。而本发明体系中的59个微单倍型在中国汉族人群和千人基因组所有人群中的平均ae值分别高达4.84和5.44。
(2)本发明筛选的微单倍型遗传标记所包含snp数量至少为6个,较已经报道的微单倍型所含snp数多,可降低因ngs技术中测序错误问题导致的假阳性分型。
(3)从分型结果看,本发明报道的59个微单倍型组合系统效能高,遗传标记数量足够,可应用于两个体的混合样本甚至多个体的复杂混合样本的分析。
附图说明
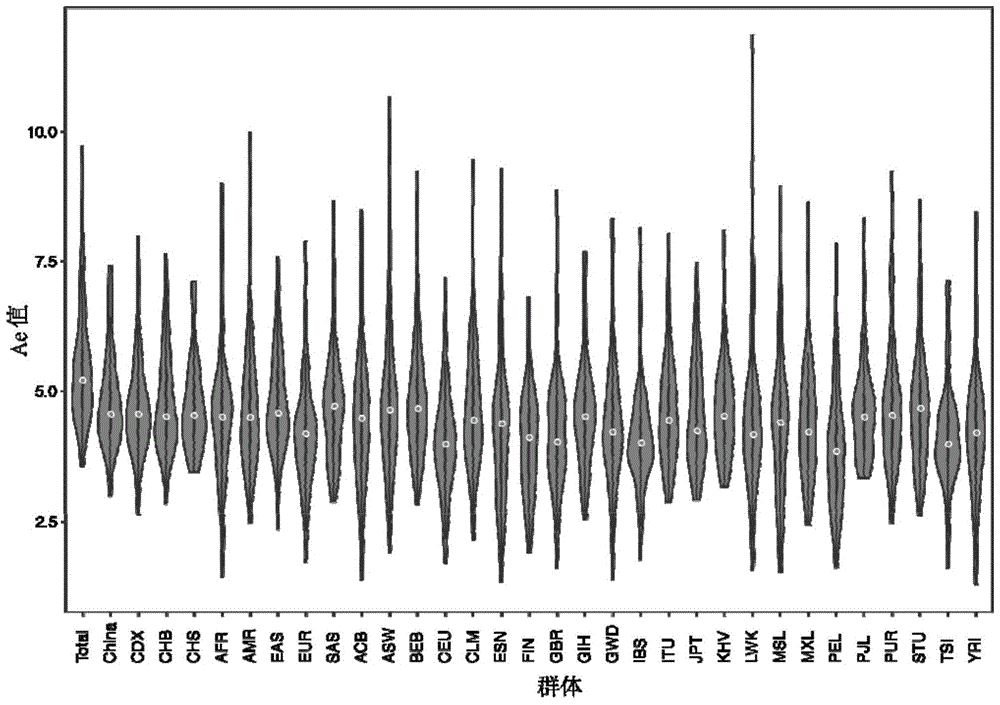
图1本发明59个微单倍型在31个千人基因组群体中的ae值分布图。
图2本发明59个微单倍型通过二代测序平台的测序深度示意图。
图3混合样本微单倍型mh02shy-001测序结果示意图。
图4混合样本微单倍型mh01shy-001测序结果示意图。
图5混合样本微单倍型mh02shy-003测序结果示意图。
图6混合样本微单倍型mh07shy-002测序结果示意图。
图7混合样本微单倍型mh02shy-002测序结果示意图。
图8混合样本微单倍型mh04shy-002测序结果示意图。
具体实施方式
为了更加简洁明了的展示本发明的技术方案、目的和优点,下面结合具体实施例及其附图对本发明做进一步的详细描述。
应用实施例
(1)通过分析千人基因组数据,获得59个微单倍型标记在千人基因组数据各个群体中的ae值分布,该分布可体现本发明中59个微单倍型标记在不同人群中的应用价值,如图1所示。
(2)59个微单倍型标记进行单一位点扩增,确保引物扩增效果,为构建复合扩增体系做准备。结果显示,59个标记均扩增效果好,非特异性扩增干扰少。
(3)通过本发明所构建的复合扩增体系(第一轮pcr体系和第二轮pcr体系)对50个无关个体进行二代测序,获得不同位点测序深度信息。结果如图2所示,结果中个别位点丰度略低,是由于该复合扩增体系的整体测序丰度较高,部分位点的丰度在相比之下略低,但其丰度完全可以满足二代测序序列认可丰度。
(4)通过本发明所构建的复合扩增体系,对由两个随机无关个体(u1和u2)组成的模拟混合样本进行二代测序。混合比例为50:50,75:25,80:20,87.5:12.5,90:10,93.75:6.25,95:5,99:1,99.5:0.5等,共9个混合样本,每个混合样dna用量为50ng。59个微单倍型位点在该混合样本检测后获得的分型如表3。从表3中可看出,除了mh02shy-002在99.5:0.5混合比和mh19shy-002在99.5:0.5混合比的混合样本中分别有1个和2个等位基因丢失外,其余所有位点的所有混合比都获得了理论分型,结果见表3和表4,可见该微单倍型复合检测体系混合样本检测效果明显。
表3:混合样本分型结果
表4:微单倍型分型与简写对照表
上述混合样本在不同微单倍型标记的分型组合可以分为以下几种类型:
(1)无相同等位基因的两个杂合子,混合后可检出4个等位基因(例如图3)。该类型的等位基因组合在混合样本检测中效能最高,且不同组分覆盖度差异明显,利于数据分析。本复合扩增体系59个标记中有20个标记表现为该类型,可见本发明用于混合样本分析的系统效能非常高。具体标记如下:mh02shy-001,mh06shy-005,mh09shy-004,mh10shy-003,mh13shy-003,mh16shy-002,mh19shy-002,mh13shy-001,mh13shy-002,mh16shy-003,mh18shy-002,mh02shy-004,mh04shy-001,mh05shy-003,mh08shy-007,mh09shy-002,mh09shy-001,mh15shy-004,mh19shy-001,mh08shy-005。
(2)具有一个相同等位基因的两个杂合子的组合。由于不同主次成分具有一个相同等位基因,即等位基因共享,该等位基因的测序深度高于其他等位基因,也可为数据分析提供线索,但两个等位基因测序深度的差异随着混合比例的变化而变化(如图4)。该类型共有24个标记,具体标记如下:mh03shy-005,mh05shy-002,mh06shy-001,mh07shy-001,mh08shy-001,mh14shy-003,mh16shy-001,mh10shy-001,mh14shy-002,mh14shy-001,mh15shy-001,mh15shy-002,mh03shy-004,mh04shy-004,mh04shy-003,mh05shy-001,mh08shy-004,mh08shy-002,mh08shy-003,mh10shy-002,mh12shy-001,mh03shy-003,mh01shy-001,mh08shy-006。
(3)一个杂合子与一个具有不同等位基因的纯合子的组合(图5、6)。属于该类型的有如下8个标记:mh02shy-003,mh06shy-003,mh06shy-004,mh11shy-001,mh15shy-003,mh03shy-002,mh07shy-002,mh03shy-001。
(4)一个杂合子和具有一个相同等位基因的纯合子的组合(图7、8)。该类型的具体标记如下:mh18shy-001,mh02shy-002,mh04shy-002,mh09shy-003,mh20shy-001,mh20shy-002。
(5)主次组分的基因型相同。这类位点无法区分混合样本,如mh06shy-002标记。
以上所述实施例仅表达了本发明的几种实施方式,其描述较为具体和详细,但并不能因此而理解为对发明专利范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。因此,本发明专利的保护范围应以所附权利要求为准。
sequencelisting
<110>中山大学
<120>一种用于法医学个体识别的59个微单倍型遗传标记分型体系及其应用
<130>9.18
<160>118
<170>patentinversion3.3
<210>1
<211>22
<212>dna
<213>人工合成
<400>1
tggctcctccttcctgatatct22
<210>2
<211>20
<212>dna
<213>人工合成
<400>2
ctgagtgaagcagaggagcc20
<210>3
<211>22
<212>dna
<213>人工合成
<400>3
agggtttttggactacttggga22
<210>4
<211>20
<212>dna
<213>人工合成
<400>4
agagggcagaacaagaccct20
<210>5
<211>20
<212>dna
<213>人工合成
<400>5
tcggttgctttgcagtaaac20
<210>6
<211>22
<212>dna
<213>人工合成
<400>6
gaaaagcagcagtgattcatgt22
<210>7
<211>20
<212>dna
<213>人工合成
<400>7
cccaatgagcaggacgttat20
<210>8
<211>29
<212>dna
<213>人工合成
<400>8
tctttctctagtagatttcaatgagtaga29
<210>9
<211>20
<212>dna
<213>人工合成
<400>9
cagcttgcagagtgacagca20
<210>10
<211>22
<212>dna
<213>人工合成
<400>10
tgcttcagggtgagtttgaaga22
<210>11
<211>20
<212>dna
<213>人工合成
<400>11
ttggaggtgcaagcctgatt20
<210>12
<211>21
<212>dna
<213>人工合成
<400>12
cacgccattctctatacagca21
<210>13
<211>28
<212>dna
<213>人工合成
<400>13
atagaatacctatagcaaacacatttct28
<210>14
<211>27
<212>dna
<213>人工合成
<400>14
tgcttatccttacctttttgaaacact27
<210>15
<211>20
<212>dna
<213>人工合成
<400>15
aagcacttaacacacccagc20
<210>16
<211>18
<212>dna
<213>人工合成
<400>16
ccctccataagcggcagc18
<210>17
<211>29
<212>dna
<213>人工合成
<400>17
tgtgtagaaataaaactactcacaattct29
<210>18
<211>35
<212>dna
<213>人工合成
<400>18
acagatatattatttaacatgtaaaattcttcctt35
<210>19
<211>21
<212>dna
<213>人工合成
<400>19
ccagagttaggctcagctcac21
<210>20
<211>21
<212>dna
<213>人工合成
<400>20
tgcctcattccagagactcag21
<210>21
<211>20
<212>dna
<213>人工合成
<400>21
atggcattggtggaatgggt20
<210>22
<211>21
<212>dna
<213>人工合成
<400>22
tgactgcagcagttgagaact21
<210>23
<211>22
<212>dna
<213>人工合成
<400>23
aggacaagaaagagctgctcat22
<210>24
<211>20
<212>dna
<213>人工合成
<400>24
gtaacttcggcagcagcact20
<210>25
<211>24
<212>dna
<213>人工合成
<400>25
cactcaatccaccaaaaataggca24
<210>26
<211>22
<212>dna
<213>人工合成
<400>26
agataaaagggggagaaatggc22
<210>27
<211>25
<212>dna
<213>人工合成
<400>27
acaaatgagaaaatggaggcttaga25
<210>28
<211>23
<212>dna
<213>人工合成
<400>28
tgtctacatcttgacaattccgt23
<210>29
<211>22
<212>dna
<213>人工合成
<400>29
tgtctcttgtaaggatgactgc22
<210>30
<211>27
<212>dna
<213>人工合成
<400>30
acatgcatataaatccttagagagagc27
<210>31
<211>22
<212>dna
<213>人工合成
<400>31
tgccctcctctttacacttgag22
<210>32
<211>27
<212>dna
<213>人工合成
<400>32
tgctcataaggaaatacaacaaagaga27
<210>33
<211>24
<212>dna
<213>人工合成
<400>33
tgaaagggctcttctttatcatgg24
<210>34
<211>23
<212>dna
<213>人工合成
<400>34
ttctgaccattgatttgcttgaa23
<210>35
<211>20
<212>dna
<213>人工合成
<400>35
tcccaccccatgaactcaga20
<210>36
<211>20
<212>dna
<213>人工合成
<400>36
tgctgctaggctaggtcctt20
<210>37
<211>25
<212>dna
<213>人工合成
<400>37
agaggatttggactcaatgaactct25
<210>38
<211>20
<212>dna
<213>人工合成
<400>38
ctgccagattccccaggaag20
<210>39
<211>20
<212>dna
<213>人工合成
<400>39
agaaacagcatgaggccagc20
<210>40
<211>22
<212>dna
<213>人工合成
<400>40
gcagcatttattgacaggctga22
<210>41
<211>20
<212>dna
<213>人工合成
<400>41
ctcatcccaccaggtcacag20
<210>42
<211>20
<212>dna
<213>人工合成
<400>42
gcttgagcccttactgagca20
<210>43
<211>20
<212>dna
<213>人工合成
<400>43
tatcagggacccaggaaggt20
<210>44
<211>20
<212>dna
<213>人工合成
<400>44
attggtggctgagttccagg20
<210>45
<211>24
<212>dna
<213>人工合成
<400>45
agatctcatgggtttattaggggt24
<210>46
<211>20
<212>dna
<213>人工合成
<400>46
cagctgggaatccacagagg20
<210>47
<211>35
<212>dna
<213>人工合成
<400>47
ctttgtttagcatataatcaagaaataactataaa35
<210>48
<211>27
<212>dna
<213>人工合成
<400>48
tggtggtgcttagaattttatttttga27
<210>49
<211>22
<212>dna
<213>人工合成
<400>49
tgaatgcgattcaggaagaagt22
<210>50
<211>29
<212>dna
<213>人工合成
<400>50
tgagattatattgtcaagttttcaaaagt29
<210>51
<211>22
<212>dna
<213>人工合成
<400>51
acaaactttcacctgcctttga22
<210>52
<211>24
<212>dna
<213>人工合成
<400>52
aaatgtgatcaagttgcatacact24
<210>53
<211>21
<212>dna
<213>人工合成
<400>53
acaccaaggattgtcagcaac21
<210>54
<211>20
<212>dna
<213>人工合成
<400>54
gccacttcctccccagactc20
<210>55
<211>26
<212>dna
<213>人工合成
<400>55
ccaagcttccatttatcaaagtacca26
<210>56
<211>18
<212>dna
<213>人工合成
<400>56
cctgcagttctggccact18
<210>57
<211>24
<212>dna
<213>人工合成
<400>57
cacatactacacaatttgcctact24
<210>58
<211>22
<212>dna
<213>人工合成
<400>58
ccaggcacacatattgtatgac22
<210>59
<211>27
<212>dna
<213>人工合成
<400>59
tccttgttgctatgtttactttaggat27
<210>60
<211>33
<212>dna
<213>人工合成
<400>60
cgaataagtctcacaagatctgatggttttatc33
<210>61
<211>26
<212>dna
<213>人工合成
<400>61
cctcctataactacaactctttcagt26
<210>62
<211>21
<212>dna
<213>人工合成
<400>62
ccaaaccaggaagtgcaagac21
<210>63
<211>20
<212>dna
<213>人工合成
<400>63
tgtcagcatggatctgtggc20
<210>64
<211>22
<212>dna
<213>人工合成
<400>64
aggaggattaagtcacaccact22
<210>65
<211>20
<212>dna
<213>人工合成
<400>65
aaggtcagagagcacaccag20
<210>66
<211>25
<212>dna
<213>人工合成
<400>66
gctggtgtttttgagtaagaaatgg25
<210>67
<211>20
<212>dna
<213>人工合成
<400>67
ctgccccctaccctccttat20
<210>68
<211>24
<212>dna
<213>人工合成
<400>68
gcatgctactgtggttaaattgga24
<210>69
<211>20
<212>dna
<213>人工合成
<400>69
ctgaggtgtggcagagatgg20
<210>70
<211>20
<212>dna
<213>人工合成
<400>70
ggtgccaggagagctttgaa20
<210>71
<211>20
<212>dna
<213>人工合成
<400>71
atggtgtccccgtgtactct20
<210>72
<211>20
<212>dna
<213>人工合成
<400>72
ggctcgtaaaagtcactggc20
<210>73
<211>20
<212>dna
<213>人工合成
<400>73
catgcgggagaatgtaggga20
<210>74
<211>20
<212>dna
<213>人工合成
<400>74
catgaggggcaacaccaagt20
<210>75
<211>20
<212>dna
<213>人工合成
<400>75
ggtctgcaaaggaaagcacc20
<210>76
<211>20
<212>dna
<213>人工合成
<400>76
caggagagccaagtccccta20
<210>77
<211>20
<212>dna
<213>人工合成
<400>77
tctgatctggaggcccaaga20
<210>78
<211>20
<212>dna
<213>人工合成
<400>78
ctacctctctgtggctctgc20
<210>79
<211>21
<212>dna
<213>人工合成
<400>79
tgaagtcacactggctgaagt21
<210>80
<211>20
<212>dna
<213>人工合成
<400>80
cccaggagcaggtggtaaag20
<210>81
<211>20
<212>dna
<213>人工合成
<400>81
tagccttcaaacctcctggc20
<210>82
<211>20
<212>dna
<213>人工合成
<400>82
gtgggtgctcagtagatgga20
<210>83
<211>22
<212>dna
<213>人工合成
<400>83
gcagctgagaagatttgctaca22
<210>84
<211>23
<212>dna
<213>人工合成
<400>84
gcacatgactcttttaaagggca23
<210>85
<211>18
<212>dna
<213>人工合成
<400>85
gcccagcctccctttcag18
<210>86
<211>26
<212>dna
<213>人工合成
<400>86
agcactaactagagtaatgcagaact26
<210>87
<211>20
<212>dna
<213>人工合成
<400>87
tcatccccacttcacagaga20
<210>88
<211>27
<212>dna
<213>人工合成
<400>88
attcctgatttattcctgtagtcattc27
<210>89
<211>23
<212>dna
<213>人工合成
<400>89
gctcctgctctaatcctttctcc23
<210>90
<211>28
<212>dna
<213>人工合成
<400>90
actgctttaattaggtatttcacttaca28
<210>91
<211>20
<212>dna
<213>人工合成
<400>91
aaagcaagcttttcaggggc20
<210>92
<211>20
<212>dna
<213>人工合成
<400>92
agaaagctggagaagggacc20
<210>93
<211>20
<212>dna
<213>人工合成
<400>93
gggatggagtggaaaaggga20
<210>94
<211>21
<212>dna
<213>人工合成
<400>94
tgcccagaggtatgatcaagc21
<210>95
<211>20
<212>dna
<213>人工合成
<400>95
tctcggcttctctgtgagca20
<210>96
<211>20
<212>dna
<213>人工合成
<400>96
ctggctgttcacagggttgt20
<210>97
<211>21
<212>dna
<213>人工合成
<400>97
catgagacaagcagacggatt21
<210>98
<211>20
<212>dna
<213>人工合成
<400>98
acccagccttgtatatggca20
<210>99
<211>22
<212>dna
<213>人工合成
<400>99
tgtgtatttccagcaaccatca22
<210>100
<211>20
<212>dna
<213>人工合成
<400>100
ctgaagggacagtgaggtgg20
<210>101
<211>20
<212>dna
<213>人工合成
<400>101
gtcttctctgttgccaaggc20
<210>102
<211>20
<212>dna
<213>人工合成
<400>102
aacttggcaggacatggtgg20
<210>103
<211>20
<212>dna
<213>人工合成
<400>103
gatcctgtaggtggtggcaa20
<210>104
<211>24
<212>dna
<213>人工合成
<400>104
tcataaatgtcacctcctcagaga24
<210>105
<211>20
<212>dna
<213>人工合成
<400>105
actatacttgcagcccaggt20
<210>106
<211>21
<212>dna
<213>人工合成
<400>106
tccccgttcacagaagaggta21
<210>107
<211>25
<212>dna
<213>人工合成
<400>107
acagttgtaacatgcatacacaagt25
<210>108
<211>22
<212>dna
<213>人工合成
<400>108
ccctgtagcctgctgaatatgt22
<210>109
<211>23
<212>dna
<213>人工合成
<400>109
ggactagaaactgacaaaaggca23
<210>110
<211>20
<212>dna
<213>人工合成
<400>110
tgcatctctaactcctgcgt20
<210>111
<211>19
<212>dna
<213>人工合成
<400>111
gtgcagggtgaagctggat19
<210>112
<211>21
<212>dna
<213>人工合成
<400>112
agcaggacctagtcttctgga21
<210>113
<211>21
<212>dna
<213>人工合成
<400>113
ctgcctcagcctctgaaattg21
<210>114
<211>22
<212>dna
<213>人工合成
<400>114
actcagaatagtggactggaca22
<210>115
<211>22
<212>dna
<213>人工合成
<400>115
aaagtgtgtgcaattggaatct22
<210>116
<211>20
<212>dna
<213>人工合成
<400>116
tgttttcccctcctggctac20
<210>117
<211>22
<212>dna
<213>人工合成
<400>117
gtgaaaccctgtctctggtaac22
<210>118
<211>20
<212>dna
<213>人工合成
<400>118
tcttgaaggtgggctggact20
- 还没有人留言评论。精彩留言会获得点赞!