膀胱癌相关的DNA甲基化生物标志物组合和检测试剂盒的制作方法
本发明属于生物
技术领域:
,具体涉及一种膀胱癌相关的dna甲基化生物标志物组合和检测试剂盒。
背景技术:
:膀胱癌是泌尿系统最常见的恶性肿瘤之一,在中国膀胱癌的2014年发病人数为7.8万人/年,并且发病率有逐年上升的趋势。膀胱癌具有发病率高、易复发的特点。血尿为膀胱癌的常见临床症状,其中约17%的血尿患者检出膀胱癌。目前,膀胱癌的主要诊断方法包括影膀胱镜检查、尿脱落细胞学检查、尿液fish检测及肿瘤标记物检查。其中,膀胱镜检查结合活检组织病理为膀胱镜诊断的金标准,但是该检查方法具有侵入性,容易产生并发症,患者依随性低。而影像学检查对于微小病灶的诊断能力有限,尿脱落细胞学检查的灵敏度低,尿液fish检测操作复杂、判读结果具有主观性。现有肿瘤标记物检查主要基于尿液中的特定蛋白质的存在进行检测,但由于尿液中测定蛋白的含量低,敏感性及特异性仍然具有局限性。通过寻找膀胱癌特异性dna甲基化生物标记物的组合,进行基于多个dna甲基化位点的检测克服了单个dna甲基化信号低的问题,提高了检测的灵敏度及特异性。同时,基于dna甲基化的检测简单易行、判读客观,避免了人为判读结果的主观性,提高了准确率。同时该检测具有非侵入性,能避免膀胱镜检查引发的并发症,提高患者依随性。技术实现要素:本发明的目的之一在于提供一种用于膀胱癌诊断的dna甲基化标志物组合。实现上述目的的技术方案如下。一种用于膀胱癌检测的dna甲基化标志物组合,其选自seqidno.1至seqidno.6序列中由【cg】指示的甲基化位点的任意两种或两种以上的组合,或选自seqidno.1至seqidno.6的完全互补的序列中所对应的甲基化位点的任意两种或两种以上的组合。在其中一些实施例中,所述用于膀胱癌检测的dna甲基化标志物组合选自seqidno.2和seqidno.4的序列中由【cg】指示的甲基化位点组合,或其完全互补序列的甲基化标志物组合;进一步选自seqidno.2、seqidno.3和seqidno.4的序列中由【cg】指示的甲基化位点组合,或其完全互补序列的甲基化标志物组合;或选自seqidno.2、seqidno.3、seqidno.4和seqidno.5的序列中由【cg】指示的甲基化位点组合,或其完全互补序列的甲基化标志物组合。在其中一些实施例中,所述用于膀胱癌检测的dna甲基化标志物组合选自seqidno.1、seqidno.2、seqidno.3、seqidno.4、seqidno.5和seqidno.6的序列中由【cg】指示的甲基化位点组合;或其完全互补序列的甲基化标志物组合。本发明的另一目的是提供上述dna甲基化标志物在制备检测膀胱癌的试剂盒中应用。本发明的另一个目的是提供一种检测膀胱癌的试剂盒。一种检测膀胱癌试剂盒,包括检测上述dna甲基化标志物组合的甲基化程度的试剂。在其中一些实施例中,采用飞行时间质谱平台检测,所述检测试剂盒包括针对每个dna甲基化标志物的扩增引物和延伸引物,所述扩增引物和延伸引物为:针对seqidno.1的seqidno.7和seqidno.13,以及seqidno.19;针对seqidno.2的seqidno.8和seqidno.14,以及seqidno.20;针对seqidno.3的seqidno.9和seqidno.15,以及seqidno.21;针对seqidno.4的seqidno.10和seqidno.16,以及seqidno.22;针对seqidno.5的seqidno.11和seqidno.17,以及seqidno.23;针对seqidno.6的seqidno.12和seqidno.18,以及seqidno.24。在其中一些实施例中,采用飞行时间质谱平台检测,所述检测试剂盒包括针对每个dna甲基化标志物的扩增引物和延伸引物,所述扩增引物和延伸引物为:针对seqidno.1的seqidno.25和seqidno.31,以及seqidno.37;针对seqidno.2的seqidno.26和seqidno.32,以及seqidno.38;针对seqidno.3的seqidno.27和seqidno.33,以及seqidno.39;针对seqidno.4的seqidno.28和seqidno.34,以及seqidno.40;针对seqidno.5的seqidno.29和seqidno.35,以及seqidno.41;针对seqidno.6的seqidno.30和seqidno.36,以及seqidno.42。在其中一些实施例中,采用飞行时间质谱平台检测,所述检测试剂盒包括针对针对每个dna甲基化标志物的扩增引物和延伸引物,所述扩增引物和延伸引物为:针对seqidno.1的seqidno.43和seqidno.49,以及seqidno.55;针对seqidno.2的seqidno.44和seqidno.50,以及seqidno.56;针对seqidno.3的seqidno.45和seqidno.51,以及seqidno.57;针对seqidno.4的seqidno.46和seqidno.52,以及seqidno.58;针对seqidno.5的seqidno.47和seqidno.53,以及seqidno.59;针对seqidno.6的seqidno.48和seqidno.54,以及seqidno.60。在其中一些实施例中,还包括虾碱性磷酸酶、pcr试剂、sap试剂、延伸反应试剂。本发明筛选到适合膀胱癌检测的甲基化位点的组合,选中seqidno.1至seqidno.6序列中由【cg】指示的甲基化位点的任意两种或两种以上的组合,比单独位点的检测具有更高的灵敏度。本发明判断膀胱癌发生的方法完全基于膀胱癌特异的甲基化位点的甲基化程度,从甲基化程度到诊断的判读完全采用统计学的数学公式,避免了任何在传统尿液fish(荧光原位杂交技术(fluorescenceinsituhybridization))检测中涉及人为判定结果的主观性,使得判读上更准确、稳定、可靠。同时,上述甲基化位点组合在判断膀胱癌的发生具有更高的灵敏度。本发明提供了所述6个dna甲基化标志物(marker,或标记物)中任意2种的检测目标甲基化位点甲基化程度的其中一种检测试剂盒。所述试剂盒中引物对的设计及其组合设计对于同时并行检测多个基因的甲基化位点起关键作用,本试剂盒的引物对组合在引物序列设计上克服了亚硫酸盐序列缺乏碱基多样性的缺点,并考虑了多至6个甲基化生物标记物引物对组合之间的相互作用。附图说明图1膀胱癌组织和正常组织的6个dna甲基化生物标记物的甲基化差异热图。图2膀胱癌人群和正常人群的尿液dna中6个甲基化生物标记物的甲基化差异热图。图3基于dna甲基化的膀胱癌风险得分在膀胱癌人群和正常人群的中的显著差异。图4三个组合的引物对对于甲基化位点序列seqidno.2所选甲基化位点的检测对比。图56个甲基化位点平均甲基化率在两组人群中的表现。具体实施方式为了便于理解本发明,下面将对本发明进行更全面的描述。本发明可以以许多不同的形式来实现,并不限于本文所描述的实施例。相反地,提供这些实施例的目的是使对本发明公开内容的理解更加透彻全面。下列实施例中未注明具体条件的实验方法,通常按照常规条件,例如sambrook等人,分子克隆:实验室手册(newyork:coldspringharborlaboratorypress,1989)中所述的条件,或按照制造厂商所建议的条件。实施例中所用到的各种常用化学试剂,均为市售产品。除非另有定义,本发明所使用的所有的技术和科学术语与属于本发明的
技术领域:
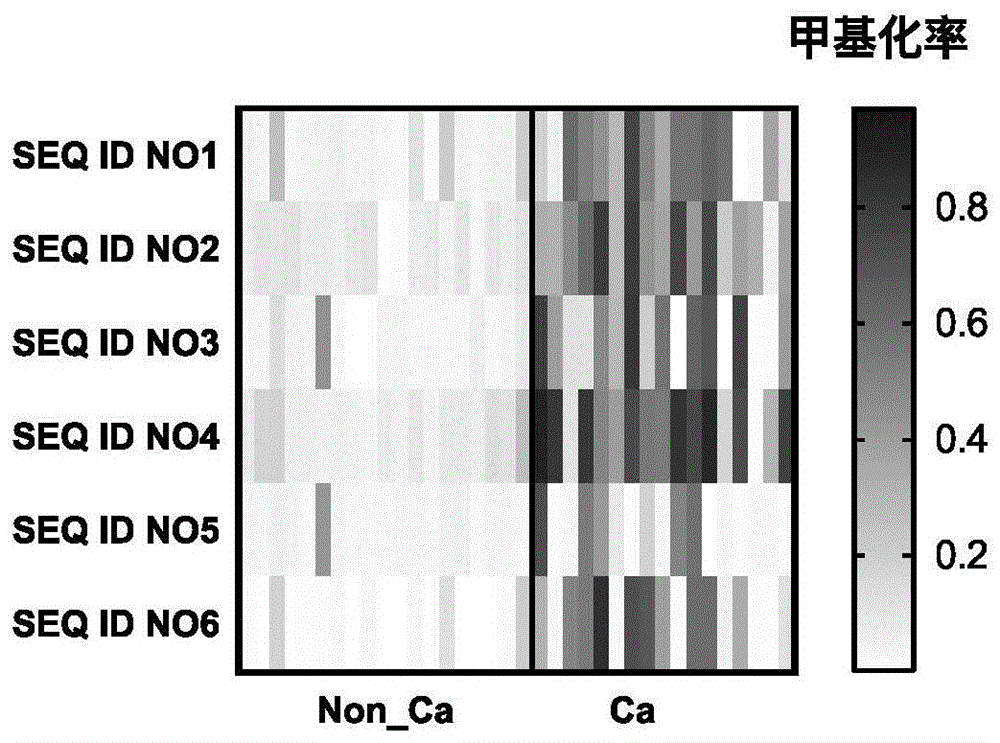
的技术人员通常理解的含义相同。本发明的说明书中所使用的术语只是为了描述具体的实施例的目的,不用于限制本发明。本发明所使用的术语“和/或”包括一个或多个相关的所列项目的任意的和所有的组合。本发明提供了一种基于挑选出来的甲基化位点甲基化程度判断膀胱癌发生的标志物。首先通过文献查阅及膀胱癌开放数据库对450000甲基化位点进行统计学及数据建模分析发现上述甲基化位点的甲基化程度与膀胱癌的发生有较强的相关性。根据长期研究和试验,我们筛选了最合适组合的6个dna甲基化生物标记物,在此基础上,提供了这6个生物标记物组合用于判断个体膀胱癌发生,采用本发明实施例3中的检测方法对上述甲基化位点在确诊的膀胱癌人群的癌组织(19例)及癌旁组织(正常组织,20例)样本进行检测,结果参见图1。通过图1可以看出,这些位点在膀胱癌的组织dna中有较高的甲基化程度,而在正常组织中甲基化程度较低,甲基化程度在癌与非癌之间存在显著差异,说明这些甲基化位点的甲基化程度可以较灵敏及特异的反映膀胱癌的发生。另一个方面,提供了所述甲基化位点的组合在利用尿液样本进行膀胱癌的发生的诊断的应用。采用实施例3中的检测方法对上述甲基化位点在膀胱癌人群(44例)及正常人群的尿液样本(53例)dna进行检测,上述甲基化位点的甲基化程度在膀胱癌及正常人群中的区分度与组织样本的结果类似,所述甲基化位点在膀胱癌人群的尿液dna中有较高的甲基化程度,而正常人群的尿液dna中甲基化程度较低,甲基化程度在两个人群中存在显著差异(图2),说明所选甲基化位点组合在尿液dna中与膀胱癌相关的信号较高,对膀胱癌的检出有优越的灵敏度。同时,采用尿液作为测试样本是非侵入的方式,可以较大程度减轻患者的负担,增加患者检测的依随性。本发明根据膀胱癌人群及正常人群的2-6个甲基化位点的甲基化程度进行逻辑回归方程拟合,通过拟合的逻辑回归方程算出膀胱癌的风险得分,该得分在膀胱癌人群和正常人群中存在显著的差异,可明显将膀胱癌人群从正常人群中检出。实施例1用于膀胱癌诊断的甲基化位点,包括表1所列核酸序列中由【cg】指示的甲基化位点的以及与表1中的由【cg】指示的核酸在序列上完全互补的核酸甲基化位点。表1dna甲基化标记物的组成实施例2一种用于膀胱癌诊断的6个甲基化生物标记物的甲基化测试试剂盒,包括3组pcr扩增引物及延伸引物组合中的任意一组,其中,每组pcr扩增引物及延伸引物组合包括6对pcr扩增引物及6条pcr延伸引物,所述3组pcr扩增引物及延伸引物如表2所示。表23组pcr扩增引物及延伸引物组合以上y为t或c;r为g或a。所述试剂盒需包括pcr扩增引物及延伸引物组合3组组合中的其中一组,3组组合对于6个甲基化位点的检测性能类似,以其中一个甲基化位点(甲基化位点seqidno.2)的甲基化程度检测为例,分别使用3个组合的引物对对系列甲基化标准品在该位点的甲基化程度按实施例3中的检测方法进行检测,检测结果如图4,组合1、2、3的引物对对于该位点的甲基化检测的线性关系分别为0.987、0.998、0.997,线性关系没有显著差异,拟合线性方程斜率分别为0.96、0.89、0.93,可判断扩增效率类似,没有显著差别。引物购于invitrogen公司,其他pcr试剂、sap试剂、延伸反应试剂均购于agenabioscience公司。图4是三个组合的引物对对于所选甲基化位点的检测对比:组合1、2、3的引物对对于该位点的甲基化检测的拟合线性类似,线性方程的斜率相近,因此扩增效率性能没有显著差异。实施例36个marker的甲基化检测对97例尿沉渣样本(其中临床确诊为膀胱癌的样本44例,正常人群样本53例)的6个甲基化位点进行检测。具体流程如下:1、dna提取方法组织提取试剂盒购自qiagen公司,按照试剂盒说明书进行。2、dna亚硫酸氢盐转化dna亚硫酸氢盐转化试剂盒购于zymo公司,按照试剂盒说明书进行。3、pcr扩增根据甲基化位点设计6对pcr引物,在1个反应孔(引物系列见表2)中进行多重pcr,扩增出含目标位点的目标序列,产物大小分别为100-200bp左右。1)配置单个引物浓度为0.5μm(每个引物)pcr引物混合物,里面包含多重反应里每个甲基化位点的f(正向)和r(反向)引物,共1个反应孔。2)pcr混合液配置:配制0.5μm(每个引物)pcr引物混合物,里面包含多重反应里每个甲基化位点的f(正向)和r(反向)引物。根据表3配制pcr混合液,dna不要加在其中。表3pcr混合液配置方案3)加入dna样本:加5μlpcr混合液到pcr反应孔,向其中加入dna,dna上样量100ng,pcr反应总体积10μl。涡旋震荡和离心。4)pcr反应程序:95℃2分钟;95℃30秒,56℃30秒,72℃60秒,45个循环;72℃5分钟;4℃保存备用。4、pcr产物消化处理1)根据表4配制sap混合液表4sap混合液配置表试剂终浓度体积(μl)nanopurewater(超纯水)/1.53sapbuffer(sap缓冲液)0.24x0.17sap酶(1.7u/ul)0.5u0.30总体积[ul]/22)向每一个pcr反应孔中加入2μlsap混液。完成后,用膜把板封好,涡旋震荡和离心。3)pcr产物消化处理:37℃40分钟;85℃5分钟。5、目标位点延伸反应1)根据表5配制iplex延伸混液:表5iplex延伸预混液配置表2)向sap消化后的产物中加入2μliplex延伸混液并混好,涡旋震荡和离心。3)pcr反应程序:94℃30秒;{94℃5秒,(52℃5秒,80℃5秒,5个循环),45个循环};72℃3分钟;4℃保存备用。6、上机前处理1)戴上手套和护眼镜。2)把洁净树脂(resin)铺平在384/6mgdimpleplate上,风干最少10分钟。3)在样本板的每一个有样本的孔里加入16ul水。如果有器材和方法,这一步可以使用液体处理器。完成后,用膜把板封好,涡旋震荡和离心。4)加入6mg洁净树脂(resin)(11235):轻轻将样本板凌空反转,放在已放树脂的dimpleplated(波浪板)上,然后将dimpleplate连样本板一起反转(过程中两快板不可水平移动),让树脂掉到孔里。用膜把板封好,放在旋转器上颠倒摇匀15分钟。以3200g(标准板离心机的4000rpm)将板离心5分钟。7、上机检测1)用点样仪进行volumecheck(体积测试)以找出合适的dispensingspeed(点样速度)。不过,点3pt-calibrant(3点标准品)的dispensingspeed最少要达90mm/sec。2)以板上的真正样本进行volumecheck,然后用massarraytmnanodispenserrs1000/fusio(rs1000/fusio点样仪)或自备点样仪将样本点到spectrochip(芯片)上。3)maldi-tofms基质辅助激光解吸电离-飞行时间质谱。4)将芯片放到massarraytyperworkstationma4或compact里打质谱,flexcontrol和spectroacquire的参数要设定为iplex(iplex.par)。备注:使用typer4plateeditor编板时样本档要以纵向导入。而且在编板前将元数据应用在样本上。5)完成实验后,导出.xml档。8、数据处理与分析通过各甲基化位点的的甲基化峰及非甲基化峰的峰噪值(snr)计算该位点的甲基化率:甲基化率=snr-m/(snr-m+snr-u);其中,snr-m为甲基化峰的峰噪值,snr-u为非甲基化峰的峰噪值。列举97例尿液样本中20例样本的6个甲基化位点的甲基化率如表6,其中样本编号1-10为癌症人群尿液样本,样本编号11-20为正常人群尿液样本(采用引物和探针的组合3进行检测,以下实施例相同)。结果显示本发明所述检测方法和试剂盒可以实现对于6个甲基化位点甲基化程度的并行检测。表697例尿液样本中20例样本的6个甲基化位点的甲基化率实施例4对97例尿沉渣样本(其中临床确诊为膀胱癌的样本44例,正常人群样本53例)的6个甲基化位点按实施例3的检测方法进行检测,根据6个甲基化位点的在两组人群中的各自平均甲基化率对比样本病理作出roc曲线,根据曲线计算auc,划分每个位点的判定阈值及计算该阈值下的灵敏度及特异性。同时,根据6个甲基化位点的甲基化率进行逻辑回归拟合,根据拟合方程计算每个样本的膀胱癌风险得分及判断膀胱癌发生的阈值,根据样本的得分与阈值相比较,将样本划分为膀胱癌阳性组及阴性组。将基于甲基化程度模型的分组与样本临床病理进行比较,得到用于判别甲基化程度模型诊断性能的roc曲线,并根据roc曲线得到auc、灵敏度及特异性。图5显示对于膀胱癌人群及正常人群的尿液样本,6个甲基化位点均有一定程度的区分。而根据图5单个甲基化位点划分的判定阀值比较单个位点模型与2-6个甲基化位点组合模型对于诊断膀胱癌发生的性能比较(表7)显示,2-6个甲基化位点的组合在判别膀胱癌的发生上与单个甲基化位点的判别相比较,灵敏度及特异性均有较大的提高,性能优于单个甲基化位点的判别。表7单个甲基化位点的模型与6个甲基化位点的模型对于诊断膀胱癌发生的比较(se/sp/auc)以上所述实施例的各技术特征可以进行任意的组合,为使描述简洁,未对上述实施例中的各个技术特征所有可能的组合都进行描述,然而,只要这些技术特征的组合不存在矛盾,都应当认为是本说明书记载的范围。以上所述实施例仅表达了本发明的几种实施方式,其描述较为具体和详细,但并不能因此而理解为对发明专利范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。因此,本发明专利的保护范围应以所附权利要求为准。序列表<110>广州市基准医疗有限责任公司中山大学孙逸仙纪念医院<120>膀胱癌相关的dna甲基化生物标志物组合和检测试剂盒<160>60<170>siposequencelisting1.0<210>1<211>50<212>dna<213>人工序列(artificialsequence)<400>1tggccggcaggcgtggagcaaaatcgagggcagctgaaggggcagcagtc50<210>2<211>50<212>dna<213>人工序列(artificialsequence)<400>2gtagcccgcgtggatgcggatatgcgtgttgagcgtggagctgcggttga50<210>3<211>50<212>dna<213>人工序列(artificialsequence)<400>3tggtaggtggttttccccaccacccggtggtgcaccacgagggctacccg50<210>4<211>50<212>dna<213>人工序列(artificialsequence)<400>4gaatagaatcgagcagttggtttgcgagcagctgggacgccgttttgagc50<210>5<211>50<212>dna<213>人工序列(artificialsequence)<400>5agcagcagctgcaggaagcggactcggcggaaaggagccccggaggggaa50<210>6<211>50<212>dna<213>人工序列(artificialsequence)<400>6gcccaggctgtaggtgcgggtggacgtagtcacgtagctccggctggagc50<210>7<211>30<212>dna<213>人工序列(artificialsequence)<400>7acgttggatggaaacgtcgggaggttttgg30<210>8<211>29<212>dna<213>人工序列(artificialsequence)<400>8acgttggatgggttcggttttgatggagg29<210>9<211>29<212>dna<213>人工序列(artificialsequence)<400>9acgttggatgtgagtttggtaggtggttt29<210>10<211>43<212>dna<213>人工序列(artificialsequence)<400>10acgttggatgggaataatattaaataagtattttaataaatag43<210>11<211>32<212>dna<213>人工序列(artificialsequence)<400>11acgttggatggtgtttggttgaaggtatttag32<210>12<211>29<212>dna<213>人工序列(artificialsequence)<400>12acgttggatggtagaggttgyggttggtg29<210>13<211>30<212>dna<213>人工序列(artificialsequence)<400>13acgttggatgatttctacccattaaaacac30<210>14<211>33<212>dna<213>人工序列(artificialsequence)<400>14acgttggatgctaaaaaatcccccacttccaac33<210>15<211>30<212>dna<213>人工序列(artificialsequence)<400>15acgttggatgtaaccrataaaccaaccata30<210>16<211>28<212>dna<213>人工序列(artificialsequence)<400>16acgttggatgactccacctcccaactca28<210>17<211>35<212>dna<213>人工序列(artificialsequence)<400>17acgttggatgccttttcaaccttctttaacactcc35<210>18<211>28<212>dna<213>人工序列(artificialsequence)<400>18acgttggatgtatcctcrtcctcctacc28<210>19<211>19<212>dna<213>人工序列(artificialsequence)<400>19ggtaggcgtggagtaaaat19<210>20<211>19<212>dna<213>人工序列(artificialsequence)<400>20ttcggggttcggtttcgag19<210>21<211>17<212>dna<213>人工序列(artificialsequence)<400>21cctcrtaatacaccacc17<210>22<211>19<212>dna<213>人工序列(artificialsequence)<400>22aacracrtcccaactactc19<210>23<211>23<212>dna<213>人工序列(artificialsequence)<400>23caacaactacaaaaaacraactc23<210>24<211>19<212>dna<213>人工序列(artificialsequence)<400>24accraaactacrtaactac19<210>25<211>30<212>dna<213>人工序列(artificialsequence)<400>25acgttggatggttttacgtttgtcggttac30<210>26<211>24<212>dna<213>人工序列(artificialsequence)<400>26acgttggatggggtgggttggggg24<210>27<211>38<212>dna<213>人工序列(artificialsequence)<400>27acgttggatggaagtaggggttttttttatggttgtag38<210>28<211>28<212>dna<213>人工序列(artificialsequence)<400>28acgttggatgggagaggggattggggag28<210>29<211>30<212>dna<213>人工序列(artificialsequence)<400>29acgttggatgttttagggttagtggggttg30<210>30<211>33<212>dna<213>人工序列(artificialsequence)<400>30acgttggatgtagttatgtttattaggttygtg33<210>31<211>30<212>dna<213>人工序列(artificialsequence)<400>31acgttggatgacaacgttcccgccgaaacg30<210>32<211>28<212>dna<213>人工序列(artificialsequence)<400>32acgttggatgccctacttcccaactcac28<210>33<211>35<212>dna<213>人工序列(artificialsequence)<400>33acgttggatgaaataaatctaataaataattttcc35<210>34<211>41<212>dna<213>人工序列(artificialsequence)<400>34acgttggatgctaataaataaccacttttttattcctactc41<210>35<211>32<212>dna<213>人工序列(artificialsequence)<400>35acgttggatgctaaaaacactcaattcccctc32<210>36<211>30<212>dna<213>人工序列(artificialsequence)<400>36acgttggatgcaacrcrctacccaaactat30<210>37<211>21<212>dna<213>人工序列(artificialsequence)<400>37gttaggttcgtcggaggagcg21<210>38<211>19<212>dna<213>人工序列(artificialsequence)<400>38taaggggttyggtttygag19<210>39<211>19<212>dna<213>人工序列(artificialsequence)<400>39ataattttccccaccaccc19<210>40<211>21<212>dna<213>人工序列(artificialsequence)<400>40cgaatcraacaattaatttac21<210>41<211>26<212>dna<213>人工序列(artificialsequence)<400>41ttaggagtagttgtaggaagyggatt26<210>42<211>23<212>dna<213>人工序列(artificialsequence)<400>42ccaaactataaatacraataaac23<210>43<211>30<212>dna<213>人工序列(artificialsequence)<400>43acgttggatgcaaactactaccccttcaac30<210>44<211>30<212>dna<213>人工序列(artificialsequence)<400>44acgttggatgaggtgaggtgagaagagaag30<210>45<211>30<212>dna<213>人工序列(artificialsequence)<400>45acgttggatggaaatgagtttggtaggtgg30<210>46<211>29<212>dna<213>人工序列(artificialsequence)<400>46acgttggatggatagtgaatagaatygag29<210>47<211>31<212>dna<213>人工序列(artificialsequence)<400>47acgttggatgggtgttagtagtagttgtagg31<210>48<211>29<212>dna<213>人工序列(artificialsequence)<400>48acgttggatggttgtttaggttgtaggtg29<210>49<211>30<212>dna<213>人工序列(artificialsequence)<400>49acgttggatgggaagtttgaattagtttag30<210>50<211>30<212>dna<213>人工序列(artificialsequence)<400>50acgttggatgcctctccttactatacactc30<210>51<211>29<212>dna<213>人工序列(artificialsequence)<400>51acgttggatgttctcctcataactacaac29<210>52<211>30<212>dna<213>人工序列(artificialsequence)<400>52acgttggatgtccacctcccaactcaaaac30<210>53<211>29<212>dna<213>人工序列(artificialsequence)<400>53acgttggatgaaacactcaattcccctcc29<210>54<211>30<212>dna<213>人工序列(artificialsequence)<400>54acgttggatgcaaccatatccaccaaatcc30<210>55<211>17<212>dna<213>人工序列(artificialsequence)<400>55ccccttcaactaccctc17<210>56<211>26<212>dna<213>人工序列(artificialsequence)<400>56atgtagttygygtggatgyggatatg26<210>57<211>26<212>dna<213>人工序列(artificialsequence)<400>57tttggtaggtggttttttttattatt26<210>58<211>24<212>dna<213>人工序列(artificialsequence)<400>58gaatagaatygagtagttggtttg24<210>59<211>26<212>dna<213>人工序列(artificialsequence)<400>59ttaggagtagttgtaggaagyggatt26<210>60<211>17<212>dna<213>人工序列(artificialsequence)<400>60ttgtaggtgygggtgga17当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1
- 一种新的多肽——dna甲基化酶13和编码这种多肽的多核苷酸的制作方法
- 具有治疗活性的环亚甲基-1,2-二羧酸酰胺,制备它们的方法和含它们的药物组合物的制作方法
- 组蛋白去乙酰化酶抑制剂的制作方法
- 基于dna聚合酶链反应的甲基化dna检测方法
- 检测Cre位点特异重组酶活性的转基因报告猪及培养方法
- 一种多肽——人含胞嘧啶特异性dna甲基化酶特征性序列片段的蛋白-9.35和编码这种 ...的制作方法
- 一种多肽——脱氧核糖核酸甲基化酶-9.35和编码这种多肽的多核苷酸的制作方法
- 利用酶活性的竞争性干扰和恢复检测被分析物的制作方法
- 一种基于化学发光反应的甲基化酶检测探针、检测试剂盒及检测方法
- 编码甲基化儿茶素生物合成酶的基因的制作方法