一种结直肠癌抑癌基因甲基化高通量检测试剂盒及其应用的制作方法
本发明属于生物
技术领域:
,具体是一种结直肠癌抑癌基因甲基化高通量检测试剂盒及其应用。
背景技术:
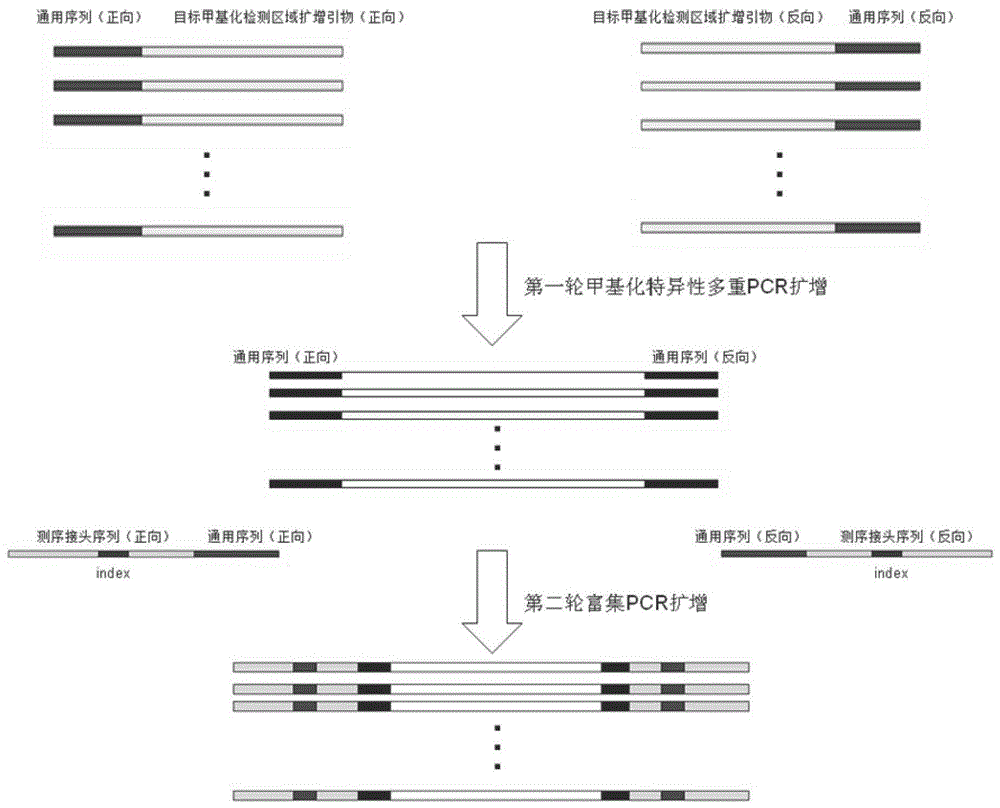
:结直肠癌(crc)发病率位居全球恶性肿瘤第三位,已成为新发肿瘤病例和死亡的第三大原因。定期筛查可以早期发现结直肠癌,从而进行早期干预,以防止疾病进展甚至治愈疾病。然而,60%~70%患者在首次确诊时即为中、晚期crc。美国预防服务工作小组(unitedstatespreventiveservicestaskforce,uspstf)的统计数据显示,如果健康人每年进行常规定期检查,实现crc的早期诊治,可避免约60%crc患者病死,平均五年生存率可以从46%提高到73%。因此,有效的早期筛查方法,可提高crc患者的五年生存率,降低病死率。目前临床主要运用两种方法进行crc筛查:粪便潜血试验(fecaloccultbloodteats,fobt)和结肠镜检查。fobt主要包括gfobt(愈创木脂粪便潜血试验)、ifobt(免疫粪便潜血试验)和fit(人体血红蛋白特异性粪便免疫化学试验)。作为常规使用的筛查方法,gfobt易受食物、药物和其他因素的影响,可导致假阳性结果,且稳定性较差;ifobt的敏感度和特异性明显优于gfobt,但此检测仍然是定性试验,如果样品采集后放置过久(超过5d),其检出率明显下降;fit其敏感度较高,但价格更昂贵。相比之下,结肠镜检查是侵入性的,需要肠道准备,以确保大肠管腔视野良好。另外,肠镜检查有一系列并发症,如肠活检部位出血、肠穿孔和感染;该检查有多种禁忌证,如严重的心脏疾病、心肺功能不全、急性腹泻、严重溃疡性结肠炎、结肠克罗恩病、腹膜炎和妊娠等。因此,无论是fobt还是结肠镜检查,患者依从性均较差。研究表明,散发性的结直肠癌往往由于遗传及表观遗传突变的积累导致正常的肠上皮细胞病变形成腺瘤进而癌变的。表观遗传的突变导致基因表达的改变,包括微卫星不稳定、组蛋白修饰及dna甲基化。其中dna甲基化与肿瘤的发生发展联系密切,其具有很好的技术优势及应用前景。dna甲基化是指生物体以s-腺普甲硫氨酸(sam)为供体,在dna甲基转移酶(dnamethyltransferase,dnmt)的催化作用下,生成5一甲基胞嘧啶(5mc)的甲基转移到5’-cpg-3’的c上的过程。该过程不改变蛋白质的一级结构和核苷酸的密码子,是一个可逆的过程,可在基因转录的过程中发挥至关重要的作用,可作为表观遗传学中调控基因最重要的机制,被认为是基因调控的表观遗传机制之一。在大多数研究中,有学者认为cpg岛基因与dna启动子区基因存在重叠,其重叠率高达60%~70%,在正常黏膜中cpg岛并未发生甲基化,该过程不需要基因的调控。而认为cpg岛作为一种肿瘤的抑制基因,其表观遗传修饰与结直肠癌的发生密切有关。在测序技术普及之前,高效液相色谱法(hplc)是经典的甲基化测定方法,但只能测定总的dna甲基化水平,对特定序列无法测定。而对特定dna序列测定的经典方法是southern杂交分析法,但此方法敏感性较低,样品需求量较大。之后,结合pcr技术与酶切技术出现了新的检测技术,例如甲基化敏感的限制性指纹法(msrf)、甲基化敏感扩增多态性法(masp)、限制性标记基因组扫描法(rlgs)和cpg岛扩增结合代表差异分析技术(mca-rda)等。随着测序技术的不断发展和哺乳动物基因组测序的完成,对哺乳动物全基因组水平dna甲基化的检测也逐渐实现。结合新一代测序技术(next-generationsequencing,ngs)的甲基化测定法是目前最新、最具前景的全基因组dna甲基化分析方法,能对dna甲基化进行定量测定且获得大量的序列信息。技术实现要素:本发明的目的是提供一种结直肠癌抑癌基因甲基化高通量检测试剂盒,本发明同时提供了应用此试剂盒结合甲基化特异性多重pcr捕获和重亚硫酸盐测序的方法,该方法能够一次性捕获一个或多个基因的目标甲基化检测区域,获得样本一个或多个基因目标区域甲基化突变序列信息,能够准确有效分析目标区域甲基化状态。本发明一方面提供了一种结直肠癌抑癌基因甲基化高通量检测试剂盒,抑癌基因有9个,包括:sept9、sdc2、wnt2、vim、sfrp2、p16、mgmt、ndrg4和bmp3;试剂盒包括以下成分:(1)10对甲基化特异性多重pcr扩增引物;(2)选自下列正向标签引物和反向标签引物组成的一对或多对,即选自由p-d501~d509中之一和p-n701~n712之一组成的标签引物序列的一对或多对:引物设计方法:确定与结直肠癌发生发展密切相关的抑癌基因启动子cpg岛区域,针对目标区域进行重亚硫酸盐测序(bisulfitesequencing,bsp)pcr引物设计,pcr产物长度选取100-300bp;引物长度选取20-30bp;在正向扩增引物和反向扩增引物的5`端分别加上一段特定的正向通用序列和反向通用序列,组成第一轮甲基化特异性多重pcr扩增引物组合,其中,所述正向通用序列与反向通用序列彼此不同,本发明的通用序列是一段扩增效率比较高的序列,设计通用序列遵循以下原则:(1)通用序列本身的gc含量在40.0%-60.0%,(2)减少通用序列添加在pcr引物上后,对pcr引物造成的严重发卡(haripin)、二聚体(dimer)情况的出现,(3)通用序列不会和模板进行非特异扩增;第二轮扩增标签引物引物:根据样本的数量设计不同的标签序列(index序列),在正向标签序列和反向标签序列的3`端分别加上正向通用序列和反向通用序列构成正向标签引物和反向标签引物,组成第二轮扩增引物(标签引物)组合,其中,设计的标签引物的数量遵循以下规则:正向标签引物的数量×反向标签引物的数量≥样本数量,以及其中,在第二轮扩增引物中添加的正向通用序列和反向通用序列分别与第一轮扩增引物中添加的正向通用序列和反向通用序列相同,以及其中,正向标签引物的标签序列与反向标签引物的标签序列可以彼此相同或不同;本发明设计标签序列遵循以下原则:(1)序列本身的gc含量在40.0%-60.0%,(2)减少标签序列添加在pcr引物上后,对pcr引物造成的严重发卡(haripin)、二聚体(dimer)情况的出现,(3)标签序列中避免3个或3个以上重复碱基。上述技术方案中,进一步地,还包括甲基化特异性扩增酶,能更好地保证重亚硫酸盐处理后的基因组dna扩增效率及特异性;还包括高保真dna聚合酶,由此减少扩增带来的错配。上述技术方案中,进一步地,引物标签序列的长度可以为6-8bp。本发明另一方面提供了一种一次性捕获一个或多个基因目标甲基化检测区域的高通量测序文库的构建方法,包括以下步骤:1)基因组dna重亚硫酸盐处理及纯化回收:将提取好的基因组dna使用qiagenepitectfastbisulfitekit进行重亚硫酸盐处理及纯化回收,处理后未甲基化的胞嘧啶(c)转化为尿嘧啶(u),而甲基化的5-甲基胞嘧啶(5mc)保持不变,接下来经过pcr放大尿嘧啶(u)转换为胸腺嘧啶(t),而5mc和5hmc重新恢复成c,经过测序可以识别目标区域的甲基化状态;2)第一轮甲基化特异性多重pcr扩增:使用前述试剂盒中甲基化特异性多重pcr扩增引物作为第一轮扩增引物对各样本的一个或多个特定基因甲基化检测区域进行扩增,以使每个基因的扩增产物的两端上都加上了通用序列;3)消化引物:用单链消化酶(核酸外切酶i)消化第一轮扩增产物中剩余的引物和引物二聚体;4)片段筛选:利用磁珠筛选对第一轮pcr产物进行片段筛选并纯化回收目标片段;5)第二轮富集扩增:使用前述试剂盒中标签引物作为第二轮扩增引物对第一轮扩增得到的产物进行扩增,以使扩增产物的两端都加上与其他样本区分的可识别的标签序列;6)混合、测序:将各样本的第二轮扩增产物按照所需测序数据量比例混合在一起,并上机测序;7)分析:基于每个样本独特的标签序列,将获得的测序结果与样本一一对应,以及根据每个基因甲基化检测区域的引物序列,将序列对应到样本的每个目标区域上,获得一个或多个基因目标区域甲基化突变序列信息,从而准确有效分析目标区域甲基化状态。上述技术方案中,进一步地,所述步骤3)中,将第一轮多重pcr扩增产物在85℃加热5分钟,然后快速冰浴冷却,使产物中的引物二聚体解链成单链引物,然后用单链消化酶进行消化,由此减少引物二聚体的存在对后续扩增的影响。上述技术方案中,进一步地,所述单链消化酶为核酸外切酶i,该酶为单链特异性3’→5’核酸外切酶,不分解双链dna及rna。上述技术方案中,进一步地,步骤6)中利用pair-end技术,如illuminahiseq2000、illuminahiseq2500和illuminamiseq进行测序,获得dna混合物的序列。本发明的有益效果:本发明提供的结直肠癌早筛dna甲基化高通量检测试剂盒及应用,能够一次性捕获一个或多个基因的目标甲基化检测区域,获得样本一个或多个基因目标区域甲基化突变序列信息,能够准确有效分析目标区域甲基化状态,使得对目标基因区域进行高通量、高灵敏度、高准确度的甲基化检测成为现实,根据大量临床样本验证结果,该方法检测结直肠癌灵敏度达83%,其中对i/ii期结直肠癌检测灵敏度可达91%,对癌前腺瘤检测灵敏度可达64%。该方法还具有成本低、操作简便快捷等技术特点,仅通过两轮pcr就可以完成样本文库构建。附图说明图1本发明甲基化特异性多重pcr文库构建方法过程示意图。具体实施方式以下结合具体实施例对本发明作进一步说明,但不以任何方式限制本发明。下面将结合实施例对本发明的实施方案进行详细的描述,但是本领域技术人员将会理解,在本发明各实施方式中,为了使读者更好地理解本申请而提出了许多技术细节。但是,即使没有这些技术细节,也可以实现本申请各权利要求保护的技术方案。以下实施例中所使用的设备和试剂如下:粪便基因组提取试剂盒(百泰克生物技术有限公司),高速离心机sigma3-30k,核酸扩增仪abi2720,甲基化特异性pcr试剂盒(天根生化科技有限公司),核酸外切酶takara(exonucleasei(e.coli))。实施例1将本发明根据已获得的结直肠癌相关的9个抑癌基因启动子cpg岛区域作为甲基化检测目标区域,设计了针对结直肠癌相关的9个抑癌基因dna甲基化突变检测试剂盒,试剂盒首先将提取到的粪便样本基因组dna经过重亚硫酸盐处理后经过甲基化特异性多重pcr、消化引物、片段筛选、第二轮富集扩增、混合测序、分析等过程获得样本检测结果。1.样本采集、运送保存本发明样本采集选取人粪便样本,采集2g粪便样本于装有保存液的离心管中。样本在常温环境中运输尽快送达实验室。采集的样本在2-8℃条件下保存不超过1周;-20℃条件保存不超过1个月;需长期保存的样本需放于-80℃条件下保存。从样品采集之日起一周之内完成基因组dna的提取。2.粪便样本基因组dna提取粪便基因组dna的提取方法参照百泰克生物技术有限公司的粪便基因组提取试剂盒,分别提取了编号为stool-s1、stool-s2、stool-s3三个人粪便样本。将得到的高质量基因组dna进行重亚硫酸盐处理后用于后续文库构建。3.引物设计确定与结直肠癌发生发展密切相关的抑癌基因启动子cpg岛区域,针对目标区域进行重亚硫酸盐测序(bisulfitesequencing,bsp)pcr引物设计,pcr产物长度选取100-300bp;引物长度选取20-30bp;在正向扩增引物和反向扩增引物的5`端分别加上一段特定的正向通用序列和反向通用序列,组成第一轮扩增引物组合,其中,所述正向通用序列与反向通用序列彼此不同,本发明的通用序列是一段扩增效率比较高的序列,设计通用序列遵循以下原则:(1)通用序列本身的gc含量在40.0%-60.0%,(2)减少通用序列添加在pcr引物上后,对pcr引物造成的严重发卡(haripin)、二聚体(dimer)情况的出现,(3)通用序列不会和模板进行非特异扩增。设计的引物如下,下划线是引入的通用序列:表1第一轮甲基化特异性多重pcr扩增引物第二轮扩增引物:根据样本的数量设计不同的标签序列(index序列),在正向标签序列和反向标签序列的3`端分别加上正向通用序列和反向通用序列构成正向标签引物和反向标签引物,组成第二轮扩增引物(标签引物)组合,其中,设计的标签引物的数量遵循以下规则:正向标签引物的数量×反向标签引物的数量≥样本数量,以及其中,在第二轮扩增引物中添加的正向通用序列和反向通用序列分别与第一轮扩增引物中添加的正向通用序列和反向通用序列相同,以及其中,正向标签引物的标签序列与反向标签引物的标签序列可以彼此相同或不同;本发明设计标签序列遵循以下原则:(1)序列本身的gc含量在40.0%-60.0%,(2)减少标签序列添加在pcr引物上后,对pcr引物造成的严重发卡(haripin)、二聚体(dimer)情况的出现,(3)标签序列中避免3个或3个以上重复碱基。第二轮扩增引物如下:表2第二轮富集扩增正向/反向标签引物4.粪便样本基因组dna重亚硫酸盐处理基因组dna重亚硫酸盐处理及纯化回收:将提取好的基因组dna使用qiagenepitectfastbisulfitekit进行重亚硫酸盐处理及纯化回收:(1)准备200μlpcr管,按照如下体系加入反应各组分:(2)盖上pcr管盖后,混匀重亚硫酸盐反应体系(颜色指示剂为蓝色确保重亚硫酸盐效果),使用pcr仪进行重亚硫酸盐甲基化处理反应:(3)重亚硫酸盐反应产物纯化:①将200μl反应产物转移到新的1.5mlep管中,向ep管中加入310μlbufferbl与250μl无水乙醇,涡旋震荡15s,瞬时离心后将混合液转移至吸附柱minelutednaspincolumn(耗材需2-8℃保存),12000rpm离心1min。②离心后倒掉收集管中废液,向吸附柱minelutednaspincolumn加入500μlbufferbw(使用前确认添加乙醇),12000rpm离心1min。③离心后倒掉收集管中废液,向吸附柱minelutednaspincolumn加入500μlbufferbd(使用前确认添加乙醇,需2-8℃保存),室温静置15min,12000rpm离心1min。④离心后倒掉收集管中废液,向吸附柱minelutednaspincolumn加入500μlbufferbw(使用前确认添加乙醇),12000rpm离心1min。⑤重复④步骤1次,共洗涤2次。⑥离心后倒掉收集管中废液,向吸附柱minelutednaspincolumn加入250μl无水乙醇,12000rpm离心1min。⑦离心后倒掉废液将吸附柱转移至新的废液收集管中,12000rpm离心1min,保证去除乙醇以免影响后续实验。⑧将吸附柱转移至新的1.5mlep管中,向吸附柱膜中央加入15μlbuffereb,室温静置1min,12000rpm离心1min回收纯化产物(产物2-8℃可保存24h,尽量避免反复冻融,长期保存至于-20℃)。3.样本文库构建(1)第一轮甲基化特异性多重pcr扩增:使用设计好的由通用序列和针对特定基因甲基化检测区域的扩增引物组成的第一轮扩增引物对目标区域进行捕获,将甲基化特异性多重pcr引物混合并稀释至每条引物工作液浓度为2μm,得到引物pool工作液,而后按照如下pcr体系及条件进行第一轮甲基化特异性多重pcr扩增:多重pcr反应体系(20μl):pcr反应条件:(2)消化引物:将第一轮多重pcr扩增产物在85℃加热5分钟,然后快速冰浴冷却,使产物中的引物二聚体解链成单链引物,然后用单链消化酶(核酸外切酶i)消化第一轮扩增产物中剩余的引物和引物二聚体,向上述20μl第一轮多重pcr反应产物中加入0.5μl的核酸外切酶i(20u/μl),37℃反应30min。(3)片段筛选:利用磁珠筛选(0.6-0.9×)对第一轮pcr产物进行片段筛选并纯化回收目标片段:磁珠室温平衡30min,并配制足够的80%乙醇,取第一轮多重pcr扩增产物20.5μl,补水至体积40μl,加入磁珠24μl,混匀后室温孵育5min,磁力悬浮至溶液变澄清,将上清转移至新管中,在上清中加入12μl磁珠,混匀后室温孵育5min,磁力悬浮至溶液变澄清弃上清。200μl80%乙醇洗涤两次,晾干后加入25μlddh2o,混匀后室温孵育5min,磁力悬浮至溶液变澄清,吸出23.5μl用于第二轮pcr富集扩增。(4)第二轮富集扩增:使用由标签(index)序列和通用序列组成的第二轮扩增引物对第一轮扩增得到的产物进行扩增,以使扩增产物的两端都加上与其他样本区分的可识别的标签序列:pcr反应体系:pcr反应条件:(5)pcr反应后进行产物纯化(0.9×纯化):磁珠室温平衡30min,并配制足够的80%乙醇,取pcr产物50μl,加入磁珠45μl,混匀后室温孵育5min,磁力悬浮至溶液变澄清,磁力悬浮至溶液变澄清弃上清。200μl80%乙醇洗涤两次,晾干后加入32μlddh2o,混匀后室温孵育5min,磁力悬浮至溶液变澄清,取出31μl产物,qubit定量及文库片段大小质控。4.文库混合、测序及分析根据样本文库qubit定量结果将各样本文库按照所需测序数据量比例混合在一起,并上机测序。获得测序数据后基于每个样本独特的标签序列,将获得的测序结果与样本一一对应,以及根据每个基因甲基化检测区域的引物序列,将序列对应到样本的每个目标区域上,获得一个或多个基因目标区域甲基化突变序列信息,从而准确有效分析目标区域甲基化状态。表3粪便样本基因组dna甲基化检测区域reads统计stool-s1stool-s2stool-s3wnt213687reads17650reads14650readsvim37093reads26259reads25463readssfrp223026reads26000reads27621readssept9-130336reads25903reads29633readssept9-245734reads50467reads43226readssdc211236reads13819reads10386readsp1610739reads11802reads11350readsndrg411589reads10261reads11690readsmgmt25493reads15386reads17856readsbmp332009reads39359reads40211reads如表3所示,通过高通量测序获得了3个粪便样本的测序数据,针对结直肠癌相关9个基因的10个甲基化检测区域进行了比对,统计出每个检测区域的reads数,结果显示各区域reads深度均达到10000×,满足肿瘤早筛应用,且区域均一性良好,大大节约检测成本。表4stool-s1样本甲基化突变结果统计甲基化检测区域区域总reads甲基化突变reads甲基化突变率(%)wnt213687reads0reads0vim37093reads0reads0sfrp223026reads1711reads7.43sept9-130336reads0reads0sept9-245734reads0reads0sdc211236reads0reads0p1610739reads0reads0ndrg411589reads0reads0mgmt25493reads0reads0bmp332009reads0reads0表5stool-s2样本甲基化突变结果统计甲基化检测区域区域总reads甲基化突变reads甲基化突变率(%)wnt217650reads0reads0vim26259reads0reads0sfrp226000reads0reads0sept9-125903reads0reads0sept9-250467reads0reads0sdc213819reads0reads0p1611802reads0reads0ndrg410261reads0reads0mgmt15386reads0reads0bmp339359reads0reads0表6stool-s3样本甲基化突变结果统计甲基化检测区域区域总reads甲基化突变reads甲基化突变率(%)wnt214650reads0reads0vim25463reads0reads0sfrp227621reads0reads0sept9-129633reads0reads0sept9-243226reads0reads0sdc210386reads0reads0p1611350reads0reads0ndrg411690reads4reads0.034mgmt17856reads0reads0bmp340211reads0reads0如表4、表5、表6分别列举了三个粪便样本的甲基化检测结果,分别统计了比对到目标甲基化检测区域的reads数和目标区域中检测到的甲基化突变的reads数,并计算得出甲基化突变频率。由表中所示,stool-s1样本中sfrp2基因甲基化突变频率高于检出限,判断dna甲基化突变检测结果为阳性;stool-s2和stool-s3样本dna甲基化突变检测结果为阴性。实施例2本发明收集了大量临床样本进行了检测方法的灵敏度性能验证,共收集得到199例临床结直肠癌/结直肠腺瘤患者粪便标本,并按照实施例1中所述实验方法进行检测,根据所得结果得到本发明所述的检测方法的灵敏度验证数据(如表7),199例临床标本中由140例i/ii期结直肠癌患者粪便样本和59例结直肠腺瘤患者粪便样本所组成,经过检测后得到检测结果为140例i/ii期结直肠癌患者中127例检测结果为阳性,本发明所述的检测试剂盒对i/ii期结直肠癌检测灵敏度可达91%;59例结直肠腺瘤患者中38例检测结果为阳性,本发明所述的检测试剂盒对癌前腺瘤检测灵敏度可达64%;本发明所述的检测试剂盒对结直肠癌/腺瘤总体检测灵敏度可达83%。表7检测方法灵敏度验证结果获得样本数检测结果为阳性样本数检测灵敏度i/ii期结直肠癌患者14012791%结直肠癌前腺瘤患者593864%总计19916583%针对本发明,本领域的普通技术人员可以理解,上述各实施方式是实现本发明的具体实施实施例,而在实际操作中可在操作细节上作出适当改变,而不偏离发明的精神和范围。本发明的保护范围由所附的权利要求定义,但同时还包含权利要求所限定的内容的等同变换形式。sequencelisting<110>大连晶泰生物技术有限公司<120>一种结直肠癌抑癌基因甲基化高通量检测试剂盒及其应用<130>2019<160>41<170>patentinversion3.5<210>1<211>42<212>dna<213>人工合成<400>1cctacacgacgctcttccgatcttggtgggggtgttagttgt42<210>2<211>49<212>dna<213>人工合成<400>2ttcagacgtgtgctcttccgatctacaaccaaacaaaattctctatcac49<210>3<211>49<212>dna<213>人工合成<400>3cctacacgacgctcttccgatcttttatttagttgagttagggggttta49<210>4<211>42<212>dna<213>人工合成<400>4ttcagacgtgtgctcttccgatctaacccaacacccaccttc42<210>5<211>53<212>dna<213>人工合成<400>5cctacacgacgctcttccgatctggaatttttagggaagttttaggttaaaga53<210>6<211>54<212>dna<213>人工合成<400>6ttcagacgtgtgctcttccgatcttacaaatactaactcttcatcccaacttca54<210>7<211>50<212>dna<213>人工合成<400>7cctacacgacgctcttccgatctagttttaggtttyggagtaggaaggtt50<210>8<211>45<212>dna<213>人工合成<400>8ttcagacgtgtgctcttccgatctaaaatcccctcccactaccat45<210>9<211>43<212>dna<213>人工合成<400>9cctacacgacgctcttccgatctaggggttggttggttattag43<210>10<211>43<212>dna<213>人工合成<400>10ttcagacgtgtgctcttccgatctccctctacccacctaaatc43<210>11<211>49<212>dna<213>人工合成<400>11cctacacgacgctcttccgatctgattatttttgtgataggaaaaggta49<210>12<211>46<212>dna<213>人工合成<400>12ttcagacgtgtgctcttccgatctacctaaaacaatctacgcatcc46<210>13<211>43<212>dna<213>人工合成<400>13cctacacgacgctcttccgatctcggttttcgttcgttttttc43<210>14<211>42<212>dna<213>人工合成<400>14ttcagacgtgtgctcttccgatctcctcgagcgtaacttccg42<210>15<211>46<212>dna<213>人工合成<400>15cctacacgacgctcttccgatctgggaggagttaatgaagggtaat46<210>16<211>49<212>dna<213>人工合成<400>16ttcagacgtgtgctcttccgatctactcatacctaacaacccaacaaaa49<210>17<211>54<212>dna<213>人工合成<400>17cctacacgacgctcttccgatcttgtcgggagtgtagaaattaataagtgagag54<210>18<211>50<212>dna<213>人工合成<400>18ttcagacgtgtgctcttccgatctacacgcaaaccaccaaacccaaaata50<210>19<211>48<212>dna<213>人工合成<400>19cctacacgacgctcttccgatctgggttagygtagtaagtggggttgg48<210>20<211>56<212>dna<213>人工合成<400>20ttcagacgtgtgctcttccgatctaacctactcrccccaaccataactaaataccc56<210>21<211>70<212>dna<213>人工合成<400>21aatgatacggcgaccaccgagatctacactagatcgcacactctttccctacacgacgct60cttccgatct70<210>22<211>70<212>dna<213>人工合成<400>22aatgatacggcgaccaccgagatctacacctctctatacactctttccctacacgacgct60cttccgatct70<210>23<211>70<212>dna<213>人工合成<400>23aatgatacggcgaccaccgagatctacactatcctctacactctttccctacacgacgct60cttccgatct70<210>24<211>70<212>dna<213>人工合成<400>24aatgatacggcgaccaccgagatctacacagagtagaacactctttccctacacgacgct60cttccgatct70<210>25<211>70<212>dna<213>人工合成<400>25aatgatacggcgaccaccgagatctacacgtaaggagacactctttccctacacgacgct60cttccgatct70<210>26<211>70<212>dna<213>人工合成<400>26aatgatacggcgaccaccgagatctacacactgcataacactctttccctacacgacgct60cttccgatct70<210>27<211>70<212>dna<213>人工合成<400>27aatgatacggcgaccaccgagatctacacaaggagtaacactctttccctacacgacgct60cttccgatct70<210>28<211>70<212>dna<213>人工合成<400>28aatgatacggcgaccaccgagatctacacctaagcctacactctttccctacacgacgct60cttccgatct70<210>29<211>70<212>dna<213>人工合成<400>29aatgatacggcgaccaccgagatctacacgcgtaagaacactctttccctacacgacgct60cttccgatct70<210>30<211>65<212>dna<213>人工合成<400>30caagcagaagacggcatacgagattaaggcgagtgactggagttcagacgtgtgctcttc60cgatc65<210>31<211>65<212>dna<213>人工合成<400>31caagcagaagacggcatacgagatcgtactaggtgactggagttcagacgtgtgctcttc60cgatc65<210>32<211>65<212>dna<213>人工合成<400>32caagcagaagacggcatacgagataggcagaagtgactggagttcagacgtgtgctcttc60cgatc65<210>33<211>65<212>dna<213>人工合成<400>33caagcagaagacggcatacgagattcctgagcgtgactggagttcagacgtgtgctcttc60cgatc65<210>34<211>65<212>dna<213>人工合成<400>34caagcagaagacggcatacgagatggactcctgtgactggagttcagacgtgtgctcttc60cgatc65<210>35<211>65<212>dna<213>人工合成<400>35caagcagaagacggcatacgagattaggcatggtgactggagttcagacgtgtgctcttc60cgatc65<210>36<211>65<212>dna<213>人工合成<400>36caagcagaagacggcatacgagatctctctacgtgactggagttcagacgtgtgctcttc60cgatc65<210>37<211>65<212>dna<213>人工合成<400>37caagcagaagacggcatacgagatcagagagggtgactggagttcagacgtgtgctcttc60cgatc65<210>38<211>65<212>dna<213>人工合成<400>38caagcagaagacggcatacgagatgctacgctgtgactggagttcagacgtgtgctcttc60cgatc65<210>39<211>65<212>dna<213>人工合成<400>39caagcagaagacggcatacgagatcgaggctggtgactggagttcagacgtgtgctcttc60cgatc65<210>40<211>65<212>dna<213>人工合成<400>40caagcagaagacggcatacgagataagaggcagtgactggagttcagacgtgtgctcttc60cgatc65<210>41<211>65<212>dna<213>人工合成<400>41caagcagaagacggcatacgagatgtagaggagtgactggagttcagacgtgtgctcttc60cgatc65当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1