一种转录组测序文库快速构建方法及其应用与流程
本申请要求了2018年12月28日提交中国专利局的,申请号为201811620280.0,发明名称为“一种转录组测序文库快速构建方法及其应用”的中国专利申请的优先权,其全部内容通过引用结合在本申请中。本发明涉及高通量测序及文库构建领域,更具体地,涉及一种转录组测序文库快速构建方法及其应用。
背景技术:
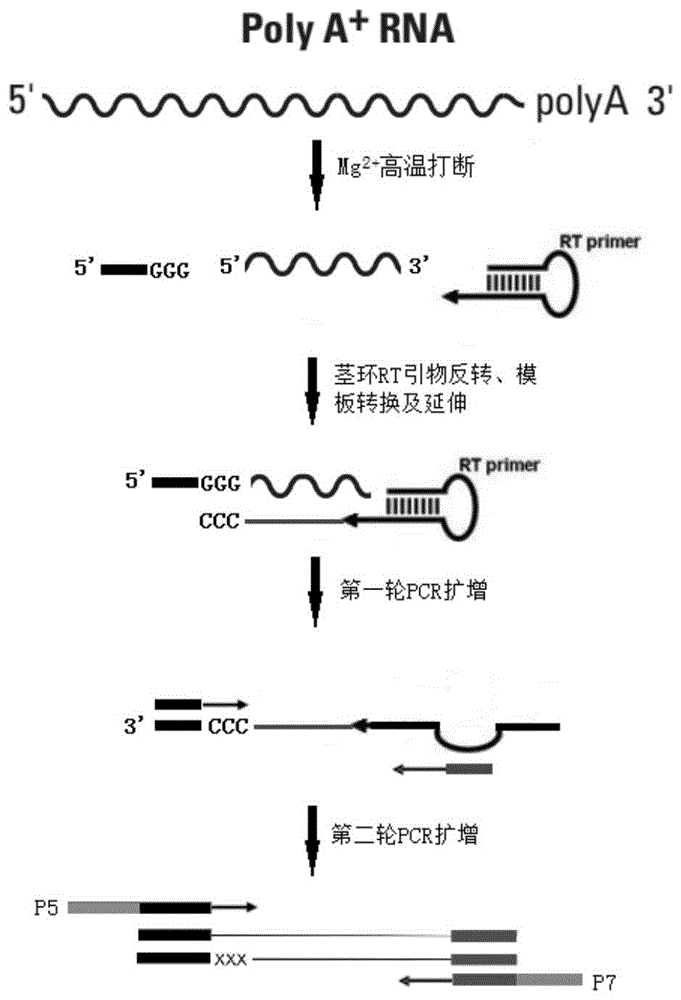
:cdna文库的构建是研究生物体功能基因组的主要技术手段。不同的rna-seq高通量测序建库方法,在覆盖度、灵敏度和多路复用能力、所建文库的全长率等方面表现各异。转录组建库时通常先去除总rna中的rrna,然后利用现有技术中的rna文库构建试剂盒进行转录组文库的构建。真核细胞的mrna3’端都带有polya。现有的转录组测序技术,一般利用逆转录酶对polya尾进行反转录,制备cdna文库。常见的做法是在合成cdna的双链后再两端连上接头,利用已知的接头序列再进行扩增,或者是利用末端转移酶在双链cdna的3’末端加上一连串的g或者c(或者a/t),再通过补齐末端,利用已知的两头序列进行扩增。转录组建库常用试剂为nebultra系列、illuminatruseq系列及kapahyper系列等,而建库试剂中的接头主要有三种经典结构,包括平端双链接头(以thermo公司的建库试剂为代表)、y型结构双链接头(以illumina公司的建库试剂为代表)和u型结构单链接头(以neb公司的建库试剂为代表)。以illuminatruseq法为例,具体的常规流程如下:1)总rna的分离和纯化:利用带有oligo(dt)的磁珠将mrna特异性吸附并分离出来,从而实现mrna的纯化;或除核糖体rna(rrna);2)将rna打断成200-600bp长度的片段;3)合成反转录cdna第一链:以片段化的rna为模板,反转录成cdna第一链,利用核酸纯化磁珠分离此杂交链;4)合成cdna第二链:rnase降解杂交链中的rna链,dna聚合酶合成第二链,磁珠纯化;5)末端修复:用酶和dntp将双链cdna末端补平;6)加腺苷酸a;7)加接头:利用t4dnaligase将通用adapter加到dna片段两端,磁珠纯化;8)pcr扩增:利用高保真dna聚合酶富集已加接头的dna双链,磁珠纯化。但是,加接头或末端修复再进行扩增,这些建库方法都存在着一些不同程度的问题,比如,接头的连接效率非常有限,会导致部分信息的丢失:平末端的双链接头效率中等,易形成接头自连;y型结构双链接头连接效率偏低,制作工艺需要额外添加退火步骤,而且退火前两条单链的浓度及比例常因为定量问题而存在批次间产生比例失衡而不稳定;u型结构单链接头由于为单链结构,天然形成部分双链的茎环结构,无需额外退火,连接效率高,不易产生接头自连,但是需要在文库pcr扩增之前使用酶把u型结构打开,增加实验的步骤及试剂成本。这些方法需要多次用不同的酶处理有限的样品,需要经过反复的纯化,会损失很多有用的信息。特别是少量样品中的低丰度信息,很大程度上会影响结果的准确性;现阶段所有的接头设计序列上仍然受到测序上游公司的制约,接头的连接效率参差,影响着最终文库得率。此外,上述文库构建方法是针对200bp以上的长rna的,此外也有针对100bp以下的小rna文库构建法,例如qiagen的mirna二代测序建库产品qiaseqmirnalibrarykits。但是并没有针对100~300bp长度rna的高效文库构建方法。有鉴于此,有必要提供一种适合100~300bp长度的rna的高效文库构建方法。技术实现要素:针对100~300bp长度的rna高效文库的构建需求,本发明提供了一种转录组测序文库快速构建方法及其应用。本发明上述目的通过以下技术方案实现:第一方面,本发明提供了一种转录组测序文库快速构建方法,包括如下步骤:1)获取样品中的mrna,磁珠捕获带polya尾的mrna;2)将1)所得产物加入含mg2+的第一缓冲体系中,在90-105℃条件下反应,获得片段化处理后的产物;3)将2)所得产物加入cdna第一链合成体系中得混合物,反应获得含有cdna第一链的产物,所述混合物中包括逆转录引物、tso引物;其中:所述逆转录引物可形成茎环结构,所述逆转录引物的序列包括:茎区-环区-茎区-poly(n)区,n选自atcg中的任一种;所述tso引物的序列包括:第一接头序列-第二接头序列-poly(g)区;所得cdna第一链的5'末端连接有所述tso引物,3'末端连接有所述逆转录引物;4)将3)所得产物加入第一轮pcr体系中,扩增获得第一轮pcr扩增产物,其中,所述第一轮pcr体系中,采用的扩增引物包括如下引物组:引物组一:fr引物、p7rf引物,其中,fr的序列包括:tso引物的第一接头序列;p7rf的序列包括:逆转录引物的环区-茎区序列;5)将4)所得第一轮pcr扩增产物进行第二轮pcr扩增,获得mrna文库;其中,所述第二轮pcr扩增中,采用的扩增引物包括如下引物组:引物组二:p5fr引物、p7rf引物,其中,p5fr的序列包括:tso引物的第一接头序列以及第二接头序列。优选地,所述步骤2)中的含mg2+的第一缓冲体系中含有逆转录引物或所述步骤3)中的cdna第一链合成体系中含有逆转录引物。优选地,所述步骤2)中,所述第一缓冲体系为:优选地,所述步骤2)中,在94℃条件下片段化处理8-12分钟获得片段化处理后的mrna。优选地,所述步骤2)中,在90-105℃条件下反应,获得片段化处理后的mrna长度为100-300bp。优选地,所述步骤3)中,所述第一链合成体系为:可以理解的是,本领域技术人员根据本领域的常规理解,可以用其他等效于episcripttmiv的逆转录酶替代episcripttmiv。优选地,所述步骤3)中,所述第一链合成体系反应条件为:优选地,所述逆转录引物序列为ggagatcgtggagttcagacgtgtgctcttccgatctccnnnnnnn。优选地,所述tso引物序列为tctttccctacacgacgctcttccgatctrgrg+g。本领域技术人员可知的,所述tso引物的poly(g)区包括:有两个核糖鸟苷(rg)和一个lna-修饰的鸟苷(+g)。优选地,所述步骤4)中,所述fr引物序列为tctttccctacacgacgctcttc。优选地,所述步骤5)中,所述p5fr引物序列为aatgatacggcgaccaccgagatctacactctttccctacacgacgctcttccgatct。优选地,所述步骤4)或步骤5)中,所述p7rf引物序列为caagcagaagacggcatacgagat-index-gtgactggagttcagacgtgtgctcttccgatct。第二方面,本发明提供了一种转录组测序文库快速构建试剂盒,包括第一方面所述的第一缓冲体系、第一链合成体系。第三方面,本发明提供了一种转录组测序文库快速构建方法或转录组测序文库快速构建试剂盒在构建转录组测序文库中的应用。本发明具有以下有益效果:1.无需加接头。在反转录时,反转录酶自动在新合成的cdna末端添加几个(dc);加入本发明设计的引物,反转录酶就会自动转换模板延伸cdna。这样得到的所有cdna单链都含有已知的引物序列,可以方便地进行pcr扩增,包括中小长度的rna。2.利用逆转录酶内的末端转移酶活性,只要单管,一步即可完成,不需要额外的cdna抽提纯化或者沉淀,或者额外的酶反应,在本发明一些实施例中,只需要少至1ng的mrna就可以得到高质量、高产量的cdna库,更重要的是得到的cdna能够代表原有样品中的rna的丰度,可以应用于直接扩增基因、构建cdna文库。3.优化的反转录酶缓冲液,能够优先反转录100~300bp长度的rna,高效合成cdna并扩增,文库得率高。综上,本发明提供了的技术方案,相比于现有报道,本发明是专门针对100~300bp中小长度的rna的高效文库构建方法,合成cdna前无需任何酶反应和化学反应处理,不会导致处理过程中rna的降解和损耗;实验过程快速、简单,整个过程没有对rna和中间产物的复杂处理,特别适用于起始样本量少的情况,实现高效的连接,最终得到浓度足够高的转录组文库,完成上机测序和后续的转录组测序相关的应用,有效实现单细胞、外泌体或珍贵临床样本的转录组测序在基础研究、临床诊断和药物研发等邻域的广泛应用。附图说明图1为本发明实施例提供的文库构建流程示意图;图2为本发明实施例提供的基因表达箱线分布图。具体实施方式以下结合说明书附图和具体实施例来进一步说明本发明,但实施例并不对本发明做任何形式的限定。除非特别说明,本发明采用的试剂、方法和设备为本
技术领域:
常规试剂、方法和设备。除非特别说明,以下实施例所用试剂和材料均为市购。未注明具体条件的实验方法,通常按照常规条件,或制造厂商所建议条件实施。在本发明一具体实施例中,结合图1所示的文库构建流程示意图,本发明提供了一种转录组测序文库快速构建方法及其应用。本发明提供的用于转录组测序文库快速构建方法,采用了本发明提供的用于转录组测序文库快速构建试剂盒,所述方法包括但不限于如下步骤的一个或多个步骤:采用mrna试剂盒提取样品中的mrna:1)用oligo(dt)磁珠捕获完整的带polya尾mrna后,加入16μl步骤1.1配置的打断buffer,94℃高温打断10min,使rna片段主峰在100-350bp范围内。1.1按照下表配置打断buffer1.2轻弹管壁混匀,短暂离心收集后置于冰上1.3按照以下程序反应,热盖105℃:2)第一链cdna合成2.1按照下表配制反应体系:2.2配置完成后,轻弹管壁混匀,短暂离心收集样本。2.3按照以下程序反应:3)第一轮pcr扩增3.1按照下表配置反应体系:3.2配置完成后,震荡混匀,短暂离心收集样本。3.3按照以下程序反应:3.4第一轮pcr产物用1x磁珠纯化,12μl水洗脱。4)第二轮pcr扩增4.1按照下表配置反应体系:4.2配置完成后,震荡混匀,短暂离心收集样本。4.3按照以下程序反应:4.4第二轮pcr产物用1x磁珠纯化,23μl水洗脱。5)qsep100全自动核酸蛋白分析系统检测文库主峰大小6)上述步骤所用引物序列如表1所示:本发明提供的技术方案用到的index序列如表2所示:序号序列序号序列s502ctctctatn701taaggcgas503tatcctctn702cgtactags505gtaaggagn703aggcagaas506actgcatan704tcctgagcs507aaggagtan705ggactccts508ctaagcctn706taggcatgs510cgtctaatn707ctctctacs511tctctccgn710cgaggctgs513tcgactagn711aagaggcas515ttctagctn712gtagaggas516cctagagtn714gctcatgas517gcgtaagan715atctcaggs518ctattaagn716actcgctas520aaggctatn718ggagctacs521gagccttan719gcgtagtas522ttatgcgan720cggagcctn721tacgctgcn722atgcgcagn723tagcgctcn724actgagcgn726cctaagacn727cgatcagtn728tgcagctan729tcgacgtc具体实验结果1基因组比对测序获得的序列数据经过质量控制以后,采用hisat2软件[1]与物种人的基因组进行比对,基因组比对率是衡量数据质量的一个标准,常规方法(test2)总rna起始量需要1μg-2μg,其基因组比对率(mappingrate)77.8%,本发明方法(test4)总rna起始量可低至1ng,其基因组比对率为82%,优于常规方法(test2),表明本发明方法可以以更少的样品起始量得到比常规方法更好的检测效果。表1基因组比对数据统计allreads:参与基因组比对的总序列数,即质量控制以后的测序序列数unmapped:与基因组未比对上的序列数mapped:即总参与比对序列中能够比对到基因组上的测序序列数mappingrate:基因组比对率,即测序序列比对到基因组占总测序过滤后序列的比值uniquemapped:总参与比对序列中能够在基因组上只有唯一比对位置的序列数uniquemappedrate:唯一位置比对率,即唯一位置比对序列占从测序过滤后序列的比值常规建库方法(test2)流程包括:mrna捕获,mrna打断,第一链cdna合成,第二链cdna合成,末端修复,接头连接,pcr富集;总rna起始量需要1μg-2μg。2表达量计算基因表达(geneexpression)是进行转录组测序研究的基础。根据基因组比对结果文件,利用htseq-count[2]计算基因的表达量,fpkm(fragmentsperkilobasemillionreads)用来进行标准化,所有基因的表达量fpkm结果分布图如图2所示,其中纵轴代表fpkm的log10对数转换值,矩形盒两端边的位置分别对应数据的上下四分位数(q1和q3),矩形盒内部的一条线段为中位数,结果显示fpkm分布的下四分位数(所有基因表达量从小到大排序后第25%的数字)位于1以上(对应图中对数转换的坐标值0),本方法基因表达量中位数高于常规方法,可以检测到fpkm大于1的基因在75%以上(1-25%=75%),箱线图表明该方法检测到了大部分基因,并且优于常规方法。生信分析参考文献[1]kimd,langmeadb,salzbergsl.hisat:afastsplicedalignerwithlowmemoryrequirements[j].naturemethods,2015,12(4):357.[2]anderss,pylpt,huberw.htseq—apythonframeworktoworkwithhigh-throughputsequencingdata[j].bioinformatics,2015,31(2):166-169.应当指出,对于本
技术领域:
的普通技术人员来说,在不脱离本发明实施例原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也视为本发明实施例的保护范围。序列表<110>广州表观生物科技有限公司<120>一种转录组测序文库快速构建方法及其应用<160>6<170>siposequencelisting1.0<210>1<211>46<212>dna<213>人工序列(artificialsequence)<400>1ggagatcgtggagttcagacgtgtgctcttccgatctccnnnnnnn46<210>2<211>32<212>dna<213>人工序列(artificialsequence)<400>2tctttccctacacgacgctcttccgatctggg32<210>3<211>23<212>dna<213>人工序列(artificialsequence)<400>3tctttccctacacgacgctcttc23<210>4<211>58<212>dna<213>人工序列(artificialsequence)<400>4aatgatacggcgaccaccgagatctacactctttccctacacgacgctcttccgatct58<210>5<211>24<212>dna<213>人工序列(artificialsequence)<400>5caagcagaagacggcatacgagat24<210>6<211>34<212>dna<213>人工序列(artificialsequence)<400>6gtgactggagttcagacgtgtgctcttccgatct34当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1