提高丝状真菌细胞在多肽的产生方面的生产力的方法与流程
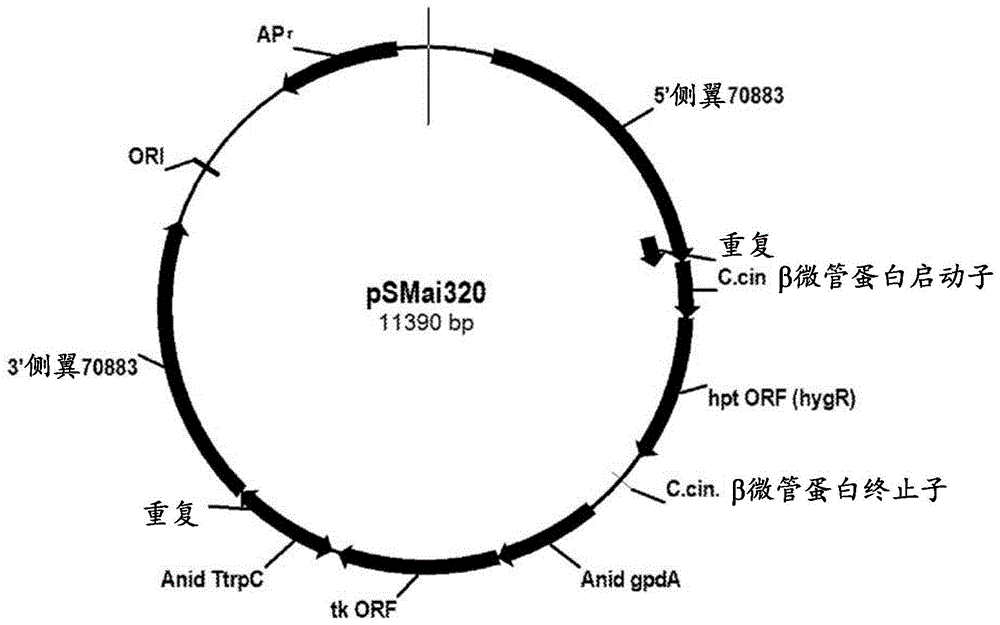
序列表的引用本申请含有计算机可读形式的序列表,将其通过引用并入本文。发明背景本发明涉及在用于产生多肽的转录因子中有缺陷的分离的突变体丝状真菌细胞。
背景技术:
:丝状真菌广泛用于生产各种工业应用的酶和其他生物制品。丝状真菌细胞在目的多肽的产生中的生产力取决于几个因素,如碳源、氮源、分泌、ph、温度和溶解氧。特别地,碳源可以确定诱导和/或抑制哪些分泌酶基因,和其的生产率。碳源通过转录因子和其相关的启动子起作用,根据碳源的水平激活或抑制这些启动子。通常由此类启动子驱动纤维素酶和半纤维素酶的表达。已知几种与纤维素酶和半纤维素酶基因的启动子区相互作用并调节其的表达的转录因子(ilmen等人,1996,mol.gen.genet.[分子与普通遗传学]251:451-460;aro等人,2001,j.biol.chem.[生物化学杂志]276:24309-24314;zeilinger等人,2001,mol.genet.genom.[分子遗传学和基因组学]266:56-63;aro等人,2003,appl.environ.microbiol.[应用与环境微生物学]69:56-65;mach等人,2003,appl.microbiol.biotechnol.[应用微生物学与生物技术]60:515-522;schmoll等人,2003,actamicrobiologicaetimmunologicahungarica[匈牙利微生物学与免疫学学报]50:125-145;strieker等人,2006,eukaryoticcell[真核细胞]5:2128-2137;strieker等人,2007,febsletters[欧洲生化学会联合会快报]581:3915-3920;seidl等人,2008,bmcgenomics[bmc基因组学]9:327-341;strieker等人,2008,appl.microbiol.biotechnol.[应用微生物学与生物技术]78:211-220;kubicek等人,2009,biotechnologyforbiofuels[用于生物燃料的生物技术]2:19-3;nakari-setala等人,2009,appl.environ.microbiol.[应用与环境微生物学]75:4853-4860)。在产生纤维素酶的丝状真菌延长时间发酵后,可以出现纤维素酶-阴性表型。本领域需要鉴定编码转录因子的基因,可以修饰这些基因以减少或消除纤维素酶-阴性表型。本发明提供了在目的多肽的产生中提高丝状真菌细胞的生产力的改进的方法,其中修饰了转录因子的活性。技术实现要素:本发明涉及亲本丝状真菌细胞的分离的突变体,该分离的突变体包含目的多肽的在启动子的转录控制下的编码序列,该启动子通过选自由以下组成的组的一种或多种转录因子调节:(a)包含氨基酸序列的转录因子,该氨基酸序列与seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、或124具有至少70%序列同一性;(b)由多核苷酸编码的转录因子,该多核苷酸包含与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123具有至少70%序列同一性的核苷酸序列;以及(c)由多核苷酸编码的转录因子,该多核苷酸在高严格条件下与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123的全长互补序列杂交;其中将该一种或多种转录因子基因在亲本丝状真菌细胞中修饰以产生该突变体,使得该突变体在该一种或多种转录因子的产生方面有部分或完全缺陷,其中(i)当在与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相同的条件下培养时,该一种或多种转录因子基因的修饰提高了该突变体在该目的多肽的产生方面的生产力,(ii)与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相比,该一种或多种转录因子基因的修饰减少或消除了所得突变体中的纤维素酶阴性表型,或(iii)该一种或多种转录因子基因的修饰导致(i)和(ii)的组合。本发明还涉及产生目的多肽的方法,该方法包括在用于产生该目的多肽的培养基中培养此类突变体丝状真菌细胞,并且任选地回收该目的多肽。本发明还涉及用于构建亲本丝状真菌细胞的突变体的方法,该方法包括修饰各自编码该亲本丝状真菌细胞中的转录因子的一种或多种基因以产生该突变体,并且任选地回收该突变体,其中该亲本丝状真菌细胞或其突变体包含在启动子的转录控制下的目的多肽编码序列,该启动子由这些转录因子中的一种或多种调节,其中该一种或多种转录因子选自由以下组成的组:(a)包含氨基酸序列的转录因子,该氨基酸序列与seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、或124具有至少70%序列同一性;(b)由多核苷酸编码的转录因子,该多核苷酸包含与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123具有至少70%序列同一性的核苷酸序列;以及(c)由多核苷酸编码的转录因子,该多核苷酸在高严格条件下与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123的全长互补序列杂交;其中将该一种或多种转录因子基因在亲本丝状真菌细胞中修饰以产生该突变体,使得该突变体在该一种或多种转录因子的产生方面有部分或完全缺陷,其中(i)当在与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相同的条件下培养时,该一种或多种转录因子基因的修饰提高了该突变体在该目的多肽的产生方面的生产力,(ii)与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相比,该一种或多种转录因子基因的修饰减少或消除了所得突变体中的纤维素酶阴性表型,或(iii)该一种或多种转录因子基因的修饰导致(i)和(ii)的组合;。本发明还涉及分离的转录因子,该分离的转录因子选自由以下组成的组:(a)包含氨基酸序列的转录因子,该氨基酸序列与seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、或124具有至少70%序列同一性;(b)由多核苷酸编码的转录因子,该多核苷酸包含与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123具有至少70%序列同一性的核苷酸序列;以及(c)由多核苷酸编码的转录因子,该多核苷酸在高严格条件下与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123的全长互补序列杂交。本发明还涉及编码本发明的转录因子的分离的多核苷酸;包含编码本发明的转录因子的多核苷酸的核酸构建体、表达载体、和重组宿主细胞;和产生此类转录因子的方法。附图说明图1示出质粒psmai320的图谱。图2示出质粒psmai321的图谱。图3示出质粒psmai322a的图谱。图4示出针对里氏木霉(t.reesei)gmer62-1a9发酵,作为时间函数,cmc板上产生纤维素酶的菌落数目的表型分析。图5示出质粒pbtp01的图谱。定义乙酰木聚糖酯酶:术语“乙酰木聚糖酯酶”意指羧基酯酶(ec3.1.1.72),其催化乙酰基基团从聚合木聚糖、乙酰化木糖、乙酰化葡萄糖、乙酸α-萘酯(alpha-napthylacetate)和对硝基苯乙酸酯(p-nitrophenylacetate)的水解。可以在含有0.01%tweentm20(聚氧乙烯脱水山梨糖醇单月桂酸酯)的50mm乙酸钠(ph5.0)中使用0.5mm对硝基苯基乙酸酯作为底物来确定乙酰木聚糖酯酶活性。将一个单位的乙酰木聚糖酯酶定义为在ph5、25℃每分钟能够释放1微摩尔对硝基酚根阴离子的酶的量。α-l-阿拉伯呋喃糖苷酶:术语“α-l-阿拉伯呋喃糖苷酶”意指α-l-阿拉伯呋喃糖苷阿拉伯呋喃水解酶(ec3.2.1.55),其催化α-l-阿拉伯糖苷中的末端非还原性α-l-阿拉伯呋喃糖苷残基的水解。该酶对α-l-阿拉伯呋喃糖苷、含有(1,3)-和/或(1,5)-键的α-l-阿拉伯聚糖、阿拉伯糖基木聚糖以及阿拉伯半乳聚糖起作用。α-l-阿拉伯呋喃糖苷酶还被称为阿拉伯糖苷酶、α-阿拉伯糖苷酶、α-l-阿拉伯糖苷酶、α-阿拉伯呋喃糖苷酶、多糖α-l-阿拉伯呋喃糖苷酶、α-l-阿拉伯呋喃糖苷水解酶、l-阿拉伯糖苷酶、或α-l-阿拉伯聚糖酶。可以使用每ml的100mm乙酸钠(ph5)中5mg的中等粘度小麦阿拉伯糖基木聚糖(麦格酶国际爱尔兰股份有限公司(megazymeinternationalireland,ltd.))以总体积200μl在40℃下持续30分钟,接着通过hpx-87h柱层析(伯乐实验室有限公司(bio-radlaboratories,inc.))进行阿拉伯糖分析来确定α-l-阿拉伯呋喃糖苷酶活性。α-葡糖醛酸糖苷酶:术语“α-葡糖醛酸糖苷酶”意指催化α-d-葡萄糖苷酸水解成为d-葡萄糖醛酸酯和醇的α-d-葡萄糖苷酸葡萄糖醛酸水解酶(ec3.2.1.139)。可以根据devries,1998,j.bacteriol.[细菌学杂志]180:243-249来确定α-葡糖醛酸糖苷酶活性。一个单位的α-葡糖醛酸糖苷酶等于能够在ph5、40℃每分钟释放1微摩尔的葡糖醛酸或4-o-甲基葡糖醛酸的酶的量。辅助活性9多肽:术语“辅助活性9多肽”或“aa9多肽”或“aa9溶解性多糖单加氧酶”意指分类为溶解性多糖单加氧酶(quinlan等人,2011,proc.natl.acad.sci.usa[美国国家科学院院刊]108:15079-15084;phillips等人,2011,acschem.biol.[acs化学生物学]6:1399-1406;li等人,2012,structure[结构]20:1051-1061)的多肽。根据henrissat,1991,biochem.j.[生物化学杂志]280:309-316以及henrissat和bairoch,1996,biochem.j.[生物化学杂志]316:695-696,aa9多肽之前被分类为糖苷水解酶家族61(gh61)。aa9多肽通过具有纤维素分解活性的酶增强纤维素材料的水解。可以通过测量在以下条件下来自由纤维素分解酶水解纤维素材料的还原糖的增加或纤维二糖与葡萄糖总量的增加来确定纤维素分解增强活性:1-50mg总蛋白/g预处理的玉米秸秆(pcs)中的纤维素,其中总蛋白包括50-99.5%w/w纤维素分解酶蛋白和0.5-50%w/waa9多肽蛋白,在适合的温度(如,40℃-80℃)和适合的ph(如,4-9)下持续1-7天,与不具有纤维素分解增强活性的相等的总蛋白负载的对照水解(1-50mg纤维素分解蛋白/gpcs中的纤维素)进行比较。可以使用celluclasttm1.5l(诺维信公司(novozymesa/s),巴格斯瓦德(bagsvaerd),丹麦)和β-葡糖苷酶的混合物作为纤维素分解活性的来源来确定aa9多肽增强活性,其中该β-葡糖苷酶是以纤维素酶蛋白负载的至少2%-5%蛋白质的重量存在的。在一方面,β-葡糖苷酶是米曲霉(aspergillusoryzae)β-葡糖苷酶(例如,根据wo02/095014,在米曲霉中重组产生的)。在另一方面,β-葡糖苷酶是烟曲霉(aspergillusfumigatus)β-葡糖苷酶(例如,如wo02/095014中所述的,在米曲霉中重组产生的)。aa9多肽增强活性还可通过以下来确定:在40℃将aa9多肽与0.5%磷酸溶胀纤维素(pasc)、100mm乙酸钠(ph5)、1mmmnso4、0.1%没食子酸、0.025mg/ml的烟曲霉β-葡糖苷酶、以及0.01%x-100(4-(1,1,3,3-四甲基丁基)苯基-聚乙二醇)一起孵育24-96小时,接着确定从pasc释放的葡萄糖。还可以根据wo2013/028928确定高温组合物的aa9多肽增强活性。aa9多肽通过将达到相同的水解程度所需要的纤维素分解酶的量降低优选至少1.01倍,例如,至少1.05倍、至少1.10倍、至少1.25倍、至少1.5倍、至少2倍、至少3倍、至少4倍、至少5倍、至少10倍、或至少20倍,来增强由具有纤维素分解活性的酶催化的纤维素材料的水解。根据wo2008/151043或wo2012/122518,aa9多肽可以在可溶性活化二价金属阳离子(例如锰或铜)的存在下使用。aa9多肽还可以在二氧化合物、二环化合物、杂环化合物、含氮化合物、醌化合物、含硫化合物、或从预处理的纤维素材料或半纤维素材料(例如预处理的玉米秸杆)获得的液体的存在下使用(wo2012/021394、wo2012/021395、wo2012/021396、wo2012/021399、wo2012/021400、wo2012/021401、wo2012/021408、以及wo2012/021410)。β-葡糖苷酶:术语“β-葡糖苷酶”意指β-d-葡糖苷葡糖水解酶(beta-d-glucosideglucohydrolase)(e.c.3.2.1.21),其催化末端非还原β-d-葡萄糖残基的水解,并释放β-d-葡萄糖。可以根据venturi等人,2002,j.basicmicrobiol.[基础微生物学杂志]42:55-66的程序使用对硝基苯基-β-d-吡喃葡萄糖苷作为底物来确定β-葡糖苷酶活性。一个单位的β-葡糖苷酶定义为在25℃,ph4.8下,在含有0.01%20的50mm柠檬酸钠中从作为底物的1mm对硝基苯基-β-d-吡喃葡萄糖苷每分钟产生1.0微摩尔的对硝基酚根阴离子。β-木糖苷酶:术语“β-木糖苷酶”意指β-d-木糖苷木糖水解酶(β-d-xylosidexylohydrolase)(e.c.3.2.1.37),其催化短β(1→4)-低聚木糖的外切水解,以将连续的d-木糖残基从非还原端移除。可以在含有0.01%20的100mm柠檬酸钠中,在ph5,40℃下,使用1mm对硝基苯基-β-d-木糖苷作为底物来确定β-木糖苷酶活性。一个单位的β-木糖苷酶定义为在40℃,ph5下,在含有0.01%20的100mm柠檬酸钠中从1mm对硝基苯基-β-d-木糖苷每分钟产生1.0微摩尔的对硝基酚根阴离子。过氧化氢酶:术语“过氧化氢酶”意指过氧化氢:过氧化氢氧化还原酶(e.c.1.11.1.6或e.c.1.11.1.21),其催化两个过氧化氢转化成氧和两个水。可以基于以下反应通过在240nm下监测过氧化氢的降解而确定过氧化氢酶活性:2h2o2→2h2o+o2在25℃,在具有10.3mm底物(h2o2)的50mm磷酸盐(ph7)中进行该反应。用分光光度计监测16-24秒内的吸光度,这应该对应于从0.45至0.4的吸光度降低。可以将一个过氧化氢酶活性单位表示为在ph7.0和25℃每分钟降解一微摩尔的h2o2。cdna:术语“cdna”意指可以通过从获得自真核或原核细胞的成熟的、剪接的,mrna分子进行反转录而制备的dna分子。cdna缺乏可以存在于对应基因组dna中的内含子序列。初始的初级rna转录物是mrna的前体,其要通过一系列的步骤(包括剪接)进行加工,然后呈现为成熟的剪接的mrna。cel-表型:术语“cel-表型”或“cel-”或“纤维素酶-阴性表型”意指当提供含有诱导纤维素酶的碳水化合物的碳源时不能产生任何纤维素酶蛋白的丝状真菌细胞。例如,展现cel-表型的丝状真菌细胞在含有纤维素底物的琼脂培养基上生长的丝状真菌菌落周围不产生消化的纤维素的晕圈(halo)或清除区。可替代地,当丝状真菌细胞在含有诱导纤维素酶的碳水化合物的液体培养基中生长时,展现cel-表型的丝状真菌细胞不分泌可测量的纤维素酶蛋白到培养基中。cel+表型:术语“cel+表型”或“cel+”或“纤维素酶-阳性表型”意指当提供含有诱导纤维素酶的碳水化合物的碳源时能产生任何纤维素酶蛋白的丝状真菌细胞。例如,展现cel+表型的丝状真菌细胞在含有纤维素底物的琼脂培养基上生长的丝状真菌菌落周围产生消化的纤维素的晕圈或清除区。可替代地,当丝状真菌细胞在含有诱导纤维素酶的碳水化合物的液体培养基中生长时,展现cel+表型的丝状真菌细胞将可测量的纤维素酶蛋白分泌到培养基中。在一方面,至少70%的菌落产生纤维素酶。在另一方面,至少75%的菌落产生纤维素酶。在另一方面,至少80%的菌落产生纤维素酶。在另一方面,至少90%的菌落产生纤维素酶。在另一方面,至少95%的菌落产生纤维素酶。纤维二糖水解酶:术语“纤维二糖水解酶”意指1,4-β-d-葡聚糖纤维二糖水解酶(e.c.3.2.1.91和e.c.3.2.1.176),其催化纤维素、纤维寡糖、或任何含β-1,4-连接的葡萄糖的聚合物中的1,4-β-d-糖苷键的水解,从该链的还原端(纤维二糖水解酶i)或非还原端(纤维二糖水解酶ii)释放纤维二糖(teeri,1997,trendsinbiotechnology[生物技术趋势]15:160-167;teeri等人,1998,biochem.soc.trans.[生化学会会刊]26:173-178)。可以根据由以下所述的程序来确定纤维二糖水解酶活性:lever等人,1972,anal.biochem.[分析生物化学]47:273-279;vantilbeurgh等人,1982,febsletters[欧洲生化学会联合会快报]149:152-156;vantilbeurgh和claeyssens,1985,febsletters[欧洲生化学会联合会快报]187:283-288;和tomme等人,1988,eur.j.biochem.[欧洲生物化学杂志],170:575-581。纤维素分解酶或纤维素酶:术语“纤维素分解酶”或“纤维素酶”意指一种或多种水解纤维素材料的酶。此类酶包括一种或多种内切葡聚糖酶、一种或多种纤维二糖水解酶、一种或多种β-葡糖苷酶、或其组合。用于测量纤维素分解酶活性的两种基本方法包括:(1)测量总纤维素分解酶活性,以及(2)测量个体纤维素分解酶活性(内切葡聚糖酶、纤维二糖水解酶和β-葡糖苷酶),如在zhang等人,2006,biotechnologyadvances[生物技术进展]24:452-481中所述的。可使用不溶性底物,包括沃特曼(whatman)№1滤纸、微晶纤维素、细菌纤维素、藻类纤维素、棉花、羧甲基纤维素、预处理的木质纤维素等,测量总纤维素分解酶活性。最常见的总纤维素分解活性测定是将沃特曼№1滤纸用作底物的滤纸测定。该测定是由国际纯粹与应用化学联合会(iupac)建立的(ghose,1987,pureappl.chem.[纯粹与应用化学]59:257-68)。可以通过测量在以下条件下,由一种或多种纤维素分解酶进行的纤维素材料水解期间,糖的产生/释放的增加来确定纤维素分解酶活性:1-50mg纤维素分解酶蛋白/g预处理的玉米秸秆(pcs)(或其他预处理的纤维素材料)中的纤维素,在适合的温度(如40℃-80℃)和适合的ph(如4-9)下持续3-7天,与未添加纤维素分解酶蛋白的对照水解相比。典型条件为:1ml反应,洗涤或未洗涤的pcs,5%不溶性固体(干重),50mm乙酸钠(ph5),0.1mmcucl2,50℃、55℃、或60℃,72小时,通过hpx-87h柱层析(伯乐实验室有限公司,赫拉克勒斯,加利福尼亚州,美国)进行糖分析。编码序列:术语“编码序列”意指直接指定多肽的氨基酸序列的多核苷酸。编码序列的边界通常由可读框确定,该可读框以起始密码子(例如atg、gtg或ttg)开始并且以终止密码子(例如taa、tag或tga)结束。编码序列可为基因组dna、cdna、合成dna或其组合。控制序列:术语“控制序列”意指对于多肽的编码序列的表达为必要的核酸序列。每个控制序列对于编码该多肽的多核苷酸来说可以是天然的(即,来自相同基因)或外源的(即,来自不同基因),或相对于彼此是天然的或外源的。此类控制序列包括但不限于前导序列、多腺苷酸化序列、前肽序列、启动子、信号肽序列、以及转录终止子。最少,控制序列包括启动子、以及转录和翻译终止信号。出于引入有利于将控制序列与编码多肽的多核苷酸的编码区连接的特异性限制性酶切位点的目的,这些控制序列可以提供有多个接头。缺陷:术语“缺陷”意指将编码本发明的转录因子的基因在亲本丝状真菌细胞中修饰以产生突变体,使得与未修饰转录因子基因的亲本丝状真菌细胞相比,当在相同条件下培养时,该突变体在转录因子的产生方面有部分缺陷(至少少25%、更优选地至少少50%、甚至更优选地至少少75%、和最优选地至少少95%的转录因子)或完全缺陷(少100%的转录因子)。可以使用本文所述的或本领域已知的方法确定由丝状真菌细胞、亲本或突变体产生的转录因子的水平。内切葡聚糖酶:术语“内切葡聚糖酶”意指4-(1,3;1,4)-β-d-葡聚糖4-葡聚糖水解酶(e.c.3.2.1.4),其催化纤维素、纤维素衍生物(如羧甲基纤维素和羟乙基纤维素)、地衣多糖中的1,4-β-d-糖苷键,混合β-1,3-1,4葡聚糖如谷类β-d-葡聚糖或木葡聚糖以及含有纤维素组分的其他植物材料中的β-1,4键的内切水解。可以通过测量底物粘度的降低或通过还原糖测定所确定的还原性末端的增加来确定内切葡聚糖酶活性(zhang等人,2006,biotechnologyadvances[生物技术进展]24:452-481)。还可以根据ghose,1987,pureandappl.chem.[纯粹与应用化学]59:257-268的程序,在ph5,40℃下,使用羧甲基纤维素(cmc)作为底物来确定内切葡聚糖酶活性。表达:术语“表达”包括涉及多肽产生的任何步骤,包括但不限于:转录、转录后修饰、翻译、翻译后修饰、以及分泌。表达载体:术语“表达载体”意指直链或环状dna分子,其包含编码多肽的多核苷酸并且可操作地连接至提供用于其表达的控制序列。阿魏酸酯酶:术语“阿魏酸酯酶”意指4-羟基-3-甲氧基肉桂酰基-糖水解酶(ec3.1.1.73),其催化4-羟基-3-甲氧基肉桂酰基(阿魏酰基)基团从酯化的糖(其在天然生物质底物中通常为阿拉伯糖)的水解,以产生阿魏酸酯(4-羟基-3-甲氧基肉桂酸酯)。阿魏酸酯酶(fae)也被称为阿魏酸酯酶(ferulicacidesterase)、羟基肉桂酰基酯酶、fae-iii、肉桂酸酯水解酶、faea、cinnae、fae-i、或fae-ii。可以在50mm乙酸钠(ph5.0)中,使用0.5mm对硝基苯基阿魏酸酯作为底物确定对阿魏酸酯酶活性。一个单位的阿魏酸酯酶等于,在ph5,25℃,每分钟能够释放1微摩尔的对硝基酚根阴离子的酶的量。片段:术语“片段”意指在转录因子的氨基和/或羧基末端缺失一个或多个氨基酸的转录因子,其中片段具有转录调节活性。在一方面,片段含有全长转录因子,例如,seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、或124的至少85%、至少90%、或至少95%的氨基酸残基。半纤维素分解酶或半纤维素酶:术语“半纤维素分解酶”或“半纤维素酶”意指可对半纤维素材料进行水解的一种或多种酶。参见例如,shallom和shoham,2003,currentopinioninmicrobiology[微生物学当前观点]6(3):219-228)。半纤维素酶是植物生物质降解中的关键组分。半纤维素酶的实例包括但不限于:乙酰甘露聚糖酯酶、乙酰木聚糖酯酶、阿拉伯聚糖酶、阿拉伯呋喃糖苷酶、香豆酸酯酶、阿魏酸酯酶、半乳糖苷酶、葡糖醛酸糖苷酶、葡糖醛酸酯酶、甘露聚糖酶、甘露糖苷酶、木聚糖酶、以及木糖苷酶。这些酶的底物(半纤维素)是支链和直链多糖的异质性组,这些多糖经由氢键与植物细胞壁中的纤维素微纤维相结合,从而将它们交联成稳健的网络。半纤维素还共价附接至木质素,从而与纤维素一起形成高度复杂的结构。半纤维素的可变结构和组织要求许多酶的协同作用以使其完全降解。半纤维素酶的催化模块是水解糖苷键的糖苷水解酶(gh),或是水解乙酸或阿魏酸侧基团的酯键的碳水化合物酯酶(ce)。这些催化模块基于其一级序列的同源性,可以分配到gh和ce家族。一些家族(具有总体上类似的折叠)可以进一步分组为宗族(clan),以字母标记(例如,gh-a)。在碳水化合物活性酶(cazy)数据库中可得到这些以及其他碳水化合物活性酶的最详实和最新的分类。半纤维素分解酶活性可根据ghose和bisaria,1987,pure&appi.chem.[理论与应用化学]59:1739-1752,在适合的温度(如40℃-80℃)和适合的ph(如4-9)下进行测量。宿主细胞:术语“宿主细胞”意指易于用包含编码多肽的多核苷酸的核酸构建体或表达载体转化、转染、转导等的任何丝状真菌细胞。术语“宿主细胞”涵盖由于复制期间出现的突变而与细胞不相同的任何细胞子代。在一方面,宿主细胞包含本发明的转录因子基因的修饰。提高的生产力:术语“提高的生产力”和其变型意指当在与未修饰的亲本丝状真菌细胞相同的培养基组成、温度、ph、细胞密度、溶解氧、和时间的条件下培养时,由具有本发明的转录因子基因的修饰的突变体丝状真菌细胞产生的目的多肽增加了至少1%、至少2%、至少3%、至少4%、至少5%、至少6%、至少7%、至少8%、至少9%、至少10%、至少11%、至少12%、至少13%、至少14%、至少15%、至少16%、至少17%、至少18%、至少19%、或至少20%。在一方面,与未修饰的亲本丝状真菌细胞相比,具有转录因子基因的修饰的突变体丝状真菌细胞在目的多肽的产生方面的生产力提高1%、2%、3%、4%、5%、6%、7%、8%、9%、10%、11%、12%、13%、14%、15%、16%、17%、18%、19%、或20%。分离的:术语“分离的”意指处于自然界中不存在的形式或环境中的物质。分离的物质的非限制性实例包括(1)任何非天然存在的物质,(2)包括但不限于任何酶、变体、核酸、蛋白质、或肽的任何物质,该物质至少部分地从与其性质相关的一种或多种或所有天然存在的成分中去除;(3)相对于自然界中发现的物质通过人工修饰的任何物质;或(4)通过相对于与其天然相关的其他组分增加该物质的量而修饰的任何物质(例如,在宿主细胞中的重组生产;编码该物质的基因的多个拷贝;以及使用比与编码该物质的基因天然相关联的启动子更强的启动子)。核酸构建体:术语“核酸构建体”意指单链或双链的核酸分子,该核酸分子是从天然存在的基因中分离的,或以原本不存在于自然界中的方式被修饰成包含核酸的区段,或者是合成的,该核酸分子包含一个或多个控制序列。可操作地连接:术语“可操作地连接”意指如下构型,在该构型中,控制序列被放置在相对于多核苷酸的编码序列适当的位置处,使得该控制序列指导该编码序列的表达。漆酶:术语“漆酶”意指催化以下反应的苯二醇:氧气氧化还原酶(e.c.1.10.3.2):1,2-或1,4-苯二醇+o2=1,2-或1,4-苯并半醌+2h2o。可以通过由漆酶将丁香醛连氮(4,4′-[连氮基双(甲基亚基)]双(2,6-二甲氧基苯酚))氧化为对应的醌4,4′-[偶氮双(甲基亚基)]双(2,6-二甲氧基环已-2,5-二烯-1-酮)来确定漆酶活性。通过在530nm下吸光度的增加来检测所述反应(如下所示)。在30℃,在具有19μm底物(丁香醛连氮)和1g/l聚乙二醇(peg)6000的23mmmes(ph5.5)中进行该反应并且在530nm下每15秒测量吸光度的变化,直至90秒。一个漆酶单位是在指定的分析条件下每分钟催化1微摩尔丁香醛连氮的转化的酶量。溶菌酶:术语“溶菌酶”意指催化在肽聚糖中的n-乙酰胞壁酸与n-乙酰-d-葡糖胺残基之间以及在壳糊精中的n-乙酰-d-葡糖胺残基之间的(1->4)-β-键的水解的肽聚糖n-乙酰基胞壁酰水解酶(acetylmuramoylhydrolase)(e.c.3.2.1.17)。溶菌酶活性可以根据实例11中所述的测定确定。过氧化物酶:术语“过氧化物酶”意指将过氧化物(例如,过氧化氢)转化为较少氧化的种类(例如,水)的酶。在此应该理解的是,过氧化物酶涵盖过氧化物分解酶。在此将术语“过氧化物分解酶”定义为供体:催化还原底物(2e-)+roor’→氧化底物+roh+r’oh反应的过氧化物氧化还原酶(e.c.编号1.11.1.x,其中x=1-3、5、7-19、或21);如催化苯酚+h2o2→醌+h2o反应的辣根过氧化物酶,和催化h2o2+h2o2→o2+2h2o反应的过氧化氢酶。除过氧化氢之外,其他过氧化物也可以被这些酶分解。在如以下所示的过氧化氢的存在下,可以通过测量由过氧化物酶氧化2,2’-连氮基-双(3-乙基苯噻唑啉-6-磺酸)abts)来确定过氧化物酶活性。反应产物abts氧化形成了在418nm下可以量化的蓝-绿颜色。h2o2+2abts还原+2h+→2h2o+2abts氧化在30℃处,在具有1.67mm底物(abts)、1.5g/lx-405、0.88mm过氧化氢、和大约0.040个单位的酶/ml的0.1m磷酸盐(ph7)中进行该反应并且在418nm下从15秒直至60秒测量吸光度的变化。一个过氧化物酶单位可以表示为在指定的分析条件下每分钟催化1微摩尔过氧化氢所需要酶的量。序列同一性:两个氨基酸序列之间或两个核苷酸序列之间的关联度通过参数“序列同一性”来描述。出于本发明的目的,使用如在emboss软件包(emboss:欧洲分子生物学开放软件包(emboss:theeuropeanmolecularbiologyopensoftwaresuite),rice等人,2000,trendsgenet.[遗传学趋势]16:276-277)(优选5.0.0版本或更新版本)的尼德尔程序中所实施的尼德曼-翁施算法(needleman-wunschalgorithm)(needleman和wunsch,1970,j.mol.biol.[分子生物学杂志]48:443-453)来确定两个氨基酸序列之间的序列同一性。使用的参数是空位开放罚分10、空位延伸罚分0.5以及eblosum62(blosum62的emboss版本)取代矩阵。使用尼德尔标记的“最长同一性”的输出(使用非简化选项获得)作为同一性百分比并且如下计算:(相同的残基x100)/(比对长度-比对中的空位总数)出于本发明的目的,使用如在emboss软件包(emboss:欧洲分子生物学开放软件包(emboss:theeuropeanmolecularbiologyopensoftwaresuite),rice等人,2000,同上)(优选5.0.0版本或更新版本)的尼德尔程序中所实施的尼德曼-翁施算法(needleman和wunsch,1970,同上)来确定两个脱氧核糖核苷酸序列之间的序列同一性。使用的参数是空位开放罚分10、空位延伸罚分0.5以及ednafull(ncbinuc4.4的emboss版本)取代矩阵。使用尼德尔标记的“最长同一性”的输出(使用非简化选项获得)作为同一性百分比并且如下计算:(相同的脱氧核糖核苷酸x100)/(比对长度-比对中的空位总数)严格条件:术语“非常低严格条件”意指对于长度为至少100个核苷酸的探针而言,遵循标准dna印迹程序,在42℃,于5xsspe,0.3%sds,200微克/ml剪切并变性的鲑鱼精子dna和25%甲酰胺中预杂交和杂交12至24小时。载体材料最终使用0.2xssc、0.2%sds,在45℃下洗涤三次,每次15分钟。术语“低严格条件”意指对于长度为至少100个核苷酸的探针而言,遵循标准dna印迹程序,在42℃,于5xsspe,0.3%sds,200微克/ml剪切并变性的鲑鱼精子dna和25%甲酰胺中预杂交和杂交12至24小时。载体材料最终使用0.2xssc、0.2%sds,在50℃下洗涤三次,每次15分钟。术语“中严格条件”意指对于长度为至少100个核苷酸的探针而言,遵循标准dna印迹程序,在42℃,于5xsspe,0.3%sds,200微克/ml剪切并变性的鲑鱼精子dna和35%甲酰胺中预杂交和杂交12至24小时。载体材料最终使用0.2xssc、0.2%sds,在55℃下洗涤三次,每次15分钟。术语“中-高严格条件”意指对于长度为至少100个核苷酸的探针而言,遵循标准dna印迹程序,在42℃,于5xsspe,0.3%sds,200微克/ml剪切并变性的鲑鱼精子dna和35%甲酰胺中预杂交和杂交12至24小时。载体材料最终使用0.2xssc、0.2%sds,在60℃下洗涤三次,每次15分钟。术语“高严格条件”意指对于长度为至少100个核苷酸的探针而言,遵循标准dna印迹程序,在42℃在5xsspe、0.3%sds、200微克/ml剪切并变性的鲑鱼精子dna和50%甲酰胺中预杂交和杂交12至24小时。载体材料最终使用0.2xssc、0.2%sds,在65℃下洗涤三次,每次15分钟。术语“非常高严格条件”意指对于长度为至少100个核苷酸的探针而言,遵循标准dna印迹程序,在42℃在5xsspe,0.3%sds,200微克/ml剪切并变性的鲑鱼精子dna和50%甲酰胺中预杂交和杂交12至24小时。载体材料最终使用0.2xssc、0.2%sds,在70℃下洗涤三次,每次15分钟。转录因子:术语“转录因子”意指具有转录调节活性的多肽。木聚糖酶:术语“木聚糖酶”意指1,4-β-d-木聚糖-木糖水解酶(1,4-β-d-xylan-xylohydrolase)(e.c.3.2.1.8),其催化木聚糖中1,4-β-d-木糖苷键的内水解。木聚糖酶活性可以在37℃在0.01%x-100和200mm磷酸钠(ph6)中用0.2%azcl-阿拉伯糖基木聚糖作为底物来确定。一个单位的木聚糖酶活性定义为在37℃,ph6,在200mm磷酸钠(ph6)中从作为底物的0.2%azcl-阿拉伯糖基木聚糖每分钟产生1.0微摩尔天青蛋白(azurine)。本文提及“约”值或参数包括针对该值或参数本身的方面。例如,提及“约x”的描述包括方面“x”。如本文和所附权利要求书中所用的,单数形式“一种/个(a/an)”、“或”以及“该(the)”包括复数指代物,除非上下文另外清楚表明。应理解的是,本文所述的本发明的这些方面包括“由方面组成”和/或“基本由方面组成”。除非另外定义或由上下文明确指示,否则本文所用的全部技术与科学术语具有如本发明所属领域的普通技术人员通常理解的相同含义。具体实施方式本发明涉及亲本丝状真菌细胞的分离的突变体,该分离的突变体包含目的多肽的在启动子的转录控制下的编码序列,该启动子通过选自由以下组成的组的一种或多种转录因子调节:(a)包含氨基酸序列的转录因子,该氨基酸序列与seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、或124具有至少70%序列同一性;(b)由多核苷酸编码的转录因子,该多核苷酸包含与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123具有至少70%序列同一性的核苷酸序列;以及(c)由多核苷酸编码的转录因子,该多核苷酸在高严格条件下与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123的全长互补序列杂交;其中将该一种或多种转录因子基因在亲本丝状真菌细胞中修饰以产生该突变体,使得该突变体在该一种或多种转录因子的产生方面有部分或完全缺陷,其中(i)当在与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相同的条件下培养时,该一种或多种转录因子基因的修饰提高了该突变体在该目的多肽的产生方面的生产力,(ii)与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相比,该一种或多种转录因子基因的修饰减少或消除了所得突变体中的纤维素酶阴性表型,或(iii)该一种或多种转录因子基因的修饰导致(i)和(ii)的组合。本发明还涉及用于构建亲本丝状真菌细胞的突变体的方法,该方法包括修饰各自编码该亲本丝状真菌细胞中的转录因子的一种或多种基因以产生该突变体,并且任选地回收该突变体,其中该亲本丝状真菌细胞或其突变体包含在启动子的转录控制下的目的多肽编码序列,该启动子由这些转录因子中的一种或多种调节,其中该一种或多种转录因子选自由以下组成的组:(a)包含氨基酸序列的转录因子,该氨基酸序列与seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、或124具有至少70%序列同一性;(b)由多核苷酸编码的转录因子,该多核苷酸包含与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123具有至少70%序列同一性的核苷酸序列;以及(c)由多核苷酸编码的转录因子,该多核苷酸在高严格条件下与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123的全长互补序列杂交;其中将该一种或多种转录因子基因在亲本丝状真菌细胞中修饰以产生该突变体,使得该突变体在该一种或多种转录因子的产生方面有部分或完全缺陷,其中(i)当在与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相同的条件下培养时,该一种或多种转录因子基因的修饰提高了该突变体在该目的多肽的产生方面的生产力,(ii)与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相比,该一种或多种转录因子基因的修饰减少或消除了所得突变体中的纤维素酶阴性表型,或(iii)该一种或多种转录因子基因的修饰导致(i)和(ii)的组合;。本发明的优点是亲本丝状真菌细胞中各自编码本发明的转录因子的一种或多种基因的修饰可以减少或消除所得突变体中的纤维素酶-阴性表型,当在与未修饰的亲本丝状真菌细胞相同的条件下培养时,其可以提高该突变体在该目的多肽的产生方面的生产力。在一方面,当在与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相同的条件下培养时,该一种或多种转录因子基因的修饰提高了该突变体在该目的多肽的产生方面的生产力。在另一方面,与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相比,该一种或多种转录因子基因的修饰减少或消除了所得突变体中的纤维素酶-阴性表型。在另一方面,一种或多种转录因子基因的修饰(i)提高了该突变体在该目的多肽的产生方面的生产力(当在与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相同的条件下培养时)并且(ii)减少或消除了所得突变体中的纤维素酶-阴性表型(与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相比)。转录因子在一方面,该转录因子与seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、或124具有至少70%,例如,至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%、或100%序列同一性。在一个实施例中,该转录因子与seqidno:2相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:4相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:6相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:8相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:10相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:12相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:14相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:16相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:18相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:20相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:22相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:24相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:26相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:28相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:30相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:32相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:34相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:36相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:38相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:40相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:42相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:44相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:46相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:48相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:50相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:52相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:54相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:56相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在一个实施例中,该转录因子与seqidno:124相差多达10个氨基酸,例如,1、2、3、4、5、6、7、8、9、或10个氨基酸。在另一个实施例中,该转录因子包含seqidno:2的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:2的氨基酸序列。在另一个实施例中,该转录因子由seqidno:2的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:4的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:4的氨基酸序列。在另一个实施例中,该转录因子由seqidno:4的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:6的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:6的氨基酸序列。在另一个实施例中,该转录因子由seqidno:6的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:8的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:8的氨基酸序列。在另一个实施例中,该转录因子由seqidno:8的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:10的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:10的氨基酸序列。在另一个实施例中,该转录因子由seqidno:10的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:12的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:12的氨基酸序列。在另一个实施例中,该转录因子由seqidno:12的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:14的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:14的氨基酸序列。在另一个实施例中,该转录因子由seqidno:14的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:16的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:16的氨基酸序列。在另一个实施例中,该转录因子由seqidno:16的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:18的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:18的氨基酸序列。在另一个实施例中,该转录因子由seqidno:18的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:20的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:20的氨基酸序列。在另一个实施例中,该转录因子由seqidno:20的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:22的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:22的氨基酸序列。在另一个实施例中,该转录因子由seqidno:22的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:24的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:24的氨基酸序列。在另一个实施例中,该转录因子由seqidno:24的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:26的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:26的氨基酸序列。在另一个实施例中,该转录因子由seqidno:26的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:28的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:28的氨基酸序列。在另一个实施例中,该转录因子由seqidno:28的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:30的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:30的氨基酸序列。在另一个实施例中,该转录因子由seqidno:30的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:32的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:32的氨基酸序列。在另一个实施例中,该转录因子由seqidno:32的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:34的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:34的氨基酸序列。在另一个实施例中,该转录因子由seqidno:34的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:36的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:36的氨基酸序列。在另一个实施例中,该转录因子由seqidno:36的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:38的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:38的氨基酸序列。在另一个实施例中,该转录因子由seqidno:38的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:40的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:40的氨基酸序列。在另一个实施例中,该转录因子由seqidno:40的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:42的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:42的氨基酸序列。在另一个实施例中,该转录因子由seqidno:42的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:44的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:44的氨基酸序列。在另一个实施例中,该转录因子由seqidno:44的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:46的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:46的氨基酸序列。在另一个实施例中,该转录因子由seqidno:46的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:48的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:48的氨基酸序列。在另一个实施例中,该转录因子由seqidno:48的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:50的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:50的氨基酸序列。在另一个实施例中,该转录因子由seqidno:50的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:52的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:52的氨基酸序列。在另一个实施例中,该转录因子由seqidno:52的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:54的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:54的氨基酸序列。在另一个实施例中,该转录因子由seqidno:54的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:56的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:56的氨基酸序列。在另一个实施例中,该转录因子由seqidno:56的氨基酸序列组成。在另一个实施例中,该转录因子包含seqidno:124的氨基酸序列或其等位基因变体。在另一个实施例中,该转录因子包含seqidno:124的氨基酸序列。在另一个实施例中,该转录因子由seqidno:124的氨基酸序列组成。在另一方面,该转录因子由多核苷酸编码,该多核苷酸与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123,或其cdna序列具有至少70%,例如,至少75%、至少80%、至少81%、至少82%、至少83%、至少84%、至少85%、至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%、或100%序列同一性。在一个实施例中,该转录因子由包含seqidno:1的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:1组成。在另一个实施例中,该转录因子由包含seqidno:3的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:3组成。在另一个实施例中,该转录因子由包含seqidno:5的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:5组成。在另一个实施例中,该转录因子由包含seqidno:7的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:7组成。在另一个实施例中,该转录因子由包含seqidno:9的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:9组成。在另一个实施例中,该转录因子由包含seqidno:11的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:11组成。在另一个实施例中,该转录因子由包含seqidno:13的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:13组成。在另一个实施例中,该转录因子由包含seqidno:15的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:15组成。在另一个实施例中,该转录因子由包含seqidno:17的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:17组成。在另一个实施例中,该转录因子由包含seqidno:19的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:19组成。在另一个实施例中,该转录因子由包含seqidno:21的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:21组成。在另一个实施例中,该转录因子由包含seqidno:23的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:23组成。在另一个实施例中,该转录因子由包含seqidno:25的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:25组成。在另一个实施例中,该转录因子由包含seqidno:27的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:27组成。在另一个实施例中,该转录因子由包含seqidno:29的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:29组成。在另一个实施例中,该转录因子由包含seqidno:31的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:31组成。在另一个实施例中,该转录因子由包含seqidno:33的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:33组成。在另一个实施例中,该转录因子由包含seqidno:35的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:35组成。在另一个实施例中,该转录因子由包含seqidno:37的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:37组成。在另一个实施例中,该转录因子由包含seqidno:39的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:39组成。在另一个实施例中,该转录因子由包含seqidno:41的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:41组成。在另一个实施例中,该转录因子由包含seqidno:43的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:43组成。在另一个实施例中,该转录因子由包含seqidno:45的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:45组成。在另一个实施例中,该转录因子由包含seqidno:47的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:47组成。在另一个实施例中,该转录因子由包含seqidno:49的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:49组成。在另一个实施例中,该转录因子由包含seqidno:51的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:51组成。在另一个实施例中,该转录因子由包含seqidno:53的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:53组成。在另一个实施例中,该转录因子由包含seqidno:55的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:55组成。在另一个实施例中,该转录因子由包含seqidno:123的多核苷酸编码。在另一个实施例中,该转录因子由多核苷酸编码,该多核苷酸由seqidno:123组成。在另一方面,该转录因子由多核苷酸编码,该多核苷酸在非常低严格条件下、低严格条件下、中严格条件下、中-高严格条件下、高严格条件下、或非常高严格条件下与(i)seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123,(ii)其cdna序列,或(iii)(i)或(ii)的全长互补序列杂交(sambrook等人,1989,molecularcloning,alaboratorymanual[分子克隆,实验室手册]第2版,冷泉港,纽约)。可以将seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123的多核苷酸,或其子序列,以及seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、或124的多肽,或其片段用于设计核酸探针以根据本领域熟知的方法来鉴定并克隆dna,所述dna编码的来自不同属或种的菌株的转录因子。特别地,可以遵循标准dna印迹程序,使用此类探针来与目的细胞的基因组dna或cdna杂交,以便鉴定和分离其中的相应基因。此类探针可明显短于完整序列,但是长度应为至少15个,例如至少25个、至少35个、或至少70个核苷酸。优选地,核酸探针长度为至少100个核苷酸,例如长度为至少200个核苷酸、至少300个核苷酸、至少400个核苷酸、至少500个核苷酸、至少600个核苷酸、至少700个核苷酸、至少800个核苷酸、或至少900个核苷酸。dna和rna探针两者都可使用。典型地将探针进行标记(例如,用32p、3h、35s、生物素、或抗生物素蛋白),用于检测相应基因。此类探针涵盖于本发明中。可以针对与上文所述的探针杂交并编码转录因子的dna来筛选由此类其他菌株制备的基因组dna或cdna文库。来自此类其他菌株的基因组dna或其他dna可通过琼脂糖或聚丙烯酰胺凝胶电泳或其他分离技术来分离。可将来自文库的dna或分离的dna转移到并固定在硝化纤维素或其他适合的运载体材料上。为了鉴定与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123或其子序列杂交的克隆或dna,将运载体材料用于dna印迹中。出于本发明的目的,杂交表明多核苷酸杂交到对应于以下的经标记的核酸探针上:(i)seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123;(ii)其cdna序列,(iii)其全长互补序列;或(iv)其子序列;杂交是在非常低的至非常高的严格条件下进行。可以使用例如x-射线胶片或本领域已知的任何其他检测手段来检测在这些条件下核酸探针杂交的分子。在一个实施例中,该核酸探针是seqidno:1或其cdna序列。在另一个实施例中,该核酸探针是seqidno:3或其cdna序列。在另一个实施例中,该核酸探针是seqidno:5或其cdna序列。在另一个实施例中,该核酸探针是seqidno:7或其cdna序列。在另一个实施例中,该核酸探针是seqidno:9或其cdna序列。在另一个实施例中,该核酸探针是seqidno:11或其cdna序列。在另一个实施例中,该核酸探针是seqidno:13或其cdna序列。在另一个实施例中,该核酸探针是seqidno:15或其cdna序列。在另一个实施例中,该核酸探针是seqidno:17或其cdna序列。在另一个实施例中,该核酸探针是seqidno:19或其cdna序列。在另一个实施例中,该核酸探针是seqidno:21或其cdna序列。在另一个实施例中,该核酸探针是seqidno:23或其cdna序列。在另一个实施例中,该核酸探针是seqidno:25或其cdna序列。在另一个实施例中,该核酸探针是seqidno:27或其cdna序列。在另一个实施例中,该核酸探针是seqidno:29或其cdna序列。在另一个实施例中,该核酸探针是seqidno:31或其cdna序列。在另一个实施例中,该核酸探针是seqidno:33或其cdna序列。在另一个实施例中,该核酸探针是seqidno:35或其cdna序列。在另一个实施例中,该核酸探针是seqidno:37或其cdna序列。在另一个实施例中,该核酸探针是seqidno:39或其cdna序列。在另一个实施例中,该核酸探针是seqidno:41或其cdna序列。在另一个实施例中,该核酸探针是seqidno:43或其cdna序列。在另一个实施例中,该核酸探针是seqidno:45或其cdna序列。在另一个实施例中,该核酸探针是seqidno:47或其cdna序列。在另一个实施例中,该核酸探针是seqidno:49或其cdna序列。在另一个实施例中,该核酸探针是seqidno:51或其cdna序列。在另一个实施例中,该核酸探针是seqidno:53或其cdna序列。在另一个实施例中,该核酸探针是seqidno:55或其cdna序列。在另一个实施例中,该核酸探针是seqidno:123或其cdna序列。在另一个实施例中,该核酸探针是编码seqidno:2的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:4的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:6的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:8的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:10的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:12的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:14的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:16的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:18的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:20的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:22的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:24的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:26的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:28的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:30的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:32的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:34的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:36的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:38的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:40的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:42的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:44的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:46的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:48的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:50的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:52的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:54的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:56的转录因子或其片段的多核苷酸。在另一个实施例中,该核酸探针是编码seqidno:124的转录因子或其片段的多核苷酸。亲本丝状真菌细胞在本发明中,该亲本丝状真菌细胞可以是任何丝状真菌细胞。该丝状真菌细胞可以是野生型细胞或其突变体。“丝状真菌”包括真菌门(eumycota)和卵菌门(oomycota)的亚门的所有丝状形式(如由hawksworth等人,1995(同上)所定义的)。丝状真菌通常的特征在于由几丁质、纤维素、葡聚糖、壳聚糖、甘露聚糖和其他复杂多糖构成的菌丝体壁。营养生长是通过菌丝延伸来进行的,而碳分解代谢是专性需氧的。在一方面,该亲本丝状真菌细胞是枝顶孢霉属(acremonium)、伞菌属(agaricus)、链格孢属(alternaria)、曲霉属、短梗霉属(aureobasidium)、葡萄座腔菌属(botryospaeria)、拟蜡菌属(ceriporiopsis)、毛喙壳属(chaetomidium)、金孢子菌属(chrysosporium)、麦角菌属(claviceps)、旋孢腔菌属(cochliobolus)、鬼伞属(coprinopsis)、乳白蚁属(coptotermes)、棒囊壳属(corynascus)、隐丛赤壳菌属(cryphonectria)、隐球菌属(cryptococcus)、色二孢属(diplodia)、黑耳属(exidia)、线黑粉酵母属(filibasidium)、镰孢属(fusarium)、赤霉属(gibberella)、全鞭毛虫属(holomastigotoides)、腐质霉属(humicola)、耙齿菌属(irpex)、香菇属(lentinula)、小腔球菌属(leptospaeria)、梨孢菌属(magnaporthe)、黑果菌属(melanocarpus)、亚灰树花菌属、毛霉属(mucor)、毁丝霉属(myceliophthora)、新美鞭菌属(neocallimastix)、链孢菌属(neurospora)、拟青霉属(paecilomyces)、青霉菌属、平革菌属(phanerochaete)、瘤胃壶菌属(piromyces)、poitrasia、假黑盘菌属(pseudoplectania)、假披发虫属(pseudotrichonympha)、根毛霉属(rhizomucor)、裂褶菌属(schizophyllum)、柱顶孢属(scytalidium)、篮状菌属(talaromyces)、嗜热子囊菌属(thermoascus)、梭孢壳霉属(thielavia)、弯颈霉属(tolypocladium)、木霉属(trichoderma)、长毛盘菌属(trichophaea)、轮枝孢属(verticillium)、小包脚菇属(volvariella)、或炭角菌属(xylaria)细胞。在实施例中,该亲本丝状真菌细胞是解纤维枝顶孢霉(acremoniumcellulolyticus)、棘孢曲霉(aspergillusaculeatus)、泡盛曲霉(aspergillusawamori)、臭曲霉(aspergillusfoetidus)、烟曲霉(aspergillusfumigatus)、日本曲霉(aspergillusjaponicus)、构巢曲霉(aspergillusnidulans)、黑曲霉(aspergillusniger)、米曲霉(aspergillusoryzae)、狭边金孢子菌(chrysosporiuminops)、嗜角质金孢子菌(chrysosporiumkeratinophilum)、拉克淖金孢子菌(chrysosporiumlucknowense)、粪状金孢子菌(chrysosporiummerdarium)、毡金孢子菌(chrysosporiumpannicola)、昆士兰金孢子菌(chrysosporiumqueenslandicum)、热带金孢子菌(chrysosporiumtropicum)、带纹金孢子菌(chrysosporiumzonatum)、杆孢状镰孢(fusariumbactridioides)、禾谷镰孢(fusariumcerealis)、库威镰孢(fusariumcrookwellense)、黄色镰孢菌(fusariumculmorum)、禾谷镰孢菌(fusariumgraminearum)、禾赤镰孢(fusariumgraminum)、异孢镰孢(fusariumheterosporum)、合欢木镰孢(fusariumnegundi)、尖孢镰孢菌(fusariumoxysporum)、多枝镰孢(fusariumreticulatum)、粉红镰孢菌(fusariumroseum)、接骨木镰孢(fusariumsambucinum)、肤色镰孢(fusariumsarcochroum)、拟枝孢镰孢菌(fusariumsporotrichioides)、硫色镰孢菌(fusariumsulphureum)、圆镰孢(fusariumtorulosum)、拟丝孢镰孢菌(fusariumtrichothecioides)、镶片镰孢菌(fusariumvenenatum)、灰腐质霉(humicolagrisea)、特异腐质霉(humicolainsolens)、疏棉状腐质霉(humicolalanuginosa)、白囊耙齿菌(irpexlacteus)、米黑毛霉(mucormiehei)、嗜热毁丝霉(myceliophthorathermophila)、粗糙脉孢菌(neurosporacrassa)、绳状青霉菌(penicilliumfuniculosum)、产紫青霉(penicilliumpurpurogenum)、黄孢原毛平革菌(phanerochaetechrysosporium)、埃默森篮状菌(talaromycesemersonii)、无色梭孢壳(thielaviaachromatica)、成层梭孢壳菌(thielaviaalbomyces)、白毛梭孢壳(thielaviaalbopilosa)、澳洲梭孢壳(thielaviaaustraleinsis)、粪梭孢壳(thielaviafimeti)、小孢梭孢壳(thielaviamicrospora)、卵孢梭孢壳(thielaviaovispora)、秘鲁梭孢壳(thielaviaperuviana)、毛梭孢壳(thielaviasetosa)、瘤孢梭孢壳(thielaviaspededonium)、耐热梭孢壳(thielaviasubthermophila)、土生梭孢壳(thielaviaterrestris)、哈茨木霉(trichodermaharzianum)、康宁木霉(trichodermakoningii)、长枝木霉(trichodermalongibrachiatum)、里氏木霉(trichodermareesei)、或绿色木霉(trichodermaviride)细胞。在另一个实施例中,该亲本丝状真菌细胞是嗜热毁丝霉细胞。在另一个实施例中,该亲本丝状真菌细胞是埃默森篮状菌细胞。在另一个实施例中,该亲本丝状真菌细胞是哈茨木霉细胞。在另一个实施例中,该亲本丝状真菌细胞是康宁木霉细胞。在另一个实施例中,该亲本丝状真菌细胞是长枝木霉细胞。在另一个实施例中,该亲本丝状真菌细胞是里氏木霉细胞。在另一个实施例中,该亲本丝状真菌细胞是绿色木霉细胞。在优选的实施例中,该亲本里氏木霉细胞是里氏木霉rut-c30。在另一个优选的实施例中,该亲本里氏木霉细胞是里氏木霉的突变体。在另一个优选的实施例中,该亲本里氏木霉细胞是里氏木霉的形态突变体(参见wo97/26330)。在另一个优选的实施例中,该亲本里氏木霉细胞是里氏木霉的蛋白酶-缺陷突变体(参见wo2011/075677)。应理解的是,对于前述物种,本发明涵盖完全和不完全阶段(perfectandimperfectstates),和其他分类学等同物(equivalent),例如无性型,而与它们已知的种名无关。本领域的技术人员会容易地识别适当等同物的身份。这些物种的菌株可容易地在许多培养物保藏中心为公众所获得,例如美国典型培养物保藏中心(atcc)、德国微生物菌种保藏中心(deutschesammlungvonmikroorganismenundzellkulturengmbh,dsmz)、荷兰菌种保藏中心(centraalbureauvoorschimmelcultures,cbs)和美国农业研究服务专利培养物保藏中心北方地区研究中心(agriculturalresearchservicepatentculturecollection,northernregionalresearchcenter,nrrl)。可通过使用本领域熟知的方法减少或消除编码转录因子的基因的表达来构建本发明的转录因子缺陷的突变体丝状真菌细胞。可以修饰该基因的一部分,如编码区或为编码区的表达所需的控制序列。该基因的此类控制序列可以是启动子序列或其功能部分,即足以影响该基因的表达的部分。例如,可以将启动子序列失活从而无表达或可以将天然启动子替换为更弱的启动子以减少编码序列的表达。可以通过基因缺失技术来构建突变体丝状真菌细胞,以消除或减少这些基因的表达。基因缺失技术使得可以部分或完全除去该基因,从而消除其表达。这类方法中,使用已经构建为邻接地含有侧翼于该基因的5'和3'区的质粒,通过同源重组完成该基因的缺失。也可以通过任何rna指导的dna内切核酸酶使用,例如,mad7(美国专利9,982,279)、mad2(美国专利9,982,279)、cas9(doudna等人,2014,science[科学]346:1258096)、“死”cas9(dcas9;qi等人,2013,cell[细胞]152(5):1173)、cas9切口酶(satomura等人2017,sci.rep.[科学报告]7(1):2095)、或cpf1内切核酸酶(zetsche等人2015,cell[细胞]163(3):759)(通过适当地设计的指导rna被引导至基因的核苷酸序列)来构建突变体丝状真菌细胞。还可以通过引入、取代和/或缺失该基因或为其转录或翻译所需的其控制序列中的一个或多个(例如若干个)核苷酸来构建突变体丝状真菌细胞。例如,可以插入或除去核苷酸,用于引入终止密码子、除去起始密码子或移码开放阅读框。可以根据本领域已知的方法,通过定点诱变或pcr产生的诱变完成这样的修饰。参见,例如botstein和shortle,1985,science[科学]229:4719;lo等人,1985,proceedingsofthenationalacademyofsciencesusa[美国国家科学院院刊]81:2285;higuchi等人,1988,nucleicacidsresearch[核酸研究]16:7351;shimada,1996,meth.mol.biol.[分子生物学方法]57:157;ho等人,1989,gene[基因]77:61;horton等人,1989,gene[基因]77:61;以及sarkar和sommer,1990,biotechniques[生物技术]8:404。还可以通过基因破坏技术,通过将破坏性核酸构建体插入进该基因中而构建突变体丝状真菌细胞,该破坏性核酸构建体包括与该基因同源的核酸片段,该片段将产生具有同源性的区域的重复并且在重复的区域之间掺入构建体dna。这样的一种基因破坏可以消除基因表达,如果插入的构建体将该基因的启动子与编码区分离或打断编码序列,这样使得产生无功能性基因产物。破坏构建体可以简单地是伴有与该基因同源的5’和3’区的选择性标记基因。该选择性标记使得可以鉴定含有破坏的基因的转化株。还可以通过基因转化过程构建突变体丝状真菌细胞(参见例如,iglesias和trautner,1983,moleculargeneralgenetics[分子普通遗传学]189:73-76)。例如,在基因转化方法中,将对应于该基因的核苷酸序列体外诱变,以产生缺陷核苷酸序列,然后将其转化进丝状真菌细胞中以产生缺陷基因。通过同源重组,该缺陷核苷酸序列替换该内源基因。该缺陷核苷酸序列还包含用于选择含有缺陷基因的转化株的标记可以是令人希望的。还可以使用与该基因的核苷酸序列互补的核苷酸序列,通过已建立的反义技术构建突变体丝状真菌细胞(parish和stoker,1997,femsmicrobiologyletters[fems微生物学快报]154:151-157)。更确切而言,可以通过引入与该基因的核苷酸序列互补的核苷酸序列来减少或失活该基因的表达,该核苷酸序列可以在该细胞中转录并且能够与在该细胞中产生的mrna杂交。由此在允许所述互补反义核苷酸序列与mrna杂交的条件下,减少或消除翻译的蛋白质的量。还可以通过已确立的rna干扰(rnai)技术来构建突变体丝状真菌细胞(参见,例如wo2005/056772和wo2008/080017)。可以使用本领域熟知的方法(包括但不限于化学诱变),通过随机或特异诱变进一步构建突变体丝状真菌细胞(参见,例如hopwood,theisolationofmutantsinmethodsinmicrobiology[微生物学方法中的突变体分离](j.r.norris和d.w.ribbons,编辑)第363-433页,academicpress[学术出版社],纽约,1970)。可以通过使亲本细胞经受诱变并筛选其中该基因的表达已经被减少或失活的突变细胞来修饰该基因。诱变可以是特异的或随机的,例如通过使用适合的物理或化学诱变剂、使用适合的寡核苷酸或使dna序列经受pcr产生的诱变来进行。此外,诱变可以通过使用这些诱变方法的任何组合来进行。适合本发明目的的物理或化学诱变剂的实例包括紫外线(uv)照射,羟胺,n-甲基-n'-硝基-n-亚硝基胍(mnng),n-甲基-n'-亚硝基胍(ntg)邻甲基羟胺,亚硝酸,乙基甲磺酸(ems),亚硫酸氢钠,甲酸和核苷酸类似物。当使用此类试剂时,诱变典型地是在适合条件下在所选的诱变剂的存在下通过孵育有待诱变的亲本细胞并选择展现出该基因的减少表达或无表达的突变体来进行的。编码本发明的转录因子的基因的表达修饰可由本领域普通技术人员通过熟知的方式,例如,定量实时pcr(qrt-pcr)、dna印迹杂交、使用cdna或寡核苷酸阵列杂交(oligoarrayhybridization)的全局基因表达谱(globalgeneexpressionprofiling)、或深度rna测序(rna-seq)分析选择的mrna或转录水平来测量。可替代地,编码本发明的转录因子的基因的修饰可以通过使用如实例9中所述的基因座特异性引物的真菌孢子pcr来确定。在一方面,当在相同条件下培养时,与未修饰的亲本丝状真菌细胞相比,突变体在转录因子的产生方面有部分缺陷。在优选的方面,当在相同条件下培养时,该突变体产生比未修饰的亲本丝状真菌细胞至少少25%、更优选地至少少50%、甚至更优选地至少少75%、和最优选地至少少95%的转录因子。在另一方面,当在相同条件下培养时,与未修饰的亲本丝状真菌细胞相比,突变体在转录因子的产生方面有完全缺陷。换句话说,编码转录因子的基因失活(例如基因的缺失、破坏等)。目的多肽目的多肽可以是任何对于突变体丝状真菌细胞(其编码序列处于由本发明的转录因子调节的启动子的转录控制之下)来说是天然或外源(异源)的多肽。该启动子对于该目的多肽的编码序列可以是天然的或异源的。该多肽可以由单个基因或两个或更多个基因编码。术语“异源多肽”在本文定义为对宿主细胞不是天然的多肽;已进行结构修饰以改变天然多肽(例如,天然多肽的蛋白质序列)的天然多肽;或由于通过重组dna技术(例如,不同的启动子、编码多肽的dna的多拷贝)操纵多核苷酸或宿主细胞使其表达发生数量上变化的天然多肽。因此,本发明术语“异源多肽”的范围还涵盖了天然多肽的重组产生,其程度使得此种表达涉及使用对于丝状真菌细胞并非天然的遗传元件,或使用经操纵以丝状真菌细胞中非天然发生的方式起作用的天然元件。在一方面,该多肽对于丝状真菌细胞是天然的。在另一方面,该多肽对于丝状真菌细胞是异源的。该多肽可以是具有目的生物活性的任何多肽。术语“多肽”在本文中不意在是指特定长度的编码产物,并且因此,涵盖肽、寡肽和蛋白。术语“多肽”还涵盖组合以形成编码产物的两个或更多个多肽。多肽还包括融合多肽,其包含自至少两种不同多肽(其中一个或多个多肽对于丝状真菌细胞可以是异源的)获得的部分或完整多肽序列的组合。多肽进一步包括杂合多肽,这些杂合多肽包含来自两个或更多个多肽的结构域,例如,来自一个多肽的结合结构域和来自另一个多肽的催化结构域。结构域可以在n-末端或c-末端融合。在一方面,该多肽是抗体、抗原、抗微生物肽、酶、生长因子、激素、免疫调节剂(immunomodulator)、神经递质、受体、报告蛋白、结构蛋白或转录因子。在另一个方面中,该多肽是一种氧化还原酶、转移酶、水解酶、裂解酶、异构酶或连接酶。在另一方面,该多肽是乙酰甘露聚糖酯酶、乙酰木聚糖酯酶、氨肽酶、α-淀粉酶、阿拉伯聚糖酶、阿拉伯呋喃糖苷酶、糖酶、羧肽酶、过氧化氢酶、纤维二糖水解酶、纤维素酶、壳多糖酶、香豆酸酯酶、环糊精糖基转移酶、角质酶、环糊精糖基转移酶、脱酰胺酶、脱氧核糖核酸酶、分散素(dispersin)、内切葡聚糖酶、酯酶、阿魏酸酯酶、aa9溶解性多糖单加氧酶、α-半乳糖苷酶、β-半乳糖苷酶、葡糖脑苷脂酶、葡糖氧化酶、α-葡糖苷酶、β-葡糖苷酶、葡糖醛酸糖苷酶、葡萄糖醛酸酯酶、卤素过氧化物酶、半纤维素酶、转化酶、异构酶、漆酶、连接酶、脂肪酶、溶菌酶、甘露聚糖酶、甘露糖苷酶、变聚糖酶、氧化酶、果胶分解酶、过氧化物酶、磷酸二酯酶、磷脂酶、植酸酶、酚氧化酶、多酚氧化酶、蛋白水解酶、核糖核酸酶、α-1,6-转葡糖苷酶、转谷氨酰胺酶、尿激酶、黄原胶酶、木聚糖酶或β-木糖苷酶。在另一方面,该多肽是纤维素酶。在另一方面,该纤维素酶选自由以下组成的组:内切葡聚糖酶、纤维二糖水解酶、和β-葡糖苷酶。在另一方面,该多肽是半纤维素酶。在另一方面,该半纤维素酶选自由以下组成的组:木聚糖酶、乙酰木聚糖酯酶、阿魏酸酯酶、阿拉伯呋喃糖苷酶、木糖苷酶、以及葡糖醛酸糖苷酶。在另一个方面中,该多肽是一种内切葡聚糖酶。在另一个方面中,该多肽是一种纤维素二糖水解酶。在另一方面,该多肽是β-葡糖苷酶。在另一方面,该多肽是aa9溶解性多糖单加氧酶。在另一个方面中,该多肽是木聚糖酶。在另一方面,该多肽是β-木糖苷酶。在另一方面,该多肽是乙酰木聚糖酯酶。在另一方面,该多肽是阿魏酸酯酶。在另一方面,该多肽是阿拉伯呋喃糖苷酶。在另一方面,该多肽是葡糖醛酸糖苷酶。在另一个方面中,该多肽是乙酰甘露聚糖酯酶。在另一个方面中,该多肽是阿拉伯聚糖酶。在另一个方面中,该多肽是香豆酸酯酶。在另一个方面中,该多肽是半乳糖苷酶。在另一个方面中,该多肽是葡萄糖醛酸酯酶。在另一个方面中,该多肽是甘露聚糖酶。在另一个方面中,该多肽是甘露糖苷酶。在本发明的方法中,突变体丝状真菌细胞是重组细胞,其包括编码异源多肽的多核苷酸,该突变体有利地用于多肽的重组产生。该细胞优选用包含编码异源多肽的多核苷酸的核酸构建体或表达载体转化,随后将该载体整合入染色体。“转化”意指将包括多核苷酸的载体引入进宿主细胞中,这样使得该载体被保持为染色体整合体或保持为自主复制的染色体外载体。通常认为整合是有利的,因为多核苷酸更有可能稳定地保持在细胞中。载体整合入染色体可以通过同源重组、非同源重组或转座发生。编码异源多肽的多核苷酸可以从任何原核、真核或其他来源例如古细菌(archaeabacteria)获得。出于本发明的目的,如在此与给定来源结合使用的术语“从……中获得”应意指该多肽由该来源或有其中已经插入来自该来源的基因的细胞产生。用来分离或克隆编码感兴趣的多肽的多核苷酸的技术在本领域中是已知的,并且包括从基因组dna分离、从cdna制备或其组合。可以例如通过使用熟知的聚合酶链式反应(pcr)实现从此类基因组dna中克隆此类多核苷酸。参见例如,innis等人,1990,pcrprotocols:aguidetomethodsandapplication[pcr:方法和应用指南],academicpress[学术出版社],纽约。克隆程序可涉及将包括编码多肽的多核苷酸的所希望的核酸片段切除并分离、将片段插入载体分子、以及将重组载体并入本发明的突变体丝状真菌细胞,其中会复制多核苷酸的多重拷贝或克隆。多核苷酸可以是基因组、cdna、rna、半合成、合成来源的,或其任何组合。可以按多种方式操纵编码异源多肽的分离的多核苷酸以提供该多肽在本发明的突变体丝状真菌细胞中的表达。依赖于表达载体,在将多核苷酸的序列插入载体之前对其进行操作可能是理想的或必需的。使用重组dna方法修饰多核苷酸序列的技术是本领域熟知的。可以将包括编码多肽的多核苷酸的核酸构建体可操作地连接至一个或多个控制序列,这些控制序列能够在与这些控制序列相容的条件下,指导编码序列在本发明的突变体丝状真菌细胞中的表达。控制序列可以是由本发明的转录因子调节的合适的启动子序列。启动子序列包括介导该多肽的表达的转录控制序列。启动子可以是在丝状真菌细胞中示出转录活性的任何核苷酸序列,包括突变型、截短型和杂合型启动子,并且可以是从编码与丝状真菌细胞天然或异源(外源)的细胞外或细胞内多肽的基因获得。在一方面,启动子是来自由本发明的转录因子调节的纤维素酶基因的启动子。在另一方面,启动子是来自由本发明的转录因子调节的半纤维素酶基因的启动子。纤维素酶基因可以是内切葡聚糖酶、纤维二糖水解酶或β-葡糖苷酶基因。半纤维素酶基因可以是乙酰甘露聚糖酯酶、乙酰木聚糖酯酶、阿拉伯聚糖酶、阿拉伯呋喃糖苷酶、香豆酸酯酶、阿魏酸酯酶、半乳糖苷酶、葡糖醛酸糖苷酶、葡糖醛酸酯酶、甘露聚糖酶、甘露糖苷酶、木聚糖酶、或木糖苷酶基因。在一方面,纤维素酶基因或半纤维素酶基因可以获得自:枝顶孢霉属(acremonium)、伞菌属(agaricus)、链格孢属(alternaria)、曲霉属、短梗霉属(aureobasidium)、葡萄座腔菌属(botryospaeria)、拟蜡菌属(ceriporiopsis)、毛喙壳属(chaetomidium)、金孢子菌属(chrysosporium)、麦角菌属(claviceps)、旋孢腔菌属(cochliobolus)、鬼伞属(coprinopsis)、乳白蚁属(coptotermes)、棒囊壳属(corynascus)、隐丛赤壳菌属(cryphonectria)、隐球菌属(cryptococcus)、色二孢属(diplodia)、黑耳属(exidia)、线黑粉酵母属(filibasidium)、镰孢属(fusarium)、赤霉属(gibberella)、全鞭毛虫属(holomastigotoides)、腐质霉属(humicola)、耙齿菌属(irpex)、香菇属(lentinula)、小腔球菌属(leptospaeria)、梨孢菌属(magnaporthe)、黑果菌属(melanocarpus)、亚灰树花菌属、毛霉属(mucor)、毁丝霉属(myceliophthora)、新美鞭菌属(neocallimastix)、链孢菌属(neurospora)、拟青霉属(paecilomyces)、青霉菌属、平革菌属(phanerochaete)、瘤胃壶菌属(piromyces)、poitrasia、假黑盘菌属(pseudoplectania)、假披发虫属(pseudotrichonympha)、根毛霉属(rhizomucor)、裂褶菌属(schizophyllum)、柱顶孢属(scytalidium)、篮状菌属(talaromyces)、嗜热子囊菌属(thermoascus)、梭孢壳霉属(thielavia)、弯颈霉属(tolypocladium)、木霉属(trichoderma)、长毛盘菌属(trichophaea)、轮枝孢属(verticillium)、小包脚菇属(volvariella)、或炭角菌属(xylaria)细胞。在另一方面,纤维素酶基因或半纤维素酶基因可以获得自:解纤维枝顶孢霉(acremoniumcellulolyticus)、棘孢曲霉(aspergillusaculeatus)、泡盛曲霉(aspergillusawamori)、臭曲霉(aspergillusfoetidus)、烟曲霉(aspergillusfumigatus)、日本曲霉(aspergillusjaponicus)、构巢曲霉(aspergillusnidulans)、黑曲霉(aspergillusniger)、米曲霉(aspergillusoryzae)、狭边金孢子菌(chrysosporiuminops)、嗜角质金孢子菌(chrysosporiumkeratinophilum)、拉克淖金孢子菌(chrysosporiumlucknowense)、粪状金孢子菌(chrysosporiummerdarium)、毡金孢子菌(chrysosporiumpannicola)、昆士兰金孢子菌(chrysosporiumqueenslandicum)、热带金孢子菌(chrysosporiumtropicum)、带纹金孢子菌(chrysosporiumzonatum)、杆孢状镰孢(fusariumbactridioides)、禾谷镰孢(fusariumcerealis)、库威镰孢(fusariumcrookwellense)、黄色镰孢菌(fusariumculmorum)、禾谷镰孢菌(fusariumgraminearum)、禾赤镰孢(fusariumgraminum)、异孢镰孢(fusariumheterosporum)、合欢木镰孢(fusariumnegundi)、尖孢镰孢菌(fusariumoxysporum)、多枝镰孢(fusariumreticulatum)、粉红镰孢菌(fusariumroseum)、接骨木镰孢(fusariumsambucinum)、肤色镰孢(fusariumsarcochroum)、拟枝孢镰孢菌(fusariumsporotrichioides)、硫色镰孢菌(fusariumsulphureum)、圆镰孢(fusariumtorulosum)、拟丝孢镰孢菌(fusariumtrichothecioides)、镶片镰孢菌(fusariumvenenatum)、灰腐质霉(humicolagrisea)、特异腐质霉(humicolainsolens)、疏棉状腐质霉(humicolalanuginosa)、白囊耙齿菌(irpexlacteus)、米黑毛霉(mucormiehei)、嗜热毁丝霉(myceliophthorathermophila)、粗糙脉孢菌(neurosporacrassa)、绳状青霉菌(penicilliumfuniculosum)、产紫青霉(penicilliumpurpurogenum)、黄孢原毛平革菌(phanerochaetechrysosporium)、埃默森篮状菌(talaromycesemersonii)、无色梭孢壳(thielaviaachromatica)、成层梭孢壳菌(thielaviaalbomyces)、白毛梭孢壳(thielaviaalbopilosa)、澳洲梭孢壳(thielaviaaustraleinsis)、粪梭孢壳(thielaviafimeti)、小孢梭孢壳(thielaviamicrospora)、卵孢梭孢壳(thielaviaovispora)、秘鲁梭孢壳(thielaviaperuviana)、毛梭孢壳(thielaviasetosa)、瘤孢梭孢壳(thielaviaspededonium)、耐热梭孢壳(thielaviasubthermophila)、土生梭孢壳(thielaviaterrestris)、哈茨木霉(trichodermaharzianum)、康宁木霉(trichodermakoningii)、长枝木霉(trichodermalongibrachiatum)、里氏木霉(trichodermareesei)、或绿色木霉(trichodermaviride)细胞。在优选的方面,该纤维二糖水解酶基因是纤维二糖水解酶i基因。在更优选地方面,该纤维二糖水解酶i基因是木霉属纤维二糖水解酶i基因。在最优选的方面,该纤维二糖水解酶i基因是里氏木霉纤维二糖水解酶i基因。在另一个优选地方面,该纤维二糖水解酶基因是纤维二糖水解酶ii基因。在更优选地方面,该纤维二糖水解酶ii基因是木霉属纤维二糖水解酶ii基因。在最优选的方面,该纤维二糖水解酶ii基因是里氏木霉纤维二糖水解酶ii基因。用于指导本发明方法中核酸构建体的转录的适合启动子的实例是从以下的基因获得的启动子:里氏木霉β-葡糖苷酶、里氏木霉aa9溶解性多糖单加氧酶、里氏木霉纤维二糖水解酶i、里氏木霉纤维二糖水解酶ii、里氏木霉内切葡聚糖酶i、里氏木霉内切葡聚糖酶ii、里氏木霉内切葡聚糖酶iii、里氏木霉内切葡聚糖酶v、里氏木霉膨胀蛋白1-、里氏木霉糖基水解酶家族11木聚糖酶i、里氏木霉糖基水解酶家族11木聚糖酶ii、里氏木霉家族10木聚糖酶i、里氏木霉家族10木聚糖酶ii、里氏木霉糖基水解酶家族10木聚糖酶iii、里氏木霉糖基水解酶家族30木聚糖酶iv、和里氏木霉β-木糖苷酶;及其突变型、截短型及杂合型启动子。控制序列也可以是合适的转录终止子序列,是由本发明的突变体丝状真菌细胞识别以终止转录的序列。终止子序列可操作地连接至编码异源多肽的核苷酸序列的3’末端。在丝状真菌细胞中有功能的任何终止子可用于本发明中。优选的终止子获得自以下的基因:米曲霉taka淀粉酶、黑曲霉葡糖淀粉酶、构巢曲霉邻氨基苯甲酸合酶、黑曲霉α-葡糖苷酶、尖孢镰孢菌胰蛋白酶样蛋白酶、里氏木霉β-葡糖苷酶、里氏木霉aa9溶解性多糖单加氧酶、里氏木霉纤维二糖水解酶i、里氏木霉纤维二糖水解酶ii、里氏木霉内切葡聚糖酶i、里氏木霉内切葡聚糖酶ii、里氏木霉内切葡聚糖酶iii、里氏木霉内切葡聚糖酶v、里氏木霉膨胀蛋白1-、里氏木霉糖基水解酶家族11木聚糖酶i、里氏木霉糖基水解酶家族11木聚糖酶ii、里氏木霉家族10木聚糖酶i、里氏木霉家族10木聚糖酶ii、里氏木霉糖基水解酶家族10木聚糖酶iii、里氏木霉糖基水解酶家族30木聚糖酶iv、和里氏木霉β-木糖苷酶。该控制序列还可以是合适的前导序列,对本发明的突变体丝状真菌细胞的翻译很重要的非翻译mrna区域。前导序列可操作地连接至编码异源多肽的核苷酸序列的5’末端。在丝状真菌细胞中有功能的任何前导序列可用于本发明中。用于丝状真菌细胞的优选前导序列从米曲霉taka淀粉酶和构巢曲霉丙糖磷酸异构酶的基因获得。控制序列也可以是聚腺苷酸化序列,其是与核苷酸序列的3’末端可操作地连接的序列,并且在转录时,丝状真菌细胞将其识别为将聚腺苷残基添加至转录的mrna的信号。在丝状真菌细胞中有功能的任何聚腺苷酸化序列可用于本发明中。丝状真菌细胞的优选的聚腺苷酸化序列获得自以下的基因:米曲霉taka淀粉酶、黑曲霉葡糖淀粉酶、构巢曲霉邻氨基苯甲酸合酶、尖孢镰刀菌胰蛋白酶样蛋白酶、和黑曲霉α-葡糖苷酶。该控制序列还可以是一种信号肽编码序列,其编码连接至多肽的氨基末端并指导编码的多肽进入细胞分泌途径的氨基酸序列。核苷酸序列的编码序列的5'端可以固有地包含在翻译阅读框中与编码分泌的多肽的编码序列的区段天然地连接的一个信号肽编码序列。可替代地,编码序列的5’端可以含有对于该编码序列是外源的信号肽编码序列。在编码序列天然地不包含信号肽编码序列的情况下,可能需要外源信号肽编码序列。或者,外源信号肽编码序列可以简单地取代天然信号肽编码序列以增强多肽的分泌。然而,可以在本发明中使用指导表达的多肽进入丝状真菌细胞分泌途径的任何信号肽编码序列,即被分泌进培养基中。丝状真菌细胞的有效信号肽编码区是获自以下各项的基因的信号肽编码区:米曲霉taka淀粉酶、黑曲霉中性淀粉酶、黑曲霉葡糖淀粉酶、曼赫根毛霉天冬氨酸蛋白酶、特异腐质霉纤维素酶、特异腐质霉内切葡聚糖酶v、和柔毛腐质霉脂肪酵。控制序列也可以是编码位于多肽氨基末端的氨基酸序列的前肽编码区。所得的多肽被称为前体酶(proenzyme)或多肽原(或在一些情况下被称为酶原(zymogen))。多肽原通常是无活性的并且可以通过催化切割或自身催化切割来自多肽原的前肽而转化为成熟的活性多肽。可以从酿酒酵母α-因子、米黑根毛霉天冬氨酸蛋白酶、以及嗜热毁丝霉的漆酶(wo95/33836)的基因获得前肽编码区。当信号肽和前肽序列二者均出现在多肽的氨基末端时,将前肽序列置于紧接着(nextto)多肽氨基末端,并且将信号肽序列置于紧接着前肽序列的氨基末端。核酸构建体还可以包含一种或多种多核苷酸,其编码有利于指导异源多肽表达的一种或多种因子,例如转录激活因子(例如反式作用因子)、分子伴侣和加工蛋白酶。在丝状真菌细胞中有功能的任何因子可用于本发明中。编码这些因子中的一种或多种的核酸不一定与编码异源多肽的核苷酸序列串联。在本发明的方法中,包含核苷酸序列、启动子和转录终止信号和翻译终止信号的重组表达载体可用于重组产生目的多肽。本文所述的多种核酸和控制序列可以结合在一起以产生重组表达载体,该重组表达载体可以包括一个或多个方便的限制位点以允许在这些位点插入或取代编码多肽的核苷酸序列。可替代地,可以通过将该核苷酸序列或包括该序列的核酸构建体插入用于表达的适当载体中来表达该核苷酸序列。在产生表达载体时,编码序列如此位于载体中,使得编码序列与用于表达的适当控制序列可操作地连接。该重组表达载体可以是可以方便地经受重组dna程序并且可以引起该核苷酸序列表达的任何载体(例如,质粒或病毒)。载体的选择将典型地取决于该载体与该载体有待引入其中的丝状真菌细胞的相容性。载体可以是直链或闭合环状质粒。载体可以是自主复制载体,即作为染色体外实体存在的载体,其复制独立于染色体复制,例如质粒、染色体外元件、微染色体或人工染色体。载体可以含有用于确保自我复制的任何手段。可替代地,载体可以是这样的载体,当它引入丝状真菌细胞中时整合入基因组中并与其中已整合了它的一个或多个染色体一起复制。此外,可以使用单独的载体或质粒或两个或更多个载体或质粒,其共同包含待引入丝状真菌细胞基因组的总dna,或可以使用转座子。载体优选地含有允许容易地选择转化丝状真菌细胞的一个或多个选择性标记。选择性标记是一种基因,其产物提供了杀生物剂抗性或病毒抗性、对重金属抗性、对营养缺陷型的原养型等。用于在丝状真菌细胞中使用的选择性标记的实例包括但不限于amds(乙酰胺酶)、argb(鸟氨酸氨甲酰基转移酶)、bar(草胺膦乙酰转移酶)、hpt(潮霉素磷酸转移酶)、niad(硝酸还原酶)、pyrg(乳清苷-5’-磷酸脱羧酶)、sc(硫酸腺苷基转移酶)、以及trpc(邻氨基苯甲酸合酶),连同其等同物。优选用在丝状真菌细胞中的是构巢曲霉的amds基因和吸水链霉菌(streptomyceshygroscopicus)的bar基因。载体优选地含有允许载体整合到宿主细胞的基因组中或载体在细胞中独立于基因组自主复制的一个或多个元件。用于整合进丝状真菌细胞的基因组中,该载体可以依赖于编码多肽的多核苷酸的序列或该载体的通过同源或非同源重组而整合进该基因组中的任何其他元件。可替代地,载体可以含有额外的核苷酸序列,用于指导通过同源重组整合入丝状真菌细胞基因组染色体中的精确位置。为了增加在精确位置整合的可能性,整合元件应优选含有足够数量的核酸,如100至10,000个碱基对、优选400至10,000个碱基对、并且最优选800至10,000个碱基对,其与相应的目标序列具有高度同一性以增强同源重组的概率。整合元件可以是与丝状真菌细胞基因组内的靶序列同源的任何序列。此外,整合元件可以是非编码或编码核苷酸序列。另一方面,载体可以通过非同源重组整合入丝状真菌细胞的基因组中。对于自主复制,载体可以进一步包含使该载体能够在丝状真菌细胞中自主复制的复制起点。复制起点可以是在细胞中发挥作用的介导自主复制的任何质粒复制子。术语“复制起点”或“质粒复制子”在本文定义为能够使质粒或载体体内复制的核苷酸序列。在丝状真菌细胞中有用的复制起点的实例是ama1和ans1(gems等人,1991,gene[基因]98:61-67;cullen等人,1987,nucleicacidsresearch[核酸研究]15:9163-9175;wo00/24883)。可根据wo00/24883中披露的方法完成ama1基因的分离和包含该基因的质粒或载体的构建。用于连接本文所述的元件以构建重组表达载体的程序是本领域的普通技术人员熟知的(参见,例如,j.sambrook,e.f.fritsch,和t.maniatis,1989,molecularcloning,alaboratorymanual[分子克隆:实验室手册],第2版,冷泉港,纽约)。载体可以通过例如转化引入该丝状真菌细胞中,这样使得该载体被维持为染色体整合物或被维持为自主复制的染色体外载体。通常认为整合是有利的,因为核苷酸序列更有可能稳定地保持在细胞中。通过同源重组、非同源重组或转座将载体整合入染色体中。将表达载体引入丝状真菌细胞中可能涉及由原生质体形成、原生质体转化和细胞壁再生组成的以本身已知的方式进行的程序。用于转化曲霉属和木霉属宿主细胞的适合程序描述于以下文献中:ep238023,yelton等人,1984,proc.natl.acad.sci.usa[美国国家科学院院刊]81:1470-1474以及christensen等人,1988,bio/technology[生物/技术]6:1419-1422。用于转化镰孢属物种的适合方法由malardier等人,1989,gene[基因]78:147-156和wo96/00787描述。产生目的多肽的方法本发明还涉及产生目的多肽的方法,该方法包括(a)培养本发明的突变体丝状真菌细胞用于产生该目的多肽,并且任选地(b)回收该目的多肽。使用本领域已知的方法在适合于产生该多肽的营养培养基中培养突变体丝状真菌细胞。例如,可以通过摇瓶培养、或在实验室或工业发酵器中小规模或大规模发酵(包括连续、分批、补料分批或固态发酵)培养丝状真菌细胞,该培养在适合的培养基中并且在允许表达和/或分离多肽的条件下进行。使用本领域中已知的程序,培养发生在包含碳和氮来源及无机盐的适合的营养培养基中。适合的培养基可从商业供应商获得或可以根据公开的组成(例如,在美国典型培养物保藏中心(americantypeculturecollection)的目录中)制备。如果多肽被分泌到该营养培养基中,那么可以直接从该培养基中回收该多肽。如果多肽不进行分泌,那么其可以从细胞裂解液中进行回收。可以使用特异性针对该多肽的本领域已知的方法来检测该目的多肽。这些检测方法包括但不限于:特异性抗体的使用、酶产物的形成或酶底物的消失。例如,可以使用酶测定来确定多肽的活性。可以使用以上方法测量突变体丝状真菌细胞的目的多肽的生产力的提高。可以通过熟知的方式,例如,定量实时pcr(qrt-pcr)、dna印迹杂交、使用cdna或寡核苷酸阵列杂交(oligoarrayhybridization)的全局基因表达谱(globalgeneexpressionprofiling)、或深度rna测序(rna-seq)分析选择的mrna或转录水平来确定编码目的多肽的基因的表达的增加。可以使用本领域已知的方法来回收多肽。例如,可通过常规方法,包括但不限于,收集、离心、过滤、提取、喷雾干燥、蒸发或沉淀,从营养培养基回收多肽。在一方面,回收包含多肽的全发酵液。可以通过本领域已知的多种程序纯化该多肽,包括但不限于色谱法(例如,离子交换色谱法、亲和色谱法、疏水色谱法、聚焦色谱法和尺寸排阻色谱法)、电泳程序(例如,制备型等电聚焦电泳)、差异性溶解(例如,硫酸铵沉淀)、sds-page或提取(参见例如,proteinpurification[蛋白质纯化],janson和ryden编辑,vchpublishers[vch出版公司],纽约,1989),以便获得基本上纯的多肽。产生转录因子的方法本发明还涉及编码本发明的转录因子的分离的多核苷酸,如本文所述。用于分离或克隆多核苷酸的技术是本领域已知的且包括从基因组dna或cdna或其组合进行分离,如以上针对目的多肽所述的。可以从木霉属菌株或相关生物中克隆多核苷酸,并且因此,例如可以是多核苷酸的转录因子编码区的等位基因变体或物种变体。本发明还涉及包含编码本发明的转录因子的、可操作地连接至一个或多个控制序列上的多核苷酸的核酸构建体和表达载体,该一个或多个控制序列在与控制序列相容的条件下指导编码序列在适合的宿主细胞中表达。编码转录因子的多核苷酸可以以如上文针对编码目的多肽的多核苷酸所述的方式操作。本发明还涉及重组宿主细胞,其包含与指导本发明转录因子产生的一个或多个控制序列可操作地连接的本发明的多核苷酸。将包含多核苷酸的核酸构建体或表达载体引入宿主细胞中,这样使得该构建体或载体作为染色体整合体或作为自主复制的染色体外载体维持,如较早前所述。该重组宿主细胞可以是任何丝状真菌细胞。该丝状真菌细胞可以是枝顶孢属(acremonium)、曲霉属(aspergillus)、短梗霉属(aureobasidium)、烟管霉属(bjerkandera)、拟腊菌属(ceriporiopsis)、金孢子菌属(chrysosporium)、鬼伞属(coprinus)、革盖菌属(coriolus)、隐球菌属(cryptococcus)、线黑粉菌科(filibasidium)、镰孢属(fusarium)、腐质霉属(humicola)、梨孢菌属(magnaporthe)、毛霉属(mucor)、毁丝霉属(myceliophthora)、新美鞭菌属(neocallimastix)、脉胞菌属(neurospora)、拟青霉属(paecilomyces)、青霉属(penicillium)、平革菌属(phanerochaete)、射脉菌属(phlebia)、瘤胃壶菌属(piromyces)、侧耳属(pleurotus)、裂褶菌属(schizophyllum)、篮状菌属(talaromyces)、嗜热子囊菌属(thermoascus)、梭孢壳属(thielavia)、弯颈霉属(tolypocladium)、栓菌属(trametes)、或木霉属(trichoderma)细胞。例如,丝状真菌宿主可以是泡盛曲霉、臭曲霉、烟曲霉、日本曲霉、构巢曲霉、黑曲霉、米曲霉、黑刺烟管霉(bjerkanderaadusta)、干拟蜡菌(ceriporiopsisaneirina)、卡内基拟蜡菌(ceriporiopsiscaregiea)、浅黄拟蜡菌(ceriporiopsisgilvescens)、潘诺希塔拟蜡菌(ceriporiopsispannocinta)、环带拟蜡菌(ceriporiopsisrivulosa)、微红拟蜡菌(ceriporiopsissubrufa)、虫拟蜡菌(ceriporiopsissubvermispora)、狭边金孢子菌(chrysosporiuminops)、嗜角质金孢子菌(chrysosporiumkeratinophilum)、卢克诺文思金孢子菌(chrysosporiumlucknowense)、粪状金孢子菌(chrysosporiummerdarium)、租金孢子菌(chrysosporiumpannicola)、女王杜香金孢子菌(chrysosporiumqueenslandicum)、热带金孢子菌(chrysosporiumtropicum)、褐薄金孢子菌(chrysosporiumzonatum)、灰盖鬼伞(coprinuscinereus)、毛革盖菌(coriolushirsutus)、杆孢状镰孢、谷类镰孢、库威镰孢、大刀镰孢、禾谷镰孢、禾赤镰孢、异孢镰孢、合欢木镰孢、尖孢镰孢、多枝镰孢、粉红镰孢、接骨木镰孢、肤色镰孢、拟分枝孢镰孢、硫色镰孢、圆镰孢、拟丝孢镰孢、镶片镰孢、特异腐质霉、疏棉状腐质霉、米黑根毛霉、嗜热毁丝霉、粗糙脉胞菌、产紫青霉、黄孢平革菌(phanerochaetechrysosporium)、射脉菌(phlebiaradiata)、刺芹侧耳(pleurotuseryngii)、埃默森篮状菌、土生梭孢霉、长域毛栓菌(trametesvillosa)、变色栓菌(trametesversicolor)、哈茨木霉、康宁木霉、长枝木霉、里氏木霉或绿色木霉细胞。可以使用以上所述的程序转化丝状真菌细胞。本发明还涉及产生本发明的转录因子的方法,这些方法包括(a)在有益于产生该转录因子的条件下培养本发明的重组宿主细胞;和任选地(b)回收该转录因子。将宿主细胞在适合产生转录因子的营养培养基中培养,使用本领域已知的方法(如上文针对目的多肽所述的那些)检测和回收转录因子。通过以下实例进一步描述本发明,这些实例不应理解为对本发明的范围进行限制。实例菌株里氏木霉btr213描述于wo2013/086633中。里氏木霉菌株agjg216-2b51是ku70被破坏且副胞菌素合成酶(pars)缺失的里氏木霉btr213菌株,其包含在里氏木霉纤维二糖水解酶i(cbh1)启动子和终止子的转录控制下的嗜碱枝顶孢(acremoniumalcalophilum)gh25溶菌酶基因(dna序列为seqidno:57并且推导的氨基酸序列为seqidno:58)的四个拷贝。里氏木霉菌株gmer62-1a9是ku70被破坏且副胞菌素合成酶(pars)基因缺失的里氏木霉btr213菌株。培养基和溶液0.2m柠檬酸由以下构成:38.424g的柠檬酸和去离子水补足至1升。将溶液过滤灭菌。cmc板由以下构成:0.5%的羧甲基纤维素钠(aqualontm,亚什兰公司(ashland、inc.))、10ml的50x的痕量元素溶液、125ml的2x的矿物盐溶液、250ml的0.1m磷酸盐柠檬酸盐缓冲液、0.5ml的5m尿素、10g的诺布尔琼脂、和去离子水补足至500ml。刚果红溶液由以下构成:2.5g的刚果红和以及去离子水补足至1升。cove板由以下构成:218g的山梨醇、20g的琼脂、20ml的cove盐溶液、10mm的乙酰胺、15mm的cscl、以及去离子水补足至1升。在高压灭菌前将溶液调节至ph7.0。cove盐溶液由以下构成:26g的kcl、26g的mgso4·7h2o、76g的kh2po4、50ml的cove痕量金属溶液、以及去离子水补足至1升。cove痕量金属溶液由以下构成:0.04g的na2b4o7·10h2o、0.4g的cuso4·5h2o、1.2g的feso4·7h2o、0.7g的mnso4·h2o、0.8g的na2moo2·2h2o、10g的znso4·7h2o、以及去离子水补足至1升。cove2板由以下构成:30g的蔗糖、20ml的cove盐溶液、10ml的1m乙酰胺、25g的诺布尔琼脂、以及去离子水补足至1升。发酵分批培养基(fermentationbatchmedium)由以下构成:24g的右旋糖、40g的大豆粉、8g的(nh4)2so4、3g的k2hpo4、8g的k2so4、3g的caco3、8g的mgso4·7h2o、1g的柠檬酸、8.8ml的85%磷酸、1ml的消泡剂、14.7ml的痕量金属溶液、以及去离子水补足至1升。痕量金属溶液由以下构成:26.1g的feso4·7h2o、5.5g的znso4·7h2o、6.6g的mnso4·h2o、2.6g的cuso4·5h2o、2g的柠檬酸、以及去离子水补足至1升。50x乳糖/蛋白胨溶液由以下构成:20g的乳糖、2g的蛋白胨、以及去离子水补足至100ml。lb+amp培养基由以下构成:10g的胰蛋白胨、5g的酵母提取物、5g的氯化钠、50mg的氨苄青霉素(过滤灭菌并且在高压灭菌之后添加)、以及去离子水补足至1升。ma培养基由以下构成:10g的乳糖、1g的细菌蛋白胨、2.8g的(nh4)2so4、4g的kh2po4、0.6g的mgso4·7h2o、0.8g的cacl2·2h2o、20ml的ma痕量元素溶液、500ml的磷酸盐柠檬酸盐缓冲液、以及去离子水补足至1升。ma痕量元素溶液由以下构成:250mg的feso4·7h2o、85mg的mnso4·h2o、70mg的znso4·7h2o、100mg的cacl2·2h2o、以及去离子水补足至1升;将ph调节至2.0。mandels-andreotti培养基由以下构成:20ml的50x痕量元素溶液、250ml的2x矿物盐溶液、500ml的0.1m磷酸盐柠檬酸盐缓冲液、1ml的5m尿素、20ml的50x乳糖/蛋白胨溶液、以及去离子水补足至1升。mex板由以下构成:15g的麦芽提取物、0.5g的蛋白胨、7.5g的诺布尔琼脂、以及去离子水补足至500ml。将溶液高压灭菌20分钟,允许冷却至约55℃,然后倒入150mm培养皿;每板50ml。2x矿物盐溶液由以下构成:5.6g的(nh4)2so4(43.8mm)、8.0gkh2po4(58.32mm)、1.2g的mgso4·7h2o(4.86mm)、1.6g的cacl2·2h2o(10.88mm)、以及去离子水补足至1升。通过高压灭菌来为溶液灭菌。pda板由以下构成:39g的马铃薯右旋糖琼脂(difco)、以及去离子水补足至1升。通过高压灭菌来为溶液灭菌。pda覆层培养基由以下构成:39g的马铃薯右旋糖琼脂(difco)、以及去离子水补足至1升。通过高压灭菌来为溶液灭菌。将高压灭菌的培养基在微波中熔化,并且然后在使用前调至55℃。peg缓冲液由以下构成:去离子水中的50%聚乙二醇(peg)4000、10mmtris-hcl(ph7.5)和10mmcacl2。0.1m磷酸盐柠檬酸盐缓冲液由溶解在500ml的去离子水中的14.19g的无水na2hpo4(0.1m)构成。将溶液的ph用0.2m柠檬酸值调节至ph5.0并过滤除菌。溶液的最终体积不调整到一升。摇瓶培养基由以下构成:20g的甘油、10g的大豆粉、1.5g的(nh4)2so4、2g的kh2po4、0.2g的cacl2、0.4g的mgso4·7h2o、0.2ml的痕量金属溶液、以及去离子水补足至1升。痕量金属溶液由以下构成:26.1g的feso4·7h2o、5.5g的znso4·7h2o、6.6g的mnso4·h2o、2.6g的cuso4·5h2o、2g的柠檬酸、以及去离子水补足至1升。soc培养基由以下构成:20g的胰蛋白胨、5g的酵母提取物、0.5g的nacl、10ml的250mmkcl、以及去离子水补足至1升。stc由以下构成:去离子水中的1m山梨醇、10mmtris(ph7.5)和10mmcacl2。tae缓冲液由以下构成:4.84g的tris碱、1.14ml的冰醋酸、2ml的0.5medta(ph8.0)、以及去离子水补足至1升。50x痕量元素溶液由以下构成:250mg的feso4·7h2o(0.9mm)、85mg的mnso4·h2o(0.31mm)、70mg的znso4·7h2o(0.24mm)、100mg的cacl2·2h2o(0.68mm)、以及去离子水补足至1升。将溶液的ph用hcl调节至ph2。通过高压灭菌来为溶液灭菌。木霉属基本培养基(trmm)板由以下构成:20ml的cove盐溶液、0.6g的cacl2.2h2o、6g的(nh4)2so4、25g的诺布尔琼脂、以及去离子水补足至480ml。将溶液高压灭菌并冷却至55℃后,添加20ml过滤除菌的50%葡萄糖。5m尿素由50ml去离子水中的15.015g尿素构成并过滤灭菌。2xyt+amp板由以下构成:16g的胰蛋白胨、10g的酵母提取物、5g的nacl、15g的细菌培养用琼脂(bactoagar)、1ml的氨苄青霉素(100mg/ml)、以及去离子水补足至1升。yp培养基是由去离子水中的1%酵母提取物和2%蛋白胨构成的。实例1:从里氏木霉提取的基因组dna使这些里氏木霉在补充有在250ml带挡板的摇瓶中的2%葡萄糖(w/v)的50ml的yp培养基中、在28℃下伴随着以200rpm的搅拌生长2天。使用衬有(emd化学品公司(emdchemicalsinc.))的漏斗收集来自培养物的菌丝体,挤压干燥,然后转移至预冷的研钵和研杵中。每个菌丝体制剂磨成细粉,并且用液氮保持冷冻。将总共1-2g的粉末转移到50ml管中,并且使用植物maxi试剂盒(凯杰有限公司(qiageninc.))从研磨菌丝体粉末中提取基因组dna。将预热至65℃的5ml的ap1缓冲液(凯杰有限公司)添加到50ml管中,随后添加10μl的rna酶a100mg/ml原液(凯杰有限公司),并且在65℃下孵育2-3小时。添加共1.8ml的ap2缓冲液(凯杰有限公司),并以3000-5000xg离心5分钟。将上清液倾析到置于50ml收集管中的qiashredder大核酸纯化柱(凯杰有限公司)中,并且在室温(15℃-25℃)下在甩平式转子中以3000-5000xg离心5分钟。将收集管中的流过液体转移到新50ml管中,同时不干扰沉淀。将1.5ml体积的ap3/e缓冲液(凯杰有限公司)添加到澄清的裂解液中,并且通过涡旋振荡立即混合。将包括可能已经形成的任何沉淀的样品(最多15ml),吸移到置于50ml收集管中的大核酸纯化柱(凯杰有限公司)中,并且在室温(15℃-20℃)下在甩平式转子中以3000-5000xg离心5分钟。弃去流过液体。将12ml的aw缓冲液(凯杰有限公司)添加到大核酸纯化柱中,并以3000-5000xg离心10分钟,以将膜干燥。弃去流过液体和收集管。将大核酸纯化柱转移到新50ml管中。将预热至65℃的0.5ml的ae缓冲液(凯杰有限公司)直接吸移到大核酸纯化柱膜中,在室温(15℃-25℃)下孵育5分钟,然后以3000-5000xg离心5分钟以洗脱基因组dna。基因组dna的浓度和纯度通过在260nm和280nm处测量吸光度确定。实例2:里氏木霉原生质体的产生与转化使用与penttila等人,1987,gene[基因]61:155-164类似的方案进行里氏木霉的原生质体制备与转化。简而言之,将里氏木霉在25ml的补充有2%(w/v)葡萄糖和10mm尿苷的yp培养基中、在27℃下伴随着以90rpm进行的轻轻搅拌培养17小时。将菌丝体通过使用真空驱动一次性过滤系统(密理博公司(millipore))过滤来收集,并且用去离子水洗涤两次并用1.2m山梨醇洗涤两次。通过在34℃下伴随90rpm的轻轻振荡使洗涤的菌丝体悬浮于100ml含有5mg/ml的yatalasetm酶(美国宝生物工程株式会社(takarabiousa,inc.))和0.36单位/ml的壳多糖酶(西格玛化工有限公司(sigmachemicalco.))的1.2m山梨醇中60-75分钟来产生原生质体。将原生质体通过以834xg离心7分钟来收集,并且用冷的1.2m山梨醇洗涤两次。将原生质体使用血球计数器来计数,并且使其重新悬浮至最终浓度为1x108个原生质体/ml的stc。将大约1-10μg的dna添加至100μl的原生质体溶液中并且轻轻混合。添加peg缓冲液(250μl),并且将反应混合并在34℃下孵育30分钟。然后添加stc(3ml),并且将反应混合,并且然后涂布在补充有1m蔗糖的pda板上(用于潮霉素选择)。在30℃下孵育16小时后,将20ml的补充有35μg/ml的潮霉素b(赛默飞世尔科技公司(thermofisherscientific))的pda覆层培养基添加至各板。将板在30℃下孵育4-7天。实例3:里氏木霉btr213的不产生纤维素酶(cel-)型和产生纤维素酶(cel+)型分离株的产生如wo2013/086633中所述,通过在14升试验性规模分批发酵罐中发酵里氏木霉btr213来产生里氏木霉btr213的不产生纤维素酶(cel-)型分离株和产生纤维素酶(cel+)型分离株。如wo2013/086633中所述,在发酵开始后168小时,收集细胞并通过在酸溶胀纤维素板上铺板来测试其产生纤维素酶的能力。将在酸溶胀纤维素板上形成透明区的细胞鉴定为产生纤维素酶(cel+)型分离株,将不形成透明区的细胞鉴定为不产生纤维素酶(cel-)型分离株。实例4:培养里氏木霉btr213不产生纤维素酶(cel-)型和产生纤维素酶(cel+)型分离株,用于rna测序分析将里氏木霉btr213不产生纤维素酶(cel-)型和产生纤维素酶(cel+)型分离株(实例3)在1升摇瓶中的200mlma培养基中在30℃下伴随180rpm的振荡生长24、48、72、96和120小时。一式三份地进行每种培养。将每个时间点和每个重复的菌丝体通过使用过滤来分别收集,在去离子水中洗涤两次,并且在液氮下冷冻。将冷冻的菌丝体通过杵和研钵磨碎至细粉。根据制造商的方案,使用plusrna纯化试剂盒(生命技术公司(lifetechnologies)/英杰(invitrogen))来分离总rna。实例5:里氏木霉btr213的不产生纤维素酶(cel-)型和产生纤维素酶(cel+)型分离株的rnaseq分析由分离自三个生物重复的rna构建truseqrnaseq文库,每个生物重复在如实例4中所述的两种条件下:cel+(48小时)和cel-(48小时)。在nextseqtm(依诺米那公司(illuminainc.))平台上使用成对的150碱基对illumina读段来测序文库。每个样品产生约1-1.2千万个读段。在clcgenomicsserver7版(clcbiogenomics,凯杰有限公司)中进行计算机模拟分析。基于质量修剪序列并移除适配体序列。使用clcgenomicsserver7版中所实施的rnaseq模块将修剪后的序列映射至里氏木霉rut-c30基因序列(里氏木霉rut-c30基因组数据库;jourdier等人,2017,biotechnol.biofuels[用于生物燃料的生物技术]10:151)(错配=2,相似度=0.8,最大错配/读段(maxmatches_per_read)=10)。将每个基因汇总的总读段计数用于下游分析。使用箱形图和主成分分析来评估重复质量。使用clc生物基因组学7版中实施的基于edger的方法,将差异表达分析(dge)作为48小时cel+相对于cel-样品之间的对比分析进行(robinson等人,2010,bioinformatics[生物信息学]26(1):139-140)。通过使用fdr校正的0.05的p值阈值过滤来评估显著性。使用k-means聚类将重要的dge基因的此子集分箱(箱=20)。将使用ec编号注释的go术语和通路映射用于添加功能注释。这些注释的源文件是从联合基因组研究所(jointgenomeinstitute)的里氏木霉qm6a基因组数据库下载的(jgi里氏木霉基因组数据库v.2.0;martinez等人,2008,naturebiotechnol.[自然生物技术]26:553-560)。将从马赫实验室(machlab)收到的定制注释用于注释已知的转录因子。将定制perl脚本和数据库查询序列(query)用于组合各种注释源,以制作注释的转录因子基因的最终列表。转录因子基因的列表示于表1中(dna序列为seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、和55并且氨基酸序列为seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、和124)。jgi蛋白质编号对应于联合基因组研究所的里氏木霉rut-c30基因组数据库的经注释的基因(jgi里氏木霉基因组数据库v.1.0;jourdier等人,2017,biotechnol.biofuels[用于生物燃料的生物技术]10:151)。将转录因子70883和132448的模型分成两个,以反映从rnaseq数据中获得的关于这些模型的冲突数据。表1实例6:里氏木霉转录因子70883基因缺失质粒psmai320的构建构建质粒psmai320以缺失里氏木霉agjg216-2b51中的转录因子(tf)70883基因(dna序列为seqidno:3并且推导的氨基酸序列为seqidno:4)。为了构建里氏木霉tf70883基因缺失盒,使用以下所示的引物1225696和1225697来pcr扩增含有里氏木霉tf70883基因的上游非编码区的2060bp片段的pcr产物(dna片段1)。正向引物1225696:gagtcgacctgcaggcatgcgtttaaacttggccacctacactgctacta(seqidno:59)反向引物1225697:cgtgaagccgtttaaatgaaactagctccagatggaaatatac(seqidno:60)扩增反应由以下构成:约180ng的里氏木霉btr213基因组dna、10mm的dntp、50pmol的正向引物、50pmol的反向引物、1xhf缓冲液(赛默飞世尔科技公司)、和2单位的热启动(hotstart)dna聚合酶(赛默飞世尔科技公司),最终体积为50μl。反应在编程为如下的热循环仪中孵育:1个循环,在98℃下持续3分钟;30个循环,各在98℃下持续10秒,58℃下持续30秒、以及72℃下持续2.5分钟;1个循环,在72℃下持续10分钟;以及在10℃下保持。将所得2,129bppcr片段通过使用tae缓冲液中的0.8%琼脂糖凝胶电泳进行纯化,从凝胶切离,并使用凝胶和pcr清洁试剂盒(pcrclean-upkit)(马歇雷-纳格尔公司(macherey-nagel))进行提取。使用以下所示的引物1225698和1225699来pcr扩增含有大肠杆菌潮霉素磷酸转移酶基因(hpt)选择标记和人单纯疱疹病毒1型胸苷激酶基因(tk)选择标记盒的pcr产物(dna片段2)。正向引物1225698:tatttccatctggagctagtttcatttaaacggcttcacggg(seqidno:61)反向引物1225699:tcgttcgaaattttcttctagagagttcaaggaagaaacagtgc(seqidno:62)扩增反应由以下构成:约10ng的pjfys1579-41-11(us20110223671a1)、10mm的dntp、50pmol的正向引物、50pmol的反向引物、1xhf缓冲液、以及2单位的热启动dna聚合酶,最终体积为50μl。反应在编程为如下的热循环仪中孵育:1个循环,在98℃下持续3分钟;30个循环,各在98℃下持续10秒,58℃下持续30秒、以及72℃下持续2.5分钟;1个循环,在72℃下持续10分钟;以及在10℃下保持。将所得4,449bppcr片段通过使用tae缓冲液中的0.8%琼脂糖凝胶电泳进行纯化,从凝胶切离,并使用凝胶和pcr清洁试剂盒进行提取。使用以下所示的引物1225700和1225701来pcr扩增含有里氏木霉tf70883基因的上游非编码区的235bp的pcr产物(dna片段3)(将作为同向重复区)。正向引物1225700:tgtttcttccttgaactctctagaagaaaatttcgaacgaaccg(seqidno:63)反向引物1225701:gaagaatcgactggctgcctactagctccagatggaaatatact(seqidno:64)扩增反应由以下构成:约180ng的里氏木霉btr213基因组dna、10mm的dntp、50pmol的正向引物、50pmol的反向引物、1xhf缓冲液、和2单位的热启动dna聚合酶,最终体积为50μl。反应在编程为如下的热循环仪中孵育:1个循环,在98℃下持续3分钟;30个循环,各在98℃下持续10秒,58℃下持续30秒、以及72℃下持续2.5分钟;1个循环,在72℃下持续10分钟;以及在10℃下保持。将所得323bppcr片段通过使用tae缓冲液中的0.8%琼脂糖凝胶电泳进行纯化,从凝胶切离,并使用凝胶和pcr清洁试剂盒进行提取。使用以下所示的引物1225702和1225703来pcr扩增含有里氏木霉tf70883基因的下游非编码区的2060bp片段的pcr产物(dna片段4)。正向引物1225702:tatttccatctggagctagtaggcagccagtcgattcttctt(seqidno:65)反向引物1225703:gctatgaccatgattacgccgtttaaaccgtccagataatgcgcacgc(seqidno:66)扩增反应由以下构成:约180ng的里氏木霉btr213基因组dna、10mm的dntp、50pmol的正向引物、50pmol的反向引物、1xhf缓冲液、和2单位的热启动dna聚合酶,最终体积为50μl。反应在编程为如下的热循环仪中孵育:1个循环,在98℃下持续3分钟;30个循环,各在98℃下持续10秒,58℃下持续30秒、以及72℃下持续2.5分钟;1个循环,在72℃下持续10分钟;以及在10℃下保持。将所得2126bppcr片段通过使用tae缓冲液中的0.8%琼脂糖凝胶电泳进行纯化,从凝胶切离,并使用凝胶和pcr清洁试剂盒进行提取。将质粒puc19(新英格兰生物实验室公司(newenglandbiolabsinc.))用hindiii进行酶切,并且通过使用tae缓冲液中的0.8%琼脂糖凝胶电泳进行纯化,其中将2,686bp片段从凝胶切离,并且使用凝胶和pcr清洁试剂盒进行提取。使用hifidna组装克隆试剂盒(新英格兰生物实验室公司)用以上所述的四种pcr产物(dna片段1、2、3和4)(总体积为20μl,由1xhifi组装主混合物(新英格兰生物实验室公司)和0.05pmol的每种pcr产物构成)来组装2,686bp片段。将反应在50℃下孵育60分钟,然后置于冰上。使用2μl的反应来转化50μl的stellartm化学感受态大肠杆菌细胞(克罗泰克实验室公司(clontechlaboratories))。将这些细胞在42℃下热振荡45秒,然后添加预热至42℃的450μl的soc培养基。将细胞在37℃下伴随200rpm的振荡孵育60分钟并且然后涂布到直径150mm的2xyt+amp板上并在37℃下孵育过夜。将所得大肠杆菌转化体单独地接种到在14ml圆底聚丙烯管中的3ml的lb+amp培养基中,并且在37℃下伴随200rpm的振荡而孵育过夜。将质粒dna使用9600(凯杰有限公司(qiageninc.))分离并通过ncoi消化筛选正确插入的片段。通过用model377xl自动dna测序仪(appliedbiosystemsinc.),使用染料终止子化学(giesecke等人,1992,j.virol.methods[病毒学方法杂志]38(1):47-60)的dna测序确认产生所希望带大小的质粒(6905bp+4485bp)。鉴定了含有插入且无pcr错误的一种质粒并将其指定为psmai320(图1)。实例7:里氏木霉转录因子(tf)92949基因缺失质粒psmai321的构建构建质粒psmai321以缺失里氏木霉agjg216-2b51中的转录因子(tf)92949基因(dna序列为seqidno:1并且推导的氨基酸序列为seqidno:2)。为了构建里氏木霉tf92949基因缺失盒,使用以下所示的引物1225708和1225709来pcr扩增含有里氏木霉tf92949基因的上游非编码区的2060bp片段的pcr产物(dna片段1)。正向引物1225708:gagtcgacctgcaggcatgcgtttaaacacacacaggggtaccgtttc(seqidno:67)反向引物1225709:cgtgaagccgtttaaatgaagttgacggttgagcagaaaacgc(seqidno:68)扩增反应由以下构成:约180ng的里氏木霉btr213基因组dna、10mm的dntp、50pmol的正向引物、50pmol的反向引物、1xhf缓冲液、和2单位的热启动dna聚合酶,最终体积为50μl。反应在编程为如下的热循环仪中孵育:1个循环,在98℃下持续3分钟;30个循环,各在98℃下持续10秒,58℃下持续30秒、以及72℃下持续2.5分钟;1个循环,在72℃下持续10分钟;以及在10℃下保持。将所得2,151bppcr片段通过使用tae缓冲液中的0.8%琼脂糖凝胶电泳进行纯化,从凝胶切离,并使用凝胶和pcr清洁试剂盒进行提取。使用以下所示的引物1225710和1225711来pcr扩增含有大肠杆菌潮霉素磷酸转移酶(hpt)选择标记和人单纯疱疹病毒1型胸苷激酶基因(hsv-1tk)选择标记盒的pcr产物(dna片段2)。正向引物1225710:ttttctgctcaaccgtcaacttcatttaaacggcttcacgggc(seqidno:69)反向引物1225711:gagtggtgggtttggtttgcgagagttcaaggaagaaacagtgc(seqidno:70)扩增反应由以下构成:约10ng的pjfys1579-41-11(us20110223671)、10mm的dntp、50pmol的正向引物、50pmol的反向引物、1xhf缓冲液、以及2单位的热启动dna聚合酶,最终体积为50μl。反应在编程为如下的热循环仪中孵育:1个循环,在98℃下持续3分钟;30个循环,各在98℃下持续10秒,58℃下持续30秒、以及72℃下持续2.5分钟;1个循环,在72℃下持续10分钟;以及在10℃下保持。将所得4,450bppcr片段通过使用tae缓冲液中的0.8%琼脂糖凝胶电泳进行纯化,从凝胶切离,并使用凝胶和pcr清洁试剂盒进行提取。使用以下所示的引物1225712和1225713来pcr扩增含有里氏木霉tf92949基因的上游非编码区的235bp的pcr产物(dna片段3)(将作为同向重复区)。正向引物1225712:tgtttcttccttgaactctcgcaaaccaaacccaccactctac(seqidno:71)反向引物1225713:gatcagttcggatacgcgctgttgacggttgagcagaaaacg(seqidno:72)扩增反应由以下构成:约180ng的里氏木霉btr213基因组dna、10mm的dntp、50pmol的正向引物、50pmol的反向引物、1xhf缓冲液、和2单位的热启动dna聚合酶,最终体积为50μl。反应在编程为如下的热循环仪中孵育:1个循环,在98℃下持续3分钟;30个循环,各在98℃下持续10秒,58℃下持续30秒、以及72℃下持续2.5分钟;1个循环,在72℃下持续10分钟;以及在10℃下保持。将所得320bppcr片段通过使用tae缓冲液中的0.8%琼脂糖凝胶电泳进行纯化,从凝胶切离,并使用凝胶和pcr清洁试剂盒进行提取。使用以下所示的引物1225714和1225715来pcr扩增含有里氏木霉tf92949基因的下游非编码区的2065bp片段的pcr产物(dna片段4)。正向引物1225714:ttttctgctcaaccgtcaacagcgcgtatccgaactgatcta(seqidno:73)反向引物1225715:gctatgaccatgattacgccgtttaaacctacctgtcgaagaaataaaagagg(seqidno:74)扩增反应由以下构成:约180ng的里氏木霉btr213基因组dna、10mm的dntp、50pmol的正向引物、50pmol的反向引物、1xhf缓冲液、和2单位的热启动dna聚合酶,最终体积为50μl。反应在编程为如下的热循环仪中孵育:1个循环,在98℃下持续3分钟;30个循环,各在98℃下持续10秒,58℃下持续30秒、以及72℃下持续2.5分钟;1个循环,在72℃下持续10分钟;以及在10℃下保持。将所得2,155bppcr片段通过使用tae缓冲液中的0.8%琼脂糖凝胶电泳进行纯化,从凝胶切离,并使用凝胶和pcr清洁试剂盒进行提取。将质粒puc19用hindiii进行酶切,并且通过使用tae缓冲液中的0.8%琼脂糖凝胶电泳进行纯化,其中将2,686bp片段从凝胶切离,并且使用凝胶和pcr清洁试剂盒进行提取。使用hifidna组装克隆试剂盒用以上所述的四种pcr产物(dna片段1、2、3和4)(总体积为20μl,由1xhifi组装主混合物和0.05pmol的每种pcr产物构成)来组装2,686bp片段。将反应在50℃下孵育60分钟,然后置于冰上。使用2μl的反应来转化50μl的stellartm化学感受态大肠杆菌细胞。将这些细胞在42℃下热振荡45秒,然后添加预热至42℃的450μl的soc培养基。将细胞在37℃下伴随200rpm的振荡孵育60分钟并且然后涂布到直径150mm的2xyt+amp板上并在37℃下孵育过夜。将所得大肠杆菌转化体单独地接种到在14ml圆底聚丙烯管中的3ml的lb+amp培养基中,并且在37℃下伴随200rpm的振荡而孵育过夜。将质粒dna使用9600分离并通过ncoi消化筛选正确插入的片段。通过用model377xl自动dna测序仪,使用染料终止子化学(giesecke等人,1992,如上)的dna测序确认产生所希望带大小的质粒(7422bp和3997bp)。鉴定了含有插入且无pcr错误的一种质粒并将其指定为psmai321(图2)。实例8:单转录因子(tf)70883缺失里氏木霉菌株qmji122-16b4-4的产生根据实例2来产生并转化里氏木霉菌株agjg216-2b51的原生质体以缺失转录因子(tf)70883基因。将原生质体转移到15个圆底聚丙烯管中,并用3μg的pmei-线性化的和凝胶纯化的psmai320(实例6)转化。在含有潮霉素b的pda板上选择七个转化体。将每个转化体转移到pda板上并且在30℃下孵育5天以产生孢子。针对在tf70883基因座处存在psmai320缺失载体,使用phiretm植物直接pcr试剂盒(赛默飞世尔科技公司)通过真菌孢子pcr方法来筛选里氏木霉agjg216-2b51的转化体。将来自每个转化体的少量孢子悬浮于20μl的稀释缓冲液(phiretm植物直接pcr试剂盒)中。将这些孢子悬浮液用作pcr中的模板,来筛选tf70883基因缺失。每个反应由以下构成:在10μl反应中的0.5μl的孢子悬浮液、10pmol的以下所示的每种引物(每个pcr中2-3种引物)、5μl的2xphiretm植物pcr缓冲液、和0.2μl的phiretm热启动iidna聚合酶。tf70883基因座5'末端的pcr筛选:正向引物1225977:tgaccgggcaggggatcgcc(seqidno:75)反向引物1225978:ctggggcgtcaagggacctgaatg(seqidno:76)反向引物1219245:ctacatcgaagctgaaagcacgaga(seqidno:77)tf70883基因座3'末端的pcr筛选:正向引物1213333:gggacgccctgctgcaacttacc(seqidno:78)反向引物1225979:cgcccttcgacgagtcggcac(seqidno:79)反应在编程为如下的热循环仪中孵育:1个循环,在98℃下持续5分钟;40个循环,各在98℃下持续10秒、65℃下持续10秒、以及72℃下持续90秒;1个循环,在72℃下持续1分钟;以及在4℃下保持。通过使用tae缓冲液中的1.5%琼脂糖凝胶电泳来分析完成的pcr。将psmai320dna正确靶向tf70883基因座的转化体产生了3kbpcr片段。选择产生正确pcr片段的一个转化体(里氏木霉qmji122-16)用于单孢子分离。在5ml的0.01%20溶液中收集来自6天时长pda板的少量孢子。将孢子溶液的2μl等分试样与100μl0.01%20溶液混合,并涂布在补充有1m蔗糖的150mmpda板上。将板在30℃下孵育2-3天。将单个菌落转移至pda板上并在30℃下孵育5-7天。如实例8中所述,利用真菌孢子pcr来鉴定具有正确靶向tf70883基因座的孢子分离株。缺失构建体psmai320含有侧翼为同向重复的可正选择的潮霉素磷酰基转移酶(hpt)基因和可负选择的胸苷激酶(tk)基因。插入同向重复以促进hpt和tk选择标记的切除并产生用作宿主的无标记菌株以缺失第二转录因子基因。将来自里氏木霉qmji122-16的孢子以1x104、1x105和1x106的浓度涂布到木霉属基本培养基板上并且在30℃下孵育5天,这些板含有1.5μm的5-氟-2’-脱氧尿苷(fdu)。将二十个分离株传代培养到pda板上并且在30℃下孵育4天。然后针对不存在hpt和tk选择标记基因,通过真菌孢子pcr方法使用phiretm植物直接pcr试剂盒筛选所有20个分离株。将来自每个分离株的少量孢子悬浮于20μl的稀释缓冲液(phiretm植物直接pcr试剂盒)中。将孢子悬浮液用作pcr中的模板来针对tf70883基因座处不存在hpt和tk选择标记基因进行筛选。每个反应由以下构成:在10μl反应中的0.5μl的孢子悬浮液、10pmol的以下所示的每种引物(3种引物)、5μl的2xphiretm植物pcr缓冲液、和0.2μl的phiretm热启动iidna聚合酶。正向引物1225977:tgaccgggcaggggatcgcc(seqidno:80)反向引物1225979:cgcccttcgacgagtcggcac(seqidno:81)反向引物1219245:ctacatcgaagctgaaagcacgaga(seqidno:82)反应在编程为如下的热循环仪中孵育:1个循环,在98℃下持续5分钟;40个循环,各在98℃下持续10秒、65℃下持续10秒、以及72℃下持续90秒;1个循环,在72℃下持续1分钟;以及在4℃下保持。通过使用tae缓冲液中的1.5%琼脂糖凝胶电泳来分析完成的pcr。切除hpt和tk选择标记基因的分离株产生了4.3kb的pcr片段。显示标记基因的正确切除的分离株经历另一轮单次分离,随后是如上所述的真菌孢子pcr。如实例1中所述的制备基因组dna,并且在nextseqtm500中使用2x150bp化学进行测序。测序鉴定转化体里氏木霉qmji122-16b4-4含有tf70883基因缺失并且不存在hpt和tk选择标记基因。实例9:单转录因子(tf)92949缺失里氏木霉菌株qmji121-8d7-3的产生根据实例2来产生并转化里氏木霉菌株agjg216-2b51的原生质体以缺失转录因子(tf)92949基因。将原生质体转移到15个圆底聚丙烯管中,并用3μg的pmei-线性化的和凝胶纯化的psmai321(实例7)转化。在含有潮霉素b的pda板上选择十二个转化体。将每个转化体转移到pda板上并且在30℃下孵育5天以产生孢子。针对在tf92949基因座处存在psmai321缺失载体,使用phiretm植物直接pcr试剂盒通过真菌孢子pcr方法来筛选里氏木霉agjg216-2b51的转化体。将来自每个转化体的少量孢子悬浮于20μl的稀释缓冲液(phiretm植物直接pcr试剂盒)中。将这些孢子悬浮液用作pcr中的模板,来筛选tf92949基因缺失。每个反应由以下构成:在10μl反应中的0.5μl的孢子悬浮液、10pmol的以下所示的每种引物(每个pcr中2-3种引物)、5μl的2xphiretm植物pcr缓冲液、和0.2μl的phiretm热启动iidna聚合酶。tf92949基因座5'末端的pcr筛选:正向引物1225980:tgccctggtttcgcgcatacgg(seqidno:83)反向引物1225981:acggataggagcagcaaagcaaaggc(seqidno:84)反向引物1219245:ctacatcgaagctgaaagcacgaga(seqidno:85)tf92949基因座3'末端的pcr筛选:正向引物1213333:gggacgccctgctgcaacttacc(seqidno:86)反向引物1225982:gagacgagactggagtcgttgccgc(seqidno:87)反应在编程为如下的热循环仪中孵育:1个循环,在98℃下持续5分钟;40个循环,各在98℃下持续10秒、65℃下持续10秒、以及72℃下持续90秒;1个循环,在72℃下持续1分钟;以及在4℃下保持。通过使用tae缓冲液中的1.5%琼脂糖凝胶电泳来分析完成的pcr。将psmai321dna正确靶向tf92949基因座的转化体产生了3kbpcr片段。选择产生正确pcr片段的一个转化体(里氏木霉qmji121-8)用于单孢子分离。在5ml的0.01%20溶液中收集来自6天时长pda板的少量孢子。将孢子溶液的2μl等分试样与100μl0.01%20溶液混合,并涂布在补充有1m蔗糖的150mmpda板上。将板在30℃下孵育2-3天。将单个菌落转移至pda板上并在30℃下孵育5-7天。利用真菌孢子pcr来鉴定具有正确靶向tf92949基因座的孢子分离株。缺失构建体psmai321含有侧翼为同向重复的可正选择的潮霉素磷酰基转移酶(hpt)基因和可负选择的胸苷激酶(tk)基因。插入同向重复以促进hpt和tk选择标记基因的切除并产生用作宿主的无标记菌株以缺失第二转录因子基因。将来自里氏木霉qmji121-8的孢子以1x104、1x105和1x106的浓度涂布到木霉属基本培养基板上并且在30℃下孵育5天,这些板含有1.5μm的5-氟-2’-脱氧尿苷(fdu)。将二十个分离株传代培养到pda板上并且在30℃下孵育4天。然后针对不存在hpt和tk选择标记基因,通过真菌孢子pcr方法使用phiretm植物直接pcr试剂盒筛选所有20个分离株。将来自每个分离株的少量孢子悬浮于20μl的稀释缓冲液(phiretm植物直接pcr试剂盒)中。将孢子悬浮液用作pcr中的模板来针对tf92949基因座处不存在hpt和tk选择标记基因进行筛选。每个反应由以下构成:在10μl反应中的0.5μl的孢子悬浮液、10pmol的以下所示的每种引物(3种引物)、5μl的2xphiretm植物pcr缓冲液、和0.2μl的phiretm热启动iidna聚合酶。正向引物1225980:tgccctggtttcgcgcatacgg(seqidno:88)反向引物1225982:gagacgagactggagtcgttgccgc(seqidno:89)反向引物1219245:ctacatcgaagctgaaagcacgaga(seqidno:90)反应在编程为如下的热循环仪中孵育:1个循环,在98℃下持续5分钟;40个循环,各在98℃下持续10秒、65℃下持续10秒、以及72℃下持续90秒;1个循环,在72℃下持续1分钟;以及在4℃下保持。通过使用tae缓冲液中的1.5%琼脂糖凝胶电泳来分析完成的pcr。切除hpt和tk选择标记基因的分离株产生了4.4kb的pcr片段。显示标记基因的正确切除的分离株经历另一轮单次分离,随后是如上所述的真菌孢子pcr。如实例1中所述的制备基因组dna,并且在nextseqtm500中使用2x150bp化学进行测序。测序鉴定转化体里氏木霉qmji121-8d7-3含有tf92949基因缺失并且不存在hpt和tk选择标记基因。实例10:双转录因子(tf)缺失里氏木霉菌株smai321-1c2-1的产生根据实例2来产生并转化里氏木霉单转录因子tf70883缺失菌株qmji122-16b4-4的原生质体以缺失第二转录因子(tf)92949基因。将原生质体转移到十个圆底聚丙烯管中,并用4μg的pmei-线性化的和凝胶纯化的psmai321(实例7)转化。在含有潮霉素b的pda板上选择二十个转化体。将每个转化体转移到pda板上并且在30℃下孵育5天以产生孢子。针对在tf92949基因座处存在psmai321缺失载体,使用phiretm植物直接pcr试剂盒通过真菌孢子pcr方法来筛选里氏木霉qmji122-16b4-4的转化体。将来自每个转化体的少量孢子悬浮于20μl的稀释缓冲液(phiretm植物直接pcr试剂盒)中。将这些孢子悬浮液用作pcr中的模板,来筛选tf92949基因缺失。每个反应由以下构成:在10μl反应中的0.5μl的孢子悬浮液、10pmol的以下所示的每种引物(每个pcr中2-3种引物)、5μl的2xphiretm植物pcr缓冲液、和0.2μl的phiretm热启动iidna聚合酶。tf92949基因座5'末端的pcr筛选:正向引物1225980:tgccctggtttcgcgcatacgg(seqidno:91)反向引物1225981:acggataggagcagcaaagcaaaggc(seqidno:92)反向引物1219245:ctacatcgaagctgaaagcacgaga(seqidno:93)tf92949基因座3'末端的pcr筛选:正向引物1213333:gggacgccctgctgcaacttacc(seqidno:94)反向引物1225982:gagacgagactggagtcgttgccgc(seqidno:95)反应在编程为如下的热循环仪中孵育:1个循环,在98℃下持续5分钟;40个循环,各在98℃下持续10秒、65℃下持续10秒、以及72℃下持续90秒;1个循环,在72℃下持续1分钟;以及在4℃下保持。通过使用tae缓冲液中的1.5%琼脂糖凝胶电泳来分析完成的pcr。将psmai321dna正确靶向tf92949基因座的转化体产生了3kbpcr片段。选择产生正确pcr片段的三种转化体(里氏木霉smai321-1、smai321-3、和smai321-14)用于单孢子分离。在5ml的0.01%20溶液中收集来自6天时长pda板的少量孢子(针对每种转化体)。将每个孢子溶液的2μl等分试样与100μl0.01%20溶液混合,并涂布在补充有1m蔗糖的150mmpda板上。将板在30℃下孵育2-3天。将单个菌落转移至pda板上并在30℃下孵育5-7天。如上所述,利用真菌孢子pcr来鉴定具有正确靶向tf92949基因座的孢子分离株。缺失构建体psmai321含有侧翼为同向重复的可正选择的潮霉素磷酰基转移酶(hpt)基因和可负选择的胸苷激酶(tk)基因。插入同向重复以促进hpt和tk可选择的标记物的切除并产生无标记菌株。将来自里氏木霉smai321-1、smai321-3、和smai321-14的孢子以1x104、1x105和1x106的浓度涂布到木霉属基本培养基板上并且在30℃下孵育,这些板含有1.5μm的5-氟-2’-脱氧尿苷(fdu)。将三十个分离株传代培养到pda板上并且在30℃下孵育4天。然后针对不存在hpt和tk选择标记基因,通过真菌孢子pcr方法使用phiretm植物直接pcr试剂盒筛选所有30个分离株。将来自每个分离株的少量孢子悬浮于20μl的稀释缓冲液(phiretm植物直接pcr试剂盒)中。将孢子悬浮液用作pcr中的模板来针对tf92949基因座处不存在hpt和tk选择标记基因进行筛选。每个反应由以下构成:在10μl反应中的0.5μl的孢子悬浮液、10pmol的以下所示的每种引物(3种引物)、5μl的2xphiretm植物pcr缓冲液、和0.2μl的phiretm热启动iidna聚合酶。正向引物1225980:tgccctggtttcgcgcatacgg(seqidno:96)反向引物1225982:gagacgagactggagtcgttgccgc(seqidno:97)反向引物1219245:ctacatcgaagctgaaagcacgaga(seqidno:98)反应在编程为如下的热循环仪中孵育:1个循环,在98℃下持续5分钟;40个循环,各在98℃下持续10秒、65℃下持续10秒、以及72℃下持续90秒;1个循环,在72℃下持续1分钟;以及在4℃下保持。通过使用tae缓冲液中的1.5%琼脂糖凝胶电泳来分析完成的pcr。切除hpt和tk选择标记基因的分离株产生了4.4kb的pcr片段。显示标记基因的正确切除的分离株经历另一轮单次分离,随后是如上所述的真菌孢子pcr。如实例1中所述的制备基因组dna,并且在nextseqtm500中使用2x150bp化学进行测序。测序鉴定转化体里氏木霉smai321-1c2-1含有tf92949基因缺失并且不存在hpt和tk选择标记基因。实例11:溶菌酶活性测定将来自2l发酵的全培养液在30℃下在旋转混合器中混合约2小时。在全培养液混合后,将所有样品在40%的尿素中稀释100x,然后使用旋转混合器再次混合约2小时。将100x稀释样品在样品缓冲液(0.1mtris、0.1mnacl、0.01%x-100缓冲液ph7.5)中通过10倍系列稀释而稀释1,000,000x,随后将稀释样品从0倍系列稀释至1/3倍至1/9倍。将溶菌酶标准品在样品缓冲液(0.1mtris、0.1mnacl、0.01%tritonx-100缓冲液ph7.5)中从0.05lsu(f)/ml浓度稀释到0.002lsu(f)/ml浓度。将包括标准品的每个稀释的共50μl转移至96孔平底板中。向各孔中添加50μl的25ug/ml荧光素缀合的细胞壁底物溶液,然后在环境温度下孵育45分钟。孵育期间,在式板读取器(分子器件有限公司(moleculardevices,llc.))上,以15分钟的间隔监测96孔板在485nm(激发)/528nm(发射)处的反应速率。通过从生成的标准曲线外推来确定样品浓度。实例12:在2升发酵中,比较里氏木霉agjg216-2b51、单转录因子70883缺失菌株qmji122-16b4-4和单转录因子92949缺失菌株qmji121-8d7-3中的嗜碱枝顶孢gh25溶菌酶生产力在2升发酵中,评估单转录因子70883缺失菌株里氏木霉qmji122-16b4-4(实例8)、单转录因子92949缺失菌株里氏木霉qmji121-8d7-3(实例9)和宿主菌株里氏木霉agjg216-2b51。使各菌株在30℃下在pda琼脂板上生长4-7天。将各自含有100ml摇瓶培养基的三个500ml摇瓶用来自pda琼脂板的两个塞子接种。将摇瓶在28℃在定轨摇床上以250rpm孵育48小时。将这些培养物用作种子进行发酵。使用共160ml的各种子培养物接种含有1.6升发酵分批培养基的3-升玻璃夹套发酵罐(欧谱生物技术公司(applikonbiotechnology))。将发酵罐维持在28℃的温度下,并且使用applikon1030控制系统(欧谱生物技术公司)将ph控制在3.5+/-0.1的设定值。将空气以2.5l/分钟的速率添加到容器中,并用以1100rpm旋转的rushton叶轮搅拌培养液。将由右旋糖和磷酸构成的发酵补料培养基以0至10g/l/小时的速率给予165小时。在发酵运行的第3、4、5、6和7天取样。将全培养液在4℃下储存。如实例11中所述,确定全培养液样品的嗜碱枝顶孢gh25溶菌酶表达水平。与宿主菌株里氏木霉agjg216-2b51相比,观察到具有tf70883缺失的菌株中嗜碱枝顶孢溶菌酶活性增加了1.36x,该菌株含有4拷贝的嗜碱枝顶孢溶菌酶基因(表2)。类似地,与宿主菌株里氏木霉agjg216-2b51相比,观察到具有tf92949缺失的菌株中嗜碱枝顶孢溶菌酶活性增加了1.27x,该菌株含有4拷贝的嗜碱枝顶孢溶菌酶基因(表2)。表2:在第7天,比较里氏木霉宿主和单转录因子缺失菌株中的相对溶菌酶活性菌株转录因子基因缺失相对溶菌酶活性agjg216-2b51(宿主)-1.00qmji122-16b4-4tf708831.36qmji121-8d7-3tf929491.27实例13:在2升发酵中,比较里氏木霉单转录因子70883缺失菌株qmji122-16b4-4和里氏木霉双转录因子缺失菌株smai321-1c2-1中的嗜碱枝顶孢gh25溶菌酶生产力在2升发酵中,评估单转录因子70883缺失菌株里氏木霉qmji122-16b4-4和双转录因子缺失菌株里氏木霉smai321-1c2-1。使各菌株在30℃下在pda琼脂板上生长4-7天。将各自含有100ml摇瓶培养基的三个500ml摇瓶用来自pda琼脂板的两个塞子接种。将摇瓶在28℃在定轨摇床上以250rpm孵育48小时。将这些培养物用作种子进行发酵。使用共160ml的各种子培养物接种含有1.6升发酵分批培养基的3-升玻璃夹套发酵罐(欧谱生物技术公司(applikonbiotechnology))。将发酵罐维持在28℃的温度下,并且使用applikon1030控制系统将ph控制在3.5+/-0.1的设定值。将空气以2.5l/min的速率添加到容器中,并用以1100rpm旋转的rushton叶轮搅拌培养液。将由右旋糖和磷酸构成的发酵补料培养基以0至10g/l/小时的速率给予165小时。在发酵运行的第3、4、5、6和7天取样。将全培养液在4℃下储存。如实例11中所述,确定全培养液样品的嗜碱枝顶孢gh25溶菌酶表达水平。与单tf70883转录因子缺失菌株,里氏木霉qmji122-16b4-4相比,观察到具有两个转录因子基因缺失的菌株(其含有4拷贝的嗜碱枝顶孢溶菌酶基因)中嗜碱枝顶孢溶菌酶活性增加了1.25x(表3)。表3:在第7天,比较里氏木霉单转录因子70883缺失和双转录因子缺失菌株中的相对溶菌酶活性实例14:crispr-mad7骨架质粒psmai322a质粒psmai322a(seqidno:99,图3)是含有直肠真杆菌(eubacteriumrectale)mad7基因密码子的crispr-mad7表达质粒,该基因密码子针对具有在构巢曲霉tef1启动子和终止子的转录控制下的sv40nls序列的米曲霉进行了优化。此外,它具有包含稻梨孢菌(magnaportheoryzae)u6-2启动子、烟曲霉trnagly(gcc)1-6序列、wa前间隔子、直肠真杆菌单指导rna序列和稻梨孢菌u6-2终止子的wa-sgrna表达盒。实例15:用于crispr-mad7介导的转录因子基因92949和70883同时基因缺失的质粒pjfys331的构建构建质粒pjfys331,以在里氏木霉中表达mad7和单指导rna,以同时靶向转录因子基因92949和70883。构建质粒pjfys331的第一步是将靶向里氏木霉转录因子基因70883的20bp前间隔子区插入mad7表达质粒psmai322a(实例14)。将mad7表达质粒psmai322a用bglii线性化,并且通过使用tae缓冲液中的1%琼脂糖凝胶电泳进行纯化,其中将8,655bp片段从凝胶切离,并且使用凝胶清洁试剂盒进行提取。使用总体积为10μl,由1xhifi组装主混合物、50ng的线性化载体和0.05pmol的寡核苷酸构成的hifidna组装克隆试剂盒,用以下所示的含有靶向里氏木霉转录因子基因70883的20bp的前间隔子区和用于插入质粒psmai322a的侧翼同源序列的单链寡核苷酸引物1227488组装8,655bp片段。引物1227488:tttaatttctactcttgtagatgcattgtcaaagcatcgcccatttttttggctcttgggttc(seqidno:100)将混合物在50℃下孵育45分钟后,将2μl的反应转化到50μl的stellartm化学感受态大肠杆菌细胞中。将这些细胞在42℃下热振荡45秒,然后添加100μl的soc培养基,并且将总体积涂布在150mm2xyt+amp板上并且在37℃下孵育过夜。将所得大肠杆菌转化体单独地接种到在14ml圆底聚丙烯管中的3ml的lb+amp培养基中,并且在37℃下伴随200rpm的振荡而孵育过夜。使用9600分离质粒dna。使用染料终止物化学,用model377xl自动dna测序仪,通过dna测序确认插入。选择一个质粒并将其指定为pjfys331a。构建质粒pjfys331的第二步是在质粒pjfys331a中插入编码烟曲霉u6-3启动子的合成dna、烟曲霉trnagly序列、靶向里氏木霉转录因子基因92949的20bp的前间隔子区、化脓性链球菌单指导rna序列、烟曲霉u6-3终止子和侧翼同源序列。含有879bp的合成dna序列由(赛默飞世尔科技公司)合成为stringtmdna片段(seqidno:101)。将由提供的含有该片段的冻干dna以30ng/μl的浓度悬浮于去离子水中。将质粒pjfsy331a用paei线性化,并且通过使用tae缓冲液中的1%琼脂糖凝胶电泳进行纯化,其中将9,494bp片段从凝胶切离,并且使用凝胶清洁试剂盒进行提取。使用hifidna组装克隆试剂盒用含有本文所述的元件的stringtmdna片段(总体积为20μl,由1xhifi组装主混合物、100ng的线性化载体、和60ng的stringtmdna片段构成)来组装9,494bp片段。将混合物在50℃下孵育15分钟后,将2μl的反应转化到50μl的stellartm化学感受态大肠杆菌细胞中。将这些细胞在42℃下热振荡45秒,然后添加100μl的soc培养基,并且将总体积涂布在150mm2xyt+amp板上并且在37℃下孵育过夜。将所得大肠杆菌转化体单独地接种到在14ml圆底聚丙烯管中的3ml的lb+amp培养基中,并且在37℃下伴随200rpm的振荡而孵育过夜。使用9600分离质粒dna。使用染料终止物化学,用model377xl自动dna测序仪,通过dna测序确认插入。选择一个质粒并将其指定为pjfys331。实例16:用于转录因子(tf)基因92949缺失的无标记修复质粒pamfs-tf92949d的构建为了使用crispr-mad7以在缺失后可以去除选择标记的方式同时靶向两个转录因子基因(用于缺失),构建质粒pamfs-tf92949d,psmai321(实例7)的无hpt-tk选择标记版。在使用的crispr-mad7多基因缺失方案中,希望仅利用来自一种质粒的hpt和tk选择标记系统(即,psmai320将包含hpt-tk选择标记)。这样做是为了确保标记环出仅需要从单个基因座发生。因此,构建质粒pamfs-tf92949d以与psmai320一起用于多路方案中,以缺失转录因子基因92949和70883。用ncoi和xhoi消化质粒psmai321(实例7)以产生以下大小的线性dna片段:5,669bp、1,753bp和3,997bp。将5,669bp片段通过使用tae缓冲液中的0.8%琼脂糖凝胶电泳进行纯化,从凝胶切离,并使用凝胶清洁试剂盒进行提取。在含有420ngpsmai321的5,669bp片段的70μl反应中,将所得dna用dnapoli,大(克列诺)片段(新英格兰生物实验室公司)处理。将反应在25℃下孵育15分钟。使用pcr清洁试剂盒从此反应混合物中纯化dna。然后使用t4dna连接酶(新英格兰生物实验室公司)在含有从上一步纯化的60ngdna的20μl反应中将dna连接成环形。将反应在16℃下孵育约18小时。使用2μl的反应来转化50μl的stellartm化学感受态大肠杆菌细胞。将这些细胞在42℃下热振荡45秒,然后添加预热至42℃的450μl的soc培养基。将细胞在37℃下伴随200rpm的振荡孵育60分钟并且然后涂布到直径150mm的2xyt+amp板上并在37℃下孵育过夜。将所得大肠杆菌转化体单独地接种到在14ml圆底聚丙烯管中的3ml的lb+amp培养基中,并且在37℃下伴随200rpm的振荡而孵育过夜。使用9600分离质粒dna。使用染料终止物化学(giesecke等人,1992,同上),用model377xl自动dna测序仪,通过dna测序确认质粒。鉴定了含有插入且无pcr错误的一个质粒并将其指定为pamfs-tf92949d。实例17:用于转化和同源重组介导的转录因子基因70883或92949缺失的线性质粒dna的制备将共100μg的质粒psmai320(实例6)或质粒psmai321(实例7)在1.7mleppendorftm管中与25μl的bamhi和100μl的缓冲液(新英格兰生物实验室公司)组合,并用无菌水定容至1ml。将反应在37℃下孵育3小时。使用minelutetm柱(凯杰有限公司)纯化dna,其中将每个反应跨十个柱划分,用10μl无菌水/柱子dna洗脱。这产生了psmai320的线性11,390kbdna和psmai321的线性11,419kbdna。实例18:单转录因子(tf)92949缺失里氏木霉纤维素酶菌株的产生根据实例2来产生并转化里氏木霉纤维素酶菌株gmer61-a19的原生质体以缺失转录因子(tf)92949基因。将原生质体转移到15个圆底聚丙烯管中,并用5μg的bamhi-线性化的和凝胶纯化的psmai321(实例7)转化。在含有潮霉素b的pda板上选择原代转化体。将每个转化体转移到pda板上并且在30℃下孵育5天以产生孢子。为确认线性化psmai321dna的正确整合已通过同源重组发生,使用phiretm植物直接pcr试剂盒通过真菌孢子pcr来筛选原代分离株。将来自每个分离株的少量孢子重悬于20μl的稀释缓冲液中。将这些孢子悬浮液用作pcr中的模板,来筛选线性化psmai321的整合。pcr由以下构成:在10μl反应中的0.5μl的孢子悬浮液、50pmol的以下所示的每种引物、5ml的2xphiretm植物pcr缓冲液、和0.2ml的phiretm热启动iidna聚合酶。正向引物1227992:gaattcaaaagcgccagtcactgcgag(seqidno:102)反向引物1219245:ctacatcgaagctgaaagcacgaga(seqidno:103)反向引物1227993:tcgtcgagtcgaagatgagagaggatgg(seqidno:104)pcr在编程为如下的热循环仪中进行:1个循环,在98℃下持续5分钟;40个循环,每个循环在98℃下持续10秒、在65℃下持续10秒并在72℃下持续1分30秒;以及1个循环,在72℃下持续1分钟。通过使用tae缓冲液中的1%琼脂糖凝胶电泳来分析完成的pcr。选择具有正确诊断dna条带的原代分离株用于单孢子纯化。在去除hpt选择标记基因之前,进行一次单孢子分离以确保菌株的纯度。将来自每个分离株的少量孢子悬浮于1ml的无菌水中。将每个孢子溶液的1μl等分试样与50μl的无菌水溶液混合,并涂布在补充有1m蔗糖的150mmpda板上。将板在30℃下孵育3-4天。将来自pda蔗糖板的单个菌落转移至pda板上并在30℃下孵育5-7天。为确认线性化psmai321dna的正确整合已通过同源重组发生,使用如上所述的phiretm植物直接pcr试剂盒通过真菌孢子pcr来筛选分离株。选择具有正确诊断dna条带的分离株用于选择标记环出。将孢子从已在30℃下孵育5-7天的pda板中收集到无菌去离子水中。计数孢子并将不同量的孢子(1x107、1x106、1x105)涂布在含有1.5μm5-氟-2’-脱氧尿苷(fdu)的木霉属基本培养基板上,并在30℃下孵育5天。将来自木霉属基本培养基fdu板上的单个菌落转移至pda板上并在30℃下孵育5-7天。为确认hpt-tk选择标记已经通过侧翼于这些标记的重复的同源重组环出菌株,使用phiretm植物直接pcr试剂盒通过真菌孢子pcr筛选分离株。将来自每个分离株的少量孢子重悬于20μl的稀释缓冲液中。将这些孢子悬浮液用作pcr中的模板来筛选标记环出。pcr由以下构成:在10μl反应中的0.5μl的孢子悬浮液、50pmol的以下所示的每种引物、5ml的2xphiretm植物pcr缓冲液、和0.2ml的phiretm热启动iidna聚合酶。正向引物1213333:gggacgccctgctgcaacttacc(seqidno:105)正向引物1227992:gaattcaaaagcgccagtcactgcgag(seqidno:106)反向引物1225982:gagacgagactggagtcgttgccgc(seqidno:107)pcr在编程为如下的热循环仪中进行:1个循环,在98℃下持续5分钟;40个循环,每个循环在98℃下持续10秒并在72℃下持续1分40秒;以及1个循环,在72℃下持续2分钟。通过使用tae缓冲液中的1%琼脂糖凝胶电泳来分析完成的pcr。选择具有正确诊断dna条带的分离株用于单孢子纯化,随后是如上所述的真菌孢子pcr。鉴定具有正确诊断dna条带的tf92949基因缺失且不存在hpt和tk选择标记基因的两个分离株。如实例1中所述的制备基因组dna,并且在nextseqtm500中使用2x150bp化学进行测序。测序鉴定转化体里氏木霉gmer62-dtf92949-5a1b和gmer62-dtf92949-10a1d含有tf92949基因缺失并且不存在hpt和tk选择标记基因。实例19:单转录因子(tf)70883缺失里氏木霉纤维素酶菌株的产生根据实例2来产生并转化里氏木霉纤维素酶菌株gmer61-a19的原生质体以缺失转录因子(tf)70883基因。将原生质体转移到15个圆底聚丙烯管中,并用5μg的bamhi-线性化的和凝胶纯化的psmai320(实例6)转化。在含有潮霉素b的pda板上选择原代转化体。将每个转化体转移到pda板上并且在30℃下孵育5天以产生孢子。为确认线性化psmai320dna的正确整合已通过同源重组发生,使用phiretm植物直接pcr试剂盒通过真菌孢子pcr来筛选原代分离株。将来自每个分离株的少量孢子重悬于20μl的稀释缓冲液中。将这些孢子悬浮液用作pcr中的模板,来筛选线性化psmai320的整合。pcr由以下构成:在10μl反应中的0.5μl的孢子悬浮液、50pmol的以下所示的每种引物、5ml的2xphiretm植物pcr缓冲液、和0.2ml的phiretm热启动iidna聚合酶。正向引物1225977:tgaccgggcaggggatcgcc(seqidno:108)反向引物1219245:ctacatcgaagctgaaagcacgaga(seqidno:109)反向引物1225978:ctggggcgtcaagggacctgaatg(seqidno:110)pcr在编程为如下的热循环仪中进行:1个循环,在98℃下持续5分钟;40个循环,每个循环在98℃下持续10秒、在65℃下持续10秒并在72℃下持续1分30秒;以及1个循环,在72℃下持续1分钟。通过使用tae缓冲液中的1%琼脂糖凝胶电泳来分析完成的pcr。选择具有正确诊断dna条带的原代分离株用于单孢子纯化。在去除hpt选择标记基因之前,进行一次单孢子分离以确保菌株的纯度。将来自每个分离株的少量孢子悬浮于1ml无菌水中。将每个孢子溶液的1μl等分试样与50μl的无菌水溶液混合,并涂布在补充有1m蔗糖的150mmpda板上。将板在30℃下孵育3-4天。将来自pda蔗糖板的单个菌落转移至pda板上并在30℃下孵育5-7天。为确认线性化psmai320dna的正确整合已通过同源重组发生,使用如上所述的phiretm植物直接pcr试剂盒通过真菌孢子pcr来筛选分离株。选择具有正确诊断dna条带的分离株用于选择标记环出。将孢子从已在30℃下孵育5-7天的pda板中收集到无菌去离子水中。计数孢子并将不同量的孢子(1x107、1x106、1x105)涂布在含有1.5μm5-氟-2’-脱氧尿苷(fdu)的木霉属基本培养基板上,并在30℃下孵育5天。将来自木霉属基本培养基fdu板上的单个菌落转移至pda板上并在30℃下孵育5-7天。为确认hpt和tk选择标记基因已经通过侧翼于这些标记的重复的同源重组环出菌株,使用phiretm植物直接pcr试剂盒通过真菌孢子pcr筛选分离株。将来自每个分离株的少量孢子重悬于20μl的稀释缓冲液中。将这些孢子悬浮液用作pcr中的模板来筛选标记环出。pcr由以下构成:在10μl反应中的0.5μl的孢子悬浮液、50pmol的以下所示的每种引物、5ml的2xphiretm植物pcr缓冲液、和0.2ml的phiretm热启动iidna聚合酶。正向引物1213333:gggacgccctgctgcaacttacc(seqidno:111)正向引物1225977:tgaccgggcaggggatcgcc(seqidno:112)反向引物1225979:cgcccttcgacgagtcggcac(seqidno:113)pcr在编程为如下的热循环仪中进行:1个循环,在98℃下持续5分钟;40个循环,每个循环在98℃下持续10秒并在72℃下持续1分40秒;以及1个循环,在72℃下持续2分钟。通过使用tae缓冲液中的1%琼脂糖凝胶电泳来分析完成的pcr。选择具有正确诊断dna条带的分离株用于单孢子纯化,随后是如上所述的真菌孢子pcr。鉴定具有正确诊断dna条带的tf92949基因缺失且不存在hpt和tk选择标记基因的两个分离株。如实例1中所述的制备基因组dna,并且在nextseqtm500中使用2x150bp化学进行测序。测序鉴定转化体里氏木霉gmer62-dtf70883-8e1d和gmer62-dtf70883-11e1a含有tf70883基因缺失并且不存在hpt和tk选择标记基因。实例20:用于转化和crispr介导的转录因子基因70883和92949的基因缺失的线性质粒dna的制备将共20μg的质粒pamfs-tf92949d(实例16)或质粒psmai320(实例6)在1.7mleppendorftm管中与10μl的pmei和20μl的缓冲液组合,并用无菌水定容至200μl。将反应在37℃下孵育3小时。使用minelutetm柱纯化dna,其中将反应跨两个柱划分,用10μl无菌水/柱子dna洗脱。这产生了pamfs-tf92949d的线性2.977kb和2.688kbdna和psmai320的线性8.702kb和2.688kbdna。实例21:双转录因子(tf)缺失纤维素酶里氏木霉菌株的产生根据实例2来产生里氏木霉纤维素酶菌株gmer61-a19的原生质体。对于每次转化,将500μl的原生质体添加至50ml管中,并与1250μl的peg溶液轻轻混合。然后将共16μg的质粒dna添加至原生质体悬浮液(5μg的psmai320pmei线性化dna;5μg的pamfs-tf92949dpmei线性化dna;6μg的pjfys331环状质粒dna)。将转化在34℃下孵育30分钟。将转化用15ml的stc稀释并通过颠倒管几次来轻轻混合。将每个转化均匀地涂布在含有补充有1m蔗糖的pda的十个板上。在34℃孵育平板。在约24小时后,用含有35μg/ml潮霉素b的20ml的pda+1m蔗糖覆盖板。然后将板在30℃下孵育5天。将来自转化板的原代分离株转移至pda板上并在30℃下孵育5-7天。为确认线性化psmai320和pamfs-tf92949dna的正确整合已通过crispr介导的基因替代整合,使用phiretm植物直接pcr试剂盒通过真菌孢子pcr来筛选原代分离株。将来自每个分离株的少量孢子重悬于20μl的稀释缓冲液中。将这些孢子悬浮液用作pcr中的模板,来筛选在目的基因处的线性化的psmai320和pamfs-tf92949d的整合。pcr由以下构成:在10μl反应中的0.5μl的孢子悬浮液、50pmol的以下所示的每种引物(每个pcr中3种引物)、5ml的2xphiretm植物pcr缓冲液、和0.2ml的phiretm热启动iidna聚合酶。tf92949基因座的pcr筛选:正向引物1227992:gaattcaaaagcgccagtcactgcgag(seqidno:114)反向引物1219245:ctacatcgaagctgaaagcacgaga(seqidno:115)反向引物1227993:tcgtcgagtcgaagatgagagaggatgg(seqidno:116)tf70883基因座的pcr筛选:正向引物1225977:tgaccgggcaggggatcgcc(seqidno:117)反向引物1219245:ctacatcgaagctgaaagcacgaga(seqidno:118)反向引物1225978:ctggggcgtcaagggacctgaatg(seqidno:119)pcr在编程为如下的热循环仪中进行:1个循环,在98℃下持续5分钟;40个循环,每个循环在98℃下持续10秒、在65℃下持续10秒并在72℃下持续1分30秒;以及1个循环,在72℃下持续1分钟。通过使用tae缓冲液中的1%琼脂糖凝胶电泳来分析pcr。选择针对两个诊断pcr反应具有正确诊断dna条带的原代分离株用于单孢子纯化。在去除hpt选择标记基因之前,进行一次单孢子分离以确保菌株的纯度。将来自每个分离株的少量孢子悬浮于1ml无菌水中。将每个孢子溶液的1μl等分试样与50μl的无菌水溶液混合,并涂布在补充有1m蔗糖的150mmpda板上。将板在30℃下孵育3-4天。将来自pda蔗糖板的单个菌落转移至pda板上并在30℃下孵育5-7天。为确认通过crispr介导的基因替代的线性化psmai320和pamfstf-92949dna的正确整合,使用如上所述的phiretm植物直接pcr试剂盒通过真菌孢子pcr来筛选分离株。选择针对两个诊断pcr反应具有正确诊断dna条带的分离株用于选择标记环出。将孢子从已在30℃下孵育5-7天的pda板中收集到无菌去离子水中。计数孢子并将不同量的孢子(1x107、1x106、1x105)铺板于含有1.5μm5-氟-2’-脱氧尿苷(fdu)的木霉属基本培养基板上,并在30℃下孵育5天。将来自木霉属基本培养基fdu板上的单个菌落转移至pda板上并在30℃下孵育5-7天。为确认hpt和tk选择标记基因已经通过侧翼于这些标记的重复的同源重组环出tf70883基因座,使用phiretm植物直接pcr试剂盒通过真菌孢子pcr筛选分离株。将来自每个分离株的少量孢子重悬于20μl的稀释缓冲液中。将这些孢子悬浮液用作pcr中的模板来筛选标记环出。pcr由以下构成:在10μl反应中的0.5μl的孢子悬浮液、50pmol的以下所示的每种引物(每个pcr中3种引物)、5ml的2xphiretm植物pcr缓冲液、和0.2ml的phiretm热启动iidna聚合酶。正向引物1213333:gggacgccctgctgcaacttacc(seqidno:120)正向引物1225977:tgaccgggcaggggatcgcc(seqidno:121反向引物1225979:cgcccttcgacgagtcggcac(seqidno:122pcr在编程为如下的热循环仪中进行:1个循环,在98℃下持续5分钟;40个循环,每个循环在98℃下持续10秒并在72℃下持续1分40秒;以及1个循环,在72℃下持续2分钟。通过使用tae缓冲液中的1%琼脂糖凝胶电泳来分析pcr。选择具有正确诊断dna条带的分离株用于单孢子纯化。在发酵中测试前,再次进行单孢子分离以确保菌株的纯度。将来自每个分离株的少量孢子悬浮于1ml无菌水中。将每个孢子溶液的1μl等分试样与50μl的无菌水溶液混合,并涂布在补充有1m蔗糖的150mmpda板上。将板在30℃下孵育3-4天。将来自补充有1m蔗糖板的pda板的单个菌落转移至pda板上并在30℃下孵育5-7天。为确认hpt和tk选择标记基因已经通过侧翼于这些标记的重复的同源重组环出菌株,使用如上所述的phiretm植物直接pcr试剂盒通过真菌孢子pcr筛选分离株。鉴定具有正确诊断dna条带的tf70883基因缺失且不存在hpt和tk选择标记基因的两个分离株。如实例1中所述的制备基因组dna,并且在nextseqtm500中使用2x150bp化学进行测序。测序鉴定转化体里氏木霉gmertfdd4d1b和里氏木霉gmertfdd7a1b含有tf70883和tf92949基因缺失并且不存在hpt和tk选择标记基因。实例22:评估摇瓶培养中转录因子缺失菌株的纤维素酶减表型的发展对于每种转录因子缺失菌株里氏木霉gmer62-dtf92949-5a1b和里氏木霉gmer62-dtf92949-10a1d(实例18)、里氏木霉gmer62-dtf70883-8e1d和里氏木霉gmer62-dtf70883-11e1a(实例19)、以及里氏木霉gmertfdd4d1b和里氏木霉gmertfdd7a1b(实例21),将孢子悬浮在0.8%的nacl,0.05%的20溶液中并且用血细胞计数器计数,将2x107个孢子一式三份地接种到125ml烧瓶中补充有0.1%的蛋白胨和1%的乳糖的20mlmandels-andreotti培养基中。将来自里氏木霉gmer62-1a9(亲本菌株)培养物的孢子用作对照。将培养物在30℃下以160rpm孵育5天。5天后,将每种培养液的10μl等分试样与2xtcep(0.05m三(2-羧乙基)膦盐酸盐、65.8mmtris-盐酸盐、26.3%甘油和2.1%十二烷基硫酸钠)混合,并在98℃下加热10分钟。冷却后,将样品装入8%-16%tgx凝胶(伯乐实验室有限公司)并且使用tris-甘氨酸缓冲系统在150v下运行直到蓝色染料到达凝胶底部。将凝胶在水中短暂洗涤并用instantbluetm(艾本德蛋白溶液公司(expedeonproteinsolutions))染色1小时。通过比较重组菌株的纤维二糖水解酶i(cbhi)和纤维二糖水解酶ii(cbhii)蛋白质的表达水平与对照菌株里氏木霉gmer62-1a9的模式来目测评估每种培养物的sds-page蛋白质模式。sds-page分析显示,在补充有0.1%的蛋白胨和1%的乳糖的mandels-andreotti培养基中培养后,重组菌株具有比原始宿主菌株更稳定的蛋白质表达模式。这些菌株始终保留cbhi和cbhii的表达。实例23:脱盐的bca测定在使用econo-10dg脱盐柱(伯乐实验室有限公司)定量脱盐后,测量里氏木霉发酵样品的蛋白质浓度。将3ml过滤培养液装入用50mm醋酸钠(ph5.0)和150mm氯化钠通过重力平衡的脱盐柱并用4ml平衡缓冲液洗脱。然后使用bca蛋白质测定试剂盒(皮尔斯公司(pierce))测量里氏木霉发酵样品的蛋白质浓度。实例24:比较里氏木霉单转录因子70883缺失菌株、单转录因子92949缺失菌株和双转录因子缺失菌株在2升发酵中的总蛋白质滴度在2升发酵中多次评估单转录因子70883缺失菌株里氏木霉gmer62-dtf70883-8e1d和gmer62-dtf70883-11e1a(实例19)、单转录因子92949缺失菌株里氏木霉gmer62-dtf92949-5a1b和gmer62-dtf92949-10a1d(实例18)、双转录因子缺失菌株里氏木霉gmertfdd4d1b和gmertfdd7a1b(实例21)以及对照菌株里氏木霉gmer62-1a9。使各菌株在30℃下在pda琼脂板上生长4-7天。将各自含有100ml摇瓶培养基的三个500ml摇瓶用来自pda琼脂板的两个塞子接种。将摇瓶在28℃在定轨摇床上以250rpm孵育48小时。将这些培养物用作种子进行发酵。使用共160ml的各种子培养物接种含有1.6升发酵分批培养基的3升玻璃夹套发酵罐(欧谱生物技术公司)。将发酵罐维持在28℃的温度下,并且使用applikon1030控制系统(欧谱生物技术公司)将ph控制在3.5+/-0.1的设定值。将空气以2.5l/分钟的速率添加到容器中,并用以1100rpm旋转的rushton叶轮搅拌培养液。将由右旋糖和磷酸构成的发酵补料培养基以0至10g/l/小时的速率给予165小时。样品在发酵运行结束时(第7天)获取,离心并过滤。如实例23中所述,在第7天的发酵样品过滤上清液中确定总蛋白质滴度。平均而言,与对照菌株里氏木霉gmer62-1a9相比,观察到具有tf70883缺失的菌株中蛋白质滴度增加了1.11x(表4)。具有单个tf92949缺失的菌株具有与对照菌株里氏木霉gmer62-1a9相似的蛋白质滴度。当单个菌株中缺失两个转录因子基因时,平均而言,与里氏木霉gmer62-1a9相比,观察到蛋白质滴度增加了1.17x(表4)。表4:在第7天,比较里氏木霉gmer62-1a9和转录因子缺失菌株中的相对蛋白质滴度实例25:评估2l发酵罐中转录因子缺失菌株的纤维素酶减表型的发展从里氏木霉gmer62-1a9(对照)、里氏木霉gmer62-dtf70883-11e1a、里氏木霉gmer62-dtf92949-5a1b、和里氏木霉gmertfdd7a1b的2l发酵罐中在第1、3、5、和7天收集一毫升原菌丝体用于在cmc板上分析,以确定纤维素酶减表型的发展动态。将第3、5和7天的菌丝体样品在0.8%nacl、0.05%20溶液中按1:5稀释。未稀释第1天的样品。对每个样品,将40μl等分试样涂布在150mmmex板上。然后将板在30℃下孵育5-7天以允许孢子形成。在5-7天后,将来自mex板的孢子悬浮在0.8%nacl,0.05%20溶液中,用血细胞计数器计数,并将约200个孢子铺板于cmc板上。将板在30℃下孵育2-3天以允许单个菌落生长。为激活真菌纤维素酶并杀死真菌,将板在50℃下孵育3-4小时。计数每个板上的菌落,然后将板用10ml刚果红溶液染色15分钟,并且用1mnacl洗涤10分钟。计数未染色和/或具有透明晕圈的菌落以确定仍活性产生纤维素酶的菌落的百分比。cmc板上里氏木霉gmer62-1a9发酵的表型分析显示,产生纤维素酶的菌落的数目随时间急剧下降。在第3天,在cmc板上的所有菌落的仅50%显示纤维素酶产生并且在第7天,在cmc板上的仅6%的菌落显示任何纤维素酶产生的迹象。另一方面,从第2天到第7天,来自菌株里氏木霉gmer62-dtf70883-11e1a(转录因子基因70883缺失)、里氏木霉gmer62-dtf92949-5a1b(转录因子基因92949缺失)、和里氏木霉gmertfdd7a1b(转录因子基因70883和92949双缺失)的菌落具有平均83%的纤维素酶产生型菌落(图4)。实例26:里氏木霉转录因子37062基因缺失质粒pbtp01的构建构建质粒pbtp01以缺失里氏木霉gmer62-1a9中的转录因子(tf)37062基因。为了构建里氏木霉tf37062基因缺失盒,使用以下所示的引物1227760和1227761来pcr扩增含有里氏木霉tf37062基因(dna序列为seqidno:123并且推导的氨基酸序列为seqidno:124)的上游非编码区的1900bp片段的pcr产物(dna片段1)。正向引物1227760:gagtcgacctgcaggcatgcttaattaacaattcctcgtgacagtttctgc(seqidno:125)反向引物1227761:cttgctcggtcctggcgtagacttatcacaaagttagccaaacagg(seqidno:126)pcr由以下构成:约180ng的里氏木霉btr213基因组dna、20mm的dntp、100pmol的正向引物1227760、100pmol的反向引物1227761、1xhf缓冲液、和2单位的热启动dna聚合酶,最终体积为100μl。pcr在编程为如下的热循环仪中孵育:1个循环,在98℃下持续3分钟;30个循环,各在98℃下持续10秒,58℃下持续30秒、以及72℃下持续2分钟;1个循环,在72℃下持续10分钟;以及在10℃下保持。将所得1949bppcr片段通过使用tae缓冲液中的0.8%琼脂糖凝胶电泳进行纯化,从凝胶切离,并使用凝胶和pcr清洁试剂盒进行提取。使用以下所示的引物1227758和1227759来pcr扩增含有编码乙酰胺酶选择标记的构巢曲霉amds基因的pcr产物(dna片段2)。正向引物1227758:tctacgccaggaccgagcaa(seqidno:127)反向引物1227759:tggaaacgcaaccctgaagg(seqidno:128)pcr由以下构成:约10ng的质粒pallo107、含有编码乙酰胺酶选择标记基因的构巢曲霉amds基因的克隆质粒、20mm的dntp、100pmol的正向引物1227758、10pmol的反向引物1227759、1xhf缓冲液、和2单位的热启动dna聚合酶,最终体积为100μl。pcr在编程为如下的热循环仪中孵育:1个循环,在98℃下持续3分钟;30个循环,各在98℃下持续10秒,58℃下持续30秒、以及72℃下持续2分钟;1个循环,在72℃下持续10分钟;以及在10℃下保持。将所得2718bppcr片段通过使用tae缓冲液中的0.8%琼脂糖凝胶电泳进行纯化,从凝胶切离,并使用凝胶和pcr清洁试剂盒进行提取。使用以下所示的引物1227762和1227763来pcr扩增含有里氏木霉tf37062基因的下游非编码区的1878bp片段的pcr产物(dna片段3)。正向引物1227762:tcccttcagggttgcgtttccatgaactaccagcatacacgac(seqidno:129)反向引物1227763:acagctatgaccatgattacgcctccttgtttgatcctagccc(seqidno:130)pcr由以下构成:约180ng的里氏木霉btr213基因组dna、20mm的dntp、100pmol的正向引物1227762、100pmol的反向引物1227763、1xhf缓冲液、和2单位的热启动dna聚合酶,最终体积为100μl。pcr在编程为如下的热循环仪中孵育:1个循环,在98℃下持续3分钟;30个循环,各在98℃下持续10秒,58℃下持续30秒、以及72℃下持续2分钟;1个循环,在72℃下持续10分钟;以及在10℃下保持。将所得1923bppcr片段通过使用tae缓冲液中的0.8%琼脂糖凝胶电泳进行纯化,从凝胶切离,并使用凝胶和pcr清洁试剂盒进行提取。将质粒puc19(新英格兰生物实验室公司(newenglandbiolabsinc.))用hindiii进行酶切,并且通过使用tae缓冲液中的0.8%琼脂糖凝胶电泳进行纯化,其中将2,686bp片段从凝胶切离,并且使用凝胶和pcr清洁试剂盒进行提取。使用hifidna组装克隆试剂盒用以上所述的三种pcr产物(dna片段1、2和3)(总体积为20μl,由1xhifi组装主混合物和0.05pmol的每种pcr产物构成)来组装2,686bp片段。将反应在50℃下孵育60分钟,然后置于冰上。使用2μl的反应来转化50μl的stellartm化学感受态大肠杆菌细胞。将这些细胞在42℃下热振荡45秒,然后添加预热至42℃的450μl的soc培养基。将细胞在37℃下伴随200rpm的振荡孵育60分钟并且然后涂布到直径150mm的2xyt+amp板上并在37℃下孵育过夜。将所得大肠杆菌转化体单独地接种到在14ml圆底聚丙烯管中的3ml的lb+amp培养基中,并且在37℃下伴随200rpm的振荡而孵育过夜。将质粒dna使用9600分离并通过ndei消化筛选正确插入的片段。通过用model377xl自动dna测序仪,使用染料终止子化学(giesecke等人,1992,如上)的dna测序确认产生所希望带大小的质粒(5836bp+3348bp)。鉴定了含有插入且无pcr错误的一种质粒并将其指定为pbtp01(图5)。实例27:转录因子(tf)37062缺失里氏木霉菌株btp1-bb1的产生根据实例2来产生并转化里氏木霉纤维素酶菌株gmer62-1a9的原生质体以缺失转录因子(tf)37062基因。将原生质体转移到15个圆底聚丙烯管中,并用4μg的paci-线性化的和凝胶纯化的pbtp01(实例26)转化。将四个转化体转移到cove2板上并且在30℃下孵育5天以产生孢子。针对乙酰胺酶(amds)标记同源整合至tf37062基因座,使用phiretm植物直接pcr试剂盒通过真菌孢子pcr方法来筛选里氏木霉gmer62-1a9的转化体。将来自每个转化体的少量孢子悬浮于20μl的稀释缓冲液(phiretm植物直接pcr试剂盒)中。将这些孢子悬浮液用作pcr中的模板,来筛选tf37062基因缺失。每个反应由以下构成:在20μl反应中的1.5μl的孢子悬浮液、10pmol的以下所示的每种引物(每个pcr中3种引物)、10μl的2xphiretm植物pcr缓冲液、和0.2μl的phiretm热启动iidna聚合酶。tf37062基因座5'末端的pcr筛选:正向引物1228448:atgcccagtcgcgaataatcactcagcc(seqidno:131)反向引物1228459:ggctgagtagtgctgccattgggtg(seqidno:132)反向引物1228447:ccataaggtggcgttgttacatctccctgagag(seqidno:133)tf37062基因座3'末端的pcr筛选:正向引物1228551:ccgtcctcggtcaggagccttgg(seqidno:134)正向引物1228564:cttacttcttcaaatccagtcatggttggcctgtg(seqidno:135)反向引物1228552:cccctatcctccttgccgtcttgctttg(seqidno:136)pcr在编程为如下的热循环仪中孵育:1个循环,在98℃下持续5分钟;40个循环,各在98℃下持续5秒以及在72℃下持续70秒;1个循环,在72℃下持续1分钟;以及在4℃下保持。通过使用tae缓冲液中的1%琼脂糖凝胶电泳来分析完成的pcr。将pbtp01构建体正确靶向tf37062基因座的转化体产生了用于5’末端和3’末端pcr的3kbpcr片段。选择产生正确pcr片段的一个转化体(里氏木霉btp1-b)用于单孢子分离。在5ml的0.01%20溶液中收集来自6天时长cove2板的少量孢子。将孢子溶液的2μl等分试样与100μl0.01%20溶液混合,并涂布在150mmcove板上。将板在30℃下孵育5-7天。将单个菌落转移至pda板上并在30℃下孵育5-7天。如上所述,利用真菌孢子pcr来鉴定具有正确靶向tf37062基因座的孢子分离株。显示标记基因的正确切除的分离株经历另一轮单次分离,随后是如上所述的真菌孢子pcr。如实例1中所述的制备基因组dna,并且在nextseqtm500中使用2x150bp化学进行测序。测序鉴定转化体里氏木霉btp1-bb1含有tf37062基因缺失。实例28:评估摇瓶培养中转录因子缺失菌株的纤维素酶减表型的发展将tf37062缺失菌株里氏木霉btp-bb1(实例27)的孢子悬浮在0.8%的nacl,0.05%的20溶液中并且用血细胞计数器计数,且将2x107个孢子一式三份地接种到125ml烧瓶中补充有0.1%的蛋白胨和1%的乳糖的20mlmandels-andreotti培养基中。将来自里氏木霉gmer62-1a9培养物的孢子用作对照。将培养物在30℃下以160rpm孵育5天。5天后,将每种培养液的10μl等分试样与2xtcep(0.05m三(2-羧乙基)膦盐酸盐、65.8mmtris-盐酸盐、26.3%甘油和2.1%十二烷基硫酸钠)混合,并在98℃下加热10分钟。冷却后,将样品装入8%-16%tgx凝胶并且使用tris-甘氨酸缓冲系统在150v下运行直到蓝色染料到达凝胶底部。将凝胶在水中短暂洗涤并用instantbluetm染色1小时。通过比较重组菌株的纤维二糖水解酶i(cbhi)和纤维二糖水解酶ii(cbhii)蛋白质的表达水平与对照菌株里氏木霉gmer62-1a9的模式来目测评估每种培养物的sds-page蛋白质模式。sds-page分析显示,在补充有0.1%的蛋白胨和1%的乳糖的mandels-andreotti培养基中培养后,里氏木霉btp-bb1具有比里氏木霉gmer62-1a9更稳定的蛋白质表达模式。里氏木霉btp-bb1始终保持cbhi和cbhii的表达。实例29:在2升发酵中,比较里氏木霉gmer62-1a9和转录因子37062缺失菌株btp1-bb1中的总蛋白质滴度在2升发酵中多次评估转录因子37062缺失菌株里氏木霉btp1-bb1(实例27)和对照菌株里氏木霉gmer62-1a9。使各菌株在30℃下在pda琼脂板上生长4-7天。将各自含有100ml摇瓶培养基的三个500ml摇瓶用来自pda琼脂板的两个塞子接种。将摇瓶在28℃在定轨摇床上以250rpm孵育48小时。将这些培养物用作种子进行发酵。使用共160ml的各种子培养物接种含有1.6升发酵分批培养基的3升玻璃夹套发酵罐(欧谱生物技术公司)。将发酵罐维持在28℃的温度下,并且使用applikon1030控制系统(欧谱生物技术公司)将ph控制在3.5+/-0.1的设定值。将空气以2.5l/分钟的速率添加到容器中,并用以1100rpm旋转的rushton叶轮搅拌培养液。将由右旋糖和磷酸构成的发酵补料培养基以0至10g/l/小时的速率给予165小时。样品在发酵运行结束时(第7天)获取,离心并过滤。如实例23中所述,在第7天的发酵样品过滤上清液中确定总蛋白质滴度。平均而言,与对照菌株里氏木霉gmer62-1a9相比,观察到具有tf37062缺失的菌株中蛋白质滴度增加了1.07x(表5)。表5:在第7天,比较里氏木霉菌株gmer62-1a9和转录因子缺失菌株btp1-bb1中的相对蛋白质滴度本发明通过以下编号的段落来进一步描述:段落1.一种亲本丝状真菌细胞的分离的突变体,该分离的突变体包含目的多肽的在启动子的转录控制下的编码序列,该启动子通过选自由以下组成的组的一种或多种转录因子调节:(a)包含氨基酸序列的转录因子,该氨基酸序列与seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、或124具有至少70%序列同一性;(b)由多核苷酸编码的转录因子,该多核苷酸包含与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123具有至少70%序列同一性的核苷酸序列;以及(c)由多核苷酸编码的转录因子,该多核苷酸在高严格条件下与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123的全长互补序列杂交;其中将该一种或多种转录因子基因在亲本丝状真菌细胞中修饰以产生该突变体,使得该突变体在该一种或多种转录因子的产生方面有部分或完全缺陷,其中(i)当在与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相同的条件下培养时,该一种或多种转录因子基因的修饰提高了该突变体在该目的多肽的产生方面的生产力,(ii)与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相比,该一种或多种转录因子基因的修饰减少或消除了所得突变体中的纤维素酶阴性表型,或(iii)该一种或多种转录因子基因的修饰导致(i)和(ii)的组合。段落2.如段落1所述的突变体,其中该转录因子包含氨基酸序列,该氨基酸序列与seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、或124具有至少80%序列同一性。段落3.如段落1所述的突变体,其中该转录因子包含氨基酸序列,该氨基酸序列与seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、或124具有至少85%序列同一性。段落4.如段落1所述的突变体,其中该转录因子包含氨基酸序列,该氨基酸序列与seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、或124具有至少90%序列同一性。段落5.如段落1所述的突变体,其中该转录因子包含氨基酸序列,该氨基酸序列与seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、或124具有至少95%序列同一性。段落6.如段落1所述的突变体,其中该转录因子包含以下或由以下组成:seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、或124。段落7.如段落1所述的突变体,其中该转录因子由多核苷酸编码,该多核苷酸包含与1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123具有至少80%序列同一性的核苷酸序列。段落8.如段落1所述的突变体,其中该转录因子由多核苷酸编码,该多核苷酸包含与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123具有至少85%序列同一性的核苷酸序列。段落9.如段落1所述的突变体,其中该转录因子由多核苷酸编码,该多核苷酸包含与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123具有至少90%序列同一性的核苷酸序列。段落10.如段落1所述的突变体,其中该转录因子由多核苷酸编码,该多核苷酸包含与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123具有至少95%序列同一性的核苷酸序列。段落11.如段落1所述的突变体,其中该转录因子由多核苷酸编码,该多核苷酸包含以下或由以下组成:seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123。段落12.如段落1所述的突变体,其中该转录因子由多核苷酸编码,该多核苷酸在非常高严格条件下与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123的全长互补序列杂交。段落13.如段落1-12中任一项所述的突变体,其中该启动子是来自纤维素酶基因的启动子。段落14.如段落13所述的突变体,其中该纤维素酶基因是纤维二糖水解酶基因。段落15.如段落14所述的突变体,其中该纤维二糖水解酶基因是纤维二糖水解酶i基因。段落16.如段落15所述的突变体,其中该纤维二糖水解酶i基因是木霉属纤维二糖水解酶i基因。段落17.如段落16所述的突变体,其中该纤维二糖水解酶i基因是里氏木霉纤维二糖水解酶i基因。段落18.如段落14所述的突变体,其中该纤维二糖水解酶基因是纤维二糖水解酶ii基因。段落19.如段落18所述的突变体,其中该纤维二糖水解酶ii基因是木霉属纤维二糖水解酶ii基因。段落20.如段落19所述的突变体,其中该纤维二糖水解酶ii基因是里氏木霉纤维二糖水解酶ii基因。段落21.如段落1-20中任一项所述的突变体,其中该启动子对于该目的多肽的编码序列是天然的。段落22.如段落1-20中任一项所述的突变体,其中该启动子对于该目的多肽的编码序列是异源的。段落23.如段落1-22中任一项所述的突变体,其中该目的多肽对于该亲本丝状真菌细胞或其突变体是天然的。段落24.如段落1-22中任一项所述的突变体,其中该目的多肽对于该亲本丝状真菌细胞或其突变体是异源的。段落25.如段落1-24中任一项所述的突变体,其中该目的多肽是抗体、抗原、抗微生物肽、酶、生长因子、激素、免疫调节剂、神经递质、受体、报告蛋白、结构蛋白或转录因子。段落26.如段落1-24中任一项所述的突变体,其中该目的多肽是纤维素酶。段落27.如段落26所述的突变体,其中该纤维素酶是内切葡聚糖酶、纤维二糖水解酶、以及β-葡糖苷酶。段落28.如段落1-24中任一项所述的突变体,其中该目的多肽是半纤维素酶。段落29.如段落28所述的突变体,其中该半纤维素酶是木聚糖酶、乙酰木聚糖酯酶、阿魏酸酯酶、阿拉伯呋喃糖苷酶、木糖苷酶和葡糖醛酸糖苷酶。段落30.如段落1-29中任一项所述的突变体,其中该亲本丝状真菌细胞是枝顶孢属(acremonium)、曲霉属(aspergillus)、短梗霉属(aureobasidium)、烟管霉属(bjerkandera)、拟腊菌属(ceriporiopsis)、金孢子菌属(chrysosporium)、鬼伞属(coprinus)、革盖菌属(coriolus)、隐球菌属(cryptococcus)、线黑粉菌科(filibasidium)、镰孢属(fusarium)、腐质霉属(humicola)、梨孢菌属(magnaporthe)、毛霉属(mucor)、毁丝霉属(myceliophthora)、新美鞭菌属(neocallimastix)、脉胞菌属(neurospora)、拟青霉属(paecilomyces)、青霉属(penicillium)、平革菌属(phanerochaete)、射脉菌属(phlebia)、瘤胃壶菌属(piromyces)、侧耳属(pleurotus)、裂褶菌属(schizophyllum)、篮状菌属(talaromyces)、嗜热子囊菌属(thermoascus)、梭孢壳属(thielavia)、弯颈霉属(tolypocladium)、栓菌属(trametes)、或木霉属(trichoderma)细胞。段落31.如段落30所述的突变体,其中该亲本丝状真菌细胞是泡盛曲霉、臭曲霉、烟曲霉、日本曲霉、构巢曲霉、黑曲霉、米曲霉、黑刺烟管霉(bjerkanderaadusta)、干拟蜡菌(ceriporiopsisaneirina)、卡内基拟蜡菌(ceriporiopsiscaregiea)、浅黄拟蜡菌(ceriporiopsisgilvescens)、潘诺希塔拟蜡菌(ceriporiopsispannocinta)、环带拟蜡菌(ceriporiopsisrivulosa)、微红拟蜡菌(ceriporiopsissubrufa)、虫拟蜡菌(ceriporiopsissubvermispora)、狭边金孢子菌(chrysosporiuminops)、嗜角质金孢子菌(chrysosporiumkeratinophilum)、卢克诺文思金孢子菌(chrysosporiumlucknowense)、粪状金孢子菌(chrysosporiummerdarium)、租金孢子菌(chrysosporiumpannicola)、女王杜香金孢子菌(chrysosporiumqueenslandicum)、热带金孢子菌(chrysosporiumtropicum)、褐薄金孢子菌(chrysosporiumzonatum)、灰盖鬼伞(coprinuscinereus)、毛革盖菌(coriolushirsutus)、杆孢状镰孢、谷类镰孢、库威镰孢、大刀镰孢、禾谷镰孢、禾赤镰孢、异孢镰孢、合欢木镰孢、尖孢镰孢、多枝镰孢、粉红镰孢、接骨木镰孢、肤色镰孢、拟分枝孢镰孢、硫色镰孢、圆镰孢、拟丝孢镰孢、镶片镰孢、特异腐质霉、疏棉状腐质霉、米黑根毛霉、嗜热毁丝霉、粗糙脉胞菌、产紫青霉、黄孢平革菌(phanerochaetechrysosporium)、射脉菌(phlebiaradiata)、刺芹侧耳(pleurotuseryngii)、埃默森篮状菌、土生梭孢霉、长域毛栓菌(trametesvillosa)、变色栓菌(trametesversicolor)、哈茨木霉、康宁木霉、长枝木霉、里氏木霉或绿色木霉细胞。段落32.如段落1-29中任一项所述的突变体,其中该亲本丝状真菌细胞是木霉属细胞。段落33.如段落32所述的突变体,其中该木霉属细胞是里氏木霉细胞。段落34.如段落1-33中任一项所述的突变体,其中该突变体在转录因子的产生方面完全缺陷。段落35.如段落1-33中任一项所述的突变体,其中该突变体在转录因子的产生方面部分缺陷。段落36.如段落1-35中任一项所述的突变体,其中当在与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相同的条件下培养时,该一种或多种转录因子基因的修饰提高了该突变体在该目的多肽的产生方面的生产力。段落37.如段落1-35中任一项所述的突变体,其中与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相比,该一种或多种转录因子基因的修饰减少或消除了所得突变体中的纤维素酶-阴性表型。段落38.如段落1-35中任一项所述的突变体,其中(i)当在与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相同的条件下培养时,该一种或多种转录因子基因的修饰提高了该突变体在该目的多肽的产生方面的生产力并且(ii)与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相比,该一种或多种转录因子基因的修饰减少或消除了所得突变体中的纤维素酶-阴性表型。段落39.如段落36或38所述的突变体,其中与该亲本丝状真菌细胞相比,该突变体在该目的多肽的产生方面的生产力提高1%、2%、3%、4%、5%6%、7%、8%、9%、10%、11%、12%、13%、14%、15%、16%、17%、18%、19%、或20%。段落40.如段落36或38所述的突变体,其中与该亲本丝状真菌细胞相比,该突变体在该目的多肽的产生方面的生产力提高至少1%、至少2%、至少3%、至少4%、至少5%、至少6%、至少7%、至少8%、至少9%、至少10%、至少11%、至少12%、至少13%、至少14%、至少15%、至少16%、至少17%、至少18%、至少19%、或至少20%。段落41.一种产生目的多肽的方法,该方法包括在用于产生该目的多肽的培养基中培养如段落1-40中任一项所述的突变体丝状真菌细胞。段落42.如段落41所述的方法,该方法进一步包括从该培养基回收该目的多肽。段落43.一种用于构建亲本丝状真菌细胞的突变体的方法,该方法包括修饰各自编码该亲本丝状真菌细胞中的转录因子的一种或多种基因以产生该突变体,并且任选地回收该突变体,其中该亲本丝状真菌细胞或其突变体包含目的多肽的在启动子的转录控制下的编码序列,该启动子由这些转录因子中的一种或多种调节,其中该一种或多种转录因子选自由以下组成的组:(a)包含氨基酸序列的转录因子,该氨基酸序列与seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、或124具有至少70%序列同一性;(b)由多核苷酸编码的转录因子,该多核苷酸包含与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123具有至少70%序列同一性的核苷酸序列;以及(c)由多核苷酸编码的转录因子,该多核苷酸在高严格条件下与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123的全长互补序列杂交;其中将该一种或多种转录因子基因在亲本丝状真菌细胞中修饰以产生该突变体,使得该突变体在该一种或多种转录因子的产生方面有部分或完全缺陷,其中(i)当在与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相同的条件下培养时,该一种或多种转录因子基因的修饰提高了该突变体在该目的多肽的产生方面的生产力,(ii)与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相比,该一种或多种转录因子基因的修饰减少或消除了所得突变体中的纤维素酶阴性表型,或(iii)该一种或多种转录因子基因的修饰导致(i)和(ii)的组合;。段落44.如段落43所述的突方法,其中该转录因子包含氨基酸序列,该氨基酸序列与seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、或124具有至少80%序列同一性。段落45.如段落43所述的突方法,其中该转录因子包含氨基酸序列,该氨基酸序列与seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、或124具有至少85%序列同一性。段落46.如段落43所述的突方法,其中该转录因子包含氨基酸序列,该氨基酸序列与seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、或124具有至少90%序列同一性。段落47.如段落43所述的突方法,其中该转录因子包含氨基酸序列,该氨基酸序列与seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、或124具有至少95%序列同一性。段落48.如段落43所述的方法,其中该转录因子包含以下或由以下组成:seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、或124。段落49.如段落43所述的方法,其中该转录因子由多核苷酸编码,该多核苷酸包含与1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123具有至少80%序列同一性的核苷酸序列。段落50.如段落43所述的方法,其中该转录因子由多核苷酸编码,该多核苷酸包含与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123具有至少85%序列同一性的核苷酸序列。段落51.如段落43所述的方法,其中该转录因子由多核苷酸编码,该多核苷酸包含与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123具有至少90%序列同一性的核苷酸序列。段落52.如段落43所述的方法,其中该转录因子由多核苷酸编码,该多核苷酸包含与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123具有至少95%序列同一性的核苷酸序列。段落53.如段落43所述的方法,其中该转录因子由多核苷酸编码,该多核苷酸包含以下或由以下组成:seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123。段落54.如段落43所述的方法,其中该转录因子由多核苷酸编码,该多核苷酸在非常高严格条件下与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123的全长互补序列杂交。段落55.如段落43-54中任一项所述的方法,其中该启动子是来自纤维素酶基因的启动子。段落56.如段落55所述的方法,其中该纤维素酶基因是纤维二糖水解酶基因。段落57.如段落56所述的方法,其中该纤维二糖水解酶基因是纤维二糖水解酶i基因。段落58.如段落57所述的方法,其中该纤维二糖水解酶i基因是木霉属纤维二糖水解酶i基因。段落59.如段落58所述的方法,其中该纤维二糖水解酶i基因是里氏木霉纤维二糖水解酶i基因。段落60.如段落56所述的方法,其中该纤维二糖水解酶基因是纤维二糖水解酶ii基因。段落61.如段落60所述的方法,其中该纤维二糖水解酶ii基因是木霉属纤维二糖水解酶ii基因。段落62.如段落61所述的方法,其中该纤维二糖水解酶ii基因是里氏木霉纤维二糖水解酶ii基因。段落63.如段落43-62中任一项所述的方法,其中该启动子对于该目的多肽的编码序列是天然的。段落64.如段落43-62中任一项所述的方法,其中该启动子对于该目的多肽的编码序列是异源的。段落65.如段落43-64中任一项所述的方法,其中该目的多肽对于该亲本丝状真菌细胞或其突变体是天然的。段落66.如段落43-64中任一项所述的方法,其中该目的多肽对于该亲本丝状真菌细胞或其突变体是异源的。段落67.如段落43-66中任一项所述的方法,其中该目的多肽是抗体、抗原、抗微生物肽、酶、生长因子、激素、免疫调节剂、神经递质、受体、报告蛋白、结构蛋白或转录因子。段落68.如段落43-66中任一项所述的方法,其中该目的多肽是纤维素酶。段落69.如段落68所述的方法,其中该纤维素酶是内切葡聚糖酶、纤维二糖水解酶、以及β-葡糖苷酶。段落70.如段落43-66中任一项所述的方法,其中该目的多肽是半纤维素酶。段落71.如段落70所述的方法,其中该半纤维素酶是木聚糖酶、乙酰木聚糖酯酶、阿魏酸酯酶、阿拉伯呋喃糖苷酶、木糖苷酶和葡糖醛酸糖苷酶。段落72.如段落43-71中任一项所述的方法,其中该亲本丝状真菌细胞是枝顶孢属(acremonium)、曲霉属(aspergillus)、短梗霉属(aureobasidium)、烟管霉属(bjerkandera)、拟腊菌属(ceriporiopsis)、金孢子菌属(chrysosporium)、鬼伞属(coprinus)、革盖菌属(coriolus)、隐球菌属(cryptococcus)、线黑粉菌科(filibasidium)、镰孢属(fusarium)、腐质霉属(humicola)、梨孢菌属(magnaporthe)、毛霉属(mucor)、毁丝霉属(myceliophthora)、新美鞭菌属(neocallimastix)、脉胞菌属(neurospora)、拟青霉属(paecilomyces)、青霉属(penicillium)、平革菌属(phanerochaete)、射脉菌属(phlebia)、瘤胃壶菌属(piromyces)、侧耳属(pleurotus)、裂褶菌属(schizophyllum)、篮状菌属(talaromyces)、嗜热子囊菌属(thermoascus)、梭孢壳属(thielavia)、弯颈霉属(tolypocladium)、栓菌属(trametes)、或木霉属(trichoderma)细胞。段落73.如段落61所述的方法,其中该亲本丝状真菌细胞是泡盛曲霉、臭曲霉、烟曲霉、日本曲霉、构巢曲霉、黑曲霉、米曲霉、黑刺烟管霉(bjerkanderaadusta)、干拟蜡菌(ceriporiopsisaneirina)、卡内基拟蜡菌(ceriporiopsiscaregiea)、浅黄拟蜡菌(ceriporiopsisgilvescens)、潘诺希塔拟蜡菌(ceriporiopsispannocinta)、环带拟蜡菌(ceriporiopsisrivulosa)、微红拟蜡菌(ceriporiopsissubrufa)、虫拟蜡菌(ceriporiopsissubvermispora)、狭边金孢子菌(chrysosporiuminops)、嗜角质金孢子菌(chrysosporiumkeratinophilum)、卢克诺文思金孢子菌(chrysosporiumlucknowense)、粪状金孢子菌(chrysosporiummerdarium)、租金孢子菌(chrysosporiumpannicola)、女王杜香金孢子菌(chrysosporiumqueenslandicum)、热带金孢子菌(chrysosporiumtropicum)、褐薄金孢子菌(chrysosporiumzonatum)、灰盖鬼伞(coprinuscinereus)、毛革盖菌(coriolushirsutus)、杆孢状镰孢、谷类镰孢、库威镰孢、大刀镰孢、禾谷镰孢、禾赤镰孢、异孢镰孢、合欢木镰孢、尖孢镰孢、多枝镰孢、粉红镰孢、接骨木镰孢、肤色镰孢、拟分枝孢镰孢、硫色镰孢、圆镰孢、拟丝孢镰孢、镶片镰孢、特异腐质霉、疏棉状腐质霉、米黑根毛霉、嗜热毁丝霉、粗糙脉胞菌、产紫青霉、黄孢平革菌(phanerochaetechrysosporium)、射脉菌(phlebiaradiata)、刺芹侧耳(pleurotuseryngii)、埃默森篮状菌、土生梭孢霉、长域毛栓菌(trametesvillosa)、变色栓菌(trametesversicolor)、哈茨木霉、康宁木霉、长枝木霉、里氏木霉或绿色木霉细胞。段落74.如段落43-71中任一项所述的方法,其中该亲本丝状真菌细胞是木霉属细胞。段落75.如段落74所述的方法,其中该木霉属细胞是里氏木霉细胞。段落76.如段落43-75中任一项所述的方法,其中该突变体丝状真菌细胞在转录因子的产生方面完全缺陷。段落77.如段落43-75中任一项所述的方法,其中该突变体丝状真菌细胞在转录因子的产生方面部分缺陷。段落78.如段落43-77中任一项所述的方法,其中当在与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相同的条件下培养时,该一种或多种转录因子基因的修饰提高了该突变体在该目的多肽的产生方面的生产力。段落79.如段落43-77中任一项所述的方法,其中与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相比,该一种或多种转录因子基因的修饰减少或消除了所得突变体中的纤维素酶-阴性表型。段落80.如段落43-77中任一项所述的方法,其中(i)当在与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相同的条件下培养时,该一种或多种转录因子基因的修饰提高了该突变体在该目的多肽的产生方面的生产力并且(ii)与未修饰该一种或多种转录因子基因的亲本丝状真菌细胞相比,该一种或多种转录因子基因的修饰减少或消除了所得突变体中的纤维素酶-阴性表型。段落81.如段落78或80所述的方法,其中与该亲本丝状真菌细胞相比,该突变体在该目的多肽的产生方面的生产力提高1%、2%、3%、4%、5%6%、7%、8%、9%、10%、11%、12%、13%、14%、15%、16%、17%、18%、19%、或20%。段落82.如段落78或80所述的方法,其中与该亲本丝状真菌细胞相比,该突变体在该目的多肽的产生方面的生产力提高至少1%、至少2%、至少3%、至少4%、至少5%、至少6%、至少7%、至少8%、至少9%、至少10%、至少11%、至少12%、至少13%、至少14%、至少15%、至少16%、至少17%、至少18%、至少19%、或至少20%。段落83.一种分离的转录因子,该分离的转录因子选自由以下组成的组:(a)包含氨基酸序列的转录因子,该氨基酸序列与seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、或124具有至少70%序列同一性;(b)由多核苷酸编码的转录因子,该多核苷酸包含与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123具有至少70%序列同一性的核苷酸序列;以及(c)由多核苷酸编码的转录因子,该多核苷酸在高严格条件下与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123的全长互补序列杂交。段落84.如段落83所述的转录因子,该转录因子与seqidno:2、4、6、8、10、12、14、16、18、20、22、24、26、28、30、32、34、36、38、40、42、44、46、48、50、52、54、56、或124具有至少至少70%、至少75%、至少80%、至少85%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或100%序列同一性。段落85.如段落83所述的转录因子,该转录因子由多核苷酸编码,该多核苷酸包含与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123具有至少70%、至少75%、至少80%、至少85%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或100%序列同一性的核苷酸序列。段落86.如段落83所述的转录因子,该转录因子由多核苷酸编码,该多核苷酸在非常高严格条件下与seqidno:1、3、5、7、9、11、13、15、17、19、21、23、25、27、29、31、33、35、37、39、41、43、45、47、49、51、53、55、或123的全长互补序列杂交。段落87.一种分离的多核苷酸,该分离的多核苷酸编码如段落83-86中任一项所述的转录因子。段落88.一种核酸构建体或表达载体,该核酸构建体或表达载体包含如段落87所述的多核苷酸,该多核苷酸可操作地连接至指导该转录因子在表达宿主中的产生的一个或多个控制序列。段落89.一种重组宿主细胞,该重组宿主细胞包含如段落87所述的多核苷酸,该多核苷酸可操作地连接至指导该转录因子的产生的一个或多个控制序列。段落90.一种产生转录因子的方法,该方法包括在有益于产生该转录因子的条件下培养如段落89所述的重组宿主细胞。本文描述和要求保护的本发明不限于本文披露的具体方面的范围,因为这些方面旨在作为本发明几方面的说明。任何等同方面旨在处于本发明的范围之内。实际上,除了本文所示和描述的那些之外,本发明的各种修改因前述说明而对本领域的技术人员变得清楚。此类修改也旨在落入所附权利要求的范围内。在冲突的情况下,以包括定义的本披露为准。序列表<110>诺维信公司(novozymesa/s)lopezdeleon,alfredomaiyuran,suchindrashasky,jeffrey<120>提高丝状真菌菌株在多肽的产生方面的生产力的方法<130>14944-wo-pct<150>us62/778,598<151>2018-12-12<160>136<170>patentin3.5版<210>1<211>2315<212>dna<213>里氏木霉<400>1atgcttcgaccggcgacgcccttgaccgtgctgcttggaattgcctttgctttgctgctc60ctatccgtcttgtcggcccccatcatctcggccatccccctcggcagttctcaggatgtc120cagtttggcgtctttggcttttgccggccctcgggctgcagcagcgtcggcattggctat180gacattggtgagaatacccatcattacatgaccccgtgcttgtgcccatgattctccgtg240tcctcctcagcctcgactccatatttgattgctgggcttgcaatgtttcccaacaaatgg300cagcgcggtaactaacaagaatgtgcgtatagcgagcgtcctcaccgacacgaagagctc360gtcgtttgatctgcccagtggcgttcgccatacgctctcttcaatcctgatacttcaccc420cgtcgccgcctttctcaccctcgtcatgcttctgatggccgttgctgcccacttccgcgc480tgcttcgcactcgtcaaggtatctgctgttcttcctcgtcttcaacctcgtcacgttgct540cgtgtgtgtggctgctttcgtcatcgacgtcctactcttcatacctcacatggcatgggg600cacctacattgttttggctgccactgttctcgttttcttgagtcttctggtctcttgcgc660catgcgacgcactctggttagcaggaaagaccgaaagcggagaattgcagagaatgccga720gatgagcggagagaactactacaaccgagagaatcaggcgccgaaatctacctttaccat780gacgacccagcctaccatgccagttgtgagtggtggcaacggcacacaagacatcctgcc840tacgttcgctacgtacgagactccaagagatgaccaagtcagcgacgagcgcatccctct900aacacagcgaaccacatccgaaagatcgccccatggaaggcagcccgcgcccgccgacat960ggctgccgttgcgggcagcgagattggcagtgcttacagcggaccgcagaggtccgcatc1020ccaggactcgtatggcaactacggcaacgtgccacccaacgcctacggacaagccggcca1080gccgtacgatggtcgctcgaatcgagtccccatcaggagagagacggaccccgtcaatgg1140ctatggcgctggcggccgtggcggctatggagcccccatgagaggacggggcggctatgg1200ccctcccggccggggtggatacggcccgcgaggcggtggacgtggaggatacggaccgcc1260atctcgcgcaggctacaatggctcatactaccacatgcccaggggcggaggacgtggccc1320ttcgccgatgaacaaccctaatgatggcgtggccgggcaatacaacagaatgccgtcgcc1380atcgcagcaaggctatgctggagcaccgcagcagccgctacaggcagacgtccccctaaa1440cccggctccgtccggcgccggcaacacatacgtcccttaccgcccgaatatcgatctgcc1500aagggctgagtcccccccaccactgccggaacctaccgaatctgccgtctcgtcggtacc1560aaacgccattgagatggatgcgacgccggctgccgcggcaagcagcaacaatggccagtt1620cggcaacctcagagatagcgacacagacgttgccggaatggtggacctccaacagggcca1680gcagcccttatccacgcagagagacacgttcgccagcgatgggagcaggtactctcaaga1740tgagtatgtattgtactgtgttgcagcctccctccacccaggtaccctcgccgttcatca1800agtaggtggctaaccaattcgaaagtacatctaggcaatttgccccctctcgtgcccagt1860ggaaccaagaggctgggagagactcacctcgggtacccgtagcaaactcgtcgcgcggac1920ctaccccgaatatgtctggcatgactgcgccagtggttgatgccaagggcaagggcgact1980actacgaagacgtcgaccctcgttttgaccaaacagtttatacacccccgcgtcgccaga2040ctcctccgccgccgctgcagctaaacacggtcgactacgaagatatacgagttcccgcca2100gcggcactcgcagccccgccgaatcggaacactccaactttacctccatctcgcaacgtg2160ggatcaacccacaatggagcccgcaacccccagtgccccaacgccgaccagtacaacagc2220gccatgacatgatcctggacaatcccgatttcaagctcccgggaggtcgagcgacggcag2280cggggaagcgcagaggaccagggatgatgccgtag2315<210>2<211>692<212>prt<213>里氏木霉<400>2metleuargproalathrproleuthrvalleuleuglyilealaphe151015alaleuleuleuleuservalleuseralaproileileseralaile202530proleuglyserserglnaspvalglnpheglyvalpheglyphecys354045argproserglycysserservalglyileglytyraspilealaser505560valleuthraspthrlysserserserpheaspleuproserglyval65707580arghisthrleuserserileleuileleuhisprovalalaalaphe859095leuthrleuvalmetleuleumetalavalalaalahispheargala100105110alaserhisserserargtyrleuleuphepheleuvalpheasnleu115120125valthrleuleuvalcysvalalaalaphevalileaspvalleuleu130135140pheileprohismetalatrpglythrtyrilevalleualaalathr145150155160valleuvalpheleuserleuleuvalsercysalametargargthr165170175leuvalserarglysasparglysargargilealagluasnalaglu180185190metserglygluasntyrtyrasnarggluasnglnalaprolysser195200205thrphethrmetthrthrglnprothrmetprovalvalserglygly210215220asnglythrglnaspileleuprothrphealathrtyrgluthrpro225230235240argaspaspglnvalseraspgluargileproleuthrglnargthr245250255thrsergluargserprohisglyargglnproalaproalaaspmet260265270alaalavalalaglysergluileglyseralatyrserglyprogln275280285argseralaserglnaspsertyrglyasntyrglyasnvalpropro290295300asnalatyrglyglnalaglyglnprotyraspglyargserasnarg305310315320valproileargarggluthraspprovalasnglytyrglyalagly325330335glyargglyglytyrglyalaprometargglyargglyglytyrgly340345350proproglyargglyglytyrglyproargglyglyglyargglygly355360365tyrglyproproserargalaglytyrasnglysertyrtyrhismet370375380proargglyglyglyargglyproserprometasnasnproasnasp385390395400glyvalalaglyglntyrasnargmetproserproserglnglngly405410415tyralaglyalaproglnglnproleuglnalaaspvalproleuasn420425430proalaproserglyalaglyasnthrtyrvalprotyrargproasn435440445ileaspleuproargalagluserproproproleuprogluprothr450455460gluseralavalserservalproasnalaileglumetaspalathr465470475480proalaalaalaalaserserasnasnglyglnpheglyasnleuarg485490495aspseraspthraspvalalaglymetvalaspleuglnglnglygln500505510glnproleuserthrglnargaspthrphealaseraspglyserarg515520525tyrserglnaspgluglnphealaproserargalaglntrpasngln530535540glualaglyargaspserproargvalprovalalaasnserserarg545550555560glyprothrproasnmetserglymetthralaprovalvalaspala565570575lysglylysglyasptyrtyrgluaspvalaspproargpheaspgln580585590thrvaltyrthrproproargargglnthrproproproproleugln595600605leuasnthrvalasptyrgluaspileargvalproalaserglythr610615620argserproalaglusergluhisserasnphethrserilesergln625630635640argglyileasnproglntrpserproglnproprovalproglnarg645650655argprovalglnglnarghisaspmetileleuaspasnproaspphe660665670lysleuproglyglyargalathralaalaglylysargargglypro675680685glymetmetpro690<210>3<211>1224<212>dna<213>里氏木霉<400>3atggtatcgccgtacgaagattcgctccgacgaaactcgtcattcaggtcccttgacgcc60ccagctaccgatgccggctcgcgctcgtaccaggagagagccacggcgcctgagccagtg120cactacaattcttcctacccgcgcagtccgcatgccatggaacccctggcgtacttgagc180atggactttagcattgtcaaagcatcgcccatgttcatggaaatcgtccacgcatcgaac240ccactggggaacaaattgagcgacatcgtcacgccacatcaggcgagtttccttaacaac300ctccagaaccagctcatcgaagagcaacgactgcacgagcccaattatctgcctcccatg360ctgggccgactggatcttgccatcaaagagtttgggtttacgagtcaagatgctgcgaaa420tttcgcctcaaccatctcgaatattttggctttgttggatacgacggttacacgaggacc480tttccgcttcgttttgggcttgcaaaggagggttcgttttatttcgttgtgttcttgttg540agtcttcagcatcacccgccgctaccgccgcccttgccatcacagcaacatgcgtcccag600acacaacagcaacggactcagcaacatcaacatcaacagcattcacagtctcgggagccc660catcagcagacacaacaacaacagcagcagcagcaacaacaacaacaacaacaacagact720cagcagcgccagcaatatccgcagcatttgcatcagagacaactttcgcagccagctcca780tacacgcagctgccgcagcactcaccggttacgcaccatacacgggaacctactgccgcc840tcctatcatcttccgccgcccccgcatcaaccggcatacggagatcggtcgagcaatccc900tcagcagatccttttcgtcatcgcctcagcgaaggatctatccggccgccacacaatccg960gctgcctcgagtgccggtacaagctcaagcctccagtcttattcacagtcacactactcc1020cggccaaattaccaggcgtcgcagaacgaacatgtctctactagccggcctgcaactcag1080ccctcctatcagctaccccctattcgggcgccgcctgaacaccggcctgcgtcccgagag1140gtcggttggcccaatggcgagcgatcaagacgagtcgatatcggtggcttgatagaacag1200cctggggattccaccaggagatga1224<210>4<211>407<212>prt<213>里氏木霉<400>4metvalserprotyrgluaspserleuargargasnserserphearg151015serleuaspalaproalathraspalaglyserargsertyrglnglu202530argalathralaprogluprovalhistyrasnsersertyrproarg354045serprohisalametgluproleualatyrleusermetasppheser505560ilevallysalaserprometphemetgluilevalhisalaserasn65707580proleuglyasnlysleuseraspilevalthrprohisglnalaser859095pheleuasnasnleuglnasnglnleuileglugluglnargleuhis100105110gluproasntyrleuproprometleuglyargleuaspleualaile115120125lysglupheglyphethrserglnaspalaalalyspheargleuasn130135140hisleuglutyrpheglyphevalglytyraspglytyrthrargthr145150155160pheproleuargpheglyleualalysgluglyserphetyrpheval165170175valpheleuleuserleuglnhishisproproleuproproproleu180185190proserglnglnhisalaserglnthrglnglnglnargthrglngln195200205hisglnhisglnglnhisserglnserarggluprohisglnglnthr210215220glnglnglnglnglnglnglnglnglnglnglnglnglnglnglnthr225230235240glnglnargglnglntyrproglnhisleuhisglnargglnleuser245250255glnproalaprotyrthrglnleuproglnhisserprovalthrhis260265270histhrarggluprothralaalasertyrhisleupropropropro275280285hisglnproalatyrglyaspargserserasnproseralaasppro290295300phearghisargleusergluglyserileargproprohisasnpro305310315320alaalaserseralaglythrserserserleuglnsertyrsergln325330335serhistyrserargproasntyrglnalaserglnasngluhisval340345350serthrserargproalathrglnprosertyrglnleuproproile355360365argalaproprogluhisargproalaserarggluvalglytrppro370375380asnglygluargserargargvalaspileglyglyleuileglugln385390395400proglyaspserthrargarg405<210>5<211>2005<212>dna<213>里氏木霉<400>5atgctgcgctactcccccgtcttacacctggatactctctccttgccaccactgaccaat60gctcttccccgcccaaagtgcgagtacctcagcgctgtcgatagctgcacgcactgccgc120gatgcccacgtgcagtgcactttcgacctgcccctggcgcgacgcggccccaaagcgagg180aagaagagcgaccagcccggccagccgcctcctgatccgagctcgctctccaccgcggct240cgacccggccagatgccgccgccgctgaccttctccggccccgcagtagccgcgctgcag300cccttcgcctcgtcgtcgctgtcgcccgacgcggcctgggagcccgtcgagccgctcagc360attgacaacggcctgccccggcagccgctgggcgacctgcccggcctctccaccatccag420aacatctcgacgcgccagcgatggatacacctggccaacgccatgacgctgcgcaacacg480acgctagagcgcgtctcgaagcgatgtatcgacctcttcttcgactacctctaccccctc540acccccctggtgtacgagccggccctccgggacgtgctcgcatacatcttctcccagccc600ttgcctggcgtcaaccaaccatcgccgctgtcacagctcacgccagacccgaccaccggc660accacccccctcaacgctgccgagtcgtgggccggctttggccagcccagcggctcgcga720accgtcggcagcaggctggctccctgggccgactcgaccttcaccctggtcacggccgtc780tgcgcagaggcagcattcatgctacccaaggacattttccccgaaggagaatccgtctct840gagatcttgctcgaagcctctcgggactgcctgcaccagcacctcgaggccgacctggag900aatccgacggccaactcgattgccattcgctacttccactccaactgcctccacgctgcg960gggaagcccaagtactcgtggcacatatttggcgaggccatccgcctggcgcaggtcatg1020cagctgcacgaggaggctgccctcgaggggctcgtccccatcgaggcagagttccgccgt1080cgctgcttttggatcctgtacttgggcgacaagtcagccgctatactcaacaatcggccc1140atcaccatccacaagtactgcttcgacgccggcatcaccacgctatacccgtcgggtatc1200gaggacgagttcctgagcacggcgtccgagccgccccggaagagcttcatatccggcttc1260aacgcaaatgtgcggctctggcagtccgcggctgatttgctgctggaaatccgcgtgctg1320caagatcagatgatgcagcactttcgagggaccatgcccccgaaccatgtgctgccctcc1380gccgacaggcagcatctcgattctctctatgtccgcttcatcacctgcttggacgatctc1440ccgccgtacctccagtcgtgcactctggcgatggcagcgatggcagaaggcaacgggtct1500gccgagtccaagcagtacgtgatacagtgcatcaacctgcaggtgacgtttcactgtctg1560cgcatggtaattacgcagaaattcgaagacctctcttattttgctcctggcgttgagcag1620gctgatctcagaaagtcggagattgtgcgagacatgctgagggtgatgaacgaggcgccc1680ttttggggcctgcaggccaatggcgagccaaacgtgagtcgtttccttgtctcttctctt1740ttctgcacacccttttcttcgacgaccccccctctctctttatatccctgcggatatgta1800tatcatcaagcctcggcacttgttgctaatctgtcctgattatgttgtctggatgctgca1860ggttgaaaagattcgccttatcggagctagtttgctggccatcatccatcgcaaccagga1920ttcacccttggctacgcgagccaggagcgacttttccgtgcttttggatattctcacgcg1980gctggactcgaaggcgtcggactaa2005<210>6<211>618<212>prt<213>里氏木霉<400>6metleuargtyrserprovalleuhisleuaspthrleuserleupro151015proleuthrasnalaleuproargprolyscysglutyrleuserala202530valaspsercysthrhiscysargaspalahisvalglncysthrphe354045aspleuproleualaargargglyprolysalaarglyslysserasp505560glnproglyglnproproproaspproserserleuserthralaala65707580argproglyglnmetproproproleuthrpheserglyproalaval859095alaalaleuglnprophealaserserserleuserproaspalaala100105110trpgluprovalgluproleuserileaspasnglyleuproarggln115120125proleuglyaspleuproglyleuserthrileglnasnileserthr130135140argglnargtrpilehisleualaasnalametthrleuargasnthr145150155160thrleugluargvalserlysargcysileaspleuphepheasptyr165170175leutyrproleuthrproleuvaltyrgluproalaleuargaspval180185190leualatyrilepheserglnproleuproglyvalasnglnproser195200205proleuserglnleuthrproaspprothrthrglythrthrproleu210215220asnalaalaglusertrpalaglypheglyglnproserglyserarg225230235240thrvalglyserargleualaprotrpalaaspserthrphethrleu245250255valthralavalcysalaglualaalaphemetleuprolysaspile260265270pheprogluglygluservalsergluileleuleuglualaserarg275280285aspcysleuhisglnhisleuglualaaspleugluasnprothrala290295300asnserilealaileargtyrphehisserasncysleuhisalaala305310315320glylysprolystyrsertrphisilepheglyglualaileargleu325330335alaglnvalmetglnleuhisgluglualaalaleugluglyleuval340345350proileglualaglupheargargargcysphetrpileleutyrleu355360365glyasplysseralaalaileleuasnasnargproilethrilehis370375380lystyrcyspheaspalaglyilethrthrleutyrproserglyile385390395400gluaspglupheleuserthralasergluproproarglysserphe405410415ileserglypheasnalaasnvalargleutrpglnseralaalaasp420425430leuleuleugluileargvalleuglnaspglnmetmetglnhisphe435440445argglythrmetproproasnhisvalleuproseralaasparggln450455460hisleuaspserleutyrvalargpheilethrcysleuaspaspleu465470475480proprotyrleuglnsercysthrleualametalaalametalaglu485490495glyasnglyseralagluserlysglntyrvalileglncysileasn500505510leuglnvalthrphehiscysleuargmetvalilethrglnlysphe515520525gluaspleusertyrphealaproglyvalgluglnalaaspleuarg530535540lyssergluilevalargaspmetleuargvalmetasnglualapro545550555560phetrpglyleuglnalaasnglygluproasnvalglulysilearg565570575leuileglyalaserleuleualaileilehisargasnglnaspser580585590proleualathrargalaargserasppheservalleuleuaspile595600605leuthrargleuaspserlysalaserasp610615<210>7<211>3789<212>dna<213>里氏木霉<400>7atggagtgcgtcatggaggacagctcggacgataccgccaaggaaccatcagctctgacc60gatggcaatttcacagaaaaagacggcgaagcagcggctgctttgggtaccagtctcacg120gcgattcaatcctggcagacctccgtcttgaccatcaatgataacaaccgttcagcaaca180cacccgtctgaccatgagtcgttgccatgtatagacacttttgaagagactccgagctct240gacgaaaccgttattgagctcgtcacatccgacctgataccgcctccatctcgtctcgtc300acatcatcacgcagttctacgctcaaagtctccgagcttgatgatgctgtgtcagacagt360ctttcgacagagacttttcgagcaacttcaccggactgggtaccttcaaccgggtccttg420agcgttgagatacccggaagcgttcagcttgtaccaaggtcatgctacgagggctttcag480cctcctgacgacctaccgatcttaccagagtggaaggctgttcagactctttctccagcc540ttggcggaaagcacgcaagattttgacgagatcatgctggacgattttgcgatatacctt600gataaggagagatgctcacaggaaatgcgatctctacaccagctcaataccaagattgga660cacaacgacttctactttgacggtgttctccgcattggcgataccaggacgtatgtgcgc720cgcattcccatcaccgcggttcccattggcaactacggctcaatctcttatcacactgtc780cgagacaacatctggctgcaatctcctctgagctacaaacgggagctatattacaggttg840gggaagccggccaaggaatatgcacgcttcttttaccctttcttatgggtggcggacctg900gcgaagcactttgtggactttctctccatcatggcggagaacgagcgtagtgtttccata960caccactttcgatctacttttgcagcatggatgaagaagagccacaaggactcgcccgag1020tttttcagctggctacgccaacacccgagcgatgactaccggacttcgatagccgccaac1080atagggtttctccataaagagactctgggggtgttgaaccatcgagaggcgtattcccat1140gctatctgggcggagatctgggaattccaatcctttacatcaatcacgagccagcccaag1200actgagactccgccgccgacgattgttacgcaatacatatttgactgctttgaacatctt1260ccttttggcgatcgcctcaaggttgtccctctgagttcacggaccacggagctccgcaac1320agcttgatccgacagcggcacctagagcttccgtcaaagctgcaccagacatctaaggac1380atctctacggcgccacaagagcggatcaagaatatccggccaggcgacacaatctcaacc1440catcgcgacgacgcagagtccggcacattgtggaaacgagaattgtccaaaggcttctct1500gacgtggacagatggtttgcgctggtgcagtcggttcacgttgacagaaaaggggtgcgt1560acctttgatgtgatatggtattacagacccgtggacacgctctgcgggttgatgaagtat1620ccgtggaacaacgagctgtttctctcagaccattgctcttgctccgagaaagccaagatc1680gaagaaagcgaggttctcggtgtccatggcgttgacttttggggcacctcagcaacaagc1740gccgagctcttttgccggcagacctaccttcaagaagagaggagatgggtgacactggat1800gggaagcacctgcaatgtaggcacacttcaccaataccagatgatgaaatgccactcgag1860tatcagccagggaagacacatctgcttcgtttgaacttgaaaagtccattttccgagcct1920tgtgagtttatgcacgcgtctgatgaaggaagccgaaggttgtattacttcaggcgacta1980ttgcggcgccgccaggttgacccaaacgctcgcgaggcgcggccaaatgaacttgtatac2040agcgagcagacctgcaaagtcagcagggatcgaatcatgggcccatgttcagtgagatgc2100ttccgagatggagagatgatccctacgccgtatgaccacgacggcacgggcaacctcttt2160tacatcactcaccgacaggtagccgccaatggacattcggtatatgtgccgcttgacgaa2220gcaccgtcctctctgcagcaaggctttgatcctctccagaagttgcagaagcttcgcggg2280atggatctcttctgtggaggcggtaactttggcagagggcttgaagacggcggaggaatc2340gagatgaaatgggcaaatgactacgacagcaaggctatccacacgtacatggccaacgtc2400aaaaaccctcaagacgtccacccgtttctgggctccattgacgacttacagcgccttgcc2460attcaaggcgacttcgcggacaacgtacccctaattggagacgttgatttcgtgtccggt2520ggcagcccatgtcctggattctcacgcctcaccaacgacaagacgacggcagcgcaaagg2580aagaaccaatcgcttgttgcggcgtttgcgtcctttgttgatctctatcggcccaaatac2640ggcgtgttggagaatgtacctgggatggtccacaagaagcacgaccgagaccgagacgtc2700ttcagccagctcatctgcgccctagtcggcttgggctatcagacgcaattcttctacctg2760gacgcatcgtcatgcgggtcgccgcagcgcagatcacgcatcttcatcgtctttgctgcc2820ccgggactcgagctgcccgacaagccggtgcagactcactcacacccccctaacacaagg2880gagcatgggatcgggtggctgcccaacggccagcacatggcaagccgagaaatgccggca2940gcaacgcccttcaagtttgtctctgcggaagaagcgaccgccgacctgccgcccatctac3000gatgccaagcccgatatatgtgttcctttccccgatcaccgctcaaccttgggtatgtca3060gacttgctgcgagctcggatatcggtcataccgacacgaccatggggcatgaacttttcg3120caagcgtggttcggggagaggaaggtcggcggctccagcacgatgacggcagccgagcgc3180gagttcttcatcgggacgaaaggcgagggcaagcagcccatgtcggtgcagccctactca3240aattcgtacggaaggatgttccccaacaggctctttgagactgtggtgacgaggcagacg3300ctgggcgacgcgaagaacgggcgactgctccactggcgagagaataggcccgtgaccatc3360atggaggcgcgcagggcgcagggcttcctcgacgaagacgtgctgctcggggaccccccg3420acgcagtacaggattgtaggcaacagcgtcgcgaggcagcctgctgtggctctcggtgtg3480acgtttcgcgaagcgtgggtggcgagcctgaagaggaacagagagctgcagcgcgcaagg3540agctgcgttgccgaggaacatggcggattcacgaacggcgttccagtgactcatgagtcc3600ctcgagaagatggcggacggcatgggcagcatatcgagagatcagcagctaagggccttt3660acgcccgcaacggacagcacactgcagcagtcgatggatatcacgatgggcagtaaacgc3720cctcgttcggccaacatggtgattgagctgctcgagtcctcgaaacgacagaggccgaac3780cccttttga3789<210>8<211>1262<212>prt<213>里氏木霉<400>8metglucysvalmetgluaspserseraspaspthralalysglupro151015seralaleuthraspglyasnphethrglulysaspglyglualaala202530alaalaleuglythrserleuthralaileglnsertrpglnthrser354045valleuthrileasnaspasnasnargseralathrhisproserasp505560hisgluserleuprocysileaspthrpheglugluthrproserser65707580aspgluthrvalilegluleuvalthrseraspleuilepropropro859095serargleuvalthrserserargserserthrleulysvalserglu100105110leuaspaspalavalseraspserleuserthrgluthrpheargala115120125thrserproasptrpvalproserthrglyserleuservalgluile130135140proglyservalglnleuvalproargsercystyrgluglyphegln145150155160proproaspaspleuproileleuproglutrplysalavalglnthr165170175leuserproalaleualagluserthrglnasppheaspgluilemet180185190leuaspaspphealailetyrleuasplysgluargcysserglnglu195200205metargserleuhisglnleuasnthrlysileglyhisasnaspphe210215220tyrpheaspglyvalleuargileglyaspthrargthrtyrvalarg225230235240argileproilethralavalproileglyasntyrglyserileser245250255tyrhisthrvalargaspasniletrpleuglnserproleusertyr260265270lysarggluleutyrtyrargleuglylysproalalysglutyrala275280285argphephetyrpropheleutrpvalalaaspleualalyshisphe290295300valasppheleuserilemetalagluasngluargservalserile305310315320hishispheargserthrphealaalatrpmetlyslysserhislys325330335aspserprogluphephesertrpleuargglnhisproseraspasp340345350tyrargthrserilealaalaasnileglypheleuhislysgluthr355360365leuglyvalleuasnhisargglualatyrserhisalailetrpala370375380gluiletrpglupheglnserphethrserilethrserglnprolys385390395400thrgluthrproproprothrilevalthrglntyrilepheaspcys405410415phegluhisleupropheglyaspargleulysvalvalproleuser420425430serargthrthrgluleuargasnserleuileargglnarghisleu435440445gluleuproserlysleuhisglnthrserlysaspileserthrala450455460proglngluargilelysasnileargproglyaspthrileserthr465470475480hisargaspaspalagluserglythrleutrplysarggluleuser485490495lysglypheseraspvalaspargtrpphealaleuvalglnserval500505510hisvalasparglysglyvalargthrpheaspvaliletrptyrtyr515520525argprovalaspthrleucysglyleumetlystyrprotrpasnasn530535540gluleupheleuserasphiscyssercysserglulysalalysile545550555560gluglusergluvalleuglyvalhisglyvalaspphetrpglythr565570575seralathrseralagluleuphecysargglnthrtyrleuglnglu580585590gluargargtrpvalthrleuaspglylyshisleuglncysarghis595600605thrserproileproaspaspglumetproleuglutyrglnprogly610615620lysthrhisleuleuargleuasnleulysserpropheserglupro625630635640cysgluphemethisalaseraspgluglyserargargleutyrtyr645650655pheargargleuleuargargargglnvalaspproasnalaargglu660665670alaargproasngluleuvaltyrsergluglnthrcyslysvalser675680685argaspargilemetglyprocysservalargcyspheargaspgly690695700glumetileprothrprotyrasphisaspglythrglyasnleuphe705710715720tyrilethrhisargglnvalalaalaasnglyhisservaltyrval725730735proleuaspglualaproserserleuglnglnglypheaspproleu740745750glnlysleuglnlysleuargglymetaspleuphecysglyglygly755760765asnpheglyargglyleugluaspglyglyglyileglumetlystrp770775780alaasnasptyraspserlysalailehisthrtyrmetalaasnval785790795800lysasnproglnaspvalhispropheleuglyserileaspaspleu805810815glnargleualaileglnglyaspphealaaspasnvalproleuile820825830glyaspvalaspphevalserglyglyserprocysproglypheser835840845argleuthrasnasplysthrthralaalaglnarglysasnglnser850855860leuvalalaalaphealaserphevalaspleutyrargprolystyr865870875880glyvalleugluasnvalproglymetvalhislyslyshisasparg885890895aspargaspvalpheserglnleuilecysalaleuvalglyleugly900905910tyrglnthrglnphephetyrleuaspalasersercysglyserpro915920925glnargargserargilepheilevalphealaalaproglyleuglu930935940leuproasplysprovalglnthrhisserhisproproasnthrarg945950955960gluhisglyileglytrpleuproasnglyglnhismetalaserarg965970975glumetproalaalathrprophelysphevalseralaglugluala980985990thralaaspleuproproiletyraspalalysproaspilecysval99510001005propheproasphisargserthrleuglymetseraspleuleu101010151020argalaargileservalileprothrargprotrpglymetasn102510301035pheserglnalatrppheglygluarglysvalglyglyserser104010451050thrmetthralaalagluarggluphepheileglythrlysgly105510601065gluglylysglnprometservalglnprotyrserasnsertyr107010751080glyargmetpheproasnargleuphegluthrvalvalthrarg108510901095glnthrleuglyaspalalysasnglyargleuleuhistrparg110011051110gluasnargprovalthrilemetglualaargargalaglngly111511201125pheleuaspgluaspvalleuleuglyaspproprothrglntyr113011351140argilevalglyasnservalalaargglnproalavalalaleu114511501155glyvalthrpheargglualatrpvalalaserleulysargasn116011651170arggluleuglnargalaargsercysvalalaglugluhisgly117511801185glyphethrasnglyvalprovalthrhisgluserleuglulys119011951200metalaaspglymetglyserileserargaspglnglnleuarg120512101215alaphethrproalathraspserthrleuglnglnsermetasp122012251230ilethrmetglyserlysargproargseralaasnmetvalile123512401245gluleuleugluserserlysargglnargproasnprophe125012551260<210>9<211>2316<212>dna<213>里氏木霉<400>9atggcccagccgccaagtttctacgcctcttccaggcccttggaagtcttcaccgacgac60atcttcttcgagaacgaagcccccatgacaagccacgcacccatgccgaatccgactagg120cctattcgtcgtcctctcagcaactcaagctcgaatgtcatcctcgagcccgcagtctcc180cagaacagcaacaagatctcaccatacaagcccaagaccgcagcacccagagcaccgctc240aagccgtcgcatggcctgaacaagttcaacggcatctcaatggctcctccctcagacaag300gcgccagtcaccgactctctgcagaagaagccgcagctatccaagttcaagaccggcctg360cagaagccagctctcgccagccctcacgacgccttctttggaaaggaaaacgttcagcct420caaatcttcccggcaccgccggcaatcaacatctccatggagcagtattaccaggattcc480aatggaaagcgaggactgatggaggcggcaccgatcaaggactctcgagtcagcagcaaa540aagcccaaggccgaggagcaggtgctcccgccgcacgactcgttccctcccattactgac600gatggcaccaaaccgcctcacagctatgcacagcttattggaatggccattcttcgatct660tcaaaacgacgcctcacccttgcccagatttacaagtggatcagcgacaactattctttc720tacaaccccaatgacgcaggctggcaaaatagcattcgacacaacctgagtctgcacaag780aactttatcaaaatcgagcgaccaaaggacgatcccggcaagggcaactactggggcatc840gagcccggcaccgagttccagttcctcaaggaaaagcccacgcgcaaagccgtcccgact900gccgagaacctccccgtcatgtccacccgcctcgagccctctcgacctacgccagcattg960atgcccgagccgtgtctgccgcctcctgctcccatccatcatcatcagcaccaccaccac1020cagcagcatcagccgcatgcgaccagcctccctccgctgccgacttctcaggcgaccttg1080tccatgcctctggagccctcatcagacgccaccattcttctgtccgacaacgcgcccgcc1140gaggacctggcggacaagggaccggacaacgaactgccattggaatcgtccttcttctcg1200ccccttcctcccgccctcaactccactccgccggttccccggccagagcgacgaaacggc1260acaccgccccctgtgagccgcaatcccgcctcgtccgcaaccaggactcacaagcgcaag1320tttgcctccatggacgacagcggctacatctcgtctctcgagtcttccgccatgcgcccg1380aacgcagccaggtcggccctcttcacatccgaggcggaccgacaccgcatcaagcgcggc1440cgggctgaggaggaaattgcacggctgagaggatcttctccctttagcccaaccaagagt1500cgctcgctctccagctatgggcttacttcgtcatctcctttccgtcagtccctcgacaac1560cagatgcctccccccctcactcccgttgtcaagaaggtcaaacccattgcgcagccacca1620ccttcggtgtctcccaacacgaacctgcgcatccatcgcgccaaggtccgccacatgctg1680cagtctcctcttcggcgtgtcgccaacctgtcgaatgaggacatgttgccgtggagcccc1740gccttccggctggatgacagcatcttcacctttgatacgcccaatgccagcagcaacctc1800cccgactttgacattttccaagacctgcccgtggatgatgaaaacgtctttgacagcatt1860ggcgctgcagatgcgggctcgcccattaagcgctcggccaagcgcgctcgcttggatcgg1920tccatttcgtcgtcagccattggggagatgtcggcttcaaagaggcttctcacatcggcc1980cctctgctcaaggcccctgacacgccttcgcagtttctggagacaccgagcaaagtcttt2040gaaggcctcagctctccctccaagatcctccaggggtcgccaatcaggggcaactcgcca2100agcaagttcgtctcgctgatagagcttccaggagatgccgactggccgtcccttcacctg2160gacaccggcgactttgcttcgggtgatacgtccgacttcaccggccttgatatccttcag2220ggtttcgaaaagattggatctggctctcagccgtccaaaggcttgaagcagagtggaaag2280cccaccctgggtcgaagctttacaaccaacttttag2316<210>10<211>771<212>prt<213>里氏木霉<400>10metalaglnproproserphetyralaserserargproleugluval151015phethraspaspilephephegluasnglualaprometthrserhis202530alaprometproasnprothrargproileargargproleuserasn354045serserserasnvalileleugluproalavalserglnasnserasn505560lysileserprotyrlysprolysthralaalaproargalaproleu65707580lysproserhisglyleuasnlyspheasnglyilesermetalapro859095proserasplysalaprovalthraspserleuglnlyslysprogln100105110leuserlysphelysthrglyleuglnlysproalaleualaserpro115120125hisaspalaphepheglylysgluasnvalglnproglnilephepro130135140alaproproalaileasnilesermetgluglntyrtyrglnaspser145150155160asnglylysargglyleumetglualaalaproilelysaspserarg165170175valserserlyslysprolysalaglugluglnvalleuproprohis180185190aspserpheproproilethraspaspglythrlysproprohisser195200205tyralaglnleuileglymetalaileleuargserserlysargarg210215220leuthrleualaglniletyrlystrpileseraspasntyrserphe225230235240tyrasnproasnaspalaglytrpglnasnserilearghisasnleu245250255serleuhislysasnpheilelysilegluargprolysaspasppro260265270glylysglyasntyrtrpglyilegluproglythrglupheglnphe275280285leulysglulysprothrarglysalavalprothralagluasnleu290295300provalmetserthrargleugluproserargprothrproalaleu305310315320metprogluprocysleuproproproalaproilehishishisgln325330335hishishishisglnglnhisglnprohisalathrserleupropro340345350leuprothrserglnalathrleusermetproleugluproserser355360365aspalathrileleuleuseraspasnalaproalagluaspleuala370375380asplysglyproaspasngluleuproleugluserserphepheser385390395400proleuproproalaleuasnserthrproprovalproargproglu405410415argargasnglythrproproprovalserargasnproalaserser420425430alathrargthrhislysarglysphealasermetaspaspsergly435440445tyrileserserleugluserseralametargproasnalaalaarg450455460seralaleuphethrserglualaasparghisargilelysarggly465470475480argalagluglugluilealaargleuargglyserserpropheser485490495prothrlysserargserleusersertyrglyleuthrserserser500505510propheargglnserleuaspasnglnmetproproproleuthrpro515520525valvallyslysvallysproilealaglnproproproservalser530535540proasnthrasnleuargilehisargalalysvalarghismetleu545550555560glnserproleuargargvalalaasnleuserasngluaspmetleu565570575protrpserproalapheargleuaspaspserilephethrpheasp580585590thrproasnalaserserasnleuproasppheaspilepheglnasp595600605leuprovalaspaspgluasnvalpheaspserileglyalaalaasp610615620alaglyserproilelysargseralalysargalaargleuasparg625630635640serileserserseralaileglyglumetseralaserlysargleu645650655leuthrseralaproleuleulysalaproaspthrproserglnphe660665670leugluthrproserlysvalphegluglyleuserserproserlys675680685ileleuglnglyserproileargglyasnserproserlyspheval690695700serleuilegluleuproglyaspalaasptrpproserleuhisleu705710715720aspthrglyaspphealaserglyaspthrseraspphethrglyleu725730735aspileleuglnglypheglulysileglyserglyserglnproser740745750lysglyleulysglnserglylysprothrleuglyargserphethr755760765thrasnphe770<210>11<211>1906<212>dna<213>里氏木霉<400>11atgccgtcaaatcaagcgagcatcgagctggaggaaatcttcaaggagctcggcatctcg60cagtatcttgacgcctttgttgaacatggctttgacacttgggacaccatcctcgatatt120caagagtctgatctgtgggtaaactgctgcggtggcgtgtgatgtagtctcagcaaggct180aactagactcccagcgatgctttaggggtcaagctcggtcatcggagagtaagtattcga240gtcgcagagccgtgagaggagtactgaccgcggacatcatcagaaacttcaacggcgaat300cgccaacgcaaggggaatcgaccccagcatctcattgtcgtcggtacaagctgccatcga360agatgaaaagtatgacgggtctcatagaaaacgagaagccccctgccatgctggaaatgg420cggaagcagtactgcgaagcgcaaataccgccggcatcctaaggtatagctcaatcagtc480aagcggccacccctcgggccagattgctaatgccttctgtccttagcccgacgaaaacgc540gcctccacggcccatgagcgcatatgtgctcttttcgaatagtgagttccaatgttgtgc600cccccgctgccctttgtgttggcaagatcgagactatttggctgaccaaacacggtagaa660ctcagagaggatttgaaaaaggacccttctctctcctttgtagcaatagccaagctcgta720gggggcaattggcaaaacctcccattgcaagagagagagtattatgaagcccaggcgaga780gcggataaggagcgctattatcgagagatggcactctacaagcagactccgcaatatcaa840gagtacatgaagtacctccaagacttcaacgagaagcaagccaaactcaggcgaggtccg900tggttccttgaatcggtattgtagccgtggggtgggtacagttgaagctgatgtttattt960ctgtgccggctagaccaagagaatgccaaacgaacagccatgcgcggctcgacctcaaca1020gcgagtggcagcggcagtagcagcgagagaatgcgagaaagcgagtctcgcgagccccct1080agccttcgacataacagctcgaattcgatgccctctccttcaaggacccatcatatcacg1140gcgggcaccgggacgtatgcagggctgacacaaccaaatggcgacatacccacacaaagc1200gaatcgcgctctcgacgacgatattcagcagagaaggagagggaacaccagaccgacacc1260ccgcagcaagttctgccctcagttttcgatctgctggccgatagacaaggtcagccgagc1320gccccagtgacaaacaactccggactcatgcaagggctggcgccagccgatcatggcagg1380cggtctatatcccacggcacaccctcgtggccagtcgactcaaggccgtcccttcatcac1440gagccatcttctgctggaagcagtataccgactgcgtcgtcaagcagtctgagtagtaga1500ttaagcgctggccatatgccaatccatgcgctcttatcgtcggatgaaggtcaaccgccc1560cagcgcattgacgatattccagcatatacgtcccactatgtttccagtccaattgagcag1620aagaggccgtccatggagtatcaggggcctaagggatacggtatccatccccccaagtgc1680ccgagtatttggagaaaagctgctaacatgaggtgccaaaggcttccagaccgcttcctc1740ggcgttccagcatatgagattcgaggagacaagcaacggggatgtgctgatgacttcggc1800aaacgaaacgccgcctagcgcctctcccatgagcgttagcgggcttgatggggtgaacgc1860tctattaaaagctggcgaaatcgtcggccatcctcgccaaatctaa1906<210>12<211>503<212>prt<213>里氏木霉<400>12metproserasnglnalaserilegluleuglugluilephelysglu151015leuglyileserglntyrleuaspalaphevalgluhisglypheasp202530thrtrpaspthrileleuaspileglngluseraspleuaspalaleu354045glyvallysleuglyhisargarglysleuglnargargilealaasn505560alaargglyileaspproserileserleuserservalglnalaala65707580ilegluaspglulystyraspglyserhisarglysargglualapro859095cyshisalaglyasnglyglyserserthralalysarglystyrarg100105110arghisprolysproaspgluasnalaproproargprometserala115120125tyrvalleupheserasnlysleuarggluaspleulyslysasppro130135140serleuserphevalalailealalysleuvalglyglyasntrpgln145150155160asnleuproleuglngluargglutyrtyrglualaglnalaargala165170175asplysgluargtyrtyrargglumetalaleutyrlysglnthrpro180185190glntyrglnglutyrmetlystyrleuglnasppheasnglulysgln195200205alalysleuargargaspglngluasnalalysargthralametarg210215220glyserthrserthralaserglyserglysersersergluargmet225230235240argglusergluserarggluproproserleuarghisasnserser245250255asnsermetproserproserargthrhishisilethralaglythr260265270glythrtyralaglyleuthrglnproasnglyaspileprothrgln275280285sergluserargserargargargtyrseralaglulysgluargglu290295300hisglnthraspthrproglnglnvalleuproservalpheaspleu305310315320leualaaspargglnglyglnproseralaprovalthrasnasnser325330335glyleumetglnglyleualaproalaasphisglyargargserile340345350serhisglythrprosertrpprovalaspserargproserleuhis355360365hisgluproserseralaglyserserileprothralaserserser370375380serleuserserargleuseralaglyhismetproilehisalaleu385390395400leuserseraspgluglyglnproproglnargileaspaspilepro405410415alatyrthrserhistyrvalserserproilegluglnlysargpro420425430sermetglutyrglnglyprolysglytyrglypheglnthralaser435440445seralapheglnhismetargpheglugluthrserasnglyaspval450455460leumetthrseralaasngluthrproproseralaserprometser465470475480valserglyleuaspglyvalasnalaleuleulysalaglygluile485490495valglyhisproargglnile500<210>13<211>3787<212>dna<213>里氏木霉<400>13atggaggctcaagttgttcccgctctgcaaagagctgctgagattggaaccctctacgat60gcaaagagagatgagttcctcagcgcttcactattccctcaaggtctgccttctagtcat120gtgtcgacacagccaacccccggcacggaaatcaccgtctccagatgcgcttcctatgga180gacgtgttcagagatctccgtatagacgaaacgactgcggtcaacattctctccgggaca240ctgcgtctccaaggagcagccatgttcctccgagacttcgaagttggcggaatggtgcgc300gctgccatcatccatcgactctctacagtgcagcagaccctcaccagcctaggcgacacc360ttcaaatcagctggcgatttggcccccttgaagacggaagactgcactcacgtcgtgatc420ggcatcaaatggggcctgcagactgttgtggccgccagctgccccacgccaggcagaaca480agttctcttaaagctgaagaggcgacctttgaacgggatctggaggttttgctgactgcc540attgagtccgcctcctttacgccgcagacggcctctcccaggctgagctctcaactggag600ctctgcaatgagttcaccatctacagcgatgcttttgaacaggagggtctaccgatgcaa660agcgtatccgaagtatgcgacttcatccgcatcgtacccgaccatcttcggcaagcaaac720agcggcaaggggttcccagtcgcatatacgttgatgcccgtcacgacgctcaaatacatc780atgcctctgccgcgcgagcttccgtacaactcaacgttcatcaggcccgattatctggcc840aacttcatccatgtcttcgacgatatcgtagcttccgcaggcaggcttcgcaagtatcac900aactacttggctggccacatcatgtatgtctccaaaagtcacgttgatgaggttttggcc960tcgattgttctcctggaggagggcaagagccggctcaccgagcaactgttcaactgcttg1020tcggacgtgcgccatggcaggcaggatccctcagttctcttcgaccttcatcgtcatgcg1080gccctcagcgagcccacaccaaggcagctctctgaccttgtcggacaggagaccgagaag1140ctcgacttcattgcaagtgtcatctctcagggggccaactacataggccacaacggactt1200tctctcgaaagggtccaaaaggcctacaaggacgctcaattctacgtctttcattttgat1260tccgcctctatgaaaggcgatcaatactggagtgacaacctggccttgttgctcgaactc1320gtcggacagcgagatgagcgcctacccgtcttcatcatggaccacgacgcgataggctcc1380gacgcctgtgttggcaattcacgtattgcacagtacaagggcggcgaagtatgcacaatc1440gatatgttggagtatcgccagtttgtctcctcgaaatgctttgctcgatgctccggcgca1500acacttgatacgagcaggtgtcaaagacctgtcaaaagacgctttgtcaaagttccctgc1560ccgagtcctgggtgtgattccggccattctcatcagtggacttgtccgaagtgctttgca1620accatcgagtatggcttcagcgacgacttcttctactgcgagtgcggccgcggactcttt1680agtgactacgagttcaagtgcaacgatgcaaagcacgacgctggcttcaagacgtatgat1740gtggaacagcttcgaaaactgctgggcaagttggatcaagatgactacatcaacatcctc1800attctgggagagaccggagtgggcaagtcgaccttcatcaatgcctttgtcaactacctg1860tcttattccacgctggacgaggccaaggatgccgactcgctcgcttctgtcattccttgc1920tcgttctctcttcagacgatggacagggaaaacccggagttgggcattcaagaattcaac1980gtcaaagttggtggacgtgacgatgaggctgacggatcgacgggaaactcggcaacccag2040aagtcggccgtgtatcccgtcatgatcggggcaaaggcgtatcgcctgattgacactcct2100ggcattggagacaccagaggcttgtcttacgacaaggagaacatggccgatattctcaag2160accatcagctcttatgacaagctgcacggcatcctggtgcttgtcaagtcgaacaatgca2220cgcctaaccgtcttgttcaaattctgcctcaaggagctcctcacgcatctacaccggagt2280gctaccaagaacatggcttttggcttcaccaacactcgcatctccaattatgcccctggc2340gatacgttcaagccgctcaaggctctccttgacgagcagtcggatattcccatctctctc2400tcgacggctacaacctactgttttgactcggaaagcttccgctaccttgctgcttataag2460cagggcatacccatggacaacgaggaagagtttcgtcgaagctgggaccactcaagcaag2520gaggcccatagactcctcaactactttgcgtctactcagccacaccctgtcaccagcaca2580ctgagcctgaacggggcacgaaaggtgatcctcgagctgacaaagcccatggccagcatc2640tcggactcgatcaagaagaacatcaagttgtgcgaagacaagaagatggagctgagtgat2700acacagttaacagcaaccaagcttcgcaggaggctgcgtctggagagaatccagttcaag2760gccgtcaagctggacaggcctcgtacggtctgcaaaaaccccgactgttgcgatttcaaa2820gacagcggtcttgatgatggagttgtcgtcaccatatacaagacgcactgccacgctgag2880tgctatcttagcaatgtcacccaagatgtcgttgctgatcccggcctcatcaactgctgg2940gcattcaacggcacgagtaattgtcaagtttgccaacaccgatggcaagagcacttgcac3000gtcctctacgagctcttcgagaccaaggtccaggttacagacaaagagatagagaggcag3060ctcaaggccaacgcggacgacgtaaccctgcgccaactggcgattactacacttgaccga3120gacattgcagagttcaacacagagttggatgaaatccgtcgtgcgtctgcccgattctgt3180ctgttcttgagagcaaacgccatcaccgtcatcaacgacgccacgctggactacctggac3240atgctcattcaggacgagcagggcactattgaggccggcagacaaagaggattctgcgtt3300gacgccaacaagaagcgtcttcaagcgctcagagaggatagaaagatccatctggagctc3360gtggaaaccttcaagcaaaacatggctcatccgacctgtcccgaggacatgctgctcgac3420gagcaaggagtcgacgccctcgtcaagaaactgtacaatctgaagcatttcggcgccaac3480ttgaggcacatcaagtatgtcattgactcctccatcgaagacacgtaccgtgagcggcct3540tatcgcgtgcccactttcaactttcgcaagcaagtcgcaagtcaccggacgccacgccat3600acgagccagcgagggacatggcagggttcgggtgaagggtatttccatcagacacagcac3660tctgtgggaaacgcggttggatttgaaggtcatgggtatccgccgctttcgtacgcggat3720gctacaaagtcgatatctcatcggttcaagtcgttttgggagaggagcattgcacgagga3780cgggtga3787<210>14<211>1243<212>prt<213>里氏木霉<400>14metglualaglnvalvalproalaleuglnargalaalagluilegly151015thrleutyraspalalysargaspglupheleuseralaserleuphe202530proglnglyleuproserserhisvalserthrglnprothrprogly354045thrgluilethrvalserargcysalasertyrglyaspvalphearg505560aspleuargileaspgluthrthralavalasnileleuserglythr65707580leuargleuglnglyalaalametpheleuargaspphegluvalgly859095glymetvalargalaalaileilehisargleuserthrvalglngln100105110thrleuthrserleuglyaspthrphelysseralaglyaspleuala115120125proleulysthrgluaspcysthrhisvalvalileglyilelystrp130135140glyleuglnthrvalvalalaalasercysprothrproglyargthr145150155160serserleulysalagluglualathrphegluargaspleugluval165170175leuleuthralailegluseralaserphethrproglnthralaser180185190proargleuserserglnleugluleucysasngluphethriletyr195200205seraspalaphegluglngluglyleuprometglnservalserglu210215220valcysasppheileargilevalproasphisleuargglnalaasn225230235240serglylysglypheprovalalatyrthrleumetprovalthrthr245250255leulystyrilemetproleuproarggluleuprotyrasnserthr260265270pheileargproasptyrleualaasnpheilehisvalpheaspasp275280285ilevalalaseralaglyargleuarglystyrhisasntyrleuala290295300glyhisilemettyrvalserlysserhisvalaspgluvalleuala305310315320serilevalleuleuglugluglylysserargleuthrgluglnleu325330335pheasncysleuseraspvalarghisglyargglnaspproserval340345350leupheaspleuhisarghisalaalaleusergluprothrproarg355360365glnleuseraspleuvalglyglngluthrglulysleuasppheile370375380alaservalileserglnglyalaasntyrileglyhisasnglyleu385390395400serleugluargvalglnlysalatyrlysaspalaglnphetyrval405410415phehispheaspseralasermetlysglyaspglntyrtrpserasp420425430asnleualaleuleuleugluleuvalglyglnargaspgluargleu435440445provalpheilemetasphisaspalaileglyseraspalacysval450455460glyasnserargilealaglntyrlysglyglygluvalcysthrile465470475480aspmetleuglutyrargglnphevalserserlyscysphealaarg485490495cysserglyalathrleuaspthrserargcysglnargprovallys500505510argargphevallysvalprocysproserproglycysaspsergly515520525hisserhisglntrpthrcysprolyscysphealathrileglutyr530535540glypheseraspaspphephetyrcysglucysglyargglyleuphe545550555560serasptyrgluphelyscysasnaspalalyshisaspalaglyphe565570575lysthrtyraspvalgluglnleuarglysleuleuglylysleuasp580585590glnaspasptyrileasnileleuileleuglygluthrglyvalgly595600605lysserthrpheileasnalaphevalasntyrleusertyrserthr610615620leuaspglualalysaspalaaspserleualaservalileprocys625630635640serpheserleuglnthrmetasparggluasnprogluleuglyile645650655glnglupheasnvallysvalglyglyargaspaspglualaaspgly660665670serthrglyasnseralathrglnlysseralavaltyrprovalmet675680685ileglyalalysalatyrargleuileaspthrproglyileglyasp690695700thrargglyleusertyrasplysgluasnmetalaaspileleulys705710715720thrilesersertyrasplysleuhisglyileleuvalleuvallys725730735serasnasnalaargleuthrvalleuphelysphecysleulysglu740745750leuleuthrhisleuhisargseralathrlysasnmetalaphegly755760765phethrasnthrargileserasntyralaproglyaspthrphelys770775780proleulysalaleuleuaspgluglnseraspileproileserleu785790795800serthralathrthrtyrcyspheaspsergluserpheargtyrleu805810815alaalatyrlysglnglyileprometaspasngluglugluphearg820825830argsertrpasphisserserlysglualahisargleuleuasntyr835840845phealaserthrglnprohisprovalthrserthrleuserleuasn850855860glyalaarglysvalileleugluleuthrlysprometalaserile865870875880seraspserilelyslysasnilelysleucysgluasplyslysmet885890895gluleuseraspthrglnleuthralathrlysleuargargargleu900905910argleugluargileglnphelysalavallysleuaspargproarg915920925thrvalcyslysasnproaspcyscysaspphelysaspserglyleu930935940aspaspglyvalvalvalthriletyrlysthrhiscyshisalaglu945950955960cystyrleuserasnvalthrglnaspvalvalalaaspproglyleu965970975ileasncystrpalapheasnglythrserasncysglnvalcysgln980985990hisargtrpglngluhisleuhisvalleutyrgluleuphegluthr99510001005lysvalglnvalthrasplysgluilegluargglnleulysala101010151020asnalaaspaspvalthrleuargglnleualailethrthrleu102510301035aspargaspilealaglupheasnthrgluleuaspgluilearg104010451050argalaseralaargphecysleupheleuargalaasnalaile105510601065thrvalileasnaspalathrleuasptyrleuaspmetleuile107010751080glnaspgluglnglythrileglualaglyargglnargglyphe108510901095cysvalaspalaasnlyslysargleuglnalaleuarggluasp110011051110arglysilehisleugluleuvalgluthrphelysglnasnmet111511201125alahisprothrcysprogluaspmetleuleuaspgluglngly113011351140valaspalaleuvallyslysleutyrasnleulyshisphegly114511501155alaasnleuarghisilelystyrvalileaspserserileglu116011651170aspthrtyrarggluargprotyrargvalprothrpheasnphe117511801185arglysglnvalalaserhisargthrproarghisthrsergln119011951200argglythrtrpglnglyserglygluglytyrphehisglnthr120512101215glnhisservalglyasnalavalglyphegluglyhisglyarg122012251230pheglyargglyalaleuhisgluaspgly12351240<210>15<211>1779<212>dna<213>里氏木霉<400>15atggcgcagtacggcccctatgttcgactctggggcatccgcccgtcacaactccctgca60gagaccgcgtcagctcaagacctcgtcccattcctccagtccatcctgagcgaagcagtt120cctttcatcagctcagtgcccctacaatcttcctcctcgtcatcatcatcatcctcctcc180tcatcgggcaaatcgaacaagtccagccaaggtggtgctggcctagacggcccctggaag240cccaagggatgcagggcgttccctcactccgccgcgccggtgcacatgtttgagcgggtc300gtctcggccgaggacctcaagctcgcggccaagagcgacaatctcccgcaggcgtcttct360tcttccggcgccgcgcccaagttccggccggaaacgtggagcctccggcgcagcgtccac420gagggcgccaggaaggccggcaccgcggactgggacgagtggaagcgctgcttcaaggac480gagcacgccgacgccgagatgaagtttacgccctcggtcatggaccacgcgctcaagaag540cagtgggattgcacgggcgccgaggtcacgctcgggggcgacacgtacgaggacctgacg600ctcaagctggaggagtcgacgcacaagatgcccgcgccgctcaacaaccgcgtcttcccc660gtgctgcagctgacggcggcggtgaggggccggcgcgagttcatcgtcgttcagatcgcg720gccgtgcccgagtcttccaaggcggaagagcagaggccggatggggcggtcaggggcgcg780tacacgagcatcgagcgcttccgggaggtggacgagcagggcaacgtcgagtggctgatg840gggacggtgtcggatgcgaggggcgtgctgccggcgtggatccagaagatggcggttccg900gggcagattgcaaaggacgttgacatgtttctggagtggattgcaaaggagagggagggc960aacccccccaggcccttcgctgacgcagacgccgtcgcccgcataaccgtcgaactcaac1020gtccgcaccgtcagcgcccagacagagcgcctggaagaggagctcgccgccctcgcggag1080tacatcgaggcggacaaggagtttcgggagaagcacgacaagcggctgcagaacctgtgc1140aacgagatcctcgcggtgaagcagcgcgtggtcgagattcaggggcccgagtggaacgag1200ggcggcggcggcggcggcgagaaggccgaggcgggacagaaggaggtggacgaggcggtt1260gagcggctgaggagggagatgggcgagatgaggggcttggtcagtgatatgtcgagtgcg1320ctggacaagctcccgacggcggcggaggcagaggctttggtgaggcgctctcaggctccg1380gcttcagcgagcaacggggacgcaacgcaagcacctgctatagataaggcctcgggaacc1440aaagaggcacgatccatcaagcaacgcatcgaagacaccatcgcctccacccgccggtgg1500aacagcgaccacaagacgaccaagctcaccgacgccgtcttcatcgcgaattacctcaag1560cagcagtccaagcgcgatccccagatggctgtctacatgcagaggacgatccagaagcac1620gttcagaacagcggtaggcgtccgcggacaagggcgcgtccaaagagcctcgagcagttt1680tgcaggatgctggtgtggaaggacgtgcttgatacggcggaaattgtgctcgtgagggat1740tggagaaggactgcgagggcgttggaggagaggggttga1779<210>16<211>592<212>prt<213>里氏木霉<400>16metalaglntyrglyprotyrvalargleutrpglyileargproser151015glnleuproalagluthralaseralaglnaspleuvalpropheleu202530glnserileleuserglualavalpropheileserservalproleu354045glnserserserserserserserserserserserserserglylys505560serasnlysserserglnglyglyalaglyleuaspglyprotrplys65707580prolysglycysargalapheprohisseralaalaprovalhismet859095phegluargvalvalseralagluaspleulysleualaalalysser100105110aspasnleuproglnalaserserserserglyalaalaprolysphe115120125argprogluthrtrpserleuargargservalhisgluglyalaarg130135140lysalaglythralaasptrpaspglutrplysargcysphelysasp145150155160gluhisalaaspalaglumetlysphethrproservalmetasphis165170175alaleulyslysglntrpaspcysthrglyalagluvalthrleugly180185190glyaspthrtyrgluaspleuthrleulysleuglugluserthrhis195200205lysmetproalaproleuasnasnargvalpheprovalleuglnleu210215220thralaalavalargglyargargglupheilevalvalglnileala225230235240alavalprogluserserlysalaglugluglnargproaspglyala245250255valargglyalatyrthrserilegluargphearggluvalaspglu260265270glnglyasnvalglutrpleumetglythrvalseraspalaarggly275280285valleuproalatrpileglnlysmetalavalproglyglnileala290295300lysaspvalaspmetpheleuglutrpilealalysgluargglugly305310315320asnproproargprophealaaspalaaspalavalalaargilethr325330335valgluleuasnvalargthrvalseralaglnthrgluargleuglu340345350glugluleualaalaleualaglutyrileglualaasplysgluphe355360365argglulyshisasplysargleuglnasnleucysasngluileleu370375380alavallysglnargvalvalgluileglnglyproglutrpasnglu385390395400glyglyglyglyglyglyglulysalaglualaglyglnlysgluval405410415aspglualavalgluargleuargargglumetglyglumetarggly420425430leuvalseraspmetserseralaleuasplysleuprothralaala435440445glualaglualaleuvalargargserglnalaproalaseralaser450455460asnglyaspalathrglnalaproalaileasplysalaserglythr465470475480lysglualaargserilelysglnargilegluaspthrilealaser485490495thrargargtrpasnserasphislysthrthrlysleuthraspala500505510valpheilealaasntyrleulysglnglnserlysargaspprogln515520525metalavaltyrmetglnargthrileglnlyshisvalglnasnser530535540glyargargproargthrargalaargprolysserleugluglnphe545550555560cysargmetleuvaltrplysaspvalleuaspthralagluileval565570575leuvalargasptrpargargthralaargalaleuglugluarggly580585590<210>17<211>2658<212>dna<213>里氏木霉<400>17atgtccaagaccttcttcggcaatgtcaagagtgtcttgagcggcgacactctggtcctc60accagcgccaacaatcccgcggccgaacgcacattttcccttgcctacgtttcggcccct120cacctcaagcgagagggagacgagccatttgccttccaatcccgcgaataccttcgaaat180ctcgtggtgggcaagcccgttcaatgcacagtcctctataccattcccacgaccggcagg240gagtttggcactgctcagctcaaggacggaactctcctgcccgatgagcttgtcaaggcc300ggttgggtcaaggtccgtgaggacgctggccgcaaggaagagtcggaggagcttctcgat360aggctagagaagcttcgtgctctggaatccgaggccaagggcgcctccaaggggctctgg420tcgggcaccgacggcaccatcgaggtacagaatgacctgggtggccccgagttcctgacg480cagtggaagggcaagaccgtggatggcatcgtcgagagggtgttgagcggtgatcgtctg540ttggtccgactgctgctgtccgataagaaacacgttcagcccctgaccctccttgccggt600atccggactccgtccaccgagcgcacgcttccttccacgggtgctacgcagcctgccgag660gaatacggaaacgaggccaaagcgttcgttgagtcaagactgcttcagcgacaggtcaag720gtcgagattgtcggcgccagcgcccagggacagctcattgccagcgtcatccacccccga780ggcaacattgccgagttcctgcttcaagagggactggccaggtgtaacgacttccactcc840actatgcttggagagaagatggcacctctgagagccgccgagaagcaggctcaggccaag900aagctccgcctacacaggcatcacgtggccaaggcagacgctggaaccaacgagatggtt960gtcaccaagatcattggcgctgatacgatcatggtcaagggcaagaacgacaacacggag1020aagaggatcagcttcagcagcatccgagggcctcgcaccaacgagccctcggagagcccc1080ttcagagacgaggccaaggagtttgtgagatcaaggctcatcggcaagcacgtcaaggtg1140agcgtcgacggaaccaagcccgcttccgagggcttcgaggccagggatgtggccaccgtc1200accgagaagggaaagaacattggtcttgcgctggtggaagccggactggcctctgtcatt1260cgacaccggaaagatgataccgacagggcgcccaactatgatgagctgcttgcggctcag1320gagaaggccaaggaggagaagaagggcatgtggtccggcaagccccaaaaggccaagcag1380tacctggacctctcagaaaacacacaaaaggcaaagattatgctcgccacgctgcagcga1440cagaagaaggtgcctgccattgtcgacttctgcaaggccggctctcgattcaccattctc1500atcccgcgcgagaacgtcaagctgacgttggttctgggcggcatccgcgcccccagggcc1560cctcgcgccgatggacaaggcggcgagccgtttggcaaggaggccctcgacctggccaac1620cgacgatgcaaccagcgagactgcgaggtggacatccacgacatggacaaggtcggcggc1680ttcattggcagtctgtacattggccgcgagaactttgccaaggtgctggtcgaggagggc1740ctggcttcggtgcacgcctactctgccgaaaagtcgggtaacgctgccgagctgttcgcg1800gcggagaagagggcaaaggaggccagaaagggcatgtggcatgactacgatccttcgcag1860gaggagaacgccgaggaggagtctggcgaggccgacgcgcccgaggccgaagtcacactg1920gacaagaagcccgccgattaccgagacgtcatcatcaccagcattgacggcaacggcaag1980ctcaagatccaggagattggaaagggcacggccgcgctggagtccctgatgagcgacttc2040cgcaagttccacatcgattccaagaacaacaagcccctggcggaggctcccaagactggc2100gagtttgtgtctgccaagttctcggccgacgaccaatggtaccgtgcgcgagtccgggcc2160aacgaccgcacagccaagatgtccgaggtcatctacgtcgactacggcaacacggagaag2220gtgccctggtccagcctgcgatccctagaccagtctcagtttggtgtgcagaggctcaag2280gcccaagccattgacgcatcgctgtcgtttgtccagctgcccacgggcgcccattacttt2340agcgaggccattgcctttatcgctgatctgaccgagggcaggcggctggttggcaacttt2400gactatgtcgatagcaaggagaacgtcagctacatcacgctgtacgacaccaaggcggac2460ggctctctgcccggacccaacgactccattaacaaggagattgtggcgagcggatacggc2520atggtgcccaagaagctcaagtcatgggagcgcagcaaggcttttgaatcatacctgaag2580cacttgagagaggttgagagccaggcgaagcaggacaggctgggcatgtgggagtatggt2640gacattactgaggactaa2658<210>18<211>885<212>prt<213>里氏木霉<400>18metserlysthrphepheglyasnvallysservalleuserglyasp151015thrleuvalleuthrseralaasnasnproalaalagluargthrphe202530serleualatyrvalseralaprohisleulysarggluglyaspglu354045prophealapheglnserargglutyrleuargasnleuvalvalgly505560lysprovalglncysthrvalleutyrthrileprothrthrglyarg65707580glupheglythralaglnleulysaspglythrleuleuproaspglu859095leuvallysalaglytrpvallysvalarggluaspalaglyarglys100105110glugluserglugluleuleuaspargleuglulysleuargalaleu115120125gluserglualalysglyalaserlysglyleutrpserglythrasp130135140glythrilegluvalglnasnaspleuglyglyproglupheleuthr145150155160glntrplysglylysthrvalaspglyilevalgluargvalleuser165170175glyaspargleuleuvalargleuleuleuserasplyslyshisval180185190glnproleuthrleuleualaglyileargthrproserthrgluarg195200205thrleuproserthrglyalathrglnproalagluglutyrglyasn210215220glualalysalaphevalgluserargleuleuglnargglnvallys225230235240valgluilevalglyalaseralaglnglyglnleuilealaserval245250255ilehisproargglyasnilealaglupheleuleuglngluglyleu260265270alaargcysasnaspphehisserthrmetleuglyglulysmetala275280285proleuargalaalaglulysglnalaglnalalyslysleuargleu290295300hisarghishisvalalalysalaaspalaglythrasnglumetval305310315320valthrlysileileglyalaaspthrilemetvallysglylysasn325330335aspasnthrglulysargileserpheserserileargglyproarg340345350thrasngluprosergluserpropheargaspglualalysgluphe355360365valargserargleuileglylyshisvallysvalservalaspgly370375380thrlysproalasergluglypheglualaargaspvalalathrval385390395400thrglulysglylysasnileglyleualaleuvalglualaglyleu405410415alaservalilearghisarglysaspaspthraspargalaproasn420425430tyraspgluleuleualaalaglnglulysalalysgluglulyslys435440445glymettrpserglylysproglnlysalalysglntyrleuaspleu450455460sergluasnthrglnlysalalysilemetleualathrleuglnarg465470475480glnlyslysvalproalailevalaspphecyslysalaglyserarg485490495phethrileleuileproarggluasnvallysleuthrleuvalleu500505510glyglyileargalaproargalaproargalaaspglyglnglygly515520525glupropheglylysglualaleuaspleualaasnargargcysasn530535540glnargaspcysgluvalaspilehisaspmetasplysvalglygly545550555560pheileglyserleutyrileglyarggluasnphealalysvalleu565570575valglugluglyleualaservalhisalatyrseralaglulysser580585590glyasnalaalagluleuphealaalaglulysargalalysgluala595600605arglysglymettrphisasptyraspproserglnglugluasnala610615620gluglugluserglyglualaaspalaproglualagluvalthrleu625630635640asplyslysproalaasptyrargaspvalileilethrserileasp645650655glyasnglylysleulysileglngluileglylysglythralaala660665670leugluserleumetseraspphearglysphehisileaspserlys675680685asnasnlysproleualaglualaprolysthrglygluphevalser690695700alalyspheseralaaspaspglntrptyrargalaargvalargala705710715720asnaspargthralalysmetsergluvaliletyrvalasptyrgly725730735asnthrglulysvalprotrpserserleuargserleuaspglnser740745750glnpheglyvalglnargleulysalaglnalaileaspalaserleu755760765serphevalglnleuprothrglyalahistyrpheserglualaile770775780alapheilealaaspleuthrgluglyargargleuvalglyasnphe785790795800asptyrvalaspserlysgluasnvalsertyrilethrleutyrasp805810815thrlysalaaspglyserleuproglyproasnaspserileasnlys820825830gluilevalalaserglytyrglymetvalprolyslysleulysser835840845trpgluargserlysalapheglusertyrleulyshisleuargglu850855860valgluserglnalalysglnaspargleuglymettrpglutyrgly865870875880aspilethrgluasp885<210>19<211>725<212>dna<213>里氏木霉<400>19atgggtgtcggaacattctttcactacttcggcacggtgctgctgcttgccgccacggcg60ctcctcatcgtcgtgtccgtgactgcgcccgtcgtcaacgacctctctctcctgaagatc120aacttcagcggcagcagctctgtcgacagaataacatttggtacctttggttactgcatc180caaaatgcaaagtacgtcttggttcatctactcattactctcaaacactaccatatgaga240aacacaatagctaacatctacaaagcggacacgatgcatgcacccactctcacatcggct300acgagggcaccgacgccttgacccaggtcacctccctcaacttctcaaagtccgaccgcg360atggcttcaggattctcacaaaggtcatgatcctgcaccccatcgccgccggcctcacct420tcttggccttcctcctctgcctgggcaccagcttcctcggctcctttgtcgcgtccttct480tctccttcctggcctttgtcgccaccgtcatcgccatggcctgcgactttgccggcttcg540agatcatcaagcacgccgtcaacaccaggggccctgccaacgtcgaggcccactggggac600ctgccgtctggtgcattctggctgctgcggcgctgacgctgattgcgaccatcttggtgt660ttgtcacttgctgtgccggtcgtgtcaagaaccgccgcactcgtcgcagcaagtccgagt720actaa725<210>20<211>216<212>prt<213>里氏木霉<400>20metglyvalglythrphephehistyrpheglythrvalleuleuleu151015alaalathralaleuleuilevalvalservalthralaprovalval202530asnaspleuserleuleulysileasnpheserglyserserserval354045aspargilethrpheglythrpheglytyrcysileglnasnalaasn505560glyhisaspalacysthrhisserhisileglytyrgluglythrasp65707580alaleuthrglnvalthrserleuasnpheserlysseraspargasp859095glypheargileleuthrlysvalmetileleuhisproilealaala100105110glyleuthrpheleualapheleuleucysleuglythrserpheleu115120125glyserphevalalaserphepheserpheleualaphevalalathr130135140valilealametalacysaspphealaglyphegluileilelyshis145150155160alavalasnthrargglyproalaasnvalglualahistrpglypro165170175alavaltrpcysileleualaalaalaalaleuthrleuilealathr180185190ileleuvalphevalthrcyscysalaglyargvallysasnargarg195200205thrargargserlysserglutyr210215<210>21<211>606<212>dna<213>里氏木霉<400>21atggctcccaagattgccatcgtctactactccatgtacggccacatccggcagctcgcc60gaggctgagaagaagggcgccgaggccgccggcgcccaggtcgacatctaccagatcccc120gagaccctccccgacgacgtcctgggcaagatgcacgcccccgccaagcccaccgacgtc180cccgtcctcgaggaccccgccaccctcacccagtacgacggcttcctgtttggcattccc240actcgttatggaaactttcccgcccagtggaaggccttctgggacaagaccggcggcatc300tgggcctcgggcggctactggggcaagaaggccggcctcttcatctccaccggcaccccc360ggcggcggccaggagtccaccgccatcgccgccctgtccaccctggcgcaccacggcatc420atctacgtgcccctcggctacgcaaagaccttccccgacatcaccaacctcgaagaggtc480cacggcggctccccctggggcgccggcacctacgccggccccaccggcgcccgccagccc540accgccctcgagctcaagatcgccaccgcccagggcgaggcctttgcccaggccctctcc600ggctga606<210>22<211>108<212>prt<213>里氏木霉<400>22metserglylysglyglyglnglyglyaspglyglyglyleuleuala151015alaalaglyglyalaglyglyaspgluglualaglyleuleualapro202530valalaalaargglyproaspalaalaglyleuvalprogluglyleu354045proleuglyglylysvalserilethrserglyasnalalysglnglu505560alavalvalleuglygluglyglyglyvalleugluaspglyaspval65707580glyglyleuglyglyglyvalhisleualaglnaspvalvalglyglu859095alaseralasercysargmettrpprotyrmetglu100105<210>23<211>2838<212>dna<213>里氏木霉<400>23atgacagacgacccggatcgcgcgtgtccggctccgacaatcgccgaggacgcggttgcg60aagagcgcgatcacgcaggcggccggcgtcgaggttccagctccagctccagctacagct120ccagctacagctcctacagctcctacagctcctacagctcctacagctcctacagctccc180acagctcctacagctccaactacggctccaaatgcggcatcagctccaacaccggctcca240gctctagctgagggcaccagtgccgagcctcaggaagcgcgctccaggaagaagcggctg300cagctgagctgcggcgagtgtcgcaggaaaaaggtaacggtaacgcggcagaggagaaga360gcaaaccgatgagagatcgcgtctggcaagttcgtgttctaaccagcaacccatcgccag420ctctcctgcgacagaggccggccgtgccgcagatgcgtcaggacgggcagagcagaccaa480tgcgagtttgagaccaacccgcgttcagcgctgctgcccatcgaccagggtgcgcagctg540gaacagatcaagtccgaccaggccgaacttcagagcctgcgggctgagatcttccagctc600aaggagctgctctcacggcctctggcgcctcctccgcctccaagtcaccctcagcctcag660cctcagccccagccccagctacatcctcaccatggcagaggctccctcacggaagactcg720cgcgtcagcgacttctcctccaaaagaagcagtgagatggaatccaaggacattgtgacc780atcagccggattcccaacagctttagtgacggcccgcctgatcccagggaaaaatcgcca840cgaggatactacagacggcacgccctgctgaggttcttttcagaggtaatctcgcgcctg900actctaccgtgcttaccgttgattgttttaacccggcgcaacagattccacagctgtttc960cattcatcaaggagattgccgaccagtggctgaagccctatggaatctacatcaagaaga1020acaaggtcgccaaagatgagagctggaggatgaagatggcagccaacgagcagcctctga1080ttgaaggcctcttgccgccaaagcatgacacggacgttctgattgcgatataccttgatc1140atttcgagcaactgcatcgcattgtccacatcccgaccttcaacagggagtatatcaact1200tctggacgcccggccgaccgcggtacccgacaatggcagcaatgatactcgccatgatct1260ccgtgtcggtctgtgcctccgtccaccttgttggctcttcgccagtccctgccagctatc1320gaaaaatggctgagacgtggatttccgccatcgatgactggctgcgactgcaaagcacaa1380agcaccgcaagctcgtgcactaccaggtggccagcttgatgtacattgccaagcggatga1440acctggtagggaagaagaggttctggaaagatacgggctccatgatccaggacgcaatca1500tggatggcttaaatcgcgatccttcactggcagataccttcttcatcagggagatgaagc1560gacggctgtggtacaccgtccgagagctggagcttcagaactcgtttgaatgcggcttgc1620ccactcttcttcatacgattgagtcgaatgtcacagcgccgctcaaccttgctgatgaag1680attttgacgacacaacgaagctgacgcccatgtcgaggccgtccaattcctacacggagt1740cttcgtaccagttccacagcttccgaagctgggagttgcgtctcgagctgtcacgacgtc1800agtttggttccggagttttcaggcctctcgattatgacgacgttttgcggtacacacacg1860agatcacacgagcattgaacgatctccctccgtggagcaatgacaggactgacagcaaaa1920ctgatggacgggtgctcctgtcgtactcgatgctggagtttcagctcaaggagtgtctcc1980tggccatccatcggccatatgtggacagggaaggcggcaaataccccatctcggaaacga2040tctgttaccagacagcgcgagaaatactgctttccaacatacagctcgcagccttgggag2100ttcaaagtctgacacaactccgcgaagaccttgcaattgcttcgctgcacatgactcgcc2160ttacactcatgcaacaccaaggtgggctcttctttcttcttttgtatatctaaggaggtc2220ttctgacttgtagaaggttccaacagcatcatcatggtcaatgccttgtcgactgtcgac2280ctgctggagcagtgcctcccggccattgaggataaatatctgcggttcgccgacccttgg2340agctttttcatgatgtgtacagccattatgttgatcaaaatacacctcggaaaagagact2400cgccaaactgccaaggcggcttgtgcacgcagatttctggatctgtactacaagagtgtt2460tggatgcatcacccctctgacctcgctcaacagcaggcaatttctcgggatatcgccaac2520caaacaagtgtgagttctatgataaccgttcaggattacgctatttcttctggcacagcc2580ttcggggatcaatctaagcttacttttacgtctatcctaggctcccgctgcctgtgctcc2640agctccttcagacggaagcgtccttcccacctcggtgtggcttgaaaacagctatcccga2700tgtatgtccaaaggcttgctaataatgctgcatgtactaatcctcccacagattgggacc2760gatccgttcgacctatccgtcgagatggacgtctgggacgaggcgggatttcctctgccg2820ggatttcccaactgctag2838<210>24<211>832<212>prt<213>里氏木霉<400>24metthraspaspproaspargalacysproalaprothrilealaglu151015aspalavalalalysseralailethrglnalaalaglyvalgluval202530proalaproalaproalathralaproalathralaprothralapro354045thralaprothralaprothralaprothralaprothralaprothr505560alaprothrthralaproasnalaalaseralaprothrproalapro65707580alaleualagluglythrseralagluproglnglualaargserarg859095lyslysargleuglnleusercysglyglucysargarglyslysleu100105110sercysaspargglyargprocysargargcysvalargthrglyarg115120125alaaspglncysgluphegluthrasnproargseralaleuleupro130135140ileaspglnglyalaglnleugluglnilelysseraspglnalaglu145150155160leuglnserleuargalagluilepheglnleulysgluleuleuser165170175argproleualaproproproproproserhisproglnproglnpro180185190glnproglnproglnleuhisprohishisglyargglyserleuthr195200205gluaspserargvalserasppheserserlysargserserglumet210215220gluserlysaspilevalthrileserargileproasnserpheser225230235240aspglyproproaspproargglulysserproargglytyrtyrarg245250255arghisalaleuleuargphephesergluglnileproglnleuphe260265270propheilelysgluilealaaspglntrpleulysprotyrglyile275280285tyrilelyslysasnlysvalalalysaspglusertrpargmetlys290295300metalaalaasngluglnproleuilegluglyleuleuproprolys305310315320hisaspthraspvalleuilealailetyrleuasphispheglugln325330335leuhisargilevalhisileprothrpheasnargglutyrileasn340345350phetrpthrproglyargproargtyrprothrmetalaalametile355360365leualametileservalservalcysalaservalhisleuvalgly370375380serserprovalproalasertyrarglysmetalagluthrtrpile385390395400seralaileaspasptrpleuargleuglnserthrlyshisarglys405410415leuvalhistyrglnvalalaserleumettyrilealalysargmet420425430asnleuvalglylyslysargphetrplysaspthrglysermetile435440445glnaspalailemetaspglyleuasnargaspproserleualaasp450455460thrphepheileargglumetlysargargleutrptyrthrvalarg465470475480gluleugluleuglnasnserpheglucysglyleuprothrleuleu485490495histhrilegluserasnvalthralaproleuasnleualaaspglu500505510asppheaspaspthrthrlysleuthrprometserargproserasn515520525sertyrthrglusersertyrglnphehisserpheargsertrpglu530535540leuargleugluleuserargargglnpheglyserglyvalphearg545550555560proleuasptyraspaspvalleuargtyrthrhisgluilethrarg565570575alaleuasnaspleuproprotrpserasnaspargthraspserlys580585590thraspglyargvalleuleusertyrsermetleuglupheglnleu595600605lysglucysleuleualailehisargprotyrvalaspargglugly610615620glylystyrproilesergluthrilecystyrglnthralaargglu625630635640ileleuleuserasnileglnleualaalaleuglyvalglnserleu645650655thrglnleuarggluaspleualailealaserleuhismetthrarg660665670leuthrleumetglnhisglnglyserasnserileilemetvalasn675680685alaleuserthrvalaspleuleugluglncysleuproalaileglu690695700asplystyrleuargphealaaspprotrpserphephemetmetcys705710715720thralailemetleuilelysilehisleuglylysgluthrarggln725730735thralalysalaalacysalaargargpheleuaspleutyrtyrlys740745750servaltrpmethishisproseraspleualaglnglnglnalaile755760765serargaspilealaasnglnthrseralaproalaalacysalapro770775780alaproseraspglyservalleuprothrservaltrpleugluasn785790795800sertyrproaspileglythrasppropheaspleuservalglumet805810815aspvaltrpaspglualaglypheproleuproglypheproasncys820825830<210>25<211>2626<212>dna<213>里氏木霉<400>25atgtcgacggctaaagcgaccaagaagagcgcgttttcctgcgagccatgtcgccgccgc60aaggtaagcctggctgcttggagatcgtgttattggcgtgtctctggctttgctgggagg120gatggagagagtgtaagagaggcatcttggagctgactctgctggtggcgttaggtcaag180tgcggcggcgagcagcccatgtgtcaacgctgcgttgctcgcaacgatgattgcgtctac240aaattgtgtgacttgatatctcactccgggcacatctacatctgcctagtagcgtctgat300cttggcggctgacacccatctgcaggaacccgacgctgtcctacacgcaacggctggagg360atcgcatcaaagagctggaggagcagcttgcacaagtcacggctgcaacaccgccaccgg420cggccattggcagcaaatcccctccatcaagtcatccaagccctacagcctcggggagcg480gtcagcaagacacccggcagcagatcgatgacagcatctcccggagcttccgcggcctca540agatagaccaaaagggcggcgtcacctaccatgggaccacgagcttcttccatctcccca600gtgaccgtagctctgcggcactaactgccgacatgcacgcagccgcgactgacattgaaa660ctcagcggagagagcgactagtttcgaacgcgtggcagcagcgcttcttggaggagactg720ctggggttcccgtacgtgtccaagtgtggtggtcgtcgcttgacagctcaagtctcatgc780cttaactctctcgcaggaaccctttcagacgcttctgaatgttcactggtgttggataca840gcccctcttcaacttcatctaccgaccagcatttacccgtacgaaacccttgtagcttgt900catgacttttcccgccccctttcttctcttccctgatatttaccaatcatgtaggcgata960tgcagtccatggggccgtactactcacatacgcttctcaatgctgttctctcccactcca1020tacgctgggcgaagagcgaccccaagacaaagcagattctcgatgagtcgtacgacggag1080gcgccgtctttggcaagcacgcacgcagcatgcttttcgacgagctcagcaggggagtat1140gcacgatcccgacggtccaaacactacttctcctaagcgcacaggaatgcagccttggga1200acacaacccaagcctggacgtatagtggtctggcgtttcggctgattgaccacttgggga1260tatgtgtggatggagatcgatatatcaactcggtccacttcaccgacgaagacttggaaa1320tccggcatcgagtcttttggtcgtgctatttctgggacaagatcatcagtctgtatcttg1380gtcgatcgccatcactcaagcacacgacggtctctccccctcagattatgtgtaagtctg1440attgtcgatactcatctcttgtcgttgtcgctcacattggcggtggttggttggtcgttg1500atgctgacggctgtttcttataagtcgacgactctgctgaaaacgagctgtgggttccct1560tcggggcgcctccactccaaaccaccccctggaagtacccacctgctactgctcattcgg1620cttcttgctttctgagcatgtgccggctgtcagtcatcttcaacgagatattgattcata1680tgtacgacccgctcatgcagaacaccgagtccgaggtgctcgagtgcctgacgacccagg1740agccggccctgcagcaatggtgggatgagcttgctccatatctcaagctcgagcctatgg1800ctctgcccagcttggccccaccgtctcacatcgtcacgaccaagtaagttcttgccgtcc1860caagtgccgaacgccgttctccgttctccgttaaccggtccaaatagctgcctctatcac1920acgttcaaggtgctattgtaccgtccaatgctcacccggagaatactggagggcgatgtt1980gcgtcgaaccagcgctacctcgtcgagtgcgtatcgtccgccacggccatcatcgctatt2040tttgatctcttctgccgaacctttaccatgaactactgtgtgctgtcgctggcttacagc2100gtctacattgcttcgtcgatattcctgctacaggttcaggcggctccgggcgacggacaa2160gctctagggcggctcaactactgtatgcaggccttgaagcaagtcaagactttcagcccg2220ggtaagtcgagtattggcgcgactttgcttcttttgtttttctgcttgtcgttcggctcg2280atcctgacgttttgtagtcatcggcagcgctatcagcgcgctgaatagggaactctcggc2340tgtcggcatctcgctggacgtgccccagcaccagcagccacccccaatccccgtcactga2400gtttcctccaatagccccaggatttccggccgtcgatgtgcccgtcgatgcgtcgtcttc2460gcagcctcagcctgctcatccgctgtttcaggttccctcaacccacagctatggtgccca2520ttcaatggccatggacccaggcatctttgaggccatgtcgtccctggagccactcagcgt2580ccgagttggggccctttcccctacggaaaatcaggactcgttttga2626<210>26<211>686<212>prt<213>里氏木霉<400>26metserthralalysalathrlyslysseralaphesercysglupro151015cysargargarglysvallyscysglyglygluglnprometcysgln202530argcysvalalaargasnaspaspcysvaltyrlysleuasnprothr354045leusertyrthrglnargleugluaspargilelysgluleugluglu505560glnleualaglnvalthralaalathrproproproalaalailegly65707580serlysserproproserserhisproserprothralaserglyser859095glyglnglnaspthrargglnglnileaspaspserileserargser100105110pheargglyleulysileaspglnlysglyglyvalthrtyrhisgly115120125thrthrserphephehisleuproseraspargserseralaalaleu130135140thralaaspmethisalaalaalathraspilegluthrglnargarg145150155160gluargleuvalserasnalatrpglnglnargpheleuglugluthr165170175alaglyvalproglupropheglnthrleuleuasnvalhistrpcys180185190trpileglnproleupheasnpheiletyrargproalaphethrarg195200205aspmetglnsermetglyprotyrtyrserhisthrleuleuasnala210215220valleuserhisserileargtrpalalysseraspprolysthrlys225230235240glnileleuaspglusertyraspglyglyalavalpheglylyshis245250255alaargsermetleupheaspgluleuserargglyvalcysthrile260265270prothrvalglnthrleuleuleuleuseralaglnglucysserleu275280285glyasnthrthrglnalatrpthrtyrserglyleualapheargleu290295300ileasphisleuglyilecysvalaspglyaspargtyrileasnser305310315320valhisphethraspgluaspleugluilearghisargvalphetrp325330335sercystyrphetrpasplysileileserleutyrleuglyargser340345350proserleulyshisthrthrvalserproproglnilemetpheasp355360365aspseralagluasngluleutrpvalpropheglyalaproproleu370375380glnthrthrprotrplystyrproproalathralahisseralaser385390395400cyspheleusermetcysargleuservalilepheasngluileleu405410415ilehismettyraspproleumetglnasnthrglusergluvalleu420425430glucysleuthrthrglngluproalaleuglnglntrptrpaspglu435440445leualaprotyrleulysleugluprometalaleuproserleuala450455460proproserhisilevalthrthrasncysleutyrhisthrphelys465470475480valleuleutyrargprometleuthrargargileleugluglyasp485490495valalaserasnglnargtyrleuvalglucysvalserseralathr500505510alaileilealailepheaspleuphecysargthrphethrmetasn515520525tyrcysvalleuserleualatyrservaltyrilealaserserile530535540pheleuleuglnvalglnalaalaproglyaspglyglnalaleugly545550555560argleuasntyrcysmetglnalaleulysglnvallysthrpheser565570575provalileglyseralaileseralaleuasnarggluleuserala580585590valglyileserleuaspvalproglnhisglnglnproproproile595600605provalthrglupheproproilealaproglypheproalavalasp610615620valprovalaspalaserserserglnproglnproalahisproleu625630635640pheglnvalproserthrhissertyrglyalahissermetalamet645650655aspproglyilepheglualametserserleugluproleuserval660665670argvalglyalaleuserprothrgluasnglnaspserphe675680685<210>27<211>2337<212>dna<213>里氏木霉<400>27atggatcaacagcagaaacaacaacagccatgtcgccgccggagacccgctctctcatgt60attgagtgccggcgccgcaagctcaagtgcgacagaaagagcccttgcagccgctgtgtc120gcgaccgagacacagtgctcttacaccacatactaccgggataagccccgagcagggcat180gggcaagatagccggatgcacgtgtcttcctcttcgatgccaagcccgtccactgtccat240ccggcatctactccgtcacatgtcgataccgatggtccaagtatcgttggtacagctact300agcgttagagagggatcactccgtgtcggtcggacaatcgaggctcgcgccgcagcgaac360tcgacactcggttcagctgagtcgtcaaccgcaccagcggctgacgagatccagattcca420aatcgccctcggcttctgaaccctgagctccaggatcttagaggtagggtgccagtgcac480gatctatcccaggcaggccgggatatgctcactggtgagccgggtttgccggacgcccgt540atcattttgaacaagacgaggattatgagatggagtcattgggttggcatagcaaaagag600gtgcgtagagtatttgtgtagatcgtctactcacatctctcgtgaattagacgtgcattg660atcgctaacattttggtcgtgcagttcgcccccatcatcacttgctacggagcagcttgc720ggctttggcgacatcaacgcctttcataacgccgagacgagagaatccattcttgaagtc780agcgagctgctccagaaatgcaaaagcgtagccagaagcctcaaaaccgggcggccgagt840agatgcctttcctcgctgaactttgatcttacaccccctactcgcgaggtggcagacaag900atgacaaccttgtactttcagtcctttgagtcaactcatcgcatcctccatcagccgacc960ttctggaaagtctacgaaaggttctggagtagcccagagagcgcttcggccaagttgcgg1020ctccaagtccttctcgtcattgcaataggctccagcctggcctcgcattcgggagctaat1080gccggacttcgcaatatggtccatcaatggatatatgcagcagaaatctggctctcaggc1140ccactggaaaaagaccgcctcgatatatccgggctccagatacactgtctggtgatgctg1200gcacgacaaatcttctccattggaggcgacctcgtctggatatcctcggggtcgctcatc1260catcgggcaatgcagattggcttgcaccgagaccccaggcaccttccaaacatgtctgtg1320ctgcagacgcagttacgcagacgattatgggctacgatattggagttggttgttcagtcg1380tccctggattcggctttaccgccgaggatctcttttgacgaatttgacacggagccgccc1440gccaattgcaatgaccaagatatagaagagccagtgcctgagctgaagcctcacagccga1500gaacggttcactggaacgtctttgcagctcattctactcgattcgttaccaacgcgttta1560cgtatcctcagcctcctgaacggcctgcactcggagttgtcctatctagacgtgcttgac1620ttgagcacggatattctcgaggcatgtcgtcgttcgaacgccttttttagggataatcaa1680gcctctggaatgactccgttccaccacaatctggttgactacctcctacgtcggttcctg1740attccccttcattgtccgtttgcgcacaaggcacggacgaacccgctgttacactattcg1800ctcaaggtcagtctagatgctgccatggctatagtctatccagagccagatgaaggtttc1860tccagtctgatggcaattggtggaggcctgttcagggagggtattcgatatgcgactagt1920atcatcagtctggagcttctgacacagattgaagcccactataaagatggaactttacat1980cgaagttcgcactccattagccacttgaagaaggcgatggaggatttgatgagtctttcc2040ctggagcgcgttcgtcaaggggagactaacgtcaaaaaccacatgttcttgtccatggtc2100atggctcaagcggaggctatggaacggggtgaatccggtcaacttcgcatcgcgaagagc2160gccagagatagtgttttgctgagcctggaactactacaaagcactcttggtgattctcct2220gtggcaacttctgtccaagagttcgattccgcaagcttcggcgaaagagaggactacaca2280tatggactggggcttgacatggacttcttcttcctcaccgactcaggctttccttga2337<210>28<211>750<212>prt<213>里氏木霉<400>28metaspglnglnglnlysglnglnglnprocysargargargargpro151015alaleusercysileglucysargargarglysleulyscysasparg202530lysserprocysserargcysvalalathrgluthrglncyssertyr354045thrthrtyrtyrargasplysproargalaglyhisglyglnaspser505560argmethisvalsersersersermetproserproserthrvalhis65707580proalaserthrproserhisvalaspthraspglyproserileval859095glythralathrservalarggluglyserleuargvalglyargthr100105110ileglualaargalaalaalaasnserthrleuglyseralagluser115120125serthralaproalaalaaspgluileglnileproasnargproarg130135140leuleuasnprogluleuglnaspleuargglyargvalprovalhis145150155160aspleuserglnalaglyargaspmetleuthrglygluproglyleu165170175proaspalaargileileleuasnlysthrargilemetargtrpser180185190histrpvalglyilealalysgluphealaproileilethrcystyr195200205glyalaalacysglypheglyaspileasnalaphehisasnalaglu210215220thrarggluserileleugluvalsergluleuleuglnlyscyslys225230235240servalalaargserleulysthrglyargproserargcysleuser245250255serleuasnpheaspleuthrproprothrarggluvalalaasplys260265270metthrthrleutyrpheglnserphegluserthrhisargileleu275280285hisglnprothrphetrplysvaltyrgluargphetrpserserpro290295300gluseralaseralalysleuargleuglnvalleuleuvalileala305310315320ileglyserserleualaserhisserglyalaasnalaglyleuarg325330335asnmetvalhisglntrpiletyralaalagluiletrpleusergly340345350proleuglulysaspargleuaspileserglyleuglnilehiscys355360365leuvalmetleualaargglnilepheserileglyglyaspleuval370375380trpileserserglyserleuilehisargalametglnileglyleu385390395400hisargaspproarghisleuproasnmetservalleuglnthrgln405410415leuargargargleutrpalathrileleugluleuvalvalglnser420425430serleuaspseralaleuproproargileserpheaspglupheasp435440445thrgluproproalaasncysasnaspglnaspileglugluproval450455460progluleulysprohisserarggluargphethrglythrserleu465470475480glnleuileleuleuaspserleuprothrargleuargileleuser485490495leuleuasnglyleuhissergluleusertyrleuaspvalleuasp500505510leuserthraspileleuglualacysargargserasnalaphephe515520525argaspasnglnalaserglymetthrprophehishisasnleuval530535540asptyrleuleuargargpheleuileproleuhiscyspropheala545550555560hislysalaargthrasnproleuleuhistyrserleulysvalser565570575leuaspalaalametalailevaltyrprogluproaspgluglyphe580585590serserleumetalaileglyglyglyleuphearggluglyilearg595600605tyralathrserileileserleugluleuleuthrglnilegluala610615620histyrlysaspglythrleuhisargserserhisserileserhis625630635640leulyslysalametgluaspleumetserleuserleugluargval645650655argglnglygluthrasnvallysasnhismetpheleusermetval660665670metalaglnalaglualametgluargglygluserglyglnleuarg675680685ilealalysseralaargaspservalleuleuserleugluleuleu690695700glnserthrleuglyaspserprovalalathrservalglngluphe705710715720aspseralaserpheglygluarggluasptyrthrtyrglyleugly725730735leuaspmetaspphephepheleuthraspserglyphepro740745750<210>29<211>2311<212>dna<213>里氏木霉<400>29atggaggagccttatctcctaccaacagggaacccgctgcctcatcgcgtgagaaagatg60cgcaagggaactcgaagctgcaccgaatgtatgtcctgtatagacgagttgtatctggta120gcagatagacactgatagcagtctttgacaaggccgaagacgcaaaacgcgctgtgtttt180ttctcccggtgacaccgtatgcgtgctgtgcaagtcccggggcagtcgctgtatcgaaca240aggttacgaggacgccagtcttccaaatcccagatctggtgcagctcggaaaagccctac300cgccgagaaggcgtctcagagtcggaatgagccatctgtagatcagccccccaagttctg360tggagatgcagagacatcagatgcccttgcctcagagagagcaaaccatgctccgattgt420atctctactcgtggatgtcaaggtcagttaagcaagattcaaaggccgactgcatccaca480tgcatgcattcacaagcgctttgaaggcgagctaatcgatggctttgcccaatgcacagc540tctcggcatcggcgaagggcggacaagacgagccctcaaatccaaaaccaatgccttggt600ccatgcagcctcttggcagcaaatctgcatatgtgtgctctacgatccgctcgatgctac660ccagctatgataccatcatatccgttctcactagaaacgggtcatggtgggacagcttcc720gctcaaaggcatatgccatttccgaggcgccttcccagacgatcgaagccttcgcgaaga780ggacctatacgagcagcaacccggccgatataggtgctctggccattgcatttgctcgga840gcttgaataagcatcgctatctgtatactctggtagatgacctcgtcatctcggacatca900acttcctgacgactatagaggggctggaatgcctcatccttctcgcaaagtcatataccg960actgcggccagccaaaaagagcctggcttgtatggagaaaaggtgcatctgcaacgcagc1020ttatggtgagtcagtccttgtttggctcagccatccggagggacgctggcctcgtctgac1080tcgtcattagggactttgctgtggtagtaactacagccatacagaaatgaggctatggtg1140ctcagtatatcacggcgacagattctgtagtatgctgctcggcttgccttatgtcctcaa1200cgacaatcactaccagtctgttatcaacgcgccggggatggccccaggcttttattttgt1260cctccgatgcgccatcatctgtggaaagatcattgaccggaatatgactgtcggcaagcc1320gtcattcgcgagagccatggaactagacgaagagatggaaagcgttgcgtcgtcacagcc1380acaggactggtgggccgttacagagaccttggaccagccgctcgaacatcttgagctcaa1440cgagctcagagagcagctattgcaacaattctacttctactgggtcaagctgttcctcca1500tctgccattcctggtggagacgacaacgagctcgccacactacttcagcagaatggcctg1560catagaggcatccaagcagattctaaagagatatcgactcttacgcaccagaatgaggtc1620gggccactgcttgttcgagtgcaagacaacagactttgcgtgcttcaccgctgctgttgt1680tcttctcattggcacgttccacaccagcgccacgtgtcactcgtccaacagcgtggagga1740tgacctagagctggtggcagacaccgataggctgttgctcgcagaagaagttgaaaacga1800ctgcaaggttgccgcgcaatgccggaaagtgctccagatgttaagtattagggaggctga1860accagcggaagcagatcgggaagtcgtaatcccgtacttcggggccgtgatccgcaaacg1920ttctcaccgcacacagccggatcctcggcctgtcactctgcctcggccgggtgggcactc1980aagtaaggacctcggcggccttgatattcgtccaccgcctccgtacagctgcgccgaaca2040gacagcggccataccagccgtggaaaatccgtggagtctggaaggattctcgctagagta2100tataagccatcgtggatttgacagggccaatttgggcggattcctaggggcggatgaggc2160cgcctgcacgcaggatgattcgacatcgtatatgacctcggcggcaatggagtgggatcc2220aggttggagcgttcttgatgatatagatgataccggcagagaccttttgacagggattga2280ggactggtctgcagaggacttcgagatgtag2311<210>30<211>694<212>prt<213>里氏木霉<400>30metglugluprotyrleuleuprothrglyasnproleuprohisarg151015valarglysmetarglysglythrargsercysthrglucysargarg202530arglysthrargcysvalpheserproglyaspthrvalcysvalleu354045cyslysserargglyserargcysilegluglnglytyrgluaspala505560serleuproasnproargserglyalaalaarglysserprothrala65707580glulysalaserglnserargasngluproservalaspglnpropro859095lysphecysglyaspalagluthrseraspalaleualasergluarg100105110alaasnhisalaproilevalserleuleuvalaspvallysleuser115120125alaseralalysglyglyglnaspgluproserasnprolyspromet130135140protrpsermetglnproleuglyserlysseralatyrvalcysser145150155160thrileargsermetleuprosertyraspthrileileservalleu165170175thrargasnglysertrptrpaspserpheargserlysalatyrala180185190ileserglualaproserglnthrileglualaphealalysargthr195200205tyrthrserserasnproalaaspileglyalaleualailealaphe210215220alaargserleuasnlyshisargtyrleutyrthrleuvalaspasp225230235240leuvalileseraspileasnpheleuthrthrilegluglyleuglu245250255cysleuileleuleualalyssertyrthraspcysglyglnprolys260265270argalatrpleuvaltrparglysglyalaseralathrglnleumet275280285glyleucyscysglyserasntyrserhisthrglumetargleutrp290295300cysservaltyrhisglyaspargphecyssermetleuleuglyleu305310315320protyrvalleuasnaspasnhistyrglnservalileasnalapro325330335glymetalaproglyphetyrphevalleuargcysalaileilecys340345350glylysileileaspargasnmetthrvalglylysproserpheala355360365argalametgluleuaspgluglumetgluservalalasersergln370375380proglnasptrptrpalavalthrgluthrleuaspglnproleuglu385390395400hisleugluleuasngluleuarggluglnleuleuglnglnphetyr405410415phetyrtrpvallysleupheleuhisleupropheleuvalgluthr420425430thrthrserserprohistyrpheserargmetalacysilegluala435440445serlysglnileleulysargtyrargleuleuargthrargmetarg450455460serglyhiscysleupheglucyslysthrthraspphealacysphe465470475480thralaalavalvalleuleuileglythrphehisthrseralathr485490495cyshisserserasnservalgluaspaspleugluleuvalalaasp500505510thraspargleuleuleualaglugluvalgluasnaspcyslysval515520525alaalaglncysarglysvalleuglnmetleuserilearggluala530535540gluproalaglualaasparggluvalvalileprotyrpheglyala545550555560valilearglysargserhisargthrglnproaspproargproval565570575thrleuproargproglyglyhisserserlysaspleuglyglyleu580585590aspileargproproproprotyrsercysalagluglnthralaala595600605ileproalavalgluasnprotrpserleugluglypheserleuglu610615620tyrileserhisargglypheaspargalaasnleuglyglypheleu625630635640glyalaaspglualaalacysthrglnaspaspserthrsertyrmet645650655thrseralaalametglutrpaspproglytrpservalleuaspasp660665670ileaspaspthrglyargaspleuleuthrglyilegluasptrpser675680685alagluasppheglumet690<210>31<211>2861<212>dna<213>里氏木霉<400>31atggagggtgcagaccgtggcaaaggtacctggatgtacccgccagcgaccgcgacgcca60taccttccccagccggtgcaggtgcgtcgatgcttccttctgtccagcaacagcaacagc120agcagcacgactccaagcttcattcttccacggcttgactttttgcatctctgcagcttc180atccacctccattgatcgtagatcgtcagattcccatcacatcaaatcaatgtcactgac240tcacacttccagctttcacccctgactctccgtccttcgctgcttccgtctctagacttg300cgcctctcaccgagtcacccccagtttttccgctcgcagcttgaagcctccacctcaacc360tcgagtctctctgctcctgcttacttgctcgccgcgcattgcgacagctccgagactcaa420tcgcctcccgcgctctccttttcgccgtcgaccgcgagctcgatcgcgacgccgttgccc480ggctcagtcgcagcaatggacctcgacccgcctcccgacgccgacatgccgtcattcgac540ctcgccggatcgtcgctcccggaagcccagccgagcctcgccaaccccgtcgagggctcc600ggcctgtcgctcgaccgccggcccaagaagtcgtcgaccacctgcgccgtctgccgcttc660cgaaaggtgcgctgcaacggccagcgcccgtcctgcggcaattgccagcgcctcggcttc720ccctgttcctacgacgatgccgacgtcgacacatggtccatgtcattgccgcgccgccgt780gtcaagcaggcctgcttgagctgccacagccgcaaggcgcgatgctccggccatctgccc840tcgtgcgagcgttgtcgagcccagggcatcgagtgcgtctaccggcccaacaagcgagcc900aagccgtcgtcggcgggagccggcattgcgggctcaaagagccccaacagcccggatcaa960gagtcggatcgcgacggcggcagggaggcacggggccatggagagagccgcgaccacgat1020gaagggcgcaatgatagcccggcattgaccgaccgtgccagctccgccagtccaggggca1080gacaaccaagggtaagtgactcggacaacaactgttttcgtgataggtacactgacggcc1140ttggaactggcagtccgcagatagatgaaagcttcagctccatcgtcagccgcgcattcg1200acctcttcttccgtcacgtccaccacatgcccatgttcaccttcctgcaccgcgcttctc1260taatggagcaatatcacgccggcaaagtcgacagggcgcttcttctggcactggtcggca1320tcacgtcgtgcttgaccaacatgggcccgggcatgcgcaaatatggaaaccgctgcatca1380acgatgccgaggccctcctccttgccgactacagccggccctcgattgtcaagatccagg1440ccctcgtcttcatcatcaaacaccgcatcctctgcaataaattctccagcgcctttgtcc1500tgcacagcttcgcctctcggtacgcctctgctttgcgcctcaactacgaggcgccccatc1560tccgcttcctggctcaagagtcccgtcggcgccttatgtgggccctgtactgcatcgaca1620cgagcatctgtggcgggtatccggactttgtcctctggagagccgaccagatccacgtct1680atttgccgtgcaacgagcgcaacttcgagtttgacctgccgcagcagacggagaagcttg1740tgccagactcacaccaaccccggccgccgctggctgaggatatcggaactctggcccttc1800atgcgcgcatcctgcacattcgccagaagatcatcgagttcacaaaggtagctcagtatg1860accgcggcatggaagccgccgagctgcagggccgtatcttcgcgctcgacaaggagctca1920atgactttgccaccaacctgccgacctcgttccagttctccgaaaactcgctccgtctcc1980gtgcctactcgccgagactatgcatctttgtcatgatccacgtctggtggcgccagtgct2040actgcgacctctatcgccttgccttggtcagcatcagtggaggtctgccgcagtccatgc2100tcgacatgttcgaccagagcttcctcgagcactgtcagaggcagtgtgtcgaccactctc2160tggcgctgactctcatcttttcactcatccagaagctgggtgcaaagccagtggcagaca2220ttgatctggccatgtgcgcgtatcagtgcgcgcggatgctcatgtaccttttccagttcg2280gcatcttcgatcggttcggcgtcacggcagaaacggtcatggagcaagctcagctatgtc2340ttcagaccatcaaggactgttgcgtggggcccgcggtggactgtatcgtggccgatttgg2400agaggctcatcaaccaggaccccaaagccgcaatacgagaagggctaccgggggggccgc2460cgaatctccaaatgacagacgggccagatgttctcagcgctcccgctgcgaccggcctgc2520agccttttagctccccttcaggcgctgtcaacaacaccgtctcgccttttactcatcccg2580tcatgactgctccctggatgtcggaaacgggctttgcctcaggactggaccagcaaagtc2640tcccgccagaggtccatgccaatcctagcaagggcggaactcccagtctgtctgcaccgc2700ggtcggagtttgggcagtctgatgtgaacgcatacgacagcacctttgcagggctcgggc2760tggacgatggatttgattatgccatgggactagacatgaacatgtgggcgaccaacgggg2820gcagctggactgcgcctggctatgggaatacctggatgtga2861<210>32<211>884<212>prt<213>里氏木霉<400>32metgluglyalaaspargglylysglythrtrpmettyrproproala151015thralathrprotyrleuproglnprovalglnleuserproleuthr202530leuargproserleuleuproserleuaspleuargleuserproser354045hisproglnphepheargserglnleuglualaserthrserthrser505560serleuseralaproalatyrleuleualaalahiscysaspserser65707580gluthrglnserproproalaleuserpheserproserthralaser859095serilealathrproleuproglyservalalaalametaspleuasp100105110proproproaspalaaspmetproserpheaspleualaglyserser115120125leuproglualaglnproserleualaasnprovalgluglysergly130135140leuserleuaspargargprolyslysserserthrthrcysalaval145150155160cysargphearglysvalargcysasnglyglnargprosercysgly165170175asncysglnargleuglypheprocyssertyraspaspalaaspval180185190aspthrtrpsermetserleuproargargargvallysglnalacys195200205leusercyshisserarglysalaargcysserglyhisleuproser210215220cysgluargcysargalaglnglyileglucysvaltyrargproasn225230235240lysargalalysproserseralaglyalaglyilealaglyserlys245250255serproasnserproaspglngluseraspargaspglyglyargglu260265270alaargglyhisglygluserargasphisaspgluglyargasnasp275280285serproalaleuthraspargalaserseralaserproglyalaasp290295300asnglnglytyrthraspglyleuglythrglyserproglnileasp305310315320gluserpheserserilevalserargalapheaspleuphephearg325330335hisvalhishismetprometphethrpheleuhisargalaserleu340345350metgluglntyrhisalaglylysvalaspargalaleuleuleuala355360365leuvalglyilethrsercysleuthrasnmetglyproglymetarg370375380lystyrglyasnargcysileasnaspalaglualaleuleuleuala385390395400asptyrserargproserilevallysileglnalaleuvalpheile405410415ilelyshisargileleucysasnlyspheserseralaphevalleu420425430hisserphealaserargtyralaseralaleuargleuasntyrglu435440445alaprohisleuargpheleualaglngluserargargargleumet450455460trpalaleutyrcysileaspthrserilecysglyglytyrproasp465470475480phevalleutrpargalaaspglnilehisvaltyrleuprocysasn485490495gluargasnpheglupheaspleuproglnglnthrglulysleuval500505510proaspserhisglnproargproproleualagluaspileglythr515520525leualaleuhisalaargileleuhisileargglnlysileileglu530535540phethrlysvalalaglntyraspargglymetglualaalagluleu545550555560glnglyargilephealaleuasplysgluleuasnaspphealathr565570575asnleuprothrserpheglnphesergluasnserleuargleuarg580585590alatyrserproargleucysilephevalmetilehisvaltrptrp595600605argglncystyrcysaspleutyrargleualaleuvalserileser610615620glyglyleuproglnsermetleuaspmetpheaspglnserpheleu625630635640gluhiscysglnargglncysvalasphisserleualaleuthrleu645650655ilepheserleuileglnlysleuglyalalysprovalalaaspile660665670aspleualametcysalatyrglncysalaargmetleumettyrleu675680685pheglnpheglyilepheaspargpheglyvalthralagluthrval690695700metgluglnalaglnleucysleuglnthrilelysaspcyscysval705710715720glyproalavalaspcysilevalalaaspleugluargleuileasn725730735glnaspprolysalaalailearggluglyleuproglyglypropro740745750asnleuglnmetthraspglyproaspvalleuseralaproalaala755760765thrglyleuglnpropheserserproserglyalavalasnasnthr770775780valserprophethrhisprovalmetthralaprotrpmetserglu785790795800thrglyphealaserglyleuaspglnglnserleuproprogluval805810815hisalaasnproserlysglyglythrproserleuseralaproarg820825830serglupheglyglnseraspvalasnalatyraspserthrpheala835840845glyleuglyleuaspaspglypheasptyralametglyleuaspmet850855860asnmettrpalathrasnglyglysertrpthralaproglytyrgly865870875880asnthrtrpmet<210>33<211>2248<212>dna<213>里氏木霉<400>33atgaagacatcatcgagtaccccacgcatccgcaagaagaggattccggtaagcatatgc60cgtctgagcagcccaggggccgctgtcatgtcacgttgacctcggtagctcggcatgctc120cgttgctgatcgtgttggtggacagaaatcctgcaccgcatgtcgccgctccaaggtgag180aggctgaactcgaagcctatagttcgtgagctcaaagttctgactgacagctaatcccag240gctaaatgcgatggtaagagaccatgctcacggtaggtcttctatgtgggctgaatcggc300tgtgcctcgatcccagattaacggttacatccagatgcagcgacctgaagaagatctgca360cttttattgatcctcccaaacaggctcatgagatgtatgcttcttgcgttaccgattcca420atttgtcgatcgcacacgcaactgaccacgctcgcctcactcatgcagccggatagagga480gcttgagcaacaggttgcggcgctcaaagataggcttcgagcggatgcattggcacaaag540cactcaagcagagaccggcttccagcacaccagccagccaggcattggcgacaccccaat600ctcatactcacctcgggccacttttgtcagccccgatagagtcgcctcagcattctgcat660acgttcatcacacacccaatcaccgaccaatacgacacttgccagaaaacacaccagatc720tcactttgagattgggtctgtgacattaccagattgcctggatgctggcctgctaagtct780cgagcaagctcgtcactacttctccatcttcttccaaggatgtgaccactttgttccggt840gctcgatcctcgctatgactcattcgaaagcattcgcgcaagaagcagtctcctgtttgg900ggcaatatgtgcagttggctgccgcgttgtgacggggtctgaaacccagcaatggcatat960gctcgatttccatgtcaagaggatgctgacttgcgccctggccagtccgtccatggcctc1020actcgagactattcaggcactgttggtccgctcgtgctatgcatcagagaggtctctcct1080tgtcgcagctgcaacgcgcatggctatcgatttggactttgcaagttcctacgatgagat1140ggtcaatcggtctgtggccccggcagcacggggagtatcctcggcaagtctggatcaaga1200tacaatcaccttgatgcgaagagtgaggacatggctccatctgtttgtgttggggcaaat1260attgcatgtcgatgctgctgacctggcgaccttcaagtttgttggggatttacggagatg1320ccgcattatactgaagagcccggccgcaactgagctcgatctgtccttgttctcccaagt1380cgagctcaatgctgtccgtggcaggatctatgactcgttgtctggcctcgtgggattcag1440cgatgaagacgcaatgatggacgtcgttcgcgaggcaaagatcgacattgctctatggta1500tgatgattgggagcacatcttcgagaagcaaagcgctcaagcgccatggcttggtgtcaa1560cttgcgcgcacaaaaatgttggtcagagaacatggccttttgccgagtcctgcgggcgtc1620tggtgtcgagaatgtggacttcatgtcgcccgctcaaaagtcagttgtcgccatggcaaa1680agatgccttggaggagcacctggatatcatgatcagagagccgaggctctatttacgcaa1740tctgcgcttcgcaatggactttgtctgggcaaagtgcgcattttgctacttactgctgct1800caaactttccattctgttgccggaaagtaaggggcgcagcagtcgagagcttgtagaaca1860cggcaatatcctcctggccgagctaagtgaagccagcggagggaatcatagcggaagccg1920gagcagcactggaaaacagtatcttcagcttctacaggtggggatcgggaagttcagctg1980tgccacccaggaagcccacgaagtacttgcgagcacggcaaatgacgacagttttgcaac2040tgctcgacggacgcctacattggcaacacagaatcgaatggagctggactcgtttgttcc2100ggaacaattcgtctttgagtgggatttccctgggctgacgttgttttcctcatcggcgac2160cagggtttcgtggcttgatgatatccttgcagaggcactcaacgggagtgaggatgcatt2220tgtttggttaccaacagacaatgacaac2248<210>34<211>650<212>prt<213>里氏木霉<400>34metlysthrserserserthrproargilearglyslysargilepro151015lyssercysthralacysargargserlysalalyscysaspglylys202530argprocysserargcysseraspleulyslysilecysthrpheile354045aspproprolysglnalahisgluileargileglugluleuglugln505560glnvalalaalaleulysaspargleuargalaaspalaleualagln65707580serthrglnalagluthrglypheglnhisthrserglnproglyile859095glyaspthrproilesertyrserproargalathrphevalserpro100105110aspargvalalaseralaphecysileargserserhisthrglnser115120125prothrasnthrthrleualaarglyshisthrargserhispheglu130135140ileglyservalthrleuproaspcysleuaspalaglyleuleuser145150155160leugluglnalaarghistyrpheserilephepheglnglycysasp165170175hisphevalprovalleuaspproargtyraspserphegluserile180185190argalaargserserleuleupheglyalailecysalavalglycys195200205argvalvalthrglysergluthrglnglntrphismetleuaspphe210215220hisvallysargmetleuthrcysalaleualaserprosermetala225230235240serleugluthrileglnalaleuleuvalargsercystyralaser245250255gluargserleuleuvalalaalaalathrargmetalaileaspleu260265270aspphealasersertyraspglumetvalasnargservalalapro275280285alaalaargglyvalserseralaserleuaspglnaspthrilethr290295300leumetargargvalargthrtrpleuhisleuphevalleuglygln305310315320ileleuhisvalaspalaalaaspleualathrphelysphevalgly325330335aspleuargargcysargileileleulysserproalaalathrglu340345350leuaspleuserleupheserglnvalgluleuasnalavalarggly355360365argiletyraspserleuserglyleuvalglypheseraspgluasp370375380alametmetaspvalvalargglualalysileaspilealaleutrp385390395400tyraspasptrpgluhisilepheglulysglnseralaglnalapro405410415trpleuglyvalasnleuargalaglnlyscystrpsergluasnmet420425430alaphecysargvalleuargalaserglyvalgluasnvalaspphe435440445metserproalaglnlysservalvalalametalalysaspalaleu450455460glugluhisleuaspilemetilearggluproargleutyrleuarg465470475480asnleuargphealametaspphevaltrpalalyscysalaphecys485490495tyrleuleuleuleulysleuserileleuleuprogluserlysgly500505510argserserarggluleuvalgluhisglyasnileleuleualaglu515520525leuserglualaserglyglyasnhisserglyserargserserthr530535540glylysglntyrleuglnleuleuglnvalglyileglylyspheser545550555560cysalathrglnglualahisgluvalleualaserthralaasnasp565570575aspserphealathralaargargthrprothrleualathrglnasn580585590argmetgluleuaspserphevalprogluglnphevalpheglutrp595600605asppheproglyleuthrleupheserserseralathrargvalser610615620trpleuaspaspileleualaglualaleuasnglysergluaspala625630635640phevaltrpleuprothraspasnaspasn645650<210>35<211>820<212>dna<213>里氏木霉<400>35atgtttaacacattcaagatcgaccccgagacgaacagcgtccaggagctcaggcgggcc60tccgatcccatctcagcccggtcgagccagcaccaggcgtgcaacaactgccacgctaaa120aaggtataaagtcgcagcaagcgcgccgcttccagtcaagtgatcaactctgttgttgtt180cctgcgctaacagtcactctcccccgattagctcaagtgcagtggtgacaagagtggctg240tgagcgctgcatcgccagccagctgcgatgcgaatacactcgctctccctctcgcagagg300cggcaggaagaccagcggcagaagctccactgacagccgtggcggattggagccggccgg360tggtgacagcagccccggcagccagtctggcaagtcgaggcacagaagcagcaggcactt420gcatgccgggactagcagcagcagcagagcccgggcctcagcgtccagggaggaagaata480tggcgatgcgttggacttgttcgaccccacgacgctcggtcctgacgatggcttcgatct540acgatccttatccttggaggcttcggacagtggttacggcaacagtgcctattacaacca600gcagcagcagcagcagcagcaacaacagcatggtgcaggaagctggcaacacgtggcctc660cagcgacccgtacggcaacttgtctggcagctcatacatgggcacgaccagctccagtgg720ccaagactacgacgtggattactacggaaactatgacgagtacggacagtatcatggcca780gcaaaacgatcctagatactggggcgggcagcaacaatag820<210>36<211>243<212>prt<213>里氏木霉<400>36metpheasnthrphelysileaspprogluthrasnservalglnglu151015leuargargalaseraspproileseralaargserserglnhisgln202530alacysasnasncyshisalalyslysleulyscysserglyasplys354045serglycysgluargcysilealaserglnleuargcysglutyrthr505560argserproserargargglyglyarglysthrserglyargserser65707580thraspserargglyglyleugluproalaglyglyaspserserpro859095glyserglnserglylysserarghisargserserarghisleuhis100105110alaglythrserserserserargalaargalaseralaserargglu115120125gluglutyrglyaspalaleuaspleupheaspprothrthrleugly130135140proaspaspglypheaspleuargserleuserleuglualaserasp145150155160serglytyrglyasnseralatyrtyrasnglnglnglnglnglngln165170175glnglnglnglnhisglyalaglysertrpglnhisvalalaserser180185190aspprotyrglyasnleuserglysersertyrmetglythrthrser195200205serserglyglnasptyraspvalasptyrtyrglyasntyraspglu210215220tyrglyglntyrhisglyglnglnasnaspproargtyrtrpglygly225230235240glnglngln<210>37<211>2317<212>dna<213>里氏木霉<400>37atggctgctcccggttccaggccggcgatcatggcctccgtcgaaggctcatatcagacc60ccagccgcaactccaaccggatcgcccgatcccgacggcatggagccgttgctgcccccg120acttcgatggctccggcgattggcggcgatggtggcggcgaggcggccaggccggccaag180cgcggacgacgaagcaaccccaaggtcaagacgggatgcttgaactgcaagtgaggacgc240tttggaataccctgattatcatgcccttccgcttccccatgaccactcactctcccatgc300atgtcctcttcctcctcctctcccttcctcttctctcgtattgacggaaacacataaagc360caccccgggcagcccactgacgagctccatccctaggcaacggcgcatcaagtgcgatga420aaagcgtccgtcttgttcgcaatgcatccgcagcaagaaggagtgcagtggctatccggc480gcccactcgcgggccccggactgccgtcgatgtccgcattgctccaaagcccttggctcc540ctcctcgacggggctcccgcaactgcagcctactcccagcagcatagcgagcgcctccac600ttcaaccactgccagcctcttgctcactggccatacgattatgctgcctcctcgacgggc660ctatcgccgcaaacgccagaccaagcctgccgcctccaatgccgcgatgccctttatgta720tgagccgtctcacaacttggccctcatgcataccgagagtttgtatttcgacctgttccg780cgtccagacggcttccgagctgtccgggtactttgactcgaccttttggacatcacgggt840tctgcaggaatgccacttcgaaccggccattcgacatgccgtcgtcgctcttggtgccct900gtacaagactctggaacagtcctgcgaacccgacctgccgccgctcgctggtgccatgag960ccgcctggactcggtcatgtgtcactggcaggttgccatcagaaagtactcggaagcctg1020caaggccatgctgcatctcagcggcgacaagctggcaaccaacaagacccggctgatggc1080cagcgtcctattagcctgcttcgactccttcattggcgatcaccgccaagccattgtcca1140gatacagaccggattgaggcttctggctcggattcagtatgatcggacacaggcccctta1200ttccaatgaacgggttgaggaggatctgctgattatcttcacacgactagccattcaagc1260caagtcgtacgacatggccttccacttcccccatccctacgtgatccatctaggccctca1320aagcctacatgatccgtcatcccccctctcagactcgggttcacctcagccatccagccc1380catcccgtataagttctcatccttacgagaagctcgcctggcttcggatcagctttgtga1440aatgctgctgcgattcatcgaacacttgcagcgcgccaagaaggaaccttcttacacttt1500acccccatcctggaggcagctcggagccacattccagagtcaaatcgactcatggtcaca1560ggccttcgagcccatcttccaatcaaggctcacgcagcccggcatgagcctccttgaaaa1620gtcgggcatcgcagcgctcaagatgttccaggtcaacaccaacgtcatcttcctcaccat1680cttctgcgacgccgaggtccagttcgacgctttcctctcccacttcaaggccattgtcag1740cctgggctgggaagtcgtcggcgacgacgagaagcgagcggccaccgagcgatgccccga1800tccgcggcggtgcaagcagcagcacggacacgcaaggaccggcggggaagaaggaggacc1860gagggcgggaagagacgcattcccaacccacaacatcaagcccagcttctccgcagacct1920gggcatcgtgccgccgctcttcgtcgtggccacaaagtgccgcgagccgaccgtccggcg1980ggaggcgatccagctgctgaggagcagtgcccggcgcgagggcatgtgggacagcgagct2040ggcggccaacattgcccaatgggtcatggagattgaggagtccgagaacccctttccgga2100gatgcagcagcagcagcagccttcgcacggtcatggtggcgcagccgcacaggccgtcgc2160gctgcccagccgggccattccggaggagaagagggtcatggtcaagtcggtcgactttga2220tttgcgagcgaggtttgccgacgtgaccgtgggctcgagggactttcgccagggggtgca2280ggatcgacgacatagggcgacgcgaatcagttggtga2317<210>38<211>716<212>prt<213>里氏木霉<400>38metalaalaproglyserargproalailemetalaservalglugly151015sertyrglnthrproalaalathrprothrglyserproaspproasp202530glymetgluproleuleuproprothrsermetalaproalailegly354045glyaspglyglyglyglualaalaargproalalysargglyargarg505560serasnprolysvallysthrglycysleuasncyslysglnargarg65707580ilelyscysaspglulysargprosercysserglncysileargser859095lyslysglucysserglytyrproalaprothrargglyproargthr100105110alavalaspvalargilealaprolysproleualaproserserthr115120125glyleuproglnleuglnprothrproserserilealaseralaser130135140thrserthrthralaserleuleuleuthrglyhisthrilemetleu145150155160proproargargalatyrargarglysargglnthrlysproalaala165170175serasnalaalametprophemettyrgluproserhisasnleuala180185190leumethisthrgluserleutyrpheaspleupheargvalglnthr195200205alasergluleuserglytyrpheaspserthrphetrpthrserarg210215220valleuglnglucyshisphegluproalailearghisalavalval225230235240alaleuglyalaleutyrlysthrleugluglnsercysgluproasp245250255leuproproleualaglyalametserargleuaspservalmetcys260265270histrpglnvalalailearglystyrserglualacyslysalamet275280285leuhisleuserglyasplysleualathrasnlysthrargleumet290295300alaservalleuleualacyspheaspserpheileglyasphisarg305310315320glnalailevalglnileglnthrglyleuargleuleualaargile325330335glntyraspargthrglnalaprotyrserasngluargvalgluglu340345350aspleuleuileilephethrargleualaileglnalalyssertyr355360365aspmetalaphehispheprohisprotyrvalilehisleuglypro370375380glnserleuhisaspproserserproleuseraspserglyserpro385390395400glnproserserproileprotyrlyspheserserleuarggluala405410415argleualaseraspglnleucysglumetleuleuargpheileglu420425430hisleuglnargalalyslysgluprosertyrthrleuproproser435440445trpargglnleuglyalathrpheglnserglnileaspsertrpser450455460glnalaphegluproilepheglnserargleuthrglnproglymet465470475480serleuleuglulysserglyilealaalaleulysmetpheglnval485490495asnthrasnvalilepheleuthrilephecysaspalagluvalgln500505510pheaspalapheleuserhisphelysalailevalserleuglytrp515520525gluvalvalglyaspaspglulysargalaalathrgluargcyspro530535540aspproargargcyslysglnglnhisglyhisalaargthrglygly545550555560glugluglyglyproargalaglyargaspalapheprothrhisasn565570575ilelysproserpheseralaaspleuglyilevalproproleuphe580585590valvalalathrlyscysarggluprothrvalargargglualaile595600605glnleuleuargserseralaargarggluglymettrpaspserglu610615620leualaalaasnilealaglntrpvalmetgluileglugluserglu625630635640asnpropheproglumetglnglnglnglnglnproserhisglyhis645650655glyglyalaalaalaglnalavalalaleuproserargalailepro660665670gluglulysargvalmetvallysservalasppheaspleuargala675680685argphealaaspvalthrvalglyserargasppheargglnglyval690695700glnaspargarghisargalathrargilesertrp705710715<210>39<211>1887<212>dna<213>里氏木霉<400>39atgagcgccgtatctaccgagaagtcacatcctaagcaagcagctgagacaacaagaccg60acttctctcaatcgtacctgtgaaggctgtcgtcagaggaagatcagatgcatcattgaa120agctctcaatcaatcagtcccccgaaatgcgcccgatgttccaagttcaacctggattgt180atctttctgcccccggctattcggaggaggcgcaataagaacgagactaggatcaaggaa240ctggagcaaaagctgcagcaactccaagatgccattgcccattctccagctgaagccgtt300gctggcaacgcgcattcagaaggatcccatcctgacagtgtctttgagtcgttccatccg360gactttcagcagccgtccacttcattcgcctattcactgccgccagaaacatttcttcca420actgcactctcttactctaccccagaagactcgatatcaactggtttggtaagccccgag480cttgcagacgatctattcacgacatttttccagactctggcgcctatttatcccctcgtg540caagttccatcggactggacatggcagcagaccagatacgccaaacctgccctcttcagg600gccattctcacagctgcttcgagtgaccgtgatccggcattcttcatgatcatgttccgc660aacacgggcatgtatgtgacggaagaggtttccataaagggcaacaagtcgcttgacctg720attcaagcacttctcgtgctatccgcgtggtactgtcccatggaggactttcgaaagctg780aagtttagccattacgcaaacttggctgggagcatggccttggacttgaggtcctcaaac840gacgagcagtattggattccaccagtcaaggactcgtttgccagctcagagcagttggta900gaaacatgcagaacgttcttggcaagttactttctttgctctagtatggcattttcttat960cgacggccgagtatcctccgctatggaccttgggttgatgattgtatacgagtacttgaa1020gccgcaccaactgcatgcctaaacgaccgccgactgatagagtggacaaagctgcagatc1080attgcggaagaatgcatgtcggcggctgggctcgacaatgagtccaacgtctgtttagcg1140gacgacagagttggtcgcgtattgaagcatggcattgaaagggcgatatcgtggaagcac1200caggtttcccctgagatcatacataaaccgatggtgatgcactaccatatggtgttgata1260agccttaatgagccggccttttacgatgcacatgatatgcaggatttccgaccgccatat1320cgactgcgtcctctgcctctggccaaaagctccgatgatcctggctcaattgccatcgcc1380aactcgcttgctcagtgcgtcagctccgcacaaacggtcatcaacacgtttctgggcatc1440ccaacggaaaccctcagagtcatgcccgtcatcgtatacacccgcatcacatacgcagca1500gtcacgctgatcaagtttgatgtctccgcccgaatgctgcagagcgttgcgtgtatgttg1560gatgacatccacctaagcccaaaggtgcttttgttacagcttcttgacaaactaatcgaa1620gtggcggggagcgagaacgttgttgtgccggttgtctttcgaggggctttggcacggatg1680atgaggtggtatgttgatcaacttgagagctttcaggagccggatcaagatgaggtcctc1740gaaccgatgatgtacgttggcgttgatgcagaaagcagtagcgggttggatgtcggttat1800ggtggtgctgaatcgcaaggatctgttgcggatccaaatgccagcttcttggatcctttg1860agaacccatcatgtagattatctatag1887<210>40<211>605<212>prt<213>里氏木霉<400>40metseralavalserthrglulysserhisprolysglnalaalaglu151015thrthrargprothrserleuasnargthrcysgluglycysarggln202530arglysileargcysileilegluserserglnserileserpropro354045lyscysalaargcysserlyspheasnleuaspcysilepheleupro505560proalaileargargargargasnlysasngluthrargilelysglu65707580leugluglnlysleuglnglnleuglnaspalailealahisserpro859095alaglualavalalaglyasnalahissergluglyserhisproasp100105110servalphegluserphehisproasppheglnglnproserthrser115120125phealatyrserleuproprogluthrpheleuprothralaleuser130135140tyrserthrprogluaspserileserthrglyleuvalserproglu145150155160leualaaspaspleuphethrthrphepheglnthrleualaproile165170175tyrproleuvalglnvalproserasptrpthrtrpglnglnthrarg180185190tyralalysproalaleupheargalaileleuthralaalaserser195200205aspargaspproalaphephemetilemetpheargasnthrglymet210215220tyrvalthrglugluvalserilelysglyasnlysserleuaspleu225230235240ileglnalaleuleuvalleuseralatrptyrcysprometgluasp245250255phearglysleulyspheserhistyralaasnleualaglysermet260265270alaleuaspleuargserserasnaspgluglntyrtrpilepropro275280285vallysaspserphealasersergluglnleuvalgluthrcysarg290295300thrpheleualasertyrpheleucyssersermetalaphesertyr305310315320argargproserileleuargtyrglyprotrpvalaspaspcysile325330335argvalleuglualaalaprothralacysleuasnaspargargleu340345350ileglutrpthrlysleuglnileilealagluglucysmetserala355360365alaglyleuaspasngluserasnvalcysleualaaspaspargval370375380glyargvalleulyshisglyilegluargalailesertrplyshis385390395400glnvalserprogluileilehislysprometvalmethistyrhis405410415metvalleuileserleuasngluproalaphetyraspalahisasp420425430metglnasppheargproprotyrargleuargproleuproleuala435440445lysserseraspaspproglyserilealailealaasnserleuala450455460glncysvalserseralaglnthrvalileasnthrpheleuglyile465470475480prothrgluthrleuargvalmetprovalilevaltyrthrargile485490495thrtyralaalavalthrleuilelyspheaspvalseralaargmet500505510leuglnservalalaleualaglysergluasnvalvalvalproval515520525valpheargglyalaleualaargmetmetargtrptyrvalaspgln530535540leugluserpheglngluproaspglnaspgluvalleuglupromet545550555560mettyrvalglyvalaspalagluserserserglyleuaspvalgly565570575tyrglyglyalagluserglnglyservalalaaspproasnalaser580585590pheleuaspproleuargthrhishisvalasptyrleu595600605<210>41<211>2066<212>dna<213>里氏木霉<400>41atgccccatatgcgcgaggtcaagacgtgtgaccgctgtcgccatttcaagcggcgctgc60gacctcctcaagccgtcgtgctcgcgatgcatccaagcgggagtgcgctgtagcttcgat120gtcaacggggtcgccgtgcccggagccgctgccaatggcggtgcgtcttcctcgcccaac180gcatcgcgacaggcgagggccggcgccttgacatcttcgtctcctgccaccaatggcgac240cctgcgctggctacaccgggtcggaacgggctcatctcgcccacggcatccaccgagagc300cccgagcccgccgtggccctcgatgccaatggcatgccgatcgagggcgcctctgcgtcc360gcgtcggccacgaaccagctgcgcgttgtccgcaagcgcaagcgcaactgcctgagctgc420cttcgctgccatcgcctcaaggtcaagtgtgacaaggaactgccgtgtggccgctgcaag480tcgagcggcaacggcagggagtgctattatagctacaacaaggggcccaacggtggaaag540ttcccttgtcccaccgccccgtcgagcaccgaggagacgaagacggtgcttgctacctgg600caggtccaacacaaggtgcgaggctccagccattggcgggacctcatgaccaaggtatgc660agagagcactttgcatggaagacttcctcgagatgtgctgactggtgggatagattggca720cattgacaacgctagactccgcgcccctggctgccgctctcgaagacgtagccaccaatg780cttgcctggccaactttacgctgccgggcaacttcccctttggcacccctggtgcgacca840agtactatactcgcgatgccgtcactcgactgttggccagcgagagggcaaactgcgatc900ggtacctccagcgatacctggatctgctcgacgtggtcaaccccatcgtggacgtacaca960tcttcacgcgcgaagttgagcgatactggatagaccccaacgcctcggacctgtgctggc1020tggcgcagttcctcatggtcatgggcctcggcagctttgcctctcccgagggggagcctc1080ccgtcgccacggagctcatgatggccgccgaggcgtgtctgatgcagacgcccttttcct1140tccggccgactctgttggcgctcaagatgctggccctcatggttgtcgccaagcaggtct1200gcaaccccacctgctggtcggtcgactcgtgctggtccctgctcggaatgctcgtgcgca1260cggccttcatctacggactgccccaggacccgtcgagcccagaggacgcggggcgcaacc1320aggacgaaaaggacgcccgacggaagctgtggctcacgatcctgtacctcgacatcaagg1380tggccatgtgtaccggaatgccgcccctcacacggccggacgagctcggcaccctcgaga1440agattcccgaatggggtccgccggacagtctccagatggtgctgtaccagtcgctgccga1500ccgtgctggccgtcatggcgcagatcaattccaacaaggagcagatctcgtaccccgacg1560tgctgcgatacaatgcccagctgcgggagctcatgagccacgcccagcgggtctgtaccg1620gacagctgcagcgtgtcacggtcgacatcttcttgcggcggtgcctcatggtgctgcatc1680gtccctttgccctgcacccagagggccccgtcatgttccccgagtcgtactggtcgtccc1740tggagtgctgtcttgcgctgctggtgcactaccgcgagatgtggtgcagcgacccggatc1800tgcgcctggacctggtcggacgggcctttgtcctcgacttcttctccgccaccttgacga1860catatatccacgtcctccgcgctgacgcccctctcacgggcgctgctgccatgggatgcg1920ccattccgccgcgacagattatcctcgacacgttgcgaagctgcgtagagatttggagct1980cggaaaaggacaagagcgtgtgctaccgcaccgggtacaacctgcttgtcgcggtgctga2040acatggttcccaagaacaagatttga2066<210>42<211>658<212>prt<213>里氏木霉<400>42metprohismetarggluvallysthrcysaspargcysarghisphe151015lysargargcysaspleuleulysprosercysserargcysilegln202530alaglyvalargcysserpheaspvalasnglyvalalavalprogly354045alaalaalaasnglyglyalaserserserproasnalaserarggln505560alaargalaglyalaleuthrserserserproalathrasnglyasp65707580proalaleualathrproglyargasnglyleuileserprothrala859095serthrgluserprogluproalavalalaleuaspalaasnglymet100105110proilegluglyalaseralaseralaseralathrasnglnleuarg115120125valvalarglysarglysargasncysleusercysleuargcyshis130135140argleulysvallyscysasplysgluleuprocysglyargcyslys145150155160serserglyasnglyargglucystyrtyrsertyrasnlysglypro165170175asnglyglylyspheprocysprothralaproserserthrgluglu180185190thrlysthrvalleualathrtrpglnvalglnhislysvalarggly195200205serserhistrpargaspleumetthrlysproleualaalaalaleu210215220gluaspvalalathrasnalacysleualaasnphethrleuprogly225230235240asnphepropheglythrproglyalathrlystyrtyrthrargasp245250255alavalthrargleuleualasergluargalaasncysaspargtyr260265270leuglnargtyrleuaspleuleuaspvalvalasnproilevalasp275280285valhisilephethrarggluvalgluargtyrtrpileaspproasn290295300alaseraspleucystrpleualaglnpheleumetvalmetglyleu305310315320glyserphealaserprogluglygluproprovalalathrgluleu325330335metmetalaalaglualacysleumetglnthrpropheserphearg340345350prothrleuleualaleulysmetleualaleumetvalvalalalys355360365glnvalcysasnprothrcystrpservalaspsercystrpserleu370375380leuglymetleuvalargthralapheiletyrglyleuproglnasp385390395400proserserprogluaspalaglyargasnglnaspglulysaspala405410415argarglysleutrpleuthrileleutyrleuaspilelysvalala420425430metcysthrglymetproproleuthrargproaspgluleuglythr435440445leuglulysileproglutrpglyproproaspserleuglnmetval450455460leutyrglnserleuprothrvalleualavalmetalaglnileasn465470475480serasnlysgluglnilesertyrproaspvalleuargtyrasnala485490495glnleuarggluleumetserhisalaglnargvalcysthrglygln500505510leuglnargvalthrvalaspilepheleuargargcysleumetval515520525leuhisargprophealaleuhisprogluglyprovalmetphepro530535540glusertyrtrpserserleuglucyscysleualaleuleuvalhis545550555560tyrargglumettrpcysseraspproaspleuargleuaspleuval565570575glyargalaphevalleuaspphepheseralathrleuthrthrtyr580585590ilehisvalleuargalaaspalaproleuthrglyalaalaalamet595600605glycysalaileproproargglnileileleuaspthrleuargser610615620cysvalgluiletrpserserglulysasplysservalcystyrarg625630635640thrglytyrasnleuleuvalalavalleuasnmetvalprolysasn645650655lysile<210>43<211>2605<212>dna<213>里氏木霉<400>43atgaccctcgactacaatgtcaacggcgggggcgatgccttactcgatctgacctacgaa60caacaccagcgggaccgggctgctgcgtacagctactcgacatcaaccactgctcactcg120agtcctggccctgcgtctaacctcgtcaccggctcgtggaatctgcccatcgacatgaac180gccaacgccggctcgtctcagggagctcgaggcagctccccggaggaataccacccaaac240tcgcctctctcacacatgtccagccctcacagccaccactcaccggctcagcactcgccc300taccagcaacccacaaacccgttgaccgactgggctcttcaccaccaccagcaacaggct360gcttccgcggcgcagcagcaccaggttttgcccgcccacgacctgacgcagtacatccag420gactccctgttgaacttcaacaacaccttttctggataccatcatgtcgactatctaccg480gccacgacgcaggctgcgatgcagacgcatttgctcgagtcgcccttcagctcgatgcct540gctctggatgataccgcacacgcaatgcactggggaggcgcgggcatgggaaattggcag600gactttcaaaacaccctgcagctggacgggctgcagaccgttggcagcaactcgcccact660ggaacctacctcgaggtgctctcattaccgtcttcgtcctctggcgatggctgggccatg720gtggactgggggcatcccggccacgagctcttccagcctacgcccagtgccgccatcttc780aacccctcccagacactgcacttgaggaccaactcggattcctccgacggccgcaactcg840gtcgaaatcggtagctttgaggaagttgccccctttccctattcgcccttctcaccagac900tctgatggccaagccgagaaccacaacaacggccaccggaactgctactcggccgatggc960catcatcaccaccatgatcacagccatagccatggtcatacccatggccacagccatagt1020cacagtcacagtcacagccacgtccacagtcccatcgcggtgactgccccagtctccctc1080aagggcgagtcgtctgcccgtcacagccctggcagcggagcgggctcggtatccccctct1140tcgtccagcaccacctcaaggaggagtgccggaagtgctggcattcgcaagagcccgatt1200gccaaggcacccaaaaccgtcgtcaggcgcgtctccaatggaaagaaggatggatctgtc1260gagaagaaggtgggccgtagaaagggccctcttttgccagagcagcgaaagcaagccagc1320gagataagaaagctgcgtgcttgcctgcgatgcaaattccttaagaagacttgcgataag1380ggagagccctgcgctggatgccagccgtctcacgcccgcctctggcaggtcccctgcacg1440cggatcgacatcaaggacattggctacttcatgaaggactggaaggccgactatgagagg1500cacatggaccgtggagtgtccgtctacaatgtcaagggcttcgcgcagaaggagacgctc1560atgtggatcacccacggctacggctttgccctgcccgtcatggtccgcgaggtctttgtt1620gccgatgacagctgcttctccgtcgagtgggtcgagtcgtacgttgcctccaacgacacc1680atcgactttgagactcggactgagaagctcgatgtcggtgccgagggcattcgcatcgac1740gctctctccgagtacctcgacaagcacatcgagggtccgtttgaggacttcatcgacgac1800cacttcgagggcactcccttcatcaccgagatcctcaagactgcccaccgctactacgtc1860aaggagaagatgcccgtcatccgaaaggccctgaagctggtccttgcctacaacctgacc1920atgcacatcaccatggtcgagagccagggatccgagatgccccttgacggccagatcgac1980gacgaggactccaagtactacggccgcaccgttgcgcctgtcatgatcaacttccagatt2040aagtgtgctctggctgacatgtggcgcgagcttcagaaggacatcctggaggagctctct2100gccctctactcgggtgtctacagcggcgagaagatgaagaactggccgaccatcttcctt2160ctggcgtccatcctgctcgccgtgtgggaggagatgcagtttgactgccactaccgcgtg2220ccagaccccgtggcggtgaacaagttctgcaacgacatggaaacgacccctgtcggtgtt2280attgtgggcctcttccacgccatctctcagaagctcccggccttcaccgactgggacacg2340cgtgagcacggccacctgcttcacaacaatgttgccgtctgtgatgccatgactgaggtg2400cgacagcatgttctcaagcatggtaagtttgcaccttttcgtgtcccgtcggacatgacc2460cgaaaagttgacgaaagtgtatgctaacaaactatgttttacagaggcttacctgcgaac2520ccggagagaagccaagtttgaccgctacgattttgattcgctttcgaacaagttcttgtc2580gaagcttgttatccgggccaactaa2605<210>44<211>840<212>prt<213>里氏木霉<400>44metthrleuasptyrasnvalasnglyglyglyaspalaleuleuasp151015leuthrtyrgluglnhisglnargaspargalaalaalatyrsertyr202530serthrserthrthralahisserserproglyproalaserasnleu354045valthrglysertrpasnleuproileaspmetasnalaasnalagly505560serserglnglyalaargglyserserprogluglutyrhisproasn65707580serproleuserhismetserserprohisserhishisserproala859095glnhisserprotyrglnglnprothrasnproleuthrasptrpala100105110leuhishishisglnglnglnalaalaseralaalaglnglnhisgln115120125valleuproalahisaspleuthrglntyrileglnaspserleuleu130135140asnpheasnasnthrpheserglytyrhishisvalasptyrleupro145150155160alathrthrglnalaalametglnthrhisleuleugluserprophe165170175sersermetproalaleuaspaspthralahisalamethistrpgly180185190glyalaglymetglyasntrpglnasppheglnasnthrleuglnleu195200205aspglyleuglnthrvalglyserasnserprothrglythrtyrleu210215220gluvalleuserleuproserserserserglyaspglytrpalamet225230235240valasptrpglyhisproglyhisgluleupheglnprothrproser245250255alaalailepheasnproserglnthrleuhisleuargthrasnser260265270aspserseraspglyargasnservalgluileglyserphegluglu275280285valalapropheprotyrserpropheserproaspseraspglygln290295300alagluasnhisasnasnglyhisargasncystyrseralaaspgly305310315320hishishishishisasphisserhisserhisglyhisthrhisgly325330335hisserhisserhisserhisserhisserhisvalhisserproile340345350alavalthralaprovalserleulysglygluserseralaarghis355360365serproglyserglyalaglyservalserproserserserserthr370375380thrserargargseralaglyseralaglyilearglysserproile385390395400alalysalaprolysthrvalvalargargvalserasnglylyslys405410415aspglyservalglulyslysvalglyargarglysglyproleuleu420425430progluglnarglysglnalasergluilearglysleuargalacys435440445leuargcyslyspheleulyslysthrcysasplysglygluprocys450455460alaglycysglnproserhisalaargleutrpglnvalprocysthr465470475480argileaspilelysaspileglytyrphemetlysasptrplysala485490495asptyrgluarghismetaspargglyvalservaltyrasnvallys500505510glyphealaglnlysgluthrleumettrpilethrhisglytyrgly515520525phealaleuprovalmetvalarggluvalphevalalaaspaspser530535540cyspheservalglutrpvalglusertyrvalalaserasnaspthr545550555560ileaspphegluthrargthrglulysleuaspvalglyalaglugly565570575ileargileaspalaleuserglutyrleuasplyshisileglugly580585590prophegluasppheileaspasphisphegluglythrpropheile595600605thrgluileleulysthralahisargtyrtyrvallysglulysmet610615620provalilearglysalaleulysleuvalleualatyrasnleuthr625630635640methisilethrmetvalgluserglnglyserglumetproleuasp645650655glyglnileaspaspgluaspserlystyrtyrglyargthrvalala660665670provalmetileasnpheglnilelyscysalaleualaaspmettrp675680685arggluleuglnlysaspileleuglugluleuseralaleutyrser690695700glyvaltyrserglyglulysmetlysasntrpprothrilepheleu705710715720leualaserileleuleualavaltrpgluglumetglnpheaspcys725730735histyrargvalproaspprovalalavalasnlysphecysasnasp740745750metgluthrthrprovalglyvalilevalglyleuphehisalaile755760765serglnlysleuproalaphethrasptrpaspthrarggluhisgly770775780hisleuleuhisasnasnvalalavalcysaspalametthrgluval785790795800argglnhisvalleulyshisglualatyrleuargthrargargglu805810815alalyspheaspargtyrasppheaspserleuserasnlyspheleu820825830serlysleuvalileargalaasn835840<210>45<211>1698<212>dna<213>里氏木霉<400>45atggatcaaaccatctccaccaagagccctcctcctcctcatcctcctcctcctcagaag60agccgccgctccaagaagggctgctacacatgccgcatcaagaaagtcaaatgcgacgaa120gtccggccccgctgcgagcgctgcgtccgtctaagacgcctctgcgactacgagcccagg180atgaagcacaacgacctcctggccatctccaggggctacataatggccctcgagagctcc240aagtcactgccgacgtgggcgcagcgcgcagcgttgacgagggctctggctcttcctact300cggcttcctgagtttccgagcagtgccagctcgctgaatctcacctcggccgatcatgag360gcgattcggtattttcggacgacgtttgcgcggcttcatcataccaagaatccggattac420tcgctcttttcaatcatcttcaccttggcacaggatgagccaatggtcatgcacgcgttg480ctggctcttggaggccacgagattgaatttcggcgcagtagcacttcagaaggatcggat540ggctcaactcagctggtaggacgagatggtaataaatggacaccagttcaacactattcg600gccgctctcggcctcttggccgatgccatcggaaatgcagacgatggtcggcaacttgag660ctggatcccatctgcgcagtcttgtatctgatgctggtctacgaacaaaagtacggagac720ggaacatgctctggtctgtcaaaccaccttgccggagcggccttgatcgtaaggcatcgc780tgccagcgcctatcagaacagatagccaagcgtggatggcaaggcacgccagtgctggcg840aggatacagccgcaaagccagtcagaagagcctggcttgtcgctcttcgtaatcaggctc900ctggtctggatcgctctctgcgatgctacggccgccagctttgggctcggcggccagttc960aacagcgagctgaaagacatcatggggccagaagatgcttcgtccatttcggggtttgat1020gtgctgcatagatactcaaactccttataccgctcaatgtggggagatgcgtatcctcaa1080gtcgaactgctggatgacgttgagaatcgggacattttcgcctttctatgcgcatccatg1140cagcttcgaagcatggtggctaatctggcgaagcttggcgcagacgagctcaagctccgt1200ttaccagctgttgaggcagcctttagacaggttgatttgcgatatggagagttgctcgag1260gtggcgtccgacctgtctctatcgaccgataactcacatcgtctggtggccaatatccgt1320gctttcatcccgatatatcacgctgcaaagctcgaactgacgcgcgtgctgcggagaaat1380gggacaactgcaaagatggaaattgtaccagaggctcatatagcagccattgtcgacttg1440gccattcaggctgataagcaccagggagtcgaaggcaccattagagtcgcatggccgctt1500ttcattgtggccctggagaccaacagcgtggttcatcaacgctggatcctcagccggttc1560caagcgatgagcagctatagcagaaatctggagcgtgcgcaccgctttctcaaagaacac1620ctcgggatgccgcttgagaagggtgaagattggttgcagagattcagcctagcaaacggg1680gagatattcgtgatatag1698<210>46<211>565<212>prt<213>里氏木霉<400>46metaspglnthrileserthrlysserproproproprohispropro151015proproglnlysserargargserlyslysglycystyrthrcysarg202530ilelyslysvallyscysaspgluvalargproargcysgluargcys354045valargleuargargleucysasptyrgluproargmetlyshisasn505560aspleuleualaileserargglytyrilemetalaleugluserser65707580lysserleuprothrtrpalaglnargalaalaleuthrargalaleu859095alaleuprothrargleuproglupheproserseralaserserleu100105110asnleuthrseralaasphisglualaileargtyrpheargthrthr115120125phealaargleuhishisthrlysasnproasptyrserleupheser130135140ileilephethrleualaglnaspgluprometvalmethisalaleu145150155160leualaleuglyglyhisgluileglupheargargserserthrser165170175gluglyseraspglyserthrglnleuvalglyargaspglyasnlys180185190trpthrprovalglnhistyrseralaalaleuglyleuleualaasp195200205alaileglyasnalaaspaspglyargglnleugluleuaspproile210215220cysalavalleutyrleumetleuvaltyrgluglnlystyrglyasp225230235240glythrcysserglyleuserasnhisleualaglyalaalaleuile245250255valarghisargcysglnargleusergluglnilealalysarggly260265270trpglnglythrprovalleualaargileglnproglnserglnser275280285glugluproglyleuserleuphevalileargleuleuvaltrpile290295300alaleucysaspalathralaalaserpheglyleuglyglyglnphe305310315320asnsergluleulysaspilemetglyprogluaspalaserserile325330335serglypheaspvalleuhisargtyrserasnserleutyrargser340345350mettrpglyaspalatyrproglnvalgluleuleuaspaspvalglu355360365asnargaspilephealapheleucysalasermetglnleuargser370375380metvalalaasnleualalysleuglyalaaspgluleulysleuarg385390395400leuproalavalglualaalapheargglnvalaspleuargtyrgly405410415gluleuleugluvalalaseraspleuserleuserthraspasnser420425430hisargleuvalalaasnileargalapheileproiletyrhisala435440445alalysleugluleuthrargvalleuargargasnglythrthrala450455460lysmetgluilevalproglualahisilealaalailevalaspleu465470475480alaileglnalaasplyshisglnglyvalgluglythrileargval485490495alatrpproleupheilevalalaleugluthrasnservalvalhis500505510glnargtrpileleuserargpheglnalametsersertyrserarg515520525asnleugluargalahisargpheleulysgluhisleuglymetpro530535540leuglulysglygluasptrpleuglnargpheserleualaasngly545550555560gluilephevalile565<210>47<211>2487<212>dna<213>里氏木霉<400>47atggaacgtacgctcgccatcttgacgaacgaacaagactttacccggccagacgataac60agcatgtccccggacgagggatcctacgacggcatggtgctgccttcaccttctcccacc120gacaccgcgtggagacctggcgtttccgcggcgcctctccagacgggccagctctcgccc180gtcagctccaacacagacgccaacatggacaacgggccgtcgaccatgcccatgatgcac240cactgctcctcctcctcctcctcgtcatgcgtggactcgtggaatgcgggctacggcaac300ccagacgagggcgaggaggacaaccagtgggagcagtgggagcagggctcggacgaggcc360ctcgccatcccgaagctcgagccgatcgacgacgacatcaacctggaggacgtgagcgcc420gcgcctctgtcacagacgccaccaaacgagctgctgggcggcgccccgatcaagcagaag480aggccacgaggacggccgaggaaacatcccctgacgcccattgtcaacgccaacaaggtg540accaagggccggtccaagaccgggtgcattacgtgcaggaagcgcaagaagaagtgcgat600gaggcaaagccgagatgtatgccagccattttctttatgtttatctccactggcctcctt660tatgctcttccctcgtgttgcgttgccacgcatgatgaacttgtgtgcattttcaaaagc720taatatgtgagattctgaacctcgataggtatgaactgtgagaagaatgccgtcgtctgc780gagggatatcacgagaagcaagtctggaagagtggcagggaacgggctgaagagggtatg840tcggacagaaaagacatgacaagactagaagtagatggacaagggacaagagaacggctt900gtggctaacccatcagcagagcgacagcggtgggaaaatctccccatcatcaccatgcag960cccatcttccacggcgtggagacggccgaggacaagatcttctggaagcactacgtcaac1020cacttcagcaacgtgctgaccgtcgagggagaggccaagaacgccttcaaggacatcatc1080ctgcagctcgccaaccggcaccagggcctgatgcactcgatcctcgccgccagcagcaag1140cacatcgactgggacacgccctacggcatcaagatcctgcagagcaacccgaccacgagc1200aaggaggcgctgcagcagcgctccgagtatcaccacgacgagggcatgaagcgcatgtac1260gccgagatgaaccaggagatggacaagagtaaccccgagtacaagacggtcctcgccgcg1320cggtacggccagatgatgtgcctcctgctgcagacgcgagcagagggcaatccacggggc1380gaccaccgggtccatctgcaggcgtacaagcacctggtgcagcactcgccgcccgaggac1440tccaacttcctgaccttcatcaccgagttcttccagtaccacatctacgccgacgacctc1500ttctggtaccccgagaagcggacggagcgcctcgcctccgaggactgggagccgtcggcg1560cccatccatcctccgcgtctgctgggcgtcgccgacggcctcttccgccacctctccgag1620atcaccagcatccgcaacgccatccgcgtcaacatggccggcgccgtcgaccccctggtg1680gactacacgagcctctacaaggccgccgagatcgacgccgccatccgggagtggacgcct1740cgctggccctcgggcgacagccgcgaccgcgtcggcctgctgtacaagcagatgctctgg1800gtctacctcttccgcacgatatacccgccctcggcgctccccggccgtcgcagcaccatt1860ggctcgctgcccacggccatgttctcctcgatgccgacgaatcctcacccccgacgggcc1920tcgatggccgcggccattggcactgccacggctggctccccgctcatgggggctgtcaat1980cccttttcccggtcgacgacgaaaacgacgcacagctgcccgtccacgcgccaaccctca2040cggaccaactccatgcatgagggcgacctgcacgcaaccaccacctctgcctctgcctct2100gcctccgcctccgagtcacacagccaacaacggcccccctcgtctccgccacccgcccgc2160cgacctgcccaggacgataagcgcatcacgctggccgtcgacgaatcgctgggcctcctc2220gagtccttcaagccgtccgatccctcgcagacgctcctcctgatcccgtgcctggtcatc2280ggcacggcgtgcttcgagcccgaccagcggtaccgcatccgcgccgccgtccgggccgtg2340cgagggtacaccggcctgaagaactgcgaccgagtcctggagctgctcgaagaggtgtgg2400gcactcatggagcagggcgactgggtggcggtatgggactggcagagggtcgcgagacga2460atgggcctcgactttccctgcacttga2487<210>48<211>756<212>prt<213>里氏木霉<400>48metgluargthrleualaileleuthrasngluglnaspphethrarg151015proaspaspasnsermetserproaspgluglysertyraspglymet202530valleuproserproserprothraspthralatrpargproglyval354045seralaalaproleuglnthrglyglnleuserprovalserserasn505560thraspalaasnmetaspasnglyproserthrmetprometmethis65707580hiscyssersersersersersersercysvalaspsertrpasnala859095glytyrglyasnproaspgluglyglugluaspasnglntrpglugln100105110trpgluglnglyseraspglualaleualaileprolysleuglupro115120125ileaspaspaspileasnleugluaspvalseralaalaproleuser130135140glnthrproproasngluleuleuglyglyalaproilelysglnlys145150155160argproargglyargproarglyshisproleuthrproilevalasn165170175alaasnlysvalthrlysglyargserlysthrglycysilethrcys180185190arglysarglyslyslyscysaspglualalysproargcysmetasn195200205cysglulysasnalavalvalcysgluglytyrhisglulysglnval210215220trplysserglyarggluargalagluglugluargglnargtrpglu225230235240asnleuproileilethrmetglnproilephehisglyvalgluthr245250255alagluasplysilephetrplyshistyrvalasnhispheserasn260265270valleuthrvalgluglyglualalysasnalaphelysaspileile275280285leuglnleualaasnarghisglnglyleumethisserileleuala290295300alaserserlyshisileasptrpaspthrprotyrglyilelysile305310315320leuglnserasnprothrthrserlysglualaleuglnglnargser325330335glutyrhishisaspgluglymetlysargmettyralaglumetasn340345350glnglumetasplysserasnproglutyrlysthrvalleualaala355360365argtyrglyglnmetmetcysleuleuleuglnthrargalaglugly370375380asnproargglyasphisargvalhisleuglnalatyrlyshisleu385390395400valglnhisserproprogluaspserasnpheleuthrpheilethr405410415gluphepheglntyrhisiletyralaaspaspleuphetrptyrpro420425430glulysargthrgluargleualasergluasptrpgluproserala435440445proilehisproproargleuleuglyvalalaaspglyleuphearg450455460hisleusergluilethrserileargasnalaileargvalasnmet465470475480alaglyalavalaspproleuvalasptyrthrserleutyrlysala485490495alagluileaspalaalaileargglutrpthrproargtrpproser500505510glyaspserargaspargvalglyleuleutyrlysglnmetleutrp515520525valtyrleupheargthriletyrproproseralaleuproglyarg530535540argserthrileglyserleuprothralametphesersermetpro545550555560thrasnprohisproargargalasermetalaalaalaileglythr565570575alathralaglyserproleumetglyalavalasnpropheserarg580585590serthrthrlysthrthrhissercysproserthrargglnproser595600605argthrasnsermethisgluglyaspleuhisalathrthrthrser610615620alaseralaseralaseralasergluserhisserglnglnargpro625630635640proserserproproproalaargargproalaglnaspasplysarg645650655ilethrleualavalaspgluserleuglyleuleugluserphelys660665670proseraspproserglnthrleuleuleuileprocysleuvalile675680685glythralacysphegluproaspglnargtyrargileargalaala690695700valargalavalargglytyrthrglyleulysasncysaspargval705710715720leugluleuleuglugluvaltrpalaleumetgluglnglyasptrp725730735valalavaltrpasptrpglnargvalalaargargmetglyleuasp740745750pheprocysthr755<210>49<211>2328<212>dna<213>里氏木霉<400>49atgtcagcagcagtaagtgatgcctcctgacagcctcgtgttacgcccctcaagctgacc60tcctacctacccaacagggggagcccctacggcctcgtgggcttctcatcccacgcctca120ttagggacccagattcctgcattacttgtcgccgcctgggaaggagatgcaagctgggca180agcccacttgtgttacctgccagaagttcaaccttgagtgcatccaaccagtcaataatc240tcttcgacgaaaacggcgccgagttcgccttggactcgggcgatggtaggaagaggcaaa300ggacgggctccatctctcaggagggggttgagagtgatgccgtgcctgccagcaagcctg360agcctgaggagtctgagcctgaacctgagtctgagtctgagtctgagtccgagctggagc420cggactcggagttggagttggagtctgatgcggagtctgacgcggagcctgacattggct480cgcagtttaacattgcgatacagatggagctttgcaatatgaccagtcacctcgagcgcc540agaatcaagaagacgatgatgattcagttgaccttgaccccaacgaggctgccattctcg600agaatctgacacgacagctcaatcgggcccagcagactcgtctccgacagatccccggct660acgttcagggccctctcggcgacgtgcctggtcctgacggcgaccaagatcacgactatg720cttgctattatgactaccgctacgacatcgactctgacgatgccagcagcacaggccgtg780acagaaagcgaagccacaccccttataccttggacgcgacttctcgcacctccaccgtgt840accaggtcaagtgcacttgggacgggcagggccctccccctctgtcctctcgtctcaacg900atttcattgcgtacatgaaggaatgtcacttgacgcatgcgccatccgccgctctacctt960tgtctctgctgccgcaagacgattacactgcggccgagatgctccagtactactcgaagc1020gcccagaggaccgccagacgaagcctgtccctgatgtgaaccccgtcaaccccaagctca1080ttgacctggcgtattccaactccctcgtcctgcagctcatcattgcccaaagggtcaatc1140accgccaggtgtcatccgccatgttgccgaccggcgaaagggctgaaggcttctacgctg1200ctgccattgccgagttcgggccaatgattgacagctacctggcggggaacgagcaggata1260tgcttccgctcactctggcaagcctggtcatttctctcaccgaggtcagtgtgtctccca1320acatcctagcgtgcccatggtcttagccccccaccccctccccctccccctttctttttc1380cctcatacgctcttcttgcaggaaccaaggactgatcggtttatacgtatcttctcacag1440agggcgcggcttgacaaacgtggtcaagctcacaaccatcccaccgcagccatgggtatc1500ttgaagacactcttgacccttccccacaagcagatgtgtaaacaggtcccgcctatcttg1560ctggagtattacatgcatgctgcttgctttgcatgtgttgcagccgatgtcaccaaggcg1620gagtcaattccatttatgagcgaggcactccgagacgccgtcgacgacctagtccaggcg1680aaatatataggcaagctctgcgggaactggctgtcgatcatggttgtcgtacagagcatc1740tttgagctgggcatgaagatgcggccgtttgccgatgacgcatcgaacgccccggcgggc1800ggtccatccaccggctacttacccaaccactttgttacctttgggcagattcaagagcgg1860ctcacgcgcttcgtacctgacaaggaccccgactgcgaaagccctgaggcggccattctc1920ttcaagaacgccgccatactatatctctggagtctcttggaatggccaaacgtctcgaag1980ccccccggatcgtataccaactttatgaagatcgcttacaagatcgcgctcctgcaactg2040agccgcatctcggaattctccagcatcaacaaggtcctgtgctggccattgctcatcgtt2100gggtgctttgccaaatcggcaaaggtcaagggcatcatcacgtcgcggctcctcagcatc2160gcgggccgcttcaaggtcggcaacgcgctcgagacccttttcctgctgcagcacgtctgg2220ggcctgccttttgagaggcgcaacccctggatggtccacaagtctatccgcgcgacgagg2280tgctgcggatgcgtctgtaccgactgcatgagtcgattgtttatctga2328<210>50<211>708<212>prt<213>里氏木霉<400>50metseralaalaglygluproleuargproargglyleuleuilepro151015argleuileargaspproaspsercysilethrcysargargleugly202530argargcyslysleuglylysprothrcysvalthrcysglnlysphe354045asnleuglucysileglnprovalasnasnleupheaspgluasngly505560alagluphealaleuaspserglyaspglyarglysargglnargthr65707580glyserileserglngluglyvalgluseraspalavalproalaser859095lysprogluprogluglusergluprogluproglusergluserglu100105110serglusergluleugluproaspsergluleugluleugluserasp115120125alagluseraspalagluproaspileglyserglnpheasnileala130135140ileglnmetgluleucysasnmetthrserhisleugluargglnasn145150155160glngluaspaspaspaspservalaspleuaspproasnglualaala165170175ileleugluasnleuthrargglnleuasnargalaglnglnthrarg180185190leuargglnileproglytyrvalglnglyproleuglyaspvalpro195200205glyproaspglyaspglnasphisasptyralacystyrtyrasptyr210215220argtyraspileaspseraspaspalaserserthrglyargasparg225230235240lysargserhisthrprotyrthrleuaspalathrserargthrser245250255thrvaltyrglnvallyscysthrtrpaspglyglnglypropropro260265270leuserserargleuasnasppheilealatyrmetlysglucyshis275280285leuthrhisalaproseralaalaleuproleuserleuleuprogln290295300aspasptyrthralaalaglumetleuglntyrtyrserlysargpro305310315320gluaspargglnthrlysprovalproaspvalasnprovalasnpro325330335lysleuileaspleualatyrserasnserleuvalleuglnleuile340345350ilealaglnargvalasnhisargglnvalserseralametleupro355360365thrglygluargalagluglyphetyralaalaalailealagluphe370375380glyprometileaspsertyrleualaglyasngluglnaspmetleu385390395400proleuthrleualaserleuvalileserleuthrgluargalaarg405410415leuasplysargglyglnalahisasnhisprothralaalametgly420425430ileleulysthrleuleuthrleuprohislysglnmetcyslysgln435440445valproproileleuleuglutyrtyrmethisalaalacyspheala450455460cysvalalaalaaspvalthrlysalagluserileprophemetser465470475480glualaleuargaspalavalaspaspleuvalglnalalystyrile485490495glylysleucysglyasntrpleuserilemetvalvalvalglnser500505510ilephegluleuglymetlysmetargprophealaaspaspalaser515520525asnalaproalaglyglyproserthrglytyrleuproasnhisphe530535540valthrpheglyglnileglngluargleuthrargphevalproasp545550555560lysaspproaspcysgluserproglualaalaileleuphelysasn565570575alaalaileleutyrleutrpserleuleuglutrpproasnvalser580585590lysproproglysertyrthrasnphemetlysilealatyrlysile595600605alaleuleuglnleuserargileserglupheserserileasnlys610615620valleucystrpproleuleuilevalglycysphealalysserala625630635640lysvallysglyileilethrserargleuleuserilealaglyarg645650655phelysvalglyasnalaleugluthrleupheleuleuglnhisval660665670trpglyleuprophegluargargasnprotrpmetvalhislysser675680685ileargalathrargcyscysglycysvalcysthraspcysmetser690695700argleupheile705<210>51<211>2436<212>dna<213>里氏木霉<400>51atgaagaagcgatacccaccgctcctcccgtctgtccctggacagccatcaggcccggca60gagccttcgtcgagctcgcagcagctcaagcgacgtcgcaccggcgtttcggtcgcctgc120aatgcttgtcgtcggaagaaaatccgggtaacttccacctgaaggttgcttgggagctca180gtccctgacgcatggagagctctcatcctaatgtttagacgttaatgctcaatgatagtg240cgatggcctgcggccaacgtgttccacatgtcgggagcttgccgtccggtgtacataccg300cgacgactacaagctgaccccggaggcgcagggtctccttgtcgaggtgatgcgtctcct360caacagcctgccggagcgagaggcgattcgcatgctgcggtatctgaagagcgagaccga420cgcggcagtgattctgtccacgctacgaggggggatatctgcgatccatcagccgtcgga480gctccgtattgccgtggcgacaatggacaattcctttcatgccctccagcttggatcaca540gaatccagttgcatacccatatctgcctccgcttcaaccgcaggctttgccgagggatga600ctttcggcggctcactacgatgggaagcaagccttcgtctccctattctccttctcgacg660gatgctctgggaccgcgatcccaccgagtcccaggcaccgctctgcgatgaacggctgca720tcagctggatatcagtcactggaccaatgctccgatcaacaacgagtcggccgcccgtgc780catctccctgtatctggagacggatcatccgctgctgggcttcttcgagcccaacctgtt840cgtatcggacctgatcaaccacaaacatgactactgctccccaatgctggtcaactcgct900gctgtattgggcctgcgtgaataatgcccgtgccgtctggtatcgaatccaccgcagctg960acttgattgaaagcaaatgtatagcgccgtcgacccaggcatcgatgctctcgcagccta1020cttctgcgccgaggccgagacgatatggaggacggaacgagagtctgactcgcttctcaa1080cctggcctccgccctcttcctaggcctgggctacctcggccaaggaagagaccatgctgt1140gctgtcgtacacttcgcaggctaccaagatggccaccaggctgggcctctttggcgtcga1200cgaacacagtcgtgcgaagcccagcatcgacaagctgtccaaagaggcagccagtgcgta1260tatgtacgccgcatggggatcattcaactggatcaggtgggtgcctcttttccccttgtt1320tcttgcgtagacatcgagaagggttcgtcgtgtgctcacggaactcgtccctttccaacg1380tgtgcagtctcatgtcgctcttctaccgccaaccgggaatattgggccctcggagccccc1440cgagcttgcccatacccggcatggaggaggacatcgaggccgcctcgtcagccacaagcc1500ctgggtctcctcgcagggaaggacccgagcccgagcctcagtcgcggtacatgggaggcg1560tatttccctacctgtgccagttctggagcatcatgtatgaggtcagtctagcgtatgacg1620acagccaatcgtctctggatagccaagggaccttgtccttcgcggagcataagttccgcc1680agctgctcgcttggagcaacactctcccgtcacgcctcctccgagccaaccagaacccgc1740attatgtccaagttcttcagtaagtttcctctgctccgtccctgtcccatctttgactcg1800caatggttcgtcttcccgctctgggccggtgttgcttaacacgtgggtctctctagcata1860tggttccatactgccgttctctgcctcttcaggccgtgcatccaggagtttggtgttgct1920cgacttcgaacgatggttaggagcatcagctccccggatattgtgtatgccgcttcggtc1980gcgcagctgaaagacctcgtcctcaatttcagactccatttcgcctcctctacgtacacc2040gtgctgtggcatacagccttgatctacattactaacgagctcctcaccggccccaaggac2100aatgactggttcttctactttttgatatgcgtgtacggctatgaacgtctgagccggtcc2160tggcgggtgaccacgtccatttccagagccctgttgtcaatggctctacgaaaggggggc2220ataaccagtaccacagcccggacaatcctgaaagatcttggaccggacgactttcgcaaa2280aagtacggcgagatccgggccacattcatggcagacttggacatggccgaggaggatccc2340agcaatgccactgttgagaggcaggctgaggactttgagcacaatgccatgctgcgagac2400tatacaaatatactagacgcggacgaggcggcatga2436<210>52<211>688<212>prt<213>里氏木霉<400>52metlyslysargtyrproproleuleuproservalproglyglnpro151015serglyproalagluproserserserserglnglnleulysargarg202530argthrglyvalservalalacysasnalacysargarglyslysile354045argcysaspglyleuargprothrcysserthrcysarggluleuala505560valargcysthrtyrargaspasptyrlysleuthrproglualagln65707580glyleuleuvalgluvalmetargleuleuasnserleuprogluarg859095glualaileargmetleuargtyrleulyssergluthraspalaala100105110valileleuserthrleuargglyglyileseralailehisglnpro115120125sergluleuargilealavalalathrmetaspasnserphehisala130135140leuglnleuglyserglnasnprovalalatyrprotyrleupropro145150155160leuglnproglnalaleuproargaspasppheargargleuthrthr165170175metglyserlysproserserprotyrserproserargargmetleu180185190trpaspargaspprothrgluserglnalaproleucysaspgluarg195200205leuhisglnleuaspileserhistrpthrasnalaproileasnasn210215220gluseralaalaargalaileserleutyrleugluthrasphispro225230235240leuleuglyphephegluproasnleuphevalseraspleuileasn245250255hislyshisasptyrcysserprometleuvalasnserleuleutyr260265270trpalacyslysglnmettyrseralavalaspproglyileaspala275280285leualaalatyrphecysalaglualagluthriletrpargthrglu290295300arggluseraspserleuleuasnleualaseralaleupheleugly305310315320leuglytyrleuglyglnglyargasphisalavalleusertyrthr325330335serglnalathrlysmetalathrargleuglyleupheglyvalasp340345350gluhisserargalalysproserileasplysleuserlysgluala355360365alaseralatyrmettyralaalatrpglyserpheasntrpileser370375380leumetserleuphetyrargglnproglyileleuglyproargser385390395400proproserleuproileproglymetglugluaspileglualaala405410415serseralathrserproglyserproargarggluglyproglupro420425430gluproglnserargtyrmetglyglyvalpheprotyrleucysgln435440445phetrpserilemettyrgluvalserleualatyraspaspsergln450455460serserleuaspserglnglythrleuserphealagluhislysphe465470475480argglnleuleualatrpserasnthrleuproserargleuleuarg485490495alaasnglnasnprohistyrvalglnvalleuglnprocysilegln500505510glupheglyvalalaargleuargthrmetvalargserileserser515520525proaspilevaltyralaalaservalalaglnleulysaspleuval530535540leuasnpheargleuhisphealaserserthrtyrthrvalleutrp545550555560histhralaleuiletyrilethrasngluleuleuthrglyprolys565570575aspasnasptrpphephetyrpheleuilecysvaltyrglytyrglu580585590argleuserargsertrpargvalthrthrserileserargalaleu595600605leusermetalaleuarglysglyglyilethrserthrthralaarg610615620thrileleulysaspleuglyproaspaspphearglyslystyrgly625630635640gluileargalathrphemetalaaspleuaspmetalaglugluasp645650655proserasnalathrvalgluargglnalagluaspphegluhisasn660665670alametleuargasptyrthrasnileleuaspalaaspglualaala675680685<210>53<211>1614<212>dna<213>里氏木霉<400>53atggcggcgtctacgctcttcatgccctctgcgggccccctctccaagaggctgtttaac60catgtcaagatgcttgctcacaaggtcacgaatgacaagtacaaatcggtagaaattgtc120ttggcgttcatgacccacatcccctggatatttccgggagaccacgcaatggacgacgag180acgtgcatgtatattgcgatggcgacaaccattgcgtttgacctgtccctccacaaggtg240ctgatgcccatggacgtcttggagtcgtcgacgacggtgtcaaagggagagtgtctggat300ccccggaccgcccttgccattgatgggttcccagacgtagatccgtggtctgagcagggc360cagctgctgctgcgagcgcgggaacgatgttacatttcgctatttgtcgtggagagaggg420tgagtgccggcttctcctcccatcttaactcctcgtctgacactgttctagaatggcatt480ggctcgcggtagaccttttatgataccaattacacgaaatatcaaggattgcgacacctg540gcatagatcaccgatagcagacgagcaggatggtcctgtggcgtccatggctgtcatgag600acgcaacttggtacgacggcgactgcgatcctggcctcgacaatctcatgctgactcttg660caggatgggcttttcaacacagttcgagcactttgcgatggctctcaggccagcaacagt720gatgggttcttggtggctcgatcgtacgtcgtcacccgagctcacttaacatcggcagaa780tcactaatcgatgcacaggattcaaggagctatagaaagattcttcgaccagtggctagc840ggaatggggcatgatgattggcttgggtcctggtaagtctcagcggaattggacttgtct900aagagacgtcacctgctgactgcaatggcagaacgtcgcctcccgccatatgtcgacatc960ctcgtcgcccacactcgcctctcgacgtacggacgcattatcaaccatccgacggcgcct1020gtcgaggtccgcatcttcttccgaaccgccgggttatcggccgccctgaacgtgatgagg1080gctgcgattcagggagagtcgcaactgcaatcgatgcccaacaacacggccattatgatt1140tcgtttgccgcatgctttgccctgaacattagcgcgtatgccccagacggctccgggctt1200gcaccgagcatcaggaggttgatcgaagaggccattggcgtgctggagcgtattggcacc1260gtcacgccgcacaggaatgggctctcggtgctgtatggaaagcatctccgacatctgctg1320cggatatcgggctcgtccaagggcacccggccgcccaaggcacagcggacaacgccgtcg1380tcgtcggagccgatgcaggacacgccgccggcgctgccgagcagcttcatggaccagcag1440atgctgtggccggacatggtgcagttttcgaccatgtcggacgaccagattgcgcaggtg1500ttgaatcagccgggcaacgtgtttgagccgtcgtcgtcgtttgggggcaacatgtcgtgg1560gaggagatgaacaactttgactggctgaactggccagctttcaatgcaaattag1614<210>54<211>464<212>prt<213>里氏木霉<400>54metalaalaserthrleuphemetproseralaglyproleuserlys151015argleupheasnhisvallysmetleualahislysvalthrasnasp202530lystyrlysservalgluilevalleualaphemetthrhisilepro354045trpilepheproglyasphisalametaspaspgluthrcysmettyr505560ilealametalathrthrilealapheaspleuserleuhislysval65707580leumetprometaspvalleugluserserthrthrvalserlysgly859095glucysleuaspproargthralaleualaileaspglypheproasp100105110valaspprotrpsergluglnglyglnleuleuleuargalaargglu115120125argcystyrileserleuphevalvalgluargglymetalaleuala130135140argglyargprophemetileproilethrargasnilelysaspcys145150155160aspthrtrphisargserproilealaaspgluglnaspglyproval165170175alasermetalavalmetargargasnleuaspglyleupheasnthr180185190valargalaleucysaspglyserglnalaserasnseraspglyphe195200205leuvalalaargserileglnglyalailegluargphepheaspgln210215220trpleualaglutrpglymetmetileglyleuglyprogluargarg225230235240leuproprotyrvalaspileleuvalalahisthrargleuserthr245250255tyrglyargileileasnhisprothralaprovalgluvalargile260265270phepheargthralaglyleuseralaalaleuasnvalmetargala275280285alaileglnglygluserglnleuglnsermetproasnasnthrala290295300ilemetileserphealaalacysphealaleuasnileseralatyr305310315320alaproaspglyserglyleualaproserileargargleuileglu325330335glualaileglyvalleugluargileglythrvalthrprohisarg340345350asnglyleuservalleutyrglylyshisleuarghisleuleuarg355360365ileserglyserserlysglythrargproprolysalaglnargthr370375380thrprosersersergluprometglnaspthrproproalaleupro385390395400serserphemetaspglnglnmetleutrpproaspmetvalglnphe405410415serthrmetseraspaspglnilealaglnvalleuasnglnprogly420425430asnvalphegluproserserserpheglyglyasnmetsertrpglu435440445glumetasnasnpheasptrpleuasntrpproalapheasnalaasn450455460<210>55<211>2678<212>dna<213>里氏木霉<400>55atggcctcaatgcatacctttccctccgtctccgagcccggggcggcgcagtcgccggcc60atgctggtcgatgcctcgaatcccagtgacggagccagtccctacgacacctcgtccatg120tctcagcagccctcgccgcgacagcagccgcaccagcatctcctgggcgcgtcttcacag180gcactgcagcagtcgcagcagcagcagcagaaccaaccacagaacgagccgtcacaacag240cagcaatcacagacccagcagcagcaacaacagcagcagcagcaacagcagcgcagcgtc300aagcggcccaggcccgtcaagtcctgcaccgagtgtcgcaaacgaaagcttcgctgcgat360cgcctctgcccctgctcgcaatgccaaaagtccaaccgtccctgcagatacgctcccgac420caagactcggccaacctgtccgatggctcggacgctgagggagccgagccgggccgccca480gccaagcgcaactactcgcacagcgcgctgcctgcgctggccgcctcgtacggcgatgct540gccgctcccagaccggccaaggcgagcgatcccgccagcctcccgttgctcgaggagctg600tccatacggatggagcgcctggagaagcagatcctcgcgcggagtccaagcagggctgag660ggcggcggcggcggcagcagcagcagcatcgtcgccggggcgccagagacgatacgggga720ttgacggtcaaacggggggccacgaggacgcggtactttgggcagaacgactcccgggtg780atgctaaacctggtaagttggccctgatgaaccattggaggtccgagtctcgatttgatc840cccccttccttgatccatcccctttgctgtggcagcaagagctgactctttctgttttct900ctctctcctagtttgacgatgccaaggcatatatagcccacaacttccggcgagagccct960tccccagcttcgagaagctacataggcatctgcgaagcgaattagcaaagtctctgaccc1020ccatcaccgtctttgtcgactctatgatgcctatccacaagagaatgatggacatcttgc1080ccagaaaggcagtctgtgaccgcctcatggcggcgtatttcgacacttcggagacgcaat1140acaggatcctccacaccccgacattttgggagcagtataaccagtactggcagggcaatc1200cgcagcccgagcagtttcttccccagatgctgtcggtgttggcggtagcgtcgaggttcg1260acaccaaatccagggggctgggccaccaggagcgcgtggagggcgtgcacatcccgactg1320cgtgcgcgctcgttcggagctggctcgacagcctcaagggcaagcagctcgtggagattg1380ccacgctgcaagtcgaagtgctgctgctgtttgcccagaggatgattatccagcggccgc1440aggattcgtggaaccatctgggcttcgtcgtgcgcatggccatgtccatgggccttcata1500gggacccctcggagtttgagccgcgcatgacgcccttccagggtgagatccgacggaggc1560tgtggttcacggtcctcgacatggacctgtacatgtcgcttgcctccaacatgccctgtc1620tcacccgagacggcgactattcctgccggccgccgcgcaacgtcgacgacagcgagctgt1680tccccgacatgacggagctgccgccgtctcggccccttgaccaggcgaccgacagccaga1740tacagatgtacaccatcatgacgcttcccacgcggatgaaggtggctcacatgatcaacc1800gcatcgataccatccgcgactaccaggaggtgctcgacgtgggcgggaagctcgaccggt1860tcatggaggacatcaactacatcttcccgcggcatgcgtccctcagcgacgcgcagaaga1920gcaggctctggcgcgaccgcgtcgtcctcgacatgcacgtgcggcgacctctgctggccc1980tgtaccgcccctttgccatgggggtgcccgacgccccggcgcaaatctcgcgcgcgtatc2040tgcggtcgtgcatggtgattctcaactacctggacgatttggaccactcgctgccgcact2100ttcgagacgtgtccgagatgtacctgcagatgttgcggcgcgacgtcatccaggccgccc2160tgagcgtctgctactttgtcaagccggccgtccgtcccgctgccgacagcgtcttgctcg2220cccagcacggcatacgggcctcgccaagccccgcggacgatttcccggcgtacgtcccgg2280acgacctgatgctgtggtcgccgtcgcggctcattggcacggtgcagaagacgattgatc2340tcctcgtgggcttcattcgcggcagcgacgtcaaggatatcctgtgcgtcgcggtcgtgc2400tggagacggtgaggaggcccgacatccgaagcgacgaaatcaccaagaatctgtttggcc2460tgctggacgcttgcctgagggcgagcagcatgaccctggacaagctccacgctttgtcac2520taggcggcggcggcggcggcagcagcagcagcaacgagtaccagcacgaggcgtatggcc2580atggcaggatgccgtttgtgcagcacggctacggcatggggggccattcggagacgacgg2640agttgggcggctggatcatgtgggatggctgggattga2678<210>56<211>852<212>prt<213>里氏木霉<400>56metalasermethisthrpheproservalsergluproglyalaala151015glnserproalametleuvalaspalaserasnproseraspglyala202530serprotyraspthrsersermetserglnglnproserproarggln354045glnprohisglnhisleuleuglyalaserserglnalaleuglngln505560serglnglnglnglnglnasnglnproglnasngluproserglngln65707580glnglnserglnthrglnglnglnglnglnglnglnglnglnglngln859095glnargservallysargproargprovallyssercysthrglucys100105110arglysarglysleuargcysaspargleucysprocysserglncys115120125glnlysserasnargprocysargtyralaproaspglnaspserala130135140asnleuseraspglyseraspalagluglyalagluproglyargpro145150155160alalysargasntyrserhisseralaleuproalaleualaalaser165170175tyrglyaspalaalaalaproargproalalysalaseraspproala180185190serleuproleuleuglugluleuserileargmetgluargleuglu195200205lysglnileleualaargserproserargalagluglyglyglygly210215220glyserserserserilevalalaglyalaprogluthrilearggly225230235240leuthrvallysargglyalathrargthrargtyrpheglyglnasn245250255aspserargvalmetleuasnleupheaspaspalalysalatyrile260265270alahisasnpheargargglupropheproserpheglulysleuhis275280285arghisleuargsergluleualalysserleuthrproilethrval290295300phevalaspsermetmetproilehislysargmetmetaspileleu305310315320proarglysalavalcysaspargleumetalaalatyrpheaspthr325330335sergluthrglntyrargileleuhisthrprothrphetrpglugln340345350tyrasnglntyrtrpglnglyasnproglnprogluglnpheleupro355360365glnmetleuservalleualavalalaserargpheaspthrlysser370375380argglyleuglyhisglngluargvalgluglyvalhisileprothr385390395400alacysalaleuvalargsertrpleuaspserleulysglylysgln405410415leuvalgluilealathrleuglnvalgluvalleuleuleupheala420425430glnargmetileileglnargproglnaspsertrpasnhisleugly435440445phevalvalargmetalametsermetglyleuhisargaspproser450455460gluphegluproargmetthrpropheglnglygluileargargarg465470475480leutrpphethrvalleuaspmetaspleutyrmetserleualaser485490495asnmetprocysleuthrargaspglyasptyrsercysargpropro500505510argasnvalaspaspsergluleupheproaspmetthrgluleupro515520525proserargproleuaspglnalathraspserglnileglnmettyr530535540thrilemetthrleuprothrargmetlysvalalahismetileasn545550555560argileaspthrileargasptyrglngluvalleuaspvalglygly565570575lysleuaspargphemetgluaspileasntyrilepheproarghis580585590alaserleuseraspalaglnlysserargleutrpargaspargval595600605valleuaspmethisvalargargproleuleualaleutyrargpro610615620phealametglyvalproaspalaproalaglnileserargalatyr625630635640leuargsercysmetvalileleuasntyrleuaspaspleuasphis645650655serleuprohispheargaspvalserglumettyrleuglnmetleu660665670argargaspvalileglnalaalaleuservalcystyrphevallys675680685proalavalargproalaalaaspservalleuleualaglnhisgly690695700ileargalaserproserproalaaspasppheproalatyrvalpro705710715720aspaspleumetleutrpserproserargleuileglythrvalgln725730735lysthrileaspleuleuvalglypheileargglyseraspvallys740745750aspileleucysvalalavalvalleugluthrvalargargproasp755760765ileargseraspgluilethrlysasnleupheglyleuleuaspala770775780cysleuargalasersermetthrleuasplysleuhisalaleuser785790795800leuglyglyglyglyglyglyserserserserasnglutyrglnhis805810815glualatyrglyhisglyargmetprophevalglnhisglytyrgly820825830metglyglyhissergluthrthrgluleuglyglytrpilemettrp835840845aspglytrpasp850<210>57<211>838<212>dna<213>嗜碱枝顶孢<400>57atgaagcttcttccctccttgattggcctggccagtctggcgtccctcgccgtcgcccgg60atccccggctttgacatttcgggctggcaaccgaccaccgactttgcaagggcgtatgct120aatggagatcgtttcgtctacatcaaggtacgttcaaccttgccaccaagttgcgaaccc180gagacaagactgtgaccgcctcctttgccctggggcagctcacgcacccagcagcatccc240atcccccggccccccacgtaccaccggaaagctaacatcaaccccctaccactgctacca300ggccaccgagggcaccacattcaagagctccgcattcagccgccagtacaccggcgcaac360gcaaaacggcttcatccgcggcgcctaccacttcgcccagcccgccgcgtcctcgggcgc420cgcgcaggcgagatacttcgccagcaacggcggcggctggtccaaggacggcatcaccct480gcccggggcgctggacatcgagtacaaccccaacggcgccacctgctacggcctctcgca540atcggccatggtgaactggatcgaggactttgtcaccacctaccacggcatcacctcccg600ctggcccgtcatctacaccaccaccgactggtggacccagtgcaccggcaactccaaccg660cttcgcgaaccgctgcccgctgtggatcgcccgctacgccagctccgtcggcactctgcc720caatggctggggcttttacaccttctggcagtacaacgacaagtatcctcagggcggtga780ttcgaactggttcaacggcgatgcgtcgcgtctcagggctctcgctaacggagactaa838<210>58<211>227<212>prt<213>嗜碱枝顶孢<400>58metlysleuleuproserleuileglyleualaserleualaserleu151015alavalalaargileproglypheaspileserglytrpglnprothr202530thraspphealaargalatyralaasnglyaspargphevaltyrile354045lysalathrgluglythrthrphelysserseralapheserarggln505560tyrthrglyalathrglnasnglypheileargglyalatyrhisphe65707580alaglnproalaalaserserglyalaalaglnalaargtyrpheala859095serasnglyglyglytrpserlysaspglyilethrleuproglyala100105110leuaspileglutyrasnproasnglyalathrcystyrglyleuser115120125glnseralametvalasntrpilegluaspphevalthrthrtyrhis130135140glyilethrserargtrpprovaliletyrthrthrthrasptrptrp145150155160thrglncysthrglyasnserasnargphealaasnargcysproleu165170175trpilealaargtyralaserservalglythrleuproasnglytrp180185190glyphetyrthrphetrpglntyrasnasplystyrproglnglygly195200205aspserasntrppheasnglyaspalaserargleuargalaleuala210215220asnglyasp225<210>59<211>50<212>dna<213>人工序列<220><223>人工dna序列<400>59gagtcgacctgcaggcatgcgtttaaacttggccacctacactgctacta50<210>60<211>43<212>dna<213>人工序列<220><223>人工dna引物<400>60cgtgaagccgtttaaatgaaactagctccagatggaaatatac43<210>61<211>42<212>dna<213>人工序列<220><223>人工dna引物<400>61tatttccatctggagctagtttcatttaaacggcttcacggg42<210>62<211>44<212>dna<213>人工序列<220><223>人工dna序列<400>62tcgttcgaaattttcttctagagagttcaaggaagaaacagtgc44<210>63<211>44<212>dna<213>人工序列<220><223>人工dna序列<400>63gaagaatcgactggctgcctactagctccagatggaaatatact44<210>64<211>44<212>dna<213>人工序列<220><223>人工dna引物<400>64tgtttcttccttgaactctctagaagaaaatttcgaacgaaccg44<210>65<211>42<212>dna<213>人工序列<220><223>人工dna引物<400>65tatttccatctggagctagtaggcagccagtcgattcttctt42<210>66<211>48<212>dna<213>人工序列<220><223>人工dna引物<400>66gctatgaccatgattacgccgtttaaaccgtccagataatgcgcacgc48<210>67<211>48<212>dna<213>人工序列<220><223>人工dna引物<400>67gagtcgacctgcaggcatgcgtttaaacacacacaggggtaccgtttc48<210>68<211>43<212>dna<213>人工序列<220><223>人工dna引物<400>68cgtgaagccgtttaaatgaagttgacggttgagcagaaaacgc43<210>69<211>43<212>dna<213>人工序列<220><223>人工dna引物<400>69ttttctgctcaaccgtcaacttcatttaaacggcttcacgggc43<210>70<211>44<212>dna<213>人工序列<220><223>人工dna引物<400>70gagtggtgggtttggtttgcgagagttcaaggaagaaacagtgc44<210>71<211>43<212>dna<213>人工序列<220><223>人工dna引物<400>71tgtttcttccttgaactctcgcaaaccaaacccaccactctac43<210>72<211>42<212>dna<213>人工序列<220><223>人工dna引物<400>72gatcagttcggatacgcgctgttgacggttgagcagaaaacg42<210>73<211>42<212>dna<213>人工序列<220><223>人工dna引物<400>73ttttctgctcaaccgtcaacagcgcgtatccgaactgatcta42<210>74<211>53<212>dna<213>人工序列<220><223>人工dna引物<400>74gctatgaccatgattacgccgtttaaacctacctgtcgaagaaataaaagagg53<210>75<211>20<212>dna<213>人工序列<220><223>人工dna引物<400>75tgaccgggcaggggatcgcc20<210>76<211>24<212>dna<213>人工序列<220><223>人工dna引物<400>76ctggggcgtcaagggacctgaatg24<210>77<211>25<212>dna<213>人工序列<220><223>人工dna引物<400>77ctacatcgaagctgaaagcacgaga25<210>78<211>23<212>dna<213>人工序列<220><223>人工dna引物<400>78gggacgccctgctgcaacttacc23<210>79<211>21<212>dna<213>人工序列<220><223>人工dna引物<400>79cgcccttcgacgagtcggcac21<210>80<211>20<212>dna<213>人工序列<220><223>人工dna引物<400>80tgaccgggcaggggatcgcc20<210>81<211>21<212>dna<213>人工序列<220><223>人工dna引物<400>81cgcccttcgacgagtcggcac21<210>82<211>25<212>dna<213>人工序列<220><223>人工dna引物<400>82ctacatcgaagctgaaagcacgaga25<210>83<211>22<212>dna<213>人工序列<220><223>人工dna引物<400>83tgccctggtttcgcgcatacgg22<210>84<211>26<212>dna<213>人工序列<220><223>人工dna引物<400>84acggataggagcagcaaagcaaaggc26<210>85<211>25<212>dna<213>人工序列<220><223>人工dna引物<400>85ctacatcgaagctgaaagcacgaga25<210>86<211>23<212>dna<213>人工序列<220><223>人工dna引物<400>86gggacgccctgctgcaacttacc23<210>87<211>25<212>dna<213>人工序列<220><223>人工dna引物<400>87gagacgagactggagtcgttgccgc25<210>88<211>22<212>dna<213>人工序列<220><223>人工dna引物<400>88tgccctggtttcgcgcatacgg22<210>89<211>25<212>dna<213>人工序列<220><223>人工dna引物<400>89gagacgagactggagtcgttgccgc25<210>90<211>25<212>dna<213>人工序列<220><223>人工dna引物<400>90ctacatcgaagctgaaagcacgaga25<210>91<211>22<212>dna<213>人工序列<220><223>人工dna引物<400>91tgccctggtttcgcgcatacgg22<210>92<211>26<212>dna<213>人工序列<220><223>人工dna引物<400>92acggataggagcagcaaagcaaaggc26<210>93<211>25<212>dna<213>人工序列<220><223>人工dna引物<400>93ctacatcgaagctgaaagcacgaga25<210>94<211>23<212>dna<213>人工序列<220><223>人工dna引物<400>94gggacgccctgctgcaacttacc23<210>95<211>25<212>dna<213>人工序列<220><223>人工dna引物<400>95gagacgagactggagtcgttgccgc25<210>96<211>22<212>dna<213>人工序列<220><223>人工dna引物<400>96tgccctggtttcgcgcatacgg22<210>97<211>25<212>dna<213>人工序列<220><223>人工dna引物<400>97gagacgagactggagtcgttgccgc25<210>98<211>25<212>dna<213>人工序列<220><223>人工dna引物<400>98ctacatcgaagctgaaagcacgaga25<210>99<211>8655<212>dna<213>人工序列<220><223>人工dna序列<400>99tcgcgcgtttcggtgatgacggtgaaaacctctgacacatgcagctcccggagacggtca60cagcttgtctgtaagcggatgccgggagcagacaagcccgtcagggcgcgtcagcgggtg120ttggcgggtgtcggggctggcttaactatgcggcatcagagcagattgtactgagagtgc180accatatgcggtgtgaaataccgcacagatgcgtaaggagaaaataccgcatcaggcgcc240attcgccattcaggctgcgcaactgttgggaagggcgatcggtgcgggcctcttcgctat300tacgccagctggcgaaagggggatgtgctgcaaggcgattaagttgggtaacgccagggt360tttcccagtcacgacgttgtaaaacgacggccagtgaattcgagctcggtacccggggat420cctctagagtcgacctgcaggcatgcaagctttctgctcgaggccatctggcttttctct480gctgtctgcctcgggaatgggatggaataccacgtacggtatttggcctccggtgccatc540cgaagcgagatgctttgagcttgaaaccccctcggcctgcacaggtgtctcatcgtgcat600ttaatccaacggcggcgagtcaaaacatcagctaattgaccaggtttctggattgtgaat660gccaactttttgggtcttgaggagttgcggggtgggaaaaaagtaaagaaatttactgag720gattttatcattgcgactataaaataaagcggcattgcaaatccttgcgttgctactatg780taaaatggactgtagttgtgctgctgaaaatagtttggcgattgtggattgtggattgtg840gattgtggattatggcaagttgtcaaggggcaagttgacgaaaatgattgtgtggtgtct900gccagcaaattgagaacgtgggtatatatttcatcttttcatgattcccttcggcttgct960tgtcaagcaatggcatcattggtctagtggtagaattcgtcgttgccatcgacgaggccc1020gtgttcgattcacggatgatgcagtcaaaagacctttttaatttctactcttgtagatct1080acaacacgtcctcgagggttttttttggctcttgggttcgaactgcccaaggcccatgtt1140ttggtcatctttttttttatgccccaccatttgggtcacccctgccaatcattccatctt1200tgttcctacccttcacgtgtgctttccgaagccaaagttcccattcaacaactctccttg1260cgttttttttttcttgaagcttgtcacccgtcgatagtttctgccatttgcaatcgagac1320agcagaatcaccgcccaagttaagcctttgtgctgatcatgctctcgaacgggccaagtt1380cgggaaaagcaaaggagcgtttagtgaggggcaatttgactcacctcccaggcaacagat1440gaggggggcaaaaagaaagaaattttcgtgagtcaatatggattccgagcatcattttct1500tgcggtctatcttgctacgtatgttgatcttgacgctgtggatcaagcaacgccactcgc1560tcgctccatcgcaggctggtcgcagacaaattaaaaggcggcaaactcgtacagccgcgg1620ggttgtccgctgcaaagtacagagtgataaaagccgccatgcgaccatcaacgcgttgat1680gcccagctttttcgatccgagaatccaccgtagaggcgatagcaagtaaagaaaagctaa1740acaaaaaaaaatttctgcccctaagccatgaaaacgagatggggtggagcagaaccaagg1800aaagagtcgcgctgggctgccgttccggaaggtgttgtaaaggctcgacgcccaaggtgg1860gagtctaggagaagaatttgcatcgggagtggggcgggttacccctccatatccaatgac1920agatatctaccagccaagggtttgagcccgcccgcttagtcatcgtcctcgcttgcccct1980ccataaaaggatttcccctccccctcccacaaaattttctttcccttcctctccttgtcc2040gcttcagtacgtatatcttcccttccctcgcttctctcctccatccttctttcatccatc2100tcctgctaacttctctgctcagcacctctacgcattactagccgtagtatctgagcactt2160ctcccttttatattccacaaaacataacacaaccttcaccatgaacaacggcacaaacaa2220cttccagaacttcattggaatctcgtcgttgcagaagactttgcgcaacgccctcatccc2280cacagaaactacccagcagttcattgtgaagaacggaatcatcaaggaagatgaactccg2340aggcgagaaccgccagattttgaaggacatcatggatgattactaccgtggtttcatctc2400ggaaacgctctcctccattgacgacatcgattggacttcgttgttcgaaaagatggaaat2460ccagctcaaaaacggcgataacaaggataccttgatcaaggagcagaccgagtatcggaa2520ggcgatccataagaagttcgccaacgatgatcggttcaagaacatgttctcggccaagtt2580gatttccgacattctccccgaattcgtgatccataacaacaactactcggcgtcggagaa2640ggaggagaagacgcaggtcatcaagttgttctcgaggttcgccacatcgttcaaagacta2700ttttaagaatcgtgcgaactgtttctcggcagatgatatctcctcgtcctcctgtcaccg2760cattgtgaacgacaacgcggaaatcttcttctcgaacgcgttggtgtataggcgcatcgt2820gaagtccctctccaacgatgacatcaacaaaatctcgggagatatgaaggattcgctcaa2880ggagatgtcgttggaggaaatctactcctatgagaagtatggcgagttcattacgcagga2940gggcatttccttctacaacgacatttgtggtaaagtcaactcgttcatgaacctctactg3000tcagaaaaacaaggagaacaaaaacctctataagctccagaagttgcataagcagatcct3060ctgtatcgcagacacctcgtacgaggtcccttacaagttcgaatccgatgaggaggtcta3120ccagtccgtcaacggattcttggacaacatctcctcgaaacacattgtcgagcggctccg3180aaagatcggcgataactacaacggctacaacttggacaaaatctatatcgtctccaagtt3240ctatgagtccgtctcgcagaaaacctatcgtgattgggagactatcaacactgcgctcga3300gattcactataacaacatcttgcctggtaacggcaaatcgaaagccgacaaggtgaagaa3360ggccgtgaaaaacgatctccagaagtcgatcacagaaatcaacgaactcgtctcgaacta3420caagctctgttcggatgataacatcaaggcggaaacgtacatccatgaaatctcgcatat3480cttgaacaacttcgaggcccaggaactcaaatacaaccccgagatccacttggtcgagtc3540ggagctcaaagcctcggagttgaagaacgtcttggatgtcatcatgaacgcattccactg3600gtgttccgtgttcatgaccgaggaactcgtcgataaagacaacaacttctacgcggaact3660cgaggaaatctacgatgaaatctatcccgtgatctccctctacaacctcgtgcgaaacta3720cgtcactcagaagccctattccaccaagaagatcaagctcaacttcggcatccccactct3780cgcagacggttggtcgaagtcgaaggagtactccaacaacgccattatcctcatgcgaga3840caacctctactacttgggtatcttcaacgcaaagaacaagccggataagaagatcattga3900aggcaacacttcggaaaacaagggagactataagaagatgatctacaacctcctccctgg3960acccaacaagatgattcctaaagtgttcctctcgtcgaagactggtgtggaaacgtataa4020gccgtcggcctacatcttggagggctacaaacagaacaagcatatcaagtcctcgaagga4080cttcgacatcactttctgtcacgacctcatcgactatttcaagaactgtattgcaatcca4140tccggaatggaagaacttcggcttcgatttctcggatacttcgacatacgaagatatctc4200gggattctaccgagaggtcgaattgcagggctataagattgattggacctacatctcgga4260aaaggatatcgacttgctccaggaaaagggccagctctacctcttccagatttacaacaa4320ggacttctccaagaagtcgacgggtaacgacaacttgcacacaatgtatctcaaaaacct4380cttctcggaggagaacttgaaggatatcgtgctcaaattgaacggagaggccgaaatctt4440cttccgtaagtcctccatcaagaacccgatcatccataagaagggatcgatcttggtcaa4500ccggacttacgaagcagaggaaaaagatcagttcggaaacatccagattgtcaggaagaa4560catccctgaaaacatctatcaggagttgtataagtacttcaacgacaagtcggataagga4620gctctccgacgaagcagccaaactcaagaacgtcgtcggacaccatgaagcagcaaccaa4680cattgtgaaggactaccggtacacttacgacaagtacttcttgcacatgccgatcactat4740caacttcaaagccaacaagaccggattcattaacgacaggatcctccagtacattgccaa4800agaaaaggacctccatgtcatcggtatcgataggggagaacggaacctcatctacgtctc4860cgtgattgacacttgtggcaacattgtcgaacagaagtcgttcaacatcgtcaacggtta4920cgattaccagattaagttgaaacagcaggaaggtgcgaggcagattgcgcgaaaggaatg4980gaaggagattggcaaaatcaaggagattaaggaaggctacttgtcgttggtcatccacga5040aatctcgaaaatggtgatcaaatacaacgccatcatcgccatggaagacctctcgtacgg5100cttcaaaaagggacggttcaaagtggagcgtcaggtgtaccagaagttcgaaacaatgtt5160gatcaacaagttgaactacttggtgttcaaggacatttccattaccgagaacggaggatt5220gctcaagggttatcagctcacgtacatccccgacaagttgaaaaacgtgggacaccagtg5280tggctgtatcttctacgtgcctgcagcctacacgtcgaaaatcgaccctacaacaggatt5340cgtgaacatcttcaagttcaaggatctcaccgtcgacgcgaagcgggagttcatcaaaaa5400gttcgactccatccgctatgattcggagaagaacttgttctgtttcacattcgactacaa5460caacttcattactcagaacaccgtgatgtccaaatcgtcgtggtccgtgtacacgtatgg5520tgtgcgcatcaaaaggcgcttcgtcaacggtcgcttctccaacgaatcggacacgatcga5580tatcacgaaagacatggagaaaacattggaaatgaccgacatcaactggcgtgacggcca5640tgacctcaggcaggacatcatcgattacgagatcgtccagcacatcttcgaaatcttccg5700tctcaccgtgcagatgaggaactccctctccgagctcgaagatcgggattacgaccggct5760catttcccctgtgttgaacgagaacaacatcttctacgactcggcaaaagcgggagatgc5820attgccgaaggacgccgatgcgaacggtgcatattgtattgcactcaagggtctctacga5880aatcaagcagatcaccgaaaactggaaggaggacggcaaattctcgagggacaagttgaa5940gatttcgaacaaggattggttcgatttcatccagaacaagaggtacttgcctccgaagaa6000gaagcgaaaggtgtgagcggacattcgatttatgccgttatgacttccttaaaaaagcct6060ttacgaatgaaagaaatggaattagacttgttatgtagttgattctacaatggattatga6120ttcctgaacttcaaatccgctgttcattattaatctcagctcttcccgtaaagccaatgt6180tgaaactattcgtaaatgtacctcgttttgcgtgtaccttgcttatcacgtgatattaca6240tgacctggacagagttctgcgcgaaagtcataacgtaaatcccgggcggtaggtgcgtcc6300cgggcggaaggtagttttctcgtccaccccaacgcgtttatcaacctcaactttcaacaa6360ccatcatgccaccaaaagcgcgtaaaacaaagcgagatttgattgagcaagagggcagga6420tggcgtaatcatggtcatagctgtttcctgtgtgaaattgttatccgctcacaattccac6480acaacatacgagccggaagcataaagtgtaaagcctggggtgcctaatgagtgagctaac6540tcacattaattgcgttgcgctcactgcccgctttccagtcgggaaacctgtcgtgccagc6600tgcattaatgaatcggccaacgcgcggggagaggcggtttgcgtattgggcgctcttccg6660cttcctcgctcactgactcgctgcgctcggtcgttcggctgcggcgagcggtatcagctc6720actcaaaggcggtaatacggttatccacagaatcaggggataacgcaggaaagaacatgt6780gagcaaaaggccagcaaaaggccaggaaccgtaaaaaggccgcgttgctggcgtttttcc6840ataggctccgcccccctgacgagcatcacaaaaatcgacgctcaagtcagaggtggcgaa6900acccgacaggactataaagataccaggcgtttccccctggaagctccctcgtgcgctctc6960ctgttccgaccctgccgcttaccggatacctgtccgcctttctcccttcgggaagcgtgg7020cgctttctcatagctcacgctgtaggtatctcagttcggtgtaggtcgttcgctccaagc7080tgggctgtgtgcacgaaccccccgttcagcccgaccgctgcgccttatccggtaactatc7140gtcttgagtccaacccggtaagacacgacttatcgccactggcagcagccactggtaaca7200ggattagcagagcgaggtatgtaggcggtgctacagagttcttgaagtggtggcctaact7260acggctacactagaaggacagtatttggtatctgcgctctgctgaagccagttaccttcg7320gaaaaagagttggtagctcttgatccggcaaacaaaccaccgctggtagcggtggttttt7380ttgtttgcaagcagcagattacgcgcagaaaaaaaggatctcaagaagatcctttgatct7440tttctacggggtctgacgctcagtggaacgaaaactcacgttaagggattttggtcatga7500gattatcaaaaaggatcttcacctagatccttttaaattaaaaatgaagttttaaatcaa7560tctaaagtatatatgagtaaacttggtctgacagttaccaatgcttaatcagtgaggcac7620ctatctcagcgatctgtctatttcgttcatccatagttgcctgactccccgtcgtgtaga7680taactacgatacgggagggcttaccatctggccccagtgctgcaatgataccgcgagacc7740cacgctcaccggctccagatttatcagcaataaaccagccagccggaagggccgagcgca7800gaagtggtcctgcaactttatccgcctccatccagtctattaattgttgccgggaagcta7860gagtaagtagttcgccagttaatagtttgcgcaacgttgttgccattgctacaggcatcg7920tggtgtcacgctcgtcgtttggtatggcttcattcagctccggttcccaacgatcaaggc7980gagttacatgatcccccatgttgtgcaaaaaagcggttagctccttcggtcctccgatcg8040ttgtcagaagtaagttggccgcagtgttatcactcatggttatggcagcactgcataatt8100ctcttactgtcatgccatccgtaagatgcttttctgtgactggtgagtactcaaccaagt8160cattctgagaatagtgtatgcggcgaccgagttgctcttgcccggcgtcaatacgggata8220ataccgcgccacatagcagaactttaaaagtgctcatcattggaaaacgttcttcggggc8280gaaaactctcaaggatcttaccgctgttgagatccagttcgatgtaacccactcgtgcac8340ccaactgatcttcagcatcttttactttcaccagcgtttctgggtgagcaaaaacaggaa8400ggcaaaatgccgcaaaaaagggaataagggcgacacggaaatgttgaatactcatactct8460tcctttttcaatattattgaagcatttatcagggttattgtctcatgagcggatacatat8520ttgaatgtatttagaaaaataaacaaataggggttccgcgcacatttccccgaaaagtgc8580cacctgacgtctaagaaaccattattatcatgacattaacctataaaaataggcgtatca8640cgaggccctttcgtc8655<210>100<211>63<212>dna<213>人工序列<220><223>人工dna引物<400>100tttaatttctactcttgtagatgcattgtcaaagcatcgcccatttttttggctcttggg60ttc63<210>101<211>879<212>dna<213>人工序列<220><223>人工dna序列<400>101agagtcgacctgcaggcatgaaccggagagtactatcaaccgagcgcctggtttttacct60ttcaatctcgaccctgtcggtgcaggtgtcggtcttgatcccaaccctgcggcttccgtg120cccagtagcgaaaatgccactccagacttatccgcgtttggcggtactggcataccactg180ggagggtatgatctgggaatgacgggaatgaatcaaaggtcccatcggtgatgggtattg240ctcctttttatttttttttttattttttttctctctttgcgagcggttctggttgggcga300atatggtgtcttggaaaagggtggggggttcacgacttctatatgctctgtatgctgaac360tgtttgtgtaactgagttgtatatccctgctttactccgtactctgatccattactttct420ttgtctgtgtcgtctaatctcgttgccatactgacccgcttaccgaccaatcatgccact480ggaaattcctttatagttcattctaatgtcttcacaagtggcttgcttgtcaagcaatgg540catcattggtctagtggtagaattcgtcgttgccatcgacgaggcccgtgttcgattcac600ggatgatgcaaatttctactcttgtagatatctgcccagtggcgttcgcctttttttttg660agcgcttacaggccacatttttaggtaagatgtcagccatgttgatggggcattgcgagt720agcggtcgttcgaaacaagatggagtctggtcaggcatggaagcaaaagtcatggcgaga780atatgtatgtcctcctttgagatcggaatatcgtggtcacatgttcaagcgtaatggtac840tgtaggatccagaaaaaaacaagctttctgctcgaggcc879<210>102<211>27<212>dna<213>人工序列<220><223>人工dna引物<400>102gaattcaaaagcgccagtcactgcgag27<210>103<211>25<212>dna<213>人工序列<220><223>人工dna引物<400>103ctacatcgaagctgaaagcacgaga25<210>104<211>28<212>dna<213>人工序列<220><223>人工dna引物<400>104tcgtcgagtcgaagatgagagaggatgg28<210>105<211>23<212>dna<213>人工序列<220><223>人工dna引物<400>105gggacgccctgctgcaacttacc23<210>106<211>27<212>dna<213>人工序列<220><223>人工dna引物<400>106gaattcaaaagcgccagtcactgcgag27<210>107<211>25<212>dna<213>人工序列<220><223>人工dna引物<400>107gagacgagactggagtcgttgccgc25<210>108<211>20<212>dna<213>人工序列<220><223>人工dna引物<400>108tgaccgggcaggggatcgcc20<210>109<211>25<212>dna<213>人工序列<220><223>人工dna引物<400>109ctacatcgaagctgaaagcacgaga25<210>110<211>24<212>dna<213>人工序列<220><223>人工dna引物<400>110ctggggcgtcaagggacctgaatg24<210>111<211>23<212>dna<213>人工序列<220><223>人工dna引物<400>111gggacgccctgctgcaacttacc23<210>112<211>20<212>dna<213>人工序列<220><223>人工dna引物<400>112tgaccgggcaggggatcgcc20<210>113<211>21<212>dna<213>人工序列<220><223>人工dna引物<400>113cgcccttcgacgagtcggcac21<210>114<211>27<212>dna<213>人工序列<220><223>人工dna引物<400>114gaattcaaaagcgccagtcactgcgag27<210>115<211>25<212>dna<213>人工序列<220><223>人工dna引物<400>115ctacatcgaagctgaaagcacgaga25<210>116<211>28<212>dna<213>人工序列<220><223>人工dna引物<400>116tcgtcgagtcgaagatgagagaggatgg28<210>117<211>20<212>dna<213>人工序列<220><223>人工dna引物<400>117tgaccgggcaggggatcgcc20<210>118<211>25<212>dna<213>人工序列<220><223>人工dna引物<400>118ctacatcgaagctgaaagcacgaga25<210>119<211>24<212>dna<213>人工序列<220><223>人工dna引物<400>119ctggggcgtcaagggacctgaatg24<210>120<211>23<212>dna<213>人工序列<220><223>人工dna引物<400>120gggacgccctgctgcaacttacc23<210>121<211>20<212>dna<213>人工序列<220><223>人工dna引物<400>121tgaccgggcaggggatcgcc20<210>122<211>21<212>dna<213>人工序列<220><223>人工dna引物<400>122cgcccttcgacgagtcggcac21<210>123<211>1409<212>dna<213>里氏木霉<400>123atgttccccgagttgaccagtggcacctggtcaccacccaatggcagcactactcagcct60ctttcgccgagggatgtggcttcttcccctggtaacaggcgcaggaggcctgtagacgaa120gcggatgaagaggaagcccatcgaccacgcaccgtgccgcgggtacatcgtgaccccatg180cagcgcgtgtcccgttctcctcaaagcccttcctccacgtcatggagctcaggaggttgg240accactaccccagcaaccgccgtctctccgacttcctcctttgccaaaccagcaccaatg300gaggtgcaggagcggtcacctgctgttcggttcgctctaccattcccgctcccgagccag360cctggtgcgcaccactcggaggtttcgccaagcaaggtccaagagtggcccagacactat420agccatgattatacccaccaccaccgcgctcagtaccacccggacagcgacagaagcgaa480tctcgagaggctaccagggcatatagcatggattacacccactggcacaaccaccatacc540tatcagtcgcaacccacaagcccccgtttgccttacgacccgacaagatactctgcgggc600gtgtatcccgctccgcatcacatggagcctaacccatacggggagtcaggggccacgtcc660ggtggcggcgccagaccacgtaggcggcgcggcaaccttcccaaggagacgacagatcag720ttgcgggcctggctcaacgcgcacctgcaccacccttatcctacagaggatgagaagcag780cagctgatgcgcacaactggacttcagatgagtaagtggcctctacatcgtccaattacc840cgatttgtgcctcgcggctaaccgcccccctctctttagatcaaatctccaactggttca900tcaacgcaagaagacgccaagtacccagcttgctcagagagcgaaatgccgagattatcg960atccaaaccgaatgatgaccagccccaacaggagatccaggtcctcgtccatcagtgacg1020gcgatctcagctcgagcgaatgggggcctgacgctcaaggccagggcgattgggcagcca1080gacgccgaagccgcagcgtataaaccttcgttgctcgagtcgaccgcattgcctagcaga1140atcaccctggcacgagaagagccatttccctccaaaaaaacagcattccctaaaacgaaa1200atggccggcctcaatggcaaaagagtccgaagagcaacggaatatacttcaggaaaaagc1260taatgggggtgtataaccctccctttttacgactacggttaattgaatgacatcacgaga1320tgttattcgatggctcttctacctttttttttttttttttttttttttcatctcttactt1380cttcaaatccagtcatggttggcctgtga1409<210>124<211>275<212>prt<213>里氏木霉<400>124metpheprogluleuthrserglythrtrpserproproasnglyser151015thrthrglnproleuserproargaspvalalaserserproglyasn202530argargargargprovalaspglualaaspglugluglualahisarg354045proargthrvalproargvalhisargaspprometglnargvalser505560argserproglnserproserserthrsertrpserserglyglytrp65707580thrthrthrproalathralavalserprothrserserphealalys859095proalaprometgluvalglngluargserproalavalargpheala100105110leupropheproleuproserglnproglyalahishissergluval115120125serproserlysvalglnglutrpproarghistyrserhisasptyr130135140thrhishishisargalaglntyrhisproaspseraspargserglu145150155160serargglualathrargalatyrsermetasptyrthrhistrphis165170175asnhishisthrtyrglnserglnprothrserproargleuprotyr180185190aspprothrargtyrseralaglyvaltyrproalaprohishismet195200205gluproasnprotyrglygluserglyalathrserglyglyglyala210215220argproargargargargglyasnleuprolysgluthrthraspgln225230235240leuargalatrpleuasnalahisleuhishisprotyrprothrglu245250255aspglulysglnglnleumetargthrthrglyleuglnmetilemet260265270valglyleu275<210>125<211>51<212>dna<213>人工序列<220><223>人工dna引物<400>125gagtcgacctgcaggcatgcttaattaacaattcctcgtgacagtttctgc51<210>126<211>46<212>dna<213>人工序列<220><223>人工dna引物<400>126cttgctcggtcctggcgtagacttatcacaaagttagccaaacagg46<210>127<211>20<212>dna<213>人工序列<220><223>人工dna引物<400>127tctacgccaggaccgagcaa20<210>128<211>20<212>dna<213>人工序列<220><223>人工dna引物<400>128tggaaacgcaaccctgaagg20<210>129<211>43<212>dna<213>人工序列<220><223>人工dna引物<400>129tcccttcagggttgcgtttccatgaactaccagcatacacgac43<210>130<211>43<212>dna<213>人工序列<220><223>人工dna引物<400>130acagctatgaccatgattacgcctccttgtttgatcctagccc43<210>131<211>28<212>dna<213>人工序列<220><223>人工dna引物<400>131atgcccagtcgcgaataatcactcagcc28<210>132<211>25<212>dna<213>人工序列<220><223>人工dna引物<400>132ggctgagtagtgctgccattgggtg25<210>133<211>33<212>dna<213>人工序列<220><223>人工dna引物<400>133ccataaggtggcgttgttacatctccctgagag33<210>134<211>23<212>dna<213>人工序列<220><223>人工dna引物<400>134ccgtcctcggtcaggagccttgg23<210>135<211>35<212>dna<213>人工序列<220><223>人工dna引物<400>135cttacttcttcaaatccagtcatggttggcctgtg35<210>136<211>35<212>dna<213>人工序列<220><223>人工dna引物<400>136cttacttcttcaaatccagtcatggttggcctgtg35当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1