一种表观遗传的DAP-seq测序建库方法与流程
本发明属于高通量测序建库技术,具体涉及一种表观遗传的dap-seq测序建库方法。
背景技术:
表观遗传学研究中,转录因子(tf,transcriptionfactors)结合位点的发掘一直是难题,chip-seq是最有效的直接研究目标tf结合位点的技术。chip-seq通过特定的tf抗体,将与tf结合的dna片段通过免疫共沉淀拉取下来,再通过高通量测序,就可以直接检测特定tf在基因组上的结合位点以及分析结合位点的motif(结合位点的特征序列)。
但是chip-seq并非对所有的物种都适用,主要存在以下的一些难点:(1)抗体制备的难点:人、小鼠等这些常见模式物种的热门转录因子,通常有商业化的chip级抗体可以用,开展chip-seq还相对容易。但对于非热门转录因子或非模式物种,则无可用的商业化抗体。而制备chip级抗体难度大,周期长,是个完全不可控的过程;(2)替代方案的实验周期长:对于没有抗体的tf,一个替代方案就是构建重组蛋白,即在目标tf序列上接上flag标签序列或者gfp(绿色荧光蛋白)序列。然后通过转基因的手段,将重组后的tf序列导入到细胞或实验动物/植物体内,表达带有flag肽段或gfp标签的tf融合蛋白。然后利用flag或gfp的抗体,就可以实现目标tf的chip-seq。这种方案依然有一个不足:对于某些物种,转基因实验难度很大或周期很长,实验门槛依然很高。
对于以上的chip-seq实验的不足,也存在一些体外实验的方法作为替代方案。比较有代表性的是指数富集的配体系统进化技术(systematicevolutionofligandsbyexponentialenrichment,selex)和蛋白结合芯片(proteinbindingmicroarrays,pbm)。这两种方法都是人工合成dna片段/探针库,然后在体外条件下与目标蛋白(tf)结合。通过对于目标蛋白结合的dna序列进行检测,获得目标tf的结合位点信息。此类方法最大的不足是dna均为人工合成,没有原始基因组dna上的修饰信息(例如dna甲基化)。而dna甲基化等信息,是影响tf结合的重要因素,所以selex、pbm等方法很难还原tf在体内真实的结合情况,与chip-seq效果上还有较大差异。
由此可知,现有技术中普遍存在效果差,破坏了原始基因组dna上的修饰信息,难以还原tf在体内真实情况的缺点。
技术实现要素:
为了解决现有技术中存在的技术问题,本发明的目的在于提供一种表观遗传的dap-seq测序建库方法。dap-seq的全称是dnaaffinitypurificationsequencing(dna亲和纯化测序),本发明公开的dap-seq测序建库方法,既可以开展体外实验,又可以得到更加接近体内实验(chip-seq)的效果。
为了达到上述目的,本发明采用的技术方案如下:
一种表观遗传的dap-seq测序建库方法,包括如下步骤:
s1、构建基因表达载体;
s2、tf体外表达预实验;
s3、dap-seq建库。
优选地,步骤s1的具体操作如下:
(1)根据tf家族的基因序列,设计合成相关基因的引物,得tf引物;
(2)用步骤s1获得的tf引物,通过pcr扩增tf基因;
(3)利用1%的琼脂糖凝胶电泳检测,跑胶鉴定目的条带,回收含目标条带的琼脂糖凝胶,得含目标条带的pcr产物;
(4)将步骤(3)所得含目标条带的pcr产物,用infusion连接方法,以pfn19k
(5)取出感受态细胞于冰上解冻,得感受态细胞悬液;
(6)向步骤(5)所得感受态细胞悬液中加入5μl的infusion连接产物及连接好的载体,于42℃热激60s,然后冰浴2min;
(7)向每个离心管中加入液体培养基,37℃复苏dh5α大肠杆菌化学感受态细胞,然后于室温,6000rpm的条件下离心5min,去掉上清液,得重悬细菌;
(8)将步骤(7)所得重悬细菌均匀涂在lb+kan(卡那霉素kanamycin,kan,10mg/ml)固体培养基上,干燥后,倒置平板,37℃过夜培养;
(9)挑选单克隆菌,并分别接菌于lb+kan(卡那霉素kanamycin,kan,10mg/ml)液体培养基中,37℃摇菌,至肉眼可见的浑浊;
(10)鉴定阳性克隆菌,并通过1%的琼脂糖凝胶电泳鉴定目的条带,挑选出阳性单克隆菌;
(11)将步骤(10)挑选出的阳性单克隆菌接种于液体培养基中,37℃过夜培养,然后通过提取构建好的目的tf的重组表达质粒,进行pcr鉴定并测序,挑选出与目标序列一致的单克隆菌,即得。
优选地,步骤(4)中的pfn19k
优选地,步骤(7),步骤(9)及步骤(11)中的液体培养基为lb液体培养基;步骤(8)中的固体培养基为lb固体培养基。
优选地,步骤s2的具体操作过程如下:
(1)配制5μl的反应体系,25℃金属浴2h;
(2)制备page胶,每个孔加1μl样品,进行电泳;
(3)用甲醇提前预处理8.5cm×4.0cm的pvdf膜,备用;
(4)将电泳完毕的凝胶去除浓缩胶后,放入8.5cm×4.0cm的pvdf膜,膜转移buffer中,平衡5min;
(5)在胶黑膜白的前提下,放入转膜槽中转膜;
(6)小心将pvdf膜取出,正面朝上,加入适量封闭液;
(7)一抗溶于封闭液,混匀后,加在8.5cm×4.0cm的pvdf膜上,孵育60min;
(8)二抗溶于封闭液,混匀后,加在8.5cm×4.0cm的pvdf膜上,孵育60min;
(9)将pvdf膜正面朝上,加入1ml的thermoscientificsupersignalwestpicopluschemiluminescentsubstrate作为荧光底物,于25℃的条件下反应2min,然后将pvdf膜放入暗盒,加入显影胶片;
(10)在暗室中曝光、显影、定影,将底片在自来水下冲洗干净后,烘干,保存,分析结果,若tf能正常体外表达,即可。
优选地,步骤(1)中的5μl的反应体系包括:3μl的
优选地,步骤(7)中的一抗指anti-halotagmonoclonalantibody,步骤(8)中的二抗指goatanti-mouseigg(h+l)hrpconjugate。
优选地,步骤s3的具体操作过程为:
(1)将dna用超声打断,然后用磁珠分选dna片段,得分选后的dna片段;
(2)在步骤(1)所得分选后的dna片段末端修复加a,同时加接头,得构建好的dna文库;
(3)体外表达带有halotag的目的蛋白,然后将该蛋白与磁珠结合,于25℃的条件下金属浴2h,得产物i;
(4)将步骤(3)所得产物i与步骤(2)所得构建好的dna文库混合,于25℃的条件下振荡1h,洗涤磁珠,洗脱样本,得洗脱液;
(5)将步骤(4)所得洗脱液进行pcr反应,获得pcr产物,用磁珠纯化,即可。
优选地,步骤(2)中的a指腺嘌呤,接头是短核苷酸序列,接头1的序列信息如seqidno.1所示;接头2的序列信息如seqidno.2所示。
p-gatcggaagagcacacgtctg(seqidno.1);
cacgacgctcttccgatct(seqidno.2);
与现有技术相比,本发明提供的dap-seq测序建库方法具有如下优点:
(1)该方法可以直接使用从细胞中提取的基因组dna与目标tf结合,从而可以保留基因组dna修饰(例如,甲基化修饰,从而更加接近chip-seq实验的效果;
(2)tf结合位点dna片段的富集过程更加简单:在表达带有halo-tag标签的tf后,将halo-tag标签与带有对应配体的磁珠连接,从而给tf带上了磁性;
(3)富集方法(磁珠富集)比传统的ip实验(抗原抗体)简单,所以便于大规模开展;
(4)不需要抗体,就可以开展转录因子结合位点研究。这对非模式物种领域,是非常重要的优势;
(5)虽然是体外实验,但可以部分保留表观修饰(dna甲基化)对tf结合的影响。
附图说明
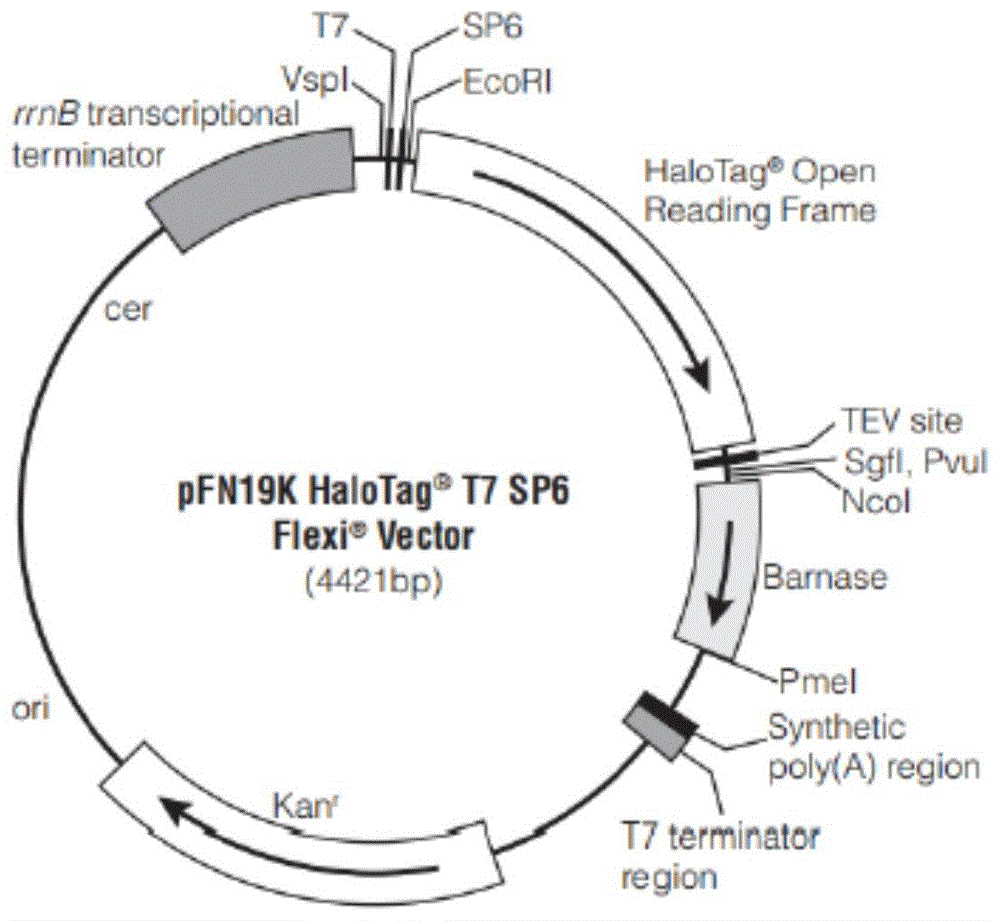
图1为原halotag表达载体图;
图2为tf体外表达预实验结果图;
图3为本发明实施例dna超声破碎结果图;
图4为本发明实施例文库质检结果图。
具体实施方式
下面结合具体实施例对本发明作进一步解释,但是应当注意的是,以下实施例仅用以解释本发明,而不能用来限制本发明,所有与本发明相同或相近的技术方案均在本发明的保护范围之内。若未特别指明,实施例中所用的技术手段为本领域技术人员所熟知的常规手段,所用原料为市售商品。
其中,本发明所用试剂均为常用试剂,均可在常规试剂生产销售公司购买。
实施例一种表观遗传的dap-seq测序建库方法
所述表观遗传的dap-seq测序建库方法,包括如下步骤:
s1、构建基因表达载体:
(1)以r2r3-myb转录因子为例,其cds序列全长如seqidno.3所述,根据infusion试剂盒说明书,设计合成r2r3-myb转录因子的引物,得tf引物,引物中加下划线的序列是载体上的末端同源重组序列;其中,tf引物的上游引物为tf-f,引物序列如seqidno.4所示,下游引物为tf-r,引物序列如seqidno.5所示;
atgaaaggggttcgtttaggaatgagaaagggtgcttggactcgggaagaagatctccttcttaggaactgcattcaaaagtatggagaaggagtttggcaccaagttcctctcagagcaggcttgaacagatgtagaaaaagctgtagattgaggtggttgaattatttgaagccaaatataaagagaggaaactttaccttcgatgaagttgatctcatcatcaagcttcataaattgttaggcaacagatggtcgctaatagcaggcagacttcccggaagaacccctaatgatgtgaaaaacttttggaacacccacatgcataagaaaatgatagctcaaagagaggaggaacgagccaaggctcataaaaaacccatgaaatataataacatcatcaaaccccagcctaggaccttctcaagaaacctacttttcccaaggggcaaaagtttcaatacagaaaacattcaaactgaaggcaatttcccaaaggcacctccagcatcattgctaggggattgtggagcaccaccatggttggatggcggcgtactcgacagtatggaaatgaatagtgaaatttcatggtccatgtatggctcagctgatcaagagccctttcaagtgccgtggctgcctgaagaacttacaacgtcaatgttggcggctagtgacaattctgctgaaggaggtcaaagtgattggattgacaatttgacttgtaatttggatctctgggattttcttaaatga(seqidno.3)
actttcagagcgataacgcgatgaaaggggttcgtttagga;(seqidno.4);
taccgagcccgaattcgttttcatttaagaaaatcccagag(seqidno.5);
(2)用步骤s1获得的tf引物,配制成10μl的pcr反应体系:mastermix5μl,上、下游引物各1μl,全基因组dna1μl,加余量超纯水;pcr反应程序为:95℃预变性3min,95℃变性30s,56℃退火30s,72℃延伸1min,循环35次;最后延伸10min,即得tf基因;
(3)利用1%的琼脂糖凝胶电泳检测,于130v的电压下跑胶20min,鉴定目的条带,回收含目标条带的琼脂糖凝胶,得含目标条带的pcr产物;
(4)将步骤(3)所得含目标条带的pcr产物进行用infusion连接方法,以pfn19k
所述halotag表达载体是在promega的pfn19khalotagt7sp6flexivector(如图1)的基础上改造而来。为方便客户构建载体,本发明用常见的致死基因ccdb替换了致死基因barnase。同时,在致死基因ccdb两端分别引入了hindiii(aagctt)和ecorv(gatatc)两个酶切位点。这样,客户可以用比较常见的大肠杆菌db3.1克隆含有致死基因ccdb的halotag表达载体。在载体线性化时,引入hindiii和ecorv两个不同的酶切位点,减少载体自连。此外,致死基因ccdb方便客户构建载体时筛选阳性克隆。
(5)取出感受态细胞于冰上解冻,得感受态细胞悬液;
(6)向步骤(5)所得感受态细胞悬液中加入连接产物,于42℃,热激60s然后冰浴2min;
(7)向每个离心管中加入lb液体培养基1ml,37℃复苏菌体,然后于室温,6000rpm的条件下离心5min,去掉上清液,得重悬细菌;
(8)将步骤(7)所得重悬细菌均匀涂在lb+kan(卡那霉素kanamycin,kan,10mg/ml)固体培养基上,干燥后,倒置平板,37℃过夜培养;
(9)挑选单克隆菌,并分别接菌于lb+kan(卡那霉素kanamycin,kan,10mg/ml)液体培养基中,37℃摇菌,至肉眼可见的浑浊;
(10)鉴定阳性克隆菌,并通过1%的琼脂糖凝胶电泳鉴定目的条带,挑选出阳性单克隆菌;
(11)将步骤(10)挑选出的阳性单克隆菌接种于lb液体培养基中,37℃过夜培养,然后然后通过提取构建好的目的tf的重组表达质粒,进行pcr鉴定并测序,挑选出与目标序列一致的单克隆菌,即得。
s2、tf体外表达预实验
(1)配制由3μl的
(2)制备1%的page胶,每个孔加1μl样品,于350ma的条件下电泳2h;
(3)用甲醇提前预处理8.5cm×4.0cm的pvdf膜30min,备用;
(4)将电泳完毕的凝胶去除浓缩胶后,放入膜转移buffer中,平衡5min;
(5)在胶黑膜白的前提下,放入转膜槽中转膜;
(6)小心将转移膜取出,正面朝上,加入适量5%脱脂奶粉的tbst溶液作为封闭液;
(7)将一抗anti-halotagmonoclonalantibody溶于封闭液,二者按1:1000混匀后,加在8.5cm×4.0cm的pvdf膜上,孵育60min;
(8)将二抗goatanti-mouseigg(h+l)hrpconjugate溶于封闭液,按体积比1:10000混匀后,加在8.5cm×4.0cm的pvdf膜上,孵育60min;
(9)将8.5cm×4.0cm的pvdf膜正面朝上,加入1ml的thermoscientificsupersignalwestpicopluschemiluminescentsubstrate作为荧光底物,于25℃的条件下反应5min,然后将pvdf膜放入暗盒,加入显影胶片;
(10)在暗室中曝光、显影、定影,将底片在自来水下冲洗干净后,烘干,保存,结果如图2所示,阳性对照组来检查整个实验流程和体系,通过蛋白marker来判定目的蛋白的表达。其中,样品obf1大小为50kda(含halotag标签蛋白),有目的条带;阳性对照组蛋白为64kda(含halotag标签蛋白),有目的条带。由此可知发现tf可以正常在体外表达。
s3、dap-seq建库:
(1)将dna用超声打断,然后用磁珠分选dna片段,得分选后的dna片段,dna分选结果如图3所示;
(2)在步骤(1)所得分选后的dna片段末端修复加a,同时加接头,得构建好的dna文库;
(3)体外表达带有halotag的目的蛋白,然后将该蛋白与磁珠结合,于25℃的条件下金属浴2h,得产物i;
(4)将步骤(3)所得产物i与步骤(2)所得构建好的dna文库混合,于25℃的条件下振荡1h,洗涤磁珠,洗脱样本,得洗脱液;
(5)将步骤(4)所得洗脱液进行pcr反应,获得pcr产物,用磁珠纯化,即可;
所述pcr反应中,配制10μl的pcr体系:affinipuredna1μl,2×kapahifi(kk2602)5μl,universalprimer(10μm)1μl,indexprimer(10μm)1μl,水2μl。其中,universal-primer引物是所有转录因子样品通用的,序列信息如seqidno.6所述:aatgatacggcgaccaccgagatctacactctttccctacacgacgctcttccgatct;(seqidno.6)而indexprimer的上游引物序列信息如seqidno.7所示,下游引物的序列信息如seqidno.8所示。
caagcagaagacggcatacgagatgcctaagtgactggagttcagacgtgtgctcttccgatct(seqidno.7);
caagcagaagacggcatacgagatttaggcgtgactggagttcagacgtgtgctcttccgatct(seqidno.8);
pcr扩增程序为:96℃,3min;96℃10s,56℃30s,72℃30s,这三步10个循环;72℃延伸2min;4℃保存。
将上述实施例所述方法制得的dap-seq文库进行质检,结果如图4所示,由图4可知,本申请方法制得的dna-seq文库质量极佳。
最后应当说明的是,以上实施例仅用以说明本发明的技术方案而非对本发明保护范围的限制,尽管参照较佳实施例对本发明做了详细说明,本领域的普通技术人员应当理解,可以对本发明的技术方案进行修改或者等同替换,而不脱离本发明技术方案的实质和范围。
- 还没有人留言评论。精彩留言会获得点赞!