包括CpG位点的发夹结构及测量CpG毗邻序列影响蛋白解离时间常数的单分子力学方法与流程
本发明属于生物物理领域,尤其是涉及一种包括cpg位点的发夹结构及测量cpg毗邻序列影响蛋白解离时间常数的单分子力学方法。
背景技术:
结合cpg位点的蛋白众多,譬如,tet1(ten-eleventranslocationmethylcytosinedioxygenase1,tet1)蛋白,又被称为cxxc6,是锌指结构蛋白之一。人tet1蛋白主要由6个结构域组成,其中cxxc结构域为锌指结构域,在辅因子锌离子的参与下,能够结合到cpg位点。tet1能够直接结合到基因的启动子区,催化5-甲基-胞嘧啶转变为5-羟甲基-胞嘧啶,募集组蛋白甲基转移酶结合到启动子区,促进基因的转录。但是cpg位点毗邻序列如何影响蛋白的底物亲和性仍然不清楚。虽然测量蛋白,譬如tet1,与不同毗邻序列包围的cpg结合位点的结合概率与解离时间常数极具挑战,但又极为重要。因此我们发明了含有多种毗邻序列包围的cpg位点的dna发夹结构,用于单分子力学方法高通量精密测量蛋白与dna的解离时间常数。
单分子技术作为生物物理方法,用于生命科学研究中实时观测单个分子动态事件,可以在纳米空间和毫秒时间尺度上精确测量单分子的三维位置、矢量、构象变化等各种动态过程。主流单分子技术有单分子光学、超分辨成像和单分子力学方法等。单分子力学方法使用高精度高灵敏度的力学操纵和检测仪器直接或间接对单分子进行主动测量,目前常用的方法有光镊、磁镊和原子力显微镜等。
单分子磁镊的基本原理依赖于控制磁场,通过磁球对目标分子施加外力。在工作池中,被测分子的一端连有磁球,另一端固定在玻璃表面。在磁场的作用下对磁球施加一个向上的作用力,从而使分子向上运动。通过倒置显微镜可以观察小球在力的作用下上升下降所形成的衍射光斑。通过建立查找表确定了衍射光斑与小球距焦平面之间距离的对应关系。由于小球的衍射光斑可被工作池下安装的倒置显微镜捕捉,因此通过小球衍射光斑的变化便可以计算小球在z轴的运动轨迹,从而实时精密探测单个分子的动态事件。
技术实现要素:
为解决上述技术问题,本发明提供一种包括cpg位点的发夹结构及测量cpg毗邻序列影响蛋白解离时间常数的单分子力学方法,具体是通过单分子磁镊精确探测一种蛋白与发夹结构茎干部分包含的多个结合位点的解离时间。
本发明采用的技术方案是:包括cpg位点的发夹结构,茎干部分包括多个等距分布的cpg位点,cpg位点为xcgy位点,其中x和y分别为c、g、a或t。
优选地,相邻的cpg位点通过随机at序列隔开,随机at序列长度为16-40bp;
优选地,随机at序列长度为18bp。
优选地,其中x和y分别为c和g、g和c、a和t或t和a;
优选地,全cpg位点组由ccgg、gcgc、acgt和tcga四个位点组成,茎干部分包括1-3组相同排列顺序的全cpg位点组。
优选地,包括生物素把手、地高辛把手、第一核酸片段、第二核酸片段、y字结构、发卡环结构和组成茎干部分的第三核酸片段和第四核酸片段;
第一核酸片段连接生物素把手和y字结构,第二核酸片段连接地高辛把手和y字结构,茎干部分连接发卡环结构和y字结构。
优选地,第三核酸片段由seqidno.13和seqidno.14退火形成,第四核酸片段由seqidno.15和seqidno.16退火形成。
包括cpg位点的发夹结构的制备方法,包括如下步骤:
pcr获得生物素把手及地高辛把手,并对制得的生物素把手及地高辛把手进行酶切;
退火反应制备y字结构、第一核酸片段、第二核酸片段、第三核酸片段、第四核酸片段和发卡环结构,并进行磷酸化;
将制得的生物素把手、地高辛把手、y字结构、第一核酸片段、第二核酸片段、第三核酸片段、第四核酸片段和发卡环结构连接得到分子尺发夹结构。
优选地,生物素把手、第一核酸片段、y字结构、第二核酸片段、地高辛把手、第三核酸片段、第四核酸片段和发卡环结构添加摩尔比例为1-9:1-3:1:1-3:1-9:1-3:1-9:1-27;
优选地,摩尔比例为6:2:1:2:6:2:4:20。
采用包括cpg位点的发夹结构测量cpg毗邻序列影响蛋白解离时间常数的单分子力学方法,包括如下步骤:
构建含有不同xcgy位点的发夹结构作为测试组;
构建含有相同xcgy位点的发夹结构作为对照组;
分别对测试组和对照组通过单分子磁镊对发夹结构施加外力,收集发夹结构在外力的作用下长度变化数据;
计算蛋白在不同结合位点的解离时间常数,对比分析测试组与对照组中相应cpg位点上蛋白解离常数之间的差异。
优选地,单分子磁镊施加外力的力学操控模式为:低力6皮牛维持55秒,测试力14.75皮牛维持10秒,高力30皮牛维持5秒,测试力14.75皮牛维持5秒,低力6皮牛维持5秒,力跳循环数范围可控制为1-100次;
优选地,采取力跳循环50次;
优选地,蛋白工作浓度优选地使用1-3μm,包括cpg位点的发夹结构工作用量优选地使用0.5-1.5ng;
优选地,蛋白工作浓度优选地使用3μm,包括cpg位点的发夹结构工作用量优选地使用0.5ng。
优选地,对照组含有与所述测试组数量相同的ccgg位点。
本发明具有的优点和积极效果是:本方案数据收集快捷,得益于单分子高通量检测方法,可在一次试验中检测到大于1000次的蛋白-dna相互作用事件,允许进行精确地统计分析,提高分析数据的准确性。
附图说明
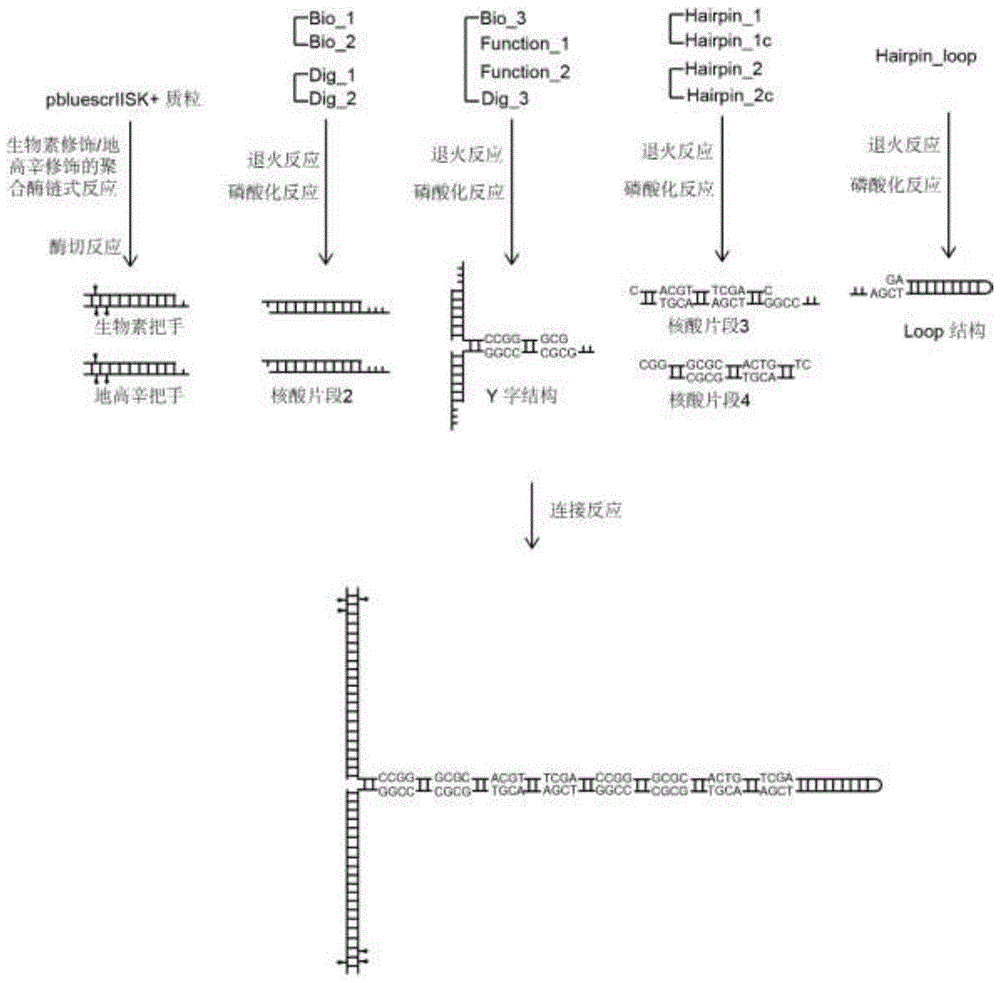
图1是合成包括cpg位点的发夹结构流程图;
图2是合成包括cpg位点的发夹结构凝胶电泳结果图;
图3实施例中包括cpg位点的发夹结构茎干部分蛋白在不同cpg结合位点的排布;
图4力跳实验中发夹结构的长度随时间的变化;
图5对211次力跳实验中蛋白从每一个cpg毗邻序列解离所用的时间;(a)对照组中蛋白从8个相同的ccgg位点解离时间统计图。(b)测试组中蛋白从上下游不同的cpg毗邻序列解离时间统计图。
图6对445次力跳实验中蛋白与不同cpg结合位点结合的可能性;
图7通过单个指数衰减函数对蛋白与不同cpg位点结合所产生的力跳实验数据进行拟合得到解离时间曲线;
图8是包括cpg位点的发夹结构结构图。
具体实施方式
下面结合附图对本发明的实施例做出说明。本发明涉及一种测量蛋白从dna上解离的时间常数的方法,特别是涉及一种基于单分子磁镊精确测量cpg毗邻序列影响蛋白解离时间常数的方法,具体是通过单分子磁镊精确探测一种蛋白与发夹结构茎干部分包含的多个结合位点的解离时间,涉及生物物理、分析化学和精密仪器测量技术领域。其中,未具体说明操作步骤的实验方法,均按照相应商品说明书进行,实施例中所用到的仪器、试剂、耗材如无特殊说明,均可从商业公司购买得到。
例如采用tet1的cxxc结构域与发夹结构中的多个cpg位点同时结合,再通过单分子磁镊施加外力,实时监测dna发夹结构的长度变化。在dna发夹结构打开的过程中,cxxc结构域与cpg位点的结合位置会出现停顿信号。运用数据处理软件分析dna发夹结构停顿信号的精确时间,解析cxxc结构域从每个cpg结合位点解离的时间常数。通过与对照dna进行对比分析,可以衡量毗邻序列如何影响cxxc结构域从cpg位点解离。
本方案设计一种茎干部分包括多个等距分布的cpg位点的发夹结构,其中各个cpg位点为形如x-cg-y的位点,xy为cpg位点的毗邻序列,其中x和y分别为c、g、a或t。相邻的x-cg-y位点通过随机at序列隔开,每个cg位点之间的at序列不能完全相同,防止错配,随机at序列长度为16-40bp,随机at序列长度优选为18bp,序列长度过短柱形图的高斯模型拟合基线无法分离,序列长度过长发夹结构合成困难。根据检测蛋白的不同,可设计不同排列组合的cpg位点,可采用茎干包括2种或2种以上类型的cpg位点的发夹结构。一般检测时可采用包括全cpg位点组的发夹结构,全cpg位点组由ccgg、gcgc、acgt和tcga四个位点组成,茎干部分包括1-3组相同排列顺序的全cpg位点组,这样的重复结构也能够进一步验证结合位点解离的时间常数。本方案以包括2个全cpg位点组的发夹结构为测试组,以包括8个ccgg位点的发夹结构为对照组为例,对本方法进一步说明,其他组合方案仅需要设计相应的核酸序列,或使用能够与cpg位点同时结合的其他蛋白均属于本方案涵盖范围。
采用包括cpg位点的发夹结构测量cpg毗邻序列影响蛋白解离时间常数的单分子力学方法包括如下步骤:
(1)构建含有不同xcgy位点的发夹结构作为测试组;
(2)构建含有相同xcgy位点的发夹结构作为对照组;
(3)分别对测试组和对照组通过单分子磁镊对发夹结构施加外力,收集发夹结构在外力的作用下长度变化数据;
(4)计算蛋白在不同结合位点的解离时间常数,对比分析测试组与对照组中相应cpg位点上蛋白解离常数之间的差异。
测试组包括多个不同的cpg毗邻序列,对照组包括数量相同的ccgg序列,测试组与对照组除了ccgg和cpg的序列不同,其他均相同,通过单分子磁镊施加外力对比测试组中的cpg序列和对照组中同一位置的ccgg的解离时间和结合概率差异。
发夹结构包括生物素把手、地高辛把手、第一核酸片段、第二核酸片段、y字结构、发卡环结构和组成茎干部分的第三核酸片段和第四核酸片段;如图1所示,分别制备各个片段,再利用连接反应将上述片段连接再一起形成分子尺发夹结构。第一核酸片段连接生物素把手和y字结构,第二核酸片段连接地高辛把手和y字结构,茎干部分连接发夹环结构和y字结构。第三核酸片段和第四核酸片段连接构成茎干部分,茎干部分含有多个等距的cpg位点,各个cpg位点由随机at序列隔开,at序列长度可选择16-40bp,当at序列长度为18bp时效果最为准确。
生物素把手及地高辛把手是通过在聚合酶链式反应体系中加入生物素修饰的尿嘧啶脱氧核糖核苷酸或地高辛修饰的尿嘧啶脱氧核糖核苷酸获得的多修饰dna片段;生物素把手通过如seqidno.1和seqidno.2所示上下游引物pcr获得,地高辛把手通过如seqidno.3和seqidno.4所示上下游引物pcr获得,生物素把手及地高辛把手均为多位点修饰,将获得的片段通过酶切形成粘性接头备用。
seqidno.1gaccgagatagggttgagtg
seqidno.2gcatcggctgaggaaagggaacaaaagctgg
seqidno.3atcgtagggtcctgaccgagatagggttgagtg
seqidno.4aaagggaacaaaagctgg
第一核酸片段、第二核酸片段、第三核酸片段、第四核酸片段、y字结构和发卡环结构均由相应的单链片段通过碱基互补配对得到。其中y字结构通过seqidno.9、seqidno.10、seqidno.11和seqidno.12退火形成,第一核酸片段通过seqidno.5和seqidno.6退火形成,第二核酸片段通过seqidno.7和seqidno.8退火形成,发夹环结构由seqidno.17退火形成;测试组发夹结构的第三核酸片段和第四核酸片段可根据需要进行设计,现以茎干部分包括两组ccgg、gcgc、acgt和tcga位点为例,测试组第三核酸片段通过seqidno.13和seqidno.14退火形成,测试组第四核酸片段通过seqidno.15和seqidno.16退火形成;对照组发夹结构茎干部分仅包括一种cpg位点,相对于前述测试组,设计对照组包括8个ccgg位点,对照组第三核酸片段通过seqidno.18和seqidno.19退火形成,对照组第四核酸片段通过seqidno.20和seqidno.21退火形成;将退火形成的各个片段磷酸化备用。
seqidno.5
tcagcaaggaaggagattttgaaaaatttatttattagatattggaaatattattagag
seqidno.6
aaatcatctcctctaataatatttccaatatctaataaataaatttttcaaaatctccttccttgc
seqidno.7
aatgatgagtgttaaaaaaagtggggaagtgagtaatgaaattattttgtatgttttttatatgaatttattttttggg
seqidno.8
gaccccaaaaaataaattcatataaaaaacatacaaaataatttcattactcacttccccactttttttaac
seqidno.9
cccttttattataccattcttcatattttttc
seqidno.10
actcatcattcaaacaaacaaaaaccacatctactccc
seqidno.11
gagatgatttgaaaaaatatgaagaatggtataataaaagggtgatttatatttatttattccggtatttaatttaattatatgcg
seqidno.12
aatatataagcgcatataattaaattaaataccggaataaataaatataaatctgggagtagatgtggtttttgtttgtttg
seqidno.13
cttatatatttatatttatacgttttatttattatttattttcgattatttataatttaattac
seqidno.14
ataataaccggtaattaaattataaataatcgaaaataaataataaataaaacgtataaatata
seqidno.15
cggttattatatattatttatgcgctatttatttaattatattacgtttatttatatatttatattc
seqidno.16
atataaataaacgtaatataattaaataaatagcgcataaataatat
seqidno.17
gattaatatttattatatttttttaaatataataaatattaatcgaatataaat
seqidno.18
gttatatatttatatttatccggtttatttattatttatttccggttatttataatttaattac
seqidno.19
ataataaccggtaattaaattataaataaccggaaataaataataaataaaccggataaatata
seqidno.20
cggttattatatattatttatccggtatttatttaattatattccggttatttatatatttatatcc
seqidno.21
atataaataaccggaatataattaaataaataccggataaataatat
将上述制备合成的生物素把手、第一核酸片段、y字结构、第二核酸片段、地高辛把手、第三核酸片段、第四核酸片段和发夹环结构按照摩尔比例为1-9:1-3:1:1-3:1-9:1-3:1-9:1-27混合,优选地,按照6:2:1:2:6:2:4:20混合连接,连接酶优选地选择t4连接酶,连接反应为循环温度梯度连接,单次温度梯度为16℃连接1小时,20℃连接1小时,25℃连接1小时,循环数为1-5次,优选地采取循环数为3次,连接得到发夹结构,分别对测试组和对照组进行连接。发夹结构可通过地高辛—地高辛抗体间相互作用固定于工作池玻璃表面,通过生物素—链霉素亲和结合与超顺磁小球连接。
单分子磁镊实验中,单次力跳实验的力学操控模式为:低力6pn维持55s,测试力14.75pn维持10s,高力30pn维持5s,测试力14.75pn维持5s,低力6pn维持5s,力跳循环数范围可控制为1-100次,优选为50次。蛋白工作浓度优选地使用3μm,dna发夹结构工作用量优选地使用0.5ng。发夹结构在外力的作用下茎干部分的双链会打开变为单链,因此发夹结构的长度会发生变化。如果有蛋白与cpg位点结合,发夹结构打开会受阻,出现停顿信号。通过单分子磁镊捕捉发夹结构在外力作用下拉伸的过程中由于蛋白的结合而导致的停顿信号所用的时间,计算蛋白在不同结合位点的解离时间常数,统计与不同位点的结合概率,对比分析不同毗邻序列对蛋白与cpg位点相互作用的影响。运用数据处理软件分析dna发夹结构停顿信号的精确时间,解析cxxc结构域从每个cpg结合位点解离的时间常数。通过与对照dna进行对比分析,可以衡量在相同的位置毗邻序列如何影响cxxc结构域从cpg位点解离。
测试用蛋白可以为tet1,tet3,mll1的cxxc结构域及对应的全蛋白,现以tet1cxxc结构域蛋白为例,通过具体实施例对本方案进一步说明。
实施例1:测试组包括cpg位点的发夹结构的构建
按照如图1所示流程制备发夹结构。
1生物素把手及地高辛把手制备
1.1生物素修饰/地高辛修饰的聚合酶链式反应
通过对pbluescriisk+质粒进行生物素修饰/地高辛修饰的聚合酶链式反应,在反应管中加入:
1μl浓度为10μm的生物素/地高辛把手上游引物;
1μl浓度为10μm的生物素/地高辛把手下游引物(引物均购自金唯智公司);
1μl浓度为10mm的datp(货号:4026q,宝日医生物技术公司);
1μl浓度为10mm的dgtp(货号:4027q,宝日医生物技术公司);
1μl浓度为10mm的dctp(货号:4028q,宝日医生物技术公司);
0.9μl浓度为10mm的dttp(货号:4029q,宝日医生物技术公司);
1μl浓度为1mm的生物素修饰的尿嘧啶脱氧核糖核苷酸(货号:11093070910,罗氏公司);
1μl浓度为1mm的地高辛修饰的尿嘧啶脱氧核糖核苷酸(货号:11093088910,罗氏公司);
1μl浓度为5ng/μl的pbluescriisk+质粒(购自生工生物工程公司);
0.25μltaqdna聚合酶(货号:ep0402,赛默飞世尔公司);
3μl25mmmgcl2溶液;
5μl10×taqkcl缓冲液;
33.85μl纯水。
聚合酶链式反应温控循环为:95℃3分钟,95℃30秒,55℃30秒,72℃50秒,循环40次,72℃5分钟,终止在12℃。
使用cyclepure试剂盒(货号:d6492-02,omega公司)进行产物纯化,所得生物素及地高辛把手片段长度为666bp。
1.2酶切制备生物素把手和地高辛把手
生物素把手酶切反应配方为:
56μl第一步所得生物素把手片段(125.0ng/μl);
18μl10×cutsmart缓冲液;
17.5μlbbvci内切酶(货号:r0601s,neb公司);
纯水88.5μl。
混匀后均分至四个反应管。
地高辛把手酶切反应配方为:
44μl第一步所得生物素把手片段(134.2ng/μl);
13.5μl10×cutsmart缓冲液;
3.5μlppumi内切酶(货号:r0506l,neb公司);
纯水74μl。
混匀后均分至三个反应管。
将上述反应管在37℃反应12小时。使用cycle-pure试剂盒进行产物纯化即得生物素把手及地高辛把手。
2构建y字结构、第一核酸片段、第二核酸片段、第三核酸片段、第四核酸片段和发夹环结构
2.1退火反应
第一核酸片段退火反应体系:
8μl浓度为50μm序列如seqidno.5所示的单链dna片段;
8μl浓度为50μm序列如seqidno.6所示的单链dna片段;
4μl的5×退火缓冲液使总体积为20μl。
第二核酸片段退火反应体系:
8μl浓度为50μm序列如seqidno.7所示的单链dna片段;
8μl浓度为50μm序列如seqidno.8所示的单链dna片段;
4μl的5×退火缓冲液使总体积为20μl。
y字结构退火反应体系:
2μl浓度为50μm序列如seqidno.9所示的单链dna片段;
2μl浓度为50μm序列如seqidno.10所示的单链dna片段;
2μl浓度为50μm序列如seqidno.11所示的单链dna片段;
2μl浓度为50μm序列如seqidno.12所示的单链dna片段;
2μl的5×退火缓冲液使总体积为10μl。
测试组第三核酸片段退火反应体系:
8μl浓度为20μm序列如seqidno.13所示的单链dna片段;
8μl浓度为20μm序列如seqidno.14所示的单链dna片段;
4μl的5×退火缓冲液使总体积为20μl。
测试组第四核酸片段退火反应体系:
8μl浓度为20μm序列如seqidno.15所示的单链dna片段;
8μl浓度为20μm序列如seqidno.16所示的单链dna片段;
4μl的5×退火缓冲液使总体积为20μl。
发卡环结构退火反应体系:
20μl浓度为50μm序列如seqidno.17所示的单链dna片段;
5μl的5×退火缓冲液;
加纯水补足到40μl。
以上退火反应温控程序为:98℃3分钟,以-0.1℃/8秒的降温速率梯度降温至25℃,25℃3分钟,终止在12℃。
2.2磷酸化反应
y字结构磷酸化反应体系:
1μly字结构退火产物;
2μl10×t4dna连接反应缓冲液;
2μlt4磷酸激酶(货号:2021a,宝日医生物技术公司);
15μl纯水。
第一核酸片段、第二核酸片段、测试组第三核酸片段、测试组第四核酸片段和发卡环结构的磷酸化反应体系为:
0.5μl对应的退火产物;
2μl10×t4dna连接反应缓冲液;
2μlt4磷酸激酶;
15.5μl纯水。
上述磷酸化反应体系磷酸化温控程序为:37℃90分钟,70℃5分钟,以-0.1℃/8秒的降温速率梯度降温至25℃,25℃3分钟,终止在12℃。
3连接测试组发夹结构
连接反应体系为:
13.6μl生物素把手(3pmole);
17.6μl地高辛把手(3pmole);
1μly字结构(0.5pmole);
2μl第一核酸片段(1pmole);
2μl第二核酸片段(1pmole);
2μl测试组第三核酸片段(1pmole);
4μl测试组第四核酸片段(2pmole);
20μl发卡环结构(20pmole);
8μl10×t4dna连接反应缓冲液;
6μlt4dna连接酶(货号:m0202s,neb公司),3.8μl纯水。
连接反应温控程序为:16℃1小时,20℃1小时,25℃1小时,循环3次,最终停止在12℃。
如图2所示,是合成dna发夹结构凝胶电泳结果图。凝胶电泳条件为1%的琼脂糖凝胶,电压100v,时间30分钟;条带1为8000bpdna标记物,条带2为dna发夹结构连接产物混合物,在2000bp左右对应的条带为dna发夹结构,其余条带均对应副产物。经1%琼脂糖电泳分离切胶后,使用gel-extraction试剂盒(货号:d2500-02,omega公司)回收dna发夹结构对应条带,至此得到磁镊所用测试组发夹结构。茎干部分蛋白在不同cpg结合位点的排布如图3所示,如图3所示,测试组发夹结构茎干部分蛋白按序排布,发夹结构如图8所示。
实施例2:对照组包括cpg位点的发夹结构的构建
与测试组cpg位点的发夹结构的构建比较,对照组需另外合成对照组第三核酸片段和对照组第四核酸片段,
对照组第三核酸片段退火反应体系为:
8μl50μm序列如seqidno.18所示核酸链;
8μl50μm序列如seqidno.19所示核酸链;
4μl5×dna退火缓冲液。
对照组第四核酸片段退火反应体系为:
8μl50μm序列如seqidno.20所示核酸链;
8μl50μm序列如seqidno.21所示核酸链;
4μl5×dna退火缓冲液。
退火反应温控程序为:98℃3分钟,以-0.1℃/8秒的降温速率梯度降温至25℃,25℃3分钟,终止在12℃。
另外对照组发夹结构的连接,连接体系如下:
24μl生物素把手(3pmole);
15.3μl地高辛把手(3pmole);
1μly字结构(0.5pmole);
2μl第一核酸片段(1pmole);
2μl第二核酸片段(1pmole);
2μl对照组第三核酸片段(1pmole);
4μl对照组第四核酸片段(1pmole);
10μl发夹环结构(10pmole);
7.5μl10×t4dna连接反应缓冲液;
6μlt4dna连接酶(货号:m0202s,neb公司);
1.2μl纯水。
连接反应温控程序为:16℃1小时,20℃1小时,25℃1小时,循环3次,最终停止在12℃。经1%琼脂糖电泳分离切胶后,使用gel-extraction试剂盒(货号:d2500-02,omega公司)回收对照组发夹结构对应条带
实施例3:使用单分子磁镊测发夹结构在受外力作用的长度变化
制作反应池:反应池由不带孔的载玻片与带孔的盖玻片组成。
1)盖玻片打孔:取60mm×24mm的玻片,用tc-169多功能打磨雕切机打两个孔,作为进水口与出水口。
2)玻片清洗:将盖玻片与载玻片竖直放置于抗体孵育盒中,加入含有清洗剂的超纯水,水浴超声30分钟,用纯水冲洗3次;加入异丙醇,水浴超声30分钟,用纯水冲洗3次;加入异丙醇,水浴超声30分钟,用纯水冲洗3次;加入纯水,水浴超声30分钟,用纯水冲洗3次;最后加入75%乙醇浸泡数小时后用n2吹干。
3)敷聚苯乙烯小球:将清洗后的聚苯乙烯小球按1:7与0.1%硝酸纤维素混合,取8μl涂于载玻片表面,100℃加热3分钟。
4)制作完整的反应池:在载玻片与盖玻片之间夹上提前切好反应孔道的双层封口膜,85℃加热4分钟使两层玻片粘合。
5)孵抗地高辛抗体:取70μl浓度为0.1mg/ml抗地高辛抗体(货号:11214667001,西格玛奥德里奇中国公司)注入反应池中于25℃孵育2小时,用工作缓冲液冲洗5次后注入70μl浓度为5mg/ml的牛血清白蛋白溶液封闭过夜。
反应体系的构建:取20μl链霉亲和素修饰的超顺磁小球(m270,货号:65305,invitrogen公司),清洗后加入0.5ng发夹结构在冰上静置10分钟,再将其注入反应池中孵育10分钟。此时发夹结构通过一端的生物素与链霉亲和素修饰的超顺磁小球相连,通过另一端的地高辛与反应池的抗地高辛抗体相连使其固定于反应池表面。
通过操控磁铁位置对超顺磁小球施加磁场,从而主动对dna发夹施加外力,力的范围可达到0.1–150皮牛。磁镊装置力学操控模式为:低力6皮牛维持55秒,测试力14.75皮牛维持10秒,高力30皮牛维持5秒,测试力14.75皮牛维持5秒,低力6皮牛维持5秒,循环次数50次。dna发夹结构工作用量为0.5ng,tet1cxxc结构域蛋白工作浓度为3μm,照相机采样频率为200hz,可实时采集磁珠空间位置,从而得到发夹结构力、时间、轨迹的实时数据,利用数据处理软件计算可得到结构长度时间的关系。
如图4所示,左侧展示的是在力跳实验中,发夹结构的长度随时间的变化;右侧展示的是对左图中方框的内容的放大,其中的虚线为蛋白与不同cpg位点结合后在外力下打开所产生停顿信号时发夹结构对应的长度。图5对211次力跳实验所产生的数据进行数理模型拟合,得到蛋白从8个ccgg位点以及8个不同的cpg毗邻序列解离需要的时间,通过对比蛋白从对照组中同一位置的解离时间,可以得知测试组中每个cpg位点与ccgg的解离时间常数差异;图6对445次力跳实验所产生的数据进行数理模型拟合,得到蛋白与8个ccgg位点以及8个不同的cpg毗邻序列结合的概率,通过对比蛋白从对照组中同一位置的结合概率,可以对比蛋白与特定cpg位点的结合概率和蛋白与对照组中同一位置ccgg的结合概率的差异;如图7所示是通过单个指数衰减函数对蛋白与不同cpg位点结合所产生的力跳实验数据进行拟合得到解离时间曲线,显示了取对数和未取对数的时间分布。产生的时间常数在底部列出,并带有标准误差。
以上对本发明的实施例进行了详细说明,但所述内容仅为本发明的较佳实施例,不能被认为用于限定本发明的实施范围。凡依本发明申请范围所作的均等变化与改进等,均应仍归属于本发明的专利涵盖范围之内。
序列表
<110>南开大学
<120>包括cpg位点的发夹结构及测量cpg毗邻序列影响蛋白解离时间常数的单分子力学方法
<130>2020
<160>21
<170>siposequencelisting1.0
<210>1
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>1
gaccgagatagggttgagtg20
<210>2
<211>31
<212>dna
<213>人工序列(artificialsequence)
<400>2
gcatcggctgaggaaagggaacaaaagctgg31
<210>3
<211>33
<212>dna
<213>人工序列(artificialsequence)
<400>3
atcgtagggtcctgaccgagatagggttgagtg33
<210>4
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>4
aaagggaacaaaagctgg18
<210>5
<211>59
<212>dna
<213>人工序列(artificialsequence)
<400>5
tcagcaaggaaggagattttgaaaaatttatttattagatattggaaatattattagag59
<210>6
<211>66
<212>dna
<213>人工序列(artificialsequence)
<400>6
aaatcatctcctctaataatatttccaatatctaataaataaatttttcaaaatctcctt60
ccttgc66
<210>7
<211>79
<212>dna
<213>人工序列(artificialsequence)
<400>7
aatgatgagtgttaaaaaaagtggggaagtgagtaatgaaattattttgtatgtttttta60
tatgaatttattttttggg79
<210>8
<211>72
<212>dna
<213>人工序列(artificialsequence)
<400>8
gaccccaaaaaataaattcatataaaaaacatacaaaataatttcattactcacttcccc60
actttttttaac72
<210>9
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>9
cccttttattataccattcttcatattttttc32
<210>10
<211>38
<212>dna
<213>人工序列(artificialsequence)
<400>10
actcatcattcaaacaaacaaaaaccacatctactccc38
<210>11
<211>86
<212>dna
<213>人工序列(artificialsequence)
<400>11
gagatgatttgaaaaaatatgaagaatggtataataaaagggtgatttatatttatttat60
tccggtatttaatttaattatatgcg86
<210>12
<211>82
<212>dna
<213>人工序列(artificialsequence)
<400>12
aatatataagcgcatataattaaattaaataccggaataaataaatataaatctgggagt60
agatgtggtttttgtttgtttg82
<210>13
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>13
cttatatatttatatttatacgttttatttattatttattttcgattatttataatttaa60
ttac64
<210>14
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>14
ataataaccggtaattaaattataaataatcgaaaataaataataaataaaacgtataaa60
tata64
<210>15
<211>67
<212>dna
<213>人工序列(artificialsequence)
<400>15
cggttattatatattatttatgcgctatttatttaattatattacgtttatttatatatt60
tatattc67
<210>16
<211>47
<212>dna
<213>人工序列(artificialsequence)
<400>16
atataaataaacgtaatataattaaataaatagcgcataaataatat47
<210>17
<211>54
<212>dna
<213>人工序列(artificialsequence)
<400>17
gattaatatttattatatttttttaaatataataaatattaatcgaatataaat54
<210>18
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>18
gttatatatttatatttatccggtttatttattatttatttccggttatttataatttaa60
ttac64
<210>19
<211>64
<212>dna
<213>人工序列(artificialsequence)
<400>19
ataataaccggtaattaaattataaataaccggaaataaataataaataaaccggataaa60
tata64
<210>20
<211>67
<212>dna
<213>人工序列(artificialsequence)
<400>20
cggttattatatattatttatccggtatttatttaattatattccggttatttatatatt60
tatatcc67
<210>21
<211>47
<212>dna
<213>人工序列(artificialsequence)
<400>21
atataaataaccggaatataattaaataaataccggataaataatat47
- 还没有人留言评论。精彩留言会获得点赞!