一种用于新冠病毒核酸快速检测的Cpf1试剂盒及其制备方法与应用与流程
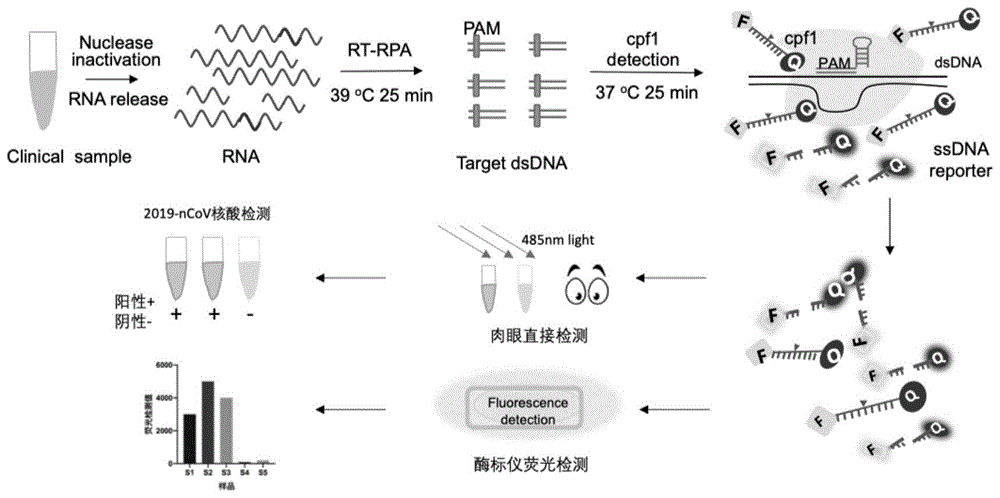
本发明涉及一种对新冠肺炎病毒核酸的快速检测方法及试剂盒,更具体的说,涉及基于cpf1荧光法检测对新冠病毒sars-cov-2病毒核酸的快速诊断方法及其试剂盒,属于生物技术领域。
背景技术:
国际病毒分类委员会(ictv)将新型冠状病毒(2019-ncov)的正式分类名为严重急性呼吸综合征冠状病毒2(severeacuterespiratorysyndromecoronavirus2,sars-cov-2)。世界卫生组织(who)宣布,由这一病毒导致的疾病的正式名称为covid-19。
目前,对于新冠肺炎的诊断主要有病毒核酸分子检测和胸部影像学检测两种。胸部影像学检测,主要依赖于胸肺部ct成像结果进行判断:早期呈现多发的小斑片影及间质改变,以肺外带明显,进而发展为双肺多发磨玻璃影、浸润影。严重者出现肺实变,甚至‘白肺’,胸腔积液少见。但是,胸肺部ct检测,需要依赖于专业。在分子生物学的手段上,目前常用的办法有两种:病毒的基因测序和核酸检测。二代测序因为可以检测微量物质,但是大多数医院没有测序的设备和生物信息团队来协助,并且成本高检测周期也比较长,不适合做筛查。
现在的诊断方法依赖病毒核酸检测:通过实时反转录聚合酶链式反应(realtimereversetranscriptionpcr,rrt-pcr)鉴定。每个病例采集上、下呼吸道标本,如支气管或肺泡灌洗液、深咳痰液,同时采集发病初期和发病14天后的血清。目前在符合疑似病例标准的基础上,对痰液、咽拭子、下呼吸道分泌物等标本进行rrt-pcr检测显示sars-cov-2核酸阳性,即可确诊sars-cov-2感染。rrt-pcr检测病毒核酸主要受制于:试剂盒质量和能够承担临床检测的p3实验室数量、p3实验室设备的准确性和稳定性、能进行熟练稳定操作的工作人员人数、需要一些昂贵的实验设备、并且检测时间较长。
目前,尚没有针对sars-cov-2感染有效治疗手段或疫苗,疾病控制主要依赖与严格的物理隔离切断传播途径,从而直接造成严重的经济损失。鉴于sars-cov-2传播途径广泛和传播速度快,迫切需要病毒的快速诊断,这对于预防和确诊该病非常重要。sars-cov-2准确快速的检测,使得防疫检疫决策可以当机立断、立即处置,达到阻断扩散风险的目的。
crispr-cas(clusteredregularlyinterspacedshortpalindromicrepeats,crisprs)是细菌中一种适应性免疫系统,cas蛋白通过rna引导的核酸酶靶向降解外来核酸[1,2]。其中,crispr-cas12a(cpf1)属于cas酶第二家族,用来引导rna指导双链dna裂解单个ruvc催化结构域。cpf1酶识别富含胸腺嘧啶(thymine,t)核苷酸的间隔区相邻基序(pam)[3],催化它们自身的指导crisprrna(crrna)成熟[4],并特异性识别和切割互补配对的双链dna(dsdna)[3]。当crispr/cpf1蛋白以序列特异性方式识别切割靶双链dna时,能诱导强大的非特异性单链dna(ssdna)反式切割活性[5]。基于cpf1上述特性,我们开发了一种快速准确的检测方法,用于检测临床标本中sars-cov-2病毒核酸。首先,对临床样品进行病毒灭活处理,释放待检样品中sars-cov-2核酸。在等温条件下,对病毒rna进行反转录(rt)获得dna,并进行重组酶聚合酶扩增(rpa)[6]。cpf1-crrna复合物结合并切割靶标dsdna,其激活ssdna的反式切割。与ssdna偶联的荧光报告分子在切割时产生荧光信号。这种称为dna核酸内切酶靶向crispr反式报告基因的新方法,为快速准确检测新冠肺炎病毒提供了强有力的平台。
参考文献
1.r,b.,etal.,crisprprovidesacquiredresistanceagainstvirusesinprokaryotes.science(newyork,n.y.),2007.315(5819):p.1709-12.
2.la,m.ands.ej,crisprinterferencelimitshorizontalgenetransferinstaphylococcibytargetingdna.science(newyork,n.y.),2008.322(5909):p.1843-5.
3.b,z.,etal.,cpf1isasinglerna-guidedendonucleaseofaclass2crispr-cassystem.cell,2015.163(3):p.759-71.
4.i,f.,etal.,thecrispr-associateddna-cleavingenzymecpf1alsoprocessesprecursorcrisprrna.nature,2016.532(7600):p.517-21.
5.js,c.,etal.,crispr-cas12atargetbindingunleashesindiscriminatesingle-strandeddnaseactivity.science(newyork,n.y.),2018.360(6387):p.436-439.
6.o,p.,etal.,dnadetectionusingrecombinationproteins.plosbiology,2006.4(7):p.e204.
技术实现要素:
本发明的目的在于针对临床新冠病毒sars-cov-2的快速检测难题,提供一种灵敏度高、特异性强、快速可视化的用于新冠病毒sars-cov-2核酸快速检测的cpf1试剂盒及其检测方法。
为了达到上述目的,本发明提供了一种用于新冠病毒sars-cov-2核酸快速检测的cpf1试剂盒,其特征在于,包括适用于新冠病毒sars-cov-2快速检测的cpf1的荧光检测体系;所述cpf1荧光检测体系包括:针对新冠病毒sars-cov-2基因orf1a、orf1b、n和e检测区段的特异性crrna、cpf1蛋白和单链dna(ssdna)报告系统;
所述特异性crrna为:针对orf1a基因的orf1a-crrna1到orf1a-crrna2,针对orf1b基因的orf1b-crrna1到orf1b-crrna3,针对n基因的n-crrna1到n-crrna2和针对e基因的e-crrna1到e-crrna2中的任意一种或多种,其序列为seqno.1到seqno.9;所述单链dna(ssdna)报告系统包括用于酶标仪荧光检测或荧光灯下肉眼直接判读的ssdnafqreporter;所述ssdnafqreporter为利用6-羧基荧光素(6-fam)和荧光淬灭剂(bhq1)标记的ssdna,标记产物如下:/56fam/tttattt/3bhq1/,命名为ssdnafqreporter/56fam/tttattt/3bhq1/。
优选地,所述特异性crrna为:等比例混合的orf1a-crrna1和orf1a-crrna2,命名为orf1a-crrnamix;或等比例混合的orf1b-crrna1、orf1b-crrna2和orf1b-crrna3,命名为orf1b-crrnamix;或等比例混合的n-crrna1和n-crrna2,命名为n-crrnamix;或等比例混合的e-crrna1和e-crrna2,命名为e-crrnamix。
本发明还提供了上述用于新冠病毒sars-cov-2核酸快速检测的cpf1试剂盒的制备方法,其特征在于,所述特异性crrna的制备方法包括:针对sars-cov-2病毒核酸orf1a、orf1b、n和e基因,其序列为seqno.10到seqno.13,寻找包含cpf1识别序列(pam)tttn的靶向序列,设计23bp长度的crrna;设计完成后,制备crrna;
所述cpf1蛋白的制备方法包括:针对cpf1蛋白核酸序列进行原核密码子优化获得序列seqno.14,构建到pet28a表达载体,进行低温诱导可溶蛋白表达,通过亲和纯化和分子筛纯化获得目的蛋白;
所述cpf1蛋白的制备方法包括:针对cpf1蛋白核酸序列进行原核密码子优化获得序列seqno.14,构建到pet28a表达载体,进行低温诱导可溶蛋白表达,通过亲和纯化和分子筛纯化获得目的蛋白。
优选地,所述crrna的制备包括构建载体puc57-t7-crrna,通过体外转录获得目的crrna;或直接合成crrna序列对应的rna。
本发明提供的用于新冠病毒sars-cov-2核酸快速检测的cpf1试剂盒既可利用酶标仪荧光检测也可以利用荧光肉眼检测。本发明中ssdnafqreporter,当使用酶标仪荧光检测时,设定检测激发光为485nm-520nm;当使用肉眼直接检测时,选用可产生485nm波长光源的发光器进行检测。
在使用荧光检测时,当cpf1检测体系中存在sars-cov-2病毒核酸,会由在sars-cov-2特异性crrna介导下特异性激活cpf1蛋白的核酸内切酶活性。激活后的cpf1蛋白会切割由荧光基团和淬灭基团标记的ssdnafqreporter,从而释放激活荧光基团,使用酶标仪可以检测到荧光读数或肉眼可见绿色反应。相对应的,待检测样品中不存在sars-cov-2病毒核酸时,不会有荧光读数或肉眼不可见绿色反应。
本发明还提供了一种新冠病毒sars-cov-2核酸快速检测方法,其特征在于,采用上述用于新冠病毒sars-cov-2核酸快速检测的cpf1试剂盒。
优选地,所述新冠病毒sars-cov-2核酸快速检测方法包括以下步骤:
步骤a:利用核酸快速释放试剂对样品病毒灭活和释放待测样品中核酸;
步骤b:利用等温扩增引物扩增待测样品中核酸:将步骤a获得产物,利用sars-cov-2病毒核酸orf1a、orf1b、n和e基因片段的特异性引物seqno.15至seqno.22,加入rt-rpa等温扩增体系,在37℃反应25min扩增获得特异性产物;
步骤c:利用cpf1检测体系识别切割sars-cov-2病毒核酸orf1a、orf1b、n和e基因片段时:将步骤b中产物加入cpf1检测体系,于37℃反应25min;
步骤d:利用荧光酶标仪或荧光灯肉眼直接检测判读待检测样品中是否存在sars-cov-2病毒核酸。
与现有技术相比,本发明的有益效果在于:
(1)本发明通过利用cpf1特异识别核酸荧光检测技术,实现对sars-cov-2核酸的高灵敏度、高特异性及快速可视化检测。基因生物信息学分析,将sars-cov-2病毒序列与ncbi数据库中sars和bat-relatedsars病毒序列,设计针对orf1a、orf1b、n和e基因片段差异性crrna。为保证检测特异性,对设计的crrna序列检索ncbi核酸数据库中包括人、动物、植物和微生物等基因,确定没有高同源性匹配。根据cpf1识别特定pam序列特点,在orf1a、orf1b、n和e基因靶序列上设计9条特异性crrna。通过检测发现9个crrna分别能特异性识别sars-cov-2病毒orf1a、orf1b、n和e的4个基因。并且,针对同一待检样品,同时对sars-cov-2病毒4基因orf1a、orf1b、n和e的检测,能有效提高检测准确性和可信性。
(2)本发明基于cpf1新冠病毒sars-cov-2的种快速检测工具,该工具包含荧光灯下肉眼直接可视检测,能实现方便快捷的结果判读。本发明为基层实验和临床一线提供了一个准确、快速、简便的检测方法。
(3)本发明公开了一系列用于新冠病毒sars-cov-2核酸检测的cpf1反应体系和crrna组合,其序列依次如seqidno.1至no.9所示。本发明系首次采用cpf1荧光法检测新冠病毒sars-cov-2,具有灵敏度高、特异性强、耗时短、高通量、不依赖大型实验设备等优势。这些优势使得本发明开发的基于cpf1荧光法检测方法方便用于基层实验和临床一线对新冠肺炎的基层快速检测和鉴定诊断。
附图说明
图1基于cpf1快速检测新冠病毒sars-cov-2核酸方法示意图;
图2不同crrna检测新冠病毒不同基因荧光检测效果;
图3检测新冠病毒不同crrna不同基因肉眼直接判读检测效果;
图4cpf1荧光检测新冠病毒sars-cov-2灵敏度;
图5cpf1检测新冠病毒sars-cov-2肉眼检测灵敏度;
图6荧光检测cpf1快速检测新冠病毒sars-cov-2与sars特异性;
图7肉眼直接检测cpf1快速检测新冠病毒sars-cov-2特异性;
图8模拟检测临床sars-cov-2核酸样品荧光检测结果;
图9模拟检测临床sars-cov-2核酸样品肉眼直接检测结果;
具体实施方式
下面结合具体实施例,进一步阐述本发明。应理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。此外应理解,在阅读了本发明讲授的内容之后,本领域技术人员可以对本发明作各种改动或修改,这些等价形式同样落于本申请所附权利要求书所限定的范围。
本发明中:rpa扩增试剂盒
本发明的总体技术示意图如附图1所示,包括以下3大部分:待检测核酸样品制备、cpf1检测成分设计制备和体系构建和荧光检测。
实施案例1:新冠病毒sars-cov-2基因片段快速灵敏检测
1.1核酸制备
本案例中新冠病毒sars-cov-2的orf1a、orf1b、n和e基因片段,是参考ncbiwuhan-hu-1毒株序列设计,由南京金斯瑞公司合成,命名为pc-orf1a、pc-orf1b、pc-n和pc-e,对应序列分别为seqno.10到seqno.13。
通过体外转录制备新冠病毒sars-cov-2的orf1a、orf1b、n和e基因对应的rna核酸样品。具体操作如下:使用megashortscriptt7转录试剂盒(thermofisherscientific),分别以pc-orf1a、pc-orf1b、pc-n和pc-e作为模板转录对应的基因rna样品pcrna-orf1a、pcrna-orf1b、pcrna-n和pcrna-e。转录后的rna用megaclear试剂盒(thermofisherscientific)纯化,并通过乙醇沉淀法回收;检测转录后rna质量和浓度,并保存在-80℃直至使用。
1.2针对sars-cov-2特异性crrna的设计制备
针对sars-cov-2特异性crrna制备按照如下方案进行,针对sars-cov-2的orf1a、orf1b、n和e基因,寻找包含cpf1识别序列(pam)tttn的靶向序列,设计23bp长度的crrna。分别命名为针对orf1a基因的orf1a-crrna1到orf1a-crrna2,针对orf1b基因的orf1b-crrna1到orf1b-crrna3,针对n基因的n-crrna1到n-crrna2和针对e基因的e-crrna1到e-crrna2。设计完成后,制备crrna。crrna的制备,可交于南京金斯瑞公司合成oligo,构建到载体puc57-t7-crrna,通过体外转录获得目的crrna,或可交由南京金斯瑞公司直接合成crrna序列对应的rna。
本发明提供的sars-cov-2的orf1a、orf1b、n和e基因的crrna包括seqno.1到seqno.9,具体信息如表1所示:
表1针对新冠病毒sars-cov-2核酸特异性crrna
1.3反转录等温扩增反应
本发明中,利用反转录等温扩增(rt-rpa)对单链rna(ssrna)病毒核酸进行预扩增,以便进行cpf1检测反应。按照等温扩增反应要求,设计并合成rt-rpa扩增引物rt-rpa-f(正向引物)和rt-rpa-r(反向引物),对应seqno.15至seqno.22(表2)。参考rt-rpa等温扩增操作步骤,扩增获得待检测样品。具体操作如下:等温扩增在50μl反应体系进行。将xμlrna样品,(18.5-x)μlddh2o,2μlrt-rpa-f,2μlrt-rpa-r和25μl反应缓冲液混匀,加入反应管溶解混匀。最后,加入2.5μl乙酸镁,混匀后在39℃下孵育25分钟。rt-rpa产物进行下一步检测。
表2针对新型冠状病毒ncov核酸特异性rt-rpa引物序列
1.4本案例中,新冠病毒cpf1检测采用20μl体系如表3所示,但不限于此,包含对相应成分的比例调整:
表3新冠病毒cpf1检测体系
1.5全波长酶标仪荧光检测
酶标仪荧光检测中,cpf1对目的基因检测体系依次加入2μlbuffer、1μlrnaseinhibitors、1μlcpf1、1μlssdnafqreporter、5μlrpasample、1μlcrrna和9μlh2o。各组分混合均匀后于37℃反应25min。其中,上述反应体系中rnaseinhibitors浓度为40u/μl,cpf1为200ng/μl,ssdnafqreporter浓度为25pm/μl,crrna浓度为1nm/μl。
首先,依次检测针对sars-cov-2病毒核酸的crrna检测效率。在cpf1检测体系中,各加入1μlcrrna,其它各组分维持一致,混合均匀,37℃反应25min,对上述反应产物进行后续结果检测判定。
利用荧光检测判定cpf1检测体系检测活性。使用全波长酶标仪用于测量检测反应的荧光,其中激发波长为485nm,发射波长为520nm,读取检测25min荧光数值为反应值。针对新冠病毒sars-cov-2不同,其反应检测结果如图2。结果表明,本发明检测体系中,针对不同基因片段的crrna均能高效特异的检测对应的orf1a、orf1b、n和e基因片段。
1.5荧光显示肉眼直接判读
同时,将上述cpf1检测反应25min后产物,置于在485nm激光灯下,可由肉眼直接进行结果判读。当crrna特异性识别靶核酸片段后,反应产物颜色由无色变为荧光绿;对应的,如果待检测无对应靶核酸,反应产物颜色仍未无色。在cpf1检测反应25min后,在荧光下进行肉眼判读,并拍照记录。如图3所示,针对不同基因片段的crrna均能高效特异的检测对应的orf1a、orf1b、n和e基因片段。
鉴于本实施案例中,针对orf1a、orf1b、n和e基因的crrna有一致的检测效果,将针对orf1a基因的orf1a-crrna1到orf1a-crrna2等比例混合获得orf1a-crrnamix;将针对orf1b基因的orf1b-crrna1到orf1b-crrna3等比例混合获得orf1b-crrnamix;针对n基因的n-crrna1到n-crrna2等比例混合获得n-crrnamix;将针对e基因的e-crrna1到e-crrna2等比例混合获得e-crrnamix。后续检测中,orf1a-crrnamix检测orf1a基因,orf1b-crrnamix检测orf1b基因,n-crrnamix检测n基因和e-crrnamix检测e基因。
实施案例2:基于cpf1荧光法高灵敏度检测sars-cov-2核酸
本案例中,为测定cpf1荧光法对sars-cov-2检测灵敏度,进行以下检测。
首先,根据根据sars-cov-2的orf1a、orf1b、n和e基因片段对应rna样品pcrna-orf1a、pcrna-orf1b、pcrna-n和pcrna-e,计算检测片段分子量,并进行10倍梯度稀释,获得每微升含有2*e7、2*e6、2*e5、2*e4、2*e3、2*e2、2*e1和2*e0拷贝数(copy/μl)的测试样品。
利用荧光法,对上述稀释样品进行cpf1特异性反应,参照实施例1中检测步骤进行检测和荧光结果判读。操作简述如下:2μl梯度稀释样品,加入到50μlrt-rpa等温扩增反应体系进行扩增。取10μl等温扩增产物,加入到20μlcpf1荧光核酸检测体系,混合均匀,37℃反应25min进行荧光结果判读。
本案例中,应用cpf1荧光法检测新冠病毒sars-cov-2,分别采用酶标仪检测(图4),可实现对4e0拷贝病毒的高灵敏度检出;采用荧光肉眼检测(图5),可实现对4e1拷贝病毒的高灵敏度检出。
实施案例3:基于cpf1荧光法高特异性检测sars-cov-2核酸
本实施案例中,为检测cpf1荧光法能否对sars-cov-2进行高特异反应,能否有效区分sars病毒和sars-cov-2病毒,进行以下检测。
首先,根据本发明中针对sars-cov-2病毒核酸检测区段pcrna-orf1a、pcrna-orf1b、pcrna-n和pcrna-e,参照实施案例1中方法,制备sars病毒对应基因序列rna样品:ncrna-orf1a、ncrna-orf1b、ncrna-n和ncrna-e。
其次,参照实施案例1,通过rt-rpa反应,分别扩增制备sars-cov-2和sars检测样品。
在荧光检测中,在cpf1检测体系中,依次加入2μlbuffer、1μlrnaseinhibitors、1μlcpf1、1μlssdnafqreporter、1μlcrrna和10μl检测样品。各组分混合均匀,在37℃反应25min。本检测体系中,rnaseinhibitors浓度为40u/μl,cpf1浓度为200ng/μl,ssdnadbreporter浓度为25pm/μl,crrna浓度为1nm/μl。
本实施案例中,利用荧光检测判定cpf1检测体系检测活性。使用全波长酶标仪用于测量检测反应的荧光,其中激发波长为485nm,发射波长为520nm,取检测25min时荧光数值为反应值。检测结果如图6,本发明中cpf1荧光法可高特异性检测sars-cov-2病毒核酸,而对sars病毒没有反应。
本实施案例中,在cpf1检测体系反应25min后,同样可以在485nm激光灯下,用肉眼对检测产物荧光反应进行判读。检测结果如图7,本发明中cpf1荧光法可高特异性检测sars-cov-2病毒核酸,而对sars病毒没有反应。
实施案例4:模拟cpf1荧光法快速检测临床sars-cov-2病毒核酸
本实施案例模拟临床组织样品的核酸进行sars-cov-2快速检测,本检测不涉及任何临床新冠病毒样品,且所有操作合格按照有关法律法规和相关规定进行。
本案例中,首先,将实施案例3中sars-cov-2病毒核酸对应rna样品(pcrna-orf1a、pcrna-orf1b和pcrna-n)和sars病毒核酸对应rna样品(pcrna-encrna-orf1a、ncrna-orf1b、ncrna-n和ncrna-e),点涂到咽拭子,模拟临床样品进行检测。
首先,对病毒灭活和病毒核酸释放。本实施例使用购自诺唯赞公司的快速核酸释放剂获得预处理的核酸。步骤如下:将咽拭子加入50μlpbs,模拟溶解病毒到液体。取3μl待检测样品加入20μl核酸裂解液,加入rna酶抑制剂,常温静置3分钟,加入20μl中和液,混匀后进行下一步检测。在cpf1检测中,每个待检测样品取5μl进行rt-rpa预扩增,步骤与实施案例1中相同,获得cpf1检测样品。
在cpf1检测体系中,依次加入2μlbuffer、1μlrnaseinhibitors、1μlcpf1、1μlssdnafqreporter、10μlrpasample、1μlcrrna。各组分混合均匀,在37℃反应25min。
本实施案例中,可通过酶标仪或荧光灯下肉眼直接判定cpf1检测结果。如图8所示,本发明中cpf1荧光法可通过酶标仪检测可有效对模拟临床阴阳性样品判定。同样的,在荧光灯下肉眼可有效对模拟临床阴阳性样品判定(图9)。
sequencelisting
<110>上海科技大学
<120>一种用于新冠病毒核酸快速检测的cpf1试剂盒及其制备方法与应用
<130>pcn1200195
<160>22
<170>patentinversion3.5
<210>1
<211>27
<212>dna
<213>artificialsequence
<220>
<223>orf1a-crrna1
<400>1
tttggtggtgcatcgtgttgtctgtac27
<210>2
<211>27
<212>dna
<213>artificialsequence
<220>
<223>orf1a-crrna2
<400>2
tttgtgacttaaaaggtaagtatgtac27
<210>3
<211>27
<212>dna
<213>artificialsequence
<220>
<223>orf1b-crrna1
<400>3
tttcactcaatacttgagcacactcat27
<210>4
<211>27
<212>dna
<213>artificialsequence
<220>
<223>orf1b-crrna2
<400>4
tttttaacatttgtcaagctgtca27
<210>5
<211>27
<212>dna
<213>artificialsequence
<220>
<223>orf1b-crrna3
<400>5
ttttaacatttgtcaagctgtcacggc27
<210>6
<211>27
<212>dna
<213>artificialsequence
<220>
<223>n-crrna1
<400>
tttcttgaactgttgcgactacgtgat27
<210>7
<211>27
<212>dna
<213>artificialsequence
<220>
<223>n-crrna2
<400>7
tttgctgctgcttgacagattgaacca27
<210>8
<211>27
<212>dna
<213>artificialsequence
<220>
<223>e-crrna1
<400>8
tttcttgctttcgtggtattcttgcta27
<210>9
<211>27
<212>dna
<213>artificialsequence
<220>
<223>e-crrna2
<400>9
tttcgtggtattcttgctagttacact27
<210>10
<211>299
<212>dna
<213>artificialsequence
<220>
<223>pc-orf1a
<400>10
actggtactggtcaggcaataacagttacaccggaagccaatatggatcaagaatccttt60
ggtggtgcatcgtgttgtctgtactgccgttgccacatagatcatccaaatcctaaagga120
ttttgtgacttaaaaggtaagtatgtacaaatacctacaacttgtgctaatgaccctgtg180
ggttttacacttaaaaacacagtctgtaccgtctgcggtatgtggaaaggttatggctgt240
agttgtgatcaactccgcgaacccatgcttcagtcagctgatgcacaatcgtttttaa299
<210>11
<211>299
<212>dna
<213>artificialsequence
<220>
<223>pc-orf1b
<400>11
cacaccgtttctatagattagctaatgagtgtgctcaagtattgagtgaaatggtcatgt60
gtggcggttcactatatgttaaaccaggtggaacctcatcaggagatgccacaactgctt120
atgctaatagtgtttttaacatttgtcaagctgtcacggccaatgttaatgcacttttat180
ctactgatggtaacaaaattgccgataagtatgtccgcaatttacaacacagactttatg240
agtgtctctatagaaatagagatgttgacacagactttgtgaatgagttttacgcatat299
<210>12
<211>228
<212>dna
<213>artificialsequence
<220>
<223>pc-e
<400>12
atgtactcattcgtttcggaagagacaggtacgttaatagttaatagcgtacttcttttt60
cttgctttcgtggtattcttgctagttacactagccatccttactgcgcttcgattgtgt120
gcgtactgctgcaatattgttaacgtgagtcttgtaaaaccttctttttacgtttactct180
cgtgttaaaaatctgaattcttctagagttcctgatcttctggtctaa228
<210>13
<211>299
<212>dna
<213>artificialsequence
<220>
<223>pc-n
<400>13
gctaacaatgctgcaatcgtgctacaacttcctcaaggaacaacattgccaaaaggcttc60
tacgcagaagggagcagaggcggcagtcaagcctcttctcgttcctcatcacgtagtcgc120
aacagttcaagaaattcaactccaggcagcagtaggggaacttctcctgctagaatggct180
ggcaatggcggtgatgctgctcttgctttgctgctgcttgacagattgaaccagcttgag240
agcaaaatgtctggtaaaggccaacaacaacaaggccaaactgtcactaagaaatctgc299
<210>14
<211>3744
<212>dna
<213>artificialsequence
<220>
<223>cpf1蛋白核酸序列
<400>14
agcaagctggaaaaatttaccaactgctacagcctgagcaagaccctgcgtttcaaagcg60
atcccggttggcaagacccaggaaaacattgacaacaaacgtctgctggttgaggacgaa120
aagcgtgcggaggattataaaggtgtgaagaaactgctggatcgttactatctgagcttt180
atcaacgacgtgctgcacagcattaagctgaaaaacctgaacaactacatcagcctgttc240
cgtaagaaaacccgtaccgagaaggaaaacaaagagctggaaaacctggaaatcaacctg300
cgtaaggagattgcgaaggcgttcaagggtaacgagggctacaagagcctgttcaagaaa360
gatatcatcgaaaccatcctgccggagttcctggacgataaggacgaaattgcgctggtt420
aacagcttcaacggttttaccaccgcgttcaccggcttctttgataaccgtgagaacatg480
tttagcgaggaagcgaaaagcaccagcatcgcgttccgttgcattaacgaaaacctgacc540
cgttacatcagcaacatggacattttcgagaaggttgacgcgatctttgataaacacgag600
gtgcaggaaatcaaggagaaaattctgaacagcgactatgatgttgaagatttctttgag660
ggtgaattctttaactttgttctgacccaagagggcatcgacgtgtacaacgcgatcatt720
ggtggcttcgtgaccgaaagcggcgagaagatcaaaggcctgaacgagtacattaacctg780
tataaccagaagaccaaacaaaagctgccgaaatttaagccgctgtataagcaggtgctg840
agcgatcgtgaaagcctgagcttctacggcgagggctataccagcgacgaggaagttctg900
gaagtgtttcgtaacaccctgaacaaaaacagcgagatcttcagcagcattaagaaactg960
gaaaagctgttcaaaaactttgacgagtacagcagcgcgggtatctttgttaagaacggc1020
ccggcgatcagcaccattagcaaagatatcttcggtgaatggaacgtgattcgtgacaag1080
tggaacgcggagtatgacgatatccacctgaagaaaaaggcggtggttaccgaaaagtac1140
gaggacgatcgtcgtaaaagcttcaaaaagattggcagctttagcctggaacagctgcaa1200
gagtacgcggacgcggatctgagcgtggttgaaaaactgaaggagatcattatccagaag1260
gttgatgaaatctacaaagtgtatggtagcagcgagaagctgttcgacgcggattttgtt1320
ctggagaagagcctgaaaaagaacgacgcggtggttgcgatcatgaaggacctgctggat1380
agcgtgaaaagcttcgaaaactacattaaggcgttctttggtgaaggcaaagagaccaac1440
cgtgacgagagcttctatggcgattttgttctggcgtacgacatcctgctgaaggtggac1500
cacatctacgatgcgattcgtaactatgttacccaaaaaccgtacagcaaggataagttc1560
aagctgtacttccagaacccgcaattcatgggtggctgggacaaggataaagagaccgac1620
tatcgtgcgaccatcctgcgttacggtagcaagtactatctggcgattatggataaaaag1680
tacgcgaaatgcctgcagaagatcgacaaagacgatgttaacggtaactacgaaaagatc1740
aactacaagctgctgccgggcccgaacaagatgctgccgaaagtgttctttagcaaaaag1800
tggatggcgtactataacccgagcgaggacatccaaaagatctacaagaacggtaccttc1860
aaaaagggcgatatgtttaacctgaacgactgccacaagctgatcgacttctttaaagat1920
agcattagccgttatccgaagtggagcaacgcgtacgatttcaactttagcgagaccgaa1980
aagtataaagacatcgcgggtttttaccgtgaggttgaggaacagggctataaagtgagc2040
ttcgaaagcgcgagcaagaaagaggtggataaactggtggaggaaggtaaactgtacatg2100
ttccaaatctacaacaaggacttcagcgataagagccacggcaccccgaacctgcacacc2160
atgtacttcaagctgctgtttgacgaaaacaaccatggtcagatccgtctgagcggtggc2220
gcggagctgttcatgcgtcgtgcgagcctgaagaaagaggagctggttgtgcacccggcg2280
aacagcccgattgcgaacaaaaacccggataacccgaaaaagaccaccaccctgagctac2340
gacgtgtataaggataaacgttttagcgaagaccaatacgagctgcacattccgatcgcg2400
attaacaagtgcccgaaaaacatcttcaagattaacaccgaagttcgtgtgctgctgaaa2460
cacgacgataacccgtatgttatcggtattgaccgtggcgagcgtaacctgctgtacatc2520
gtggttgtggacggtaaaggcaacattgtggaacagtatagcctgaacgagattatcaac2580
aactttaacggtatccgtattaagaccgattaccacagcctgctggacaaaaaggagaag2640
gaacgtttcgaggcgcgtcagaactggaccagcatcgaaaacattaaggagctgaaagcg2700
ggctatatcagccaagttgtgcacaagatttgcgaactggttgagaaatacgatgcggtg2760
atcgcgctggaggacctgaacagcggttttaagaacagccgtgttaaggtggaaaagcag2820
gtttaccaaaagttcgagaagatgctgatcgataagctgaactacatggtggacaaaaag2880
agcaacccgtgcgcgaccggtggcgcgctgaaaggttatcagattaccaacaagttcgaa2940
agctttaaaagcatgagcacccaaaacggcttcatcttttacattccggcgtggctgacc3000
agcaaaatcgatccgagcaccggttttgttaacctgctgaagaccaaatataccagcatt3060
gcggatagcaaaaagttcatcagcagctttgaccgtattatgtacgtgccggaggaagac3120
ctgttcgagtttgcgctggactataagaacttcagccgtaccgacgcggactacatcaaa3180
aagtggaaactgtacagctatggtaaccgtatccgtattttccgtaacccgaaaaagaac3240
aacgtttttgactgggaggaagtgtgcctgaccagcgcgtataaggaactgttcaacaaa3300
tacggtatcaactatcagcaaggcgatattcgtgcgctgctgtgcgagcagagcgacaag3360
gcgttctacagcagctttatggcgctgatgagcctgatgctgcaaatgcgtaacagcatc3420
accggtcgtaccgatgttgattttctgatcagcccggtgaaaaacagcgacggcattttc3480
tacgatagccgtaactatgaagcgcaggagaacgcgattctgccgaagaacgcggacgcg3540
aacggtgcgtataacatcgcgcgtaaagttctgtgggcgattggccagttcaaaaaggcg3600
gaggacgaaaagctggataaggtgaaaatcgcgattagcaacaaagaatggctggagtac3660
gcgcaaaccagcgttaagcacgagaacctgtacttccaatcccaccaccaccaccaccac3720
caccaccaccaccaccaccactga3744
<210>15
<211>33
<212>dna
<213>artificialsequence
<220>
<223>ofr1a-c-rt-rpa-f
<400>15
actggtactggtcaggcaataacagttacaccg33
<210>16
<211>32
<212>dna
<213>artificialsequence
<220>
<223>ofr1a-c-rt-rpa-r
<400>16
ttgtgcatcagctgactgaagcatgggttcgc32
<210>17
<211>34
<212>dna
<213>artificialsequence
<220>
<223>orf1b-n-rt-rpa-f
<400>17
cacaccgtttctatagattagctaatgagtgtgc34
<210>18
<211>33
<212>dna
<213>artificialsequence
<220>
<223>orf1b-n-rt-rpa-r
<400>18
tgcgtaaaactcattcacaaagtctgtgtcaac33
<210>19
<211>32
<212>dna
<213>artificialsequence
<220>
<223>e-rt-rpa-f
<400>19
tgtactcattcgtttcggaagagacaggtacg32
<210>20
<211>35
<212>dna
<213>artificialsequence
<220>
<223>e-rt-rpa-r
<400>20
tagaccagaagatcaggaactctagaagaattcag35
<210>21
<211>33
<212>dna
<213>artificialsequence
<220>
<223>n-rt-rpa-f
<400>21
ctaacaatgctgcaatcgtgctacaacttcct33
<210>22
<211>33
<212>dna
<213>artificialsequence
<220>
<223>n-rt-rpa-r
<400>22
ttcttagtgacagtttggccttgttgttgttgg33
- 还没有人留言评论。精彩留言会获得点赞!