一种梨形番茄材料的制备方法
本发明属于农业生物技术领域,具体涉及一种梨形番茄材料的制备方法。
背景技术:
番茄是全球广泛种植的蔬菜和水果兼用类作物,深受消费者喜欢。
番茄的果形是重要的经济性状。目前大部分鲜食番茄果实都是圆形。番茄果实的形状能很大程度上影响消费者的选择,是重要的商品性状,奇特的形状会吸引消费者购买,增加农民的收入,因此培育形状奇特的番茄果实是重要的育种方向。
番茄品种培育主要是利用传统的杂交、回交的手段来改良番茄果形,该法周期长,一般需要5-10年,且受连锁累赘等因素的限制。
技术实现要素:
本发明的目的是提供一种梨形番茄材料的制备方法。
本发明提供了一种蛋白质,获自番茄,命名为slovate蛋白,为如下(a1)或(a2)或(a3):
(a1)序列表中序列1所示的蛋白质;
(a2)将序列表中序列1所示的蛋白质经过一个或几个氨基酸残基的取代和/或缺失和/或添加且与番茄果形相关的由其衍生的蛋白质;
(a3)来源于番茄且与(a1)所限定的氨基酸序列至少具有75%、至少具有80%、至少具有85%、至少具有90%、至少具有95%、至少具有96%、至少具有97%、至少具有98%或至少具有99%以上同一性且与番茄果形相关的蛋白质。
编码slovate蛋白的基因,命名为slovate基因,也属于本发明的保护范围。
slovate基因具体为如下(b1)或(b2)或(b3)或(b4):
(b1)编码区如序列表中序列2所示的dna分子;
(b2)序列表中序列3所示的dna分子;
(b3)来源于番茄且与(b1)或(b2)至少具有75%、至少具有80%、至少具有85%、至少具有90%、至少具有95%、至少具有96%、至少具有97%、至少具有98%或至少具有99%以上同一性且编码所述蛋白质的dna分子;
(b4)在严格条件下与(b1)或(b2)限定的核苷酸序列杂交且编码所述蛋白质的dna分子。
所述严格条件可为如下:50℃,在7%十二烷基硫酸钠(sds)、0.5mnapo4和1mmedta的混合溶液中杂交,在50℃,2×ssc,0.1%sds中漂洗;还可为:50℃,在7%sds、0.5mnapo4和1mmedta的混合溶液中杂交,在50℃,1×ssc,0.1%sds中漂洗;还可为:50℃,在7%sds、0.5mnapo4和1mmedta的混合溶液中杂交,在50℃,0.5×ssc,0.1%sds中漂洗;还可为:50℃,在7%sds、0.5mnapo4和1mmedta的混合溶液中杂交,在50℃,0.1×ssc,0.1%sds中漂洗;还可为:50℃,在7%sds、0.5mnapo4和1mmedta的混合溶液中杂交,在65℃,0.1×ssc,0.1%sds中漂洗;也可为:在6×ssc,0.5%sds的溶液中,在65℃下杂交,然后用2×ssc,0.1%sds和1×ssc,0.1%sds各洗膜一次;也可为:2×ssc,0.1%sds的溶液中,在68℃下杂交并洗膜2次,每次5min,又于0.5×ssc,0.1%sds的溶液中,在68℃下杂交并洗膜2次,每次15min;也可为:0.1×sspe(或0.1×ssc)、0.1%sds的溶液中,65℃条件下杂交并洗膜。
含有slovate基因的表达盒、重组载体或重组微生物均属于本发明的保护范围。
本发明还保护slovate蛋白的应用,为如下(c1)或(c2):
(c1)调控植物的果形;
(c2)调控番茄的果形。
本发明还保护一种植物育种方法,包括如下步骤:对植物中slovate基因进行基因编辑,从而得到果实形状为梨形的植物。所述植物可为茄科植物。所述植物可为番茄属植物。所述植物为果实形状为圆形的植物。所述植物具体可为番茄品种ac。所述基因编辑具体借助cas9技术实现。sgrna中的靶序列结合区如序列表的序列4第2-21位核苷酸所示。sgrna具体如序列表中的序列4所示。
本发明还保护一种植物育种方法,包括如下步骤:抑制植物中slovate基因的表达,从而得到果实形状为梨形的植物。所述植物可为茄科植物。所述植物可为番茄属植物。所述植物为果实形状为圆形的植物。所述植物具体可为番茄品种ac。
本发明还保护一种植物育种方法,包括如下步骤:降低植物中slovate蛋白的含量和/或活性,从而得到果实形状为梨形的植物。所述植物可为茄科植物。所述植物可为番茄属植物。所述植物为果实形状为圆形的植物。所述植物具体可为番茄品种ac。
本发明还保护一种制备基因编辑植物的方法,包括如下步骤:在受体植物中导入重组质粒,得到转基因植物,从转基因植物中筛选得到基因编辑植物;所述重组质粒中具有cas9蛋白的编码基因和sgrna的编码基因;所述sgrna的靶序列位于slovate基因;所述基因编辑植物的果实形状为梨形。所述植物可为茄科植物。所述植物可为番茄属植物。所述植物为果实形状为圆形的植物。所述植物具体可为番茄品种ac。sgrna中的靶序列结合区如序列表的序列4第2-21位核苷酸所示。sgrna具体如序列表中的序列4所示。所述重组质粒具体可为在crispr/cas9载体的bsai酶切位点插入所述靶序列结合区的编码dna得到的。
本发明还保护一种制备基因编辑植物的方法,包括如下步骤:在受体植物中导入表达sgrna基因的重组质粒和表达cas9基因的重组质粒,得到转基因植物,从转基因植物中筛选得到基因编辑植物;所述sgrna的靶序列位于slovate基因;与所述受体植物相比,所述基因编辑植物的果实形状为梨形。所述植物可为茄科植物。所述植物可为番茄属植物。所述植物为果实形状为圆形的植物。所述植物具体可为番茄品种ac。sgrna中的靶序列结合区如序列表的序列4第2-21位核苷酸所示。sgrna具体如序列表中的序列4所示。
果实形状为梨形指的是果实形状类似梨(鸭梨)的外型。
本发明利用crispr/cas9方法对圆果番茄的slovate基因进行基因编辑,得到了梨形番茄。本发明可以在1年以内得到一个果型改变的植物新品种,而利用传统育种手段需要连续回交、自交,至少需要3-4年时间,本发明的方法操作简单,成本低,大大加快了育种进程。本发明对于梨形番茄育种具有重要的应用推广价值。
附图说明
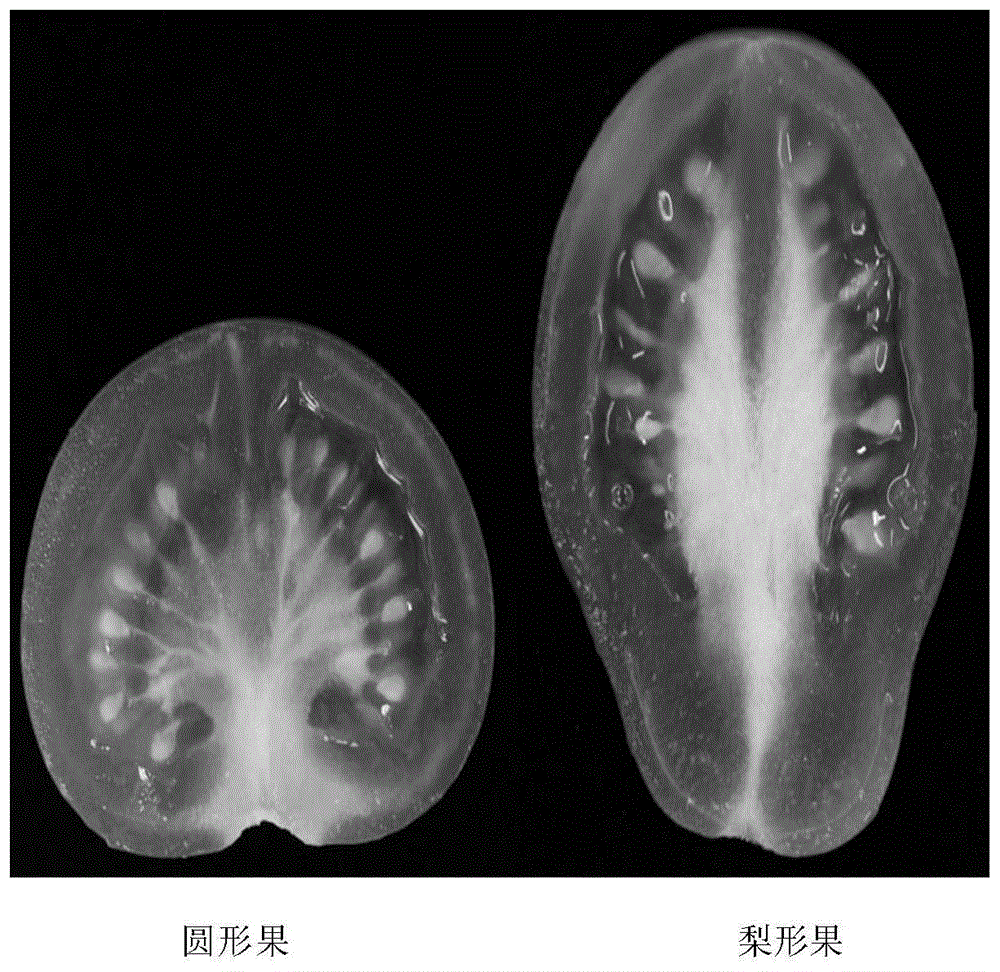
图1为供试植株果实的照片。
具体实施方式
以下的实施例便于更好地理解本发明,但并不限定本发明。下述实施例中的实验方法,如无特殊说明,均为常规方法。下述实施例中所用的试验材料,如无特殊说明,均为自常规生化试剂商店购买得到的。如无特殊说明,以下实施例中的定量试验,均设置三次重复实验,结果取平均值。
crispr/cas9载体:即文献“dengetal.,efficientgenerationofpink-fruitedtomatoesusingcrispr/cas9system,journalofgeneticsandgenomics45(2018)51-54”中的ptx041。
番茄品种ac,又称为番茄品种ailsacraig,也称为野生型番茄,来源于美国番茄遗传资源中心(tgrc,http://tgrc.ucdavis.edu/),编号为la2838a。
从番茄中发现一个新蛋白,命名为slovate蛋白,如序列表的序列1所示。番茄中,编码slovate蛋白的基因命名为slovate基因,cdna中的开放阅读框如序列表的序列2所示,基因组dna中起始密码子至终止密码子之间的部分如序列表的序列3所示(序列3中第1-694位和第1242-1384位为外显子)。
实施例1、利用crispr/cas9方法对番茄进行slovate基因编辑获得梨形番茄
一、重组载体的构建
1、分别制备两条单链dna分子,然后一起进行退火,得到两端具有粘末端的双链dna分子。
两条单链dna分子的核苷酸序列分别如下:
5’-tttgaatccacgatgatctccgtt-3’;
5’-aaacaacggagatcatcgtggatt-3’。
2、取crispr/cas9载体,用限制性内切酶bsai进行酶切,回收载体骨架。
3、将步骤1得到的两端具有粘末端的双链dna分子和步骤2得到的载体骨架连接,得到重组质粒。该重组质粒表达序列表中序列4所示的sgrna。该sgrna中的靶序列结合区如序列表的序列4第2-21位核苷酸所示。
二、基因编辑植株的制备
1、取步骤一制备的重组质粒,导入农杆菌gv3101,得到重组农杆菌。
2、取步骤1得到的重组农杆菌,通过农杆菌介导的方法对野生型番茄进行遗传转化,得到t0代植株。
(1)取野生型番茄,挑选饱满、粒大的种子,用75%乙醇水溶液浸泡2min,然后用10%naclo水溶液浸泡10min,然后用无菌水冲洗7遍,然后将种子播种于种子生长培养基并培养8天。取子叶,在无菌条件下将子叶切成小方块,将子叶块接种于预培养培养基并培养2天,得到的子叶块作为外植体。
种子生长培养基:将2.2gms盐、30g蔗糖和水混合,用水定容至1l,用1mol/lkoh调ph至5.8~6.0之间,加入琼脂并使其浓度为0.8%,高压灭菌。
预培养培养基:将4.4gms盐、1.0mg玉米素(zeatin)和30g蔗糖溶于水中,用水定容至1l,用1mol/lkoh调ph至5.8~6.0之间,加入琼脂并使其浓度为0.8%,高压灭菌。
(2)取步骤1得到得到重组农杆菌,用50ml液体ms培养基重悬,得到od600nm=0.4的菌悬液,然后加入50μl0.074mol/l乙酰丁香酮水溶液,即为侵染液。
液体ms培养基:将4.4gms盐、30g蔗糖和水混合,用水定容至1l,用1mol/lkoh调ph至5.8~6.0之间,高压灭菌。
(3)取步骤(1)得到的外植体,在步骤(2)得到的浸染液中浸没10min,然后接种至预培养培养基上培养2天。
(4)完成步骤(3)后,取外植体,接种至筛选分化培养基培养8周(每2周继代一次),此时外植体长出抗性芽。
筛选分化培养基:将4.4gms盐、2.0mg玉米素、50mg卡那霉素、100mg肌醇、0.5mg叶酸和20g蔗糖溶于水中,用水定容至1l,用1mol/lkoh调ph至5.8~6.0之间,加入琼脂并使其浓度为0.8%,高压灭菌。
(5)步骤(4)中抗性芽芽长为3cm时,切取抗性芽,转接至生根培养基进行培养,生根的植株即为t0代植株。
生根培养基:将4.4gms盐、50mg卡那霉素、0.5mg叶酸、0.5mg吲哚丁酸和30g蔗糖溶于水中,用水定容至1l,用1mol/lkoh调ph至5.8~6.0之间,加入琼脂并使其浓度为0.8%,高压灭菌。
整个过程的培养条件:25℃、16h光照/8h黑暗。
3、从t0代植株中筛选纯合突变的植株。
筛选方法:取植株叶片的基因组dna,采用f1和r1组成的引物对进行pcr扩增,回收pcr扩增产物并进行测序。
f1:5′-cggttctccagagttattgc-3′;
r1:5′-gctgtactcatcagctgagg-3′。
筛选到发生纯合突变的植株(突变均位于slovate基因中f1和r1组成的引物对的靶序列)。与野生型番茄相比,该纯合突变植株中的“aatccacgatgatctccgtt”替换为了“aatccacgagatctccgtt”,且为纯合型替换(即两条染色体发生了相同替换)。上述替换导致基因cds序列从第203位发生移码突变,提前产生终止密码子,翻译出丧失dna结合结构域的截短蛋白,以致slovate蛋白质功能的丧失(植株中不含有slovate蛋白)。
4、正常培养步骤3得到的纯合突变植株,自交获得种子,即为t1代种子,将t1代种子培育为植株,即为t1代植株。
5、从步骤4得到的t1代植株中筛选不含外源dna的植株。
筛选方法:取植株叶片的基因组dna,采用f2和r2组成的引物对进行pcr扩增(f2和r2组成的引物对的靶序列位于crispr/cas9载体中的cas9基因,预期扩增产物约400bp),如果得到扩增产物说明植株中含有外源dna,如果没有得到扩增产物说明植株中不含外源dna。
f2:5′-ttgacaagctgttcatccag-3′;
r2:5′-ccttcgtaatctcggtgttc-3′。
t1代植株群体中约1/4的个体不含外源dna。
6、取步骤5筛选得到的植株,提取叶片的基因组dna,采用f1和r1组成的引物对进行pcr扩增,回收pcr扩增产物并进行测序。
从t1代植株中筛选得到的不含外源dna的植株,slovate基因均发生了与步骤3得到的纯合突变植株相同的纯合突变。结果表明,将步骤一制备的重组质粒导入野生型番茄产生的突变能够从t0代稳定遗传到t1代。
将从t1代植株中筛选得到的不含外源dna的植株均命名为slovate基因编辑植株。
三、果实观察
供试植株为:slovate基因编辑植株、野生型番茄植株。
1、25℃、16h光照/8h黑暗条件下培养供试植株幼苗,直至植株长至4-5个叶片。
2、完成步骤1后,将植株(每种供试植株10株,且长势一致)移栽到大棚中。株距、行距在60cm以上;供试植株随机分布;正常的水肥管理,保证所有植株水肥条件基本一致。
移栽3个月后,植株开始进入结果期,结果期持续约6个月。整个结果期,收集成熟果实。
slovate基因编辑植株,平均每株植株在整个结果期收获的成熟果实数量约为50个,均为梨形果。
野生型番茄植株,平均每株植株在整个结果期收获的成熟果实数量约为50个,均为圆形果。
示例性的梨形果和圆形果的照片见图1。
以上结果表明,slovate基因及其编码的蛋白质可以调控番茄的果形,通过对slovate基因进行基因编辑使基因功能丧失可以使圆形番茄材料转变成梨形番茄材料。
以上对本发明进行了详述。对于本领域技术人员来说,在不脱离本发明的宗旨和范围,以及无需进行不必要的实验情况下,可在等同参数、浓度和条件下,在较宽范围内实施本发明。虽然本发明给出了特殊的实施例,应该理解为,可以对本发明作进一步的改进。总之,按本发明的原理,本申请欲包括任何变更、用途或对本发明的改进,包括脱离了本申请中已公开范围,而用本领域已知的常规技术进行的改变。按以下附带的权利要求的范围,可以进行一些基本特征的应用。
sequencelisting
<110>中国科学院遗传与发育生物学研究所
青岛市农业科学研究院
<120>一种梨形番茄材料的制备方法
<130>gncyx200500
<160>4
<170>patentinversion3.5
<210>1
<211>278
<212>prt
<213>solanumlycopersicum
<400>1
metglylysserleulysleuargpheserargvalilealaserphe
151015
asnsercysargserlysasnproserserleuproglnasnproasn
202530
phepheprohislysleuthrserthrlyshisileserproaspphe
354045
proleuileaspglnasnglnasnglnasnhisargasntyrvalpro
505560
gluserthrmetileservalglycyscysargsergluphelystrp
65707580
glulysgluglulysphehisvalvalserserserphevalserglu
859095
glugluglucysgluglugluileasnleualaleuargproproleu
100105110
thrproproargpheserargilevalvalglulyslyslyslyslys
115120125
glnglnargvallyslysthrlysthrlysserargileileargmet
130135140
serthrserseralaaspglutyrserglyileleuserglythrasn
145150155160
thrasptrpaspasnasngluglugluthrgluserleuvalserser
165170175
serargsercystyrasppheserseraspaspserserthraspphe
180185190
asnprohisleugluthrilecysgluthrthrthrmetargargarg
195200205
hislysargasnalaasnthrlysargargserilelysglnserarg
210215220
proserpheserserserlysglyargargserservalserthrser
225230235240
seraspsergluleuproalaargleuservalphelyslysleuile
245250255
procysservalaspglylysvallysgluserphealailevallys
260265270
lysserglnaspprotyr
275
<210>2
<211>837
<212>dna
<213>solanumlycopersicum
<400>2
atgggaaaaagtttgaagcttcggttctccagagttattgcttctttcaattcgtgccgt60
tcgaaaaacccttcttctcttccccaaaatcctaatttcttcccacataagctcactagt120
acaaaacacatttcccccgatttccctcttattgatcaaaatcaaaatcaaaatcaccgt180
aattacgtgccagaatccacgatgatctccgttgggtgttgtagatcagaattcaagtgg240
gagaaagaagagaagtttcacgtggtttctagttccttcgtgtctgaagaagaagaatgt300
gaagaggagatcaatttggccttacgacctcctcttacacctccgcgattcagtagaatt360
gttgttgagaagaagaagaagaaacaacagcgagttaaaaaaacgaaaacaaaaagtaga420
atcatccgaatgagtacttcctcagctgatgagtacagcgggatattaagcggtactaat480
actgattgggataataatgaagaggaaactgaatctttagtttcatcttccagaagctgt540
tacgatttctcaagcgatgactcatctactgatttcaaccctcacttagaaaccatatgt600
gagaccactacaatgaggcgtcgtcacaagagaaatgccaacaccaagaggagatcaatc660
aagcaatccagaccaagtttttcctcttcaaaaggtagaagatcgtcggtttctacgtca720
tcagatagcgagctaccggcaaggttatcggtgtttaagaagctgataccgtgtagtgtg780
gatgggaaagtgaaggagagtttcgcgatagtgaagaaatctcaggacccgtactaa837
<210>3
<211>1384
<212>dna
<213>solanumlycopersicum
<400>3
atgggaaaaagtttgaagcttcggttctccagagttattgcttctttcaattcgtgccgt60
tcgaaaaacccttcttctcttccccaaaatcctaatttcttcccacataagctcactagt120
acaaaacacatttcccccgatttccctcttattgatcaaaatcaaaatcaaaatcaccgt180
aattacgtgccagaatccacgatgatctccgttgggtgttgtagatcagaattcaagtgg240
gagaaagaagagaagtttcacgtggtttctagttccttcgtgtctgaagaagaagaatgt300
gaagaggagatcaatttggccttacgacctcctcttacacctccgcgattcagtagaatt360
gttgttgagaagaagaagaagaaacaacagcgagttaaaaaaacgaaaacaaaaagtaga420
atcatccgaatgagtacttcctcagctgatgagtacagcgggatattaagcggtactaat480
actgattgggataataatgaagaggaaactgaatctttagtttcatcttccagaagctgt540
tacgatttctcaagcgatgactcatctactgatttcaaccctcacttagaaaccatatgt600
gagaccactacaatgaggcgtcgtcacaagagaaatgccaacaccaagaggagatcaatc660
aagcaatccagaccaagtttttcctcttcaaaaggtaaaagtactatttgtgcgatcaaa720
tcttttatatttacagataattttacagtcacattaataagtattgcttttcgcgtgtgg780
aatttggagaggacagagtaaacatagatcttatagctattttgtggagagattaatttc840
aaaagacacccaataacaataacatattcaataaaattccactaactagagtagaaaaag900
gatagagtttacacggagcttaccgttcaaaaattctggctcaaagcaaatgcatatctt960
atttagagatcaataattttattttaaatatttttatcgtctttttctttgggaaatagt1020
gtatatgtaattgtcgtacaaaaatatatgaagcttaatcaaaattcattacattaatta1080
aaacggaaaactatttaatttataattatgtctcaacatgcatgcttcctttgttacttg1140
tttattattgggtcgaatatatgaaaatatattttggaaagacttttgttgcgctagtat1200
ttaaaaaactagtactgattaattatattatgtttgaacaggtagaagatcgtcggtttc1260
tacgtcatcagatagcgagctaccggcaaggttatcggtgtttaagaagctgataccgtg1320
tagtgtggatgggaaagtgaaggagagtttcgcgatagtgaagaaatctcaggacccgta1380
ctaa1384
<210>4
<211>97
<212>rna
<213>artificialsequence
<400>4
gaauccacgaugaucuccguuguuuuagagcuagaaauagcaaguuaaaauaaggcuagu60
ccguuaucaacuugaaaaaguggcaccgagucggugc97
- 还没有人留言评论。精彩留言会获得点赞!