一种鉴定甘蓝品种真实性的方法及其专用SSR引物组合与流程
本发明属于分子标记及其检测领域,具体涉及一种鉴定甘蓝品种真实性的方法及其专用ssr引物组合。
背景技术:
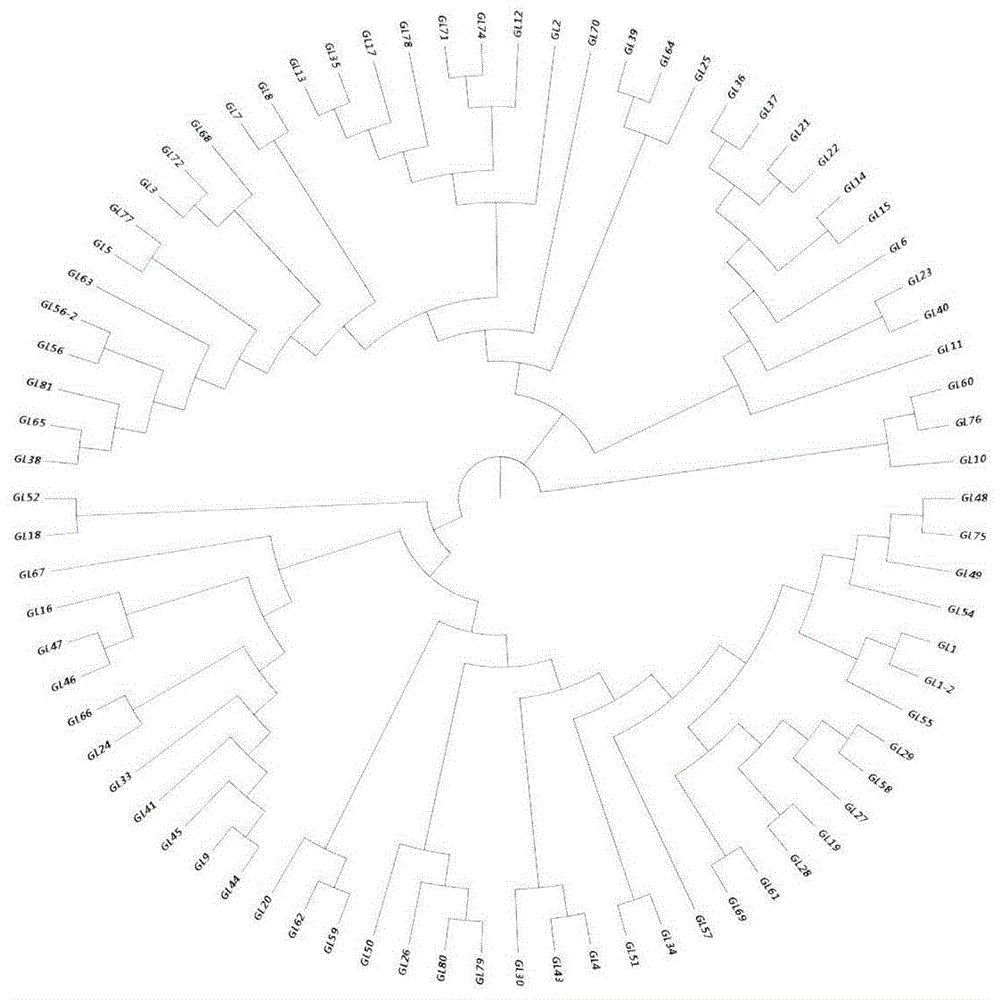
:甘蓝是世界范围内种植规模最大的叶菜类蔬菜作物,并在我国有悠久的栽培历史。改革开放以来随着甘蓝产业的快速发展,通过国家和各省审定的品种逐年增多。自2017年《非主要农作物品种登记办法》实施以来,新品种登记数量呈现出了一种井喷式的爆发显现。这种爆发现象为开展登记品种管理和市场监管,特别是对依赖田间鉴定的方式提出了前所未有的挑战。由于甘蓝育种企业较为分散,各省市之间品种审定相互不协调,甘蓝的品种管理未能有效跟进,存在很多假冒伪劣品种,其中很多为衍生、派生近似品种。加之良种生产基地管理混乱,盗育、套购品种现象猖獗,同名异种现象严重,种子质量事故时有发生。从2017年开始,农业农村部开始执行非主要农作物品种登记制度,现阶段甘蓝申请品种已经达到927份,而公告品种只有475份,由于申请品种过多无法在短时间内完成真实性鉴定,我国急需一种可以快速准确鉴定甘蓝作物品种的方法。根据《非主要农作物品种登记指南》的要求,品种特性说明和品种dus测试报告中涉及的有关性状如有明确关联基因,可以直接提交dna检测结果。因此,我国急需建立与当前甘蓝品种管理需求相匹配的dna快速检测技术。近年来,ssr分子标记以其数量多、变异丰富、遗传稳定等优点广泛应用在品种真实性鉴定方面。目前,我国甘蓝品种鉴定的dna分子检测主要依据ssr分子标记。但是,当时的ssr引物研发没有相关参考基因组与变异组数据支撑,对扩增片段及ssrmotif内部的序列真实变异情况没有掌握,存在不真实变异情况;ssr引物筛选所用的品种数量较少,且不能代表我国当前市场的销售品种。因此现行标准容易造成扩增不成功,真实性、稳定性不佳等问题,进而造成分子鉴定判定结果呈现出不同程度的假阳性、假阴性误判。因此,在利用ssr分子标记鉴定甘蓝品种的工作中,需要一种基于大规模基因组重测序和变异组数据分析的、稳定高效的ssr位点和测定方法。技术实现要素:本发明提供了一种鉴定甘蓝品种真实性的方法及其专用ssr引物组合,可以得出稳定、高效的鉴定结果:待测甘蓝品种是否属于标准甘蓝品种中的某一种,以及具体是哪一种。本发明是通过以下的技术方案实现的:鉴定甘蓝品种真实性的ssr位点,所述ssr位点选自如下第一ssr位点到第二十ssr位点中的任1到20种:第一ssr位点,位于甘蓝参考基因组第1染色体第28107690-28107707位,或其种间同源基因组片段;第二ssr位点,位于甘蓝参考基因组第3染色体第1716131-1716145位,或其种间同源基因组片段;第三ssr位点,位于甘蓝参考基因组第7染色体第45714507-45714532位,或其种间同源基因组片段;第四ssr位点,位于甘蓝参考基因组第3染色体第4812097-4812110位,或其种间同源基因组片段;第五ssr位点,位于甘蓝参考基因组第6染色体第22832474-22832503位,或其种间同源基因组片段;第六ssr位点,位于甘蓝参考基因组第8染色体第41048816-41048837位,或其种间同源基因组片段;第七ssr位点,位于甘蓝参考基因组第9染色体第554615-554630位,或其种间同源基因组片段;第八ssr位点,位于甘蓝参考基因组第2染色体第51127987-51128007位,或其种间同源基因组片段;第九ssr位点,位于甘蓝参考基因组第3染色体第51332896-51332919位,或其种间同源基因组片段;第十ssr位点,位于甘蓝参考基因组第3染色体第57313406-57313429位,或其种间同源基因组片段;第十一ssr位点,位于甘蓝参考基因组第5染色体第2844992-2845013位,或其种间同源基因组片段;第十二ssr位点,位于甘蓝参考基因组第7染色体第24720924-24720947位,或其种间同源基因组片段;第十三ssr位点,位于甘蓝参考基因组第5染色体第4488018-4488029位,或其种间同源基因组片段;第十四ssr位点,位于甘蓝参考基因组第5染色体第42709888-42709899位,或其种间同源基因组片段;第十五ssr位点,位于甘蓝参考基因组第6染色体第25743451-25743474位,或其种间同源基因组片段;第十六ssr位点,位于甘蓝参考基因组第1染色体第39859153-39859176位,或其种间同源基因组片段;第十七ssr位点,位于甘蓝参考基因组第8染色体第40732487-40732501位,或其种间同源基因组片段;第十八ssr位点,位于甘蓝参考基因组第7染色体第43515151-43515168位,或其种间同源基因组片段;第十九ssr位点,位于甘蓝参考基因组第9染色体第11333972-11333983位,或其种间同源基因组片段;第二十ssr位点,位于甘蓝参考基因组第1染色体第13200279-13200299位,或其种间同源基因组片段。所述参考基因组为甘蓝参考基因组brassicaolereceav2.1。鉴定甘蓝品种真实性的ssr引物组,所述ssr引物组用于分别扩增所述的ssr位点,所述ssr引物组包括:第一ssr引物对,用于扩增所述第一ssr位点;第二ssr引物对,用于扩增所述第二ssr位点;第三ssr引物对,用于扩增所述第三ssr位点;第四ssr引物对,用于扩增所述第四ssr位点;第五ssr引物对,用于扩增所述第五ssr位点;第六ssr引物对,用于扩增所述第六ssr位点;第七ssr引物对,用于扩增所述第七ssr位点;第八ssr引物对,用于扩增所述第八ssr位点;第九ssr引物对,用于扩增所述第九ssr位点;第十ssr引物对,用于扩增所述第十ssr位点;第十一ssr引物对,用于扩增所述第十一ssr位点;第十二ssr引物对,用于扩增所述第十二ssr位点;第十三ssr引物对,用于扩增所述第十三ssr位点;第十四ssr引物对,用于扩增所述第十四ssr位点;第十五ssr引物对,用于扩增所述第十五ssr位点;第十六ssr引物对,用于扩增所述第十六ssr位点;第十七ssr引物对,用于扩增所述第十七ssr位点;第十八ssr引物对,用于扩增所述第十八ssr位点;第十九ssr引物对,用于扩增所述第十九ssr位点;第二十ssr引物对,用于扩增所述第二十ssr位点。在一些实施方式中,所述ssr引物组,包括:所述第一ssr引物对,与序列表中的seqidno:1和seqidno:2的核苷酸序列的同一性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;所述第二ssr引物对,与序列表中的seqidno:3和seqidno:4的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;所述第三ssr引物对,与序列表中的seqidno:5和seqidno:6的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;所述第四ssr引物对,与序列表中的seqidno:7和seqidno:8的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;所述第五ssr引物对,与序列表中的seqidno:9和seqidno:10的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;所述第六ssr引物对,与序列表中的seqidno:11和seqidno:12的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;所述第七ssr引物对,与序列表中的seqidno:13和seqidno:14的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;所述第八ssr引物对,与序列表中的seqidno:15和seqidno:16的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;所述第九ssr引物对,与序列表中的seqidno:17和seqidno:18的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;所述第十ssr引物对,与序列表中的seqidno:19和seqidno:20的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;所述第十一ssr引物对,与序列表中的seqidno:21和seqidno:22的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;所述第十二ssr引物对,与序列表中的seqidno:23和seqidno:24的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;所述第十三ssr引物对,与序列表中的seqidno:25和seqidno:26的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;所述第十四ssr引物对,与序列表中的seqidno:27和seqidno:28的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;所述第十五ssr引物对,与序列表中的seqidno:29和seqidno:30的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;所述第十六ssr引物对,与序列表中的seqidno:31和seqidno:32的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;所述第十七ssr引物对,与序列表中的seqidno:33和seqidno:34的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;所述第十八ssr引物对,与序列表中的seqidno:35和seqidno:36的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;所述第十九ssr引物对,与序列表中的seqidno:37和seqidno:38的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;所述第二十ssr引物对,与序列表中的seqidno:39和seqidno:40的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;优选地,每对所述引物对的一条引物连接有荧光分子,更优选,所述荧光分子选自rox、tamra、fam、hex。鉴定甘蓝品种真实性的ssr试剂盒,所述ssr试剂盒配制为pcr反应体系;所述pcr反应体系包括:所述ssr引物组,优选地,所述ssr引物组中的每一对的上游引物和下游引物在所述体系中浓度之比为1:1;所述上游引物和下游引物在体系中终浓度均优选为0.25μmol/l;优选地,所述体系还包括:dntps:体系中终浓度为每种0.15mmol/l,氯化镁:体系中终浓度为2.5mmol/l,dna聚合酶:体系中终浓度为0.05u/μl,pcr缓冲液:由体系中终浓度为10-50mmol/l的氯化钾与体系中终浓度为1-10mmol/l的tris-hcl(ph7.5-9.0)配制而成。一种鉴定甘蓝品种真实性的检测方法,包括以下步骤:步骤一:检测待测甘蓝的如所述ssr位点的基因型;步骤二:所述待测甘蓝的品种判定:如果所述待测甘蓝基于所述20个ssr位点的基因型,和甘蓝标准品种中的某指定品种的基于所述20个ssr位点的基因型的差异位点数为0-2,则待测甘蓝与所述甘蓝标准品种的该指定品种判定为相似的品种;如果所述待测甘蓝基于所述20个ssr位点的基因型,和甘蓝标准品种中的某指定品种的基于所述20个ssr位点的基因型的差异位点数为大于2,则待测甘蓝与所述甘蓝标准品种的该指定品种判定为不同的品种;优选地,所述判定的结果是根据聚类分析得到的。在一些实施方式中,所述检测待测甘蓝的ssr位点基因型步骤包括以下分步骤:分步骤一:分别以所述待测甘蓝的基因组dna和甘蓝标准品种的基因组dna为模板,分别采用所述ssr引物组合中的引物组的进行pcr扩增,得到pcr扩增产物;分步骤二:对所述pcr扩增产物进行检测,获得所述待测甘蓝和所述甘蓝标准品种基于20个所述ssr位点的基因型。在一些实施方式中,所述分步骤二的检测方法包括:荧光信号检测:检测所述pcr扩增产物的荧光信号,获得所述待测甘蓝和所述标准甘蓝品种基于所述20个ssr位点的基因型;或:扩增产物片段检测:检测所述pcr扩增产物的片段大小,获得待测甘蓝和标准甘蓝品种基于所述20个ssr位点的基因型。在一些实施方式中,所述标准甘蓝品种选自以下78个甘蓝品种:中甘21,苏甘8号,早夏16号,铁头八号,西园10号,苏甘19,瑞甘50,黔甘3号,华耐0903,瑞甘48,春甘2号,宿甘六号,中甘22,苏甘20,秀美,寒美,先甘382,探春,苏甘3号,华耐g1103,瑞甘55,瑞甘21,瑞甘37,苏甘27,福兰,华耐g1202,y2,绿宝石,锦绣,碧浪,华耐g1301,满月55,满月56,苏甘65,瑞甘22,名图,瑞甘16,瑞甘17,千美5号,双环56,满月51,铁头46,满月4号,先甘097,先甘011,冬丽42,秦甘1652,双环49,双环50,欧美来,中甘15号,中甘21号,中甘101号,京丰一号,先甘101,中甘56号,中甘192号,中甘103号,中甘301号,甘蓝628号,中甘19号,中甘96号,中甘011号,中甘588号,中甘596号,晚丰,中甘18号,中甘9号,怡春,夏光,争春,8398,惠丰7号,西园4号,春丰,惠丰5号,惠丰4号,苏晨1号。一种鉴定甘蓝品种是否相同的的检测方法,待测甘蓝为两种未知品种的甘蓝;所述检测方法包括:步骤一:检测所述待测甘蓝的所述ssr位点的基因型;步骤二:所述待测甘蓝的品种是否相同的判定:如果所述待测甘蓝基于所述20个ssr位点的基因型的差异位点数为0-2,则待测甘蓝判定为相似的品种;如果所述待测甘蓝基于所述20个ssr位点的基因型的差异位点大于2,则待测甘蓝判定为不同的品种。所述ssr位点,或所述ssr引物组合,或所述ssr试剂盒,或所述检测方法,在以下x1或x2或x3中的应用:x1:鉴定待测甘蓝的品种是否属于标准甘蓝品种中的某一种;x2:鉴定待测甘蓝的品种具体为标准甘蓝品种中的哪一种;x3:鉴定待测甘蓝样本是否为相同的品种。本发明相比现有技术具有以下有益效果:1、本发明提供了一种可以快速,准确,稳定地进行品种真实性检测的ssr分子标记鉴定方法。本发明ssr引物组合可用于对甘蓝品种在种子或幼苗期进行早期鉴定,也可以进行自种子开始的全生命周期的真实性鉴定,保证品种的真实性,可以解决国内种子种苗市场的同名异种与同种异名问题,切实保护生产者和育种家的权益,并为甘蓝种质资源和新品种保护提供强有力的技术支持。2、本发明提供的方法可以鉴定得出:待测甘蓝品种是否属于标准甘蓝品种中的某一种,以及具体是哪一种。故此方法可以对已知品种进行真实性鉴定,也可以对有标准样品的未知甘蓝品种进行真实性鉴定;还可以鉴定两种未知的品种是否属于相似品种。3、本发明提供的方法具有高通量、准确、低成本、操作简单、节约人力、物力等优点,具有十分广阔的应用前景。4、本发明相比现有技术中其他ssr相关技术方案还有以下区别:①首先,植物大规模测序刚兴起没几年,之前的绝大部分ssr相关技术是没有参考基因组作为数据基础的,所以只能盲选,随机选,选出来的标记完全是看运气的,其潜在鉴别能力根本无从谈起。②本发明投入的大量劳动,对20份品种进行了重测序,并以这些数据作为数据基础,与参考基因组联合使用进行大数据分析,进行了海量的数据挖掘与计算,与其现有技术引用别人文献种使用过的标记或者从某些免费数据库选标记是有本质区别的。③本发明采用育种专家提供的品种作为验证材料,使ssr标记的鉴别能力有充分保障。④由于本发明使用的数据挖掘方法,所选出的ssr标记可以非常有效的代表全基因组信息,同时采用的甘蓝品种材料涵盖了所有市售品种的类型,故而本发明所选出的ssr标记具有非常强的潜在鉴别未知甘蓝品种的能力。5、本发明还有以下特点:①快速,通过本发明进行品种鉴定的时候,可以使用幼苗甚至种子直接提取的dna进行检测,区别于dus鉴定必须有成熟植株才能进行鉴定,将鉴定时间从几个月缩短到几个小时。②相对于表型鉴定方法,本发明基于dna检测的鉴定方法不受外在环境的影响,不会因环境条件的变化而变化,其结果稳定可靠。③用本发明的方法进行鉴定,在甘蓝的各个物候期进行鉴定,其结果稳定一致。④本发明的20个ssr位点均通过target-seq测序检测,3730荧光毛细管检测以及聚丙烯酰胺凝胶电泳检测,多平台检测结果的高度一致,充分证明了本发明各ssr位点的可靠性。⑤相对于dus测试,本发明的操作不需要长时间的经验积累。附图说明图1为实施例1中建立在20个引物组上的78个供试甘蓝品种的聚类图。图2-图21为实施例2中20个引物组在部分供试甘蓝品种中的ssr分型效果图。其中,图2:引物为bossr_2_001,采用的品种为中甘21,图3:引物为bossr_2_002,采用的品种为瑞甘48,图4:引物为bossr_2_016,采用的品种为早夏16号,图5:引物为bossr_2_021,采用的品种为秀美,图6:引物为bossr_2_038,采用的品种为京丰一号,图7:引物为bossr_2_042,采用的品种为晚丰,图8:引物为bossr_2_043,采用的品种为夏光,图9:引物为bossr_2_045,采用的品种为西园4号,图10:引物为bossr_2_051,采用的品种为苏晨1号,图11:引物为bossr_2_058,采用的品种为千美5号,图12:引物为bossr_2_061,采用的品种为中甘19,图13:引物为bossr_2_063,采用的品种为名图,图14:引物为bossr_2_080,采用的品种为满月55,图15:引物为bossr_2_084,采用的品种为探春,图16:引物为bossr_2_088,采用的品种为福兰,图17:引物为bossr_2_094,采用的品种为碧浪,图18:引物为bossr_2_108,采用的品种为锦绣,图19:引物为bossr_2_109,采用的品种为双环,图20:引物为bossr_2_110,采用的品种为铁头,图21:引物为bossr_2_114,采用的品种为先甘。图22为实施例2为ssr标记个数(即ssr位点个数)与区分78个供试甘蓝品种的差异标记图。具体实施方式定义如下:甘蓝品种真实性:本质上是指一个甘蓝品种与其遗传背景的真实对应性;在实际工作中,某供检品种是否具有真实性,是该指供检品种与文件记录(如品种说明书、标签等)是否相符。种间同源基因组片段:是指在brassicaoleracea参考基因组序列的版本号为v2.1之外,其他品种的甘蓝中与brassicaoleracea参考基因组序列的版本号为v2.1同源的基因组片段。比如,就特定基因组片段,在本发明的78个标准品种中与brassicaoleracea参考基因组序列的版本号为v2.1存在相同的基因组片段。第一方面,本发明提供鉴定甘蓝品种真实性的ssr位点,分别位于甘蓝基因组,数量为20个,可选用其中的1个或多个,具体信息如表1所示。上述ssr位点选自如下第一ssr位点到第二十ssr位点中的任1到20种:第一ssr位点,位于甘蓝参考基因组第1染色体第28107690-28107707位,或其种间同源基因组片段;第二ssr位点,位于甘蓝参考基因组第3染色体第1716131-1716145位,或其种间同源基因组片段;第三ssr位点,位于甘蓝参考基因组第7染色体第45714507-45714532位,或其种间同源基因组片段;第四ssr位点,位于甘蓝参考基因组第3染色体第4812097-4812110位,或其种间同源基因组片段;第五ssr位点,位于甘蓝参考基因组第6染色体第22832474-22832503位,或其种间同源基因组片段;第六ssr位点,位于甘蓝参考基因组第8染色体第41048816-41048837位,或其种间同源基因组片段;第七ssr位点,位于甘蓝参考基因组第9染色体第554615-554630位,或其种间同源基因组片段;第八ssr位点,位于甘蓝参考基因组第2染色体第51127987-51128007位,或其种间同源基因组片段;第九ssr位点,位于甘蓝参考基因组第3染色体第51332896-51332919位,或其种间同源基因组片段;第十ssr位点,位于甘蓝参考基因组第3染色体第57313406-57313429位,或其种间同源基因组片段;第十一ssr位点,位于甘蓝参考基因组第5染色体第2844992-2845013位,或其种间同源基因组片段;第十二ssr位点,位于甘蓝参考基因组第7染色体第24720924-24720947位,或其种间同源基因组片段;第十三ssr位点,位于甘蓝参考基因组第5染色体第4488018-4488029位,或其种间同源基因组片段;第十四ssr位点,位于甘蓝参考基因组第5染色体第42709888-42709899位,或其种间同源基因组片段;第十五ssr位点,位于甘蓝参考基因组第6染色体第25743451-25743474位,或其种间同源基因组片段;第十六ssr位点,位于甘蓝参考基因组第1染色体第39859153-39859176位,或其种间同源基因组片段;第十七ssr位点,位于甘蓝参考基因组第8染色体第40732487-40732501位,或其种间同源基因组片段;第十八ssr位点,位于甘蓝参考基因组第7染色体第43515151-43515168位,或其种间同源基因组片段;第十九ssr位点,位于甘蓝参考基因组第9染色体第11333972-11333983位,或其种间同源基因组片段;第二十ssr位点,位于甘蓝参考基因组第1染色体第13200279-13200299位,或其种间同源基因组片段。所述甘蓝参考基因组为brassicaolereceav2.1。第二方面,本发明提供鉴定甘蓝品种真实性的ssr引物组,可以通过pcr扩增反应得基于上述ssr位点的pcr扩增产物。上述ssr引物组合选自:第一ssr引物对至第二十ssr引物对,分别用于pcr扩增上述第一ssr位点至第二十ssr位点。上述第一ssr引物对,与序列表中的seqidno:1和seqidno:2的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;上述第二ssr引物对,与序列表中的seqidno:3和seqidno:4的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;上述第三ssr引物对,与序列表中的seqidno:5和seqidno:6的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;上述第四ssr引物对,与序列表中的seqidno:7和seqidno:8的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;上述第五ssr引物对,与序列表中的seqidno:9和seqidno:10的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;上述第六ssr引物对,与序列表中的seqidno:11和seqidno:12的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;上述第七ssr引物对,与序列表中的seqidno:13和seqidno:14的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;上述第八ssr引物对,与序列表中的seqidno:15和seqidno:16的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;上述第九ssr引物对,与序列表中的seqidno:17和seqidno:18的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;上述第十ssr引物对,与序列表中的seqidno:19和seqidno:20的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;上述第十一ssr引物对,与序列表中的seqidno:21和seqidno:22的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;上述第十二ssr引物对,与序列表中的seqidno:23和seqidno:24的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;上述第十三ssr引物对,与序列表中的seqidno:25和seqidno:26的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;上述第十四ssr引物对,与序列表中的seqidno:27和seqidno:28的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;上述第十五ssr引物对,与序列表中的seqidno:29和seqidno:30的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;上述第十六ssr引物对,与序列表中的seqidno:31和seqidno:32的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;上述第十七ssr引物对,与序列表中的seqidno:33和seqidno:34的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;上述第十八ssr引物对,与序列表中的seqidno:35和seqidno:36的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;上述第十九ssr引物对,与序列表中的seqidno:37和seqidno:38的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%;上述第二十ssr引物对,与序列表中的seqidno:39和seqidno:40的核苷酸序列的同源性大于等于85%、90%、95%、96%、97%、98%或99%,优选为100%。在优选的实施方式中,上述ssr引物组合选自引物组01-20中的一组或多组;上述引物组01-20的dna序列信息如序列表seqid:1-40所示,参见表2。上述引物组中,上游引物的5′端可带有荧光标签序列以便荧光pcr检测,例如fam荧光标签序列的荧光信号为蓝色,hex荧光标签序列的荧光信号为绿色。第三方面,本发明提供鉴定甘蓝品种真实性的ssr试剂盒,ssr试剂配制为pcr反应体系,该体系优选包括:上述ssr引物组,上游引物和下游引物的终浓度之比为1:1。第四方面,本发明提供一种鉴定甘蓝品种真实性的检测方法,包括以下步骤:步骤一:检测待测甘蓝的ssr位点基因型。分步骤一:分别以上述待测甘蓝的基因组dna和甘蓝标准品种的基因组dna为模板,分别采用上述ssr引物组合中的引物组的进行pcr扩增反应,得到pcr扩增产物;分步骤二:对上述pcr扩增产物进行检测,获得待测甘蓝和甘蓝标准品种基于20个上述ssr位点的基因型。所述检测可以为荧光信号检测:检测上述pcr扩增产物的荧光信号,获得待测甘蓝和标准甘蓝品种基于上述20个ssr位点的基因型;上述检测也可以为扩增产物片段检测:利用毛细管电泳检测上述pcr扩增产物的片段大小,获得待测甘蓝和标准甘蓝品种基于上述20个ssr位点的基因型。步骤二:待测甘蓝的品种判定步骤:通过聚类分析待测甘蓝和标准甘蓝品种,判断标准参考gb/t3543.1、gb/t3543.5、ny/t2594,基于上述20个ssr位点的基因型得出结论以下结果:如果所述待测甘蓝基于所述20个ssr位点的基因型,和甘蓝标准品种中的某指定品种的基于所述20个ssr位点的基因型的差异位点数为0-2,则待测甘蓝与所述甘蓝标准品种的该品种属于相似的品种;如果所述待测甘蓝基于所述20个ssr位点的基因型,和甘蓝标准品种中的某指定品种的基于所述20个ssr位点的基因型的差异位点数为大于2,则待测甘蓝与所述甘蓝标准品种的该指定品种判定为不同的品种。上述pcr扩增反应的程序优选为:94℃预变性5min;94℃变性30s,60℃退火45s,72℃延伸45s,每循环降0.8℃,共12个循环;94℃变性30s,50℃退火45s,72℃延伸45s,共25个循环;72℃终延伸10min。扩增产物电泳前于-20℃或冰上保存。上述标准菜心品种包括以下78个甘蓝品种:中甘21,苏甘8号,早夏16号,铁头八号,西园10号,苏甘19,瑞甘50,黔甘3号,华耐0903,瑞甘48,春甘2号,宿甘六号,中甘22,苏甘20,秀美,寒美,先甘382,探春,苏甘3号,华耐g1103,瑞甘55,瑞甘21,瑞甘37,苏甘27,福兰,华耐g1202,y2,绿宝石,锦绣,碧浪,华耐g1301,满月55,满月56,苏甘65,瑞甘22,名图,瑞甘16,瑞甘17,千美5号,双环56,满月51,铁头46,满月4号,先甘097,先甘011,冬丽42,秦甘1652,双环49,双环50,欧美来,中甘15号,中甘21号,中甘101号,京丰一号,先甘101,中甘56号,中甘192号,中甘103号,中甘301号,甘蓝628号,中甘19号,中甘96号,中甘011号,中甘588号,中甘596号,晚丰,中甘18号,中甘9号,怡春,夏光,争春,8398,惠丰7号,西园4号,春丰,惠丰5号,惠丰4号,苏晨1号。第五方面,本发明提供一种鉴定甘蓝品种是否相同的的检测方法。其中,待测甘蓝为两种未知品种的甘蓝;该检测方法包括:步骤一:检测待测甘蓝的上述20个ssr位点的基因型;检测方法如第四方面所述。步骤二:待测甘蓝的品种(两个未知的甘蓝品种)是否相同的判定:如果两个未知的甘蓝品种基于上述20个ssr位点的基因型的差异位点数为0-2,则两个未知的甘蓝判定为相似的品种;如果两个未知的甘蓝品种基于所述20个ssr位点的基因型的差异位点大于2,则两个未知的甘蓝判定为不同的品种。第六方面,本发明提供上述ssr位点,ssr引物组合,ssr试剂盒,述检测方法,在以下x1或x2或x3中的应用:x1:鉴定待测甘蓝的品种是否属于标准甘蓝品种中的某一种;x2:鉴定待测甘蓝的品种具体为标准甘蓝品种中的哪一种;x3:鉴定待测甘蓝样本是否为相同的品种。x1、x2和x3均属于鉴定甘蓝品种真实性的应用。以下的实施例便于更好地理解本发明,但并不限定本发明。下述实施例中的实验方法,如无特殊说明,均为常规方法。下述实施例中所用的试验材料,如无特殊说明,均为自常规生化试剂商店购买得到的。实施例1用于鉴定甘蓝品种真实性的ssr引物组合的获得一、20个ssr位点的发现:这些位点是根据甘蓝的参考基因组brassicaolereceav2.1与20份种质资源全基因组重测序数据进行数据挖掘所获得的。1、区别于其他分子标记鉴定方法中,随机盲目选取ssr标记的方法,本发明使用甘蓝参考基因组,并使用独有的,具有代表性的,20份甘蓝品种重测序数据(重测序结果未公开),通过特定的大数据分析手段,对全基因组的ssr位点进行分析,通过pic、maf、序列保守性等一系列的筛选条件进行筛选,最终获得了一个ssr位点的集合,通过数据降维等手段选取出能充分代表本作物基因组的ssr位点。通过target-seq简化测序技术,abi3730荧光毛细管电泳等不同检测手段进行验证,最终结合20份实际材料选出了20个鉴别能力最强的ssr位点作为本发明中所使用的ssr位点。本发明基于20份甘蓝代表资源的重测序数据,首次发现并获得20个ssr位点。这20份甘蓝资源类型丰富,涵盖了市场上现有的甘蓝主要的生态类型与农艺性状,尽可能多地体现了种质代表性,具有较高的遗传多样性。2、本发明的ssr位点的筛选包括以下步骤:首先通过20份甘蓝材料的重测序结果,与brassicaoleracea参考基因组v2.1序列进行比对一共找到10119个ssr位点,筛选pic大于0.35的之后剩余7804个ssr位点,去除碱基重复次数小于5的剩余4784个ssr位点,去除缺失比率大于10%的剩余3721个ssr位点,选取基因型个数小于等于4,长度大于等于3的,剩余231个ssr位点,最后按照基因组位置均匀分布选取ssr位点,最终剩余128个ssr位点。对78个品种(参见实施例2)使用这128对ssr引物进行扩增后,通过targetseq技术获得其ssr序列信息,并进行数据分析,去掉缺失比例大于10%的ssr位点数据剩余103个ssr位点作为备选位点。再使用minimalmarker算法选出20个ssr位点,此算法优先考虑其对78个甘蓝品种的鉴别能力,并综合考虑ssr位点多态性,均匀分布于10对染色体等条件。为了确定这20个ssr位点鉴别能力的可靠性,发明人重新设计适用于荧光毛细管电泳检测的引物,使用这20对引物对78个甘蓝品种进行了pcr扩增,并使用ab3730荧光毛细管电泳系统进行片段长度检测,其结果证明所选20个ssr位点具有非常高的鉴别能力与可靠性。3、具体的,ssr位点的筛选标准如下:在全基因组范围内选择位置均匀、多态性好、杂合度小、maf>0.3、pca聚类效果好、区分度高且两翼150bp序列保守(无indel,无snp,无其它ssr)的ssr位点。20个ssr位点的基本信息详见表1。其中ssr位点在染色体上的位置和motif信息是基于甘蓝参考基因组序列比对确定的,所述甘蓝brassicaolerecea参考基因组序列的版本号为v2.1,网址参见:网址1:ftp://ftp.ensemblgenomes.org/pub/plants/release-38/fasta/brassica_oleracea/dna/:gff3;网址2:ftp://ftp.ensemblgenomes.org/pub/plants/release-38/gff3/brassica_oleracea/。表1中pic值和主要等位变异扩增长度是根据实施例2的78个品种得到的。表1.20个ssr位点的基本信息二、用于鉴定甘蓝品种真实性的ssr引物组合的获得基于步骤一发现的20个ssr位点,本发明的发明人开发了具有较高的多态性信息量(即pic值,pic值指的是一个标记,用于在群体检测多态性的价值;pic值取决于检测的等位基因的数目和等位基因它们的频率分布;pic值等于1减去所有等位基因频率的平方的总和)的用于鉴定甘蓝品种真实性的ssr引物组合。基于前述ssr位点在甘蓝brassicaolerecea参考基因组序列的版本号为v2.1中的上下游序列设计引物,ssr引物组合由20个引物组组成。每个引物组由2条引物序列组成,用于扩增一个ssr位点。20个引物组中各个引物的核苷酸序列如表2。表2.20个引物组的信息实施例2本实施例为实施例1开发的ssr引物组合的有效性检验。本实施例的78个供试甘蓝品种均为常见的优良品种或部分国外引进品种,来源均为北京市农林科学院蔬菜中心种质库。具体品种如下:编号品种名称来源编号品种名称来源gl01中甘21蔬菜中心种质库gl41双环56蔬菜中心种质库gl02苏甘8号蔬菜中心种质库gl43满月51蔬菜中心种质库gl03早夏16号蔬菜中心种质库gl44铁头46蔬菜中心种质库gl04铁头八号蔬菜中心种质库gl45满月4号蔬菜中心种质库gl05西园10号蔬菜中心种质库gl46先甘097蔬菜中心种质库gl06苏甘19蔬菜中心种质库gl47先甘011蔬菜中心种质库gl07瑞甘50蔬菜中心种质库gl48冬丽42蔬菜中心种质库gl08黔甘3号蔬菜中心种质库gl49秦甘1652蔬菜中心种质库gl09华耐0903蔬菜中心种质库gl50双环49蔬菜中心种质库gl10瑞甘48蔬菜中心种质库gl51双环50蔬菜中心种质库gl11春甘2号蔬菜中心种质库gl52欧美来蔬菜中心种质库gl12宿甘六号蔬菜中心种质库gl54中甘15号蔬菜中心种质库gl1-2中甘22蔬菜中心种质库gl55中甘21号蔬菜中心种质库gl13苏甘20蔬菜中心种质库gl56中甘101号蔬菜中心种质库gl14秀美蔬菜中心种质库gl56-2京丰一号蔬菜中心种质库gl15寒美蔬菜中心种质库gl57先甘101蔬菜中心种质库gl16先甘382蔬菜中心种质库gl58中甘56号蔬菜中心种质库gl17探春蔬菜中心种质库gl59中甘192号蔬菜中心种质库gl18苏甘3号蔬菜中心种质库gl60中甘103号蔬菜中心种质库gl19华耐g1103蔬菜中心种质库gl61中甘301号蔬菜中心种质库gl20瑞甘55蔬菜中心种质库gl62甘蓝628号蔬菜中心种质库gl21瑞甘21蔬菜中心种质库gl63中甘19号蔬菜中心种质库gl22瑞甘37蔬菜中心种质库gl64中甘96号蔬菜中心种质库gl23苏甘27蔬菜中心种质库gl65中甘011号蔬菜中心种质库gl24福兰蔬菜中心种质库gl66中甘588号蔬菜中心种质库gl25华耐g1202蔬菜中心种质库gl67中甘596号蔬菜中心种质库gl26y2蔬菜中心种质库gl68晚丰蔬菜中心种质库gl27绿宝石蔬菜中心种质库gl69中甘18号蔬菜中心种质库gl28锦绣蔬菜中心种质库gl70中甘9号蔬菜中心种质库gl29碧浪蔬菜中心种质库gl71怡春蔬菜中心种质库gl30华耐g1301蔬菜中心种质库gl72夏光蔬菜中心种质库gl33满月55蔬菜中心种质库gl74争春蔬菜中心种质库gl34满月56蔬菜中心种质库gl758398蔬菜中心种质库gl35苏甘65蔬菜中心种质库gl76惠丰7号蔬菜中心种质库gl36瑞甘22蔬菜中心种质库gl77西园4号蔬菜中心种质库gl37名图蔬菜中心种质库gl78春丰蔬菜中心种质库gl38瑞甘16蔬菜中心种质库gl79惠丰5号蔬菜中心种质库gl39瑞甘17蔬菜中心种质库gl80惠丰4号蔬菜中心种质库gl40千美5号蔬菜中心种质库gl81苏晨1号蔬菜中心种质库1、供试甘蓝品种的基因组dna的获得采用ctab法分别提取78个供试甘蓝品种的叶片(混取30粒种子的真叶,即每个品种取30粒种子长出来的真叶,指的是:同一品种取30个不同植株的真叶混合)的基因组dna,得到供试甘蓝品种的基因组dna。上述ctab法的操作具体为:分别摘取幼苗期的上述78个品种的叶片,置于冷冻干燥仪(coolsafe55-4)中脱水;然后用高通量研磨仪(geno/grind6875)打碎叶片,再取200mg叶片干粉,向其中加入800μlctab提取液(2%ctab,1.4mmnacl,100mmtris-hclph8.0,20mmedtaph8.0,1%pvp-40,0.2%β-巯基乙醇),混匀,于65℃水浴30min,加入等体积的氯仿/异戊醇(v:v=24:1),在10000rpm/min条件下离心10分钟,吸取上清液转移至新的离心管中,加入0.8倍体积预冷的异丙醇,轻轻颠倒混匀,-20℃放置30min后在4℃、12,000r/min离心10min。弃上清液,加入70%乙醇溶液洗涤2遍,自然条件下干燥加100μlddh2o溶解dna,得到供试甘蓝品种基因组dna,检测浓度后4℃备用。供试甘蓝品种的基因组dna的质量和浓度均须满足pcr要求,达标标准为:紫外分光光度计nanodrop2000(thermo)检测a260/a280比值在1.8左右,a260/a230比值大于1.8;供试甘蓝品种的基因组dna的浓度在30-50ng/μl。2、分别以78个供试甘蓝品种的基因组dna为模板,分别采用20个引物组进行pcr扩增,得到pcr扩增产物。各个pcr反应体系中,名称中含有“f”的引物和名称中含有“r”的引物浓度比为1:1。反应体系包括:上游引物(名称中含有“f”)和下游引物(名称中含有“r”)在体系中浓度之比为1:1。反应程序为:预变性:94℃5min;扩增:94℃变性30s,60℃退火45s,72℃延伸45s,每循环降0.8℃,共12个循环;94℃变性30s,50℃退火45s,72℃延伸45s,共25个循环;终延伸:72℃10min。形成的扩增产物电泳前于4℃保存。3、荧光毛细管电泳完成步骤2后,根据ssr分子标记扩增片段大小的不同,可以根据不同仪器选用多个引物组合进行电泳。按照预先确定的组合引物,分别取等体积同一组合引物的不同荧光标记的扩增产物,tamra稀释50倍,其它荧光产物稀释100倍后充分混匀。从混合液中吸取1μl,加入到dna分析仪专用上样板孔中。各孔再分别加入0.1μl分子量内标和8.9μl去离子甲酰胺,在pcr仪上95℃变性5min,取出立即置-20℃冰箱内或冰上,冷却5min。瞬时离心10s后置放到dna分析仪上。开始检测。部分结果见图2-21。结果表明,各个引物组在供试甘蓝品种中可以得到很好的分型效果。4、聚类分析根据78个供试甘蓝品种基于20个ssr位点的基因型,利用mega7软件对78个供试甘蓝品种进行聚类分析。建立在20个引物组上的78个供试甘蓝品种的聚类图如图1所示。结果表明,20个引物组可以完全区分78个供试甘蓝品种。由此可见,实施例1开发的ssr引物组合可以应用于甘蓝品种dna指纹数据库构建和品种真实性鉴定。5、效率评价品种真实性鉴定可以采用序贯分析方式减少工作量。本发明的发明人比较了ssr标记个数(即引物组数)与区分78个供试甘蓝品种区分率的关系。如图22所示的78个品种两两之间比较并统计的差异标记数,其中两两之间比较的结果的个数为个;在这些结果中,差异位点个数为3的约占总数的2.0%,差异位点个数为4的约占总数的3.0%,差异位点个数为5的约占总数的3.4%,差异位点个数为6的约占总数的3.7%,差异位点个数为7的约占总数的4.1%,差异位点个数为8的约占总数的5.6%,差异位点个数为9的约占总数的8.3%,差异位点个数为10的约占总数的12.4%,差异位点个数为11的约占总数的15.6%,差异位点个数为12的约占总数的14.9%,差异位点个数为13的约占总数的11.8%,差异位点个数为14的约占总数的7.9%,差异位点个数为15的约占总数的4.4%,差异位点个数为16的约占总数的1.8%,差异位点个数为17的约占总数的0.4%;此结果说明用这些标记在78个品种的多态性好;20个引物组(即20个ssr标记个数)在78个供试大白菜品种中的区分率达到100%。实施例3本实施例是检测待测甘蓝品种是否属于78个供试甘蓝品种中的品种的方法,该待测甘蓝的品种未知,需要经过本实施例的检测方法得到该待测甘蓝的品种是否是这78个品种之一。1、待测甘蓝品种的基因组dna的获得待测甘蓝品种的叶片取自北京市农林科学院蔬菜研究中心试验基地。按照实施例2中的步骤1的方法,将“供试甘蓝品种的叶片”替换为“待测甘蓝品种的叶片”,其它步骤均不变,得到待测甘蓝品种的基因组dna。2、ssr引物及pcr反应体系的配置按照实施例2中的步骤2的方法,将“供试甘蓝品种的基因组dna”替换为“待测甘蓝品种的基因组dna”,其它步骤均不变,得到待测甘蓝品种的pcr产物。3、荧光毛细管电泳检测取待测甘蓝品种的pcr产物。将待测甘蓝品种的20个ssr扩增产物的片段大小分别与实施例2的78个供试甘蓝品种的20个ssr位点进行对比,统计待测甘蓝品种和20个标准甘蓝品种的差异位点数,然后进行如下判断:如果待测甘蓝品种与某标准甘蓝品种的差异位点数为2个以上,则待测甘蓝品种和该标准甘蓝品种判定为不同的甘蓝品种;差异位点数越多,遗传亲缘关系越远。如果待测甘蓝品种与某标准甘蓝品种的差异位点数为0-2,则待测甘蓝品种和该标准甘蓝品种判定为相似的甘蓝品种。结果表明,待测甘蓝品种在20个ssr位点上与78个供试甘蓝品种的差异位点数均为2个以上,因此,待测甘蓝品种不属于78个供试甘蓝品种中的任何一个品种,即待测甘蓝品种并不是上述78个供试甘蓝品种之一。实施例4本实施例是采用毛细管电泳对比片段大小来判定甘蓝品种,而不是采用荧光信号判定。本案例是以abi3730荧光毛细管检测平台为参考,如使用其他平台则根据设备操作要求做相应调整。根据ssr分子标记扩增片段大小的不同,可以根据不同仪器选用多个引物组合进行电泳。s1:按照预先确定的组合引物,分别取等体积同一组合引物的不同荧光标记的扩增产物,tamra稀释50倍,其它荧光产物稀释100倍后充分混匀。从混合液中吸取1μl,加入到dna分析仪专用上样板孔中。各孔再分别加入0.1μl分子量内标和8.9μl去离子甲酰胺,在pcr仪上95℃变性1min,取出立即置冰上,冷却5min。瞬时离心10s后置放到dna分析仪上。s2:打开abi3730dna分析仪,检查仪器工作状态和试剂状态。将装有样品的上样板放置于样品架基座上,将装有电极缓冲液的buffer板放置于buffer板架基座上,打开数据收集软件,按照dna分析仪的使用手册进行操作。dna分析仪将自动运行参数,并保存电泳原始数据。检测荧光引物所用激发波长及颜色参照仪器默认值。s3:导出电泳原始数据文件,采用数据分析软件按照下列步骤进行数据甄别:在数据分析软件中预先设置好ssr引物名称及荧光类别、分子量内标、相应引物的扩增片段大小;将电泳原始数据文件导入分析软件,选择panel、分子量内标、bin、质量控制参数等进行分析;分析软件会对检测质量赋以颜色标志进行评分,绿色表示质量可靠无需干预,红色表示质量不过关或未落入规定片段大小范围内,黄色表示有疑问需要查验原始图像进行确认。s4:分别通过同时进行试验的标准样品、参照样品(依据引物选择少量的对照),校准不同电泳板间的数据偏差后再读取扩增片段大小。甄别后的特异峰如果落入规定的片段大小范围内,则直接读取扩增片段大小;若其峰大多不在规定范围内,可将其整体平移尽量使峰落设置范围内后读取数据。s5:将待测甘蓝品种的20个ssr扩增产物的片段大小分别与78个供试甘蓝品种的20个ssr位点进行对比,统计待测甘蓝品种和20个标准甘蓝品种的差异位点数,然后进行如下判断:如果待测甘蓝品种与某标准甘蓝品种的差异位点数为2个以上,则待测甘蓝品种和该标准甘蓝品种判定为不同的甘蓝品种;差异位点数越多,遗传亲缘关系越远。如果待测甘蓝品种与某标准甘蓝品种的差异位点数为0-2个,则待测甘蓝品种和该标准甘蓝品种判定为甘蓝相似的品种。实施例5本实施例是检测两个未知的甘蓝品种是否为同一种,本实施例的待测甘蓝品种为两个未知的甘蓝品种。1、待测甘蓝品种的基因组dna的获得分别取两个待测甘蓝品种的叶片,采用ctab法分别提取这两个待测甘蓝品种的基因组dna,该ctab法的操作见实施例2,得到待测甘蓝品种的基因组dna。2、ssr引物及pcr反应体系的配置按照实施例2中的步骤2的方法,将“供试甘蓝品种的基因组dna”替换为“待测甘蓝品种的基因组dna”,其它步骤均不变,得到待测甘蓝品种的pcr产物。3、荧光毛细管电泳检测和数据记录步骤2的pcr反应完成后,对pcr产物根据不同引物的主要等未变异扩增长度的不同,结合检测设备进行分组电泳,检测其pcr产物的片段大小并进行数据记录,其记录方法为:某样品在某个位点仅出现1个等位变异,大小为150bp,则在该位点的主要等位变异的基因型写作150/150;某样品在某个位点有两个等位变异,大小分别为141bp、150bp,则在该位点的主要等位变异的基因型写作141/150。4、在步骤3中分别获得两个未知甘蓝品种(待测甘蓝品种)的20个ssr位点数据,分别对比两个未知甘蓝品种在相同上述20个ssr位点中每个ssr位点上的数据,记录有差异位点的个数。如果两个未知甘蓝品种在上述20个ssr位点的差异位点数为2个以上,则两个未知甘蓝品种判定为不同的甘蓝品种;差异位点数越多,遗传亲缘关系越远;如果两个未知甘蓝品种在上述20个ssr位点的异位点数为0-2个,则两个未知甘蓝品种判定为相似的甘蓝品种。最后所应说明的是,以上具体实施方式仅用以说明本发明的技术方案而非限制,尽管参照较佳实施例对本发明进行了详细说明,本领域的普通技术人员应当理解,可以对本发明的技术方案进行修改或者等同替换,而不脱离本发明技术方案的精神和范围,其均应涵盖在本发明的权利要求范围当中。序列表<110>北京市农林科学院<120>一种鉴定甘蓝品种真实性的方法及其专用ssr引物组合<141>2020-02-28<160>40<170>siposequencelisting1.0<210>1<211>34<212>dna<213>artificialsequence<400>1aagaggtaatttaaataaactcttaaattttgaa34<210>2<211>25<212>dna<213>artificialsequence<400>2tctaatcataatatgaggtgggcgt25<210>3<211>29<212>dna<213>artificialsequence<400>3tcaaaagtaaagaaactttgaaagaagca29<210>4<211>25<212>dna<213>artificialsequence<400>4ggtttgatgaagcattctgagacaa25<210>5<211>23<212>dna<213>artificialsequence<400>5ctgagttctcgctttcactctct23<210>6<211>25<212>dna<213>artificialsequence<400>6tgcgcaacaaaagagatgataacaa25<210>7<211>21<212>dna<213>artificialsequence<400>7gctcgtgcagattcgaaatcg21<210>8<211>25<212>dna<213>artificialsequence<400>8cacgcgattcaaaagagtccattat25<210>9<211>25<212>dna<213>artificialsequence<400>9ccacacatgcaacatactcttcttt25<210>10<211>25<212>dna<213>artificialsequence<400>10tcccatatagaatcaatccttccct25<210>11<211>25<212>dna<213>artificialsequence<400>11cagaacctatcgtgtgtgtttcttg25<210>12<211>25<212>dna<213>artificialsequence<400>12cgagtagtagagagtgaaactttcg25<210>13<211>27<212>dna<213>artificialsequence<400>13tgaagaaatagaagaaactgatacaca27<210>14<211>25<212>dna<213>artificialsequence<400>14atacggtcagttattcttcggaata25<210>15<211>25<212>dna<213>artificialsequence<400>15tgctcgaaaaccttaaattccaaca25<210>16<211>25<212>dna<213>artificialsequence<400>16acggagatggatcggaacttattag25<210>17<211>25<212>dna<213>artificialsequence<400>17agtttgcagaatcaaaagagagagc25<210>18<211>25<212>dna<213>artificialsequence<400>18tgaagctagttccgttacagatcaa25<210>19<211>25<212>dna<213>artificialsequence<400>19tacttttgataggatgcgtgaatca25<210>20<211>25<212>dna<213>artificialsequence<400>20catgcaactgagaccataacatgta25<210>21<211>25<212>dna<213>artificialsequence<400>21aagggatgtgatgaaggtaacatga25<210>22<211>25<212>dna<213>artificialsequence<400>22gcttcatttcctagtaattgggtgg25<210>23<211>25<212>dna<213>artificialsequence<400>23atgactaagtcaacttgtcagacga25<210>24<211>25<212>dna<213>artificialsequence<400>24tagtttatatagacacccagcccag25<210>25<211>25<212>dna<213>artificialsequence<400>25tcaaagggtctagaaatctcccaat25<210>26<211>23<212>dna<213>artificialsequence<400>26ggatgaggtcagctttgcatttt23<210>27<211>25<212>dna<213>artificialsequence<400>27aaacatcaccaccagatctcaatct25<210>28<211>25<212>dna<213>artificialsequence<400>28catcttggacactgcctctttaatg25<210>29<211>25<212>dna<213>artificialsequence<400>29agttacaagtaggtgacacaactca25<210>30<211>25<212>dna<213>artificialsequence<400>30gccagaagaagttagagagttttgt25<210>31<211>25<212>dna<213>artificialsequence<400>31tcgatcttctgttcttccaatttgg25<210>32<211>25<212>dna<213>artificialsequence<400>32taaaccaactctatcaatccggtga25<210>33<211>25<212>dna<213>artificialsequence<400>33catgaaatcgagaaacagaatccga25<210>34<211>25<212>dna<213>artificialsequence<400>34tgaagttgcctttgaaatcagtgaa25<210>35<211>22<212>dna<213>artificialsequence<400>35gtgaaccaactgtagcaaaccg22<210>36<211>25<212>dna<213>artificialsequence<400>36gcctgcacatacatatcaaaccata25<210>37<211>25<212>dna<213>artificialsequence<400>37gaagaacacgaagaagacaattggt25<210>38<211>25<212>dna<213>artificialsequence<400>38aaaagaaaacacaacaaagcaacgg25<210>39<211>24<212>dna<213>artificialsequence<400>39caaaacaacggggaattcacagag24<210>40<211>25<212>dna<213>artificialsequence<400>40ccaaccggttgaattgaatctatga25当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1