一种单细胞转录组测序文库的构建方法及其应用
1.本发明涉及生物技术领域,具体涉及单细胞测序领域,特别是涉及一种单细胞转录组测序文库的构建方法及其应用。
背景技术:
2.过去的十几年中,rna测序(rna-seq)技术飞速发展,已成为差异基因表达,mrna剪接分析等研究不可或缺的工具,在生物医学领域得到广泛应用。现有的rna-seq方法大都基于细胞群体,其结果仅反映了基因在多细胞群体中的平均表达水平,并不能呈现出每个细胞中基因表达的差异。近来发展起来的单细胞rna-seq是在单个细胞水平对rna进行测序的一项新技术,它不仅能有效解决组织样本细胞异质性和常规rna-seq所掩盖的细胞群体的转录组异质性难题,还为解析单细胞的行为、机制以及与机体的关系提供了新的研究方向,成为时下单细胞研究的一种新途径。
3.目前,单细胞rna文库构建的方法通常需要对单个细胞内极少量的rna进行预扩增,如将rna反转录成cdna,然后在其两端加上已知序列进行pcr扩增,或使用ivt技术对cdna进行体外转录扩增,但这两种建库方式都无法避免由pcr扩增引入的偏差,造成转录组覆盖不均一、背景噪声大以及定量不准确等问题;同样,这些方法会因某些转录本在扩增和捕获过程中丢失而造成基因dropout,特别是对于中低表达水平的基因,dropout现象会更加明显,而这些中低表达量的基因对于分析基因与基因之间共表达,以及中低表达基因对生物通路的贡献非常重要。大多数单细胞转录组测序方法如scrb-seq、drop-seq等虽然可以检测较多数量的细胞,但只能对转录本的3
′
端进行测序,每个细胞只能检测较少数量的基因;smart-seq方法可以利用模板转换对全长cdna进行扩增,提高了基因检出数量,但检测细胞数量少,步骤复杂,相较于其他方法成本较高。另外,传统的超声、酶切片段化文库构建方法,需要通过末端修复、加接头等复杂步骤实现文库构建。为了能够有效的获得的单个细胞的准确转录组特征,有必要开发一种新的单细胞转录组测序技术。
技术实现要素:
4.针对以上问题,本发明提供了一种单细胞转录组测序文库的构建方法及其应用,可以快速有效的对单细胞转录组进行建库,本发明利用tn5转座酶直接对双链cdna进行dna片段化和连接反应,无需对cdna进行pcr预扩增,可以快速高效的进行单细胞转录组测序文库的构建。本方法可检测到更多的基因,有效降低中低丰度基因dropout的比例,获得更准确的转录组信息,减少建库时间和成本。
5.为实现上述目的及其他相关目的,本发明第一方面提供一种单细胞转录组测序文库的构建方法,包括以下步骤:
6.(a)在孔板中对分选的单细胞进行裂解;
7.(b)利用逆转录引物进行逆转录合成第一链edna;
8.(c)利用置换合成反应合成第二链cdna;
transcription and tagmentation sequencing)技术,利用tn5转座酶直接对双链cdna进行酶切,无需对cdna模板进行pcr预扩增,可有效减少文库偏好性,获得更准确的转录组信息;使用in-house tn5转座酶将dna打断和接头连接同步完成,可减少起始模板量和样品处理时间,简化复杂的实验操作,有效提高建库效率,降低建库成本;本方法建库步骤简单,可检测更多的基因,有效降低中低丰度基因dropout的比例,更真实的体现单细胞基因表达情况,有利于在单细胞层面上进行更精细的研究,更加符合单细胞测序发展的要求。
附图说明
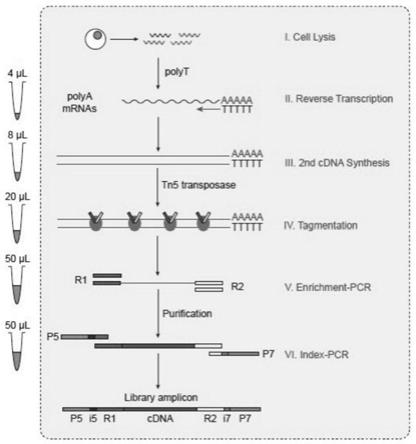
28.图1显示为本发明实施例的建库流程图。
29.图2显示为本发明实施例的文库片段分布图。
30.图3显示为本发明实施例与smart-seq2方法比较的基因数量分布小提琴图。
31.图4显示为本发明实施例与smart-seq2方法比较的测序饱和曲线图。
32.图5显示为本发明实施例与smart-seq2方法比较的基因dropout图。
具体实施方式
33.以下通过特定的具体实例说明本发明的实施方式,本领域技术人员可由本说明书所揭露的内容轻易地了解本发明的其他优点与功效。本发明还可以通过另外不同的具体实施方式加以实施或应用,本说明书中的各项细节也可以基于不同观点与应用,在没有背离本发明的精神下进行各种修饰或改变。
34.需要说明的是,本发明中的文库构建是基于小鼠单核巨噬细胞白血病细胞raw 264.7来进行的。
35.本发明中使用的主要试剂、仪器及材料如下表1中所示。
36.表1
[0037][0038]
具体实施过程如下:
[0039]
本实施例以96孔板的单细胞建库为例,如需对更多的单细胞进行建库,只需设计
合成更多的index pcr引物即可。通过以下步骤来具体说明本发明的单细胞转录组的建库方法:
[0040]
一、裂解细胞
[0041]
1、按照如下组成配制溶液,并将该溶液分配至96个孔中,溶液组成如下:
[0042]
1%ca630
-----------------------------------------
0.6μl
[0043]
rnase out核糖核酸酶抑制剂
------------------------
0.025μl
[0044]
无核酸酶水
---------------------------------------
1.175μμl
[0045]
脱氧核糖核苷三磷酸预混液
-------------------------
0.1μl
[0046]
逆转录引物(2μm)
----------------------------------
0.1μl
[0047]
总体积
-------------------------------------------
2μl
[0048]
其中,逆转录引物为按5’至3’依次为ttttttttttttttttttttttttttttttvn的固定结构,该固定结构用于与mrna的poly(a)尾进行特异性结合以保证逆转录的特异性。
[0049]
2、利用将raw 264.7细胞重悬至pbs溶液中,流式细胞仪分选单细胞至96孔板。
[0050]
3、冰上裂解15分钟。
[0051]
二、逆转录
[0052]
1、配制溶液,并将溶液分配至含裂解细胞的96个孔中,溶液组成如下:
[0053]
二硫苏糖醇(0.1m)
-------------------------------------
0.2μl
[0054]
superscript iii第一链缓冲液(5x)
----------------------
0.8μl
[0055]
rnase out核糖核酸酶抑制剂(40units/ul)
----------------
0.2μl
[0056]
superscript iii逆转录酶(200units/ul)
-----------------
0.2μl
[0057]
无核酸酶水
-------------------------------------------
0.6μl
[0058]
总体积
-----------------------------------------------
2μl
[0059]
2、进行逆转录反应:25℃10分钟,35℃10分钟,37℃10分钟,50℃60分钟,70℃15分钟,4℃保持。
[0060]
三、第二链cdna合成
[0061]
1、配制溶液,并将该溶液分配至含逆转录产物的96个孔中,溶液组成如下:
[0062]
nebnext第二链合成反应缓冲液
---------------------------
0.8μl
[0063]
nebnext第二链合成酶混合物
-----------------------------
0.4μl
[0064]
无核酸酶水
--------------------------------------------
2.8μl
[0065]
总体积
------------------------------------------------
4μl
[0066]
2、进行第二链cdna合成程序:16℃60分钟,4℃保持。
[0067]
四、双链cdna模板片段化
[0068]
1、配制溶液,并将该溶液分配至含双链cdna的96个孔中,溶液组成如下:
[0069]
5x反应缓冲液
--------------------------------------------
4μμl
[0070]
tn5转座酶
-----------------------------------------------
1μl
[0071]
无核酸酶水
----------------------------------------------
7μl
[0072]
总体积
--------------------------------------------------
l2μl
[0073]
其中,tn5转座酶为本实验室制备的in-house tn5转座酶,in-house tn5转座酶的制备方法如下:tn5重组质粒转化细菌,经iptg诱导表达,破碎菌体,使用ni柱纯化tn5转座
酶蛋白;组装后转移至蛋白超滤浓缩管中超滤纯化,获得高活性高纯度的tn5转座酶。
[0074]
2、进行片段化程序:55℃10分钟,10℃保持。
[0075]
3、完成片段化程序后,立即添加5μl0.2%sds终止反应,使用移液枪吹打混匀,并在室温下放置5分钟。
[0076]
五、pcr富集片段化的模板
[0077]
1、配制溶液,并将该溶液分配至含片段化模板的96个孔中,溶液组成如下:
[0078]
5xtrueprep缓冲液
----------------------------------
10μl
[0079]
trueprep扩增酶
------------------------------------
1μl
[0080]
read1引物(1μm)(seq id no:21)
----------------------
1μl
[0081]
read2引物(1μm)(seq id no:22)
----------------------
1μl
[0082]
无核酸酶水
----------------------------------------
12μl
[0083]
总体积
--------------------------------------------
25μl
[0084]
2、进行片段pcr富集程序:72℃3分钟,98℃30秒,25个循环(98℃15秒,60℃30秒,72℃3分钟),72℃5分钟,4℃保持。
[0085]
3、片段纯化:每孔添加50μl vahts dna纯化磁珠,使用移液枪吹打混匀,室温下放置5分钟。将样本放置在磁力架分钟后去除上清液。用100μl80%乙醇洗涤2次。添加23μl无核酸酶水,使用移液枪吹打混匀,室温下放置5分钟。
[0086]
六、indexpcr
[0087]
1、配制溶液,并将该溶液分配至含磁珠纯化模板的96个孔中,溶液组成如下:
[0088]
kapa热启动hifi高保真酶混合物
------------------------
25μl
[0089]
p5引物(1μm)(seq id no:1~8)
-------------------------
1μl
[0090]
p7引物(1μm)(seq id no:9~20)
------------------------
1μl
[0091]
总体积
----------------------------------------------
27μl
[0092]
其中,一个p5与一个p7组成一对引物,该引物适配于illumina测序平台测序,利用96对引物对96孔的细胞进行index标记。
[0093]
p5和p7的引物序列如下:
[0094]
p5:aatgatacggcgaccaccgagatctacac[index]tcgtcggcagcgtc,
[0095]
p7:caagcagaagacggcatacgagat[index]gtctcgtgggctcgg。
[0096]
其中index为长度为8bp的核酸序列,关于index的具体序列,本领域技术人员可以根据实际需要进行设计更多种类进行使用。
[0097]
本实施例中,p5和p7引物来自于illumina建库试剂盒,p5引物包括p501、p502、p503、p504、p505、p506、p507、p508,p501~p508的序列如seq id no.1~seq id no.8所示;p7引物包括p701、p702、p703、p704、p705、p706、p707、p708、p709、p710、p711、p712,p701~p712的序列如seq id no.9~seq id no.20所示。
[0098]
2、进行index pcr程序:95℃30秒,15个循环(98℃20秒,60℃15秒,72℃1分钟),72℃5分钟,4℃保持。
[0099]
3、吸出96孔板中的index pcr产物,使用qiaquick pcr纯化试剂盒进行纯化,获得illumina测序平台兼容的测序文库。
[0100]
七、对构建好的文库进行测序。
[0101]
1、文库纯化结束后,利用安捷伦fragment analyzer检测文库的片段分布,检测结果如图2所示。
[0102]
2、将上述所得的文库使用illumina x10平台进行测序,并与smart-seq2文库进行比较,通过生物信息学分析测序结果如图3、4、5所示。以上所用引物序列如表2所示。
[0103]
表2引物序列表
[0104][0105]
从图3可以看出,在raw 264.7细胞中可以找到平均多达9000个基因,平均比smart-seq2多3000个。
[0106]
从图4可以看出,尽管smart-seq2文库(蓝色曲线)在约50万reads/cell时达到饱和,scstat-seq文库(红色曲线)稍晚达到饱和;但在相同深度下,scstat-seq文库比smart-seq2文库平均多检测出约2000个基因。
[0107]
从图5可以看出,scstat-seq文库(蓝色曲线)在线性回归分析中表现出比smart-seq2文库(红色曲线)更低的dropout水平。
[0108]
综上所述,同现有的单细胞测序技术相比,本发明的scstat-seq(single-cell streamlined transcription and tagmentation sequencing)技术,利用tn5转座酶直接对双链cdna进行酶切,无需对cdna模板进行pcr预扩增,可有效减少文库偏好性,获得更准确的转录组信息;使用in-house tn5转座酶将dna打断和接头连接同步完成,可减少起始模板量和样品处理时间,简化复杂的实验操作,有效提高建库效率,降低建库成本;本方法建库步骤简单,可检测更多的基因,有效降低中低丰度基因dropout的比例,更真实的体现单细胞基因表达情况,有利于在单细胞层面上进行更精细的研究,更加符合单细胞测序发展的要求。
[0109]
上述实施例仅例示性说明本发明的原理及其功效,而非用于限制本发明。任何熟
悉此技术的人士皆可在不违背本发明的精神及范畴下,对上述实施例进行修饰或改变。因此,举凡所属技术领域中具有通常知识者在未脱离本发明所揭示的精神与技术思想下所完成的一切等效修饰或改变,仍应由本发明的权利要求所涵盖。
[0110]
[0111]
[0112]
[0113]
[0114]
[0115]
[0116]
[0117]
[0118]
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1