一种植物粒型相关蛋白及其编码基因与应用的制作方法
本发明属于基因工程领域,涉及一种植物株型生长发育相关蛋白及其编码基因与应用。
背景技术:
水稻是世界上重要的粮食作物,作为主食养育了世界上近一半的人口。伴随着世界人口的不断增长、自然环境的严重恶化和全球耕地面积持续减少,不断培育高产优质适应性好的水稻,对于保证全球粮食安全和维护世界和平稳定,具有重要意义。水稻的产量构成主要由穗粒数、每株穗数以及粒重等因素决定,其中影响粒重的主要依赖于种子粒型。种子粒型是一个复杂的性状,其中粒长,粒宽和粒厚是影响粒重的重要因素。种子大小及形状不但影响其外观品质,还对其品质性状包括蒸煮品质,加工品质等产生重要影响。以水稻为食的不同国家以及同一国家的不同地区会根据自身喜好和饮食习惯选择不同粒型的水稻品种。比如在欧洲和亚洲的大部分的国家喜爱细长的稻米,中国的南方也同样如此,但在中国的北方喜欢使用相对短圆的稻米。因此进一步阐明控制水稻粒型的分子机制与调控网络,利用重要的粒型基因进行育种应用,对于提高水稻产量和品质以及保证粮食安全具有重大意义。
目前在水稻中已经克隆了许多控制籽粒大小的关键基因,主要是参与泛素蛋白酶体信号途径,g蛋白信号通路,mapk信号和植物激素信号等途径,影响细胞分裂或者细胞大小来调控种子大小。大多数已被克隆基因主要是通过构建遗传群体检测qtls,进一步通过构建回交群体克隆基因,如gw2,gs3等;但也不乏许多基因通过物理诱变,化学诱变和t-dna插入等方法得到粒型发生变化的突变体,通过图位克隆,插入位点检测或全基因组重测序等方法获得基因。虽然克隆到许多粒型相关基因,但真正运用到生产实际中的还是较少,因此进一步发掘新的粒型基因,全面深入解析粒型的调控的分子机制显得尤为重要。
技术实现要素:
本发明的目的是一种植物粒型相关蛋白及其编码基因与应用。
本发明提供的水稻粒型相关蛋白ossg2,来源于水稻品种宁粳1号,是如下(a)或(b)的蛋白质:
(a)由序列表中seqidno.1所示的氨基酸序列组成的蛋白质;
(b)将seqidno.1的氨基酸序列经过一个或几个氨基酸残基的取代和/或缺失和/或添加且与粒型相关的由seqidno.1衍生的蛋白质。
序列表中的seqidno.1由810个氨基酸残基组成。
上述(b)中的ossg2可人工合成,也可先合成其编码基因,再进行生物表达得到。上述(b)中的ossg2的编码基因可通过将序列表中seqidno.2或seqidno.3所示的dna序列中缺失一个或几个氨基酸残基的密码子,和/或进行一个或几个碱基对的错义突变,和/或在其5′端和/或3′端连上表1所示的标签的编码序列得到。
编码上述粒型相关蛋白的基因(ossg2)也属于本发明的保护范围。
所述基因ossg2可为如下1)或2)或3)或4)的dna分子:
1)序列表中seqidno.2所示的dna分子;
2)序列表中seqidno.3所示的dna分子;
3)在严格条件下与1)或2)限定的dna序列杂交且编码所述蛋白的dna分子;
4)与1)或2)或3)限定的dna序列具有90%以上同源性,且编码粒型相关蛋白的dna分子。
序列表中的seqidno.2由2433个核苷酸组成。
所述严格条件可为在0.1×sspe(或0.1×ssc),0.1%sds的溶液中,在65。c下杂交并洗膜。
含有以上任一所述基因的重组表达载体也属于本发明的保护范围。
可用现有的植物表达载体构建含有所述基因的重组表达载体。
所述植物表达载体包括双元农杆菌载体和可用于植物微弹轰击的载体等。所述植物表达载体还可包含外源基因的3’端非翻译区域,即包含聚腺苷酸信号和任何其它参与mrna加工或基因表达的dna片段。所述聚腺苷酸信号可引导聚腺苷酸加入到mrna前体的3’端,如农杆菌冠瘿瘤诱导(ti)质粒基因(如胭脂合成酶nos基因)、植物基因(如大豆贮存蛋白基因)3’端转录的非翻译区均具有类似功能。
使用所述基因构建重组植物表达载体时,在其转录起始核苷酸前可加上任何一种增强型启动子或组成型启动子,如花椰菜花叶病毒(camv)35s启动子、玉米的泛素启动子(ubiquitin),它们可单独使用或与其它的植物启动子结合使用;此外,使用本发明的基因构建植物表达载体时,还可使用增强子,包括翻译增强子或转录增强子,这些增强子区域可以是atg起始密码子或邻接区域起始密码子等,但必需与编码序列的阅读框相同,以保证整个序列的正确翻译。所述翻译控制信号和起始密码子的来源是广泛的,可以是天然的,也可以是合成的。翻译起始区域可以来自转录起始区域或结构基因。
为了便于对转基因植物细胞或植物进行鉴定及筛选,可对所用植物表达载体进行加工,如加入可在植物中表达的编码可产生颜色变化的酶或发光化合物的基因(gus基因、萤光素酶基因等)、具有抗性的抗生素标记物(庆大霉素标记物、卡那霉素标记物等)或是抗化学试剂标记基因(如抗除莠剂基因)等。从转基因植物的安全性考虑,可不加任何选择性标记基因,直接以逆境筛选转化植株。
所述重组表达载体可为在pcubi1390载体的多克隆位点hindiii和bamhi之间重组插入所述基因(ossg2)得到的重组质粒。所述重组质粒具体可为pcubi1390-ossg2;所述pcubi1390-ossg2是将将ossg2基因组编码序列通过重组技术插入pcubi1390多克隆位点hindiii和bamhi之间得到的(clontech公司,infusion重组试剂盒)。
将含有ossg2的pcubi1390命名为pcubi1390-ossg2。
含有以上任一所述基因(ossg2)的表达盒、转基因细胞系及重组菌均属于本发明的保护范围。
扩增所述基因(ossg2)全长或任一片段的引物对也属于本发明的保护范围。
本发明的另一个目的是提供一种培育粒型改变的转基因植物的方法。
本发明提供的培育粒型改变的转基因植物的方法,是将所述基因导入粒型长度变短的植物中,得到粒型正常的转基因植物;将所述蛋白的编码基因在正常植物中进行敲除,可以得到粒型变短的转基因植物,具体来说,所述基因通过所述重组表达载体导入粒型长度变短植物中;所述粒型长度变短植物为sg2。
所述蛋白、所述基因、所述重组表达载体、表达盒、转基因细胞系或重组菌或所述方法均可应用于水稻育种。
将本发明所述蛋白的编码基因导入粒型生长异常的植物中,可以培育粒型生长发育成正常的植物;在粒型正常植物中敲除该基因,可以获得粒型变小的植物。所述蛋白及其编码基因可以应用于水稻粒型的遗传改良。
利用任何一种可以引导外源基因在植物中表达的载体,将编码所述蛋白的基因导入植物细胞,可获转基因细胞系及转基因植株。携带有所述基因的表达载体可通过使用ti质粒、ri质粒、植物病毒载体、直接dna转化、显微注射、电导、农杆菌介导等常规生物学方法转化植物细胞或组织,并将转化的植物组织培育成植株。被转化的植物宿主既可以是单子叶植物,也可以是双子叶植物,如:烟草、百脉根、拟南芥、水稻、小麦、玉米、黄瓜、番茄、杨树、草坪草、苜宿等,特别是在水稻中的应用。
本发明首次发现、定位并克隆得到一个新的粒型相关蛋白的基因ossg2。将所述蛋白的编码基因导入粒型变短的植物中,可以得到粒型发育正常的转基因植物;将所述蛋白的编码基因在正常植物中进行敲除,可以得到粒型变短的转基因植物。所述蛋白及其编码基因可以应用于作物籽粒的遗传改良。
附图说明
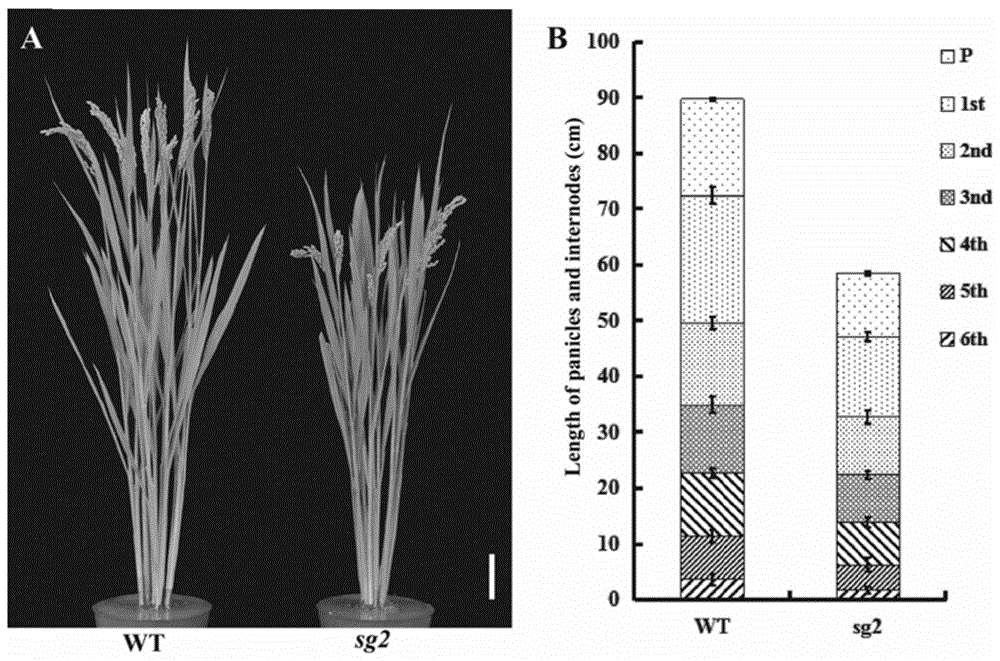
图1为野生型宁粳1号(wt)和突变体sg2成熟株型的表型图(a野生型wt和突变体sg2成熟株型对比图;b野生型wt和突变体sg2主茎各节间和穗对比图)。
图2为野生型宁粳1号(wt)和突变体sg2籽粒的表型分析图(a野生型宁粳1号(wt)和突变体sg2粒长对比图;b野生型宁粳1号(wt)和突变体sg2体式镜观察图;c野生型宁粳1号(wt)和突变体sg2粒宽对比图;d野生型宁粳1号(wt)和突变体sg2平均粒长;e野生型宁粳1号(wt)和突变体sg2平均千粒重;f野生型宁粳1号(wt)和突变体sg2平均粒宽)。
图3为野生型宁粳1号(wt)和突变体sg2颖壳的扫描电镜观察(a野生型宁粳1号(wt)和突变体sg2外颖外表皮细胞扫描电镜观察;b野生型宁粳1号(wt)和突变体sg2外颖外表皮细胞平均长度;c野生型宁粳1号(wt)和突变体sg2外颖外表皮细胞平均宽度;d野生型宁粳1号(wt)和突变体sg2外颖内表皮细胞的扫描电镜观察;e野生型宁粳1号(wt)和突变体sg2外颖内表皮细胞平均长度)。
图4为ossg2初定位结果和mutmap关联分析结果(a为ossg2基因初定位结果;b为ossg2基因mutmap关联分析结果)。
图5为转基因敲除结果(a野生型宁粳1号(wt),突变体sg2和敲除加系cr-1的株型观察;b野生型宁粳1号(wt),突变体sg2和敲除加系cr-1的测序鉴定和粒型观察)。
图6为转基因互补结果。
具体实施方式
以下的实施例便于更好地理解本发明,但并不限定本发明。下述实施例中的实验方法,如无特殊说明,均为常规方法。下述实施例中所用的试验材料,如无特殊说明,均为自常规生化试剂商店购买得到的。
实施例1、一种植物粒型相关蛋白及其编码基因的发现
一、水稻粒型突变体的获得
粳稻品种宁粳1号经转基因组培产生筛选出种子籽粒变小的突变体,命名为sg2。
与野生型比较,sg2在成熟后株型紧凑,株高显著变矮,其中倒一节间长度显著变短(图1a,b)、籽粒长度变短,宽度变窄,千粒重明显变小(图2a-f)、种子外颖内外表皮观察表明sg2籽粒长度变短和宽度变小是由于细胞大小引起的(图3a-e)。
综上所述,在突变体sg2中由于细胞大小改变,引起籽粒长度变短的表型。
二、突变基因定位
1、突变基因初步定位
首先我们构建野生型宁粳1号和突变体sg2正反交,所得f1自交后后代中正常型和突变型的种子符合3:1的分离比例,因此,sg2中籽粒变小的表型是由单个隐性核基因控制的。
突变体sg2与籼稻品种93-11杂交构建定位群体,在sg2与93-11构建的f2分离群体(5000株)中选取籽粒表型与突变体表型非常相似的极端单株,利用其叶片提dna。首先,用覆盖水稻全基因组的565对ssr引物在宁粳1号和93-11之间进行多态性分析,挑选在12条染色体均匀分布的多态性标记进行连锁分析。利用公共图谱上的分子标记和基于水稻基因组序列数据自行开发的ssr、indel分子标记对突变位点进行进一步定位,最后将粒型关键基因ossg2初定位在第2号染色体标记dbb16和hsp38两个分子标记之间。
上述ssr标记分析的方法如下所述:
(1)提取上述选取单株的总dna作为模板,具体方法如下:
①取0.2克左右的水稻幼嫩叶片,置于eppendorf管中,管中放置一粒钢珠,把装好样品的eppendorf管在液氮中冷冻5min,置于2000型geno/grinder仪器上粉碎样品1min。
②加入660μl提取液(含100mmtris-hcl(ph8.0),20mmedta(ph8.0),1.4mnacl,0.2g/mlctab的溶液),漩涡器上剧烈涡旋混匀,65℃水浴30min,每隔10分钟摇一次。
③加入40μl20%sds,65℃温浴10min,每隔两分钟轻轻上下颠倒混匀。
④加入100μl5mnacl,温和混匀。
⑤加入100μl10×ctab,65℃温浴10min,间断轻轻上下颠倒混匀。
⑥加入900μl氯仿,充分混匀,12000rpm离心3min。
⑦转移上清液至1.5mleppendorf管中,加入600μl异丙醇,混匀,12000rpm离心5min。
⑧弃上清液,沉淀用70%(体积百分含量)乙醇漂洗一次,室温凉干。
⑨加入100μl1×te(121克tris溶于1升水中,用盐酸调ph值至8.0得到的溶液)溶解dna。
⑩取2μl电泳检测dna质量,并用du800分光光度仪测定浓度(bechmaninstrumentinc.u.s.a)。
(2)将上述提取的dna稀释成约20ng/μl,作为模板进行pcr扩增;
pcr反应体系(10μl):dna(20ng/μl)1μl,上游引物(2pmol/μl)1μl,下游引物(2pmol/μl)1μl,10xbuffer(mgcl2free)1μl,dntp(10mm)0.2μl,mgcl2(25mm)0.6μl,rtaq(5u/μl)0.1μl,ddh2o5.1μl,共10μl。
pcr反应程序:94.0℃变性5min;94.0℃变性30s、55℃退火30s、72℃延伸1min,共循环35次;72℃延伸7min;10℃保存。pcr反应在mjresearchptc-225热循环仪中进行。
(3)ssr标记的pcr产物检测
扩增产物用8%非变性聚丙烯酰胺凝胶电泳分析。以50bp的dnaladder为对照比较扩增产物的分子量大小,银染显色。
上述ssr和indel标记开发如下:
(1)将公共图谱的ssr标记与水稻基因组序列进行整合,下载突变位点附近的bac/pac克隆序列。用ssrhunter(李强等,遗传,2005,27(5):808-810)或ssrit软件搜索克隆中潜在的ssr序列(重复次数≥6);将这些ssr及其邻近400~500bp的序列在ncbi通过blast程序在线与相应的籼稻序列进行比较,如果两者的ssr重复次数有差异,初步推断该ssr引物的pcr产物在籼、粳间存在多态性;再利用primerpremier5.0软件设计ssr引物,并由上海英俊/南京金斯瑞生物技术有限公司合成。将自行设计的ssr成对引物等比例混合,检测其在宁粳1号和93-11之间的多态性,具有多态的分子标记用作定位ossg2基因的分子标记。用于定位的分子标记见表1。
(2)indel标记的开发
indel引物设计:下载定位区间附近日本晴的克隆序列,在工数据库中与籼稻品种93-11序列进行比对,寻找两者之间存在的3个及3个以上碱基缺失的位置,运用primerpremier5.0软件设计含有缺失位点的引物,从而获得具有多态性的引物见表1。
indel标记分析的pcr反应体系:dna(20ng/ul)2ul,primer1(10pmol/ul)2ul,primer2(10pmol/ul)2ul,10xbuffer(mgcl2free)2ul,dntp(10mm)0.4ul,mgcl2(25mm)1.2ul,rtaq(5u/ul)0.4ul,ddh2o10ul,总体积20ul。扩增反应在ptc-200(mjresearchinc.)pcr仪上进行:94℃3min;94℃30sec,55℃(引物不同,有所调整)45sec,72℃2.5min,35个循环;72℃5min。
pcr产物纯化回收,按试剂盒(北京tiangen公司)步骤进行。pcr产物酶切消化过夜后,用1-4%的琼脂糖凝胶中电泳分离,经eb染色后于紫外灯下观察拍照。dcaps用8%的非变性page胶分离,银染。
表1用于初定位的分子标记
2、突变基因的mutmap分析
(1)突变基因的关联分析
从遗传分析结果表明在sg2突变体中籽粒变小的表型是由于单隐性核基因控制的,从初定位结果表明sg2的定位区间跨着丝粒,无法进行进一步精细定位。所以利用mutmap的方法进行进一步克隆,方法如下:
首先从sg2/wt的f2群体中,筛选出30株与野生型籽粒表型一样的的单株分别等量混合dna构成野生型混池,30株与突变体籽粒表型一样的单株分别等量混合dna构成突变体混池,然后对野生型宁粳1号,突变体sg2,野生型混池和突变体混池四个样品运用hiseq2500测序平台进行测序,然后运用mutmap方法进行关联分析结果如图4。
(2)突变基因的获得
经过初定位结果和mutmap分析的结果,在初定位区间发现了ossg2基因存在一个8个碱基的缺失(图5b)
根据定位的位点设计引物,序列如下所述:
primer1:5'-gagaattccacgccgctacg-3'(seqidno.4)
primer2:5'-gaagaaatttctaggaggca-3'(seqidno.5)
以primer1和primer2为引物,以宁粳1号的cdna为模板,进行pcr扩增获得目的基因。
扩增反应在ptc-200(mjresearchinc.)pcr仪上进行:94℃3min;94℃30sec,60℃45sec,72℃3min,35个循环;72℃5min。将pcr产物回收纯化后克隆到载体peasy(北京全式金公司),转化大肠杆菌dh5α感受态细胞(北京tiangen公司cb101),挑选阳性克隆后,进行测序。序列测定结果表明,pcr反应获得的片段具有序列表中seqidno.2所示的核苷酸序列,编码810个氨基酸残基组成的蛋白质(从atg到tga)(见序列表的seqidno.1)。将seqidno.1所示的蛋白命名为ossg2(即为基因定位中所述的ossg2基因),将seqidno.1所示的蛋白的编码基因命名ossg2。
实施例2、转基因植物的获得和鉴定
一、重组表达载体构建
以宁粳1号的cdna为模板,进行pcr扩增获得ossg2基因的编码序列,pcr引物序列如下:
primer3(下划线所示序列为hindiii的酶切位点):
5'-ccggcgcgccaagcttattaatcactatgcctacttcatagtatattat-3'(seqidno.6)
primer4(下划线所示序列为bamhi的酶切位点):
5'-gaattcccggggatcctgtcttaacacccctcacagcatcatattccca-3'(seqidno.7)
上述引物位于seqidno.3所示基因的启动子区和编码区,将pcr产物回收纯化。采用hdcloningkit重组试剂盒(takara公司)将pcr产物克隆到载体pcambia1390中。
上述引物分别位于seqidno.3所示序列,扩增产物包含了该基因的全部编码区部分,将pcr产物回收纯化。采用infusion重组试剂盒(clontech)将pcr产物克隆到载体pcubi1390中,构建成pcubi1390-ossg2;重组反应体系(10.0μl):pcr产物5.4μl(50-100ng),pcubi1390载体1.6μl(30-50ng),5×infusionbuffer2.0μl,infusionenzymemix1μl。短暂离心后将混合体系37℃水浴0.5h以上,取2.5μl反应体系用热激法转化大肠杆菌dh5α感受态细胞(北京tiangen公司;cb101)。将全部转化细胞均匀涂布在含50mg/l卡那霉素的lb固体培养基上。
37℃培养16h后,挑取克隆阳性克隆,进行测序。测序结果表明,得到了含有seqidno.3所示基因的重组表达载体,将含有sg2的pcubi1390命名为pcubi1390-ossg2,ossg2基因插入在多克隆位点hindiii和bamhi之间。
根据seqidno.3的序列,利用crispr技术进行定点敲除,引物如下:
primer5:
5'-ggcagcggtacgaggggctcctca-3'(seqidno.8)
primer6:
5'-aaactgaggagcccctcgtaccgc-3'(seqidno.9)
将上述两引物融入pcambia1305.1载体中,将该基因进行定点敲除,命名为pcambia1305.1-crispr-ossg2.
二、重组农杆菌的获得
用热击法将pcambia1390-ossg2和pcambia1305.1-crispr-ossg2分别转化农杆菌eha105菌株(购自美国英俊公司),得到重组菌株,提取质粒进行pcr及酶切鉴定。将pcr及酶切鉴定正确的重组菌株分别命名为eh-pcambia1390-ossg2和eh-pcambia1305.1-crispr-ossg2。
三、转基因植物的获得
将eh-pcambia1390-ossg2转化水稻突变体sg2,eh-pcambia1305.1-crispr-ossg2转化水稻宁粳1号,具体方法为:
(1)28℃培养eh-pcambia1390-ossg2和eh-pcambia1305.1-crispr-ossg216小时,收集菌体,并稀释到含有100μmol/l的n6液体培养基(sigma公司,c1416)中至浓度为od600≈0.5,获得菌液;
(2)将培养至一个月的突变体sg2和宁粳1号水稻成熟胚胚性愈伤组织与步骤(1)的菌液混合侵染30min,滤纸吸干菌液后转入共培养培养基(n6固体共培养培养基,sigma公司)中,24℃共培养3天;
(3)将步骤(2)的愈伤接种在含有100mg/l巴龙霉素(phytotechnologylaboratories公司)的n6固体筛选培养基上第一次筛选(16天);
(4)挑取健康愈伤转入含有100mg/l巴龙霉素的n6固体筛选培养基上第二次筛选,每15天继代一次;
(5)挑取健康愈伤转入含有50mg/l巴龙霉素的n6固体筛选培养基上第三次筛选,每15天继代一次;
(6)挑取抗性愈伤转入分化培养基上分化;
得到分化成苗的t0代阳性植株。以突变体sg2和宁粳1号为阴性对照。
四、转基因植株的鉴定
1、pcr分子鉴定
将步骤三获得的t1代互补阳性植株提取基因组dna,以基因组dna为模板,利用引物primer7和primer8作为引物对进行扩增(primer7:5'—gctccaccatgttatcacatcaat—3'(seqidno.10)和primer8:5'—cacagaagtgatacgagtgc—3'(seqidno.11))。将步骤三获得的t1代定点敲除阳性植株提取基因组dna,以基因组dna为模板,引物primer9和primer10作为引物对进行扩增(primer9:5'—ttgagaattccacgccgcta—3'(seqidno.12)和primer10:5'—cggggatgagcaccggctcc—3'(seqidno.13))。pcr反应体系:dna(20ng/ul)2ul,primer5(10pmol/ul)2ul,primer6(10pmol/ul)2ul,10xbuffer(mgcl2free)2ul,dntp(10mm)0.4ul,mgcl2(25mm)1.2ul,rtaq(5u/ul)0.4ul,ddh2o10ul,总体积20ul。扩增反应在ptc-200(mjresearchinc.)pcr仪上进行:94℃3min;94℃30sec,55℃45sec,72℃1min,35个循环;72℃5min。
用试剂盒(北京tiangen公司)纯化回收pcr产物。pcr产物用1%的琼脂糖电泳检测。
2、表型鉴定
分别将t1代转pcubi1390-ossg2和pcambia1305.1-crispr-ossg2的转基因植株,野生型宁粳1号和突变体sg2种植在南京农业大学土桥实验基地大田情况下。鉴定到转基因互补家系恢复到了野生型的表型,转基因定点敲除家系与突变体表型高度相同。由此验证粒型异常的表型是由ossg2基因控制的,即该ossg2基因为粒型相关基因(图5-图6)。
序列表
<110>南京农业大学
<120>一种植物粒型相关蛋白及其编码基因与应用
<160>13
<170>siposequencelisting1.0
<210>1
<211>810
<212>prt
<213>粳稻品种宁粳1号(ningjing1)
<400>1
metserthralaalalysgluglualaalaalaseralaproalapro
151015
alametglyglygluglualaalaalaargalaalaglnlysargtyr
202530
gluglyleuleuthrvalargalalysalavallysglylysglyala
354045
trptyrtrpalahisleugluprovalleuileproalaalaaspthr
505560
glymetproprolysalavallysleuargcysglyleucysserala
65707580
valpheseralaserasnproserargthralasergluhisleulys
859095
argglythrcysproasnpheseralaproproproglyalaalaala
100105110
alaserglyserserglyserglnhisglnglnglnthrproglnala
115120125
alaleuglnalaleuproproproasnserthralaserserproile
130135140
proileserserilealaproserserproarghisprohishishis
145150155160
serglnproglnglnproglnserhishishishishishishisser
165170175
glyserarglysarghissermetproproalatyrthralaalaglu
180185190
provalserhishishishisleuvalvalvalaspproserthrval
195200205
tyrserproproleuproalaleuproproproproproglnglnpro
210215220
glnseralaleuvalleuserglyglylysgluaspleuglyalaleu
225230235240
alametleugluaspservallysargleulysserprolysalaser
245250255
proglyalametleuprolysproglnalaaspalaalaleualaleu
260265270
leualaglutrppheleugluserserglyglyvalserleuserala
275280285
valalaasnprolysleuargserpheleuarghisvalglyleupro
290295300
gluleuglnargthraspleualaglyalaargleuaspalaargphe
305310315320
alaglualaargalaaspalathralaargvalargaspalaleuphe
325330335
pheglnleualaalaaspglytrparggluglnvalvalthrleucys
340345350
valasnleuproasnglythrservalphehisargglyvalproval
355360365
proalaproalaproserasptyralaglugluvalleuleuaspala
370375380
valalaservalseralaserglyserserasnaspleuhishiscys
385390395400
alaglyilevalalaaspargphelysserlysalaleuargaspleu
405410415
gluasnlyshishistrpmetvalasnleusercysglnilehisgly
420425430
phethrargleuvalargaspphealaarggluleuproleuphearg
435440445
seralaalaalalysseralalysleualaalatyrpheasnalalys
450455460
prothrvalargserleuleuhislyshisglnileglngluleugly
465470475480
hisalaserleuleuargvalalahisvalpropheasnsersergly
485490495
serasptyrargalaalapheglumetleugluaspvalleuthrser
500505510
alaargproleuglnleualavalleugluglusertyrlysleuval
515520525
cysileaspaspseralaalaargglumetalaaspmetleuglnasp
530535540
glyserphetrpsergluvalglualavalhisleuleuvallysleu
545550555560
ilemetaspmetvallysglumetgluthraspargproleuvalgly
565570575
glncysleuproleutrpgluaspleuargglylysvalargasptrp
580585590
cysasplyspheasnthraspgluglyalaalaleuasnvalvalglu
595600605
lysargphearglysasntyrhisproalatrpseralaalapheile
610615620
leuaspproleutyrleuilelysaspalaserglyargtyrleupro
625630635640
prophelyspheleuthrproaspglnglulysaspvalaspmetleu
645650655
ilethrargmetvalserarggluglualahisilealavalmetglu
660665670
leumetlystrpargthrgluglyleuaspproleutyralaglnala
675680685
valglnvalargglnproaspproserthrglylysmetlysvalala
690695700
asnlysglnserserargleuvaltrpgluthrcysleusergluleu
705710715720
lysserleuglylysvalalavalargleuilepheleuhisalathr
725730735
alaargglypheargcysserprosermetleuargtrpleuserala
740745750
proglyserleualaglyglyileaspargalahisargleuvalphe
755760765
valalaalaasnserlysleugluargargasppheserseraspglu
770775780
asplysaspalagluleuleuthrgluglyaspaspaspvalleuasn
785790795800
gluproglyserleugluargserserval
805810
<210>2
<211>2433
<212>dna
<213>粳稻品种宁粳1号(ningjing1)
<400>2
atgtcgacggccgccaaggaggaggcggcagcgtcggctccggcgccggcaatgggcggc60
gaggaggcggcggcgcgggcggcgcagaagcggtacgaggggctcctcacggtgcgggcc120
aaggcggtcaagggcaagggcgcctggtactgggcgcacctggagccggtgctcatcccc180
gccgccgacaccgggatgccgcccaaggccgtcaagctgcgctgcggcctctgctccgcc240
gtcttctccgcctccaacccatcgcgcacggcttcggagcacctcaagcggggcacctgc300
cccaacttctccgcgccaccgccgggcgccgccgcggcatccggttcgtccggttcgcag360
catcagcagcagacgccacaggcggcgctgcaggcgttgccgccgcccaactcgacggca420
tcgtcccctatcccaatctcgtccattgcgccctcgtcgccgcgccacccgcaccaccac480
tcgcagccccaacagccgcagtcgcatcaccaccatcaccatcactccggcagccggaag540
cgccactcgatgcctccggcgtacacggccgcggagcccgtgtcccaccaccaccatctc600
gtcgtcgtcgacccgtcgacggtttactcccctccgctgcccgccctgccgccgccacca660
ccgcaacaaccgcagtcggcgctcgtgctctccggcggcaaggaggatctcggcgcgttg720
gccatgctggaggacagcgtgaagcggctcaagtcgcccaaggcgtcgccgggggcgatg780
ctgccgaagccgcaggccgacgcggcgctcgccctgctcgccgaatggtttctcgagtcg840
tcgggcggcgtgtcgctgtcggccgtggcgaacccgaaactccggtccttcctccgccac900
gtcggcctgccggagctgcagcggacagacctggcgggggctcggctggatgcgcggttc960
gccgaggcccgcgccgacgccaccgcgcgcgtccgcgacgcgctctttttccagctcgcg1020
gcggacgggtggcgggagcaggtggtaactctgtgtgtcaacctcccaaatggaacatcc1080
gtgttccaccgcggcgtgccggtccccgccccggcgccctcggactacgccgaggaggtg1140
ttgctggacgcggtggcgtccgtgtccgcgtctggctcctccaacgacctgcaccattgc1200
gcgggcatcgtggctgaccgcttcaagtcaaaggcccttcgtgatctggagaacaagcac1260
cattggatggtgaacctgtcctgccaaatccatggcttcacccggctggttcgggacttc1320
gcgcgggagctccctctgttccgctccgctgcggctaaatccgccaagctggccgcctac1380
ttcaatgccaagccgacggtgcggtccctgctgcacaagcaccaaatccaggagctcggg1440
catgcgtcgcttctccgtgtggcgcatgtgccattcaatagcagcggcagcgactaccgt1500
gcggccttcgagatgctggaggacgtgttgacctctgctcgcccgctccagctcgccgtc1560
ttggaagagtcctataagctggtctgcatcgacgattcggccgccagggagatggcggac1620
atgttgcaggatgggtcattctggagtgaggttgaggccgtgcatctgctcgtgaagctg1680
atcatggacatggtgaaagagatggagactgacaggcctctggttggccaatgcctgcct1740
ctctgggaggacctacgtggcaaggttagggattggtgcgataaattcaacactgatgag1800
ggcgctgccctgaatgtggtggagaaaaggttcaggaagaactaccatccagcctggtca1860
gcggctttcatattggatcctctctacctaatcaaggatgccagtgggcgctacctcccg1920
cccttcaagttcttgactcctgatcaagagaaggatgtggacatgctcatcaccaggatg1980
gtgtcgcgggaggaggcgcacattgcggtcatggagctcatgaaatggcggacggaaggg2040
ctggacccgttgtatgcgcaggcggtgcaggtccggcagcctgacccgtccaccgggaag2100
atgaaggtcgcaaacaagcagagcagccgccttgtctgggagacgtgcctgagcgagctc2160
aagtccctcgggaaggtggctgtacggctcatcttcctccatgccactgctagggggttc2220
aggtgctcaccctccatgctgcgttggctctctgctcctggcagtttagccggtggcatt2280
gatcgtgcgcaccggctagtgttcgttgctgcaaattccaagctagagaggagggatttc2340
tcaagtgatgaagacaaggatgctgagttactcactgaaggggacgatgatgtgctaaac2400
gagcctggcagtttggagcgctcctcagtgtaa2433
<210>3
<211>8494
<212>dna
<213>粳稻品种宁粳1号(ningjing1)
<400>3
attaatcactatgcctacttcatagtatattattgttcattgccttccggatgtcttttc60
aacaccacatgtttactgaggcttctaatttctattgtggtgcatactttaaatgtataa120
tttgtgaaaattgacatattcttttggcaaggagtttgtaccacgtgcatttctaagtac180
aatattcaatcactaattttagagtaaatttcataaaactacaactatttagacaaaact240
atattttgctgtaacttcatatgaacatttctaaaggctgcaactatattgcatgcgagc300
gattttataaaaaataaaactacaacttaaaagtgatatgagtgtttgacatgtggttct360
ttgacaaagttgcatttttctcataaattgtcaactatggttgtatgaccaatcaaagtt420
gtagcaaattaattgttttgacatatagttttctgaaattatcataaatctaaattatca480
tatagttttctgaaattacttatttggtttgtagcatatttgttcacacacacacacaca540
catatatctatatatattcgaaagatcttaagttttattaggccagccaccgagttataa600
tgtttattgtcctatattagttcttgaagcacaatcaacaacttcagaaggtctacatat660
atagccagattggaaagctcatcggtgtattcactattggtactcattcactattggtac720
tgtcagcaccatttgttgtggtctgtaaatccatgttcatgagaataccgtgtctaagca780
ctcgtatcacttctgtgatatgagtgataaatttttcaaacacagtacagttttgcacta840
tttatgtccaacgtttcaccgttcgttttatttgaaaactttttcaaataagatggacgg900
tcaaacattagacacggattttcacagctgtacttatttttagaacggaggtattaacat960
ttcttttatctagcgcatatattttaatagaccaggcaagaaaaggaaaaatgataggta1020
gagtgtcgatagactgtatagtacgtacagttgccaacagtccccgtttcttcagttata1080
gacttttatatggctactggttacaatatctttatctgacgtccaactcttttcatatta1140
ctacagtggaggaaaaacagaaaaaaaaaggagaactattttttttaatctaacctgcct1200
aatccaaaatatatccaaaccttattaattaattacagctgtgatggtatatacatacac1260
acacaattattttcttgtgcatgctaatctactatatacaaatggctctgtttgagtaca1320
aatgcaaataattgtggttgaatggacttaattttttggtcctgtgtgttatccagcgga1380
gttacgttacagctataccgcattgatgaaaattaatgttttacatcaaccccgctatag1440
aaaagtctctctctccggaatgatattgatatatatggtgctgcatgcatgcatgttctt1500
gcgcaatatattacggtgatttttattcttctctgatcagtgatcactccatacattgcg1560
gggtactagtaattaatcaaccgtctggctgttgcagctagcaacgcatgcaaatcttac1620
tgtgtgttttgtgatgctcaaatctaactgttacagtcgtacttaccgttgcgccagaca1680
agcacaagccacagaaaaggtcatacgcagtacagtactatatacagtaaccgttccatg1740
catgatgctaattatctgcatgtacttttacttggctaatgactccagattaatttgtgt1800
ataccacatgcatgtataccagcgtacgtattttcctgcagtatatactactactagtag1860
ttaggtacgtacgtacgtatgtatagccactagccagattggaaagctagctgcagcctg1920
caggtgtagcgtatgagagcatgtgaaattcctaaataattgagggtacacagccttttg1980
ctgtcctagaaacagtcgagaaattcaaccaaacttgccagctttttcaaacgcttattt2040
atcataatgtggatgggttagctctagcttgcttgcttactcgctttctccccagtggag2100
gagggaagtattgtacgccgcccttaattgtctgtctagcttgcctgcaaatactcacta2160
ctgctacgtgtataatgcgtgcagttcatacgctgtccggcgcctcaccaaaactggtac2220
cccaccccacccttctatatatgtttagccacgcacgccattgacaactactacgtcctt2280
tttttccccatatagacgccacagcaaaatactcaatgctactaagtatacgcgattgat2340
aactattagtatgtttggtaaaatgccactgtcacacggttaataatgacgatgaatttg2400
gcttgatgctaaaaaatgccctagattttactgactttgtcagagagatttgtagctttt2460
cagtcgtaaccatttgtttttatgacttttgaattaactgatcatctggacttgagattt2520
tctttctccacccacaggaaaaaaatgaataattctatgttaaataagcgatgtgctgtt2580
actattgcggaggtacacgtatcatgggagcaggtcaggtagccgaaatggaggttttgt2640
aaggtgtttatacggcagcggcgcggcggcaaatttgcgctgccgccggccggggggaac2700
gaaaagggagcgagaggtaggaaaggctaaacaagggggagggcgtgggctctcacgcca2760
cggcctctagggccaccaactattccaccgcaataattgtgggagagaggagaggagaag2820
agaggtgtgtggtggtggtcatgcatagtagccgaacacaatacacttgtacaatcctcc2880
cccttattttacatataggcctttgtaccataaagaatagactaggcattataccgagag2940
gcattcctcttgtgggagtgggcagtcaaccctgccccaaagaccagaccaaaaaaaatt3000
aaagaaaagaaaaagggtgtagagaagtagtctggatagagagagagatggtggctctca3060
ggctctcaagcttgctcctcctcttcttcttccataaattcctaggttgcccctctcctg3120
ctttctcttcttcttcaatctccatccccatcatctatctatccatcatccatcagacca3180
gacccttcctcctctcccctgcctaccggcagcagcacccagcagtaagcccccaagcag3240
taagctatagctgcctgtgtgaggccccaaaatctctctactctccctttctccatcccc3300
acacacattcctccgatatctagtatatctccctactacaccatcatccaggctacatta3360
gtttggtctcttttttttttcttttataagttttgggtttttcctcaccaccaccacaag3420
cgaggagttgcacaaccatccaaagggaggggagtgcaagaattgtgagctgccacaagt3480
gatatttttccaattttgtttttgggcaagtgtagtgcactcactcacacactaggcagg3540
ggcaggggcagggcggcctataaataaaaagaaaaaaaaagagagagaagatatattact3600
agtagtatttgctattggaagaagccaaaggcaggggaggaggaagagtcttcttctgct3660
gctctctctcttctcttctcaattcatcctccttttgtccctatcaaaccctttttttct3720
gtctctctctttctaccttggcattgttttctcgcaaatcttgctttctaaacccccaaa3780
acctcccggaaaacaacccacccaccccgcgcgtggatggacggcccgcggccgccgccg3840
ccgcccaatgcgctgcgccgatcgcggttgtttccgacgaccacgccgccgccgtccgcc3900
accgggggtctccggcattagaacccgggcagttgagaattccacgccgctacggcggga3960
ttggattagcgggcgggcggcggtcagggaagaggaggttctctcttgtggtcgttgccg4020
ctggaggtgatcgatcgatcggtcggtcgtctcgcggggcgatcggtcggcatgtcgacg4080
gccgccaaggaggaggcggcagcgtcggctccggcgccggcaatgggcggcgaggaggcg4140
gcggcgcgggcggcgcagaagcggtacgaggggctcctcacggtgcgggccaaggcggtc4200
aagggcaagggcgcctggtactgggcgcacctggagccggtgctcatccccgccgccgac4260
accgggatgccgcccaaggccgtcaagctgcgctgcggcctctgctccgccgtcttctcc4320
gcctccaacccatcgcgcacggcttcggagcacctcaagcggggcacctgccccaacttc4380
tccgcgccaccgccgggcgccgccgcggcatccggttcgtccggttcgcagcatcagcag4440
cagacgccacaggcggcgctgcaggcgttgccgccgcccaactcgacggcatcgtcccct4500
atcccaatctcgtccattgcgccctcgtcgccgcgccacccgcaccaccactcgcagccc4560
caacagccgcagtcgcatcaccaccatcaccatcactccggcagccggaagcgccactcg4620
atgcctccggcgtacacggccgcggagcccgtgtcccaccaccaccatctcgtcgtcgtc4680
gacccgtcgacggtttactcccctccgctgcccgccctgccgccgccaccaccgcaacaa4740
ccgcagtcggcgctcgtgctctccggcggcaaggaggatctcggcgcgttggccatgctg4800
gaggacagcgtgaagcggctcaagtcgcccaaggcgtcgccgggggcgatgctgccgaag4860
ccgcaggccgacgcggcgctcgccctgctcgccgaatggtttctcgagtcgtcgggcggc4920
gtgtcgctgtcggccgtggcgaacccgaaactccggtccttcctccgccacgtcggcctg4980
ccggagctgcagcggacagacctggcgggggctcggctggatgcgcggttcgccgaggcc5040
cgcgccgacgccaccgcgcgcgtccgcgacgcgctctttttccagctcgcggcggacggg5100
tggcgggagcaggtggtaactctgtgtgtcaacctcccaaatggaacatccgtgttccac5160
cgcggcgtgccggtccccgccccggcgccctcggactacgccgaggaggtgttgctggac5220
gcggtggcgtccgtgtccgcgtctggctcctccaacgacctgcaccattgcgcgggcatc5280
gtggctgaccgcttcaagtcaaaggcccttcgtgatctggagaacaagcaccattggatg5340
gtgaacctgtcctgccaaatccatggcttcacccggctggttcgggacttcgcgcgggag5400
ctccctctgttccgctccgctgcggctaaatccgccaagctggccgcctacttcaatgcc5460
aagccgacggtgcggtccctgctgcacaagcaccaaatccaggagctcgggcatgcgtcg5520
cttctccgtgtggcgcatgtgccattcaatagcagcggcagcgactaccgtgcggccttc5580
gagatgctggaggacgtgttgacctctgctcgcccgctccagctcgccgtcttggaagag5640
tcctataagctggtctgcatcgacgattcggccgccagggagatggcggacatgttgcag5700
gatgggtcattctggagtgaggttgaggccgtgcatctgctcgtgaagctgatcatggac5760
atggtgaaagagatggagactgacaggcctctggttggccaatgcctgcctctctgggag5820
gacctacgtggcaaggttagggattggtgcgataaattcaacactgatgagggcgctgcc5880
ctgaatgtggtggagaaaaggttcaggaagaactaccatccagcctggtcagcggctttc5940
atattggatcctctctacctaatcaaggatgccagtgggcgctacctcccgcccttcaag6000
ttcttgactcctgatcaagagaaggatgtggacatgctcatcaccaggatggtgtcgcgg6060
gaggaggcgcacattgcggtcatggagctcatgaaatggcggacggaagggctggacccg6120
ttgtatgcgcaggcggtgcaggtccggcagcctgacccgtccaccgggaagatgaaggtc6180
gcaaacaagcagagcagccgccttgtctgggagacgtgcctgagcgagctcaagtccctc6240
gggaaggtggctgtacggctcatcttcctccatgccactgctagggggttcaggtgctca6300
ccctccatgctgcgttggctctctgctcctggcagtttagccggtggcattgatcgtgcg6360
caccggctagtgttcgttgctgcaaattccaagctagagaggagggatttctcaagtgat6420
gaagacaaggatgctgagttactcactgaaggggacgatgatgtgctaaacgagcctggc6480
agtttggagcgctcctcagtgtaacacccttatttaattgttcgtttctttttctctatt6540
agccttttttagaagttaatccctaggaatttgctagaatttccaccccctacctatttt6600
tttgcctcctagaaatttcttcctcgtgcccatgctgttaattcttacttcctcactcta6660
gattttggtggggtggggagtggggcatgagatggagatatgcatcctttgttcatcaag6720
tttggttaagaatgactgtctcttgccatatatgattccatggggccattgaacttgcag6780
agattaatggaagtctttctgtcaatatgttcatagtcggtggtacaccgaaagcgggaa6840
gaatgtgtaaccatttttttttgtgaaaattaataatattgatatgcaattgaatgcaat6900
ctgactgccgtctatatatctattattgctttcttcaaattgcactgttacttcttttac6960
attgttcttgaacgtgtggtgctctgttcctagttaatactaccatacatcactccactc7020
aacagtccacactgagcaggcttcgaacaaagagtaataacatccctttattgttttgac7080
cgtgattaactcaacaccaggaactcttgctactctgaaagagaaatgctcaccggatgc7140
taattaataatatccttgtgtggtttcttctaacgcgcctggccttgtttctggaggcat7200
tgtgcatcattggagcttggcagcttctgtgatcctgtcatgactcatgaaggcgaggct7260
cactttgcaacagcatcagccaagaaggtacatatctcatcttctaggatgttctttcgg7320
ttctactccatggcatttggcttttcagctggacctgctatgctggaagtttaaacagta7380
gcagcagcagaagcagctccagctcggtttattacgaggtgagcggtgccaactttcaca7440
ttttgccttttctcagtgttactcgtagcatttactgtttttctccagagtaggcacaac7500
ggcaatgccgcaatgggtttttctgcattgtgtgatcatgtgagattatcatgagcacgg7560
tcgttgtcgtcgtcgtcatcttcttcttcgtgctccttgccttggtcgtcgcccaaccgg7620
gcgatgattgcgccggttcgcatggcttcatctcgacggtatcggaaaggctccggtccg7680
gagagggatgaaaagccggagttgaaggtgcagacacggtaggctttttgctcctccccc7740
atccccatgcccatcaccacggccagggcctttgacaatcatgccttgcatcaaaacatg7800
tccttttagtttgcagggggtacaagcttgccaaggcagtgatcgattccctttcctatc7860
attgggatgggcacctcggttgctgcctaccggagagctcgccgtgctttcgatccaatc7920
tgccatgagagctcctctccttgcccttctggtcttatggtttgatggagaaaagagcgt7980
ttgattggagcggcagggatgtgttcttggacaggagaagaggaaagaaaagcctgtgta8040
atacaggctgttcctgcatccgtaccaattggggtttttcttttttcgctcggcttctct8100
gttgctcctggctcctgctgctctctcctcccctcagctgtccaagcacatgctttacct8160
gactaatcattaccatttgagctttattaggcgtcgtcgcagagaacggaggagatgatt8220
attggacataatgcagtcagtattcagcaagtgatggtgagtatcgttggtgctggcaca8280
tctccttgtttttccttaattttcctcctgtgtcagtactgctggggctgtgtagttgtt8340
gctaaagtagtagtagcagcagcagcagtacaaactattgctggaatgggtgtattctgc8400
tttttctcagtcgcttttgcaacttgatgtactatgttttgtacacatgttgtgtagtag8460
ttgggaatatgatgctgtgaggggtgttaagaca8494
<210>4
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>4
gagaattccacgccgctacg20
<210>5
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>5
gaagaaatttctaggaggca20
<210>6
<211>49
<212>dna
<213>人工序列(artificialsequence)
<400>6
ccggcgcgccaagcttattaatcactatgcctacttcatagtatattat49
<210>7
<211>49
<212>dna
<213>人工序列(artificialsequence)
<400>7
gaattcccggggatcctgtcttaacacccctcacagcatcatattccca49
<210>8
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>8
ggcagcggtacgaggggctcctca24
<210>9
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>9
aaactgaggagcccctcgtaccgc24
<210>10
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>10
gctccaccatgttatcacatcaat24
<210>11
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>11
cacagaagtgatacgagtgc20
<210>12
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>12
ttgagaattccacgccgcta20
<210>13
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>13
cggggatgagcaccggctcc20
- 还没有人留言评论。精彩留言会获得点赞!
- 降低种子重量的白桦AP2基因及其编码蛋白的制造方法与工艺
- 可编码ArtRD蛋白基因及其蛋白和应用的制造方法与工艺
- 蝎神经毒素AaIT及其编码基因与应用的制造方法与工艺
- 腈水解酶突变体及其编码基因和应用的制造方法与工艺
- 植物抗旱相关蛋白AsTMK1及其编码基因和应用的制造方法与工艺
- 植物抗旱相关蛋白EtSnRK2.2及其编码基因和应用的制造方法与工艺
- OsSAPK7蛋白及其编码基因在提高水稻白叶枯病抗性中的应用的制造方法与工艺
- 一种抗除草剂抗虫融合基因、编码蛋白及应用的制造方法与工艺
- 毛果杨PtrZFP103基因及其编码蛋白和应用的制造方法与工艺
- 大豆bHLH转录因子基因GmFER及其编码蛋白和应用的制造方法与工艺
- 一种新的多肽——鸟苷三磷酸酶激活蛋白10.12和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——Ras GTP 酶激活蛋白11.22和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——Ras GTP酶激活蛋白18和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——Ras GTP酶激活蛋白10和编码这种多肽的多核苷酸的制作方法
- 一种猪p21蛋白抗体及其制备方法与应用的制作方法
- 应用microRNA-7抑制前列腺肿瘤生长及其肿瘤进程的制作方法
- 一种抗p21ras蛋白的单链抗体及其应用的制作方法
- 一种协同抑癌的前列腺癌药物组合物的制作方法
- 用于癌症治疗的包含p21蛋白质的缀合物的制作方法
- 一种新的多肽——Ras GTP酶激活蛋白12和编码这种多肽的多核苷酸的制作方法