鉴定长芒苋、糙果苋和沙地苋单粒种子的基于SNP位点的CAPs分子标记及其应用的制作方法
本发明涉及分子生物学技术领域,具体涉及鉴定长芒苋、糙果苋和沙地苋单粒种子的基于snp位点的caps分子标记及其应用。
背景技术:
检疫性杂草长芒苋、糙果苋和沙地苋隶属苋科苋属异株苋亚属,原产北美,是美国大豆、玉米农田的超级杂草。苋属植物的种子小,仅1mm左右,同属不同种植物的种子形态相近,难以依据种子形态进行有效区分。因此,开发能够实现快速高效鉴定长芒苋、糙果苋和沙地苋单粒种子的分子生物学检测方法对于口岸检测具有重要意义。
单核苷酸多态性(singlenucleotidepolymorphism,snp)是指由于单个核苷酸的变异所形成的遗传标记,其数量多、多态性丰富、适于快速、自动化分析。snp的检测方法有多种,但许多方法由于技术难度高、成本费用高、检测时间较久,阻碍了应用。caps(cleavedamplificationpolymorphismsequence-taggedsites)技术又称为pcr-rflp,即限制性片段长度多态性聚合酶链反应技术。是使用特异设计的pcr引物扩增目标基因,用专一性的限制性内切酶切割获得特异性条带并进行rflp分析。pcr-rflp的应用降低了snp位点检测成本和难度,同时缩短了检测时间,在2小时内即能获得检测结果,是目前最适用于口岸实验室的检测方法。
技术实现要素:
本发明的目的之一是提供鉴定长芒苋、糙果苋和沙地苋单粒种子的caps分子标记,本发明的另一目的是提供鉴定长芒苋、糙果苋和沙地苋单粒种子的引物和试剂盒。
本发明通过对苋属(amaranthusl.)18个种52份样本的大量关键基因进行扩增、序列分析和比对,分析其系统进化关系及序列差异,发现位于多拷贝核基因its序列的snp位点可区分长芒苋、糙果苋、沙地苋与其他苋属植物种子。基于该snp位点,结合18s、its、26s的序列分析,本发明开发了caps分子标记,确定以位于18s以及下游its的片段(seqidno.1)作为区分鉴定长芒苋和其它苋属植物单粒种子的分子标记,以位于26s及其上游its的片段(seqidno.2)作为区分鉴定糙果苋/沙地苋与其它苋属植物单粒种子的分子标记。
具体地,本发明的技术方案如下:
本发明提供用于鉴定长芒苋、糙果苋和沙地苋单粒种子的caps分子标记,包括分子标记1和/或分子标记2;分子标记1的核苷酸序列如seqidno.1所示,snp位点位于如seqidno.1所示序列的第233位,该位点为a/c;分子标记2的核苷酸序列如seqidno.2所示;snp位点位于如seqidno.2所示序列的第200位,该位点为c/a/t。
上述caps分子标记中,对于分子标记1的所述snp位点,长芒苋种子为a,其它苋属种子为c;对于分子标记2所述snp位点,糙果苋和沙地苋种子为c,其它苋属种子为a/t。
上述caps分子标记中,分子标记1位于18s以及下游its,分子标记2位于26s及其上游its。分子标记1可有效地将长芒苋与其他苋属植物的单粒种子区分,分子标记2可有效地将糙果苋/沙地苋与其他苋属植物的单粒种子区分。上述caps分子标记联用可在苋属植物单粒种子中鉴定出长芒苋、糙果苋、沙地苋的种子。
本发明提供所述caps分子标记或其检测试剂在鉴定长芒苋、糙果苋和沙地苋的单粒种子中的应用。
本发明还提供所述caps分子标记或其检测试剂在植物检验检疫中的应用。
在上述caps分子标记的基础上,本发明提供所述caps分子标记的检测引物,其包括引物对1和引物对2;引物对1的核苷酸序列如seqidno.3-4所示,引物对2的核苷酸序列如seqidno.5-6所示。上述两对引物可用于在苋属植物单粒种子中鉴定长芒苋、糙果苋、沙地苋的种子。
引物对1:
正向引物78f:f5'-gtccctgccctttgtacaca-3;
反向引物419r:r5'-gggttcgttgttgcatcgag-3'。
引物对2:
290f:5'-ttgcagaatcccgtgaacca-3';
762r:5'-agaggacgcttctccagact-3'。
上述检测引物中,引物对1用于检测分子标记1,引物对2用于检测分子标记2。
本发明还提供包含所述检测引物的试剂盒。所述试剂盒可用于在苋属植物单粒种子中鉴定长芒苋、糙果苋、沙地苋的种子。
优选地,所述试剂盒还包括dna聚合酶、pcr反应缓冲液、dntps、标准阳性模板、限制性内切酶(如hpych4v限制性内切酶、hhai限制性内切酶)中的一种或多种。
本发明提供所述检测引物或包含所述检测引物的试剂盒在鉴定长芒苋、糙果苋和沙地苋的单粒种子中的应用。
本发明提供所述检测引物或包含所述检测引物的试剂盒在植物检验检疫中的应用。
本发明提供用于区分鉴定长芒苋、糙果苋、沙地苋与其他苋属植物单粒种子的方法,其为对待测苋属植物单粒种子中所述caps分子标记进行分型,根据分型结果判断待测苋属植物单粒种子是否为长芒苋的种子,或者是否为糙果苋或沙地苋的种子。
优选地,所述方法包括:以待测苋属植物单粒种子的基因组dna为模板,利用所述检测引物进行pcr扩增,利用hpych4v对seqidno.3-4所示检测引物扩增得到的pcr产物进行pcr-rflp限制酶切反应,利用hhai对seqidno.5-6所示检测引物扩增得到的pcr产物进行pcr-rflp限制酶切反应,根据得到的酶切片段长度判断待测苋属植物单粒种子是否为长芒苋的种子,或者是否为糙果苋、沙地苋的种子。
作为本发明的优选方案,所述方法包括如下步骤:
(1)提取待测苋属植物单粒种子的基因组dna;
(2)以步骤(1)的基因组dna为模版,分别利用seqidno.3-4所示引物和seqidno.5-6所示引物进行pcr扩增反应;
(3)利用hpych4v对步骤(2)中seqidno.3-4所示检测引物扩增得到的pcr产物进行pcr-rflp限制酶切反应,利用hhai对步骤(2)中seqidno.5-6所示检测引物扩增得到的pcr产物进行pcr-rflp限制酶切反应;
(4)电泳检测pcr-rflp反应产物,若hpych4v酶切产物显示为主条带在233bp,则待测样品为长芒苋,若显示为主条带在284-286bp,则待测样品为除长芒苋外的苋属其它物种;若hhai酶切产物显示为主条带在213bp,则待测样品为糙果苋/沙地苋,若显示为主条带在426-429bp,则待测样品为除糙果苋、沙地苋外的苋属其它物种。
上述方法中,步骤(2)的pcr扩增反应体系以50μl计为:30ng/μl模板dna1μl,10pmol/μl正、反向引物各2μl,5u/μltaqdna聚合酶mix预混液(含酶、dntp等)25μl,余量为水。pcr扩增反应的条件为:98℃3分钟;98℃10秒,54℃10秒,72℃10秒,38个循环;72℃5分钟,4℃保存。
步骤(3)中酶切反应体系以50μl计为:1μg/μl模板dna1μl,10×buffer5.0μl,hpych4v酶或hhai酶1μl,余量为水。pcr-rflp酶切反应条件为:37℃水浴10分钟。
本发明的有益效果在于:本发明通过对苋属(amaranthusl.)18个种52份样本及1个外类群(鸡冠花)的基因序列进行分析比对,分析其序列差异,得到用于区分鉴定长芒苋、糙果苋、沙地苋与其他苋属植物的snp位点,基于该snp位点,本发明开发了区分鉴定长芒苋、糙果苋、沙地苋与其他苋属植物单粒种子的caps分子标记,其特异性检测引物和pcr-rflp检测方法。利用本发明提供的caps分子标记及方法可实现对长芒苋、糙果苋、沙地苋单粒种子的快速、准确检测鉴定,与现有技术中对于取样量需求较大的多粒种子提取方法相比,本发明提供的单粒种子鉴定方法更加准确有效,为口岸快速鉴定检疫性杂草长芒苋、糙果苋、沙地苋提供了可靠的检测方法。
附图说明
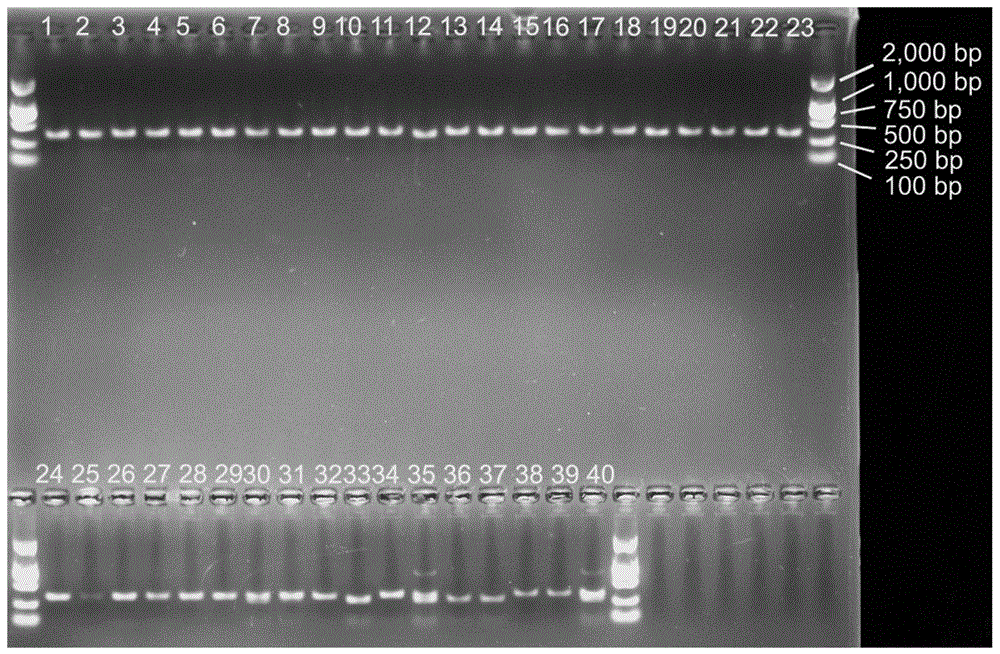
图1为本发明实施例3中seqidno.1所示caps分子标记的扩增片段的hpych4v限制性内切酶酶切结果;其中,1:白苋12332;2:白苋12538;3:白苋12771;4:北美苋12303;5:北美苋code1;6:南非苋;7:皱果苋4112;8:皱果苋11987;9:苋12743;10:苋4621;11:苋3683;12:皱叶苋;13:菱叶苋11350;14:菱叶苋12127;15:合被苋;16:地中海苋7367;17:地中海苋7469;18:地中海苋2170;19:凹头苋130;20:凹头苋10100206;21:凹头苋4167;22:绿穗苋11504;23:绿穗苋7659;24:绿穗苋7491;25:鲍氏苋;26:刺苋;27:糙果苋cq;28:糙果苋09014;29:沙地苋;30:绿穗苋、皱果苋、苋和长芒苋混合样;31:北美苋、反枝苋和长芒苋混合样;32:凹头苋、菱叶苋和长芒苋混合样;33:长芒苋;34:糙果苋;35:反枝苋、糙果苋和长芒苋;36:长芒苋12167;37:长芒苋11907;38:反枝苋;39:糙果苋;40:糙果苋、长芒苋及其他苋属混合样;dna条带标记使用的为dl2000。
图2为本发明实施例3中seqidno.2所示caps分子标记的扩增片段的hhai限制性内切酶酶切结果;其中,1:皱叶苋;2:苋;3:菱叶苋;4:南非苋;5:皱果苋;6:凹头苋;7:绿穗苋;8:白苋;9:地中海苋;10:膨果苋;11:北美苋;12:沙地苋;13:刺苋;14:白苋;15:合被苋;16:刺苋;17:糙果苋;18:鸡冠花;19:糙果苋;20-23:长芒苋;dna条带标记使用的为dl2000。
图3为本发明实施例3中seqidno.1所示caps分子标记的扩增片段的hpych4v限制性内切酶酶切结果以及seqidno.2所示caps分子标记的扩增片段的hhai限制性内切酶酶切结果;其中,1-9为hhai限制性内切酶酶切结果;1:糙果苋12014;2:糙果苋js;3:糙果苋12164;4:糙果苋12022;5:糙果苋12165;6:长芒苋12167;7:糙果苋2012009;8:糙果苋和其他苋属种子的混合样品;9:长芒苋11907;10-17为hpych4v限制性内切酶酶切结果,10:长芒苋120914-6和其他苋属种子的混合样品;11:长芒苋120914-7和其他苋属种子的混合样品;12:长芒苋120914-8和其他苋属种子的混合样品;13:长芒苋120914-9和其他苋属种子的混合样品;14:长芒苋11914;15:长芒苋12167;16:糙果苋11976;17:长芒苋12195;dna条带标记使用的为dl2000。
具体实施方式
下面将结合实施例对本发明的优选实施方式进行详细说明。需要理解的是以下实施例的给出仅是为了起到说明的目的,并不是用于对本发明的范围进行限制。本领域的技术人员在不背离本发明的宗旨和精神的情况下,可以对本发明进行各种修改和替换。
下述实施例中所使用的实验方法如无特殊说明,均为常规方法。
下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。以下实施例中pcr扩增所用试剂均购自中检国研,引物生工合成及测序均由上海生物工程技术服务有限公司完成。
实施例1snp位点以及基于snp位点的caps分子标记的获得
通过对苋属(amaranthusl.)18个种52份样本的18s、its、26s等基因的序列进行分析比对,分析其系统进化关系及序列差异,得到用于区分长芒苋、糙果苋、沙地苋与其他苋属植物的snp位点,其中,用于区分长芒苋与其他苋属植物的snp位点位于如seqidno.1所示序列的第233位,长芒苋的该位点为a,其它苋属植物的该位点为c。用于区分糙果苋/沙地苋与其他苋属植物的snp位点位于如seqidno.2所示序列的第200位,糙果苋和沙地苋的该位点为c,其它苋属植物的该位点为a/t。
用于基因扩增和序列分析的52份植物材料的具体名称及材料来源见表1,用于实验的种子来自经形态鉴定后的植株标本。部分植物材料的snp位点序列比对结果如表2和表3所示。
表152份植物材料及来源
表2seqidno.1中长芒苋与苋属其它种相区分的snp位点(部分结果)
表3seqidno.2中糙果苋和沙地苋与苋属其它种相区分的snp位点(部分结果)
基于上述snp位点以及18s、its、26s的序列分析结果,确定以seqidno.1所示序列作为区分鉴定长芒苋与其他苋属植物的caps分子标记,确定以seqidno.2所示序列作为区分鉴定糙果苋/沙地苋与其他苋属植物的caps分子标记。
用于扩增检测上述caps分子标记的特异性引物的具体序列如下:
用于扩增seqidno.1所示caps分子标记:
正向引物78f:f5'-gtccctgccctttgtacaca-3;
反向引物419r:r5'-gggttcgttgttgcatcgag-3'。
用于扩增seqidno.2所示caps分子标记:
290f:5'-ttgcagaatcccgtgaacca-3';
762r:5'-agaggacgcttctccagact-3'。
实施例2区分鉴定长芒苋、糙果苋、沙地苋与其他苋属植物单粒种子的方法
利用实施例1提供的caps分子标记及其特异性检测引物鉴定长芒苋、糙果苋、沙地苋,具体方法如下:
1、dna提取
将待测苋属植物的单粒种子研磨后置于1.5ml离心管中,使用lysasea在95℃水浴10分钟,后离心2分钟待用。
2、pcr扩增
(1)seqidno.1所示caps分子标记的扩增
以步骤1得到的dna为模板,采用正向引物78f:5'-gtccctgccctttgtacaca-3'和反向引物419r:5'-gggttcgttgttgcatcgag-3'进行pcr扩增。
pcr反应体系以50μl计为:30ng/μl模板dna1μl,10pmol/μl引物f和r各2μl,5u/μltaqdna聚合酶mix预混液(含酶、dntp等)25μl,余量为水。
pcr扩增程序为:98℃3分钟;98℃10秒,54℃10秒,72℃10秒,38个循环;72℃5分钟。
(2)seqidno.2所示caps分子标记的扩增
以步骤1得到的dna为模板,采用正向引物290f:5'-ttgcagaatcccgtgaacca-3'和反向引物762r:5'-agaggacgcttctccagact-3'进行pcr扩增。
pcr反应体系以50μl计为:30ng/μl模板dna1μl,10pmol/μl引物f和r各2μl,5u/μltaqdna聚合酶mix预混液(含酶、dntp等)25μl,余量为水。
pcr扩增程序为:98℃3分钟;98℃10秒,54℃10秒,72℃10秒,38个循环;72℃5分钟。
3、pcr-rflp检测
(1)采用限制内切酶hpych4v对seqidno.1所示caps分子标记的扩增产物进行酶切,酶切反应体系为50μl:1μg/μl模板dna1μl,10×buffer5.0μl,hpych4v酶1μl,余量为水。
pcr-rflp酶切反应条件为:37℃水浴10分钟。
(2)采用限制内切酶hhai对seqidno.2所示caps分子标记的扩增产物进行酶切,酶切反应体系为50μl:1μg/μl模板dna1μl,10×buffer5.0μl,hhai酶1μl,余量为水。
pcr-rflp酶切反应条件为:37℃水浴10分钟。
4、结果分析
3%琼脂糖凝胶电泳检测步骤3的酶切反应产物。若hpych4v酶切产物显示为233bp单一主条带,则待测样品为长芒苋,若显示为284-286bp单一主条带,则待测样品为苋属其它物种;若hhai酶切产物显示条带为213bp单一主条带,则待测样品为糙果苋/沙地苋,若显示为426-429bp的单一主条带,则待测样品为苋属其它物种。
实施例3长芒苋、糙果苋、沙地苋的区分鉴定
以表1所示的52份植物材料样本的单粒种子作为待测样品,利用实施例2提供的区分鉴定长芒苋、糙果苋、沙地苋与其他苋属植物的方法进行长芒苋、糙果苋、沙地苋种子的区分鉴定。
以表1所示的52份植物材料样本中不同植物材料单粒种子的混合样品作为对照。
用特异性引物78f、419r,对长芒苋以及其它苋进行pcr-rflp检测,结果如图1、图3和表4所示。特异性引物78f、419r对长芒苋以及其它苋属植物的单粒种子的扩增成功率为100%。经限制内切酶hpych4v酶切后,结果显示,长芒苋的单粒种子的酶切产物的主条带在233bp,其他苋属植物的单粒种子的主条带在284-286bp。长芒苋与其他苋属植物种子的混合样品中部分有两条主带(284-286bp、233bp),也有部分有一条284-286bpbp主带。切胶纯化酶切产物284-286bp和233bp条带后进行dna测序,测序所得结果为seqidno.1所示全部或部分序列,表明利用本发明的鉴定方法可实现长芒苋和其它苋属种单粒种子的准确区分。
用特异引物290f、762r,对糙果苋和沙地苋以及其它苋进行pcr-rflp检测,结果如图2、图3和表4所示。特异性引物290f、762r对糙果苋和沙地苋以及其它苋属植物的单粒种子样本的扩增成功率为100%。经限制内切酶hhai酶切后,糙果苋和沙地苋的单粒种子的酶切产物的主条带在213bp,其他苋属植物的单粒种子的主条带在426-429bp。糙果苋、沙地苋与其他苋属植物种子的混合样品中部分有两条主带(426-429bp、213bp)。切胶纯化酶切产物426-429bp和213bp条带进行测序分析,糙果苋和沙地苋样品条带的测序结果均与seqidno.2所示的全部或部分序列一致,表明利用本发明的鉴定方法可实现糙果苋、沙地苋与其它苋属种单粒种子的准确区分。
以上不同植物材料单粒种子的混合样品的鉴定中,部分混合样品由于阳性样本(长芒苋、糙果苋和沙地苋)含量有限,pcr产物的特异性片段以及酶切产物都无法得到清晰准确的结果,例如:混合样品30和31在seqidno.1的233位变异位点上的测序结果为杂合子m,而混合样品32在该位点为纯合子c。相应地,在酶切产物的电泳检测中,混合样品31和32仅可见一条284-286bp的主条带,无法判断是否含有长芒苋(见表4和图1)。以上结果表明,本发明的caps分子标记及其检测方法可在苋属植物单粒种子中准确有效的检测、鉴定出长芒苋、糙果苋和沙地苋。
此外,采用本发明实施例2中的caps分子标记及其检测方法对表4中的各样品对应的母体植株的叶片进行pcr-rflp检测,也能够获得与以种子为样本相同的检测结果。
表4苋属单粒种子或混合样品hpych4v和hhai限制性内切酶酶切结果
虽然,上文中已经用一般性说明及具体实施方案对本发明作了详尽的描述,但在本发明基础上,可以对之作一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。
序列表
<110>中国检验检疫科学研究院
<120>鉴定长芒苋、糙果苋和沙地苋单粒种子的基于snp位点的caps分子标记及其应用
<130>khp201111495.4
<160>6
<170>siposequencelisting1.0
<210>1
<211>285
<212>dna
<213>人工序列(artificialsequence)
<400>1
atggtccggtgaagtgttcggatcgcgccgacgtgggcggttcgccgccggcgacgtcgc60
gagaagttcactgaaccttatcatttagaggaaggagaagtcgtaacaaggtttccgtag120
gtgaacctgcggaaggatcattgtcgaaacctgccaagcagattgaccagcgaacatgtt180
tatcatacgtggagcggggtgccctagcgaagccttacggacgagctattgcaccctcct240
cccaacgtcgggcggtgctcctttgtgaggggtgctgctcgatgc285
<210>2
<211>426
<212>dna
<213>人工序列(artificialsequence)
<400>2
agcctttggccagggcacgtctgcctgggcgtcacgcactgcgtctcccccaacccgcct60
agctgtgggaggggcgaggaggatggtctcccatgcctcaccgggcgtggatggcctaaa120
acaggagcccacggtttcgagctgctgcggcgattggtggtgtgcaaggcctagcctaga180
atgcaatcgcgtcgtacagcgcgtggaccttgtggccttgaggaccctagagtgttgccc240
gagggcgaccaaccactgcgaccccaggtcaggcgggactacccgctgagtttaagcata300
tcaataagcggaggaaaagaaacttacaaggattcccctagtaacggcgagcgaaccggg360
aatagcccagctttaaaatcgggcggctttgctgtctgaattgtagtctggaggcccttc420
ctctaa426
<210>3
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>3
gtccctgccctttgtacaca20
<210>4
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>4
gggttcgttgttgcatcgag20
<210>5
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>5
ttgcagaatcccgtgaacca20
<210>6
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>6
agaggacgcttctccagact20
- 还没有人留言评论。精彩留言会获得点赞!