一种T7RNA聚合酶和T7启动子的表达系统及使用其在真核生物中表达蛋白质的方法与流程
本申请涉及一种基于hiv-1的vpu和rev-rre元件的t7rna聚合酶(t7rnap)和t7启动子表达系统,该表达系统可用于在真核细胞中持续稳定高效地表达蛋白。
背景技术:
t7表达系统已被广泛地应用于蛋白质的表达,其中在原核生物中的应用最为普遍(参见,专利文献1)。最典型的代表是novagen公司研发的pet系列质粒,这类质粒目前已被广泛地应用在大肠杆菌中高效地表达蛋白质。
对于真核生物,有研究者利用酵母菌株开展了t7表达系统的研究,但却没有得到积极的结果,只检测到t7rnap的表达,没有检测到t7启动子控制的目的蛋白的合成(参见,非专利文献1和2)。
还有研究者在哺乳动物细胞中以病毒(如牛痘病毒)作为载体构建存在于细胞质中的t7表达系统,存在于细胞质中的牛痘病毒载体会随着细胞分裂而丢失;同时,大量复制的牛痘病毒最终也会杀死宿主细胞。因此,该t7表达系统只能实现蛋白质在细胞质中的瞬时表达,难以实现持续稳定地表达目标蛋白(参见,非专利文献1和3)。
为实现真核细胞内持续稳定地表达蛋白质,关键点在于将细胞核内转录后再加工的真核mrna通过核膜成功转运至细胞质,然后才能实现mrna在细胞质内翻译成蛋白质。但是,由于细胞核内t7rnap转录出的mrna没有5′帽子结构和3′poly(a)尾巴,不能被运输出细胞核,因此影响了细胞质内蛋白质的翻译合成,不能高效表达蛋白质。
对于t7rnap转录的缺乏5′cap结构的mrna,有研究发现,在细胞质内利用ires结构可以帮助缺乏5′cap结构的真核生物mrna翻译合成蛋白质(参见,非专利文献4),但是,该研究并未涉及细胞核内的t7rnap转录的缺乏5′cap结构的mrna运输到细胞质的问题。同样,还有研究表明,在t7启动子启动的转录单元中添加真核生物终止子序列可以帮助t7rnap转录产物产生3′poly(a)尾巴(参见,非专利文献5),但是该研究也未解决如何将缺乏5′cap结构的mrna从细胞核运输到细胞质的问题。
现有技术文献:
专利文献1:cn102643851a
非专利文献1:elroy-steino,fuersttr,mossb.cap-independenttranslationofmrnaconferredbyencephalomyocarditisvirus5'sequenceimprovestheperformanceofthevacciniavirus/bacteriophaget7hybridexpressionsystem[j].proceedingsofthenationalacademyofsciences,1989,86(16):6126-30.
非专利文献2:bentonbm,engw-k,dunnjj,etal.signal-mediatedimportofbacteriophaget7rnapolymeraseintothesaccharomycescerevisiaenucleusandspecifictranscriptionoftargetgenes[j].molecularandcellularbiology,1990,10(1):353-60.
非专利文献3:wangw,liy,wangy,etal.bacteriophaget7transcriptionsystem:anenablingtoolinsyntheticbiology[j].biotechnologyadvances,2018.
非专利文献4:elroy-steino,mossb.cytoplasmicexpressionsystembasedonconstitutivesynthesisofbacteriophaget7rnapolymeraseinmammaliancells[j].proceedingsofthenationalacademyofsciences,1990,87(17):6743-7.
非专利文献5:kendower,michaelrosbash,t7rnapolymerase-directedtranscriptsareprocessedinyeastandlink3′endformationtomrnanuclearexport[j].rna,2002,8:686–697.
技术实现要素:
发明要解决的问题
本发明主要解决真核生物中t7表达系统转录的无5′cap结构的mrna跨核膜运输到细胞质,连续稳定高效地表达重组蛋白的问题。
用于解决问题的方案
本发明人发现,人类免疫缺陷病毒(hiv-1)逆转录病毒基因组上存在编码rev蛋白的基因,与rev蛋白特异性结合的rna称为rev响应元件(rev-responsiveelement,rre)。rev蛋白可以结合rre,通过与剪接因子、剪接体相互作用抑制剪接,同时与核孔蛋白相互作用,将带有rre的不完全剪接mrna从细胞核运至细胞质。
hiv-1逆转录病毒还编码小的调节蛋白,其中vpu蛋白是由81个氨基酸寡聚形成的膜蛋白,折叠成两个不同的结构域,即n端跨膜疏水性结构域和c端细胞质结构域。由于具有该结构,vpu蛋白以寡聚体形式穿入细胞膜形成通道,能够改善细胞膜的通透性。
核定位信号(nuclearlocalizationsingal,nls)通常为短的氨基酸序列,它能辅助目标蛋白进入细胞核,核定位信号的引入可以使蛋白进入细胞核。当在膜蛋白上游添加nls,膜蛋白会定位在细胞核核膜上。
本发明人基于以上事实,为解决t7表达系统转录mrna跨核膜转运到细胞质的问题,提出如下两种设计方案:
1)在t7表达系统中引入hiv-1逆转录病毒中的rev-rre调控元件,rev蛋白可以特异性结合rre序列,将带有rre序列的缺乏5`cap结构t7rnap转录的mrna从细胞核运输至细胞质;
2)在t7表达系统中引入带有核定位序列的vpu基因,使其蛋白定位在细胞核核膜上,从而改变核膜的通透性,使t7rnap转录的产物通过渗透扩散进入细胞质。
上述设计方案对于所适用的真核生物不进行特别限制,只要能实现t7rnap转录的缺乏5′cap结构的mrna跨核膜运输到细胞质即可。优选地,适用于酵母、哺乳动物、昆虫。更优选地,适用于酿酒酵母(sachromycescerevisiae)、毕赤酵母(pichiapastoris)、马克斯鲁维酵母(kluyveromycesmarxianus)、仓鼠、人肾细胞、人hela细胞、cho、cos、bhk、sp2/0、nih3t3、蚕、果蝇。最优选地,适用于酿酒酵母(sachromycescerevisiae)和人hela细胞。
优选地,针对不同的真核生物可使用如下两种表达系统:
(一)对于酵母,构建基于hiv-1的vpu的t7表达系统。
(二)对于哺乳动物,构建基于hiv-1的vpu或rev-rre元件的t7表达系统。
为验证上述t7表达系统在真核生物中是否表达目标蛋白,本发明使用了潮霉素抗性蛋白(hph)、nanoluc萤光素酶(nluc)或增强型绿色荧光蛋白(egfp),将其构建于t7启动子启动的转录单元。
验证实验证明了上述两种设计方案的合理性,两种t7表达系统均实现了预期的效果。结果表明,vpu蛋白起到了改变核膜通透性的作用,帮助t7rnap转录的mrna出核,完成了翻译过程;携带rre的mrna也与rev蛋白结合,帮助t7rnap转录的mrna出核,完成了翻译过程。
具体地,本发明的技术方案包括如下:
[1]、一种t7rna聚合酶和t7启动子的表达系统,其中,所述表达系统包含hiv-1逆转录病毒中的vpu蛋白和/或rev-rre调控元件。
[2]、根据[1]所述的表达系统,其中,所述t7rna聚合酶由seqidno:1所表示的序列编码。
[3]、根据[1]或[2]所述的表达系统,其中,所述t7rna聚合酶的基因还含有核定位序列。
[4]、一种使用t7rna聚合酶和t7启动子的表达系统在真核生物中表达蛋白质的方法,其中,所述表达系统包含hiv-1逆转录病毒中的vpu蛋白和/或rev-rre调控元件。
[5]、根据[4]所述的方法,其中,所述方法包括:
1)合成所述带有核定位序列的t7rna聚合酶基因序列,所述t7rna聚合酶由seqidno:1所表示的序列编码,将所述带有核定位序列的t7rna聚合酶基因插入到整合型载体中;
2)将所述整合型载体线性化,回收线性化片段,转化到真核菌株中,通过抗性筛选得到基因组上整合了带有核定位序列的t7rna聚合酶基因的真核重组菌株;
3)构建具有携带核定位序列的vpu基因的载体,以含有目标蛋白基因的质粒为模板扩增包含有t7启动子的目标蛋白dna片段,回收所述包含有t7启动子的目标蛋白片段,将其插入具有前述携带核定位序列的vpu基因的载体中,得到含有所述vpu基因和目标蛋白基因的载体;和
4)将3)得到的所述载体转化到2)中的真核重组菌株中,构建基于hiv-1逆转录病毒中的vpu蛋白的t7rna聚合酶的表达系统。
[6]、根据[4]所述的方法,其中,所述方法包括:
1)构建表达t7rna聚合酶和rev的载体,所述t7rna聚合酶由seqidno:1所表示的序列编码;
2)将含有t7启动子、t7终止子、ires、rre和目标蛋白基因的dna片段转移至所述载体,构建能够表达t7rna聚合酶、rev和目标蛋白的载体;
3)用2)中构建的载体进行转染,完成慢病毒包装;和
4)用所述慢病毒感染目标真核细胞,通过抗性筛选得到目标转化子,通过验证目标蛋白确定所述表达系统。
[7]、根据[4]所述的方法,其中,所述方法包括:
1)构建表达t7rna聚合酶和vpu的载体,所述t7rna聚合酶由seqidno:1所表示的序列编码;
2)将含有t7启动子、t7终止子、ires和目标蛋白基因的dna片段转移至所述载体,构建能够表达t7rna聚合酶、vpu、目标蛋白的载体;
3)用2)中构建的载体进行转染,完成慢病毒包装;和
4)用所述慢病毒感染目标真核细胞,通过抗性筛选得到目标转化子,通过验证目标蛋白确定所述表达系统。
[8]、根据[6]所述的方法,其中,在所述步骤1)中,选取哺乳动物的启动子作为表达t7rna聚合酶和rev的启动子,构建含有2a或者ires的真核多顺反子结构,得到同时表达t7rna聚合酶和rev的载体。
[9]、根据[7]所述的方法,其中,在所述步骤1)中,选取哺乳动物的启动子作为表达t7rna聚合酶和vpu的启动子,构建含有2a或者ires的真核多顺反子结构,得到同时表达t7rna聚合酶和vpu蛋白的载体。
[10]、根据[4]~[9]任一项所述的方法,其中,使用潮霉素抗性蛋白、nanoluc萤光素酶或增强型绿色荧光蛋白验证所述表达系统表达的目标蛋白。
[11]、根据[4]~[10]任一项所述的方法,其中,所述核定位序列为sv40t-antigen核定位序列、nucleoplasmin核定位序列、egl-13核定位序列、c-myc核定位序列或tus-protein核定位序列。
[12]、根据[1]~[11]任一项所述的方法,其中,所述真核生物为酵母、哺乳动物或昆虫。
发明的效果
本发明通过构建基于hiv-1的vpu和/或rev-rre元件的t7表达系统,显著改善了t7rnap转录的缺乏5′cap结构的mrna从细胞核跨膜向细胞质的运输,促进其翻译合成蛋白质,实现了基于t7表达系统的重组蛋白在真核细胞内的持续稳定高效地表达。
附图说明
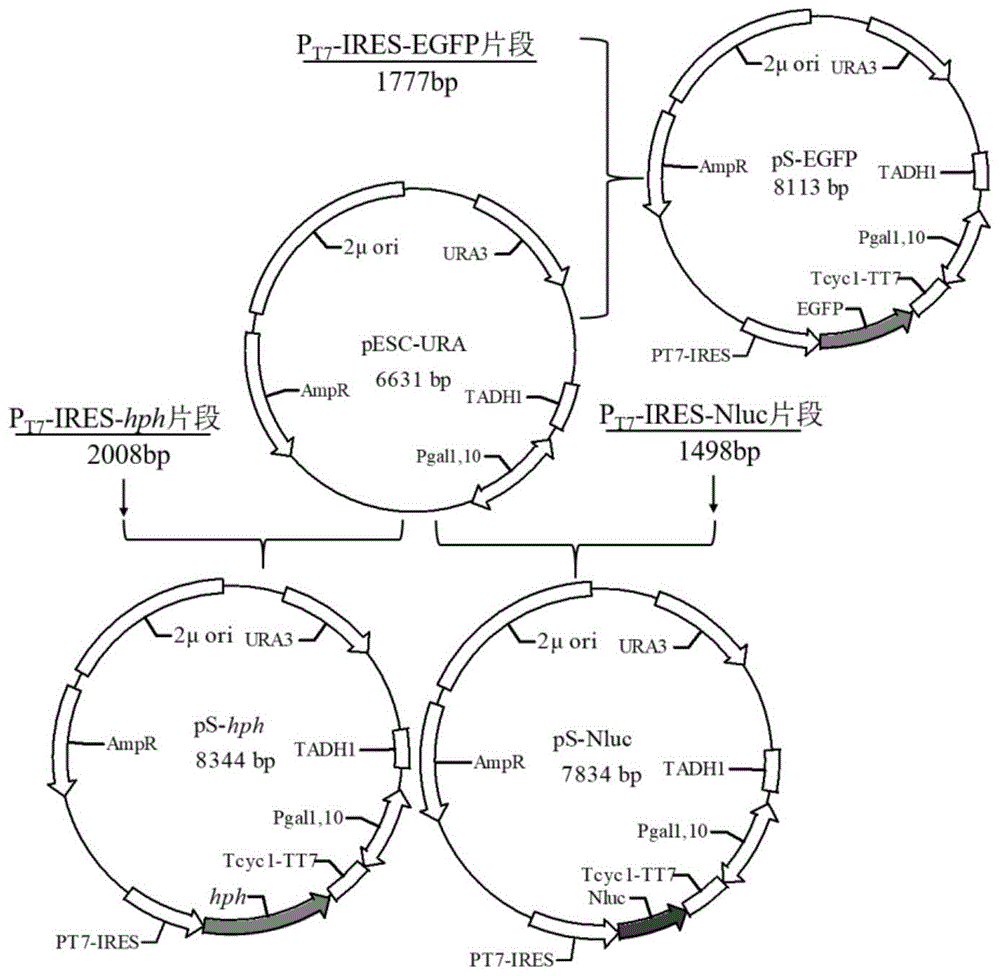
图1为ps-t7rnap质粒构建的示意图。
图2为酿酒酵母基因工程菌株by4741(ho::nls-t7rnap)的构建结果的pcr验证图。其中,泳道1是对照菌by4741基因组pcr结果的电泳图;泳道2是by4741(ho::nls-t7rnap)基因组pcr结果的电泳图。
图3为ps-hph质粒构建的示意图。
图4为ps-vpu质粒构建的示意图。
图5为ps-vpu/hph质粒构建的示意图。
图6为潮霉素对不同酿酒酵母重组菌株的生长速度影响的效果图。其中,control代表by4741(ho::nls-t7rnap,pesc-ura)菌株;yhph代表by4741(ho::nls-t7rnap,ps-hph)菌株;yvpu-hph代表by4741(ho::nls-t7rnap,ps-vpu/hph)菌株。
图7为潮霉素对不同酿酒酵母重组菌株的菌落生长影响的效果图。其中,control代表by4741(ho::nls-t7rnap,pesc-ura)菌株;yhph代表by4741(ho::nls-t7rnap,ps-hph)菌株;yvpu-hph代表by4741(ho::nls-t7rnap,ps-vpu/hph)菌株,潮霉素浓度分别为0μg/ml,50μg/ml,100μg/ml,150μg/ml,200μg/ml。
图8为vpu对t7表达系统在酿酒酵母中表达nluc影响的效果图。其中,control代表by4741(ho::nls-t7rnap,pesc-ura)菌株;ynluc代表by4741(ho::nls-t7rnap,ps-nluc)菌株;yvpu-nluc代表by4741(ho::nls-t7rnap,ps-vpu/nluc)菌株。
图9为vpu对t7表达系统在酿酒酵母中表达egfp影响的效果图。其中,control代表by4741(ho::nls-t7rnap,pesc-ura)菌株;yegfp代表by4741(ho::nls-t7rnap,ps-egfp)菌株;yvpu-egfp代表by4741(ho::nls-t7rnap,ps-vpu/egfp)菌株。
图10为pmscvpuro-t7rnap和pmscvpuro-t7rnap-rev质粒构建的示意图。
图11为pmscvpuro-t7rnap-nluc-rre和pmscvpuro-t7rnap-rev-nluc-rre质粒构建的示意图。
图12为rev-rre对t7表达系统在人hela细胞株中表达nluc影响的效果图。其中,w是野生型人hela细胞株;w-t7rp是引入pmscvpuro-t7rnap-rev的人hela细胞株;w-rev-t7rp是引入pmscvpuro-t7rnap-rev-nluc-rre的人hela细胞株。
图13为pmscvpuro-t7rnap-vpu质粒构建的示意图。
图14为pmscvpuro-t7rnap-nluc和pmscvpuro-t7rnap-vpu-nluc质粒构建的示意图。
图15为vpu对t7表达系统在人hela细胞株中表达nluc影响的效果图。其中,w是野生型人hela细胞株;w-t7rp是引入pmscvpuro-t7rnap-nluc的人hela细胞株;w-vpu-t7rp是引入pmscvpuro-t7rnap-vpu-nluc的人hela细胞株。
图16为ps-rev/egfp/rre质粒构建的示意图。
图17为pmscvpuro-t7rnap-egfp和pmscvpuro-t7rnap-rev-egfp-rre质粒构建的示意图。
具体实施方式
以下结合实施例进一步解释本发明,需要说明的是,以下实施例仅用于说明本发明,本发明的保护范围不限于此。
<实验材料>
载体:pmri31(genbank:kj502281.1);pesc-ura(genbank:af063585.2);pmscvpuro,购自上海酶研生物科技有限公司;pumvc,购自上海酶研生物科技有限公司;pmd2.g,购自addgene(catalog:12259)公司。
菌株:酿酒酵母菌株by4741,购自invitrogen公司。
细胞株:293t细胞,购自北京北纳创联生物技术研究院;hela细胞,购自北京北纳创联生物技术研究院。
培养基:ypd培养基,购自北京索莱宝科技有限公司;sd-ura培养基,购自北京索莱宝科技有限公司;deme培养基,购自北京索莱宝科技有限公司;opti-mem减血清培养基购自北京索莱宝科技有限公司。
试剂:胎牛血清(fbs),购自北京全式金生物技术有限公司;10×pbs缓冲液,购自北京索莱宝科技有限公司;
<质粒和载体的构建以及蛋白的表达方法>
需要说明的是,本申请中涉及的常规的质粒和载体的构建、蛋白的表达方法以及慢病毒包装等方法,可参照本领域公知的分子生物学及遗传学方法,例如,可参照“本领域的常规生物学方法”、“最新分子生物学实验方法汇编(currentprotocolsinmolecularbiology,wiley出版)”、“分子克隆实验指南(molecularcloning:alaboratorymanual,冷泉港实验室出版)”等公开出版物中记载的相应方法。
<实施例1>基于hiv-1vpu蛋白的t7表达系统在酿酒酵母中的构建和表达
1.ps-t7rnap质粒的构建
设计合成带有核定位信号(nuclearlocalizationsignal,nls)的t7rnap(genbank:ky484013.1),其中t7rnap的核苷酸序列如seqidno:1所示,带有核定位信号(nls)的核苷酸序列为如seqidno:2所示。
seqidno:2:atgcccaagaagaagcggaaggtc
将nls-t7rnap基因片段插入到pmri31载体gal1,10诱导型启动子(genbank:k02115.1)的下游,构建质粒命名为ps-t7rnap,所用的tcyc1核苷酸序列如seqidno:3所示,构建过程如图1。
2.基因工程菌株by4741(ho::nls-t7rnap)的构建
将质粒ps-t7rnap使用sfiⅰ核酸内切酶线性化。回收线性化质粒片段,电转入酿酒酵母菌株by4741,通过遗传霉素g418抗性筛选得到整合了t7rnap基因的酿酒酵母重组菌株by4741(ho::nls-t7rnap)。为了确保是阳性转化子,提取该酿酒酵母重组菌株和空白菌株的基因组并用特定引物ho3验证t7rnap基因是否正确插入到酿酒酵母基因组ho区域上,验证结果如图2。
ho3的核苷酸序列如下:
hof3:cgtgcctgcgatgagatac(seqidno:4)
hor3:ggcgtatttctactccagca(seqidno:5)
对照组扩增出来的大小为2947bp的片段,表明没有插入片段,而实验组扩出了7672bp的片段,说明所述t7rnap基因成功插入到了酿酒酵母by4741染色体上。
3.质粒ps-vpu/hph的构建
根据提供的基因序列合成pt7-ires-hph片段,pt7序列为seqidno:6所示的序列,hph的序列为seqidno:7所示的序列,tt7序列为seqidno:8所示的序列,将此片段通过无缝克隆的方法插入到载体pesc-ura中,完成质粒ps-hph的构建,构建流程如图3。同时,将携带核定位序列(nls)的vpu基因(如seqidno:9所示)插入载体pesc-ura中gal双向启动子的下游,构建质粒ps-vpu,构建流程如图4。
以质粒ps-hph为模板扩增pt7-ires-hphdna片段,所使用的引物如下:
f:tcaaggagaaaaaaccaaaaaacccctcaaggccc(seqidno:10)
r:ttaatgcagctggattaatacgactcactataggt(seqidno:11)
最后将回收的pt7-ires-hph片段同样采用无缝克隆的方法插入质粒载体ps-vpu,构建质粒ps-vpu/hph,载体构建过程如图5。
4.基因工程菌株by4741(ho::nls-t7rnap,ps-vpu/hph)的构建
将最终目标质粒ps-vpu/hph转入酿酒酵母菌by4741(ho::nls-t7rnap),得到酿酒酵母重组菌株by4741(ho::nls-t7rnap,ps-vpu/hph)。具体操作参照以下酿酒酵母感受态的制备及转化步骤。
酿酒酵母感受态的制备过程如下所示:
(1)从平板上挑单一酵母菌落于5ml的ypd液体培养基中,30℃下培养12h。再从中吸取500μl培养液至50ml的ypd液体培养基,继续30℃培养18-24h至od600=2左右即可。
(2)菌液转至灭菌的50ml离心管,5000rpm离心5min后去上清,然后用30ml的预冷灭菌水重悬菌体,然后5000rpm离心5min去上清。
(3)用20ml预冷灭菌的1m山梨醇重悬菌体,去上清。最后再使用200μl-500μl的1m山梨醇重悬细胞转移至1.5ml离心管,即得到酿酒酵母感受态细胞。每次做酵母转化时都需制备新鲜的感受态以保证高转化率。
酿酒酵母电转转化步骤如下:
(1)取3~5μl的线性质粒或质粒(浓度≥300ng/μl)和40μl的酿酒酵母感受态细胞于预冷过的1.5ml离心管混合均匀,并转移至2mm的电转杯中,冰上预冷5min。
(2)用纸擦净电转杯上的水滴,采用电转仪在1500v的电击强度下电击。
(3)在电转杯中加入1ml的ypd培养基,轻轻悬浮转移至灭菌的1.5ml离心管,30℃下静置复苏2h。待复苏后,用灭菌水清洗3遍,涂适量的细胞于相应的平板上。30℃下培养2-3天直至出现单菌落。
5.所构建的t7表达系统在酿酒酵母中表达重组蛋白效果的检测
控制t7rnap基因表达的是gal启动子,该启动子需要在以半乳糖为碳源的诱导条件下所表达,所以酿酒酵母重组菌株可首先在sd-ura中培养24h后再用sg-ura诱导培养48h,否则t7rna聚合酶不会参与t7表达系统发挥自身的功能。
潮霉素抗性蛋白(hph)报告基因在酿酒酵母重组菌株中的表达检测
<菌株生长速率>
参照实施例1中4的方法将载体pesc-ura和ps-hph分别转入酿酒酵母重组菌株by4741(ho::nls-t7rnap),得到对照菌株by4741(ho::nls-t7rnap,pesc-ura)和菌株by4741(ho::nls-t7rnap,ps-hph)。将这两个对照菌和实验组菌株by4741(ho::nls-t7rnap,ps-vpu/hph)分别在sd-ura液体培养基中培养24h后,稀释调整od600,使三种菌株的od600值一致,按1%的转接量接菌至sg-ura液体培养基,并添加潮霉素终浓度为400μg/ml,每隔12h测菌液od600,通过菌株的生长情况来反应表达抗性蛋白的量,结果如图6。
从图6中可以看出,对照菌株by4741(ho::nls-t7rnap,pesc-ura)(以control表示)在400μg/ml潮霉素浓度中基本不生长,携带vpu基因的菌株by4741(ho::nls-t7rnap,ps-vpu/hph)(以yvpu-hph表示)比不携带vpu基因的菌株by4741(ho::nls-t7rnap,ps-hph)(以yhph表示)生长要快,说明携带vpu基因的菌株能表达更多的抗性蛋白,因而菌株生长快。该实验结果说明含有vpu蛋白的t7表达系统利用vpu蛋白提高细胞核核膜的通透性,使更多的t7rnap转录的mrna运输到细胞质,完成翻译过程,合成较多抗性蛋白。
<菌落大小和生长状态>
将酿酒酵母菌by4741(ho::nls-t7rnap,pesc-ura)、重组菌by4741(ho::nls-t7rnap,ps-hph)和重组菌株by4741(ho::nls-t7rnap,ps-vpu/hph)、在sd-ura液体培养基中培养24h后,稀释调节三种菌株的菌体浓度,使其od600值保持一致,稀释后点菌在含不同浓度潮霉素的sg-ura固体平板,潮霉素浓度依次为0μg/ml,50μg/ml,100μg/ml,150μg/ml,200μg/ml(参见图7,最上端数字代表潮霉素的浓度)。3天后观察菌落的大小和生长状态,结果如图7所示。
从图7中可以看出,潮霉素浓度为0μg/ml时,菌株都能正常生长;随着潮霉素浓度的增加,不携带抗性基因的by4741(ho::nls-t7rnap,pesc-ura)(以control表示)生长受到抑制,直至不能生长。携带vpu基因的by4741(ho::nls-t7rnap,ps-vpu/hph)(以yvpu-hph表示)比不携带vpu基因的by4741(ho::nls-t7rnap,ps-hph)(以yhph表示)的菌落数要多,说明vpu蛋白的引入能提高细胞核核膜的通透性,使更多的t7rnap转录的mrna穿过细胞核核膜进入细胞质,合成潮霉素抗性蛋白质。
nanoluctm萤光素酶(nluc)报告基因在重组酿酒酵母菌株中的表达检测
参照实施例1中3和4的方法构建质粒和酿酒酵母重组菌株,得到酵母菌株by4741(ho::nls-t7rnap,pesc-ura),by4741(ho::nls-t7rnap,ps-nluc)、by4741(ho::nls-t7rnap,ps-vpu/nluc)。nluc的核苷酸序列如seqidno:12所示。
将上述三株菌株分别进行如下操作:在sd-ura液体培养基中培养24h后,按1%的接菌量转接到sg-ura液体培养基中培养至一定浓度后,离心收集菌体,用过滤除菌的pbs洗涤3次,重悬于无菌的pbs。
检测方法:将nano-glotm底物与试剂盒提供的裂解缓冲液以1:50稀释,并与酵母细胞以1:10混合,转移200μl样品到白色96孔板中,立即使用多功能酶标仪的luminescence模式测定生物发光强度,200μl样品转移到96孔透明板用于测定od600。为了比较不同菌之间的差异,实验中的平均生物发光强度用生物发光强度除以od600。样品在测定之前要稀释到合适的浓度(od600=0.3~0.8),结果如图8所示。
图8中可以看出,不含萤光素酶基因的对照菌株(以control表示)基本没有检测到生物发光信号,携带vpu基因的菌株by4741(ho::nls-t7rnap,ps-vpu/nluc)(以yvpu-nluc表示)生物发光强度是不携带vpu基因的菌株by4741(ho::nls-t7rnap,ps-nluc)(以ynluc表示)的2.0倍。该结果表明vpu蛋白提高了细胞核核膜的通透性,从而促进t7rnap转录的mrna跨膜运输到细胞质中,因此比未携带vpu蛋白的t7表达系统合成更多的蛋白质,实现了高效表达重组蛋白的目的。
增强型绿色荧光蛋白(egfp)报告基因在重组酿酒酵母菌株中的表达检测
参照实施例1中3和4的方法构建质粒和酿酒酵母重组菌株,得到酵母菌株by4741(ho::nls-t7rnap,pesc-ura)、by4741(ho::nls-t7rnap,ps-egfp)和by4741(ho::nls-t7rnap,ps-vpu/egfp)。egfp的核苷酸序列如seqidno:13所示。
对上述三株菌株分别进行如下操作:在sd-ura液体培养基中培养24h后,按1%的接菌量转接到sg-ura液体培养基中培养至一定浓度后,离心收集菌体,用无菌水洗涤3次,重悬于无菌水,通过多功能酶标仪检测荧光基因表达,结果如表1和图9所示。
表1不同酿酒酵母重组菌株的平均荧光强度
表1中,control代表by4741(ho::nls-t7rnap,pesc-ura)菌株;yegfp代表by4741(ho::nls-t7rnap,ps-egfp)菌株;yvpu-egfp代表by4741(ho::nls-t7rnap,ps-vpu/egfp)菌株。
从图9中也可以看出,by4741(ho::nls-t7rnap,ps-vpu/egfp)菌株的荧光强度显著高于by4741(ho::nls-t7rnap,pesc-ura)菌株和by4741(ho::nls-t7rnap,ps-egfp),说明vpu蛋白提高了细胞核核膜的通透性,从而促进t7rnap转录的mrna跨膜运输到细胞质中,因此比未携带vpu蛋白的t7表达系统合成更多的蛋白质,实现了高效表达重组蛋白的目的。
<实施例2>基于hiv-1rev-rre调控元件的t7表达系统在哺乳动物细胞中的构建和表达
1.慢病毒表达载体pmscvpuro-t7rnap-rev-nluc-rre的构建
选取人源磷酸甘油酸激酶1(pgk1)基因(如seqidno:14所示)的启动子作为表达t7rnap和rev的启动子,t7rnap和rev(如seqidno:15所示)蛋白的共表达通过2a(如seqidno:16所示)或者ires(如seqidno:17所示)连接来实现,最终构建得到pmscvpuro-t7rnap和pmscvpuro-t7rnap-rev质粒。以2a连接为例,构建流程如图10所示。
分别合成pgk1、nls-t7rnap、sv40poly(a)(如seqidno:18所示)、2a-rev-sv40poly(a)片段,采用融合pcr技术将片段融合得到大片段pgk1-t7rnap-sv40poly(a)和pgk1-t7rnap-2a-rev-sv40poly(a),最后将dna片段pgk1-t7rnap-sv40poly(a)和pgk1-t7rnap-2a-rev-sv40poly(a)转移至慢病毒表达载体pmscvpuro上,完成质粒pmscvpuro-t7rnap和pmscvpuro-t7rnap-rev的构建。
合成pt7-ires-nluc-rre片段,rre的序列为seqidno:19所示的序列。最后将dna片段pt7-ires-nluc-rre转移至载体pmscvpuro-t7rnap和pmscvpuro-t7rnap-rev上,完成质粒pmscvpuro-t7rnap-nluc-rre和pmscvpuro-t7rnap-rev-nluc-rre构建,具体构建流程如图11所示。
2.293t细胞包装慢病毒,感染人hela细胞
将上述所构建载体pmscvpuro-t7rnap-nluc-rre和pmscvpuro-t7rnap-rev-nluc-rre转染293t细胞,完成慢病毒包装。通过嘌呤霉素筛选成功转化的转化子。具体操作参照以下293t细胞包装慢病毒及慢病毒感染目的细胞的步骤。
293t细胞包装慢病毒过程如下所示:
(1)当细胞生长至九成满时,弃去细胞培养基,用无菌pbs轻柔润洗一次,加入1ml0.25%胰酶消化。在显微镜下观察到细胞被消化变圆后,弃去胰酶,加入10%fbs的dmem培养基重悬293t细胞,计数5×105个细胞分至6孔细胞培养板的每个孔。
(2)24h后,转染293t细胞:
a:用470μlopti-mem稀释30ulfugene9,并充分轻轻地混匀,室温下静置5min。
b:准备目标质粒和包装载体质粒:
两个包装载体质粒pumvc:1μg;pmd2.g:0.5μg
目标质粒:1μg。
c:将上述准备的质粒混合液滴加到opti-mem与fugene9的混合液中,室温静置20min;在接种有细胞、含100μl培养基的6孔板的每个孔中加入2–10μl上述混合物。吹吸或使用振荡器振荡10~30s进行混匀,将细胞放回培养箱继续培养。
d:6~8h后对293t细胞换液,注意加液时,不要吹起细胞(沿着皿壁,轻轻地加新培养液),置37℃细胞培养箱中继续培养48h。
(3)目的细胞的铺板:
目的细胞需要提前一天铺板至六孔细胞培养板中(细胞接种个数是2×105~3×105个),以保证感染时的密度为30~40%;同时准备一个不感染病毒的对照组。第二天弃上清液,加入含有病毒的培养基进行感染。
(4)慢病毒液的收集:
转染48h后(此时长满293t细胞),收集上清液于15ml离心管中,3000rpm离心20min,将上清液用0.45μm滤头过滤。再向六孔板加适量培养液,24h之后,可以再次收集上清液,按照每次用量分装,保存于-80℃冰箱中。
慢病毒感染目的细胞(人hela细胞)的步骤如下:
(1)配置3ml培养基1640(10%fbs),然后加入3ml病毒上清液(剩余的病毒上清液放-80℃冰箱中保存),24h之后补加4ml1640培养液,保证细胞处于良好状态;
(2)细胞培养板置于细胞培养箱中再培养24h;
(3)48h后,弃上清,更换新鲜培养基6~8ml,并加入相应浓度的嘌呤霉素筛选,杀死未转入dna的细胞;
(4)72h后,选择阴性对照组全部死亡且转染dna组有适量存活的细胞进行单克隆培养,将孔内细胞用有限稀释法分单个细胞到细胞培养用96孔板继续培养。
(5)培养10d左右,挑选细胞单克隆,进行检测。
3.所构建的t7表达系统在哺乳动物细胞中表达重组蛋白效果的检测。
nluc报告基因在人hela细胞株中的表达检测
野生人hela细胞株作为对照,用胰蛋白酶消化野生hela细胞和感染慢病毒的细胞至悬浮,用无菌pbs洗涤3次,并将细胞重悬于无菌pbs。
检测方法:将
从图12中可以看出,野生人hela细胞株(以w表示)基本没有检测到生物发光信号。携带rev-rre元件的人hela细胞株(以w-rev-t7rp表示)生物发光强度是不携带rev-rre元件的人hela细胞株(以w-t7rp表示)生物发光强度的4.3倍,说明携带rre元件的mrna可以与rev蛋白结合,促进t7rnap转录的mrna出核,完成翻译过程,连续稳定地表达目标蛋白。
<实施例3>基于hiv-1vpu蛋白的t7表达系统在哺乳动物细胞中的构建和表达
1.慢病毒表达载体pmscvpuro-t7rnap-vpu-nluc的构建
选取人源磷酸甘油酸激酶基因的启动子pgk1作为表达t7rnap和vpu的启动子,t7rnap和vpu蛋白的表达通过2a或者ires连接来实现,最终构建得到pmscvpuro-t7rnap-vpu。以2a连接为例,构建流程如图13所示。
分别合成片段pgk1、nls-t7rnap、2a-vpu-nls-sv40poly(a),采用融合pcr技术将三个片段融合得到大片段pgk1-t7rnap-2a-vpu-sv40poly(a),最后通过无缝克隆方法将dna片段pgk1-t7rnap-2a-vpu-sv40poly(a)转移至慢病毒表达载体pmscvpuro上,完成质粒pmscvpuro-t7rnap-vpu的构建。
合成pt7-ires-nluc片段,通过无缝克隆的方法将dna片段pt7-ires-nluc转移至载体pmscvpuro-t7rnap和pmscvpuro-t7rnap-vpu,完成质粒pmscvpuro-t7rnap-nluc和pmscvpuro-t7rnap-vpu-nluc的构建,具体构建流程如图14所示。
2.293t细胞包装慢病毒,感染人hela细胞
将上述所构建载体pmscvpuro-t7rnap-nluc和pmscvpuro-t7rnap-vpu-nluc转染293t细胞,完成慢病毒包装。通过嘌呤霉素筛选成功转化的转化子,具体操作参照实施例2中293t细胞包装慢病毒及慢病毒感染目的细胞的步骤。
3.所构建的t7表达系统在哺乳动物细胞中表达重组蛋白效果的检测。
nluc报告基因在人hela细胞中的表达检测
野生人hela细胞株作为对照,用胰酶消化野生人hela细胞和感染慢病毒的细胞至悬浮,用无菌pbs洗涤3次,并将细胞重悬于无菌pbs。
检测方法:将
从图15可以看出,野生人hela细胞株(以w表示)基本没有检测到生物发光信号。携带vpu蛋白的人hela细胞株(以w-vpu-t7rp表示)生物发光强度是不携带vpu蛋白的人hela细胞株(以w-t7rp表示)生物发光强度的4.5倍,说明vpu蛋白可以提高细胞核核膜通透性,帮助t7rnap转录的mrna出核,完成翻译过程,实现连续稳定高效地表达目标蛋白。
<实施例4>基于rev-rre的t7表达系统在酿酒酵母与人hela细胞中表达蛋白的差异
参照实施例1构建适用于酿酒酵母的基于rev-rre的t7表达系统,首先将rev基因片段(参见,序列表中的seqidno:3)插入载体pesc-uragal双向启动子的下游,构建质粒载体ps-rev。合成pt7-ires-egfp-rre片段,用无缝克隆的方法将前述片段插入质载体ps-rev,构建质粒ps-rev/egfp/rre,载体构建过程如图16所示。参照实施例1中4的方法构建酿酒酵母菌株by4741(ho::nls-t7rnap,ps-rev/egfp/rre)。参照实施例2以egfp为报告基因,构建适用于人hela细胞的基于rev-rre的t7表达载体pmscvpuro-t7rnap-rev-egfp-rre,构建过程如图17所示。以增强型绿色荧光蛋白(egfp)为报告基因按照实施例1的方法进行测定,比较基于rev-rre调控元件的t7表达系统在酿酒酵母和人hela细胞中表达重组蛋白的作用,结果如表2所示。
表2酿酒酵母与人hela细胞的平均荧光强度
表2中,control1代表酿酒酵母by4741(ho::nls-t7rnap,pesc-ura)菌株;control2代表野生人hela细胞株;yrev-rre-egfp代表酿酒酵母by4741(ho::nls-t7rnap,ps-rev/egfp/rre)菌株;w-t7rp-rev-egfp-rre代表引入pmscvpuro-t7rnap-rev-egfp-rre人hela细胞株。
从表2可以看出,基于rev-rre调控元件的t7表达系统,在酿酒酵母中control1和yrev-rre-egfp间荧光强度差异不大,说明yrev-rre-egfp基本没有绿色荧光蛋白的表达。在人hela细胞中,w-t7rp-rev-egfp-rre的荧光强度是control2的21.3倍,说明基于rev-rre元件的t7表达系统在哺乳动物细胞的蛋白表达优于酿酒酵母。
产业上的可利用性
本申请开发了一种用于真核生物生产蛋白质的表达系统,通过hiv-1vpu膜蛋白和rev-rre调控元件帮助t7rnap转录的缺乏5′cap结构的mrna运输到细胞质,从而实现在真核细胞中持续稳定高效地表达重组蛋白。
序列表
<110>北京化工大学
<120>一种t7rna聚合酶和t7启动子的表达系统及使用其在真核生物中表达蛋白质的方法
<160>19
<170>siposequencelisting1.0
<210>1
<211>2652
<212>dna
<213>人工序列(artificialsequence)
<400>1
atgaacacgattaacatcgctaagaacgacttctctgacatcgaactggctgctatcccg60
ttcaacactctggctgaccattacggtgagcgtttagctcgcgaacagttggcccttgag120
catgagtcttacgagatgggtgaagcacgcttccgcaagatgtttgagcgtcaacttaaa180
gctggtgaggttgcggataacgctgccgccaagcctctcatcactaccctactccctaag240
atgattgcacgcatcaacgactggtttgaggaagtgaaagctaagcgcggcaagcgcccg300
acagccttccagttcctgcaagaaatcaagccggaagccgtagcgtacatcaccattaag360
accactctggcttgcctaaccagtgctgacaatacaaccgttcaggctgtagcaagcgca420
atcggtcgggccattgaggacgaggctcgcttcggtcgtatccgtgaccttgaagctaag480
cacttcaagaaaaacgttgaggaacaactcaacaagcgcgtagggcacgtctacaagaaa540
gcatttatgcaagttgtcgaggctgacatgctctctaagggtctactcggtggcgaggcg600
tggtcttcgtggcataaggaagactctattcatgtaggagtacgctgcatcgagatgctc660
attgagtcaaccggaatggttagcttacaccgccaaaatgctggcgtagtaggtcaagac720
tctgagactatcgaactcgcacctgaatacgctgaggctatcgcaacccgtgcaggtgcg780
ctggctggcatctctccgatgttccaaccttgcgtagttcctcctaagccgtggactggc840
attactggtggtggctattgggctaacggtcgtcgtcctctggcgctggtgcgtactcac900
agtaagaaagcactgatgcgctacgaagacgtttacatgcctgaggtgtacaaagcgatt960
aacattgcgcaaaacaccgcatggaaaatcaacaagaaagtcctagcggtcgccaacgta1020
atcaccaagtggaagcattgtccggtcgaggacatccctgcgattgagcgtgaagaactc1080
ccgatgaaaccggaagacatcgacatgaatcctgaggctctcaccgcgtggaaacgtgct1140
gccgctgctgtgtaccgcaaggacaaggctcgcaagtctcgccgtatcagccttgagttc1200
atgcttgagcaagccaataagtttgctaaccataaggccatctggttcccttacaacatg1260
gactggcgcggtcgtgtttacgctgtgtcaatgttcaacccgcaaggtaacgatatgacc1320
aaaggactgcttacgctggcgaaaggtaaaccaatcggtaaggaaggttactactggctg1380
aaaatccacggtgcaaactgtgcgggtgtcgataaggttccgttccctgagcgcatcaag1440
ttcattgaggaaaaccacgagaacatcatggcttgcgctaagtctccactggagaacact1500
tggtgggctgagcaagattctccgttctgcttccttgcgttctgctttgagtacgctggg1560
gtacagcaccacggcctgagctataactgctcccttccgctggcgtttgacgggtcttgc1620
tctggcatccagcacttctccgcgatgctccgagatgaggtaggtggtcgcgcggttaac1680
ttgcttcctagtgaaaccgttcaggacatctacgggattgttgctaagaaagtcaacgag1740
attctacaagcagacgcaatcaatgggaccgataacgaagtagttaccgtgaccgatgag1800
aacactggtgaaatctctgagaaagtcaagctgggcactaaggcactggctggtcaatgg1860
ctggcttacggtgttactcgcagtgtgactaagcgttcagtcatgacgctggcttacggg1920
tccaaagagttcggcttccgtcaacaagtgctggaagataccattcagccagctattgat1980
tccggcaagggtctgatgttcactcagccgaatcaggctgctggatacatggctaagctg2040
atttgggaatctgtgagcgtgacggtggtagctgcggttgaagcaatgaactggcttaag2100
tctgctgctaagctgctggctgctgaggtcaaagataagaagactggagagattcttcgc2160
aagcgttgcgctgtgcattgggtaactcctgatggtttccctgtgtggcaggaatacaag2220
aagcctattcagacgcgcttgaacctgatgttcctcggtcagttccgcttacagcctacc2280
attaacaccaacaaagatagcgagattgatgcacacaaacaggagtctggtatcgctcct2340
aactttgtacacagccaagacggtagccaccttcgtaagactgtagtgtgggcacacgag2400
aagtacggaatcgaatcttttgcactgattcacgactccttcggtaccattccggctgac2460
gctgcgaacctgttcaaagcagtgcgcgaaactatggttgacacatatgagtcttgtgat2520
gtactggctgatttctacgaccagttcgctgaccagttgcacgagtctcaattggacaaa2580
atgccagcacttccggctaaaggtaacttgaacctccgtgacatcttagagtcggacttc2640
gcgttcgcgtaa2652
<210>2
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>2
atgcccaagaagaagcggaaggtc24
<210>3
<211>246
<212>dna
<213>人工序列(artificialsequence)
<400>3
ttatgtcacgcttacattcacgccctccccccacatccgctctaaccgaaaaggaaggag60
ttagacaacctgaagtctaggtccctatttatttttttatagttatgttagtattaagaa120
cgttatttatatttcaaatttttcttttttttctgtacagacgcgtgtacgcatgtaaca180
ttatactgaaaaccttgcttgagaaggttttgggacgctcgaaggctttaatttgcggcc240
ggtacc246
<210>4
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>4
cgtgcctgcgatgagatac19
<210>5
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>5
ggcgtatttctactccagca20
<210>6
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>6
taatacgactcactatagg19
<210>7
<211>1026
<212>dna
<213>人工序列(artificialsequence)
<400>7
atgaaaaagcctgaactcaccgcgacgtctgtcgagaagtttctgatcgaaaagttcgac60
agcgtctccgacctgatgcagctctcggagggcgaagaatctcgtgctttcagcttcgat120
gtaggagggcgtggatatgtcctgcgggtaaatagctgcgccgatggtttctacaaagat180
cgttatgtttatcggcactttgcatcggccgcgctcccgattccggaagtgcttgacatt240
ggggaattcagcgagagcctgacctattgcatctcccgccgtgcacagggtgtcacgttg300
caagacctgcctgaaaccgaactgcccgctgttctgcagccggtcgcggaggccatggat360
gcgatcgctgcggccgatcttagccagacgagcgggttcggcccattcggaccgcaagga420
atcggtcaatacactacatggcgtgatttcatatgcgcgattgctgatccccatgtgtat480
cactggcaaactgtgatggacgacaccgtcagtgcgtccgtcgcgcaggctctcgatgag540
ctgatgctttgggccgaggactgccccgaagtccggcacctcgtgcacgcggatttcggc600
tccaacaatgtcctgacggacaatggccgcataacagcggtcattgactggagcgaggcg660
atgttcggggattcccaatacgaggtcgccaacatcttcttctggaggccgtggttggct720
tgtatggagcagcagacgcgctacttcgagcggaggcatccggagcttgcaggatcgccg780
cggctccgggcgtatatgctccgcattggtcttgaccaactctatcagagcttggttgac840
ggcaatttcgatgatgcagcttgggcgcagggtcgatgcgacgcaatcgtccgatccgga900
gccgggactgtcgggcgtacacaaatcgcccgcagaagcgcggccgtctggaccgatggc960
tgtgtagaagtactcgccgatagtggaaaccgacgccccagcactcgtccgagggcaaag1020
gaatag1026
<210>8
<211>48
<212>dna
<213>人工序列(artificialsequence)
<400>8
ctagcataaccccttggggcctctaaacgggccttgaggggttttttg48
<210>9
<211>243
<212>dna
<213>人工序列(artificialsequence)
<400>9
atgcaacctatacaaatagcaatagtagcattagtagtagcaataataatagcaatagtt60
gtgtggtccatagtaatcatagaatataggaaaatattaagacaaagaaaaatagacagg120
ttaattgatagactaatagaaagagcagaagacagtggcaatgagagtgaaggaaaaata180
tcagcacttgtggagatgggggtggagatggggcaccatgctccttgggatgttgatgat240
ctg243
<210>10
<211>35
<212>dna
<213>人工序列(artificialsequence)
<400>10
tcaaggagaaaaaaccaaaaaacccctcaaggccc35
<210>11
<211>35
<212>dna
<213>人工序列(artificialsequence)
<400>11
ttaatgcagctggattaatacgactcactataggt35
<210>12
<211>516
<212>dna
<213>人工序列(artificialsequence)
<400>12
atggtcttcacactcgaagatttcgttggggactggcgacagacagccggctacaacctg60
gaccaagtccttgaacagggaggtgtgtccagtttgtttcagaatctcggggtgtccgta120
actccgatccaaaggattgtcctgagcggtgaaaatgggctgaagatcgacatccatgtc180
atcatcccgtatgaaggtctgagcggcgaccaaatgggccagatcgaaaaaatttttaag240
gtggtgtaccctgtggatgatcatcactttaaggtgatcctgcactatggcacactggta300
atcgacggggttacgccgaacatgatcgactatttcggacggccgtatgaaggcatcgcc360
gtgttcgacggcaaaaagatcactgtaacagggaccctgtggaacggcaacaaaattatc420
gacgagcgcctgatcaaccccgacggctccctgctgttccgagtaaccatcaacggagtg480
accggctggcggctgtgcgaacgcattctggcgtaa516
<210>13
<211>795
<212>dna
<213>人工序列(artificialsequence)
<400>13
atgtctaaaggtgaagaattattcactggtgttgtcccaattttggttgaattagatggt60
gatgttaatggtcacaaattttctgtctccggtgaaggtgaaggtgatgctacttacggt120
aaattgaccttaaaatttatttgtactactggtaaattgccagttccatggccaacctta180
gtcactactttcggttatggtgttcaatgttttgctagatacccagatcatatgaaacaa240
catgactttttcaagtctgccatgccagaaggttatgttcaagaaagaactatttttttc300
aaagatgacggtaactacaagaccagagctgaagtcaagtttgaaggtgataccttagtt360
aatagaatcgaattaaaaggtattgattttaaagaagatggtaacattttaggtcacaaa420
ttggaatacaactataactctcacaatgtttacatcatggctgacaaacaaaagaatggt480
atcaaagttaacttcaaaattagacacaacattgaagatggttctgttcaattagctgac540
cattatcaacaaaatactccaattggtgatggtccagtcttgttaccagacaaccattac600
ttatccactcaatctgccttatccaaagatccaaacgaaaagagagaccacatggtcttg660
ttagaatttgttactgctgctggtattacccatggtatggatgaattgtacaaatctaga720
actagtggatcccccgggctgcaggaattcgatatcaagcttatcgataccgtcgacctc780
gagtcatgtaattag795
<210>14
<211>500
<212>dna
<213>人工序列(artificialsequence)
<400>14
gggtaggggaggcgcttttcccaaggcagtctggagcatgcgctttagcagccccgctgg60
gcacttggcgctacacaagtggcctctggcctcgcacacattccacatccaccggtaggc120
gccaaccggctccgttctttggtggccccttcgcgccaccttctactcctcccctagtca180
ggaagttcccccccgccccgcagctcgcgtcgtgcaggacgtgacaaatggaagtagcac240
gtctcactagtctcgtgcagatggacagcaccgctgagcaatggaagcgggtaggccttt300
ggggcagcggccaatagcagctttgctccttcgctttctgggctcagaggctgggaaggg360
gtgggtccgggggcgggctcaggggcgggctcaggggcggggcgggcgcccgaaggtcct420
ccggaggcccggcattctgcacgcttcaaaagcgcacgtctgccgcgctgttctcctctt480
cctcatctccgggcctttcg500
<210>15
<211>348
<212>dna
<213>人工序列(artificialsequence)
<400>15
atggcaggaagaagcggagacagcgacgaagaactcctcaaggcagtcagactgatcaag60
tttctctaccaaagcaacccacctcccagcccagaggggacccgacaggcccgaaggaat120
cgaagaagaaggtggagagagagacagagacagatccgagcacttagtggatggattctt180
agcactcatctgggtcgatctgcggagcctgtgcctcttcagctaccaccgcttgagaga240
cttactcttgattgtaacgaggattgtggaaattctgggacgcagggggtgggaaatcat300
caagtattggtggagtctcctacaatattggagtcaggaactaaagaa348
<210>16
<211>51
<212>dna
<213>人工序列(artificialsequence)
<400>16
aattttgacctgctcaagttggccggagacgttgagtccaaccctgggccc51
<210>17
<211>627
<212>dna
<213>人工序列(artificialsequence)
<400>17
tgtttattcaagtggaagcagatttgtacgctcaagcggttgaataaactagttaacgtt60
actggccgaagtcgcttggaataaggccggtgtgcgtttgtctatatgttattttccacc120
atattgccgtcttttggcaatgtgagggcccggaaacctggccctgtcttcttgacgagc180
attcctaggggtctttcccctctcgccaaaggaatgcaaggtctgttgaatgtcgtgaag240
gaagcagttcctctggaagcttcttgaagacaaacaacgtctgtagcgaccctttgcagg300
cagcggaaccccccacctggcgacaggtgcctctgcggccaaaagccacgtgtataagat360
acacctgcaaaggcggcacaaccccagtgccacgttgtgagttggatagttgtggaaaga420
gtcaaatggctctcctcaagcgtattcaacaaggggctgaaggatgcccagaaggtaccc480
cattgtatgggatctgatctggggcctcggtgcacatgctttacatgtgtttagtcgagg540
ttaaaaaaacgtctaggccccccgaaccacggggacgtggttttcctttgaaaaacacga600
tgataatatggccacaacgtcgatatg627
<210>18
<211>122
<212>dna
<213>人工序列(artificialsequence)
<400>18
aacttgtttattgcagcttataatggttacaaataaagcaatagcatcacaaatttcaca60
aataaagcatttttttcactgcattctagttgtggtttgtccaaactcatcaatgtatct120
ta122
<210>19
<211>234
<212>dna
<213>人工序列(artificialsequence)
<400>19
aggagctatgttccttgggttcttgggagcagcaggaagcactatgggctcagcgtcaat60
ggcgctgacggtacaggccaggctattgttgtctggtatagtgcaacagcagaacaattt120
gctgagggctattgaggcgcaacagcatctgttgcaactcacagtctggggcatcaagca180
gctccaggcaagaatcctggctgtggaaagatacctaaaggaccaacagctcct234
- 还没有人留言评论。精彩留言会获得点赞!