一种基于病原菌线粒体基因组序列检测西瓜枯萎病、番茄枯萎病和莲藕腐败病病原的方法
1.本发明采用基于聚合酶链式反应(polymerase chain reaction,缩写为pcr)和环介导等温扩增(loop-mediated isothermal amplification,缩写为lamp)技术快速检测西瓜枯萎病、番茄枯萎病和莲藕腐败病病原菌的方法,属于生物技术研究领域。
技术背景
2.莲藕腐败病又称为藕瘟病,是莲藕种植过程中的重要病害之一,藕田发病后减产10~20%,严重的可达60%~80%。在1983年,该病最初报道由真菌尖刀镰孢菌莲专化型【fusarium oxysporum schl.f.sp.nelumbicola(nis.&wat.)booth】侵染引起,但近些年国内同行的相关研究表明,另一种镰刀菌属的病原真菌f.commune,同样可以侵染莲藕并且导致莲藕腐败病的相似症状。有报道,f.commune和f.oxysporum侵染植物的机理基本相似,都通过菌丝体及厚垣孢子随病残体在藕田土壤中越冬,在条件合适的时候,从地下茎或根系侵入,然后在茎内和整个植株内蔓延,导致茎节及根系变色腐烂,地上部叶片、叶柄以及莲蓬枯死。
3.在遗传学方面,f.commune和f.oxysporum非常相近,它们基因组当中的内转录间隔区(internal transcribed spacer,缩写为its)一致性为100%,无法对其进行区分;在形态学方面,它们互为姐妹菌株,如果不是从事真菌分类和鉴定的人员很难快速而准确地区分它们。目前,常用的区分手段就是对其看家基因(例如:mtssu,ef1α等)的保守序列进行pcr扩增,完成sanger法测序后,将序列比对后实现病原菌的鉴定。但这种方法步骤多,从真菌样本的准备,提取基因组到最后测序完成,需要至少等待3天以上的时间,样本量增多之后容易出错,消耗的试剂成本也不低。
4.在陆地种植的西瓜和番茄也同样受到镰刀菌属病原物的威胁,其中最主要的就是西瓜枯萎病和番茄枯萎病这两种土传病害。造成西瓜枯萎病的病原物主要有三种尖孢镰刀菌专化型引起,分别是f.oxysporum f.sp.niveum,f.oxysporum f.sp.cucumerinum和f.oxysporum f.sp.radicis-cucumerinum;导致番茄枯萎病的病原物则主要是f.oxysporum f.sp.lycopersici和f.oxysporum f.sp.radicis-lycopersici。
5.上述病菌都属于土传病原真菌,在侵染初期症状不明显。因此,建立一种快速而准确的方法用来评估种苗以及在发病初期(或尚未显症时期)的植株当中是否存在上述的病原物就显得非常重要。依据上述评估和检测结果,植保人员和农户可以及时地进行病害防控等补救措施,并对后续的田间管理以及病害防治提供依据和预案。
6.线粒体(mitochondrion)是一种广泛存在于真核生物细胞中的细胞器,它由两层膜包被,是细胞进行有氧呼吸的主要场所。它通过电子传递偶联氧化磷酸化过程,产生生物细胞可以直接利用的高能化合物腺苷三磷酸(adenosine triphosphate,atp)。此外,线粒体也参与了其他的细胞生物学过程,包括离子平衡、中间代谢以及细胞凋亡等(ion homeostasis,intermediary metabolism and apoptosis)。在线粒体中,存在一套独立于
细胞核染色体基因组的脱氧核糖核酸(deoxyribonucleicacid,缩写为dna),即线粒体基因组dna。线粒体基因组dna的大小在不同的物种当中变异很大,从几kbp到几百kbp,通常包含编码14种蛋白的基因,以及合成22-26种trna、2种rrna的dna序列。线粒体dna与基因组dna相比,在单个细胞内,其拷贝数量更多,而且更容易通过普通物理作用(冷冻或高温)被释放出来,因此以线粒体dna上的特异序列作为检测靶标是一种灵敏度非常高的方法。
7.在播种或移栽前对设施土壤以及种苗当中的病原物种类和数量进行精确检测是土传病害预测预报的核心技术。最传统的土壤病原物检测方法是采用平板稀释培养法(scherm等,1998),但是该方法费时费力,且平板上生长的杂菌对病原菌真实数量的统计会有干扰。随着生物信息学和分子生物学技术的不断发展,基于聚合酶链式反应(polymerase chain reaction,pcr)、环介导等温扩增(loop-mediated isothermal amplification,lamp)以及荧光定量pcr等技术常常被应用于土传病原物的检测和定量分析(li等,2014;mbofung等,2011)。它们的基本原理都是扩增靶标物种基因组上看家基因的特异序列,从而实现特定病原物的鉴定及其定量分析(zitnick-anderson等,2018;li等,2014)。然而,获得高质量的土壤微生物总基因组dna并不容易,加之上述技术存在检测下限,部分检测结果会出现“假阴性”的情况。因此,如何进一步提升检测方法的灵敏度进而对主要土传病害发生进行精准预测成为当前研究的热点。
8.在线粒体中,存在一套独立于细胞核基因组的核酸dna,即线粒体基因组dna。线粒体基因组dna在不同的物种当中变异很大,但在同一物种中其序列非常保守(burger等,2003)。单个真核细胞当中,基因组dna只有一份拷贝,而线粒体基因组dna则有数十个甚至数百个拷贝。因此以线粒体dna上的特异序列作为检测靶标将是一种更加灵敏和准确的检测方法。
9.聚合酶链式反应(pcr)是一种用于扩增特定的dna片段的分子生物学技术。理论上,它可将微量的靶标dna片段以2的n次方倍进行放大。pcr基本原理是利用dna在体外95℃高温时变性形成单链,低温时与互补的单链dna按碱基配对的原则结合,在72℃左右的温度下,反应体系中的dna聚合酶会将互补dna链从引物结合的位置开始,沿着核酸链5
′-3′
的方向合成并延伸至配对模板的终点。
10.环介导等温扩增(lamp)技术最早发表在2000年的生物学杂志《nucleic acids research》上。该技术对靶标核酸序列的扩增具有高特异性、高敏感性,而且操作非常简便。目前,在人类临床疾病的诊断、动植物病原微生物的定性定量检测等领域,基于lamp的检测技术都是一个重要的辅助手段。其原理可以简要的概括如下:整个反应至少需要4种特异引物,分别为fip(上游内部引物),bip(下游内部引物),f3(上游外部引物)和b3(下游外部引物),如果需要提高扩增效率,还可以增加两条loop引物。这些引物和模板、高活性链置换dna聚合酶按照一定的浓度比例混合,在65℃的含有必须的离子和底物的反应液中,靶标dna链形成单链置换dna,然后在fip和bip的位置形成颈环结构。利用颈环结构以及链置换dna聚合酶,链置换型dna合成在不停地自我循环,并逐步延长和放大。在dna合成反应时,特殊的化合物在反应液中逐渐积累,在达到一定浓度后,会在反应液中呈现特殊的指示状态,比如变色或变浑浊。最后,通过肉眼或者浊度仪,即可以判断在检测样品当中是否有靶标dna片段存在。该方法由于对实验条件和平台要求不高,在农业生产当中已经得到了广泛的应用。
技术实现要素:
11.本发明利用线粒体dna序列组装软件grabb对菌株的二代测序结果进行组装,这些菌株核酸序列的来源包括:本实验室分离并进行二代测序的2个莲藕致病菌,9个已公开的序列信息的尖孢镰刀菌(f.oxysporum)和1个f.commune菌,共计12个菌株;通过对这12个线粒体基因组部分序列的比对和聚类分析,发现在线粒体序列上有多处差异,其中3处差异序列适合用于进行检测分析,包括2个f.commune特异位点和1个f.oxysporum特异位点;利用这些差异序列,我们设计了2套lamp检测引物,经过对发病西瓜苗、番茄苗植株以及人工接种的莲藕藕节的活体检测,证实它们在不需要提取核酸的前提下就可以很快地鉴定相关病原菌。
12.相关技术路线如下:
13.1.下载对应f.commune和f.oxysporum菌株的线粒体基因组序列或对应的二代基因组测序数据。
14.2.用grabb软件对二代基因组测序数据进行线粒体基因组序列的组装和拼接。
15.3.将上述直接下载的线粒体基因组序列以及通过拼接获得的线粒体基因组序列进行omega软件的比对,比对工具的网址为https://www.ebi.ac.uk/tools/msa/clustalo/。
16.4.利用mega7软件对上述omega得到的结果进行分析,在比对图中找到差异位点,然后根据差异位点的序列设计引物。普通pcr引物采用在线引物设计软件https://www.ncbi.nlm.nih.gov/tools/primer-blast/获得,引物的名字分别为:fc特异1-s,fc特异1-a,fc特异2-s,fc特异2-a,fo特异3-s,fo特异3-a。其中,两套引物为f.commune特异检测引物,一套引物为f.oxysporum特异检测引物。另外,lamp引物设计则通过http://primerexplorer.jp/e/网站获得最适合的引物组合。引物包括fip(上游内部引物),bip(下游内部引物),f3(上游外部引物),b3(下游外部引物),lf(上游环引物)和lb(下游环引物)。
17.5.在进行pcr和lamp检测前,根据不同的样品和实验目的按照不同的步骤进行预处理,详见具体实施方式中的步骤。
18.6.最后在冰盒上,按照pcr酶的说明书,配置总体积为25μl的常规pcr反应试剂;取上述1μl冻融裂解后的培养液作为模板加入pcr反应体系当中并混匀,然后设置pcr仪进行pcr扩增反应,扩增反应的pcr条件除了第一步95℃或98℃预变性5分钟,其它步骤按照pcr酶的说明书进行设置。pcr完成后用2%的琼脂糖凝胶电泳进行分析,并采用紫外成像仪观察电泳条带。
19.7.对于lamp反应,以靶标样品为模板,将其加入已经按照如下比例配置好的反应液当中(反应液的配置详见具体实施方式部分)。混匀后,将pcr管静置于62℃(检测f.oxysporum)或66℃(检测f.commune)条件下反应60~120min(注:部分无热盖的反应条件需要在pcr管内加入矿物油,防止水分蒸发至盖子上),然后观察反应管的浑浊度和颜色。如果反应管内的反应液变浑浊、且颜色由紫色变为天蓝色,则说明反应管对应样品当中存在靶标核酸片段;如果反应管的颜色为紫色,说明无靶标片段。有条件的实验室可以用2%的琼脂糖凝胶电泳进行分析,并采用紫外成像仪观察对应样品泳道是否出现阶梯状、由小到大的多个条带,如有则说明发生了扩增反应。
20.8.对于上述所有pcr反应、荧光定量pcr反应和lamp反应均应当设置无模板的阴性
对照,如有必要,可以加入含有靶标模板的阳性对照。
21.有益效果:本发明设计的引物和检测方法可以快速、准确的检测发病植株当中的f.oxysporum或(和)f.commune,这避免了大量的病原菌分离鉴定的繁琐工作以及缩短了发病植物样本的检测和诊断的时间,为相关病害的及时防控赢得宝贵时间,防止病害进一步扩展导致更大的损失。
附图说明
22.图1.实例中f.oxysporum菌株和f.commune菌株的线粒体差异序列
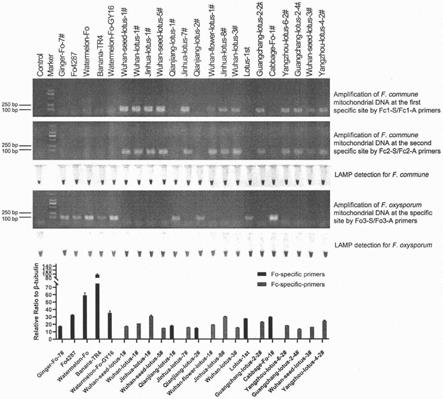
23.图2.实例中使用的常规pcr、荧光定量pcr和lamp方法对多个f.oxysporum菌株和f.commune菌株的鉴定结果
24.图3.用于检测f.oxysporum菌株和f.commune菌株的lamp引物灵敏度分析
25.图4.应用实例中lamp方法检测发病西瓜苗中枯萎病菌(f.oxysporum)的结果。
26.图5.应用实例中lamp方法检测发病番茄苗中枯萎病菌(f.oxysporum)的结果。
27.图6.应用实例中lamp方法检测在藕节上人工接种莲藕腐败病菌(f.commune)的结果。
具体实施方式
28.1.相关菌株的线粒体基因组序列比对分析
29.从national center for biotechnology information(ncbi)网站上下载或者通过grabb软件组装包含f.oxysporum和f.commune共12种不同来源菌株的线粒体基因组序列,其中包括f.oxysporum,f.oxysporum f.sp.niveum,f.oxysporum f.sp.cucumerinum,f.oxysporum f.sp.cubense tr4,f.oxysporum f.sp.lycopersici,f.oxysporum f.sp.cumini,f.oxysporum f.sp.conglutinans,f.commune,以及本实验室从莲藕上分离的f.oxysporum和f.commune菌株。将上述真菌的线粒体基因组信息放入在线软件https://www.ebi.ac.uk/tools/msa/clustalo/中进行分析,分析结束后,导出clustalw属性文件。然后,利用mega7将该文件打开,寻找上述序列当中f.oxysporum和f.commune存在差异的位置和序列,并记录下来,同时将该差异序列附近的核酸区域的比对结果另存为fas和max属性文件。经过比对,获得了2个在f.commune特异存在,1个在f.oxysporum特异存在的位点(图1)。相关序列信息如下:
30.f.commune特异序列1#:
[0031][0032]
f.commune特异序列2#:
[0033][0034]
f.oxysporum特异序列3#:
[0035]
[0036]
经过ncbi核酸数据库的比对,发现f.oxysporum特异序列3#仅在f.oxysporum种内高度保守,在其他物种当中,序列相似度较低;对于f.commune特异序列1#和f.commune特异序列2#,除了一个可侵染莴苣的尖孢镰刀菌内f.oxysporum f.sp.lactucae strain 09-002发现了高度同源的序列,尚未在除f.commune以外的其它物种当中发现高度相似的序列。绝大部分的f.oxysporum植物病原菌都有严格的寄主专化性,而f.oxysporum f.sp.lactucae strain 09-002菌株不能侵染除菊科以外的作物,因此,在莲藕、荸荠等能够被f.commune侵染的作物上,f.commune特异序列1#和f.commune特异序列2#就是唯一在f.commune当中存在的。
[0037]
2.针对上述特异性位点设计pcr引物、荧光定量引物和lamp专用引物用于区分并检测f.commune和f.oxysporum
[0038]
普通pcr引物和荧光定量引物采用在线引物设计软件https://www.ncbi.nlm.nih.gov/tools/primer-blast/获得,其引物名字、序列以及对应的扩增区域如表1所示:
[0039][0040][0041]
表1普通pcr引物和荧光定量引物
[0042]
lamp检测引物设计通过http://primerexplorer.jp/e/网站获得,包括fip(上游内部引物),bip(下游内部引物),f3(上游外部引物),b3(下游外部引物),lf(上游环引物)和lb(下游环引物)。进入http://primerexplorer.jp/e/网站,选择primerexplorer v5版本,分别导入需要检测的f.commune特异序列1#和f.oxysporum特异序列3#,然后分别对其设计引物。根据实际情况综合考虑,在列出的引物当中,分别选择出一套最适合的引物组合,如表2所示。考虑到lamp检测非常灵敏,建议引物最好采用准确度较高的page或者hplc方法合成。
[0043][0044]
表2针对f.commune和f.oxysporum的特异性lamp检测引物
[0045]
3.待测样品的准备
[0046]
3.1靶标为待测菌株总dna:取实验室保存的f.commune和f.oxysporum菌株共计22个(菌株信息顺序如表3所示),培养后,分别收集其菌丝,液氮研磨破碎后,用ctab法取对应的总dna,该dna稀释后,其浓度应该在100ng/μl~1ng/μl之间。
[0047]
菌株编号中文菌株名称英文菌株名称菌株种属1生姜7#ginger-fo-7#fo2fo4287fo4287fo3西瓜枯萎watermelon-fofo4香蕉枯萎(南农)banana-tr4fo5gy16watermelon-fo-gy16fo6子莲-1wuhan-seed-lotus-1#fc7武汉藕早-1wuhan-lotus-1#fc8藕腐-1jinhua-lotus-1#fc9子莲-5wuhan-seed-lotus-5#fc10武汉藕-1qianjiang-lotus-1#fo11藕腐-7jinhua-lotus-7#fc12武汉-2qianjiang-lotus-2#fo13花莲-1wuhan-flower-lotus-1#fc14金华藕腐-8jinhua-lotus-8#fc15武汉藕早-3wuhan-lotus-3#fc
16藕1stlotus-1stfo17广昌2-2测序6#guangchang-lotus-2-2#fc18flg原始1号cabbage-fo-1#fo19扬州6-2
-①
yangzhou-lotus-6-2#fc20广昌2-4guangchang-lotus-2-4#fc21子莲-3wuhan-seed-lotus-3#fc22扬州4-2
①-
1yangzhou-lotus-4-2#fc
[0048]
表3实例中使用的部分菌株列表
[0049]
(注:表中f.commune和f.oxysporum分别简写为fc和fo)
[0050]
3.2靶标为待测菌株的培养物:转接待测菌株至200μl(2ml灭菌离心管)液体potato dextrose broth(pdb)中25℃180rpm培养2天,然后放于-20℃冰箱或液氮内让其冻融一次;解冻后后直接沸水浴10分钟(对于某些菌株,冻融还不能释放足够的线粒体核酸,可以增加此步骤)。最后,用灭菌双蒸馏水稀释10倍后,取0.5μl作为后续试验的模板。
[0051]
3.3靶标为疑似被侵染的植物茎秆:将田间出现类似枯萎病症状或实验室内已人工接种的植株取回(注:部分植株在接种后尚未显症),截取其茎秆基部约2-5cm(或挖取莲藕藕节上的接种部位),用自来水反复冲洗,去除表皮的杂质、土壤或培养基质。对于幼嫩或者木质化程度不高的茎秆,可以直接在截断处挤出茎秆内部的汁液,用移液器和无污染的200μl pcr离心管收集5-10μl即可,然后加入10倍体积的灭菌双蒸馏水,混匀后沸水浴或者95度高温处理10分钟,取0.5~1μl上清液作为lamp的反应模板。对于较硬或者木质化较高的样品,可以选择维管束系统变色或者组织褐化的样品0.05~0.1g,用液氮研磨(要小心样品间的交叉污染)或者破碎机器震荡破碎,之后加入10倍体积的灭菌双蒸馏水,沸水浴后,取0.5~1μl上清液作为lamp的反应模板。
[0052]
4.普通pcr、荧光定量pcr以及lamp的特异性验证
[0053]
普通pcr反应体系的配置:购买南京擎科生物科技有限公司的直扩pcr酶(t5 direct pcr kit for plant)。在冰盒上按照要求配置25μl的pcr反应体系,加入1μl对应的总dna(经过3.1处理的)或者0.5μl对应的培养物上清(经过3.1处理的),混匀后,按照酶提供的说明书进行pcr仪的设置,其中,退火温度设置为58℃。
[0054]
荧光定量pcr反应体系的配置:购买takara公司的定量pcr试剂盒(tbpremix ex taq
tm ii),采用3步法进行扩增,40个循环,退火温度设定为58℃。
[0055]
lamp反应体系的配置:按照lamp反应试剂盒(bst dna polymerase large fragment,p701)说明书的要求,在冰盒上制备反应液:将除酶和模板以外的各反应物先加入反应管中;加入终浓度为0.8mol
·
l-1
甜菜碱和180μmol
·
l-1
羟基萘酚蓝(hnb);最后,加入反应酶和适量的模板(0.25~1μl)。初始反应液为紫色澄清状态,在62℃(对于f.oxysporum)或66℃(对于f.commune)反应60~120min后,含有靶标模板的反应管则变为天蓝色,并且呈现浑浊状态。
[0056]
对表3中的22个菌株进行检测,其中,普通pcr和lamp可以使用培养物制备的模板(3.2),而对于荧光定量,则必须使用提取的总dna作为模板(3.1)。从图2中可以看出,三种方法均可以准确鉴定靶标菌株,而且从荧光定量的结果来看,若以核基因组中的β-tubulin序列作为内参,以线粒体中特异序列扩增的模板拷贝数为该内参的10倍或以上,说明在相
同的反应条件下,利用线粒体基因组做为检测靶标至少可以提高一个数量级的检测灵敏度(其拷贝数是核基因组的10倍或以上)。
[0057]
5.lamp检测的灵敏度验证
[0058]
为了进一步分析lamp检测方法的灵敏度,将ctab法提取的lotus-1st和guangchang-lotus-2-2#总dna进行梯度稀释,包括如下核酸浓度:200ng/μl、10ng/μl、1ng/μl、100pg/μl、10pg/μl、1pg/μl、100fg/μl和10fg/μl。根据检查引物的情况,设置各自的反应条件,90分钟后观察反应管的颜色,并进行琼脂糖核酸电泳检测。由图3可知,在90分钟的反应时间内,f.commune的引物可以实现在最低10pg/μl的总核酸浓度下检出相关靶标样品,而f.oxysporum则可以在1pg/μl的总核酸浓度下检出靶标样品。
[0059]
6.lamp检测方法在发病西瓜、番茄植株(或莲藕藕节组织)当中的应用
[0060]
取回植物样本后,先按照具体实施方法的3.3步骤对样品进行预处理,然后根据要检测的病原物选择对应的引物和设置lamp反应条件,待反应120分钟后,观察反应管的颜色变化以及进行琼脂糖凝胶电泳分析。由图4可以看出,在生病的西瓜苗上,仅通过检测茎秆挤出的渗出液即可检测到尖孢镰刀菌(枯萎病菌)的存在;而在番茄植株上,茎秆的渗出液同样可以检测到枯萎病菌的存在(尽管有的植株尚未显症),但其健康植株的对照反应管则没有发生变化。对于人工接种的莲藕藕节,针对f.commune的检测引物可以检测到接种部位上存在相关病原菌。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1