长日照抑制基因变异在培育栽培稻中的应用
1.本发明属于生物技术领域,具体涉及长日照抑制基因变异在培育栽培稻中的应用。
背景技术:
2.培育抽穗期适中的品种是扩大水稻生产面积保证水稻稳产的关键因素(itoh,et al.2018)。栽培稻的祖先种(oryza rufipogon griff.)主要生长在热带和亚热带低纬度地区,是短日照敏感植物,在短日照条件下开花。经过自然选择和人工驯化,栽培稻在长日照条件下也可以开花,其地理分布范围逐渐扩张北达45
°
n,南抵23
°
s(monfreda,et al.2008;huang,et al.2012),成为在长短日均能生长的作物。在整个扩张过程中,培育抽穗期适宜的栽培稻品种至关重要,所以解析栽培稻抽穗期多样性的遗传机制对于解析栽培稻的驯化历史,培育广适品种至关重要。
3.先前的研究已经表明光周期的敏性是影响栽培稻抽穗期长短的重要因素(shang,et al.2012)。到目前为止,水稻光周期基因调控网络内的很多基因已经被克隆(gao,et al.2014)。其中,heading date 3a(hd3a)和rice flowering-locus t 1(rft1)是抽穗期光周期基因调控网络中的两个成花素基因,形成信号完成从营养生长向生殖生长的转变(kojima,et al.2002;komiya,et al.2008)。heading date 1(hd1)和early heading date 1(ehd1)是光周期信号通路中两个枢纽基因,它们通过在不同的日照长度下接收来自上游基因的多个调控信号来调控成花素基因hd3a和rft1的表达水平,进而影响水稻的抽穗期(masahiro yano 2000;yano,et al.2000;wei,et al.2016)。hd1影响水稻对光周期的敏感性,它在长日照的条件下抑制水稻开花,但是短日照的条件下促进水稻开花(yano,et al.2000)。独立于hd1的光周期信号通路中的另一个基因ehd1在长日照和短日照的条件下都促进水稻开花(wei,et al.2016)。其中在短日照的条件下,ehd1受其上游调控因子osgigantea(osgi),early heading date 2(ehd2)和mads box gene 51(osmad51)等多个基因的调控,促进水稻开花(matsubara,et al.2008;ryu,et al.2009)。然而在长日照条件下,以ehd1为枢纽的基因调控,其中mads box gene50(osmad50),early heading date 2(ehd2),early heading date 3(ehd3)和early heading date 4(ehd4)通过直接或者间接促进ehd1的表达来达到促进水稻开花的目的。heading date 6(hd6)、heading date(qtl)-5(t)(dth8)、(heading date 2)dth7、constans-like gene 4(oscol4)、heading date 7(ghd7),phytochrome b(phyb)和photosensitivity 5(se5)通过直接或者间接下调ehd1的表达抑制水稻开花(dehesh k 1991;takahashi,et al.2001;xue,et al.2008;lee,et al.2010;wei,et al.2010;hyuk,et al.2013;du,et al.2017)。综上所述,栽培稻光周期基因调控网络可以分为短日照促进途径、长日照促进途径和长日照抑制途径。
技术实现要素:
4.本发明的一个目的是提供检测dth8、dth7、ghd7、hd1、phyb和oscol4基因单倍型类
型的物质的用途。
5.本发明提供了检测dth8、dth7、ghd7、hd1、phyb和oscol4基因单倍型类型的物质在检测或辅助检测待测栽培稻是否来源于高纬度稻作区中的应用;
6.或,本发明提供了检测dth8、dth7、ghd7、hd1、phyb和oscol4基因的单倍型类型的物质在制备检测或辅助检测待测栽培稻是否来源于高纬度稻作区产品中的应用;
7.所述各个基因单倍型类型包括各个基因groupa单倍型和各自基因groupb单倍型。
8.本发明还提供了检测dth8、dth7、ghd7、hd1、phyb和oscol4基因的单倍型类型的物质在检测待测或辅助检测栽培稻是否适于栽种在高纬度地区中的应用;
9.或,本发明还提供了检测dth8、dth7、ghd7、hd1、phyb和oscol4基因的单倍型类型的物质在制备检测或辅助检测待测栽培稻是否适于栽种在高纬度地区产品中的应用;
10.所述各个基因单倍型类型包括各个基因groupa单倍型和各自基因groupb单倍型。
11.本发明还提供了检测dth8、dth7、ghd7、hd1、phyb和oscol4基因的单倍型类型的物质在筛选待测或辅助筛选高纬度栽培稻中的应用;
12.或,本发明还提供了检测dth8、dth7、ghd7、hd1、phyb和oscol4基因的单倍型类型的物质在制备筛选待测或辅助筛选高纬度栽培稻产品中的应用;
13.所述各个基因单倍型类型包括各个基因groupa单倍型和各自基因groupb单倍型。
14.本发明还提供了检测dth8、dth7、ghd7、hd1、phyb和oscol4基因的单倍型类型的物质在检测或辅助检测待测栽培稻抽穗期情况中的应用;
15.所述各个基因单倍型类型包括各个基因groupa单倍型和各自基因groupb单倍型;
16.所述dth8基因groupa单倍型为符合如下情况的dth8基因:编码区第56位为t、第74为a、第96位为t、第256位为a、306为a、第461位为a、第539-541位缺失、第569位为a、第651位为c、第659位为a和第893位为a;
17.所述dth8基因groupb单倍型为符合如下情况的dth8基因:编码区第56位为c、第74为c、第96位为g、第256位为g、306为c、第461位为c、第539-541位为gcg、第569位为c、第651位为t、第659位为c和第893位为g;
18.所述ghd7基因groupa单倍型为符合如下情况的ghd7基因:编码区第368位为a、第409为g、第527位为a、第703位为c;
19.所述ghd7基因groupb单倍型为符合如下情况的ghd7基因:编码区第368位为g、第409为a、第527位为t、第703位为g;
20.所述hd1基因groupa单倍型为符合如下情况的hd1基因:编码区第251-252位为aa、第443为g、第469位为a、第472位为a、490为a、第1215位为c、第1477位为a;
21.所述hd1基因groupb单倍型为符合如下情况的hd1基因:编码区第251-252位为gc、第443为a、第469位为c、第472位为c、490为g、第1215位为a、第1477位为g;
22.所述oscol4基因groupa单倍型为符合如下情况的oscol4基因:编码区第385位为c、第438为t、第733位为t;
23.所述oscol4基因groupb单倍型为符合如下情况的oscol4基因:编码区第385位为a、第438为c、第733位为g;
24.所述dth7基因groupa单倍型为符合如下情况的dth7基因:编码区第125位为t、第204为t、第641位为a、第1147位为g、1266为c、第1369位为a、第1385位t;
25.所述dth7基因groupb单倍型为符合如下情况的dth7基因:编码区第125位为c、第204为g、第641位为g、第1147位为a、1266为t、第1369位为g、第1385位为c;
26.所述phyb基因groupa单倍型为符合如下情况的phyb基因:编码区第282位为g、第525为c、第808位为t、第899位为a、1183为t、第1606位为c、第1638位t、第1833位为g;
27.所述phyb基因groupb单倍型为符合如下情况的phyb基因:编码区第282位为c、第525为t、第808位为c、第899位为g、1183为g、第1606位为t、第1638位为c、第1833位为a。
28.上述应用中,所述检测dth8、dth7、ghd7、hd1、phyb和oscol4基因的单倍型类型的物质包括分别用于扩增dth8、dth7、ghd7、hd1、phyb和oscol4基因的引物。
29.上述应用为在长日照条件下培育栽培稻进行检测,在本发明的实施例是在长日照地区如北京培育。
30.本发明另一个目的是提供一种检测或辅助检测待测栽培稻是否来源于高纬度地区的方法。
31.本发明提供的方法,包括如下步骤:检测或辅助检测待测栽培稻中dth8、dth7、ghd7、hd1、phyb和oscol4这6个基因的单倍型,
32.若符合如下条件:其中4个或4个以上基因属于各自group a单倍型时,则所述待测栽培稻来源于或候选来源于高纬度地区;若不符合所述条件,则所述待测栽培稻不来源于或候选不来源于高纬度地区。
33.本发明还有一个目的是提供一种检测或辅助检测待测栽培稻是否适于栽种在高纬度地区的方法。
34.本发明提供的方法,包括如下步骤:检测或辅助检测待测栽培稻中dth8、dth7、ghd7、hd1、phyb和oscol4这6个基因的单倍型,
35.若符合如下条件:其中4个或4个以上基因属于各自group a单倍型时,则所述待测栽培稻适于或候选适于栽种在高纬度地区;若不符合所述条件,则所述待测栽培稻不适于或候选不适于栽种在高纬度地区。
36.本发明第4个目的是提供一种筛选高纬度栽培稻的方法。
37.本发明提供的方法,包括如下步骤:检测或辅助检测待测栽培稻中dth8、dth7、ghd7、hd1、phyb和oscol4这6个基因的基因型,选取4个或4个以上基因属于各自group a单倍型的栽培稻作为高纬度栽培稻。
38.本发明第4个目的一种检测栽培稻抽穗期情况的方法。
39.本发明提供的方法,包括如下步骤:检测或辅助检测待测栽培稻中dth8、dth7、ghd7、hd1、phyb和oscol4基因的基因型,
40.具有某个基因属于group a单倍型的待测栽培稻的抽穗期早于具有该个基因属于group b单倍型的待测栽培稻。
41.上述高纬度地区为热带或亚热带以北的稻作区。
42.上述方法中,所述检测或辅助检测待测栽培稻中dth8、dth7、ghd7、hd1、phyb和oscol4这6个基因的单倍型的方法如下:
43.1)播种所述待测栽培稻的种子,在长日照条件下培育,得到待测栽培稻植株;
44.2)提取所述待测栽培稻的基因组dna,扩增所dth8、dth7、ghd7、hd1、phyb和oscol4这6个基因,测序,得到各个基因的基因型。
45.本研究首先收集广布于世界不同稻作区的栽培稻品种,然后对8个重要的长日照抑制基因hd1,dth8,ghd7,oscol4,dth7,hd6,se5和phyb进行遗传多样性分析,解析单个基因单倍型或者多个基因单倍型组合对栽培稻抽穗期多样性和地理分布区域的影响,以期阐释栽培稻抽穗期多样性形成的分子机制,为不同稻作区育种提供理论指导。本发明证实栽培稻dth8、dth7、ghd7、hd1、phyb和oscol4这6个基因group a中的单倍型分别都与栽培稻早花期关联,group b中单倍型分别都与晚花期单倍型关联。本发明证实当栽培稻4个或4个以上长日照抑制基因属于group a中的单倍型时,则这一类栽培稻主要分布在30
°
n以北的稻作区,这对栽培稻的区域性育种具有理论指导意义。
附图说明
46.图1为长日照抑制基因遗传变异分析;每个基因最上方是其结构示意图,矩形表示基因的外显子,有几个矩形就代表有几外显子。连接矩形之间的细线代表基因的内含子和非翻译区。外显子上的每一条垂直的细线代表的是外显子上碱基变异的位置。每个基因图中最下方的表格第一行代表其编码区突变位点的具体位置,第二行代表的是与突变位点对应的氨基酸变异的具体位置;每个基因最左边一列代表其不同单倍型,例如:dth8_1~dth8_18分别代表的是dth8的18个单倍型,其它7个基因以此类推;每个基因的图形最上方的基因结构示意图上碱基变异位置与该基因最下方表格第一行碱基变异的位置通过中间的斜线对应起来;每个基因图形最右边三列分别代表该基因的每个单倍型包含水稻三个亚种的样本个数,i:indica,j:japonica,w:野生稻;每个基因最左列单倍型名称与最右三列单倍型亚种个数之间的碱基代表相应基因编码区的snps、indel和stop code信息。r/nf:功能/非功能单倍型。(a)dth8.碱基位置为80的这一列中a代表cggcggcggctgcggctgg,19bp代表在这个位置为cggcggcggctgcggctgg这样一个19bp的缺失;碱基位置为451这一列中的8bp代表在这个位置存在gagaaggc这样8bp的缺失;碱基位置为721这一列中的3bp代表在该位置处存在gcg这样一个3bp的缺失;碱基位置为819这一列中的6bp代表在这个位置为aggcgg这样一个6bp的缺失;碱基位置为825这一列中的gcgg4代表在这个位置的碱基为gcgg重复4次,12bp代表在这个位置为gcgggcgggcgggcgg这样一个12bp的缺失;(b)ghd7.碱基位置为190这一列中的3bp代表在该位置为cga这样3bp的缺失;碱基位置为448的这一列中3bp代表在该位置为gtg这样3bp的缺失。(c)hd1.碱基位置为58这一列中的agg1代表agg这样一个重复,6bp代表在该位置存在aggagg这样6bp的缺失。碱基位置为331这一列中的m代表ccgctcgccaggcgccaccagcgcgtccccgtcgcg,36bp代表在该位置存在ccgctcgccaggcgccaccagcgcgtccccgtcgcg这样36bp缺失。碱基位置为499这一列中的3bp代表在该位置处存在aac这样3bp的缺失。碱基位置为961这一列中的9bp代表在该位置存在cagcagcag这样9bp的缺失。碱基位置为1021这一列中的n代表gtggtaccttcacagatcacaatgctgagtgagcagcagcata,43bp代表在这个位置存在gtggtaccttcacagatcacaatgctgagtgagcagcagcata这样43bp的缺失。碱基位置为1153这一列中的2bp代表在该位置存在tt这样2bp的缺失。碱基位置为1324这一列中的3bp代表aga这样3bp的缺失。碱基位置为1369这一列中的4bp代表在该位置存在aaga这样4bp的缺失。(d)dth7.碱基位置为934这一列中的2bp代表在这个位置存在tt这样2bp的缺失。碱基位置为1516这一列中的8bp代表在该位置存在aacgttgt这样8bp的缺失。碱基位置为1546这一列中的2bp代表在该位置存在ga这样2bp的缺失。碱基位置为2221
这一列中的a和b分别代表ctcgacctacctacctacctacgcgatggctttggactc和cttgacctacctacctacctacgcgatggctttggactc,39bp代表在该位置处存在a或b这样39bp的缺失。(e)phyb.碱基位置为124这一列中的6bp代表在这个位置存在ggcggg这样6bp的缺失。碱基位置为436这一列中的9bp代表在这个位置存在ctgtcgccc这样9bp的缺失。碱基位置2947这一列中的1bp代表在该位置处存在1bp a这样的缺失。(f)oscol4.碱基位置为919这一列中的6bp代表在该位置存在gccgac这样6bp的缺失。(g)hd6.碱基位置为727这一列中的29bp代表在这个位置存在gtacttggaacagaagcactaaatgctta这样29bp的缺失。(h)se5。
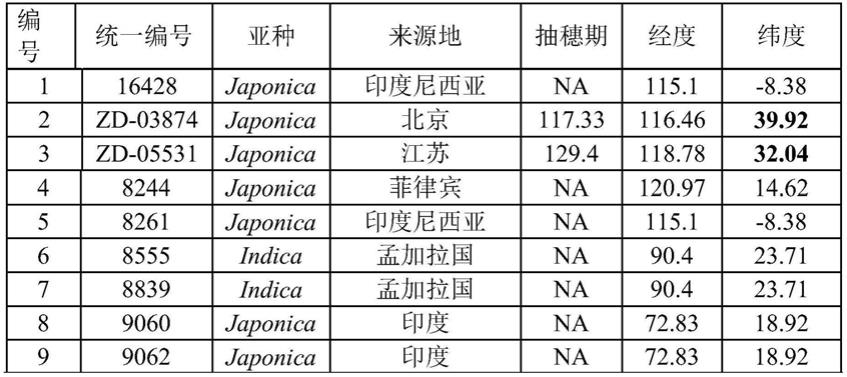
47.图2为栽培稻六个长日照抑制基因的networktrees;其中,h为单倍型,如oscol4:h_2表示oscol4的第二个单倍型oscol4_2;a.oscol4:h_2,h_6,h_11,h_13和h_18是该基因的高频单倍型。b.phyb:h_5和h_17是该基因的高频单倍型。c.dth8:h_3,h_4,h_6,h_14,h_15和h_17是该基因的高频单倍型。d.ghd7:h_23和h_41是该基因的高频单倍型。e.dth7:h_7,h_12,h_27,h_29,h_32是该基因的高频单倍型。f.hd1:h_8,h_11,h_12,h_17,h_27和h_31是该基因的高频单倍型。每个基因networktree中圈的大小代表单倍型频率的高低。每个基因networktree对应下方的基因结构图上的snp,代表长日照抑制基因group a与group b组间特异的snps。
48.图3为栽培稻六个长日照抑制基因单倍型与其抽穗期和地理分布样式的关系;6个基因的group a和group b中单倍型boxplot分别代表每个基因单倍型与抽穗期的关联性,其中深灰色代表group a中的单倍型,浅灰色代表group b中的单倍型。六个基因的group a和group b中单倍型heatmap分别代表每个基因在不同的纬度出现频率,灰度越深表示出现频率越高。六个基因的单倍型地理分布图代表每个基因group a和group b中单倍型的地理分布区域,其中深灰色点代表group a中的单倍型,浅灰色纹理的点代表group b中的单倍型。a.oscol4 b.dth7 c.phyb d.ghd7 e.dth8 f.hd1。
49.图4为长日照抑制基因dth8、dth7、ghd7、phyb、oscol4单倍型组合与抽穗期的关系及其地理分布式样;a.长日照抑制基因dth8、dth7、ghd7、phyb、oscol4单倍型组合与抽穗期的关联性:横轴代表携带0~5个group a中单倍型的栽培稻,纵轴代表携带不同个数group a中单倍型栽培稻的抽穗期。b.长日照抑制基因dth8、dth7、ghd7、phyb、oscol4单倍型组合在最冷月最低温密度分布图上的分布。c.长日照抑制基因dth8、dth7、ghd7、phyb、oscol4单倍型组合与其地理分布式样的关联性:横轴代表栽培稻携带0~5个group a中单倍型,纵轴代表携带不同单倍型栽培稻的分布纬度。
具体实施方式
50.下述实施例中所使用的实验方法如无特殊说明,均为常规方法。
51.下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
52.实施例1、水稻长日照抑制开花基因变异在培育栽培稻中的应用。
53.一、水稻长日照抑制开花基因变异与抽穗期和栽培区域的研究发现。
54.1、样本的种植和表型统计
55.种植地点:长日照地区北京(日照时长≥14小时/天)。
56.抽穗期统计:从播种日期到第一个抽穗之间的天数;
57.具体如下:
58.将表1所示的83份来源于中国华南双季稻稻作区、华中双季稻稻作区、西南高原的单双季稻稻作区、华北单季稻稻作区、东北早熟单季稻稻作区和西北干燥区单季稻稻作区等6大稻作区,以及东亚、南亚和东南亚等水稻主要种植区的栽培稻分别于2010年、2011年5月播种在北京,每个样本种一行,每行10株。
59.分别统计每个植株从播种日期到抽穗之间的天数,然后对每个品种连续两年总计20株抽穗期求平均值,则这个平均值计为该品种的抽穗期。
60.具体列表如表1所示:
61.表1
62.63.64.65.[0066][0067]
上表中,第1列为品种编号;第2列品种编号15-19为中国农科院水稻所提供,其余的为国际水稻研究中心(irri)编号;第3列为亚种属名;第4列为品种来源地;第5列为抽穗期;第6列为品种来源地的经度;第7列为品种来源地的纬度,其中
“-”
表示南纬,单独数字表示北纬;na表示信息缺失。
[0068]
2、遗传变异分析
[0069]
首先以常规方法提取上述表1所示抽穗后的各个样本的基因组dna作为模板,然后从ncbi上获取长日照抑制基因hd1,dth8,ghd7,oscol4,dth7,hd6,se5和phyb基因的niponbare(o.sativa ssp.japonica)参考序列,并在primer3(http://www.embnet.sk/cgi-bin/primer3_www.cgi)分别上输入这些基因序列设计引物(表2),接着分别用表2所示的引物扩增长日照抑制基因hd1,dth8,ghd7,oscol4,dth7,hd6,se5和phyb的编码区序列,然后进行sanger测序得到每个品种8个长日照抑制基因的编码区序列。
[0070]
pcr扩增所需引物序列如下表2:
[0071]
表2
[0072][0073]
上表中,第一列为基因名称,第二列为引物名称,第3列从上到下依次为序列1-序列19,第4列从上到下依次为序列20-序列40。
[0074]
上述pcr扩增所需引物组合如下:
[0075]
扩增hd1基因的引物为hd1正向/hd1反向和hd1正向/hd1-1r反向;
[0076]
扩增dth7基因的引物为dth7-1正向/dth7-1反向、dth7-2正向/dth7-2反向、dth7-3正向/dth7-3反向、dth7-4正向/dth7-4反向;
[0077]
扩增phyb基因的引物为:phyb-1正向/phyb-1反向、phyb-2正向/phyb-2反向、phyb-3正向/phyb-3反向,phyb-1-1f/phyb-1-3r、phyb-1-1f/phyb-1-4r、phyb-1-1f/phyb-1-5r、phyb-1-2f/phyb-1-3r、phyb-1-2f/phyb-1-4r、phyb-1-2f/phyb-1-5r;
[0078]
扩增se5基因的引物为:se5-1正向/se5-1反向、se5-2正向/se5-2反向;
[0079]
扩增hd6基因的引物为hd6-1正向/hd6-1反向、hd6-2正向/hd6-2反向、hd6-3正向/hd6-3反向;
[0080]
扩增oscol4基因的引物为oscol4正向/oscol4反向。
[0081]
将上述每个品种8个长日照抑制基因的编码区序列的测序结果采用bioedit软件,分别将148个品种的8个长日照抑制基因的编码序列进行校正,然后采用clustalx对每个基因的所有样本进行序列比对。
[0082]
结果如图1所示,8个基因中外显子的个数是从1~10,其中dth8只有一个外显子,hd6外显子数目hd6为10个。8个基因序列比对后长度变化范围是774bp~3516bp。8个基因的
基因结构示意图参见图1。dth8,ghd7,hd1,dth7,phyb,oscol4,hd6和se5比对后分别共鉴定了25,47,41,59,30,19,10和2snps,其中这8个基因中分别发现14,37,29,42,15,12,6和0个snps导致了基因编码蛋白氨基酸的改变。另外还发现在所有导致氨基酸改变的snps中,有一半的有意义突变是位于第一个外显子上。在dth8、dth7、ghd7、hd1和hd6 5个基因上分别鉴定了3,2,1,5,和1个snp形成终止密码子。同时,还在dth8,dth7,ghd7,hd1、phyb,oscol4和hd6分别鉴定了3、4,2、6、3、1和1个indels(图1),在se5中没有鉴定到indel。通过对这8个基因的遗传多样性的比较,发现dth8,dth7,ghd7,hd1、phyb和oscol4的这六个基因的遗传多样性明显高于hd6和se5。
[0083]
3、单倍型分析
[0084]
利用dnasp5.0和network 4.0软件,在83个栽培稻和65个野生稻个体中一共发现dth8,dth7,ghd7,hd1、phyb,oscol4,se5和hd6分别有18,41,41,42,28,22,3和13个单倍型(图1和表3)。同时,对每个基因的频率大于5%的单倍型进行统计,发现每个基因频率大于5%的单倍型个数都不超过6个,且频率变化范围是从5.10%到86%,同时每个基因高频单倍型的总和占基因中所有单倍型的比率的变化范围是从45.8%到99.6%(表4)。然后根据中介法(median-joining,mj),利用network4.0软件为这8个基因分别构建了networktrees。根据networktrees上单倍型亲缘关系,发现dth8,dth7,ghd7,hd1、phyb,oscol4这6个基因能显著的被分成group a和group b两组(图2),其中groupa和groupb两组单倍型分别特有的snps具体如下表5所示。同时还发现这6个基因group b中的祖先单倍型主要由indica和野生稻组成,group a中的祖先单倍型主要由japonica和分布于中国地区的野生稻组成(图3)。且这些基因networktrees group a和group b中都有的高频单倍型的分布(表4),其中group a中栽培稻高频单倍型主要出现在中国的黑龙江、吉林、辽宁、山西、北京和天津等地,以及日本和韩国等温带高纬度稻作区,且主要由japonica组成,然而group b中的栽培稻高频单倍型主要出现在中国的南方地区,以及南亚和东南亚等热带和亚热带稻作区,且主要有indica组成(图2、表1和表3)。但是对于另外两个基因hd6和se5,networktrees分别主要分别由其祖先单倍型hd6_1和se5_3构成,且这两个单倍型都由野生稻、indica和japonica组成,分别占这两个基因总单倍型的81%和86%(表4)。
[0085]
表3
[0086]
[0087]
[0088]
[0089][0090]
上表中,第1列为品种编号(与表1对应);第2-9列依次为该品种dth8、ghd7、hd1、hd6、oscol4、osprr37、phyb、se5的单倍型(具体与各个基因编码区相比的变异位点见图1所示);na表示信息缺失。
[0091]
表4
[0092]
[0093]
[0094]
[0095]
[0096][0097]
上表中hap_1等为某个基因的单倍型1,例如dth8的hap_1可以简称为dth8_1或者h1,其它基因以此类推。
[0098]
表3和表4的研究结果说明,长日照抑制基因dth8,dth7,ghd7,hd1,phyb,oscol4这6个基因发生了大量遗传变异,这种差异可能与栽培稻在不同地区的适应性相关。
[0099]
将这6个基因中的不同单倍型分为2组group a和group b(各个基因单倍型的遗传变异如图1所示):
[0100]
dth8的groupa包括:dth8_1至dth8_5;
[0101]
dth8的groupb包括:dth8_6至dth8_18;
[0102]
dth7的groupa包括:dth7_1至dth7_20;
[0103]
dth7的groupb包括:dth7_21至dth7_41;
[0104]
ghd7的groupa包括:ghd7_35至ghd7_39、ghd7_41;
[0105]
ghd7的groupb包括:ghd1_34至ghd1_39、ghd1_40;
[0106]
hd1的groupa包括:hd1_1至hd1_25、hd1_35至hd1_38;
[0107]
hd1的groupb包括:hd1_26至hd1_34、hd1_39至hd1_42;
[0108]
phyb的groupa包括:phyb_2至phyb_5、phyb_7、phyb_28;
[0109]
phyb的groupb包括:phyb_1、phyb_6、phyb_8至phyb_27;
[0110]
oscol4的groupa包括:oscol_16至oscol_18;
[0111]
oscol4的groupa包括:oscol1_15、oscol1_19至oscol1_22。
[0112]
将表4中的长日照抑制基因dth8,dth7,ghd7,hd1,phyb,oscol4这6个基因group a中单倍型区别于group b中单倍型的遗传变异总结如表5所示。
[0113]
表5
[0114][0115]
表5中第1行为每个基因突变碱基的编号,
[0116]
第2行为用于区分dth8_a和dth8_b的突变碱基在dth8基因编码区的位置(基因的rapdb_locus编号是os08g0174500,提交年份是2008年,niponbare(o.sativa ssp.japonica)染色体上的位置是chr08:4332106
–
4334829)序列的位置;
[0117]
第3行和第4行为dth8_a和dth8_b的突变位置的碱基;
[0118]
第5行为用于区分ghd7_a和ghd7_b的突变碱基在ghd7基因编码区的位置(基因的rapdb_locus编号是os07g0261200,提交年份是2008年,niponbare(o.sativa ssp.japonica)染色体上的位置是chr07:9152377-9155030)序列的位置;
[0119]
第6行和第7行为ghd7_a和ghd7_b的突变位置的碱基;
[0120]
第8行为用于区分hd1_a和hd1_b的突变碱基在hd1基因编码区的位置在hd1基因编码区((基因的rapdb_locus编号是os06g0275000,提交年份是2008年,niponbare(o.sativa ssp.japonica)染色体上的位置是chr06:9336376-9338569)序列的位置;
[0121]
第9行和第10行为hd1_a和hd1_b的突变位置的碱基;
[0122]
第11行为用于区分oscol4_a和oscol4_b的突变碱基在oscol4基因编码区的位置(基因的rapdb_locus编号是os02g0610500,提交年份是2008年,niponbare(o.sativa ssp.japonica)染色体上的位置是chr02:23989803-23991271)序列的位置;
[0123]
第12行和第13行为oscol4_a和oscol4_b的突变位置的碱基;
[0124]
第14行为用于区分dth7_a和dth7_b的突变碱基在dth7基因编码区的位置(基因的rapdb_locus编号是os07g0695100,提交年份是2008年,niponbare(o.sativa ssp.japonica)染色体上的位置是chr07:29616705-29629215)序列的位置;
[0125]
第15行和第16行为dth7_a和dth7_b的突变位置的碱基;
[0126]
第17行为用于区分phyb_a和phyb_b的突变碱基在phyb基因编码区的位置(基因的rapdb_locus编号是os03g0309200,提交年份是2008年,niponbare(o.sativa ssp.japonica)染色体上的位置是chr03:11020131-11028178)序列的位置;
[0127]
第18行和第19行为phyb_a和phyb_b的突变位置的碱基。
[0128]
4、单倍型与抽穗期关联性分析
[0129]
为了研究上述3栽培稻长日照抑制基因group a和group b中单倍型与上述1栽培稻抽穗期的关联性,采用r version 3.5.2构建抽穗期与group a和groupb的boxblot,并用paired t-test评估分别携带groupa和groupb单倍型栽培稻抽穗期差异的显著性。(由于se5和hd6遗传多样性低,且生物学进化保守,所以只对dth8、dth7、ghd7、phyb、oscol4、hd1 6个基因的单倍型与抽穗期多样性进行关联分析。)
[0130]
结果从图3的boxplot和表1与表3可以看出,当栽培稻携带的任何一个长日照抑制开花基因单倍型属于其相应networktree group a时,抽穗期的变化范围是73.8~192天。与之相反,当栽培稻携带的任何一个长日照抑制开花基因单倍型属于其相应networktree group b时,栽培稻抽穗期变化范围为77.5~203天。这说明当栽培稻携带的任何一个长日照抑制开花基因单倍型属于其相应networktree group a时,那么这个栽培稻的抽穗期要明显早于基因单倍型属于group b的栽培稻,反之亦然。其中dth8(t=-10.802p=1.263e-13),dth7(t=-2.1884,p=0.03813),和ghd7(t=-2.7768,p=0.01078)三个基因的遗传多样性与抽穗期的多样性显著相关,phyb的遗传多样性与抽穗期的相关性接近极显著(t=-1.9479,p=0.06525)(图3)。由此可以看出栽培稻长日照抑制基因的遗传多样性促进了其抽穗期的多样性。
[0131]
此外,为了解析在不同的地理学区域多个长日照抑制基因是如何共同调节栽培稻抽穗期多样性的,还对不同基因进行单倍型进行组合分析,发现如果栽培稻的这6个长日照抑制基因同时有一个或一个以上基因的单倍型都属于其相应基因networktreegroup a时,那么这个栽培稻抽穗期要明显早于不携带group a中单倍型的栽培稻抽穗期,且差异极显著(图4a)。这些研究结果表明在栽培稻向高纬度长日照区域扩张的过程中,为了适应高纬度稻作区气温和日照长度要求,不同长日抑制基因networktree group a中的单倍型都受到了强烈的选择,长日照抑制基因group a中单倍型的累积是促进栽培稻抽穗期逐渐提前,使不同地理区域稻作区的栽培稻最终获得了最佳抽穗期的遗传基础。
[0132]
5、栽培稻长日照抑制基因的地理学进化模式
[0133]
为了解析3中栽培稻长日照抑制基因group a和group b中单倍型在1中从热带低纬度地区到温带高纬度区域(23
°
s到45
°
n)的地理学进化模式,对长基因单倍型的地理学分布区域和频率进行分析。由于遗传多样性分析表明基因se5和hd6遗传多样性低,且生物学进化保守,所以依然只分析了oscol4、dth7、phyb、ghd7、dth8、hd1这6个基因单倍型的地理学分布区域和出现频率。首先利用栽培稻单倍型进纬度分布信息,采用excel软件和r version 3.5.2的leaflet功能绘,分别为6个长日照抑制基因绘制groupa和groupb中单倍型的地理学分布频率热图heatmap和单倍型地理学分布图。
[0134]
然后从worldclim version 2database下载最冷月最低温度的原始数据(30s/km的分辨率),然后使用r version 3.5.2的sp和rgdal软件包,将气候因子原始数据转化为气
候因子密度分布图,最后根据栽培稻的单倍型信息和经纬度信息,将同时携带不同个数长日照抑制基因单倍型组合的栽培稻地理位置在气候因子密度分布图上标示出来。
[0135]
最后,为了确定单倍型组合与抽穗和地理学分布的关联性,采用r分别构建了单倍型组合与栽培稻抽穗期和地理学分布的boxplot,同是采用r中的anova和lsd软件包,评估了不同单倍型组合对栽培稻抽穗期和地理学分布影响的显著性。
[0136]
结果从图3的热图(heatmap),可以看出hd1 networktree group a中单倍型遍布于45
°
n~23
°
s的所有水稻种植区域,所以推测hd1在栽培稻抽穗期多样性进化的过程中最先受到了选择。另外5个基因的研究结果发现,如果栽培稻携带5个基因中任何一个基因的networktree group b中单倍型,那么其以高频率的状态出现在30
°
n以南的热带或者亚热带低纬度地理区域。同时,从这5个基因单倍型的地理学分布区域和频率也可以很清晰看出,有小部分栽培稻携带这这5个基因networktree group a中单倍型也开始出现在30
°
n~20
°
n这一地理区域,但是在这一区域内仍以group b中单倍型为主,即这5个基因group b中单倍型出现的频率要大于group a中出现的频率(图3)。与之相反,随着栽培稻种植区域继续北扩至30
°
n以北的温带高纬度稻作区,栽培稻长日照抑制基因的单倍型分布就以networktree group a中的单倍型为主了。这就说明随着栽培稻的北扩,为了适应温带高纬度地区的自然环境,这5个长日照抑制基因networktree group a中的单倍型开始逐渐取代其group b中的祖先单倍型。而且当栽培稻地理学分布范围跨越30
°
n并继续向北扩展后,networktree group a中的单倍型基本上完全取代了group b中单倍型,陆续在30
°
n的地理区域占主导地位(图3)。
[0137]
此外,为了研究多个长日照基因是如何同时控制栽培稻地理学分布区域的出现频率,对不同基因单倍型进行组合分析,结果从图4可以看出栽培稻同时有4个或4个以上的长日照基因的单倍型都属于其相应networktree group a时,这些栽培稻的地理学分布范围基本上完全位于30
°
n以北的温带高纬度地理区域。反之,栽培稻出现在30
°
n以南地理区域,且差异极显著(图4a,4c)。这就形成了长日照基因不同单倍型在30
°
n的地理学分界线。同时最冷月最低温度(min temperature of coldest month)的地理学温差密度分布图也显示30
°
n是地球南北温差的一条分界线,且这条温差分界线与这上述单倍型30
°
n分界线重合。这也再一次支持了在栽培稻北扩的过程中,为了适应高纬度地理区域的温度变异,不同长日照抑制基因networktree group a中的单倍型受到选择,然后逐渐累积,最后在30
°
n形成长日照基因不同单倍型的地理学分界线(图4b)。
[0138]
因此,上述栽培稻dth8、dth7、ghd7、hd1、phyb,oscol4这6个长日照抑制基因中有4个或4个以上长日照抑制基因单倍型为group a型时,则这一类栽培稻来源于或适于栽种在30
°
n或者30
°
n以北的稻作区。
[0139]
从上述可以看出,可以通过检测待测栽培稻dth8、dth7、ghd7、hd1、phyb,oscol4这6个基因的单倍型是否分别属于该基因的group a中单倍型,以及有几个基因同时属于其group a中单倍型判断待测栽培稻来源于或适于栽种的稻作区;具体如下:
[0140]
在长日照条件下培育待测栽培稻,培育至抽穗,检测该待测水稻中dth8、dth7、ghd7、hd1、phyb和oscol4这6个基因的单倍型;
[0141]
所述检测该待测水稻中dth8、dth7、ghd7、hd1、phyb、oscol4这6个基因的单倍型为提取抽穗期的待测栽培稻植株的基因组dna,扩增dth8、dth7、ghd7、hd1、phyb,oscol4这6个
基因,并进行测序。
[0142]
若符合如下条件:4个或4个以上基因的单倍型属于group a(表4所示)时,则待测栽培稻来源于或候选栽培稻来源于30
°
n以北;或待测栽培稻适于或候选栽培稻适于栽种在30
°
n以北;
[0143]
若不符合上述条件,则待测栽培稻不来源于或候选不来源于30
°
n以北;或待测栽培稻不适于或候选不适于栽种在30
°
n以北。
[0144]
也可以通过检测待测栽培稻中dth8、dth7、ghd7、hd1、phyb,oscol4这6个基因的单倍型判断待测栽培稻的抽穗期情况;具体如下:
[0145]
在长日照条件下培育待测栽培稻,培育至抽穗,检测该待测水稻中dth8、dth7、ghd7、hd1、phyb,oscol4这6个基因。
[0146]
所述检测该待测水稻中dth8、dth7、ghd7、hd1、phyb,oscol4这6个基因的单倍型为提取抽穗期的待测栽培稻植株的基因组dna,扩增dth8、dth7、ghd7、hd1、phyb,oscol4这6个基因,并进行测序。
[0147]
dth8、dth7、ghd7、hd1、phyb,oscol4这6个基因中某个基因的单倍型属于group a的待测植物抽穗期早于该基因单倍型属于group b的待测植物。
[0148]
实施例2、水稻长日照抑制开花基因在溯源水稻品种来源中的应用
[0149]
在长日照条件下(日照时长≥14小时/天)于2010年、2011年5月将表1所示的148个不同来源的栽培稻播种,培育至抽穗期。
[0150]
提取处于抽穗期的栽培稻植株的基因组dna作为模板,分别扩增对6个的长日照抑制基因dth8、dth7、ghd7、hd1、phyb、oscol4,得到目的基因。
[0151]
所需引物序列如下表2所示。
[0152]
将目的基因测序,若待测栽培稻中dth8、dth7、ghd7、hd1、phyb、oscol4这6个基因符合如下条件:4个或4个以上基因属于其各自基因的group a中的单倍型时,则待测栽培稻来源于或候选来源于30
°
n或30
°
n以北地区;若不符合上述条件,则待测栽培稻不来源于或候选不来源于30
°
n以北地区。
[0153]
结果如表3和表1所示,显示148个不同来源的样本中,用本发明的方法检测,全部来源溯源正确。
[0154]
实施例3、水稻长日照抑制开花基因在检测栽培稻是否适合栽种在30
°
n以北地区中的应用
[0155]
在长日照条件下(日照时长≥14小时/天)于2010年、2011年5月年将148个不同来源的栽培稻播种,培育至抽穗期。
[0156]
提取处于抽穗期的栽培稻植株的基因组dna作为模板,分别扩增6个的长日照抑制基因dth8、dth7、ghd7、hd1、phyb、oscol4,得到目的基因。
[0157]
所需引物序列如下表2所示。
[0158]
将目的基因测序,若待测栽培稻中dth8、dth7、ghd7、hd1、phyb、oscol4这6个基因符合如下条件:4个或4个以上基因属于group a单倍型时,则待测栽培稻适于栽种在30
°
n以北地区;若不符合上述条件,则待测栽培稻不适于或候选不适于栽种在30
°
n以北地区。
[0159]
结果如表3和表1所示,显示148个不同来源的样本中,用本发明的方法检测,其中31个待测栽培稻适于栽种在30
°
n以北。
[0160]
这与事实相符合,这一类栽培稻的确种植在30
°
n以北,而无法生长在30
°
n以南。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1