一种嵌合硫酸软骨素蛋白多糖4受体的T淋巴细胞及其制备方法和用途与流程
本发明属于生物工程领域,涉及一种用于治疗肿瘤的药物,具体来说是一种嵌合硫酸软骨素蛋白多糖4受体的t淋巴细胞(cspg4.car-t)及其制备方法和用途。
背景技术:
头颈部癌是指原发于上呼吸消化道包括鼻腔、副鼻窦、口腔、咽腔(包括鼻咽、口咽、喉咽)以及喉等部位的上皮源性恶性肿瘤。头颈部癌是人类常见的恶性肿瘤之一,发病率居所有恶性肿瘤第6位,多发于男性,且与年龄相关,好发于60岁以上人群。美国的最新肿瘤统计发现最近1年新发头颈癌约6.8万例,其中70.4%为男性。而在我国,每年约有13万例头颈癌新发,并有约7万例患者因头颈癌死亡。头颈癌病理类型主要为鳞癌,占95%以上,因此头颈部癌中绝大多数均是头颈部鳞状细胞癌(hnscc),而hnscc也成为头颈部癌的主要研究对象。外科手术、放化疗仍是目前hnscc治疗的主要方法。但由于头颈部重要血管神经密集,手术难度大,而放化疗对正常组织损伤大,且存在耐药复发的难题,目前总体的治疗效果仍不理想,5年生存率仅50%左右。与此同时,患者对术后生活质量的要求不断提升,疗效显著而副作用低的治疗手段亟待开发。
免疫治疗是肿瘤的第四大疗法,是指通过相应的免疫学手段来提高免疫效应细胞如自然杀伤细胞、cd8+t细胞等对肿瘤细胞的特异性识别和杀伤作用,最终达到治疗肿瘤的目的。肿瘤免疫治疗不会产生类似放化疗后的耐药性,而且疗效持久甚至有免疫记忆功能,还可以根据肿瘤患者特定的自身条件设计特异性的靶向治疗,契合精准治疗的理念。肿瘤免疫治疗主要包括单克隆抗体治疗、t细胞疗法、肿瘤疫苗疗法、细胞因子疗法等,其中单克隆抗体和t细胞疗法在肿瘤治疗中表现出的优越性使其越来越受到重视,有成为肿瘤一线治疗的潜力。
嵌合抗原受体(car)t细胞疗法是目前肿瘤免疫治疗研究的热门。car主要由2部分偶联形成,一段是能特异性结合肿瘤相关抗原的单链抗体(scfv),以及一个能激活t细胞发挥免疫效应功能的激活性受体。针对b细胞表面cd19抗原的car-t疗法在临床上治疗b细胞源性的血液肿瘤已经取得了巨大成功,但在治疗实体瘤的研究中却陷入挣扎,其中最主要的原因之一就是实体瘤具有高度异质性,缺乏一个稳定表达的肿瘤特异性抗原。因此,挖掘肿瘤特异性抗原以探索新型有效的治疗靶点,是目前所有实体瘤包括hnscc免疫治疗研究的难点与关键。
硫酸软骨素蛋白多糖4(cspg4)是一种细胞表面蛋白多糖,在正常组织细胞中的分布中受到严格调控,主要表达于软骨等正常组织,主要功能可能与细胞的生长、迁移、黏附及与细胞外基质的相互作用有关。cspg4最初是在黑色素瘤中被发现,后来研究发现在中枢神经系统中存在的神经-胶质细胞抗原2(ng2)是它的同源蛋白。有研究对多种实体瘤包括恶性黑色素瘤、乳腺癌、间皮瘤、胰腺癌、头颈鳞癌等的术后标本进行检测,发现cspg4在许多患者的肿瘤中表达比正常组织明显增高,但是它在肿瘤发展中的具体作用及机制尚不清晰。肿瘤干细胞是指肿瘤中具有自我更新能力并能产生异质性肿瘤细胞的细胞,对肿瘤的发生发展有着重要作用,而且对目前常用的放化疗手段不敏感,是放化疗后肿瘤复发的关键因素之一。有研究发现,恶性黑色素瘤、脑胶质瘤、结肠癌的肿瘤干细胞中时常发现有cspg4表达,提示靶向cspg4的治疗有可能成为杀伤肿瘤干细胞、根治肿瘤复发的新型有效武器。
嵌合硫酸软骨素蛋白多糖4受体的t淋巴细胞,其胞外段结构由信号肽序列、763.74或优化后的scfv序列、人免疫球蛋白g1(igg1)或优化后的铰链序列串联构成;跨膜区序列为cd28的跨膜区序列;胞内段结构由人的41bb胞内段共刺激域序列或cd28胞内段共刺激域序列串联人的cd3ζ胞内段序列构成。结构示意图如图12所示:
信号肽序列:mefglswlflvailkgvqcsr(seqidno.43)。
cd28跨膜序列:fwvlvvvggvlacysllvtvafiifwv(seqidno.44)。
cd3ζ序列:rvkfsrsadapayqqgqnqlynelnlgrreeydvldkrrgrdpemggkprrknpqeglynelqkdkmaeayseigmkgerrrgkghdglyqglstatkdtydalhmqalppr(seqidno.45)。
我们首先在头颈部肿瘤组织和细胞系中检测了cspg4的表达水平及与头颈部肿瘤干细胞标志物cd44的关系,探索cspg4作为头颈部癌免疫治疗靶点的可行性与价值。同时,目前针对cspg4靶点设计的cspg4.cart细胞已经被初步证实对多种肿瘤的细胞系有效。然而,目前报道的cspg4.cart的疗效不够显著,结构有待优化,而且对hnscc的具体疗效也尚无进一步的研究。基于以上研究报道,我们进一步设计并优化了一系列靶向cspg4蛋白的car,并观察其对hnscc细胞系的体内外的治疗效果。
技术实现要素:
针对现有技术中的上述技术问题,本发明提供了一种嵌合硫酸软骨素蛋白多糖4受体的t淋巴细胞(cspg4.car-t)及其制备方法和用途,所述的这种嵌合硫酸软骨素蛋白多糖4受体的t淋巴细胞(cspg4.car-t)及其制备方法和用途要解决现有技术中的针对cspg4靶点设计的cspg4.cart细胞治疗肿瘤的效果不显著的技术问题。
本发明提供了一种嵌合硫酸软骨素蛋白多糖4受体的t淋巴细胞,包括胞外段结构、人的cd28跨膜序列、胞内段结构;所述的胞外段结构由信号肽序列、优化的cspg4单链抗体序列、人免疫球蛋白g2突变序列为铰链区的序列依次串联构成;所述的优化的cspg4单链抗体的基因序列如seqidno.33-42中任一所示;所述的铰链区的基因序列如seqidno.31所示;所述的胞内段结构由人的cd28或41bb胞内段共刺激域序列、人的cd3ζ胞内段序列串联构成。
进一步的,所述的信号肽序列如seqidno.43所示。
进一步的,所述的人的cd28跨膜序列如seqidno.44所示。
进一步的,所述的人的cd3ζ胞内段序列如seqidno.45所示。
进一步的,所述的人的cd28胞内段共刺激域序列如seqidno.26所示。
进一步的,所述的人的41bb胞内段共刺激域序列如seqidno.27所示。
本发明还提供了上述一种嵌合硫酸软骨素蛋白多糖4受体的t淋巴细胞的制备方法,包括如下步骤:
1)分别合成优化的cspg4单链抗体序列、人免疫球蛋白g2突变序列;所述的优化的cspg4单链抗体的基因序列如seqidno.33-42中任一所示示;所述的铰链区的基因序列如seqidno.31所示;
2)将优化的cspg4单链抗体序列、人免疫球蛋白g2突变序列、人的cd28或41bb胞内段共刺激域序列、人的cd3ζ胞内段序列连接后克隆至载体中;
3)将人肾上皮细胞系重悬于含质量百分比浓度为10%胎牛血清的dmem培养基中,利用脂质体转染系统,将慢病毒包装质粒与步骤2)的载体加入至人肾上皮细胞系培养基中,48小时后收集细胞上清冻存在-80℃冰箱中备用;
4)将原代t细胞培养在含有质量百分比浓度为10%的胎牛血清、100u/ml的青霉素和100μg/ml的硫酸链霉素的rpmi-1640培养基中;使用包被了抗cd3和抗cd28抗体的磁珠激活t细胞,共孵育后加入步骤3)制备好的慢病毒进行感染,感染后再将细胞上清去掉并置换成新鲜的t细胞完全培养基;培养后获得嵌合硫酸软骨素蛋白多糖4受体的t淋巴细胞。
本发明还提供了上述嵌合硫酸软骨素蛋白多糖4受体的t淋巴细胞在制备治疗头颈部肿瘤的药物中的用途。
本发明还提供了一种载体,含有胞外段结构、人的cd28跨膜序列、胞内段结构;所述的胞外段结构由信号肽序列、优化的cspg4单链抗体序列、人免疫球蛋白g2突变序列为铰链区的序列依次串联构成;所述的优化的cspg4单链抗体的基因序列如seqidno.33-42中任一所示;所述的铰链区的基因序列如seqidno.31所示;所述的胞内段结构由人的cd28或41bb胞内段共刺激域序列、人的cd3ζ胞内段序列串联构成。
本发明还提供了一种慢病毒,含有上述的载体。
我们首先发现在许多头颈恶性肿瘤标本和细胞系中cspg4蛋白存在高表达,靶向cspg4治疗头颈鳞癌具有可行性。同时,我们发现cspg4与头颈部肿瘤干细胞标志物cd44存在共表达情况,提示靶向cspg4治疗头颈鳞癌具有重要意义。
我们发现目前研究的以cspg4单抗763.74作为单链抗体(scfv)序列,以人免疫球蛋白g1(higg1)为铰链区,以cd28或者41bb胞内段序列构成cspg4.car-t细胞在体外可以较好的杀伤cspg4阳性的靶细胞,但在体内杀伤靶向肿瘤细胞的能力较差。
一方面,本发明通过优化car结构中的铰链区igg1序列,以降低cspg4.car对iggfc受体(fcγr)的亲和力。我们尝试将igg1的ch2ch3序列截除(chc)、截短(chs)、替换成突变的igg2(chm)或者替换成在cd44.car中应用的突变截短的低亲和力神经生长因子受体(nms)序列,发现只有cspg4.chm.car能有效地在细胞膜表面表达car并且识别cspg4蛋白。进一步的体内外研究证实,cspg4.chm.car-t的体内抗肿瘤活性显著提高。
另一方面,本发明将cspg4单抗763.74的scfv序列进行优化,发现优化后的cspg4.car-t的同样能特异性地识别cspg4蛋白。相较于之前以763.74为scfv的car-t,这种优化的car-t的自激活信号和耗竭信号显著减弱,并且在体外试验中表现出更强的杀伤活性。在肿瘤小鼠模型上,优化的cspg4.car-t细胞能更好的控制住肿瘤的增殖,并在肿瘤切片中发现优化的cspg4.car-t细胞能更多的浸润到肿瘤微环境中。
本发明和已有技术相比,其技术进步是显著的。本发明设计并优化了cspg4.car-t细胞,并证实了优化后的cspg4.car-t细胞能在体内外有效的杀伤cspg4阳性的肿瘤细胞,为临床治疗靶向治疗cspg4阳性的实体肿瘤提供了新的方法和思路。
附图说明
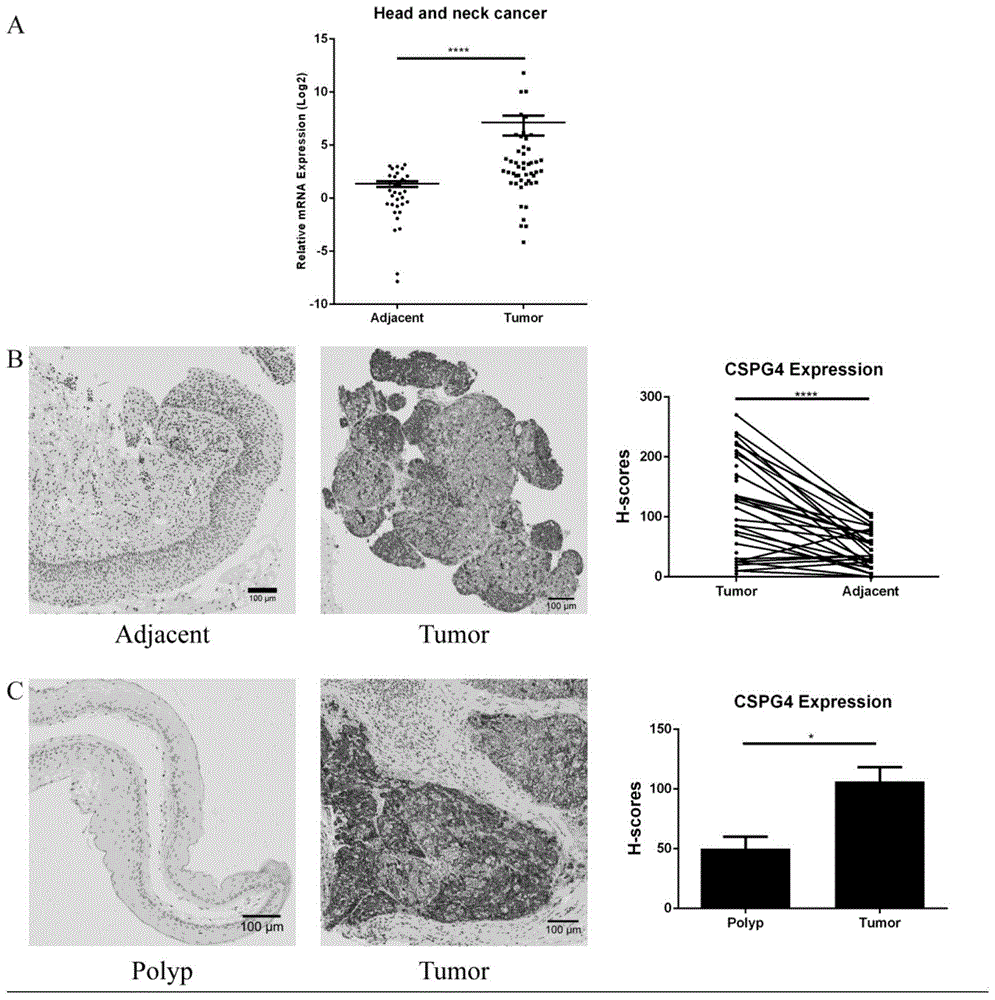
图1.a:q-pcr检测头颈部肿瘤及癌旁对照组织中cspg4mrna的转录水平。b:免疫组化检测头颈部肿瘤及癌旁对照组织中cspg4蛋白表达水平。c:免疫组化检测头颈部肿瘤及头颈部炎症息肉组织中cspg4蛋白表达水平。
图2.a:q-pcr检测头颈部肿瘤多个细胞系和鼻咽正常鳞状上皮细胞系np69中cspg4mrna转录水平。b:westernblot检测头颈部肿瘤多个细胞系和鼻咽正常鳞状上皮细胞系np69中cspg4总蛋白表达水平。c:流式技术检测头颈部肿瘤多个细胞系和鼻咽正常鳞状上皮细胞系np69中cspg4蛋白在细胞膜表面的表达水平。
图3.a:tcga大数据库分析头颈鳞癌标本中cspg4mrna和cd44mrna转录水平的相关性。b:免疫荧光检测头颈肿瘤标本中cspg4蛋白与cd44蛋白的共表达情况。c:流式技术检测头颈部肿瘤细胞系中cspg4和cd44蛋白的共表达情况。
图4.a:cspg4.igg1.cd28.car和cspg4.igg1.41bb.car对靶细胞的体外杀伤试验。b:cspg4.igg1.cd28.car和cspg4.igg1.41bb.car对靶细胞的体内杀伤试验。
图5.a:流式技术检测铰链区改造后cspg4.car的表达情况。b:流式技术检测铰链区改造后的cspg4.car对cspg4蛋白的识别能力。
图6.a:以chm作为铰链区的cspg4.car.t的体外杀伤试验。b:以chm作为铰链区的cspg4.car.t细胞的体内杀伤试验。
图7.流式技术检测cspg4.cd28.car和cspg4.41bb.cart细胞在无抗原刺激的情况下激活相关受体cd69和icos及t细胞耗竭相关受体pd-1的表达。
图8.a:流式技术检测763.74scfv序列优化以后的cspg4.car的表达情况。b:流式技术检测scfv序列优化以后的cspg4.car对cspg4抗原的特异性识别能力。c:流式技术检测scfv序列优化以后的cspg4.car的激活相关受体cd69和t细胞耗竭相关受体pd-1的表达。
图9.a:挑选了2个优化后的cspg4.car感染人原代t细胞,流式技术检测相应原代t细胞激活相关受体cd69和t细胞耗竭相关受体pd-1的表达。b:优化以后的cspg4.car对靶细胞的体外杀伤试验。c:elisa检测优化以后的cspg4.car在杀伤肿瘤时分泌相关细胞炎症因子的能力。
图10.a:优化以后的cspg4.car对靶细胞的体内杀伤试验。b:优化以后的cspg4.car.t细胞治疗后荷瘤小鼠的生存曲线。
图11.a:westernblot检测优化以后的cspg4.car在静息状态下的4g10蛋白表达水平。b:免疫荧光检测优化以后的cspg4.car.t细胞治疗后肿瘤组织中的t细胞浸润情况。
图12为嵌合硫酸软骨素蛋白多糖4受体的t淋巴细胞的结构示意图。
具体实施方式
实施例1:头颈部鳞癌中cspg4的表达
肿瘤中cspg4mrna检测:取新鲜或-80℃冻存的肿瘤组织剪成碎块,约50mg组织中加入1mltrizon试剂,再在匀浆仪上匀浆处理,然后用康维世纪cw0581s试剂盒按照说明书提取mrna。提取的mrna测浓度后用takararr036a试剂盒按说明书操作逆转录成cdna保存。之后再用实时荧光定量试剂盒(takararr420a)按说明书操作检测cdna中cspg4的表达水平,所用cspg4引物:
former:ctggagaatggtggaagag(seqidno.1)
reverse:ggacagtgacagtgaagg(seqidno.2)
gapdh引物:
former:caaggtcatccatgacaactttg(seqidno.3)
reverse:gtccaccaccctgttgctgtag(seqidno.4)。
肿瘤组织切片染色:首先将肿瘤组织浸没在4%的多聚甲醛中4℃固定,经过包埋、切片(片厚4μm)后获得白片。后按照即用型免疫组织化学试剂盒(上海生工,c506333-0020)说明书操作。cspg4抗体:atlasantibodieshpa002951。染色完成后组织切片在显微镜明场下拍摄记录。免疫组化结果由经验丰富的病理科医师进行组织化学评分(histochemistryscore,h-score),具体方法为对细胞染色强度分为无、弱、中、强4个等级,无黄色颗粒样沉淀为无,记0分;淡黄色为弱,记1分;棕黄色为中,记2分;深棕黄色为强,记3分。再将该分数分别乘以对应细胞所占的百分比,相加的总值即为评分值,范围为0-300。
如图1所示,a:头颈部肿瘤中cspg4mrna转录水平显著高于癌旁对照组织。b:头颈部肿瘤中cspg4蛋白表达水平显著高于癌旁对照组织。c:头颈部肿瘤中cspg4蛋白表达水平显著高于息肉组织。
结论:在头颈鳞癌临床标本中cspg4转录和蛋白表达水平异常增高。
实施例2:人头颈鳞癌细胞系中cspg4的转录和表达
细胞系中cspg4mrna检测:将检测细胞用pbs洗涤后收集沉淀,每1,000,000个细胞加入1mltrizon裂解,后续检测方法同实施例1中的肿瘤mrna检测方法。
westernblot检测细胞系中cspg4蛋白表达:收集细胞,用蛋白抽提试剂盒(碧云天,p0013)按照说明书抽提总蛋白,bca法测定蛋白浓度,加入上样缓冲液制备电泳样品,聚丙烯酰胺凝胶电泳,经转膜、封闭、一抗及二抗孵育后滴入ecl显影试剂在凝胶图象处理系统上显影分析。所用cspg4抗体:abcamab139408。
流式细胞术分析细胞系中cspg4蛋白表达:将检测细胞用facs缓冲液(磷酸缓冲液pbs+2%胎牛血清)洗涤后用经一抗、二抗孵育及dapi染色后在bdlsrfortessa流式细胞仪(bdbioscience)上检测然后用flowjosoftware软件(treestar)进行数据分析。所用cspg4抗体:santacruzsc53389。
如图2所示,a:头颈部肿瘤多个细胞系中cspg4mrna转录水平显著高于正常鳞状上皮细胞系np69。b:头颈部肿瘤多个细胞系中cspg4总蛋白表达水平显著高于正常鳞状上皮细胞系np69。c:头颈部肿瘤多个细胞系中cspg4蛋白主要表达在细胞膜表面。
结论:在头颈鳞癌细胞系中cspg4转录显著高于正常鼻咽上皮细胞np69,cspg4总蛋白表达水平也显著增高,且主要位于细胞膜外,为car-t靶向治疗提供了可能。
实施例3:在头颈鳞癌中cspg4与肿瘤干细胞标志物cd44具有共表达特征
癌症和基因肿瘤图谱(tcga)大数据分析:从tcga大数据库中(网址:https://cancergenome.nih.gov)下载所有头颈鳞癌标本的基因转录本信息,pearson检验分析cspg4和cd44基因转录水平的相关性。
肿瘤组织切片cspg4与cd44共染色:肿瘤组织白片的获取方法同实施例1。白片经烤片、脱蜡、水化、抗原修复、酶灭活、封闭、一抗二抗孵育以及dapi复染后封片,最后在荧光显微镜下观察分析。所用抗体:cspg4抗体:atlasantibodieshpa002951;cd44抗体:biolegend103006。
流式细胞术分析头颈鳞癌细胞系中cspg4和cd44蛋白共表达:具体试验操作方法见实施例2。所用cspg4抗体:santacruzsc53389;cd44抗体:miltenyibiotec130-095-177。
如图3所示,a:tcga大数据库分析发现头颈鳞癌标本中cspg4mrna转录水平和cd44mrna转录水平正相关。b:免疫荧光显示头颈肿瘤标本中cspg4蛋白与cd44蛋白存在广泛共表达。c:头颈部肿瘤中表达cd44蛋白的细胞系同时也表达cspg4蛋白。
结论:头颈鳞癌中表达肿瘤干细胞标志物cd44的肿瘤细胞往往同时也表达cspg4蛋白,靶向治疗cspg4阳性细胞有望杀伤头颈肿瘤的肿瘤干细胞。
实施例4:cspg4.car的初步构建及杀伤效果
cspg4.car质粒和慢病毒的初步构建:我们取cspg4单克隆抗体763.74的单链抗体(scfv)序列,以人免疫球蛋白g1(higg1)作为铰链序列,取人cd28的跨膜序列作为跨膜区,以人的cd28或41bb的胞内段序列作为共刺激分子,最后串联人的cd3ζ胞内段序列构成初步的cspg4.car,即cspg4.higg1.cd28和cspg4.higg1.41bbcar。氨基酸序列经过密码子优化后转换成碱基序列并由公司合成(qinglanbiotech)。氨基酸序列见下表:
所有car的碱基序列最终通过gibson连接的方式(neb#e2611l)克隆至phr-sffv-ires-egfp载体中。将293t细胞(takara#632180)重悬于含10%胎牛血清的dmem培养基(gibco#11995-065)中,利用脂质体转染系统(mirus#2300),将500ng慢病毒包装质粒pcmvdr8.92(addgene#8455)和pmd2.g(addgene#12259)与500ngcspg4.car质粒按照脂质体转染说明书的操作步骤加入至293t细胞培养基中。48小时后收集细胞上清冻存在-80℃冰箱中备用。
人的原代t细胞培养及慢病毒感染:人的原代t细胞采自健康的知情志愿者。原代t细胞培养在含有10%的胎牛血清、100u/ml的青霉素和100μg/ml的硫酸链霉素的rpmi-1640培养基中(以上试剂均购自gibco)。为了维持t细胞的增殖,培养基中加入100u/ml的hil-2(sigma-aldrich)。使用包被了抗cd3和抗cd28抗体的磁珠(lifetechnologies#11132d)激活t细胞。t细胞与磁珠在1:3的比例下共孵育24小时后加入制备好的慢病毒进行感染。18小时后将细胞上清去掉并置换成新鲜的t细胞完全培养基。t细胞经磁珠刺激5天后需将磁珠撤去并每隔2-3天补充新鲜的t细胞完全培养基。
car-t体外杀伤试验:将表达cspg4和mcherry荧光双阳性的k562靶细胞与不表达cspg4和mcherry荧光的k562非靶细胞按照1:1的细胞比率混合后与car-t细胞按照具体实验安排的细胞比例共孵育3天后上流式细胞仪检测。以不含t细胞的k562混合细胞群作为对照组,分析得到靶细胞所占细胞的比例(ck%);在含t细胞的实验组中通过cd3ε染色将t细胞区分出k562细胞群,然后分析k562靶细胞所占k562细胞群的比例(ex%),t细胞的杀伤效率=(1-ex%/ck%)×100%
car-t体内杀伤试验:取5至8周大的免疫缺陷型(nsg)小鼠,在小鼠背部皮下接种1×106个表达有萤火虫荧光素酶(luc)基因的k562-cspg4靶细胞,待靶细胞在体内稳定4天后向nsg小鼠尾静脉注射入5×106个car-t细胞进行治疗。接下来每周通过动物活体成像技术检测小鼠体内肿瘤增殖情况。具体实施操作:向荷瘤小鼠的腹腔注射萤火虫荧光素底物,底物用量按照1.5mg/g小鼠体重施用;10分钟后待底物充分循环至小鼠全身,用2.5%-3.5%的异氟烷气体麻醉小鼠进行成像。生物发光成像通过ivis光谱成像系统(perkinelmer)执行,荧光定量数据通过活体成像软件(perkinelmer)获得。
如图4所示,a:cspg4.igg1.cd28.car和cspg4.igg1.41bb.car在体外能有效杀伤肿瘤细胞。b:cspg4.igg1.cd28.car和cspg4.igg1.41bb.car在体内不能有效控制肿瘤的增殖。
结论:cspg4.higg1.cd28和cspg4.higg1.41bbcart在体外试验中能较好的杀伤靶细胞,但在体内试验中几乎完全不能控制肿瘤的增殖。
实施例5:cspg4.car铰链区结构的优化
cspg4.car铰链区结构的优化:因有报道称higg1抗体的可结晶片段(fc)功能阈ch2ch3序列与其他细胞的fcγ受体亲和力较高,可因此引起相应cart细胞的非特异性激活,导致其在体内复杂细胞环境中抑瘤效果减弱,我们尝试优化cspg4.car结构的higg1铰链区ch2ch3序列。我们尝试将ch2ch3序列去除(ch2ch3cut,chc)、截短(ch2ch3short,chs)、突变(ch2ch3mutant,chm)以及更换为突变截短的低亲和力神经生长因子受体(nms),以未改造的ch2ch3(ch2ch3widetype,chw)作为对照,并进行后续的检测验证,具体质粒构建方法见实施例4。所用氨基酸序列见下表:
铰链区改造后cspg4.car的表达和抗原识别能力检测验证:首先我们用识别higg或nms的抗体通过流式技术检测cspg4.car的表达水平。随后我们用crispr/cas9技术构建了cspg4基因敲除细胞系(cspg4-ko),再分别用cspg4野生型(cspg4-wt)和cspg4-ko与cspg4.cart细胞以3:1的比例共孵育24小时,通过流式技术检测t细胞表面激活性受体cd69的表达水平,检测cspg4.cart对cspg4抗原的特异性识别能力。流式检测方法参考实施例2,所用higg抗体:jacksonimmunoresearch109-605-098;nms抗体:bd560326;cd69抗体:biolegend310906。
如图5所示,a:改造了铰链区igg1的序列后的cspg4.car的表达情况。b:改造了铰链区igg1的序列后的cspg4.car对cspg4蛋白的识别能力,只有突变后的chm作为铰链区的cspg4.car能特异性识别cspg4抗原。
结论:上述改造的cspg4.car中只有以chm作为铰链区的cspg4.car可以正常的表达cspg4.car同时能特异性识别cspg4抗原。
实施例6:cspg4.chm.car的体内外杀伤效果
我们分别构建cspg4.chm.cd28.car和cspg4.chm.41bb.cart检测其对靶细胞的体内外杀伤效果,具体操作方法参照实施例4。
如图6所示,a:以chm作为铰链区的cspg4.car.t细胞在体外能有效杀伤肿瘤细胞。b:以chm作为铰链区的cspg4.car.t细胞在体内能显著抑制但是不能完全控制肿瘤的增殖。
结论:cspg4.chm.cd28和cspg4.chm.41bbcart在体外试验中能较好的杀伤靶细胞,在体内试验中可以抑制但不能完全控制住肿瘤的增殖。
实施例7:cspg4.car在静息状态下具有较强的激活和耗竭信号
为了找到cspg4.cart体内肿瘤杀伤效果不佳的原因,我们用流式技术检测了cspg4.cart在静息状态下激活相关蛋白cd69、icos和耗竭相关蛋白pd-1的表达水平,具体操作方法参考实施例2。所用cd69抗体:biolegend310906;icos抗体:biolegend313520;pd-1抗体:ebioscience12-2799-42。
如图7所示,cspg4.cd28.car和cspg4.41bb.cart细胞在无抗原刺激的情况下也表达激活相关受体cd69和icos,同时还表达t细胞耗竭相关受体pd-1。
结论:cspg4.cd28.car和cspg4.41bb.cart在静息状态下表达较强的激活和耗竭信号,这可能与它们体内杀伤肿瘤效果不佳有关。
实施例8:cspg4.carscfv序列的优化
我们对763.74scfv序列的框架结构进行改造优化,并对优化后的cspg4.car的表达能力、特异性抗原识别能力以及静息状态下的自激活和耗竭信号进行检测评估,具体方法参考实施例5和7。优化后cspg4.carscfv氨基酸序列见下表。
如图8所示,a:将763.74的scfv序列优化以后的cspg4.car的表达情况,发现各种优化版本上cspg4.car均能较好表达且表达水平增高。b:将763.74的scfv序列优化以后的cspg4.car均能特异性的识别cspg4抗原。c:将763.74的scfv序列优化以后的cspg4.car的自激活信号cd69和耗竭信号pd-1的表达均显著下降。
结论:优化后的cspg4.car均能在t细胞膜上正常表达cspg4.car并能特异性识别cspg4抗原,并且在静息状态下的自激活和耗竭信号显著下降。
实施例9:scfv优化后cspg4.car的体外杀伤效果
我们选取了2种代表性的优化scfv,即h-cspg4和m-cspg4-5,然后制备慢病毒感染人原代t细胞,流式技术检测它们的自激活和耗竭信号,以及体外对靶细胞的杀伤效果(参照实施例4)。同时我们还用elisa方法检测了杀伤过程种t细胞分泌的细胞炎症因子,取细胞上清液按照试剂盒说明书操作处理后在在酶标仪下于450nm处检测吸光度,记录分析数据。所用elisa试剂盒:干扰素γ(ifn-γ):bd555142;肿瘤坏死因子α(tnf-α):bd555212;白细胞介素2(il-2):bd555190。
如图9所示,a:挑选了2个优化后的cspg4.car感染人原代t细胞,发现其自激活信号cd69和耗竭信号pd-1的表达均显著下降。b:优化以后的cspg4.car体外杀伤肿瘤的效果更强。c:优化以后的cspg4.car在杀伤肿瘤时具有更好的分泌细胞因子和炎症因子的能力。
结论:h-cspg4.chm.41bb.car和m-cspg4-5.chm.41bb.cart细胞在静息状态下自激活信号cd69和耗竭信号pd-1显著下降,体外杀伤能力和细胞炎症因子分泌能力显著增强。
实施例10:scfv优化后cspg4.car的体内杀伤效果
我们进一步在nsg小鼠上检测h-cspg4.chm.41bb.car和m-cspg4-5.chm.41bb.cart细胞对肿瘤的体内杀伤效果,定量分析肿瘤的生长曲线和小鼠的生存时间,具体操作参考实施例4。
如图10所示,a:优化以后的cspg4.car体内杀伤肿瘤的能力显著增强。b:优化以后的cspg4.car.t细胞能显著延长荷瘤小鼠的生存时间。
结论:h-cspg4.chm.41bb.car和m-cspg4-5.chm.41bb.cart细胞在体内试验种可以显著控制肿瘤的增殖,延长荷瘤小鼠的生存时间。
实施例11:scfv优化后cspg4.car杀伤效果提升的机制分析
我们进一步使用抗磷酸化酪氨酸抗体克隆4g10通过westernblot方法检测分析h-cspg4.chm.41bb.car和m-cspg4-5.chm.41bb.cart细胞静息状态下的自磷酸化水平,操作方法参考实施例2。同时我们取出体内试验中小鼠体内的肿瘤进行免疫荧光分析t细胞的浸润情况,ki67反应肿瘤的增殖情况,gfp和cd3提示t细胞的浸润情况,具体操作方法参考实施例3。
如图11所示,a:优化以后的cspg4.car在静息状态下的磷酸化水平显著下降。b:优化以后的cspg4.car.t细胞能更好地浸润在肿瘤微环境中。
结论:h-cspg4.chm.41bb.car和m-cspg4-5.chm.41bb.cart细胞在未刺激状态下的自磷酸化水平显著低于优化前,能更广泛的在肿瘤中增殖浸润,有助于它们体内肿瘤中更好的发挥抑瘤效果。
序列表
<110>复旦大学附属眼耳鼻喉科医院
<120>一种嵌合硫酸软骨素蛋白多糖4受体的t淋巴细胞及其制备方法和用途
<160>45
<170>siposequencelisting1.0
<210>1
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>1
ctggagaatggtggaagag19
<210>2
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>2
ggacagtgacagtgaagg18
<210>3
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>3
caaggtcatccatgacaactttg23
<210>4
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>4
gtccaccaccctgttgctgtag22
<210>5
<211>238
<212>prt
<213>homosapiens
<400>5
metalaglnvallysleulysgluserglyprogluleulyslyspro
151015
glygluthrvallysilesercyslysalaserglytyrthrphethr
202530
asptyrsermethistrpvallyslysthrproglylysglyleulys
354045
trpleuglytrpileasnthralathrglygluprothrtyralaasp
505560
aspphelysglyargphealaileserleugluthrseralaargthr
65707580
valtyrleuglnileasnasnleuargasngluaspthralathrtyr
859095
phecysphesertyrtyrasptyrtrpglyglnglythrthrvalthr
100105110
valserserglyglyglyglyserglyglyglyglyserglyglygly
115120125
glyleuaspilelysleuserglnserproserileleuservalthr
130135140
proglygluthrvalserleusercysargalaserglnthriletyr
145150155160
lysasnleuhistrptyrglnglnlysserhisargserproargleu
165170175
leuilelystyrglyseraspserileserglyileproserargphe
180185190
thrglyserglyserglythrasptyrthrleuasnileasnserval
195200205
lysprogluaspgluglyiletyrtyrcysleuglnglytyrserthr
210215220
protrpthrpheglyglyglythrlysleugluilelysarg
225230235
<210>6
<211>231
<212>prt
<213>homosapiens
<400>6
prolyssercysasplysthrhisthrcysproprocysproalapro
151015
gluleuleuglyglyproservalpheleupheproprolysprolys
202530
aspthrleumetileserargthrprogluvalthrcysvalvalval
354045
aspvalserhisgluaspprogluvallyspheasntrptyrvalasp
505560
glyvalgluvalhisasnalalysthrlysproargglugluglntyr
65707580
asnserthrtyrargvalvalservalleuthrvalleuhisglnasp
859095
trpleuasnglylysglutyrlyscyslysvalserasnlysalaleu
100105110
proalaproileglulysthrileserlysalalysglyglnproarg
115120125
gluproglnvaltyrthrleuproproserargaspgluleuthrlys
130135140
asnglnvalserleuthrcysleuvallysglyphetyrproserasp
145150155160
ilealavalglutrpgluserasnglyglnprogluasnasntyrlys
165170175
thrthrproprovalleuaspseraspglyserphepheleutyrser
180185190
lysleuthrvalasplysserargtrpglnglnglyasnvalpheser
195200205
cysservalmethisglualaleuhisasnhistyrthrglnlysser
210215220
leuserleuserproglylys
225230
<210>7
<211>41
<212>prt
<213>homosapiens
<400>7
argserlysargserargleuleuhisserasptyrmetasnmetthr
151015
proargargproglyprothrarglyshistyrglnprotyralapro
202530
proargaspphealaalatyrargser
3540
<210>8
<211>42
<212>prt
<213>homosapiens
<400>8
lysargglyarglyslysleuleutyrilephelysglnprophemet
151015
argprovalglnthrthrglnglugluaspglycyssercysargphe
202530
proglugluglugluglyglycysgluleu
3540
<210>9
<211>231
<212>prt
<213>homosapiens
<400>9
prolyssercysasplysthrhisthrcysproprocysproalapro
151015
gluleuleuglyglyproservalpheleupheproprolysprolys
202530
aspthrleumetileserargthrprogluvalthrcysvalvalval
354045
aspvalserhisgluaspprogluvallyspheasntrptyrvalasp
505560
glyvalgluvalhisasnalalysthrlysproargglugluglntyr
65707580
asnserthrtyrargvalvalservalleuthrvalleuhisglnasp
859095
trpleuasnglylysglutyrlyscyslysvalserasnlysalaleu
100105110
proalaproileglulysthrileserlysalalysglyglnproarg
115120125
gluproglnvaltyrthrleuproproserargaspgluleuthrlys
130135140
asnglnvalserleuthrcysleuvallysglyphetyrproserasp
145150155160
ilealavalglutrpgluserasnglyglnprogluasnasntyrlys
165170175
thrthrproprovalleuaspseraspglyserphepheleutyrser
180185190
lysleuthrvalasplysserargtrpglnglnglyasnvalpheser
195200205
cysservalmethisglualaleuhisasnhistyrthrglnlysser
210215220
leuserleuserproglylys
225230
<210>10
<211>14
<212>prt
<213>homosapiens
<400>10
prolyssercysasplysthrhisthrcysproprocyspro
1510
<210>11
<211>124
<212>prt
<213>homosapiens
<400>11
prolyssercysasplysthrhisthrcysproprocysproalapro
151015
gluleuleuglyglyproservalpheleupheproprolysprolys
202530
aspthrleumetileserargthrprogluvalthrcysvalvalval
354045
aspvalserhisgluaspprogluvallyspheasntrptyrvalasp
505560
glyvalgluvalhisasnalalysthrlysproargglugluglntyr
65707580
asnserthrtyrargvalvalservalleuthrvalleuhisglnasp
859095
trpleuasnglylysglutyrlyscyslysvalserasnlysalaleu
100105110
proalaproileglulysthrileserlysalalys
115120
<210>12
<211>228
<212>prt
<213>homosapiens
<400>12
gluarglyscyscysvalglucysproprocysproalaproproval
151015
alaglyproservalpheleupheproprolysprolysaspthrleu
202530
metileserargthrprogluvalthrcysvalvalvalaspvalser
354045
hisgluaspprogluvalglnpheasntrptyrvalaspglyvalglu
505560
valhisasnalalysthrlysproargglugluglnpheglnserthr
65707580
pheargvalvalservalleuthrvalvalhisglnasptrpleuasn
859095
glylysglutyrlyscyslysvalserasnlysglyleuproalapro
100105110
ileglulysthrileserlysthrlysglyglnproarggluprogln
115120125
valtyrthrleuproproserargaspgluleuthrlysasnglnval
130135140
serleuthrcysleuvallysglyphetyrproseraspilealaval
145150155160
glutrpgluserasnglyglnprogluasnasntyrlysthrthrpro
165170175
provalleuaspseraspglyserphepheleutyrserlysleuthr
180185190
valasplysserargtrpglnglnglyasnvalphesercysserval
195200205
methisglualaleuhisasnhistyrthrglnlysserleuserleu
210215220
serproglylys
225
<210>13
<211>140
<212>prt
<213>homosapiens
<400>13
lysglualacysprothrglyleutyrthrhisserglyglucyscys
151015
lysalacysasnleuglygluglyvalalaglnprocysglyalaasn
202530
glnthrvalcysgluprocysleuaspservalthrpheseraspval
354045
valseralathrgluprocyslysprocysthrglucysvalglyleu
505560
glnsermetseralaprocysvalglualaaspaspalavalcysarg
65707580
cysalatyrglytyrtyrglnaspgluthrthrglyargcysgluala
859095
cysargvalcysglualaglyserglyleuvalphesercysglnasp
100105110
lysglnasnthrvalcysgluglucysproaspglythrtyrserasp
115120125
glualaalaargalaalaaspalaglucysgluglu
130135140
<210>14
<211>234
<212>prt
<213>homosapiens
<400>14
glnvalglnleuvalglnserglysergluleulyslysproglyala
151015
servallysvalsercyslysalaserglytyrthrphethrasptyr
202530
sermethistrpvalargglnalaproglyglnglyleuglutrpmet
354045
glytrpileasnthralathrglygluprothrtyralaaspaspphe
505560
lysglyargphevalpheserleuaspthrservalserthralatyr
65707580
leuglnileserserleulysalagluaspthralavaltyrtyrcys
859095
phesertyrtyrasptyrtrpglyglnglythrthrvalthrvalser
100105110
serglyglyglyglyserglyglyglyglyserglyglyglyglyglu
115120125
ilevalleuthrglnserproasppheglnservalthrprolysglu
130135140
lysvalthrilethrcysargalaserglnthriletyrlysasnleu
145150155160
histrptyrglnglnlysproaspglnserprolysleuleuilelys
165170175
tyrglyseraspserileserglyileproserargpheserglyser
180185190
glyserglythraspphethrleuthrileasnserleuglualaglu
195200205
aspalaalathrtyrtyrcysleuglnglytyrserthrprotrpthr
210215220
pheglyglyglythrlysvalgluilelys
225230
<210>15
<211>234
<212>prt
<213>homosapiens
<400>15
glnileglnleuvalglnserglyprogluleulyslysproglyglu
151015
thrvallysilesercyslysalaserglytyrthrphethrasptyr
202530
sermetasntrpvallysglnalaproglylysglyleulystrpmet
354045
glytrpileasnthralathrglygluprothrtyralaaspaspphe
505560
lysglyargphealapheserleugluthrseralaserthralatyr
65707580
leuglnileasnasnleulysasngluaspthralathrtyrphecys
859095
phesertyrtyrasptyrtrpglyglnglythrthrleuthrvalser
100105110
serglyglyglyglyserglyglyglyglyserglyglyglyglyasp
115120125
ilevalleuthrglnserproalathrleuservalthrproglyasp
130135140
argvalserleusercysargalaserglnthriletyrlysasnleu
145150155160
histrptyrglnglnlysserhisgluserproargleuleuilelys
165170175
tyrglyseraspserileserglyileproserargpheserglyser
180185190
glyserglythraspphethrleuserileasnservalgluthrglu
195200205
asppheglymettyrphecysleuglnglytyrserthrprotrpthr
210215220
pheglyglyglythrlysleugluilelys
225230
<210>16
<211>234
<212>prt
<213>homosapiens
<400>16
glnileglnleuvalglnserglyprogluleulyslysproglyglu
151015
thrvallysilesercyslysalaserglytyrthrphethrasptyr
202530
sermethistrpvallysglnalaproglylysglyleulystrpmet
354045
glytrpileasnthralathrglygluprothrtyralaaspaspphe
505560
lysglyargphealapheserleugluthrseralaserthralatyr
65707580
leuglnileasnasnleulysasngluaspthralathrtyrphecys
859095
phesertyrtyrasptyrtrpglyglnglythrthrleuthrvalser
100105110
serglyglyglyglyserglyglyglyglyserglyglyglyglyasp
115120125
ilevalleuthrglnserproalathrleuservalthrproglyasp
130135140
argvalserleusercysargalaserglnthriletyrlysasnleu
145150155160
histrptyrglnglnlysserhisgluserproargleuleuilelys
165170175
tyrglyseraspserileserglyileproserargpheserglyser
180185190
glyserglythraspphethrleuserileasnservalgluthrglu
195200205
asppheglymettyrphecysleuglnglytyrserthrprotrpthr
210215220
pheglyglyglythrlysleugluilelys
225230
<210>17
<211>234
<212>prt
<213>homosapiens
<400>17
glnileglnleuvalglnserglyprogluleulyslysproglyglu
151015
thrvallysilesercyslysalaserglytyrthrphethrasptyr
202530
sermetasntrpvallysglnalaproglylysglyleulystrpmet
354045
glytrpileasnthralathrglygluprothrtyralaaspaspphe
505560
lysglyargphealapheserleugluthrseralaserthralatyr
65707580
leuglnileasnasnleulysasngluaspmetalathrtyrphecys
859095
phesertyrtyrasptyrtrpglyglnglythrthrleuthrvalser
100105110
serglyglyglyglyserglyglyglyglyserglyglyglyglyasp
115120125
ilevalleuthrglnserproalathrleuservalthrproglyasp
130135140
argvalserleusercysargalaserglnthriletyrlysasnleu
145150155160
histrptyrglnglnlysserhisgluserproargleuleuilelys
165170175
tyrglyseraspserileserglyileproserargpheserglyser
180185190
glyserglythraspphethrleuserileasnservalgluthrglu
195200205
asppheglymettyrphecysleuglnglytyrserthrprotrpthr
210215220
pheglyglyglythrlysleugluilelys
225230
<210>18
<211>234
<212>prt
<213>homosapiens
<400>18
glnileglnleuvalglnserglyprogluleulyslysproglyglu
151015
thrvallysilesercyslysalaserglytyrthrphethrasptyr
202530
sermetasntrpvallysglnalaproglylysglyleulystrpmet
354045
glytrpileasnthralathrglygluprothrtyralaaspaspphe
505560
lysglyargphealapheserleugluthrseralaserthralatyr
65707580
leuglnileasnasnleulysasngluaspthralathrtyrphecys
859095
phesertyrtyrasptyrtrpglyglnglythrthrleuthrvalser
100105110
serglyglyglyglyserglyglyglyglyserglyglyglyglyasp
115120125
ilevalleuthrglnserproalathrleuservalthrproglyasp
130135140
servalserleusercysargalaserglnthriletyrlysasnleu
145150155160
histrptyrglnglnlysserhisgluserproargleuleuilelys
165170175
tyrglyseraspserileserglyileproserargpheserglyser
180185190
glyserglythraspphethrleuserileasnservalgluthrglu
195200205
asppheglymettyrphecysleuglnglytyrserthrprotrpthr
210215220
pheglyglyglythrlysleugluilelys
225230
<210>19
<211>234
<212>prt
<213>homosapiens
<400>19
glnileglnleuvalglnserglyprogluleulyslysproglyglu
151015
thrvallysilesercyslysalaserglytyrthrphethrasptyr
202530
sermetasntrpvallysglnalaproglylysglyleulystrpmet
354045
glytrpileasnthralathrglygluprothrtyralaaspaspphe
505560
lysglyargphealapheserleugluthrseralaserthralatyr
65707580
leuglnileasnasnleulysasngluaspthralathrtyrphecys
859095
phesertyrtyrasptyrtrpglyglnglythrthrleuthrvalser
100105110
serglyglyglyglyserglyglyglyglyserglyglyglyglyasp
115120125
ilevalmetthrglnserproalathrleuservalthrproglyasp
130135140
argvalserleusercysargalaserglnthriletyrlysasnleu
145150155160
histrptyrglnglnlysserhisgluserproargleuleuilelys
165170175
tyrglyseraspserileserglyileproserargpheserglyser
180185190
glyserglyseraspphethrleuserileasnservalgluproglu
195200205
aspvalglyvaltyrtyrcysleuglnglytyrserthrprotrpthr
210215220
pheglyglyglythrlysleugluilelys
225230
<210>20
<211>234
<212>prt
<213>homosapiens
<400>20
glnileglnleuvalglnserglyprogluleulyslysproglyglu
151015
thrvallysilesercyslysalaserglytyrthrphethrasptyr
202530
sermethistrpvallysglnalaproglylysglyleulystrpmet
354045
glytrpileasnthralathrglygluprothrtyralaaspaspphe
505560
lysglyargphealapheserleugluthrseralaserthralatyr
65707580
leuglnileasnasnleulysasngluaspthralathrtyrphecys
859095
phesertyrtyrasptyrtrpglyglnglythrthrleuthrvalser
100105110
serglyglyglyglyserglyglyglyglyserglyglyglyglyasp
115120125
ilevalmetthrglnserproalathrleuservalthrproglyasp
130135140
argvalserleusercysargalaserglnthriletyrlysasnleu
145150155160
histrptyrglnglnlysserhisgluserproargleuleuilelys
165170175
tyrglyseraspserileserglyileproserargpheserglyser
180185190
glyserglyseraspphethrleuserileasnservalgluproglu
195200205
aspvalglyvaltyrtyrcysleuglnglytyrserthrprotrpthr
210215220
pheglyglyglythrlysleugluilelys
225230
<210>21
<211>234
<212>prt
<213>homosapiens
<400>21
glnileglnleuvalglnserglyprogluleulyslysproglyglu
151015
thrvallysilesercyslysalaserglytyrthrphethrasptyr
202530
sermetasntrpvallysglnalaproglylysglyleulystrpmet
354045
glytrpileasnthralathrglygluprothrtyralaaspaspphe
505560
lysglyargphealapheserleugluthrseralaserthralatyr
65707580
leuglnileasnasnleulysasngluaspmetalathrtyrphecys
859095
phesertyrtyrasptyrtrpglyglnglythrthrleuthrvalser
100105110
serglyglyglyglyserglyglyglyglyserglyglyglyglyasp
115120125
ilevalmetthrglnserproalathrleuservalthrproglyasp
130135140
argvalserleusercysargalaserglnthriletyrlysasnleu
145150155160
histrptyrglnglnlysserhisgluserproargleuleuilelys
165170175
tyrglyseraspserileserglyileproserargpheserglyser
180185190
glyserglyseraspphethrleuserileasnservalgluproglu
195200205
aspvalglyvaltyrtyrcysleuglnglytyrserthrprotrpthr
210215220
pheglyglyglythrlysleugluilelys
225230
<210>22
<211>234
<212>prt
<213>homosapiens
<400>22
glnileglnleuvalglnserglyprogluleulyslysproglyglu
151015
thrvallysilesercyslysalaserglytyrthrphethrasptyr
202530
sermethistrpvallysglnalaproglylysglyleulystrpmet
354045
glytrpileasnthralathrglygluprothrtyralaaspaspphe
505560
lysglyargphealapheserleugluthrseralaserthralatyr
65707580
leuglnileasnasnleulysasngluaspthralathrtyrphecys
859095
phesertyrtyrasptyrtrpglyglnglythrthrleuthrvalser
100105110
serglyglyglyglyserglyglyglyglyserglyglyglyglyasp
115120125
ilevalleuthrglnserproalathrleuservalthrproglyasp
130135140
servalserleusercysargalaserglnthriletyrlysasnleu
145150155160
histrptyrglnglnlysserhisgluserproargleuleuilelys
165170175
tyrglyseraspserileserglyileproserargpheserglyser
180185190
glyserglythraspphethrleuserileasnservalgluthrglu
195200205
asppheglymettyrphecysleuglnglytyrserthrprotrpthr
210215220
pheglyglyglythrlysleugluilelys
225230
<210>23
<211>234
<212>prt
<213>homosapiens
<400>23
glnileglnleuvalglnserglyprogluleulyslysproglyglu
151015
thrvallysilesercyslysalaserglytyrthrphethrasptyr
202530
sermetasntrpvallysglnalaproglylysglyleulystrpmet
354045
glytrpileasnthralathrglygluprothrtyralaaspaspphe
505560
lysglyargphealapheserleugluthrseralaserthralatyr
65707580
leuglnileasnasnleulysasngluaspmetalathrtyrphecys
859095
phesertyrtyrasptyrtrpglyglnglythrthrleuthrvalser
100105110
serglyglyglyglyserglyglyglyglyserglyglyglyglyasp
115120125
ilevalleuthrglnserproalathrleuservalthrproglyasp
130135140
servalserleusercysargalaserglnthriletyrlysasnleu
145150155160
histrptyrglnglnlysserhisgluserproargleuleuilelys
165170175
tyrglyseraspserileserglyileproserargpheserglyser
180185190
glyserglythraspphethrleuserileasnservalgluthrglu
195200205
asppheglymettyrphecysleuglnglytyrserthrprotrpthr
210215220
pheglyglyglythrlysleugluilelys
225230
<210>24
<211>714
<212>dna
<213>homosapiens
<400>24
atggcccaggtcaaactgaaggagtctggacctgagctgaagaagcctggagagacagtc60
aagatctcctgcaaggcttctggttataccttcacagactattcaatgcactgggtgaag120
aagactccaggaaagggtttaaagtggctgggctggataaacactgcgactggtgagcca180
acatatgcagatgacttcaagggacggtttgccatctctttggaaacctctgccaggact240
gtctatttgcagatcaataatctcagaaatgaggacacggctacatatttctgttttagt300
tactacgactactggggccaaggcaccacggtcaccgtctcctcaggtgggggcggttca360
ggcggaggtggctctggcggtggcggattggacatcaagctcagtcagtctccatccatc420
ctgtctgtgactccaggtgaaacagtcagtctttcctgtagggccagccagactatttac480
aagaacctacactggtatcaacagaaatcacatcggtctccaaggcttctcatcaagtat540
ggttctgattccatctctggcatcccctccaggttcactggcagtggatcagggacagat600
tacactctcaatatcaacagtgtgaagcccgaagatgaaggaatatattactgtcttcaa660
ggttacagtacaccttggacgttcggtggagggaccaagctggaaataaaacgg714
<210>25
<211>684
<212>dna
<213>homosapiens
<400>25
gagcgcaaatgttgtgtcgagtgcccaccgtgcccagcaccacctgtggcaggaccgtca60
gtcttcctcttccccccaaaacccaaggacaccctcatgatctcccggacccctgaggtc120
acgtgcgtggtggtggacgtgagccacgaagaccccgaggtccagttcaactggtacgtg180
gacggcgtggaggtgcataatgccaagacaaagccacgggaggagcagttccaaagcacg240
ttccgtgtggtcagcgtcctcaccgttgtgcaccaggactggctgaacggcaaggagtac300
aagtgcaaggtctccaacaaaggcctcccagcccccatcgagaaaaccatctccaaaacc360
aaagggcagccccgagaaccacaggtgtacaccctgcccccatcccgggatgagctgacc420
aagaaccaggtcagcctgacctgcctggtcaaaggcttctatcccagcgacatcgccgtg480
gagtgggagagcaatgggcagccggagaacaactacaagaccacgcctcccgtgctggac540
tccgacggctccttcttcctctacagcaagctcaccgtggacaagagcaggtggcagcag600
gggaacgtcttctcatgctccgtgatgcatgaggctctgcacaaccactacacgcagaag660
agcctctccctgtctccgggtaaa684
<210>26
<211>123
<212>dna
<213>homosapiens
<400>26
aggagtaagaggagcaggctcctgcacagtgactacatgaacatgactccccgccgcccc60
gggcccacccgcaagcattaccagccctatgccccaccacgcgacttcgcagcctatcgc120
tcc123
<210>27
<211>158
<212>dna
<213>homosapiens
<400>27
cagatccagctggtgcagagcggccccgagctaaacggggcagaaagaaactcctgtata60
tattcaaacaaccatttatgagaccagtacaaactactcaagaggaagatggctgtagct120
gccgatttccagaagaagaagaaggaggatgtgaactg158
<210>28
<211>684
<212>dna
<213>homosapiens
<400>28
gagcgcaaatgttgtgtcgagtgcccaccgtgcccagcaccacctgtggcaggaccgtca60
gtcttcctcttccccccaaaacccaaggacaccctcatgatctcccggacccctgaggtc120
acgtgcgtggtggtggacgtgagccacgaagaccccgaggtccagttcaactggtacgtg180
gacggcgtggaggtgcataatgccaagacaaagccacgggaggagcagttccaaagcacg240
ttccgtgtggtcagcgtcctcaccgttgtgcaccaggactggctgaacggcaaggagtac300
aagtgcaaggtctccaacaaaggcctcccagcccccatcgagaaaaccatctccaaaacc360
aaagggcagccccgagaaccacaggtgtacaccctgcccccatcccgggatgagctgacc420
aagaaccaggtcagcctgacctgcctggtcaaaggcttctatcccagcgacatcgccgtg480
gagtgggagagcaatgggcagccggagaacaactacaagaccacgcctcccgtgctggac540
tccgacggctccttcttcctctacagcaagctcaccgtggacaagagcaggtggcagcag600
gggaacgtcttctcatgctccgtgatgcatgaggctctgcacaaccactacacgcagaag660
agcctctccctgtctccgggtaaa684
<210>29
<211>42
<212>dna
<213>homosapiens
<400>29
cccaaatcttgtgacaaaactcacacatgcccaccgtgccca42
<210>30
<211>372
<212>dna
<213>homosapiens
<400>30
cccaaatcttgtgacaaaactcacacatgcccaccgtgcccagcacctgaactcctgggg60
ggaccgtcagtcttcctcttccccccaaaacccaaggacaccctcatgatctcccggacc120
cctgaggtcacatgcgtggtggtggacgtgagccacgaagaccctgaggtcaagttcaac180
tggtacgtggacggcgtggaggtgcataatgccaagacaaagccgcgggaggagcagtac240
aacagcacgtaccgtgtggtcagcgtcctcaccgtcctgcaccaggactggctgaatggc300
aaggagtacaagtgcaaggtctccaacaaagccctcccagcccccatcgagaaaaccatc360
tccaaagccaaa372
<210>31
<211>684
<212>dna
<213>homosapiens
<400>31
gagcgcaaatgttgtgtcgagtgcccaccgtgcccagcaccacctgtggcaggaccgtca60
gtcttcctcttccccccaaaacccaaggacaccctcatgatctcccggacccctgaggtc120
acgtgcgtggtggtggacgtgagccacgaagaccccgaggtccagttcaactggtacgtg180
gacggcgtggaggtgcataatgccaagacaaagccacgggaggagcagttccaaagcacg240
ttccgtgtggtcagcgtcctcaccgttgtgcaccaggactggctgaacggcaaggagtac300
aagtgcaaggtctccaacaaaggcctcccagcccccatcgagaaaaccatctccaaaacc360
aaagggcagccccgagaaccacaggtgtacaccctgcccccatcccgggatgagctgacc420
aagaaccaggtcagcctgacctgcctggtcaaaggcttctatcccagcgacatcgccgtg480
gagtgggagagcaatgggcagccggagaacaactacaagaccacgcctcccgtgctggac540
tccgacggctccttcttcctctacagcaagctcaccgtggacaagagcaggtggcagcag600
gggaacgtcttctcatgctccgtgatgcatgaggctctgcacaaccactacacgcagaag660
agcctctccctgtctccgggtaaa684
<210>32
<211>420
<212>dna
<213>homosapiens
<400>32
aaagaggcctgccccaccggcctgtacacccacagcggagagtgctgcaaggcctgcaac60
ctgggagagggcgtggcccagccttgcggcgccaatcagaccgtgtgcgagccctgcctg120
gacagcgtgaccttcagcgacgtggtgtccgccaccgagccctgcaagccttgcaccgag180
tgtgtgggcctgcagagcatgagcgccccctgcgtggaagccgacgacgccgtgtgtaga240
tgcgcctacggctactaccaggacgagacaaccggcagatgcgaggcctgtagagtgtgc300
gaggccggcagcggcctggtgttcagttgtcaggacaagcagaacaccgtgtgtgaagag360
tgccccgacggcacctacagcgacgaggccgcccgggccgccgacgccgagtgcgaggaa420
<210>33
<211>702
<212>dna
<213>homosapiens
<400>33
caggtgcagctggtgcagagcggcagcgagctgaagaagcccggcgccagcgtgaaggtg60
agctgcaaggccagcggctacaccttcaccgactacagcatgcactgggtgaggcaggcc120
cccggccagggcctggagtggatgggctggatcaacaccgccaccggcgagcccacctac180
gccgacgacttcaagggcaggttcgtgttcagcctggacaccagcgtgagcaccgcctac240
ctgcagatcagcagcctgaaggccgaggacaccgccgtgtactactgcttcagctactac300
gactactggggccagggcaccaccgtgaccgtgagcagcggtgggggcggttcaggcgga360
ggtggctctggcggtggcggagagatcgtgctgacccagagccccgacttccagagcgtg420
acccccaaggagaaggtgaccatcacctgcagggccagccagaccatctacaagaacctg480
cactggtaccagcagaagcccgaccagagccccaagctgctgatcaagtacggcagcgac540
agcatcagcggcatccccagcaggttcagcggcagcggcagcggcaccgacttcaccctg600
accatcaacagcctggaggccgaggacgccgccacctactactgcctgcagggctacagc660
accccctggaccttcggcggcggcaccaaggtggagatcaag702
<210>34
<211>702
<212>dna
<213>homosapiens
<400>34
cagatccagctggtgcagagcggccccgagctgaagaagcccggcgagaccgtgaagatc60
agctgcaaggccagcggctacaccttcaccgactacagcatgaactgggtgaagcaggcc120
cccggcaagggcctgaagtggatgggctggatcaacaccgccaccggcgagcccacctac180
gccgacgacttcaagggcaggttcgccttcagcctggagaccagcgccagcaccgcctac240
ctgcagatcaacaacctgaagaacgaggacaccgccacctacttctgcttcagctactac300
gactactggggccagggcaccaccctgaccgtgagcagcggtgggggcggttcaggcgga360
ggtggctctggcggtggcggagacatcgtgctgacccagagccccgccaccctgagcgtg420
acccccggcgacagggtgagcctgagctgcagggccagccagaccatctacaagaacctg480
cactggtaccagcagaagagccacgagagccccaggctgctgatcaagtacggcagcgac540
agcatcagcggcatccccagcaggttcagcggcagcggcagcggcaccgacttcaccctg600
agcatcaacagcgtggagaccgaggacttcggcatgtacttctgcctgcagggctacagc660
accccctggaccttcggcggcggcaccaagctggagatcaag702
<210>35
<211>702
<212>dna
<213>homosapiens
<400>35
cagatccagctggtgcagagcggccccgagctgaagaagcccggcgagaccgtgaagatc60
agctgcaaggccagcggctacaccttcaccgactacagcatgcactgggtgaagcaggcc120
cccggcaagggcctgaagtggatgggctggatcaacaccgccaccggcgagcccacctac180
gccgacgacttcaagggcaggttcgccttcagcctggagaccagcgccagcaccgcctac240
ctgcagatcaacaacctgaagaacgaggacaccgccacctacttctgcttcagctactac300
gactactggggccagggcaccaccctgaccgtgagcagcggtgggggcggttcaggcgga360
ggtggctctggcggtggcggagacatcgtgctgacccagagccccgccaccctgagcgtg420
acccccggcgacagggtgagcctgagctgcagggccagccagaccatctacaagaacctg480
cactggtaccagcagaagagccacgagagccccaggctgctgatcaagtacggcagcgac540
agcatcagcggcatccccagcaggttcagcggcagcggcagcggcaccgacttcaccctg600
agcatcaacagcgtggagaccgaggacttcggcatgtacttctgcctgcagggctacagc660
accccctggaccttcggcggcggcaccaagctggagatcaag702
<210>36
<211>702
<212>dna
<213>homosapiens
<400>36
cagatccagctggtgcagagcggccccgagctgaagaagcccggcgagaccgtgaagatc60
agctgcaaggccagcggctacaccttcaccgactacagcatgaactgggtgaagcaggcc120
cccggcaagggcctgaagtggatgggctggatcaacaccgccaccggcgagcccacctac180
gccgacgacttcaagggcaggttcgccttcagcctggagaccagcgccagcaccgcctac240
ctgcagatcaacaacctgaagaacgaggacatggccacctacttctgcttcagctactac300
gactactggggccagggcaccaccctgaccgtgagcagcggtgggggcggttcaggcgga360
ggtggctctggcggtggcggagacatcgtgctgacccagagccccgccaccctgagcgtg420
acccccggcgacagggtgagcctgagctgcagggccagccagaccatctacaagaacctg480
cactggtaccagcagaagagccacgagagccccaggctgctgatcaagtacggcagcgac540
agcatcagcggcatccccagcaggttcagcggcagcggcagcggcaccgacttcaccctg600
agcatcaacagcgtggagaccgaggacttcggcatgtacttctgcctgcagggctacagc660
accccctggaccttcggcggcggcaccaagctggagatcaag702
<210>37
<211>702
<212>dna
<213>homosapiens
<400>37
cagatccagctggtgcagagcggccccgagctgaagaagcccggcgagaccgtgaagatc60
agctgcaaggccagcggctacaccttcaccgactacagcatgaactgggtgaagcaggcc120
cccggcaagggcctgaagtggatgggctggatcaacaccgccaccggcgagcccacctac180
gccgacgacttcaagggcaggttcgccttcagcctggagaccagcgccagcaccgcctac240
ctgcagatcaacaacctgaagaacgaggacaccgccacctacttctgcttcagctactac300
gactactggggccagggcaccaccctgaccgtgagcagcggtgggggcggttcaggcgga360
ggtggctctggcggtggcggagacatcgtgctgacccagagccccgccaccctgagcgtg420
acccccggcgacagcgtgagcctgagctgcagggccagccagaccatctacaagaacctg480
cactggtaccagcagaagagccacgagagccccaggctgctgatcaagtacggcagcgac540
agcatcagcggcatccccagcaggttcagcggcagcggcagcggcaccgacttcaccctg600
agcatcaacagcgtggagaccgaggacttcggcatgtacttctgcctgcagggctacagc660
accccctggaccttcggcggcggcaccaagctggagatcaag702
<210>38
<211>702
<212>dna
<213>homosapiens
<400>38
cagatccagctggtgcagagcggccccgagctgaagaagcccggcgagaccgtgaagatc60
agctgcaaggccagcggctacaccttcaccgactacagcatgaactgggtgaagcaggcc120
cccggcaagggcctgaagtggatgggctggatcaacaccgccaccggcgagcccacctac180
gccgacgacttcaagggcaggttcgccttcagcctggagaccagcgccagcaccgcctac240
ctgcagatcaacaacctgaagaacgaggacaccgccacctacttctgcttcagctactac300
gactactggggccagggcaccaccctgaccgtgagcagcggtgggggcggttcaggcgga360
ggtggctctggcggtggcggagacatcgtgatgacccagagccccgccaccctgagcgtg420
acccccggcgacagggtgagcctgagctgcagggccagccagaccatctacaagaacctg480
cactggtaccagcagaagagccacgagagccccaggctgctgatcaagtacggcagcgac540
agcatcagcggcatccccagcaggttcagcggcagcggcagcggcagcgacttcaccctg600
agcatcaacagcgtggagcccgaggacgtcggcgtgtactactgcctgcagggctacagc660
accccctggaccttcggcggcggcaccaagctggagatcaag702
<210>39
<211>702
<212>dna
<213>homosapiens
<400>39
cagatccagctggtgcagagcggccccgagctgaagaagcccggcgagaccgtgaagatc60
agctgcaaggccagcggctacaccttcaccgactacagcatgcactgggtgaagcaggcc120
cccggcaagggcctgaagtggatgggctggatcaacaccgccaccggcgagcccacctac180
gccgacgacttcaagggcaggttcgccttcagcctggagaccagcgccagcaccgcctac240
ctgcagatcaacaacctgaagaacgaggacaccgccacctacttctgcttcagctactac300
gactactggggccagggcaccaccctgaccgtgagcagcggtgggggcggttcaggcgga360
ggtggctctggcggtggcggagacatcgtgatgacccagagccccgccaccctgagcgtg420
acccccggcgacagggtgagcctgagctgcagggccagccagaccatctacaagaacctg480
cactggtaccagcagaagagccacgagagccccaggctgctgatcaagtacggcagcgac540
agcatcagcggcatccccagcaggttcagcggcagcggcagcggcagcgacttcaccctg600
agcatcaacagcgtggagcccgaggacgtcggcgtgtactactgcctgcagggctacagc660
accccctggaccttcggcggcggcaccaagctggagatcaag702
<210>40
<211>702
<212>dna
<213>homosapiens
<400>40
cagatccagctggtgcagagcggccccgagctgaagaagcccggcgagaccgtgaagatc60
agctgcaaggccagcggctacaccttcaccgactacagcatgaactgggtgaagcaggcc120
cccggcaagggcctgaagtggatgggctggatcaacaccgccaccggcgagcccacctac180
gccgacgacttcaagggcaggttcgccttcagcctggagaccagcgccagcaccgcctac240
ctgcagatcaacaacctgaagaacgaggacatggccacctacttctgcttcagctactac300
gactactggggccagggcaccaccctgaccgtgagcagcggtgggggcggttcaggcgga360
ggtggctctggcggtggcggagacatcgtgatgacccagagccccgccaccctgagcgtg420
acccccggcgacagggtgagcctgagctgcagggccagccagaccatctacaagaacctg480
cactggtaccagcagaagagccacgagagccccaggctgctgatcaagtacggcagcgac540
agcatcagcggcatccccagcaggttcagcggcagcggcagcggcagcgacttcaccctg600
agcatcaacagcgtggagcccgaggacgtcggcgtgtactactgcctgcagggctacagc660
accccctggaccttcggcggcggcaccaagctggagatcaag702
<210>41
<211>702
<212>dna
<213>homosapiens
<400>41
cagatccagctggtgcagagcggccccgagctgaagaagcccggcgagaccgtgaagatc60
agctgcaaggccagcggctacaccttcaccgactacagcatgcactgggtgaagcaggcc120
cccggcaagggcctgaagtggatgggctggatcaacaccgccaccggcgagcccacctac180
gccgacgacttcaagggcaggttcgccttcagcctggagaccagcgccagcaccgcctac240
ctgcagatcaacaacctgaagaacgaggacaccgccacctacttctgcttcagctactac300
gactactggggccagggcaccaccctgaccgtgagcagcggtgggggcggttcaggcgga360
ggtggctctggcggtggcggagacatcgtgctgacccagagccccgccaccctgagcgtg420
acccccggcgacagcgtgagcctgagctgcagggccagccagaccatctacaagaacctg480
cactggtaccagcagaagagccacgagagccccaggctgctgatcaagtacggcagcgac540
agcatcagcggcatccccagcaggttcagcggcagcggcagcggcaccgacttcaccctg600
agcatcaacagcgtggagaccgaggacttcggcatgtacttctgcctgcagggctacagc660
accccctggaccttcggcggcggcaccaagctggagatcaag702
<210>42
<211>702
<212>dna
<213>homosapiens
<400>42
cagatccagctggtgcagagcggccccgagctgaagaagcccggcgagaccgtgaagatc60
agctgcaaggccagcggctacaccttcaccgactacagcatgaactgggtgaagcaggcc120
cccggcaagggcctgaagtggatgggctggatcaacaccgccaccggcgagcccacctac180
gccgacgacttcaagggcaggttcgccttcagcctggagaccagcgccagcaccgcctac240
ctgcagatcaacaacctgaagaacgaggacatggccacctacttctgcttcagctactac300
gactactggggccagggcaccaccctgaccgtgagcagcggtgggggcggttcaggcgga360
ggtggctctggcggtggcggagacatcgtgctgacccagagccccgccaccctgagcgtg420
acccccggcgacagcgtgagcctgagctgcagggccagccagaccatctacaagaacctg480
cactggtaccagcagaagagccacgagagccccaggctgctgatcaagtacggcagcgac540
agcatcagcggcatccccagcaggttcagcggcagcggcagcggcaccgacttcaccctg600
agcatcaacagcgtggagaccgaggacttcggcatgtacttctgcctgcagggctacagc660
accccctggaccttcggcggcggcaccaagctggagatcaag702
<210>43
<211>21
<212>prt
<213>homosapiens
<400>43
metglupheglyleusertrpleupheleuvalalaileleulysgly
151015
valglncysserarg
20
<210>44
<211>27
<212>prt
<213>homosapiens
<400>44
phetrpvalleuvalvalvalglyglyvalleualacystyrserleu
151015
leuvalthrvalalapheileilephetrpval
2025
<210>45
<211>112
<212>prt
<213>homosapiens
<400>45
argvallyspheserargseralaaspalaproalatyrglnglngly
151015
glnasnglnleutyrasngluleuasnleuglyargarggluglutyr
202530
aspvalleuasplysargargglyargaspproglumetglyglylys
354045
proargarglysasnproglngluglyleutyrasngluleuglnlys
505560
asplysmetalaglualatyrsergluileglymetlysglygluarg
65707580
argargglylysglyhisaspglyleutyrglnglyleuserthrala
859095
thrlysaspthrtyraspalaleuhismetglnalaleuproproarg
100105110
- 还没有人留言评论。精彩留言会获得点赞!